Молекулярная биология, 2020, T. 54, № 4, стр. 688-698
Выявление наследственных перестроек в гене BRCA1 методом NGS-секвенирования опухолевой ДНК
A. Minucci a, *, **, G. Mazzuccato a, C. Marchetti b, c, A. Pietragalla b, G. Scambia b, c, A. Fagotti b, c, A. Urbani a, c, ***
a Molecular Diagnostic and Genomics Laboratory, Fondazione Policlinico Universitario A. Gemelli IRCCS
00168 Rome, Italy
b Division of Oncological Gynecology, Department of Women’s and Children’s Health, Fondazione Policlinico Universitario
A. Gemelli IRCCS
00168 Rome, Italy
c Catholic University of the Sacred Heart
00168 Rome, Italy
* E-mail: angelo.minucci@policlinicogemelli.it
** E-mail: angelo.minucci@virgilio.it
*** E-mail: andrea.urbani@policlinicogemelli.it
Поступила в редакцию 30.09.2019
После доработки 16.12.2019
Принята к публикации 25.12.2019
Аннотация
Большинство патогенных вариантов генов BRCA1/2 содержат однонуклеотидные замены или небольшие инсерции/делеции. Однако в генах BRCA идентифицированы и такие крупные геномные перестройки, как вариации числа копий (CNV). Обнаружение крупных геномных перестроек в семьях с наследственным раком молочной железы и яичников обязательно при условии, что секвенирование не выявило предрасполагающих патогенных вариантов. Изменения в структуре гена можно детектировать с помощью методов секвенирования нового поколения (NGS), поскольку количественный анализ чтений в сочетании с биоинформатическими инструментами позволяет оценивать и прогнозировать крупные наследственные геномные перестройки. Однако анализ ДНК опухолевой ткани с помощью NGS представляет сложную задачу, и методики определения CNV еще предстоит оптимизировать. Нами изучена применимость технологии NGS для обнаружения крупных наследственных перестроек локуса гена BRCA1 в постоперационных образцах опухолевых тканей. В постоперационных образцах высокозлокачественного серозного рака яичников нами выявлены семь различных крупных геномных перестроек BRCA1, обнаруженных ранее в этих опухолях. Полученные результаты показывают, что с помощью NGS можно точно определять наследственные крупные геномные перестройки BRCA1 в первичных опухолях. Предполагается, что оценку таких перестроек в локусе BRCA1 следует проводить в тех случаях, когда анализ начинается с образца опухолевой ткани, а не крови. NGS-секвенирование образцов опухолевой ДНК может стать предпочтительным методом обнаружения как соматических, так и наследственных крупных геномных перестроек в локусах BRCA.
ВВЕДЕНИЕ
Основным критерием, определяющим необходимость выявления генеративного аллеля BRCA1/2 (gBRCA) и проведения генетического консультирования, является наследственная предрасположенность к развитию опухолей молочной железы и/или яичников. За последнее десятилетие многочисленные клинические исследования показали, что пациентки с высокозлокачественным серозным раком яичников (HGSOC) и патогенными вариантами gBRCA чувствительны к ингибиторам поли(АDP-рибозa)-полимеразы (PARPi) и химиотерапии препаратами на основе платины, что повышает общую выживаемость [1]. Кроме того, ингибиторы PARP эффективны у пациенток с BRCA-специфическими патогенными вариантами в опухолевой ДНК [2, 3]. Тестирование BRCA в фиксированных формалином образцах, заключенных в парафин, или в образцах замороженной ткани позволяет оценивать одновременно как соматические, так и наследственные изменения в структуре гена, и имеет большое диагностическое и клиническое значение при HGSOC. В настоящее время во многих лабораториях, где для тестирования gBRCA применяют технологии секвенирования нового поколения (NGS), внедрен анализ BRCA в тканях опухолей (tBRCA) [4].
В генах BRCA обнаружены вариации числа копий (CNV), известные также как крупные геномные перестройки (LGR), включающие делеции, дупликации или вставки размером более 1000 т.п.н. В популяциях с сильным эффектом основателя частота CNV значительно варьирует (от менее 1 до более 24%) [5]. В гене BRCA1 LGR встречаются чаще, вероятно, из-за более высокой частоты Alu-элементов и/или из-за событий гомологичной рекомбинации между BRCA1 и его псевдогеном [6]. Следовательно, LGR составляют значительную часть патогенных вариантов BRCA, и их исследование включено в молекулярный анализ BRCA. Методы лабораторной диагностики LGR BRCA включают Саузерн-блотинг, количественную ПЦР, конкурентный анализ кривых плавления с высоким разрешением, анализ на ДНК-микрочипах и мультиплексную амплификацию лигированных зондов (MLPA) [7–11]. На сегодняшний день NGS применяют для выявления структурных изменений ДНК, поскольку генерирование миллионов одинаковых последовательностей геномных областей-мишеней в сочетании с соответствующими биоинформатическими инструментами и количественным анализом чтений (ридов) секвенирования может использоваться для оценки и прогнозирования наследственных LGR (gLGR) [12–14].
Тем не менее, обнаружение LGR в целевых генах, основанное на данных NGS и наиболее часто используемое в диагностических протоколах, остается несовершенным, главным образом, в силу того, что все методики обнаружения LGR в данных целевого секвенирования базируются на подходе, основанном на глубине покрытия. Этот подход предполагает, что количество целевых прочтений коррелирует, как правило, с количеством копий последовательности в соответствующем районе генома. При этом LGR детектируют путем расчета покрытия каждого ампликона и обнаружения выбросов. Однако оценка глубины покрытия имеет ряд недостатков, которые искажают покрытие, включая: 1) неравномерную амплификацию; 2) влияние GC-состава 3) неоднозначное картирование коротких фрагментов. Следует отметить, что перед введением в клиническую практику метод обнаружения LGR с помощью NGS должен быть валидирован.
Применение NGS для анализа LGR в опухоли осложняется гетерогенностью опухолевой ткани, загрязнением образца нормальными клетками и низким количеством клеток. Как следствие, некоторые риды, картированные на определенную область, могут не принадлежать опухолевым клеткам, а значения числа прочтений могут не совсем точно отражать CNV в опухолевых клетках, что создает трудности при определении сегментов, содержащих CNV.
Мутационные события в генах BRCA полностью соответствуют модели развития опухоли в рамках теории двойного удара Кнудсона. Соответственно, потеря немутантного аллеля локуса BRCA1 или BRCA2, называемая локус-специфической потерей гетерозиготности, обычно встречается в опухолях с мутациями в BRCA [15, 16]. Фактически, в первичной опухоли наследственные патогенные варианты могут появляться при инактивации обоих аллелей, вызывая нестабильность генома и нарушение роста [17]. Это относится и к патогенным gLGR, особенно к LGR, возникновение которых обусловлено делециями экзонов в BRCA.
Нами оптимизирован и валидирован протокол тестирования gBRCA, который включает набор Devyser BRCA NGS (“Devyser”, Швеция) для подготовки библиотеки ДНК, технологию Illumina MiSeq (“Illumina, США) для NGS и программное обеспечение Amplicon Suite (“SmartSeq”, Италия) для анализа данных. Подготовка библиотеки ДНК для секвенирования включала использование метода целевого обогащения, позволяющего получать перекрывающиеся ампликоны и обеспечивающего полное и равномерное покрытие всех экзонов и экзон-интронных соединений генов BRCA, что позволяет получить более высокое покрытие инсерций/делеций и провести точный анализ CNV. Нами показано, что эта методика позволяет со 100%-ной чувствительностью и специфичностью выявлять gCNV, она удовлетворяет клиническую потребность в обнаружении всех патогенных вариантов BRCA, включая LGR [11, 12].
В нашей работе оценена возможность использования разработанной методики NGS/BRCA для выявления семи различных gLGR BRCA1 в постоперационных образцах тканей HGSOC.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Выбор и подготовка образцов. Всего были отобраны семь пациентов с HGSOC с CNV BRCA1, ранее идентифицированными в образцах крови с помощью NGS и подтвержденными методом MLPA. От каждого пациента с помощью диагностической биопсии на первых стадиях заболевания или при проведении циторедуктивной хирургии получены образцы ткани, которые сразу же заморозили и проанализировали методом NGTS. Образцы ДНК выделяли из периферической крови и из срезов замороженной ткани HGSOC (содержание неопластических клеток не менее 70%) с использованием автоматического устройства MagCore HF16 Plus (“Diatech Lab Line”, Италия) в соответствии с руководством производителя. Концентрацию и качество дцДНК определяли с помощью высокочувствительного анализа с использованием флуориметра Qubit (“Thermo Fisher Scientific”, США). Каждый образец ДНК удовлетворял следующим требованиям: отношение OD260/280 ≥ 1.7, концентрация ≥15 нг/мкл, отсутствие деградации по данным электрофореза в агарозном геле. Образцы ДНК хранили при –20°C. Все пациенты подписали информированное согласие на тестирование BRCA в образцах крови и опухолевой ткани в соответствии с принципами Хельсинкской декларации.
Анализ BRCA. BRCA анализировали с использованием набора Devyser BRCA (“Devyser”), как сообщалось ранее [18]. Реакции секвенирования проводили с использованием набора реагентов V2, 500 Cycles и системы Illumina MiSeq (“Illumina”). Результаты NGS интерпретировали при помощи программного обеспечения Amplicon Suite (SmartSeq s.r.l., Италия).
Анализ MLPA и номенклатура CNV BRCA1. Анализ MLPA проводили в соответствии с рекомендациями производителя (MRC-Holland, Нидерланды) с использованием наборов SALSA P002 BRCA1 и SALSA P045 BRCA2 MLPA. CNV BRCA1 называли в соответствии с требованиями руководства Human Genome Variation Society (http://www.HGVS.org/varnomen) для CNV (табл. 1). В качестве референсных последовательностей BRCA1 и BRCA2 использовали NG_005905.2, NM_007294.3 и NG_012772.3, NM_000059.3 соответственно.
Таблица 1.
Уровень идентичности герминальных gLGR BRCA1, идентифицированных у семи пациентов с HGSOC и подтвержденных на образцах первичных опухолей методом NGTS
| Паци-ент | Геномная мутация (экзон//NM_007294.3) | Изменение ДНК (номенклатура HGVS/e/NM_007294.3) | Изменение ДНК (геномная сборка/hg19-GRCh37) | Определение CNV по данным NGTS* | Потеря гетерозиготности по данным NGTS ** |
|---|---|---|---|---|---|
| s1 | Del экзон 1–2 | c.(?_-232)_(80+1_81-1) del | g.(41267797_41276033)_(41277500_?)del | 0.4 < 0.5 | Возможно |
| s2 | Del экзон 1–11 | c.(?_-232)_(4185+1_4186-1) del | g.(41234593_41242960)_(41277500_?) del | <0.2 | Да |
| s3 | Del экзон 14 | c.(4357+1_4358-1)_(4484+1_4485-1) del | g.(41226539_41228504)_(41228632_41234420) del | <0.3 | Да |
| s4 | Del экзон 15–16 | c.(4484+1_4485-1)_(4986+1_4987-1) del | g.(41219713_41222944)_(41226539_41228504) del | 0.3 < 0.4 | Да |
| s5 | Del экзон 17–18 | c.(4986+1_4987-1)_(5152+1_5153-1) del | g.(41215391_41215890)_(41219713_41222944) del | <0.3 | Да |
| s6 | Del экзон 19 | c.(5152+1_5153-1)_(5193+1_5194-1) del | g.(41209153_41215349)_(41215391_41215890) del | 0.3 | Да |
| s7 | Del экзон 20–21 | c.(5277+1_5278-1)_(5332+1_5333-1) del | g.(41201212_41203079)_(41203135_41209068) del | 0.4 < 0.5 | Возможно |
Определение CNV BRCA1 с помощью NGS. CNV идентифицировали по данным секвенирования Illumina путем загрузки файлов FASTQ в программное обеспечение Amplicon Suite. Это программное обеспечение обнаруживает CNV на основе покрытия в определенных целевых областях, соответствующих каждому сегменту BRCA, после дизайна амплификации. Amplicon Suite Software использует собственный алгоритм для обнаружения ампликонов, покрытых несколькими прочтениями со значительными отличиями от ожидаемых значений. Тот же алгоритм использовали для каждого экзона с последующим объединением данных для всех перекрывающихся ампликонов. При таком подходе эффект от одного экзона минимизируется и не влияет на обнаружение CNV, если покрытие одного ампликона изменено из-за затрудненного отжига.
Соотношение, вычисленное по данным NGTS 100 образцов замороженной ткани с нормальным gCNV, составило 0.6–1.3. Этот диапазон соответствует части нормальной вариации. Как следствие, целевые области с такими соотношениями не считались делетированными или дублированными, что позволило избежать ложноположительных результатов из-за возможной нестабильности генома в образцах опухоли. Делеции BRCA1 идентифицировали только при средних соотношениях менее 0.6. Наконец, образцы опухолей с целевыми областями и средним соотношением 0.4 или менее считали гомозиготными делециями, возникающими в результате гетерозиготной герминальной делеции с потерей гетерозиготности другого аллеля. Для точной идентификации CNV анализировали не менее шести образцов.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Чтобы обеспечить высокую точность определения CNV BRCA1, мы удостоверились в том, что все образцы, использованные нами, соответствовали техническим характеристикам с высоким средним показателем качества прочтения (показатель Q > 35) и минимальным покрытием 2000 прочтений на ампликон. Соответствие этим требованиям оценивали с помощью калькулятора покрытия последовательности (https://devyser.com/resources/tools/calculator/), который использовали при планировании каждого секвенирования. В частности, калькулятор покрытия использовали для оптимизации глубины прочтения каждого образца и емкости проточной ячейки для определения оптимальной глубины прочтения на основе количества образцов.
В образцах ткани больных HGSOC, полученных путем диагностической биопсии на начальных стадиях опухолевого процесса или при циторедуктивной хирургии, замороженных и проанализированных с помощью NGTS, выявлены семь различных gLGR BRCA1, идентифицированных ранее с помощью NGS и подтвержденных методом MLPA. Пять из этих LGR идентифицированы абсолютно однозначно, что указывает на потерю гетерозиготности de novo в опухоли. На рис. 1 приведены результаты нормирования данных определения CNV в экзонах BRCA1 с помощью программы Amplicon Suite в соответствии со ссылкой NM_007300.4. Данные визуализации CNV во всех ампликонах BRCA1, покрываемых набором Devyser, приведены в Приложении (рисунок).
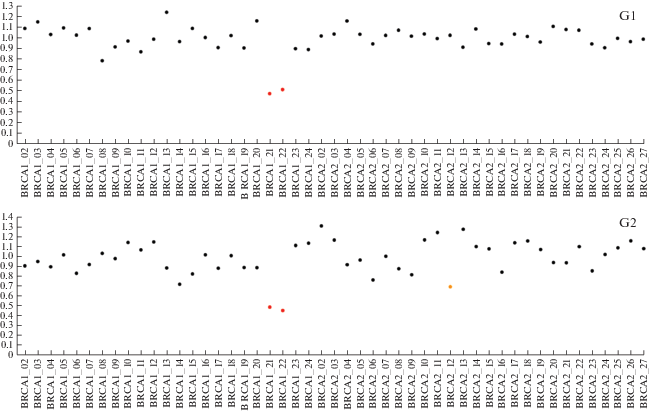
Рис. 1.
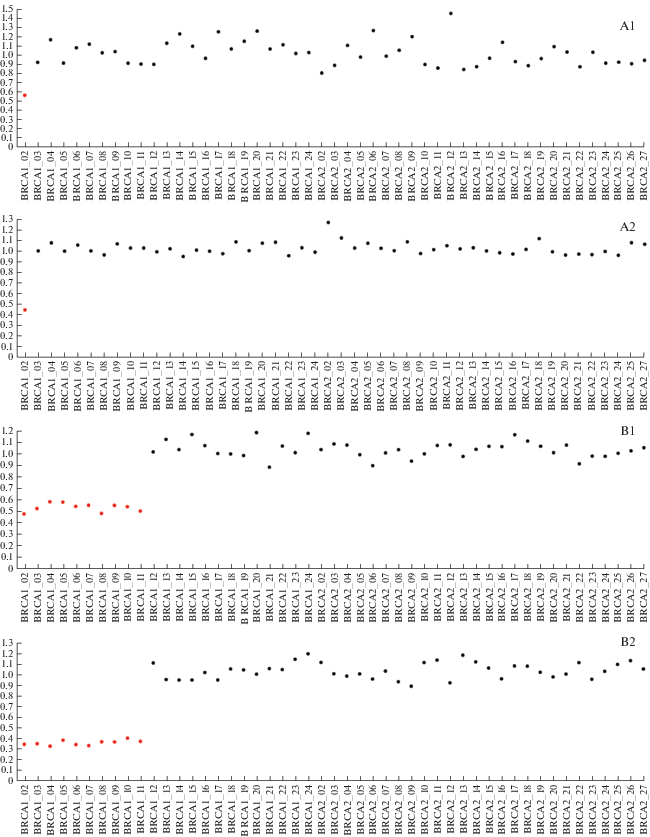
Рис. 1.
Продолжение.
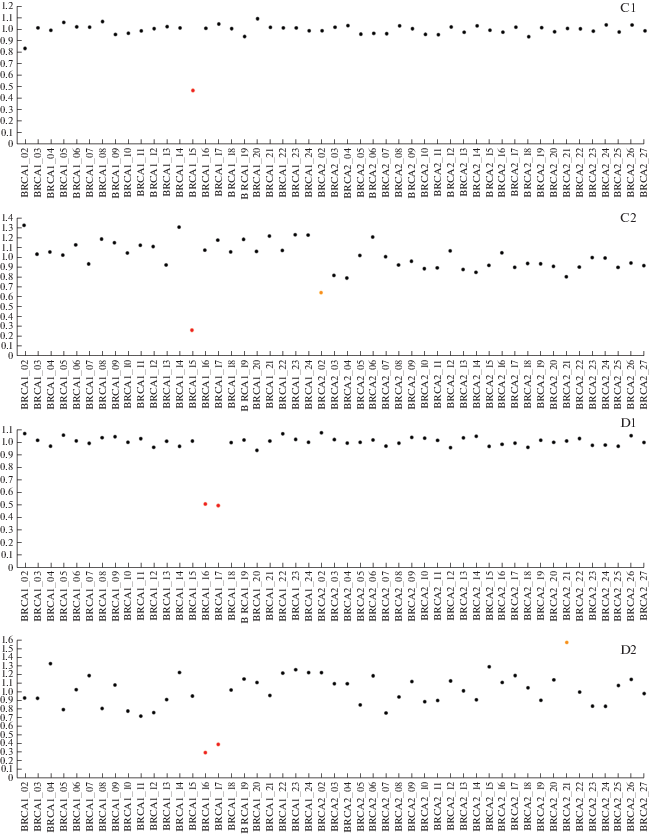
Рис. 1.
Продолжение.
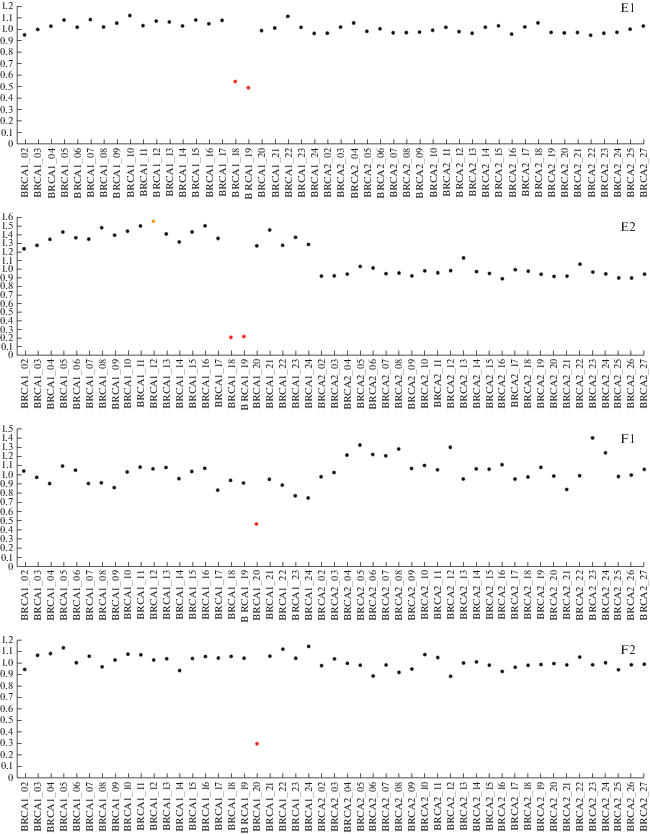
Рис. 1.
CNV гена BRCA1 в образцах крови и опухолевой ткани семи пациенток с HGSOC, выявленные методом NGS. A1 (кровь) – A2 (ткань) экзоны 1–2; B1 (кровь) – B2 (ткань) экзоны 1–11; C1 (кровь) – C2 (ткань) экзон 14; D1 (кровь) – D2 (ткань) экзоны 15–16; E1 (кровь) – E2 (ткань) экзоны 17–18; F1 (кровь) – F2 (ткань) экзон 19; G1 (кровь) – G2 (ткань): экзоны 20–21. Показаны результаты выявления CNV генов BRCA1/2 полученные методом NGS и программного обеспечения Amplicon Suite. CNV в гене BRCA1 обнаружены в образцах крови и опухолевой ткани. Программное обеспечение Amplicon Suite нормирует данные CNV в экзонах BRCA1 по референсной последовательности NM_007300.4. CNV считали делецией только при среднем соотношении менее 0.6. Образцы опухолей с областями-мишенями со средним соотношением 0.4 или менее считали гомозиготной делецией (0 копий гена) в результате наследственной делеции одного аллеля с потерей гетерозиготности другого. Согласно этим критериям, в образцах B, C, D, E и F возможна потеря гетерозиготности.
В гене BRCA1 выявлены следующие патогенные делеции (gLGR): делеции экзонов 1–2, 1–11, 14, 15–16, 17–18, 19 и 20–21.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В нашей работе показаны возможности выявления делеций экзонов в гене BRCA1 в опухолевых тканях методами целевого обогащения и NGS. Проанализированы образцы, полученные от семи пациенток с HGSOC, у которых LGR в гене BRCA1 были обнаружены ранее и подтверждены методами NGS и MLPA. Замороженные образцы первичных опухолей проанализированы нами методом NGTS. Этот метод обеспечил детекцию gLGR BRCA1 во всех использованных образцах опухолевой ткани. Уменьшение глубины покрытия отдельных экзонов приблизительно вдвое предполагает существование гетерозиготных экзонных делеций. Кроме того, в пяти образцах выявлена потеря гетерозиготности, что указывает на гомозиготную экзонную делецию и на высокую эффективность идентификации LGR с помощью NGTS, поскольку патогенный gLGR ведет себя, как и любой патогенный вариант BRCA. Следует отметить, что для получения столь хороших результатов необходима точная оценка качества образцов ДНК.
NGS BRCA-кодирующих локусов широко используется в клинической практике. Этот метод стал обычным средством молекулярного анализа при определении стратегии персонализированной терапии рака яичников и молочной железы [19, 20]. Около двух третей патогенных вариантов BRCA, выявляемых при раке яичников, относятся к наследственным. В одной трети случаев патогенные варианты встречаются исключительно в опухолевой ткани [21, 22]. Как следствие, сравнение соматических и наследственных изменений структуры tBRCA, выявляемых методом NGS в образцах, полученных при диагностической биопсии или на ранних стадиях опухолевого процесса, или при циторедуктивной хирургии и в образцах нормальной ткани приобретает диагностическое значение. Однако LGR в BRCA, даже если они составляют значительную долю от общего количества патогенных вариантов в образцах опухоли, редко учитываются из-за несовершенства методики.
Все протоколы определения LGR с помощью NGS основаны на использовании глубины покрытия, критерия, действительно важного для определения LGR [12, 23]. Секвенирование на основе NGS–CNV имеет ряд преимуществ перед секвенированием по Сэнгеру и MLPA. Наиболее очевидно, что для полного скрининга генов BRCA необходим один метод, который позволит анализировать одновременно однонуклеотидные полиморфизмы/инсерции, делеции и CNV. В течение последних 15 лет золотым стандартом идентификации всех клинически значимых патогенных вариантов BRCA считалось секвенирование по Сэнгеру и MLPA [24]. Однако эти дорогостоящие, пошаговые и трудоемкие технологии постепенно были заменены NGS, методом, имеющим более высокую пропускную способность и масштабируемость, более короткое время проведения анализа, а также более дешевым в пересчете на образец. Также стоит упомянуть, что NGS требует меньшего количества ДНК (30–50 нг), чем секвенирование по Сэнгеру или MLPA. Более того, определение LGR с использованием прочтений, полученных с помощью NGS, позволяет избежать ложноположительных результатов из-за мутаций в сайте гибридизации праймеров MLPA [23]. Даже в том случае, когда мутации и/или полиморфизмы приводят к ошибкам при отжиге праймеров, потенциально вызывая изменения покрытия конкретного ампликона, маловероятно, что весь экзон будет покрыт одним ампликоном NGS, минимизируя таким образом эффекты полиморфизма нуклеотидов на уровне экзона, и способствуя сохранению точности обнаружения CNV [11, 12].
Для анализа структуры BRCA и обнаружения CNV стратегия NGS с использованием метода целевого обогащения превосходит NGS на основе мультиплексной ПЦР, при которой возможно выпадение ампликонов, что создает недостаточное покрытие области и приводит к отсутствию гомогенности нормированного покрытия в целевых регионах. Таким образом, использование стратегии мультиплексной ПЦР, которая способна охватить целевые области с помощью максимального количества ампликонов, имеет первостепенное значение. Набор Devyser, используемый в этом исследовании, охватывает 188 ампликонов со средней длиной прочтения 196 п.н. (диапазон от 120 до 278 п.н.). Более того, он создает библиотеки секвенирования полных генов BRCA в одной пробирке, обеспечивая полное перекрывание ампликонов в каждой таргетной области [24]. Оценка определения LGR BRCA с помощью набора Devyser показала лишь немного более низкую точность в случае ампликонов экзона 2 BRCA1, что свидетельствует о наличии сложной области и, следовательно, о склонности к ложноположительным результатам определения CNV [25].
Основанное на NGS определение CNV позволяет точно анализировать аллели в герминальных/диплоидных клетках, но имеет ограничения при анализе гетерогенных соматических образцов. Связанная с этим биоинформатическая проблема остается открытой. Фактически, контаминация нормальными клетками, плоидность опухолевых клеток и гетерогенность опухоли искажают взаимосвязь между количеством прочтений и наличием CNV. Не все прочтения, сопоставленные с определенной областью, относятся к опухолевым клеткам. В результате количества прочтений могут не коррелировать с наличием CNV в опухоли, что затрудняет определение CNV и приводит к ложноположительным результатам. Однако, даже если CNV, обнаруженные в образцах опухоли, действительно оказываются ложноположительными, то анализ крови пациентов позволит в дальнейшем исключить их наследственное происхождение, ограничивая ложное обнаружение только приобретенными перестройками.
Фиксированные формалином и заключенные в парафин образцы ткани стабильны при комнатной температуре, хорошо хранятся и пригодны для создания больших коллекций образцов. Из-за этого фиксация формалином и заключение в парафин является стандартным методом получения образца ткани, который в дальнейшем можно использовать в молекулярных тестах.
Однако в таких образцах образуются белок-белковые и белково-нуклеиновые сшивки [26, 27], что связано с фиксацией формалином и заключением в парафин [28]. Секвенирование и последующий анализ ДНК, выделенной из заключенных в парафин образцов, представляет серьезную задачу. В этой связи в лабораториях должны независимо тестировать различные протоколы выделения и амплификации ДНК и выбирать метод, наиболее подходящий для данной лаборатории.
Замороженные образцы трудно собирать и транспортировать, дорого хранить. Однако такие образцы содержат минимально поврежденную ДНК, что позволяет получать препараты, сопоставимые по качеству с ДНК, выделенной из крови, что исключено при использовании фиксированных формалином образцов. В ДНК, выделенных из замороженных образцов, ожидаются меньшие колебания покрытия таргетной последовательности, что снижает количество ложноположительных результатов. Исходя из этого, мы предпочитали использовать замороженные образцы для анализа структуры генов. Идентификация LGR BRCA методом NGTS в замороженном опухолевом материале остается сложной задачей, однако мы смогли показать возможность ее решения. Учитывая полученные нами результаты и постоянное совершенствование технологии секвенирования и биоинформатических инструментов, мы подчеркиваем, что NGTS можно рассматривать как эффективную стратегию обнаружения всех патогенных вариантов BRCA. По-видимому, NGTS может стать предпочтительным методом обнаружения как соматических, так и наследственных патогенных вариантов BRCA, включая gLGR.
Мы благодарны Franziska M. Lohmeyer за критические замечания и редактирование текста статьи.
Концепция и дизайн: A. Minucci, C. Marchetti, G. Scambia, A. Fagotti. Сбор данных: осуществлен A. Minucci и G. Mazzuccato, анализ и интерпретация A. Minucci. Написание рукописи – A. Minucci. В окончательном утверждении рукописи участвовали все авторы.
От всех пациентов получено письменное информированное согласие.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Lee J.M., Ledermann J.A., Kohn E.C. (2014) PARP Inhibitors for BRCA1/2 mutation-associated and BRCA-like malignancies. Ann. Oncol. 25, 32–40.
Pujade-Lauraine E., Ledermann J.A., Selle F., Gebski V., Penson R.T., Oza A.M., Korach J., Huzarski T., Poveda A., Pignata S., Friedlander M., Colombo N., Harter P., Fujiwara K., Ray-Coquard I., Banerjee S., Liu J., Lowe E.S., Bloomfield R., Pautier P.; SOLO2/ENGOT-Ov21 investigators. (2017) Olaparib tablets as maintenance therapy in patients with platinum-sensitive, relapsed ovarian cancer and a BRCA1/2 mutation (SOLO2/ENGOT-Ov21): a double-blind, randomised, placebo-controlled, phase 3 trial. Lancet Oncol. 18, 1274–1284.
Ledermann J., Harter P., Gourley C., Friedlander M., Vergote I., Rustin G., Scott C.L., Meier W., Shapira-Frommer R., Safra T., Matei D., Fielding A., Spencer S., Dougherty B., Orr M., Hodgson D., Barrett J.C., Matulonis U. (2014) Olaparib maintenance therapy in patients with platinum-sensitive relapsed serous ovarian cancer: a preplanned retrospective analysis of outcomes by BRCA status in a randomised phase 2 trial. Lancet Oncol. 15, 852–861.
Endris V., Stenzinger A., Pfarr N., Penzel R., Möbs M., Lenze D., Darb-Esfahani S., Hummel M., Sabine-Merkelbach-Bruse., Jung A., Lehmann U., Kreipe H., Kirchner T., Büttner R., Jochum W., Höfler G., Dietel M., Weichert W., Schirmacher P. (2016) NGS-based BRCA1/2 mutation testing of high-grade serous ovarian cancer tissue: results and conclusions of the first international round robin trial. Virchows Arch. 468, 697–705.
Ewald I.P., Ribeiro P.L., Palmero E.I., Cossio S.L, Giugliani R., Ashton-Prolla P. (2009) Genomic rearrangements in BRCA1 and BRCA2: a literature review. Genet. Mol. Biol. 32, 437–446.
Concolino P., Rizza R., Hackmann K., Paris I., Minucci A., De Paolis E., Scambia G., Zuppi C., Schrock E., Capoluongo E. (2017) Characterization of a new BRCA1 rearrangement in an Italian woman with hereditary breast and ovarian cancer syndrome. Breast Cancer Res. Treat. 164, 497–503.
Puget N., Stoppa-Lyonnet D., Sinilnikova O.M., Pagès S., Lynch H.T., Lenoir G.M., Mazoyer S. (1999) Screening for germ-line rearrangements and regulatory mutations in BRCA1 led to the identification of four new deletions. Cancer Res. 59, 455–461.
Barrois M., Bièche I., Mazoyer S., Champème M.H., Bressac-de Paillerets B., Lidereau R. (2004) Real-time PCR-based gene dosage assay for detecting BRCA1 rearrangements in breast-ovarian cancer families. Clin. Genet. 65, 131–136.
Minucci A., De Paolis E., Concolino P., De Bonis M., Rizza R., Canu G., Scaglione G.L., Mignone F., Scambia G., Zuppi C., Capoluongo E. (2017) Competitive PCR-high resolution melting analysis (C-PCR-HRMA) for large genomic rearrangements (LGRs) detection: a new approach to assess quantitative status of BRCA1 gene in a reference laboratory. Clin. Chim. Acta. 470, 83–92.
Frolov A., Prowse A.H., Vanderveer L., Bove B., Wu H., Godwin A.K. (2002) DNA array-based method for detection of large rearrangements in the BRCA1 gene. Genes Chromosomes Cancer. 35, 232–241.
Concolino P., Mello E., Minucci A., Santonocito C., Scambia G., Giardina B., Capoluongo E. (2014) Advanced tools for BRCA1/2 mutational screening: comparison between two methods for large genomic rearrangements (LGRs) detection. Clin. Chem. Lab. Med. 52, 1119–1127.
Concolino P., Rizza R., Mignone F., Costella A., Guarino D., Carboni I., Capoluongo E., Santonocito C., Urbani A., Minucci A. (2018) A comprehensive BRCA1/2 NGS pipeline for an immediate Copy Number Variation (CNV) detection in breast and ovarian cancer molecular diagnosis. Clin. Chim. Acta. 480, 173–179.
Enyedi M., Jaksa G., Pintér L., Sükösd F., Gyuris Z., Hajdu A., Határvölgyi E., Priskin K., Haracska L. (2016) Simultaneous detection of BRCA mutations and large genomic rearrangements in germline DNA and FFPE tumor samples. Oncotarget. 20, 61845–61859.
Mafficini A., Simbolo M., Parisi A., Rusev B., Luchini C., Cataldo I., Piazzola E., Sperandio N., Turri G., Franchi M., Tortora G., Bovo C., Lawlor R.T., Scarpa A. (2016) BRCA somatic and germline mutation detection in paraffin embedded ovarian cancers by next-generation sequencing. Oncotarget. 7, 1076–1083.
Smith S.A., Easton D.F., Evans D.G., Ponder B.A. (1992) Allele losses in the region 17q12-21 in familial breast and ovarian cancer involve the wild-type chromosome. Nat. Genet. 2, 128–131.
Gudmundsson J., Johannesdottir G., Bergthorsson J.T., Arason A., Ingvarsson S., Egilsson V., Barkardottir R.B. (1995) Different tumor types from BRCA2 carriers show wild-type chromosome deletions on 13q12–q13. Cancer Res. 55, 4830–4832.
Maxwell K.N., Wubbenhorst B., Wenz B.M., De Sloover D., Pluta J., Emery L., Barrett A., Kraya A.A., Anastopoulos I.N., Yu S., Jiang Y., Chen H., Zhang N.R., Hackman N., D’Andrea K., Daber R., Morrissette J.J.D., Mitra N., Feldman M., Domchek S.M., Nathanson K.L. (2017) BRCA locus-specific loss of heterozygosity in germline BRCA1 and BRCA2 carriers. Nat. Commun. 8, 319.
Costella A., De Leo R., Guarino D., D’Indinosante M., Concolino P., Mazzuccato G., Urbani A., Scambia G., Capoluongo E., Fagotti A., Minucci A. (2018) High-resolution melting analysis coupled with next-generation sequencing as a simple tool for the identification of a novel somatic BRCA2 variant: a case report. Hum. Genome Var. 5, 10.
Minucci A., Scambia G., Santonocito C., Concolino P., Canu G., Mignone F., Saggese I., Guarino D., Cos-tella A., Molinario R., De Bonis M., Ferrandina G., Petrillo M., Scaglione G.L., Capoluongo E. (2015) Clinical impact on ovarian cancer patients of massive parallel sequencing for BRCA mutation detection: the experience at Gemelli hospital and a literature review. Exp. Rev. Mol. Diagn. 15, 1383–1403.
Percival N., George A., Gyertson J., Hamill M., Fernandes A., Davies E., Rahman N., Banerjee S. (2016) The integration of BRCA testing into oncology clinics. Br. J. Nurs. 25, 690–694.
George J., Alsop K., Etemadmoghadam D., Hondow H., Mikeska T., Dobrovic A., deFazio A; Australian Ovarian Cancer Study Group, Smyth G.K., Levine D.A., Mitchell G., Bowtell D.D. (2013) Nonequivalent gene expression and copy number alterations in high-grade serous ovarian cancers with BRCA1 and BRCA2 mutations. Clin. Cancer Res. 19, 3474–3484.
Cancer Genome Atlas Research Network. (2011) Integrated genomic analyses of ovarian carcinoma. Nature. 474, 609–615.
Schmidt A.Y., Hansen T.V.O., Ahlborn L.B., Jønson L., Yde C.W., Nielsen F.C. (2017) Next-generation sequencing-based detection of germline copy number variations in BRCA1/BRCA2: validation of a one-step diagnostic workflow. J. Mol. Diagn. 19, 809–816.
Wallace A.J. (2016) New challenges for BRCA testing: a view from the diagnostic laboratory. Eur. J. Hum. Genet. 24, S10–S8.
Capone G.L., Putignano A.L., Trujillo Saavedra S., Paganini I., Sestini R., Gensini F., De Rienzo I., Papi L., Porfirio B. (2018) Evaluation of a Next-generation sequencing assay for BRCA1 and BRCA2 mutation detection. J. Mol. Diagn. 20, 87–94.
Pikor L.A., Enfield K.S., Cameron H., Lam W.L. (2011) DNA extraction from paraffin embedded material for genetic and epigenetic analyses. J. Vis. Exp. 26, 49.
Tang W., David F.B., Wilson M.M., Barwick B.G., Leyland-Jones B.R., Bouzyk M.M. (2009) DNA extraction from formalin-fixed, paraffin-embedded tissue. Cold Spring Harb. Protoc. 2, pdb.prot513.
Ferrer I., Armstrong J., Capellari S., Parchi P., Arzberger T., Bell J., Budka H., Strobel T., Giaccone G., Rossi G., Bogdanovic N., Fakai P., Schmitt A., Riederers P., Al-Sarraj S., Ravid R., Kretzschmar H. (2007) Effects of formalin fixation, paraffin embedding, and time of storage on DNA preservation in brain tissue: a Brain Net Europe study. Brain Pathol. 17, 297–303.
Дополнительные материалы
- скачать ESM-Page_1.tiff
- Page 1. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_2.tiff
- Page 2. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_3.tiff
- Page 3. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_4.tiff
- Page 4. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_5.tiff
- Page 5. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_6.tiff
- Page 6. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
- скачать ESM-Page_7.tiff
- Page 7. Supplementary Figure. A1 (blood)-A2 (tissue): BRCA1 exons 1-2; B1 (blood)-B2 (tissue): BRCA1 exons 1 to 11; C1 (blood)-C2 (tissue): BRCA1 exon 14; D1 (blood)-D2 (tissue): BRCA1 exons 15-16; E1 (blood)-E2 (tissue): BRCA1 exons 17-18; F1 (blood)-F2 (tissue): BRCA1 exon 19; G1 (blood)-G2 (tissue): BRCA1 exons 20-21.This figure shows NGS/CNV data of all amplicons covering BRCA genes.
Инструменты
Молекулярная биология


