Молекулярная биология, 2020, T. 54, № 4, стр. 531-541
Расширяя генетический код: искусственные основания в биологических системах
С. А. Мукба a, b, c, П. К. Власов d, П. М. Колосов e, Е. Ю. Шувалова a, Т. В. Егорова a, Е. З. Алкалаева a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Физический факультет Московского государственного университета им. М.В. Ломоносова
119992 Москва, Россия
c Сколковский институт науки и технологий
121205 Московская обл., Сколково, Россия
d Institute of Science and Technology
3400 Klosterneuburg, Vienna, Austria
e Институт высшей нервной деятельности и нейрофизиологии Российской академии наук
117485 Москва, Россия
* E-mail: alkalaeva@eimb.ru
Поступила в редакцию 18.02.2020
После доработки 18.02.2020
Принята к публикации 18.02.2020
Аннотация
Генетический код предполагает использование пяти нуклеотидов (аденина, гуанина, цитозина, тимина и урацила), составляющих две пары для кодирования информации в ДНК и две пары для кодирования в РНК. Однако в последние годы для расширения генетического кода разрабатываются дополнительные пары оснований, использование которых увеличивает информационную емкость и разнообразие последовательностей нуклеиновых кислот, обеспечивая тем самым платформу для сайт-специфического включения функциональных компонентов в ДНК и РНК. Разработка таких расширенных систем позволила синтезировать многие искусственные пары оснований и протестировать их в различных условиях. Синтетические пары оснований в дальнейшем были модифицированы таким образом, чтобы устранить слабые места и позволить использовать их в конкретных целях. Более того, успешными оказались первые попытки создать полусинтетический организм, содержащий ДНК с искусственными парами оснований. Это еще больше расширило возможности применения искусственных пар оснований. В данном обзоре мы рассмотрим наиболее значимые из существующих синтетических пар оснований, а также их практическое применение.
ВВЕДЕНИЕ
Создание искусственных пар оснований (unnatural base pairs, UBP) – одно из наиболее значительных достижений синтетической биологии. Более 50 лет назад Александр Рич (A. Rich) впервые предположил возможность синтеза UBP [1], и 30 лет прошло с того момента, когда в лаборатории Стива Беннера (S.A. Benner) появилась первая пара искусственных нуклеотидов, специфично включающихся в ДНК [2]. Последние 20 лет разработка UBP стремительно развивается, и все более доступным становится их практическое применение и широкое использование в различных методах. Помимо практического значения, UBP позволили расширить возможности исследования фундаментальных процессов, лежащих в основе взаимодействия ДНК, РНК и полимераз.
Основные критерии, которые следует учитывать при создании UBP – эффективность и селективность репликации ДНК-полимеразой, например, фрагментом Кленова или Taq-полимеразой; легкость, с которой фрагменты ДНК, содержащие UBP, могут быть амплифицированы с помощью ПЦР; возможность транскрипции с этими парами; их стабильность и доступность. В настоящее время удалось синтезировать различные типы UBP, соответствующих этим критериям.
Все полученные на сегодняшний день UBP можно разделить на две группы: UBP, образующие водородные связи между основаниями (водородная комплементарность); и UBP с гидрофобными взаимодействиями внутри пары (структурная комплементарность). Первые синтезированные UBP обладали только водородными связями. Их топология отличалась от имеющейся в природных нуклеотидах, что обеспечивало специфичность при энзиматическом синтезе. Затем были получены гидрофобные UBP. Это доказало, что водородная связь не является единственным механизмом, способным обеспечить хранение и передачу генетической информации. На данный момент активно ведутся разработки и улучшение обоих видов UBP.
UBP С ВОДОРОДНЫМИ СВЯЗЯМИ
isoG–isoC
Первая пара синтетических нуклеотидов – изогуанин (isoG, 6-амино-2-кетопурин) и изоцитозин (isoC, 2-амино-4-кетопиримидин) была предложена А. Ричем еще в 1962 г. [1]. В этой паре используется нестандартная топология водородных связей между донором и акцептором протонов, которые отличаются от топологии в природных парах (рис. 1а, табл. 1). Однако впервые эта пара оснований была синтезирована под руководством С. Беннера [2]. Высокий потенциал этой искусственной пары стал ясен благодаря последующим экспериментам, посвященным репликации, транскрипции и трансляции нуклеиновых кислот, содержащих isoC–isoG [2–7]. Тем не менее, остались некоторые ограничения, связанные с таутомерной формой isoG, способной взаимодействовать с тимином, что приводит к ошибке репликации во время повторных циклов ПЦР [3]. Для решения этой проблемы вместо природного тимидинтрифосфата использовали 2-тиотимидинтрифосфат, TS (рис. 1б, табл. 1) [4]. Помимо проблемы, связанной с таутомеризацией isoG, обнаружено, что 2-аминогруппа isoC, соответствующая 2-кетогруппе в природных основаниях, хуже взаимодействует с ДНК- и РНК-полимеразами [4–7]. Кроме того, нуклеозид isoC нестабилен в щелочных и кислых условиях, что затрудняет получение содержащих его олигонуклеотидов путем химического синтеза. Группа Беннера решила эту проблему, введя метильную группу в пятое положение пиримидинового кольца isoC (isoCМе). В результате точность воспроизведения за один цикл ПЦР достигла 98%, но этого все еще было недостаточно, поскольку после 20 циклов ПЦР коэффициент удержания искусственной пары достигает примерно 67% (0.9820 = 0.67) [5].
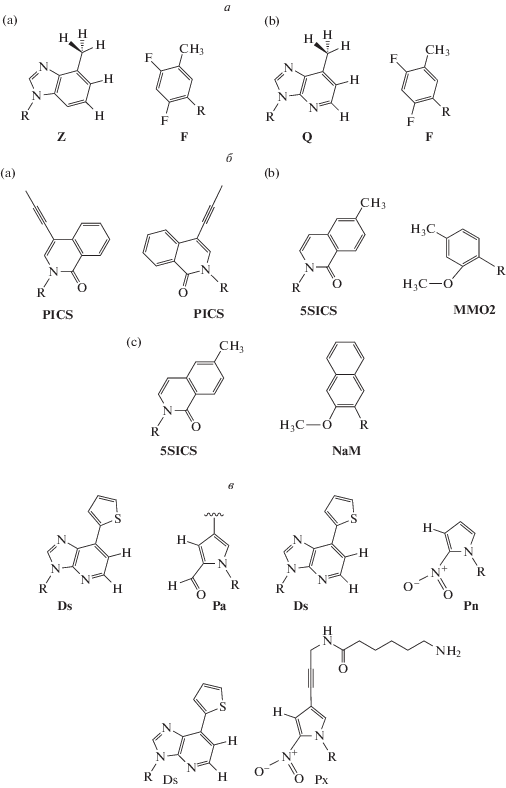
Рис. 1.
Искусственные пары оснований, образующих водородные связи, отличные от связей в естественных парах. а – Пара isoG–isoC [2]; б – стерическое наложение пары оснований 2-тиоT-isoG. Для решения проблемы возникновения связи между isoG и dT/U, вызванной таутомеризацией isoGИ, вместо природного dTTP предложено использовать 2-тиотимидиндезоксирибонуклеозидтрифосфат [4]; в – пара Z–P [10]; г – 5-met–isoC (S) и isoG (B), которые образуют hachimoji ДНК вместе с парой Z–P и природными нуклеотидами [17]; д – пары x–y, s–y и v–y [11–15].
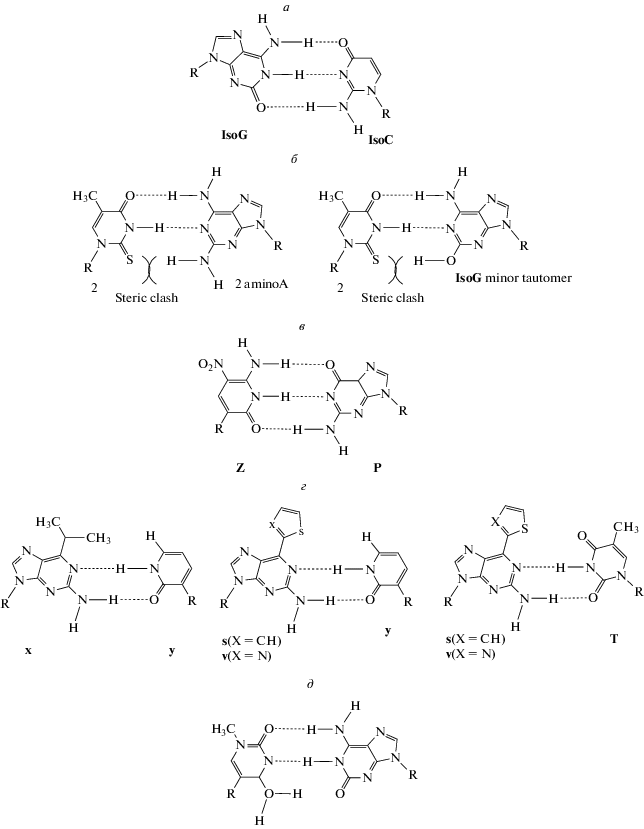
Таблица 1.
Искусственные пары оснований (UPB) и их применение
| UBP | Тип связи | Свойства и применение |
|---|---|---|
| isoG-met–isoC (B–S) | Водородная | Селективность в одном цикле ПЦР до 98%, что составляет приблизительно 67% в ПЦР с 20 циклами. Транскрипция возможна с определенными модификациями. Используется в количественной гибридизации. B и S также применяют в качестве строительных блоков в ДНК hachimoji |
| P–Z | Водородная | Селективность достигает за цикл ПЦР 99.8%; может использоваться в качестве молекулярного маяка. Одна из пар, из которой состоит ДНК hachimoji |
| x–y, s–y, v–y | Водородная | Низкая селективность, но возможны синтез белка и транскрипция |
| 5SICS–NaM | Гидрофобная | Селективность в ПЦР варьирует от 98 до 99.8%. Используется в амплификации при повреждениях ДНК, а также для создания первого полусинтетического организма |
| Ds–Pa | Гидрофобная | Высокая селективность в репликации (>99%). Транскрипция возможна с использованием Ds и модифицированного Pa |
| Ds–Px | Гидрофобная | Селективность в репликации составляет 99.8–99.9%. Модифицированные пары оснований Px: FAM-hx-Px и NH2-hx-Px являются флуоресцентными. Эти пары можно использовать как молекулярный маяк |
| Dss–Pn | Гидрофобная | Селективность в репликации составляет более 99.5%. Может использоваться как молекулярный маяк |
P–Z
После получения пары isoG–isoC и исследования ее свойств стало ясно, что для разработки синтетических оснований важно учитывать взаимодействие между парами оснований и полимеразами. Для взаимодействия со специфическими аминокислотными остатками в полимеразах в качестве акцепторов водородных связей необходимы N3 атомы аденина и гуанина и 2-кетогруппа цитозина и тимина, ориентированные в направлении малой бороздки. В результате дальнейших исследований была получена пара оснований Z–P (рис. 1в, табл. 1) [8, 9], что устранило большинство проблем, возникших при использовании isoG–isoC. В частности, ПЦР с новой парой P–Z не требовал пары A–TS для обеспечения специфичности, а основание P не образовывало нежелательные таутомеры в растворе, в отличие от isoG. Также были оптимизированы условия ПЦР для достижения 99.8% селективности за счет увеличения концентрации dCTP (от 0.2 до 0.6 мМ), что, по существу, устраняет неправильное связывание dZTP с гуанином. Встраивание dPTP напротив Z, обеспечивается увеличением концентрации dPTP до 0.6 мМ, при этом концентрацию dATP, dTTP и dGTP снижали до 0.1 мМ [10].
hachimoji
В 2019 году Стив Беннер и его коллеги объявили об успешном создании и анализе ДНК, состоящей из четырех природных и четырех синтетических нуклеотидов (dP, dZ, dS и dB, где S представляет собой met–isoC, а B – isoG) (рис. 1г, табл. 1). Показано, что dP–dZ и dS–dB ведут себя практически также, как природные пары оснований. Эта ДНК–РНК-подобная система была названа “hachimoji” (в переводе с японского это означает восемь букв) [11].
Стабильность цепей ДНК hachimoji оценивали по 40 термодинамическим параметрам. C этой целью были разработаны 94 hachimoji-дуплекса со всеми восемью нуклеотидами. Показано, что ДНК hachimoji может использоваться как информационная система, так как при молекулярном распознавании она ведет себя как стандартная 4-буквенная ДНК. Структурный анализ также показал, что ДНК-дуплексы hachimoji имели B-форму, аналогичную GC-богатой ДНК с небольшими отклонениями [11].
Для доказательства способности ДНК hachimoji передавать информацию необходимо было энзиматически получить соответствующую РНК-молекулу hachimoji. Оказалось, что Т7 РНК-полимераза, которую обычно используют для исследования способности синтетических нуклеотидов участвовать в транскрипции, не могла включать rSTP напротив dB, находящейся в ДНК-матрице. Поэтому использовали мутантную Т7 РНК-полимеразу (с заменами Y639F/H784A/P266L, названную FAL), способную транскрибировать весь набор нуклеотидов hachimoji. Этот фермент использовали для транскрипции матрицы, содержащей синтетические дезоксинуклеотиды, которая кодировала РНК-аптамер Spinach. В результате получили молекулу РНК, содержащую искусственные нуклеотиды и способную формировать флуоресцирующий аптамер, т.е. она транскрибировалась без существенных ошибок [11].
x–y, s–y, v–y
Альтернативные синтетические основания, образующие пары с водородными связями, были синтезированы в лаборатории Ичиро Хирао (I. Hirao) [12–15]. В этих парах с одной стороны находились 2-амино-6-модифицированные пурины, такие как 2-амино-6-(N,N-диметиламино)пурин (x) [12], 2-амино-6-(2-тиенил)пурин (s) [13, 14] и 2-амино-6-(2-тиазоил)пурин (v) [15], а с другой стороны – пиридин-2-он (y) [12] (рис. 1д, табл. 1). Селективность и эффективность репликации этих пар оснований уменьшается в ряду v–y > s–y > > x–y. Еще одно преимущество пар s–y и v–y состоит в том, что они обладают более высокой термостабильностью, чем пара х–y. Пара s–y оказалась наиболее подходящим кандидатом для использования в системе транскрипции, так как при нормальных условиях нуклеозидные производные v химически менее стабильны по сравнению с s. В то время как основание s казалось идеальным кандидатом для использования в транскрипции, основание y, образующее с ним водо-родную связь, имело ряд существенных недостатков. Во-первых, включение s в РНК комплементарно y, находящейся в ДНК, менее эффективно, чем включение y в РНК комплементарно s. Во-вторых, выяснилось, что у может встраиваться комплементарно не только s, но и А.
Поэтому было предложено новое основание, комплементарное s – имидазолин-2-он (z) [16], которое позволило решить проблему образования неправильной пары у–A. Однако эксперименты по репликации и транскрипции показали, что zTP недостаточно гидрофобен и поэтому имеет низкую способность к стэкинговым взаимодействиям, что, в свою очередь, снижает аффинность между ДНК-полимеразой и zTP. Кроме того, оказалось, что термическая стабильность дуплексных фрагментов ДНК, содержащих пару s–z, была ниже, чем у фрагментов, содержащих пару s–y. Хотя результаты экспериментов по транскрипции и трансляции казались убедительными, селективность репликации этих UBP все еще была недостаточной.
ГИДРОФОБНЫЕ СИНТЕТИЧЕСКИЕ ПАРЫ ОСНОВАНИЙ
Последующие эксперименты показали, что комплементарные взаимодействия между нуклеотидными парами могут образовываться не только за счет водородных связей, а для обеспечения селективности при репликации вполне достаточно структурного соответствия оснований друг другу [17].
F–Z, F–Q
Первая пара оснований, не образующих водородную связь, F–Z (рис. 2а, табл. 1), была синтезирована J.C. Morales и E.T. Kool [18]. Первоначально показали, что F встраивается напротив аденина с высокой эффективностью и точностью; пара F–A очень близка в этом отношении к паре Т–A [17, 18]. Эти результаты позволяют предположить, что геометрическое соответствие, даже в отсутствие водородной связи между парами оснований, может играть важную роль в направлении отбора нуклеотидов для репликации и транскрипции. ДНК со встроенным F тестировали в качестве матрицы для репликации фрагментом Кленова. dATP эффективно встраивался напротив F, в отличие от dCTP, dTTP, dGTP. Кроме того, показано встраивание dFTP напротив А.
Для образования пары с F было синтезировано основание Z, не образующее водородные связи [18]. Однако у этого основания отсутствует атом-акцептор водорода, соответствующий азоту в положении 3 у пуринов, который необходим для взаимодействия со специфическими аминокислотными остатками в полимеразах, т.е. распознавание полимеразами было подавлено. Задачу распознавания решили с помощью основания Q, у которого в этой позиции находится азот (рис. 2а, табл. 1). Действительно, фрагмент Кленова реплицировал пару Q–F с большей эффективностью, чем пару Z–F [18].
5SICS–NaM
Предложено также другое гидрофобное основание, названное PICS, которое комплементарно само себе в дцДНК [19]. Фрагмент Кленова может включать PICS в строящуюся цепочку ДНК напротив PICS в ДНК-матрице. К сожалению, большой размер пары PICS–PICS препятствует последующему удлинению цепи во время репликации: структура, образованная в результате сближения молекул PICS друг с другом при формировании пары, не распознается полимеразами [19–21]. Эта проблема решена с помощью изобретения и модификации гидрофобных пар оснований 5SICS–MMO2 и 5SICS–NaM, которые могут функционировать в качестве третьей пары оснований при амплификации ДНК и транскрипции с помощью Т7 РНК-полимеразы [19, 22]. В настоящее время наибольшая степень специфичности в ПЦР достигается с помощью пары оснований 5SICS–NaM – 99.8% (рис. 2б, табл. 1), результат, которому предшествуют огромные усилия группы F.E. Romesberg (Ф.Е. Ромсберг). Сосредоточившись на замене природных пуриновых или пиримидиновых оснований преимущественно гидрофобными аналогами, полученными на основе бензольных, нафталиновых, изокарбостирольных, пиридиновых и пиридоновых каркасов, они обнаружили, что количество UBP-кандидатов, образованных этими нуклеотидами, настолько большое, что индивидуальный анализ оказался нецелесообразным. Поэтому был проведен скрининг с анализом 3600 UBP-кандидатов. С помощью скрининга удалось идентифицировать пару, образованную dSICS и dMMO2 (dSICS–dMMO2), которая после оптимизации стала парой d5SICS–dMMO2 [21]. Впоследствии усилия по оптимизации UBP были сосредоточены на улучшении dMMO2 как пары для d5SICS, что привело к созданию пары d5SICS–dNaM [19, 22].
Ds–Pa, Ds–Px
В 2003 году обнаружили, что структурная комплементарность пары Q–F несовершенна, и атомы водорода в центре образования пары сталкиваются друг с другом. Эту проблему устранили путем замены шестичленного кольца F-основания на пятичленное. Таким образом был разработан Pa (пиррол-2-карбальдегид) [23]. Однако при использовании UBP Q–Pa обнаружились еще две проблемы: возможное образование пар Q–T и Q–Q вместо Q–Pa при ферментативном синтезе ДНК.
Следующая попытка репликации с гидрофобными нуклеотидами была осуществлена с использованием оснований s и Pa. Оказалось, что эта пара хорошо функционирует при сайт-специфическом включении sTP в РНК напротив Pa. Однако эту пару нельзя применять при репликации из-за способности s-основания образовывать водородные связи с природными основаниями. Поэтому пара s–Pa стала отправной точкой для дальнейших модификаций, что привело к созданию пар Ds–Pa и Ds–Px (рис. 2в, табл. 1) [24].
Был также разработан еще один, образующий пары с Pa, нуклеотид, Ds (7-(2-тиенил)имидазо (4,5-b)пиридин), где N1 атом в s заменен на СН, а 2-аминогруппа удалена (рис. 2в, табл. 1). Проблему образования пары Ds–Ds решили путем модификации субстрата Ds: обычный трифосфатный субстрат Ds был заменен на γ-амидотрифосфат Ds (dDsTPNH2). В результате, в 2006 году группой И. Хирао достигнута точность ПЦР за цикл >99% для пары оснований Ds–Pa. В этой работе использовали 3'-5'-экзонуклеазную активность ДНК-полимераз в сочетании с обычными трифосфатами и γ-амидотрифосфатами (модифицированными трифосфатами). Однако γ-амидотрифосфаты снижают эффективность амплификации ПЦР, что ограничивает применение этих оснований [25].
Поэтому на следующем этапе была создана система, не требующая γ-амидотрифосфатов. Сначала синтезировали 2-нитропиррол (Pn) (рис. 2в, табл. 1). Нитрогруппу в новое основание добавили для предотвращения неправильного встраивания A напротив Pa в результате электростатического отталкивания между кислородом нитрогруппы и N1 атомом A. Затем в положение 4 основания Pn ввели пропинильную группу и в качестве партнера по спариванию с Ds получили 4-[3-(6-аминогексанамидо)-1-пропинил]-2-нитропиррол (Px) (рис. 2в, табл. 1) [23]. С помощью пары Ds–Px достигнута беспрецедентная точность репликации фрагментов ДНК, содержащих искусственные основания, >99.9% [24].
Также обнаружили, что существенную роль в устранении синтетических нуклеотидов, неправильно встроенных во время репликации, играет экзонуклеазная активность полимеразы. Кроме того, выявлена зависимость эффективности и селективности пары Ds–Px в ПЦР от контекста последовательности вокруг этой пары: ДНК-матрицы, содержащие 3'-пурин-Ds-пурин-5', менее эффективны и селективны, чем последовательности, содержащие 3'-пиримидин-Ds-пиримидин-5'. Разработана пара UBP Ds–Diol–Px, селективность которой при репликации составляет 99.77–99.92% в зависимости от контекста последовательности, окружающей искусственный нуклеотид [26, 27].
ПРАКТИЧЕСКОЕ ПРИМЕНЕНИЕ ИСКУССТВЕННЫХ ПАР ОСНОВАНИЙ
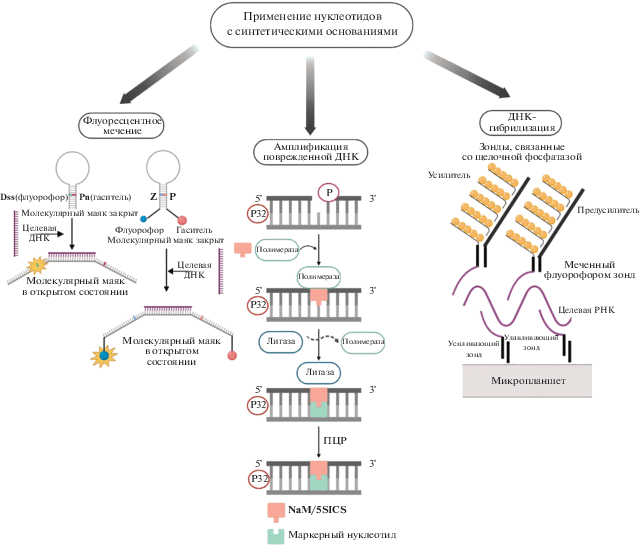
UBP, которые функционируют в качестве третьей пары оснований при репликации, транскрипции и трансляции, расширяют генетический алфавит и могут использоваться в широком спектре приложений, таких как сайт-специфическое флуоресцентное мечение, иммобилизация нуклеиновых кислот, анализ молекул ДНК и РНК. UBP, изначально синтезированные для расширения генетического алфавита, стали мощными инструментами мониторинга структурных изменений нуклеиновых кислот и их взаимодействия с другими молекулами, они используются также для обнаружения специфических последовательностей ДНК.
Одно из потенциальных применений UBP – флуоресцентное мечение нуклеиновых кислот (рис. 3, табл. 1) [27–33]. Обычное флуоресцентное мечение фрагментов ДНК имеет некоторые ограничения, связанные со случайным и частичным включением модифицированных субстратов в ДНК. Таким образом сложно добиться специфичного мечения мишени. Эту проблему можно решить с помощью UBP. Так, основания Ds и Px по своей природе не являются флуоресцентными, однако, используя некоторые модификации Ds, можно добиться их флуоресценции. С этой целью был разработан флуоресцентный аналог Ds, 7-(2,2'-битиен-5-ил)-имидазо[4,5-b] пиридин (Dss), который сильно флуоресцирует в составе олигонуклеотидов при 456 нм после возбуждения при 385 нм [27]. Другое синтетическое основание (Pn) действует как гаситель флуоресценции [29, 30]. Позднее сконструировали систему молекулярного маяка (molecular beacon) с парой Dss–Pn, действующей как пара флуорофор–гаситель.
Также были химически синтезированы два производных основания Px, модифицированные в положении 4-пропинила Px аминогексанамидной группой (NH2-hx-dPxTP) или (флуоресцеин-5-карбоксамидо) гексанамидной группой (FAM-hx-dPxTP) [30]. Эти модифицированные субстраты Px могут сайт-специфически включаться в ходе амплификации ДНК комплементарно Ds, что позволяет обнаруживать матрицы ДНК, содержащие Ds, по флуоресценции и далее выделять их на шариках с иммобилизованными антителами к FAM [24]. Используя этот метод, удалось выделить малое количество целевого фрагмента ДНК из большого количества контаминирующих фрагментов ДНК со случайными последовательностями после 30 циклов ПЦР. С помощью парных систем Dss–Pn, Dss–Px и Ds-(FAM-hx-dPxTP) показана способность UBP служить молекулярными маяками для обнаружения специфических последовательностей нуклеиновых кислот при ПЦР в реальном времени.
В 2008 году был рассмотрен альтернативный подход к проектированию молекулярных маяков. Такие молекулярные маяки представляют собой короткие олигонуклеотидные зонды, которые несут флуоресцентную молекулу на одном конце и гаситель флуоресценции на другом. Флуорофор и гаситель расположены близко друг к другу, когда зонд имеет вторичную структуру в виде шпильки. Когда же олигонуклеотид связывается с зондом, флуорофор и гаситель удаляются друг от друга. Это приводит к возникновению открытого состояния молекулярного маяка, обеспечивая таким образом генерацию сигнала флуоресценции, свободной от гашения. Одна из проблем использования данного метода – получение ложноположительных результатов. В частности, причиной возникновения такого рода ошибок может быть связывание олигонуклеотида не с петлей, а со стеблевой областью зонда. Для решения этой проблемы P. Sheng (П. Шенг) и соавт. предложили подход, опирающийся на искусственно расширенную генетическую информационную систему, которая включает UBP с водородными связями, а именно Z и P [34]. Они выбрали эту пару потому, что она стабильнее и дешевле, чем пара isoG–isoC. Кроме того, пара dZ–dP легко узнается многими нативными ДНК-полимеразами. Z и P, помещенные в область стебля, не уменьшают стабильность структуры зонда, тогда как присутствие этой синтетической пары в стебле не позволяет олигонуклеотиду, состоящему исключительно из природных нуклеотидов, связаться с данной областью. Все эти факторы делают пару Z–P одним из лучших кандидатов для создания последовательностей, содержащих синтетические нуклеотиды.
UBP можно использовать также в методе разветвленной ДНК-гибридизации (branched DNA assay) (рис. 3, табл. 1). Встраивание isoC и isoG в ДНК-зонд уменьшает уровень неспецифической гибридизации. Этот подход применен для количественного выявления ДНК вируса ВИЧ, а также вирусов гепатита B и C в крови [35–37].
J. Riedl (Дж Ридль.) и соавт. предложили новый метод детекции поврежденных участков ДНК (рис. 3, табл. 1). В этом методе критически важно встраивание искусственных нуклеотидов (dNaM или d5SICS) в месте повреждения в ходе эксцизионной репарации оснований. Этот метод позволяет синтезировать большие количества ДНК, содержащей синтетические нуклеотиды, с помощью ПЦР. С помощью этого метода исследованы повреждения в гене KRAS, и показана его эффективность и специфичность [38].
Одной из основных целей разработки UBP, помимо использования в различных in vitro подходах, является создание полусинтетических организмов, которые хранят, реализуют и передают расширенную генетическую информацию [39]. Первая попытка получить полусинтетический организм, который содержит UBP в ДНК, была успешно реализована группой F.E. Romesberg. Для включения в ДНК они выбрали пару синтетических гидрофобных нуклеотидов dNaM–d5SICS, которую синтезировали ранее [19–22]. Эта пара нуклеотидов удовлетворяла всем необходимым условиям: она могла встраиваться с высокой эффективностью и точностью в последовательность с любым контекстом, а также эффективно транскрибироваться в РНК. В качестве потенциального первого полусинтетического организма была выбрана Escherichia coli [39]. Авторы ожидаемо столкнулись с проблемой доставки синтетических нуклеотидов внутрь клетки, связанной с прохождением через две мембраны. Для решения этой проблемы прибегли к двум подходам. Первый включает пассивную диффузию свободных нуклеозидов в клетку с последующим их трифосфорилированием посредством последовательного действия киназ в пути восстановления нуклеозидов [40, 41]. Такой подход не оправдал ожиданий: уровня искусственных нуклеотидов в клетке было недостаточно для поддержания их в структуре ДНК. Поэтому они разработали альтернативный подход – исследовали способность восьми переносчиков нуклеозидтрифосфата из различных микроорганизмов транспортировать искусственные нуклеозидтрифосфаты в клетки. В результате один переносчик из Phaeodactylum tricornutum (PtNTT2) оказался активным как с d5SICSTP, так и с dNaMTP и мог импортировать их в клетки. В итоге впервые удалось создать полусинтетический организм, имеющий шестибуквенный генетический код [42–45].
ЗАКЛЮЧЕНИЕ
Системы UBP, описанные в нашем обзоре, имеют большой потенциал для использования в различных приложениях работы с нуклеиновыми кислотами. К искусственным основаниям могут быть присоединены практически любые функциональные группы, что позволяет наделять нуклеиновые кислоты желаемыми функциями. Так исследователи достигли впечатляющих результатов, модифицируя основания и используя их в качестве флуорофоров для различных типов секвенирования и ПЦР в реальном времени. А создание полусинтетических организмов с расширенным генетическим кодом позволяет моделировать в них совершенно новые функции, что дает широчайший простор для исследований. Кроме того, использование UBP также дает возможность увеличения функциональности белков путем включения нестандартных аминокислот в ходе транскрипции и трансляции. Нет сомнений в том, что будут найдены и другие возможности использования UBP, а уже известные UBP определенно не являются последними.
Список литературы
Rich A. (1962) Problems of evolution and biochemical information transfer. In: Horizons in Biochemistry. Eds Kasha M., Pullman B. New York: Acad. Press, pp. 103–126.
Switzer C., Moroney S.E., Benner S.A. (1989) Enzymatic incorporation of a new base pair into DNA and RNA. J. Am. Chem. Soc. 111, 8322–8323.
Piccirilli J.A., Krauch T., Moroney S.E., Benner S.A. (1990) Enzymatic incorporation of a new base pair into DNA and RNA extends the genetic alphabet. Nature. 343, 33–37.
Sismour A.M., Benner S.A. (2005) The use of thymidine analogs to improve the replication of an extra DNA base pair: a synthetic biological system. Nucl. Acids Res. 33, 5640–5646.
Switzer C.Y., Moroney S.E., Benner S.A. (1993) Enzymatic recognition of the base pair between isocytidine and isoguanosine. Biochemistry. 32, 10489–10496.
Horlacher J., Hottiger M., Podust V.N., Hubscher U., Benner S.A. (2006) Recognition by viral and cellular DNA polymerases of nucleosides bearing bases with nonstandard hydrogen bonding patterns. Proc. Natl. Acad. Sci. USA. 92, 6329–6333.
Benner S.A. (2004) Understanding nucleic acids using synthetic chemistry. Acc. Chem. Res. 37, 784–797.
Martinot T.A., Benner S.A. (2004) Artificial genetic systems: exploiting the ‘aromaticity’ formalism to improve the tautomeric ratio for isoguanosine derivatives. J. Org. Chem. 69, 3972–3975.
Yang Z., Hutter D., Sheng P., Sismour A.M., Benner S.A. (2006) Artificially expanded genetic information system: A new base pair with an alternative hydrogen bonding pattern. Nucl. Acids Res. 34, 6095–6101.
Yang Z., Sismour A.M., Sheng P., Puskar N.L., Benner S.A. (2007) Enzymatic incorporation of a third nucleobase pair. Nucl. Acids Res. 35, 4238–4249.
Hoshika S., Leal N.A., Kim M., Kim M., Karalkar N.B., Kim H., Bates A.M., Watkins N.E. Jr, SantaLucia H.A., Meyer A.J., DasGupta S., Piccirilli J.A., Ellington A.D., SantaLucia J. Jr, Georgiadis M.M., Benner S.A. (2019) Hachimoji DNA and RNA: system with eight building blocks. Science. 363, 884–887.
Ohtsuki T., Kimoto M., Ishikawa M., Mitsui T., Hirao I., Yokoyama S. (2001) Unnatural base pairs for specific transcription. Proc. Natl. Acad. Sci. USA. 98, 4922–4925.
Ishikawa M., Hirao I., Yokoyama S. (2000) Synthesis of 3-(2-deoxy-β-D-ribofuranosyl)pyridin-2-one and 2‑amino-6-(N,N-dimethylamino)-9-(2-deoxy-β-D-ribofuranosyl)purine derivatives for an unnatural base pair. Tetrahedron Lett. 41, 3931–3934.
Fujiwara T., Kimoto M., Sugiyama H., Hirao I., Yokoyama S. (2001) Synthesis of 6-(2-thienyl)purine nucleoside derivatives that form unnatural base pairs with pyridin-2-one nucleosides. Bioorganic Med. Chem. Lett. 11, 2221–2223.
Hirao I., Ohtsuki T., Fujiwara T., Mitsui T., Yokogawa T., Okuni T., Nakayama H., Takio K., Yabuki T., Kigawa T., Kodama K., Yokogawa T., Nishikawa K., Yokoyama S. (2002) An unnatural base pair for incorporating amino acid analogs into proteins. Nat. Biotechnol. 20, 177–182.
Mitsui T., Kimoto M., Harada Y., Yokoyama S., Hirao I. (2005) An efficient unnatural base pair for a base-pair-expanded transcription system. J. Am. Chem. Soc. 127, 8652–8658.
Johar Z., Zahn A., Leumann C.J., Jaun B. (2008) Solution structure of a DNA duplex containing a biphenyl pair. Chemistry. 14, 1080–1086.
Morales J.C., Kool E.T. (1998) Efficient replication between non-hydrogen-bonded nucleoside shape analogs. Nat. Struct. Biol. 5, 950–954.
Leconte A.M., Hwang G.T., Matsuda S., Capek P., Hari Y., Romesberg F.E. (2008) Discovery, characterization, and optimization of an unnatural base pair for expansion of the genetic alphabet. J. Am. Chem. Soc. 130, 2336–2343.
Seo Y.J., Hwang G.T., Ordoukhanian P., Romesberg F.E. (2009) Optimization of an unnatural base pair toward natural-like replication. J. Am. Chem. Soc. 131, 3246–3252.
Matsuda S., Fillo J.D., Henry A.A., Rai P., Wilkens S.J., Dwyer T.J., Geierstanger B.H., Wemmer D.E., Schultz P.G., Spraggon G., Romesberg F.E. (2007) Efforts toward expansion of the genetic alphabet: structure and replication of unnatural base pairs. J. Am. Chem. Soc. 129, 10466–10473.
Seo Y.J., Romesberg F.E. (2009) Major groove derivatization of an unnatural base pair. Chembiochem. 10, 2394–2400.
Mitsui T., Kitamura A., Kimoto M., To T., Sato A., Hirao I., YokoyamaS. (2003) An unnatural hydrophobic base pair with shape complementarity between pyrrole-2-carbaldehyde and 9-methylimidazo[(4,5)-b]pyridine. J. Am. Chem. Soc. 125, 5298–5307.
Kimoto M., Kawai R., Mitsui T., Yokoyama S., Hirao I. (2009) An unnatural base pair system for efficient PCR amplification and functionalization of DNA molecules. Nucl. Acids Res. 37(2), e14. https://doi.org/10.1093/nar/gkn956
Hirao I., Mitsui T., Kimoto M., Yokoyama S. (2007) Development of an unnatural base pair for efficient PCR amplification. Nucl. Acids Symp. Ser. (Oxf). 51, 9–10.
Hirao I., Kimoto M., Mitsui T., Fujiwara T., Kawai R., Sato A., Harada Y., Yokoyama S. (2006) An unnatural hydrophobic base pair system: site-specific incorporation of nucleotide analogs into DNA and RNA. Nat. Methods. 3, 729–735.
Yamashige R., Kimoto M., Takezawa Y., Sato A., Mitsui T., Yokoyama S., Hirao I. (2012) Highly specific unnatural base pair systems as a third base pair for PCR amplification. Nucl. Acids Res. 40, 2793–2806.
Collins M.L., Irvine B., Tyner D., Fine E., Zayati C., Chang C.A., Horn T., Ahle D., Detmer J., Shen L.P., Kolberg J., Bushnell S., Urdea M.S., Ho D.D. (1997) A branched DNA signal amplification assay for quantification of nucleic acid targets below 100 molecules/mL. Nucl. Acids Res. 25, 2979–2984.
Tyagi S., Kramer F.R. (1996) Molecular beacons: probes that fluoresce upon hybridization. Nat. Biotechnol. 14, 303–308.
Kimoto M., Mitsui T., Yokoyama S., Hirao I. (2010) A unique fluorescent base analogue for the expansion of the genetic alphabet. J. Am. Chem. Soc. 132, 4988–4989.
Kimoto M., Mitsui T., Yamashige R., Sato A., Yokoyama S., Hirao I. (2010) A new unnatural base pair system between fluorophore and quencher base analogues for nucleic acid-based imaging technology. J. Am. Chem. Soc. 132, 15418–15426.
Someya T., Ando A., Kimoto M., Hirao I. (2015) Site-specific labeling of RNA by combining genetic alphabet expansion transcription and copper-free click chemistry. Nucl. Acids Res. 43, 6665–6676.
Yamashige R., Kimoto M., Okumura R., Hirao I. (2018) Visual detection of amplified DNA by polymerase chain reaction using a genetic alphabet expansion system. J. Am. Chem. Soc. 140, 14038–14041.
Sheng P., Yang Z., Kim Y., Wu Y., Tan W., Benner S.A. (2008) Design of a novel molecular beacon: modification of the stem with artificially genetic alphabet. Chem. Commun. 41, 5128–5130.
Sherrill C.B., Marshall D.J., Moser M.J., Larsen C.A., Daudé-Snow L., Prudent J.R. (2004) Nucleic acid analysis using an expanded genetic alphabet to quench fluorescence. J. Am. Chem. Soc. 126, 4550–4556.
Moser M.J., Christensen D.R., Norwood D., Prudent J.R. (2006) Multiplexed detection of anthrax-related toxin genes. J. Mol. Diagnostics. 8, 89–96.
Johnson S.C., Marshall D.J., Harms G., Miller C.M., Sherrill C.B., Beaty E.L., Lederer S.A., Roesch E.B., Madsen G., Hoffman G.L., Laessig R.H., Kopish G.J., Baker M.W., Benner S.A., Farrell P.M., Prudent J.R. (2004) Multiplexed genetic analysis using an expanded genetic alphabet. Clin. Chem. 50, 2019–2027.
Riedl J., Ding Y., Fleming A.M., Burrows C.J. (2015) Identification of DNA lesions using a third base pair for amplification and nanopore sequencing. Nat. Commun. 6, 1–11.
Malyshev D.A., Romesberg F.E. (2015) The expanded genetic alphabet. Angew. Chemie Int. Ed. 54, 11930–11944.
Yan H., Tsai M.D. (1999) Nucleoside monophosphate kinases: structure, mechanism, and substrate specificity. Adv. Enzymol. Relat. Areas Mol. Biol. 73, 103–104.
Wu Y., Fa M., Tae E.L., Schultz P.G., Romesberg F.E. (2002) Enzymatic phosphorylation of unnatural nucleosides. J. Am. Chem. Soc. 124, 14626–14630.
Zhang Y., Ptacin J.L., Fischer E.C., Aerni H.R., Caffaro C.E., San Jose K., Feldman A.W., Turner C.R., Romesberg F.E. (2017) A semi-synthetic organism that stores and retrieves increased genetic information. Nature. 551, 644–647.
Malyshev D.A., Dhami K., Lavergne T., Chen T., Dai N., Foster J.M., Corrêa I.R., Romesberg F.E. (2014) A semi-synthetic organism with an expanded genetic alphabet. Nature. 509, 385–388.
Feldman A.W., Dien V.T., Karadeema R.J., Fischer E.C., You Y., Anderson B.A., Krishnamurthy R., Chen J.S., Li L., Romesberg F.E. (2019) Optimization of replication, transcription, and translation in a semi-synthetic organism. J. Am. Chem. Soc. 141(27), 10644–10653.https://doi.org/10.1021/jacs.9b02075
Feldman A.W., Romesberg F.E. (2017) In vivo structure-activity relationships and optimization of an unnatural base pair for replication in a semi-synthetic organism. J. Am. Chem. Soc. 139, 11427–11433.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология