Молекулярная биология, 2020, T. 54, № 4, стр. 542-561
Деградация РНК в эукариотических клетках
К. А. Татосян a, И. Г. Устьянцев a, Д. А. Крамеров a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: kramerov@eimb.ru
Поступила в редакцию 16.03.2020
После доработки 16.03.2020
Принята к публикации 19.03.2020
Аннотация
РНК – один из важнейших компонентов живых организмов, необходимый для экспрессии генов и ее регуляции в клетке. Механизмам синтеза РНК, особенно мРНК, всегда уделялось большое внимание, тогда как обстоятельные исследования путей распада РНК – процесса, не менее важного для жизнедеятельности клетки, начались сравнительно недавно. К настоящему времени многое известно о различных системах деградации РНК и их роли в жизнедеятельности эукариотических клеток. Установлено, что наряду с синтезом, распад РНК может быть важным механизмом, регулирующим ее уровень в клетке. В данном обзоре описаны основные белковые системы, обеспечивающие разрушение молекул РНК, пути распада кодирующих и некодирующих РНК, механизмы выявления дефектных молекул в ядре и цитоплазме, особенности структуры, обеспечивающие стабильность или короткое время жизни РНК в клетке.
ВВЕДЕНИЕ
Деградацию РНК в клетке осуществляют ферментные системы, расщепляющие фосфодиэфирные связи с образованием, в конечном итоге, мононуклеотидов. Как в ядре, так и в цитоплазме существует множество механизмов, направленных на быстрое выявление и удаление нефункциональных (дефектных) РНК. Время жизни таких молекул в клетке столь коротко, что их можно обнаружить только при инактивации факторов клеточных систем деградации. Стабильность же полноценных РНК в клетке значительно варьирует; она связана с функцией этих РНК и регулируется различными структурными элементами, а также специфическими химическими модификациями. Функция таких структурных элементов и модификаций в значительной мере состоит в защите молекул РНК от быстрого распада. По нашим оценкам время полужизни РНК, не имеющих защитных структур, в клетках млекопитающих составляет порядка 20–40 мин [1, 2]. Предлагается считать РНК с периодом полужизни менее 2 ч нестабильными, от 2 до 16 ч – стабильными, свыше 16 ч – высокостабильными [3].
Стабильность мРНК в цитоплазме обеспечивает кеп, локализованный на 5′-конце, и поли(А)-последовательность (хвост), взаимодействующая с поли(А)-связывающими белками (PABP), на 3′‑конце. Именно эти структуры обеспечивают физическое сближение 5′- и 3′-концов транслируемой мРНК и ее циркуляризацию, что препятствует доступу деаденилаз, декепирующих факторов и экзонуклеаз к мРНК [4]. Исключение составляют мРНК гистонов, которые лишены поли(А)-хвоста; их стабильность регулируется стебельно–петлевой структурой, расположенной на 3′-конце. У многих “типичных” мРНК в 3′-части также обнаруживаются определенные мотивы, повышающие стабильность и, вероятнее всего, тоже складывающиеся в стебельно–петлевые структуры [5]. Поли(A)-хвост способен увеличивать время жизни не только мРНК и длинных нкРНК, но и определенных транскриптов РНК-полимеразы III – РНК некоторых SINE (коротких мобильных генетических элементов, не кодирующих белки) [6].
Показано, что время жизни мРНК может определяться составом кодонов. Частота встречаемости разных тРНК, связывающихся с синонимичными кодонами, довольно сильно различается. Молекулы мРНК, включающие много кодонов, для расшифровки которых необходимы редкие тРНК, транслируются медленно и в большей степени подвержены распаду. Напротив, мРНК с оптимальным набором кодонов, транслируются быстрее и дольше не распадаются, хотя они имеют более короткий поли(A)-хвост, чем мРНК, у которых состав кодонов менее оптимален [7, 8].
Стабильность молекулы РНК часто зависит от устойчивости ее вторичной или третичной структуры. Так, установлено, что гипомодифицированные молекулы тРНК обладают менее устойчивой вторичной структурой и быстрее деградируют [9]. На примере малой нкРНК грызунов – 4.5SI РНК – показано, что такая сравнительно простая структура, как двухцепочечный стебель, образованный благодаря комплементарному взаимодействию 5′- и 3′-концевых участков молекулы, обеспечивает ее стабильность в клетке [1]. Длинные некодирующие РНК MALAT1 и NEAT1 избегают деградации в ядре благодаря стабильной трехспиральной структуре на ее 3′-конце [10].
Стабильность малых РНК может возрастать за счет специфических модификаций. Так, 2′-O-метилирование 3′-концевой рибозы – часто встречающаяся модификация, которая значительно повышает стабильность микроРНК, siРНК и piРНК. Такая модификация препятствует синтезу на 3′-конце молекулы олиго(U)-последовательности, которая считается распространенным сигналом деградации РНК [11].
Важным способом защиты РНК от клеточных нуклеаз является их постоянная ассоциация с белками. Например, рРНК входят в состав рибосомы, мяРНК – сплайсосомы [12], 7SL РНК – SRP-частицы [13], 7SK РНК – малого ядерного РНП [14].
ФЕРМЕНТЫ И КОМПЛЕКСЫ, ОБЕСПЕЧИВАЮЩИЕ РАСПАД РНК
По типу ферментативной активности выделяют три основных класса ферментов, разрушающих РНК (рибонуклеаз, или РНКаз): эндонуклеазы разрезают цепь РНК внутри, 5'-3'-экзонуклеазы осуществляют ее гидролиз с 5'-конца, а 3'-5'-экзонуклеазы разрушают молекулы РНК с 3'-конца. Клетка активно использует ферменты всех трех типов для деградации РНК. Конечными участниками очень многих метаболических путей распада РНК являются либо экзосомы, для которых характерны 3'-5'-экзонуклеазная и эндонуклеазная активности, либо экзонуклеазы Xrn, проявляющие 5'-3'-экзонуклеазную активность. Недавние исследования также указывают на 3'-5'-экзонуклеазу Dis3L2, как на “финального” участника сразу нескольких важных путей деградации РНК в цитоплазме [15, 16]. Основные пути деградации различных клеточных РНК и белки, обеспечивающие их распад, описаны ниже и отображены на схеме (рис. 1 ).
Экзосома. Экзосома – это основной рибонуклеазный комплекс который участвует как в контроле качества РНК и ее деградации, так и в процессинге некоторых типов РНК в клетках эукариот. Экзосомы консервативны, они обнаружены у всех исследованных на данный момент эукариот; схожие по структуре и функции белковые комплексы выявлены также в клетках эубактерий и архей. Коровая часть экзосомы состоит из девяти субъединиц и представляет собой двухслойное кольцо, образованное белковым гексамером (полипептиды Rrp41, Rrp42, Rrp43, Rrp45, Rrp46 и Mtr3), и “шапкой”, содержащей три белка (Rrp4, Rrp40 и Csl4). За нуклеазную функцию экзосомы отвечают субъединицы Rrp44 (обладает 3'-5'-экзонуклеазной и эндонуклеазной активностями, у человека – Dis3) и Rrp6 (3'-5'-экзонуклеаза, у человека – PM-Scl 100 или EXOSC10) [17].
Экзонуклеаза Rrp44/Dis3 высокопроцессивна: связавшись с субстратом, она полностью разрушает его, освобождая нуклеозид-5′-монофосфаты. 3′‑конец деградируемой РНК должен пройти сквозь коровое кольцо, чтобы достичь экзонуклеазного активного центра Rrp44/Dis3, поэтому длина неструктурированного одноцепочечного 3′-конца должна составлять не менее 35 н. [18]. Эндонуклеазный каталитический центр Rrp44/Dis3 расположен более поверхностно, поэтому РНК может его достичь как изнутри, пройдя сквозь коровый канал, так и снаружи комплекса (в этом случае эндонуклеаза может разрезать петли и другие структурированные участки РНК, подготавливая субстрат к расщеплению экзонуклеазами) [19]. Экзонуклеаза Rrp6 также осуществляет гидролиз РНК, высвобождая нуклеозид-5′-монофосфаты, однако, в отличие от Rrp44/Dis3, она дистрибутивна, т.е. отсоединяется от субстрата после катализа одной реакции [20]. Rrp6 связывается с “шапкой” коровой части экзосомы. Предполагается, что 3′-конец РНК проходит сквозь отверстие “шапки” и достигает каталитического центра Rrp6 через щель между “шапкой” и гексамером. Пока непонятно, от чего зависит будет ли конкретная РНК подвергаться деградации с помощью Rrp44/Dis3 или Rrp6. Ядерные экзосомы состоят из 11 субъединиц, они содержат и Rrp44/Dis3, и Rrp6; цитоплазматические же состоят из 10 субъединиц и лишены Rrp6 [21].
Ключевым вспомогательным фактором ядерных экзосом является хеликаза Mtr4. У дрожжей она входит в состав комплекса TRAMP (Trf4/5–Air1/2–Mtr4 polyadenylation complex). Этот комплекс включает неканоническую поли(А)-полимеразу Trf4 или Trf5, РНК-связывающий белок Air1 или Air2 и хеликазу Mtr4 [22–24]. Вариант TRAMP4 (Trf4–Air2–Mtr4) участвует, в основном, в контроле качества РНК в ядре, вариант TRAMP5 (Trf5–Air1–Mtr4) – в процессинге РНК [25]. TRAMP облегчает и усиливает активность ядерной экзосомы: с одной стороны, хеликаза Mtr4 разворачивает вторичные структуры в составе деградируемой РНК, с другой, Trf4/5 синтезирует на 3′-конце этой РНК олиго(А)-последовательность, которая служит удобным субстратом для посадки экзосомы. Trf4/5 и Air1/2 активируют Mtr4, которой для связывания субстрата требуется одноцепочечный концевой фрагмент из 5–6 н., а Mtr4, в свою очередь, регулирует длину олиго(А)-участка, не позволяя Trf4/5 синтезировать длинные поли(А)-хвосты [26]. У дрожжей комплекс TRAMP обеспечивает быструю деградацию практически всех РНК, служащих субстратами ядерных экзосом: аберрантных мРНК, рРНК, тРНК, мя/мякРНК и продуктов “повсеместной” транскрипции РНК-полимеразой II – CUT (cryptic unstable transcripts), а также участвует в процессинге пре-рРНК и пре-мя/мякРНК [27].
У млекопитающих хеликаза Mtr4 формирует два разных комплекса в ядрышках и в нуклеоплазме. В ядрышках Mtr4 связывается с белками Trf4-2 (гомолог Trf4 дрожжей) и ZCCHC7 (ближайший гомолог Air1/2), формируя аналог дрожжевого TRAMP – hTRAMP (human TRAMP). В нуклеоплазме Mtr4 вместе с РНК-связывающими белками RBM7 и ZCCHC8 образует другой комплекс – NEXT (Nuclear Exosome Targeting). hTRAMP и NEXT отличаются не только расположением в ядре, но и функцией: hTRAMP вовлечен в олигоаденилирование рРНК и тРНК, обеспечивая их деградацию экзосомой, а NEXT участвует в процессинге и деградации транскриптов РНК-полимеразы II (мРНК, мя/мякРНК, PROMPT – promoter upstream transcripts) [28].
В цитоплазме экзосома функционирует в комплексе со SKI (superkiller complex), который состоит из РНК-хеликазы Ski2 и РНК-связывающих белков Ski3 и Ski8 [29]. У дрожжей взаимодействие между SKI-комплексом и экзосомой осуществляет белок Ski7. Однако у млекопитающих он отсутствует; видимо, его функции берет на себя белок Hbs1 [30].
Экзонуклеаза Dis3L2. Dis3L2 (Dis3-like 3′-5′ exoribonuclease 2) является свободным цитоплазматическим ферментом, ответственным за деградацию многих РНК в 3′-5′-направлении. Эта нуклеаза сходна с экзосомной Dis3, но лишена N-концевого домена PIN, ответственного за эндонуклеазную активность и за связывание с остальной экзосомой. Каталитический домен Dis3L2 находится в средней части молекулы, в то время как в N-концевой части располагаются два домена холодового шока. Показана способность этого фермента in vitro разрушать как одно-, так и двухцепочечные РНК-субстраты. Олиго(U) на 3′-конце РНК служит сигналом к ее деградации Dis3L2 [16].
Экзонуклеазы Xrn. Основные 5′-3′-экзонуклеазы в клетках эукариот – это ферменты группы Xrn: цитоплазматическая Xrn1 (Pacman у Drosophila melanogaster) и ядерная Xrn2 (Rat1 у дрожжей). Они осуществляют процессивную деградацию 5′‑монофосфорилированных РНК-субстратов. Механизм работы Xrn1 исследован хорошо, в частности, известно, что субстратами этого фермента могут быть только некепированные (кеп слишком велик для активного центра фермента) РНК с одноцепочечным участком не менее чем из четырех нуклеотидов на 5′-конце. При этом сложная вторичная структура уже связавшейся с этим ферментом РНК разрушается по мере прохода в активный центр. Структуры Xrn1 и Xrn2 сходны в N-концевой части, особенно в области активного центра, и различаются в C-концевых участках [31]. У дрожжей для работы ядерной экзонуклеазы Rat1 необходим кофактор – пирофосфогидролаза Rai1, которая превращает 5′-трифосфат в монофосфат; у млекопитающих аналогичный кофактор Xrn2 пока не обнаружен [32].
ПРОЦЕССИНГ, КОНТРОЛЬ КАЧЕСТВА И ДЕГРАДАЦИЯ мРНК В ЯДРЕ
При процессинге пре-мРНК в ядре происходит присоединение кепа к 5′-концу, удаление некодирующих участков – интронов (сплайсинг), полиаденилирование 3′-конца РНК, связывание ее с упаковочными и экспортирующими факторами. Системы контроля качества мРНК работают на всех этих этапах и обеспечивают быструю деградацию дефектных молекул.
Кеп защищает мРНК от 5′-3′-экзонуклеазной деградации, обеспечивает процессинг пре-мРНК, экспорт в цитоплазму и инициацию трансляции [33]. Кепирование происходит вскоре после инициации транскрипции, когда синтезированная мРНК достигает длины 25–50 н., и напрямую связано с переходом РНК-полимеразы II в состояние элонгации [34]. Таким образом контролируется переход фермента к элонгации только в случае удачного кепирования, некепированные же транскрипты сразу подвергаются деградации 5′-3′-экзонуклеазой Xrn2/Rat1.
Сплайсинг пре-мРНК, как и кепирование, происходит котранскрипционно и осуществляется рибонуклеопротеидным комплексом – сплайсосомой. Удаляемые в результате сплайсинга интроны имеют форму лассо, поэтому сначала они разрезаются эндонуклеазой Dbr1 [35], а затем уже разрушаются с обоих концов молекулы ядерной экзосомой и экзонуклеазой Xrn2/Rat1. Пре-мРНК, которые по какой-то причине оказались несплайсированными, деградируют с помощью все тех же Xrn2/Rat1 (с предварительным декепированием комплексом Dcp1/Dcp2 с участием ядерного кофактора декепирования Lsm2-8) и ядерной экзосомы.
В клетках эукариот мРНК (кроме мРНК гистонов) подвергаются 3′-концевому полиаденилированию. Поли(А)-хвост выполняет несколько функций: он обеспечивает защиту 3′-конца молекулы от деградации экзосомой, участвует в транспорте мРНК из ядра в цитоплазму и в трансляции. Сигналом полиаденилирования служит гексануклеотид AAUAAA в 3′-части транскрибируемой области мРНК. Белок CPSF (Cleavage and Polyadenylation Specificiy Factor) связывается с ним, стимулирует разрезание мРНК и привлечение полиаденилполимеразы – PAP [36]. В клетках дрожжей поли(А)-хвост достигает длины 70–80 н., у млекопитающих – около 250 н. Он взаимодействует с поли(А)-связывающими белками (PABP, poly(A)-binding protein), молекулы которых присоединяются к РНК по ходу полиаденилирования. Минимальный сайт ассоциации белка PABP с поли(А)-хвостом – 12 расположенных подряд остатков аденозина; мРНК без поли(А)-хвоста, достаточного для связывания хотя бы одной молекулы PABP, подвергаются быстрой деградации [37, 38]. Поэтому деаденилирование является первым этапом практически всех механизмов деградации зрелой мРНК.
После AAUAAA-зависимого разрезания мРНК РНК-полимераза II продолжает синтезировать оставшуюся часть транскрипта. При этом 5′-конец этого транскрипта остается незащищенным и привлекает 5′-3′-экзонуклеазу Xrn2/Rat1, которая разрушает РНК. В процессе деградации РНК нуклеаза догоняет РНК-полимеразу II и сталкивается с ней, что стимулирует терминацию транскрипции по модели “торпедо” [39]. Транскрипты, которые не подверглись вовремя AAUAAA-зависимому разрезанию, разрушаются ядерной экзосомой [40].
В процессе синтеза мРНК связывается с белками, обеспечивающими ее правильную упаковку и экспорт. Комплекс THO, состоящий из белков Mft1, Tho2, Hpr1, Thp2 и Tex1, вместе с РНК-хеликазой Sub2 осуществляет экспорт мРНК из ядра дрожжей. Мутации в THO/Sub2 приводят к деградации транскриптов, причем причиной этого, по-видимому, является нарушение их полиаденилирования. Вероятно, AAUAAA-зависимое разрезание и полиаденилирование возможно только при сборке правильного мРНП-комплекса [41]. Дефекты в сборке мРНП приводят к удерживанию соответствующей мРНК около сайта транскрипции и последующей деградации с 5′- и/или 3′-конца [42].
Все описанные системы контроля качества синтезируемых мРНК и у дрожжей, и у высших эукариот работают котранскрипционно в непосредственной близости от комплекса элонгации транскрипции. После терминации транскрипции контроль качества мРНК становится значительно менее эффективным [43].
Наиболее популярная гипотеза, объясняющая принцип, по которому осуществляется контроль качества РНК – кинетическая [44]. Факторы деградации конкурируют с факторами процессинга, упаковки и экспорта за связь с мРНК, причем в случае полноценных молекул связывание с “правильными” белками энергетически выгодно и стимулирует переход к следующему этапу созревания. Дефектные же молекулы с меньшей вероятностью смогут быстро и правильно связаться с нужными белками, а значит, с большей вероятностью подвергнутся разрушению. Таким образом, факторы деградации не обязательно должны обладать способностью к выявлению разнообразных дефектов мРНК, так как они по умолчанию разрушают любые молекулы, которые не успели вовремя связаться с нужными белками и перейти к следующему этапу процессинга или экспортироваться из ядра [45]. При этом среди разрушенных таким образом мРНК имеется существенная доля нормальных молекул, которые просто по каким-то причинам также не смогли быстро связаться с нужными факторами [40].
Деградация РНК, опосредуемая PABPN1 и PAPα/γ (PPD). Относительно недавно был описан путь распада мРНК в ядре клеток млекопитающих, сопряженный с “избыточным” полиаденилированием таких мРНК канонической поли(A)-полимеразой (PAP, в одной из двух форм — PAPα или PAPγ), опосредуемый ядерным поли(A)-связывающим белком (PABPN1) и приводящий к деградации в ядерной экзосоме [46]. Сигналом к деградации в данном случае служит поли(A)-хвост длиной 300–800 н. Этот путь назван PPD (PABPN1 and PAPα/γ-mediated decay), он участвует в деградации большей части неверно сплайсированных мРНК, в составе которых оказался интрон, а также мРНК, которым не удалось образовать мРНП и экспортироваться из ядра. Некоторые нкРНК (см. ниже) также деградируют с помощью PPD [47].
ПУТИ РАСПАДА мРНК В ЦИТОПЛАЗМЕ
Деаденилирование и декепирование мРНК. Деаденилирование – первый шаг на пути деградации большинства мРНК. При этом длинный поли(А)-хвост укорачивается до размеров небольшого олиго(А)-участка. Деаденилирование обеспечивается поли(А)-специфичными 3′-5′-экзонуклеазами в составе комплексов Pan2–Pan3 и Ccr4–Not [38, 48]. У дрожжей комплекс Pan2–Pan3, по-видимому, не играет большой роли и работает в основном в ядре; деаденилирование в цитоплазме, предшествующее деградации мРНК, осуществляет комплекс Ccr4–Not. Деаденилирование же мРНК млекопитающих происходит в цитоплазме в два этапа с участием двух белковых комплексов соответственно.
Первый этап – укорачивание поли(А)-хвоста комплексом Pan2–Pan3, который связывается с PABP через Pan3. Pan2 – каталитическая субъединица. Pan2–Pan3 работает дистрибутивно и довольно медленно, постепенно укорачивая поли(А)-хвост до длины примерно 110 н. Затем PABP и некоторые факторы инициации трансляции уходят, уступая место репрессорам трансляции. мРНК становится нетранслируемой и может отправляться в Р-тельца (см. ниже) или претерпевать второй этап деаденилирования с помощью комплекса Ccr4–Not, который работает процессивно и быстро. Not является структурообразующим белком, тогда как каталитическая функция осуществляется деаденилазами Ccr4 и Caf/Pop2. Pan2–Pan3 и Ccr4–Not могут образовывать суперкомплекс, благодаря чему координируются два этапа деаденилирования [49]. Стоит отметить, что первый этап деаденилирования фактически начинается, как только мРНК попадает в цитоплазму. Именно он по умолчанию определяет время жизни мРНК, не содержащей какие-либо детерминанты короткого времени жизни [50].
После деаденилирования мРНК подвергается одному из двух путей деградации: с 5′- или с 3′‑конца. В первом случае с олиго(А) связывается комплекс Lsm1-7, который привлекает декепирующие белки Dcp1/Dcp2. Декепированная мРНК деградирует в 5′-3′-направлении с помощью экзонуклеазы Xrn1. По-видимому, это самый частый путь распада мРНК в цитоплазме [31]. Второй вариант – это деградация с 3′-конца с помощью цитоплазматической экзосомы с участием SKI-комплекса. В этом случае 5′-концевой участок (не более 10 н.) с кепом разрушается с помощью декепирующего белка DcpS [51]. Известно, что комплекс Lsm1-7, связываясь с олиго(А), ингибирует экзосомный путь деградации, являясь регулятором выбора пути распада мРНК. Пока, правда, неизвестно, что влияет на этот выбор [52].
В клетках дрожжей обнаружены специфические цитоплазматические гранулы, содержащие факторы декепирования (Lsm1, Dcp1/Dcp2), 5′-3′-экзонуклеазу Xrn1, интермедиаты деградации мРНК. Эти скопления названы процессирующими тельцами, или Р-тельцами. В них происходит декепирование и 5′-3′-деградация мРНК. Позже в Р-тельцах были обнаружены ферменты деаденилирования (Pan2, Pan3, Ccr4 и Caf1) и деаденилированные мРНК. Р-тельца клеток млекопитающих часто называют GW-тельцами. Р-тельца представляют собой специфические сайты, обеспечивающие не только разрушение мРНК, но и накопление, а также временное хранение нетранслируемых мРНК [53]. Такая изоляция в Р‑тельцах защищает клетку от случайной трансляции молекул не успевших подвергнуться деградации [49]. Таким образом, в цитоплазме мРНК может пребывать в двух пространственно и функционально дискретных сайтах: в составе полирибосом (транслируемое состояние) и Р-телец (нетранслируемое состояние) [54].
Уридилирование мРНК. Второй по частоте встречаемости модификацией 3′-концов клеточных РНК после полиаденилирования является уридилирование. Подобно полиаденилированию, оно осуществляется путем нематричного синтеза на 3′-конце РНК олиго(U)-, а изредка поли(U)-последовательности терминальными уридилтрансферазами (TUTases, ТУТазами). ТУТазы по структуре схожи с поли(A)-полимеразами и обычно лишены классического функционального РНК-узнающего мотива (RRM, RNA-recognition motif). Они либо способны взаимодействовать с любой РНК, либо нуждаются во вспомогательных факторах, и характеризуются различной субстратной специфичностью. Уридилированию подвергаются транскрипты всех трех РНК-полимераз эукариот, и одно из важнейших его назначений – регуляция стабильности мРНК. Молекулы мРНК уридилируются ТУТазами, в первую очередь, TUT4 (ZCCHC6) и TUT7 (ZCCHC11). Практически все ТУТазы выполняют свои функции в цитоплазме. Уридилированные РНК распознаются и утилизируются свободными нуклеазами Dis3L2 и 3′hExo (Eri1), а также экзосомой [55–57].
Полностью функциональные типичные мРНК обычно не уридилируются. Видимо, этому препятствует достаточно длинный поли(A)-хвост, поэтому для осуществления этой модификации требуется хотя бы частичное деаденилирование. Однако у дрожжей поли(A)-хвост относительно короткий, и у них уридилированию может подвергаться как деаденилированная, так и аденилированная мРНК. Показано, что укороченный олиго(A)-хвост, содержащий хотя бы два остатка уридина на конце, уже хорошо распознается комплексом Lsm1-7, направляющим декепирование, и эффективность этого связывания возрастает, если длина олиго(U)-последовательности достигает 5 н., а затем выходит на “плато”. У человека уридилировано порядка 20% мРНК с поли(A)-хвостом длиной менее 25 н., и такие мРНК подвергаются деградации [58–60]. В некоторых случаях уридилирование приводит к подавлению трансляции конкретной мРНК без ее деградации: олиго(U)-последовательность связывается с вышележащей частью молекулы, и трансляция останавливается [61, 62]. Интересно, что у Arabidopsis thaliana описан механизм, когда уридилтрансфераза Urt1 защищает деаденилированные (т.е., содержащие олиго(A)-хвосты) мРНК от деградации с 3′-конца. Однако и в данном случае уридилирование, скорее всего, не повышает стабильность мРНК в клетке, а требуется для того, чтобы обеспечить ее деградацию только в направлении от 5′- к 3′-концу [63].
Уридилирование мРНК с последующей деградацией Dis3L2 – это важный и относительно недавно описанный путь распада РНК в 3′-5′-направлении, в который не вовлечена экзосома. TUT4/7 и Dis3L2, функционируя вместе, по-видимому, ответственны за деградацию значительной части мРНК в клетках человека и других организмов [15, 64]. Они также активно участвуют в деградации большинства мРНК в ходе апоптоза. Dis3L2 Schizosaccharomyces pombe, вероятно, функционирует согласованно с Xrn1. Мишенями обоих ферментов зачастую служат одни и те же мРНК, причем оба они локализуются в P-тельцах [65].
Уридилирование также вовлечено в распад фрагментов мРНК, которые могут образовываться на предыдущих этапах деградации, таких как NMD (см. ниже), РНК-интерференция или при остановке рибосомы. В частности, в клетках человека фрагменты, остающиеся после разрезания, опосредованного микроРНК, уридилируются с помощью TUT2 и TUT3 (до 15 остатков), а затем подвергаются деградации, точный механизм которой пока неизвестен [57]. TUT4 и TUT7 ответственны за деградацию не только клеточных, но и вирусных РНК, участвуя, тем самым, в иммунном ответе. Активность этих ферментов наблюдается также при инфицировании человека вирусом гриппа [66].
Деградация мРНК гистонов. мРНК гистонов уникальны, потому что они не полиаденилированы. У млекопитающих 3′-концы этих мРНК содержат стебельно-петлевую структуру, которая защищает их от клеточных экзонуклеаз в отсутствие поли(А)-хвоста и определяет время жизни [67]. С этим элементом связываются белок SLBP (stem loop binding protein) и 3′-5′-экзонуклеаза Eri1 [68]. Стебельно-петлевая структура должна располагаться на расстоянии не более 45–80 н. от терминирующего кодона, что говорит о том, что при терминации рибосома должна находиться в непосредственной близости от SLBP. Важно, что мРНК гистонов транслируются только во время репликации ДНК и должны подвергаться деградации к концу S-фазы. Показано, что к концу S‑фазы или при ингибировании репликации ДНК мРНК гистонов подвергаются олигоуридилированию, вероятно, с помощью TUT7, которая привлекается белком SLBP [69]. Олиго(U)-хвост служит платформой для связывания комплекса Lsm1-7 с последующим декепированием и деградацией мРНК гистонов с помощью Xrn1 с 5′-конца или затравкой для экзосомной деградации с 3′‑конца [70]. В последнем случае в распаде мРНК также участвуют ТУТазы и Eri1, которые осуществляют несколько последовательных раундов олигоуридилирования и распада стебельно-петлевой структуры, после чего оставшаяся мРНК разрушается с помощью экзосомы. SLBP может взаимодействовать с хеликазой Upf1, которая расплетает стебельно-петлевую структуру, облегчая ее деградацию [71].
Деградация мРНК, опосредованная AU-богатым элементом (AMD, AU-rich element-mediated decay, ARE-mediated decay). AU-богатые элементы (ARE, AU-rich element) представляют собой последовательности длиной 50–150 н., часто содержащие несколько AUUUA-мотивов. Они расположены в 3′-нетранслируемой области (3′-НТО) многих эукариотических мРНК, имеющих короткое время жизни. Бóльшая часть таких мРНК кодирует белки, регулирующие клеточный рост или ответ клетки на внешние факторы. Экспрессиия генов именно таких белков нуждается в наиболее строгом контроле, в том числе, с помощью механизма быстрой деградации мРНК. Многие из этих генов являются протоонкогенами [72]. Стабильность ряда длинных нкРНК также негативно коррелирует с наличием AU-богатых элементов [3]. AU-богатый элемент обнаружен также в 5′-концевой области одной короткоживущей вирусной РНК (HSUR1, Herpesvirus saimiri U РНК1) [73].
Существуют клеточные факторы, которые связываются с АRE-BP (ARE-binding proteins) и регулируют стабильность мРНК как стимулируя деградацию, так и подавляя ее. Наиболее изученные АRE-связывающие белки, стимулирующие деградацию мРНК, – TTP, BRF1 и KSRP [74, 75]. АRE-связывающие белки, по-видимому, стимулируют быстрое деаденилирование АRE-содержащих мРНК. Далее мРНК подвергаются декепированию и экзонуклеазной деградации с 3′- или с 5′-конца [76, 77]. АRE-связывающий белок HuR, наоборот, стабилизирует мРНК. АRE-содержащие мРНК не деградируют в течение короткого времени до трансляции. Их защиту от дестабилизирующих белков осуществляет именно белок HuR. АRE-связывающий белок AUF1 может вызывать как быструю деградацию, так и стабилизацию АRE-содержащих мРНК в зависимости от конкретного субстрата [78].
mCRD-зависимая деградация мРНК c-fos. Интересно, что многие из короткоживущих мРНК, содержащих описанные АU-богатые элементы, включают и другие дестабилизирующие элементы, находящиеся в белоккодирующих регионах или в 3′-НТО. Таким образом, в клетках млекопитающих возник механизм двойной регуляции времени жизни мРНК. Существование двух детерминант быстрой деградации мРНК обеспечивает большую гибкость при клеточном ответе на внешние и внутренние сигналы.
Известны два независимых пути быстрой деградации мРНК c-fos в клетке: ARE-зависимый и ARE-независимый. Вторая детерминанта быстрой деградации мРНК c-fos, mCRD (major protein-coding-region determinant of instability), находится в белоккодирующей части. Интересно, что действие актиномицина Д и других ингибиторов транскрипции не влияет на функцию mCRD, хотя подавляет дестабилизирующий эффект ARE. Это подтверждает, что каждая из этих детерминант использует свой независимый путь деградации [79].
mCRD c-fos представляет собой участок из 320 н. Функциональный элемент mCRD – это 87-нуклеотидная богатая пуринами последовательность в его 5′-части; минимальное расстояние между mCRD и поли(А)-хвостом должно быть около 450 н. [80]. Механизм mCRD-зависимой деградации связан с процессом трансляции мРНК. С mCRD связывается Unr и другие вспомогательные факторы, которые также взаимодействуют с PABP на поли(А)-хвосте мРНК, циклизуя мРНК, и с деаденилазой Ccr4, активность которой при этом блокирована. Для активации деградации необходимо прохождение рибосомы по mCRD. Рибосома сбрасывает Unr и другие белки, освобождая конец поли(А)-хвоста и активируя деаденилазу [80, 81].
Похожий механизм ARE-независимой деградации присущ и мРНК c-myc, которая также содержит детерминанту быстрой деградации в белоккодирующей области помимо AU-богатого элемента в 3′-НТО. Как и у c-fos, этот ARE-независимый путь деградации c-myc, зависит от трансляции [82, 83].
Деградация мРНК, опосредованная GU-богатым элементом (GMD, GU-rich element-mediated decay, GRE-mediated decay). GU-богатые элементы обычно представляют собой GU-повторы или последовательности, содержащие UGUUUGUUUGU, найденные у дрозофилы и млекопитающих. Как и AU-богатые элементы, GRE расположены в 3′-НТО многих короткоживущих мРНК. К таким мРНК относятся матрицы белков, участвующих в дифференцировке, пролиферации, транскрипции, передаче сигналов и других процессах. В среднем GRE-содержащие мРНК более стабильны, чем ARE-содержащие. В редких случаях мРНК могут содержать оба типа элементов. Основные GRE-связывающие белки относятся к семейству CELF – CELF1, CELF2 и др. Интересно, что CELF1 и CELF2, а также HuR могут связываться и с GU-богатыми, и с AU-богатыми элементами. Предполагается, что CELF1 стимулируют быстрое деаденилирование и деградацию мРНК млекопитающих, привлекая деаденилазу PARN. Показано, что при стрессе с GRE-содержащими мРНК связывается CELF2 и, наоборот, стабилизирует их, транспортируя в стресс-гранулы. В клетках дрозофилы PARN не найдена, однако они содержат белок Cup, который взаимодействует с кеп-связывающим белком eIF4E и способен привлекать деаденилазу Ccr4. Подробности GRE-зависимой деградации мРНК пока не выяснены [84].
Нонсенс-опосредованный распад (NMD, nonsense-mediated decay). NMD – это механизм контроля мРНК, с помощью которого происходит быстрая деградация аберрантных (дефектных) молекул, содержащих кодоны преждевременной терминации трансляции – PTC (premature termination codon). В результате предотвращается накопление потенциально опасных для клетки белков с недосинтезированными С-концами. PTC могут возникать в результате геномных мутаций, ошибок транскрипции или неправильного сплайсинга мРНК. Возможно, NMD возник параллельно с появлением сплайсинга у эукариот как механизм, выявляющий и разрушающий попавшие в цитоплазму молекулы мРНК, содержащие неудаленные интроны (у млекопитающих большая часть таких мРНК разрушается в ядре с помощью PPD) [85]. Сейчас известно, что механизм NMD часто используется не только для разрушения аберрантных молекул в клетке, но и для регуляции экспрессии ряда генов. Субстратами NMD могут быть мРНК с дополнительной короткой открытой рамкой считывания, расположенной выше основной рамки, интрон в 3′-НТО или просто очень длинная 3′-НТО [86, 87]. Среди мРНК, деградирующих с помощью NMD, – мРНК факторов, участвующих в клеточном ответе на разные типы стресса [88–90], и самих факторов NMD [91]. Кроме того, с помощью альтернативного сплайсинга, сопряженного с механизмом NMD [92], осуществляется авторегуляция экспрессии некоторых клеточных белков: hnRNP L [93], PTB [94], факторов, ремоделирующих хроматин [95, 96] и др.
Деградация мРНК с помощью NMD выявлена у дрожжей [97], растений [98] и животных [86]. Белки, участвующие в этом процессе, довольно сильно различаются у разных групп организмов. Основные факторы, необходимые для NMD у животных – Upf1, Upf2, Upf3, Smg1, Smg5, Smg6, Smg7 и PNRC2. Upf1 – это АТР-связывающий белок с РНК-хеликазной активностью. Согласно большинству моделей NMD именно этот белок обеспечивает узнавание субстрата деградации [99]. Upf1 взаимодействует с факторами eRF1 и eRF3, что говорит о его участии и в терминации трансляции [100]. В комплексе с Upf2 и Upf3 белок Upf1 подвергается фосфорилированию киназой Smg1 [101]. Факторы Smg5, Smg6, Smg7 и PNRC2, взаимодействуя с фосфорилированным Upf1, непосредственно запускают деградацию мРНК. Существуют два варианта деградации мРНК после запуска NMD в клетках животных. Чаще всего Smg6 разрезает мРНК около PTC [102]. Образующиеся при этом 5′- и 3′-фрагменты мРНК подвергаются экзонуклеазному распаду (5′-концевые фрагменты – в экзосомах, 3′-концевые – с помощью экзонуклеазы Xrn1) без деаденилирования и декепирования [103]. Альтернативно Smg5 и Smg7 привлекают деаденилазы и экзонуклеазы; в этом случае путь NMD начинается с деаденилирования (Ccr4–Caf1), вслед за которым запускается экзосомная деградация в 3′-5′-направлении [104] или PNRC2 рекрутирует факторы декепирования Dcp1/Dcp2, после чего происходит распад мРНК с 5′-конца с помощью Xrn1 [105]. Последние данные указывают на то, что при NMD фрагменты мРНК могут уридилироваться TUT4 и TUT7, а затем деградировать с участием экзосомы или экзонуклеазы Dis3L2 [106].
Предложено несколько моделей, объясняющих, каким образом выявляется субстрат NMD, но ни одна из них пока не может полностью объяснить механизм. [107]. В модели маркера, расположенного ниже по ходу транскрипции (downstream marker model), основная роль принадлежит маркерным белкам, которые расположены на мРНК по ходу транскрипции ниже, чем PTC, и выше, чем нормальный терминирующий кодон. Пример такого маркера в клетках млекопитающих – большой белковый комплекс EJC (exon junction protein complex), связывающийся с мРНК в ядре примерно на 20–25 н. выше места слияния экзонов [108]. Модель ложного 3′-НТО основана на предположении, что терминация трансляции на преждевременном стоп-кодоне отличается от терминации на нормальном кодоне, так как происходит на увеличенном расстоянии от 3′-конца мРНК [109]. Предложена также модель [107], согласно которой NMD вообще не предполагает механизм выявления мРНК, содержащих PTC, и является последствием преждевременной терминации трансляции, из-за которой большая часть мРНК освобождается от-рибосом и становится доступной для клеточных нуклеаз.
Деградация мРНК, опосредованная Staufen1 (SMD, Staufen1-mediated decay). Staufen1 (Stau1) – это РНК-связывающий белок, который участвует в транспорте и трансляционном контроле мРНК. Обнаружено, что Stau1 обеспечивает короткое время жизни мРНК Arf1 (фактор АDP-рибозилирования) в клетке [110]. Stau1 связывается с определенным участком – 3′-НТО мРНК, называемым SBS (Stau1-binding site). SBS Arf1 представляет собой 19-нуклеотидную шпильку со 100-нуклеотидной петлей, расположенную на расстоянии 20–25 н. от терминирующего кодона [111]. SMD представляет собой механизм деградации, зависящий от трансляции: он осуществляется при нормальной терминации рибосомы на стоп-кодоне [110].
Рис. 1.
Основные пути деградации клеточных РНК. Гены различных типов РНК транскрибируются в ядре с помощью одной из трех РНК-полимераз. Как в ядре, так и в цитоплазме зрелые и незрелые транскрипты проходят контроль качества, и аберрантные молекулы РНК уничтожаются (PPD, RTD, NRD, NSD, NGD, NMD). Механизмы распада зрелых функциональных мРНК (SMD, NMD, РНК-интерференция) используются клеткой для регуляции экспрессии генов. Практически все пути распада РНК в итоге приводят к их деградации экзосомой (с участием кофакторов TRAMP, NEXT или SKI) либо к разрушению экзонуклеазами Xrn1/2 или Dis3L2.
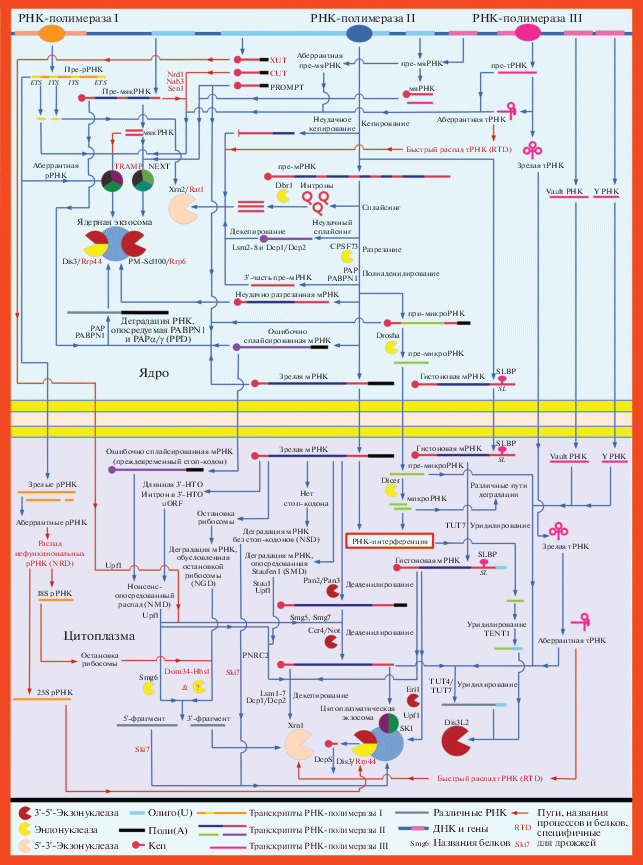
В клетке существуют и другие мРНК, служащие мишенями SMD. В основном они кодируют белки, участвующие в контроле клеточного метаболизма, транспорте органелл, делении клетки [110]. Однако в изученных мРНК-мишенях SMD, за исключением Arf1, SBS не обнаружены. Доказано [112], что образование сайта связывания Stau1 может быть обусловлено комплементарным связыванием SINE Alu внутри 3′-НТО мРНК с другим Alu-элементом в составе цитоплазматической полиаденилированной длинной нкРНК. Такие РНК были названы 1/2-sbsRNA, так как они содержат одну из комплементарных цепей, образующих сайт связывания белка Stau1. Одна такая нкРНК может дестабилизировать несколько мРНК [112]. Позже аналогичный механизм деградации мРНК обнаружили и у грызунов. В 3′-НТО таких мРНК находят SINE (B1, B2, B4 и ID), которые посредством комплементарного взаимодействия с такими элементами в составе длинных нкРНК формируют двухцепочечный сайт связывания с белком Stau1 и обеспечивают быструю деградацию мРНК [113]. Предполагается, что этот процесс вовлечен в регуляцию миогенеза [114]. Показано, что наряду со Stau1 в SMD может участвовать его паралог – белок Stau2 [115].
Механизм распада РНК при SMD сходен с NMD. Stau1/Stau2 связывается с белком Upf1, который фосфорилируется Smg1 и взаимодействует с PNRC2, который, в свою очередь, привлекает декепирующие белки Dcp1/Dcp2, после чего запускается процесс распада РНК с 5′-конца с помощью Xrn1. Пока неизвестно, участвуют ли белки Smg5, Smg6 и Smg7 в SMD [116].
Деградация мРНК без стоп-кодонов (NSD, nonstop decay). NSD – это путь распада РНК в клетке, направленный на удаление мРНК, в которых отсутствует стоп-кодон в открытой рамке считывания [117, 118]. Такие мРНК могут возникать из-за прерванной транскрипции, преждевременного полиаденилирования и, возможно, неполной экзосомной деградации. Также NSD помогает диссоциации связанной с аберрантной мРНК рибосомы, чтобы дать ей возможность садиться на другие матрицы.
Последовательность событий при NSD зависит от наличия поли(А)-хвоста и от того, где именно на мРНК остановилась рибосома. Если мРНК без стоп-кодона полиаденилирована, то рибосома продолжает трансляцию поли(А)-хвоста, что приводит к синтезу полилизиновой последовательности на конце пептида. Присутствие положительно заряженного полилизинового пептида в выходном туннеле рибосомы может привести к ее остановке. В этом случае деградация происходит по механизму NGD (см. ниже). Если же рибосоме удалось достичь конца поли(А)-хвоста, то с пустым А-сайтом рибосомы связывается белок Ski7, чей GTPазный домен напоминает аналогичный домен факторов трансляции EF1A и eRF3. Ski7 связывается с комплексом SKI и привлекает экзосому, которая осуществляет деградацию мРНК с 3′-конца без предварительного деаденилирования; диссоциацию рибосомы при этом обеспечивает белковый комплекс Dom34–Hbs1 [119]. Если мРНК не полиаденилирована, то после диссоциации рибосомы к незащищенному 3′-концу мРНК сразу привлекается экзосома (без участия Ski7 и SKI-комплекса). Полипептид также разрушается, подвергаясь убиквитинированию и протеасомной деградации [117].
Деградация мРНК, обусловленная остановкой рибосомы (NGD, no-go decay). NGD – это механизм деградации мРНК, вызванный остановкой рибосомы и невозможностью дальнейшей трансляции [120]. Причиной остановки может оказаться вторичная структура мРНК, редкий кодон, трансляция поли(А)-хвоста или нарушения в составе самой рибосомы. Этот путь деградации включает разрезание мРНК эндонуклеазой около места остановки рибосомы, которое узнается комплексом Dom34–Hbs1. Он привлекает неизвестную пока эндонуклеазу, которая производит разрез. Комплекс Dom34–Hbs1 также стимулирует диссоциацию рибосомы от мРНК. Далее происходит деградация 5′-фрагмента мРНК экзосомой (подобно механизму NSD) и распад 3′‑фрагмента с помощью экзонуклеазы Xrn1 (часто в Р-тельцах). Полипептид подвергается деградации [117].
Исследования NSD и NGD в основном проводили на дрожжах, однако на данный момент известно о существовании этих путей деградации мРНК в клетках и растений, и животных [121–124].
РНК-интерференция. РНК-интерференция – это механизм регуляции (подавления) экспрессии генов, основанный на комплементарном связывании небольших молекул РНК (микроРНК или эндогенных siРНК) с целевыми мРНК. Если комплементарность полная, то мРНК подвергается эндонуклеазному разрезанию комплексом RISC (RNA-induced silencing complex), если неполная, то трансляция репрессируется, но мРНК не разрезается. Сами микроРНК кодируются отдельными генами, первичные транскрипты которых, называемые при-микроРНК, кепированы и полиаденилированы подобно мРНК. При-микроРНК процессируются эндонуклеазой Drosha до пре-микроРНК, которые экспортируются из ядра и в цитоплазме разрезаются эндонуклеазой Dicer до зрелых микроРНК. РНК-интерференция – хорошо изученный процесс, сильно отличающийся от большинства других путей деградации РНК. Подробно он рассмотрен во многих обзорах, например, в [125–128].
МЕХАНИЗМЫ ДЕГРАДАЦИИ НЕКОДИРУЮЩИХ РНК
рРНК. Практически все РНК синтезируются в ядре в виде более длинных предшественников, которые затем подвергаются процессингу. РНК-полимераза I синтезирует полицистронную РНК, кодирующую три из четырех рРНК эукариот. Зрелые рРНК образуются путем ряда последовательных разрезаний и укорочений первичного транскрипта [129]. Вырезаются и деградируют два крайних фрагмента ETS (external transcribed spacer) и два внутренних ITS (internal transcribed spacer). Известно, что 5′-концевой фрагмент ETS разрушается ядерной экзосомой с участием TRAMP [130], ITS1 – экзонуклеазой Xrn2 [131].
Процесс деградации рРНК пока мало изучен из-за высокой стабильности этих молекул. Контроль качества зрелых рРНК в ядре осуществляет ядерная экзосома в комплексе с TRAMP. Пока неизвестно, по каким признакам факторы ядерной деградации РНК отличают аберрантные молекулы. Возможно, сигналом к разрушению рРНК служит невозможность ее правильного связывания с рибосомными белками. Также известно, что нарушение экспорта рибосомных субъединиц в цитоплазму приводит к высокому уровню деградации рРНК в ядре [132].
Распад нефункциональных рРНК (NRD, nonfunctional rRNA decay) – это механизм распознавания и разрушения дефектных зрелых рРНК, обнаруженый в клетках дрожжей [22, 133]. NRD происходит в цитоплазме и включает в себя два независимых пути: распад 25S рРНК, содержащих мутации, в пептидил-трансферазном центре (25S NRD), и распад 18S рРНК с мутациями в декодирующем центре (18S NRD). Субстраты 25S NRD накапливаются в цитоплазме около ядерной оболочки, и их деградация не зависит от трансляции, в то время как мутантные 18S рРНК рассеяны по цитоплазме, и их деградация прекращается в отсутствие синтеза белка в клетке.
Деградацию дефектных 18S рРНК обеспечивают те же белки, что и NGD мРНК: 5′-3′-экзонуклеаза Xrn1, цитоплазматическая экзосома, фактор Ski7, комплекс Dom34–Hbs1. Субстраты 18S NRD, как и NGD, часто локализуются в Р-тельцах. Причина сходства 18S NRD и NGD заключается, видимо, в том, что в обоих процессах сигналом деградации является рибосома, остановившаяся на мРНК. В случае 18S NRD причина этой остановки – ошибка в декодирующем центре 18S рРНК, в случае NGD – барьер на мРНК, препятствующий транслокации. В обоих случаях остановившаяся рибосома содержит значащий кодон в А-сайте. Возможно, что при остановке рибосомы как мРНК, так и 18S рРНК отправляются в Р‑тельца и подвергаются деградации [117, 134].
25S NRD не имеет сходства с другими механизмами деградации. Более того, этот процесс не зависит не только от трансляции, но и от всех белков, участвующих в различных типах распада РНК, кроме экзосомной экзонуклеазы Dis3. Предполагается, что механизм 25S NRD обусловлен невозможностью ассоциации рибосомных белков с дефектной 25S рРНК и связан с распадом белков [117, 134].
тРНК. В ядре тРНК подвергаются экзосомной деградации с предварительным олигоаденилированием комплексом TRAMP [23]. Бóльшая часть разрушаемых таким образом молекул – аберрантные предшественники и гипомодифицированные тРНК. Однако показано, что деградации в ядре также подвергаются и нормальные предшественники. При инактивации экзосомы уровень зрелых тРНК в таких клетках увеличивается, указывая на то, что немалая доля разрушаемых в норме пре-тРНК – функциональные молекулы, способные пройти нормальный процессинг [135]. Возможно, как и в случае мРНК, такая “неразборчивость” систем деградации связана с тем, что при контроле качества РНК сигналом к разрушению служат не столько ошибки, сколько кинетический фактор: если предшественник не успел быстро перейти к следующему этапу процессинга (т.е. связаться с соответствующими белками), он должен быть деградирован. Тем не менее, есть данные, что поли(А)-полимераза Trf4, входящая в соcтав TRAMP, может отличать гипомодифицированные тРНК от полностью модифицированных [136].
Функциональные тРНК очень стабильны: время их жизни измеряется часами и днями. В течение этого времени они проходят через цикл трансляции около 40 раз. Каждый тип тРНК содержит специфические модификации, которые обеспечивают их правильное узнавание и аминоацилирование. В клетках дрожжей качество зрелых тРНК контролируется механизмом, называемым “быстрый распад тРНК” (RTD, rapid tRNA decay). Гипомодифицированные молекулы быстро деацилируются и деградируют [137]. В этом процессе участвуют 5′-3′-экзонуклеазы Rat1 (в ядре) и Xrn1 (в цитоплазме), а также белок Met22 [138]. Предполагается, что ферменты деградации узнают гипомодифицированные тРНК по уменьшению стабильности вторичной структуры тРНК, особенно это касается акцепторного и T-стеблей [9]. Кроме того, сигналом к быстрой деградации путем RTD служит CCACCA, локализованный на 3′‑конце акцепторного стебля структурно нестабильных тРНК, вместо CCA у стабильных. Добавление CCA – важный этап созревания тРНК в ядре, который осуществляет тРНК-нуклеотидилтрансфераза. По-видимому, этот фермент “пытается” присоединить второй CCA-триплет ко всем тРНК, стимулируя при этом временное разворачивание акцепторного стебля. Стабильные молекулы достаточно прочны, они избегают этого разворачивания в отличие от нестабильных, которые оказываются помеченными CCACCA-сигналом и разрушаются. Стоит отметить, что такое CCACCA-мечение нестабильных тРНК характерно для всех царств живых организмов [139, 140].
Показано, что при окислительном стрессе клеток дрожжей, человека и растений тРНК могут разрезаться в антикодоновой петле. У дрожжей этот процесс осуществляет эндонуклеаза Rny1, которая при стрессе выходит из вакуоли в цитозоль [141].
Малые ядерные и малые ядрышковые РНК. Малые ядерные РНК (мяРНК) – это транскрипты РНК-полимераз II и III, участвующих в сплайсинге клеточных мРНК. Механизмы деградации этих РНК изучены недостаточно. За распад мяРНК, как и за их созревание, по-видимому, ответственны комплекс TRAMP и ядерная экзосома [142, 143]. В деградации зрелых U6 мяРНК у Saccharomyces cerevisiae также участвуют TRAMP и ядерная экзосома [144]. Имеются указания на то, что и у млекопитающих один из аналогов TRAMP, комплекс NEXT, способствует деградации U6 мяРНК, связываясь с U-богатыми последовательностями ее зрелых и незрелых вариантов [145]. В клетках человека при процессинге пре-U6 мяРНК уридилируется с помощью ядерной ТУТазы TENT1 (TUT6), в результате чего к четырем остаткам уридина на 3′-конце присоединяются еще 16. Затем 15 из них гидролизуются экзонуклеазой Usb1, а в остальных остатках уридина образуется связь между третьим и вторым фосфатами, что стабилизирует U6 мяРНК. Пре-U6 мяРНК без этой модификации распадается в ядерной экзосоме [146].
Дефектные U1 мяРНК дрожжей, неспособные сформировать правильный комплекс с белками, могут деградировать как в ядре с 3′-конца с помощью экзосомной экзонуклеазы Rrp6, так и в цитоплазме с участием декепирующего белка Dcp2 и 5′-3′-экзонуклеазы Xrn1. Аналогичный механизм деградации U1 мяРНК в цитоплазме работает и у млекопитающих [147]. Показано также, что неправильно процессированные предшественники мяРНК подвергаются уридилированию в цитоплазме с помощью TUT4/7 и последующей деградации экзонуклеазой Dis3L2 [148].
О деградации малых ядрышковых РНК (мякРНК), участвующих в модификации клеточных рРНК, тРНК и мяРНК, известно немного. В дрожжах мякРНК кодируются автономными генами, в то время как у высших эукариот мякРНК закодированы в интронах генов и вырезаются из соответствующих транскриптов. Как и в случае мяРНК, в их созревание вовлечена ядерная экзосома [149]. Показано, что у дрожжей предшественники этих РНК деградируют в экзосоме после олигоаденилирования комплексом TRAMP; процесс деградации находится в равновесии с процессом созревания предшественников мякРНК, опосредуемого белками Pab2 и Rrp6 [150, 151]. По-видимому, как и в случае мяРНК, в клетках млекопитающих в распаде мякРНК участвуют комплекс NEXT и ядерная экзосома. Также показано, что некодирующие транскрипты генов, которые содержат интроны, кодирующие мякРНК млекопитающих, могут деградировать путем PPD [47].
Vault РНК и Y РНК. Vault РНК и Y РНК – это транскрипты РНК-полимеразы III длиной порядка 100 н., которые участвуют в процессах аутофагии и инициации репликации ДНК соответственно [152, 153]. Геном человека содержит по четыре гена каждой из этих групп РНК. Vault РНК входят в состав крупных цитоплазматических рибонуклеопротеидных комплексов Vault. Значительная часть Y РНК, которые встречаются как в цитоплазме (cYtopasmic), так и в ядре, связаны в клетке с белками La и Ro60. Эти белки, вероятно, предохраняют Y РНК от деградации [15]. Как Vault, так и Y РНК уридилируются ТУТазами TUT4/7, а затем уничтожаются цитоплазматической экзонуклеазой Dis3L2 [148]. Однако, недавно проведенное исследование показало, что уровень Y РНК снижается в ответ на нокдаун деаденилазы PARN, которая отвечает за деградацию олиго(A)-хвоста, синтезируемого на 3′-конце этих РНК неканонической поли(A)-полимеразой PAPD5. Такой олиго(A)-хвост опосредует деградацию Y РНК с помощью экзосомной нуклеазы Dis3 [147]. Также известно, что и Vault, и Y РНК процессируются до более мелких фрагментов, называемых svRNA и YsRNA, деградация которых пока не исследована [15].
МикроРНК, эндогенные siРНК и piРНК. Деградация предшественников микроРНК, синтезируемых РНК-полимеразой II, пока плохо изучена. У млекопитающих пре-микроРНК могут уридилироваться цитоплазматическими ТУТазами TUT4/7. После уридилирования они подвергаются деградации с помощью экзосомных нуклеаз Rrp6 и Dis3 [15, 154]. Лучше всего изучены особенности функционирования и “жизненного цикла” микроРНК семейства let-7. Показано, что белковый фактор плюрипотентности Lin28 привлекает TUT4/7 к РНК-предшественнику let-7 (pre-let-7), в результате чего такая РНК больше не может взаимодействовать с Dicer и уничтожается цитоплазматической Dis3L2. Некоторые мутации в гене экзонуклеазы Dis3L2 вызывают синдром Перлмана, при котором наблюдается эмбриональный гигантизм. Вероятно, именно нарушения метаболизма let-7 приводят к развитию этого синдрома [155]. TUT4/7 уридилируют также и другие аберрантные пре-микроРНК, что приводит к их деградации все той же экзонуклеазой Dis3L2, причем в этом случае синтез олиго(U)-последовательности дистрибутивный из-за отсутствия вспомогательных факторов наподобие Lin28 [57]. Ядерные при-микроРНК, предшественники самих пре-микроРНК, могут подвергаться полиаденилированию и деградации путем PPD [47].
Что касается большинства зрелых малых РНК, регулирующих трансляцию мРНК (т.е. микроРНК, siРНК, piРНК), то описаны разные механизмы распада этих РНК у разных организмов. Широко распространено уридилирование, которое приводит к деградации этих РНК и “противостоит” 2′‑O-метилированию 3′-концевой рибозы, которое, напротив, стабилизирует эти РНК [56]. МикроРНК состоят из “гидовой” и “пассажирской” цепей, причем первая, как правило, отличается крайне высокой стабильностью (у мышей время полужизни составляет в среднем 119 ч), а вторая весьма уязвима для нуклеаз [156]. Известно множество путей деградации зрелых микроРНК – как с 5′-, так и с 3′-конца. В первом случае за это отвечают нуклеазы Xrn (цитоплазматическая Xrn1 – у человека и Caenorhabditis elegans, ядерная Xrn2 – только у C. elegans). С 3′-конца микроРНК могут подвергаться деградации экзосомой (человек и дрозофила), полинуклеотидфосфорилазой (человек), экзонуклеазой Eri1 (мышь), PARN (человек), Atrimmer2 и SDN (Small RNA Degrading Nucleases) (Arabidopsis) [15, 157, 158]. Зрелые микроРНК, связавшиеся с целевыми мРНК посредством комплементарного взаимодействия, распадаются с помощью механизма TDMD (target RNA-directed miRNA degradation), который сопряжен с уридилированием. Его осуществляет ТУТаза TENT1 (хотя не исключены и другие), а за деградацию отвечает экзонуклеаза Dis3L2 [61].
Зрелые piРНК у большинства организмов 2′‑O-метилированы по 3′-концу, за что отвечает метилтрансфераза Hen1. В ее отсутствие такие РНК уничтожаются неизвестной нуклеазой с 3′‑конца [159]. Эндогенные siРНК распадаются схожим образом; нуклеаза, ответственная за деградацию уридилированных форм этих РНК, также не выявлена [15].
Короткоживущие транскрипты РНК-полимеразы II. За последнее время в клетках эукариот обнаружено большое число разнообразных типов короткоживущих транскриптов РНК-полимеразы II [160, 161]. Как и мРНК, эти транскрипты кепированы по 5′-концу. Есть данные, что некоторые из этих РНК могут участвовать в регуляции экспрессии других генов. С другой стороны, большая часть этих транскриптов может оказаться нефункциональной, будучи лишь продуктом “повсеместной” работы РНК-полимеразы II.
Механизм деградации одного типа таких РНК у дрожжей – CUT (Cryptic unstable transcripts) – изучен довольно хорошо. CUT – это транскрипты длиной до 400 н., которые транскрибируются с межгенных участков вблизи промоторов генов мРНК и сразу разрушаются. CUT содержат сайты связывания с NNS-комплексом, который состоит из РНК-связывающих белков Nrd1 и Nab3 и хеликазы Sen1. Nrd1 и Nab3 связываются со специфическими мотивами в составе синтезируемых РНК (GUAA/G и UCUUG соответственно). Nrd1 также взаимодействует с C-концевым доменом РНК-полимеразы II и стимулирует терминацию транскрипции. NNS-комплекс привлекает TRAMP-комплекс и ядерную экзосому, с помощью которых происходит 3′-5′-экзонуклеазная деградация CUT [162]. Показано, что отдельные транскрипты CUT (в частности, SRG1) экспортируются из ядра и деградируют с участием декепирующего комплекса Dcp1/Dcp2 и экзонуклеазы Xrn1 [163].
Деградация короткоживущих транскриптов UNT (upstream non-coding transcripts) растений и PROMPT млекопитающих происходит в ядре и также зависит от ядерной экзосомы. У млекопитающих кофактором, привлекающим экзосому, является комплекс NEXT [164–167]. Однако по крайней мере часть PROMPT полиаденилируется и деградирует путем PPD. Кроме того, имеются данные о возможности котранскрипционного декепирования PROMPT с последующей деградацией Xrn2. Неясно, требуются ли различные пути деградации для разных “подмножеств” PROMPT, или все они могут подвергаться распаду любым из путей [47].
XUT (Xrn1 unstable transcripts), найденные в клетках дрожжей, кепируются и полиаденилируются в ядре подобно мРНК, а затем экспортируются в цитоплазму, где подвергаются деградации в 5′-3′-направлении экзонуклеазой Xrn1 [168]. Недавно показали, что XUT разрушаются в цитоплазме с помощью механизма NMD [169]. Оказалось, что эти РНК содержат короткие открытые рамки считывания, на которые садятся рибосомы и приступают к трансляции. Остановка этих рибосом на довольно большом расстоянии от 3′-конца молекулы приводит к запуску NMD с помощью Upf1, который стимулирует декепирование транскрипта и его разрушение экзонуклеазой Xrn1 [170]. Многие XUT образуют в ядре дуплексы с мРНК, что после транспортировки в цитоплазму защищает их от посадки рибосомы, а значит, и от деградации по механизму NMD. Однако хеликазы Mtr4 и Dbp2 могут расплетать такой дуплекс, если после его образования остается одноцепочечный 3′-концевой участок. Он, по-видимому, и служит платформой для посадки хеликазы [169].
ЗАКЛЮЧЕНИЕ
Деградации в клетке, в конечном счете, подвергаются все РНК, но время жизни конкретной молекулы зависит от ее типа. Значительная часть синтезируемой в клетке РНК разрушается очень быстро еще в ядре. Прежде всего, это фрагменты молекул, вырезаемые при процессинге различных типов РНК, которые разрушаются котранскрипционно, чаще всего экзонуклеазой Xrn2/Rat1, в непосредственной близости от места синтеза РНК. Многочисленные и, по-видимому, нефункциональные молекулы РНК, которые являются побочным продуктом активности РНК-полимеразы II (CUT, PROMPT, UNT), распадаются в ядре при помощи комплекса TRAMP (у млекопитающих – NEXT) и экзосомы. Кроме того, быстрой деградации в ядре подвергаются аберрантные РНК. Такие молекулы содержат ошибки, возникшие вследствие нарушений во время синтеза или процессинга РНК, поэтому они потенциально опасны для клетки. Эти РНК обычно подвергаются олигоаденилированию с помощью комплекса TRAMP и последующей деградации ядерной экзосомой. Аберрантные РНК, которым удалось избежать разрушения с помощью систем контроля качества в ядре, подвергаются деградации уже в цитоплазме. В последнее время становится ясно, что самые разнообразные высокоструктурированные аберрантные некодирующие РНК в цитоплазме уридилируются TUT4/7 и разрушаются экзонуклеазой Dis3L2 [61, 148, 171]. В клетках дрожжей для удаления дефектных рРНК в цитоплазме работает механизм NRD, а тРНК с недостаточным числом модификаций нуклеотидов распадаются при помощи RTD. Дефекты мРНК выявляются в процессе трансляции. Молекулы мРНК, содержащие преждевременные стоп-кодоны и интроны, разрушаются при помощи механизма NMD. Отсутствие стоп-кодона в мРНК приводит к ее распаду через NSD, а препятствие на пути рибосомы – к NGD. Механизмы запуска перечисленных путей деградации различны, но во всех случаях на конечном этапе молекулы дефектных РНК разрушаются с 3′- и/или с 5′-концов клеточными экзонуклеазами Dis3 (в составе экзосомы) и Xrn1 соответственно.
В клетке существуют и механизмы, которые направлены на деградацию полноценных функциональных РНК. Стабильные долгоживущие РНК, такие как рРНК или тРНК, разрушаются лишь тогда, когда теряют свою функциональность в результате накопления дефектов; в этом случае они удаляются благодаря механизмам NRD и RTD. Согласно последним исследованиям, ряд малых некодирующих РНК, таких как Y РНК, Vault РНК, BC1 и BC200 РНК, распадаются с участием TUT4/7 и Dis3L2 [148]. Деградация микроРНК, siРНК и piРНК также происходит путем уридилирования с последующим экзонуклеазным распадом с 3′-конца. Процесс распада мРНК чаще всего включает этапы деаденилирования и декепирования с последующей экзонуклеазной деградацией с 5′-конца (под действием Xrn1) или с 3′-конца (в результате активности цитоплазматической экзосомы). Также распространен вариант распада олигоаденилированных мРНК путем уридилирования TUT4/7, после которого следует декепирование и разрушение с 5′-конца нуклеазой Xrn1 или деградация с 3′-конца Dis3L2.
Деградация мРНК – это один из важнейших механизмов регуляции экспрессии генов. Молекулы мРНК часто содержат элементы первичной или вторичной структуры, которые обеспечивают их быструю деградацию или, напротив, высокую стабильность. Самыми распространенными сигналами быстрой деградации мРНК многих клеточных регуляторных факторов являются AU- или GU-богатые элементы; эти же элементы могут привлекать и стабилизирующие факторы. Регуляция экспрессии некоторых белков осуществляется благодаря изменению времени жизни их мРНК путем деградации через механизм NMD. Сигналами быстрого распада таких мРНК могут выступать длинная 3′-НТО, дополнительная короткая открытая рамка считывания выше основной рамки или неудаленный в результате альтернативного сплайсинга интрон. Реже в роли детерминанты, регулирующей время жизни мРНК, выступают элементы не первичной, а вторичной структуры, например, стебельно-петлевая структура в составе мРНК гистонов или двухцепочечная структура SBS, определяющая распад ряда мРНК через SMD.
Таким образом, в эукариотической клетке существует разветвленная сеть путей деградации РНК, функционирование которой направлено, с одной стороны, на защиту клетки от потенциально опасных нефункциональных молекул РНК, а с другой, на регуляцию экспрессии генов.
Работа выполнена при поддержке Российского фонда фундаментальных исследований (18-34-00247мол_а) и Российского научного фонда (19-14-00327).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Koval A.P., Gogolevskaya I.K., Tatosyan K.A., Kramerov D.A. (2012) Complementarity of end regions increases the lifetime of small RNAs in mammalian cells. PLoS One. 7, e44157.
Koval A.P., Gogolevskaya I.K., Tatosyan K.A., Kramerov D.A. (2015) A 5'-3'-terminal stem in small non-coding RNAs extends their lifetime. Gene. 555, 464–468.
Clark M.B., Johnston R.L., Inostroza-Ponta M., Fox A.H., Fortini E., Moscato P., Dinger M.E., Mattick J.S. (2012) Genome-wide analysis of long noncoding RNA stability. Genome Res. 22, 885–898.
Gorgoni B., Gray N.K. (2004) The roles of cytoplasmic poly(A)-binding proteins in regulating gene expression: a developmental perspective. Brief. Funct. Genomic Proteomic. 3, 125–141.
Goodarzi H., Najafabadi H.S., Oikonomou P., Gre-co T.M., Fish L., Salavati R., Cristea I.M., Tavazoie S. (2012) Systematic discovery of structural elements governing stability of mammalian messenger RNAs. Nature. 485, 264–268.
Устьянцев И.Г., Татосян К.А., Стасенко Д.В., Кочанова Н.Ю., Бородулина О.Р., Крамеров Д.А. (2020) Полиаденилирование транскриптов, считываемых РНК-полимеразой III с SINE, значительно удлиняет время их жизни в клетке. Молекуляр. биология. 54, 78–86.
Mugridge J.S., Coller J., Gross J.D. (2018) Structural and molecular mechanisms for the control of eukaryotic 5'-3' mRNA decay. Nat. Struct.Mol. Biol. 25, 1077–1085.
Nicholson A.L., Pasquinelli A.E. (2019) Tales of detailed poly(A) tails. Trends Cell Biol. 29, 191–200.
Whipple J.M., Lane E.A., Chernyakov I., D’Silva S., Phizicky E.M. (2011) The yeast rapid tRNA decay pathway primarily monitors the structural integrity of the acceptor and T-stems of mature tRNA. Genes Dev. 25, 1173–1184.
Brown J.A., Valenstein M.L., Yario T.A., Tycowski K.T., Steitz J.A. (2012) Formation of triple-helical structures by the 3'-end sequences of MALAT1 and MENbeta noncoding RNAs. Proc. Natl. Acad. Sci. USA. 109, 19202–19207.
Ji L., Chen X. (2012) Regulation of small RNA stability: methylation and beyond. Cell Res. 22, 624–636.
Papasaikas P., Valcarcel J. (2016) The spliceosome: the ultimate RNA chaperone and sculptor. Trends Biochem. Sci. 41, 33–45.
Wild K., Weichenrieder O., Strub K., Sinning I., Cusack S. (2002) Towards the structure of the mammalian signal recognition particle. Curr. Opin. Struct. Biol. 12, 72–81.
Peterlin B.M., Brogie J.E., Price D.H. (2012) 7SK snRNA: a noncoding RNA that plays a major role in regulating eukaryotic transcription. Wiley Interdiscip. Rev. RNA. 3, 92–103.
Labno A., Tomecki R., Dziembowski A. (2016) Cytoplasmic RNA decay pathways – Enzymes and mechanisms. Biochim. Biophys. Acta. 1863, 3125–3147.
Luan S., Luo J., Liu H., Li Z. (2019) Regulation of RNA decay and cellular function by 3'-5' exoribonuclease DIS3L2. RNA Biol. 16, 160–165.
Januszyk K., Lima C.D. (2014) The eukaryotic RNA exosome. Curr. Opin. Struct. Biol. 24, 132–140.
Bonneau F., Basquin J., Ebert J., Lorentzen E., Conti E. (2009) The yeast exosome functions as a macromolecular cage to channel RNA substrates for degradation. Cell. 139, 547–559.
Wasmuth E.V., Lima C.D. (2012) Exo- and endoribonucleolytic activities of yeast cytoplasmic and nuclear RNA exosomes are dependent on the noncatalytic core and central channel. Mol. Cell. 48, 133–144.
Januszyk K., Liu Q., Lima C.D. (2011) Activities of human RRP6 and structure of the human RRP6 catalytic domain. RNA. 17, 1566–1577.
Chlebowski A., Lubas M., Jensen T.H., Dziembowski A. (2013) RNA decay machines: the exosome. Biochim. Biophys. Acta. 1829, 552–560.
Doma M.K., Parker R. (2007) RNA quality control in eukaryotes. Cell. 131, 660–668.
Kadaba S., Wang X., Anderson J.T. (2006) Nuclear RNA surveillance in Saccharomyces cerevisiae: Trf4p-dependent polyadenylation of nascent hypomethylated tRNA and an aberrant form of 5S rRNA. RNA. 12, 508–521.
Vanacova S., Wolf J., Martin G., Blank D., Dettwiler S., Friedlein A., Langen H., Keith G., Keller W. (2005) A new yeast poly(A) polymerase complex involved in RNA quality control. PLoS Biol. 3, e189.
San Paolo S., Vanacova S., Schenk L., Scherrer T., Blank D., Keller W., Gerber A.P. (2009) Distinct roles of non-canonical poly(A) polymerases in RNA metabolism. PLoS Genet. 5, e1000555.
Jia H., Wang X., Anderson J.T., Jankowsky E. (2012) RNA unwinding by the Trf4/Air2/Mtr4 polyadenylation (TRAMP) complex. Proc. Natl. Acad. Sci. USA. 109, 7292–7297.
Tudek A., Lloret-Llinares M., Jensen T.H. (2018) The multitasking polyA tail: nuclear RNA maturation, degradation and export. Phil. Transact. Royal Soc. London. Ser. B, Biol. Sci. 373, 20180169.
Lubas M., Andersen P.R., Schein A., Dziembowski A., Kudla G., Jensen T.H. (2015) The human nuclear exosome targeting complex is loaded onto newly synthesized RNA to direct early ribonucleolysis. Cell Rept. 10, 178–192.
Halbach F., Reichelt P., Rode M., Conti E. (2013) The yeast ski complex: crystal structure and RNA channeling to the exosome complex. Cell. 154, 814–826.
Kalisiak K., Kulinski T.M., Tomecki R., Cysewski D., Pietras Z., Chlebowski A., Kowalska K., Dziembowski A. (2017) A short splicing isoform of HBS1L links the cytoplasmic exosome and SKI complexes in humans. Nucl. Acids Res. 45, 2068–2080.
Nagarajan V.K., Jones C.I., Newbury S.F., Green P.J. (2013) XRN 5'→3' exoribonucleases: structure, mechanisms and functions. Biochim. Biophys. Acta. 1829, 590–603.
Xiang S., Cooper-Morgan A., Jiao X., Kiledjian M., Manley J.L., Tong L. (2009) Structure and function of the 5'→3' exoribonuclease Rat1 and its activating partner Rai1. Nature. 458, 784–788.
Cougot N., van Dijk E., Babajko S., Seraphin B. (2004) “Cap-tabolism”. Trends Biochem. Sci. 29, 436–444.
Perales R., Bentley D. (2009) “Cotranscriptionality”: the transcription elongation complex as a nexus for nuclear transactions. Mol. Cell. 36, 178–191.
Chapman K.B., Boeke J.D. (1991) Isolation and characterization of the gene encoding yeast debranching enzyme. Cell. 65, 483–492.
Устьянцев И.Г., Голубчикова Ю.С., Бородулина О.Р., Крамеров Д.А. (2017) Каноническое и неканоническое полиаденилирование РНК. Молекуляр. биология. 51, 262–273.
Eckmann C.R., Rammelt C., Wahle E. (2011) Control of poly(A) tail length. Wiley Interdiscip. Rev. RNA. 2, 348–361.
Wilusz C.J., Wilusz J. (2008) New ways to meet your (3') end oligouridylation as a step on the path to destruction. Genes Dev. 22, 1–7.
Tollervey D. (2004) Molecular biology: termination by torpedo. Nature. 432, 456–457.
Porrua O., Libri D. (2013) RNA quality control in the nucleus: the Angels’ share of RNA. Biochim. Biophys. Acta. 1829, 604–611.
Saguez C., Schmid M., Olesen J.R., Ghazy M.A., Qu X., Poulsen M.B., Nasser T., Moore C., Jensen T.H. (2008) Nuclear mRNA surveillance in THO/sub2 mutants is triggered by inefficient polyadenylation. Mol. Cell. 31, 91–103.
Rougemaille M., Villa T., Gudipati R.K., Libri D. (2008) mRNA journey to the cytoplasm: attire required. Biol. Cell. 100, 327–342.
Schmid M., Jensen T.H. (2010) Nuclear quality control of RNA polymerase II transcripts. Wiley Interdiscip. Rev. RNA. 1, 474–485.
Hopfield J.J. (1974) Kinetic proofreading: a new mechanism for reducing errors in biosynthetic processes requiring high specificity. Proc. Natl. Acad. Sci. USA. 71, 4135–4139.
Houseley J., Tollervey D. (2009) The many pathways of RNA degradation. Cell. 136, 763–776.
Bresson S.M., Conrad N.K. (2013) The human nuclear poly(a)-binding protein promotes RNA hyperadenylation and decay. PLoS Genet. 9, e1003893.
Bresson S.M., Hunter O.V., Hunter A.C., Conrad N.K. (2015) Canonical poly(A) polymerase activity promotes the decay of a wide variety of mammalian nuclear RNAs. PLoS Genet. 11, e1005610.
Wahle E., Winkler G.S. (2013) RNA decay machines: deadenylation by the Ccr4-not and Pan2-Pan3 complexes. Biochim. Biophys. Acta. 1829, 561–570.
Zheng D., Ezzeddine N., Chen C.Y., Zhu W., He X., Shyu A.B. (2008) Deadenylation is prerequisite for P‑body formation and mRNA decay in mammalian cells. J. Cell Biol. 182, 89–101.
Chen C.Y., Shyu A.B. (2011) Mechanisms of deadenylation-dependent decay. Wiley Interdiscip. Rev. RNA. 2, 167–183.
Milac A.L., Bojarska E., Wypijewska del Nogal A. (2014) Decapping scavenger (DcpS) enzyme: advances in its structure, activity and roles in the cap-dependent mRNA metabolism. Biochim. Biophys. Acta. 1839, 452–462.
Chowdhury A., Mukhopadhyay J., Tharun S. (2007) The decapping activator Lsm1p-7p-Pat1p complex has the intrinsic ability to distinguish between oligoadenylated and polyadenylated RNAs. RNA. 13, 998–1016.
Luo Y., Na Z., Slavoff S.A. (2018) P-bodies: composition, properties, and functions. Biochemistry. 57, 2424–2431.
Sheth U., Parker R. (2003) Decapping and decay of messenger RNA occur in cytoplasmic processing bodies. Science. 300, 805–808.
Lim J., Ha M., Chang H., Kwon S.C., Simanshu D.K., Patel D.J., Kim V.N. (2014) Uridylation by TUT4 and TUT7 marks mRNA for degradation. Cell. 159, 1365–1376.
Munoz-Tello P., Rajappa L., Coquille S., Thore S. (2015) Polyuridylation in eukaryotes: a 3'-end modification regulating RNA life. BioMed Res. Internat. 2015, 968127.
Zigackova D., Vanacova S. (2018) The role of 3' end uridylation in RNA metabolism and cellular physiology. Phil. Transact. Royal Soc. London. Ser. B, Biol. Sci. 373, 20180171.
Chang H., Lim J., Ha M., Kim V.N. (2014) TAIL-seq: genome-wide determination of poly(A) tail length and 3' end modifications. Mol. Cell. 53, 1044–1052.
Rissland O.S., Norbury C.J. (2009) Decapping is preceded by 3' uridylation in a novel pathway of bulk mRNA turnover. Nat. Struct. Mol. Biol. 16, 616–623.
Song M.G., Kiledjian M. (2007) 3' Terminal oligo U-tract-mediated stimulation of decapping. RNA. 13, 2356–2365.
Haas G., Cetin S., Messmer M., Chane-Woon-Ming B., Terenzi O., Chicher J., Kuhn L., Hammann P., Pfeffer S. (2016) Identification of factors involved in target RNA-directed microRNA degradation. Nucl. Acids Res. 44, 2873–2887.
Lapointe C.P., Wickens M. (2013) The nucleic acid-binding domain and translational repression activity of a Xenopus terminal uridylyl transferase. J. Biol. Chem. 288, 20723–20733.
Sement F.M., Ferrier E., Zuber H., Merret R., Alioua M., Deragon J.M., Bousquet-Antonelli C., Lange H., Gagliardi D. (2013) Uridylation prevents 3' trimming of oligoadenylated mRNAs. Nucl. Acids Res. 41, 7115–7127.
Thomas M.P., Liu X., Whangbo J., McCrossan G., Sanborn K.B., Basar E., Walch M., Lieberman J. (2015) Apoptosis triggers specific, rapid, and global mRNA decay with 3' uridylated intermediates degraded by DIS3L2. Cell Repts. 11, 1079–1089.
Malecki M., Viegas S.C., Carneiro T., Golik P., Dressaire C., Ferreira M.G., Arraiano C.M. (2013) The exoribonuclease Dis3L2 defines a novel eukaryotic RNA degradation pathway. EMBO J. 32, 1842–1854.
Le Pen J., Jiang H., Di Domenico T., Kneuss E., Kosalka J., Leung C., Morgan M., Much C., Rudolph K.L.M., Enright A.J., O’Carroll D., Wang D., Miska E.A. (2018) Terminal uridylyltransferases target RNA viruses as part of the innate immune system. Nat. Struct. Mol. Biol. 25, 778–786.
Pandey N.B., Marzluff W.F. (1987) The stem-loop structure at the 3' end of histone mRNA is necessary and sufficient for regulation of histone mRNA stability. Mol. Cell. Biol. 7, 4557–4559.
Tan D., Marzluff W.F., Dominski Z., Tong L. (2013) Structure of histone mRNA stem-loop, human stem-loop binding protein, and 3'hExo ternary complex. Science. 339, 318–321.
Lackey P.E., Welch J.D., Marzluff W.F. (2016) TUT7 catalyzes the uridylation of the 3' end for rapid degradation of histone mRNA. RNA. 22, 1673–1688.
Mullen T.E., Marzluff W.F. (2008) Degradation of histone mRNA requires oligouridylation followed by decapping and simultaneous degradation of the mRNA both 5' to 3' and 3' to 5'. Genes Dev. 22, 50–65.
Hoefig K.P., Rath N., Heinz G.A., Wolf C., Dameris J., Schepers A., Kremmer E., Ansel K.M., Heissmeyer V. (2013) Eri1 degrades the stem-loop of oligouridylated histone mRNAs to induce replication-dependent decay. Nat. Struct. Mol. Biol. 20, 73–81.
Barreau C., Paillard L., Osborne H.B. (2005) AU-rich elements and associated factors: are there unifying principles? Nucl. Acids Res. 33, 7138–7150.
Fan X.C., Myer V.E., Steitz J.A. (1997) AU-rich elements target small nuclear RNAs as well as mRNAs for rapid degradation. Genes Dev. 11, 2557–2568.
Gherzi R., Lee K.Y., Briata P., Wegmuller D., Moroni C., Karin M., Chen C.Y. (2004) A KH domain RNA binding protein, KSRP, promotes ARE-directed mRNA turnover by recruiting the degradation machinery. Mol. Cell. 14, 571–583.
Sanduja S., Blanco F.F., Dixon D.A. (2011) The roles of TTP and BRF proteins in regulated mRNA decay. Wiley Interdiscip. Rev. RNA. 2, 42–57.
Mukherjee D., Gao M., O’Connor J.P., Raijmakers R., Pruijn G., Lutz C.S., Wilusz J. (2002) The mammalian exosome mediates the efficient degradation of mRNAs that contain AU-rich elements. EMBO J. 21, 165–174.
Stoecklin G., Mayo T., Anderson P. (2006) ARE-mRNA degradation requires the 5'-3' decay pathway. EMBO Repts. 7, 72–77.
Gratacos F.M., Brewer G. (2010) The role of AUF1 in regulated mRNA decay. Wiley Interdiscip. Rev. RNA. 1, 457–473.
Shyu A.B., Belasco J.G., Greenberg M.E. (1991) Two distinct destabilizing elements in the c-fos message trigger deadenylation as a first step in rapid mRNA decay. Genes Dev. 5, 221–231.
Grosset C., Chen C.Y., Xu N., Sonenberg N., Jacquemin-Sablon H., Shyu A.B. (2000) A mechanism for translationally coupled mRNA turnover: interaction between the poly(A) tail and a c-fos RNA coding determinant via a protein complex. Cell. 103, 29–40.
Chang T.C., Yamashita A., Chen C.Y., Yamashita Y., Zhu W., Durdan S., Kahvejian A., Sonenberg N., Shyu A.B. (2004) UNR, a new partner of poly(A)-binding protein, plays a key role in translationally coupled mRNA turnover mediated by the c-fos major coding-region determinant. Genes Dev. 18, 2010–2023.
Lemm I., Ross J. (2002) Regulation of c-myc mRNA decay by translational pausing in a coding region instability determinant. Mol.Cell. Biol. 22, 3959–3969.
Wisdom R., Lee W. (1991) The protein-coding region of c-myc mRNA contains a sequence that specifies rapid mRNA turnover and induction by protein synthesis inhibitors. Genes Dev. 5, 232–243.
Vlasova-St Louis I., Dickson A.M., Bohjanen P.R., Wilusz C.J. (2013) CELFish ways to modulate mRNA decay. Biochim. Biophys. Acta. 1829, 695–707.
Jaillon O., Bouhouche K., Gout J.F., Aury J.M., Noel B., Saudemont B., Nowacki M., Serrano V., Porcel B.M., Segurens B., Le Mouel A., Lepere G., Schachter V., Betermier M., Cohen J., Wincker P., Sperling L., Duret L., Meyer E. (2008) Translational control of intron splicing in eukaryotes. Nature. 451, 359–362.
Mendell J.T., Sharifi N.A., Meyers J.L., Martinez-Murillo F., Dietz H.C. (2004) Nonsense surveillance regulates expression of diverse classes of mammalian transcripts and mutes genomic noise. Nat. Genet. 36, 1073–1078.
Schweingruber C., Rufener S.C., Zund D., Yama-shita A., Muhlemann O. (2013) Nonsense-mediated mRNA decay – mechanisms of substrate mRNA recognition and degradation in mammalian cells. Biochim. Biophys. Acta. 1829, 612–623.
Gardner L.B. (2008) Hypoxic inhibition of nonsense-mediated RNA decay regulates gene expression and the integrated stress response. Mol. Cell. Biol. 28, 3729–3741.
Li Z., Vuong J.K., Zhang M., Stork C., Zheng S. (2017) Inhibition of nonsense-mediated RNA decay by ER stress. RNA. 23, 378–394.
Martin L., Gardner L.B. (2015) Stress-induced inhibition of nonsense-mediated RNA decay regulates intracellular cystine transport and intracellular glutathione through regulation of the cystine/glutamate exchanger SLC7A11. Oncogene. 34, 4211–4218.
Yepiskoposyan H., Aeschimann F., Nilsson D., Oko-niewski M., Muhlemann O. (2011) Autoregulation of the nonsense-mediated mRNA decay pathway in human cells. RNA. 17, 2108–2118.
McGlincy N.J., Smith C.W. (2008) Alternative splicing resulting in nonsense-mediated mRNA decay: what is the meaning of nonsense? Trends Biochem. Sci. 33, 385–393.
Rossbach O., Hung L.H., Schreiner S., Grishina I., Heiner M., Hui J., Bindereif A. (2009) Auto- and cross-regulation of the hnRNP L proteins by alternative splicing. Mol. Cell Biol. 29, 1442–1451.
Wollerton M.C., Gooding C., Wagner E.J., Garcia-Blanco M.A., Smith C.W. (2004) Autoregulation of polypyrimidine tract binding protein by alternative splicing leading to nonsense-mediated decay. Mol. Cell. 13, 91–100.
Izumikawa K., Yoshikawa H., Ishikawa H., Nobe Y., Yamauchi Y., Philipsen S., Simpson R.J., Isobe T., Takahashi N. (2016) Chtop (Chromatin target of Prmt1) auto-regulates its expression level via intron retention and nonsense-mediated decay of its own mRNA. Nucl. Acids Res. 44, 9847–9859.
Yan Q., Weyn-Vanhentenryck S.M., Wu J., Sloan S.A., Zhang Y., Chen K., Wu J.Q., Barres B.A., Zhang C. (2015) Systematic discovery of regulated and conserved alternative exons in the mammalian brain reveals NMD modulating chromatin regulators. Proc. Natl. Acad. Sci. USA. 112, 3445–3450.
Guan Q., Zheng W., Tang S., Liu X., Zinkel R.A., Tsui K.W., Yandell B.S., Culbertson M.R. (2006) Impact of nonsense-mediated mRNA decay on the global expression profile of budding yeast. PLoS Genet. 2, e203.
Шварц А.М., Комарова Т.В., Скулачев М.В., Зверева А.С., Дорохов Ю.Л., Атабеков И.Г. (2007) Стабильность мРНК в растениях зависит от длины 3'-нетранслируемой области. Биохимия. 72, 260–269.
Schell T., Kocher T., Wilm M., Seraphin B., Kulozik A.E., Hentze M.W. (2003) Complexes between the nonsense-mediated mRNA decay pathway factor human upf1 (up-frameshift protein 1) and essential nonsense-mediated mRNA decay factors in HeLa cells. Biochem. J. 373, 775–783.
He F., Jacobson A. (2015) Nonsense-mediated mRNA decay: degradation of defective transcripts is only part of the story. Annu. Rev. Genet. 49, 339–366.
Grimson A., O’Connor S., Newman C.L., Anderson P. (2004) SMG-1 is a phosphatidylinositol kinase-related protein kinase required for nonsense-mediated mRNA decay in Caenorhabditis elegans. Mol. Cell. Biol. 24, 7483–7490.
Huntzinger E., Kashima I., Fauser M., Sauliere J., Izaurralde E. (2008) SMG6 is the catalytic endonuclease that cleaves mRNAs containing nonsense codons in metazoan. RNA. 14, 2609–1267.
Lykke-Andersen S., Chen Y., Ardal B.R., Lilje B., Waage J., Sandelin A., Jensen T.H. (2014) Human nonsense-mediated RNA decay initiates widely by endonucleolysis and targets snoRNA host genes. Genes Dev. 28, 2498–2517.
Loh B., Jonas S., Izaurralde E. (2013) The SMG5-SMG7 heterodimer directly recruits the CCR4-NOT deadenylase complex to mRNAs containing nonsense codons via interaction with POP2. Genes Dev. 27, 2125–2138.
Nicholson P., Gkratsou A., Josi C., Colombo M., Muhlemann O. (2018) Dissecting the functions of SMG5, SMG7, and PNRC2 in nonsense-mediated mRNA decay of human cells. RNA. 24, 557–573.
Kurosaki T., Miyoshi K., Myers J.R., Maquat L.E. (2018) NMD-degradome sequencing reveals ribosome-bound intermediates with 3'-end non-templated nucleotides. Nat. Struct. Mol. Biol. 25, 940–950.
Brogna S., McLeod T., Petric M. (2016) The meaning of NMD: translate or perish. Trends Genet.: TIG. 32, 395–407.
Chamieh H., Ballut L., Bonneau F., Le Hir H. (2008) NMD factors UPF2 and UPF3 bridge UPF1 to the exon junction complex and stimulate its RNA helicase activity. Nat. Struct. Mol. Biol. 15, 85–93.
Amrani N., Ganesan R., Kervestin S., Mangus D.A., Ghosh S., Jacobson A. (2004) A faux 3'-UTR promotes aberrant termination and triggers nonsense-mediated mRNA decay. Nature. 432, 112–118.
Kim Y.K., Furic L., Desgroseillers L., Maquat L.E. (2005) Mammalian Staufen1 recruits Upf1 to specific mRNA 3'UTRs so as to elicit mRNA decay. Cell. 120, 195–208.
Kim Y.K., Furic L., Parisien M., Major F., DesGroseillers L., Maquat L.E. (2007) Staufen1 regulates diverse classes of mammalian transcripts. EMBO J. 26, 2670–2681.
Gong C., Maquat L.E. (2011) lncRNAs transactivate STAU1-mediated mRNA decay by duplexing with 3' UTRs via Alu elements. Nature. 470, 284–288.
Lucas B.A., Lavi E., Shiue L., Cho H., Katzman S., Miyoshi K., Siomi M.C., Carmel L., Ares M., Jr., Maquat L.E. (2018) Evidence for convergent evolution of SINE-directed Staufen-mediated mRNA decay. Proc. Natl. Acad. Sci. USA. 115, 968–973.
Wang J., Gong C., Maquat L.E. (2013) Control of myogenesis by rodent SINE-containing lncRNAs. Genes Dev. 27, 793–804.
Park E., Gleghorn M.L., Maquat L.E. (2013) Staufen2 functions in Staufen1-mediated mRNA decay by binding to itself and its paralog and promoting UPF1 helicase but not ATPase activity. Proc. Natl. Acad. Sci. USA. 110, 405–412.
Kim Y.K., Maquat L.E. (2019) UPFront and center in RNA decay: UPF1 in nonsense-mediated mRNA decay and beyond. RNA. 25, 407–422.
Inada T. (2013) Quality control systems for aberrant mRNAs induced by aberrant translation elongation and termination. Biochim. Biophys. Acta. 1829, 634–642.
Klauer A.A., van Hoof A. (2012) Degradation of mRNAs that lack a stop codon: a decade of nonstop progress. Wiley Interdiscip. Kev. RNA. 3, 649–660.
Tsuboi T., Kuroha K., Kudo K., Makino S., Inoue E., Kashima I., Inada T. (2012) Dom34:hbs1 plays a general role in quality-control systems by dissociation of a stalled ribosome at the 3' end of aberrant mRNA. Mol. Cell. 46, 518–529.
Harigaya Y., Parker R. (2010) No-go decay: a quality control mechanism for RNA in translation. Wiley Interdiscip. Rev. RNA. 1, 132–141.
Passos D.O., Doma M.K., Shoemaker C.J., Muhlrad D., Green R., Weissman J., Hollien J., Parker R. (2009) Analysis of Dom34 and its function in no-go decay. Mol. Biol. Cell. 20, 3025–3032.
Saito S., Hosoda N., Hoshino S. (2013) The Hbs1–Dom34 protein complex functions in non-stop mRNA decay in mammalian cells. J. Biol. Chem. 288, 17832–17843.
Szadeczky-Kardoss I., Csorba T., Auber A., Schamberger A., Nyiko T., Taller J., Orban T.I., Burgyan J., Silhavy D. (2018) The nonstop decay and the RNA silencing systems operate cooperatively in plants. Nucl. Acids Res. 46, 4632–4648.
Szadeczky-Kardoss I., Gal L., Auber A., Taller J., Silhavy D. (2018) The no-go decay system degrades plant mRNAs that contain a long A-stretch in the coding region. Plant Sci.: Internat. J. Exp. Plant Biol. 275, 19–27.
Agrawal N., Dasaradhi P.V., Mohmmed A., Malhotra P., Bhatnagar R.K., Mukherjee S.K. (2003) RNA interference: biology, mechanism, and applications. Microbiol. Mol. Biol. Rev.: MMBR. 67, 657–685.
Fang X., Qi Y. (2016) RNAi in plants: an argonaute-centered view. Plant Cell. 28, 272–285.
Fischer S.E.J. (2015) RNA interference and microRNA-mediated silencing. Curr. Protocols Mol. Biol. 112, 2611–2615.
Вильгельм А.Э., Чумаков С.П., Прасолов В.С. (2006) Интерференция РНК: биология и перспективы применения в биомедицине и биотехнологии. Молекуляр. биология. 40, 387–403.
Fatica A., Tollervey D. (2002) Making ribosomes. Curr. Opin. Cell Biol. 14, 313–318.
Briggs M.W., Burkard K.T., Butler J.S. (1998) Rrp6p, the yeast homologue of the human PM-Scl 100-kDa autoantigen, is essential for efficient 5.8 S rRNA 3' end formation. J. Biol. Chem. 273, 13255–13263.
Wang M., Pestov D.G. (2011) 5'-end surveillance by Xrn2 acts as a shared mechanism for mammalian pre-rRNA maturation and decay. Nucl. Acids Res. 39, 1811–1822.
Dez C., Houseley J., Tollervey D. (2006) Surveillance of nuclear-restricted pre-ribosomes within a subnucleolar region of Saccharomyces cerevisiae. EMBO J. 25, 1534–1546.
LaRiviere F.J., Cole S.E., Ferullo D.J., Moore M.J. (2006) A late-acting quality control process for mature eukaryotic rRNAs. Mol. Cell. 24, 619–626.
Cole S.E., LaRiviere F.J., Merrikh C.N., Moore M.J. (2009) A convergence of rRNA and mRNA quality control pathways revealed by mechanistic analysis of nonfunctional rRNA decay. Mol. Cell. 34, 440–450.
Gudipati R.K., Xu Z., Lebreton A., Seraphin B., Steinmetz L.M., Jacquier A., Libri D. (2012) Extensive degradation of RNA precursors by the exosome in wild-type cells. Mol. Cell. 48, 409–421.
Hamill S., Wolin S.L., Reinisch K.M. (2010) Structure and function of the polymerase core of TRAMP, a RNA surveillance complex. Proc. Natl. Acad. Sci. USA. 107, 15045–15050.
Alexandrov A., Chernyakov I., Gu W., Hiley S.L., Hughes T.R., Grayhack E.J., Phizicky E.M. (2006) Rapid tRNA decay can result from lack of nonessential modifications. Mol. Cell. 21, 87–96.
Chernyakov I., Whipple J.M., Kotelawala L., Grayhack E.J., Phizicky E.M. (2008) Degradation of several hypomodified mature tRNA species in Saccharomyces cerevisiae is mediated by Met22 and the 5'-3' exonucleases Rat1 and Xrn1. Genes Dev. 22, 1369–1680.
Betat H., Morl M. (2015) The CCA-adding enzyme: a central scrutinizer in tRNA quality control. BioEssays: News Rev. Mol., Cell. Dev. Biol. 37, 975–982.
Wilusz J.E., Whipple J.M., Phizicky E.M., Sharp P.A. (2011) tRNAs marked with CCACCA are targeted for degradation. Science. 334, 817–821.
Thompson D.M., Parker R. (2009) The RNase Rny1p cleaves tRNAs and promotes cell death during oxidative stress in Saccharomyces cerevisiae. J. Cell Biol. 185, 43–50.
Allmang C., Kufel J., Chanfreau G., Mitchell P., Petfalski E., Tollervey D. (1999) Functions of the exosome in rRNA, snoRNA and snRNA synthesis. EMBO J. 18, 5399–5410.
Zhang L., Wan Y., Huang G., Wang D., Yu X., Huang G., Guo J. (2015) The exosome controls alternative splicing by mediating the gene expression and assembly of the spliceosome complex. Sci. Repts. 5, 13403.
Didychuk A.L., Butcher S.E., Brow D.A. (2018) The life of U6 small nuclear RNA, from cradle to grave. RNA. 24, 437–460.
Hrossova D., Sikorsky T., Potesil D., Bartosovic M., Pasulka J., Zdrahal Z., Stefl R., Vanacova S. (2015) RBM7 subunit of the NEXT complex binds U-rich sequences and targets 3'-end extended forms of snRNAs. Nucl. Acids Res. 43, 4236–4248.
Hilcenko C., Simpson P.J., Finch A.J., Bowler F.R., Churcher M.J., Jin L., Packman L.C., Shlien A., Campbell P., Kirwan M., Dokal I., Warren A.J. (2013) Aberrant 3' oligoadenylation of spliceosomal U6 small nuclear RNA in poikiloderma with neutropenia. Blood. 121, 1028–1038.
Shukla S., Parker R. (2014) Quality control of assembly-defective U1 snRNAs by decapping and 5'-to-3' exonucleolytic digestion. Proc. Natl. Acad. Sci. USA. 111, E3277–E3786.
Labno A., Warkocki Z., Kulinski T., Krawczyk P.S., Bijata K., Tomecki R., Dziembowski A. (2016) Perlman syndrome nuclease DIS3L2 controls cytoplasmic non-coding RNAs and provides surveillance pathway for maturing snRNAs. Nucl. Acids Res. 44, 10437–10453.
van Hoof A., Lennertz P., Parker R. (2000) Yeast exosome mutants accumulate 3'-extended polyadenylated forms of U4 small nuclear RNA and small nucleolar RNAs. Mol. Cell. Biol. 20, 441–452.
Larochelle M., Lemay J.F., Bachand F. (2012) The THO complex cooperates with the nuclear RNA surveillance machinery to control small nucleolar RNA expression. Nucl. Acids Res. 40, 10240–10253.
Lubas M., Christensen M.S., Kristiansen M.S., Domanski M., Falkenby L.G., Lykke-Andersen S., Andersen J.S., Dziembowski A., Jensen T.H. (2011) Interaction profiling identifies the human nuclear exosome targeting complex. Mol. Cell. 43, 624–637.
Buscher M., Horos R., Hentze M.W. (2020) “High vault-age”: non-coding RNA control of autophagy. Open Biol. 10, 190307.
Horos R., Buscher M., Sachse C., Hentze M.W. (2019) Vault RNA emerges as a regulator of selective autophagy. Autophagy. 15, 1463–1464.
Liu X., Zheng Q., Vrettos N., Maragkakis M., Ale-xiou P., Gregory B.D., Mourelatos Z. (2014) A microRNA precursor surveillance system in quality control of microRNA synthesis. Mol. Cell. 55, 868–879.
Chang H.M., Triboulet R., Thornton J.E., Gregory R.I. (2013) A role for the Perlman syndrome exonuclease Dis3l2 in the Lin28-let-7 pathway. Nature. 497, 244–248.
Gantier M.P., McCoy C.E., Rusinova I., Saulep D., Wang D., Xu D., Irving A.T., Behlke M.A., Hertzog P.J., Mackay F., Williams B.R. (2011) Analysis of microRNA turnover in mammalian cells following Dicer1 ablation. Nucl. Acids Res. 39, 5692–5703.
Sanei M., Chen X. (2015) Mechanisms of microRNA turnover. Curr. Opin. Plant Biol. 27, 199–206.
Wang X., Wang Y., Dou Y., Chen L., Wang J., Jiang N., Guo C., Yao Q., Wang C., Liu L., Yu B., Zheng B., Chekanova J.A., Ma J., Ren G. (2018) Degradation of unmethylated miRNA/miRNA*s by a DEDDy-type 3' to 5' exoribonuclease Atrimmer 2 in Arabidopsis. Proc. Natl. Acad. Sci. USA. 115, E6659–E6667.
Kamminga L.M., Luteijn M.J., den Broeder M.J., Redl S., Kaaij L.J., Roovers E.F., Ladurner P., Berezikov E., Ketting R.F. (2010) Hen1 is required for oocyte development and piRNA stability in zebrafish. EMBO J. 29, 3688–3700.
Szczepinska T., Kalisiak K., Tomecki R., Labno A., Borowski L.S., Kulinski T.M., Adamska D., Kosinska J., Dziembowski A. (2015) DIS3 shapes the RNA polymerase II transcriptome in humans by degrading a variety of unwanted transcripts. Genome Res. 25, 1622–1633.
Tisseur M., Kwapisz M., Morillon A. (2011) Pervasive transcription – lessons from yeast. Biochimie. 93, 1889–1896.
Thiebaut M., Kisseleva-Romanova E., Rougemaille M., Boulay J., Libri D. (2006) Transcription termination and nuclear degradation of cryptic unstable transcripts: a role for the Nrd1–Nab3 pathway in genome surveillance. Mol. Cell. 23, 853–864.
Thompson D.M., Parker R. (2007) Cytoplasmic decay of intergenic transcripts in Saccharomyces cerevisiae. Mol. Cell. Biol. 27, 92–101.
Belostotsky D. (2009) Exosome complex and pervasive transcription in eukaryotic genomes. Curr. Opin. Cell Biol. 21, 352–358.
Chekanova J.A., Gregory B.D., Reverdatto S.V., Chen H., Kumar R., Hooker T., Yazaki J., Li P., Skiba N., Peng Q., Alonso J., Brukhin V., Grossniklaus U., Ecker J.R., Belostotsky D.A. (2007) Genome-wide high-resolution mapping of exosome substrates reveals hidden features in the Arabidopsis transcriptome. Cell. 131, 1340–1353.
Davidson L., Francis L., Cordiner R.A., Eaton J.D., Estell C., Macias S., Caceres J.F., West S. (2019) Rapid depletion of DIS3, EXOSC10, or XRN2 reveals the immediate impact of exoribonucleolysis on nuclear RNA metabolism and transcriptional control. Cell Rept. 26, 2779–2791 e5.
Preker P., Nielsen J., Kammler S., Lykke-Andersen S., Christensen M.S., Mapendano C.K., Schierup M.H., Jensen T.H. (2008) RNA exosome depletion reveals transcription upstream of active human promoters. Science. 322, 1851–1854.
van Dijk E.L., Chen C.L., d’Aubenton-Carafa Y., Gourvennec S., Kwapisz M., Roche V., Bertrand C., Silvain M., Legoix-Ne P., Loeillet S., Nicolas A., Thermes C., Morillon A. (2011) XUTs are a class of Xrn1-sensitive antisense regulatory non-coding RNA in yeast. Nature. 475, 114–117.
Wery M., Descrimes M., Vogt N., Dallongeville A.S., Gautheret D., Morillon A. (2016) Nonsense-mediated decay restricts lncRNA levels in yeast unless blocked by double-stranded RNA structure. Mol. Cell. 61, 379–392.
Muhlrad D., Parker R. (1999) Aberrant mRNAs with extended 3' UTRs are substrates for rapid degradation by mRNA surveillance. RNA. 5, 1299–1307.
Ustianenko D., Pasulka J., Feketova Z., Bednarik L., Zigackova D., Fortova A., Zavolan M., Vanacova S. (2016) TUT-DIS3L2 is a mammalian surveillance pathway for aberrant structured non-coding RNAs. EMBO J. 35, 2179–2191.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


