Молекулярная биология, 2020, T. 54, № 5, стр. 858-871
Сверхустойчивость штамма Bacillus licheniformis 24 к окислительному стрессу ассоциирована со сверхэкспрессией генов ферментативной антиоксидантной системы
Д. С. Карпов a, *, П. Д. Осипова b, А. И. Домашин b, Н. Б. Поляков c, d, А. И. Соловьев c, М. В. Зубашева c, В. Г. Жуховицкий c, e, В. Л. Карпов a, С. В. Поддубко b, Н. Д. Новикова b
a Центр высокоточного редактирования и генетических технологий для биомедицины, Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Институт медико-биологических проблем Российской академии наук
123007 Москва, Россия
c Национальный исследовательский центр эпидемиологии и микробиологии им. почетного акад. Н.Ф. Гамалеи Министерства здравоохранения России
123098 Москва, Россия
d Институт геохимии и аналитической химии им. В.И. Вернадского Российской академии наук
119334 Москва, Россия
e Первый Московский государственный медицинский университет им. И.М. Сеченова
Министерства здравоохранения России
119991 Москва, Россия
* E-mail: aleom@yandex.ru
Поступила в редакцию 20.04.2020
После доработки 15.05.2020
Принята к публикации 15.05.2020
Аннотация
На Международной космической станции (МКС) создаются и поддерживаются искусственные условия жизнедеятельности, адекватные потребностям человека, которые одновременно считаются благоприятными для развития многочисленных микроорганизмов: плесневых грибов и бактерий. Среди обнаруженных на МКС микроорганизмов, наряду с представителями аутомикрофлоры членов экипажей, выявлено значительное число видов спорообразующих бактерий. В большинстве случаев именно из этой группы микроорганизмов формируются резиденты, способные к заселению, росту и размножению на материалах интерьера и оборудования станций, а также к участию в процессах биоповреждения последних. И именно эти бактерии проявляют повышенную устойчивость к действию различных стрессовых факторов, например, ДНК-повреждающих и окислительных агентов. Молекулярные механизмы устойчивости таких бактериальных штаммов к стрессовым воздействиям мало изучены. В рамках санитарно-микробиологического мониторинга среды обитания МКС обнаружен и выделен штамм Bacillus licheniformis 24. В результате проведенного исследования нами показано, что этот штамм обладает повышенной устойчивостью к перекиси водорода и параквату по сравнению с “земным” штаммом B. licheniformis В-10956. У штамма B. licheniformis 24 наблюдается сверхэкспрессия генов ферментов, нейтрализующих активные формы кислорода: каталазы (katX) и супероксиддисмутаз (sodA и sodF). Обнаружено, что B. licheniformis 24 продуцирует меньше сероводорода, чем B. licheniformis В-10956, и выявлена ассоциация этого явления с резко сниженной экспрессией оперона cysIJ, кодирующего сульфитредуктазу. На основании полученных результатов можно предполагать, что ферментативные системы антиоксидантной защиты вносят более значимый вклад в сверхустойчивость штаммов B. licheniformis к окислительным агентам, чем компоненты неферментативных систем (например, сероводород).
ВВЕДЕНИЕ
Орбитальные космические станции создают своеобразные условия для жизнедеятельности микроорганизмов. Так, на орбите Международной космической станции (МКС) повышен уровень ионизирующего излучения, что ведет к более интенсивному повреждению наследственного материала микробных клеток [1]. Кроме того, наблюдается недостаток питательных веществ [2], а внутренние поверхности интерьера и оборудования станции периодически подвергаются обработке антимикробными и спороцидными реагентами [3]. Несмотря на действие этих факторов, губительных для большинства видов прокариот, микробы все же заселяют внутренние объемы космической станции [4, 5], хотя их видовое разнообразие значительно снижено [6]. Видовой состав микробиома станции изучают в рамках мероприятий по оценке санитарно-микробиологического состояния среды обитания членов экипажа МКС [5‒8]. Вполне ожидаемо, что обнаруживаемые внутри МКС бактериальные штаммы обладают повышенной по сравнению с земными штаммами устойчивостью к действию различных стрессовых факторов. Так, ранее нами описан штамм Bacillus subtilis 20, сверхустойчивый к действию ДНК-повреждающих факторов и некоторых окислительных агентов [9]. Кроме МКС представителей рода Bacillus, сверхустойчивых к ультрафиолетовому излучению и окислительному стрессу, находят также в регулярно стерилизуемых помещениях, где осуществляется сборка космических аппаратов [2]. Особой устойчивостью к окислительному стрессу отличается штамм B. pumilus SAFR-032 [3]. В настоящее время молекулярные механизмы сверхустойчивости к окислительному стрессу у бактериальных штаммов, выделенных как на МКС, так и из стерильных помещений, остаются во многом неясными.
В ходе 24 экспедиции на МКС в ее российском сегменте выделен штамм B. licheniformis 24 [10, 11]. Нами исследована устойчивость этого штамма к различным окислительным агентам, проанализирована экспрессия генов, ассоциированных с клеточным ответом на окислительный стресс, и уровень продуцируемого сероводорода.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы. Штамм Bacillus licheniformis В-10956 получен из Всероссийской коллекции промышленных микроорганизмов. Штамм B. licheniformis 24 выделен из внутренней среды МКС в ходе 24 экспедиции. Клетки растили на жидкой или агаризованной среде LB (1% бактотриптон, 0.5% дрожжевой экстракт, 1.5% агар, 1% NaCl, рН 7.0‒7.1) или агаризованной среде J (0.5% бактотриптон, 1.5% дрожжевой экстракт, 1.5% агар, рН 7.3‒7.5) при 37°С. В жидкой среде LB бактерии культивировали при встряхивании (200 об/мин).
Масс-спектрометрия с матричноактивированной лазерной десорбцией и ионизацией (масс-спектрометрия MALDI-TOF). Экстракцию белков и пептидов проводили, как описано Sauer и соавт. [12], с незначительными модификациями. Отдельные колонии микроорганизмов собирали с поверхности агара пластиковой микробиологической петлей объемом 1 мкл и ресуспендировали в 300 мкл деионизированной воды. К суспензии добавляли 900 мкл 96%-ного этанола, полученную смесь тщательно перемешивали и центрифугировали при 18 894 × g в течение 2 мин. Супернатант количественно отбирали, и осадок подсушивали на воздухе. К высушенному осадку добавляли от 5 до 40 мкл 70%-ной муравьиной кислоты (в зависимости от объема осадка) и равный объем ацетонитрила. Полученную смесь центрифугировали при 18 894 × g в течение 2 мин. Супернатант, содержащий белковый экстракт, использовали в масс-спектрометрическом анализе.
На 386-луночную стальную мишень (“Bruker Daltonics”, Германия) наносили 1 мкл супернатанта и высушивали при комнатной температуре. На поверхность высушенного экстракта наносили 1 мкл раствора матрицы: насыщенный раствор α-циано-4-гидроксикоричной кислоты (“Sigma Aldrich”, США), содержащий 50% ацетонитрила и 2.5% трифторуксусной кислоты (“Panreac”, США), ‒ который также высушивали при комнатной температуре.
Масс-спектрометрический анализ проводили на приборе UltrafleXtreme (“Bruker daltonics”), оснащенном Nd:Yag-лазером (355 нм) в линейном режиме. Детектировали положительно заряженные ионы в диапазоне от 2000 до 20 000 Th при следующих настройках ионного источника: напряжение на IS1 20 кВ, на IS2 19 кВ, на линзах (параметр “Lens”) 4.5 кВ, напряжение питания детектора 2885 В, коэффициент усиления 12.6.
Спектры снимали в автоматическом режиме с использованием программы Flex Control (v.3.4, build 135). Точки обстрела лазером на мишени выбирали случайным образом. Суммировали 1200 спектров с 200 точек обстрела при частоте лазера 2 кГц.
Библиотеки спектров для каждого образца получали при анализе трипликатов 8 технических повторов. Калибровочным стандартом и положительным контролем служил белковый экстракт Escherichia coli DH5α с дополнительными белками (РНКаза A [M + H]+ 13683.2 Да, миоглобин [M + H]+ 16952.3 Да) (кат. номер 255343, “Bruker Daltonics”).
Полученные спектры обрабатывали с помощью программного комплекса MALDI biotyper Compass Explorer 4.1 (“Bruker Daltonics”) с использованием стандартного метода препроцессинга данных (Biotyper Preprocessing Standard Method). Обработанные спектры образцов сравнивали с референсной базой характеристических спектральных профилей, включающей 8817 записей. Результаты поиска характеристического профиля выражали как логарифм значений. Значения ниже 1.699 соответствовали ненадежному определению рода, значения 1.700–1.999 ‒ надежному определению рода и, возможно, вида, значения 2.000–2.299 ‒ надежному определению рода и с высокой вероятностью вида и, наконец, значения 2.300–3.000 ‒ надежной идентификации вида.
Дендрограммы строили с помощью программы MALDI biotyper Compass Explorer 4.1 (“Bruker Daltonics”), используя стандартный метод (Biotyper MSP Dendrogam Creation Standart Method), и программы IDBac (версия 1.1.10) [13]. Результат работы программы IDBac экспортировали в формате newick [14] и визуализировали c помощью функции Tree Explorer программы MEGA (версия 10.1.7) [15]. Конвертирование масс-спектров в формат mzML проводили c помощью программы MSConvert (версия 3.0.19217-f7f3a630b) [16]. Спектральные данные в виде гелей представляли с использованием программного обеспечения mmass 5.5.0 [17].
Выделение геномной ДНК. Геномную ДНК из клеток, выращенных до стационарной фазы, выделяли по ранее описанной методике [9]. Клетки из культуры осаждали центрифугированием, промывали в буфере Tрис-HCl (pH 8.0) и ресуспендировали в 300 мкл лизирующего буфера (2% Tритон X-100, 1% SDS, 100 мM NaCI, 10 мM Tрис-HCI, pH 8.0). Добавляли равный объем смеси фенол : хлороформ : изоамиловый спирт в соотношении 25 : 24 : 1 (pH 8.0) и около 100 мкл стеклянных шариков (“Sigma-Aldrich”). Клетки разрушали, встряхиванием на приборе Precellys 2400 (“Bertin Technologies”, Франция) при 6800 об/мин. Фазы разделяли центрифугированием и нуклеиновые кислоты осаждали из водной фазы этиловым спиртом в присутствии ацетата натрия. Полученную суспензию центрифугировали при 20 000 × g, осадок ресуспендировали в буфере TE (10 мM Tрис-HCl, pH 8.0, 1 мM EDTA) и обрабатывали РНКазой А (“Thermo Fisher Scientific”, США) в соответствии с рекомендациями производителя. Очищенную ДНК осаждали центрифугированием при 20 000 × g и ресуспендировали в 10 мM буфере Tрис-HCl (pH 8.0).
Секвенирование рибосомной ДНК (рДНК). ДНК выделяли из клеток, выращенных до стационарной фазы, как описано выше, и использовали в качестве матрицы для амплификации внутреннего транскрибируемого спейсера 16S‒23S рибосомных генов. В реакции использовали праймеры L516SF и L523SR (табл. 1) и условия, описанные в работе Xu & Cote [18]. ПЦР-продукт очищали с использованием набора PCR Clean Up (“Thermo Fisher Scientific”). ПЦР-фрагменты секвенировали с праймером L516SF и набором реагентов ABI PRISM® BigDye™ Terminator v. 3.1 с последующим анализом продуктов реакции на автоматическом анализаторе ДНК Applied Biosystems 3730 (“Applied Biosystems”, США).
Таблица 1.
Олигонуклеотиды, использованные в работе
| Название олигонуклеотида | Нуклеотидная последовательность 5' → 3' | Назначение |
|---|---|---|
| L516SF | TCGCTAGTAATCGCGGATCAGC | Идентификация штаммов Bacillusа |
| L523SR | GCATATCGGTGTTAGTCCCGTCC | |
| BlrpsJ-ex-RT-F | CCGGAGCCAGCGTTTCTGGTC | Референсный ген rpsJ в ОТ-ПЦР |
| BlrpsJ-ex-RT-R | GTGGTGTTGGGTTCACAATGTCG | |
| BlperR-ex-RT-F | TAACCTGAAAGAAGCGTTGG | Определение уровня мРНК гена perR |
| BlperR-ex-RT-R | CTGCAGTTGGATGAGACATAG | |
| BlmrgA-ex-RT-F | AAGCGATTACCGTCAAATCC | Определение уровня мРНК гена mrgA |
| BlmrgA-ex-RT-R | CTCTGTCAGGGCGATATAAAG | |
| Blfur-ex-RT-F | CGACAGTATATCGGACTCTTG | Определение уровня мРНК гена fur |
| Blfur-ex-RT-R | TTCTTTGCGGAGGTCATATC | |
| BlkatX_ex-RT-F | GAACCGAAGCAGGGCATAAA | Определение уровня мРНК гена katX |
| BlkatX_ex-RT-R | GGATAATCTCCGCGCTCAATC | |
| BlsodA_ex-RT-F | GCAAACCTTGATGCAGTACC | Определение уровня мРНК гена sodA |
| BlsodA_ex-RT-R | CTCCTCCGTTTGGAGAAAGA | |
| BlsodF_ex-RT-F | CCTGCCCTACTCGTATTCAG | Определение уровня мРНК гена sodF |
| BlsodF_ex-RT-R | GTTCAAACCGTCGACATAGC | |
| BlcysI_ex-RT-F | ATGTACGGACCGCTGTATC | Определение уровня мРНК гена cysI |
| BlcysI_ex-RT-R | GCTTGTCATCCTCCACAATC | |
| BlcysJ_ex-RT-F | GCTTGAAGGCTCAGGTTTG | Определение уровня мРНК гена cysJ |
| BlcysJ_ex-RT-R | GCATTCGGATCCCATTTCAG | |
| BlcysK-ex-RT-F | AATCCCAGGCAGCTTTATTC | Определение уровня мРНК гена cysK |
| BlcysK-ex-RT-R | AGAGGTTTGCCGATTTCTTC | |
| BlMetI-ex-RT-F | GAAGAGCTGTCTGAGGAAATG | Определение уровня мРНК гена metI |
| BlMetI-ex-RT-R | CGTCTTCAACCCTCTCATTAAG |
а Использованы последовательности олигонуклеотидов из работы [18].
Определение кинетики роста бактерий в среде LB. Кинетику роста бактериальных культур определяли с использованием прибора Bioscreen C (“Oy Growth Curves Ab Ltd”, Финляндия). Начальная оптическая плотность культур бактерий составляла 0.01 при длине волны 600 нм (OD600). В приборе Bioscreen C клетки культивировали в 96-луночном планшете в 150 мкл среды LB при температуре 37°С в течение 16 ч и встряхивании при 200 об/мин. Оптическую плотность культуры в каждой лунке измеряли каждый час.
Определение продукции сероводорода. Штаммы культивировали в жидкой среде LB в течение 16 ч в пробирках, под крышками которых прикрепляли полоску фильтровальной бумаги, смоченной 2%-ным раствором ацетата свинца. Уровень продукции H2S штаммами оценивали по интенсивности почернения фильтровальной бумаги в результате образования соединения PbS.
Выделение суммарной РНК. РНК из клеток, выращенных до средней и поздней логарифмической фазы, выделяли, как описано ранее [9]. Клетки осаждали центрифугированием и ресуспендировали в буфере, содержащем 10 мМ Tрис-HCl (pH 8.0), 100 мМ NaCl и 1 мМ EDTA. Клетки лизировали в буфере, содержащем 50 мМ Tрис-HCl (pH 8.0), 8% сахарозы, 0.5% Тритона X-100, 10 мМ EDTA, 4 мг/мл лизоцима. Лизат смешивали с равным объемом фенола (pH 4.5) и инкубировали при 65ºС в течение 15 мин. Фазы разделяли центрифугированием при 20 000 × g. Водную фазу смешивали с равным объемом смеси фенол : хлороформ : изоамиловый спирт (25 : 24 : 1), pH 4.5. Фазы разделяли центрифугированием и высаживали РНК из водной фазы, добавляя ацетат натрия и этиловый спирт. РНК из смеси осаждали центрифугированием, промывали 70%-ным этанолом, подсушивали и ресуспендировали в деионизированной воде. Примеси геномной ДНК из препарата РНК удаляли обработкой ДНКазой I (“Thermo Fisher Scientific”) по протоколу, рекомендованному производителем. РНК осаждали центрифугированием и ресуспендировали в деионизованной воде качества mQ, обработанной диэтилпирокарбонатом.
Определение уровня экспрессии генов. Уровни мРНК анализируемых генов определяли, как описано ранее [9]. кДНК синтезировали с использованием обратной транскриптазы RevertAid H-minus (“Thermo Fisher Scientific”) и рассеянной затравки по протоколу, рекомендованному производителем. Относительное количество мРНК исследуемых генов определяли методом ПЦР в реальном времени с флуоресцентным красителем Eva Green (“Синтол”, Россия) на приборе LightCycler-480-II (“Roshe Life Science”, США) в соответствии с рекомендациями производителя. Референсным геном служил rpsJ [19]. Чтобы исключить влияние случайных мутаций и возможную вариацию в копийности исследуемых генов, относительный сигнал, полученный при анализе кДНК, нормировали на относительный сигнал, полученный при амплификации соответствующих фрагментов с геномной ДНК. Первичную обработку данных проводили с помощью программного обеспечения, поставляемого с прибором. Дальнейшую обработку проводили с помощью Microsoft Excel. Олигонуклеотиды, последовательности которых представлены в табл. 1, подбирали с помощью сервера mFold [20].
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Повторная идентификация B. licheniformis 24
Впервые штамм B. licheniformis 24 идентифицирован в 2007 году Новиковой и соавт. [11] с помощью системы автоматизированной идентификации микробных штаммов Vitek-60 (“Bio Merieux”, Франция). Теперь нами проведена повторная идентификация штамма с использованием секвенирования участка внутреннего транскрибируемого спейсера рДНК и MALDI-TOF-анализа. Согласно результатам выравнивания фрагмент рДНК штамма B. licheniformis 24 на 93.4% идентичен соответствующему участку штамма B. licheniformis B-10956 (рис. 1). Эту же последовательность участка рДНК штамма B. licheniformis 24 использовали в поиске сходных последовательностей по всей базе невырожденных нуклеотидных последовательностей NCBI с помощью BLAST. По результатам поиска наиболее близким B. licheniformis 24 оказался вид B. paralicheniformis (табл. 2). Известно, что некоторые виды рода Bacillus (например, B. cereus, B. anthracis и B. thuringiensis) практически невозможно отличить только на основании последовательности рДНК [22]. Кроме того, следует учитывать, что штамм выделен из среды МКС с повышенным радиационным фоном, поэтому можно ожидать наличия множества мутаций в его геноме, включая и рибосомные гены.
Рис. 1.
Выравнивание участков рДНК штаммов B. licheniformis. Выравнивание нуклеотидных последовательностей выполнено при помощи программы EMBOSS-Stretcher (http://www.ebi.ac.uk/Tools/psa/emboss_stretcher/) [21] с параметрами по умолчанию. Точками указаны нуклеотиды, по которым рДНК B. licheniformis 24 отличается от B. licheniformis B-10956.

Таблица 2.
Результат поиска сходных последовательностей в базе данных NCBI с помощью BLAST
| Штамм | Значение Еа | Степень идентичности, % | Идентификатор NCBI |
|---|---|---|---|
| B. paralicheniformis A4-3 | 4 × 10‒177 | 95.05 | CP043501.1 |
| B. paralicheniformis 14DA11 | 4 × 10‒177 | 95.05 | CP023168.1 |
| B. paralicheniformis Bac48 | 4 × 10‒177 | 95.05 | CP023666.1 |
| B. paralicheniformis FA6 | 2 × 10‒175 | 94.80 | CP033198.1 |
| B. paralicheniformis MDJK30 | 2 × 10‒175 | 94.80 | CP020352.1 |
| B. paralicheniformis CBMAI 1303 | 2 × 10‒175 | 94.80 | CP033389.1 |
| B. paralicheniformis ATCC 9945a | 2 × 10‒175 | 94.80 | CP005965.1 |
| B. licheniformis SRCM100141 | 8 × 10‒174 | 94.57 | CP021669.1 |
| B. licheniformis MCC 2514 | 8 × 10‒174 | 94.57 | CP038186.1 |
| B. licheniformis NCTC10341 | 8 × 10‒174 | 94.57 | LR134392.1 |
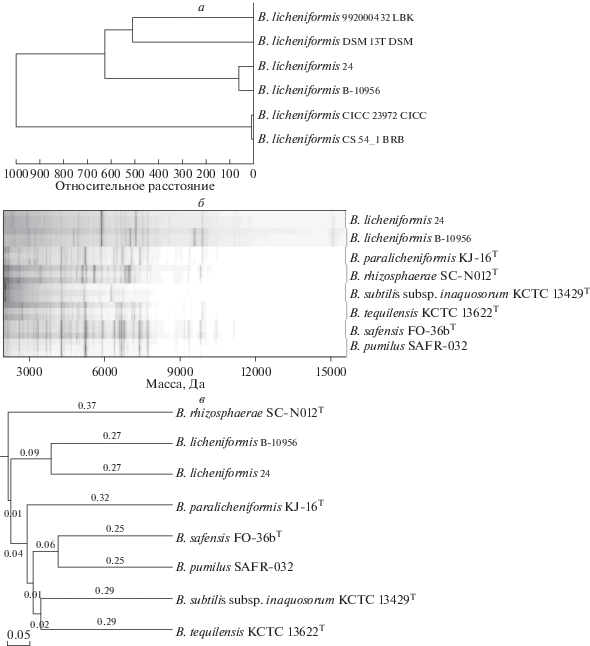
В качестве дополнительного инструмента идентификации исследуемого штамма использовали масс-спектрометрический анализ. Согласно данным MALDI-TOF, видовая принадлежность исследуемого штамма не может быть надежно определена с использованием немодифицированной референсной базы данных (табл. 3). На основании сравнительного анализа спектральных характеристик всех имеющихся в базе данных MALDI Biotyper 4.1 штаммов B. licheniformis, а также штамма сравнения B. licheniformis B-10956, отсутствующего в базе, анализируемый штамм гораздо ближе к B. licheniformis B-10956 (рис. 2а). На основании этих результатов спектры штамма B. licheniformis B-10956 внесли в базу MALDI Biotyper 4.1 и провели повторный масс-спектрометрический анализ. Согласно данным, полученным с использованием модифицированной базы, штамм B. licheniformis 24, действительно, с большой степенью надежности близок к штамму B. licheniformis B-10956 (табл. 3). Однако база данных не содержит спектров B. paralicheniformis и некоторых других видов бацилл, поэтому для более детального анализа использовали спектральные данные в формате mzML, полученные для типового штамма B. paralicheniformis KJ-16 и других, включая штаммы B. pumilus SAFR-032 и B. safensis FO-36bT [23]. При визуальном анализе спектрограмм B. licheniformis 24 обнаружено бόльшее сходство с B. licheniformis В-10956 (рис. 2б), чем с другими штаммами. Это подтверждено и при анализе сходства штаммов с построением дендрограммы (рис. 2в). Следует также отметить, что анализируемый штамм не похож на сверхустойчивые к окислительному стрессу штаммы B. pumilus SAFR-032 и B. safensis FO-36bT. Таким образом, можно заключить, что анализируемый штамм B. licheniformis 24 ближе к виду B. licheniformis, чем к B. paralicheniformis и другим известным видам бацилл, ассоциированным с космическими аппаратами.
Таблица 3.
Результат идентификации штаммов B. licheniformis 24 методом MALDI-TOF
| Состояние референсной базы данных | Анализируемый штамм | Наиболее близкий штамм B. licheniformis |
Надежность идентификацииа |
|---|---|---|---|
| До внесения спектров B. licheniformis B-10956 в базу данных |
B. licheniformis 24 | B. licheniformis DSM 13T DSM | 1.42 ± 0.08 |
| B. licheniformis B-10956 |
B. licheniformis DSM 13T DSM | 1.89 ± 0.13 | |
| После внесения спектров B. licheniformis B-10956 в базу данных |
B. licheniformis 24 | B. licheniformis DSM 13T DSM | 1.96 ± 0.08 |
| B. licheniformis B-10956 | B. licheniformis B-10956 | 2.70 ± 0.05 |
Рис. 2.
Результаты анализа сходства B. licheniformis 24 с некоторыми представителями рода Bacillus с использованием данных масс-спектрометрии MALDI-TOF. а – Дендрограмма сходства штаммов B. licheniformis на основе анализа MALDI-TOF спектров. Кластеризация выполнена на основе референсных спектров (MSP) базы данных MALDI Biotyper 4.1 и спектров исследуемых штаммов. б – Представление результатов масс-спектрометрии штаммов рода Bacillus в виде гелевой спектрограммы. Для каждого штамма представлены данные по трем техническим повторам. в – Дендрограмма сходства штамма B. licheniformis с другими видами рода Bacillus, построенная с использованием данных, полученных нами и в работе Strejcek и др. [23], c помощью программы IDBac и MEGA. Числами указаны относительные расстояния, отражающие степень различия спектрограмм штаммов.
Гиперустойчивость B. licheniformis 24 к перекиси водорода
При анализе устойчивости к действию перекиси водорода обнаружено, что штамм B. licheniformis 24 практически не формирует зону ингибирования роста вокруг источника стрессового агента в отличие от штамма B. licheniformis В-10956 (рис. 3а). Логично предположить, что сверхустойчивость к перекиси водорода связана с гиперактивацией защитных антиоксидантных систем клетки. Судить о гиперактивации систем можно по повышению уровня экспрессии генов, кодирующих компоненты этих систем. Ген perR кодирует сенсор перекиси водорода и транскрипционного регулятора пероксидного регулона [24]. В штамме B. licheniformis 24 обнаружено увеличение экспрессии гена perR в среднем в 7 раз по сравнению со штаммом B. licheniformis В-10956 (рис. 3б). Кроме того, в штамме B. licheniformis 24 обнаружено более чем четырехкратное увеличение экспрессии гена спороспецифичной каталазы katX (рис. 3б), которая необходима для устойчивости прорастающих спор к перекиси водорода [25]. По данным световой микроскопии, в этом эксперименте бόльшая часть культуры не находилась на стадии споруляции к 16 ч культивирования (данные не показаны). Споруляция начинается не ранее 24 ч культивирования. Можно предположить, что наблюдаемая сверхэкспрессия гена katX связана с нарушением регуляции генов споруляции.
Рис. 3.
Сверхустойчивость штамма B. licheniformis 24 к перекиси водорода ассоциирована с повышенной экспрессией генов perR и katX. а ‒ Тест на устойчивость к действию перекиси водорода. На диск наносили 5 мкл 35%-ного водного раствора пероксида водорода. Представлена характерная совмещенная фотография зон ингибирования роста штаммов B. licheniformis В-10956 (В-10956) и B. licheniformis 24 (24). б ‒ Относительный уровень мРНК генов, вовлеченных в ответ на окислительный стресс. За единицу принят уровень экспрессии генов в штамме B. licheniformis В-10956. Разброс данных представлен стандартным отклонением для средних значений в трех независимых экспериментах. ***Статистически значимое отличие при p < 0.001. Статистическая значимость рассчитана с помощью двустороннего критерия Стьюдента для сравнения двух независимых средних.
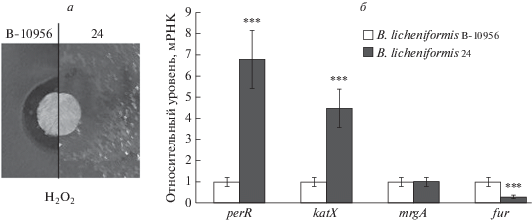
Один из внутриклеточных источников перекиси водорода ‒ реакция окисления двухвалентного железа (реакция Фентона) [26]. Показано, что в условиях действия экзогенных источников окислительного стресса клетки при участии PerR и других факторов транскрипции, например Fur, активируют внутриклеточные системы, понижающие содержание железа ‒ Fe(II) [27]. Определены уровни мРНК генов, имеющих отношение к снижению внутриклеточной концентрации свободных ионов Fe2+ (рис. 3б). Ген fur кодирует репрессор транскрипции генов транспорта железа [28]. Ген mrgA кодирует ферритинподобный белок из семейства Dps мультифункциональных белков, связывающих ионы железа [29]. Согласно результатам ПЦР в реальном времени, уровень мРНК fur более чем в три раза ниже в B. licheniformis 24 по сравнению с B. licheniformis В-10956 (рис. 3б). Несмотря на индукцию perR, уровень мРНК его мишени ‒ mrgA ‒ не отличается между двумя исследуемыми штаммами (рис. 3б).
Повышенная устойчивость штамма B. licheniformis 24 к параквату ассоциирована со сверхэкспрессией генов супероксиддисмутаз
Еще одно соединение, широко используемое в моделях окислительного стресса, ‒ паракват. Это органическое соединение индуцирует образование активных форм кислорода, преимущественно супероксидрадикалов [30, 31]. Устойчивость B. licheniformis 24 к параквату оценивали по кинетике роста в стандартной питательной среде LB и в питательной среде с 50 мкМ параквата. В ходе анализа кривых роста штаммов B. licheniformis в стандартных условиях выяснилось, что оптическая плотность культуры B. licheniformis 24 после типичного экспоненциального роста через несколько часов резко падает в два раза и держится на этом уровне в течение 10 ч ‒ вплоть до окончания 20-часового эксперимента (рис. 4а). Снижение оптической плотности культуры B. licheniformis 24, по-видимому, связано с лизисом клеток, наблюдаемым при осмотре культуры в микроскоп. Эти данные согласуются с тем, что колонии B. licheniformis 24, формирующиеся на твердой питательной среде, содержат полупрозрачные и водянистые “капли” (рис. 4б). Нами обнаружено, что при выращивании B. licheniformis 24 на среде J, близкой по составу LB, но не содержащей 1% NaCl, происходит формирование непрозрачных колоний неоднородной консистенции с относительно небольшими слизистыми включениями (рис. 4в). Возможно, лизис колоний B. licheniformis 24 на среде LB связан с чувствительностью штамма к высоким концентрациям солей. Ранее Shu и др. сообщали, что колонии со слизистыми включениями на среде LB формируют штаммы B. vallismortis [32]. Это может означать сходство видов Bacillus по фенотипическим проявлениям чувствительности к высокой солености среды.
Рис. 4.
Повышенная устойчивость B. licheniformis 24 к параквату ассоциирована со сверхэкспрессией генов супероксиддисмутаз. а ‒ Кинетика роста бактериальных культур в стандартной среде LB в отсутствие и в присутствии 50 мкМ параквата (прибор Bioscreen C). б ‒ Внешний вид колоний B. licheniformis 24 на агаризованной среде LB. в ‒ Внешний вид колоний B. licheniformis 24 на агаризованной среде J. г ‒ Относительный уровень мРНК супероксиддисмутаз. За единицу принят уровень экспрессии соответствующих генов в штамме B. licheniformis В-10956. Разброс данных представлен стандартным отклонением для средних значений в трех независимых экспериментах. ** Статистически значимое отличие при p < 0.01. Статистическая значимость рассчитана с помощью двустороннего критерия Стьюдента для сравнения двух независимых средних.
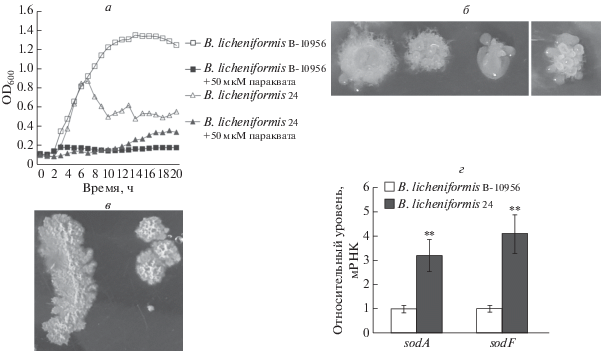
В присутствии 50 мкМ параквата роста культуры штамма B. licheniformis В-10956 (рис. 4а) не зарегистрировано в течение 20-часового эксперимента. В тоже время оптическая плотность культуры B. licheniformis 24 после 11 ч инкубации медленно увеличивалась и почти вдвое превосходила таковую для B. licheniformis В-10956 к 19 ч инкубации. На основании этих данных можно сделать вывод о повышенной адаптационной способности B. lichenifor-mis 24 к ингибирующему действию параквата по сравнению с B. licheniformis В-10956.
Известно, что фермент супероксиддисмутаза катализирует восстановление супероксидрадикалов до перекиси водорода и тем самым вносит вклад в устойчивость к параквату [33]. Представлялось логичным определить активность генов супероксиддисмутаз. По результатам определения уровня мРНК методом ПЦР в реальном времени в штамме B. licheniformis 24, по сравнению с В-10956, экспрессия генов супероксиддисмутаз sodA и sodF повышена почти в 3.5 и 4.5 раза соответственно (рис. 4г).
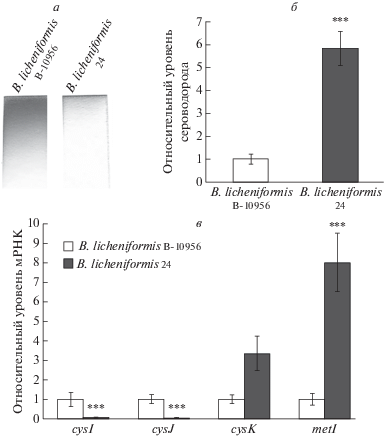
B. licheniformis 24 продуцирует значительно меньше сероводорода, чем B. licheniformis B-10956
Помимо ферментов, нейтрализующих активные формы кислорода, этим действием обладает сероводород [34]. Согласно полученным нами данным, B. licheniformis 24 продуцирует гораздо меньше сероводорода, чем B. licheniformis B-10956 (рис. 5а, б). Определены уровни экспрессии генов, кодирующих ферменты, связанные с метаболизмом сероводорода. Оперон cysIJ кодирует α- и β-субъединицы сульфитредуктазы. Ген cysK кодирует цистеинсинтазу, потребляющую сероводород при синтезе цистеина из О-ацетил-L-серина [35]. Ген metI кодирует фермент с активностями цистатион-γ-лиазы/О-ацетилгомосеринтиолазы, потребляющий сероводород в пути биосинтеза метионина [36]. Установлено, что уровень мРНК оперона cysIJ более чем на порядок снижен в B. licheniformis 24 по сравнению с B. licheniformis B-10956 (рис. 5в), тогда как уровень экспрессии генов cysK и metI, наоборот, в несколько раз выше у B. licheniformis 24 (рис. 5в).
Рис. 5.
Уровень продукции сероводорода штаммом B. licheniformis 24 ассоциирован с низкой экспрессией оперона cysIJ, кодирующего сульфитредуктазу. а ‒ Анализ продукции сероводорода с помощью ацетата свинца. Представлены характерные фотографии фильтровальной бумаги, смоченной 2%-ным раствором ацетата свинца, после 16-часовой выдержки в пробирке с жидкими культурами исследуемых штаммов. б ‒ Денситометрический анализ интенсивности окрашивания полосок фильтровальной бумаги, представленных на рис. 5а. Анализ выполнен с помощью программы ImageJ (https://imagej.nih.gov/ij/). Разброс данных представлен стандартным отклонением для средних значений, ***p < 0.001. в ‒ Относительные уровни экспрессии генов метаболизма сероводорода. За единицу принят уровень экспрессии генов в штамме B. licheniformis В-10956. Разброс данных представлен стандартным отклонением для средних значений в трех независимых экспериментах. **p < 0.01, ***p < 0.001. Статистическая значимость рассчитана с помощью двустороннего критерия Стьюдента для сравнения двух независимых средних.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Повышенный радиационный фон на космических станциях действует не только напрямую, нарушая целостность генетической информации и вызывая мутации, но и опосредованно, индуцируя радиолиз воды, сопровождаемый генерацией перекиси водорода и активных форм кислорода ‒ в основном радикалов гидроксила ($^{\centerdot }{\text{OH}}$) и супероксид анионов (${\text{O}}_{2}^{{ - \centerdot }}$) [37]. Следовательно, адаптация микробиологического сообщества к условиям космических станций включает не только усиление механизмов репарации ДНК, но и систем ответа на окислительный стресс. Исследованный нами штамм B. licheniformis 24, а также ранее охарактеризованный B. subtilis 20 [9] выделены из внутренних объемов МКС и, как оказалось, проявляют сверхустойчивость к окислительным агентам. Механизмы устойчивости к окислительному стрессу можно условно разделить на две группы: ферментативную (или активную), то есть механизмы с участием ферментов, нейтрализующих активные формы кислорода путем их восстановления, и неферментативную (или пассивную), включающую органические и неорганические соединения, а также белки и пептиды, которые инактивируют активные формы кислорода, вступая с ними в химическую реакцию [38‒40]. В штамме B. subtilis 20 нами выявлена активация компонентов обоих систем, а именно сверхэкспрессия гена каталазы (katA) и повышение продукции сероводорода [9]. Участие сероводорода в устойчивости к окислительному стрессу, вызванному действием антибиотиков, для бактерий хорошо известно [41]. И для нас стало неожиданностью, что в B. licheniformis 24 его продукция снижена в несколько раз по сравнению со штаммом B. licheniformis В-10956 (рис. 5а). Однако штамм B. licheniformis 24 оказался более устойчив к действию перекиси водорода, чем B. subtilis 20, сверхпродуцирующий сероводород [9]. Это проявляется в отсутствии зоны ингибирования роста у B. licheniformis 24, тогда как у штамма B. subtilis 20 эта зона присутствует (см. [9]). По-видимому, сероводород важный, но не единственный компонент клеточной защиты от активных форм кислорода. Согласно литературным данным, сероводород не играет важной роли в устойчивости к перекиси водорода грамотрицательной бактерии Shewanella oneidensis [42]. В штамме B. licheniformis 24 сверхэкспрессированы гены нескольких ферментов антиоксидантной защиты, а именно: каталазы (katX) и супероксиддисмутаз (sodA и sodF), ‒ а также ген одного из регуляторов клеточного ответа на окислительный стресс ‒ perR. Можно предположить, что коллективная активация генов perR регулона и других антиоксидантных систем компенсирует низкую продукцию сероводорода и обеспечивает наблюдаемую гиперустойчивость B. licheniformis 24 к окислительным агентам. В некоторых работах обнаружена сверхэкспрессия perR-зависимого гена mrgA, кодирующего ферритинподобный белок, связывающий Fe(II) в процессе прорастания спор B. subtilis, экспонированных в условиях открытого космоса, и при симуляции условий Марса [43]. Однако показано, что mrgA играет незначительную роль в устойчивости к окислительному стрессу, сопутствующему облучению ионизирующей радиацией [44]. Эти данные хорошо согласуются с тем, что в B. licheniformis 24 уровень экспрессии mrgA не отличается от такового для штамма B. licheniformis В-10956. На основании данных по сниженному уровню экспрессии гена негативного регулятора обмена железа fur (рис. 2б) в B. licheniformis 24 по сравнению с B. licheniformis В-10956 можно предположить более активный импорт ионов Fe2+ у B. licheniformis 24, что может способствовать более интенсивной продукции активных форм кислорода через реакцию Фентона. В тоже время в литературе есть данные о том, что белок Fur инактивируется во время окислительного стресса [28, 45]. Показано, что у B. licheniformis в условиях действия перекиси водорода происходит активация экспрессии Fur-зависимых генов [46]. Высказано предположение, что активация Fur-зависимых генов связана с восполнением недостатка ионов Fe2+, возникающего как следствие окислительных реакций. Возможно, изменения в активности Fur и зависимых от него генов не следует рассматривать как важную часть непосредственно антиоксидатной защиты клеток.
Нельзя исключать и участие других механизмов устойчивости к окислительному стрессу. Например, на основе сравнения геномов двух штаммов, изолированных из чистых помещений, где проводится сборка космических аппаратов, и сверхустойчивых к окислительному стрессу B. pumilus SAFR32 и B. safensis FO-36b с геномами чувствительных к перекиси водорода штаммов B. pumilus ATCC7061T и B. safensis JPL-MERTA-8-2 выдвинута гипотеза, что гены репарации ДНК могут быть ассоциированы с наблюдаемым фенотипом гиперустойчивости к стрессовым воздействиям [47, 48]. Очевидно, что репарация ДНК нужна при исправлении вызванных окислительным стрессом повреждений в геномной ДНК.
Таким образом, на основании полученных данных можно заключить, что сверхустойчивость штамма B. licheniformis 24 к окислительному стрессу ассоциирована с гиперактивностью ферментативных систем антиоксидантной защиты.
Часть работы выполнена на оборудовании ЦКП “Геном” ИМБ РАН (http://www.eimb.ru/ RUSSIAN_NEW/INSTITUTE/ccu_genome_c.php).
Работа выполнена при финансовой поддержке Российского научного фонда (№ 17-74-30030) (табл. 1, рис. 3‒5), Российского фонда фундаментальных исследований (№ 18-29-07021) (рис. 2), гранта Министерства науки и высшего образования Российской Федерации (№ 075-15-2019-1660) (табл. 2), Программ Российской академии наук № 01201367566 (рис. 1) и № 01201373016 (табл. 3).
В представленной работе не использовали животных в качестве объектов исследования.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Horneck G., Klaus D.M., Mancinelli R.L. (2010) Space microbiology. Microbiol. Mol. Biol. Rev. 74, 121‒156.
Kempf M.J., Chen F., Kern R., Venkateswaran K. (2005) Recurrent isolation of hydrogen peroxide-resistant spores of Bacillus pumilus from a spacecraft assembly facility. Astrobiology. 5, 391–405.
Gioia J., Yerrapragada S., Qin X., Jiang H., Igboeli O.C., Muzny D., Dugan-Rocha S., Ding Y., Hawes A., Liu W., Perez L., Kovar C., Dinh H., Lee S., Nazareth L., Blyth P., Holder M., Buhay C., Tirumalai M.R., Liu Y., Dasgup-ta I., Bokhetache L., Fujita M., Karouia F., Eswara Moorthy P., Siefert J., Uzman A., Buzumbo P., Verma A., Zwiya H., McWilliams B.D., Olowu A., Clinkenbeard K.D., Newcombe D., Golebiewski L., Petro-sino J.F., Nicholson W.L., Fox G.E., Venkateswaran K., Highlander S.K., Weinstock G.M. (2007) Paradoxical DNA repair and peroxide resistance gene conservation in Bacillus pumilus SAFR-032. PloS One. 2, e928.
Novikova N.D., Polikarpov N.A., Poddubko S.V., Deshevaya E.A. (2001) The results of microbiological research of environmental microflora of orbital station MIR. Proceedings of the 31st International Conference on Environmental Systems, Orlando, FL, USA, (2001), #2001-2001-2310.
Пирсон Д.Л., Мак Джиннис М.Р., Викторов А.Н. (1994) Микробиологическая обсемененность. Космическая биология и медицина: обитаемость космических летательных аппаратов (Т2). Москва: Наука.
Lang J.M., Coil D.A., Neches R.Y., Brown W.E., Cavalier D., Severance M., Hampton-Marcell J.T., Gilbert J.A., Eisen J.A. (2017) A microbial survey of the International Space Station (ISS). Peer J. 5, e4029.
Новикова Н.Д., Пирсон Д.Л., Поддубко С.В., Дешевая Е.А., Отт С.М., Кастро В.А., Брюс Р.Д. (2009) Микробиологическая характеристика среды обитания. Космическая биология и медицина: российско-американское сотрудничество в области космической биологии и медицины (Т5). Москва: Наука.
chijo T., Yamaguchi N., Tanigaki F., Shirakawa M., Nasu M. (2016) Four-year bacterial monitoring in the International space station-Japanese experiment module “Kibo” with culture-independent approach. NPJ Microgravity. 2, 16007
Карпов Д.С., Домашин А.И., Котлов М.И., Осипова П.Д., Киселева С.В., Серегина Т.А., Гончаренко А.В., Миронов А.С., Карпов В.Л., Поддубко С.В. (2020) Биотехнологический потенциал штамма Bacillus subtilis 20. Молекуляр. биология. 54, 137‒145
Баранов В.М., Новикова Н.Д., Поликарпов Н.А., Сычев В.Н., Левинских М.А., Алексеев В.Р., Окуда Т., Сугимото М., Гусев О.А., Григорьев А.И. (2009) Эксперимент “Биориск”: 13-месячная экспозиция покоящихся форм организмов на внешней стороне Российского сегмента Международной космической станции (предварительные результаты). ДАН. 426, 706–709
Новикова Н.Д., Поликарпов Н.А., Дешевая Е.А., Свистунова Ю.В., Григорьев А.И. (2007) Результаты исследований в эксперименте по длительному экспонированию микроорганизмов в условиях открытого космоса. Авиакосмическая и экологическая медицина. 41, 14‒20.
Sauer S., Freiwald A., Maier T., Kube M., Reinhardt R., Kostrzewa M., Geider K. (2008) Classification and identification of bacteria by mass spectrometry and computational analysis. PloS One. 3, e2843.
Clark C.M., Costa M.S., Sanchez L.M., Murphy B.T. (2018) Coupling MALDI-TOF mass spectrometry protein and specialized metabolite analyses to rapidly discriminate bacterial function. Proc. Natl. Acad. Sci. USA. 115, 4981‒4986.
Felsenstein J., Sinauer A. (2007) Inferring Phylogenies. Sinauer Associates, Inc., Sunderland, Mass.
Kumar S., Stecher G., Li M., Knyaz C., Tamura K. (2018) MEGA X: molecular evolutionary genetics ana-lysis across computing platforms. Mol. Biol. Evol. 35, 1547‒1549.
Chambers M.C., Maclean B., Burke R., Amodei D., Ruderman D.L., Neumann S., Gatto L., Fischer B., Pratt B., Egertson J., Hoff K., Kessner D., Tasman N., Shulman N., Frewen B., Baker T.A., Brusniak M.Y., Paulse C., Creasy D., Flashner L., Kani K., Moulding C., Seymour S.L., Nuwaysir L.M., Lefebvre B., Kuhlmann F., Roark J., Rainer P., Detlev S., Hemenway T., Huhmer A., Langridge J., Connolly B., Chadick T., Holly K., Eckels J., Deutsch E.W., Moritz R.L., Katz J.E., Agus D.B., MacCoss M., Tabb D.L., Mallick P. (2012) A cross-platform toolkit for mass spectrometry and proteomics. Nat. Biotechnol. 30, 918–920.
Niedermeyer T.H., Strohalm M. (2012) mMass as a software tool for the annotation of cyclic peptide tandem mass spectra. PloS One. 7, e44913.
Xu D., Cote J.-C. (2003) Phylogenetic relationships between Bacillus species and related genera inferred from comparison of 39 end 16S rDNA and 5' end 16S–23S ITS nucleotide sequences. Int. J. Syst. Evol. Microbiol. 53, 695–704.
Velho R.V., Caldas D.G., Medina L.F., Tsai S.M., Brandelli A. (2011) Real-time PCR investigation on the expression of sboA and ituD genes in Bacillus spp. Lett. Appl. Microbiol. 52, 660‒666.
Zuker M. (2003) Mfold web server for nucleic acid fol-ding and hybridization prediction. Nucleic Acids Res. 31, 3406‒3415.
McWilliam H., Li W., Uludag M., Squizzato S., Park Y.M., Buso N., Cowley A.P., Lopez R. (2013) Analysis tool web services from the EMBL-EBI. Nucleic Acids Res. 41, W597‒W600.
Bourque S.N., Valero J.R., Lavoie M.C., Levesque R.C. (1995) Comparative analysis of the 16S to 23S ribosomal intergenic spacer sequences of Bacillus thuringiensis strains and subspecies and of closely related species. Appl. Environ. Microbiol. 61, 1623–1626.
Strejcek M., Smrhova T., Junkova P., Uhlik O. (2018) Whole-cell MALDI-TOF MS versus 16S rRNA gene analysis for identification and dereplication of recurrent bacterial isolates. Front. Microbiol. 9, 1294.
Lee J.W., Helmann J.D. (2006) The PerR transcription factor senses H2O2 by metal-catalysed histidine oxidation. Nature. 440, 363–367.
Bagyan I., Casillas-Martinez L., Setlow P. (1998) The katX gene, which codes for the catalase in spores of Bacillus subtilis, is a forespore-specific gene controlled by sigmaF, and KatX is essential for hydrogen peroxide resistance of the germinating spore. J. Bacteriol. 180, 2057–2062.
Walling C. (1975) Fenton’s reagent revisited. Acc. Chem. Res. 8, 125–131.
Faulkner M.J., Helmann J.D. (2011) Peroxide stress elicits adaptive changes in bacterial metal ion homeostasis. Antioxid. Redox. Signal. 15, 175–189.
Pinochet-Barros A., Helmann J.D. (2018) Redox sensing by Fe2+ in bacterial Fur family metalloregulators. Antioxid. Redox. Signal. 29, 1858–1871.
Chiancone E., Ceci P. (2010) The multifaceted capacity of Dps proteins to combat bacterial stress conditions: detoxification of iron and hydrogen peroxide and DNA binding. Biochim. Biophys Acta. 1800, 798–805.
Jimenez-Del-Rio M., Suarez-Cedeno G., Velez-Pardo C. (2010) Using paraquat to generate anion free radicals and hydrogen peroxide in in vitro: antioxidant effect of vitamin E: a procedure to teach theoretical and experimental principles of reactive oxygen species biochemistry. Biochem. Mol. Biol. Educ. 38, 104–109.
Samai M., Sharpe M.A., Gard P.R., Chatterjee P.K. (2007) Comparison of the effects of the superoxide dismutase mimetics EUK-134 and tempol on paraquat-induced nephrotoxicity. Free Radic. Biol. Med. 43, 528–534.
Shu L.J., Yang Y.L. (2017) Bacillus classification based on matrix-assisted laser desorption ionization time-of-flight mass spectrometry-effects of culture conditions. Sci. Rep. 7, 15546.
Inaoka T., Matsumura Y., Tsuchido T. (1999) SodA and manganese are essential for resistance to oxidative stress in growing and sporulating cells of Bacillus subtilis. J. Bacteriol. 181, 1939‒1943.
Nagy P., Winterbourn C.C. (2010) Rapid reaction of hydrogen sulfide with the neutrophil oxidant hypochlorous acid to generate polysulfides. Chem. Res.Toxicol. 23, 1541‒1543.
Li K., Xin Y., Xuan G., Zhao R., Liu H., Xia Y., Xun L. (2019) Escherichia coli uses separate enzymes to produce H2S and reactive sulfane sulfur from L-cysteine. Front. Microbiol. 10, 298.
Auger S., Yuen W.H., Danchin A., Martin-Verstraete I. (2002) The metIC operon involved in methionine biosynthesis in Bacillus subtilis is controlled by transcription antitermination. Microbiology. 148, 507–518.
Lomax M.E., Gulston M.K., O’Neill P. (2002) Chemical aspects of clustered DNA damage induction by ionising radiation. Radiat. Prot. Dosimetry. 99, 63–68.
Riezzo I., Cerretani D., Fiore C., Bello S., Centini F., D’Errico S., Fiaschi A.I., Giorgi G., Neri M., Pomara C., Turillazzi E., Fineschi V. (2010) Enzymatic-nonenzymatic cellular antioxidant defense systems response and immunohistochemical detection of MDMA, VMAT2, HSP70, and apoptosis as biomarkers for MDMA (Ecstasy) neurotoxicity. J. Neurosci. Res. 88, 905–916.
Panda S.K. (2012) Assay Guided Comparison for Enzymatic and Non-enzymatic Antioxidant Activities with Special Reference to Medicinal Plants. IntechOpen. https://doi.org/10.5772/50782; https://www.intechopen.com/books/antioxidant-enzyme/assay-guided-comparison-for-enzymatic-and-non-enzymatic-antioxidant-activities-with-special-referenc
Abel C.-G., Emmanuel A.-H., Irasema V.-A., Miguel Angel M.-T. (2012) Oligoglucan Elicitor Effects during Plant Oxidative Stress. IntechOpen. https://doi.org/10.5772/26057; https://www.intechopen.com/books/cell-metabolism-cell-homeostasis-and-stress-response/oligoglucan-elicitor-effects-during-plant-oxidative-stress
Mironov A., Seregina T., Nagornykh M., Luhachack L.G., Korolkova N., Lopes L.E., Kotova V., Zavilgelsky G., Shakulov R., Shatalin K., Nudler E. (2017) Mechanism of H2S-mediated protection against oxidative stress in Escherichia coli. Proc. Natl. Acad. Sci. USA. 114, 6022–6027.
Wu G., Wan F., Fu H., Li N., Gao H. (2015) A matter of timing: contrasting effects of hydrogen sulfide on oxidative stress response in Shewanella oneidensis. J. Bacteriol. 197, 3563–3572.
Nicholson W.L., Moeller R., Team P., Horneck G. (2012) Transcriptomic responses of germinating Bacillus subtilis spores exposed to 1.5 years of space and simulated martian conditions on the EXPOSE-E experiment PROTECT. Astrobiology. 12, 469–486.
Moeller R., Raguse M., Reitz G., Okayasu R., Li Z., Klein S., Setlow P., Nicholson W.L. (2014) Resistance of Bacillus subtilis spore DNA to lethal ionizing radiation damage relies primarily on spore core components and DNA repair, with minor effects of oxygen radical detoxification. Appl. Environ. Microbiol. 80, 104–109.
Varghese S., Wu A., Park S., Imlay K.R., Imlay J.A. (2007) Submicromolar hydrogen peroxide disrupts the ability of Fur protein to control free-iron levels in Escherichia coli. Mol. Microbiol. 64, 822–830.
Schroeter R., Voigt B., Jurgen B., Methling K., Pother D.C., Schafer H., Albrecht D., Mostertz J., Mader U., Evers S., Maurer K.H., Lalk M., Mascher T., Hecker M., Schweder T. (2011) The peroxide stress response of Bacillus licheniformis. Proteomics. 11, 2851–2866.
Tirumalai M.R., Rastogi R., Zamani N., O’Bryant Williams E., Allen S., Diouf F., Kwende S., Weinstock G.M., Venkateswaran K.J., Fox G.E. (2013) Candidate genes that may be responsible for the unusual resistances exhibited by Bacillus pumilus SAFR-032 spores. PloS One. 8, e66012.
Tirumalai M.R., Stepanov V.G., Wunsche A., Montazari S., Gonzalez R.O., Venkateswaran K., Fox G.E. (2018) Bacillus safensis FO-36b and Bacillus pumilus SAFR-032: a whole genome comparison of two spacecraft assembly facility isolates. BMC Microbiol. 18, 57.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


