Молекулярная биология, 2020, T. 54, № 5, стр. 718-724
Функциональные особенности длинных некодирующих РНК, содержащих последовательности мобильных генетических элементов
К. А. Татосян a, Л. С. Зиневич b, Д. Э. Демин a, c, А. М. Шварц a, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Институт биологии развития им. Н.К. Кольцова Российской академии наук
119334 Москва, Россия
c Московский физико-технический институт (Национальный исследовательский университет)
141701 Московская обл., Долгопрудный, Россия
* E-mail: shvarec@yandex.ru
Поступила в редакцию 21.04.2020
После доработки 21.04.2020
Принята к публикации 27.04.2020
Аннотация
Длинные некодирующие РНК (днРНК) могут выполнять различные функции: участвовать в организации хроматина и регуляции экспрессии генов на транскрипционном и посттранскрипционном уровнях, в регуляции активности и стабильности некоторых белков. Общая черта большинства известных днРНК – наличие в их составе последовательностей мобильных генетических элементов в смысловой или инвертированной ориентации. Согласно ряду исследований мобильные генетические элементы могут служить функциональными модулями, отвечающими за взаимодействие днРНК с определенными белками, участками ДНК или других РНК. Имеющиеся данные позволяют описывать группы днРНК, обладающих сходными чертами строения и содержащих определенные мобильные генетические элементы, и предсказывать свойства новых днРНК. В данном обзоре обобщены результаты исследований, посвященных роли мобильных генетических элементов в реализации функций днРНК.
ВВЕДЕНИЕ
Длинные некодирующие РНК (днРНК) – это транскрипты длиной более 200 н., не участвующие в процессе синтеза белков в качестве матрицы [1]. В клетках человека синтезируется по разным оценкам от 30 до 270 тысяч разных днРНК [2, 3]. Несмотря на меньшую, в среднем, консервативность генов днРНК по сравнению с белоккодирующими генами, некоторые исследователи отмечают консервативность их расположения относительно других генов, а также консервативность вторичных структур днРНК [4–6]. Показано, что днРНК играют важную роль в регуляции экспрессии генов на транскрипционном и посттранскрипционном уровнях, в организации хроматина, регуляции стабильности разных типов РНК и некоторых белков [7, 8].
Современные данные указывают на высокую тканевую специфичность экспрессии и функционирования днРНК. Так, примерно 78% днРНК экспрессируется только в одном или нескольких типах тканей. Больше всего разнообразных днРНК экспрессируется в тканях нервной системы и семенников [9, 10]. Масштабный скрининг функций днРНК человека, проведенный на клетках разного типа, показал, что экспрессия 499 днРНК важна для их нормальной пролиферации. При этом эффект подавляющего большинство таких днРНК наблюдался только в клетках одного типа [11]. днРНК играют важную роль в регуляции клеточной дифференцировки [12], а также в развитии различных заболеваний, в том числе нейродегенеративных [13] и онкологических [14].
Более 80% известных днРНК содержат последовательности мобильных генетических элементов (МГЭ). Такие последовательности занимают в среднем более 40% общей длины днРНК [15]. МГЭ могут играть важную роль в функционировании днРНК: содержать регуляторные последовательности [16], обеспечивать взаимодействие с другими РНК, содержащими комплементарную последовательность сходного элемента [17], позволять днРНК взаимодействовать с различными участками ДНК, содержащими МГЭ, как с помощью комплементарных взаимодействий, так и за счет образования триплексов ДНК : ДНК : РНК [18]. В данном обзоре описаны результаты исследований, показавших важную роль последовательностей МГЭ в реализации днРНК своих биологических функций.
СТИМУЛЯЦИЯ ТРАНСЛЯЦИИ
В работе Carrieri и соавт. [16] изучено взаимодействие мРНК гена Uchl1 (ubiquitin carboxyterminal hydrolase L1, С-концевая убиквитингидролаза L1) и днРНК AS Uchl1, которые синтезируются с разных цепей одного локуса ДНК, благодаря чему первый и второй экзоны мРНК могут комплементарно взаимодействовать с первым экзоном днРНК (рис. 1а). Оказалось, что днРНК AS Uchl1 способна активировать трансляцию взаимодействующей с ней мРНК Uchl1, причем ключевую роль в этом взаимодействии играет инвертированная последовательность МГЭ SINE B2, локализованная в днРНК AS Uchl1. Этот элемент принадлежит к классу SINE (Short INterspersed Elements) – относительно коротких (от 100 до 600 н.) ретротранспозонов, и филогенетически происходит от молекулы тРНК [19]. Описанное взаимодействие мРНК–днРНК наблюдается преимущественно в условиях подавления сигнального пути mTORС1, когда активируется экспорт днРНК AS Uchl1 из ядра в цитоплазму [16].
Рис. 1.
Функциональные особенности днРНК, в реализации которых могут участвовать последовательности мобильных генетических элементов.
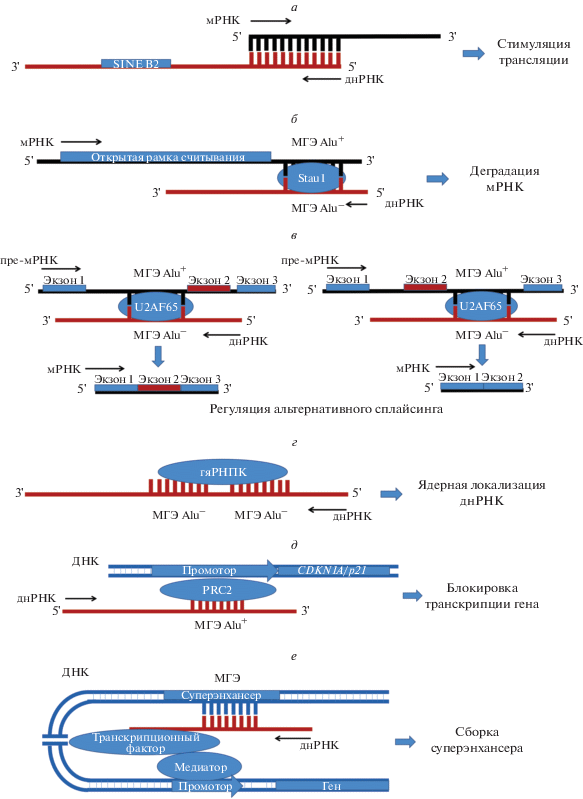
Проведен также поиск других днРНК, содержащих последовательность SINE B2 и обладающих 5′-концевым участком, комплементарным 5′-концевой области какой-либо мРНК. Cтимулирующее действие днРНК на трансляцию парной мРНК показано еще для одной пары – мРНК Utx–днРНК AS Utx [16].
Эти данные позволили создать технологию трансляционных энхансеров, представляющих собой искусственную днРНК, содержащую 5′-концевую последовательность длиной 72 н., комплементарную 5′-концевой области целевой мРНК, а также инвертированную последовательность SINE B2 и полиА-последовательность на 3′-конце. Такие днРНК позволяют до 10 раз увеличить эффективность трансляции целевой мРНК [20].
АКТИВАЦИЯ ДЕГРАДАЦИИ мРНК
В ряде работ, проведенных под руководством Lynne Maquat, описано существование нового механизма деградации мРНК – SMD (Staufen1-mediated mRNA decay, Staufen1-опосредованный распад мРНК) [21–23]. Этот механизм, в частности, уничтожает мРНК, которые содержат в 3'-некодирующей области (3'-НТО) последовательности Alu, наиболее распространенного у человека семейства SINE, происходящего из 7SL РНК. Для запуска этого механизма Alu в 3'-НТО мРНК должна соединиться с комплементарной последовательностью такого же элемента в составе днРНК [17]. Белок Stau1 связывается с двухцепочечным участком, который образуется благодаря этому комплементарному взаимодействию и запускает процесс деградации мРНК (рис. 1б). Более точные условия реализации данного механизма в настоящий момент не установлены, однако показано, что к активации SMD приводит комплементарное взаимодействие мРНК–днРНК с расчетным значением ∆G в диапазоне от –151.7 до –444.2 ккал/моль, тогда как взаимодействие, характеризующееся меньшим ∆G (от 0 до –108.2 ккал/моль), недостаточно стабильно. Однако в отдельных случаях даже взаимодействие с расчетным ∆G = –416 ккал/моль оказалось недостаточным для запуска деградации мРНК. Чтобы выявить гены, мРНК которых подвержены деградации по механизму SMD, проведен поиск матриц, содержащих Alu-последовательность в 3'-НТО, уровень которых повышается при подавлении экспрессии STAU1. Такой подход позволил найти у человека 24 гена, экспрессия которых может контролироваться этим механизмом, включая гены таких важных участников процессов канцерогенеза, как TP53, SERPINE1, AKT2 [23].
Позже аналогичный механизм деградации мРНК обнаружили не только у человека, но и у грызунов. В 3'-НТО мРНК грызунов, деградирующих с помощью SMD, нашли SINE B1, B2, B4 и ID, которые, благодаря комплементарному взаимодействию с такими элементами в составе днРНК, формируют двухцепочечный сайт связывания с белком Stau1, обеспечивая деградацию соответствующей мРНК [24].
КОНТРОЛЬ СПЛАЙСИНГА мРНК
Ген, кодирующий днРНК 5S-OT, частично перекрывается с геном 5S рРНК, однако транскрипция этих генов идет в противоположных направлениях. Подавление экспрессии днРНК 5S-OT в клетках человека приводит к изменению сплайсинга более 200 экзонов различных транскриптов. Влияние этой днРНК на сплайсинг обусловлено ее взаимодействием с фактором сплайсинга U2AF65. Известно, что днРНК 5S-OT содержит антисмысловую Alu-последовательность, поэтому все гены, транскрипты которых меняются при снижении уровня этой днРНК, были проверены на наличие в их составе смысловой Alu-последовательности. Оказалось, что 90% таких генов содержат Alu-повтор, причем чаще всего в районе ±2000 н. от границ экзона, сплайсинг которого зависит от днРНК 5S-OT. Как правило, последовательность Alu располагалась перед экзонами, которые под действием днРНК 5S-OT включались в состав зрелой мРНК и после экзонов, которые исключались из зрелой мРНК (рис. 1в). Данная днРНК может играть важную роль в активации и дифференцировке макрофагов. Так, при активации моноцитарной линии клеток человека THP-1 проболовым эфиром повышался уровень экспрессии как днРНК 5S-OT, так и маркера дифференцировки макрофагов CD11b. Подавление экспрессии днРНК 5S-OT с помощью коротких интерферирующих РНК (киРНК) приводило к снижению уровня CD11b в активированных клетках THP-1 [25].
ЯДЕРНАЯ ЛОКАЛИЗАЦИЯ днРНК
Недавно был проведен поиск сигнальной последовательности, определяющей ядерную локализацию РНК [24]. С этой целью изготовлена библиотека тестовых РНК, содержащих более 5500 разных фрагментов РНК с показанной ядерной локализацией. С использованием этого подхода установлено, что ядерная локализация характерна для РНК, содержащих несколько близкорасположенных С/U-гексамеров. Эти последовательности взаимодействуют с ядерным белком гяРНПК (Heterogeneous nuclear ribonucleoprotein K), что и определяет их локализацию (рис. 1г). Такие последовательности в днРНК преимущественно встречаются в составе антисмысловых Alu-повторов. Показано, что днРНК, содержащие один или несколько антисмысловых Alu-повторов, локализуются главным образом в ядре, однако это правило касается не всех днРНК, что указывает на недостаточную универсальность данного сигнала локализации. Важно отметить, что различия в локализации днРНК, содержащих и не содержащих эту сигнальную последовательность, наблюдались преимущественно в клетках эпителиального происхождения: MCF7, HeLa, HUVEC, HepG2, но практически отсутствовали в линии эмбриональных стволовых клеток Н1 [26].
ПОДАВЛЕНИЕ ЭКСПРЕССИИ ГЕНОВ
С помощью библиотеки киРНК проведен поиск днРНК, стимулирующих пролиферацию линии клеток MCF10A молочной железы человека [27]. Показано, что подавление экспрессии днРНК APTR приводит к выраженному замедлению пролиферации клеток. РНК APTR длиной 2303 н. содержит две последовательности SINE-элемента Alu, расположенные в антисмысловой ориентации. днРНК APTR, с одной стороны, способна взаимодействовать с комплексом подавления транскрипции PRC2, с другой, с промотором гена CDKN1A/p21, продукт которого подавляет пролиферацию (рис. 1д). Экспрессия этой днРНК снижается в условиях теплового или генотоксического стресса, что приводит к активации экспрессии гена CDKN1A/p21. Оказалось, что для взаимодействия с промотором гена CDKN1A/p21 днРНК APTR должна содержать антисмысловые Alu-последовательности. Однако удаление Alu-повторов из промотора гена CDKN1A/p21 не приводило к снижению способности днРНК APTR контролировать его активность. Это указывает на то, что для взаимодействия этой днРНК с промотором гена CDKN1A/p21 важно не столько комплементарное взаимодействие этих последовательностей, сколько белковые факторы, взаимодействующие с антисмысловыми Alu-повторами в составе днРНК [27].
ОРГАНИЗАЦИЯ СУПЕРЭНХАНСЕРОВ
Суперэнхансеры представляют собой организованную группу энхансеров, содержащих большое количество коактиваторов, в частности, медиаторный комплекс Med1. Эти структуры играют важную роль в регуляции дифференцировки клеток млекопитающих [28]. Предполагается, что структура таких регуляторных комплексов может поддерживаться за счет днРНК, в том числе, посредством триплексов ДНК : ДНК : РНК [18] (рис. 1е). Правильность этой гипотезы проверена с использованием днРНК, образующих триплексы с известными областями суперэнхансеров из 27 различных типов клеток и тканей человека. Способность ряда днРНК образовывать триплекс проверена с помощью метода ChiRP-Seq (Chromatin Isolation by RNA purification). В данной работе обнаружено 442 днРНК, отвечающих этим условиям. Экспрессия этих днРНК положительно коррелировала с экспрессией генов, связанных с соответствующими энхансерами [18]. Ранее было показано, что одна из выбранных днРНК, CCAT1, необходима для активации транскрипции гена MYC с помощью суперэнхансера, с которым взаимодействует данная днРНК [29]. Оказалось, что области отобранных днРНК, которые могут взаимодействовать с ДНК, примерно в половине случаев представляют собой последовательности МГЭ, преимущественно SINE (34%). Области взаимодействия данных днРНК и ДНК не пересекаются с сайтами связывания факторов транскрипции, характерных для суперэнхансеров, что может косвенно указывать на структурную роль днРНК [18].
ВОЗМОЖНЫЕ ФУНКЦИИ ПОСЛЕДОВАТЕЛЬНОСТЕЙ МГЭ В СОСТАВЕ днРНК
Описанные выше роли последовательностей МГЭ в составе днРНК подтверждены экспериментально. Однако можно предположить, что многочисленные МГЭ, разбросанные по геному и входящие в состав различных типов РНК, способны выполнять и другие функции.
Присутствие как в днРНК, так и в ряде других типов РНК последовательностей МГЭ в прямой либо в обратной ориентации указывает на потенциальную возможность комплементарного взаимодействия широкого спектра РНК, в том числе, транскрибируемых непосредственно с МГЭ. Образующиеся в этом случае двухцепочечные участки РНК могут служить как мишенью для различных систем деградации РНК, так и приводить к активации механизмов врожденного противовирусного иммунитета [23, 30]. Можно предположить, что экспрессия транскриптов МГЭ, наблюдаемая при некоторых типах стрессов [31], может участвовать в активации клеточного иммунного ответа [30] и приводить к деградации днРНК, содержащих комплементарную последовательность.
Имеются данные, свидетельствующие об участии ряда днРНК в организации хроматина [29, 32]. Мы уже упоминали, что ключевую роль в связывании днРНК с хроматином могут играть последовательности МГЭ, локализованные внутри этих РНК [18]. Ранее показали, что частота хромосомных перестроек между двумя областями генома зависит от их взаимного расположения в ядре [33], а экспрессия мРНК, содержащей последовательности матричных цепей двух отдаленных генов, способствует хромосомной перестройке между этими генами [34]. Можно предположить, что днРНК, в состав которой входят последовательности двух МГЭ, расположенных в интронах разных генов, может стимулировать хромосомные перестройки между ними, в том числе, приводящие к образованию онкогенов.
ЗАКЛЮЧЕНИЕ
Последовательности МГЭ могут как участвовать во взаимодействиях между днРНК и другими типами РНК и геномной ДНК, так и связываться с рядом белковых факторов. Причем зачастую одни и те же элементы в составе разных днРНК вовлечены в разные варианты взаимодействий. Можно предположить, что конкретная функция, которую последовательность МГЭ выполняет в составе днРНК, обусловлена особенностями структуры транскрипта. В ряде исследований выявлены ключевые особенности структуры днРНК, определяющие их роль, благодаря чему удалось воссоздать искусственные днРНК с аналогичными функциями. Так, сконструирована днРНК, способная усиливать экспрессию конкретного гена благодаря 5′-концевой последовательности, комплементарной 5′-концевой области соответствующей мРНК, и инвертированной последовательности SINE B2 [20]. Найдена также целая группа природных днРНК, содержащих инвертированные Alu-повторы, способные эффективно соединяться с Alu-элементами в 3′-НТО определенных мРНК и вызывать их деградацию. Однако не все днРНК, отвечающие выбранным критериям, обладают такой способностью [23]. Причиной этого предположительно могут быть разноплановые свойства Alu-повторов. Так, согласно [22], днРНК, содержащие такие последовательности, способны активировать деградацию мРНК в цитоплазме, в то время как наличие нескольких инвертированных Alu-повторов может определять ядерную локализацию днРНК [26]. Стоит отметить, что влияние инвертированных Alu-повторов на ядерную локализацию днРНК проявляется не во всех клеточных линиях. Этот эффект наблюдали преимущественно в клетках эпителиального происхождения (MCF7, HeLa, HUVEC, HepG2), но практически не отмечали в линии эмбриональных стволовых клеток Н1 или лимфобластоидной линии GM12878 [26]. Любопытно также, что в клетках эпителиального происхождения предсказано большее число днРНК, потенциально способных участвовать в организации суперэнхансеров, по сравнению с клетками Н1 или GM12878 [18]. Это может указывать на большую вовлеченность днРНК в контроль и организацию хроматина именно в эпителиальных клетках.
Таким образом, результаты опубликованных на сегодняшний день исследований свидетельствуют о важной роли последовательностей МГЭ в реализации различных биологических функций днРНК, причем большая часть работ посвящена самому распространенному МГЭ человека – SINE Alu. Однако лишь полученные в нескольких исследованиях данные позволяют установить однозначную связь между структурой днРНК, последовательностью определенной МГЭ и выполняемой функцией.
Исследование выполнено при финансовой поддержке Российского научного фонда (проект № 19-74-10083).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Perkel J.M. (2013) Visiting “noncodarnia”. BioTechniques. 54(301), 303–304.
Hon C.C., Ramilowski J.A., Harshbarger J., Bertin N., Rackham O.J., Gough J., Denisenko E., Schmeier S., Poulsen T.M., Severin J., Lizio M., Kawaji H., Kasukawa T., Itoh M., Burroughs A.M., Noma S., Djebali S., Alam T., Medvedeva Y.A., Testa A.C., Lipovich L., Yip C.W., Abugessaisa I., Mendez M., Hasegawa A., Tang D., Lassmann T., Heutink P., Babina M., Wells C.A., Kojima S., Nakamura Y., Suzuki H., Daub C.O., de Hoon M.J., Arner E., Hayashizaki Y., Carninci P., Forrest A.R. (2017) An atlas of human long non-coding RNAs with accurate 5' ends. Nature. 543, 199–204.
Ma L., Cao J., Liu L., Du Q., Li Z., Zou D., Bajic V.B., Zhang Z. (2019) LncBook: a curated knowledgebase of human long non-coding RNAs. Nucl. Acids Res. 47, D128–D134.
Ponjavic J., Ponting C.P., Lunter G. (2007) Functionality or transcriptional noise? Evidence for selection within long noncoding RNAs. Genome Res. 17, 556–565.
Guttman M., Amit I., Garber M., French C., Lin M.F., Feldser D., Huarte M., Zuk O., Carey B.W., Cassady J.P., Cabili M.N., Jaenisch R., Mikkelsen T.S., Jacks T., Hacohen N., Bernstein B.E., Kellis M., Regev A., Rinn J.L., Lander E.S. (2009) Chromatin signature reveals over a thousand highly conserved large non-coding RNAs in mammals. Nature. 458, 223–227.
Ulitsky I. (2016) Evolution to the rescue: using comparative genomics to understand long non-coding RNAs. Nat. Rev. Genet. 17, 601–614.
Буре И.В., Кузнецова Е.Б., Залетаев Д.В. (2018) Длинные некодирующие РНК и их роль в онкогенезе. Молекуляр. биология. 52, 907–920.
Peng W.X., Koirala P., Mo Y.Y. (2017) LncRNA-mediated regulation of cell signaling in cancer. Oncogene. 36, 5661–5667.
Cabili M.N., Trapnell C., Goff L., Koziol M., Tazon-Vega B., Regev A., Rinn J.L. (2011) Integrative annotation of human large intergenic noncoding RNAs reveals global properties and specific subclasses. Genes Dev. 25, 1915–1927.
Liu S.J., Nowakowski T.J., Pollen A.A., Lui J.H., Horlbeck M.A., Attenello F.J., He D., Weissman J.S., Kriegstein A.R., Diaz A.A., Lim D.A. (2016) Single-cell analysis of long non-coding RNAs in the developing human neocortex. Genome Biol. 17, 67.
Koch L. (2017) Functional genomics: Screening for lncRNA function. Nat. Rev. Genet. 18, 70.
Fatica A., Bozzoni I. (2014) Long non-coding RNAs: new players in cell differentiation and development. Nat. Rev. Genet. 15, 7–21.
Riva P., Ratti A., Venturin M. (2016) The long non-coding RNAs in neurodegenerative diseases: novel mechanisms of pathogenesis. Curr. Alzheimer Res. 13, 1219–1231.
Bhan A., Soleimani M., Mandal S.S. (2017) Long noncoding RNA and cancer: a new paradigm. Cancer Res. 77, 3965–3981.
Kelley D., Rinn J. (2012) Transposable elements reveal a stem cell-specific class of long noncoding RNAs. Genome Biol. 13, R107.
Carrieri C., Cimatti L., Biagioli M., Beugnet A., Zucchelli S., Fedele S., Pesce E., Ferrer I., Collavin L., Santoro C., Forrest A.R., Carninci P., Biffo S., Stupka E., Gustincich S. (2012) Long non-coding antisense RNA controls Uchl1 translation through an embedded SINEB2 repeat. Nature. 491, 454–457.
Gong C., Maquat L.E. (2011) lncRNAs transactivate STAU1-mediated mRNA decay by duplexing with 3' UTRs via Alu elements. Nature. 470, 284–288.
Soibam B. (2017) Super-lncRNAs: identification of lncRNAs that target super-enhancers via RNA:DNA:DNA triplex formation. RNA. 23, 1729–1742.
Krayev A.S., Markusheva T.V., Kramerov D.A., Ryskov A.P., Skryabin K.G., Bayev A.A., Georgiev G.P. (1982) Ubiquitous transposon-like repeats B1 and B2 of the mouse genome: B2 sequencing. Nucl. Acids Res. 10, 7461–7475.
Yao Y., Jin S., Long H., Yu Y., Zhang Z., Cheng G., Xu C., Ding Y., Guan Q., Li N., Fu S., Chen X.J., Yan Y.B., Zhang H., Tong P., Tan Y., Yu Y., Fu S., Li J., He G.J., Wu Q. (2015) RNAe: an effective method for targeted protein translation enhancement by artificial non-coding RNA with SINEB2 repeat. Nucl. Acids Res. 43, e58.
Gleghorn M.L., Gong C., Kielkopf C.L., Maquat L.E. (2013) Staufen1 dimerizes through a conserved motif and a degenerate dsRNA-binding domain to promote mRNA decay. Nat. Struct. Mol. Biol. 20, 515–524.
Gong C., Tang Y., Maquat L.E. (2013) mRNA-mRNA duplexes that autoelicit Staufen1-mediated mRNA decay. Nat. Struct. Mol. Biol. 20, 1214–1220.
Park E., Maquat L.E. (2013) Staufen-mediated mRNA decay. Wiley Interdisciplinary Rev. RNA. 4, 423–435.
Lucas B.A., Lavi E., Shiue L., Cho H., Katzman S., Miyoshi K., Siomi M.C., Carmel L., Ares M., Jr., Maquat L.E. (2018) Evidence for convergent evolution of SINE-directed Staufen-mediated mRNA decay. Proc. Natl. Acad. Sci. USA. 115, 968–973.
Hu S., Wang X., Shan G. (2016) Insertion of an Alu element in a lncRNA leads to primate-specific modulation of alternative splicing. Nat. Struct. Mol. Biol. 23, 1011–1019.
Lubelsky Y., Ulitsky I. (2018) Sequences enriched in Alu repeats drive nuclear localization of long RNAs in human cells. Nature. 555, 107–111.
Negishi M., Wongpalee S.P., Sarkar S., Park J., Lee K.Y., Shibata Y., Reon B.J., Abounader R., Suzuki Y., Sugano S., Dutta A. (2014) A new lncRNA, APTR, associates with and represses the CDKN1A/p21 promoter by recruiting polycomb proteins. PLoS One. 9, e95216.
Дидыч Д.А., Тюлькина Д.В., Плешкан В.В., Алексеенко И.В., Свердлов Е.Д. (2015) Суперэнхансеры–регуляторы регуляторных генов развития и рака? Молекуляр. биология. 49, 915–922.
Xiang J.F., Yin Q.F., Chen T., Zhang Y., Zhang X.O., Wu Z., Zhang S., Wang H.B., Ge J., Lu X., Yang L., Chen L.L. (2014) Human colorectal cancer-specific CCAT1-L lncRNA regulates long-range chromatin interactions at the MYC locus. Cell Res. 24, 513–531.
Ahmad S., Mu X., Yang F., Greenwald E., Park J.W., Jacob E., Zhang C.Z., Hur S. (2018) Breaching self-tolerance to Alu duplex RNA underlies MDA5-mediated inflammation. Cell. 172, 797–810. e13.
Dunker W., Zhao Y., Song Y., Karijolich J. (2017) Recognizing the SINEs of infection: regulation of retrotransposon expression and modulation of host cell processes. Viruses. 9, 386.
Herzing L.B., Romer J.T., Horn J.M., Ashworth A. (1997) Xist has properties of the X-chromosome inactivation centre. Nature. 386, 272–275.
Schwartz M., Hakim O. (2014) 3D view of chromosomes, DNA damage, and translocations. Curr. Opin. Genet. Dev. 25, 118–125.
Gupta S.K., Luo L., Yen L. (2018) RNA-mediated gene fusion in mammalian cells. Proc. Natl. Acad. Sci. USA. 115, E12295–E12304.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


