Молекулярная биология, 2021, T. 55, № 1, стр. 96-106
Бетасателлит курчавости листьев хлопка мультана как инструмент для изучения локализации геминивирусов в растениях
Z. Iqbal a, *, **, M. N. Sattar b, M. Khurshid c
a Central Laboratories, King Faisal University
31982 Al-Ahsa, Kingdom of Saudi Arabia
b Department of Agricultural Biotechnology, College of Agriculture and Food Sciences, King Faisal University
31982 Al-Ahsa, Kingdom of Saudi Arabia
c Institute of Biochemistry and Biotechnology, University of the Punjab, Quaid-e-Azam Campus
54590 Lahore, Pakistan
* E-mail: zafar@kfu.edu.sa
** E-mail: zafariqbal2009@gmail.com
Поступила в редакцию 11.03.2020
После доработки 30.04.2020
Принята к публикации 27.05.2020
Аннотация
Бетасателлит курчавости листьев хлопка Мультана (CLCuMB) – распространенный бетасателлит, который обычно встречается вместе с бегомовирусами, ассоциированными с заболеванием курчавости листьев хлопка (CLCuD), в Старом Свете. Этот бетасателлит имеет малоизбирательный характер репликации и транс-реплицируется широким кругом геминивирусов. Бетасателлит CLCuMB кодирует единственную открытую рамку считывания (ORF) βC1, расположенную в комплементарном направлении, продукт которой определяет патогенность, выраженность симптомов и подавляет посттранскрипционный и транскрипционный сайленсинг генов. Бетасателлит успешно используют в качестве вектора для доставки генов путем замены гена βС1 на целевой ген. В представленной работе для замены гена βC1 на ген зеленого флуоресцентного белка (GFP) использован этот же подход и рассмотрено возможное применение полученного конструкта в качестве репортерного вектора, позволяющего изучать локализацию геминивирусов in planta. С этой целью рекомбинантный CLCuMB, продуцирующий GFP (CLCuMB-GFP), был использован для совместного заражения растений Nicotiana benthamiana либо только вирусом курчавости листьев хлопка Кохрана (cotton leaf curl Kokharan virus, CLCuKoV), либо в комбинации с бетасателлитом CLCuMB дикого типа. Показано, что CLCuKoV поддерживал репликацию и системное распространение CLCuMB‑GFP как сам по себе, так и в сочетании с CLCuMB дикого типа. Наличие CLCuMB-GFP было четко показано методами ПЦР и Саузерн-блотинга. На основании полученных результатов можно сделать вывод, что модифицированный бетасателлит CLCuMB может быть использован как удобный инструмент для изучения локализации геминивирусов in planta.
ВВЕДЕНИЕ
Бетасателлиты (род Betasatellite, семейство Tolecusatellitidae) – это кольцевые одноцепочечные сателлиты, геном которых представлен молекулой ДНК длиной около 1.3 т.н. Бетасателлиты часто ассоциированы с геминивирусами (особенно с бегомовирусами). Впервые бетасателлиты описаны в 1999 году [1], а к настоящему времени охарактеризован 61 вид и около 1 300 их полноразмерных изолятов из 37 стран (https://talk.ictvonline.org/taxonomy/) [2]. Бетасателлиты не относятся к типичным сателлитным ДНК-вирусам и используют вирус-помощник для распространения, размножения и упаковки [3]. Все функции бетасателлитов реализованы в единственной комплементарной открытой рамке считывания (ORF), кодирующей продукт βC1 (рис. 1), который способствует повышенному накоплению вируса-помощника в растениях [4, 5], проявлению симптомов [1, 4, 6] и подавлению транскрипционного и посттранскрипционного сайленсинга генов в ответе растения-хозяина [7]. Белок βC1 локализован по периферии клетки и в эндоплазматическом ретикулуме (ЭПР), предположительно участвуя в передвижении вируса [8]. Он также регулирует уровни микроРНК, что играет роль в процессах развития [9], связывается с ДНК и РНК [10], взаимодействует с факторами растения-хозяина [11], подавляет выработку жасмоновой кислоты [12], взаимодействует с белком PsbP, нарушая противовирусный ответ хозяина [13], а также взаимодействует с белком snRK1 [14].
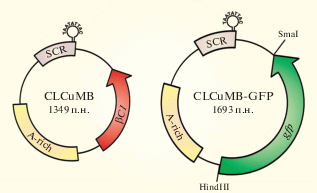
Рис. 1.
Организация генома и примерный размер молекул CLCuMB и CLCuMB-GFP. Молекула CLCuMB кодирует ген единственного продукта, βC1, в комплементарном направлении, который заменили на ген белка GFP (gfp) с получением конструкции CLCuMB-GFP. Сайты рестрикции (SmaI и HindIII) для замены гена βС1 на ген gfp введены с помощью специального набора праймеров. SCR ‒ консервативная область сателлитов; A-rich ‒ обогащенная по аденину область.
Бетасателлит курчавости листьев хлопка Мультана (сotton leaf curl Multan betasatellite, CLCuMB) – единственный из бетасателлитов, часто встречающийся в Азии при заболевании курчавости листьев хлопка (cotton leaf curl disease, CLCuD), ассоциированном с бегомовирусом. CLCuMB транс-реплицируется с разнообразными геминивирусами, в том числе c вирусом курчавости листьев капусты (cabbage leaf curl virus, CabLCV) [15], вирусом курчавости листьев хлопка Кохрана (cotton leaf curl Kokharan virus, CLCuKoV) [5], вирусом курчавости листьев хлопка Гезиры (cotton leaf curl Gezira virus, CLCuGeV) [16], вирусом курчавости листьев хлопка Мультана (cotton leaf curl Multan virus, CLCuMuV), вирусом желтой курчавости листьев томата (tomato yellow leaf curl virus, TYLCV), вирусом желтой скрученности бамии (okra yellow crinkle virus, OYCrV) [17], вирусом курчавости томата Бангалора (tomato leaf curl Bangalore virus, ToLCBaV) [18], вирусом деформирующей курчавости листьев бамии (okra enation leaf curl virus, OELCuV) [19], вирусом курчавости листьев томата (tomato leaf curl virus, ToLCV) [20, 21], вирусом курчавости верхушки свеклы (beet curly top virus, BCTV) [22], вирусом курчавости листьев томата Нью-Дели (tomato leaf curl New Delhi virus, ToLCNDV) [23, 24] и вирусом хлоротической карликовости нута (chickpea chlorotic dwarf virus, CpCDV) [25]. Кодируемый хелперным вирусом белок Rep обеспечивает транс-репликацию бетасателлита после связывания с 9-нуклеотидной последовательностью TAATATTAC, которая является единственной гомологичной последовательностью, общей для бетасателлита и вируса-помощника [26]. Малоизбирательный характер репликации CLCuMB разнообразными геминивирусами указывает на то, что бетасателлит соответствует “гипотезе универсального итерона” и имеет уникальную последовательность, которая имитирует последовательности итеронов и допускает связывание с Rep [27]. Эта способность CLCuMB позволяет считать его чрезвычайно полезным инструментом в молекулярной биологии, который можно использовать в качестве экспрессионного вектора для изучения локализации белков in planta. К настоящему времени бетасателлит CLCuMB уже успешно модифицируют для продукции генов животных в растениях и для индуцированного вирусами сайленсинга генов путем замены ORF гена βC1 на целевой ген [22, 28, 29].
Бегомовирусы (род Begomovirus, семейство Geminiviridae) представляют серьезную угрозу для двудольных растений, как для травянистых, так и для деревьев. Бегомовирусы переносятся белокрылкой; их геном, либо представленный одной молекулой (и тогда называемый монопартитным), либо состоящий из двух компонентов (и называемый бипартитным), упакован в капсид сдвоенной формы и представлен одноцепочечной молекулой ДНК размером около 2800 нуклеотидов [30]. Ассоциированные с вирусами одноцепочечные молекулы ДНК-сателлитов были обнаружены для подавляющего большинства монопартитных бегомовирусов и лишь для некоторых бипартитных бегомовирусов и мастревирусов (род Mastrevirus). К настоящему времени для геминивирусных инфекций охарактеризованы три различных типа одноцепочечных ДНК‑сателлитных молекул: альфасателлиты, бетасателлиты и дельтасателлиты (обнаруживаемые как по отдельности, так и в различных комбинациях) [25, 31, 32].
Понимание и изучение роли бетасателлитов стало важной областью исследований в вирусологии растений за последние два десятилетия. Видоизменение и использование векторов на основе вирусов растений стало многообещающим инструментом молекулярной биологии для экспрессии целевых генов в растениях. Вирусные векторы уже широко используются при продукции полезных белков в коммерческих масштабах, при доставке генетического материала, в генной инженерии, генной терапии и в производстве вакцин [33‒35].
В представленной работе ORF гена βC1 бетасателлита CLCuMB мы заменили на ген gfp для изучения клеточной и субклеточной локализации вируса CLCuKoV в присутствии и в отсутствие бетасателлита CLCuMB дикого типа.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Продукция инфекционных клонов. Геном CLCuMB (регистрационный номер #AJ298903) был использован для замены гена βC1 на ген gfp без нарушения рамки считывания. Для создания рекомбинантного бетасателлита CLCuMB, экспрессирующего белок GFP (CLCuMB-GFP), был разработан специальный набор праймеров к фланкирующей области гена βC1, которые содержали сайт для эндонуклеазы HindIII в прямом праймере и сайты HindIII и SmaI в обратном (табл. 1). Продукт, полученный с использованием этих праймеров, клонировали в вектор pTZ57R/T, используя набор InsTAclone PCR cloning kit (“Thermo Fisher Scientific, Inc.”, США), секвенировали, обрабатывали эндонуклеазой рестрикции HindIII, очищали электрофоретически, лигировали для получения кольцевой молекулы и, наконец, использовали в качестве матрицы для амплификации остова CLCuMB (несущего введенные сайты рестрикции) с праймерами β01/ β02 [36]. Продукт амплификации клонировали в вектор pTZ57R/T с получением конструкции pTZCββC1‒. Область с частичными повторами длиной около 200 п.о. (включающая ориджин репликации), полученную из плазмиды pTZCββC1‒, клонировали в бинарный вектор pGreen0029 по сайтам BamHI и KpnI. Затем фрагмент длиной около 1000 п.о., полученный из pTZCββC1‒, встраивали по сайтам KpnI с получением тандемных повторов (pGNCββC1‒1.2). Ген белка GFP (фрагмент около 700 п.о.), обладающего сильным флуоресцентным сигналом [37], амплифицировали с использованием специфичных праймеров (табл. 1) и клонировали в вектор pGNCββC1‒1.2 по сайтам HindIII и SmaI с получением конструкции CββC1‒/GFP+ (рис. 1). Инфекционные клоны CLCuKoV (регистрационный номер # AJ496286) и CLCuMB описаны ранее [38].
Таблица 1.
Олигонуклеотидные праймеры, использованные в работе
| Праймер | Последовательность 5' → 3' | Примечаниеa |
|---|---|---|
| CLCK CP 35sF CLCK CP 35sR |
GGTCGACGAATTATGTCGAAGCGACCAG GGATCCAATTCAATATCTATTAATTTGTCACG |
Амплификация CP CLCuKoV |
| BETA GFP F BETA GFP R |
GATAAGCTTATGAGTAAAGGAGAAG GGCCCGGGTTATTTGTATAGTTCATC |
Клонирование и детекция gfp |
| BetaC1 F BetaC1 R |
ATAAATCGATATGACAACGAGCGGAACAAA TGTTCCCGGGTTAAACGGTGAACTTTTATT |
Детекция Cβ |
| CBBF CBBR |
TCGGAAGCTTTTCTGCTTATTTGATGGAAATG ATAAAAGCTTCACCCCCGGGATTCCAAACAC AAACCAGCAATGCAT |
Амплификация остова Cβ (без βC1) |
| β01 β02 |
GGTACCACTACGCTACGCAGCAGCC GGTACCTACCCTCCCAGGGGTACA |
Детекция и амплификация Cβ |
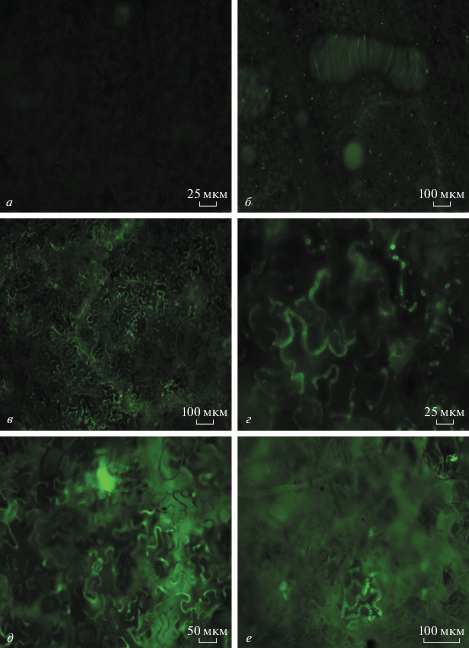
Инфильтрация растений Nicotiana benthamiana. Все конструкции в бинарных векторах использовали для трансформации агробактерий Agrobacterium tumifaciens (штамм LBA 4404) и инфильтрации растений N. benthamiana на 06‒08 стадии развития листьев, как описано ранее [39]. Растения выдерживали в климатической камере при отсутствии насекомых [5]. Растения ежедневно осматривали, проводили фотосъемку и регистрацию симптомов на 20 сутки после инокуляции (dpi). Продукцию белка GFP в обработанных растениях анализировали с помощью флуоресцентного микроскопа LAS AF (“Leica”, Германия). Образцы листьев отбирали на 25 dpi для выделения геномной ДНК, чтобы определить наличие введенных вирусных компонентов с помощью ПЦР и Саузерн-блотинга.
Анализ инфекционности CLCuKoV методами ПЦР и гибридизации Саузерн-блот. Выделенные экстракты геномов [40] анализировали методами диагностической ПЦР и гибридизации Саузерн-блот. Для определения ДНК CLCuKoV (Ko), CLCuMB (Cβ) и CLCuMB‑GFP (CββC1‒/GFP+) были использованы праймеры к гену белка оболочки CP (CLCKCP35sF/CLCKCP35sR), праймеры β01/β02 и праймеры BETA GFP F/R (табл. 1) соответственно. Наблюдаемое in vivo наличие GFP в случае CββC1‒/GFP+ было подтверждено как в обработанных, так и в системно пораженных листьях с использованием праймеров, специфичных к гену gfp (BETA GFP F/R; табл. 1). Для детекции Ko, Cβ и CββC1‒/GFP+ была использована гибридизация Саузерн-блот, как описано ранее [5]. ДНК GFP детектировали с помощью амплифицированного методом ПЦР радиоактивно меченного зонда к гену gfp, содержащего α-[32P]dCTP. Для детектирования сигнала гибридизации использовали прибор Phosphor imager (Personal Molecular Imager FX; “Bio-Rad”, США).
Визуализация GFP. Продукцию белка GFP в обработанных растениях N. benthamiana сначала анализировали с помощью ручной УФ-лампы, а затем использовали флуоресцентный микроскоп LAS AF (“Leica”) для наблюдения за флуоресценцией в обработанных и системно заразившихся листьях.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Анализ инфекционности и симптомов, ассоциированных с вирусом CLCuKoV, в отсутствие или в присутствии бетасателлита CLCuMB
Заражение вирусом CLCuKoV (ДНК-конструкция Ko) методом агробактериальной инфильтрации вызывало скручивание листьев и утолщение жилок у растений N. benthamiana на 12 dpi (рис. 2, табл. 2). Впоследствии степень выраженности фенотипа, ассоциированного с заражением, усиливалась и рост этих растений замедлялся по сравнению с необработанными (рис. 2). Наличие инфекции подтверждено с помощью как ПЦР, так и гибридизации Саузерн-блот (рис. 3а).
Рис. 2.
Фенотип растений N. benthamiana, инокулированных вирусом CLCuKoV и конструкцией CLCuMB-GFP, в отсутствие и в присутствии бетасателлита CLCuMB. Необработанные (здоровые; а) и обработанные контрольным раствором (б), инокулированные Ko (в), Ko и Cβ (г), Ko и CββC1-/GFP+ (д) или Ko, Cβ и CββC1–/GFP+ (е). Растения сфотографированы на 25 сутки после инокуляции (dpi).

Таблица 2.
Анализ инфекционной способности и выраженности симптомов, вызванных вирусом CLCuKoV, в присутствии и в отсутствие CLCuMB и CLCuMB-GFP
| Инокулята | Анализ инфекционности | Симптомыc | Латент-ный период, сутки | ||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| ПЦР-диагностика (инфицированные растения/инокулированные растения) | Саузерн-блотингb | ||||||||||
| эксперимент I | эксперимент II | Ko | Cβ | CββC1–/GFP+ | |||||||
| Ko | Cβ | CββC1–/GFP+ | Ko | Cβ | CββC1/GFP+ | ||||||
| Ko | 3/3 | ‒/‒ | ‒/‒ | 3/3 | ‒/‒ | ‒/‒ | (+) | ‒/‒ | n.t. | LC и VT | 12 |
| Ko + Cβ | 5/5 | 5/5 | ‒/‒ | 5/5 | 5/5 | ‒/‒ | (+) | (+) | n.t. | DLC, VT и ST | 10 |
| Ko + + CββC1‒/GFP+ | 5/5 | ‒/‒ | 5/5 | 5/5 | ‒/‒ | 5/5 | (+) | n.t. | (+) | LC и VT | 12 |
| Ko + Cβ + + CββC1‒/GFP+ | 5/5 | 5/5 | 5/5 | 5/5 | 5/5 | 5/5 | (+) | (+) | (+) | SLC, VT и ST | 10 |
| Контроль | 0/3 | ‒/‒ | ‒/‒ | 0/3 | ‒/‒ | ‒/‒ | (‒) | (‒) | (‒) | NS | |
a Ko ‒ ДНК вируса CLCuKoV, Cβ ‒ ДНК бетасателлита CLCuMB, CββC1‒/GFP+ ‒ конструкция на основе Cβ, экспрессирующая GFP. b Детекция Ko, Cβ and CββC1‒/GFP+ гибридизацией Саузерн-блот обозначена как позитивная (+), негативная (‒) или неанализированная (n.t.). c LC ‒ скручивание листьев, DLC ‒ закручивание листьев книзу, SLC ‒ сильное закручивание листьев, VT ‒утолщение жилок, ST ‒ отставание в росте, NS ‒ отсутствие симптомов.
Рис. 3.
Детекция ДНК CLCuKoV, CLCuMB, и CLCuMB-GFP гибридизацией Саузерн-блот. ДНК CLCuKoV обнаруживали с помощью радиоактивно меченного ампликона гена CP, ДНК CLCuMB – с использованием радиоактивно меченного ампликона гена βC1, а ДНК GFP – c помощью амплифицированного методом ПЦР радиоактивно меченного гена gfp. Все зонды содержали радиоактивную метку α-[32P]dCTP. Образцы ДНК, нанесенные на гель, были получены из верхних листьев ложнообработанных (контрольных) растений (дорожка M), либо необработанных растений (H). а ‒ ДНК CLCuKoV (0.5 нг) нанесена как положительный контроль (дорожка 1), далее нанесены образцы ДНК, выделенные из растений, обработанных Ko (дорожки 2 и 3), Ko и Cβ (дорожка 4), Ko и CββC1‒/GFP+ (дорожки 5 и 6), а также одновременно Ko, Cβ и CββC1‒/GFP+ (дорожки 7 и 8). б ‒ ДНК CLCuMB (0.5 нг) нанесена как положительный контроль (дорожка 1), Ko и Cβ (дорожка 2), Ko, Cβ и CββC1‒/GFP+ (дорожка 3). в ‒ Ko и CββC1‒/GFP+ (дорожка 1), Ko, Cβ и CββC1‒/GFP+ (дорожки 2 и 3), ДНК CββC1‒/GFP+ (1 нг) в качестве положительного контроля (дорожка 4). Репликативные формы вирусной ДНК обозначены как SS ‒ одноцепочечная, SC ‒ суперскрученная, OC ‒ открытая кольцевая.
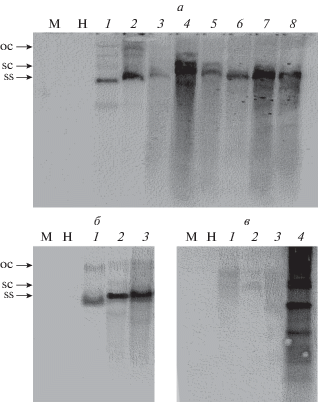
У растений N. benthamiana, обработанных одновременно ДНК-конструкциями Ko и Cβ, латентный период сокращался до 10 суток, а заражение выражалось в закручивании листьев книзу, задержке роста и утолщении жилок (рис. 2, табл. 2). У этих растений выраженность симптомов нарастала с течением времени и развивался хлороз (пожелтение листовых пластинок), чего не наблюдалось в случае обработки растений только конструкцией Ko (рис. 2). Методом гибридизации Саузерн-блот в листьях растений выявлены как Ko, так и Cβ, при этом уровень Ko был выше, чем в растениях, обработанных только Ko (рис. 3а, б).
Анализ инфекционности и симптомов, ассоциированных с вирусом CLCuKoV, в присутствии CLCuMB-GFP
Совместная обработка Ko и CββC1–/GFP+ приводила к ослаблению фенотипических проявлений по сравнению с растениями, обработанными только конструкцией Ko. У всех обработанных растений N. benthamiana на 12 dpi проявлялся фенотип некоторой скрученности листьев и утолщения жилок (рис. 2, табл. 2). Диагностика методом ПЦР показала, что Ko успешно транс-реплицирует бетасателлитную конструкцию CββC1–/GFP+ и формирует жизнеспособный комплекс (хотя ген βC1 отсутствует), причем обе конструкции обнаружены во вновь появляющихся системно зараженных листьях (табл. 2). Методом Саузерн-блотинга показано, что накопление Ko в этом случае сравнимо с таковым для растений, инокулированных только Ko (рис. 3а, в). Хотя конструкт CββC1‒/GFP+ достоверно детектировали методом ПЦР, по данным Саузер-блотинга уровень CββC1‒/GFP+ был очень низким (рис. 3в).
Анализ инфекционности и симптомов, ассоциированных с CLCuKoV, в присутствии CLCuMB и CLCuMB-GFP
По сравнению с растениями, обработанными Ko и CββC1–/GFP+, у растений N. benthamiana, инокулированных одновременно Ko, Cβ и CββC1–/GFP+, были более выражены следующие симптомы: закручивание листьев книзу, уменьшение размера листьев и утолщение жилок (рис. 2, табл. 2). По сравнению же с растениями, обработанными только Ko, в присутствии Cβ сокращался латентный период (который составлял 10 суток), а степень выраженности симптомов усиливалась вплоть до начала цветения и развивался хлороз, чего не наблюдалось у растений, инокулированных только Ko. Транс-репликация Cβ и CββC1–/GFP+, жизнеспособность и стабильность CββC1–/GFP+ подтверждены методом ПЦР. Показано, что CββC1–/GFP+ сохраняется вплоть до 25 dpi (данные не приведены). Саузерн-блот-анализ выявил более высокое накопление Ko и Cβ (рис. 3а, б), в то время как CββC1–/GFP+ накапливался на более низком уровне (рис. 3в).
Флуоресценция GFP в растениях N. benthamiana
Для всех растений, инокулированных gfp-содержащей конструкцией, флуоресценцию белка GFP в сайтах инокуляции наблюдали сначала при помощи УФ‑лампы, а затем методом флуоресцентной микроскопии. В необработанных (системно зараженных) листьях с помощью УФ-лампы флуоресценция не детектировалась. Впоследствии как инокулированные, так и неинокулированные листья исследовали методом флуоресцентной микроскопии. Во всех инокулированных листьях детектировали заметный уровень флуоресценции GFP по сравнению с фрагментами ложно обработанного листа (рис. 4). Тем не менее, в системно зараженных листьях выявлены лишь разрозненные участки флуоресценции GFP в растениях, обработанных конструкцией CLCuMB-GFP совместно с Ko или Ko + CLCuMB (рис. 4).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Геминивирусы реплицируются с помощью интермедиатной двухцепочечной ДНК в ядрах инфицированных клеток растений [26, 41]. Много усилий было приложено для того, чтобы установить точную локализацию геминивирусов в клетках растения-хозяина. Уже показано, что ДНК геминивируов локализуется исключительно в ядре зараженной клетки хозяина. Например, в ходе инфекции вирусом золотистой мозаики томата (tomato golden mosaic virus; TGMV) растений N. benthamiana вирионы аккумулировались в виде паракристаллических скоплений в ядрах ‒ как в клетках сосудистой системы, так и в других клетках [42]. Конденсация хроматина и изменения в структуре ядра, наблюдаемые при заражении TGMV, также были обусловлены присутствием вирусной ДНК в ядре [43]. Кроме того, исследования гибридизации in situ как монопартитных [44, 45], так и бипартитных [46, 47] геминивирусов показали, что вирусная ДНК накапливается в ядрах инфицированных клеток, но обычно ограничивается тканями проводящей системы. До настоящего времени в исследованиях по локализации белков с использованием GFP его ген присоединяли к отдельным генам бегомовируса или бетасателлита, таким как V2, CP, C4 или βC1 [48‒50], с целью выяснить роль этих генов в перемещении вируса или в его локализации. До настоящего момента не было исследований, в которых для выяснения локализации бегомовируса в растении-хозяине кодируемая вирусом ORF была бы заменена на ген gfp.
Геминивирусы реплицируются самостоятельно с очень высокой скоростью, тем самым обеспечивая эффективную амплификацию любого гена, клонированного в их геном [51]. Однако замена вирусного гена на чужеродный может повлиять на основные функции вируса, а встраивание дополнительного гена приведет к увеличению размера вирусного генома, что может нарушить процесс сборки вирионов. Но эти проблемы можно обойти, если использовать в качестве носителя гена ДНК-сателлиты [52, 53].
Бетасателлит CLCuMB использован нами для экспрессии гена gfp вместо βC1 (рис. 1). Полученный рекомбинантный CLCuMB-GFP успешно реплицировался и системно перемещался по растению с помощью родственного хелперного бегомовируса CLCuKoV. Ранее CLCuMB был использован с близким бегомовирусом CLCuMuV и неродственным куртовирусом BCTV для сайленсирования гена GUS и экспрессии гена петунии ChsA, причем тоже путем замены ORF βС1 [28]. В результате этого исследования было показано, что совместное введение рекомбинантного бетасателлита CLCuMB и хелперного вируса приводит к сайленсированию гена GUS в растениях петунии, а в трансгенных растениях табаках была достигнута гиперпродукция белка ChsA. Полученные нами результаты согласуются с этими данными. Таким образом, бетасателлит CLCuMB может быть использован как инструмент для эффективной доставки целевых генов в растения. В аналогичном исследовании авторы использовали возможности CLCuMB для доставки гена B‑клеточной лимфомы-2 (Bcl-2) наряду с двумя генами оболочки ВИЧ-1, gag и p24, в растения N. benthamiana и N. glutinosa, в результате показав успешную экспрессию генов p24 и Bcl-2 в растениях [22, 54]. Бетасателлит CLCuMB использовали и для развития у растений устойчивости к хелперным бегомовирусам. Для этого ген барназы разрезали и встраивали во фланкирующие области CLCuMB таким образом, чтобы он активировался белком Rep, который кодируется хелперным бегомовирусом. Когда растения, обладающие кассетой с бетасателлитом и разделенным геном барназы, заражали вирусом ToLCV, молекулы бетасателлита реплицировались, приводя к экспрессии активного гена барназы, что в свою очередь приводило к разрушению инфицированной клетки [55].
Известно несколько исследований, в которых определяли характер локализации белка βC1 в растительной клетке путем создания химерного белка βC1 и репортерных генов [11, 56‒59]. В них показано, что белок βC1 формирует мультимерные комплексы: точечные или гранулярные тельца ‒ и локализуется по периферии клетки и в ядре. Изучению различных бетасателлитов уделяется большое внимание с целью понять их роль в развитии геминивирусных инфекций. Несмотря на это, только в одной работе описана локализация бегомовируса в присутствии бетасателлита, что удалось выявить методом гибридизации in situ [21]. Поскольку CLCuMB транс-реплицируется разными геминивирусами, использование GFP‑продуцирующей конструкции на основе этого бетасателлита позволит выяснить как локализацию геминивирусов в различных тканях растения, так и лучше понять механизм их репликации.
Обработка растений одновременно рекомбинантной конструкцией CLCuMB-GFP, вирусом CLCuKoV и бетасателлитом дикого типа CLCuMB приводила к более выраженной симптоматике по сравнению с растениями, инокулированными CLCuKoV и CLCuMB. Ранее сообщалось, что делеция A-обогащенного участка китайского бетасателлита желтой курчавости листьев томата приводила к смягчению симптомов у растений N. benthamiana [60]. Следовательно, серьезность симптомов, вероятно, может быть связана с наличием двух A-богатых участков, один из которых содержится в конструкции CLCuMB‑GFP, а второй – в бетасателлите CLCuMB дикого типа. Однако эту гипотезу еще предстоит подтвердить.
В листьях системно зараженных геминивирусом растений N. benthamiana, инокулированных конструкцией CLCuMB-GFP, флуоресценцию наблюдали только в отдельных участках листа. Возможно, это определяется паттерном локализации геминивирусов, так как их репликация ограничена флоэмой и они остаются связанными с тканями проводящей системы [46]. С этими данными согласуются полученные нами результаты: методом гибридизации Саузерн-блот выявлен очень низкий уровень продукта CLCuMB-GFP. Кроме того, раньше показано, что CLCuMB локализован в клетках флоэмы и не способен высвобождать монопартитный бегомовирус из этих клеток, в отличие от некоторых бипартитных бегомовирусов [21]. Низкое содержание GFP может быть связано и с размером конструкции CLCuMB-GFP, так как она содержит почти на 350 нуклеотидов больше, чем CLCuMB дикого типа. Хотя нет прямых сведений относительно влияния размера на продуктивность ДНК-сателлитов, ранее показано, что при введении вставки в геномную последовательность вируса полосатости кукурузы или африканского вируса мозаики маниока сильно снижается эффективность заражения [61, 62], а увеличенный в размере вирусный геном не выдерживает давления отбора на упаковку и транспорт вирусных частиц. Логично предположить, что это справедливо и в случае CLCuMB-GFP.
ЗАКЛЮЧЕНИЕ
Используя световую микроскопию, мы исследовали транс-репликацию CLCuMB-GFP с геминивирусом CLCuKoV. На основании полученных результатов можно рассматривать конструкт CLCuMB-GFP в качестве перспективного репортерного вектора для исследования локализации геминивирусов in planta. В дальнейшем планируется продолжить это исследование с использованием конфокальной микроскопии, что позволит выявить новые возможности CLCuMB-GFP и расширить наши знания по локализации геминивирусов в клетках растения.
Авторы выражают искреннюю благодарность Деканату научных исследований Университета им. Короля Фейсала, Королевство Саудовская Аравия (Deanship of Scientific Research, King Faisal University, Kingdom of Saudia Arabia) за публикацию этой исследовательской работы в рамках гранта Nasher track Grant No. 186401.
Статья не содержит исследований с участием людей или животных.
Авторы заявляют об отсутствии конфликта интересов.
Концепция предложена и экспериментально реализована Z.I. Саузерн-блот-гибридизацию проводили Z.I. и M.N.S, они же подготовили первую рукопись. Корректура выполнена М.К. Окончательный вариант манускрипта прочитан и одобрен всеми авторами.
Список литературы
Saunders K., Stanley J. (1999) A nanovirus-like component associated with yellow vein disease of Ageratum conyzoides: evidence for interfamilial recombination between plant DNA viruses. Virology. 264, 142‒152.
Silva J.C.F., Carvalho T.F.M., Basso M.F., Deguchi M., Pereira W.A., Sobrinho R.R., Vidigal P.M.P., Brustolini O.J.B., Silva F.F., Dal-Bianco M., Fontes R.L.F., Santos A.A., Zerbini F.M., Cerqueira F.R., Fontes E.P.B. (2017) Geminivirus data warehouse: a database enriched with machine learning approaches. BMC Bioinf. 18, 240‒240.
Briddon R.W., Bull S.E., Amin I., Idris A.M., Mansoor S., Bedford I.D., Dhawan P., Rishi N., Siwatch S.S., Abdel-Salam A.M., Brown J.K., Zafar Y., Markham P.G. (2003) Diversity of DNA β, a satellite molecule associated with some monopartite begomoviruses. Virology. 312, 106‒121.
Briddon R.W., Mansoor S., Bedford I.D., Pinner M.S., Saunders K., Stanley J., Zafar Y., Malik K.A., Markham P.G. (2001) Identification of DNA components required for induction of cotton leaf curl disease. Virology. 285, 234‒243.
Iqbal Z., Sattar M.N., Kvarnheden A., Mansoor S., Briddon R.W. (2012) Effects of the mutation of selected genes of cotton leaf curl Kokhran virus on infectivity, symptoms and the maintenance of cotton leaf curl Multan betasatellite. Virus Res. 169, 107‒116.
Qazi J., Amin I., Mansoor S., Iqbal J., Briddon R.W. (2007) Contribution of the satellite encoded gene βC1 to cotton leaf curl disease symptoms. Virus Res. 128, 135‒139.
Zhou X. (2013) Advances in understanding begomovirus satellites. Ann. Rev. Phytopathol. 51, 357–381.
Saeed M. (2007) The role of geminiviral DNA β−satellite in viral pathogenicity and movement. In: School of Agriculture, Food and Wine. University of Adelaide: Urrbrae.
Amin I., Patil B.L., Briddon R.W., Mansoor S., Fauquet C.M. (2011) A common set of developmental miRNAs are upregulated in Nicotiana benthamiana by diverse begomoviruses. Virol. J. 8, 143.
Cui X., Li G., Wang D., Hu D., Zhou X. (2005) A begomovirus DNAβ-encoded protein binds DNA, functions as a suppressor of RNA silencing, and targets the cell nucleus. J. Virol. 79, 10764‒10775.
Cheng X., Wang X., Wu J., Briddon R.W., Zhou X. (2011) βC1 encoded by tomato yellow leaf curl China betasatellite forms multimeric complexes in vitro and in vivo. Virology. 409, 156‒162.
Zhang T., Luan J.B., Qi J.F., Huang C.J., Li M., Zhou X.P., Liu S.S. (2012) Begomovirus–whitefly mutualism is achieved through repression of plant defences by a virus pathogenicity factor. Mol. Ecol. 21, 1294‒1304.
Gnanasekaran P., Ponnusamy K., Chakraborty S. (2019) A geminivirus betasatellite encoded βC1 protein interacts with PsbP and subverts PsbP-mediated antiviral defence in plants. Mol. Plant Pathol. 20, 943‒960.
Kamal H., Minhas F.U.A., Farooq M., Tripathi D., Hamza M., Mustafa R., Khan M.Z., Mansoor S., Pappu H.R., Amin I. (2019) In silico prediction and validations of domains involved in Gossypium hirsutum SnRK1 protein interaction with cotton leaf curl Multan betasatellite encoded βC1. Front. Plant Sci. 10.
Nawaz-ul-Rehman M.S., Briddon R.W., Fauquet C.M. (2012) A melting pot of Old World begomoviruses and their satellites infecting a collection of Gossypium species in Pakistan. PLoS One. 7, e40050.
Tahir M.N., Amin I., Briddon R.W., Mansoor S. (2011) The merging of two dynasties ‒ identification of an African cotton leaf curl disease-associated begomovirus with cotton in Pakistan. PLoS One. 6, e20366.
Sattar M.N., Ligthart M., Kvarnheden A. (2019) Compatibility and interaction of begomoviruses and DNA-satellites causing leaf curl disease in Asia, Africa and Mediterranean Region. Eur. J. Plant Pathol. 155, 111–124.
Tiwari N., Padmalatha K., Singh V., Haq Q., Malathi V. (2010) Tomato leaf curl Bangalore virus (ToLCBV): infectivity and enhanced pathogenicity with diverse betasatellites. Arch. Virol. 155, 1343‒1347.
Hameed U., Zia-Ur-Rehman M., Herrmann H.-W., Haider M.S., Brown J.K. (2014) First report of okra enation leaf curl virus and associated cotton leaf curl Multan betasatellite and cotton leaf curl Multan alphasatellite infecting cotton in Pakistan: a new member of the cotton leaf curl disease complex. Plant Dis. 98, 1447‒1447.
Borah B., Cheema G., Gill C., Dasgupta I. (2010) A geminivirus-satellite complex is associated with leaf deformity of mentha (Mint) plants in Punjab. Indian J. Virol. 21, 103‒109.
Eini O., Rasheed M., Randles J. (2017) In situ hybri-dization and promoter analysis reveal that cotton leaf curl Multan betasatellite localizes in the phloem. Acta Virol. 61, 23‒31.
Kharazmi S., Ataie Kachoie E., Behjatnia S.A.A. (2016) Cotton leaf curl Multan betasatellite DNA as a tool to deliver and express the human B-cell lymphoma 2 (Bcl-2) gene in plants. Mol. Biotechnol. 58, 362‒372.
Saeed M. (2010) Tomato leaf curl New Delhi virus DNA A component and cotton leaf curl Multan betasatellite can cause mild transient symptoms in cotton. Acta Virol. 54, 317‒318.
Iqbal Z., Shafiq M., Ali I., Mansoor S., Briddon R.W. (2017) Maintenance of cotton leaf curl Multan betasa-tellite by tomato leaf curl New Delhi virus-analysis by mutation. Front. Plant Sci. 8, 2208.
Hamza M., Tahir M.N., Mustafa R., Kamal H., Khan M.Z., Mansoor S., Briddon R.W., Amin I. (2018) Identification of a dicot infecting mastrevirus along with alpha- and betasatellite associated with leaf curl disease of spinach (Spinacia oleracea) in Pakistan. Virus Res. 256, 174‒182.
Hanley-Bowdoin L. Settlage S.B. Orozco B.M. Nagar S. Robertson D. (2000) Geminviruses: models for plant DNA replication, transcription, and cell cycle regulation. Crit. Rev. Plant Sci. 35, 105‒140.
Nawaz-ul-Rehman M.S., Mansoor S., Briddon R.W., Fauquet C.M. (2009) Maintenance of an Old World betasatellite by a New World helper begomovirus and possible rapid adaptation of the betasatellite. J. Virol. 83, 9347‒9355.
Kharazmi S., Behjatnia S.A.A., Hamzehzarghani H., Niazi A. (2012) Cotton leaf curl Multan betasatellite as a plant gene delivery vector trans-activated by taxonomically diverse geminiviruses. Arch. Virol. 157, 1269‒1279.
Kumar J., Gunapati S., Kumar J., Kumari A., Kumar A., Tuli R., Singh S.P. (2014) Virus-induced gene silencing using a modified betasatellite: a potential candidate for functional genomics of crops. Arch. Virol. 159, 2109‒2113.
Hanley-Bowdoin L., Bejarano E.R., Robertson D., Mansoor S. (2013) Geminiviruses: masters at redirecting and reprogramming plant processes. Nat. Rev. Microbiol. 11, 777‒788.
Kumar J., Kumar J., Singh S.P., Tuli R. (2014) Association of satellites with a mastrevirus in natural infection: complexity of Wheat dwarf India virus disease. J. Virol. 88, 7093‒7104.
Fiallo-Olivé E., Tovar R., Navas-Castillo J. (2016) Deciphering the biology of deltasatellites from the New World: maintenance by New World begomoviruses and whitefly transmission. New Phytol. 212, 680‒692.
Rybicki E.P. (2009) Plant-produced vaccines: promise and reality. Drug Discov. Today. 14, 16‒24.
Chung H.Y., Lee H.H., Kim K.I., Chung H.Y., Hwang-Bo J., Park J.H., Sunter G., Kim J.B., Shon D.H., Kim W., Chung I.S. (2011) Expression of a recombinant chimeric protein of hepatitis A virus VP1-Fc using a replicating vector based on Beet curly top virus in tobacco leaves and its immunogenicity in mice. Plant Cell Rep. 30, 1513‒1521.
Baulcombe D.C., Chapman S., Santa Cruz S. (1995) Jellyfish green flourescent protein as a reporter for virus infections. Plant J. 7, 1045‒1053.
Briddon R.W., Bull S.E., Mansoor S., Amin I., Markham P.G. (2002) Universal primers for the PCR-mediated amplification of DNA β: a molecule associated with some monopartite begomoviruses. Mol. Biotechnol. 20, 315‒318.
Siemering K.R., Golbik R., Sever R., Haseloff J. (1996) Mutations that suppress the thermosensitivity of green fluorescent protein. Curr. Biol. 6, 1653‒1663.
Mansoor S., Briddon R.W., Bull S.E., Bedford I.D., Bashir A., Hussain M., Saeed M., Zafar M.Y., Malik K.A., Fauquet C., Markham P.G. (2003) Cotton leaf curl di-sease is associated with multiple monopartite begomoviruses supported by single DNA β. Arch. Virol. 148, 1969‒1986.
Hussain M., Mansoor S., Iram S., Zafar Y., Briddon R.W. (2007) The hypersensitive response to tomato leaf curl New Delhi virus nuclear shuttle protein is inhibited by transcriptional activator protein. Mol. Plant-Microbe Interact. 20, 1581‒1588.
Dellaporta S.L., Wood J., Hicks J.B. (1983) A plant DNA minipreparation: version II. Plant Mol. Biol. Rep. 1, 19‒21.
Gutierrez C. (2000) DNA replication and cell cycle in plants: learning from geminiviruses. EMBO J. 19, 792‒799.
Rushing A.E., Sunter G., Gardiner W.E., Dute R.R., Bisaro D.M. (1987) Ultrastructural aspects of tomato golden mosaic virus infection in tobacco. Phytopathology. 77, 1231‒1236.
Bass H.W., Nagar S., Hanley-Bowdoin L., Robertson D. (2000) Chromosome condensation induced by geminivirus infection of mature plant cells. J. Cell Sci. 113, 1149‒1160.
Morilla G., Krenz B., Jeske H., Bejarano E.R., Wege C. (2004) Tête à tête of tomato yellow leaf curl virus and tomato yellow leaf curl Sardinia virus in single nuclei. J. Virol. 78, 10715‒10723.
Rojas M.R., Jiang H., Salati R., Xoconostle-Cázares B., Sudarshana M.R., Lucas W.J., Gilbertson R.L. (2001) Functional analysis of proteins involved in movement of the monopartite begomovirus, tomato yellow leaf curl virus. Virology. 291, 110‒125.
Morra M.R., Petty I.T.D. (2000) Tissue specificity of geminivirus infection is genetically determined. Plant Cell. 12, 2259‒2270.
Wege C., Saunders K., Stanley J., Jeske H. (2001) Comparative analysis of the tissue tropism of bipartite geminiviruses. J. Phytopathol. 149, 359‒368.
Poornima Priyadarshini C.G., Ambika M.V., Tippeswamy R., Savithri H.S. (2011) Functional characterization of coat protein and V2 involved in cell to cell movement of cotton leaf curl Kokhran virus-Dabawali. PLoS One. 6, e26929.
Chowda-Reddy R.V., Achenjang F., Felton C., Etarock M.T., Anangfac M.-T., Nugent P., Fondong V.N. (2008) Role of a geminivirus AV2 protein putative protein kinase C motif on subcellular localization and pathogenicity. Virus Res. 135, 115‒124.
Teng K., Chen H., Lai J., Zhang Z., Fang Y., Xia R., Zhou X., Guo H., Xie Q. (2010) Involvement of C4 protein of beet severe curly top virus (family Geminiviridae) in virus movement. PLoS One. 5, e11280.
Hayes R.J., Petty I.T.D., Coutts R.H.A., Buck K.W. (1988) Gene amplification and expression in plants by a replicating geminivirus vector. Nature. 334, 179‒182.
Scholthof H.B., Scholthof K.-B.G., Jackson A.O. (1996) Plant virus gene vectors for transient expression of foreign proteins in plants. Ann. Rev. Phytopathol. 34, 299‒323.
Peretz Y., Mozes-Koch R., Akad F., Tanne E., Czosnek H., Sela I. (2007) A universal expression/silencing vector in plants. Plant Physiol. 145, 1251‒1263.
Kachoie A.E. Behjatnia S.A.A., Kharazmi S. (2018) Expression of human immunodeficiency virus type 1 (HIV-1) coat protein genes in plants using cotton leaf curl Multan betasatellite-based vector. PLoS One. 13, e0190403.
Pakniat-Jahromy A., Behjatnia S.A.A., Dry I.B., Izadpanah K., Rezaian M.A. (2010) A new strategy for generating geminivirus resistant plants using a DNA betasatellite/split barnase construct. J. Virol. Methods. 170, 57‒66.
Kumar P.P., Usha R., Zrachya A., Levy Y., Spanov H., Gafni Y. (2006) Protein-protein interactions and nuclear trafficking of coat protein and βC1 protein associated with Bhendi yellow vein mosaic disease. Virus Res. 122, 127‒136.
Saeed M., Y. Zafar Y., Randles J.W., Rezaian M.A. (2007) A monopartite begomovirus-associated DNA β satellite substitutes for the DNA B of a bipartite begomovirus to permit systemic infection. J. Gen. Virol. 88, 2881‒2889.
Sharma P., Ikegami M. (2010) Identification of the virulence factors and suppressors of posttranscriptional gene silencing encoded by Ageratum yellow vein virus, a monopartite begomovirus. Virus Res. 149, 19‒27.
Zhang S.C., Wege C., Jeske H. (2001) Movement proteins (BC1 and BV1) of abutilon mosaic geminivirus are cotransported in and between cells of sink but not of source leaves as detected by green flourescent protein tagging. Virology. 290, 249‒260.
Tao X., Qing L., Zhou X. (2004) Function of A-rich region in DNAβ associated with tomato yellow leaf curl China virus. Chin. Sci. Bull. 49, 1490‒1493.
Etessami P., Watts J., Stanley J. (1989) Size reversion of african cassava mosaic virus coat protein gene deletion mutants during infection of Nicotiana benthamiana. J. Gen. Virol. 70, 277‒289.
Shen W.-H., Hohn B. (1991) Mutational analysis of the small intergenic region of maize streak virus. Virology. 183, 721‒730.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология