Молекулярная биология, 2021, T. 55, № 1, стр. 75-85
Молекулярный полиморфизм генов β-галактозидазы LAC4 у молочных и природных штаммов дрожжей Kluyveromyces
Л. В. Лютова a, b, Г. И. Наумов a, А. В. Шнырева b, Е. С. Наумова a, *
a Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117545 Москва, Россия
b Биологический факультет Московского государственного университета им. М.В. Ломоносова
119234 Москва, Россия
* E-mail: lena_naumova@yahoo.com
Поступила в редакцию 17.06.2020
После доработки 28.07.2020
Принята к публикации 04.08.2020
Аннотация
Характерной особенностью молочных дрожжей Kluyveromyces lactis является способность ферментировать лактозу, тогда как подавляющее большинство остальных видов дрожжей не способны даже ассимилировать этот дисахарид. Молекулярный полиморфизм генов LAC4, кодирующих β-галактозидазу, контролирующую ферментацию лактозы, практически не изучен, а опубликованные данные касаются только одного штамма K. lactis var. lactis – NRRL Y-1140, выделенного из сливок в США. С помощью молекулярного кариотипирования, Саузерн-гибридизации и секвенирования мы изучили гены β-галактозидазы у сбраживающих лактозу штаммов K. lactis, выделенных из молочных продуктов и природных источников в разных регионах мира. Установлено, что у молочных дрожжей K. lactis var. lactis способность ферментировать лактозу контролируется по крайней мере тремя полимерными локусами LAC различной хромосомной локализации: LAC1 (хромосома III), LAC2 (II) и LAC3 (IV). Большинство изученных нами штаммов обладали локусом LAC2. Впервые проведен сравнительный анализ β-галактозидаз дрожжей рода Kluyveromyces и этих ферментов из других дрожжей. Филогенетический анализ выявил значительные различия между белками LAC4 дрожжей рода Kluyveromyces (K. lactis, K. marxianus, К. aestuarii, K. nonfermentans, K. wickerhamii), Scheffersomyces stipitis, Sugiyamaella lignohabitans и Debaryomyces hansenii. Обнаружена корреляция между последовательностями β-галактозидаз и экологическим происхождением штаммов Kluyveromyces: молочные продукты и природные источники. Группа молочных штаммов гетерогенна и включает дрожжи K. lactis var. lactis и K. marxianus (99.80–100% сходства), что указывает на общее происхождение их генов LAC4. Филогенетический анализ β-галактозидаз указывает на близкое генетическое родство молочных и госпитальных штаммов K. lactis var. lactis и K. marxianus. Клинические изоляты способны сбраживать лактозу и, по-видимому, происходят от молочных дрожжей.
ВВЕДЕНИЕ
Наряду с культурными растениями и домашними животными человечество на протяжении многих тысячелетий использует микроорганизмы, прежде всего дрожжи-сахаромицеты, в хлебопечении, виноделии, пивоварении, производстве спирта. К “одомашненным” микроорганизмам можно отнести и молочные дрожжи Kluyveromyces, которые часто выделяются из различных молочных продуктов (сыр, молоко, кисломолочные продукты) и придают им приятный аромат и вкус.
Гидролиз дисахарида лактозы до галактозы и глюкозы осуществляется при участии фермента β-галактозидазы [К.Ф 3.2.1.23]. У дрожжей Kluyveromyces лактоза гидролизуется внутриклеточной β-галактозидазой, поэтому для транспорта сахара в клетку необходимы соответствующие пермеазы. Следует отметить, что известно более 700 видов дрожжей-аскомицетов, из которых лактозу способны утилизировать только около 1%. Помимо дрожжей Kluyveromyces, этим свойством обладают только некоторые виды Candida, Debaryomyces и Scheffersomyces.
В настоящее время род Kluyveromyces включает семь видов: наземные K. lactis, K. marxianus, K. dobzhanskii и K. wickerhamii, а также морские K. aestuarii, K. nonfermentas и K. siamensis [1, 2]. В свою очередь, вид K. lactis имеет сложный состав и включает две разновидности: молочные дрожжи K. lactis var. lactis и не сбраживающие лактозу природные изоляты K. lactis var. drosophilarum. Виды рода Kluyveromyces существенно различаются по способности утилизировать лактозу. Дрожжи K. lactis var. lactis и молочные штаммы K. marxianus способны ферментировать лактозу, тогда как К. aestuarii, K. siamensis, K. nonfermentans, K. wickerhamii и природные изоляты K. marxianus ассимилируют лактозу, но не способны ее ферментировать из-за зависимого от дыхания низкоаффинного транспорта лактозы [3–5]. Дрожжи K. dobzhanskii и K. lactis var. drosophilarum не утилизируют лактозу и не имеют даже молчащих генов LAC [6]. Система генов ферментации лактозы хорошо изучена на одном штамме дрожжей K. lactis var. lactis NRRL Y-1140, который является родоначальником генетических линий [7–11]. У этого штамма идентифицированы тесно сцепленные гены LAC4 и LAC12, кодирующие, соответственно, β-галактозидазу и пермеазу лактозы, а также регуляторные гены. Ранее при скрещивании штаммов NRRL Y-1118 и NRRL Y-1140 (оба выделены из сливок в США) обнаружили полимерные лактозные локусы LAC1 и LAC2 [12]. У гибридов этих штаммов в мейозе наблюдалось дигенное полимерное расщепление по способности ферментировать лактозу. Сцепление генов LAC4 и LAC12 в локусах LAC1 и LAC2 позднее установили путем тетрадного анализа гибридов штаммов NRRL Y-1118 (LAC1) и NRRL Y-1140 (LAC2) с природными штаммами K. lactis var. drosophillarum, у которых нет этих генов [4]. Эти локусы имеют различную хромосомную локализацию: LAC1 (хромосома II) и LAC2 (хромосома III) [4, 13]. Третий полимерный локус LAC3, расположенный в хромосоме IV, обнаружен нами у штаммов K. lactis var. lactis, выделенных из молочных продуктов в Советском Союзе [14]. Полиморфизм генов β-галактозидазы дрожжей K. lactis ранее не изучали. В базе данных GenBank представлены нуклеотидные последовательности гена LAC4 только трех штаммов K. lactis var. lactis: NRRL Y-1140 (выделен из сливок в США), F61 (молоко) и GG799 (пищевая промышленность, США). Все эти штаммы содержат локус LAC2.
В настоящей работе молекулярно-генетическое изучение генов β-галактозидазы LAC4 дрожжей Kluyveromyces проведено на материале штаммов различного происхождения.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Объекты исследования. Используемые в работе штаммы KLuyveromyces приведены в табл. 1. Дрожжи культивировали при 28°С на полной среде YPD, г/л: бактоагар – 20, глюкоза – 20, дрожжевой экстракт – 10 и пептон – 20.
Таблица 1.
Происхождение штаммов дрожжей Kluyveromyces
| Штамма и его номер | Источник и место выделения | Локус LAC |
|---|---|---|
| Kluyveromyces lactis var. lactis | ||
| CBS 683 (T) | Мягкий сыр, Великобритания | LAC2 |
| NRRL Y-1140 | Сливки, США | То же |
| SM 3.8 | Сыр Камамбер, Франция | » |
| SM 5.8 | То же | » |
| SM 6.7 | » | » |
| SM 16.9 | » | » |
| SM 48.7 | » | » |
| CBS 762 | Сливки, США | » |
| CBS 845 | Молоко, Великобритания | » |
| CBS 1065 | Молоко | » |
| CBS 1797 | Мокрота, Норвегия | » |
| CBS 2360 | Молоко | » |
| CBS 2619 | Сливки, США | » |
| CBS 5618 | Мокрота, Норвегия | » |
| CBS 8043 | Кишечник ребенка, Новая Зеландия | » |
| ВКПМ Y-3737 | Почва, Измайловский парк, Москва, Россия | » |
| H1 | Почва, Измайловский парк, Москва, Россия | » |
| H2 | Почва, Измайловский парк, Москва, Россия | » |
| H3 | Почва, Измайловский парк, Москва, Россия | » |
| GG799 | Пищевая промышленность, США | » |
| F61 | Молоко | » |
| NRRL Y-1118 | Сливки, США | LAC1 |
| CBS 1067 | Молоко, Нидерланды | То же |
| ВКМ Y-762 | Сливки, США | » |
| ВКМ Y-869 | Кислое молоко, Кольский полуостров, Россия | » |
| ВКМ Y-870 | Чал, Туркмения | » |
| ВКМ Y-896 | Мягкий сыр, Италия | » |
| ВКМ Y-1527 | Мокрота, Испания | » |
| ВКМ Y-1186 | Молоко, Киев, Украина | LAC3 |
| ВКМ Y-1333 | Кислое молоко, Ставропольский край, Россия | То же |
| ВКМ Y-1339 | Сметана, Санкт-Петербург, Россия | » |
| ВКМ Y-1343 | Молоко, Гомельская обл., Беларусь | » |
| ВКМ Y-1868 | Чал, Туркмения | LAC1/LAC3 |
| UCM Y-328 | Кефир, Киев, Украина | LAC1/LAC2 |
| ВКПМ Y-492 | Молочная сыворотка, Украина | LAC1/LAC2 |
| Kluyveromyces marxianus | ||
| CBS 397 | Йогурт, Нидерланды | LAC |
| B0399 | Творог, Италия | То же |
| UFV-3 | Молоко, Бразилия | » |
| L03 | Молочный продукт | » |
| 100656-19 | Кровь человека, Нидерланды | » |
| NBRC 1777 | Почва, Япония | » |
| CBS 6556 | Ферментированное кукурузное тесто, Мексика | » |
| DMKU3-1042 | Почва, Тайланд | » |
| FIM1 | Спина коровы, Нидерланды | » |
| DMB1 | Гидролизат сахарного тростника | » |
| UFS-Y2791 | Сок Agave americana, Южная Африка | » |
| Kluyveromyces dobzhanskii | ||
| CBS 2104 (T) | Drosophila pseudoobscura, Калифорния, США | » |
| Kluyveromyces wickerhamii | ||
| CBS 2745 (T) | Drosophila sp., Калифорния, США | » |
| Kluyveromyces aestuarii | ||
| CBS 4438 (T) | Морская грязь, Флорида, США | » |
| Kluyveromyces nonfermentans | ||
| CBS 8778 (T) | Ил, залив Сагами, Япония | » |
| Kluyveromyces siamensis | ||
| CBS 10860 (T) | Вода мангрового леса, Тайланд | » |
| Scheffersomyces stipitis | ||
| CBS 6054 | Неизвестно | » |
| Sugiyamaella lignohabitans | ||
| CBS 10342 (T) | Гнилое бревно, Иллинойс, США | » |
| Debaryomyces hansenii | ||
| CBS 767 (T) | Неизвестно | » |
Примечание. Сокращенные названия коллекций: ВКМ – Всероссийская коллекция микроорганизмов, Пущино, Москва; ВКПМ – Всероссийская коллекция промышленных микроорганизмов, Москва, Россия; CBS – The Westerdijk Fungal Biodiversity Institute, Утрехт, Нидерланды; SM – J.P. Schmidt, Institut National Agronomique, Париж-Гриньон, Франция; NRRL – USDA-ARS Culture Collection, National Center for Agricultural Utilization Research, Пеория, США; UCM – Украинская коллекция микроорганизмов, Институт микробиологии и вирусологии НАН, Киев, Украина; NBRC/IFO – National Institute of Technology and Evaluation, Токио, Япония; UCDFST – Phaff Yeast Collection, University of California, Дэвис, США. Соответствие штаммов различных коллекций: CBS 683=ВКМ Y-868, NRRL Y-1140 = CBS 2359, NRRL Y-1118 = CBS 6315, CBS 6556 = KCTC 17555. FIM1 = CBS 4857, CBS 2745 = UCDFST 54-210, CBS 4438 = NRRL YB-4510, CBS 8778 = NRRL Y-27343. T – типовая культура.
ПЦР. Дрожжевую ДНК выделяли при помощи набора Genomic DNA Purification Kit (“Fermentas”, Литва). Дизайн олигонуклеотидных праймеров для амплификации и секвенирования генов LAC4 осуществляли онлайн на сайте https:// www.yeastgenome.org. Для амплификации генов LAC4 использовали праймеры MR11 (5'-ATGTCTTGCCTTATTCCTG-3') и MR14 (5'-GATCTCGCTTTTGAATAA-3'). ПЦР проводили в 30 мкл буфера, содержащего 2.5 мМ MgCl2, 0.1 мМ каждого dNTP, 50 пмоль каждого праймера, 2.5 ед. акт Taq-полимеразы (“Helicon”, Россия), 20–200 нг ДНК по следующей схеме: начальная денатурация при 94°С в течение 3 мин; затем 30 циклов в режиме: денатурация при 94°С – 30 с, отжиг праймеров при 56°С – 30 с, синтез ДНК при 72°С – 60 с; конечная достройка при 72°С – 10 мин. Продукты амплификации разделяли путем электрофореза в 1%-ном агарозном геле при 60–65 В в 0.5 × TBE-буфере (45 мМ Трис-НСl рН 8.0, 10 мМ EDTA, 45 мМ борная кислота) в течение 1–1.5 ч. Гель окрашивали бромистым этидием, промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете на трансиллюминаторе Vilber Lourmat (Франция). В качестве маркера молекулярных масс использовали препарат “1 kb DNA Ladder” (“Thermo Fisher”, США).
Определение нуклеотидных последовательностей и филогенетический анализ. Амплифицированныe фрагменты генов LAC4 элюировали из геля при помощи набора Cleanup Mini (“Евроген”, Москва) согласно протоколу фирмы-изготовителя. Нуклеотидные последовательности генов LAC4 определяли по двум цепям при помощи двух пар праймеров: MR11/MR14 и MR12 (5'-CGAAGCTTGTTATGGCAG-3')/MR13 (5'-CGCGTACTTAGACAGAGC-3') с помощью прямого секвенирования по методу Сенгера на автоматическом секвенаторе “Applied Biosystems 3730” (США). Последовательности анализировали, используя программу SeqMan package (“DNA Star Inc.”, США).
Поиск гомологии с известными нуклеотидными последовательностями генов LAC4 проводили с помощью программы BLAST в базе данных GenBank. Множественные выравнивания изученных нуклеотидных и аминокислотных последовательностей проводили, используя программу BioEdit (http://www.mbio.ncsu.edu/BioEdit/bioedit.html). Филогенетические деревья строили методом объединения соседей (Neighbour-Joining) в программе MEGA 7 [15].
Пульс-электрофорез хромосомных ДНК (молекулярное кариотипирование) и Саузерн-гибридизация. Условия приготовления препаратов хромосомной ДНК описаны ранее [16]. Электрофоретическое разделение хромосомных ДНК проводили на аппарате CHEF-DR III фирмы “Bio-Rad” (США) в 0.8%-ном агарозном геле с использованием трехступенчатого режима: 1) 175 В, 8 ч, время переключения полей 40–120 с; 2) 130 В, 24 ч, время переключения полей 120–360 с; 3) 100 В, 8 ч, время переключения полей 360–1200 с. В качестве буфера использовали 0.5 × TBE, охлажденный до 14°С. В качестве кариотипических стандартов использовали коммерческие препараты ДНК штаммов Saccharomyces cerevisiae YNN 295 (=ATCC 20358) и Wickerhamomyces canadensis (syn. Hasenula wingei) YB-4662-VIA (=ATCC 28162) (“Bio-Rad”, США). После электрофореза гель окрашивали бромистым этидием, промывали в дистиллированной воде и фотографировали в ультрафиолетовом свете.
Хромосомную ДНК переносили на нитроцеллюлозную мембрану, используя аппарат Vacuum blotter (“BioRad”). ДНК фиксировали на мембране путем отжига при 80°С в течение 2 ч. В качестве зонда использовали ПЦР-амплифицированный фрагмент гена LAC4 штамма K. lactis var. lactis NRRL Y-1140. Метку вводили нерадиоактивным методом с использованием дигоксигенина (dig-II-dUTP) из набора “DIG High Prime DNA Labeling Detection Starter Kit I” (“Roche”, Швейцария). Гибридизацию и детекцию гибридизационных сигналов проводили по протоколу фирмы-изготовителя.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Молекулярное кариотипирование и Саузерн-гибридизация
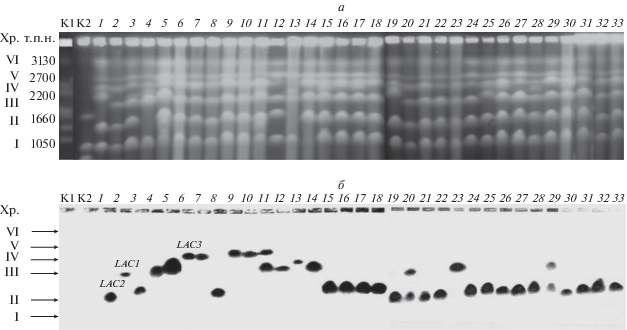
С помощью пульс-электрофореза и Саузерн-гибридизации хромосомной ДНК с зондом LAC4 мы провели молекулярный скрининг генов β-галактозидазы у 33 сбраживающих лактозу штаммов дрожжей K. lactis var. lactis различного экологического и географического происхождения (табл. 1). Штаммы выделены в различных регионах бывшего СССР, Европы, США и Новой Зеландии из молочных продуктов, почвы и клинических источников (мокрота, кишечник). На рис. 1а приведены кариотипические профили 33 изученных штаммов. Размеры отдельных хромосом определяли по кариотипам стандартных штаммов W. canadensis YB-4662-VIA и S. cerevisiae YNN 295 (рис. 1а, дорожки 1 и 2). Изученные штаммы характеризуются сходными молекулярными кариотипами с шестью хромосомными полосами размером от 1050 до 2800 т.п.н. (рис. 1а). Отмечен некоторый полиморфизм размеров хромосом II и III. Гибридизация с зондом LAC4 выявила локус LAC1 у семи штаммов, LAC2 – у 19 и LAC3 у четырех (рис. 1б). У штаммов UCM Y-328 и ВКПМ Y-492, выделенных из молочных продуктов на Украине, обнаружены полимерные локусы LAC1 и LAC2 (рис. 1б, дорожки 20 и 29). Выделенный из чала в Туркмении штамм ВКМ Y-1868 имеет полимерные гены LAC1 и LAC3 (рис. 1б, дорожка 11). Нами не отмечена корреляция между происхождением штаммов и присутствием определенных локусов LAC (табл. 1). Например, клинические изоляты CBS 5618 и CBS 8043, выделенные, соответственно, из мокроты в Норвегии и кишечника ребенка в Новой Зеландии, содержат локус LAC2 (табл. 1). Также европейский штамм ВКМ Y-1527 (мокрота, Испания) содержит локус LAC1. Независимо от источника выделения (молочные продукты, почва и клинические изоляты) большинство изученных штаммов обладали локусом LAC2 (табл. 1).
Рис. 1.
Пульс-электрофорез хромосомных ДНК дрожжей K. lactis var. lactis (а) и Саузерн-гибридизация с зондом LAC4 (б). Дорожки: K1 – W. canadensis (хромосомный стандарт); K2 – S. cerevisiae YNN 295 (хромосомный стандарт); K. lactis var. lactis: 1 – NRRL Y-1140; 2 –NRRL Y-1118; 3 – CBS 683; 4 – ВКМ Y-869; 5 – ВКМ Y-870, 6 – ВКМ Y-1186; 7 – ВКМ Y-1333; 8 – CBS 762; 9 – ВКМ Y-1339; 10 – ВКМ Y-1343; 11 – ВКМ Y-1868; 12 – ВКМ Y-762; 13 – ВКМ Y-896; 14 – ВКМ Y-1527; 15 – SM 3.8; 16 – SM 5.8; 17 – SM 6.7; 18 – SM 16.9; 19 – SM 48.7; 20 – UCM Y-328; 21 – CBS 845; 22 – CBS 1065; 23 – CBS 1067; 24 – CBS 1797; 25 – CBS 2360; 26 – CBS 2619; 27 – CBS 5618; 28 – CBS 8043; 29 – ВКПМ Y-492; 30 – ВКПМ Y-3737; 31 – H1; 32 – H2; 33 – H3. Размеры хромосом (т.п.н.) приведены по кариотипическим стандартам S. cerevisiae YNN 235 и W. canadensis YB-4662-VIA. Хр. – хромосома.
Нуклеотидный полиморфизм генов LAC4 дрожжей Kluyveromyces
Мы провели секвенирование генов LAC4 различной хромосомной локализации у 11 штаммов K. lactis var. lactis: локус LAC1 (NRRL Y-1118, ВКМ Y-869, ВКМ Y-870 и ВКМ Y-1527), LAC2 (CBS 683, ВКПМ Y-3737 и SM 48.7) и LAC3 (ВКМ Y-1186, ВКМ Y-1333, ВКМ Y-1339 и ВКМ Y-1343) (табл. 1). В анализ вошли штаммы, выделенные из молочных продуктов, почвы и в клиниках. Полученные нуклеотидные последовательности сравнили между собой и с последовательностями генов LAC4 локуса LAC2 штаммов NRRL Y-1140, F61 и GG799, депонированными в базу данных GenBank. Последовательности генов LAC4 у штаммов, обладающих локусом LAC1, были идентичными или отличались 1–2 нуклеотидами. У штаммов с локусом LAC2 обнаружено от 0 до 3 замен в нуклеотидных последовательностях генов LAC4. Все четыре изученных нами штамма с локусом LAC3 имели идентичные последовательности генов β-галактозидазы. Гены LAC4 локусов LAC1, LAC2 и LAC3 различались 1–5 нуклеотидами, наибольшее количество замен выявлено в гене β-галактозидазы локуса LAC3. Анализ спектра нуклеотидных замен показал, что наиболее часто встречаются транзиции типа C → T, большинство из которых представлены расположенными в третьем положении кодона молчащими заменами, не вызывающими изменений в аминокислотной последовательности кодируемого белка. Мы также секвенировали ген LAC4 молочного штамма K. marxianus CBS 397, выделенного из йогурта в Нидерландах. Сравнительный анализ нуклеотидных последовательностей генов LAC4 дрожжей K. lactis var. lactis и K. marxianus CBS 397 выявил их большое сходство и всего 1–3 нуклеотидные замены.
В базе данных GenBank представлены нуклеотидные последовательности геномов типовых культур К. aestuarii, K. nonfermentans и K. wickerhamii, а также 10 штаммов K. marxianus, выделенных из молочных продуктов и различных природных источников (табл. 1). Нуклеотидные последовательности генов β-галактозидазы штаммов K. marxianus молочного происхождения (B0399, UFV-3 и L03) и клинического изолята 100656-19, выделенного из крови человека, были идентичными и не отличались от последовательности штамма CBS 397. С другой стороны, сходство нуклеотидных последовательностей генов LAC4 природных штаммов K. marxianus составило 92.88–98.34%. Последовательности генов LAC4 молочных и природных штаммов K. marxianus различались более 60 заменами. В целом, нуклеотидные последовательности генов β-галактозидаз дрожжей K. lactis var. lactis и K. marxianus сходны на 89.94–99.97%, тогда как уровень их сходства с генами LAC4 типовых культур K. wickerhamii CBS 2745, К. aestuarii CBS 4438 и K. nonfermentans CBS 8778 не превышал 70%.
Сравнительный анализ аминокислотных последовательностей β-галактозидаз дрожжей Kluyveromyces
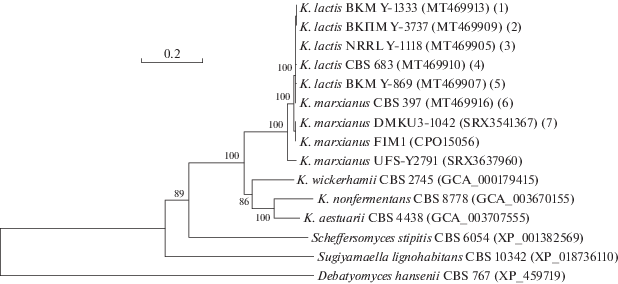
По полученным нуклеотидным последовательностям генов LAC4 были определены первичные структуры соответствующих белков, состоящих из 1025 аминокислотных остатков. В сравнительный анализ были также включены β-галактозидазы дрожжей Scheffersomyces stipitis CBS 6054, Sugiyamaella lignohabitans CBS 10342 и Debaryomyces hansenii CBS 767. На основании анализа изученных аминокислотных последовательностей построено филогенетическое дерево (рис. 2). β-Галактозидазы дрожжей рода Kluyveromyces сформировали отдельный кластер со 100%-ной статистической поддержкой. Внутри этого кластера выделяют два основных подкластера. Первый представлен аминокислотными последовательностями LAC4 штаммов K. lactis var. lactis и K. marxianus, идентичными на 94.00–100%. Этот подкластер включает две группы штаммов: молочные дрожжи K. lactis var. lactis и K. marxianus (99.80–100% сходства) и природные изоляты, β-галактозидазы которых идентичны на 94.25–98.34%. Наиболее дивергентной является β-галактозидаза штамма K. marxianus UFS-Y2791, выделенного из Agave americana в Южной Африке. Второй подкластер включает белки LAC4 дрожжей K. wickerhamii, K. aestuarii и K. nonfermentans. Наибольшее сходство имеют β-галактозидазы K. aestuarii и K. nonfermentans (79.98%), идентичные белку LAC4 K. wickerhamii на 72.77 и 69.60% соответственно. Сходство β-галактозидаз двух подкластеров составило 63.67–70.37%. Отдельное положение на дереве занимают лактазы других родов дрожжей: Scheffersomyces, Sugiyamaella и Debaryomyces, сходство которых с β-галактозидазами дрожжей Kluyveromyces не превышало 45%.
Рис. 2.
Филогенетическое дерево аминокислотных последовательностей β-галактозидаз дрожжей рода Kluyveromyces. В качестве внешней группы использовали β-галактозидазы дрожжей Scheffersomyces stipitis CBS 6054, Sugiyamaella lignohabitans CBS 10342, Debaryomyces hansenii CBS 767. Приведены значения бутстрепа >70%. Шкала соответствует 20 заменам на 100 аминокислотных позиций. Цифрами в скобках обозначены группы штаммов с идентичными аминокислотными последовательностями: (1) –ВКМ Y-1186 (MT469912), ВКМ Y-1339 (MT469915), ВКМ Y-1343 (MT469914); (2) – SM 48.7 (MT469911); (3) – NRRL Y-1140 (XP_452194.1), ВКМ Y-1527-7A (MT469908), F61 (KF420203.1); (4) – GG799; (5) – ВКМ Y-870 (MT469906); (6) – B0399 (CM004407.1), 100656-19 (CABJCX010000079.1), UFV-3 (SRX3637959), L03 (SRX3541362); (7) – DMB1 (BBIL00000000.1), NBRC 1777 (AP014601.1), CBS 6556 (KQ039400.1). После номера штамма приведены регистрационные номера последовательностей в GenBank.
Филогенетический анализ нуклеотидных последовательностей домена D1/D2 и ITS-участка рДНК
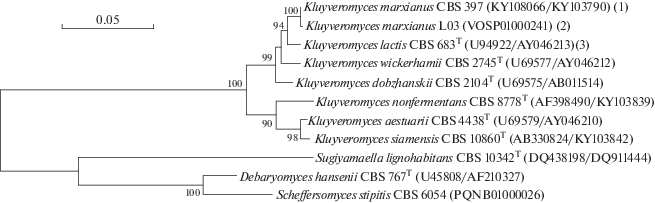
Современная классификация дрожжей-аскомицетов основана на филогенетическом анализе ряда молекулярных маркеров, прежде всего домена D1/D2 гена 26S рРНК и 5.8S-ITS-фрагмента, включающего ген 5.8S РНК и внутренние транскрибируемые спейсеры ITS1/ITS2. На основании депонированных в GenBank нуклеотидных последовательностей домена D1/D2 и ITS-участка дрожжей Kluyveromyces построено филогенетическое дерево (рис. 3). Дрожжи Kluyveromyces со 100%-ной статистической значимостью сформировали отдельный кластер, который, в свою очередь, разделен на два подкластера. Первый включает сухопутные виды K. lactis, K. marxianus, K. dobzhanskii и K. wickerhamii, а второй – морские виды K. aestuarii, K. nonfermentas и K. siamensis. Дрожжи K. lactis и K. marxianus имеют практически идентичные домены D1/D2, но значимо различаются последовательностями ITS1-участка: 23 нуклеотидные замены. Штаммы K. lactis var. lactis, молочные (CBS 683 и NRRL Y-1140) и не молочные (GG799), имели идентичные последовательности D1/D2 и ITS. Штаммы K. marxianus разделились на две группы, ITS1 которых различались двумя нуклеотидными заменами. Первая группа включает молочные штаммы (CBS 397 и B0399), клинический изолят 100656-19 и штаммы не молочного происхождения (NBRC 1777, CBS 6556, DMKU3-1042, FIM1 и DMB1). Два молочных штамма LO3 и UFV-3 составили вторую группу. Деление на группы не связано с географическим происхождением штаммов и способностью ферментировать лактозу.
Рис. 3.
Филогенетическое дерево нуклеотидных последовательностей домена D1/D2 гена 26S рРНК и участка ITS1-5.8S-ITS2 дрожжей Kluyveromyces. В качестве внешней группы использовали последовательности дрожжей Scheffersomyces stipitis CBS 6054, Sugiyamaella lignohabitans CBS 10342 (T), Debaryomyces hansenii CBS 767 (T). Приведены значения бутстрепа >70%. Шкала соответствует 50 нуклеотидным заменам на 1000 позиций. Цифрами в скобках обозначены группы штаммов, имеющие идентичные последовательности: (1) – CBS 712T (U94924/AY046214), B0399 (CM004409), 100656-19 (CABJCX010000020), NBRC 1777 (AB771427/AB771426), CBS 6556 (KY108061/KY103826), DMKU3-1042 (AP012217), FIM1 (KY108083/KY103823), DMB1 (GCA_000747785); (2) – UFV-3 (CP009307); (3) – NRRL Y-1140 (KY108038/ KY103771), GG799 (KY103771/CP021242); После номера штамма приводены регистрационные номера последовательностей в GenBank. T – типовая культура.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
На материале штаммов K. lactis var. lactis различного происхождения впервые проведен молекулярный скрининг генов LAC4, контролирующих ферментацию лактозы. У этих дрожжей способность ферментировать лактозу контролируется по крайней мере тремя полимерными локусами LAC различной хромосомной локализации: LAC1 (хромосома III), LAC2 (хромосома II) и LAC3 (хромосома IV).
В целом, топологии деревьев, построенных на основании аминокислотных последовательностей β-галактозидаз и нуклеотидных последовательностей домена D1/D2 и ITS-участка рДНК хорошо согласуются: со 100%-ной статистической значимостью выделяются кластеры, объединяющие виды рода Kluyveromyces, а молочные дрожжи K. lactis и K. marxianus являются наиболее близкородственными (рис. 2 и 3). В то же время имеются некоторые различия, связанные, по-видимому, с отсутствием видов K. dobzhanskii и K. siamensis на дереве, построенном по аминокислотным последовательностям β-галактозидаз. Так, на дереве рибосомных последовательностей наземные и морские виды Kluyveromyces разделены на два четких подкластера, тогда как на втором дереве β-галактозидаза наземных дрожжей K. wickerhamii примыкает к β-галактозидазам морских видов К. aestuarii и K. nonfermentans.
Филогенетический анализ выявил значительные различия между белками LAC4 дрожжей рода Kluyveromyces (K. lactis, K. marxianus, К. aestuarii, K. nonfermentans, K. wickerhamii), Scheffersomyces stipitis, Sugiyamaella lignohabitans и Debaryomyces hansenii. β-Галактозидазы Kluyveromyces идентичны на 63.67–100%. Наибольшим сходством обладают β-галактозидазы штаммов K. lactis var. lactis и K. marxianus: 94.00–100%. С другой стороны, белки LAC4 дрожжей K. marxianus разделились на две группы, соответствующие происхождению штаммов: молочные продукты и природные источники. Следует отметить близкое генетическое родство молочных и госпитальных штаммов K. lactis var. lactis и K. marxianus. Ранее мы показали, что клинические изоляты K. lactis var. lactis по многим молекулярным маркерам не отличаются от штаммов, выделенных из молочных продуктов [16]. Принимая по внимание способность клинических штаммов сбраживать лактозу, они, очевидно, происходят от молочных дрожжей. Ряд характеристик, свойственных патогенным дрожжам, уже присущи госпитальным штаммам K. marxianus: образование псевдомицелия, устойчивость к повышенной температуре и высокая пектолитическая активность [17]. Обнаружены также клинические изоляты S. cerevisiae, по многим молекулярным маркерам сходные с пекарскими штаммами [18–21].
С помощью комплементационного анализа при гибридизации с lac-тестерами K. lactis var. lactis ранее установлено, что молочные, клинические и природные штаммы K. marxianus обладают активными генами LAC4, но имеют различные типы пермеаз лактозы [3]. На специальной среде с ингибитором дыхания антимицином А показано, что молочные и клинические штаммы содержат не зависящую от дыхания сильную пермеазу лактозы, тогда как природные изоляты характеризуются зависящей от дыхания слабой пермеазной активностью. Феномен ассимиляции сахаров дрожжами в аэробных условиях и неспособность их сбраживать в анаэробных условиях получил название эффекта Клюйвера [22, 23]. С помощью антимицина А установлена зависимость многих ферментов K. lactis от дыхания, в том числе и пермеазы глюкозы [24]. Низкой активностью пермеазы лактозы обладают также дрожжи K. wickerhamii, способные только ассимилировать лактозу [25]. В то же время эти дрожжи имеют активный ген LAC4. Генетические данные очень хорошо согласуются с результатами недавно проведенного сравнительного анализа пермеазных генов LAC12 у штаммов K. marxianus различного происхождения [5, 26]. На основании геномного анализа штаммов Kluyveromyces, депонированных в GenBank, реконструирована история эволюции генов утилизации дисахаридов – лактозы и целлобиозы. Показано, что предковый белок LAC12 был бифункциональным и участвовал в транспорте обоих дисахаридов внутрь клетки, затем LAC4 гидролизовал лактозу, а CEL2 – целлобиозу. В процессе эволюции дрожжи K. marxianus и K. nonfermentans утратили собственно транспортер целлобиозы CEL1, который сохранился у остальных видов рода Kluyveromyces. У дрожжей K. marxianus вместе с потерей CEL1 произошла дупликация гена LAC12, в результате которой образовались четыре копии, локализованные в субтеломерных районах различных хромосом: 8 и 2 и оба плеча хромосомы 3. Однако только расположенный в левом плече хромосомы 3 предковый ген LAC12 кодирует функциональную пермеазу лактозы, а остальные копии участвуют в транспорте другого дисахарида – целлобиозы [5]. У неспособных ферментировать лактозу природных штаммов K. marxianus белок LAC12 отличается 13 заменами от соответствующего белка молочных и госпитальных штаммов [26]. Независимо от источника выделения, все изученные штаммы K. marxianus обладали функциональным геном LAC4 (хромосома 3L), тогда как остальные копии (хромосомы 8, 2 и правое плечо хромосомы 3) были утрачены или вырождены в псевдогены [26].
Дрожжи K. lactis var. lactis и K. marxianus совместно обитают в молочных продуктах и, обладая общей системой типов спаривания, могут образовывать межвидовые гибриды [27]. По-видимому, доместикация молочных дрожжей K. lactis var. lactis произошла на основе приобретения генного кластера LAC4–LAC12 от молочных штаммов K. marxianus [6]. В свою очередь, дрожжи K. marxianus могли приобрести “лактозные” гены в результате горизонтального переноса соответствующих генов от бактерий [28]. Наше предположение подтверждено Varela и соавт. [5]. Проведенное ранее секвенирование и аннотация генома типовой культуры K. lactis var. drosophilarum CBS 2105, выделенной из Drosophila sp. (США), не обнаружило последовательностей генов LAC4 и LAC12, что полностью согласуется с проведенным нами Саузерн-блотингом [6]. Известно, что у штамма K. lactis var. lactis NRRL Y-1140 и дрожжей K. marxianus генный кластер LAC4–LAC12 расположен, соответственно, в субтеломерной области хромосом IIR и 3L [11, 26, 29]. С помощью точечно-матричного анализа (dot matrix plots) проведено сравнение субтеломерных областей указанных хромосом у K. lactis var. lactis NRRL Y-1140, K. lactis var. drosophilarum CBS 2105 и трех штаммов К. marxianus: L03 (молочный продукт), NBRC 1777 (почва) и UFS Y-2791 (сок Agave americana) [5]. Если сходство нуклеотидных последовательностей хромосом IIR и 3L на большей части длины составляет всего 63.4–64.5%, то, начиная с кластера LAC4–LAC12, оно резко возрастает до 99.8% (штамм L03), 96.1% (NBRC 1777) и 85.1% (UFS Y-2791). С другой стороны, у разновидностей K. lactis нуклеотидные последовательности правого плеча второй хромосомы идентичны на 94.1%, за исключением субтеломерного участка длиной около 15 т.п.н., в котором расположен генный кластер LAC4–LAC12. По-видимому, в результате межвидовой гибридизации произошел перенос лактозного кластера из молочного штамма K. marxianus в правое плечо хромосомы II дрожжей K. lactis. Следует отметить, что большинство изученных нами штаммов K. lactis var lactis содержали локус LAC2 (рис. 1б). Причем этим локусом обладали как молочные, госпитальные, так и четыре природных штамма (ВКПМ Y-3737, H1, H2 и H3), выделенные нами в свое время из почвы в Измайловском парке Москвы [30]. Локусы LAC1 и LAC3 могли произойти от локуса LAC2 в процессе внутривидовой гибридизации за счет рекомбинации гомологичных субтеломерных последовательностей различных хромосом. Хорошо известно, что повторяющиеся субтеломерные последовательности являются горячими точками внутри- и межхромосомных рекомбинационных событий. На дрожжах Saccharomyces показан природный межвидовой перенос многих субтеломерных генов, включая гены ферментации различных сахаров [31, 32]. Интрогрессия кластера генов β-галактозидазы (LAC4) и пермеазы лактозы (LAC12) в геном дрожжей K. lactis var. lactis, по-видимому, совпала по времени с одомашниванием “молочных” видов животных [5, 33].
Анализ нуклеотидных последовательностей генов пермеазы лактозы у разных штаммов K. lactis var. lactis позволит прояснить происхождение их полимерных локусов LAC и получить ценную информацию в области генетики адаптивных ферментационных признаков. В настоящее время мы проводим гибридологический анализ штаммов, обладающих разными локусами LAC, и секвенирование пермеазных генов LAC12.
Работа поддержана российско-тайваньским грантом Российского фонда фундаментальных исследований (№ 18-54-52002 МНТ_а).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Kurtzman C.P. (2003) Phylogenetic circumscription of Saccharomyces, Kluyveromyces and other members of the Saccharomycetaceae, and the proposal of the new genera Lachancea, Nakaseomyces, Naumovia, Vanderwaltozyma and Zygotorulospora. FEMS Yeast Res. 4, 233–245.
Lachance M.-A., Kurtzman C.P., Fell J.W., Boekhout T. (2011) Kluyveromyces van der Walt (1971). In: The Yeasts. A Taxonomic Study. Eds Kurtzman C.P., Fell J.W. Amsterdam: Elsevier, 471–482.
Наумов Г.И. (2006) Генетика полиморфизма утилизации лактозы у дрожжей Kluyveromyces marxianus. ДАН. 409(3), 422–424.
Наумов Г.И. (2008) Обнаружение суперсемейств лактозных генов LAC у дрожжей Kluyveromyces. ДАН. 420(6), 832–834.
Varela J.A., Puricelli M., Ortiz-Merino R.A., Giacomobono R., Braun-Galleani S., Wolfe K.H., Morrissey J.P. (2019) Origin of lactose fermentation in Kluyveromyces lactis by interspecies transfer of a neofunctionalized gene cluster during domestication. Curr. Biol. 29, 4284–4290.
Наумов Г.И., Наумова Е.С., Баррио Е., Керол А. (2006) Генетическое и молекулярное изучение неспособности дрожжей Kluyveromyces lactis var. drosophilarum сбраживать лактозу. Микробиология. 75, 299–304.
Riley M.I., Sreekrishna K., Bhairi S., Dickson R.C. (1987) Isolation and characterization of mutants of Kluyveromyces lactis defective in lactose transport. Mol. Gen. Genet. 208, 145–151.
Dickson R.C., Riley M.I. (1989) The lactose-galactose regulon of Kluyveromyces lactis. in: Yeast Genetic Engineering. Eds Barr P.J., Brake A.J., Valenzuela P. Boston: Butterworth Publ., 19–40.
Gödecke A., Zachariae W., Arvanitidis A., Breunig K.D. (1991) Coregulation of the Kluyveromyces lactis lactose permease and β-galactosidase genes is achieved by interaction of multiple LAC9 binding sites in a 2.6 kbp divergent promoter. Nucl. Acids Res. 19, 5351–5358.
Breunig K.D., Bolotin-Fukuhara M., Bianchi M.M., Bourgarel D., Falcone C., Ferrero I., Frontali L., Goffrini P., Krijger J.J., Mazzoni C., Milkowski C., Steensma H.Y., Wésolowski-Louvel M., Zeeman A.M. (2000) Regulation of primary carbon metabolism in Kluyveromyces lactis. Enz. Microb. Techn. 26, 771–780.
Fairhead C., Dujon B. (2006). Structure of Kluyveromyces lactis subtelomeres: duplications and gene content. FEMS Yeast Res. 6, 428–441.
Herman A., Halvorson H. (1963) Identification of the structural gene for beta-glucosidase in Saccharomyces lactis. J. Bacteriol. 85(4), 895–900.
Wesolowski-Louvel M., Fukuhara H. (1995) A map of the Kluyveromyces lactis genome. Yeast. 11, 211–218.
Наумов Г.И., Наумова Е.С. (2014) Полимерные гены ферментации лактозы у дрожжей Kluyveromyces lactis: новый локус LAC3. ДАН. 455(3), 363–365.
Kumar S., Stecher G., Tamura K. (2016) MEGA7: molecular evolutionary genetics analysis version 7.0 for bigger datasets. Mol. Biol. Evol. 33(7), 1870–1874.
Наумова Е.С., Сухотина Н.Н., Наумов Г.И. (2005) Молекулярные маркеры, дифференцирующие молочные дрожжи Kluyveromyces lactis var. lactis от их ближайших диких родственников – европейской популяции “krassilnikovii”. Микробиология. 74(3), 387–393.
Çuhadar T., Kalkancı A. (2017) Emerging pathogen: Candida kefyr (Kluvyeromyces marxianus). Mikrobiyol. Bul. 51(4), 387–395. https://doi.org/10.5578/mb.61813
Hennequin C., Thierry A., Richard G.F., Lecointre G., Nguyen H.-V., Gaillardin C., Dujon B. (2001) Microsatellite typing as a new tool for identification of Saccharomyces cerevisiae strains. J. Clin. Microbiol. 3, 551–559.
de Llanos R., Querol A., Planes A.M., Fernandez-Espinar M.T. (2004) Molecular characterization of clinical Saccharomyces cerevisiae isolates and their association with non-clinical strains. Syst. Appl. Microbiol. 27(4), 427–435.
Imre A., Rácz H.V., Antunovics Z., Rádai Z., Kovács R., Lopandic K., Pócsi I., Pfliegler W. P. (2019) A new, rapid multiplex PCR method identifies frequent probiotic origin among clinical Saccharomyces isolates. Microbiol. Res. 227, 126298. https://doi.org/10.1016/j.micres.2019.126298
Pfliegler W.P., Boros E., Pázmándi K., Jakab Á., Zsuga I., Kovács R., Urbán E., Antunovics Z., Bácsi A., Sipic-zki M., Majoros L., Pócsi I. (2017) Commercial strain-derived clinical Saccharomyces cerevisiae can evolve new phenotypes without higher pathogenicity. Mol. Nutr. Food Res. 61(11). https://doi.org/10.1002/mnfr.201601099
Fukuhara H. (2003) The Kluyver effect revisited. FEMS Yeast Res. 3, 327–331.
Fukuhara H. (2006) Kluyveromyces lactis – a retrospective. FEMS Yeast Res. 6, 323–324.
Wesolowski-Louvel M., Goffrini P., Ferrero I., Fukuhara H. (1992) Glucose transport in the yeast Kluyveromyces lactis. I. Properties of an inducible low-affinity glucose transporter gene. Mol. Gen. Genet. 333, 89–96.
Наумов Г.И. (2005) Почему дрожжи Kluyveromyces wickerhamii ассимилируют, но не сбраживают лактозу? ДАН. 403, 847–849.
Varela J.A., Montini N., Scully D., Van der Ploeg R., Oreb M., Boles E., Hirota J., Akada R., Hoshida H., Morrissey J.P. (2017) Polymorphisms in the LAC12 gene explain lactose utilisation variability in Kluyveromyces marxianus strains. FEMS Yeast Res. 17(3). https://doi.org/10.1093/femsyr/fox021
Наумов Г.И. (2005) Доместикация молочных дрожжей Kluyveromyces lactis: перенос кластера генов β-галактозидазы (LAC4) и лактозной пермеазы (LAC12)? ДАН. 401(2), 279–281.
Poch O., L’Hôte H., Dallery V., Debeaux F., Fleer R., Sodoyer R. (1992) Sequence of the Kluyveromyces lactis β-galactosidase: comparison with prokaryotic enzymes and secondary structure analysis. Gene. 118, 55–63.
Bussereau F., Casaregola S., Lafay J.-F., Bolotin-Fukuhara M. (2006) The Kluyveromyces lactis repertoire of transcriptional regulators. FEMS Yeast Res. 6, 325–335.
Наумов Г.И., Наумова Е.С., Глушакова А.М., Качалкин А.В., Чернов И.Ю. (2014) Обнаружение молочных дрожжей Kluyveromyces lactis var. lactis в природе. Микробиология. 83, 677–681.
Naumova E.S., Naumov G.I., Michailova Yu.V., Martynenko N.N., Masneuf-Pomarède I. (2011) Genetic diversity study of the yeast Saccharomyces bayanus var. uvarum reveals introgressed subtelomeric Saccharomyces cerevisiae genes. Res. Microbiol. 162(2), 204–213.
Peter J., De Chiara M., Friedrich A., Yue J.-X., Pflie-ger D., Bergström A., Sigwalt A., Barre B., Freel K., Llored A., Cruaud C., Labadie K., Aury J.-M., Istace B., Lebrigand K., Barbry P., Engelen S., Lemainque A., Wincker P., Liti G., Schacherer J. (2018) Genome evolution across 1.011 Saccharomyces cerevisiae isolates. Nature. 556 (7701), 339–344
Larson G., Fuller D.Q. (2014) The evolution of animal domestication. Annu. Rev. Ecol. Evol. Syst. 45, 115–136.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


