Молекулярная биология, 2021, T. 55, № 1, стр. 20-41
Особенности регуляции синтеза рибосомных белков у прокариот
А. О. Михайлина a, *, Е. Ю. Никонова a, О. С. Костарева a, С. В. Тищенко a
a Институт белка Российской академии наук
142290 Московская обл., Пущино, Россия
* E-mail: alisamikhaylina15@gmail.com
Поступила в редакцию 17.06.2020
После доработки 12.07.2020
Принята к публикации 20.07.2020
Аннотация
Биосинтез белка, проходящий на рибосомах, считается основным процессом в жизни клетки. В сбалансированном синтезе белков и РНК в биогенезе рибосом важную роль играет регуляция экспрессии генов рибосомных белков. В представленном обзоре рассмотрены некоторые особенности процессов авторегуляции синтеза рибосомных белков у прокариот. Обсуждаются также механизмы ингибирования синтеза рибосомных белков, кодируемых 12 оперонами, такие как конкурентное ингибирование, механизмы “ловушки” и ретрорегуляции. Приведены примеры регуляции синтеза белков как индивидуальными рибосомными белками, так и их комплексами.
ВВЕДЕНИЕ
Регуляция синтеза рибосомных белков в клетках Escherichia coli была открыта более 40 лет назад и за это время хорошо изучена. Гены белков у прокариот, как правило, организованы в функциональные единицы – опероны, которые находятся под контролем оператора. В состав оперонов генов рибосомных белков (р-белков) могут входить и гены нерибосомных белков. Так, у E. coli это гены, кодирующие компоненты репликационного комплекса (dnaG – праймаза, priB – праймосомный белок N), субъединицы РНК-полимеразы (rpoA, rpoB, rpoC, rpoD), факторы трансляции (tsf, fus, tufA), гены, продукты которых участвуют в созревании рРНК (rimM), в процессинге (trmD) и модификации (rnpA) тРНК и экспорте белков через мембрану (secY). Для биогенеза рибосом требуется приблизительно эквимолярное количество р-белков и рРНК. Многие р-белки прокариот регулируют экспрессию как своих собственных генов, так и генов других белков своего оперона (рис. 1). К белкам-регуляторам относятся как белки малой (Small) рибосомной субчастицы (S1, S4, S7, S8, S15 и S20), так и большой (Large) субчастицы (L1, L4, L10, L12, L20).
Рис. 1.
Организация генов р-белков E. coli в оперонах. Белки-репрессоры показаны окружностями; участки мРНК, с которыми они взаимодействуют, обозначены символом ┴. Гены, экспрессия которых регулируется белком-репрессором, обозначены белым цветом; гены, которые не регулируются соответствующим р-белком, выделены серым цветом. P – промоторы; t и att – терминатор транскрипции и аттенюатор соответственно. а – S10-оперон. б – L11-оперон. в – S1-оперон. г – S2-оперон. д – L20-оперон. е – S6-оперон. ж – L10-оперон. з – S15-оперон. и – S4/α-оперон. к – S8/spc-оперон. л – S7/str-оперон. м – S20-оперон.
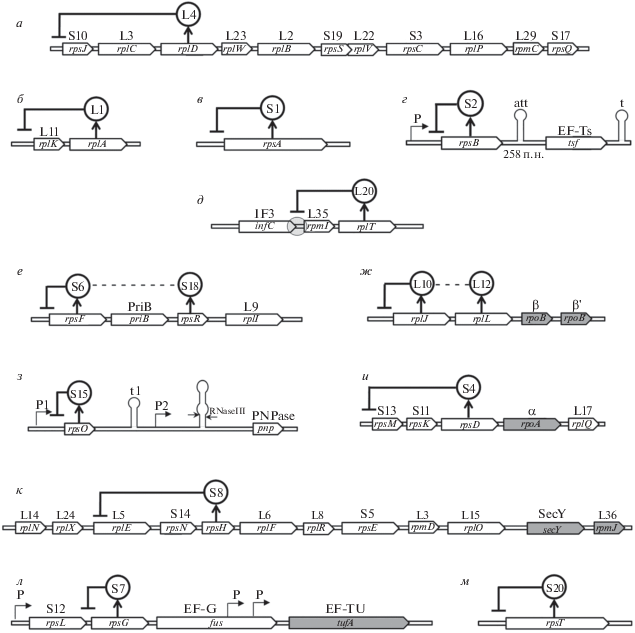
Основной принцип регуляции синтеза р-белков – принцип обратной связи, когда один из р-белков, кодируемых опероном, при его избыточном синтезе действует как репрессор трансляции мРНК всего оперона. Причем оператор может находиться как выше первого гена, так и между генами оперона. Участки связывания на мРНК некоторых белков-репрессоров гомологичны участкам связывания этих белков на рРНК, однако, многие р-белки регулируют синтез белков, связываясь с участком мРНК, не имеющим явной гомологии со специфическим участком на рРНК.
Авторегуляция синтеза р-белков может осуществляться по различным механизмам. Механизм конкурентного ингибирования предполагает конкуренцию между специфическими участками связывания белка-регулятора на мРНК и рРНК. При регуляции по механизму “ловушки” взаимодействие белка-репрессора с мРНК приводит к формированию структур, затрудняющих инициацию трансляции мРНК. Механизм ретрорегуляции подразумевает дестабилизацию мРНК при взаимодействии с белком-репрессором. Кроме того, регуляция может осуществляться как одиночными белками, так и их комплексами.
РЕГУЛЯЦИЯ СИНТЕЗА РИБОСОМНЫХ БЕЛКОВ НА ОСНОВЕ КОНКУРЕНТНОГО ИНГИБИРОВАНИЯ
Конкурентный механизм авторегуляции синтеза р-белков предполагает конкуренцию мРНК и рРНК за связывание с белком-репрессором. Как правило, сродство репрессора к мРНК значительно ниже, чем к рРНК.
S10-оперон
Экспрессия S10-оперона E. coli, содержащего гены 11 р-белков, контролируется р-белком L4 (рис. 1а) [1]. L4 регулирует как трансляцию, так и транскрипцию генов своего оперона [2].
В бактериальной рибосоме L4 связывается в основном с доменом I 23S рРНК, а его протяженная петля формирует часть туннеля выхода полипептида [3]. Считается, что C-концевая часть белка отвечает за регуляторные свойства, а центральный участок необходим для встраивания в рибосому [4].
Участки связывания белка L4 на 23S рРНК и мРНК не имеют явного сходства (рис. 2) [5]. Основной участок взаимодействия белка L4 с 23S рРНК – это место пересечения четырех спиралей, содержащее петлевой участок (рис. 2б) [6]. Участок мРНК S10-оперона, связавшись с которым белок L4 осуществляет контроль синтеза белков S10-оперона как на уровне транскрипции, так и на уровне трансляции, находится в 5'-нетранслируемой области (5'-НТО) первого гена оперона rpsJ и содержит шпильки HD–HG (рис. 2а).
Рис. 2.
Вторичная структура участков связывания белка L4 на РНК в E. coli. а – 5'-НТО гена белка S10 (rpsJ). Голубым выделены нуклеотиды терминатора транскрипции; последовательность SD и старт-кодон rpsJ – зеленым. Минимальный фрагмент мРНК, обладающий высоким сродством к белку L4, выделен штриховой рамкой. б – Минимальный фрагмент 23S рРНК, специфически связывающийся с белками L4 и L24. Красным выделены участки, с которыми взаимодействует белок L4.
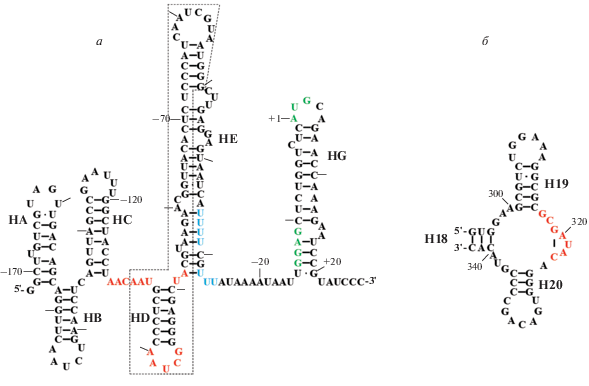
Известно, что участки мРНК S10-оперона E. coli, отвечающие за терминацию транскрипции и ингибирование трансляции, частично перекрываются, но не идентичны [7, 8]. 5'-НТО оперона содержит шесть шпилек (рис. 2а), однако первые три шпильки несущественны для регуляции экспрессии оперона in vivo. Шпильки HD и HE необходимы для контроля транскрипции, а для контроля трансляции наиболее важны шпильки HE и HG [7, 9, 10]. Шпилька HE и расположенная за ней уридин-богатая последовательность образуют сайт ρ-независимого терминатора транскрипции. Минимальный фрагмент мРНК S10-оперона E. coli, обладающий высоким сродством к белку L4, содержит шпильку HD и часть шпильки НЕ [5]. На основании биоинформатического анализа предсказана консервативная структура регуляторного участка мРНК S10-оперона [11]. Предполагается, что основным участком связывания L4 является петля шпильки HD и консервативные неспаренные нуклеотиды, фланкирующие эту шпильку (рис. 2а) [12].
Регуляция транскрипции осуществляется в результате взаимодействия белка L4 с 5'-НТО мРНК, фактором транскрипции NusA и РНК-полимеразой [1]. Белок L4, взаимодействуя с NusA, увеличивает время паузы РНК-полимеразы на терминаторном сайте шпильки HE, что приводит к преждевременной терминации транскрипции [13–15].
У некоторых бактерий L4-опосредованная регуляция отсутствует. Так, белок L4 Pseudomonas aeruginosa не ингибирует синтез белков своего оперона, и в области 5'-НТО мРНК белка S10 отсутствуют детерминанты, похожие на участок связывания этого белка в E. coli. Оперон P. aeruginosa, несущий ген белка L4, содержит также ген белка L24, а область в районе 5'-НТО мРНК гена rplC похожа на участок связывания этих белков в рибосоме. В связи с этим предполагается, что в регуляции S10-оперона P. aeruginosa участвуют оба белка – L4 и L24. По-видимому, возможные механизмы регуляции этого оперона отличаются у разных организмов [16].
S10-подобный оперон археи Methanocaldococcus jannaschii кодирует не 11, как у E. coli, а пять генов р-белков (L3, L4, L23, L2, S19), первым геном этого оперона является ген белка L3, а не S10. Синтез белков этого оперона у M. jannaschii также регулируется белком L4. L4-связывающий участок включает 5'-НТО и начало кодирующей части мРНК белка L3 [17].
L11-оперон
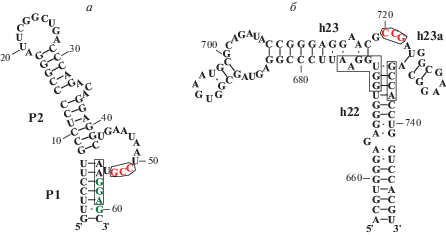
Консервативный двухдоменный р-белок L1 регулирует трансляцию мРНК L11-оперона бактерий. Этот белок участвует в формировании L1-выступа 50S субчастицы рибосомы, прочно и специфически связываясь с 23S рРНК в районе спиралей 76–78. При недостатке 23S рРНК белок L1 связывается со специфическим участком мРНК своего оперона и препятствует ее трансляции. Подробно изучена регуляция L11-оперона E. coli [18–20] и L1-оперона архей рода Methanocaldococcus [21, 22].
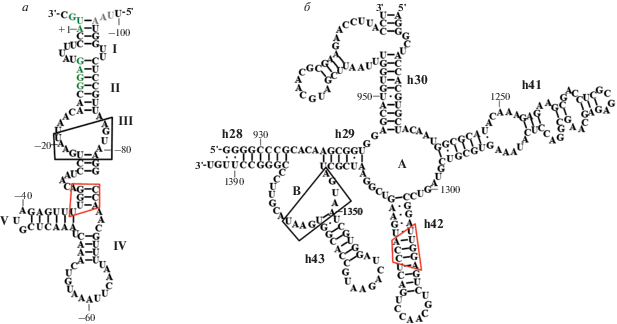
Участок связывания белка L1 на мРНК L11-оперона E. coli располагается в 5'-НТО мРНК белка L11 (рис. 3). Сопряженность трансляции генов оперона приводит к сопряжению репрессии трансляции [23, 24]. У архей M. vannielii и M. jannaschii участок связывания белка L1 находится в 5'-НТО его собственной мРНК (рис. 3). Авторегуляция синтеза белка L1 M. vannielii происходит либо до формирования первой пептидной связи L1, либо на этой стадии [25].
Рис. 3.
Организация генов L11-оперона у бактерий и L1-оперона архей. Белок-репрессор обозначен окружностью, участки мРНК, с которыми он взаимодействует, показаны символом ┴.
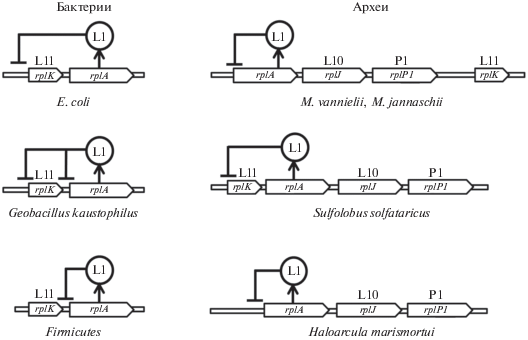
Поиск участков связывания белка L1 на мРНК различных бактерий показал, что положение L1-связывающего сайта не строго консервативно [11]. В бактериях одних групп эти сайты располагаются перед геном белка L11 (Proteobacteria, Spirochaetes, Thermotogae и Tenericutes), в других – перед геном белка L1 (Cyanobacteria, Actinobacteria и Chloroflexi), а в 40% геномов Firmicute участки связывания белка L1 обнаружены перед обоими генами (рис. 3). Недавно на мРНК L11-оперона Thermotoga maritimа найдены два L1-регуляторных участка: первый в 5'-НТО мРНК белка L11, как в E. coli, а второй включает лидерный и кодирующий участки мРНК белка L1 [26].
Структуры участков связывания белка L1 на рРНК консервативны во всех доменах жизни [27, 28]. Сайты связывания белка L1 на мРНК имеют высокую гомологию первичной и вторичной структуры с его участком связывания на рРНК [22, 29] (рис. 4). Однако константы связывания белка L1 с рРНК и мРНК отличаются примерно на порядок, в результате чего регуляция трансляции L11-оперона осуществляется по классическому принципу обратной связи [24].
Рис. 4.
Участки связывания белка L1 на 23S рРНК (T. thermophilus) и мРНК (E. coli и M. jannaschii). На фрагментах РНК красным выделены нуклеотиды, образующие консервативные контакты с белком L1.
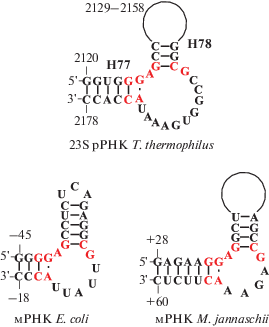
Поскольку участки связывания белка L1 на мРНК и рРНК гомологичны у бактерий и архей (рис. 4), L1 археи M. vannielii способен функционально замещать белок L1 E. coli как в составе рибосомы, так и в качестве репрессора трансляции, а L1 E. coli ингибирует трансляцию мРНК L1-оперона M. vannielii in vitro [22, 30]. Белок L1 бактерии T. thermophilus способен регулировать синтез белков L1-оперона археи M. vannielii in vitro [31].
Структурные и биохимические исследования свидетельствуют о ведущей роли домена I белка L1 во взаимодействии с РНК в бактериях [31–33] и археях [34].
S1-оперон
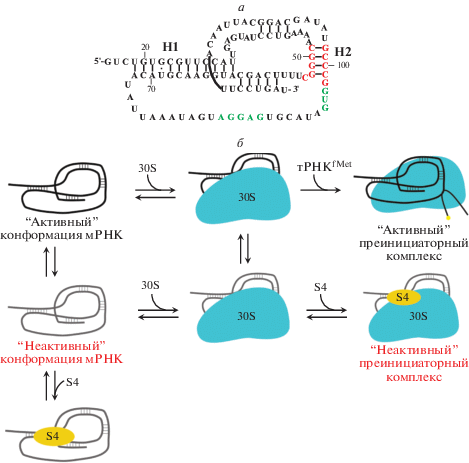
Р-белок S1, кодируемый геном rpsA, регулирует свой собственный синтез на уровне трансляции (рис. 1в) [35, 36]. S1 – один из белков малой рибосомной субчастицы, расположен между головкой и платформой 30S субчастицы и контактирует с мРНК, р-белками [37, 38] и РНК-полимеразой [39, 40].
Известно, что белок S1 необходим для трансляции некоторых мРНК [35], включая свою собственную [41]. Белок содержит шесть доменов. Три N-концевых домена (D1–D3) взаимодействуют с р-белками, а также с различными мРНК в составе рибосомы и обладают РНК-шаперонной активностью [42]. Эти домены участвуют в формировании преинициаторного комплекса 30S рибосомной субчастицы с мРНК. С-концевые домены D3–D6 обеспечивают специфичность узнавания одноцепочечных участков различных мРНК [43, 44]. Известно, что участок связывания белка S1 на его мРНК находится в 5'-НТО, он образован тремя шпильками (I–III), разделенными AU-богатыми одноцепочечными участками (ss1 и ss2) (рис. 5а). Шпилька III содержит старт-кодон и SD-подобный элемент (GAAG) (рис. 5а), формирующий только три комплементарные пары оснований c анти-SD 16S рРНК [45]. Тем не менее, S1-оперон – один из наиболее сильных оперонов в E. сoli с эффективным отрицательным аутогенным контролем [36]. Последовательности GG(A) в петлях шпилек I и II мРНК совместно со слабой SD могут образовывать разделенный в пространстве общий SD-элемент (рис. 5б) [35]. Регуляция синтеза белка S1 происходит на уровне формирования 30S преинициаторного комплекса. Так, 30S рибосомная субчастица без белка S1 не способна образовывать преинициаторный комплекс со своей мРНК in vitro. Добавление белка S1 к 30S субчастицам с дефицитом S1 в молярном соотношении 1 : 1 восстанавливает их способность связывать мРНК rpsA, тогда как избыток белка ингибирует это связывание (рис. 5б).
Рис. 5.
Регуляторный участок мРНК rpsA. а – Вторичная структура участка инициации трансляции белка S1 на мРНК rpsA. Серым отмечены консервативные нуклеотиды GG. Рамкой выделена SD-последовательность. б – Схема аутогенной регуляции синтеза белка S1.
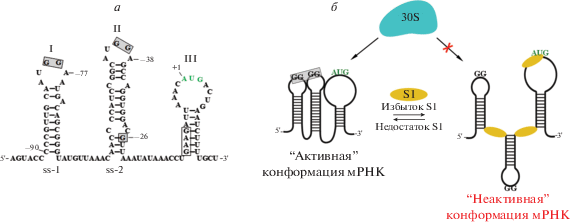
Белок S1 взаимодействует с одноцепочечными участками ss-1 и ss-2 на мРНК rpsA (рис. 5а). Это взаимодействие изменяет структуру участка инициации трансляции rpsA, нарушая его активную конформацию, и предотвращает формирование преинициаторного комплекса с 30S субчастицей [35] (рис. 5б).
Вторичная структура 5'-НТО мРНК rpsA сходна в пяти семействах γ-протеобактерий (Enterobacteriaceae, Pasterellaceae, Vibrionaceae, Erwiniaceae, Shewanellaceae) [45]. Спирали II и III довольно консервативны, межспиральный участок, как правило, содержит AU-богатую последовательность [11]. Слабая спираль III всегда содержит SD-подобный элемент и AUG в петле, а в петлях шпилек I и II имеются триплеты GGA (рис. 5а).
S2-оперон
S2-оперон (оперон rpsB–tsf) бактерий кодирует р-белок S2 и фактор элонгации Ts (рис. 1г). В клетках E. coli регуляция этого оперона осуществляется на уровне трансляции белком S2 [46].
Глобулярный домен белка S2 взаимодействует с “телом” 30S субчастицы рибосомы, а двухцепочечный домен направлен к “головке” малой субчастицы. Показано, что белок S2 на рибосоме участвует в связывании SD на этапе инициации трансляции [47]. Гомологии S2-связывающего участка на мРНК и на 16S рРНК (h26 и h35–h37) [48] не обнаружено.
Регуляторный участок, с которым взаимодействует белок S2, находится в 5'-НТО мРНК rpsB. Наиболее важны для регуляции участки CR и RH мРНК rpsB (рис. 6). Интересно, что белок S2 более эффективно регулирует экспрессию rpsB–lacZ в присутствии белка S1 [46]. Белок S2 требуется для встраивания S1 в рибосому [40] и может образовывать комплекс с S1.
Рис. 6.
Вторичная структура 5'-НТО мРНК rpsB E. coli. LH и RH – консервативные шпилечно-петельные структуры, CR – центральный, слабоструктурированный участок. Консервативные нуклеотиды выделены красным.
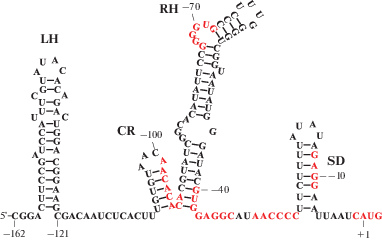
Удлиненный последовательностью TGTG “–10”-промотор гена rpsB и вторичная структура 5'-НТО мРНК rpsB консервативны у γ-протеобактерий [49]. Ген tsf не имеет своего промотора, и EF-Ts синтезируется с бицистронной мРНК rpsB–tsf [50, 51]. Гены разделены протяженным участком, содержащим инвертированные повторы, за которыми следует аттенюатор [52]. При связывании белка S2 с участком мРНК rpsB синтез EF-Ts также ингибируется [46]. Активность промотора S2-оперона снижается при аминокислотном голодании in vivo или при увеличении концентрации алармона ppGpp in vitro. Для этой регуляции важна GC-богатая нуклеотидная последовательность, отделяющая “–10”-элемент от старта транскрипции [49].
Таким образом, регуляция синтеза белков S2 и EF-Ts осуществляется как белком S2 на уровне трансляции по принципу обратной связи, так и на уровне транскрипции – глобальным регулятором ppGpp. Причем для эффективной и регулируемой транскрипции S2-оперона необходимо сочетание всех консервативных элементов промотора rpsB [49].
L20-оперон
L20-оперон (оперон rpmI–rplT) включает гены р-белков L35 (rpmI) и L20 (rplT), и фактора инициации трансляции 3 – IF3 (infC) (рис. 1д). Белок L20 прямо ингибирует трансляцию цистрона L35 и сопряженно – трансляцию своего собственного цистрона [53]. Это первично связывающийся с рРНК белок 50S субчастицы [54], взаимодействующий с участком между спиралями 40 и 41 23S рРНК.
Для репрессии трансляции L20-оперона in vivo достаточно С-концевого домена белка [55]. В межцистронной области infC–rpmI мРНК E. coli обнаружены два регуляторных сайта, с которыми L20 взаимодействует с одинаковым сродством (рис. 7). Первый сайт включает псевдоузел, сформированный участками infC и rpmI (рис. 7а, б) [55] и способствующий сопряжению трансляции IF3 и цистронов р-белков [56]. Второй сайт (рис. 7а, в) содержит центральную часть спирали t1, структура которой похожа на сайт связывания белка L20 на 23S рРНК (рис. 7г) [57]. Наличие двух L20-связывающих сайтов подтверждено in vivo мутационным анализом мРНК L20-оперона [58]. Эти участки находятся вблизи псевдоузла, образуя один участок в трехмерной структуре оператора. Минимальный участок мРНК, необходимый для репрессии трансляции, включает псевдоузел и нижние 2/3 спирали t1 (рис. 7) [55, 58].
Рис. 7.
Вторичная структура трех L20-связывающих сайтов на РНК. Консервативные нуклеотиды на всех участках выделены красным. а – Участок infC–rpmI мРНК E. coli. б – Первый сайт связывания L20 с мРНК – псевдоузел, в котором спираль S2 образуется удаленными друг от друга участками мРНК (показано сходящимися стрелками). в – Второй L20-связывающий сайт на мРНК. Рамками выделены старт-кодон гена rpmI и стоп-кодон infC. г – L20-связывающий сайт на 23S рРНК E. coli.
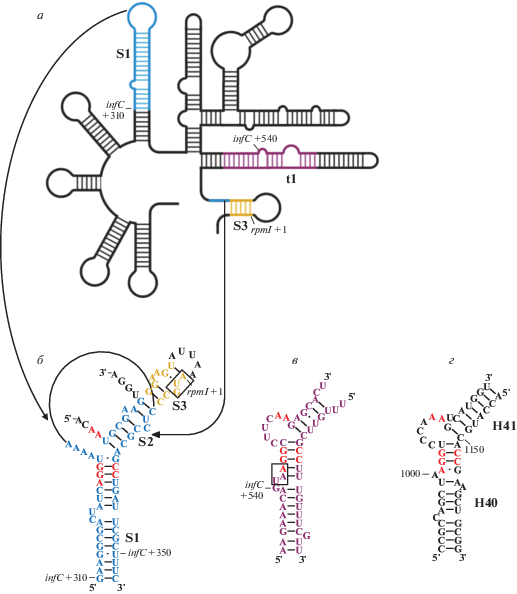
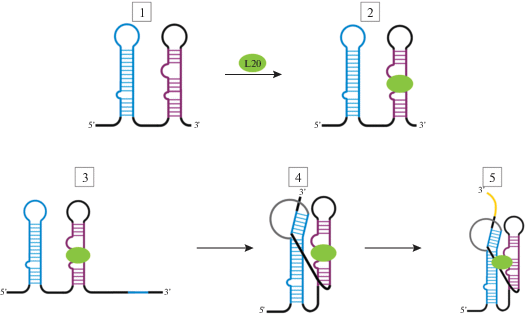
Предполагается, что белок L20 регулирует синтез белков своего оперона по механизму конкуренции между репрессором и рибосомой за связывание с мРНК [59]. При транскрипции оператора rpmI сначала синтезируется шпилька S1 мРНК (показана синим на рис. 8, шаг 1), затем шпилька t1 (показана фиолетовым на рис. 8, шаг 1). После формирования шпильки мРНК белок L20 прочно связывается с участком на этой шпильке (рис. 8, шаг 2). Далее синтезируется участок мРНК, соответствующий 3'-концу шпильки S2 (рис. 8, шаг 3, линейный участок 3'-конца мРНК показан синим), что приводит к формированию псевдоузла (рис. 8, шаг 4). После того, как операторный участок принял необходимую конформацию, связавшаяся ранее молекула белка L20 меняет свое положение на мРНК (рис. 8, шаг 5), блокируя участок взаимодействия с рибосомой. Предполагается, что взаимодействие L20 со шпилькой t1 мРНК позволяет временно увеличивать локальную концентрацию белка L20 вблизи мРНК до тех пор, пока не сформируется псевдоузел мРНК. Структура комплекса L20 с операторной частью мРНК к настоящему времени неизвестна, однако показано, что белок связывается с этой областью с молярным соотношением 1 : 1 [59].
Рис. 8.
Предполагаемая модель регуляции генов rpmI–rplT белком L20. Участки мРНК, формирующие псевдоузел – шпилька S1 мРНК infC и участок 5'-НТО мРНК rpmI, формирующий спираль S2, выделены синим. Шпилька t1 мРНК infC показана фиолетовым. Желтым обозначен участок гена rpmI, зеленым овалом – р-белок L20.
В B. subtilis найден только один участок, с которым взаимодействует белок L20, и он отличается от регуляторного участка в E. coli. Эта область находится в 5'-НТО мРНК infC, а регуляция осуществляется на уровне транскрипции, но не трансляции. Тем не менее, данный участок мРНК также имеет сходство с L20-связывающим сайтом 23S рРНК B. subtilis [60]. Таким образом, несмотря на отличие в структурах L20-связывающих участков РНК у разных организмов, белок имеет схожие детерминанты для взаимодействия на мРНК и рРНК.
S6-оперон
В геномах многих видов бактерий ген rpsF, кодирующий р-белок S6, локализован рядом с генами priB (компонент праймосомы) и rpsR (р-белок S18) (рис. 1е). S6-оперон (rpsF-оперон) E. coli включает также ген rplI (р-белок L9) [61]. Белки S6 и S18 не относятся к первично-связывающимся с рРНК белкам, они формируют гетеродимер, взаимодействующий с участком 16S рРНК, связанным с р-белком S15 [62–64]. Нуклеотиды ССR (R = A/G) в 16S рРНК – единственный консервативный участок, специфически взаимодействующий с комплексом S6∙S18 (рис. 9б). Аналогичная последовательность обнаруживается и в S6∙S18-связывающем сайте на мРНК rpsF (рис. 9а), что указывает на ключевую роль этого элемента во взаимодействии белкового комплекса с РНК [65], причем сродство S6∙S18 к рРНК выше, чем к мРНК [66].
Рис. 9.
Вторичная структура S6∙S18-связывающих участков РНК. а – Предположительная вторичная структура участка мРНК rpsF E. coli. Последовательность SD выделена зеленым. б – Фрагмент 16S рРНК T. thermophilus. Рамками выделены нуклеотиды, образующие S6∙S18-связывающий сайт, красным – консервативный CCR-мотив.
РНК-белковые контакты в рибосомном и регуляторном комплексах S6∙S18 консервативны. Замены аминокислотных остатков в белке S18, которые приводят к потере сродства белка к рРНК, ослабляют и его взаимодействие с мРНК [65, 67]. Мутации, нарушающие белок-белковые контакты в комплексе S6∙S18, также приводят к снижению уровня регуляции синтеза белков S6‑оперона [63, 67]. Анализ 5'-НТО S6-оперона у γ‑протеобактерий, Firmicutes и Tenericutes показал, что ССR-мотив входит в состав петли консервативной шпильки Р1 мРНК [65], на 3'-конце которой находится SD (рис. 9а). В модели комплекса с мРНК белок S6, взаимодействующий в рибосоме с малым желобком h22 и h23b рРНК, образует связи со спиралью P1. Белок S18 может также контактировать со спиралью P1 и с CCR-мотивом (рис. 9а).
Связывание S6∙S18 с мРНК стабилизирует ее структуру, делая SD недоступным для взаимодействия с рибосомой и ингибируя трансляцию мРНК S6-оперона по принципу обратной связи. Мутационный анализ показал, что последовательность спирали P1 мРНК rpsF важна для эффективности трансляции как в клетках E. coli, так и у B. subtilis [66, 67], однако белок S18 B. subtilis имеет слабое сродство к мРНК в отсутствие белка S6 [66].
L10-оперон
L10-оперон (rplJL-оперон) E. coli содержит гены р-белков L10 (rplJ) и L12 (rplL) и β-, β'-субчастиц РНК-полимеразы (рис. 1ж). В результате аттенюации транскрипции и процессинга мРНК rplJL образуются два отдельных транскрипта, один из которых содержит цистроны р-белков, а второй – цистроны субъединиц РНК-полимеразы. Трансляция цистронов rplL и rplJ сопряжена [23] и регулируется по принципу конкурентного ингибирования комплексом, состоящим из одной молекулы белка L10 и четырех молекул L12 (L10∙(L12)4) [68–72].
У представителей семи родов энтеробактерий, включая E. coli, гены rplКАJL разделены на два оперона: L11 (rplКА) – регулируется белком L1, и L10 (rplJL) – регулируется белком L10 [23, 24]. У архебактерий гены rplАJL транскрибируются в виде трицистронной мРНК, трансляция которой регулируется белком L1 [73].
Белок L12 на рибосоме взаимодействует только с белком L10, не связывается с рРНК, и не может самостоятельно регулировать трансляцию [23, 74]. Это единственный р-белок, который представлен на рибосоме в нескольких копиях. В E. coli и других мезофильных бактериях образуется пентамерный комплекс L10∙(L12)4, тогда как термофильные бактерии содержат гептамерный комплекс L10∙(L12)6 [75–77].
Основной вклад в узнавание белка L10 на рибосоме вносит консенсусный мотив “излом–поворот” (H42–44 23S рРНК) в районе GTPазного центра (рис. 10б) [57, 78], похожий на L10-связывающий участок мРНК rplJ E. coli (рис. 10а) [79]. Важную роль во взаимодействии с белком играет вторичная структура мРНК, удаление любого участка этой спирали снижает эффективность регуляции [80, 81]. Известно, что в связывании многих р-белков с РНК ведущую роль играют неспаренные нуклеотиды и выпетливания [82–86].
Рис. 10.
Вторичная структура L10∙(L12)4-связывающих сайтов РНК E. coli. а – Вторичная структура L10∙(L12)4-связывающего сайта мРНК rplJL. б – Вторичная структура L10∙(L12)4-связывающего участка 23S рРНК. Красным показаны L10∙(L12)4-связывающие участки. Старт-кодон мРНК rplJ выделен зеленым.
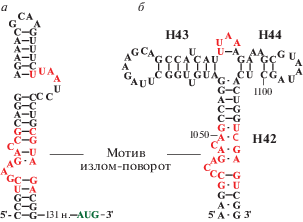
Показано, что два консервативных аденина в выпетливании UUAA мРНК защищаются комплексом L10·(L12)4 от химических реагентов (рис. 10а) [87]. У всех организмов выпетливание находится на расстоянии 4 п.н. от консенсусного мотива “излом–поворот”. Консервативная петля UAA 23S рРНК имеет такое же расположение относительно мотива “излом–поворот”, как и консервативные аденины в выпетливании мРНК (рис. 10) [79]. Замены этих аденинов как в мРНК, так и в рРНК, снижают сродство L10·(L12)4 к РНК.
Модель регуляции L10-оперона основана на возможности существования двух альтернативных конформаций 5'-НТО мРНК rplJ. Предполагается, что регуляция оперона осуществляется в результате конкуренции рибосомы с репрессором за связывание с мРНК в “открытой” или “закрытой” конформации (рис. 11a). Связывание L10(L12)4 приводит к изменению вторичной структуры мРНК – формируется двойная спираль между SD цистрона rplL и 5'-проксимальным участком мРНК и образуется “закрытая” конформация мРНК (SD недоступна) [88] (рис. 11a). Такая структура может стать мишенью для РНКаз, специфичных к двухцепочечным РНК (РНКаза III), что приводит к снижению стабильности мРНК.
Рис. 11.
Схемы регуляции экспрессии мРНК rplJL. а – Модель координированной экспрессии трансляции мРНК rplJL E. coli. Последовательности SD обозначены зеленым. б – Модель аутогенной аттенюации транскрипции L10-оперона B. subtilis. Серым квадратом обозначен участок терминации. Нуклеотидные остатки, переходящие между шпильками АТ и ААТ выделены желтым, между шпильками АТ и Т – голубым. Зеленым обозначены SD гена, кодирующего лидерный пептид и rplJ. Консервативный мотив “излом–поворот”, с которым взаимодействует комплекс L10(L12)4, выделен красным.
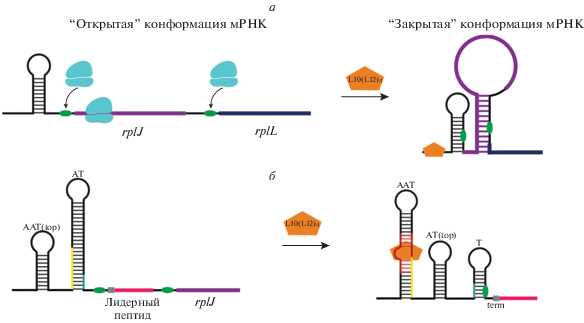
Регуляция синтеза мРНК rplJL B. subtilis проходит по механизму аттенюации транскрипции [89, 90]. Лидерный участок этой мРНК содержит три перекрывающиеся шпильки. Внутренний теминатор транскрипции и находящаяся перед ним шпилька AT действуют как антитерминатор транскрипции, а расположенная выше структура AAT, работает как анти-антитерминатор (рис. 11б). L10(L12)4B. subtilis стабилизирует AAT-шпильку мРНК (рис. 11б), предотвращая образование AT-шпильки и обеспечивая терминацию транскрипции. Лидерный участок мРНК rplJL B. subtilis кодирует лидерный пептид, SD которого находится с 3'-конца терминаторной шпильки (рис. 11б). Трансляция лидерного пептида может увеличивать экспрессию rplJL B. subtilis за счет блокирования доступности транскрипта для ρ-фактора. Таким образом, L10-оперон B. subtilis регулируется как по механизму аттенюации, так и по механизму антитерминации, осуществляемой лидерным пептидом. Такой двойной посттранскрипционный контроль может обеспечить тонкую настройку экспрессии rplJL B. subtilis в зависимости от фазы роста клеток. Аналогичная структура лидерного участка rplJL, включающего последовательность лидерного пептида, обнаружена и у других видов Bacillus [89].
РЕГУЛЯЦИЯ СИНТЕЗА РИБОСОМНЫХ БЕЛКОВ ПО МЕХАНИЗМУ “ЛОВУШКИ”
Механизм регуляции синтеза р-белков по принципу “ловушки” предполагает формирование “неактивной” структуры мРНК при связывании белка-репрессора, что блокирует рибосому на преинициаторной стадии. Наиболее хорошо изучена авторегуляция по механизму “ловушки” синтеза р-белка S15 E. coli. Предполагается, что механизм “ловушки” действует при более низком сродстве белка к мРНК, чем при конкурентном механизме, так как репрессору нужно только стабилизировать непродуктивный инициаторный комплекс, тогда как при конкурентном механизме репрессор и рибосома должны конкурировать за связывание с мРНК [91].
S15-оперон
S15-оперон состоит из двух генов – rpsO (кодирует р-белок S15) и pnp (кодирует полинуклеотидфосфорилазу). В E. coli эти два гена котранскрибируются. При избыточном синтезе S15 ингибирует трансляцию своей мРНК. Оперон содержит два промотора: P1 – перед геном rpsO, и слабый промотор P2 – между генами rpsO и pnp (рис. 1з). Кроме того, в участке между генами есть последовательность, образующая ρ-независимый терминатор, а в области ниже P2 – шпилька, содержащая участок узнавания РНКазой III (рис. 1з) [92]. При разрушении этой шпильки образуется дуплексная структура с неупорядоченной 3'-концевой частью, которая расщепляется самой полинуклеотидфосфорилазой, что приводит к дестабилизации мРНК pnp. Таким образом, полинуклеотидфосфорилаза регулирует свой синтез на посттранскрипционном уровне РНКаза III-зависимым путем [93].
Однодоменный белок S15 взаимодействует с 16S рРНК в двух местах (рис. 12а), играя основную роль в сборке центрального домена 30S субчастицы рибосомы [63]. Основной сайт связывания белка сформирован спиралями 20–22 16S рРНК и узлом GGC в месте их сочленения. Другой сайт представляет собой консервативный мотив G·U/G-C, расположенный на расстоянии одного витка спирали от места пересечения трех спиралей.
Рис. 12.
Схемы S15 -связывающих участков на РНК. а – Фрагмент рРНК E. coli, сайт 1 (GGC) отмечен красным, сайт 2 (G·U/G-C-мотив) – синим. б–д – Схемы S15 -связывающих участков мРНК E. coli (б), R. radiobacter (в), T. thermophilus (г), G. kaustophilus (д). Отмечены старт-кодон и последовательность SD. Синим цветом обведен G·U/G-C-мотив, красным – места пересечения трех спиралей, зеленым – дополнительные сайты связывания белка S15 на мРНК.
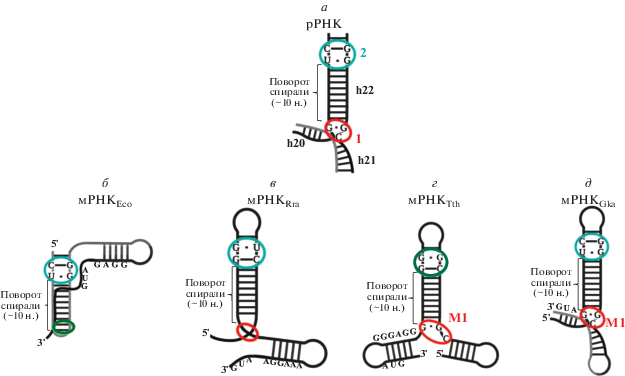
5'-НТО мРНК rpsO в отличие от S15-связывающего участка на рРНК имеет неконсервативную структуру. Различия между гомологами белка в E. coli (мРНКEco, EcoS15), Geobacillus kaustophilus (мРНКGka, GkaS15), T. thermophilus (мРНКTth, TthS15) и Rhizobium radiobacter (мРНКRra) обуславливают специфичность узнавания различных структур мРНК этих организмов (рис. 12б–д). Таким образом, даже консервативные РНК-связывающие белки разных бактерий могут иметь отличающиеся РНК-узнающие модули, что свидетельствует о коэволюции бактериальных гомологов белка S15 и специфических участков на мРНК. Не все мРНК rpsO регулируются любым из гомологов белка S15. Например, трансляция мРНКEco не меняется в ответ на добавление GkaS15, а мутации в мРНКGka и мРНКRra не влияют на взаимодействие с белком S15 других организмов [94].
S15-связывающий участок мРНК E. coli, как и рРНК, имеет два пространственно разнесенных сайта связывания. Основной сайт мРНКEco схож с G·U/G-C-мотивом 16S рРНК, другой сайт лишь немного напоминает место пересечения трех спиралей. В T. thermophilus, напротив, основной сайт связывания схож с участком сочленения трех спиралей 16S рРНК, а G·U/G-C-мотив имеет замену – G·G/G-C (рис. 12г). Сайты связывания мРНКGka с белком S15 сходны с обоими связывающими участками на 16S рРНК (рис. 12д). Четвертая разновидность – мРНКRra, имеет консервативный участок G·U/G-C и структуру, напоминающую тройной узел (рис. 12в) [94]. Введение мутаций в G·U/G-C-мотив у E. coli и G. kaustophilus и в сайт, напоминающий структуру тройного узла у T. thermophilus и R. radiobacter, приводит к инактивации регуляторной функции белка S15 [95]. Аминокислотные остатки EcoS15, которые узнают малый желобок h22 16S рРНК, участвуют и в узнавании соответствующей спирали псевдоузла мРНК [96].
В отсутствие белка S15 rpsO мРНК может принимать либо конформацию псевдоузла, либо двойной шпильки, но с рибосомой связывается только псевдоузел мРНК (рис. 13а). Ингибирование трансляции мРНК rpsO E. coli осуществляется при переходе тройственного комплекса (S15 · 30S · мРНК) в неактивную конформацию [97]. Участок псевдоузла мРНК взаимодействует с N-концевым доменом белка S2 на рибосоме, а участок перед псевдоузлом контактирует с h26 16S рРНК. При этом SD остается доступной для взаимодействия с рибосомой, так как находится внутри большой петли псевдоузла. Связываясь с рибосомой, S15 стабилизирует такое состояние мРНК, блокируя рибосому в преинициаторном состоянии и предотвращая переход инициаторного кодона в декодирующий центр (рис. 13а), что тормозит взаимодействие кодон–антикодон в P-сайте [98]. Это классический пример регуляции по принципу “ловушки”.
Рис. 13.
Иллюстрация механизма регуляции синтеза белка S15. a – Механизм “ловушки”, по которому осуществляется ингибирование трансляции мРНК rpsO у E. coli. б – Модель конкурентного механизма регуляции трансляции р-белка S15 T. thermophilus. мРНК rpsO либо связывается с TthS15 и разрушается, либо связывается с 30S субчастицей, образуя активный инициаторный комплекс.
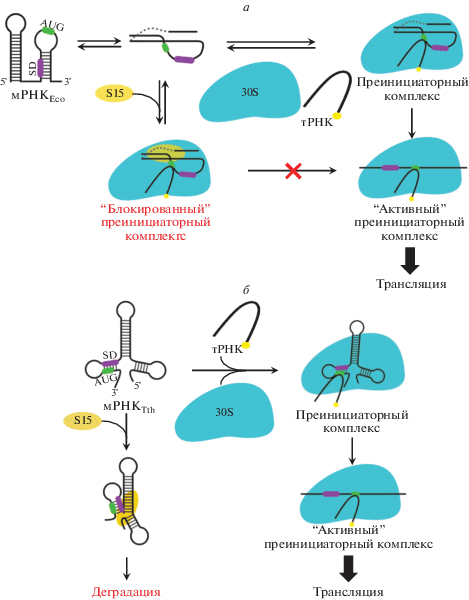
Регуляторный сайт S15 мРНК в T. thermophilus образован тремя спирально-петельными участками (рис. 12г), которые не имеют структурного сходства с псевдоузлом мРНК в E. coli (рис. 12б). При взаимодействии с белком TthS15 структура мРНК претерпевает конформационные изменения, в результате которых формируется структура мРНК, сходная с участком связывания белка на 16S рРНК. В результате участок связывания с 30S рибосомной субчастицей на мРНК становится недоступным (рис. 13б) [95], и трансляция ингибируется [99]. Сродство EcoS15 к своей мРНК на два порядка ниже, чем сродство TthS15 к своей мРНК. Считается, что регуляция трансляции мРНК rpsO в T. thermophilus проходит по конкурентному механизму, а не по механизму “ловушки”, как у E. coli.
S4/α-оперон
α-Oперон E. coli включает пять генов р-белков и ген α-субъединицы РНК-полимеразы (рис. 1и). Синтез белков оперона, кроме α-субъединицы РНК-полимеразы, регулирует белок S4 [100, 101]. S4 вместе с белком S7 инициирует сборку 30S рибосомной субчастицы [102, 103]. Он взаимодействует с 5'-концевым доменом 16S рРНК в месте пересечения пяти спиралей (h3, h4, h16–h18) [104–106]. Известно, что для образования функциональных комплексов белка S4 как с мРНК, так и с рРНК необходима лишь его N-концевая часть [107].
Структуры участков связывания белка S4 E. coli на мРНК и рРНК отличаются. Регуляторный участок на мРНК α-оперона представляет собой двойной псевдоузел (рис. 10a) [108], включающий 5'-НТО и начало кодирующего участка мРНК rpsM [109]. Спираль H1 мРНК почти не имеет консервативных нуклеотидов, для регуляции наиболее важна ее пространственная укладка (рис. 10a). Старт-кодон мРНК rpsM (GUG) в 4 раза менее эффективен, чем AUG, однако замена GUG на AUG снижает экспрессию генов α-оперона in vivo в 6 раз [110]. Видимо, инициация трансляции мРНК α-оперона, как и ее репрессия, зависит от структуры этого участка мРНК.
Механизм регуляции α-оперона по принципу “ловушки”, как и в случае S15-оперона, основан на конформационном переключении между двумя структурами псевдоузла мРНК и формировании “неактивного” преинициаторного комплекса (рис. 14б) [91, 109, 111]. Преинициаторный комплекс формируется только с “активной” конформацией псевдоузла мРНК. “Неактивная” конформация мРНК образует комплекс с 30S субчастицей, но не может связывать тРНКfMet. Белок S4 играет роль аллостерического репрессора, сдвигая равновесие между двумя конформациями мРНК в сторону “неактивной” формы [109, 112]. При регуляции трансляции по механизму “ловушки” нет необходимости в высокой аффинности репрессора к мРНК, сродство белка S4 E. coli к мРНК и рРНК примерно одинаково [113, 114].
Рис. 14.
Регуляция α-оперона. a – Вторичная структура S4-связывающего псевдоузла мРНК rpsM E. coli. Последовательности SD и старт-кодона выделены зеленым. Красным выделены консервативные нуклеотиды. б – Схема репрессии трансляции α-оперона E. coli белком S4.
У многих эубактерий ген белка S4 (rpsD) не входит в состав α-оперона. Так, ген rpsD B. subtilis представляет собой авторегулируемую транскрипционную единицу [115]. Вторичная структура S4-связывающего участка мРНК B. subtilis не имеет конформации псевдоузла [11], что свидетельствует о различиях в принципах узнавания белком S4 регуляторного участка у B. subtilis и у E. coli.
Необходимо отметить, что на ρ-зависимых терминаторах белок S4 выполняет такую же функцию, как и фактор транскрипции NusA при антитерминации транскрипции [116]. Таким образом, этот белок не только ингибирует трансляцию α-оперона, но и стимулирует транскрипцию рРНК, поддерживая сбалансированный синтез рРНК и р-белков.
РЕГУЛЯЦИЯ СИНТЕЗА РИБОСОМНЫХ БЕЛКОВ ПО МЕХАНИЗМУ РЕТРОРЕГУЛЯЦИИ
Механизм ретрорегуляции подразумевает деградацию мРНК рибонуклеазами в результате взаимодействия белка-репрессора с дистальным участком мРНК. В качестве хорошо изученного примера такой регуляции можно привести ингибирование синтеза белков L14–L24 S8/spc-оперона.
S8/spc-оперон
S8/spc-оперон E. coli состоит из 12 генов, экспрессия 10 из которых ингибируется при связывании р-белка S8 с межцистронным участком мРНК, включающим старт-кодон гена rplE (рис. 1к).
Консервативный двухдоменный белок S8 играет важную роль в сборке 30S субчастицы рибосомы, связываясь, в основном, со спиралью 21 16S рРНК [48]. Участки связывания белка S8 на мРНК и 16S рРНК E. coli очень похожи [117, 118] (рис. 15а, б), но сродство к мРНК в 5 раз ниже, чем к специфическому фрагменту 16S рРНК [117].
Рис. 15.
Регуляция S8/spc-оперона. а, б – Вторичные структуры участков связывания белка S8 на мРНК (а) и рРНК (б) E. сoli. Рамкой выделен участок, с которым взаимодействует белок S8. SD и cтарт-кодон мРНК белка L5 выделены зеленым. Красным показаны консервативные нуклеотиды мРНК. в – Модель “ретрорегуляции” синтеза белков L14 и L24 белком S8 (показана часть S8/spc-оперона).
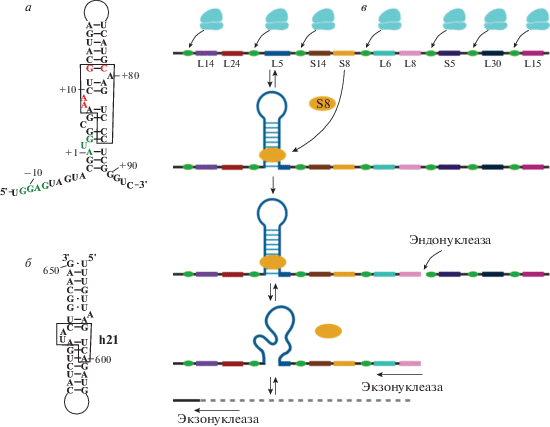
Регуляторный участок расположен в 5'-концевой части мРНК белка L5 (рис. 15в) [119, 120]. В регуляторном комплексе с белком взаимодействует преимущественно внутренняя петля спирали мРНК [117]. Неспаренные A8 и А9 и пара G12–C79 мРНК (рис. 15а) консервативны [11]. Контакты белка S8 с внутренней петлей специфического фрагмента мРНК аналогичны контактам с 16S рРНК [48, 117].
Связываясь с мРНК в начале гена белка L5, белок S8 напрямую блокирует его трансляцию. Нарушение трансляционного сопряжения приводит к ингибированию трансляции последующих цистронов белков (рис. 15в) [121]. Однако лишь у трех генов оперона отсутствуют SD, и некоторые из них располагаются с 3'-конца от места связывания S8 (рис. 15в). Предложен механизм регуляции экспрессии генов spc-оперона с участием эндонуклеаз и последующей деградацией мРНК экзонуклеазами. Согласно этой модели после действия эндонуклеазы (например, РНКазы III) происходит деградация мРНК L14–L24 3'-, 5'-экзонуклеазами (полинуклеотидфосфорилазой и/или РНКазой II) (рис. 15в). Таким образом, ингибирование трансляции цистронов белков L14 и L24 происходит по механизму ретрорегуляции, что приводит к дестабилизации мРНК [122]. О регуляции трансляции самых удаленных цистронов secY и rpmJ (рис. 1к) сведений нет.
S7/str-оперон
S7/str-оперон E. coli состоит из четырех генов, кодирующих р-белки S12, S7 и факторы элонгации EF-G и EF-Tu (рис. 1л). Белок S7 ингибирует синтез р-белков S12 и S7 [123] и EF-G, причем, трансляция мРНК белков S7 и S12 сопряжена [124]. Белок S7 E. coli инициирует сборку 30S рибосомной субчастицы, организуя сворачивание 3'-концевого домена 16S рРНК и способствуя связыванию других р-белков, формирующих головку 30S субчастицы [103]. S7 взаимодействует с коротким двухпетлевым фрагментом 16S рРНК в нижней части 3'-концевого домена (рис. 16б) [125].
Рис. 16.
Вторичные структуры S7-связывающих фрагментов мРНК (а) и рРНК (б). Идентичные последовательности, необходимые для связывания, выделены рамками. Стоп-кодон белка S12 (UAA) на фрагменте мРНК выделен серым, последовательность SD и инициаторный кодон S7 – зеленым.
Участок связывания белка на мРНК S7/str-оперона E. coli находится между генами rpsL и rpsG [123] и содержит нерегулярную шпильку (рис. 16а). Минимальный фрагмент мРНК, который сохраняет сродство к S7, включает межцистронную область мРНК str. Вторичные структуры сайтов связывания белка на мРНК и рРНК различаются, при этом обе РНК содержат два идентичных S7-связывающих участка (рис. 16). Первый участок – спираль 42 16S рРНК и трехспиральное сочленение мРНК str [124], второй – петли В и А рРНК и нижняя часть спирали III мРНК [126].
При взаимодействии с белком S7 спираль V мРНК дестабилизируется и репрессируется трансляция мРНК белков S7 и EF-G, трансляция цистрона S12 может ингибироваться по механизму ретрорегуляции [124, 127].
Структура участка мРНК S7/str-оперона не консервативна. S7/str-оперон T. thermophilus не содержит протяженного участка между генами rpsL и rpsG, а у Cyanobacteria эта последовательность больше похожа на специфический участок 16S рРНК [11].
Авторегуляция S20-оперона
Механизм авторегуляции S20-оперона к настоящему времени не установлен, и данные о конкретном участке связывания белка S20 на мРНК отсутствуют. Показано, что при дефиците аминокислот имеет место ppGpp-зависимая регуляция синтеза белка S20, причем для этой регуляции необходима лидерная последовательность мРНК [128].
Белок S20 – один из шести первично-связывающихся с 16S рРНК белков 30S субчастицы рибосомы. S20 взаимодействует с двумя участками 16S рРНК: спиралями 9, 11, 13 и спиралью 44, соединяя таким образом 5'-домен и 3'-минорный домен 16S рРНК [129]. Удаление белка S20 приводит к снижению скорости и эффективности связывания мРНК с рибосомой, а также к нарушениям сборки 30S субчастицы [130].
Синтез р-белка S20 авторегулируется на постранскрипционном уровне (рис. 1м) [131]. Предполагается, что S20 связывается с участком в 5'‑НТО своей мРНК, блокируя SD и инициаторный кодон. Стоит отметить, что инициаторным кодоном гена rpsT, кодирующего р-белок S20, является последовательность UUG [132]. Минимальный участок, необходимый для эффективной регуляции синтеза S20, – инициаторный кодон UUG мРНК rpsT и окружающие его нуклеотиды. Замена UUG на AUG приводит к снижению ингибирующего эффекта белка S20 [133].
Несмотря на информацию о способности белка S20 ингибировать свой синтез как in vivo, так и in vitro, данных о его взаимодействии с фрагментами rpsT мРНК получить не удалось [133, 134]. Предполагается, что для регуляции синтеза белка S20 необходимо его взаимодействие не только с участком своей мРНК, а с преинициаторным комплексом мРНК-30S субчастица [133].
ЗАКЛЮЧЕНИЕ
Синтез р-белков регулируется посредством двух довольно близких механизмов – конкуренции между участками связывания белка на рРНК и мРНК и механизму “ловушки”. При конкурентном ингибировании трансляции участок связывания репрессора на мРНК может быть похож на участок связывания этого белка на рРНК (белок L1), а может и не иметь практически никакой гомологии (белок S2). Регуляция синтеза белков по механизму “ловушки” (S15-оперон и S4/α-оперон) осуществляется за счет блокировки рибосомы в преинициаторном состоянии при связывании белка-регулятора. Причем регуляторные участки связывания как белка S4, так и S15 на мРНК E. coli имеют структуру псевдоузла. Регуляторная область мРНК L20-оперона E. coli тоже имеет структуру псевдоузла, однако, регуляция синтеза белков этого оперона проходит не по механизму “ловушки”, а по механизму конкуренции между репрессором и рибосомой за связывание с мРНК. Несмотря на то, что механизм регуляции по принципу “ловушки” более эффективен, чем механизм конкуренции, поскольку он может функционировать, даже если концентрация репрессора или его сродство к мРНК малы, регуляция трансляции большинства бактериальных мРНК подчиняется простому механизму конкуренции между репрессором и 30S субчастицей за взаимодействие с мРНК. При экспрессии генов р-белков может использоваться также механизм ретрорегуляции, подразумевающий дестабилизацию мРНК при взаимодействии с белком-репрессором (регуляция синтеза белков L14 и L24 белком S8 и, вероятно, синтеза белка S12 белком S7).
Несмотря на консервативность свойств рибосомных белков-регуляторов участки их связывания на мРНК и механизмы регуляции синтеза белков могут отличаться у разных организмов. Так, белок S15 T. thermophilus регулирует трансляцию S15-оперона по конкурентному типу, а не по механизму “ловушки”, как у E. coli. У B. subtilis и некоторых других бактерий участки связывания р-белков L20, S15, S7 и S4 на мРНК отличаются от регуляторных участков этих белков у E. coli. Экспрессия генов L10-оперона B. subtilis может регулироваться не только посредством механизма конкуренции рибосомы с белковым комплексом за связывание с мРНК, как у E. coli, но и путем антитерминации.
Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований в рамках научного проекта № 19-14-50124.
Настоящая статья не содержит каких-либо исследований, выполненных с использованием биологических материалов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Zengel J.M., Lindahl L. (1994) Diverse mechanisms for regulating ribosomal protein synthesis in Escherichia coli. Prog. Nucl. Acids Res. Mol. Biol. 47, 331–370.
Zengel J.M., Mueckl D., Lindahl L. (1980) Protein L4 of the E. coli ribosome regulates an eleven gene r protein operon. Cell. 21(2), 523–535.
Schuwirth B.S., Borovinskaya M.A., Hau C.W., Zhang W., Vila-Sanjurjo A., Holton J.M., Cate J.H. (2005) Structures of the bacterial ribosome at 3.5 A resolution. Science. 310(5749), 827–834.
Li X., Lindahl L., Zengel J.M. (1996) Ribosomal protein L4 from Escherichia coli utilizes nonidentical determinants for its structural and regulatory functions. RNA. 2(1), 24–37.
Stelzl U., Zengel J.M., Tovbina M., Walker M., Nierhaus K.H., Lindahl L., Patel D.J. (2003) RNA-structural mimicry in Escherichia coli ribosomal protein L4-dependent regulation of the S10 operon. J. Biol. Chem. 278(30), 28237–28245.
Stelzl U., Nierhaus K.H. (2001) A short fragment of 23S rRNA containing the binding sites for two ribosomal proteins, L24 and L4, is a key element for rRNA folding during early assembly. RNA. 7(4), 598–609.
Freedman L.P., Zengel J.M., Archer R.H., Lindahl L. (1987) Autogenous control of the S10 ribosomal protein operon of Escherichia coli: genetic dissection of transcriptional and posttranscriptional regulation. Proc. Natl. Acad. Sci. USA. 84(18), 6516–6520.
Zengel J.M., Lindahl L. (1990) Escherichia coli ribosomal protein L4 stimulates transcription termination at a specific site in the leader of the S10 operon independent of L4-mediated inhibition of translation. J. Mol. Biol. 213(1), 67–78.
Zengel J.M., Lindahl L. (1996) A hairpin structure upstream of the terminator hairpin required for ribosomal protein L4-mediated attenuation control of the S10 operon of Escherichia coli J. Bacteriol. 178(8), 2383–2387.
Sha Y., Lindahl L., Zengel J.M. (1995) RNA determinants required for L4-mediated attenuation control of the S10 r-protein operon of Escherichia coli. J. Mol. Biol. 245(5), 486–498.
Fu Y., Deiorio-Haggar K., Anthony J., Meyer M.M. (2013) Most RNAs regulating ribosomal protein biosynthesis in Escherichia coli are narrowly distributed to Gammaproteobacteria. Nucl. Acids Res. 41(6), 3491–3503.
Zengel J.M., Sha Y., Lindahl L. (2002) Surprising flexibility of leader RNA determinants for r-protein L4-mediated transcription termination in the Escherichia coli S10 operon. RNA. 8(5), 572–578.
Zengel J.M., Lindahl L. (1990) Ribosomal protein L4 stimulates in vitro termination of transcription at a NusA-dependent terminator in the S10 operon leader. Proc. Natl. Acad. Sci. USA. 87(7), 2675–2679.
Zengel J.M., Lindahl L. (1992) Ribosomal protein L4 and transcription factor NusA have separable roles in mediating terminating of transcription within the leader of the S10 operon of Escherichia coli. Genes Dev. 6(12B), 2655–2662.
Sha Y., Lindahl L., Zengel J.M. (1995) Role of NusA in L4-mediated attenuation control of the S10 r-protein operon of Escherichia coli. J. Mol. Biol. 245(5), 474–485.
Михайлина А.О., Костарева О.С., Сарских А.В., Федоров Р.В., Пиндл В., Гарбер М.Б., Тищенко С.В. (2014) Исследование регуляторных свойств архейного рибосомного белка L4. Биохимия. 79(1), 87–95.
Williams K.P. (2008) Strong mimicry of an rRNA binding site for two proteins by the mRNA encoding both proteins. RNA Biol. 5(3), 145–148.
Yates J.L., Arfsten A.E., Nomura M. (1980) In vitro expression of Escherichia coli ribosomal protein genes: autogenous inhibition of translation. Proc. Natl. Acad. Sci. USA. 77(4), 1837–1841.
Dean D., Nomura M. (1980) Feedback regulation of ribosomal protein gene expression in Escherichia coli. Proc. Natl. Acad. Sci. USA. 77(6), 3590–3594.
Brot N., Caldwell P., Weissbach H. (1981) Regulation of synthesis of Escherichia coli ribosomal proteins L1 and L11. Arch. Biochem. Biophys. 206(1), 51–53.
Kohrer C., Mayer C., Neumair O., Grobner P., Piendl W. (1998) Interaction of ribosomal L1 proteins from mesophilic and thermophilic archaea and bacteria with specific L1-binding sites on 23S rRNA and mRNA. Eur. J. Biochem. 256(1), 97–105.
Hanner M., Mayer C., Köhrer C., Golderer G., Gröbner P., Piendl W. (1994) Autogenous translational regulation of the ribosomal MvaL1 operon in the archaebacterium Methanococcus vannielii. J. Bacteriol. 176(2), 409–418.
Yates J.L., Dean D., Strycharz W.A., Nomura M. (1981) E. coli ribosomal protein L10 inhibits translation of L10 and L7/L12 mRNAs by acting at a single site. Nature. 294(5837), 190–192.
Baughman G., Nomura M. (1983) Localization of the target site for translational regulation of the L11 operon and direct evidence for translational coupling in Escherichia coli. Cell. 34(3), 979–988.
Mayer C., Kohrer C., Grobner P., Piendel W. (1998) MvaL1 autoregulates the synthesis of the three ribosomal proteins encoded on the MvaL1 operon of the archaeon Methanococcus vannielii by inhibiting its own translation before or at the formation of the first peptide bond. Mol. Microbiol. 27(2), 455–468.
Михайлина А.О., Костарева О.С., Никонова Е.Ю., Гарбер М.Б., Тищенко С.В. (2018) Идентификация сайтов связывания рибосомного белка L1 на мРНК Thermus thermophilus и Thermotoga maritima. Молекуляр. биология. 52(1), 98–105.
Zimmermann R.A., Thurlow D.L., Finn R.S., Marsch T.L., Ferret L.K. (1980) Genetics and evolution of RNA polymerase, tRNA and ribosomes. Univ. Tokyo Press, Tokyo. 1, 569–584.
Gourse R.L., Thurlow D.L., Gerbi S.A., Zimmermenn R.A. (1981) Specific binding of a procaryot ribosomal protein to an eukaryotic ribosomal RNA: Implications for evolution and autoregulation. Proc. Natl. Acad. Sci. USA. 78(5), 2722–2726.
Draper D.E. (1989) How do proteins recognize specific RNA sites? New clues from autogenously regulated ribosomal proteins. Trends Biochem. Sci. 14(8), 335–338.
Baier G., Hohenwarter O., Hofbauer C., Hummel H., Stoffler-Mailicke M., Stoffler G. (1989) Structural and functional equivalence between ribosomal proteins of Escherichia coli L1 and Methanococcus vannielii L6. Syst. Appl. Microbiol. 12, 119–126.
Korepanov A.P., Kostareva O.S., Bazhenova M.V., Bubunenko M.G., Garber M.B., Tishchenko S.V. (2015) Studying the properties of domain I of the ribosomal protein L1: incorporation into ribosome and regulation of the L1 operon expression. Protein J. 34(2), 103–110.
Nevskaya N., Tishchenko S., Volchkov S., Kljastorny V., Nikonova E., Nikonov O., Nikulin A., Kohrer C., Piendl W., Zimmermann R., Stockley, P., Garber, M., Nikonov S. (2006) New insights into the interaction of ribosomal protein L1 with RNA. J. Mol. Biol. 355(4), 747–759.
Tishchenko S., Gabdulkhakov A., Nevskaya N., Sarskikh A., Kostareva O., Nikonova E., Sycheva A., Moshkovskii S., Garber M., Nikonov S. (2012) High-resolution crystal structure of the isolated ribosomal L1 stalk. Acta Crystallogr. D. 68(8), 1051–1057.
Gabdulkhakov A., Tishchenko S., Mikhaylina A., Garber M., Nevskaya N., Nikonov S. (2017) Crystal structure of the 23S rRNA fragment specific to r-protein L1 and designed model of the ribosomal L1 stalk from Haloarcula marismortui. Crystals. 7(2), 37.
Boni I.V., Artamonova V.S., Tzareva N.V., Dreyfus M. (2001) Non-canonical mechanism for translational control in bacteria: synthesis of ribosomal protein S1. EMBO J. 20(15), 4222–4232.
Boni I.V., Artamonova V.S., Dreyfus M. (2000) The last RNA binding repeat of the Escherichia coli ribosomal protein S1 is specifically involved in autogenous control. J. Bacteriol. 182(20), 5872–5879.
Sengupta J., Agrawal R.K., Frank J. (2001) Visualization of protein S1 within the 30S ribosomal subunit and its interaction with messenger RNA. Proc. Natl. Acad. Sci. USA. 98(21), 11991–11996.
Byrgazov K., Manoharadas S., Kaberdina A.C., Vesper O., Moll I. (2012) Direct interaction of the N-terminal domain of ribosomal protein S1 with protein S2 in Escherichia coli. PLoS One. 7, e32702.https://doi.org/10.1371/journal.pone.0032702
Sukhodolets M.V., Garges S., Adhya S. (2006) Ribosomal protein S1 promotes transcriptional cycling. RNA. 12(8), 1505–1513.
Demo G., Rasouly A., Vasilyev N., Svetlov V., Loveland A.B., Diaz-Avalos R., Grigorieff N., Nudler E., Korostelev A.A. (2017) Structure of RNA polymerase bound to ribosomal 30S subunit. eLife. 6, e28560. https://doi.org/10.7554/eLife.28560
Sorensen M.A., Fricke J., Pedersen S. (1998) Ribosomal protein S1 is required for translation of most, if not all, natural mRNAs in Escherichia coli in vivo. J. Mol. Biol. 280(4), 561–569.
Duval M., Korepanov A., Fuchsbauer O., Fechter P., Haller A., Fabbretti A., Choulier L., Micura R., Klaholz B.P., Romby P. (2013) Escherichia coli ribosomal protein S1 unfolds structured mRNAs onto the ribosome for active translation initiation. PLoS Biol. 11(12), e1001731.
Lu Y., Lim L., Song J. (2017) NMR studies reveal that protein dynamics critically mediate aggregation of the well-folded and very soluble E. coli S1 ribosomal protein. bioRXiv. https://doi.org/10.1101/178459
Hajnsdorf E., Boni I.V. (2012) Multiple activities of RNA-binding proteins S1 and Hfq. Biochimie. 94(7), 1544–1553.
Dunn J.J., Buzash-Pollert E., Studier F.W. (1978) Mutations of bacteriophage T7 that affect initiation of synthesis of the gene 0.3 protein. Proc. Natl. Acad. Sci. USA. 75(6), 2741–2745.
Aseev L.V., Levandovskaya A.A., Tchufistova L.S., Skaptsova N.V., Boni I.V. (2008) A new regulatory circuit in ribosomal protein operons: S2-mediated control of the rpsB’tsf expression in vivo. RNA. 14(9), 1882–1894.
Yusupova G., Jenner L., Rees B., Moras D., Yusupov M. (2006) Structural basis for messenger RNA movement on the ribosome. Nature. 444(7117), 391–394.
Brodersen D.E., Clemons Jr., W.M., Carter A.P., Wimberly B.T., Ramakrishnan V. (2002) Crystal structure of the 30S ribosomal subunit from Thermus thermophilus: Structure of the proteins and their interactions with 16S RNA. J. Mol. Biol. 316(3), 725–768.
Асеев Л.В., Колединская Л.С., Бони И.В. (2014) Анализ активности и регуляции удлиненного “–10” rpsB-промотора Escherichia coli in vivo. Биохимия. 79(8), 974–984.
An G., Bendiak D.S., Mamelak L.A., Friesen J.D. (1981) Organization and nucleotide sequence of a new ribosomal operon in Escherichia coli containing the genes for ribosomal protein S2 and elongation factor Ts. Nucl. Acids Res. 9(16), 4163–4172.
Bendiak D.S., Friesen J.D. (1981) Organization of genes in the four minute region of the Escherichia coli chromosome: evidence that rpsB and tsf are co-transcribed. Mol. Gen. Genet. 181(3), 356–362.
Merino E., Yanofsky C. (2005) Regulation by termination-antitermination: a genomic approach. Trends Genet. 21(5), 260–264.
Lesage P., Chiaruttini C., Graffe M., Dondon J., Milet M., Springer M. (1992) Messenger RNA secondary structure and translational coupling in the Escherichia coli operon encoding translation initiation factor IF3 and the ribosomal proteins, L35 and L20. J. Mol. Biol. 228(2), 366–386.
Spillmann S., Dohme F., Nierhaus K.H. (1977) Assembly in vitro of the 50S subunit from Escherichia coli ribosomes: proteins essential for the first heat dependent conformational change. J. Mol. Biol. 115(3), 513–523.
Chiaruttini C., Milet M., Springer M. (1996) A long-range RNA-RNA interaction forms a pseudoknot required for translational control of the IF3-L35-L20 ribosomal protein operon in Escherichia coli. EMBO J. 15(16), 4402–4413.
Allemand F., Haentjens J., Chiaruttini C., Royer C., Springer M. (2007) Escherichia coli ribosomal protein L20 binds as a single monomer to its own mRNA bearing two potential binding sites. Nucl. Acids Res. 35(9), 3016–3031.
Harms J., Schluenzen F., Zarivach R., Bashan A., Gat S., Agmon I., Bartels H., Franceschi F., Yonath A. (2001) High resolution structure of the large ribosomal subunit from a mesophilic eubacterium. Cell. 107(5), 679–688.
Guillier M., Allemand F., Raibaud S., Dardel F., Springer M., Chiaruttini C. (2002) Translational feedback regulation of the gene for L35 in Escherichia coli requires binding of ribosomal protein L20 to two sites in its leader mRNA: a possible case of ribosomal RNA-messenger RNA molecular mimicry. RNA. 8(7), 878–889.
Haentjens-Sitri J., Allemand F., Springer M., Chiaruttini C. (2008) A competition mechanism regulates the translation of the Escherichia coli operon encoding ribosomal proteins L35 and L20. J. Mol. Biol. 375(3), 612–625.
Choonee N., Even S., Zig L., Putzer H. (2007) Ribosomal protein L20 controls expression of the Bacillus subtilis infC operon via a transcription attenuation mechanism. Nucl. Acids Res. 35(5), 1578–1588.
Isono K., Kitakawa M. (1978) Cluster of ribosomal protein genes in Escherichia coli containing gene for proteins S6, S18, and L9. Proc. Natl. Acad. Sci. USA. 75(12), 6163–6167.
Held W., Ballou B., Mizushima S., Nomura M. (1974) Assembly mapping of 30 S ribosomal proteins from Escherichia coli. J. Biol. Chem. 249, 3103–3111.
Agalarov S.C., Sridhar Prasad G., Funke P.M., Stout C.D., Williamson J.R. (2000) Structure of the S15, S6, S18-rRNA complex: assembly of the 30S ribosome central domain. Science. 288(5463), 107–113.
Recht M.I., Williamson J.R. (2001) Central domain assembly: thermodynamics and kinetics of S6 and S18 binding to an S15-RNA complex. J. Mol. Biol. 313(1), 35–48.
Matelska D., Purta E., Panek S., Boniecki M.J., Bujnicki J.M., Dunin-Horkawicz S. (2013) S6:S18 ribosomal protein complex interacts with a structural motif present in its own mRNA. RNA. 19(10), 1341–1348.
Fu Y., Deiorio-Haggar K., Soo M.W., Meyer M.M. (2014) Bacterial RNA motif in the 5′ UTR of rpsF interacts with an S6:S18 complex. RNA. 20(2), 168–176.
Babina A.M., Soo M.W., Fu Y., Meyer M.M. (2015) An S6:S18 complex inhibits translation of E. coli rpsF. RNA. 21(12), 2039–2046.
Dennis P.P., Fiil N.P. (1979) Transcriptional and post-transcriptional control of RNA polymerase and ribosomal protein genes cloned on composite ColE1 plasmids in the bacterium Escherichia coli. J. Biol. Chem. 254(16), 7540–7547.
Fukuda R. (1980) Autogenous regulation of the synthesis of ribosomal proteins, L10 and L7/12, in Escherichia coli. Mol. Gen. Genet. 178(2), 483–486.
Brot N., Caldwell P., Weissbach H. (1980) Autogenous control of Escherichia coli ribosomal protein L10 synthesis in vitro. Proc. Natl. Acad. Sci. USA. 77(5), 2592–2595.
Holowachuk E.W., Friesen J.D., Fiil N.P. (1980) Bacteriophage lambda vehicle for the direct cloning of Escherichia coli promoter DNA sequences: feedback regulation of the rplJL-rpoBC operon. Proc. Natl. Acad. Sci. USA. 77(4), 2124–2128.
Friesen J.I., Tropak M., An C. (1983) Mutations in the rplJ leader of Escherichia coli that abolish feedback regulation. Cell. 32(2), 361–369.
Shimmin L.C., Dennis P.P. (1989) Characterization of the L11, L1, L10 and L12 equivalent ribosomal protein gene cluster of the halophilic archaebacterium Halobacterium cutirubrum. EMBO J. 8(4), 1252–1235.
Johnsen M., Christensen T., Dennis P.P., Fiil N.P. (1982) Autogenous control: ribosomal protein L10-L12 complex binds to the leader sequence of its mRNA. EMBO J. 1(8), 999–1004.
Diaconu M., Kothe U., Schlünzen F., Fischer N., Harms J.M., Tonevitsky, Stark H., Rodnina M.V., Wahl M.C. (2005) Structural basis for the function of the ribosomal L7/L12 stalk in factor binding and GTPase activation. Cell. 121(7), 991–1004.
Gordiyenko Y., Videler H., Zhou M., McKay A.R., Fucini P., Biegel E., Müller V., Robinson C.V. (2010) Mass spectrometry defines the stoichiometry of ribosomal stalk complexes across the phylogenetic tree. Mol. Cell Prot. 9(8), 1774–1783.
Ilag L.L., Videler H., McKay A.R., Sobott F., Fucini P., Nierhaus K.H., Robinson, C.V. (2005) Heptameric (L12)6/L10 rather than canonical pentameric complexes are found by tandem MS of intact ribosomes from thermophilic bacteria. Proc. Natl. Acad. Sci. USA. 102(23), 8192–8197.
Klein D.J., Schmeing T.M., Moore P.B., Steitz T.A. (2001) The kink-turn: A new RNA secondary structure motif. EMBO J. 20(15), 4214–4221.
Iben J.R., Draper D.E. (2008) Specific interactions of the L10(L12)4 ribosomal protein complex with mRNA, rRNA, and L11. Biochemistry. 47(9), 2721–2731.
Climie S.C., Friesen J.D. (1987) Feedback regulation of the rplJL-rpoBC ribosomal protein operon of Escherichia coli requires a region of mRNA secondary structure. J. Mol. Biol. 198(3), 371–381.
Babitzke P., Baker C.S., Romeo T. (2009) Regulation of translation initiation by RNA binding proteins. Annu. Rev. Microbiol. 63, 27–44.
Peattie D.A., Douthwaite S., Garrett R.A., Noller H.F. (1981) A “bulged” double helix in a RNA-protein contact site. Proc. Natl. Acad. Sci. USA. 7(6), 1697–1701.
Douthwaite S., Christensen A., Garrett R.A. (1982) Binding site of ribosomal proteins on prokaryotic 5S ribonucleic acids: a study with ribonucleases. Biochemistry. 21(10), 2313–2320.
Thurlow D.L., Ehresmann C., Ehresmann B. (1983) Nucleotides in 16S rRNA that are required in unmodified form for features recognized by ribosomal protein S8. Nucl. Acids Res. 11(19), 6787–6802.
Gregory R.J., Zeller M.L., Thurlow D.L., Gourse R.L., Stark M. J.R., Dahlberg A.E., Zimmermann R.A. (1984) Interaction of ribosomal proteins S6, S8, S15 and S18 with the central domain of 16 S ribosomal RNA from Escherichia coli. J. Mol. Biol. 178(2), 287–302.
Stern S., Wilson R.C., Noller H.F. (1986) Localization of the binding site for protein S4 on 16S ribosomal RNA by chemical and enzymatic probing and primer extension. J. Mol. Biol. 192(1), 101–110.
Climie S.C., Friesen J.D. (1988) In vivo and in vitro structural analysis of the rplJ mRNA leader of Escherichia coli. Protection by bound L10-L7/L12. J. Biol. Chem. 263(29), 15166–15175.
Christensen T., Johnsen M., Fiil N.P., Friesen J.D. (1984) RNA secondary structure and translation inhibition: analysis of mutants in the rplJ leader. EMBO J. 3(7), 1609–1612.
Yakhnin H., Yakhnin A.V., Babitzke P. (2015) Ribosomal protein L10(L12)4 autoregulates expression of the Bacillus subtilis rplJL operon by a transcription attenuation mechanism. Nucl. Acids Res. 43(14), 7032–7043.
Merino E., Yanofsky C. (2002) Regulation by termination-antitermination: a genomic approach. Bacillus subtilis and its closest relatives: from genes to cells. Washington DC: ASM Press, 1, 323–336.
Draper D.E., Gluick T.C., Schlax P.J. (1998) Pseudoknots, RNA folding, and translational regulation. In RNA Structure and Function. Cold Spring Harbor, NY: Cold Spring Harbor Lab. Press, 1, 415–436.
Régnier P., Portier C. (1986) Initiation, attenuation and RNase III processing of transcripts from the Escherichia coli operon encoding ribosomal protein S15 and polynucleotide phosphorylase. J. Mol. Biol. 187(1), 23–32.
Jarrige A.C., Mathy N., Portier C. (2001) PNPase autocontrols its expression by degrading a double-stranded structure in the pnp mRNA leader. EMBO J. 20(23), 6845–6855.
Slinger B.L., Newman H., Lee Y., Pei S., Meyer M.M. (2015) Co-evolution of bacterial ribosomal protein S15 with diverse mRNA regulatory structures. PLoS Genet. 11(12), e1005720.
Serganov A., Polonskaia A., Ehresmann B., Ehresmann C., Patel D.J. (2003) Ribosomal protein S15 represses its own translation via adaptation of an rRNA-like fold within its mRNA. EMBO J. 22(8), 1898–1908.
Mathy N., Pellegrini O., Serganov A., Patel D.J., Ehresmann C., Portier C. (2004) Specific recognition of rpsO mRNA and 16S rRNA by Escherichia coli ribosomal protein S15 relies on both mimicry and site differentiation. Mol. Microbiol. 52(3), 661–675.
Philippe C., Eyermann F., Bénard L., Portier C., Ehresmann B., Ehresmann C. (1993) Ribosomal protein S15 from Escherichia coli modulates its own translation by trapping the ribosome on the mRNA initiation loading site. Proc. Natl. Acad. Sci. USA. 90(10), 4394–4398.
Marzi S., Myasnikov A.G., Serganov A., Ehresmann C., Romby P., Yusupov M., Klaholz B.P. (2007) Structured mRNAs regulate translation initiation by binding to the platform of the ribosome. Cell. 130(6), 1019–1031.
Ehresmann C., Ehresmann B., Ennifar E., Dumas P., Garber M., Mathy N., Nikulin A., Portier C., Patel D., Serganov A. (2004) Molecular mimicry in translational regulation: the case of ribosomal protein S15. RNA Biol. 1(1), 66–73.
Jinks-Robertson S., Nomura M. (1982) Ribosomal protein S4 acts in trans as a translational repressor to regulate expression of the alpha operon in Escherichia coli. Bacteriol. 151(1), 193–202.
Thomas M., Bedwell D.M., Nomura M. (1987) Regulation of alpha operon gene expression in Escherichia coli. A novel form of translational coupling. J. Mol. Biol. 196(2), 333–345.
Nomura M., Held W.A. (1974) Reconstitution of ribosomes: Studies of ribosome structure, function and assembly. In Ribosomes. Cold Spring Harbor, New York: Cold Spring Harbor Lab. Press, 1, 193–224.
Nowotny V., Nierhaus K.H. (1988) Assembly of the 30S subunit from Escherichia coli ribosomes occurs via two assembly domains which are initiated by S4 and S7. Biochemistry. 27(18), 7051–7055.
Schluenzen F., Tocilj A., Zarivach R., Harms J., Gluehmann M., Janell D., Bashan A., Bartels H., Agmon I., Franceschi F., Yonath, A. (2000) Structure of functionally activated small ribosomal subunit at 3.3 Å resolution. Cell. 102(5), 615–623.
Wimberly B.T., Brodersen D.E., Clemons Jr. W.M., Morgan-Warren R.J., Carter A.P., Vonrhein C., Hartsch T., Ramakrishnan V. (2000) Structure of the 30S ribosomal subunit. Nature. 407(6802), 327–339.
Bellur D.L., Woodson S.A. (2009) A minimized rRNA binding site for ribosomal protein S4 and its implications for 30S assembly. Nucl. Acids Res. 37(6), 1886–1896.
Baker A., Draper D. (1995) Messenger RNA recognition by fragments of ribosomal protein S4. J. Biol. Chem. 270(39), 22939–22945.
Tang C.K., Draper D.E. (1989) Unusual mRNA pseudoknot structure is recognized by a protein translational repressor. Cell. 57(4), 531–536.
Spedding G.S., Gluick T.C., Draper D.E. (1993) Ribosome initiation complex formation with the pseudoknotted alpha operon messenger RNA. J. Mol. Biol. 229(3), 609–622.
Tang C.K., Draper D.E. (1990) Evidence for allosteric coupling between the ribosome and repressor binding sites of a translationally regulated mRNA. Biochemistry. 29(18), 4434–4439.
Schlax P.J., Xavier K.A., Gluick T.C., Draper D.E. (2001) Translational repression of the Escherichia coli alpha operon mRNA: importance of an mRNA conformational switch and a ternary entrapment complex. J. Biol. Chem. 276(42), 38494–38501.
Spedding G.S., Draper D.E. (1993) Allosteric mechanism for translational repression in the Escherichia coli alpha operon. Proc. Natl. Acad. Sci. USA. 90(10), 4399–4403.
Deckman I.C., Draper D.E., Thomas M.S. (1987) S4-alpha mRNA translation repression complex. I. Thermodynamics of formation. J. Mol. Biol. 196(2), 313–322.
Vartikar J.V., Draper D.E. (1989) S4-16 S ribosomal RNA complex. Binding constant measurements and specific recognition of a 460-nucleotide region. J. Mol. Biol. 209(2), 221–234.
Grundy F.J., Henkin T.M. (1991) The rpsD gene, encoding ribosomal protein S4 is autogeneously regulated in Bacillus subtilis. J. Bacteriol. 173(15), 4595–4602.
Torres M., Condon C., Balada J.M., Squires C., Squires C.L. (2001) Ribosomal protein S4 is a transcription factor with properties remarkably similar to NusA, a protein involved in both nonribosomal and ribosomal RNA termination. EMBO J. 20(14), 3811–3820.
Merianos H.J., Wang J., Moore P.B. (2004) The structure of a ribosomal protein S8/spc-operon mRNA complex. RNA. 10(6), 954–964.
Zimmermann R.A., Alimov A., Uma K., Wu H., Wower I., Nikonowicz E.P., Drygin D., Dong P., Jiang L. (2000) How proteins and RNA recognize one another. In The Ribosome: Structure, Function, Antibiotics, and Cellular Interactions. Eds Garrett R.A., Dowthwaite S.A., Liljas A., Marbeson A.T., Moore P.B., Noller H. Washington DC.: ASM Press, 1, 93–104.
Cerretti D.P., Mattheakis L.C., Kearney K.R., Vu L., Nomura M. (1988) Translational regulation of the spc operon in Escherichia coli: Identification and structural analysis of the target site for S8 repressor protein. J. Mol. Biol. 204(2), 309–329.
Gregory R.J., Cahill P.B., Thurlow D.L., Zimmermann R.A. (1988) Interaction of Escherichia coli ribosomal protein S8 with its binding sites in ribosomal RNA and messenger RNA. J. Mol. Biol. 204(2), 295–307.
Mattheakis L., Nomura M. (1988) Feedback regulation of the spc operon in Escherichia coli: Translational coupling and mRNA processing. J. Bacteriol. 170(10), 4482–4492.
Mattheakis L., Vu L., Sor F., Nomura M. (1989) Retroregulation of the synthesis of ribosomal proteins L14 and L24 by feedback repressor S8 in Escherichia coli. Proc. Natl. Acad. Sci. USA. 86(2), 448–452.
Dean D., Yates J.L., Nomura M. (1981) Identification of ribosomal protein S7 as a repressor of translation within the str operon of E. coli. Cell. 24(2), 413–419.
Saito K., Mattheakis L.C., Nomura M. (1994) Post-transcriptional regulation of the str operon in Escherichia coli. Ribosomal protein S7 inhibits coupled translation of S7 but not its independent translation. J. Mol. Biol. 235(1), 111–124.
Dragon F., Brakier-Gingras L. (1993) Interaction of Escherichia coli ribosomal protein S7 with 16S rRNA. Nucl. Acids Res. 21(5), 1199–1203.
Robert F., Brakier-Gingras L. (2001) Ribosomal protein S7 from Escherichia coli uses the same determinants to bind 16S ribosomal RNA and its messenger RNA. Nucl. Acids Res. 29(3), 677–682.
Golovin A., Spiridonova V., Kopylov A. (2006) Mapping contacts of the S12–S7 intercistronic region of str-operon mRNA with ribosomal protein S7 of E. coli. FEBS Lett. 580(25), 5858–5862.
Burgos H.L., O’Connor K., SanchezVazquez P., Gourse R.L. (2017) Roles of transcriptional and translational control mechanisms in regulation of ribosomal protein synthesis in Escherichia coli. J. Bacteriol. 199, e00407–17.
Hendrick E.G., Hill W.E. (2010) Protein S20 binds two 16S rRNA sites as assembly is initiated. J. Mol. Biol. 401, 493–502.
Tobin C., Mandava C.S., Ehrenberg M., Andersson D.I., Sanyal S. (2010) Ribosomes lacking protein S20 are defective in mRNA binding and subunit association. J. Mol. Biol. 397, 767–776.
Parsons G.D., Mackie G.A. (1983) Expression of the gene for ribosomal protein S20: effects of gene dosage. J. Bacteriol. 154, 152–160.
Mackie G.A. (1981) Nucleotide sequence of the gene for ribosomal protein S20 and its flanking regions. J. Biol. Chem. 256, 8177–8182.
Parsons G.D., Donly B.C., Mackie G.A. (1988) Mutations in the leader sequence and initiation codon of the gene for ribosomal protein S20 (rpsT) affect both translational efficiency and autoregulation. J. Bacteriol. 170, 2485–2492.
Donly B.C., Mackie G.A. (1988) Affinities of ribosomal protein S20 and C-tenninal deletion mutants for 16S rRNA and S20 mRNA. Nucl. Acids Res. 16(3), 997–1010.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


