Молекулярная биология, 2021, T. 55, № 1, стр. 86-95
Уровень sgРНК как основной фактор, влияющий на эффективность CRISPRi нокдауна в клеточной линии K562
Y. Wang a, Y. Xie b, Z. C. Dong a, X. J. Jiang a, P. Gong a, J. Lu a, F. Wan a, *
a College of Life Sciences, Inner Mongolia Agricultural University
010010 Inner, Mongolia, China
b College of Science, Inner Mongolia Agricultural University
010010 Inner, Mongolia, China
* E-mail: fwan@imau.edu.cn
Поступила в редакцию 10.02.2020
После доработки 07.04.2020
Принята к публикации 27.04.2020
Аннотация
В работе определяли вклад уровня экспрессии неактивной нуклеазы Cas9 (dCas9) и гид-РНК (sgРНК) в эффективность нокдауна системой CRISPR-интерференции (CRISPRi). С этой целью получены клоны клеток K562, продуцирующие белок KRAB-dCas9 на различных уровнях под контролем индуцибельной системы Tet-on или конститутивного промотора SFFV. Отдельные клоны были отобраны с помощью сортировки клеток с активацией флуоресценции (FACS) для дальнейшего изучения. В качестве мишеней sgРНК-экспрессирующих лентивирусных векторов из двух библиотек использовали шесть генов в четырех клеточных линиях с различными уровнями экспрессии KRAB-dCas9. Уровни экспрессии KRAB-dCas9/sgРНК и эффективность нокдауна оценивали методом проточной цитометрии. В клоне клеток с наивысшим уровнем экспрессии KRAB-dCas9 воспроизводимо и статистически значимо наблюдали наиболее эффективный нокдаун. Эти данные указывают на то, что уровень KRAB-dCas9 может определять эффективность CRISPRi. В этом клоне меняли уровень экспрессии sgРНК, манипулируя с множественностью инфекции (MOI) при лентивирусной трансдукции. Обнаружено, что эффективность нокдауна не коррелировала ни с уровнем экспрессии целевого гена, ни с уровнем KRAB-dCas9, который оставался относительно постоянным (коэффициент вариации 2.2%). В случае генов mmadhc, rpia и znf148 эффективность нокдауна составила 74.72, 72.28 и 39.08% соответственно и коррелировала с уровнем экспрессии sgРНК. В ходе анализа линейной регрессионной модели установлено, что на эффективность нокдауна влияет уровень экспрессии как KRAB-dCas9, так и sgРНК. Однако детальные расчеты показывают, что наибольший вклад вносит уровень sgРНК, что позволяет считать его основным фактором, влияющим на эффективность CRISPRi.
ВВЕДЕНИЕ
Технология CRISPRi постепенно вытесняет методы, основанные на малых интерферирующих РНК (siРНК), в механистических исследованиях функций генов [1]. Однако пока нет исследований по выявлению факторов, определяющих эффективность системы CRISPRi. В целом, на эффективность нокдауна могут влиять уровни двух основных игроков системы: белка dCas9 и направляющей РНК (single-guide RNA; sgРНК).
При репрессии транскрипции гена-мишени обычно используют один из трех белков: KRAB, MeCP2, dCas9 и его синтетические производные [2, 3]. KRAB привлекает KAP1, который служит каркасом для различных факторов, индуцирующих образование гетерохроматина [4]; MeCP2 связывается с метилированной ДНК и привлекает гистондеацетилазу и корепрессор SIN3A [5]. Показано, что комбинация dCas9 с KRAB и MeCP2 повышает эффективность нокдауна [2, 3]. Наиболее часто в CRISPRi используют следующие комбинации репрессорных факторов: dCas9, KRAB-dCas9 и dCas9-KRAB. Хотя процесс KRAB-dCas9 и sgРНК-опосредованного нокдауна CRISPRi не изучен досконально, можно предполагать, что его механизм такой же, как и в случае с эндонуклеазной системой CRISPR: сначала происходит экспрессия компонентов системы sgРНК и Cas9, затем образование комплекса sgРНК с белком Cas9, который транспортируется в ядро, где ищет мишень и после связывания с ней вносит двухцепочечный разрыв в ДНК [6]. Cas9 обеспечивает стабильность sgРНК, а уровень содержания комплексов Cas9 с sgРНК в ядре определяет эффективность редактирования генома. Следует ожидать, что подобные процессы происходят и при CRISPRi. Представляется важным установить, как уровни содержания репрессоров на основе dCas9 и sgРНК влияют на эффективность CRISPRi.
В исследовании для оценки эффективности CRISPRi использованы различные клоны клеток с различным уровнем белка KRAB-dCas9 и суб-клоны с различным уровнем экспрессии sgРНК. Для идентификации основного фактора, влияющего на эффективность CRISPRi, применен линейный регрессионный анализ.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Культуры клеток и получение плазмид. Линии клеток HEK-293T и K562 приобретены у National Infrastructure of Cell Line Resource (Китай). Клетки HEK-293T культивировали при 37°C в среде Игла, модифицированной по способу Дульбекко (DMEM), содержащей 4.5 г/л глюкозы (“Gibco”, США) с добавлением 10% эмбриональной телячьей сыворотки (FBS; “Gibco”). Клетки K562 культивировали в модифицированной по Искову среде Дульбекко (IMDM; “Gibco”) с добавлением 10% FBS. Клеточные культуры проверяли каждые 3 месяца на контаминацию микоплазмами и каждый раз получали отрицательный результат. Чтобы создать вектор с доксициклининдуцируемой экспрессией белка KRAB-dCas9, промотор в плазмиде SFFV-KRAB-dCas9 (плазмида #60954, “Addgene”, США; любезно предоставлена Jonathan S. Weissman) заменили на промотор pTRE3G (плазмида #96964, “Addgene”; любезно предоставлена Elena Cattaneo) с помощью классического метода клонирования. Праймеры указаны в табл. S1 Приложения (см. Приложение на сайте http://www.molecbio.ru/downloads/2021/1/supp_ Wang_rus.pdf).
Упаковка лентивирусных частиц. За 1 сутки до трансфекции во флаконы Т175 высевали 2.1 × 107 клеток HEK-293T. Клетки трансфицировали упаковывающими плазмидами pSPAX2 (11.6 мкг) и pMD2.G (2.8 мкг) (#12260 и #12259 “Addgene” соответственно; любезно предоставлены Didier Trono) и целевыми плазмидами (11.6 мкг) с использованием полиэтиленимина (PEI; 1 мг/мл), имеющего соотношение азота к фосфору (N/P) 20 [2, 7]. Супернатанты, содержащие вирусные частицы, собирали через 48 и 72 ч после трансфекции и фильтровали с использованием 0.45-мкм фильтров. После этого на дно центрифужной пробирки, содержащей 32 мл вируссодержащего супернатанта, вносили 4 мл 20%-ного раствора сахарозы и проводили ультрацентрифугирование при 82 700 × g и 4°C в течение 2 ч [8]. Осадок ресуспендировали в 100 мкл предварительно охлажденного PBS без Ca2+/Mg2+. Титр препарата лентивирусных частиц определяли методом проточной цитометрии, как описано ранее [8].
Клональная селекция после лентивирусной трансдукции. С целью получить единичные клоны с конститутивной экспрессией белка KRAB-dCas9 1 × × 105 клеток K562 трансдуцировали лентивирусными частицами, содержащими SFFV-KRAB-dCas9, с использованием полибрена (конечная концентрация 10 мкг/мл) при MOI 20 в 6-луночных планшетах. Через 72 ч после трансдукции клетки анализировали и сортировали в 96-луночные планшеты с использованием сортировщика клеток BD FACSAria II (“BD Biosciences”, США). Единичные клоны, появившиеся через 14 суток, культивировали для дальнейшего анализа.
Для получения единичных клонов с индуцибельной экспрессией KRAB-dCas9 1 × 105 клеток K562 инфицировали лентивирусными частицами, экспрессирующими трансактиватор промотора pTRE3G (pLVX-Tet3G). Трансдуцированные клетки растили в течение 10 суток в присутствии G418 (конечная концентрация 100 мкг/мл). Полученные клоны трансдуцировали лентивирусными частицами, содержащими pTRE3G-KRAB-dCas9, с использованием полибрена и выделяли отдельные клоны, как описано выше.
Проточная цитометрия. Проточную цитометрию проводили на приборе Beckman CytoFLEX S (“Beckman Coulter”, США). Данные анализировали с использованием программного обеспечения Flowjo 10 и CytoExpert 2.0.
Иммуноблотинг и иммуноферментный анализ (ИФА). Для определения содержания белка KRAB-dCas9 клетки клона K562-idCas9 #49 индуцировали доксициклином в концентрациях 200 нМ и 2000 нМ в течение 48 ч. Общий белок выделяли в лизисном буфере RIPA с 1 мМ PMSF и определяли концентрацию с использованием BCA Protein Assay kit (#PC0020, “Solarbio Life Science”, Китай). Для иммуноблотинга белковые лизаты (20 мкг) разделяли электрофорезом в 8%-ном SDS-PAAG и переносили на нитроцеллюлозную мембрану 0.22 мкм при 150 В в течение 90 мин. Мембрану инкубировали сначала с поликлональными антителами против Cas9 (#632607, “Clontech Laboratories Inc.”, США) и α-тубулина (#AC012, “ABclonal”, США), а затем со вторичными антителами IRDyeTM-800CW (Lot No. C50113-06, “BD Biosciences”). Мембрану сканировали с помощью системы визуализации OdysseyTM CLx (“Li-COR Bioscience”, США). Содержание белка KRAB-dCas9 количественно определяли с использованием набора CRISPR/Cas9 для ИФА (#P-4060-48, “EpiGentek”, США) в соответствии с протоколом производителя.
Оценка эффективности CRISPRi нокдауна. Одиночные клоны инфицировали лентивирусом, экспрессирующим sgРНК, (MOI 20, если не указано иное) с полибреном (конечная концентрация 10 мкг/мл). После 72-часовой трансдукции клоны отбирали пуромицином (конечная концентрация 5 мкг/мл) в течение 72 ч. Уровень экспрессии генов-мишеней определяли количественной ПЦР с использованием метода относительной количественной оценки. Праймеры для ПЦР указаны в табл. S1 Приложения (см. Приложение на сайте http://www.molecbio.ru/downloads/2021/1/supp_Wang_rus.pdf). В первую очередь специфичность праймеров проверяли путем анализа кривых плавления продуктов ПЦР и их разделения электрофорезом в агарозном геле. Далее определяли эффективность реакции ПЦР, анализируя серию разведений кДНК в качестве матрицы. Уровень экспрессии оценивали с помощью пакета программ, поставляемого с прибором LightCycler 96 (“Roche”, Швейцария). Относительный уровень экспрессии целевого гена определяли путем деления на усредненный уровень мРНК GAPDH и β-актина, используемых в качестве референсных генов. Эффективность нокадуна рассчитывали по формуле:
где мРНКц ‒ относительный уровень мРНК целевого гена в опыте; мРНКк ‒ относительный уровень мРНК в отрицательном контроле ‒ клеточном клоне, не инфицированном лентивирусом.
Статистический анализ. Дисперсионный анализ (ANOVA) выполняли при сравнении данных, полученных от четырех клеточных клонов. Линейный регрессионный анализ выполняли с целью выявления взаимосвязи между эффективностью нокдауна и уровнем экспрессии либо KRAB-dCas9, либо sgРНК. Все статистические анализы проводили с использованием пакета программ SPSS.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
С целью понять, какой из факторов: уровень белка KRAB-dCas9 или уровень sgРНК ‒ оказывает наибольшее влияние на эффективность нокдауна CRISPRi, мы применили иерархическую схему экспериментов, согласно которой сначала сравнивали эффективность нокдауна в клонах с различным уровнем содержания белка KRAB-dCas9, а затем сравнивали эффективность нокдауна в субклонах клеток с различным уровнем экспрессии sgРНК (рис. 1а). В первой серии экспериментов получены клоны клеток K562 с конститутивной или индуцибельной экспрессией гена химерного белка KRAB-dCas9-mCherry.
Рис. 1.
Схемы лентивирусных конструкций и отбора клеточных клонов с индуцибельной экспрессией компонентов системы CRISPRi. Получение и селекция клонов с индуцируемой экспрессией dCas9. а ‒ Схема эксперимента. #B ‒ K562-dCas9 #B; #C ‒ K562-dCas9 #C; #49 ‒ K562-idCas9 #49; #60 ‒ K562-idCas9 #60. #B1, #B2, #B3, #B4 и #B5 обозначают субклоны клона K562-dCas9 #B, инфицированные лентивирусным вектором, несущим sgРНК, при различных MOI. б – Схема лентивирусных векторов, кодирующих KRAB-dCas9 под контролем индуцибельных и конститутивных промоторов. в ‒ 60 клонов с индуцибельной экспрессией dCas9 ранжированы по соотношению индуцированный MFI/неиндуцированный MFI, красными ромбиками отмечены отдельные клоны, использованные в дальнейшем анализе. Экспрессию индуцировали доксицилином в конечной концентрации 2 000 нМ в течение 48 ч.
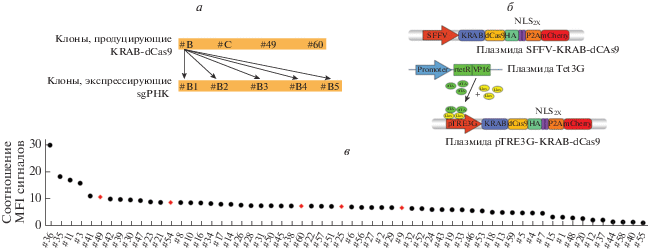
Систему Tet-on использовали при создании клеточных линий, экспрессия химерного белка KRAB-dCas9-mCherry в которых индуцировалась доксициклином: сначала с помощью лентивирусной трансдукции и последующей селекцией G418 в клетки K562 ввели конструкцию, кодирующую транс-активатор (плазмида Tet3G) промотора pTRE3G, далее в эти клетки путем лентивирусной трансдукции ввели конструкцию, экспрессирующую KRAB-dCas9-mCherry под контролем индуцибельного промотора pTRE3G (рис. 1б). Из двенадцати 96-луночных планшетов путем FACS с использованием mCherry в качестве репортерного белка случайным образом выбраны 60 клонов (K562-idCas9 от #1 до #60) для дальнейших экспериментов. Индукцию экспрессии трансгена определяли с помощью проточной цитометрии в присутствии доксициклина (рис. S1 и S2 Приложения, см. на сайте http://www.molecbio.ru/downloads/2021/1/supp_Wang_rus.pdf). На основании степени изменения уровня экспрессии трансгена, вычисленного как отношение средней интенсивности флуоресценции (MFI) клеток после индукции доксициклина к интенсивности без индукции доксициклином (индуцированный MFI/неиндуцированный MFI), провели ранжировку выбранных 60 клонов (рис. 1в) и для дальнейшего анализа выбрали 5 клонов (K562-idCas9 #9, #25, #49, #54, #60) с различной степенью изменения уровня экспрессии трансгена.
Сначала подтвердили индуцибельную экспрессию KRAB-dCas9-mCherry в K562-idCas9 #49 и #60 по содержанию mCherry, коэкспрессируемого с KRAB-dCas9, с помощью проточной цитометрии (рис. 2а). Далее детектировали dCas9 с помощью иммуноблоттинга и ИФА (рис. 2б, в). Белок KRAB-dCas9, как и ожидалось, в основном локализовался в ядре. Сигнал MFI mCherry соответствовал сигналу ИФА. Это значит, что mCherry служит адекватным репортером уровня экспрессии KRAB-dCas9. Для проведения CRISPRi экспериментов у компании “Addgene” были приобретены две библиотеки sgРНК: hCRISPRi-v2 [9] и Dolcetto [10]. Каждая библиотека содержит несколько sgРНК, нацеленных на интересующие нас гены. Ранжированные sgРНК представлены в табл. 1. Пилотные эксперименты по CRISPRi нокдауну проводили с использованием лентивирусных частиц, несущих sgРНК высшего ранга против UBE4A и DPH2 из библиотеки hCRISPRi-v2. K562-idCas9 #9, #25, #54 были исключены из дальнейшего анализа на основании низкой эффективности CRISPRi нокдауна в пилотных экспериментах (рис. S3 а‒г Приложения, см. на сайте http://www.molecbio.ru/downloads/2021/1/supp_Wang_rus.pdf). В клоне K562-idCas9 #49, обладающем относительно высоким уровнем экспрессии KRAB-dCas9-mCherry, эффективность нокдауна гена DPH2 в присутствии 100 нМ доксициклина составила 24% (рис. S3 д Приложения). Клоны K562-idCas9 #49, а также #60, который имеет относительно низкую экспрессию KRAB-dCas9-mCherry (рис. 2г), выбрали для дальнейшего анализа.
Рис. 2.
Оценка уровня KRAB-dCas9 в клеточных клонах с индуцибельной и конститутивной экспрессией соответствующего гена. а ‒ MFI-сигнал mCherry в клонах K562-idCas9 #49 и K562-idCas9 #60 в ответ на различные концентрации доксициклина. Треугольниками обозначен клон K562-idCas9 #49, кружками – клон K562-idCas9 #60. б, в – Результаты определения содержания KRAB-dCas9 в ядерной и цитоплазматической фракциях клеток методами иммуноблотинга и ИФА в клоне K562-idCas9 #49, обработанном доксициклином в конечной концентрации 200 нМ и 2 000 нМ. г ‒ MFI-сигнал mCherry в двух клонах с конститутивной экспрессией dCas9 и в двух клонах с индуцибельной экспрессией dCas9 после обработки доксициклином в конечной концентрации 100 нМ. Обозначения клонов: #B ‒ K562-dCas9 #B; #C ‒ K562-dCas9 #C; #49 ‒ K562-idCas9 #49; #60 ‒ K562-idCas9 #60.
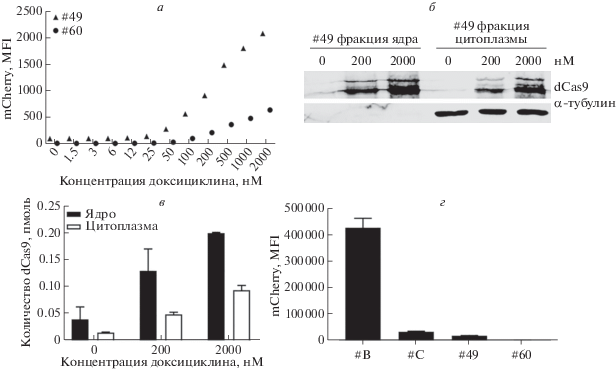
Таблица 1.
Последовательности спейсеров sgРНК
| sgРНК | Последовательность (5' → 3') | Ранг | Библиотека |
|---|---|---|---|
| DPH2 | GATGTTTAGCAGCCCTGCCG | 2/10 | hCRISPRi-v2 |
| UBE4A | GACAGGGTCGAAGTTCAAGA | 1/10 | hCRISPRi-v2 |
| MMADHC | GAAAATACGGGCAATACCGA | 1/10 | hCRISPRi-v2 |
| RPIA | GGTGCACAGTCTGGGACCCG | 1/10 | hCRISPRi-v2 |
| ZNF148 | GCAGGCCGAGCGCTTTACCG | 1/10 | hCRISPRi-v2 |
| LSM4 | CAGTTGCCCTGCCCTCACCA | 1/3 | Dolcetto |
| NPEPPS | TGGCGGGCGGGCTGCCTACG | 3/3 | Dolcetto |
| LIMA1 | GATGCTTTCTCCATGTGGCA | 1/3 | Dolcetto |
Аналогичным образом анализировали клоны клеток, конститутивно экспрессирующие KRAB-dCas9-mCherry, и выбрали три клона: K562-dCas9 #A, K562-dCas9 #B и K562-dCas9 #C. В пилотных экспериментах по нокдауну установлено, что в клоне K562-dCas9 #A нокдауна генов UBE4A и DPH2 не наблюдается (рис. S3 е Приложения, см. на сайте http://www.molecbio.ru/downloads/2021/1/ supp_Wang_rus.pdf), поэтому он был исключен из дальнейшего анализа.
Чтобы оценить влияние уровня экспрессии KRAB-dCas9 на эффективность нокдауна в CRISPRi-системе использовали клетки с конститутивной K562-dCas9 #B, #C и индуцибельной K562-idCas9 #49, #60 экспрессией KRAB-dCas9. В индуцибельных клонах уровни KRAB-dCas9 были ниже, чем в конститутивных клонах (рис. 2г). Для CRISPRi-экспериментов из каждой библиотеки случайным образом выбрали по три sgРНК против целевых генов с высоким рейтингом (табл. 1). После лентивирусной трансдукции sgРНК векторами проводили селекцию клонов клеток с помощью пуромицина и оценивали эффективность нокдауна методом количественной ПЦР. Наиболее эффективное подавление экспрессии генов mmadhc, rpia, znf148, lsm4 и lima1 наблюдали в клоне K562-dCas9 #B с наивысшим уровнем экспрессии KRAB-dCas9 (рис. 3а). В остальных клонах наблюдали относительно низкую эффективность нокдауна или его отсутствие, что выражалось в относительно высоком уровне экспрессии целевого гена (рис. 3а). Эти данные указывают на то, что высокий уровень экспрессии KRAB-dCas9 ‒ важное условие эффективного процесса CRISPRi.
Рис. 3.
Для эффективного нокдауна CRISPRi необходим достаточный уровень KRAB-dCas9. а – Эффективность нокдауна 6 генов в четырех клонах. Обозначения клонов: #B ‒ K562-dCas9 #B; #C ‒ K562-dCas9 #C; #49 ‒ K562-idCas9 #49; #60 ‒ K562-idCas9 #60. б ‒ Влияние уровня экспрессии целевого гена на эффективность CRISPRi нокдауна в клоне K562-dCas9 #B. в ‒ Относительный уровень экспрессии 6 целевых генов в четырех клонах. Обозначения клонов, как на виде (а).
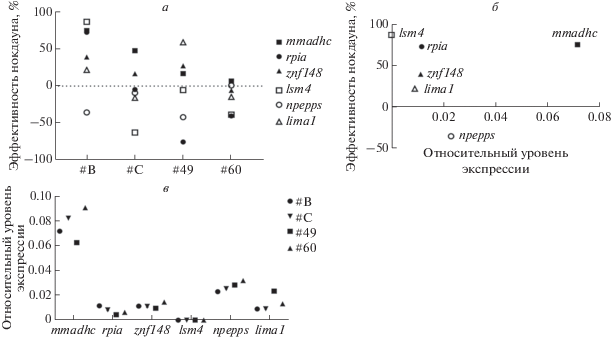
Для оценки статистической значимости различий между четырьмя клеточными клонами использовали ANOVA. Установлено, что K562-dCas9 #B значимо отличался от K562-dCas9 #C и K562-idCas9 #60, а p-value при сравнении K562-dCas9 #B и K562-idCas9 #49 составляло 0.054, из чего можно сделать вывод, что K562-dCas9 #B отличается также и от K562-idCas9 #49. Это еще раз подтверждает наш вывод о том, что K562-dCas9 #B работает как эффективный компонент CRISPRi-системы (табл. 2).
Таблица 2.
Эффективность нокдауна в четырех клеточных клонах
| Клеточный клон | Средняя эффективность нокдауна (%) | Стандартное отклонение (%) |
|---|---|---|
| K562-dCas9 #B | 43.16a | 45.55 |
| K562-idCas9 #49 | –3.24ab | 48.65 |
| K562-dCas9 #C | –4.63b | 36.48 |
| K562-idCas9 #60 | –15.24b | 19.92 |
Мы проанализировали также, влияет ли уровень экспрессии целевого гена на эффективность нокдауна, и не обнаружили значимой корреляции (рис. 3б). Относительный уровень экспрессии генов-мишеней в четырех клеточных клонах отличался друг от друга, вероятно, как следствие процедуры клонирования [11] (рис. 3в). Непрерывный мониторинг уровня экспрессии KRAB-dCas9 показал, что K562-dCas9 #C теряет экспрессию трансгена с течением времени, тогда как в других клонах этого не происходило (табл. S2 Приложения).
Используя клон K562-dCas9 #B, мы оценили влияние уровня экспрессии sgРНК на эффективность нокдауна. Перед выполнением основных экспериментов подобрали условия переноса трансгена, обеспечивающие наибольшую эффективность трансдукции клеток, путем измерения MFI синего флуоресцентного белка (BFP), коэкспрессируемого с sgРНК в полицистронном транскрипте. Согласно полученным данным, значения MFI BFP коррелировали с MOI и соответствовали полиномиальной кривой (рис. 4а). По результатам анализа этой кривой скорость переноса трансгена составила 34.1% при MOI 0.5, что близко к ранее сообщавшейся скорости переноса гена: 28.6% при MOI 0.5 в клетках K562 [12]. Эти данные подтверждают эффективную упаковку лентивируса и трансдукцию.
Рис. 4.
Эффективность CRISPRi нокдауна коррелирует с уровнем экспрессии sgРНК. а ‒ Зависимость эффективности переноса sgРНК против гена mmadhc от MOI, определенная по MFI-сигналу BFP в клоне K562-dCas9 #B. б – Диаграмма рассеяния для клона K562-dCas9 #B, экспрессирующего sgРНК против гена mmadhc. Красной рамкой выделена область клеток с положительным сигналом флуоресценции BFP. в ‒ Зависимость MFI-сигнала BFP в клетках, экспрессирующих sgРНК, от MOI лентивируса, несущего sgРНК против гена mmadhc, в клоне K562-dCas9 #B. Клетки, экспрессирующие sgРНК, детектировали с помощью проточной цитофлуориметрии. г ‒ Зависимость эффективности нокдауна от MOI лентивируса, кодирующего sgРНК против гена mmadhc, в клоне K562-dCas9 #B.
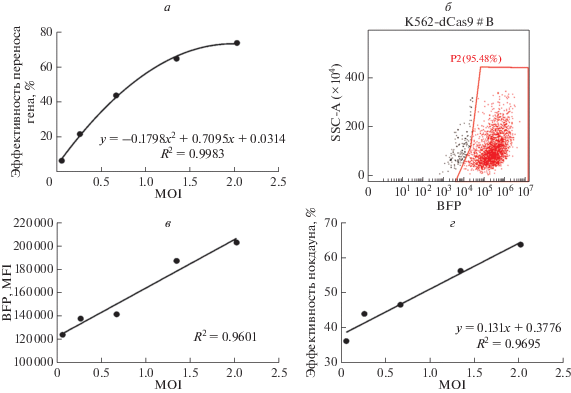
Затем с использованием проточной цитометрии подтвердили положительную корреляцию MOI с уровнем экспрессии sgРНК (рис. 4б). Обнаружили линейную корреляцию BFP MFI с MOI (рис. 4в). Более того, наблюдали линейную зависимость между MOI и эффективностью нокдауна (рис. 4г), из чего следует, что уровень экспрессии sgРНК положительно коррелирует с эффективностью нокдауна. На примере гена mmadhc показано, что при MOI 0.5, обычно используемом при скрининге библиотек CRISPRi, может быть достигнута эффективностью нокдауна около 45% (рис. 4г).
С целью оценить индивидуальный вклад уровней экспрессии dCas9 и sgРНК в эффективность нокдауна использовали модель линейной регрессии с MFI dCas9 и sgРНК в качестве независимых переменных. Подтверждено, что соблюдается условие равной дисперсии у сравниваемых групп данных. Согласно полученным данным, когда dCas9 служит независимой переменной в линейной модели, R2 = 0.243, F = 8.367, p = 0.008, а значит уровень содержания dCas9 значимо влияет на эффективность нокдауна. По значению стандартизованного коэффициента, β1 = 0.525, можно сделать вывод о том, что при изменении уровня dCas9 на единицу стандартного отклонения эффективность нокдауна изменится на 0.525 стандартного отклонения. Результаты аналогичных расчетов, выполненных для sgРНК в качестве независимой переменной в линейной модели, следующие: R2 = 0.949, F = 75.84, p = 0.003 и стандартизованный коэффициент β2 = 0.981. Это значит, что уровень sgРНК тоже сильно влияет на эффективность нокдауна, и изменение уровня sgРНК на единицу стандартного отклонения соответствует изменению эффективности нокдауна на 0.981 стандартного отклонения. Таким образом, на основании значений стандартизованного коэффициента β2 можно сделать вывод о том, что уровень sgРНК оказывает бóльшее влияние на эффективность нокдауна, чем уровень dCas9.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
На первом этапа исследований динамики процесса CRISPRi, опосредованного искусственным репрессором KRAB-dCas9, мы оценили влияние уровней химерного белка на основе dCas9 и sgРНК на эффективность нокдауна. Полученные данные могут служить основой для дальнейшей оптимизации скрининга библиотек CRISPRi.
Обнаружено, что уровень экспрессии KRAB-dCas9 значительно влияет на эффективность CRISPRi-системы. На основании полученных результатов, несмотря на ограниченное число проверенных клонов, можно полагать, что для эффективного нокдауна необходим определенный уровень химерного белка dCas9. Безусловно, только после проведения дополнительных исследований на большем числе клеточных клонов можно выяснить, действительно ли существует пороговый уровень содержания KRAB-dCas9, выше которого гарантирован эффективный нокдаун.
Требование по определенному уровню KRAB-dCas9 имеет большое значение при скрининге библиотек CRISPRi. Дело в том, что после лентивирусной инфекции обязательно проводят отбор клеток, а проблема состоит в том, что их требуется много, так как число клеток в каждой группе должно тысячекратно превышать количество sgРНК в используемой библиотеке. Именно поэтому при работе с библиотеками большого размера требуется высокоскоростной сортировщик клеток, который, к сожалению, недоступен в большинстве лабораторий. В отсутствие подобного инструмента скрининг библиотеки CRISPRi следует проводить в клеточных клонах, обеспечивающих стабильный и высокий уровень содержания KRAB-dCas9. Однако промотор интегрированного лентивирусного вектора может метилироваться, что приводит к выключению экспрессии трансгена при непрерывном культивировании клеточных линий, что, по-видимому, произошло в одном из клеточных клонов ‒ K562-dCas9 #C. На основании вышеизложенного имеет смыл вести непрерывный мониторинг уровня KRAB-dCas9 и использовать в эксперименте несколько клонов.
Известно, что в отдельных изолированных клонах изменен транскрипционный паттерн клеток, экспрессирующих KRAB-dCas9, поэтому CRISPRi стоит проводить в поликлональных популяциях клеток [11]. Однако влияние изменения паттерна транскрипции, как следствие процедуры клеточного клонирования, можно нивелировать использованием одного и того же клона в дальнейших исследованиях. Помимо использования клонов с высоким уровнем содержания химерного белка dCas9 мощность скрининга CRISPRi можно увеличить за счет постоянной селекции клеток в присутствии антибиотиков ‒ при условии наличия гена устойчивости к ним в используемых трансгенных конструкциях. Альтернативой может быть использование более эффективного химерного репрессора dCas9-KRAB-MeCP2 [13].
Из шести генов, случайно выбранных для CRISPRi, эффективность репрессии npepps и rpia была самой низкой в большинстве или во всех клонах. Согласно аннотации, экспрессия гена npepps чувствительна к пуромицину, поэтому вполне вероятно, что при селекции sgРНК трансдуцированных клонов в присутствии пуромицина произошла активация его экспрессии. В случае rpia также есть сообщения о сверхэкспрессии этого гена в ответ на обработку доксициклином [14].
Нами обнаружено, что при высоком уровне содержания KRAB-dCas9 эффективность CRISPRi хорошо коррелирует с MOI. В то же время эффективность CRISPRi хорошо коррелировала и с экспрессией sgРНК. Несмотря на отсутствие данных, мы предполагаем, что в CRISPRi, как и в случае системы CRISPR, накопление направляющей РНК в ядре служит ограничивающим фактором для эффективного действия эффектора Cas9 [6, 15]. Нами обнаружено, что уровень sgРНК в большей степени влияет на эффективность CRISPRi, чем уровень dCas9. Логично предположить, что динамика образования рибонуклеопротеидных комплексов и поиска мишени на ДНК сходна в системах CRISPR и CRISPRi.
Для повышения эффективности нокдауна критически важно достигать высокого уровеня экспрессии sgРНК, учитывая ее нестабильность в свободном состоянии ‒ когда она не связана с белком Cas9 [6]. Действительно, увеличение стабильности sgРНК повышает эффективность ДНК-интерференции [16]. Использование сильного промотора также должно способствовать увеличению уровня sgРНК.
Корреляция между уровнем экспрессии sgРНК и эффективностью нокдауна в системе CRISPRi может быть использована для скрининга библиотек CRISPRi, причем индуцибельная экспрессия sgРНК позволит лучше контролировать эффективность нокдауна. Это же сообщалось для системы CRISPR, где на платформе индуцибельной sgРНК удается проводить эффективный контроль нокдауна [17], что опять же указывает на сходство между двумя системами: CRISPR и CRISPRi.
Авторы выражают благодарность проф. Jonathan S. Weissman, David E. Root и John G. Doench за любезно предоставленную через “Addgene” библиотеку CRISPRi, а также д-ру Luke A. Gilbert за советы по использованию библиотеки CRISPRi.
Работа выполнена при финансовой поддержке National Natural Science Foundation of China (81860652).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Evers B., Jastrzebski K., Heijmans J., Grernrum W., Beijersbergen R.L., Bernards R. (2016) CRISPR knockout screening outperforms shRNA and CRISPRi in identifying essential genes. Nat. Biotechnol. 34, 631–633.
Gilbert L.A., Horlbeck M.A., Adamson B., Villalta J.E., Chen Y.W., Whitehead E.H., Guimaraes C.P., Panning B., Ploegh H.L., Bassik M.C., Qi L.S., Kampmann M., Weissman J.S. (2014) Genome-scale CRISPR-mediated control of gene repression and activation. Cell. 159, 647‒661.
Yeo N.C., Chavez A., Lancebyrne A., Chan Y.C., Menn D.J., Milanova D., Kuo C.C., Guo X.G., Sharma S., Tung A., Cecchi R.J., Tuttle M., Pradhan S., Lim E.T., Davidsohn N., Ebrahimkhani M.R., Collins J.J., Lewis N.E., Kiani S., Church G.M. (2018) An enhanced CRISPR repressor for targeted mammalian gene regulation. Nat. Methods. 15, 611‒616.
Peng H.Z., Ivanov A.V., Oh H.J., Lau Y.C., Rauscher F.J. (2009) Epigenetic gene silencing by the SRY protein is mediated by a KRAB-O protein that recruits the KAP1 co-repressor machinery. J. Biol. Chem. 284, 35670‒35680.
Hansen J.C., Ghosh R.P., Woodcock C.L. (2010) Binding of the Rett syndrome protein, MeCP2, to methylated and unmethylated DNA and chromatin. IUBMB Life. 62, 732‒738.
Ma H.H., Tu L.C., Naseri A., Huisman M., Zhang S.J., Grunwald D., Pederson T. (2016) CRISPR-Cas9 nuclear dynamics and target recognition in living cells. J. Cell Biol. 214, 529‒537.
Yang S.Z., Shi H.J., Chu X.R., Zhou X.L., Sun P.N. (2016) A rapid and efficient polyethyleneimine-based transfection method to prepare lentiviral or retroviral vectors: useful for making iPS cells and transduction of primary cells. Biotechnol. Lett. 38, 1631‒1641.
Kutner R.H., Zhang X.Y., Reiser J. (2009) Production, concentration and titration of pseudotyped HIV-1-based lentiviral vectors. Nat. Protoc. 4, 495‒505.
Horlbeck M.A., Gilbert L.A., Villalta J.E., Adamson B.S., Pak R.A., Chen Y.W., Fields A.P., Park C.Y., Corn J.E., Kampmann M., Weissman J.S. (2016) Compact and highly active next-generation libraries for CRISPR-mediated gene repression and activation. Elife. 5, e19760.
Sanson K.R., Hanna R.E., Hegde M., Donovan K.F., Strand C., Sullender M.E., Vaimberg E.W., Goodale A., Root D.E., Piccioni F., Doench J.G. (2018) Optimized libraries for CRISPR-Cas9 genetic screens with multiple modalities. Nat. Commun. 9, 5416.
Stojic L., Lun A.T., Mangei J., Mascalchi P., Quarantotti V., Barr A.R., Bakal C., Marioni J.C., Gergely F., Odom D.T. (2018) Specificity of RNAi, LNA and CRISPRi as loss-of-function methods in transcriptional analysis. Nucleic Acids Res. 46, 5950‒5966.
Kustikova O.S., Wahlers A., Kuhlcke K., Stahle B., Zander A.R., Baum C., Fehse B. (2003) Dose finding with retroviral vectors: correlation of retroviral vector copy numbers in single cells with gene transfer efficiency in a cell population. Blood. 102, 3934‒3937.
Martella A., Firth M., Taylor B.J., Goppert A., Cuomo E.M., Roth R.G., Dickson A.J., Fisher D.I. (2019) Systematic evaluation of CRISPRa and CRISPRi modalities enables development of a multiplexed, orthogonal gene activation and repression system. ACS Synth. Biol. 8, 1998‒2006.
Ahler E., Sullivan W.J., Cass A., Braas D., York A.G., Bensinger S.J., Graeber T.G., Christofk H.R. (2013) Doxycycline alters metabolism and proliferation of human cell lines. PLoS One. 8, e64561.
Yuen G., Khan F.J., Gao S.J., Stommel J.M., Batchelor E., Wu X.L., Luo J. (2017) CRISPR/Cas9-mediated gene knockout is insensitive to target copy number but is dependent on guide RNA potency and Cas9/sgRNA threshold expression level. Nucleic Acids Res. 45, 12039‒12053.
Mu W., Zhang Y.P., Xue X.T., Liu L., Wei X.F., Wang H.Y. (2019) 5′ capped and 3′ polyA-tailed sgRNAs enhance the efficiency of CRISPR-Cas9 system. Protein Cell. 10, 223‒228.
Aubrey B.J., Kelly G.L., Kueh A.J., Brennan M.S., Oconnor L., Milla L., Wilcox S., Tai L., Strasser A., Herold M.J. (2015) An inducible lentiviral guide RNA platform enables the identification of tumor-essential genes and tumor-promoting mutations in vivo. Cell Rep. 10, 1422‒1432.
Дополнительные материалы
- скачать ESM_1.docx
- Supplementary Table 1. The sequences of primers
Supplementary Table 2. The mCherry MFI in four clones
Supplementary Fig. 1. The histogram of #1 to #30 inducible single clones.
The blue peak is the mCherry fluorescent histogram without induction, and the red peak is with 2000 nM doxycycline induction for 48 h.
Supplementary Fig. 2. The histogram of #31 to #60 inducible single clones.
The blue peak is the mCherry fluorescent histogram without induction, and the red peak is with 2000 nM doxycycline induction for 48 h.
Supplementary Fig. 3. Pilot CRISPRi experiments targeting UBE4A and DPH2 in cell clones.
(a) The relative expression level of UBE4A mRNA in K562-idCas9 #9.
(b)The relative expression level of UBE4A mRNA in K562-idCas9 #25.
(c) The relative expression level of UBE4A mRNA in K562-idCas9 #49.
(d)The relative expression level of UBE4A mRNA in K562-idCas9#54. Neg. indicated none sgRNA transduction.
(e) The relative expression level of DPH2 mRNA in K562-idCas9 #49.
(f) The relative expression level of UBE4A and DPH2 mRNA in K562-dCas9 #A.
Инструменты
Молекулярная биология


