Молекулярная биология, 2021, T. 55, № 2, стр. 194-209
Начальные стадии эксцизионной репарации оснований ДНК в нуклеосомах
О. А. Кладова a, Н. А. Кузнецов a, О. С. Федорова a, *
a Институт химической биологии и фундаментальной медицины Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
* E-mail: fedorova@niboch.nsc.ru
Поступила в редакцию 29.07.2020
После доработки 21.08.2020
Принята к публикации 26.08.2020
Аннотация
Эксцизионная репарация оснований – основной путь, ответственный за коррекцию большого числа химически модифицированных оснований ДНК в клетках млекопитающих. Упаковка ДНК в хроматине влияет на доступность поврежденных участков для ферментов, вовлеченных в процессы репарации. В обзоре представлены данные о ферментах, участвующих в эксцизионной репарации оснований, свидетельствующие о том, что в коровой частице нуклеосомы доступность поврежденной ДНК затрудняет октамер гистонов. При этом исправление повреждений в ДНК в значительной степени зависит от их ротационного и трансляционного позиционирования в коровой частице нуклеосомы, а также от особенностей действия ферментов.
СТРОЕНИЕ НУКЛЕОСОМ
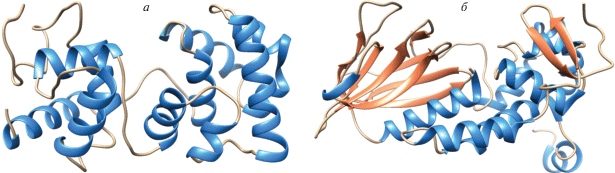
В клетках эукариот ДНК находится в плотно упакованном состоянии в комплексе c белками-гистонами в составе хроматина. Структурная единица хроматина – нуклеосома, содержит кор, образованный двумя копиями четырех гистоновых белков (Н2А, Н2В, Н3 и Н4), т.е. восемью молекулами белка. Каждый гистоновый белок, состоящий из ~100–150 аминокислотных остатков (10–15 кДа), содержит структурированный домен и неструктурированный N-концевой “хвост” (иногда С-концевой хвост). Структурированные домены гистоновых белков попарно взаимодействуют друг с другом, образуя октамер, обладающий осью псевдосимметрии 2-го порядка (диадная ось), на который ~1.67 раза наматывается цепь ДНК длиной 145–147 п.н. с образованием коровой частицы нуклеосомы (nucleosome core particle, NCP) [1, 2] (рис. 1).
Рис. 1.
Строение коровой частицы нуклеосомы. Вид сверху (а) и сбоку (б). ДНК выделена синим цветом, гистоновый октамер красным (PDB ID: 3LEL).
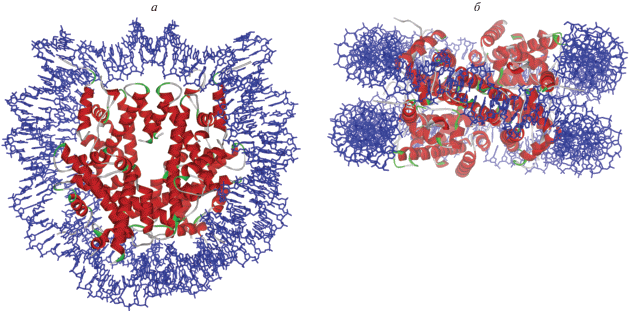
В 1977 году Finch и соавт. [3] показали, что NCP представляет собой диск диаметром 110 Å и высотой 57 Å. В кристаллической структуре NCP, определенной в 1984 году с разрешением 7 Å [4], ДНК в нуклеосоме намотана на гистоновый октамер, образуя плоскую левозакрученную суперспираль. В 1997 году Luger и соавт. [1] установили кристаллическую структуру гистонового кора с разрешением 2.8 Å. Детали структуры NCP выявили при более высоком разрешении (1.9 Å) [5, 6]. В дальнейшем определили структуры нуклеосом у таких видов, как Homo sapiens, Mus musculus, Gallus gallus, Drosophila melanogaster и Saccharomyces cerevisiae [7–12]. В большинстве случаев структуры нуклеосом, полученные с помощью рентгеновской кристаллографии, созданы с использованием ДНК Widom 601 [2, 13]. Эта ДНК, полученная путем отбора из комбинаторной библиотеки фрагментов ДНК, предназначена для фиксации положения ДНК в нуклеосоме, поскольку имеет наибольшее сродство к гистоновому октамеру [14]. В составе нуклеосомы ДНК Widom 601 обладает наименьшей подвижностью из всех других случайных последовательностей ДНК и поэтому менее доступна для ферментов рестрикции [15]. Другая позиционирующая нуклеосомы последовательность ДНК, часто используемая в экспериментальных работах, это фрагмент гена, кодирующего 5S рРНК морского ежа Lytechinus variegatus [16], консервативного у многих организмов [17].
В NCP в отличие от нуклеосом отсутствует линкерный участок длиной 10–90 п.н., который может быть связан с гистоном Н1. Расположение ДНК в NCP определяется в основном 14 электростатическими контактами. Основными среди них являются контакты с боковыми заместителями остатков аргинина на поверхности гистонов, которые вставляются в малую бороздку ДНК [18, 19]. При этом N-хвосты гистонов также участвуют во внутри- и межнуклеосомных взаимодействиях с ДНК [6, 18]. Кроме того, они играют важную регуляторную роль, поскольку содержат различные модификации, служащие сигналами к началу процессов транскрипции и репарации [19].
С внешней стороны нуклеосомы расположен линкерный гистон Н1, который фиксирует в нуклеосоме цепь ДНК. Нуклеосомы являются мобильными структурами, позволяющими белкам извне взаимодействовать с внутренними участками ДНК in vitro. Так, РНК-полимераза способна транскрибировать нуклеосомную ДНК без ее диссоциации из комплекса с гистоновым октамером [20–22]. In vivo транскрипция ДНК протекает при участии ремоделирующих факторов, направляемых с помощью регуляторных белков и действующих непосредственно на гистоновый октамер, увеличивая мобильность нуклеосом. По-видимому, такие процессы протекают благодаря способности нуклеосом “скользить” по ДНК без еe диссоциации [19].
СТРУКТУРНОЕ ПОЛОЖЕНИЕ ПОВРЕЖДЕННЫХ НУКЛЕОТИДОВ В СОСТАВЕ НУКЛЕОСОМЫ
Клеточная ДНК постоянно повреждается в результате негативного воздействия различных внешних и внутренних факторов, включая активные клеточные метаболиты, УФ- и ионизирующее излучение, загрязняющие агенты окружающей среды, лекарственные средства. Среди повреждений ДНК наиболее распространены модификации гетероциклических оснований, обусловленные их окислением, алкилированием и дезаминированием, а также одноцепочечные разрывы и апуриновые/апиримидиновые (AP) сайты, возникающие как спонтанно, так и под действием ДНК-гликозилаз. Эти повреждения исправляются с помощью системы эксцизионной репарации оснований (BER, base excision repair), которая протекает по короткозаплаточному (short-patch, SP) или длиннозаплаточному пути (long-patch, LP) [23]. Процесс BER включает несколько последовательных ферментативных реакций. На первой стадии специфические ДНК-гликозилазы находят и удаляют модифицированные основания. Поиск ДНК-гликозилазами таких оснований происходит путем как 3D-диффузии, так и “ускоренной” 1D-диффузии фермента по цепи ДНК [24]. Узнавание модифицированного основания инициирует протекание N-гликозилазной реакции, в ходе которой происходит гидролиз N–С1′-гликозидной связи между основанием и остатком рибозы и образование АР-сайта. Далее, в случае монофункциональных ДНК-гликозилаз, АР-эндонуклеаза (АРЕ1 у человека) катализирует гидролиз 5′-фосфодиэфирной связи с образованием на концах разрыва рибозофосфатного остова 3′-гидро-ксила и 5′-дезоксирибозофосфата (5′-dRP). Бифункциональные ДНК-гликозилазы обладают собственной АР-лиазной активностью, поэтому после гликозилазной реакции они катализируют реакцию, приводящую к разрыву рибозофосфатного остова с 3′-стороны от АР-сайта с образованием 5′-фосфата и 3′-производного рибозы.
В SP-пути BER остаток 5′-dRP может быть удален с помощью полимеразы β (Polβ), а 3′-производное рибозы, оставшееся после АР-лиазной реакции, расщепляется с помощью 3′-экзонуклеазной активности АР-эндонуклеазы с образованием 3′-ОН на конце. Затем Polβ заполняет образовавшуюся однонуклеотидную брешь (gap), а ДНК-лигаза I (LigI) или комплекс ДНК-лигазы III (LigIII) с XCCR1 (X-ray cross complementation group protein 1) сшивают разорванную цепь.
В LP-пути BER принимают участие такие дополнительные факторы, как FEN1, PCNA и RSC. После образования АР-сайта АР-эндонуклеаза, Polδ или Polε достраивают цепь ДНК с 3′-OH-стороны, замещая цепь, содержащую 5′-dRP, несколькими нуклеотидами. Образующийся флэп (flap = откидная заслонка) удаляется с помощью FEN1, а получившийся разрыв цепи сшивается с помощью LigI. Активность Polδ и Polε в этом пути репарации может быть усилена дополнительными факторами PCNA и RFC [25, 26].
Повреждение ДНК в составе хроматина должно приводить к возникновению модифицированных оснований в различных участках ДНК. Положение азотистых оснований по отношению к гистоновому октамеру в NCP различают по их ориентации в спирали ДНК – в сторону раствора или в сторону гистона (ротационное положение: Out, Mid и In), а также по положению относительно диадной оси (трансляционное) (рис. 2).
Рис. 2.
Ротационное положение гетероциклических оснований ДНК по отношению к гистоновому октамеру. Красным выделено положение Out – наружу, зеленым – Mid – промежуточное, синим – In – внутрь.
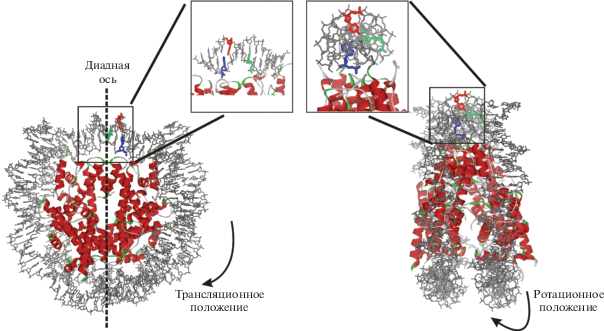
В нуклеосомах фрагмент ДНК по-разному связывается с разными участками гистонового октамера. Например, тетрамер Н3-Н4, фланкированный двумя димерами Н2А и Н2В, связывается с 60 п.н. фрагмента ДНК более прочно, чем два димера Н2А и Н2В с концами ДНК [1, 5]. Поэтому ДНК на концах фрагмента обладает большей подвижностью, за счет чего она способна перемещаться относительно поперечной оси симметрии и спонтанно разматываться из гистонового октамера [27, 28]. При этом доступность ранее закрытых районов ДНК возрастает на порядок, что также зависит от посттрансляционной модификации гистонов [29, 30]. Это имеет большое значение для протекания процессов репарации ДНК по пути BER.
УДАЛЕНИЕ УРАЦИЛА
К появлению остатков урацила в составе молекулы ДНК может приводить как внедрение dUMP в процессе репликации, так и спонтанное дезаминирование цитозина [31–33]. Несмотря на достаточно малоe количество остатков урацила в геномной ДНК [34], образующиеся пары U:G являются мутагенными, поскольку в ходе репликации приводят к трансверсии C:G в T:A [33].
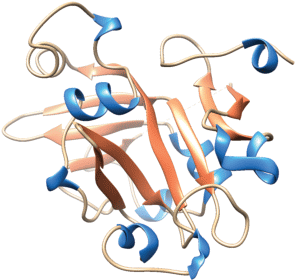
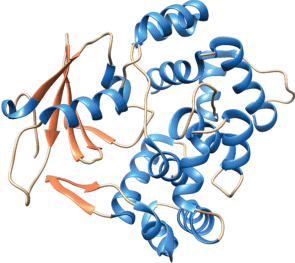
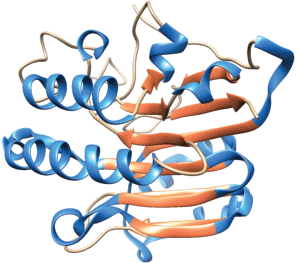
В клетках человека удаление урацила из ДНК осуществляют несколько монофункциональных ДНК-гликозилаз: UNG, TDG, SMUG1 и MBD4 (рис. 3). Монофункциональные ДНК-гликозилазы способны гидролизовать N-гликозидную связь, используя активированную молекулу воды для нуклеофильной атаки с образованием AP-сайта в качестве конечного продукта, который в дальнейшем узнается AP-эндонуклеазами.
Рис. 3.
Структуры ферментов: hUNG (PDB ID: 1AKZ) (а), hTDG (PDB ID: 3UFJ) (б), xSMUG1 (PDB ID: 1OE4) (в), hMBD4 (каталитический домен, PDB ID: 4E9G) (г).
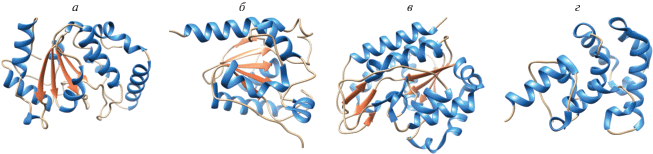
Основная урацил-ДНК-гликозилаза человека – фермент UNG, представлена двумя формами: митохондриальной (UNG1) и ядерной (UNG2). В отличие от UNG, способной удалять урацил, расположенный напротив любого азотистого основания, фермент TDG работает только с остатками урацила и тимина, расположенными напротив гуанина [35]. Показано, что TDG человека способен удалять тимин из пар T:G, T:C и T:T [36], а также бромурацил из пары с гуанином [37]. Фермент SMUG1 (single-strand specific monofunctional uracil-DNA-glycosylase) обнаружен только в клетках эукариот. Первоначально SMUG1 описали как фермент, специфичный к урацилу, расположенному в оцДНК. Позднее показали, что в качестве субстрата SMUG1 предпочитает дцДНК, если в системе присутствует AP-эндонуклеаза, способная стимулировать число оборотов фермента [33, 38]. SMUG1 также удаляет 5-гидроксиметилурацил, 5-формилурацил и 5-гидроксиурацил (5-OHU) из ДНК [39]. Фермент MBD4 впервые охарактеризовали как белок, связывающийся с метилированными CpG в ДНК [40], а затем показали, что MBD4 способен удалять урацил из ДНК [41].
Изучено действие урацил-ДНК-гликозилаз человека UNG2 и SMUG1, а также других ферментов, принимающих участие в процессе BER (АРЕ1, Polβ и LigIII), на удаление остатка урацила из ДНК в составе реконструированных нуклеосом in vitro [42]. Гистоновые октамеры выделяли из эритроцитов курицы, а в качестве ДНК-субстрата использовали участок гена 5S рРНК L. variegatus длиной 146 п.н. Урацил-ДНК-гликозилазы UNG2 и SMUG1 удаляли остатки урацила из такой ДНК, однако их активность была в 3–9 раз ниже, чем на свободной ДНК, и практически не зависела от ротационного положения остатка урацила относительно гистонового октамера (т.е. In или Out). Кроме того, оказалось, что и другие участники BER (АРЕ1, Polβ и LigIII) активны по отношению к ДНК-субстрату, входящему в состав нуклеосом. Присутствие белка XRCC1 не приводило к возрастанию активности Polβ и LigIII.
Используя модельные нуклеосомы, полученные из эритроцитов курицы и содержащие в составе ДНК участок GRE (glucocorticoid hormone receptor response element), Smerdon и соавт. [43] изучили влияние ротационного положения остатка уридина в нуклеосомной ДНК на активность ферментов, участвующих в BER, а именно, урацил-ДНК-гликозилазы UNG, АР-эндонуклеазы АРЕ1 и ДНК-полимеразы Polβ. Установлено, что активность и UNG, и АРЕ1 в отношении нуклеосомного субстрата, содержащего пару G:U, составляет лишь ~10% от активности в отношении свободной ДНК, что согласуется с данными [42]. Однако в отличие от [42] два ротационно различающихся положения остатка уридина (т.е. “внутрь” и “наружу” по отношению к гистоновому октамеру) удаляются с разными скоростями, которые отличаются в 2–3 раза и возрастают с увеличением концентрации фермента. Скорость удаления урацила ферментом UNG возрастала в присутствии АРЕ1. В отличие от UNG и АРЕ1 фермент Polβ был полностью неактивен в отношении ДНК, связанной c нуклеосомой. Поэтому сделан вывод [43], что первые стадии BER с участием UNG и АРЕ1 могут протекать на ДНК, входящей в состав хроматина, тогда как чтобы сделать участки ДНК, требующие репарации, доступными для Polβ, на следующих стадиях BER необходимы хроматин-ремоделирующие факторы.
В ходе дальнейших исследований установлено [44], что удаление N-концевых хвостов гистоновых белков в составе нуклеосом не влияет значительно на эффективность удаления остатков уридина из ДНК с помощью UNG и АРЕ1. Однако в этом случае эффект ингибирования активности Polβ нуклеосомами не исчезает, хотя N-концевые хвосты гистоновых белков могут взаимодействовать с ДНК в области мисматча G:U. Более того, присутствие белков XRCC1 или WRN (Werner syndrome protein) также не оказывало влияния на процесс BER. Поэтому был сделан вывод, что какие-то другие факторы должны иметь значение для репарации ДНК в составе хроматина.
Различие результатов, полученных в работах [42, 43], касается активности Polβ. Если в [42] обнаружено лишь уменьшение активности Polβ, то в [43] наблюдали полное ингибирование Polβ нуклеосомными частицами независимо от ротационной ориентации урацила относительно гистонового октамера. Это различие могло быть связано со структурой использованных субстратов. Кроме разницы в общей последовательности нуклеотидов, имелись различия и в структуре специфической пары: в [42] это уотсон-криковская пара U:A, аналогичная паре Т:А, у которой отсутствует лишь 5-СН3-группа. Поэтому пара U:A не должна влиять на структуру и стабильность ДНК. Использовали также ДНК-субстрат, содержащий неканоническую пару G:U, в которой остаток урацила сдвинут в сторону большой бороздки, что нарушает стэкинг-взаимодействие и, вероятно, влияет на доступность остатка урацила в процессах его репарации [43].
В этих исследованиях показано, что первые стадии BER могут протекать в нуклеосомах, однако для завершения процесса необходима какая-либо дестабилизация структуры, ремоделирование или частичное разрушение нуклеосом. При этом непонятно, каким образом ДНК-гликозилазы получают доступ к стерически закрытым повреждениям. Ранее предполагалось (см., например [28]), что частичное разматывание ДНК с гистонового октамера помогает факторам транскрипции и другим факторам связываться со своими сайтами на ДНК.
Активность фермента MBD4 в нуклеосомных частицах изучали с использованием фрагмента гена 5S рРНК L. variegatus, содержащего мисматч T:G [45]. Гистоновые белки выделяли из эритроцитов курицы. Активность MBD4 при взаимодействии со свободной ДНК была выше, чем с ДНК в составе NCP. Когда же мисматч T:G помещали в два разных положения относительно диадной оси на противоположных концах ДНК, то не обнаруживали различий в изменении активности MBD4. Показано также, что активность MBD4 возрастает при взаимодействии с нуклеосомными частицами, содержащими ацетилированные гистоновые белки.
Подробно изучено влияние расположения модифицированного основания в нуклеосоме на скорость его удаления ДНК-гликозилазой Udg E. coli [46]. Субстратами фермента служили фрагменты ДНК длиной 154 п.н., содержащие нуклеосом-позиционирующий элемент гена 5S РНК Xenopus borealis. Остатки уридина располагались в одном из положений фрагмента ДНК (–78…+76), считая от диадной оси, в составе пары U:A, причем их ориентация была “внутрь, In” или “наружу, Out” относительно поверхности гистонов. Гистоновые белки H3/H4 и H2A/H2B были выделены из эритроцитов курицы. Обнаружено, что скорость удаления остатков уридина из ДНК в составе нуклеосом составляла (1.1–3.6) × 10–4 с–1 в случае ориентации “внутрь” и 0.16–0.33 с–1 при ориентации “наружу”. Эти величины возрастали по мере удаления остатка U от диадной оси в сторону концов ДНК в пределах каждого диапазона. Если же в более длинном фрагменте ДНК (227 п.н.) U находится за пределами нуклеосомы в составе линкерной области, то он удалялся с такой же скоростью, как и при взаимодействии со свободной ДНК. При этом в присутствии линкерного гистона Н1 скорость ферментативного процесса уменьшается. Несмотря на то, что при ферментативном удалении остатков U из ДНК, находящейся в составе нуклеосом, скорость процесса могла быть снижена в 3000–10 000 раз, репарация таких повреждений в клетке возможна при более высоких концентрациях Udg. Большое отличие от данных [43] может быть обусловлено тем, что используемая ранее ДНК не имела прочно связывающегося с гистоновым октамером участка, который позволял строго фиксировать ее. Поэтому для основания, ориентированного “внутрь”, возможна и другая ориентация вследствие перемещения цепи ДНК относительно гистонового октамера.
Чтобы надежнее оценить влияние ротационной динамики ДНК на поверхности нуклеосом на стадию узнавания повреждения в процессе BER, Smerdon и соавт. [47] изучали взаимодействие фермента UNG человека c ДНК (в присутствии и в отсутствие АРЕ1), содержащей остатки U, строго позиционированные в трех ориентациях относительно диадной оси нуклеосомной частицы: U-In (в сторону гистонового ядра), U-Out (в сторону растворителя) и U-Mid (промежуточная). Гистоновые белки выделяли из эритроцитов курицы. Чтобы обеспечить строгое позиционирование, ДНК длиной 150 п.н. содержала каноническую последовательность GRE, фланкированную хорошо охарактеризованными TG-мотивами, позиционирующими нуклеосомы [48]. Ориентация остатков U подтверждена с помощью футпринтинга гидроксильными радикалами. Установлено, что при положении субстрата U-Out скорость каталитической реакции была почти такой же, как на свободной ДНК, и существенно более низкой при положениях U-In и U-Mid. ДНК, с целью выяснения роли ее ротационной динамики в составе нуклеосом, сшивали с гистонами с помощью формальдегида, что также предотвращало ее перемещение относительно гистонового октамера. Сшивание U-In ДНК с гистонами приводило к еще более сильному снижению скорости реакции, тогда как в положения U-Out скорость даже возрастала. Кроме того, фермент АРЕ1, следующий за UNG в процессе BER, стимулировал активность UNG в отношении уридина в составе как свободной ДНК, так и в положении Out в нуклеосомной ДНК. При этом стимуляция активности UNG ферментом APE1 была примерно в 2 раза ниже, чем при взаимодействии с ДНК в составе нуклеосомной частицы. Не обнаружено стимуляции активности UNG ферментом APE1 при взаимодействии с уридином в положениях Mid и In. Эти данные свидетельствуют о том, что UNG взаимодействует с U на поверхности нуклеосом. Сделан вывод, что, по меньшей мере, вблизи диадной оси, доступ UNG к урацилу обеспечивается за счет ротационной динамики ДНК на поверхности гистонов, а не за счет кратковременного разматывания. При этом благодаря ротационной динамике ДНК происходит образование промежуточного состояния, ориентация U в котором делает его доступным для связывания с ферментом.
Изучение влияния трансляционной и ротационной динамики ДНК в нуклеосомах на скорость протекания отдельных стадий процесса BER продолжили Rodriguez и Smerdon [49]. Ранее эти авторы показали, что UNG взаимодействует с U на поверхности нуклеосом. Сделан вывод о том, что доступность урацила для UNG (по меньшей мере вблизи диадной оси) обеспечивается за счет ротационной динамики ДНК на поверхности гистонов, а не за счет кратковременного разматывания ДНК. При этом благодаря ротационной динамике ДНК образуется промежуточное состояние, в котором ориентация U делает его доступным для связывания с ферментом. На примере позиционирующей нуклеосому ДНК Widom 601, содержащей в разных положениях остаток U или АР-сайт, показано, что скорость удаления ориентированного “наружу” урацила ДНК-гликозилазой UNG в 210 раз больше, чем при ориентации “внутрь”. При ориентации “внутрь” ферментам более доступны основания, находящиеся как можно дальше от диадной оси. Ключевым фактором является трансляционное положение ориентированных наружу брешей цепи ДНК. Ориентированная наружу брешь, расположенная вблизи концов ДНК, заполняется в 3 раза быстрее, чем брешь вблизи диадной оси, имеющая то же ротационное положение. Удаление ориентированного “наружу” остатка уридина вблизи диадной оси в присутствии ферментов UNG и АРЕ1 происходит очень эффективно, однако активность Polβ значительно подавлена. Таким образом, динамика NCP очень важна для процессов репарации в присутствии UNG/APE1.
Чтобы лучше понять, как влияют структурные свойства фермента и субстрата на каталитическую активность, изучено поведение трех урацил-ДНК-гликозилаз одного структурного суперсемейства – Udg E. coli, SMUG1 и TDG, в отношении нуклеосом, содержащих поврежденный сайт U:G [50]. Вместо UNG человека использовали его бактериальный гомолог Udg, сходный как по аминокислотной последовательности, так и по общему строению белковой глобулы [51, 52]. Субстратом служила ДНК Widom 601 длиной 145 п.н., содержащая пару U:G в позиции 75, т.е. вблизи диадной оси, в ориентации Out. Согласно [13, 53], ДНК Widom 601 связывается с гистоновым октамером в единственной ориентации. Нуклеосомные частицы получали с использованием рекомбинантных гистоновых белков X. laevis. Чтобы предотвратить ингибирование реакции продуктом, наблюдаемое в случае SMUG1 и TDG, эксперименты проводили в условиях “одного оборота фермента”, т.е. при избытке фермента. В этих условиях наблюдаемая константа скорости (kobs), соответствующая самой медленной кинетической стадии, отражает химическую реакцию разрыва гликозидной связи. Обнаружено, что Udg проявляет высокую активность в процессе удаления урацила из пары U:G, SMUG1 почти неактивен на NCP, тогда как TDG имеет промежуточную активность, однако она пропадает в случае пары T:G.
Кинетика накопления продукта в случае Udg была однофазной, и наблюдаемая константа скорости была только в 4 раза ниже регистрируемой для ДНК-дуплекса, не связанного с гистоновым октамером. У TDG и SMUG1 кинетика была двухфазной, что, возможно, указывало на присутствие в субстрате двух типов сайтов, различающихся доступностью для фермента. Причиной разной активности этих трех гликозилаз могут быть разные факторы. Хотя Udg, SMUG1 и TDG содержат в структуре интеркалирующую петлю, SMUG1 уникален тем, что он имеет дополнительную спираль, которая действует как “клин”, который способствует деформации ДНК-цепи и выворачиванию из нее остатка U. В отличие от Udg и TDG, которые нарушают структуру ДНК только в районе урацил-содержащего нуклеотида, SMUG1 нарушает также пару оснований с 5′‑стороны от U, внося тем самым большее возмущение в структуру спирали ДНК. Это может быть причиной снижения торсионной подвижности ДНК по сравнению с реакцией в растворе и служить барьером для дестабилизации двойной спирали ДНК, необходимой для протекания гликозилазной реакции.
Позднее S. Delaney и E.D. Olmon [54] проверили способность Udg удалять остатки урацила и 5‑гидроксиурацила (5-OHU), расположенные в разных ротационных положениях в составе нуклеосомных частиц. Оказалось, что при наиболее доступном для растворителя положении остатка U-Out, фермент Udg удаляет это повреждение всего на порядок медленнее, чем из несвязанного с NCP ДНК-дуплекса, практически со 100% эффективностью. Если остаток урацила находился в стерически затрудненных положениях Mid и In, то наблюдалась двухфазная кинетика накопления продукта, а общая эффективность удаления урацила составляла 19 и 15% соответственно. Появление двух фаз при накоплении продукта реакции связывают с возможным нахождением в системе небольшого остаточного количества ДНК, не связанной с гистоновым октамером [54]. При взаимодействии Udg с 5-OHU эффективность удаления поврежденного основания не зависела от его ротационного положения в нуклеосомной частице и составляла около 10%.
УДАЛЕНИЕ ТИМИНГЛИКОЛЯ
В клетках человека удаление поврежденных пиримидиновых оснований из ДНК катализирует несколько ферментов. NTHL1 и NEIL1 узнают и удаляют такие повреждения, как 5,6-дигидроурацил, 5-гидроксиурацил, тимингликоль (Tg) и другие [55–60] (рис. 4).
С использованием гистонов, выделенных из эритроцитов курицы, и позиционирующей нуклеосому последовательности, содержащей фрагмент 5S рДНК из L. variegatus, показано, что фермент NTHL1 может взаимодействовать с остатками Tg, которые располагаются снаружи гистонового октамера [61]. Процесс взаимодействия проходит без значительного разрушения нуклеосом, но если остатки Tg располагаются со стороны гистонов, то их процессинг протекает значительно хуже и усиливается при возрастании концентрации фермента до физиологического значения, равного в ядрах ~2.3 мкМ [62]. При этом эффективность удаления поврежденных оснований была ниже, если они располагались ближе к центру (т.е. к диадной оси), чем к концевому участку ДНК на нуклеосоме. Поэтому сделано заключение, что репарация таких повреждений ферментом NTH1 включает спонтанное, обратимое, частичное разматывание ДНК с гистонового октамера (pис. 5).
Аналогичное исследование выполнено для ДНК-гликозилазы человека NEIL1 [63]. В отличие от NTHL1 фермент NEIL1 (human Endonuclease III like) принадлежит к другому структурному суперсемейству – Fpg/Nei. Хотя NEIL1, как и NTHL1, способен удалять из ДНК остатки Tg, между ними имеются различия. Во-первых, только NEIL1 удаляет основания из одноцепочечной или содержащей расплетенный участок (“пузырь”, bubble) ДНК [64]. Это делает NEIL1 ферментом, наиболее подходящим для удаления повреждений, блокирующих ДНК-полимеразы, таких как Tg, в вилке репликации. Во-вторых, после действия NTHL1 остаток рибозы удаляется с 3′-концевого нуклеотида с помощью АРЕ1, тогда как в случае NEIL1 он удаляется с помощью полинуклеотидкиназы (PNK). NEIL1 взаимодействует с факторами PCNA и FEN1, участвующими как в репликации ДНК, так и в LP-пути BER [65, 66]. Следовательно, эти две ДНК-гликозилазы катализируют различные каналы BER. В-третьих, количество NTHL1 не изменяется в течение клеточного цикла, тогда как содержание NEIL1 увеличивается в S-фазе [55]. Поэтому высказана гипотеза о том, что NEIL1 участвует в удалении повреждений в репликационной вилке, тогда как действие NTHL1 является более глобальным и не зависит от репликации [63].
Эти ДНК-гликозилазы, несмотря на различия в функциях, обладают одинаковой каталитической активностью по отношению к Tg, а их концентрация в ядрах клеток тоже примерно одинакова и составляет 25–80 нМ [63]. Однако эти ферменты по-разному связываются с неспецифической ДНК – NEIL1 намного более прочно, чем NTHL1. Поэтому сделан вывод, что у NEIL1 гораздо меньше возможности, чем у NTHL1, обнаружить поврежденное основание в хроматине без помощи других белков-партнеров, которые будут либо способствовать связыванию NEIL1 с повреждением, либо снизят эффективность неспецифического связывания с ДНК [63]. В противоположность этому NTHL1, вероятно, способен узнавать повреждения в хроматине без помощи дополнительных факторов.
Изучено также влияние нуклеосомных частиц на протекание каждой из четырех стадий “короткозаплаточного” пути BER [67]. Используя в качестве ДНК-субстратов последовательности ДНК Widom 601, позиционирующие нуклеосомы [14], и 5S рДНК L. variegatus, содержащие специфический сайт Tg, показано, что участники первых трех стадий процесса BER (ДНК-гликозилаза NTHL1, АР-эндонуклеаза АРЕ1 и Polβ) способны процессировать свои субстраты, входящие в состав нуклеосом. В целом, полагают [67], что первые две стадии “короткозаплаточного” пути BER протекают в интактных нуклеосомах, когда специфический сайт Tg кратковременно экспонирован в раствор в результате разматывания ДНК с гистонового октамера. Polβ, катализирующая третью стадию BER, также активна по отношению к сайту, ориентированному наружу нуклеосомы. Этот фермент в каталитически активном комплексе изгибает ДНК на 90°. Вследствие этого для протекания ферментативной реакции ДНК вблизи бреши должна отсоединяться от гистонового октамера. Все три участника BER последовательно замещают друг друга в ходе процесса. В отличие от них LigIIIα и XRCC1, которые участвуют на четвертой стадии и заканчивают процесс BER, проявляют активность при достаточно высоких концентрациях, приводящих к разрушению нуклеосом. Следует отметить, что в результате разрушения нуклеосом происходит также ускорение реакции с участием Polβ.
Рис. 5.
Схематичное изображение путей удаления поврежденного основания в составе NCP при прямом взаимодействии с ДНК-гликозилазой или частичном разворачивании ДНК для эффективного удаления повреждения. ДНК показана серым, гистоновый октамер красным, поврежденный нуклеотид синим, ДНК-гликозилаза зеленым.
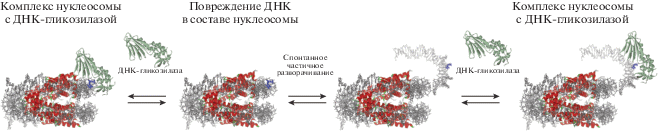
Проведено количественное измерение скоростей разматывания ДНК в нуклеосомах и репарации повреждений, а также их сравнение [68]. В случае взаимодействия фермента NTHL1 с нуклеосомными частицами, содержащими ДНК Widom 601 или 5S рДНК, в которые вводили остаток Tg в стерически недоступное для фермента положение, значения наблюдаемой константы скорости реакции kobs составили 0.0014 ± 0.0004 и 0.003 ± ± 0.0009 с−1 соответственно, что в ~100 раз меньше каталитической константы скорости kcat = 0.19 с−1 и в 10 раз меньше наблюдаемой константы скорости для свободной ДНК Widom 601, равной 0.03 ± ± 0.005 с−1. Поскольку скорость каталитической стадии гидролиза N-гликозидной связи больше наблюдаемой константы скорости (kcat > kobs), сделан вывод, что kobs характеризует стадию спонтанного разматывания ДНК, при которой поврежденное основание экспонировано наружу и стерически доступно для взаимодействия с ферментом. Однако скорость спонтанного разматывания ДНК была недостаточной для того, чтобы получить наблюдаемую скорость репарации in vivo. Поэтому эффективное протекание процесса BER в клетках требует участия факторов, ремоделирующих хроматин и специфичных для BER или принимающих участие в транскрипции, репликации, или других процессах репарации.
Поскольку в процессах BER происходит согласованная “передача” каждого ДНК-интермедиата от одного фермента к другому, то в случае взаимодействия ДНК-гликозилазы NTHL1 с АР-эндонуклеазой АРЕ1 такой процесс изучали на нуклеосомных частицах [69]. В отличие от свободного ДНК-субстрата, интермедиат которого, образующийся в гликозилазной реакции, быстро утилизируется в реакции с АРЕ1, в реакции на NCP такой интермедиат накапливается до значительно более высокого уровня и продолжает какое-то время существовать. Это связано с более высокой активностью АРЕ1 по сравнению с NTHL1 при взаимодействии со свободной ДНК, тогда как в случае ДНК, связанной с нуклеосомой, активность АРЕ1 снижается настолько, что становится заметной АР-лиазная активность NTHL1. При этом АРЕ1 взаимодействует с продуктом АР-лиазной реакции в качестве 3′-диэстеразы. Проверена способность двух бактериальных ферментов (EndoIII и Fpg) удалять 5-гидроксиурацил (5-OHU) из различных положений (In, Mid, Out) в составе нуклеосомных частиц [54]. EndoIII является функциональным и структурным гомологом NTHL1, в то время как Fpg – это структурный гомолог NEIL1, но их субстратная специфичность во многом различна. Показано, что при взаимодействии EndoIII и Fpg с нуклеосомами, содержащими 5-OHU, образуется лишь 10% продукта, причем эффективность расщепления не зависит от ротационного положения поврежденного основания. Такой небольшой процент расщепленного субстрата может быть связан с особенностями действия этих ДНК-гликозилаз. Из рентгеноструктурных данных известно, что EndoIII и Fpg значительно изгибают ДНК-субстрат при связывании – примерно на 55° и 66° соответственно [70, 71], что становится стерически затрудненным для ДНК в прочном комплексе с гистоновыми белками.
УДАЛЕНИЕ 1,N6-ЭТЕНОАДЕНИНА
Алкилирующие агенты присутствуют в окружающей среде, возникают внутри клетки и могут вызывать различные повреждения в ДНК [72–75], например такие, как этенопроизводные гетероциклических оснований: 1,N6-этеноаденин, 3,N4-этеноцитозин, N2,3-этеногуанин и 1,N2-этеногуанин. В клетках эукариот подобные повреждения из ДНК удаляет алкиладенин-ДНК-гликозилаза AAG [76, 77] (рис. 6).
Изучена активность AAG в отношении нуклеосомных частиц, реконструированных из гистоновых белков X. laevis и содержащих остаток 1,N6‑этеноаденина (εA) и гипоксантина (Hx) в различных ротационных положениях (In, Mid и Out) [54]. В качестве последовательности, позиционирующей нуклеосомы, использовали ДНК Widom 601 длиной 145 п.н. Как и у некоторых других ДНК-гликозилаз, константы скорости гидролиза N-гликозидной связи были выше на свободной ДНК, не связанной с гистоновым октамером. В случае NCP, содержащих остаток Hx в положении Out, образовывалось 10% продукта реакции, в положении Mid и In – примерно одинаковое количество продукта – 6 и 5% соответственно. При взаимодействии AAG с NCP, содержащей остаток εA в положении Out, образовывалось 84% продукта, в положении Mid – 16%. При этом не удалось зарегистрировать образование продукта реакции, когда остаток εA находился в позиции In. Это позволяет заключить, что чем менее поврежденное основание доступно для растворителя, а значит и для фермента, тем ниже скорость образования продукта реакции.
Хорошо изучена эффективность гликозилазной реакции, катализируемой алкиладенин-ДНК-гликозилазой AAG, в случае ДНК-субстратов в составе NCP, реконструированных из гистонов X. laevis и содержащих остаток 1,N6-этеноаденина (εA) в одном из 49 положений ДНК Widom 601 длиной 145 п.н. [78]. Доступность каждого из оснований ДНК для взаимодействия в водном растворе определяли с помощью футпринтинга гидроксильными радикалами. Определена наблюдаемая константа скорости N-гликозилазной реакции для каждого из положений εA. В целом активность AAG по отношению к отдельным сайтам коррелировала с их доступностью для раствора. Однако некоторые легкодоступные для растворителя положения εA подвергались репарации хуже, чем другие. Возможно, они находились в тех участках ДНК, в которых имелись структурные искажения спирали из-за взаимодействия с гистонами [2, 13]. Кроме того, в некоторых положениях цепи ДНК εA могли экранироваться неструктурированными хвостами гистонов, например Н2А и/или Н2В. В то же время показано, что в NCP концы ДНК могли спонтанно на короткое время разматываться с гистонового октамера, делая их доступными для взаимодействия [29].
УДАЛЕНИЕ 8-ОКСОГУАНИНА
Пуриновые и пиримидиновые основания в ДНК окисляются активными формами кислорода, которые образуются внутри клетки как в ходе собственного метаболизма, так и под действием различных факторов, например ионизирующей радиации или некоторых химических агентов [79–82]. Из четырех оснований ДНК самый низкий окислительно-восстановительный потенциал имеет гуанин, поэтому он легко окисляется с образованием 8-оксогуанина (8-oxoG). Несмотря на то, что это повреждение практически не блокирует репликативные ДНК-полимеразы, за исключением ДНК-полимеразы-йота [83], его образование может приводить к возникновению мутаций, чаще всего трансверсии G:C в T:A, поскольку при синтезе ДНК репликативные либо репаративные ДНК-полимеразы встраивают напротив 8-oxoG дезоксиаденозинмонофосфат либо дезоксицитидинмонофосфат [84]. В клетках млекопитающих 8-oxoG удаляется с помощью 8-оксогуанин-ДНК-гликозилазы OGG1 (рис. 7).
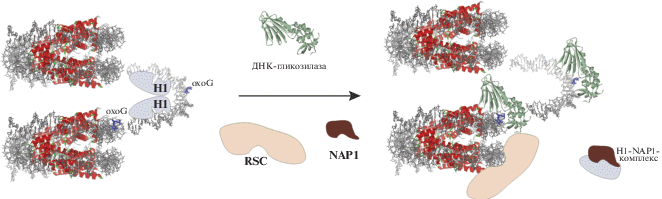
На более приближенной к хроматину модельной системе изучены динуклеосомы, полученные из рекомбинантных гистоновых белков X. laevis, ассоциированные с линкерным гистоном Н1 и содержащие строго позиционированную ДНК, сконструированную на основе ДНК Widom 601 [85]. Специфический сайт 8-oxoG помещали либо в NCP, либо в линкерный район ДНК (pис. 8). Показано, что в отсутствие гистона Н1 ДНК-гликозилаза человека OGG1 удаляет 8-oxoG из линкерного района ДНК и разрывает ДНК с такой же эффективностью, как и свободную ДНК. В присутствии гистона Н1 эффективность инициации репарации 8-oxoG уменьшается в 10 раз независимо от длины линкера. В присутствии шаперона NAP1, индуцирующего связывание Н1, эффективность удаления 8-oxoG восстанавливается до прежнего значения, если повреждение находилось в линкере, или остается низкой, если 8-oxoG находится в NCP. Удаление 8-oxoG из NCP возможно только тогда, когда кроме NAP1 присутствует белок RSC, ремоделирующий нуклеосомы. Предложена модель протекания BER при удалении остатков 8-oxoG в хроматине (рис. 8).
Рис. 8.
Модель удаления поврежденного основания в составе динуклеосомы. ДНК показана серым, гистоновый октамер красным, поврежденный нуклеотид синим, ДНК-гликозилаза зеленым. Схематично изображены гистон H1, шаперон NAP1 и белок RSC, ремодeлирующий нуклеосомы.
Показано, что фермент OGG1 удаляет 8-oxoG из мононуклеосомных частиц с эффективностью примерно 10% вне зависимости от того, в каком ротационном положении находится поврежденное основание (In, Mid или Out) [54]. Предполагается, что репарация 8-oxoG может полностью ингибироваться нуклеосомными частицами, а небольшая степень накопления продукта связана с присутствием остаточной ДНК, не связанной с гистоновым октамером. Такие же данные получены и для бактериального фермента Fpg, функционального гомолога OGG1.
Помимо одиночных повреждений в ДНК могут возникать кластерные окислительные повреждения, например, вызванные ионизирующим излучением. Если эти повреждения находятся в обеих цепях ДНК и репарируются ферментами системы BER (ДНК-гликозилазами и АР-эндонуклеазами), то образуются двойные разрывы цепей (DSB, double strand breaks), что может привести к лабилизации и распаду нуклеосом. На примере ДНК-гликозилаз NTHL1 (и NTHL1Δ55) и OGG1 установлено [86], что нуклеосомы способны частично подавлять образование двухцепочечных разрывов ДНК, однако эффективность подавления зависит от: 1) типа повреждения и ДНК-гликозилазы; 2) нуклеотидного контекста вблизи повреждений и их взаимного расположения в обеих цепях; 3) ориентации повреждений относительно гистонового октамера; 4) расстояния между кластером повреждений и краем нуклеосом. Если два повреждения расположены в обеих цепях напротив друг друга, то одно из них может иметь ориентацию, благоприятную для взаимодействия с ферментами системы BER, а другое – неблагоприятную. Образование двухцепочечных разрывов не обязательно должно приводить к разрушению нуклеосомы. В некоторых случаях разорванные концы ДНК остаются связанными с гистоновым октамером. Таким образом, можно предположить, что большинство двухцепочечных разрывов, сгенерированных в процессе BER, должно образовываться в линкерных областях, а также в районах ДНК, ассоциированных с повышенными скоростями ремоделирования нуклеосом.
РАСЩЕПЛЕНИЕ АР-САЙТА
Изучена также способность АР-эндонуклеазы АРЕ1 дикого типа (WT) и мутантных форм (Q51H, I64V, P112L, D148E, R237C, G241R, P311S, и A317V) расщеплять АР-сайты в составе свободной ДНК и NCP (рис. 9) [87].
С этой целью использовали последовательность ДНК Widom 601 и нуклеосомы, полученные из эритроцитов курицы. Активность АРЕ1 по отношению к NCP-связанной ДНК была в ~10 раз ниже, чем у свободной ДНК. Такое заметное снижение активности АРЕ1 дикого типа при взаимодействии с ДНК в составе NCP, вероятно, вызвано тем, что в фермент-субстратном комплексе цепь ДНК должна сильно изогнуться, что стерически затруднено. Эффективность удаления AP-сайта в положении Out была в 10 раз выше, чем в положении In. Инцизионная активность опухолеассоциированного варианта R237C и редкого варианта G241R в составе NCP была ниже, чем у АРЕ1 дикого типа, и не зависела от ориентации АР-сайта (In или Out). Активность АРЕ1 также оказалась сверхчувствительной к свойствам нуклеосомного комплекса вблизи АР-сайта. Более часто встречающиеся варианты Q51H, I64V и D148E АРЕ1 проявляли активность, близкую к активности фермента дикого типа. Полагают, что существует механизм взаимодействия АРЕ1 с ДНК в составе нуклеосом, не зависящий от классического ремоделирования хроматина [87]. Этот механизм состоит в том, что во взаимодействие АРЕ1 с нуклеосомной ДНК вовлекается участок поверхности фермента, содержащий определенные аминокислоты, среди которых наиболее важную роль играют R237 и G241, и который способствует связыванию с ДНК в составе комплексов с белками.
Ранее показали, что первые три стадии SP-пути BER могут протекать на ДНК в составе хроматина без участия ремоделирующих факторов и без разрушения нуклеосом. Этот же вывод сделан и для четвертой стадии [88], в которой участвуют белок XRCC1 и LigIIIα. LigIIIα отличается от ферментов, катализирующих первые три стадии (ДНК-гликозилазы, АР-эндонуклеазы и Polβ), тем, что в комплексе с ДНК он полностью охватывает одноцепочечный разрыв, что должно нарушить взаимодействие ДНК с гистонами и разрушить нуклеосому. Однако доказано, что комплекс LigIIIα-XRCC1 связывает ник в ДНК, когда происходит периодическое спонтанное разматывание ДНК из нуклеосомы [88]. Этот комплекс распадается после завершения лигирования ДНК, и нуклеосома возвращается в свое исходное состояние.
МОДИФИКАЦИИ ГИСТОНОВ
Известно [89], что в составе нуклеосом гистоновые белки могут подвергаться модификациям, таким как ацетилироваание лизинов, метилирование лизина и аргинина, фосфорилирование серина, треонина и тирозина, убиквитинирование и SUMO-илирование лизина (SUMO = Small Ubiquitin-like Modifier). ДНК подвергается метилированию метилтрансферазами по С5-атомам цитозина. Кроме того, в гистоновых белках могут возникать мутации. Большинство этих модификаций служат сигналами регуляции активности генов. Некоторые модификации влияют на кинетические и термодинамические параметры связывания ДНК с гистоновыми белками, структуру нуклеосом и вызывают их ремоделирование.
Изучено влияние мутаций в гистоне Н2А на процесс BER [90]. Семейство белков Н2А содержит наибольшее число вариантов. В структуру H2A входят три области, важные для интра- и интермолекулярной ассоциации: это докинг-домен, находящийся на С-конце; L1-петля и кислотный участок (the acidic patch). Один из самых изученных вариантов гистона Н2А – H2A.Z, найден почти у всех эукариот. Хотя этот вариант только на 60% идентичен каноническому Н2А, он высококонсервативен у различных организмов, что указывает на его значимость. H2A.Z вовлечен в различные биологические процессы, включая транскрипцию, репарацию двойных разрывов, эксцизионную репарацию нуклеотидов (NER) и прохождение клеточного цикла [91–93]. МакроH2A – это другой вариант H2A, который представляет особый интерес. МакроН2А содержит H2A-подобный гистоновый домен, связанный лизин-богатым линкером с C-концевым макродоменом, и участвует в инактивации Х-хромосомы, регуляции экспрессии генов и репарации двухцепочечных разрывов. Изучена также роль H2A.Z и макроH2A в процессах BER, а именно, в удалении остатков уридина урацил-ДНК-гликозилазами UDG и SMUG1 [90]. Активность UDG в отношении остатков U, малодоступных для растворителя, была ограниченной в канонических NCP и более высокой в NCP, содержащих варианты Н2А. Тем самым можно заключить, что варианты гистоновых белков способствуют удалению модифицированных оснований из малодоступных положений. В случае макроН2А в пределах концевых участков ДНК длиной 35–40 п.н. активность обеих ДНК-гликозилаз была сравнимой с активностью, наблюдаемой в случае свободных ДНК-дуплексов.
Как уже отмечено, модификация гистонов повышает доступность поврежденных оснований для ферментов репарации [94]. Как показано ранее, ацетилирование лизина-56 в гистоне Н3 влияет на стабильность NCP [95]. Если в клеточной ДНК нет повреждений, то фермент SIRT6 деацетилирует этот сайт на стадии G2 и более поздних стадиях клеточного цикла [96]. Если же ДНК содержит повреждения, то уровень K56Ac в H3 сохраняется. Это может способствовать репарации ДНК благодаря нейтрализации заряда на входе/выходе NCP [97]. Для подтверждения этого предположения в лаборатории S.H. Wilson проведено кинетическое исследование активности АРЕ1 при взаимодействии с NCP, содержащей H3K56Ac и H3K14Ac [98], и с ДНК Widom 601. Показано, что в Н33К56Ас-NCP разрезание цепи ДНК ферментом АРЕ1 протекает более эффективно, чем в неацетилированных NCP. Ацетилирование же альтернативного сайта H3K14Ac никак не влияло на активность АРЕ1. Ассоциированный с хроматином фактор HMGB1 (the high-mobility group box 1 protein) также усиливал действие АРЕ1. HMGB1 на порядок увеличивает активность АРЕ1 при взаимодействии со свободной ДНК в стационарных условиях [99]. Таким образом, этот белок выполняет двойственную функцию, влияя как на сам процесс BER, так и на структуру хроматина.
ЗАКЛЮЧЕНИЕ
Изучение процессов BER на нуклеосомах позволяет понять, какую роль играют позиционирование и природа повреждения, динамические свойства NCP и свойства фермента в репарации ДНК в составе хроматина. Репарация большинства повреждений в BER определяется их ротационной ориентацией и трансляционным положением, которые влияют на доступность поврежденного сайта для с фермента. Во многих работах показано, что большинство ДНК-гликозилаз наиболее эффективно удаляют поврежденное основание, ориентированное “наружу”, т.е. наиболее доступное для растворителя. Такие заключения сделаны для ферментов Udg, EndoIII и Fpg E. coli, UNG, NTHL1, NEIL1, AAG, APE1 человека. Изучена реакционная способность различных ДНК-гликозилаз, принадлежащих к четырем структурным суперсемействам из известных шести [54], в том числе как монофункциональных, так и бифункциональных, имеющих различную молекулярную массу. Показано, что некоторые из перечисленных ДНК-гликозилаз проявляли высокую активность по отношению к повреждениям, доступным для растворителя, другие были полностью неактивны. Чтобы понять причину таких различий в активности ДНК-гликозилаз при взаимодействии с нуклеосомами, сравнили их размеры, структуру и механизм действия [54]. Можно предположить возможность существования обратно пропорциональной зависимости между эффективностью удаления поврежденного основания и размером фермента, обусловленной стерическими причинами. Действительно, среди исследованных ферментов наиболее активно с NCP взаимодействуют небольшие ферменты Udg и AAG, имеющие массу 25.6 [100] и 24.3 кДа [101] соответственно. Меньше был только EndoIII – 23.5 кДа [102]. Форма и размеры ДНК-гликозилаз также могут быть важны для взаимодействия с NCP, поскольку гистоновый кор способен стерически препятствовать связыванию фермента со специфическим сайтом даже при Out-ориентации поврежденного основания. Моделирование возможных структур комплексов ДНК-гликозилаз с нуклеосомами и расчет расстояний между гистоновым октамером и ферментами выявили отсутствие ближних контактов между белками в случае Udg и AAG. Например, в UNG человека наименьшее расстояние (~10 Å) наблюдается между аминокислотными остатками Y275, R276 и гистонами. В AAG наименьшее расстояние было между остатком М164 и гистоном Н3 (7.8 Å). В другом предельном случае расстояние между К88 в ферменте Fpg и С-концом гистона Н2А составляет 2 Å, а между R31 и Н4 – 4.2 Å. Такие короткие расстояния свидетельствуют о возможном влиянии гистонового кора на взаимодействие Fpg со своим специфическим сайтом узнавания, что приводит к низкой активности фермента.
Эффективность взаимодействия ДНК-гликозилаз с NCP может зависеть не только от структурных характеристик фермента, но и от особенностей механизма катализа. Например, AAG и Udg, проявляющие высокую активность на NCP, изгибают свои ДНК-субстраты на 22° и 45°, тогда как малоактивные либо совсем неактивные EndoIII, Fpg и OGG1 изгибают ДНК на 55°, 66° и 70° соответственно. Очевидно, что чем больше угол изгиба, тем большее число связей между остовом ДНК и гистоновым кором нужно дестабилизировать. Поэтому ферментам Fpg и OGG1 для осуществления каталитического процесса требуется затратить больше энергии, чем AAG и UDG, которые изгибают ДНК-дуплекс не столь значительно. Это обстоятельство можно рассматривать как одну из причин различий в активности ферментов этих двух групп.
Кроме того, большое значение имеет собственная динамика нуклеосом и возможность их ремоделирования. Динамические свойства нуклеосом зависят как от свойств ДНК (нуклеотидной последовательности, природы повреждения), так и от свойств гистоновых белков (посттрансляционных модификаций, наличия мутаций) и влияния N-хвостов гистоновых белков на эффективность процессов, протекающих с участием ДНК, связанной с нуклеосомами [87, 103, 104].
Структура и принцип связывания BER-ферментов с ДНК определяют их способность узнавать и удалять повреждения в NCP. ДНК-гликозилазы и АР-эндонуклеаза АРЕ1 взаимодействуют преимущественно с одной цепью ДНК. Поэтому их активность зависит от ротационной ориентации поврежденного основания. Собственная динамика нуклеосом может иметь существенное значение при репарации повреждений вблизи концов ДНК. Для удаления повреждений, расположенных около диадной оси, ДНК-гликозилазами, эндонуклеазой АРЕ1 и полимеразой Polβ требуются ремоделирующие факторы. Активность этих ферментов на первых трех стадиях BER ниже, чем при взаимодействии со свободной ДНК, но при этом они способны “работать” с минимальным нарушением NCP. Однако лигирование рибозофосфатного остова LigIII-XRCC1, не столь сильно зависящее от ротационной ориентации, требует разрушения NCP [67].
Поскольку перечисленные параметры, определяющие доступность нуклеосомной ДНК, не являются взаимоисключающими, общая эффективность репарации каждого данного повреждения определяется совокупностью всех параметров.
Работа выполнена при поддержке гранта Министерства науки и высшего образования MD-3775.2019.4 и при частичной поддержке бюджетного финансирования (№ АААА-А17-117020210022-4).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Luger K., Mäder A.W., Richmond R.K., Sargent D.F., Richmond T.J. (1997) Crystal structure of the nucleosome core particle at 2.8 Å resolution. Nature. 389, 251–260.
Vasudevan D., Chua E.Y.D., Davey C.A. (2010) Crystal structures of nucleosome core particles containing the “601” strong positioning sequence. J. Mol. Biol. 403, 1–10.
Finch J.T., Lutter L.C., Rhodes D., Brown R.S., Rushton B., Levitt M., Klug A. (1977) Structure of nucleosome core particles of chromatin. Nature. 269, 29–36.
Richmond T.J., Finch J.T., Rushton B., Rhodes D., Klug A. (1984) Structure of the nucleosome core particle at 7 resolution. Nature. 311, 532–537.
Davey C.A., Sargent D.F., Luger K., Maeder A.W., Richmond T.J. (2002) Solvent mediated interactions in the structure of the nucleosome core particle at 1.9 Å resolution. J. Mol. Biol. 319, 1097–1113.
Richmond T.J., Davey C.A. (2003) The structure of DNA in the nucleosome core. Nature. 423, 145–150.
Tsunaka Y., Kajimura N., Tate S., Morikawa K. (2005) Alteration of the nucleosomal DNA path in the crystal structure of a human nucleosome core particle. Nucl. Acids Res. 33, 3424–3434.
Tachiwana H., Kagawa W., Osakabe A., Kawaguchi K., Shiga T., Hayashi-Takanaka Y., Kimura H., Kurumizaka H. (2010) Structural basis of instability of the nucleosome containing a testis-specific histone variant, human H3T. Proc. Natl. Acad. Sci. USA. 107, 10454–10459.
Ueda J., Harada A., Urahama T., Machida S., Maehara K., Hada M., Makino Y., Nogami J., Horikoshi N., Osakabe A., Taguchi H., Tanaka H., Tachiwana H., Yao T., Yamada M., Iwamoto T., Isotani A., Ikawa M., Tachibana T., Okada Y., Kimura H., Ohkawa Y., Kurumizaka H., Yamagat K. (2017) Testis-specific histone variant H3t gene is essential for entry into spermatogenesis. Cell Rep. 18, 593–600.
Harp J.M., Hanson B.L., Timm D.E., Bunick G.J. (2000) Asymmetries in the nucleosome core particle at 2.5 Å resolution. Acta Crystallogr. Sect. D: Biol. Crystallogr. 56, 1513–1534.
White C.L., Suto R.K., Luger K. (2001) Structure of the yeast nucleosome core particle reveals fundamental changes in internucleosome interactions. EMBO J. 20, 5207–5218.
Clapier C.R., Chakravarthy S., Petosa C., Fernández-Tornero C., Luger K., Müller C.W. (2008) Structure of the Drosophila nucleosome core particle highlights evolutionary constraints on the H2A-H2B histone dimer. Proteins Struct. Funct. Genet. 71, 1–7.
Chua E.Y.D., Vasudevan D., Davey G.E., Wu B., Davey C.A. (2012) The mechanics behind DNA sequence-dependent properties of the nucleosome. Nucl. Acids Res. 40, 6338–6352.
Lowary P.T., Widom J. (1998) New DNA sequence rules for high affinity binding to histone octamer and sequence-directed nucleosome positioning. J. Mol. Biol. 276, 19–42.
Polach K.J., Widom J. (1999) Restriction enzymes as probes of nucleosome stability and dynamics. Methods Enzymol. 304, 278–298.
Simpson R.T., Stafford D.W. (1983) Structural features of a phased nucleosome core particle. Proc. Natl. Acad. Sci. USA. 80, 51–55.
Long E.O., Dawid I.B. (1980) Repeated genes in eukaryotes. Annu. Rev. Biochem. 49, 727–764.
Pepenella S., Murphy K.J., Hayes J.J. (2014) Intra- and inter-nucleosome interactions of the core histone tail domains in higher-order chromatin structure. Chromosoma. 123, 3–13.
Mellor J. (2006) Dynamic nucleosomes and gene transcription. Trends Genet. 22(6), 320–329. https://doi.org/10.1016/j.tig.2006.03.008
Studitsky V.M., Kassavetis G.A., Geiduschek E.P., Felsenfeldt G. (1997) Mechanism of transcription through the nucleosome by eukaryotic RNA polymerase. Science. 278, 1960–1963.
Kulaeva O.I., Gaykalova D.A., Pestov N.A., Golovastov V.V., Vassylyev D.G., Artsimovitch I., Studitsky V.M. (2009) Mechanism of chromatin remodeling and recovery during passage of RNA polymerase II. Nat. Struct. Mol. Biol. 16, 1272–1278.
Nacheva G.A., Guschin D.Y., Preobrazhenskaya O.V., Karpov V.L., Ebralidse K.K., Mirzabekov A.D. (1989) Change in the pattern of histone binding to DNA upon transcriptional activation. Cell. 58, 27–36.
Liu Y., Wilson S.H. (2012) DNA base excision repair: A mechanism of trinucleotide repeat expansion. Trends Biochem. Sci. 37, 162–172.
Esadze A., Stivers J.T. (2018) Facilitated diffusion mechanisms in DNA base excision repair and transcriptional activation. Chem. Rev. 118, 11298–11323.
Rodriguez Y., Hinz J.M., Smerdon M.J. (2015) Accessing DNA damage in chromatin: Preparing the chromatin landscape for base excision repair. DNA Repair (Amst). 32, 113–119.
Jagannathan I., Cole H.A., Hayes J.J. (2006) Base excision repair in nucleosome substrates. Chromosom. Res. 14, 27–37.
Polach K.J., Widom J. (1995) Mechanism of protein access to specific DNA sequences in chromatin: a dynamic equilibrium model for gene regulation. J. Mol. Biol. 254, 130–149.
Li G., Levitus M., Bustamante C., Widom J. (2005) Rapid spontaneous accessibility of nucleosomal DNA. Nat. Struct. Mol. Biol. 12, 46–53.
Anderson J.D., Widom J. (2000) Sequence and position-dependence of the equilibrium accessibility of nucleosomal DNA target sites. J. Mol. Biol. 296, 979–987.
Tims H.S., Gurunathan K., Levitus M., Widom J. (2011) Dynamics of nucleosome invasion by DNA binding proteins. J. Mol. Biol. 411, 430–448.
Krokan H.E., Drabløs F., Slupphaug G. (2002) Uracil in DNA – occurrence, consequences and repair. Oncogene. 21, 8935–8948.
Andersen S., Heine T., Sneve R., König I., Krokan H.E., Epe B., Nilsen H. (2004) Incorporation of dUMP into DNA is a major source of spontaneous DNA damage, while excision of uracil is not required for cytotoxicity of fluoropyrimidines in mouse embryonic fibroblasts. Carcinogenesis. 26, 547–555.
Kavli B., Otterlei M., Slupphaug G., Krokan H. (2007) Uracil in DNA–general mutagen, but normal intermediate in acquired immunity. DNA Repair (Amst.). 6, 505–516.
Lindahl T. (1993) Instability and decay of the primary structure of DNA. Nature. 362, 709–715.
Cortazar D., Kunz C., Saito Y., Steinacher R., Schar P. (2007) The enigmatic thymine DNA glycosylase. DNA Repair (Amst.). 6, 489–504.
Neddermann P., Jiricny J. (1993) The purification of a mismatch-specific thymine-DNA glycosylase from HeLa cells. J. Biol. Chem. 268, 21218–21224.
Neddermann P., Jiricny J. (1994) Efficient removal of uracil from G.U mispairs by the mismatch-specific thymine DNA glycosylase from HeLa cells. Proc. Natl. Acad. Sci. USA. 91, 1642–1646.
Wibley J.E.A., Waters T.R., Haushalter K., Verdine G.L., Pearl L.H. (2003) Structure and specificity of the vertebrate anti-mutator uracil-DNA glycosylase SMUG1. Mol. Cell. 11, 1647–1659.
Masaoka A., Matsubara M., Hasegawa R., Tanaka T., Kurisu S., Terato H., Ohyama Y., Karino N., Matsuda A., Ide H. (2003) Mammalian 5-formyluracil-DNA glycosylase. 2. role of SMUG1 uracil-DNA glycosylase in repair of 5-formyluracil and other oxidized and deaminated base lesions. Biochemistry. 42, 5003–5012.
Hendrich B., Bird A. (1998) Identification and characterization of a family of mammalian methyl-CpG binding proteins. Mol. Cell. Biol. 18, 6538–6547.
Hendrich B., Hardeland U., Ng H.H., Jiricny J., Bird A. (1999) The thymine glycosylase MBD4 can bind to the product of deamination at methylated CpG sites. Nature. 401, 301–304.
Nilsen H., Lindahl T., Verreault A. (2002) DNA base excision repair of uracil residues in reconstituted nucleosome core particles. EMBO J. 21, 5943–5952.
Beard B.C., Wilson S.H., Smerdon M.J. (2003) Suppressed catalytic activity of base excision repair enzymes on rotationally positioned uracil in nucleosomes. Proc. Natl. Acad. Sci. USA. 100, 7465–7470.
Beard B.C., Stevenson J.J., Wilson S.H., Smerdon M.J. (2005) Base excision repair in nucleosomes lacking histone tails. DNA Repair (Amst.). 4, 203–209.
Ishibashi T., So K., Cupples C.G., Ausio J. (2008) MBD4-mediated glycosylase activity on a chromatin template is enhanced by acetylation. Mol. Cell. Biol. 28, 4734–4744.
Cole H.A., Tabor-Godwin J.M., Hayes J.J. (2010) Uracil DNA glycosylase activity on nucleosomal DNA depends on rotational orientation of targets. J. Biol. Chem. 285, 2876–2885
Hinz J.M., Rodriguez Y., Smerdon M.J. (2010) Rotational dynamics of DNA on the nucleosome surface markedly impact accessibility to a DNA repair enzyme. Proc. Natl. Acad. Sci. USA. 107, 4646–4651.
Kosmoski J.V., Smerdon M.J. (1999) Synthesis and nucleosome structure of DNA containing a UV photoproduct at a specific site. Biochemistry. 38, 9485–9494.
Rodriguez Y., Smerdon M.J. (2013) The structural location of DNA lesions in nucleosome core particles determines accessibility by base excision repair enzymes. J. Biol. Chem. 288, 13863–13875.
Tarantino M.E., Dow B.J., Drohat A.C., Delaney S. (2018) Nucleosomes and the three glycosylases: high, medium, and low levels of excision by the uracil DNA glycosylase superfamily. DNA Repair (Amst.). 72, 56–63.
Olsen L.C., Aasland R., Wittwer C.U., Krokan H.E., Helland D.E. (1989) Molecular cloning of human uracil-DNA glycosylase, a highly conserved DNA repair enzyme. EMBO J. 8, 3121–3125.
Xiao G., Tordova M., Jagadeesh J., Drohat A.C., Stivers J.T., Gilliland G.L. (1999) Crystal structure of Escherichia coli uracil DNA glycosylase and its complexes with uracil and glycerol: Structure and glycosylase mechanism revisited. Proteins Struct. Funct. Genet. 35, 13–24.
Wu B., Mohideen K., Vasudevan D., Davey C.A. (2010) Structural insight into the sequence dependence of nucleosome positioning. Structure. 18, 528–536.
Olmon E.D., Delaney S. (2017) Differential ability of five DNA glycosylases to recognize and repair damage on nucleosomal DNA. ACS Chem. Biol. 12, 692–701.
Hazra T.K., Izumi T., Boldogh I., Imhoff B., Kow Y.W., Jaruga P., Dizdaroglu M., Mitra S. (2002) Identification and characterization of a human DNA glycosylase for repair of modified bases in oxidatively damaged DNA. Proc. Natl. Acad. Sci. USA. 99, 3523–3528.
Bandaru V., Sunkara S., Wallace S.S., Bond J.P. (2002) A novel human DNA glycosylase that removes oxidative DNA damage and is homologous to Escherichia coli endonuclease VIII. DNA Repair (Amst.). 1, 517–529.
Doublie S., Bandaru V., Bond J.P., Wallace S.S. (2004) The crystal structure of human endonuclease VIII-like 1 (NEIL1) reveals a zincless finger motif required for glycosylase activity. Proc. Natl. Acad. Sci. USA. 101, 10284–10289.
Yang N., Chaudhry M.A., Wallace S.S. (2006) Base excision repair by hNTH1 and hOGG1: a two edged sword in the processing of DNA damage in γ-irradiated human cells. DNA Repair (Amst.). 5, 43–51.
Ide H., Kotera M. (2004) Human DNA glycosylases involved in the repair of oxidatively damaged DNA. Biol. Pharm. Bull. 27, 480–485.
Miyabe I., Zhang Q.-M., Kino K., Sugiyama H., Takao M., Yasui A., Yonei S. (2002) Identification of 5-formyluracil DNA glycosylase activity of human hNTH1 protein. Nucl. Acids Res. 30, 3443–3448.
Prasad A., Wallace S.S., Pederson D.S. (2007) Initiation of base excision repair of oxidative lesions in nucleosomes by the human, bifunctional DNA glycosylase NTH1. Mol. Cell. Biol. 27, 8442–8453.
Liu X., Choudhury S., Roy R. (2003) In vitro and in vivo dimerization of human endonuclease III stimulates its activity. J. Biol. Chem. 278, 50061–50069.
Odell I.D., Newick K., Heintz N.H., Wallace S.S., Pederson D.S. (2010) Non-specific DNA binding interferes with the efficient excision of oxidative lesions from chromatin by the human DNA glycosylase, NEIL1. DNA Repair (Amst.). 9, 134–143.
Dou H., Mitra S., Hazra T.K. (2003) Repair of oxidized bases in DNA bubble structures by human dNA glycosylases NEIL1 and NEIL2. J. Biol. Chem. 278, 49679–49684.
Dou H., Theriot C.A., Das A., Hegde M.L., Matsumoto Y., Boldogh I., Hazra T.K., Bhakat K.K., Mitra S. (2008) Interaction of the human DNA glycosylase NEIL1 with proliferating cell nuclear antigen: the potential for replication-associated repair of oxidized bases in mammalian genomes. J. Biol. Chem. 283, 3130–3140.
Hegde M.L., Theriot C.A., Das A., Hegde P.M., Guo Z., Gary R.K., Hazra T.K., Shen B., Mitra S. (2008) Physical and functional interaction between human oxidized base-specific DNA glycosylase NEIL1 and flap endonuclease. J. Biol. Chem. 283, 27028–27037.
Odell I.D., Barbour J.-E., Murphy D.L., Della-Maria J.A., Sweasy J.B., Tomkinson A E., Wallace S.S., Pederson D.S. (2011) Nucleosome disruption by DNA ligase III-XRCC1 promotes efficient base excision repair. Mol. Cell. Biol. 31, 4623–4632.
Maher R.L., Prasad A., Rizvanova O., Wallace S.S., Pederson D.S. (2013) Contribution of DNA unwrapping from histone octamers to the repair of oxidatively damaged DNA in nucleosomes. DNA Repair (Amst.). 12, 964–971.
Maher R.L., Wallace S.S., Pederson D.S. (2019) The lyase activity of bifunctional DNA glycosylases and the 3'-diesterase activity of APE1 contribute to the repair of oxidized bases in nucleosomes. Nucl. Acids Res. 47, 2922–2931.
Fromme J.C., Bruner S.D., Yang W., Karplus M., Verdine G.L. (2003) Product-assisted catalysis in base-excision DNA repair. Nat. Struct. Mol. Biol. 10, 204–211.
Fromme J.C., Verdine G.L. (2003) DNA lesion recognition by the bacterial repair enzyme MutM. J. Biol. Chem. 278, 51543–51548.
Rydberg B., Lindahl T. (1982) Nonenzymatic methylation of DNA by the intracellular methyl group donor S-adenosyl-L-methionine is a potentially mutagenic reaction. EMBO J. 1, 211–216.
Marnett L.J. (2002) Oxy radicals, lipid peroxidation and DNA damage. Toxicology. 181–182, 219–222.
Dosanjh M.K., Chenna A., Kim E., Fraenkel-Conrat H., Samson L., Singer B. (1994) All four known cyclic adducts formed in DNA by the vinyl chloride metabolite chloroacetaldehyde are released by a human DNA glycosylase. Proc. Natl. Acad. Sci. USA. 91, 1024–1028.
Rydberg B., Qiu Z.H., Dosanjh M.K., Singer B. (1992) Partial purification of a human DNA glycosylase acting on the cyclic carcinogen adduct 1,N6-ethenodeoxyadenosine. Cancer Res. 52, 1377–1179.
O’Brien P.J., Ellenberger T. (2004) Dissecting the broad substrate specificity of human 3-methyladenine-DNA glycosylase. J. Biol. Chem. 279, 9750–9757.
O’Connor T.R. (1993) Purification and characterization of human 3-methyladenine-DNA glycosylase. Nucl. Acids Res. 21, 5561–5569.
Kennedy E.E., Li C., Delaney S. (2019) Global repair profile of human alkyladenine DNA glycosylase on nucleosomes reveals DNA packaging effects. ACS Chem. Biol. 14, 1687–1692.
Wang Y. (2008) Bulky DNA lesions induced by reactive oxygen species. Chem. Res. Toxicol. 21, 276–281.
Zuo S., Boorstein R.J., Teebor G.W. (1995) Oxidative damage to 5-methylcytosine in DNA. Nucl. Acids Res. 23, 3239–3243.
Douki T., Delatour T., Paganon F., Cadet J. (1996) Measurement of oxidative damage at pyrimidine bases in γ-irradiated DNA. Chem. Res. Toxicol. 9, 1145–1151.
Bjelland S., Seeberg E. (2003) Mutagenicity, toxicity and repair of DNA base damage induced by oxidation. Mutat. Res. 531, 37–80.
Zhang Y., Yuan F., Wu X., Taylor J.S., Wang Z. (2001) Response of human DNA polymerase iota to DNA lesions. Nucl. Acids Res. 29, 928–935.
Hanes J.W., Thal D.M., Johnson K.A. (2006) Incorporation and replication of 8-oxo-deoxyguanosine by the human mitochondrial DNA polymerase. J. Biol. Chem. 281, 36241–36248.
Menoni H., Shukla M.S., Gerson V., Dimitrov S., Angelov D. (2012) Base excision repair of 8-oxoG in dinucleosomes. Nucl. Acids Res. 40, 692–700.
Cannan W.J., Tsang B.P., Wallace S.S., Pederson D.S. (2014) Nucleosomes suppress the formation of double-strand DNA breaks during attempted base excision repair of clustered oxidative damages. J. Biol. Chem. 289, 19881–19893.
Hinz J.M., Mao P., McNeill D.R., Wilson D.M. (2015) Reduced nuclease activity of apurinic/apyrimidinic endonuclease (APE1) variants on nucleosomes: identification of access residues. J. Biol. Chem. 290, 21067–21075.
Cannan W.J., Rashid I., Tomkinson A.E., Wallace S.S., Pederson D.S. (2017) The human ligase IIIα-XRCC1 protein complex performs DNA nick repair after transient unwrapping of nucleosomal DNA. J. Biol. Chem. 292, 5227–5238.
Lee T.H. (2019) Physical chemistry of epigenetics: single-molecule investigations. J. Phys. Chem. B. 123, 8351–8362.
Li C., Delaney S. (2019) Histone H2A variants enhance the initiation of base excision repair in nucleosomes. ACS Chem. Biol. 14, 1041–1050.
Zlatanova J., Thakar A. (2008) H2A.Z: view from the top. Structure. 16, 166–179.
Billon P., Côté J. (2012) Precise deposition of histone H2A.Z in chromatin for genome expression and maintenance. Biochim. Biophys. Acta – Gene Regul. Mech. 1819, 290–302.
Yu Y., Deng Y., Reed S.H., Millar C.B., Waters R. (2013) Histone variant Htz1 promotes histone H3 acetylation to enhance nucleotide excision repair in Htz1 nucleosomes. Nucl. Acids Res. 41, 9006–9019.
Bowman G.D., Poirier M.G. (2015) Post-translational modifications of histones that influence nucleosome dynamics. Chem. Rev. 115, 2274–2295.
North J.A., Shimko J.C., Javaid S., Mooney A.M., Shoffner M.A., Rose S.D., Bundschuh R., Fishel R., Ottesen J.J., Poirier M.G. (2012) Regulation of the nucleosome unwrapping rate controls DNA accessibility. Nucl. Acids Res. 40, 10215–10227.
Michishita E., McCord R.A., Boxer L.D., Barber M.F., Hong T., Gozani O., Chua K.F. (2009) Cell cycle-dependent deacetylation of telomeric histone H3 lysine K56 by human SIRT6. Cell Cycle. 8, 2664–2666.
Maas N.L., Miller K.M., DeFazio L.G., Toczyski D.P. (2006) Cell cycle and checkpoint regulation of histone H3 K56 acetylation by Hst3 and Hst4. Mol. Cell. 23, 109–119.
Rodriguez Y., Horton J.K., Wilson S.H. (2019) Histone H3 lysine 56 acetylation enhances AP endonuclease 1-mediated repair of AP sites in nucleosome core particles. Biochemistry. 58, 3646–3655.
Prasad R., Liu Y., Deterding L.J., Poltoratsky V.P., Kedar P.S., Horton J.K., Kanno S.I., Asagoshi K., Hou E.W., Khodyreva S.N., Lavrik O.I., Tomer K.B., Yasui A., Wilson S.H. (2007) HMGB1 is a cofactor in mammalian base excision repair. Mol. Cell. 27, 829–841.
Bennett S.E., Sanderson R.J., Mosbaugh D.W. (1995) Processivity of Escherichia coli and rat liver mitochondrial uracil-DNA glycosylase is affected by NaCl concentration. Biochemistry. 34, 6109–6119.
Lau A.Y., Wyatt M.D., Glassner B.J., Samson L.D., Ellenberger T. (2000) Molecular basis for discriminating between normal and damaged bases by the human alkyladenine glycosylase AAG. Proc. Natl. Acad. Sci. USA. 97, 13573–13578.
Asahara H., Wistort P.M., Bank J.F., Bakerian R.H., Cunningham R.P. (1989) Purification and characterization of Escherichia coli endonuclease III from the cloned nth gene. Biochemistry. https://doi.org/10.1021/bi00436a048
Widlund H.R., Vitolo J.M., Thiriet C., Hayes J.J. (2000) DNA sequence-dependent contributions of core histone tails to nucleosome stability: differential effects of acetylation and proteolytic tail removal. Biochemistry. 39, 3835–3841.
Yang Z., Zheng C., Hayes J.J. (2007) The core histone tail domains contribute to sequence-dependent nucleosome positioning. J. Biol. Chem. 282, 7930–7938.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология