Молекулярная биология, 2021, T. 55, № 2, стр. 305-311
Матричные свойства 5-метил-2'-дезоксицитидина и 5-гидроксиметил-2'-дезоксицитидина в реакциях с транслезионными и репаративными ДНК-полимеразами человека
Е. С. Шилкин a, Д. В. Петрова b, В. А. Полтораченко a, Е. О. Болдинова a, Д. О. Жарков b, c, *, А. В. Макарова a, **
a Научно-исследовательский центр “Курчатовский институт” – Институт молекулярной генетики
123182 Москва, Россия
b Институт химической биологии и фундаментальной медицины Сибирского отделения
Российской академии наук
630090 Новосибирск, Россия
c Новосибирский государственный университет
630090 Новосибирск, Россия
* E-mail: dzharkov@niboch.nsc.ru
** E-mail: amakarova-img@yandex.ru
Поступила в редакцию 30.08.2020
После доработки 18.09.2020
Принята к публикации 24.09.2020
Аннотация
5-Метил-2'-дезоксицитидин (mC) и продукт его регулируемого окисления 5-гидроксиметил-2'-дезоксицитидин (hmC) играют ключевую роль в эпигенетической регуляции экспрессии генов, в клеточной дифференцировке и канцерогенезе. Геномные СpG-сайты, содержащие mC и hmC, представляют собой горячие точки мутагенеза. Основной механизм мутагенеза в CpG-сайтах – спонтанное дезаминирование модифицированных остатков цитозина. Однако дополнительным источником мутаций в районах СpG-сайтов, содержащих повреждения, потенциально могут быть высокоошибочные транслезионные и репаративные ДНК-полимеразы. В настоящей работе проанализирована точность включения нуклеотидов напротив mC и hmC ДНК-полимеразами человека Polβ, Polλ, Polη, Polι, Polκ и праймазой-полимеразой PrimPol in vitro. Результаты исследования указывают на высокую точность копирования mC и hmC репаративными ДНК-полимеразами Polβ и Polλ. Polη, Polι, Polκ и PrimPol копировали mC и hmC с меньшей точностью и включали напротив них dAMP и dTMP. Такой же спектр ошибочного включения dNMP наблюдали и в сайтах с немодифицированными остатками цитозина.
ВВЕДЕНИЕ
Эпигенетическое метилирование ДНК по положению C5 2'-дезоксицитидина с образованием 5-метил-2'-дезоксицитидина (mC) – один из ключевых механизмов, регулирующих компактизацию хроматина и экспрессию генов высших эукариот [1–3]. Метилированию подвергаются преимущественно динуклеотиды CpG, и 70–80% сайтов CpG в геноме человека содержат mC [4, 5]. В эпигенетической регуляции участвует и продукт регулируемого окисления mC – 5-гидроксиметил-2'-дезоксицитидин (hmC) [6, 7]. mC превращается в hmC под действием диоксигеназ семейства TET [8, 9].
Динуклеотиды CpG представляют собой горячие точки мутагенеза. Основной тип мутаций в CpG-сайтах – транзиции СG → TA, образуются в результате спонтанного дезаминирования mC до T и последующего включения dAMP при репликации [10, 11]. Известно, что эффективность репарации пар TG ниже, чем dUG [10, 11]. Мутации может вызывать также 5-гидроксиметил-2'-дезоксиуридин (hmU), образующийся в результате последовательного окисления mC до hmC и дезаминирования hmC [12].
Первичными мутагенными повреждениями в динуклеотидах CpG могут быть не только основания Thy и hmUra, образующиеся в ходе дезаминирования. Присутствие mC может повышать способность комплементарного или соседнего с ним G к окислению [13] и образованию аддуктов с канцерогенами [14, 15], а сам mC под действием ультрафиолетового излучения дает ряд промутагенных продуктов [16–18]. В последние годы накапливаются данные о том, что часть мутаций, образующихся в горячих точках мутагенеза в области CpG-сайтов, возникает в результате работы ДНК-полимераз в ходе нормальной репликации ДНК и не связана с дезаминированием mC [19, 20].
Помимо высокоточных репликативных ДНК-полимераз α, δ и ε (Polα, Polδ, Polε) в клетках человека присутствуют специализированные ДНК-полимеразы. Репаративные полимеразы β, θ, λ и μ (Polβ, Polθ, Polλ, Polμ), принадлежащие к семействам X и А, отвечают за синтез ДНК при эксцизионной репарации нуклеотидов и негомологичном соединении концов ДНК [21]. Транслезионные ДНК-полимеразы η, ι и κ (Polη, Polι, Polκ) семейства Y и ДНК-полимераза ζ (Polζ) семейства B включают нуклеотиды напротив повреждений, предотвращая коллапс движущейся репликативной вилки на поврежденной матрице [22–24]. Праймаза-ДНК-полимераза PrimPol выполняет функцию реинициации синтеза ДНК после поврежденных участков [25]. Точность специализированных ДНК-полимераз при синтезе на неповрежденной ДНК-матрице значительно ниже, чем у репликативных ДНК-полимераз [23, 26].
По частоте возникновения спонтанных повреждений, затрагивающих динуклеотиды CpG, можно выделить дезаминирование C и mC и окисление G с образованием 8-оксо-7,8-дигидро-2'-дезоксигуанозина (oxoG) [27]. Репарация продуктов повреждения в составе пар dUG, dU-oxoG и T-oxoG протекает по пути эксцизионной репарации оснований с вовлечением Polβ или Polλ. Если oxoG не был удален до начала репликации, то для его “прохождения” привлекаются транслезионные ДНК-полимеразы [28]. Таким образом, как mC, так и hmC могут выступать в качестве матричных нуклеотидов для включения dNMP репаративными и транслезионными ДНК-полимеразами. Однако вопрос о точности включения нуклеотидов напротив mC и hmC ДНК-полимеразой человека не исследован. В настоящей работе изучена точность и эффективность включения dNMP напротив mC и hmC панелью репаративных и транслезионных ДНК-полимераз человека – Polβ, Polλ, Polη, Polι, Polκ и PrimPol in vitro.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Белки. Polι выделяли из клеток Saccharomyces cerevisiae согласно [29]. Polη экспрессировали с использованием плазмиды, полученной в [29], и выделяли из клеток S. cerevisiae, аналогично Polζ [30] и Rev1 [31]. PrimPol экспрессировали в клетках Escherichia coli и выделяли согласно [32]. Polβ и Polλ, содержащие на N-конце дополнительную последовательность с шестью остатками His, выделяли из клеток E. coli методом аффинной хроматографии на Ni-NTA (“Qiagen”, Германия) с последующей очисткой на гепарин-сефарозе (“GE Healthcare”, США). Препарат Polκ, выделенный из клеток E. coli, любезно предоставлен Л.В. Генингом (НИЦ Курчатовский институт – ИМГ).
ДНК-субстраты. Праймер и ДНК-матрицы, содержащие С, mC и hmC, синтезированы в ИХБФМ СО РАН из коммерчески доступных амидофосфитов (“Glen Research”, США). Олигонуклеотиды, содержащие модифицированные основания, при гидролизе метилзависимой эндонуклеазой рестрикции GlaI (“Сибэнзим”, Россия) давали продукт с ожидаемой электрофоретической подвижностью [33]. Для получения ДНК-субстратов [5′-32P]-меченый праймер гибридизовали с немеченными олигонуклеотидными матрицами в соотношении 1 : 1.1, нагревая раствор до 73°C и медленно охлаждая до 24°C. Последовательности олигонуклеотидов представлены на рис. 1.
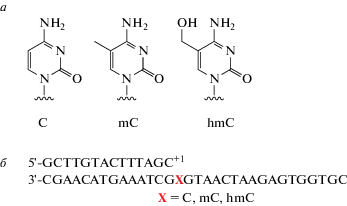
Рис. 1.
Структура использованного ДНК-субстрата. a – Структуры С, mC и hmC. б – Нуклеотидная последовательность цепей ДНК-субстрата.
Анализ активности ДНК-полимераз. Активность ДНК-полимераз оценивали в реакции удлинения праймера в смеси объемом 20 мкл, содержащей 30 мМ HEPES–KOH (рН 7.0), 8% глицерина, 0.1 мг/мл бычьего сывороточного альбумина, 20 нМ ДНК-субстрат, MgCl2, dNTP и ДНК-полимеразы в концентрациях, указанных в табл. 1. Реакции инициировали добавлением dNTP и инкубировали при 37°C в течение времени, указанного в табл. 1. Реакции останавливали, добавляя 20 мкл буфера для нанесения (95% формамида, 10 мМ EDТА и 0.1% бромфенолового синего). Продукты реакции разделяли в денатурирующем 21%-ном полиакриламидном геле с добавлением 7 М мочевины и детектировали с помощью радиолюминесцентного сканера Typhoon 9400 (“GE Healthcare”). Об эффективности включения dNTP судили по проценту удлиненного праймера (PrEx) – соотношению между удлиненными молекулами ДНК (продуктами реакции) и всеми молекулами ДНК в дорожке. Эксперименты повторяли 3–4 раза, рассчитывали стандартные ошибки средних значений. Данные сравнивали с помощью t-критерия Стьюдента, различия считали статистически значимыми при p < 0.05.
Таблица 1.
Условия определения ДНК-полимеразной активности
| ДНК-полимераза | MgCl2, мМ | dNTP, мкМ | ДНК-полимераза, нМ | Время реакции, мин |
|---|---|---|---|---|
| Polη | 10 | 50 | 5 | 4 |
| Polκ | 10 | 50 | 5 | 2 |
| Polι | 1 | 50 | 10 | 8 |
| Polβ | 10 | 50 | 5 | 5 |
| Polλ | 1 | 50 | 100 | 10 |
| PrimPol | 10 | 200 | 200 | 15 |
Анализ включения индивидуальных нуклеотидов в условиях стационарной кинетики. Для измерения эффективности включения индивидуальных dNTP концентрации dNTP в реакционной смеси варьировали от 1 до 1000 мкМ. Реакции проводили при 37°С в течение 4–30 мин (включение dGTP напротив C, mC и hmC – 4 мин, включение dCTP напротив С – 10 мин, включение dCTP напротив mC и hmC – 30 мин). Полученные данные использовали для расчета параметров kcat и KM в уравнении Михаэлиса–Ментен kobs = kcat × × [dNTP])/(KM + [dNTP]) с помощью нелинейной регрессии в программе GraFit 5 (Erithacus Software, Великобритания), где kobs и kcat – наблюдаемая и максимальная скорости реакции, соответственно, KM – кажущаяся константа Михаэлиса. Значения параметров KM и kcat использовали для определения эффективности включения нуклеотида (kcat/KM) и коэффициента эффективности включения нуклеотида напротив модифицированного С (Finc – отношение kcat/KM включения dNМP напротив модифицированного С и kcat/KM включения dNMP напротив С). Эксперименты повторяли 3 раза и рассчитывали ошибку среднего.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
В работе использовали двухцепочечный ДНК-субстрат с выступающим одноцепочечным 5′‑участком, в котором С, mC или hmC (рис. 1а) находились в положении +1 матричной цепи в нуклеотидном окружении –G(X)G– (рис. 1б). Точность включения нуклеотидов напротив mC и hmC оценивали с использованием индивидуальных dNMP в реакциях удлинения праймера. Транслезионные ДНК-полимеразы семейства Y (Polη, Polι и Polκ) показали наименьшую точность копирования неповрежденного матричного С. Хотя все ДНК-полимеразы семейства Y включали напротив С преимущественно комплементарный dGMP (рис. 2а, дорожка 3; рис. 2б, дорожка 3; рис. 2в, дорожка 3), наблюдалось также включение некомплементарных dAMP (рис. 2а, дорожка 2; рис. 2б дорожка 2; рис. 2в, дорожка 2) и dTMP (рис. 2а, дорожка 4; рис. 2б, дорожка 4; рис. 2в, дорожка 4). Polι включала нуклеотидные субстраты напротив С с наименьшей точностью (рис. 2б). Полученные результаты согласуются с данными о невысокой точности копирования неповрежденной матрицы ДНК-полимеразами семейства Y [34–36].
Рис. 2.
Включение dNTP ДНК-полимеразами человека: Polη (а), Polι (б), Polκ (в), Polβ (г), Polλ (д) и праймазой-полимеразой PrimPol (е) напротив С, mC и hmC. Показаны значения удлинения праймера (PrEx, %) и стандартные ошибки средних значений.
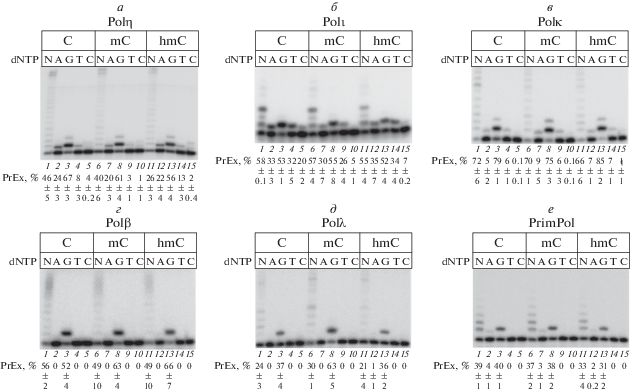
Все ДНК-полимеразы семейства Y эффективно включали dNMP напротив С, mC и hmC (рис. 2а–в). Спектр включения dNMP напротив mC и hmC не отличался существенно от спектра включения нуклеотидных субстратов напротив С полимеразами Polη и Polκ (рис. 2а, в). Однако Polι включала dCMP напротив mC и hmC с меньшей эффективностью по сравнению с немодифицированным С (рис. 2б). Включение комплементарного dGMP и некомплементарного dCMP Polι изучено более детально c помощью стационарной кинетики (табл. 2). Оказалось, что Polι включает dCMP напротив mC и hmC в 4–7.1 раза менее эффективно по сравнению с С. Кроме того, Polι в 3.1–5 раз менее эффективно включала напротив mC и hmC комплементарный dGMP (табл. 2). Таким образом, наличие 5-метильной и 5-гидроксиметильной групп в несколько раз снижает активность Polι как при нормальном, так и при мутагенном включении dNMP напротив С.
Таблица 2.
Включение dGMP и dCMP напротив С, mC и hmC Polι человека
| Субстратный dNTP | Матричный нуклеотид | kcat, мин−1 | KM, мкM | kcat/KM, мкМ−1 · мин−1 | Finc |
|---|---|---|---|---|---|
| dGTP | C | 0.199 ± 0.035 | 20.4 ± 2 | 9.7 × 10−3 | 1 |
| mC | 0.188 ± 0.026 | 95 ± 12.8 | 2 × 10−3 | 0.2 | |
| hmC | 0.163 ± 0.027 | 54.6 ± 6.6 | 3.1 × 10−3 | 0.32 | |
| dCTP | C | 0.135 ± 0.027 | 164.6 ± 0.4 | 8.2 × 10−4 | 1 |
| mC | 0.035 ± 0.001 | 315 ± 58.3 | 1.1 × 10−4 | 0.14 | |
| hmC | 0.043 ± 0.011 | 214.6 ± 74.5 | 2.1 × 10−4 | 0.25 |
Репаративные ДНК-полимеразы Polβ и Polλ семейства X включали напротив матричного С только комплементарный dGMP (рис. 2г, дорожка 3 и рис. 2д, дорожка 3). Метильная или гидроксиметильная группы в положении 5 пиримидинового кольца не влияли на точность включения нуклеотидных субстратов напротив С (рис. 2г, дорожки 6–15 и рис. 2д, дорожки 6–15). Интересно отметить, что Polλ включала dGMP напротив mC (рис. 2д, дорожка 8) примерно в 2 раза эффективнее, чем напротив немодифицированного С (p = 0.01) и hmC (p = 0.0004) (рис. 2д, дорожки 3 и 13). Таким образом, репаративные Polβ и Polλ копировали mC и hmC с высокой точностью и эффективностью.
PrimPol включала напротив С преимущественно комплементарный dGMP (рис. 2е, дорожка 3) и небольшое количество некомплементарного dAMP (рис. 2е, дорожка 2). Спектр включения dNMP напротив mC и hmC существенно не отличался от немодифицированного С (рис. 2е, дорожки 6–15).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
ДНК-полимеразы семейств Y и X и праймаза-полимераза PrimPol, участвующие в репарации и репликации поврежденной ДНК, не обладают корректирующей 3′ → 5′-экзонуклеазной активностью и копируют неповрежденную ДНК с невысокой точностью [23, 26, 37]. В ходе репарации повреждений в динуклеотидах CpG или репликации таких матриц репаративные и транслезионные ДНК-полимеразы могут встретить в матрице mC и hmC. Ошибки при копировании mC и hmC могут быть одним из источников мутагенеза, ассоциированного с CpG. Действительно, некоторые недавние исследования указывают на участие ДНК-полимераз (в частности, репликативной Polε и транслезионной Polη) в мутагенезе по динуклеотидам CpG независимо от дезаминирования mС [19, 38–41].
Нами впервые оценена точность и эффективность включения dNTP напротив mC и hmC шестью специализированными ДНК-полимеразами человека. Показано, что репаративные ДНК-полимеразы семейства X копируют mC и hmC с высокой точностью. Polλ включала комплементарный dGMP напротив mC даже более эффективно, чем напротив немодифицированного С.
Транслезионные ДНК-полимеразы семейства Y и PrimPol включали нуклеотидные субстраты с меньшей точностью. В мутационном спектре динуклеотидов CpG доминируют транзиции СG → → TA [42, 43]. Все ДНК-полимеразы семейства Y и PrimPol включали напротив mC и hmC некомплементарный dATP, что может быть причиной таких мутаций. Эффективность включения dATP напротив mC и hmC убывала в ряду Polι > Polη > > Polκ ~ PrimPol. Однако общий спектр включения dNTP напротив mC и hmC существенно не отличался от немодифицированного С.
Таким образом, полученные данные свидетельствуют о том, что, по крайней мере в отсутствие дезаминирования матричного C или его производных mC и hmC, репаративные ДНК-полимеразы не вносят заметного вклада в мутагенез по CpG-сайтам. Транслезионные ДНК-полимеразы Polη, Polι и Polκ и праймаза-полимераза PrimPol могут быть источником мутаций в CpG-сайтах. В клетке такая ситуация может возникать при прохождении репликативной вилкой остатка oxoG в контексте CpG с привлечением транслезионных ДНК-полимераз [28]. Оценки встречаемости динуклеотида CpG [4, 5] и частоты oxoG в геноме человека [44, 45] позволяют утверждать, что каждая клетка содержит около 100 Cp(oxoG)-сайтов. Поскольку включение некомплементарных dNMP полимеразами семейства Y оказалось сопоставимым для C, mC и hmC, их промутагенное действие должно быть независимым от статуса метилирования ДНК.
Следует отметить, что нами изучены ДНК-матрицы, содержащие C, mC или hmC только в одном нуклеотидном контексте – GCG. Возможно, в других контекстах или в случае повреждений точность и эффективность включения dNMP ДНК-полимеразами может отличаться. Например, спектры соматических мутаций в некоторых опухолях указывают на возможную роль Polη в мутагенезе по остаткам C в контексте YCG (где Y – пиримидин, но чаще T), которая не зависит от дезаминирования цитозина [39]. Таким образом, для установления механизмов мутагенеза в динуклеотидах CpG перспективным представляется изучение точности транслезионных полимераз при копировании эпигенетически модифицированных оснований ДНК в расширенном наборе нуклеотидных контекстов.
Авторы выражают благодарность Л.В. Генингу (ИМГ) за предоставление Polκ, О.И. Лаврик (ИХБФМ СО РАН) за экспрессионный вектор Polλ и К.А. Бондаренко за помощь в выделении Polη.
Работа поддержана грантами Российского фонда фундаментальных исследований (№ 17-00-00264-комфи (АВМ) и № 17-00-00261-комфи (ДОЖ)). Тестирование активности PrimPol поддержано Российским научным фондом (№ 18-14-00354 (АВМ)).
Работа не содержит исследований с использованием животных или биологических материалов, полученных от людей, в качестве объектов.
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Weber M., Schübeler D. (2007) Genomic patterns of DNA methylation: Targets and function of an epigenetic mark. Curr. Opin. Cell Biol. 19, 273–280.
Law J.A., Jacobsen S.E. (2010) Establishing, maintaining and modifying DNA methylation patterns in plants and animals. Nat. Rev. Genet. 11, 204–220.
Szulwach K.E., Jin P. (2014) Integrating DNA methylation dynamics into a framework for understanding epigenetic codes. Bioessays. 36, 107–117.
Jabbari K., Bernardi G. (2004) Cytosine methylation and CpG, TpG (CpA) and TpA frequencies. Gene. 333, 143–149.
Stevens M., Cheng J.B., Li D., Xie M., Hong C., Maire C.L., Ligon K.L., Hirst M., Marra M.A., Costello J.F., Wang T. (2013) Estimating absolute methylation levels at single-CpG resolution from methylation enrichment and restriction enzyme sequencing methods. Genome Res. 23, 1541–1553.
Branco M.R., Ficz G., Reik W. (2011) Uncovering the role of 5-hydroxymethylcytosine in the epigenome. Nat. Rev. Genet. 13, 7–13.
Shen L., Zhang Y. (2013) 5-Hydroxymethylcytosine: generation, fate, and genomic distribution. Curr. Opin. Cell Biol. 25, 289–296.
Lu X., Zhao B.S., He C. (2015) TET family proteins: oxidation activity, interacting molecules, and functions in diseases. Chem. Rev. 115, 2225–2239.
Wu X., Zhang Y. (2017) TET-mediated active DNA demethylation: mechanism, function and beyond. Nat. Rev. Genet. 18, 517–534.
Waters T.R., Swann P.F. (2000) Thymine-DNA glycosylase and G to A transition mutations at CpG sites. Mutat. Res. 462, 137–147.
Pfeifer G.P. (2006) Mutagenesis at methylated CpG sequences. Curr. Top. Microbiol. Immunol. 301, 259–281.
Olinski R., Starczak M., Gackowski D. (2016) Enigmatic 5-hydroxymethyluracil: oxidatively modified base, epigenetic mark or both? Mutat. Res. 767, 59–66.
Kawai K., Wata Y., Hara M., Tojo S., Majima T. (2002) Regulation of one-electron oxidation rate of guanine by base pairing with cytosine derivatives. J. Am. Chem. Soc. 124, 3586–3590.
Denissenko M.F., Pao A., Tang M.-S., Pfeifer G.P. (1996) Preferential formation of benzo[a]pyrene adducts at lung cancer mutational hotspots in P53. Science. 274, 430–432.
Hu W., Feng Z., Tang M.-S. (2003) Preferential carcinogen-DNA adduct formation at codons 12 and 14 in the human K-ras gene and their possible mechanisms. Biochemistry. 42, 10012–10023.
Vairapandi M., Duker H.J. (1994) Excision of ultraviolet-induced photoproducts of 5-methylcytosine from DNA. Mutat. Res. 315, 85–94.
You Y.-H., Li C., Pfeifer G.P. (1999) Involvement of 5‑methylcytosine in sunlight-induced mutagenesis. J. Mol. Biol. 293, 493–503.
Lee D.-H., Pfeifer G.P. (2003) Deamination of 5‑methylcytosines within cyclobutane pyrimidine dimers is an important component of UVB mutagenesis. J. Biol. Chem. 278, 10314–10321.
Tomkova M., McClellan M., Kriaucionis S., Schuster-Böckler B. (2018) DNA replication and associated repair pathways are involved in the mutagenesis of methy-lated cytosine. DNA Repair. 62, 1–7.
Tomkova M., Schuster-Böckler B. (2018) DNA modifications: naturally more error prone? Trends Genet. 34, 627–638.
Yamtich J., Sweasy J.B. (2010) DNA polymerase family X: function, structure, and cellular roles. Biochim. Biophys. Acta. 1804, 1136–1150.
Pata J.D. (2010) Structural diversity of the Y-family DNA polymerases. Biochim. Biophys. Acta. 1804, 1124–1135.
Yang W. (2014) An overview of Y-family DNA polymerases and a case study of human DNA polymerase η. Biochemistry. 53, 2793–2803.
Makarova A.V., Burgers P.M. (2015) Eukaryotic DNA polymerase ζ. DNA Repair. 29, 47–55.
García-Gómez S., Reyes A., Martínez-Jiménez M.I., Chocrón E.S., Mourón S., Terrados G., Powell C., Salido E., Méndez J., Holt I.J., Blanco L. (2013) PrimPol, an archaic primase/polymerase operating in human cells. Mol. Cell. 52, 541–553.
Белоусова Е.А., Лаврик О.И. (2010) ДНК-полимеразы β и λ и их роль в репликации и репарации ДНК. Молекуляр. биология. 44, 947–965.
Lindahl T., Barnes D.E. (2000) Repair of endogenous DNA damage. Cold Spring Harb. Symp. Quant. Biol. 65, 127–133.
Maga G., Villani G., Crespan E., Wimmer U., Ferrari E., Bertocci B., Hübscher U. (2007) 8-oxo-guanine bypass by human DNA polymerases in the presence of auxiliary proteins. Nature. 447, 606–608.
Makarova A.V., Grabow C., Gening L.V., Tarantul V.Z., Tahirov T.H., Bessho T., Pavlov Y.I. (2011) Inaccurate DNA synthesis in cell extracts of yeast producing active human DNA polymerase iota. PLoS One. 6, e16612.
Makarova A.V., Stodola J.L., Burgers P.M. (2012) A four-subunit DNA polymerase ζ complex containing Polδ accessory subunits is essential for PCNA-mediated mutagenesis. Nucl. Acids Res. 40, 11618–11626.
Makarova A.V., Nick McElhinny S.A., Watts B.E., Kunkel T.A., Burgers P.M. (2014) Ribonucleotide incorporation by yeast DNA polymerase ζ. DNA Repair. 18, 63–67.
Boldinova E.O., Stojkovič G., Khairullin R., Wanrooij S., Makarova A.V. (2017) Optimization of the expression, purification and polymerase activity reaction conditions of recombinant human PrimPol. PLoS One. 12, e0184489.
Petrova D.V., Naumenko M.B., Khantakova D.V., Grin I.R., Zharkov D.O. (2019) Relative efficiency of recognition of 5-methylcytosine and 5-hydroxymethylcytosine by methyl-dependent DNA endonuclease GlaI. Russ. J. Bioorg. Chem. 45, 625–629.
Johnson R.E., Washington M.T., Prakash S., Prakash L. (2000) Fidelity of human DNA polymerase η. J. Biol. Chem. 275, 7447–7450.
Zhang Y., Yuan F., Wu X., Wang Z. (2000) Preferential incorporation of G opposite template T by the low-fidelity human DNA polymerase ι. Mol. Cell. Biol. 20, 7099–7108.
Zhang Y., Yuan F., Xin H., Wu X., Rajpal D.K., Yang D., Wang Z. (2000) Human DNA polymerase κ synthesizes DNA with extraordinarily low fidelity. Nucl. Acids Res. 28, 4147–4156.
Boldinova E.O., Wanrooij P.H., Shilkin E.S., Wanrooij S., Makarova A.V. (2017) DNA damage tolerance by eukaryotic DNA polymerase and primase PrimPol. Int. J. Mol. Sci. 18, 1584.
Poulos R.C., Olivier J., Wong J.W.H. (2017) The interaction between cytosine methylation and processes of DNA replication and repair shape the mutational landscape of cancer genomes. Nucl. Acids Res. 45, 7786–7795.
Rogozin I.B., Goncearenco A., Lada A.G., De S., Yurchenko V., Nudelman G., Panchenko A.R., Cooper D.N., Pavlov Y.I. (2018) DNA polymerase η mutational signatures are found in a variety of different types of cancer. Cell Cycle. 17, 348–355.
Tomkova M., Tomek J., Kriaucionis S., Schuster-Böckler B. (2018) Mutational signature distribution varies with DNA replication timing and strand asymmetry. Genome Biol. 19, 129.
Fang H., Barbour J.A., Poulos R.C., Katainen R., Aaltonen L.A., Wong J.W.H. (2020) Mutational processes of distinct POLE exonuclease domain mutants drive an enrichment of a specific TP53 mutation in colorectal cancer. PLoS Genet. 16, e1008572.
Alexandrov L.B., Nik-Zainal S., Wedge D.C., Aparicio S.A.J.R., Behjati S., Biankin A.V., Bignell G.R., Bolli N., Borg A., Børresen-Dale A.-L., Boyault S., Burkhardt B., Butler A.P., Caldas C., Davies H.R., Desmedt C., Eils R., Eyfjörd J.E., Foekens J.A., Greaves M., Hosoda F., Hutter B., Ilicic T., Imbeaud S., Imielinski M., Jäger N., Jones D.T.W., Jones D., Knappskog S., Kool M., Lakhani S.R., López-Otín C., Martin S., Munshi N.C., Nakamura H., Northcott P.A., Pajic M., Papaemmanuil E., Paradiso A., Pearson J.V., Puente X.S., Raine K., Ramakrishna M., Richardson A.L., Richter J., Rosenstiel P., Schlesner M., Schumacher T.N., Span P.N., Teague J.W., Totoki Y., Tutt A.N.J., Valdés-Mas R., van Buuren M.M., van ’t Veer L., Vincent-Salomon A., Waddell N., Yates L.R., Australian Pancreatic Cancer Genome Initiative, ICGC Breast Cancer Consortium, ICGC MMML-Seq Consortium, ICGC PedBrain, Zucman-Rossi J., Futreal P.A., McDermott U., Lichter P., Meyerson M., Grimmond S.M., Siebert R., Campo E., Shibata T., Pfister S.M., Campbell P.J., Stratton M.R. (2013) Signatures of mutational processes in human cancer. Nature. 500, 415–421.
Alsøe L., Sarno A., Carracedo S., Domanska D., Dingler F., Lirussi L., SenGupta T., Tekin N.B., Jobert L., Alexandrov L.B., Galashevskaya A., Rada C., San-dve G.K., Rognes T., Krokan H.E., Nilsen H. (2017) Uracil accumulation and mutagenesis dominated by cytosine deamination in CpG dinucleotides in mice lacking UNG and SMUG1. Sci. Rep. 7, 7199.
ESCODD (European Standards Committee on Oxidative DNA Damage) (2002) Comparative analysis of baseline 8-oxo-7,8-dihydroguanine in mammalian cell DNA, by different methods in different laboratories: an approach to consensus. Carcinogenesis. 23, 2129–2133.
ESCODD (European Standards Committee on Oxidative DNA Damage), Gedik C.M., Collins A. (2005) Establishing the background level of base oxidation in human lymphocyte DNA: results of an interlaboratory validation study. FASEB J. 19, 82–84.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


