Молекулярная биология, 2021, T. 55, № 3, стр. 491-499
N-домен антирестрикционных белков ArdA ингибирует репрессорную активность гистоноподобного белка H-NS
О. Е. Мелькина a, Г. Б. Завильгельский a, *
a Государственный научно-исследовательский институт генетики и селекции промышленных микроорганизмов Национального исследовательского центра “Курчатовский институт”
117545 Москва, Россия
* E-mail: zavilgel@genetika.ru
Поступила в редакцию 03.09.2020
После доработки 03.09.2020
Принята к публикации 01.10.2020
Аннотация
ДНК-мимикрирующие антирестрикционные белки ArdA специфически ингибируют рестрикционную и модификационную активности систем рестрикции–модификации типа I. В пространственной структуре мономера ArdA фиксируются три домена: N-домен, центральный домен и С-домен. Анализ аминокислотных последовательностей большой группы белков ArdA, кодируемых хромосомными генами грамположительных и грамотрицательных бактерий, а также генами, локализованными в конъюгативных плазмидах и транспозонах, выявил в N-домене консервативный мотив из 20 аминокислотных остатков. Показано, что N-домены белков ArdA бактерий Arthrobacter sp., Bifidobacterium longum и Pseudomonas plecoglossicida ингибируют репрессорную активность глобального регулятора – белка H-NS, в клетках Escherichia coli. Наличие в структуре ArdA N-домена как ингибитора H-NS дает возможность мобильным элементам (конъюгативным плазмидам и транспозонам) не только преодолевать в процессе горизонтального переноса генов межклеточные рестрикционнные барьеры, но и быстрее адаптироваться к условиям нового генома бактерии-реципиента.
Белки семейства ArdA эффективно ингибируют рестрикционную и модификационную активности ферментов рестрикции–модификации типа I, что позволяет мобильным элементам – конъюгативным плазмидам и транспозонам, преодолевать межклеточные рестрикционные барьеры [1–4]. По данным рентгеноструктурного анализа в пространственной структуре белка-мономера ArdA (ORF18 Tn916) (165 а.к.) выделяются три домена: 1) N-домен размером (3–61 а.к.), 2) центральный домен (62–103 а.к.) и 3) С-концевой домен (104–165 а.к.) [5]. В настоящей работе рассмотрены центральный и С-концевой домены, объединенные в единый домен, далее С-домен, так как по своей структуре он подобен B-форме дцДНК (ДНК-мимикрия) и содержит консервативный участок размером 14 а.к., так называемый антирестрикционный мотив [6], определяющий контакт между мономерами [5]. Пространственная структура N-домена значительно отличается от структуры С-домена: он содержит β-структуру из трех расположенных антипараллельно отрезков и одной короткой α-спирали, связанных петлей из 10 и более остатков [5]. Расположение структурных элементов в N-домене сходно с их расположением в B-домене биотинкарбоксилазы [7]. Ранее было показано, что высокий уровень антирестрикционной активности, примерно равный активности цельного ArdA, в белке ArdA ColIb-P9 (166 а.к.) проявляет С-концевой фрагмент, состоящий из 76 а.к. (90–166). [2, 8]. О роли N-домена в функционировании белков ArdA в настоящее время ничего не известно. Естественно предположить, что N-домен помогает решать белку ArdA (помимо ингибирования систем рестрикции–модификации типа I) другие, возможно, не менее важные, задачи, возникающие у трансмиссивных плазмид и транспозонов при переходе из клетки-донора в клетку-реципиент. Иначе трудно объяснить сохранение этого домена в составе ArdA, участвующего в процессе горизонтального переноса генов. Например, ДНК-мимикрирующий белок Ocr вирулентного бактериофага T7 ингибирует клеточные системы рестрикции–модификации типа I значительно эффективнее, чем белки ArdA [9]. При этом Ocr не содержит элемента, подобного N-домену ArdA [10].
Нами показано, что белки ArdA ингибируют не только ферменты рестрикции–модификации типа I, но и репрессорную активность гистоноподобного белка H-NS [11]. Поэтому мы решили проверить, способны ли N-домены белков ArdA снижать репрессорную активность H-NS.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Бактериальные штаммы и плазмиды. Бактериальные штаммы и плазмиды, используемые в настоящей работе, представлены в табл. 1.
Таблица 1.
Бактериальные штаммы и плазмиды
| Штамм/плазмида | Характеристика | Источник/ ссылка |
|---|---|---|
| Штамм | ||
| MG1655 | F¯ilvG rfb-50 rph-1 | Национальный биоресурсный центр, Всероссийская коллекция промышленных микроорганизмов (ВКПМ) |
| AB1157 | F¯ thr-1 leu-6 proA2 his-4 thi-1 argE3 LacY1 galK2 ara14 xyl-5 mtl-1tsx-33 rpsL31 supE44 r+m+ | ВКПМ |
| TG-1 | thi-1 supE44 hadD5 D(lac-ProAB) [F’traD36 proAB+ lacIq lacZDM15] | То же |
| Ac-1227 | Arthrobacter sp. (655) | – |
| Ас-1636 | Bifidobacterium longum (553) | – |
| B-13282 | Pseudomonas plecoglossicida (613) | – |
| Плазмида | ||
| pUC18 | Вектор, Apr | “Fermentas”, Литва |
| p15ara | В вектор pACYC184 по сайту HindIII встроен арабинозный промотор ParaB из плазмиды pBADHisB, Cmr | [12] |
| pDEW201 | ori репликации pBR322, в вектор встроена беспромоторная кассета luxCDABEP. luminescens; Apr | [15] |
| pDWluxC | В вектор pDEW201 встроен фрагмент ДНК A. fischeri размером 124 п.н, который в lux-опероне расположен между генами luxI и luxC; фрагмент расположен перед кассетой luxICDABEG P. luminescens; Apr | [11] |
| p15ara:ardA655 | Cодержит в векторе p15ara ген ardAArthrobacter sp. под арабинозным промотором; Cmr | Настоящая работа |
| p15ara:ardA553 | Cодержит в векторе p15ara ген ardAB. longum под арабинозным промотором; Cmr | То же |
| p15ara:ardA613 | Cодержит в векторе p15ara ген ardAP. plecoglossicida под арабинозным промотором; Cmr | – |
| p15ara:N655 | Cодержит в векторе p15ara N-домен (66 а.к.) гена ardAArthrobacter sp. под арабинозным промотором; Cmr | – |
| p15ara:N553 | Cодержит в векторе p15ara N-домен (64 а.к.) гена ardA B. longum под арабинозным промотором; Cmr | – |
| p15ara:N613 | Cодержит в векторе p15ara N-домен (60 а.к.) гена ardA P. plecoglossicida под арабинозным промотором; Cmr | – |
| p15ara:C655 | Cодержит в векторе p15ara С-домен (118 а.к.) гена ardAArthrobacter sp. под арабинозным промотором; Cmr | – |
| p15ara:C553 | Cодержит в векторе p15ara С-домен (92 а.к.) гена ardA B. longum под арабинозным промотором; Cmr | – |
| p15ara:C613 | Cодержит в векторе p15ara С-домен (110 а.к.) гена ardA P. plecoglossicida под арабинозным промотором; Cmr | – |
| pardA655 | Cодержит в векторе pUC18 ген ardAArthrobacter sp. под lac-промотором; Apr | – |
| pardA553 | Cодержит в векторе pUC18 ген ardAB. longum под lac-промотором; Apr | – |
| pardA613 | Cодержит в векторе pUC18 ген ardAP. plecoglossicida под промотором lac; Apr | – |
| pN655 | Cодержит в векторе pUC18 N-домен (66 а.к.) гена ardAArthrobacter sp. под lac-промотором; Apr | – |
| pN553 | Cодержит в векторе pUC18 N-домен (64 а.к.) гена ardA B. longum под lac-промотором; Apr | – |
| pN613 | Cодержит в векторе pUC18 N-домен (60 а.к.) гена ardA P. plecoglossicida под lac-промотором; Apr | – |
| pC655 | Cодержит в векторе pUC18 С-домен (118 а.к.) гена ardAArthrobacter sp. под lac-промотором; Apr | – |
| pC553 | Cодержит в векторе pUC18 С-домен (92 а.к.) гена ardA B. longum под lac-промотором; Apr | – |
| pC613 | Cодержит в векторе pUC18 С-домен (110 а.к.) гена ardA P. plecoglossicida под lac-промотором; Apr | – |
Бактериофаг λvir (далее λ) любезно предоставлен Р. Деворе (R. Devoret, Франция). Немодифицированные фаги, выращенные на штамме E. coli TG-1, у которого отсутствует система рестрикции–модификации типа I, обозначены как λ.0.
Для измерения эффективности ингибирования репрессорной активности белка H-NS мы использовали N- и C-домены белков ArdA грамположительных бактерий Arthrobacter sp. и Bifidobacterium longum, а также грамотрицательной бактерии Pseudomonas plecoglossicida. На рис. 1 представлены аминокислотные последовательности белков ArdA, обозначенных далее 655, 553 и 613.
Рис. 1.
Аминокислотные последовательности ArdA 655, ArdA 553 и ArdA 613. N-домены белков подчеркнуты, курсивом отмечена консервативная последовательность (20 а.к.) в N-доменах.

Белок ArdA 613 – ген ardA расположен в транспозоне, встроенном в хромосому грамотрицательных аэробов P. plecoglossicida (ВКПМ В-13282).
Фрагменты, содержащие гены ardA 655, ardA 553 и ardA 613, амплифицировали с использованием в качестве матриц хромосомных ДНК соответствующих бактерий и следующих праймеров:
655EcoRI_NdeI_dir 5′–CGCGAATTCCATATGCGGAGGAGACCCGCCATGACCACAC–3′
655PstI_rev 5′–GGCCTGCAGCTCAGCGCTCATGTTCGCTCTCGTCAC-3′
553EcoRI_NdeI_dir 5′–GGCGAATTCCATATGAGGAGCATCATCATGGCCGGATTG–3′
553PstI_rev 5′–GATCTGCAGGCTCACCCATGGTTCACGGCGGTC–3'
613EcoRI_NdeI dir 5′–CGCGAATTCCATATGACGAAGGAGCGAAAGCCATGAGC–3′
613PstI rev 5′–GGGCTGCAGCGGTGCCTTCTGCTGGTTTAGCGAT–3′.
N-домены выделяли с использованием следующих праймеров:
655Nd_rev 5′–GAACTGCAGTCAGATGTTCTCGTGGTCCATGACC–3′
553Nd_rev 5′–GAACTGCAGTCA TTC CCC GCC CTC GTA GTC GT–3′
613Nd_rev 5′–GAACTGCAGTCAACCCTCGAAGTCGTGGATG–3′.
С-домены выделяли с использованием следующих праймеров:
655Cd _dir 5′–CGCCATATGCCCACCGCGGGCGAGATG–3′
553Cd _dir 5′–CGCCATATGGGACTGCTCGCCACCGTG–3′
613Cd _dir 5′–CGCCATATGTTCGGCGGCTACCGTCTC–3′.
Фрагменты ДНК, содержащие гены ardA 655 (617 п.н.), ardA 553 (509 п.н.) и ardA 613 (571 п.н.), а также кодирующие N- и С-домены, были встроены в вектор p15ara под контроль арабинозного промотора (araBAD), используя сайты рестрикции NdeI и PstI. В вектор pUC18 данные фрагменты встроены под lac-промотор по сайтам рестрикции EcoRI и PstI.
Среды и условия культивирования бактерий. LB-среда: 1% триптона, 0.5% дрожжевого экстракта и 0.5% NaCl; для верхнего и нижнего слоев чашек Петри использовали 1.8 и 0.7%-ный LB-агар. Бактерии растили в LB-среде, содержащей 100 мкг/мл ампициллина, 25 мкг/мл хлорамфеникола при 30 или 37°С. Ночные культуры использовали для инокуляции жидкой среды LB c последующим ростом с аэрацией при 30°C до ранней экспоненциальной фазы. После добавления при необходимости 20 мМ L-арабинозы инкубацию продолжали при той же температуре. Оптическую плотность клеточной суспензии измеряли при 600 нм на колориметре КФК-2МР.
Реактивы, ферменты, эндонуклеазное расщепление, лигирование, трансформация. Эндонуклеазное расщепление, лигирование фрагментов ДНК, электрофорез в агарозном геле, выделение фрагментов ДНК из геля проводили согласно [16]. В реакциях расщепления и лигирования использовали ферменты фирмы “Promega” (США). Трансформацию клеток проводили кальциевым методом.
Измерение антирестрикции. Антирестрикционную активность белков ArdA измеряли в штамме E. coli K-12 AB1157 с активной системой рестрикции–модификации типа I EcoKI. Методика измерения антирестрикционной активности белков семейства Ard описана в [2].
Измерение ингибирования H-NS-репрессии. Ингибирование H-NS-репрессии измеряли, используя промотор гена luxC Aliivibrio fischeri, транскрипция которого сильно репрессирована белком H-NS [11, 13, 14]. Клетки штамма E. coli MG655 трансформировали плазмидой pDWluxC, сконструированной на основе беспромоторного вектора pDEW201 [15]. Эта плазмида содержит промотор перед геном luxC A. fischeri, встроенный перед кассетой luxCDABE P. luminescens (гены-репортеры, кодирующие люциферазу) [11]. Бактерии росли в среде LB при температуре 30°C. Интенсивность биолюминесценции клеточной суспензии и ее оптическую плотность измеряли через определенные промежутки времени.
Интенсивность биолюминесценции суспензии клеток (200 мкл) измеряли с помощью люминометра LM-01А (“Beckman”) при комнатной температуре.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Консервативный участок N-домена ArdA
Сравнительный анализ аминокислотных последовательностей гомологов ArdA выявил в области N-домена четко выраженный консервативный участок размером 20 а.к. Этот участок особенно консервативен в белках ArdA, кодируемых хромосомными генами грамположительных бактерий.
На рис. 2 представлены аминокислотные последовательности консервативного участка из 20 а.к. (в квадрате) в N-домене белков ArdA.
Рис. 2.
Аминокислотные последовательности начальной части N-доменов белков ArdA, кодируемых генами грамположительных и грамотрицательных бактерий. Квадратом выделен консервативный участок размером 20 а.к. Особо выделены четыре аминокислотных остатка (l, y, g, w), найденные во всех представленных белках.

Ингибирование рестрикционной активности фермента EcoKI
Антирестрикционную активность белков ArdA измеряли с использованием фаговой техники [2]: фаг λ.0 (содержащий немодифицированную ДНК) титровали на штамме E. coli K12 AB1157 в качестве хозяина с активной системой рестрикции–модификации EcoKI. В табл. 2 представлены результаты измерения антирестрикционной активности. Белки ArdA 655 и 613 эффективно ингибируют фермент EcoKI, снижая его рестрикционную активность примерно на четыре порядка. Белок ArdA 553 лишь частично ингибирует фермент EcoKI, понижая его рестрикционную активность в 10 раз. N-домены 655, 553 и 613 не обладают антирестрикционной активностью.
Таблица 2.
Антирестрикционная активность белков ArdA и их N-доменов
| Плазмида | Разведение исходного титра фага λ.0 | Ослабление рестрикции*** | ||
|---|---|---|---|---|
| 10–3 | 10–4 | 10–6 | ||
| pUC18 (вектор) | 140** | – | 1 | |
| pArdA 655* | – | – | 1260 | 8.100 |
| pArdA 553 | – | 152 | – | 10.1 |
| pN655 | 136 | – | – | 1 |
| pN553 | 128 | – | – | 1 |
| pArdA613 | – | – | 1148 | 8.053 |
| pN613 | 142 | – | – | 1 |
* Фрагменты с генами ardA 655, ardA 553 ardA 613 и фрагменты, кодирующие N-домены белков ArdA 655, 553 и 613, встроены в вектор pUC18 под lac-промотор. Вектор pUC18 и плазмиды c генами ardA (pArdA 655, pArdA 553, pArdA 613) и с фрагментами, кодирующими N-домены 655, 553 и 613 (pN-домены), трансформировали в бактерии E. coli K12 AB1157 c активной системой рестрикции–модификации типа I EcoKI. ** Количество негативных колоний фага λ.0, образующихся на газоне с бактериями AB1157, содержащими плазмиды pArdA 655, pArdA 553, pArdA 613 или pN-домены 655, 553 и 613, а также вектор pUC18 в качестве контроля. *** Ослабление EcoKI-рестрикции определяется как эффективность посева λ.0 на штаммах, содержащих плазмиды, отнесенная к эффективности на штамме, содержащем вектор pUC18. Представлены усредненные результаты пяти независимых опытов.
Ингибирование репрессорной активности белка H-NS
На рис. 3–5 представлены результаты эксперимента, в котором определяли способность белков-гомологов ArdA ингибировать H-NS-зависимую репрессию транскрипции бактериальных генов. С этой целью бактерии E. coli MG1655 трансформировали двумя совместимыми плазмидами: 1) pDWluxC, содержащей репрессируемый H-NS промотор перед геном luxC (выделен из генома морских бактерий A. fischeri) и кассету luxCDABE Photorhabdus luminescens (гены-репортеры, кодируют люциферазу), и 2) p15ara-613, 655, 553 с генами ardA под арабинозным промотором. Бактерии росли в среде LB с антибиотиками при температуре 30°C. Через определенные промежутки времени измеряли интенсивность биолюминесценции клеточной суспензии и ее оптическую плотность. Стрелкой указан момент добавления в среду 20 мM L-арабинозы.
Рис. 3.
Влияние цельного белка ArdA 655, его N- и C-доменов на транскрипцию с промотора перед геном luxC A. fischeri. По оси ординат отложена интенсивность биолюминесценции бактерий E. coli MG1655, содержащих плазмиду pDWluxC, в зависимости от времени инкубации в среде LB при 30°C. Бактериальные клетки росли в присутствии 20 мM L-арабинозы (светлые символы) или в отсутствие L-арабинозы (темные символы). Стрелкой указан момент добавления в среду 20 мM L-арабинозы. Эксперименты, результаты которых представлены на рис. 3–5, проведены не менее 6 раз, причем с использованием в каждом опыте нескольких независимо полученных колоний.
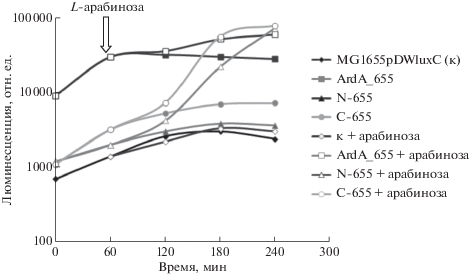
Рис. 4.
Влияние полного белка ArdA 553, его N- и C-доменов на транскрипцию с промотора перед геном luxC A. fischeri. По оси ординат отложена интенсивность биолюминесценции бактерий E. coli MG1655, содержащих плазмиду pDWluxC, в зависимости от времени инкубации в среде LB при 30°C. Бактериальные клетки росли в присутствии 20 мM L-арабинозы (светлые символы) или в отсутствие L-арабинозы (темные символы). Стрелкой указан момент добавления в среду 20 мM L-арабинозы.
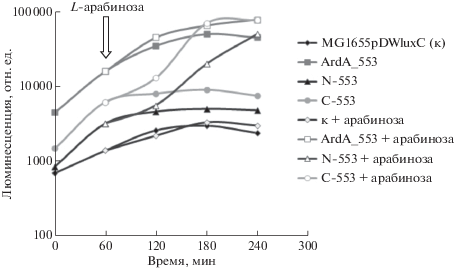
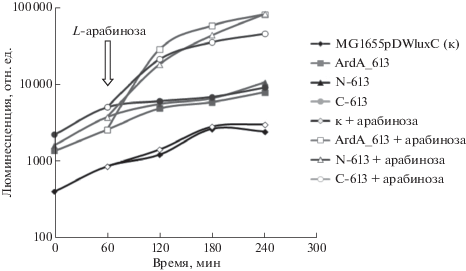
Рис. 5.
Влияние цельного белка ArdA 613, его N- и C-доменов на транскрипцию с промотора перед геном luxC A. fischeri. По оси ординат отложена интенсивность биолюминесценции бактерий E. coli MG1655, содержащих плазмиду pDWluxC, в зависимости от времени инкубации в LB при 30°C. Бактериальные клетки росли в присутствии 20 мM L-арабинозы (светлые символы) или в отсутствие L-арабинозы (темные символы). Стрелкой указан момент добавления в среду 20 мM L-арабинозы.
Как видим, ArdA 655 эффективно ингибирует репрессор H-NS практически независимо от присутствия L-арабинозы в клетке (рис. 3). Однако N- и С-домены ArdA 655 эффективно ингибируют белок H-NS лишь при добавлении в среду индуктора арабинозного промотора – L-арабинозы.
ArdA 553 ингибирует H-NS также практически независимо от присутствия L-арабинозы в среде (рис. 4). Высокие ингибирующие активности N- и С-доменов 553, достигающие уровня активности полного белка ArdA, проявляются также лишь в присутствии индуктора L-арабинозы, причем при более высоких внутриклеточных концентрациях доменов по сравнению с цельным белком ArdA.
В отличие от ArdA 655 и ArdA553 белок ArdA613 ингибирует репрессорную активность H-NS практически с такой же эффективностью, как и N-, и С-домены (рис. 5).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
N-домен белков ArdA обладает собственной активностью – способностью ингибировать белок H-NS, один из основных регуляторных белков, который участвует в компактизации бактериальной хромосомы. На примере белков ArdA 655 и ArdA 553 можно видеть, что присутствие N-домена в составе белка значительно усиливает способность С-домена ингибировать H-NS. Однако антирестрикционной активностью N-домен не обладает.
Ранее было показано, что белок 5.5 бактериофага Т7, ингибирует H-NS-репрессию [17]. Установлено, что белок 5.5 образует комплекс с центральным доменом 2, участвующим в образовании димера H-NS, и ингибирует процесс олигомеризации белков H-NS [18]. Согласно данным, представленным в GenBank CAA6800530, белок 5.5 бактериофага Т7 (99 а.к.) содержит β-структуру с двумя расположенными антипараллельно отрезками, связанную петлями с короткими α-спиралями [19], что в определенной степени сходно с пространственной структурой N-домена ArdA. Однако, как показано [20], белок 5.5 Т7 на первой стадии формирует комплекс с бактериальной тРНК, и лишь на второй стадии происходит дополнительное присоединение H-NS и образование тройного комплекса “белок 5.5:тРНК:H-NS”. Роль белка 5.5 при инфицировании клетки бактериофагом T7 состоит в отвлечении клеточного белка H-NS от фаговой ДНК, так как иначе большое количество H-NS образует с ней прочный комплекс, блокирующий репликацию и, соответственно, размножение фага. В случае с белком ArdA, гены которого в основном расположены в мобильных элементах – конъюгативных плазмидах и транспозонах, ингибирование H-NS помогает мобильным элементам открывать транскрипцию собственных генов, особенно тех, у которых регуляторные области содержат много АТ-пар, и которые эффективно репрессируются белком H-NS.
Белок H-NS большинства грамотрицательных бактерий является глобальным репрессором (silencer), например, блокирует транскрипцию около 15% генов в геномах E. coli и Salmonella enterica, а также всех богатых AT-парами генов, которые попадают в бактерии в составе мобильных элементов при горизонтальном переносе [21, 22]. На основании данных, представленных в настоящей работе, можно предположить, что присутствие в структуре антирестрикционных белков ArdA N-домена, дополнительно к основному ДНК-мимикрирующему С-домену, дает возможность трансмиссивным элементам в процессе конъюгативного горизонтального переноса генов не только преодолевать межклеточный рестрикционнный барьер, но и быстрее адаптироваться к условиям нового генома бактерии-реципиента, например, повышая транскрипционную активность переносимых генов.
Авторы выражают благодарность сотрудникам НИЦ Курчатовский институт Сергею Расторгуеву и Алексею Коженкову за помощь в полногеномном секвенировании бактерий и поиск нуклеотидных последовательностей генов ardA.
Работа поддержана Российским фондом фундаментальных исследований (грант 19-04-00495).
Работа не содержит исследований с участием людей или животных.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Belogurov A.A., Yussifov T.N., Kotova V.Yu., Zavilgelsky G.B. (1985) The novel gene(s) (ard) of plasmid pKM101: alleviation of EcoKI restriction. Mol. Gen. Genet. 198, 509–513.
Delver E.P., Kotova V.Yu., Zavilgelsky G.B., Belogurov A.A. (1991) Nucleotide sequence of the gene (ard) encoding the antirestriction protein of plasmid ColIb-P9. J. Bacteriol. 173 (18), 5887–5892.
Chilley P.M., Wilkins B.M. (1995) Distribution of the ardA family of antirestriction genes on conjugative plasmids. Microbiology. 141, 2157–2164.
Serfiotis-Mitsa D., Roberts G.A., Cooper L.P., White J.H., Nutley M., Cooper A., Blakely G.W., Dryden D.T.F. (2008) The Orf18 gene product from conjugative transposon Tn916 is an ArdA antirestriction protein that inhibits type I DNA restriction-modification systems. J. Mol. Biol. 383, 970–981.
McMahon S.A., Roberts G.A., Johnson K.A., Cooper L.P., Liu H., White J.H., Carter L.G., Sanghvi B., Oke M., Walkinshaw M.D., Blakely G.W., Naismith J.H., Dryden D.T.F. (2009) Extensive DNA mimicry by the ArdA anti-restriction protein and its role in the spread of antibiotic resistance. Nucl. Acids Res. 37, 4887–4897.
Belogurov A.A., Delver E.P. (1995) A motif conserved among the type I restriction-modification enzymes and antirestriction proteins: a possible basis for mechanism of action of plasmid-encoded anti-restriction functions. Nucl. Acids Res. 23, 785–787.
Shen Y., Volrath S.L., Weatherly S.C., Elich T.D., Tong L. (2004) A mechanism for the potent inhibition of eukaryotic acetyl-coenzyme A carboxylase by soraphen A, a macrocyclic polyketide natural product. Mol. Cell. 16, 881–891.
Nekrasov S.V., Agafonova O.V., Belogurova N.G., Delver E.P., Belogurov A.A. (2007) Plasmid-encoded antirestriction protein ArdA can discriminate between type I methyltransferase and complete restriction-modification system. J. Mol. Biol. 365, 281–297.
Завильгельский Г.Б., Котова В.Ю., Расторгуев С.М. (2008) Сравнительный анализ антирестрикционной активности белков ArdA (ColIb-P9) и Ocr (T7). Биохимия. 73, 1124–1130.
Walkinshaw M.D., Tylor P., Sturrock S.S., Atanasiu C., Berge T., Henderson R.M., Edwardson J.M., Dry-den D.T.F. (2002) Structure of Ocr from bacteriophage T7, a protein that mimics B-form of DNA. Mol. Cell. 9, 187–194.
Melkina O.E., Goryanin I.I., Zavilgelsky G.B. (2016) The DNA-mimic antirestriction proteins ArdA ColIb-P9, Arn T4, and Ocr T7 as activators of H-NS-dependent gene transcription. Microbiol. Res. 192, 283–291.
Robin S., Togashi D., Ryder A.G., Wall J.G. (2009) Trigger factor from psychrophilic bacterium Psychrobacter frigidicola is a monomeric chaperone. J. Bacteriol. 191, 1162–1169.
Ulitzur S., Matin A., Fraley C., Meighen E.(1997) H-NS protein represses transcription of the lux systems of Vibrio fischeri and other luminous bacteria cloned into Escherichia coli. Curr. Microbiol. 35, 336–342.
Ulitzur S. (1998) H-NS controls the transcription of three promoters of Vibrio fischeri lux cloned in Escherichia coli. J. Biolumin. Chemilumin. 13, 185–188.
Van Dyk T., Rosson R.A. (1998) Photorhabdus luminescens luxCDABE promoter probe vectors. In: Methods in Molecular Biology. Ed LaRossa R.A. Totowa, New York: Humana Press Inc., 102, 85–95.
Sambrook J., Fritsch E.F., Maniatis T. (1989). Molecular Cloning: a laboratory Manual, 2nd ed. New York: Cold Spring Harbor Lab. Press.
Liu Q., Richardson C.C. (1993) Gene 5.5 protein of bacteriophage T7 inhibits the nucleoid protein H-NS of Escherichia coli. Proc. Natl. Acad. Sci. USA. 90, 1761–1765.
Ali S.S., Beckett E., Bac S.J., Nawarre W.W. (2011) The 5.5 protein of phage T7 inhibits H-NS through interactions with the central oligomerization domain. J. Bacteriol. 193, 4881–4892.
Redgwell R.T., Michniewski S., Harrison D.C., Millard A. (2020) GenBank CAA6800530. University of Warwick, Coventry, United Kingdom.
Zhu B., Lee S.-J., Tan M., Wang E.-D., Richardson C.C. (2012) Gene 5.5 of bacteriophage T7 in complex with Escherichia coli nucleoid protein H-NS and transfer RNA masks transfer RNA priming in T7 replication. Proc. Natl. Acad. Sci. USA. 109, 8050–8059.
Dorman C.J. (2007) H-NS, the genome sentinel. Nat. Rev. Microbiol. 5, 157–161.
Dillon S.C., Dorman C.J. (2010) Bacterial nucleoid-associated proteins, nucleoid structure and gene expression. Nat. Rev. Microbiol. 8, 185–195.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


