Молекулярная биология, 2021, T. 55, № 4, стр. 598-605
Идентификация новых дифференциально экспрессирующихся длинных некодирующих РНК с онкогенным потенциалом
О. И. Бровкина a, b, *, И. В. Пронина a, Л. А. Урошлев a, c, М. В. Фридман c, В. И. Логинов a, Т. П. Казубская d, Д. О. Уткин d, Н. Е. Кушлинский d, Э. А. Брага a
a Научно-исследовательский институт общей патологии и патофизиологии
125315 Москва, Россия
b Федеральный научно-клинический центр специализированных видов медицинской помощи и медицинских технологий Федерального медико-биологического агентства России
115682 Москва, Россия
c Институт общей генетики им. Н.И. Вавилова Российской академии наук
117971 Москва, Россия
d Национальный медицинский исследовательский центр онкологии им. Н.Н. Блохина
Министерства здравоохранения России
115478 Москва, Россия
* E-mail: brov.olia@gmail.com
Поступила в редакцию 17.11.2020
После доработки 05.12.2020
Принята к публикации 07.12.2020
Аннотация
В последнее время накапливается все больше данных, свидетельствующих о роли длинных некодирующих РНК (lncРНК) как важнейшего регулятора транскрипции в клетках. С помощью метода глубинного машинного обучения определены четыре новых lncРНК, дифференциально экспрессирующихся в опухолях яичников: TMEM92-AS1, FAM222A-AS, TXLNB и lnc-CCL28. Представленность транскриптов lnc-CCL28 была достаточной, как в опухолях, так и в нормальной ткани, для дальнейшего анализа методом ПЦР с обратной транскрипцией (ОТ-ПЦР). На основании литературных данных, а также предыдущих исследований нашей лаборатории, помимо lnc-CCL28 для ОТ-ПЦР анализа были выбраны также lncРНК LINC00152, NEAT1 и SNHG17 как наиболее перспективные потенциальные маркеры для диагностики и прогноза рака яичников. Впервые в опухолях яичников обнаружено повышение уровня lnc-CCL28 и SNHG17 и подтверждена гиперэкспрессия LINC00152 и NEAT1. Для lnc-CCL28 впервые показана вовлеченность не только в патогенез яичников, но и в канцерогенез в целом. Выявлено, что гиперэкспрессия LINC00152 и lnc-CCL28 статистически значимо ассоциирована с более поздними стадиями и метастазированием. Таким образом, идентифицированы новые lncРНК, дифференциально экспрессирующиеся в опухолях яичников, а также обнаружены lncРНК с онкогенным и прогностическим потенциалом.
В последнее время накапливается все больше данных, свидетельствующих о роли длинных некодирующих РНК (lncРНК) в регуляции биологических процессов в клетке, а также в качестве потенциального биомаркера в онкологии [1]. Размер lncРНК составляет не менее 200 п.о., и на них не происходит трансляции белка. Долгое время считалось, что lncРНК не несет биологической функции. Позже обнаружили, что транскрипционная активность генов lncРНК иногда достигает высоких уровней и lncРНК может связываться с гистонами и транскрипционными факторами [2, 3]. В дальнейшем показали, что lncРНК ‒ важнейший регулятор транскрипции в клетках [4‒6]. Эти результаты стимулировали исследования по выяснению роли lncРНК в развитии злокачественных опухолей, в том числе рака яичников (РЯ). Следует заметить, что для РЯ вопрос поиска биомаркеров до сих пор стоит очень остро. РЯ лидирует среди онкологических заболеваний по смертности среди женщин. Ранняя диагностика и подобранная с учетом генетических и внешних факторов терапия позволяют существенно минимизировать риски осложнений и летального исхода. Одним из наиболее вероятных механизмов нарушения уровня lncРНК при РЯ считается дисбаланс тройной связи: lncРНК-микроРНК-мРНК. Чаще всего lncРНК комплементарно связывается с микроРНК (miРНК), что сопровождается повышением уровня соответствующей мРНК в клетке [7].
Появление новых высокопроизводительных методов транскриптомного анализа привело к накоплению больших массивов данных, которые представлены в открытых репозиториях. Анализ таких данных позволяет выявить новые перспективные lncРНК и сравнить их специфичность с уже найденными lncРНК и miРНК. Именно такой подход использован нами для решения одной из задач представленной работы ‒ скрининга lncРНК, предположительно связанных с miРНК, вовлеченных в патогенез РЯ [8]. Работу проводили с применением доступных баз данных и математического моделирования.
Второй задачей исследования был анализ экспрессии lncРНК, найденных по результатам скрининга и в опубликованных работах, на выборке опухолей и парной гистологически неизмененной ткани яичников больных РЯ. На основании данных литературы мы выбрали LINC00152, NEAT1, SNHG17 ‒ lncРНК, способные (по данным базы PathCards, https://pathcards.genecards.org/) к связыванию тех супрессорных miРНК, роль которых в развитии и прогрессии РЯ установлена нами ранее (например, miR-124 и miR-125b) [8].
Выбранные нами lncРНК считаются онкогенными и отличаются повышенным уровнем экспрессии в образцах опухолей разной локализации. Так, повышенная экспрессия LINC00152 характерна для многих типов рака, таких как гепатоцеллюлярная карцинома, светлоклеточная почечно-клеточная карцинома, рак желудка, рак желчного пузыря, а также обнаружена в опухолях больных РЯ [9‒12]. Гиперэкспрессия LINC00152 ассоциирована с ростом злокачественных новообразований, что может быть объяснено тем, что эта lncРНК регулирует экспрессию гена MCL1, кодирующего антиапоптотический белок MCL-1 [13].
NEAT1 ‒ одна из наиболее представленных lncРНК в опухолевых клетках, согласно последним работам, включающим транскриптомный анализ методом RNA-Seq [14, 15]. Дисрегуляция гена NEAT1 приводит к гиперэкспрессии Hu-антигена, который индуцирует воспаление и действует как антиапоптотический фактор [16].
В отличие от указанных выше lncРНК, роль SNHG17 в развитии РЯ еще не установлена, хотя была показана связь гиперэкспрессии SNHG17 с ростом опухоли и эпителиально-мезенхимальным переходом у больных раком молочной железы [17].
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Биоинформатический анализ баз данных. Для скрининга генов lncРНК со статистически значимой дифференциальной экспрессией проанализированы базы данных GEO (Gene Expression Omnibus; https://www.ncbi.nlm.nih.gov/gds) и TCGA (The Cancer Genome Atlas; https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga). Из первой базы собрана информация по 50 образцам РЯ, для которых выполнено секвенирование lncРНК. Из второй базы данных собрана информация по 500 образцам РЯ (из 739, имеющихся в базе), для которых выполнено секвенирование lncРНК.
Для образцов из базы данных GEO проведена нормализация. С помощью процедуры removeBatchEffect из пакета limma в R-studio [18] выполнено устранение систематических ошибок, возникающих на этапе выравнивания последовательностей.
С целью разбить полученную совокупность lncРНК на отдельные кластеры использованы алгоритмы метода главных компонент (principal component analysis; PCA) [19] и метод визуализации данных высокой размерности с помощью представления каждой точки данных в двух- или трехмерном пространстве (стохастическое вложение соседей с t‑распределением, t-distributed stochastic neighbor embedding; t-SNE) [20]. Далее lncРНК отбирали по дифференциальной экспрессии со значением p‑параметра < 0.05 с поправкой на множественные данные.
Для выявления взаимосвязи между lncРНК и miРНК применена модель математического обучения, основанная на технике классификации и регрессии в форме ансамбля слабых предсказывающих моделей ‒ обычно деревьев решений [21].
Клинико-морфологические данные. Образцы опухолей и парной гистологически не измененной ткани яичников больных РЯ были собраны и морфологически охарактеризованы в ФГБУ “НМИЦ онкологии им. Н.Н. Блохина” Минздрава России. Исследование было инициировано после одобрения Комиссии по этике и получения Информированного согласия на добровольное участие. Использованы образцы, полученные от 35 больных РЯ; клинико-морфологические данные приведены в табл. 1.
Таблица 1.
Клиническая характеристика пациентов с РЯ и взятых у них образцов опухолей
| Клинико-морфологические параметры | Число, % | |
|---|---|---|
| Возраст пациентов, лет | <40 | 5 (14) |
| 40‒60 | 17 (49) | |
| >60 | 13 (37) | |
| Стадия заболевания | I | 8 (22) |
| II | 5 (14) | |
| III | 20 (59) | |
| IV | 2 (5) | |
| Размер первичной опухоли | Т1 | 8 (23) |
| Т2 | 6 (17) | |
| Т3 | 21 (60) | |
| Лимфогенное метастазирование | Nx | 5 (14) |
| N0 | 20 (57) | |
| N1 | 10 (29) | |
| Наличие метастазова | есть | 29 (83) |
| нет | 6 (17) | |
Пациенты до операции не получали ни химиотерапевтического, ни лучевого лечения. Все образцы были классифицированы в соответствии с TNM-классификацией [22] и гистологически верифицированы на основании критериев классификации ВОЗ [23]. Образцы тканей хранили при ‒70°С. Полученные в ходе биопсии или оперативного вмешательства образцы объемом до 5 мм3 измельчали с использованием гомогенизатора T 10 basic Ultra-Turrax (“IKA”, Германия).
Выделение РНК. Тотальную РНК выделяли с использованием протокола гуанидин-тиоцианат-фенол-хлороформной экстракции [24]. Концентрацию суммарной РНК определяли спектрофотометрически при длине волны 260 нм. Качество РНК также оценивали спектрофотометрически с использованием коэффициентов поглощения при 260 против 230 нм и 260 против 280 нм. Сохранность РНК определяли по соотношению интенсивностей полос 28S рРНК и 18S рРНК при электрофорезе в 1%-ном денатурирующем агарозном геле. Перед использованием все образцы РНК обрабатывали ДНКазой, свободной от РНКазы. Концентрацию суммарной РНК в водном растворе оценивали на спектрофотометре NanoVue Plus (“GE Healthcare”, Великобритания).
Анализ экспрессии lncРНК. Для обратной транскрипцию (ОТ) РНК использовали обратную транскриптазу MMLV (“Евроген”, Россия) с добавлением случайных гексануклеотидных праймеров. При каждой постановке реакции ОТ включали отрицательные контроли, не содержащие РНК.
ПЦР в реальном времени проводили в 96-луночном планшете в реакционной смеси объемом 20 мкл, содержащей интеркалирующий краситель SYBR Green, ‒ qPCRmix-HS SYBR (“Евроген”). В табл. 2 представлены праймеры, использованные в ОТ-ПЦР.
Таблица 2.
Исследованные методом ОТ-ПЦР lncРНК и праймеры для их анализа
| lncРНК | Длина ампликона, п.н. | Последовательности праймерова, 5' → 3' |
|---|---|---|
| NEAT1 | 118 | CCATCCTGGTGTGCCATATT |
| GCCAACACTCTCTGGCTATAC | ||
| LINC00152 | 97 | GAAGGCTGTGTGCCTCTATT |
| TCAGGTTGGTGCTATTGGTATC | ||
| SNHG17 | 107 | GGGATCTGGGTTTGCTGATATT |
| GTAGCCTCACTCTCCATTCTCT | ||
| lnc-CCL28 | 85 | TGGACTTGACCACATGACTAAC |
| CAAGCACCACTGCACAAATAC |
Условия амплификации фрагментов ДНК: 1 мин при 95°C (1 цикл); 20 с при 95°C, 20 с при 62°C (40 циклов). Для оценки экспрессии целевых генов в качестве контроля использовали экспрессию гена B2M, в качестве нормы ‒ парные непораженные ткани. Для ПЦР в реальном времени использовали термоциклер CFX96 (“Bio-Rad”, США). ПЦР в реальном времени проводили в трех повторах. Наличие и длину продукта анализировали методом электрофореза в 1%-ном агарозном геле с интеркалирующим красителем бромидом этидия.
Статистическая обработка результатов. График амплификации обрабатывали с помощью программного обеспечения Bio-Rad CFX Manager v3.1. Применен ∆∆СT-метод для относительного количественного анализа c учетом образцов сравнения (парных непораженных тканей). Данные всех экспериментов были объединены в одно исследование с помощью пакета программ Bio-Rad CFX Manager v3.1. Непараметрический U-критерий Манна–Уитни для определения различий между двумя группами рассчитан с помощью программы R-Studio.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Результаты скрининга lncРНК, выбранных на основании анализа баз данных
Методом снижения размерности t-SNE выделено несколько кластеров, содержащих lncРНК, и несколько кластеров, содержащих miРНК (рис. 1).
Рис. 1.
Кластеризация lncРНК методом главных компонент. Знаками “минус” обозначены miРНК, знаками “плюс” – lncРНК. Синим цветом выделены кластеры miРНК, красным – lncРНК. По осям Х и Y находятся значения, соответственно первой и второй главной компоненты в методе PCA.
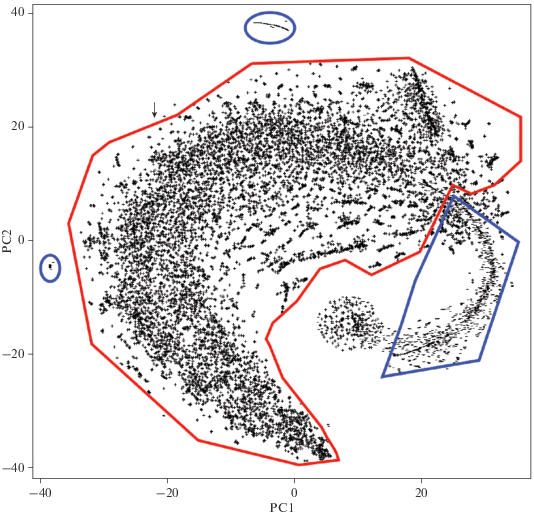
С помощью анализа кластеров, полученных в результате построения математической модели методом глубинного машинного обучения, удалось выявить четыре lncРНК со статистически значимой дифференциальной экспрессией (табл. 3).
Таблица 3.
Дифференциально экспрессирующиеся lncРНК и корреляция с экспрессией miРНК, потенциально связанных с патогенезом РЯ
| lncРНК | Доля образцов с экспрессией в опухоли (слева) и в норме (справа)a | log2FCb | p-value | miРНКc | Корреляцияd, rs/p-value |
|---|---|---|---|---|---|
| TMEM92-AS1 | 730/758, 23/58 | 13.1 | 0.001 | miR-34b-3p | ‒0.80/0.01 |
| FAM222A-AS | 736/758, 18/58 | 14.1 | 0.001 | miR-339-3p | ‒0.91/0.02 |
| lnc-CCL28 | 752/758, 3/58 | 17.5 | 0.001 | miR-148a-3p | ‒0.30/0.10 |
| TXLNB | 692/758, 0/58 | >105 | <0.001 | miR-375 | ‒0.34/0.30 |
Таким образом, биоинформатически отобраны lncРНК, потенциально способные к взаимодействию с miРНК (miR-34b, miR-339, miR-148a и miR-375), которые, как нами показано ранее [8, 9], вовлечены в патогенез РЯ.
Анализ экспрессии lncРНК методом ОТ-ПЦР
Из четырех дифференциально экспрессирующихся lncРНК, отобранных при биоинформатическом скрининге баз данных, для дальнейшего исследования методом ОТ-ПЦР выбрана lnc-CCL28 (табл. 2), представленность которой была достаточной для определения методом ОТ-ПЦР как в опухолях, так и в нормальной ткани. В результате анализа баз данных выявлена отрицательная корреляция между уровнями lnc-CCL28 и miR-148a-3p (rs = ‒0.30, p = 0.1, табл. 3), что указывает на возможность прямого или опосредованного взаимодействия lnc-CCL28 с miR-148a-3p в опухолях яичников. Ранее нами показано, что miРНК miR-148a-3p вовлечена в патогенез РЯ [8].
С помощью биоинформатического скрининга и данных литературы отобрано четыре lncРНК: lnc-CCL28, LINC00152, NEAT1 и SNHG17. Все они связываются с miРНК, роль которых в развитии и прогрессии РЯ установлена нами ранее (miR-148a, miR-124, miR-125b) [8, 9]. Методом ОТ-ПЦР мы проанализировали экспрессию этих четырех lncРНК и выявили статистически значимые различия в образцах опухолей и нормальной ткани (рис. 2). Анализ результатов ОТ-ПЦР выполнен с применением пакета программ Bio-Rad CFX Manager v3.1. и программы R-Studio.
Рис. 2.
Экспрессия lnc-CCL28, LINC00152, NEAT1 и SNHG17 в образцах опухолей РЯ и в нормальной ткани. По оси ординат приведены средние значения уровней исследованных lncРНК в нормальных и опухолевых образцах по отношению к экспрессии гена B2M. Вверху приведены значения p, полученные по результатам теста Манна–Уитни.
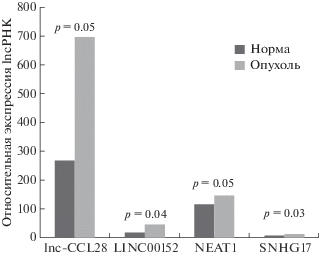
Таким образом, методом ОТ-ПЦР зарегистрирована гиперэкспрессия четырех lncРНК: lnc-CCL28, LINC00152, NEAT1 и SNHG17 ‒ в образцах опухолей РЯ, из чего можно предполагать их участие в онкогенезе. Следует подчеркнуть, что повышение экспрессии lnc-CCL28 и SNHG17 обнаружено при РЯ впервые, а о дифференциальной экспрессии lnc-CCL28 ранее вообще не сообщалось.
Установлено, что экспрессия lnc-CCL28 и LINC00152 значимо выше в опухолях, размер которых более 5 см (табл. 4). Кроме того, для этих двух lncРНК обнаружено значимое повышение экспрессии на более поздних, III и IV, стадиях в сравнении с I и II стадиями заболевания (табл. 4). Впервые выявлена значимая связь гиперэкспрессии lnc-CCL28 и LINC00152 с наличием метастазов у пациентов (табл. 4).
Таблица 4.
Корреляция уровней экспрессии lnc-CCL28, LINC00152, NEAT1 и SNHG17 с клинико-морфологической характеристикой образцов опухолей больных РЯ
| lncРНК | p-value (CI 95)a | ||
|---|---|---|---|
| размер опухоли Т1/T2‒3 | стадии I‒II/III‒IV | метастазы +/‒ | |
| lnc-CCL28 | 0.07 (‒11.1‒5.1) | 0.04 (‒14.4‒7.9) | 0.02 (‒32.2‒10.4) |
| LINC00152 | 0.02 (‒1.3‒0.4) | 0.03 (‒1.7‒0.6) | 0.02 (‒3.9‒1.3) |
| NEAT1 | 0.05 (‒26.2‒33.6) | 0.90 (‒16.2‒27.0) | 0.61 (‒33.1‒25.6) |
| SNHG17 | 0.05 (‒20.2‒49.2) | 0.08 (‒19.7‒29.1) | 0.63 (‒19.8‒9.4) |
Экспрессия NEAT1 и SNHG17 была значимо повышена у пациентов с размером опухоли более 5 см, однако не выявлена зависимость экспрессии этих lncРНК от стадии и наличия метастазов (табл. 4).
Полученные нами результаты для LINC00152, NEAT1 и SNHG17 согласуются с данными других авторов по экспрессии генов этих lncРНК в опухолях разных локализаций [11, 17, 25]. Заметим, что для SNHG17 впервые выявлена потенциально онкогенная роль при РЯ, а для lnc-CCL28 – при канцерогенезе вообще.
Кроме того, нами обнаружено, что повышенная экспрессия lnc-CCL28 и LINC00152 ассоциирована с неблагоприятным прогнозом при РЯ. В случае LINC00152 это может быть связано с тем, что эта lncРНК негативно регулирует уровень miR-125b (табл. 5). Эта miРНК, в свою очередь, контролирует экспрессию гена миелоидноклеточного лейкоза-1, MCL1, стимулирующего клеточный рост и пролиферацию [26]. Таким образом, высокая экспрессия LINC00152 может активировать транскрипцию гена MCL1 за счет тройной связи: lncРНК-miРНК-мРНК ‒ и тем самым выполнять роль конкурентной эндогенной РНК.
Таблица 5.
Характеристика исследованных lncРНКa
| lncРНК | Локализация | Биологическая роль | Связь с miРНК | Белковая мишень |
|---|---|---|---|---|
| lnc-CCL28 | Не установлена | Не установлена | miR-455-3p miR-27b-3p miR-148a-3p |
Не установлена |
| LINC00152 | Внеклеточная, цитоплазма, цитоскелет | Способствует делению клеток и росту метастазов | miR-139-5p miR-125b |
MCL-1 PI3K/AKT |
| NEAT1 | Ядерная, цитоскелет, внеклеточная | Положительная регуляция воспалительных процессов | miR-124 | ELAVL1 |
| SNHG17 | Внеклеточная | Способствует делению клеток и росту метастазов | miR-124 | CDKN1C |
a Приведена информация по данным базы PathCards (https://pathcards.genecards.org/).
Биологическая роль lnc-CCL28 пока остается неясной, однако информация в базах данных miRBase (http://www.mirbase.org/) и DIANA (http://diana. imis.athena-innovation.gr/) свидетельствует о том, что эта lncРНК ингибирует экспрессию miR-27b-3p за счет комплементарного спаривания оснований (табл. 5). miR-27b-3p, в свою очередь, подавляет рост опухолевых клеток, что было показано на клеточных линиях рака молочной железы [27].
Полученные результаты требуют подтверждения на более представительной коллекции образцов. Кроме того, планируется исследовать и сопоставить уровни экспрессии lncРНК и потенциально связанных с ними miРНК на общей выборке образцов.
На основании полученных результатов можно заключить, что скрининг открытых баз данных позволяет обнаружить lncРНК, экспрессия которых ассоциирована с развитием РЯ. Проанализированные нами lncРНК отобраны на основании данных о их возможном взаимодействии с miРНК, вовлеченными в патогенез РЯ. Показано, что гиперэкспрессия четырех lncРНК: lnc-CCL28, LINC00152, NEAT1 и SNHG17 ‒ ассоциирована с развитием РЯ. Экспрессия lnc-CCL28 исследована впервые, при этом обнаружена ее возможная связь с патогенезом и прогрессией РЯ. Также впервые выявлена гиперэкспрессия SNHG17 в опухолях яичников и прогностический потенциал lnc-CCL28 и LINC00152 при РЯ.
Работа выполнена за счет средств Российского научного фонда (грант № 20-15-00368).
Все процедуры, выполненные в данной работе, соответствуют этическим стандартам Институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики. У всех пациентов получено письменное информированное согласие.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Kung J.T.Y., Colognori D., Lee J.T. (2013) Long noncoding RNAs: past, present, and future. Genetics. 193(3), 651–669.
Brannan C.I., Dees E.C., Ingram R.S., Tilghman S.M. (1990) The product of the H19 gene may function as an RNA. Mol. Cell. Biol. 10(1), 28–36.
Wunderlich Z., Mirny L.A. (2009) Different gene regulation strategies revealed by analysis of binding motifs. Trends Genet. 25(10), 434–440.
Yao R.-W., Wang Y., Chen L.-L. (2019) Cellular functions of long noncoding RNAs. Nat. Cell Biol. 21(5), 542–551.
Fang Y., Fullwood M.J. (2016) Roles, functions, and mechanisms of long non-coding RNAs in cancer. Genomics Proteomics Bioinformatics. 14(1), 42–54.
Mercer T.R., Dinger M.E., Mattick J.S. (2009) Long non-coding RNAs: insights into functions. Nat. Rev. Genet. 10(3), 155–159.
López-Urrutia E., Bustamante Montes L.P., Ladrón de Guevara Cervantes D., Pérez-Plasencia C., Campos-Parra A.D. (2019) Crosstalk between long non-coding RNAs, micro-RNAs and mRNAs: deciphering molecular mechanisms of master regulators in cancer. Front. Oncol. 9, 669.
Loginov V.I., Pronina I.V., Burdennyy A.M., Filippova E.A., Kazubskaya T.P., Kushlinsky D.N., Utkin D.O., Khodyrev D.S., Kushlinskii N.E., Dmitriev A.A., Braga E.A. (2018) Novel miRNA genes deregulated by aberrant methylation in ovarian carcinoma are involved in metastasis. Gene. 662, 28–36.
Филиппова Е.А., Логинов В.И., Бурденный А.М., Брага Э.А., Пронина И.В., Казубская Т.П., Куш-линский Д.Н., Уткин Д.О., Фридман М.В., Ходырев Д.С., Кушлинский Н.Е. (2019) Гиперметилированные гены микроРНК в карциномах яичника: системы маркеров прогноза метастазирования. Бюлл. эксп. биологии и медицины. 167(1), 86–90.
Zhou J., Zhi X., Wang L., Wang W., Li Z., Tang J., Wang J., Zhang Q., Xu Z. (2016) Erratum to: Linc00152 promotes proliferation in gastric cancer through the EGFR-dependent pathway. J. Exp. Clin. Cancer Res. 35, 30.
Seo D., Kim D., Kim W. (2019) Long non-coding RNA linc00152 acting as a promising oncogene in cancer progression. Genomics Inform. 17(4), e36.
Cai Q., Wang Z.-Q., Wang S.-H., Li C., Zhu Z.-G., Quan Z.-W., Zhang W.-J. (2016) Upregulation of long non-coding RNA LINC00152 by SP1 contributes to gallbladder cancer cell growth and tumor metastasis via PI3K/AKT pathway. Am. J. Transl. Res. 8(10), 4068–4081.
Shigemasa K., Katoh O., Shiroyama Y., Mihara S., Mukai K., Nagai N., Ohama K. (2002) Increased MCL-1 expression is associated with poor prognosis in ovarian carcinomas. Jpn. J. Cancer Res. Gann. 93(5), 542–550.
Chakravarty D., Sboner A., Nair S.S., Giannopoulou E., Li R., Hennig S., Mosquera J.M., Pauwels J., Park K., Kossai M., MacDonald T.Y., Fontugne J., Erho N., Vergara I.A., Ghadessi M., Davicioni E., Jenkins R.B., Palanisamy N., Chen Z., Nakagawa S., Hirose T., Bander N.H., Beltran H., Fox A.H., Elemento O., Rubin M.A. (2014) The oestrogen receptor alpha-regulated lncRNA NEAT1 is a critical modulator of prostate cancer. Nat. Commun. 5, 5383.
Lebedeva S., Jens M., Theil K., Schwanhäusser B., Selbach M., Landthaler M., Rajewsky N. (2011) Transcriptome-wide analysis of regulatory interactions of the RNA-binding protein HuR. Mol. Cell. 43(3), 340–352.
Mitsunari K., Miyata Y., Asai A., Matsuo T., Shida Y., Hakariya T., Sakai H. (2016) Human antigen R is po-sitively associated with malignant aggressiveness via upregulation of cell proliferation, migration, and vascular endothelial growth factors and cyclooxygenase-2 in prostate cancer. Transl. Res. 175, 116–128.
Du Y., Wei N., Hong J., Pan W. (2020) Long non-coding RNASNHG17 promotes the progression of breast cancer by sponging miR-124-3p. Cancer Cell Int. 20, 40.
Smyth G.K. (2005) limma: Linear Models for Microarray Data. In: Bioinformatics and Computational Biology Solutions Using R and Bioconductor. Statistics for Biology and Health. Eds. Gentleman R., Carey V.J., Huber W., Irizarry R.A., Dudoit S. New York: Springer. 397–420. https://doi.org/10.1007/0-387-29362-0_23
Pearson K.F.R.S (1901) LIII. On lines and planes of closest fit to systems of points in space. Lond. Edinb. Dublin Philos. Mag. J. Sci. 2(11), 559–572. https://doi.org/10.1080/14786440109462720
Hinton G.E., Roweis S. (2002) Stochastic neighbor embedding. Adv. Neural Inf. Process. Syst. 15, 857–864.
Chen T., Guestrin C. (2016) XGBoost: A Scalable Tree Boosting System. Proceedings of the 22nd ACM SIGKDD International Conference on Knowledge Discovery and Data Mining. 785–794. https://doi.org/10.1145/2939672.2939785
(1987) TNM Classification of Malignant Tumours, 4th edition. Eds Hermanek P., Sobin L.H. Berlin, Heidelberg: Springer-Verlag.
(2014) WHO Classification of Tumours of Female Reproductive Organs, vol. 6, 4th edition. Eds Kurman R., Carcangiu M.L., Herrington C.S., Young R.H. Lyon: IARC Press.
Пронина И.В., Логинов В.И., Ходырев Д.С., Казубская Т.П., Брага Э.А. (2012) Уровень экспрессии гена RASSF1А в первичных эпителиальных опухолях разной локализации. Молекуляр. биология. 46(2), 260‒268. https://doi.org/10.1134/S0026893312010189
Chai Y., Liu J., Zhang Z., Liu L. (2016) HuR-regulated lncRNA NEAT1 stability in tumorigenesis and progression of ovarian cancer. Cancer Med. 5(7), 1588–1598.
Gong J., Zhang J.-P., Li B., Zeng C., You K., Chen M.-X., Yuan Y., Zhuang S.-M. (2013) MicroRNA-125b promotes apoptosis by regulating the expression of Mcl-1, Bcl-w and IL-6R. Oncogene. 32(25), 3071–3079.
Chen D., Si W., Shen J., Du C., Lou W., Bao C., Zheng H., Pan J., Zhong G., Xu L., Fu P., Fan W. (2018) miR-27b-3p inhibits proliferation and potentially reverses multi-chemoresistance by targeting CBLB/GRB2 in breast cancer cells. Cell Death Dis. 9(2), 188. https://doi.org/10.1038/s41419-017-0211-4
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


