Молекулярная биология, 2021, T. 55, № 5, стр. 796-828
Адресация, транспорт и локальная трансляция мРНК в клетках эукариот: от классического взгляда к многообразию современных концепций
К. А. Лашкевич a, b, С. Е. Дмитриев a, b, c, *
a Научно-исследовательский институт физико-химической биологии имени А.Н. Белозерского,
Московский государственный университет имени М.В. Ломоносова
119234 Москва, Россия
b Факультет биоинженерии и биоинформатики, Московский государственный университет
имени М.В. Ломоносова
119234 Москва, Россия
c Институт молекулярной биологии имени В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: sergey.dmitriev@belozersky.msu.ru
Поступила в редакцию 26.02.2021
После доработки 26.02.2021
Принята к публикации 12.03.2021
Аннотация
Пространственная организация процесса биосинтеза белка в эукариотической клетке изучается вот уже более 50 лет, и многие факты успели войти в учебники. Так, согласно классическим представлениям, мРНК секретируемых и мембранных белков транслируются рибосомами, ассоциированными с мембранами эндоплазматического ретикулума, в то время как растворимые белки цитоплазмы синтезируются на свободных полисомах. Однако в последние годы появилось много новых данных об избирательной трансляции мРНК также на митохондриях и пластидах, вблизи пероксисом и эндосом, в различных гранулах и на элементах цитоскелета (актиновой сети, виментиновых промежуточных филаментах, микротрубочках и на центросомах); периодически возобновляются споры о возможности синтеза белка в ядре. Локализованная трансляция может определяться как сигналами адресации в синтезируемом белковом продукте, так и нуклеотидными последовательностями в самой мРНК, а иногда и тем, и другим. Благодаря РНК-связывающим белкам многие транскрипты объединяются в специфичные РНК-конденсаты и формируют РНП-частицы, которые могут транспортироваться по элементам цитоскелета к месту трансляции, образовывать гранулы и провоцировать фазовые переходы участков цитоплазмы – как в нормальных условиях, так и при клеточном стрессе. Трансляция некоторых мРНК происходит в специализированных “трансляционных фабриках”, ассемблисомах, трансперонах и других структурах, необходимых для правильного сворачивания белков, взаимодействия с функциональными партнерами и формирования олигомерных комплексов. Внутриклеточная локализация мРНК оказывает существенное влияние на эффективность синтеза белкового продукта и, возможно, определяет характер трансляционного ответа на стрессовые воздействия. Компартментализация мРНК и трансляционного аппарата играет большую роль также при вирусных инфекциях. Многие вирусы провоцируют формирование специальных внутриклеточных структур ‒ “вирусных фабрик” ‒ для продукции своих белков. В обзоре рассмотрены сложившиеся к настоящему моменту представления об избирательной субклеточной локализации клеточных и вирусных мРНК, о молекулярных механизмах локальной трансляции, ее влиянии на адресацию и топогенез белков и на особенности регуляции биосинтеза белка в разных компартментах эукариотической клетки. Особое внимание уделено современным методам системной биологии, появление которых дало новый импульс изучению локализованной трансляции.
ВВЕДЕНИЕ
Клетка эукариот имеет сложную ультраструктуру, включающую ядро, мембранные органеллы, элементы цитоскелета, белковые и нуклеопротеидные гранулы. Все это обеспечивает компартментализацию и пространственную регуляцию большинства внутриклеточных процессов. Биогенез и функционирование органелл требуют синтеза новых белков, каждому из которых необходимо занять правильную нишу для эффективной работы. Из-за огромного функционального и структурного многообразия полипептидов и большой энергозатратности их производства в клетке выработались диверсифицированные механизмы адресации белков в места их локализации и функционирования. Десятилетия исследований раскрыли фундаментальные основы этих процессов. В зависимости от того, на какой стадии происходит доставка полипептида в нужную органеллу, выделяют два способа локализации: посттрансляционный и котрансляционный. Первый путь, безусловно, очень важен, а иногда даже незаменим, как в случае ядерных белков ‒ для их импорта из цитоплазмы в ядро. Однако в данном обзоре мы сосредоточимся на втором способе локализации ‒ когда белок оказывается в нужном месте сразу по окончании его синтеза.
Классический пример такого пути ‒ синтез секретируемых и мембранных белков, который в основном осуществляется на мембране эндоплазматического ретикулума (ЭПР). Долгое время считалось, что это едва ли не единственный пример локализованной трансляции. Однако данные, полученные в последние годы, раскрывают куда более сложную пространственную организацию биосинтеза белка в эукариотической клетке. Обнаружено множество альтернативных механизмов, включающих избирательную трансляцию мРНК на поверхности других внутриклеточных структур: на внешних мембранах митохондрий, пластид и других мембранных органелл, в специализированных гранулах и на элементах цитоскелета. Все эти процессы изучены гораздо хуже, однако уже сейчас ясно, что в их основе лежит узнавание специальных сигналов либо в последовательности самой мРНК, либо в структуре синтезируемого с нее продукта. Нередко эти два механизма сочетаются. В редких случаях, возможно, определенную роль играют также особые свойства специализированных рибосом.
В многочисленных исследованиях выявлена широкая распространенность таких случаев, когда мРНК сначала транспортируются к нужному компартменту в виде неактивных рибонуклеопротеидных комплексов (РНП) и только потом наступает фаза ее активной трансляции, приводящая к синтезу белков в строго определенном участке клетки. Этот способ имеет ряд преимуществ перед доставкой зрелого белка, так как при его реализации снижаются энергетические затраты на транспортировку и конкуренцию с другими сортирующими механизмами, облегчается сборка мультикомпонентных систем и организация локальных регуляторных “хабов”.
Разнообразие и точность механизмов адресации белков и локализации процесса их синтеза обеспечивается эффективным кооперативным действием различных клеточных систем, включая цитоскелет, работу встроенных в мембраны транслокационных комплексов, а также сети специфичных РНК-белковых и белок-белковых взаимодействий. Нарушение нормального трафика белков может приводить к их неверной локализации и инактивации, а в худшем случае – к проявлению токсических эффектов, образованию агрегатов и другим пагубным для клетки и организма последствиям, включая тяжелые патологии.
В обзоре рассмотрены различные стратегии адресации белков в компартменты эукариотической клетки, связанные с локализацией мРНК и котрансляционными механизмами. Помимо широко известных классических моделей, обсуждаются альтернативные или дополнительные, менее изученные пути, которые позволяют значительно расширить наши представления о пространственной организации процесса биосинтеза белка в клетке.
ТРАНСЛЯЦИЯ, АССОЦИИРОВАННАЯ С МЕМБРАНАМИ ЭПР
Идеи разграниченной трансляции у эукариот были выдвинуты еще в 50-х годах прошлого века, когда Дж. Паладе (G. Palade), используя методы электронной микроскопии, обнаружил несколько популяций клеточных рибосом (тогда еще “гранул Паладе”) – они были либо связаны с мембраной ЭПР, либо свободно рассеяны в цитоплазме [1]. Различную функциональную роль этих рибосом подтвердили позднее в работах по изучению механизма синтеза секретируемых и цитоплазматических белков. Возник вопрос: если существует деление рибосом по типу производимого ими белка, то каким образом принимается решение, где будет происходить синтез: на мембране ЭПР или в цитоплазме? В результате последующих исследований обнаружили существование особой популяции ЭПР-ассоциированных мРНК [2‒5], большая часть которых кодирует секретируемые или мембранные белки. Однако сейчас известно, что на ЭПР могут синтезироваться и растворимые белки цитозоля [6‒11], а способов адресации мРНК на мембраны ЭПР может быть несколько [12‒14]. Кроме того, недавно заявлено о существовании целого клеточного компартмента – TIGER-домена, ассоциированного с ЭПР и организующего специфические мРНК-белковые взаимодействия вблизи мембраны [15].
Продуктзависимые механизмы ЭПР-локализованной трансляции: классический SRP-зависимый путь и другие способы
Первой попыткой объяснить локализованный синтез секретируемых и мембранных белков стала “сигнальная гипотеза”, предложенная Blobel и соавт. [16, 17]. Согласно ей, специальная аминокислотная последовательность на N-конце растущего белка, сигнальный пептид (SP), несет информацию, благодаря которой и устанавливается связь между транслирующей рибосомой и мембраной ЭПР. Позднее, в начале 80-х годов, удалось идентифицировать растворимый посредник, узнающий SP, – им оказалась РНП-частица, названная SRP (signal recognition particle – сигналраспознающая частица) [18]. Также был найден второй компонент этой системы – рецептор SRP на мембране ЭПР, SR (SRP-receptor – рецептор сигналраспознающей частицы) [19]. В итоге появилась классическая модель SRP-зависимой мембранной локализации рибосом, синтезирующих мембранные и секретируемые белки (рис. 1, I).
Рис. 1.
Разнообразие механизмов адресации белков в ЭПР и локализации мРНК на мембране ЭПР. I ‒ Классический SRP-зависимый путь: SRP-частица, преассоциированная с рибосомой, узнает появляющуюся из рибосомного тоннеля сигнальную последовательность (SP) или трансмембранный домен, останавливает элонгационный цикл, после чего происходит транспортировка комплекса мРНК/рибосома/новосинтезированный пептид/SRP на мембрану (М) ЭПР, где SRP связывается со своим мембранным рецептором, SR. Транслокон (Sec-комплекс) участвует в посадке рибосомы на ЭПР и транслокации пептида через мембрану в люмен (Л), в этом ему помогают ассоциированные с транслоконом белки (TRAP, TRAM, OST и др.). II ‒ Посттрансляционный механизм импорта белков в ЭПР, опосредуемый шаперонами и другими белками. III ‒ Локализация синтезируемого продукта на мембране ЭПР посредством его взаимодействия с резидентным белком ЭПР. IV ‒ Удерживание мРНК на мембране ЭПР в комплексе с полисомой. V ‒ Опосредованная 3'-нетранслируемой областью (3'-НТО) локализация мРНК, не зависящая от трансляции. Показаны некоторые из известных мРНК-связывающих белков, вовлеченных в различные пути локализации мРНК. VI ‒ Ассоциация трансляционного комплекса с ЭПР посредством мембранных рецепторов рибосомы и мРНК-связывающих белков.
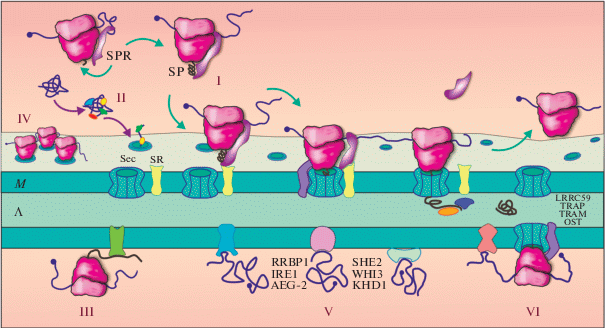
Механизм избирательного узнавания SRP-частицей основан на особом аминокислотном “коде”, составляющем SP и включающем положительно заряженные N-концевые аминокислоты, гидрофобную коровую часть (которая играет главную роль в связывании) и следующие за ней полярные остатки [20]. Аналогичным образом узнаются трансмембранные домены (ТМД) мембранных белков [21]. Узнавание и связывание SP или ТМД происходит котрансляционно ‒ по мере выхода синтезируемого пептида из рибосомного канала. Возможно, в этом процессе задействованы и другие белки (см. ниже) [22]. После того как произошло распознавание, один из доменов SRP блокирует элонгационный цикл, а комплекс мРНК/рибосома/новосинтезированный пептид/ SRP транспортируется на мембрану ЭПР, где SRP связывается со своим мембранным рецептором, SR. Специальный мультисубъединичный белковый комплекс – транслокон (Sec61-комплекс), образующий канал в мембране ЭПР, участвует в посадке рибосомы на ЭПР и транслокации пептида через мембрану [23, 24]. После прикрепления рибосомы к транслокону SRP диссоциирует, возобновляется элонгация и происходит дальнейшая транслокация растущего пептида в люмен или встраивание в мембрану ЭПР [25, 26].
Интересной вариацией классического пути служит пример необычной мРНК, которая кодирует транскрипционный фактор XBP1 животных (а также его растительные и дрожжевые аналоги bZIP60 и HAC1 соответственно). Он является важным элементом стрессового ответа клетки на накопление в ЭПР несвернутых белков (UPR; unfolded protein response) [27]. Белок XBP1u, кодируемый несплайсированной формой этой мРНК, котрансляционно рекрутирует рибосомный комплекс на мембрану ЭПР по SRP-зависимому пути [28]. Однако окончанию синтеза и высвобождению XBP1u препятствует его С-концевой участок ‒ он содержит особую последовательность аминокислот, вызывающую остановку рибосомы [29, 30]. Комплексы рибосом с пептидом и мРНК плотно удерживаются на мембране за счет высококонсервативного гидрофобного домена XBP1u, взаимодействующего с транслоконом, однако полноценной транслокации при этом не происходит [28, 31, 32]. Детали этого явления не до конца понятны и вызывают споры. Так, в некоторых работах показано высвобождение белка XBP1u и его активная роль в контроле стрессового ответа (см. дискуссию в статье [33]), в то время как в других ‒ уничтожение XBP1u системой “рибосомного контроля качества” (RQC), распознающей остановившиеся трансляционные комплексы [34]. В случае UPR происходит неканонический сплайсинг мРНК XBP1 специфичной нуклеазой IRE1, локализованной в мембране ЭПР вблизи транслокона и активирующейся при этом виде стресса. Вырезание 26-нуклеотидного интрона приводит к сдвигу открытой рамки считывания в мРНК и продукции с нее активного белка XBP1s, который проникает в ядро и активирует экспрессию генов, направленных на борьбу с последствиями UPR [27].
Последующие исследования внесли дополнения в классическую модель. Оказалось, что инактивация SRP и/или SR в дрожжах [35], трипаносомах [36] и клетках человека [37] не приводила к фатальным последствиям, при этом часть белков продолжала встраиваться в мембраны или транслоцироваться в люмен ЭПР, хотя и не так эффективно, как в норме. Это подтолкнуло к идее существования альтернативных, SRP-независимых механизмов доставки белков в ЭПР [38‒40]. Большую роль в этом играют посттрансляционные механизмы (рис. 1, II): классический GET/TRC40-зависимый импорт и недавно открытый путь SND (SRP-independent targeting) [41], ‒ описание которых выходит за рамки данного обзора. Однако, помимо них, описаны продуктнезависимые котрансляционные механизмы, основанные на локализации самих мРНК (они подробно рассмотрены в следующем разделе). Известны также случаи, когда локализованная трансляция на ЭПР все-таки обусловлена свойствами полипептида, но не зависит от SP и SRP. Например, мРНК цитозольного белка DIAPH1/Dia1 млекопитающих и птиц, не имеющего SP, при трансляции локализована на мембранах перинуклеарного ЭПР, однако это происходит не благодаря связыванию растущего белка с транслоконом, а посредством взаимодействия его N-концевой части с ассоциированным с мембраной белком семейства Rho GTPаз [42] (рис. 1, III).
В заключение следует обратить внимание на одно не совсем очевидное обстоятельство. Длинная мРНК, кодирующая большой мембранный или секретируемый белок, может транслироваться одновременно несколькими рибосомами. В этом случае она непрерывно удерживается вблизи мембраны благодаря тому, что в каждый конкретный момент времени находится как минимум одна рибосома, ассоциированная с транслоконом (рис. 1, IV), даже если остальные компоненты полисомы при этом не связаны с мембраной [8, 10]. Это довольно важное обстоятельство, поскольку оно рождает идею о существовании некого постоянного пула транскриптов, ассоциированных с мембранами. Более того, нельзя исключать, что и сами рибосомы также могут обладать “мембранной” специализацией. Полисомы на мембране ЭПР часто имеют кольцевую форму [43], что подразумевает повторяющиеся циклы трансляции конкретной мРНК одними и теми же рибосомами, переходящими со стоп-кодона на стартовый кодон посредством CLAR (closed-loop assisted reinitiation) [44]. Таким образом, уже на этом этапе появляется гипотеза о существовании специализированного трансляционного аппарата, занимающегося синтезом мембранных и секретируемых белков.
Продуктнезависимая локализация мРНК и рибосом на мембране ЭПР
Описанные выше механизмы локализованной трансляции мРНК связаны со структурой или функцией закодированных в них белковых продуктов. Однако во многих случаях в последовательностях полипептидов не обнаруживают ни SP, ни ТМД, иногда это просто растворимые цитозольные белки, никак не связанные по своей функции с ЭПР. И все же кодирующие их мРНК оказываются среди транскриптов, ассоциированных с ЭПР [3‒14]. Связывание таких мРНК с мембраной может происходить независимо от их трансляции (рис. 1, V) и обусловлено наличием в их нуклеотидной последовательности собственных сигналов локализации (цис-элементов), чаще всего расположенных в 3'-нетранслируемой области (3'-НТО) [14, 45]. В качестве примера можно привести мРНК PMP1 дрожжей Saccharomyces cerevisiae, содержащую в 3'-НТО UG-богатый мотив, который способствует формированию характерной шпилечной структуры и обеспечивает взаимодействие с ЭПР [46]. Посредниками таких взаимодействий могут быть специальные РНК-связывающие белки (см. ниже). Дрожжевые мРНК USE1 и SUC2, кодирующие соответственно заякоренный в мембране ЭПР и секретируемый белки, также продолжают пребывать на ЭПР при инактивации SRP и блокировании трансляции [12]. Недавно в мРНК, кодирующих секреторные и мембранные белки (в том числе в вышеупомянутой мРНК SUC2), обнаружен особый мотив SECReTE, способствующий удержанию и повышению стабильности мРНК на мембране ЭПР и усиливающий секрецию закодированных в них белков [47]. Многие мРНК, кодирующие ассоциированные с мембраной белки, содержат AU-богатые повторы (ARE) в 3'-НТО и при посредничестве белка TIS11B формируют на мембране ЭПР особые TIS-гранулы, объединенные в отдельный субклеточный компартмент ‒ TIGER-домен (TIS granule-ER) [15, 48]. По некоторым данным, мРНК может также связываться с ЭПР неспецифически ‒ за счет взаимодействия поли(А)-хвоста с мембранными рецепторами [9, 49].
Рецепторы мРНК на мембране ЭПР – предмет активных поисков. Так, большая роль в этом вопросе отводится многофункциональному мембранному белку RRBP1/p180, который участвует в поддержании целостности сети ЭПР внутри клетки, взаимодействует с микротрубочками, неспецифически связывает мРНК на поверхности ЭПР за счет наличия у него лизинбогатого домена, регулирует стабильность транскриптов, способствует сборке и удержанию полисом на мембране [49‒51]. мРНК-связывающими свойствами обладает также онкобелок MTDH/AEG-1 – он взаимодействует с мРНК, кодирующими преимущественно трансмембранные и секретируемые белки [52]. Интересно, что сайты связывания AEG-1 находятся в основном в кодирующих частях мРНК, реже – в 5'-НТО, а в 3'-НТО они практически отсутствуют. Недавно способность связывать с высокой аффинностью ЭПР-ассоциированные мРНК, рибосомы и тРНК выявлена также у IRE1 [53]. В дрожжах ассоциацию мРНК с мембраной ЭПР, вероятно, обеспечивает целый ряд немембранных белков: SHE2, WHI3, KHD1 и другие [54‒56]. Современные высокопроизводительные методы выявляют среди резидентов ЭПР десятки потенциальных РНК-связывающих белков [9, 57‒59], однако их роль в локализации мРНК требует тщательной проверки. У многих из них отсутствуют канонические РНК-связывающие домены, поэтому механизмы их взаимодействия с РНК малопонятны. Другая сложность заключается в том, что зачастую эти белки оказываются полифункциональными, что затрудняет их изучение.
В работе, выполненной группой К. Ничитты (C. Nicchitta) [9], список потенциальных РНК-связывающих мембранных белков ЭПР расширен до двух десятков представителей. Более того, как показали авторы, связывание с ЭПР мРНК-рибосомных комплексов, синтезирующих мембранные и секретируемые белки, опосредовано разными белками. В другом исследовании [11] эта же группа проанализировала репертуар транскриптов, связанных с рибосомами, ассоциированными либо с SEC61B/Sec61β (субъединица транслокона), либо с LRRC59 (резидентный белок ЭПР). Авторы выявили как общие для них мРНК-мишени (в частности, мРНК растворимых белков цитозоля), так и специфичные: например, транскрипты, кодирующие белки ЭПР, оказались связаны преимущественно с SEC61B, а с мРНК интегральных белков плазматической мембраны – с LRRC59. Это подтверждает вышеприведенную гипотезу о различных механизмах связывания мРНК, кодирующих мембранные и секретируемые белки.
Отдельный крайне интригующий вопрос касается мембранных рецепторов самих рибосом. В вышеупомянутом исследовании [11] изучены четыре потенциальных рецептора рибосом на мембране ЭПР: LRRC59, RPN1/рибофорин I (субъединица олигосахарилтрансферазного комплекса ЭПР, OST) и два компонента транслокона, SEC61B и SEC62. Доказана способность SEC61B, LRRC59 и, в меньшей степени, рибофорина I связывать рибосомы в культивируемых клетках человека. Помимо транслокона и OST, другие белки ЭПР также могут быть кандидатами на эту роль. Например, гетеротетрамерный комплекс TRAP и связанный с транслоцируемой цепью мембранный белок TRAM взаимодействуют с рибосомой на этапах ее связывания с транслоконом и последующей транслокации полипептида в люмен (рис. 1, VI) [9, 11, 60–62]. Кроме того, вышеупомянутый мембранный РНК-связывающий белок RRBP1/p180 был первоначально идентифицирован именно как рецептор рибосомы на мембране ER [50].
Таким образом, в случае ЭПР реализуется оба основных способа обеспечить локализованную трансляцию мРНК: котрансляционное рекрутирование посредством узнавания продукта и независимая от трансляции локализация мРНК. Скорее всего, эти два механизма действуют кооперативно.
ТРАНСЛЯЦИЯ, АССОЦИИРОВАННАЯ С ВНЕШНЕЙ МЕМБРАНОЙ МИТОХОНДРИЙ
Менее изучены процессы трансляции на мембранах другой важнейшей органеллы – митохондрии. Как известно, митохондрия имеет две мембраны: внешнюю и внутреннюю. Трансляция может происходить в непосредственной близости или на поверхности обеих ‒ в зависимости от того, где закодирован транскрипт: в ядерном или в митохондриальном геноме [63]. Митохондрии самостоятельно синтезируют лишь небольшой набор белков (компонентов дыхательной цепи), для чего используют митохондриальный трансляционный аппарат бактериального типа. Биосинтез и встраивание во внутреннюю мембрану таких белков, по всей видимости, происходит котрансляционно. Остальные митохондриальные белки, которых насчитывается более тысячи, закодированы в ядерном геноме, производятся цитозольными рибосомами и нуждаются в импорте в митохондрии [64]. Основным механизмом импорта митохондриальных белков принято считать посттрансляционную транслокацию (рис. 2, I), основанную на узнавании шаперонами специальных митохондриальных адресных пептидов на N-конце – MTS (mitochondrial targeting sequence) и транслокации через внешнюю и внутреннюю мембраны при участии митохондриальных поровых комплексов TOM/TIM [65]. Как и в случае ЭПР, рассмотрение посттрансляционного транспорта белков в митохондрию выходит за рамки нашего обзора, однако этот материал подробно изложен в нескольких работах [63‒65].
Рис. 2.
Разнообразие механизмов адресации белков в митохондрии. I – Посттрансляционная адресация, опосредованная шаперонами. II – Котрансляционная адресация комплекса мРНК/рибосома/MTS-cодержащий пептид на поровый комплекс TOM/TIM, опосредованная NAC (nascent polypeptide-associated complex). III – Непосредственное связывание NAC с белками внешней мембраны митохондрий. IV – Связывание транскриптов с внешней мембраной митохондрий с помощью мРНК-связывающих белков. V – Путь ER-SURF: DJP1-опосредованное встраивание белков, синтезированных на мембране ЭПР, в мембрану митохондрии. Подробности в тексте.
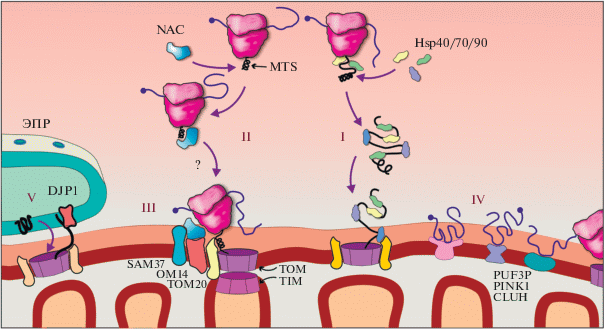
Котрансляционный импорт белков в митохондрии
Связывание цитоплазматических рибосом и полисом с внешней мембраной митохондрий было обнаружено еще в 70-е годы прошлого века [66, 67]. Чуть позже было показано, что эти полисомы обогащены транскриптами, кодирующими митохондриальные белки [68], что они способны синтезировать эти белки и встраивать их в органеллу [69]. Существование котрансляционного импорта белков в митохондрии ‒ процесса, в котором трансляция сопряжена с транслокацией синтезируемого продукта через мембрану (рис. 2, II), ‒ было доказано в 90-е годы. В эксперименте с дрожжами ингибирование посттрансляционного импорта не приводило к полной блокировке транслокации белков в митохондрии, в то время как инактивация цитозольных рибосом незамедлительно блокировала импорт [70]. Со временем эта концепция стала подкрепляться новыми экспериментальными данными. В одной из работ [71] в культивируемых клетках человека экспрессировали искусственную конструкцию, кодирующую флуоресцентный белок EGFP с двумя разными адресными последовательностями: на N‑конце был сигнал локализации в митохондриях, на С-конце – в ЭПР. Продукт обнаруживали исключительно в митохондриях, из чего был сделан вывод о том, что локализация белка определяется еще до завершения трансляции. Тем не менее для большинства белков митохондрий два пути импорта, по-видимому, дополняют друг друга, хотя в некоторых случаях (например, для фермента фумаразы [72]) котрансляционный путь доминирует. Современные методы, включая рибосомный профайлинг, позволили выяснить, что транслокация около 30% митохондриальных белков происходит, скорее всего, в сопряжении с трансляцией [73]. Особенно это характерно для белков внутренней мембраны митохондрий [73].
Природу связывания рибосом с митохондриями изучали как биохимическими, так и микроскопическими методами. Выявлена важная роль взаимодействия белков внешней мембраны с компонентами рибосомы [74]. Позднее с помощью криоэлектронной томографии удалось визуализировать это связывание [75] и выявить решающую роль взаимодействия адресного пептида с транслоконом внешней мембраны ТОМ ‒ по аналогии со взаимодействием SP или ТМД с Sec-комплексом транслокона ЭПР. Важность компонентов TOM для распознавания MTS при котрансляционном импорте показана и в другой работе [76]. Также обнаружено, что рибосомы на мембране митохондрий расположены особым образом ‒ в виде кластеров в специальных областях, где внешняя и внутренняя мембраны сближены и образуют участки контактов транслоконов TOM и TIM [75].
Точный механизм рекрутирования цитозольных рибосом на внешнюю мембрану митохондрий пока не выяснен, хотя известно, что белковый комплекс NAC (nascent polypeptide-associated complex) взаимодействует с новосинтезированным пептидом на рибосоме, выполняя роль связующего звена в этом процессе [77, 78]. Удаление NAC у нематод Caenorhabditis elegans вызывает ошибочную релокализацию рибосом, синтезирующих митохондриальные белки, на мембрану ЭПР, что приводит к нарушению протеостаза обеих органелл и к сокращению продолжительности жизни животных [79]. На дрожжах показано и обратное явление: удаление SRP из клеток приводило к ошибочной адресации белков ЭПР в митохондрию [21]. Эти данные позволяют заключить, что NAC препятствует посадке рибосом на транслокон ЭПР. Такой вывод согласуется с ранними наблюдениями о конкуренции между NAC и SRP за связывание с растущей цепью синтезируемого полипептида и/или с самой рибосомой [80, 81]. В более поздних работах внесены дополнительные коррективы в эти представления. Согласно нынешней модели, оба комплекса могут находиться на рибосоме одновременно, при этом присутствие NAC вызывает структурные перестройки в SRP, которые влияют на активность частицы (см. обсуждение в работе [82]).
Хотя NAC обычно отводится всего лишь роль “конкурента” SRP, есть данные и о его активном участии в адресации синтезирующихся белков в митохондрии (рис. 2, III). Например, прямое участие дрожжевого NAC в котрансляционном импорте продемонстрировано в системе in vitro [83]. Также на дрожжах показано, что OM14 ‒ белок внешней мембраны митохондрий ‒ функционирует как рецептор NAC, а нокаут его гена приводит к снижению доли рибосом, ассоциированных с внешней мембраной митохондрий, и к нарушению импорта белков в органеллу [84]. В качестве партнера NAC охарактеризован еще один митохондриальный белок ‒ SAM37, компонент дрожжевого мембранного комплекса сортировки и сборки белков (SAM-комплекса), динамически ассоциированный с транслоконом ТОМ [85].
Совсем недавно картина еще более усложнилась из-за появления нового игрока – белка HEL2/ZNF598, компонента системы RQC [22]. У дрожжей этот белок, вероятно, осуществляет дополнительный контроль узнавания SP частицей SRP и доставки соответствующих мРНК на транслокон.
Таким образом, полного понимания механизмов котрансляционного импорта белков в митохондрии, роли NAC и SRP в этом процессе пока нет ‒ данные довольно ограниченны и по количеству, и по набору модельных организмов, – однако интерес к этой теме неуклонно растет [64, 86, 87].
Локализация мРНК на мембране митохондрий
С использованием различных методов и в разных модельных системах достоверно показано, что множество транскриптов ядерных генов, кодирующих митохондриальные белки, локализовано на внешней мембране митохондрий [4, 5, 73, 88‒92]. Это указывает на большую роль локальной трансляции в адресации митохондриальных белков [93]. Как и в случае с ЭПР, за локализацию мРНК на митохондрии могут отвечать как детерминанты в составе самой мРНК, так и сигналы в синтезируемом полипептиде (рис. 2, IV). Примером первого способа может служить мРНК OXA1, которая как у человека, так и у дрожжей содержит в 3'-НТО функционально консервативный цис-действующий элемент, необходимый для локализации этой мРНК на митохондрии и правильной адресации соответствующего белка [94]. Второй способ, зависимый от трансляции, может быть опосредован взаимодействием MTS с компонентами транслокона TOM или другими сигналами в кодируемом полипептиде [95, 96]. Для мРНК дрожжевого белка ATM1, АВС-транспортера внутренней мембраны митохондрий, показан независимый вклад обоих механизмов [97].
Недавно Poulsen и соавт. [98] проанализировали общие свойства дрожжевых мРНК, ассоциированных с митохондриями, и пришли к неожиданному выводу, что такие транскрипты обычно имеют черты, препятствующие эффективной инициации трансляции, и связывают меньше рибосом. В результате авторы предложили модель, согласно которой митохондриальная локализация мРНК определяется медленной инициацией и большей подвижностью транскрипта, в то время как активно транслируемые мРНК менее подвижны и реже достигают поверхности митохондрий. Еще одна недавняя находка [99] увязывает особые трансляционные свойства мРНК, кодирующих митохондриальные и мембранные белки, с участием фактора инициации eIF3 в первых циклах элонгации полипептида. Недостаток eIF3 приводил к дефекту биогенеза митохондрий в мышцах мышей из-за сложности при синтезе митохондриальных белков на стадии включения первых нескольких десятков аминокислот [99].
Репертуар охарактеризованных РНК-связывающих белков внешней мембраны митохондрий пока весьма ограничен, но их роль в локализации и судьбе транскриптов митохондриальных белков, в биогенезе и поддержании функций органеллы прослеживается довольно четко [59, 100]. В пользу этого говорит, например, обнаружение определенных нуклеотидных мотивов в мРНК, кодирующих митохондриальные белки дрожжей [101]. Функцию рецепторов мРНК на внешней мембране митохондрий дрожжей могут, вероятно, выполнять некоторые представители семейства белков Puf (например PUF3), которые способствуют локализации таких мРНК на мембране органеллы, регулируют их стабильность и трансляцию [102‒108]. Еще один РНК-связывающий белок, который может выполнять роль “митохондриального якоря” для некоторых мРНК в клетках человека, ‒ это SYNJ2BP [109]. Нокаут гена SYNJ2BP показал, что этот белок необходим для быстрого возобновления трансляции этих мРНК после стресса.
У дрозофил белки PINK1 и Parkin распознают и связывают мРНК, кодирующие компоненты дыхательной цепи, на мембране митохондрий, обеспечивая их локальную трансляцию и импорт продуктов. Ассоциированная с митохондриями киназа PINK1 рекрутирует Е3-убиквитинлигазу Parkin, которая смещает трансляционные репрессоры (в частности Dcp1, POP2, Pum-1, Glorund/ hnRNP-F/H) с локализованных мРНК и способствует привлечению активаторов трансляции (eIF4A и eIF4G) [110]. В ооцитах же PINK1 преимущественно локализуется на дефектных митохондриях и путем локального фосфорилирования Larp обеспечивает их “блокаду”, прекращая синтез новых белков [111]. Это приводит к выбраковыванию митохондрий с дефектным геномом из внутриклеточной популяции, в результате чего они не передаются потомкам. Важную роль в локальной трансляции на внешней мембране митохондрий играет также регулятор AKAP1/MDI, который привлекает мРНК-связывающий белок Larp, стимулирующий трансляцию [111, 112].
мРНК-связывающий цитозольный белок CLUH, представленный у всех эукариот ‒ от дрожжей до млекопитающих, ‒ связывается преимущественно с транскриптами, кодирующими митохондриальные белки, включая компоненты комплексов окислительного фосфорилирования и цикла трикарбоновых кислот [113]. При дефиците CLUH наблюдаются серьезные дисфункции клеточного дыхания и энергетического обмена, вызванные изменениями стабильности и трансляции ассоциированных с ним мРНК [114]. Гомолог CLUH у дрозофилы (Cluless) обнаружен в митохондриальной фракции, где он ассоциирован с мембранными белками (TOM20), а также в комплексе с рибосомами [115, 116]. На основании этих данных можно построить функциональную модель, в которой CLUH высших эукариот, подобно дрожжевому PUF3, участвует в связывании и локализации ядерных транскриптов митохондриальных белков на мембране митохондрий, регулирует их стабильность и трансляцию.
Подводя итог этого раздела, хотелось бы еще раз обратить внимание на сходство принципов локализованной трансляции на внешней мембране митохондрий и на мембранах ЭПР. На самом деле эти органеллы связывают намного более тесные взаимодействия, чем принято считать. Места близкого контакта между мембранами митохондрий и ЭПР играют ключевую роль в поддержании гомеостаза липидов и кальция, в инициации аутофагии, делении митохондрий и других процессах [117, 118]. Существенная доля транскриптов, локализованных на митохондриях и ЭПР, оказывается ассоциированной и с той, и с другой органеллой, а значит, трансляция, скорее всего, идет в области их контактов [5]. Как пример – мРНК OSM1, которая имеет двойную локализацию. В основе этого явления лежит использование альтернативных стартов трансляции, дающих разные сигнальные последовательности в белке [73]. Явление двойной локализации самих полипептидов также далеко не редкость [119]. В дрожжах недавно открыт новый механизм импорта белков в митохондрии – ER-SURF (ER surface-mediated protein targeting): митохондриальные белки изначально встраиваются в ЭПР, но затем перенаправляются с его поверхности в митохондрии с помощью ЭПР-локализованного шаперона DJP1 (рис. 2, V) [120].
РОЛЬ ЛОКАЛИЗОВАННОЙ ТРАНСЛЯЦИИ В ИМПОРТЕ БЕЛКОВ В ПЕРОКСИСОМЫ И ПРОЧИЕ МЕМБРАННЫЕ ОРГАНЕЛЛЫ
Важную структурную и функциональную связь с ЭПР и митохондриями имеют пероксисомы – одномембранные органеллы, ответственные за окисление жирных кислот, детоксикацию опасных форм кислорода и азота, а также ряд других важных функций [121, 122]. Единственный пока известный путь импорта белков в пероксисомы – посттрансляционный. В отличие от ЭПР и митохондрий, в эти органеллы полипептиды транспортируются в полностью свернутой форме, зачастую в составе олигомеров [121]. Адресация опосредована особыми сигнальными последовательностями ‒ PTS (peroxisomal targeting sequences), которые находятся либо на C-конце (PTS1), либо на N-конце (PTS2) белков. И все же механизм импорта, сопряженный с трансляцией, для некоторых пероксисомных белков описан [123], однако синтез происходит не на самой органелле. Первоначальное встраивание белков в этом случае происходит в мембрану ЭПР, после чего они доставляются в пероксисомы направленным везикулярным транспортом. Такой ЭПР-опосредованный топогенез пероксисомных белков служит основой формирования пероксисом de novo и биогенеза их мембраны [121]. Еще одна особенность некоторых пероксисомных белков, имеющая отношение к трансляции, ‒ их двойственная локализация, при которой одни изоформы оказываются в пероксисомах, а другие остаются в цитозоле [124]. Зачастую эти изоформы, отличающиеся наличием PTS1 на С-конце, синтезируются с одной и той же мРНК благодаря необычно высокому уровню сквозного прочтения стоп-кодона [125, 126].
Значительная роль в адресации белков в пероксисомы отводится локализации кодирующих их транскриптов в непосредственной близости от этих органелл или прямо на их поверхности. Работ по изучению локализации мРНК на пероксисомах пока очень немного, однако это направление исследований бурно развивается [127]. Так, Zipor и соавт. [128] проанализировали локализацию 50 дрожжевых мРНК, кодирующих пероксисомные белки, и для 12 из них (в том числе мРНК 8 пероксинов и 4 ферментов матрикса) обнаружили высокий уровень колокализации с пероксисомами: от 50 до 80%. Масштабный анализ транскриптома, ассоциированного с пероксисомами у мышей [129], выявил обогащение представителями, кодирующими пероксисомные белки, в том числе пероксины и ферменты матрикса, вовлеченные в β-окисление жирных кислот и биосинтез желчных кислот. Кроме того, пероксисомная фракция содержала мРНК, кодирующие митохондриальные и секретируемые белки.
В литературе иногда можно встретить данные по связи трансляции и с другими мембранными органеллами, а также по специфичной локализации мРНК на них. Так, недавно в скрининговом исследовании обнаружена избирательная локализация нескольких видов мРНК на эндосомах, аппарате Гольджи и на внешней стороне ядерной оболочки [130]. Кроме того, в гифах грибов показана локализованная трансляция мРНК септина на эндосомах [131, 132], а в нейронах поздние эндосомы, часто ассоциированные с митохондриями, служат платформой для трансляции многих мРНК и необходимы для эффективного синтеза ламина B2 [133].
ТРАНСЛЯЦИЯ В ЯДРЕ?
Пожалуй, наиболее спорный момент в истории изучения локализации трансляции связан с обнаружением 80S рибосомных комплексов и даже, предположительно, транслирующих рибосом в клеточном ядре. История этих исследований представляет собой цепочку работ, в которых авторы пытаются доказать или опровергнуть эти тезисы [134‒136]. Впервые о “ядерной трансляции” заговорили в 50-х годах прошлого века, когда в работах Allfrey & Mirsky [137, 138] было показано включение меченых аминокислот в выделенные ядра тимоцитов теленка. Однако наличие рибосом на внешней ядерной мембране (которая непосредственно переходит в мембрану ЭПР) общеизвестно, а степень очистки ядер от цитоплазматической фракции в те времена была явно недостаточной, чтобы из этих работ можно было сделать однозначные выводы. Тем не менее в дальнейшем опыты по включению меченых аминокислот ядрами разных клеток многократно повторяли с использованием различных способов очистки и продемонстрировали энергозависимость этого процесса, чувствительность к ингибиторам белкового синтеза, ДНКазе и РНКазе (подробный разбор этих многочисленных работ 50–70-х годов можно найти в обзоре [139]). Из очищенных ядер удалось выделить функционально активные полисомы [140]. Изолированные ядрышки также активно включали аминокислоты [134, 141], что выглядит особенно правдоподобно с учетом наличия в них созревающих рибосомных субчастиц.
И все же по мере развития молекулярной биологии приходило понимание, что “легитимным” местом синтеза как цитозольных, так и ядерных белков может быть исключительно цитоплазма. Выяснилось, что созревание и ядерный экспорт большой и малой рибосомных субчастиц происходит по отдельности, причем финальные стадии биогенеза и “контроль качества” рибосомы проходят уже вне ядра [142], а до этого момента субчастицы связаны со специальными инактивирующими факторами, препятствующими инициации трансляции [143]. Однако не может не вызывать удивления тот факт, что многие трансляционные факторы, тРНК и сопутствующие ферменты обнаруживают в ядре (хотя данные на этот счет несколько противоречивы) [144]. Факт отсутствия 80S рибосом в ядре также подвергается сомнению. Так, Al-Jubran и соавт. [145] применили метод бимолекулярной флуоресцентной комплементации, присоединив разные части флуоресцентного белка к компонентам малой и большой субчастиц, и наблюдали свечение в ядре. Совсем недавно Chouaib и др. [130] показали, что мРНК небольшого числа конкретных белков локализуются почти исключительно в ядре.
Кроме того, со временем стали появляться факты, свидетельствующие о наличии разнообразных “неканонических” трансляционных событий в клетке, – и их почти сразу начали связывать с гипотетической “ядерной трансляцией” [146]. В данный момент эта тема в основном ассоциируется с малопонятным пока процессом синтеза гипотетических “дефектных рибосомных продуктов” (DRiP; defective ribosomal products), или “продуктов пионерной трансляции” (PTP; pioneer translation products), которые считают результатом трансляции несплайсированных пре-мРНК и одним из источников пептидов для презентации в главном комплексе гистосовместимости I класса (MHC I) [147‒149]. Другая ветвь исследований увязывает “ядерную трансляцию” с некоторыми проявлениями нонсенсопосредованной деградации мРНК (NMD; nonsense-mediated mRNA decay) – важного механизма контроля качества транскриптов, элиминирующего мРНК с преждевременными стоп-кодонами [150‒152]. Основным аргументом в пользу такой связи служит то, что многие факторы NMD имеют ядерную или перинуклеарную локализацию [153, 154]. Обе эти концепции, в принципе, уживаются друг с другом, поскольку продукция PTP может быть связана именно с “пионерным раундом” трансляции неправильно сплайсированных транскриптов, из которых не были удалены интроны. Вопрос заключается только в том, действительно ли эти события происходят в ядре.
Появление этих аргументов спровоцировало новую волну работ сторонников “ядерной трансляции” с применением современных методов мечения белков в сочетании с электронной и конфокальной микроскопией. Существенный вклад в формирование обновленной концепции внесли работы лаборатории П. Кука (P. Cook) [150, 152, 155], в которых использованы разные типы пульс-мечения синтезируемых полипептидов, позволяющие детектировать места встраивания метки с помощью микроскопа. Другая группа авторов с помощью метода, основанного на встраивании в растущую полипептидную цепь антибиотика пуромицина (с последующей визуализацией продукта с помощью пуромицинспецифичных антител), также обнаружила интенсивное накопление метки в ядре [156]. Этот факт долгое время считался очень серьезным аргументом в пользу “ядерной трансляции”, однако в двух недавно опубликованных работах [157, 158] метод пуромицилирования подвергся серьезной критике. Авторы выяснили, что в тех условиях, которые обычно используют при проведении реакции, продукты встраивания пуромицина покидают рибосому и быстро диффундируют от места синтеза, зачастую оказываясь в том числе и в ядре. Не исключено, что и другие типы мечения не лишены подобного недостатка. Таким образом, на сегодняшний день едва ли можно с уверенностью говорить о том, что существуют какие-либо неопровержимые аргументы в пользу существования “ядерной трансляции”.
ЦИТОЗОЛЬНЫЕ РНП-ГРАНУЛЫ И “ТРАНСЛЯЦИОННЫЕ ФАБРИКИ” КАК ПЛАТФОРМА ДЛЯ ЛОКАЛИЗАЦИИ И ТРАНСЛЯЦИИ мРНК
Стресс-гранулы, процессинговые тельца и другие виды РНП-гранул
Помимо конститутивно присутствующих органелл в цитоплазме клетки в различных условиях можно наблюдать временные немембранные образования, в частности РНП-гранулы ‒ относительно крупные конденсаты, представляющие собой многокомпонентные мультифункциональные комплексы. К наиболее известным и изученным типам таких комплексов относятся стресс-гранулы (СГ) и процессинговые тельца (ПТ) [159, 160]. ПТ (рис. 3, I) существуют в клетке как в нормальных, так и в стрессовых условиях и обогащены факторами, участвующими в деградации РНК. Однако, по современным воззрениям, уничтожение мРНК не является основным предназначением ПТ. В отличие от ПТ, СГ (рис. 3, II) преимущественно формируются при определенных видах стресса и в них сконцентрированы мРНК в комплексе с малыми рибосомными субчастицами, факторами инициации и терминации трансляции, а также многочисленными РНК-связывающими белками [159]. Основной функцией как СГ, так и ПТ сейчас считается регуляция РНК-метаболизма: прежде всего избирательная трансляционная репрессия, а также контролируемая деградация и другие процессы [159, 160]. Принято считать, что в образовании ПТ, СГ, других крупных РНП-конденсатов и немембранных органелл важную роль играет явление разделения фаз жидкость–жидкость (LLPS; liquid-liquid phase separation), связанное со свойствами самих биополимеров: РНК и белков, имеющих неструктурированные или повторяющиеся участки [161‒164]. Скорее всего, “зачатки” СГ – высокомолекулярные РНП (пре-СГ) и РНК-РНК-конденсаты, находящиеся в динамическом равновесии между разборкой и ростом, – присутствуют в клетке и в нормальных условиях [165, 166]. При наступлении же стресса они используются как “точки роста” для образования полноценных СГ за счет LLPS.
Рис. 3.
Трансляция, ассоциированная с немембранными органеллами клетки. Схематически представлены: процессинговые тельца (I); стресс-гранула (II); “трансляционная фабрика” (III); трансперон (IV); ассемблисома (V); рибосомы и нуклеопротеидные комплексы, ассоциированные с элементами цитоскелета (центросомой и микротрубочками) (VI). Подробности в тексте.
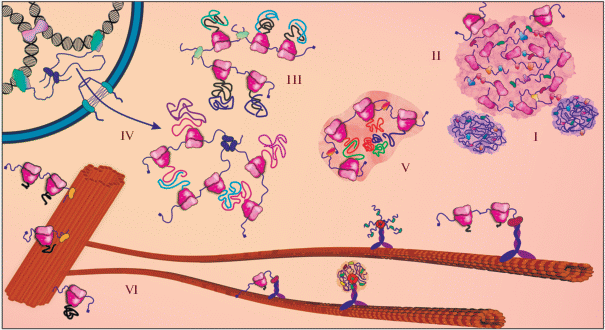
СГ и ПТ традиционно считаются трансляционно неактивными компартментами (хотя бы потому, что в составе ПТ не обнаруживают рибосомы, а в СГ – 60S субчастицы). Однако, согласно некоторым данным, обе структуры иногда могут также выступать “платформой” для локализованной трансляции мРНК. Например, в ооцитах дрозофилы мРНК gurken транслируется непосредственно на поверхности ПТ [167]. Эта мРНК синтезируется в трофоцитах и в комплексе с трансляционными репрессорами транспортируется в ооциты, где связывается с поверхностью ПТ и активаторами трансляции. Другая мРНК, bicoid, на ранних стадиях развития ооцита локализуется в коровой части ПТ, а по мере его созревания перемещается на их периферию, где происходит ее трансляция [168]. Локализация факторов инициации трансляции и полисом на ближней периферии ПТ обнаружена и в клетках млекопитающих [169]. Возможно, такое соседство важно для быстрого возобновления трансляции мРНК сразу после их высвобождения из ПТ. Таким образом, данная структура, изначально считавшаяся местом хранения “приговоренных к деградации” транскриптов, на самом деле может играть роль в динамической регуляции биосинтеза белка.
Вопрос о трансляционной активности, ассоциированной с СГ, также весьма неоднозначен. Классической считается модель, согласно которой мРНК в СГ находятся в полностью репрессированном состоянии и возобновление их трансляции происходит только после разборки СГ [159]. Однако недавно выяснилось, что активно транслируемые мРНК могут временно взаимодействовать с СГ [170]. Новые данные свидетельствуют также о возможности трансляции транскриптов, локализованных непосредственно в СГ [171], а также в неких своеобразных СГ-подобных гранулах, находящихся в клеточных протрузиях (выпячиваниях цитоплазмы) [172, 173]. Эти факты заставляют задуматься о необходимости пересмотра устоявшейся модели.
Помимо ПТ и СГ, в клетках в разных условиях находят и другие типы РНК-гранул, которые носят ткане- и мРНК-специфичный характер [172, 174‒177]. Например, гранулярные высокомолекулярные РНК-белковые комплексы широко представлены в нервных клетках [178, 179], где они служат транспортной формой неактивных полисом при перемещении мРНК вдоль нейритов [180, 181]. Активная трансляция может происходить непосредственно в таких гранулах [182]. Некоторые нейрональные гранулы, вероятно, представляют собой одиночные полисомы, “замороженные” в неактивном состоянии [183] и способные быстро возобновить элонгацию при активации синапса [181]. В клетках зародышевой линии животных широко представлен другой тип образований – так называемые герминальные гранулы. В зависимости от организма и типа клеток (половые клетки или их предшественники, клетки окружающих тканей или клетки зародышевой линии эмбрионов) под этим термином могут подразумеваться немного разные структуры: полярные гранулы, P-гранулы, губчатые и хроматоидные тельца, тельца Бальбиани, гранулы в “митохондриальных облаках” или особый приядерный компартмент nuage [184]. Считается, что эти гранулы, помимо прочих их функций, служат хранилищем нетранслируемых родительских транскриптов, которые активируются при мейозе, после оплодотворения или на определенной стадии раннего эмбриогенеза [185, 186]. В некоторых случаях динамика этих гранул тесно связана с необычными пертурбациями, которые претерпевают во время мейоза митохондрии [184]. Локализация многих кодирующих и некодирующих РНК – компонентов этих гранул – имеет сложную динамику [187, 188]. Любопытно, что с гранулами ассоциирована также рРНК митохондриальных рибосом, которую обнаруживают в этот момент в цитоплазме [189, 190]. Не исключено, что это как-то связано с ее кодирующим потенциалом [191]. Сложный паттерн и динамика распределения мРНК обеспечивают, по-видимому, регуляцию трансляции при созревании половых клеток и на ранних стадиях эмбриогенеза [185].
“Трансляционные фабрики”, ассемблисомы, транспероны и другие трансляционно активные мРНК-конденсаты
В последнее время стали появляться сведения о том, что и активно транслируемые транскрипты могут образовывать в клетке некие конденсаты, иногда именуемые “трансляционными фабриками” (рис. 3, III). Так, у дрожжей мРНК некоторых ферментов гликолиза в нормальных условиях формируют трансляционно активные гранулы, которые в случае стресса служат основой формирования ПТ [192, 193]. Эта особенность, по-видимому, характерна и для клеток человека [193]. Авторы решили назвать этот новый вид гранул “CoFe-гранулами” (core fermentation). Та же группа показала существование у дрожжей еще одного типа трансляционно активных частиц, в которых сосредоточены мРНК, кодирующие ряд трансляционных факторов [194]. Эти гранулы специфически наследуются дочерними клетками и, по-видимому, играют роль в фокусировке трансляционной активности в области поляризованного роста. Колокализация нескольких копий мРНК одного гена также показана напрямую в клетках человека: при визуализации транскриптов, с которых шла трансляция тяжелой цепи динеина, обнаружена их колокализация в 3‒7-членных кластерах [195]. Согласно опубликованным недавно результатам крупномасштабного (более 500 мРНК) скрининга, около 6% мРНК человека присуща четкая локализация в клетке [130]. В большинстве случаев это гранулярная локализация или концентрация мРНК в определенных клеточных компартментах и на органеллах, хотя возможно образование конденсатов и в виде равномерно распределенной сети, как в случае уже упоминавшихся выше TIS-гранул, ассоциированных с мембраной ЭПР [15, 48].
Таким образом, нуклеопротеидные гранулы могут не только выступать негативными регуляторами трансляции, “маскируя” мРНК с помощью РНК-связывающих белков, но и служить платформой для активной трансляции. Механизм образования таких гранул и принципы рекрутирования в них мРНК изучены пока недостаточно. Возможно, конденсаты мРНК могут формироваться отчасти благодаря взаимодействию самих РНК – в особенности их длинных неструктурированных участков [48, 161]. Кроме того, в литературе обсуждается влияние ядерных событий на колокализацию функционально сходных мРНК. Возможно, соседство генов в ядре и их совместная транскрипция обеспечивают впоследствии соседство мРНК в цитоплазме, опосредованное ядерно-цитоплазматическими РНК-связывающими белками [196], – такие группы мРНК получили название “трансперонов” (рис. 3, IV), по аналогии с оперонами прокариот.
Однако, как минимум в некоторых случаях, образование цитоплазматических конденсатов мРНК связано с белок-белковыми взаимодействиями кодируемых ими продуктов. Котрансляционное связывание синтезируемого белка с его будущими функциональными партнерами давно известно для полицистронных мРНК прокариот. Теперь стало ясно, что этот механизм довольно распространен и в эукариотическом мире [197]. Взаимодействуют не только продукты, синтезируемые соседними рибосомами с одной мРНК [198], но и – что гораздо важнее – продукты разных мРНК. Впервые это было показано для нескольких гетероолигомерных комплексов дрожжей [199‒201], затем подтверждено для мембранных [202], ядерных [203] и цитоплазматических [204‒206] белковых комплексов в клетках человека. Котрансляционное взаимодействие продуктов не может не влиять на локализацию мРНК. В случае таких крупных продуктов-олигомеров, как протеасома, котрансляционные конденсаты мРНК и их продуктов могут даже формировать особые компартменты, названные ассемблисомами (рис. 3, V) [204]. Протеасомная ассемблисома формируется не только благодаря взаимодействию друг с другом продуктов трансляции, но и при помощи специального транскрипт-специфичного механизма сегрегации мРНК, опосредованного белком NOT1 ‒ компонентом комплекса Ccr4-Not. Комплекс Ccr4-Not участвует в формировании и других котрансляционных конденсатов [201, 207].
Интересно, что после начала синтеза двух компонентов протеасомы, RPT1 и RPT2, синтезирующие их рибосомы останавливаются. Эта остановка происходит в четко детерминированных местах, позволяя появившимся из рибосомного тоннеля участкам белков связаться друг с другом. Трансляция возобновляется только после установления взаимодействия и переноса мРНК в ассемблисому [204]. Как легко заметить, эта картина сильно напоминает описанный в первых главах обзора SRP-опосредованный контроль синтеза секретируемых или мембранных белков. Таким образом, в регуляции сходных процессов в клетке задействованы одинаковые механизмы. Более того, сходный принцип “заморозки” мРНК-рибосомных комплексов и их последующей активации “по сигналу” используется, вероятно, и в других случаях – например, при транспортировке неактивных мРНК в нейронах, как описано ниже.
ЛОКАЛИЗАЦИЯ ТРАНСЛЯЦИОННЫХ ФАКТОРОВ В КЛЕТКЕ
Говоря о локализованной трансляции, о гипотетических “трансляционных фабриках” и трансперонах, нельзя не рассмотреть вопрос о внутриклеточной локализации компонентов самого трансляционного аппарата. Хорошо известно, что во время стресса 40S субчастицы рибосом, многие факторы инициации и мРНК-связывающие белки релокализуются в специальные немембранные органеллы ‒ СГ, описанные выше. Однако некоторые факты указывают на то, что компоненты трансляционного аппарата могут быть неслучайно распределены в цитоплазме и в нормальных условиях. Так, методом фракционирования и иммуноцитохимии обнаружено неслучайное распределение факторов eIF4E и eIF4G в мышиных фибробластах: помимо диффузного распределения в цитоплазме, наблюдалось их сосредоточение в районе ЭПР, аппарата Гольджи, на лидирующем крае и в клеточных протрузиях [208, 209]. Эта локализация совпадала с распределением рибосомных белков и наблюдалась только при активном белковом синтезе [209]. Сложная и динамическая локализация выявлена также для поли(А)-связывающего белка PABP [210]. Наиболее яркий паттерн распределения имеет дрожжевой фактор eIF2B. При активной трансляции существенная доля его молекул локализована в специфических гранулах, называемых “eIF2B-тельцами” [211]; через эти тельца рециркулирует фактор eIF2, производя обмен GDP на GTP. В нейронах трансляционная машинерия может быть локально сконцентрирована в синапсах или ассоциирована с рецепторами стимулирующих лигандов, что необходимо для своевременной локальной активации трансляции определенных мРНК [212‒214]. Кроме того, известно, что многие компоненты трансляционного аппарата (например, eIF3, eIF4G, eEF1A, eEF2, eRF3 и, возможно, сами рибосомы) взаимодействуют с элементами цитоскелета: микротрубочками, промежуточными и актиновыми филаментами, а также с соответствующими моторами [215, 216], ‒ участвуя в активном транспорте трансляционных комплексов.
ВНУТРИКЛЕТОЧНЫЙ ТРАНСПОРТ мРНК И ИХ ВЗАИМОДЕЙСТВИЕ С ЭЛЕМЕНТАМИ ЦИТОСКЕЛЕТА
Локализация мРНК для ее последующей компартментализованной трансляции не обязательно бывает связана с органеллами или гранулами. Часто в качестве места локализации выступает какая-то область клетки: например, ламеллоподии фибробластов и участки фокальных контактов обогащены мРНК β-актина и актинсвязывающих белков [217‒219], мРНК ASH1 дрожжей локализована исключительно в почке готовящейся к делению материнской клетки [220], а транскрипты морфогенов и гомеозисных генов oskar, nanos, bicoid и gurken после транспортировки из трофоцитов располагаются на определенных полюсах ооцита дрозофилы, что в дальнейшем определяет дорзо-вентральную и антериор-постериорную полярность эмбриона [221, 222]. Эти модели можно назвать классическими, они описаны в учебниках и во множестве обзоров. Другой популярный объект подобных исследований – это нейроны. В этих клетках именно локализация мРНК в значительной степени определяет протеомы нейритов и сомы [223], а в аксонах нейронов локальный синтез рибосомных белков необходим для “ремонта” рибосом на месте, поскольку транспорт новых рибосом из ядра, находящегося в соме, занимает в этих клетках слишком много времени [224]. Системный анализ локализации мРНК в культивируемых клетках человека [130] и эмбрионах дрозофилы [225, 226] показывает, что четко выраженная субклеточная локализация относится, скорее, к правилам, а не исключениям.
В большинстве случаев локализация мРНК определяет локализацию продукта, но иногда она нужна для неких промежуточных стадий его созревания или функционирования. Например, GTPаза RAB13 синтезируется с мРНК, локализованных в клеточных протрузиях, и, хотя сам продукт имеет перинуклеарную локализацию, его синтез в протрузиях важен для активации GTPазы, поскольку именно там синтезируемый белок RAB13 взаимодействует со своим фактором GDP/GTP-обмена ‒ RABIF [205]. В клетках человека локализация мРНК рибосомных белков на лидирующем крае движущихся клеток, опосредованная белком LARP6, вероятно, играет важную роль в регуляции их трансляции и в контроле клеточной миграции [227]. И это несмотря на то, что для реализации своей функции эти белки, скорее всего, должны сначала попасть в ядрышко. Впрочем, их участия в локальном “ремонте” рибосом, как в нейронах [224], также нельзя исключить.
Тем не менее смысл локализации мРНК в определенных участках клеток не всегда очевиден [130]. Возможно, пространственное расположение трансляционных комплексов относительно других структур клетки может служить фактором, который задает нужную ориентацию белковых комплексов более высокого порядка. Недавно такой механизм воссоздан искусственно в клеточном лизате [228]. Локализовав с помощью магнитных наночастиц в определенном месте мРНК, кодирующую актинсвязывающий белок, авторы смогли организовать нужным образом структуру актиновых филаментов в месте локализации транскрипта.
Транспортировка мРНК к месту локализации происходит, как правило, в составе РНП-комплексов, трансляция в которых угнетена специфичными репрессорами (рис. 3, VI). Адрес доставки закодирован в последовательности мРНК – в цис-действующих элементах локализации, иногда образно называемых “зип-кодами” (zipcodes, почтовые индексы). Чаще всего эти последовательности располагаются в 3'-НТО мРНК, но встречаются и в кодирующей части или 5'-НТО; они могут значительно варьировать как по длине (от нескольких нуклеотидов до нескольких сотен), так и по структуре [220, 229, 230]. С элементами локализации в мРНК взаимодействуют различные белковые факторы, образуя РНП-комплексы, которые транспортируются по элементам цитоскелета – как правило, посредством направленного активного транспорта по микротрубочкам или, значительно реже, по промежуточным или актиновым филаментам с участием молекулярных моторов [218, 231, 232].
Классический пример мРНК-связывающего белка, обеспечивающего адресацию транскриптов с “зип-кодами”, ‒ ZBP1/IGF2BP1/IMP1. Он узнает участок в 3'-НТО мРНК β-актина (а также в целом ряде других транскриптов) и одновременно формирует комплекс с кинезинподобным моторным белком KIF11 [219, 233, 234]. В состав этой РНП-частицы входят и другие мРНК-связывающие белки, а также 40S субчастицы рибосом и ряд компонентов “пионерного раунда трансляции” [235]. Это позволяет предположить, что транспортировка происходит сразу после экспорта из ядра ‒ еще до первого раунда трансляции. Сходные принципы лежат и в основе механизма транспортировки вышеупомянутых мРНК из ооцитов дрозофилы (комплекс РНК-связывающего белка Egalitarian и грузового адаптера динеина Bicaudal либо аналогичных им по функциям Staufen и кинезина-1), а также мРНК ASH1 дрожжей (РНК-связывающий белок SHE2 и миозиновый мотор MYO4). С многочисленными работами по идентификации этих компонентов можно ознакомиться в обзорах [221, 236]. В транспорт мРНК в нейронах позвоночных вовлечено, по-видимому, множество разных мРНК-связывающих белков, в том числе: STAU1 и STAU2 (гомологи Staufen), FMRP, TDP-43 и важный компонент СГ ‒ G3BP1 [236, 237]. В некоторых случаях известны РНК-связывающие белки, которые объединяют несколько мРНК конкретных генов в специфичные транспортировочные агрегаты [238, 239]. А вот ситуация с моторами в случае нейронального транспорта пока менее понятна, хотя недавно удалось выявить ключевую роль белка APC и кинезинового комплекса KIF3A/B/KAP3 в специфичном транспорте мРНК вдоль аксонов [173, 240].
Кроме транспортировки в составе индивидуального РНП и “замороженного” трансляционного комплекса, мРНК могут перемещаться по элементам цитоскелета и в составе крупных структур (СГ, ПТ или других гранул), а также на мембранных органеллах (митохондриях, эндосомах, лизосомах и т.д.). Последний способ, по-видимому, особенно распространен в аксонах и для его описания все чаще используют образное выражение “путешествие автостопом” (hitchhiking) [133, 236, 237].
В конечной локации мРНК высвобождается из комплексов с транспортными и репрессорными белками и оказывается доступной для трансляционного аппарата [241‒243]. Например, в случае уже упоминавшихся мРНК β-актина человека и ASH1 дрожжей такое высвобождение регулируется фосфорилированием компонентов РНП, которые до этого взаимодействуют с трансляционными факторами, препятствуя инициации трансляции [234, 244]. Для нейронов также предложены два оригинальных механизма активации мРНК: путем разрезания 3'-НТО [245] и путем высвобождения неактивных мРНК-рибосомных комплексов из специфичных ассоциатов с трансмембранными рецепторами нейронстимулирующих лигандов [212, 213]. Впрочем, правило “молчаливой” транспортировки выполняется не всегда: например, при APC-зависимом транспорте в клеточные протрузии мРНК в процессе доставки могут транслироваться [246]. Локализация мРНК может быть также и результатом пассивной диффузии и “заякоривания” на определенных клеточных структурах [247]. Гораздо реже ассиметричное распределение мРНК в клетке достигается с помощью локальной защиты от деградации [248]. Интересно, что на локализацию мРНК в цитоплазме могут влиять события, происходившие с ней еще в ядре: тип промотора, с которого был синтезирован транскрипт [242], или вхождение гена в определенный трансперон [196].
Помимо транспортной магистрали, элементы цитоскелета могут выступать в качестве мест постоянной дислокации мРНК и некодирующих транскриптов [130, 249, 250], обеспечивая тем самым локальный синтез транспортных белков и другие функции [251‒254]. Так, локализованная трансляция мРНК циклина B на митотическом веретене в быстро делящихся клетках эмбрионов амфибий помогает согласовать производство этого регулятора с циклами клеточного деления [255]. Локализованная трансляция мРНК аксонемального динеина в основании цилии (клеточной антенны) критична для сперматогенеза у дрозофилы [256]. Важным трансляционным “хабом” могут быть центросомы (рис. 3, VI): они аккумулируют на себе различные трансляционные компоненты [257‒260] и специфические РНК [130, 226, 250, 255, 260, 261], хотя смысл и значение такой локализации пока неясны. Известно только, что во время клеточного деления некоторые мРНК направляются по микротрубочкам в прицентриольный матрикс, а затем перераспределяются между дочерними клетками посредством актинового транспорта, обеспечивая таким образом асимметричную сегрегацию транскриптов [261]. Нарушение центросомной локализации всего одной мРНК (cen) приводит к ошибкам в сборке веретена деления и геномной нестабильности на модели эмбриона дрозофилы [262]. Интересно, что синтез ряда белков самих центросом также происходит прямо на этих органеллах и обусловлено это продуктзависимой локализацией соответствующих мРНК [263]. Эта локализация регулируется в клеточном цикле и консервативна от дрозофилы до человека.
ЧАСТНЫЕ СЛУЧАИ ЛОКАЛИЗАЦИИ И ТРАНСЛЯЦИИ мРНК ВИРУСОВ И ТРАНСПОЗОНОВ
мРНК многих вирусов (особенно тех из них, у которых весь жизненный цикл проходит в цитоплазме) транслируются в специальных структурах – в составе частиц, гранул или в мембранных компартментах, которые вирусы формируют в ходе инфекции и где протекают определенные стадии их жизненного цикла [264]. Некое подобие таких структур могут образовывать и клеточные мРНК, транскрибируемые с ДНК-копий некоторых ретротранспозонов. Хотя подробное рассмотрение этой темы потребовало бы написания отдельного обзора, мы коснемся некоторых ее аспектов, проиллюстрировав их на конкретных примерах.
Мембрана ЭПР, в частности, служит “полигоном” для трансляции мРНК вирусов семейства Flaviviridae, включающего вирусы Зика, денге, гепатита С и другие [265‒267]. На протяжении большей части жизненного цикла этих РНК-содержащих вирусов их плюс- и минус-цепи ассоциированы с мембранами ЭПР и с происходящими из них специальными мембранными структурами ‒ “репликативными фабриками”. По мере развития инфекции происходит реорганизация ЭПР и аппарата Гольджи, а трансляционные свойства мембран изменяются таким образом, что они становятся местом преимущественной трансляции вирусных мРНК, забирающих ресурсы у транскриптов клетки-хозяина. Благодаря пространственной изоляции все это происходит без значительной активации интерферонового ответа, UPR и других сигнальных путей, направленных на подавление вирусной инфекции. Сложные мембранные структуры, необходимые для репликации и происходящие из ЭПР, аппарата Гольджи, лизосом и реже митохондрий, иногда двухслойные, формируют и другие животные и растительные (+)РНК-содержащие вирусы: пикорнавирусы, коронавирусы, тогавирусы, калицивирусы и т.д. [264]. Так, в клетках, зараженных коронавирусами, было найдено от двух до шести новых типов мембранных структур, из которых наиболее хорошо охарактеризованы “запутанные мембраны” (convoluted membranes) и двумембранные везикулы (double-membrane vesicles) [268, 269]. В транскриптах многих РНК-содержащих вирусов содержатся множественные копии мотива SECReTE [270]. Например, в геноме циркулирующего сейчас в человеческой популяции коронавируса ‒ SARS-CoV-2, вызывающего COVID-19, ‒ обнаружено 40 таких участков, а также сигналы возможной митохондриальной локализации РНК [270, 271]. В случае же геномной мРНК вируса иммунодефицита человека, ВИЧ-1, ее локализация на митохондриях была показана экспериментально [272].
К сожалению, о роли таких структур в трансляции обычно мало что известно. В отдельных случаях (например, при инфекции вирусом Синдбис) выявлено обогащение этих фокусов одними трансляционными факторами (eIF3, eEF2) и обеднение другими (eIF2, eIF4G) [273]. Ортореовирусы имеют специальный механизм для активного привлечения компонентов трансляционного аппарата на границу и внутрь своих вирусных фабрик [274], при коронавирусной инфекции также наблюдается обогащение мембранных структур трансляционными компонентами [275]. В случае полиовируса локализация мРНК на мембранах может придавать трансляции дополнительную устойчивость благодаря CReP-опосредованному привлечению туда eIF2 [276].
Многие ДНК-содержащие вирусы характеризуются еще более внушительной реорганизацией мембранных компартментов клетки. Например, сложный репродуктивный цикл поксвирусов реализуется в особых околоядерных цитоплазматических локусах клетки-хозяина (виросомах, или тельцах Гварниери), в которых концентрируются факторы инициации трансляции (eIF4E, eIF4G, PABP) и другие важные трансляционные компоненты [277]. В то же время для сборки вирионов используется специальный гелеподобный компартмент (ATI-тельца), содержащий многочисленные копии белка ATI. Интересно, что трансляция мРНК, кодирующей ATI, происходит в этих тельцах, а не в виросомах [278], а для ее локализации используется, по-видимому, тот же принцип, что и в клеточных ассемблисомах: растущие из рибосомы ATI-пептиды взаимодействуют с другими молекулами ATI, локализованными в тельцах, обеспечивая заякоривание мРНК.
Одновременно с ремоделированием мембранных структур вирусы могут оказывать сильное влияние и на формирование клеточных РНП-гранул: СГ и ПТ. Интересно, что одни вирусы (например, гриппа и везикулярного стоматита, вирус Зика и пикорнавирусы) активно препятствуют формированию СГ, в то время как другие (вирус бешенства), наоборот, стимулируют их образование, используя этот механизм для подавления клеточной трансляции или для регуляции собственного цикла репликации [279‒281]. SARS-CoV-2 при заражении клеток индуцирует разборку ПТ и препятствует сборке СГ, которые мешают продуктивной инфекции [282, 283]. Вместе с тем этот коронавирус использует LLPS для создания собственных РНП-конденсатов (см. работы [284, 285] и ссылки в них). Литература о взаимоотношениях вирусов с СГ и ПТ настолько обширна, что лучший способ детально в этом разобраться ‒ обратиться к специализированным обзорам на эту тему [286, 287].
Ретротранспозон Ty1 дрожжей использует еще более изощренный путь, неразрывно связанный с ЭПР, для сборки вирусоподобных частиц, в которых реплицируется его геном [288]. РНК Ty1 по мере трансляции узнается SRP-частицей клетки-хозяина и доставляется на мембрану ЭПР, а синтезируемый с этой РНК белок оболочки Gag поступает в люмен ЭПР. Тем не менее Gag обнаруживают в цитоплазме в ассоциации с комплексом РНК Ty1 и SPR, хотя в отсутствие транслокации в ЭПР весь синтезируемый Gag быстро деградирует. Согласно предложенной модели, при транслокации в люмен ЭПР Gag принимает стабильную конформацию, а затем снова возвращается в цитоплазму и связывается с комплексом РНК и SRP, инициируя сборку вирусоподобных частиц [288].
РНК других известных ретротранспозонов также могут проявлять характерные паттерны локализации в клетке. Например, уникальная бицистронная мРНК автономного ретротранспозона LINE-1 (L1) локализуется в особых нуклеопротеидных частицах, которые обогащены белками – продуктами ее трансляции (ORF1p и ORF2p), необходимыми для транспозиции, а также маркерами СГ [289, 290]. Кроме того, состав этих частиц меняется в зависимости от жизненного цикла LINE-1 и от локализации в клетке – цитоплазматической или ядерной [291]. Интересно, что РНК неавтономных ретротранспозонов, которые используют ревертазу LINE-1 для транспозиции (Alu-повторов и SVA-элементов), находят в составе иных структур (см. [292]) – впрочем, рассмотрение локализации некодирующих РНК, к которым относятся транскрипты этих элементов, выходит за рамки данного обзора.
ОСОБЕННОСТИ РЕГУЛЯЦИИ ЛОКАЛИЗОВАННОЙ ТРАНСЛЯЦИИ
Большой интерес в области изучения локальной трансляции вызывает вопрос о том, различаются ли механизмы регуляции биосинтеза белков в зависимости от места локализации и трансляции кодирующих их мРНК. В частности неясно, одинаково ли регулируется трансляция свободными цитоплазматическими рибосомами и рибосомами, ассоциированными с мембранами органелл, существуют ли какие-то особенности в работе и циркуляции рибосом, трансляционных факторов и мРНК внутри и между разными компартментами. Совокупность предполагаемых на этот счет гипотез довольно обширна и включает крайние утверждения. Одни авторы доказывают, что для локализованной трансляции характерен особый, компартментзависимый способ контроля, который может значительно отличаться от классических внутриклеточных механизмов регуляции биосинтеза белка. Локализованные на мембране полисомы отличаются от цитозольных по динамике и структуре. Так, показано, что эти полисомы гораздо менее подвижны [10], а число рибосом в них в среднем больше, чем в составе свободных полисом [7, 10]. Это наводит на мысль, что такая раздельная регуляция тех и других вполне может иметь место. Однако другие авторы настаивают на отсутствии каких-либо значимых различий (см. ниже). Истина, вероятно, находится где-то посередине, но ее поиск осложняется недостатком экспериментальных данных по этой тематике, не всегда однозначной их трактовкой, а также наличием публикаций с противоречащими друг другу результатами. Ниже мы предлагаем рассмотреть аргументы обеих сторон.
Аргументы в пользу гипотезы компартментзависимой регуляции трансляции
К ярым сторонникам гипотезы компартментспецифичной трансляции принадлежит К. Ничитта (C. Nicchitta). В ряде работ, выполненных его группой, показано, что мРНК может проходить весь трансляционный цикл ‒ от инициации до терминации и рециклинга ‒ на мембране ЭПР без обмена рибосомами и мРНК с цитозолем [293‒296]. Происходить это может за счет того, что рибосомы, ассоциированные с транслоконом ЭПР или другими рибосомными рецепторами на ЭПР, после терминации способны оставаться на мембране и инициировать трансляцию соседних мРНК, которые также могут быть связаны с мембраной за счет разных механизмов, описанных ранее (рис. 1). Этот принцип справедлив для мРНК, кодирующих не только секретируемые или мембранные, но и цитозольные белки [7, 8, 45], высвобождающиеся после синтеза в цитоплазму и никак не связанные с транслоконом. Таким образом, авторы фактически утверждают, что процессы трансляции в цитозоле и на мембране ЭПР могут идти независимо друг от друга и это, в свою очередь, создает возможность их компартментзависимой регуляции.
Факты такого разграниченного контроля Ничитта с соавторами раскрывают в своих работах. Например, они показали, что в случае некоторых видов стресса (стресс ЭПР и вирусная инфекция) трансляция в цитозоле подавлялась, в то время как на мембране ЭПР она оставалась на прежнем уровне или даже стимулировалась [297, 298]. В другой работе [299] продемонстрирована избирательная релокализация мРНК мембранных и секретируемых белков с мембраны ЭПР в цитозоль во время стресса ЭПР, в то время как находящиеся на мембране мРНК цитозольных белков, а также мРНК белков-участников стрессового ответа локализацию не изменяли. Перераспределение мРНК происходило в первые полчаса после стресса, а к концу первого часа транскрипты, покинувшие мембрану ЭПР, возвращались обратно. Такой способ уменьшить скорость поступления белков в ЭПР путем селективной релокализации мРНК предложен в качестве механизма, альтернативного или дополнительного к деградации ассоциированных с мембраной мРНК во время ответа на стресс [299]. Авторы этих работ выдвигают новую парадигму, в которой мембранам ЭПР отводится первоочередная роль в локализации трансляции большинства мРНК (включая те, которые кодируют цитозольные белки) и в создании особых условий для регуляции их трансляции [296]. При стрессе мембраны ЭПР могут служить платформой для синтеза антистрессовых белков, даже если сами эти белки имеют цитозольную локализацию.
Эта идея находит подтверждение и в работах других научных групп. Так, в исследовании Staudacher и др. [300] показана селективная релокализация ряда мРНК на мембрану ЭПР и стимуляция их трансляции на мембране в условиях гипоксии. В число таких транскриптов вошли мРНК, кодирующие элементы адаптационного ответа на гипоксию, ферменты гликолиза и другие мишени транскрипционного фактора HIF1A. В их 5'- и 3'‑НТО обнаружены консервативные мотивы, необходимые для локализации на ЭПР и способствующие трансляции ЭПР-ассоциированными рибосомами.
Специфическая локализация мРНК может определять также быстрое возобновление их трансляции по окончании стресса. Например, в клетках с утраченным “митохондриальным якорем” SYNJ2BP некоторые мРНК, ассоциированные с внешней мембраной митохондрий, при возвращении в нормальные условия вовлекаются в трансляцию гораздо медленнее [109].
При анализе дифференцированных эффектов стресса на трансляцию в разных компартментах нельзя не учитывать их разную обогащенность трансляционными факторами. Так, недавно в работе группы M. Gromeier [276] показано, что ключевой фактор инициации eIF2 удерживается на мембранах ЭПР посредством взаимодействия с белком CReP. По мнению авторов, это обеспечивает селективную устойчивость трансляции некоторых транскриптов (например, мРНК полиовируса или мРНК резидентного шаперона ЭПР BiP) к стрессам, вызывающим инактивацию eIF2 или eIF4F. Кроме того, нельзя забывать, что локализация отдельных компонентов может сильно меняться в зависимости от условий – например, релокализация целого ряда трансляционных факторов в СГ может приводить к изменению их распределения между компартментами во время стресса.
С исторической точки зрения, идеи пространственно опосредованной регуляции синтеза белка не новы: подобные вопросы поднимались еще первооткрывателями локализованной трансляции, включая самого Паладе. Однако в современной науке эти идеи находят как положительные отклики, так и критику.
Аргументы против компартментзависимой регуляции и противоречивые данные
Тема трансляции мРНК, кодирующих цитозольные белки, на мембране ЭПР остается дискуссионной, так как в разных работах приводится различная оценка доли таких транскриптов. По данным рибосомного профайлинга клеток дрожжей и млекопитающих [8], они составляют не более 7‒15%, с поправкой на возможные загрязнения. Однако Reid & Nicchitta [7] считают, что не менее 20% “цитозольных” (согласно официальной аннотации) белков синтезируется ЭПР-ассоциированными рибосомами. Используя другой методический подход, основанный на визуализации трансляции единичных молекул мРНК, Voigt и соавт. [10] насчитали на мембране ЭПР порядка 7‒8% от общего количества мРНК репортерного белка с цитоплазматической локализацией. Для другого репортера, секретируемого, 60% кодирующей его мРНК оказалось ассоциированной с ЭПР, из чего можно сделать вывод о динамическом характере связывания мРНК с ЭПР и возможности ее циркуляции между мембраной и цитоплазмой.
В работе Jan и др. [8] модель стабильной ассоциации рибосом с ЭПР после терминации трансляции опровергается. Авторы утверждают, что рибосомы после терминации быстро диссоциируют с мембраны, возвращаясь в пул цитозольных рибосом. Эта работа вызвала спор между двумя группами ученых [301, 302], который ярко охарактеризовал ситуацию, сложившуюся в области изучения локализованной трансляции, где в силу недостаточной разработанности темы возможны совершенно различные интерпретации одних и тех же результатов. Новые данные, опубликованные группой Nicchitta [11], внесли некоторую ясность в обсуждаемый вопрос, продемонстрировав возможность развития событий по обоим сценариям: в условиях ингибирования глобальной клеточной трансляции рибосомы могут диссоциировать с мембраны ЭПР как быстро, так и медленно, а определяется это видом рецептора, с которым связана рибосома.
Сразу два интересных наблюдения сделано еще в одной работе [303]. Unsworth и соавт. показали, что, во-первых, при окислительном стрессе, индуцированном арсенитом натрия, ЭПР-ассоциированные мРНК не уходят в СГ, как это делают “цитозольные” транскрипты. Во-вторых, в клетках, подвергнутых окислительному стрессу, ЭПР-ассоциированные полисомы устойчивы к обработке пуромицином, в то время как полисомы, синтезирующие цитозольные белки, при добавлении пуромицина распадаются. Авторы увидели здесь аналогию с “тяжелыми” полисомами, формирующимися во время митоза: они трансляционно менее активны, чем полисомы в активной фазе роста, и также не чувствительны к пуромицину [304]. При делении клеток это необходимо для защиты мРНК от деградации и быстрого возобновления трансляции по окончании митоза. В подтверждение этой аналогии можно вновь привести работу Reid & Nicchitta [7], где показано, что трансляция химерного транскрипта, направленного на ЭПР, восстанавливается после отмены стресса быстрее, чем трансляция “цитозольного” репортера.
Структурные и функциональные особенности рибосом в зависимости от вида транслируемых мРНК
В заключение этого раздела рассмотрим свидетельства о различиях в динамике и структуре самих рибосом, ассоциированных с мембранами органелл и локализованных в цитоплазме, а также о функциональной специализации рибосом, транслирующих различные пулы мРНК.
На разных этапах трансляционного цикла с рибосомой взаимодействуют десятки различных лигандов и партнеров. В случае двух популяций рибосом: свободных и ассоциированных с мембраной – состав ассоциированных с ними белков и прочих компонентов трансляционного аппарата может различаться. SRP-частица, которая взаимодействует с рибосомой на этапах распознавания SP или ТМД и транспортировки комплекса на мембрану ЭПР, относится к одному из таких компонентов. Согласно классическим представлениям, SRP распознает SP или ТМД по мере их появления из рибосомного тоннеля. Однако количество SRP в клетке крайне невелико, поэтому возникает вопрос, как обеспечивается эффективное сканирование всех транслирующих рибосом на наличие SP или ТМД. Chartron и соавт. [305] оригинально разрешают этот парадокс: SRP может быть ассоциирован с рибосомой, синтезирующей секретируемый белок, еще до появления N-концевого SP из рибосомного тоннеля. Согласно этой модели, первичное рекрутирование SPR на рибосому происходит независимо от сигнала в растущем пептиде, так как она предварительно связывается со специальными участками в 3'-НТО таких мРНК.
По мере синтеза и появления пептида из рибосомы с ним также может взаимодействовать уже упоминавшийся выше комплекс NAC. Для дрожжей показано [85, 306], что разные формы NAC (гетеродимеры αβ-NAC и αβ'-NAC) могут быть вовлечены в биогенез разных наборов белков за счет связывания различных субстратов. Комплексу αβ'-NAC отводится специфическая роль в биогенезе митохондриальных белков, поскольку он преимущественно связан с рибосомами, активно синтезирующими именно их.
Эти данные согласуются с гипотезой “рибосомного фильтра” и концепцией “специализированных рибосом”, согласно которым рибосомы и рибосомные комплексы гетерогенны по своему составу, что может обеспечивать селективную трансляцию различных пулов мРНК [307‒310]. Помимо классических компонентов, упомянутых выше, гетерогенность может касаться и других ассоциированных с рибосомами белков. Например, в составе локализованных на мембране рибосом обнаружен белок PKM (мышечная пируваткиназа), проявляющий активность неканонического РНК-связывающего белка, специфичного для локализованных на ЭПР транскриптов [311]. Нельзя не упомянуть также идею “рибосомного кода” [312, 313], согласно которой разные паралоги рибосомных белков могут по-разному регулировать клеточные процессы – в том числе трансляцию локализованных мРНК [312]. У дрожжей 59 из 79 рибосомных белков кодируются паралогичными парами генов, и хотя их последовательности обычно характеризуются высоким процентом идентичности, удаление разных паралогов может приводить к различающимся фенотипам. Segev & Gerst [314] сообщали о мРНК-специфичной регуляции синтеза митохондриальных белков, которая обеспечивается пулом рибосом с разным составом белков-паралогов. Впрочем, некоторые авторы справедливо указывают на то, что аргументов в пользу концепции “специализированных рибосом” пока явно недостаточно [315].
МЕТОДЫ СИСТЕМНОЙ БИОЛОГИИ В ИЗУЧЕНИИ ЛОКАЛИЗОВАННОЙ ТРАНСЛЯЦИИ
Многие из ярких открытий в области изучения локализованной трансляции, сделанных в последние годы и спровоцировавших взрывной рост интереса к этой теме, были бы невозможны без появления целого ряда новых экспериментальных подходов, основанных на методах системной биологии. Эти подходы позволяют проводить анализ локализации сразу большого числа мРНК и выявлять картину трансляции в целом. Появление микрочипов, а затем и высокопроизводительного секвенирования позволило определять набор мРНК, ассоциированных с теми или иными органеллами. Сначала это делали путем фракционирования цитоплазмы. Так был проведен полногеномный анализ транскриптома, ассоциированного с мембранами в клетках дрожжей и человека [2, 3], с митохондриями этих же организмов [88, 316, 317] и с пероксисомами мышей [129], а также определен набор транскриптов, локализованных в псевдоподиях мигрирующих фибробластов [173] и в отростках нейронов [318‒320]. Позже был сделан рибосомный профайлинг (выделение и секвенирование фрагментов мРНК, заключенных в транслирующих рибосомах) мембранной фракции клеток или разных частей нейронов [7, 223].
Следующее поколение методов связано с прижизненной модификацией РНК, находящейся в интересующем компартменте, с последующим ее выделением и секвенированием. Такую локальную модификацию (в том числе биотинилирование или окисление оснований) можно осуществить прямо в живой клетке, если ввести в нее ген модифицирующего фермента (например, APEX или miniSOG), обеспечив белку соответствующую локализацию [5, 321, 322]. Подвергнуть локальному биотинилированию можно также РНК-связывающие белки, что после химической сшивки позволяет выделить РНК-белковые комплексы и получить таким образом еще больше информации [4, 323].
Крайне интересен метод проксимальноспецифичного рибосомного профайлинга, разработанный в лаборатории Дж. Вайсмана (J.S. Weissman) [8, 73]. Он позволяет пометить биотином и выделить только те рибосомы, которые в данный момент находятся в интересующем компартменте, а затем секвенировать заключенные в них фрагменты мРНК, которые они в этот момент транслировали. Используя такой вариант рибосомного профайлинга, авторы оценили динамику трансляции мРНК рибосомами, локализованными на мембранах ЭПР [8] и митохондрий [73].
Своеобразным апогеем “локальной транскриптомики” стал недавно разработанный метод секвенирования фрагментов РНК in situ, то есть прямо на фиксированном препарате клеток или тканей [324‒326]. Так удается получать качественную и количественную информацию о распределении транскриптов на срезе или даже в трехмерном формате, хотя метод ограничен разрешением микроскопа.
Принципиально иной подход связан с развитием метода флуоресцентной гибридизации in situ (FISH). Этот метод даже в классическом своем варианте позволял собирать информацию по субклеточной локализации сотен и даже тысяч индивидуальных транскриптов [225], хотя для этого требовалось изготовление индивидуальных фиксированных препаратов. Современные же варианты FISH, с применением мультиплексирования, позволяют детектировать сотни и тысячи индивидуальных транскриптов одновременно и сразу в большом числе клеток [130, 226, 327‒329]. В совокупности с прогрессивными методами флуоресцентной визуализации единичных молекул репортерных мРНК, появившимися в последнее время [330], это дает возможность оценить неоднородность в характере трансляции индивидуальных транскриптов. К недостаткам этих методов, по сравнению с подходами с использованием секвенирования, относится “аналоговый” принцип формирования сигнала, хотя именно технологии на основе FISH позволяют напрямую визуализировать расположение транскриптов в клетке.
Применение методов системной биологии позволило убедиться, что существенная доля мРНК (в зависимости от организма, вида клеток, стадии развития, условий и примененного метода – от нескольких процентов до ~3/4 выявляемых транскриптов) имеет в клетке четко детерминированную субклеточную локализацию: апикально-базальную, в протрузиях и местах клеточных контактов, связанную с мембранами органелл, с центросомой, микротрубочками, различными видами гранул и т.д. [13, 225, 331‒333]. Таким образом, адресная доставка мРНК с ее последующей локализованной трансляцией представляет исключительно важный механизм формирования внутриклеточных структур, компартментов и функционально отличающихся полюсов клеток.
ЗАКЛЮЧЕНИЕ
Приведенные выше факты позволяют заключить, что роль внутриклеточной локализации мРНК и локальной трансляции в поддержании целостности протеома и в нормальном функционировании эукариотической клетки, несомненно, высока. Изучение этих аспектов биосинтеза белка вызывает большой интерес в современной науке и активно развивается. Этому способствует постоянно расширяющийся арсенал разнообразных методов. Тем не менее нерешенных вопросов пока остается довольно много. Например, в то время как процессы котрансляционного рекрутирования рибосом на мембрану ЭПР и транслокация пептида в люмен описаны достаточно подробно, аналогичные события на митохондриях, а тем более в окрестностях других органелл нуждаются в более глубоком изучении. Неясен вклад “трансляционных фабрик” и прочих подобных РНК-конденсатов в процессы жизнедеятельности клетки, не изучена их структурная подоплека, практически отсутствуют данные о том, что происходит с этими элементами клетки под действием стресса, вирусной инфекции и других воздействий. Остаются нерешенными вопросы особенностей структуры трансляционных комплексов и регуляции биосинтеза белка в разных субклеточных компартментах. Тем не менее постоянно увеличивающееся число работ в области локализованной трансляции позволяет надеяться, что в ближайшее время многие из этих загадок будут решены и нас ждет еще много волнующих открытий.
Авторы благодарны Ларисе Мочаловой за обсуждение текста и ценные замечания. Работа выполнена при финансовой поддержке РФФИ (программа “Экспансия”, грант № 19-14-50309). Авторы являются членами Междисциплинарной научно-образовательной школы Московского университета “Молекулярные технологии живых систем и синтетическая биология”.
Настоящая статья не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Palade G.E. (1955) A small particulate component of the cytoplasm. J. Biophys. Biochem. Cytol. 1, 59‒68. https://doi.org/10.1083/jcb.1.1.59
Diehn M., Bhattacharya R., Botstein D., Brown P.O. (2006) Genome-scale identification of membrane-associated human mRNAs. PLoS Genet. 2, e11. https://doi.org/10.1371/journal.pgen.0020011
Diehn M., Eisen M.B., Botstein D., Brown P.O. (2000) Large-scale identification of secreted and membrane-associated gene products using DNA microarrays. Nat. Genet. 25, 58–62. https://doi.org/10.1038/75603
Kaewsapsak P., Shechner D.M., Mallard W., Rinn J.L., Ting A.Y. (2017) Live-cell mapping of organelle-associated RNAs via proximity biotinylation combined with protein-RNA crosslinking. eLife. 6, e29224. https://doi.org/10.7554/eLife.29224
Fazal F.M., Han S., Parker K.R., Kaewsapsak P., Xu J., Boettiger A.N., Chang H.Y., Ting A.Y. (2019) Atlas of subcellular RNA localization revealed by APEX-Seq. Cell. 178, 473‒490.e426. https://doi.org/10.1016/j.cell.2019.05.027
Lerner R.S., Seiser R.M., Zheng T., Lager P.J., Reedy M.C., Keene J.D., Nicchitta C.V. (2003) Partitioning and translation of mRNAs encoding soluble proteins on membrane-bound ribosomes. RNA. 9, 1123‒1137. https://doi.org/10.1261/rna.5610403
Reid D.W., Nicchitta C.V. (2012) Primary role for endoplasmic reticulum-bound ribosomes in cellular translation identified by ribosome profiling. J. Biol. Chem. 287, 5518‒5527. https://doi.org/10.1074/jbc.M111.312280
Jan C.H., Williams C.C., Weissman J.S. (2014) Principles of ER cotranslational translocation revealed by proximity-specific ribosome profiling. Science. 346, 1257521. https://doi.org/10.1126/science.1257521
Jagannathan S., Hsu J.C., Reid D.W., Chen Q., Thompson W.J., Moseley A.M., Nicchitta C.V. (2014) Multifunctional roles for the protein translocation machinery in RNA anchoring to the endoplasmic reticulum. J. Biol. Chem. 289, 25907‒25924. https://doi.org/10.1074/jbc.M114.580688
Voigt F., Zhang H., Cui X.A., Triebold D., Liu A.X., Eglinger J., Lee E.S., Chao J.A., Palazzo A.F. (2017) Single-molecule quantification of translation-dependent association of mRNAs with the endoplasmic reticulum. Cell Rep. 21, 3740‒3753. https://doi.org/10.1016/j.celrep.2017.12.008
Hoffman A.M., Chen Q., Zheng T., Nicchitta C.V. (2019) Heterogeneous translational landscape of the endoplasmic reticulum revealed by ribosome proximity labeling and transcriptome analysis. J. Biol. Chem. 294, 8942‒8958. https://doi.org/10.1074/jbc.RA119.007996
Kraut-Cohen J., Afanasieva E., Haim-Vilmovsky L., Slobodin B., Yosef I., Bibi E., Gerst J.E. (2013) Translation- and SRP-independent mRNA targeting to the endoplasmic reticulum in the yeast Saccharomyces cerevisiae. Mol. Biol. Cell. 24, 3069‒3084. https://doi.org/10.1091/mbc.E13-01-0038
Weis B.L., Schleiff E., Zerges W. (2013) Protein targeting to subcellular organelles via MRNA localization. Biochim. Biophys. Acta. 1833, 260‒273. https://doi.org/10.1016/j.bbamcr.2012.04.004
Kraut-Cohen J., Gerst J.E. (2010) Addressing mRNAs to the ER: cis sequences act up! Trends Biochem. Sci. 35, 459‒469. https://doi.org/10.1016/j.tibs.2010.02.006
Ma W., Mayr C. (2018) A membraneless organelle associated with the endoplasmic reticulum enables 3'UTR-mediated protein–protein interactions. Cell. 175, 1492‒1506, e1419. https://doi.org/10.1016/j.cell.2018.10.007
Blobel G., Sabatini D.D. (1971) Ribosome-membrane interaction in eukaryotic cells. In: Biomembranes. Ed. Manson L.A. Boston, MA.: Springer, 193‒195. https://doi.org/10.1007/978-1-4684-3330-2_16
Blobel G., Dobberstein B. (1975) Transfer of proteins across membranes. I. Presence of proteolytically processed and unprocessed nascent immunoglobulin light chains on membrane-bound ribosomes of murine myeloma. J. Cell Biol. 67, 835‒851. https://doi.org/10.1083/jcb.67.3.835
Walter P., Ibrahimi I., Blobel G. (1981) Translocation of proteins across the endoplasmic reticulum. I. Signal recognition protein (SRP) binds to in-vitro-assembled polysomes synthesizing secretory protein. J. Cell Biol. 91, 545‒550. https://doi.org/10.1083/jcb.91.2.545
Gilmore R., Blobel G., Walter P. (1982) Protein translocation across the endoplasmic reticulum. I. Detection in the microsomal membrane of a receptor for the signal recognition particle. J. Cell Biol. 95, 463‒469. https://doi.org/10.1083/jcb.95.2.463
Nilsson I., Lara P., Hessa T., Johnson A.E., von Heijne G., Karamyshev A.L. (2015) The code for directing proteins for translocation across ER membrane: SRP cotranslationally recognizes specific features of a signal sequence. J. Mol. Biol. 427, 1191–1201. https://doi.org/10.1016/j.jmb.2014.06.014
Costa E.A., Subramanian K., Nunnari J., Weissman J.S. (2018) Defining the physiological role of SRP in protein-targeting efficiency and specificity. Science. 359, 689–692. https://doi.org/10.1126/science.aar3607
Matsuo Y., Inada T. (2021) The ribosome collision sensor Hel2 functions as preventive quality control in the secretory pathway. Cell Rep. 34, 108877. https://doi.org/10.1016/j.celrep.2021.108877
Pfeffer S., Burbaum L., Unverdorben P., Pech M., Chen Y., Zimmermann R., Beckmann, R., Forster F. (2015) Structure of the native Sec61 protein-conducting channel. Nat. Commun. 6, 8403. https://doi.org/10.1038/ncomms9403
Voorhees R.M., Hegde R.S. (2016) Structure of the Sec61 channel opened by a signal sequence. Science. 351, 88–91. https://doi.org/10.1126/science.aad4992
Nyathi Y., Wilkinson B.M., Pool M.R. (2013) Co-translational targeting and translocation of proteins to the endoplasmic reticulum. Biochim. Biophys. Acta. 1833, 2392–2402. https://doi.org/10.1016/j.bbamcr.2013.02.021
Aviram N., Schuldiner M. (2017) Targeting and translocation of proteins to the endoplasmic reticulum at a glance. J. Cell Sci. 130, 4079–4085. https://doi.org/10.1242/jcs.204396
Hetz C., Papa F.R. (2018) The unfolded protein response and cell fate control. Mol. Cell. 69, 169–181. https://doi.org/10.1016/j.molcel.2017.06.017
Yanagitani K., Imagawa Y., Iwawaki T., Hosoda A., Saito M., Kimata Y., Kohno K. (2009) Cotranslational targeting of XBP1 protein to the membrane promotes cytoplasmic splicing of its own mRNA. Mol. Cell. 34, 191–200. https://doi.org/10.1016/j.molcel.2009.02.033
Yanagitani K., Kimata Y., Kadokura H., Kohno K. (2011) Translational pausing ensures membrane targeting and cytoplasmic splicing of XBP1u mRNA. Science. 331, 586–589. https://doi.org/10.1126/science.1197142
Shanmuganathan V., Schiller N., Magoulopoulou A., Cheng J., Braunger K., Cymer F., Berninghausen O., Beatrix B., Kohno K., von Heijne G., Beckmann R. (2019) Structural and mutational analysis of the ribosome-arresting human XBP1u. eLife. 8, e46267. https://doi.org/10.7554/eLife.46267
Kanda S., Yanagitani K., Yokota Y., Esaki Y., Kohno K. (2016) Autonomous translational pausing is required for XBP1u mRNA recruitment to the ER via the SRP pathway. Proc. Natl. Acad. Sci. USA. 113, E5886–E5895. https://doi.org/10.1073/pnas.1604435113
Plumb R., Zhang Z.R., Appathurai S., Mariappan M. (2015) A functional link between the co-translational protein translocation pathway and the UPR. eLife. 4, e07426. https://doi.org/10.7554/eLife.07426
Chen C.Y., Malchus N.S., Hehn B., Stelzer W., Avci D., Langosch D., Lemberg M.K. (2014) Signal peptide peptidase functions in ERAD to cleave the unfolded protein response regulator XBP1u. EMBO J. 33, 2492–2506. https://doi.org/10.15252/embj.201488208
Han P., Shichino Y., Schneider-Poetsch T., Mito M., Hashimoto S., Udagawa T., Kohno K., Yoshida M., Mishima Y., Inada T., Iwasaki S. (2020) Genome-wide survey of ribosome collision. Cell Rep. 31, 107610. https://doi.org/10.1016/j.celrep.2020.107610
Mutka S.C., Walter P. (2001) Multifaceted physiological response allows yeast to adapt to the loss of the signal recognition particle-dependent protein-targeting pathway. Mol. Biol. Cell. 12, 577–588. https://doi.org/10.1091/mbc.12.3.577
Liu L., Liang X.H., Uliel S., Unger R., Ullu E., Michaeli S. (2002) RNA interference of signal peptide-binding protein SRP54 elicits deleterious effects and protein sorting defects in trypanosomes. J. Biol. Chem. 277, 47348–47357. https://doi.org/10.1074/jbc.M207736200
Ren Y.G., Wagner K.W., Knee D.A., Aza-Blanc P., Nasoff M., Deveraux Q.L. (2004) Differential regulation of the TRAIL death receptors DR4 and DR5 by the signal recognition particle. Mol. Biol. Cell. 15, 5064–5074. https://doi.org/10.1091/mbc.e04-03-0184
Johnson N., Powis K., High S. (2013) Post-translational translocation into the endoplasmic reticulum. Biochim. Biophys. Acta. 1833, 2403–2409. https://doi.org/10.1016/j.bbamcr.2012.12.008
Shao S., Hegde R.S. (2011) A calmodulin-dependent translocation pathway for small secretory proteins. Cell. 147, 1576–1588. https://doi.org/10.1016/j.cell.2011.11.048
Ast T., Schuldiner M. (2013) All roads lead to Rome (but some may be harder to travel): SRP-independent translocation into the endoplasmic reticulum. Crit. Rev. Biochem. Mol. Biol. 48, 273–288. https://doi.org/10.3109/10409238.2013.782999
Borgese N., Coy-Vergara J., Colombo S.F., Schwappach B. (2019) The ways of tails: the GET pathway and more. Protein J. 38, 289–305. https://doi.org/10.1007/s10930-019-09845-4
Liao G., Ma X., Liu G. (2011) An RNA-zipcode-independent mechanism that localizes Dia1 mRNA to the perinuclear ER through interactions between Dia1 nascent peptide and Rho-GTP. J. Cell Sci. 124, 589–599. https://doi.org/10.1242/jcs.072421
Christensen A.K., Kahn L.E., Bourne C.M. (1987) Circular polysomes predominate on the rough endoplasmic reticulum of somatotropes and mammotropes in the rat anterior pituitary. Am. J. Anat. 178, 1–10. https://doi.org/10.1002/aja.1001780102
Alekhina O.M., Terenin I.M., Dmitriev S.E., Vassilenko K.S. (2020) Functional cyclization of eukaryotic mRNAs. Int. J. Mol. Sci. 21, 1677. https://doi.org/10.3390/ijms21051677
Pyhtila B., Zheng T., Lager P.J., Keene J.D., Reedy M.C., Nicchitta C.V. (2008) Signal sequence- and translation-independent mRNA localization to the endoplasmic reticulum. RNA. 14, 445–453. https://doi.org/10.1261/rna.721108
Loya A., Pnueli L., Yosefzon Y., Wexler Y., Ziv-Ukelson M., Arava Y. (2008) The 3'-UTR mediates the cellular localization of an mRNA encoding a short plasma membrane protein. RNA. 14, 1352–1365. https://doi.org/10.1261/rna.867208
Cohen-Zontag O., Baez C., Lim L.Q.J., Olender T., Schirman D., Dahary D., Pilpel Y., Gerst J.E. (2019) A secretion-enhancing cis regulatory targeting element (SECReTE) involved in mRNA localization and protein synthesis. PLoS Genet. 15, e1008248. https://doi.org/10.1371/journal.pgen.1008248
Ma W., Zhen G., Xie W., Mayr C. (2020) Unstructured mRNAs form multivalent RNA-RNA interactions to generate TIS granule networks. bioRxiv. 2020.02.14.949503. https://doi.org/10.1101/2020.02.14.949503
Cui X.A., Zhang H., Palazzo A.F. (2012) p180 promotes the ribosome-independent localization of a subset of mRNA to the endoplasmic reticulum. PLoS Biol. 10, e1001336. https://doi.org/10.1371/journal.pbio.1001336
Savitz A.J., Meyer D.I. (1990) Identification of a ribosome receptor in the rough endoplasmic reticulum. Nature. 346, 540–544. https://doi.org/10.1038/346540a0
Ueno T., Kaneko K., Sata T., Hattori S., Ogawa-Goto K. (2012) Regulation of polysome assembly on the endoplasmic reticulum by a coiled-coil protein, p180. Nucleic Acids Res. 40, 3006–3017. https://doi.org/10.1093/nar/gkr1197
Hsu J.C., Reid D.W., Hoffman A.M., Sarkar D., Nicchitta C.V. (2018) Oncoprotein AEG-1 is an endoplasmic reticulum RNA-binding protein whose interactome is enriched in organelle resident protein-encoding mRNAs. RNA. 24, 688–703. https://doi.org/10.1261/rna.063313.117
Acosta-Alvear D., Karagoz G.E., Frohlich F., Li H., Walther T.C., Walter P. (2018) The unfolded protein response and endoplasmic reticulum protein targeting machineries converge on the stress sensor IRE1. eLife. 7, e43036. https://doi.org/10.7554/eLife.43036
Genz C., Fundakowski J., Hermesh O., Schmid M., Jansen R.P. (2013) Association of the yeast RNA-binding protein She2p with the tubular endoplasmic reticulum depends on membrane curvature. J. Biol. Chem. 288, 32384–32393. https://doi.org/10.1074/jbc.M113.486431
Colomina N., Ferrezuelo F., Wang H., Aldea M., Gari E. (2008) Whi3, a developmental regulator of budding yeast, binds a large set of mRNAs functionally related to the endoplasmic reticulum. J. Biol. Chem. 283, 28670–28679. https://doi.org/10.1074/jbc.M804604200
Syed M.I., Moorthy B.T., Jenner A., Fetka I., Jansen R.P. (2018) Signal sequence-independent targeting of MID2 mRNA to the endoplasmic reticulum by the yeast RNA-binding protein Khd1p. FEBS Lett. 592, 1870–1881. https://doi.org/10.1002/1873-3468.13098
Castello A., Fischer B., Frese C.K., Horos R., Alleaume A.M., Foehr S., Curk T., Krijgsveld J., Hentze M.W. (2016) Comprehensive identification of RNA-binding domains in human cells. Mol. Cell. 63, 696–710. https://doi.org/10.1016/j.molcel.2016.06.029
Baltz A.G., Munschauer M., Schwanhausser B., Vasile A., Murakawa Y., Schueler M., Youngs N., Penfold-Brown D., Drew K., Milek M., Wyler E., Bonneau R., Selbach M., Dieterich C., Landthaler M. (2012) The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Mol. Cell. 46, 674–690. https://doi.org/10.1016/j.molcel.2012.05.021
Bethune J., Jansen R.P., Feldbrugge M., Zarnack K. (2019) Membrane-associated RNA-binding proteins orchestrate organelle-coupled translation. Trends Cell Biol. 29, 178–188. https://doi.org/10.1016/j.tcb.2018.10.005
Kalies K.U., Gorlich D., Rapoport T.A. (1994) Binding of ribosomes to the rough endoplasmic reticulum mediated by the Sec61p-complex. J. Cell Biol. 126, 925–934.https://doi.org/10.1083/jcb.126.4.925
Harada Y., Li H., Li H., Lennarz W.J. (2009) Oligosaccharyltransferase directly binds to ribosome at a location near the translocon-binding site. Proc. Natl. Acad. Sci. USA. 106, 6945–6949. https://doi.org/10.1073/pnas.0812489106
Pfeffer S., Dudek J., Zimmermann R., Forster F. (2016) Organization of the native ribosome-translocon complex at the mammalian endoplasmic reticulum membrane. Biochim. Biophys. Acta. 1860, 2122–2129. https://doi.org/10.1016/j.bbagen.2016.06.024
Hansen K.G., Herrmann J.M. (2019) Transport of proteins into mitochondria. Protein J. 38, 330–342. https://doi.org/10.1007/s10930-019-09819-6
Becker T., Song J., a Pfanner N. (2019) Versatility of preprotein transfer from the cytosol to mitochondria. Trends Cell Biol. 29, 534–548. https://doi.org/10.1016/j.tcb.2019.03.007
Wiedemann N., Pfanner N. (2017) Mitochondrial machineries for protein import and assembly. Ann. Rev. Biochem. 86, 685–714. https://doi.org/10.1146/annurev-biochem-060815-014352
Kellems R.E., Butow R.A. (1972) Cytoplasmic-type 80 S ribosomes associated with yeast mitochondria. I. Evidence for ribosome binding sites on yeast mitochondria. J. Biol. Chem. 247, 8043–8050.
Kellems R.E., Allison V.F., Butow R.A. (1974) Cytoplasmic type 80 S ribosomes associated with yeast mitochondria. II. Evidence for the association of cytoplasmic ribosomes with the outer mitochondrial membrane in situ. J. Biol. Chem. 249, 3297–3303.
Suissa M., Schatz G. (1982) Import of proteins into mitochondria. Translatable mRNAs for imported mitochondrial proteins are present in free as well as mitochondria-bound cytoplasmic polysomes. J. Biol. Chem. 257, 13048–13055.
Ades I.Z., Butow R.A. (1980) The products of mitochondria-bound cytoplasmic polysomes in yeast. J. Biol. Chem. 255, 9918–9924.
Fujiki M., Verner K. (1993) Coupling of cytosolic protein synthesis and mitochondrial protein import in yeast. Evidence for cotranslational import in vivo. J. Biol. Chem. 268, 1914–1920.
Mukhopadhyay A., Ni L., Weiner H. (2004) A co-translational model to explain the in vivo import of proteins into HeLa cell mitochondria. Biochem. J. 382, 385–392. https://doi.org/10.1042/BJ20040065
Yogev O., Karniely S., Pines O. (2007) Translation-coupled translocation of yeast fumarase into mitochondria in vivo. J. Biol. Chem. 282, 29222–29229. https://doi.org/10.1074/jbc.M704201200
Williams C.C., Jan C.H., Weissman J.S. (2014) Targeting and plasticity of mitochondrial proteins revealed by proximity-specific ribosome profiling. Science. 346, 748–751. https://doi.org/10.1126/science.1257522
MacKenzie J.A., Payne R.M. (2004) Ribosomes specifically bind to mammalian mitochondria via protease-sensitive proteins on the outer membrane. J. Biol. Chem. 279, 9803–9810. https://doi.org/10.1074/jbc.M307167200
Gold V.A., Chroscicki P., Bragoszewski P., Chacinska A. (2017) Visualization of cytosolic ribosomes on the surface of mitochondria by electron cryo-tomography. EMBO Rep. 18, 1786–1800. https://doi.org/10.15252/embr.201744261
Eliyahu E., Pnueli L., Melamed D., Scherrer T., Gerber A.P., Pines O., Rapaport D., Arava Y. (2010) Tom20 mediates localization of mRNAs to mitochondria in a translation-dependent manner. Mol. Cell. Biol. 30, 284–294. https://doi.org/10.1128/MCB.00651-09
George R., Beddoe T., Landl K., Lithgow T. (1998) The yeast nascent polypeptide-associated complex initiates protein targeting to mitochondria in vivo. Proc. Natl. Acad. Sci. USA. 95, 2296–2301. https://doi.org/10.1073/pnas.95.5.2296
George R., Walsh P., Beddoe T., Lithgow T. (2002) The nascent polypeptide-associated complex (NAC) promotes interaction of ribosomes with the mitochondrial surface in vivo. FEBS Lett. 516, 213–216. https://doi.org/10.1016/s0014-5793(02)02528-0
Gamerdinger M., Hanebuth M.A., Frickey T., Deuerling E. (2015) The principle of antagonism ensures protein targeting specificity at the endoplasmic reticulum. Science. 348, 201–207. https://doi.org/10.1126/science.aaa5335
Wiedmann B., Sakai H., Davis T.A., Wiedmann M. (1994) A protein complex required for signal-sequence-specific sorting and translocation. Nature. 370, 434–440. https://doi.org/10.1038/370434a0
Powers T., Walter P. (1996) The nascent polypeptide-associated complex modulates interactions between the signal recognition particle and the ribosome. Curr. Biol. 6, 331–338. https://doi.org/10.1016/s0960-9822(02)00484-0
Hsieh H.H., Lee J.H., Chandrasekar S., Shan S.O. (2020) A ribosome-associated chaperone enables substrate triage in a cotranslational protein targeting complex. Nat. Commun. 11, 5840. https://doi.org/10.1038/s41467-020-19548-5
Funfschilling U., Rospert S. (1999) Nascent polypeptide-associated complex stimulates protein import into yeast mitochondria. Mol. Biol. Cell. 10, 3289–3299. https://doi.org/10.1091/mbc.10.10.3289
Lesnik C., Cohen Y., Atir-Lande A., Schuldiner M., Arava Y. (2014) OM14 is a mitochondrial receptor for cytosolic ribosomes that supports co-translational import into mitochondria. Nat. Commun. 5, 5711. https://doi.org/10.1038/ncomms6711
Ponce-Rojas J.C., Avendano-Monsalve M.C., Yanez-Falcon A.R., Jaimes-Miranda F., Garay E., Torres-Quiroz F., DeLuna A., Funes S. (2017) αβ'-NAC cooperates with Sam37 to mediate early stages of mitochondrial protein import. FEBS J. 284, 814–830. https://doi.org/10.1111/febs.14024
Ahmed A.U., Fisher P.R. (2009) Import of nuclear-encoded mitochondrial proteins: a cotranslational perspective. Int. Rev. Cell Mol. Biol. 273, 49‒68. https://doi.org/10.1016/S1937-6448(08)01802-9
Lesnik C., Golani-Armon A., Arava Y. (2015) Loca-lized translation near the mitochondrial outer membrane: an update. RNA Biol. 12, 801–809. https://doi.org/10.1080/15476286.2015.1058686
Sylvestre J., Vialette S., Corral Debrinski M., Jacq C. (2003) Long mRNAs coding for yeast mitochondrial proteins of prokaryotic origin preferentially localize to the vicinity of mitochondria. Genome Biol. 4, R44. https://doi.org/10.1186/gb-2003-4-7-r44
Gadir N., Haim-Vilmovsky L., Kraut-Cohen J., Gerst J.E. (2011) Localization of mRNAs coding for mitochondrial proteins in the yeast Saccharomyces cerevisiae. RNA. 17, 1551–1565. https://doi.org/10.1261/rna.2621111
Matsumoto, S., Uchiumi T., Saito T., Yagi M., Takazaki S., Kanki T., Kang D. (2012) Localization of mRNAs encoding human mitochondrial oxidative phosphorylation proteins. Mitochondrion. 12, 391–398. https://doi.org/10.1016/j.mito.2012.02.004
Zabezhinsky D., Slobodin B., Rapaport D., Gerst J.E. (2016) An essential role for COPI in mRNA localization to mitochondria and mitochondrial function. Cell Rep. 15, 540–549. https://doi.org/10.1016/j.celrep.2016.03.053
Sabharwal A., Sharma D., Vellarikkal S.K., Jayarajan R., Verma A., Senthivel V., Scaria V., Sivasubbu S. (2019) Organellar transcriptome sequencing reveals mitochondrial localization of nuclear encoded transcripts. Mitochondrion. 46, 59–68. https://doi.org/10.1016/j.mito.2018.02.007
Golani-Armon A., Arava Y. (2016) Localization of nuclear-encoded mRNAs to mitochondria outer surface. Biochemistry (Mosc.). 81, 1038–1043. https://doi.org/10.1134/S0006297916100023
Sylvestre J., Margeot A., Jacq C., Dujardin G., Corral-Debrinski M. (2003) The role of the 3' untransla-ted region in mRNA sorting to the vicinity of mitochondria is conserved from yeast to human cells. Mol. Biol. Cell. 14, 3848–3856. https://doi.org/10.1091/mbc.e03-02-0074
Garcia M., Delaveau T., Goussard S., Jacq C. (2010) Mitochondrial presequence and open reading frame mediate asymmetric localization of messenger RNA. EMBO Rep. 11, 285–291. https://doi.org/10.1038/embor.2010.17
Eliyahu E., Lesnik C., Arava Y. (2012) The protein chaperone Ssa1 affects mRNA localization to the mitochondria. FEBS Lett. 586, 64–69. https://doi.org/10.1016/j.febslet.2011.11.025
Corral-Debrinski M., Blugeon C., Jacq C. (2000) In yeast, the 3' untranslated region or the presequence of ATM1 is required for the exclusive localization of its mRNA to the vicinity of mitochondria. Mol. Cell. Biol. 20, 7881–7892. https://doi.org/10.1128/mcb.20.21.7881-7892.2000
Poulsen T.M., Imai K., Frith M.C., Horton P. (2019) Hallmarks of slow translation initiation revealed in mitochondrially localizing mRNA sequences. bioRxiv. 614255. https://doi.org/10.1101/614255
Lin Y., Li F., Huang L., Polte C., Duan H., Fang J., Sun L., Xing X., Tian G., Cheng Y., Ignatova Z., Yang X., Wolf D.A. (2020) eIF3 associates with 80S ribosomes to promote translation elongation, mitochondrial homeostasis, and muscle health. Mol. Cell. 79, 575–587.e7. https://doi.org/10.1016/j.molcel.2020.06.003
Schatton D., Rugarli E.I. (2018) A concert of RNA-binding proteins coordinates mitochondrial function. Crit. Rev. Biochem. Mol. Biol. 53, 652–666. https://doi.org/10.1080/10409238.2018.1553927
Gerber A.P., Herschlag D., Brown P.O. (2004) Extensive association of functionally and cytotopically related mRNAs with Puf family RNA-binding proteins in yeast. PLoS Biol. 2, E79. https://doi.org/10.1371/journal.pbio.0020079
Jacobs Anderson J.S., Parker R. (2000) Computational identification of cis-acting elements affecting post-transcriptional control of gene expression in Saccharomyces cerevisiae. Nucleic Acids Res. 28, 1604–1617. https://doi.org/10.1093/nar/28.7.1604
Saint-Georges Y., Garcia M., Delaveau, T., Jourdren L., Le Crom S., Lemoine S., Tanty V., Devaux F., Jacq C. (2008) Yeast mitochondrial biogenesis: a role for the PUF RNA-binding protein Puf3p in mRNA localization. PLoS One. 3, e2293. https://doi.org/10.1371/journal.pone.0002293
Quenault T., Lithgow T., Traven A. (2011) PUF proteins: repression, activation and mRNA localization. Trends Cell Biol. 21, 104–112. https://doi.org/10.1016/j.tcb.2010.09.013
Lee C.D., Tu B.P. (2015) Glucose-regulated phosphorylation of the PUF protein Puf3 regulates the translational fate of its bound mRNAs and association with RNA granules. Cell Rep. 11, 1638–1650. https://doi.org/10.1016/j.celrep.2015.05.014
Kershaw C.J., Costello J.L., Talavera D., Rowe W., Castelli L.M., Sims P.F., Grant C.M., Ashe M.P., Hubbard S.J., Pavitt G.D. (2015) Integrated multi-omics analyses reveal the pleiotropic nature of the control of gene expression by Puf3p. Sci. Rep. 5, 15518. https://doi.org/10.1038/srep15518
Lapointe C.P., Stefely J.A., Jochem A., Hutchins P.D., Wilson G.M., Kwiecien N.W., Coon J.J., Wickens M., Pagliarini D.J. (2018) Multi-omics reveal specific targets of the RNA-binding protein Puf3p and its orchestration of mitochondrial biogenesis. Cell Syst. 6, 125–135.e6. https://doi.org/10.1016/j.cels.2017.11.012
Wang Z., Sun X., Wee J., Guo X., Gu Z. (2019) Novel insights into global translational regulation through Pumilio family RNA-binding protein Puf3p revealed by ribosomal profiling. Curr. Genet. 65, 201–212. https://doi.org/10.1007/s00294-018-0862-4
Qin W., Myers S.A., Carey D.K., Carr S.A., Ting A.Y. (2020) Functional proximity mapping of RNA binding proteins uncovers a mitochondrial mRNA anchor that promotes stress recovery. bioRxiv. 2020.11.17.387209. https://doi.org/10.1101/2020.11.17.387209
Gehrke S., Wu Z., Klinkenberg M., Sun Y., Auburger G., Guo S., Lu B. (2015) PINK1 and Parkin control loca-lized translation of respiratory chain component mRNAs on mitochondria outer membrane. Cell Metab. 21, 95–108. https://doi.org/10.1016/j.cmet.2014.12.007
Zhang Y., Wang Z. H., Liu Y., Chen Y., Sun N., Gucek M., Zhang, F., Xu H. (2019) PINK1 inhibits local protein synthesis to limit transmission of deleterious mitochondrial DNA mutations. Mol. Cell. 73, 1127–1137.e1125. https://doi.org/10.1016/j.molcel.2019.01.013
Zhang Y., Chen Y., Gucek M., Xu H. (2016) The mitochondrial outer membrane protein MDI promotes local protein synthesis and mtDNA replication. EMBO J. 35, 1045–1057. https://doi.org/10.15252/embj.201592994
Gao J., Schatton D., Martinelli P., Hansen H., Pla-Martin D., Barth E., Becker C., Altmueller J., Frommolt P., Sardiello M., Rugarli E.I. (2014) CLUH re-gulates mitochondrial biogenesis by binding mRNAs of nuclear-encoded mitochondrial proteins. J. Cell Biol. 207, 213–223. https://doi.org/10.1083/jcb.201403129
Schatton D., Pla-Martin D., Marx M.C., Hansen H., Mourier A., Nemazanyy I., Pessia A., Zentis P., Corona T., Kondylis V., Barth E., Schauss A.C., Velagapudi V., Rugarli E.I. (2017) CLUH regulates mitochondrial metabolism by controlling translation and decay of target mRNAs. J. Cell Biol. 216, 675‒693. https://doi.org/10.1083/jcb.201607019
Sen A., Kalvakuri S., Bodmer R., Cox R.T. (2015) Clueless, a protein required for mitochondrial function, interacts with the PINK1-Parkin complex in Drosophila. Dis. Model. Mech. 8, 577–589. https://doi.org/10.1242/dmm.019208
Sen A., Cox R.T. (2016) Clueless is a conserved ribonucleoprotein that binds the ribosome at the mitochondrial outer membrane. Biol. Open. 5, 195–203. https://doi.org/10.1242/bio.015313
Giacomello M., Pellegrini L. (2016) The coming of age of the mitochondria-ER contact: a matter of thickness. Cell Death Differ. 23, 1417–1427. https://doi.org/10.1038/cdd.2016.52
Shimizu S. (2019) Organelle zones in mitochondria. J. Biochem. 165, 101–107. https://doi.org/10.1093/jb/mvy068
Yogev O., Pines O. (2011) Dual targeting of mitochondrial proteins: mechanism, regulation and function. Biochim. Biophys. Acta. 1808, 1012–1020. https://doi.org/10.1016/j.bbamem.2010.07.004
Hansen K.G., Aviram N., Laborenz J., Bibi C., Meyer M., Spang A., Schuldiner M., Herrmann J.M. (2018) An ER surface retrieval pathway safeguards the import of mitochondrial membrane proteins in yeast. Science. 361, 1118–1122. https://doi.org/10.1126/science.aar8174
Farre J.C., Mahalingam S.S., Proietto M., Subramani S. (2019) Peroxisome biogenesis, membrane contact sites, and quality control. EMBO Rep. 20(1), e46864. https://doi.org/10.15252/embr.201846864
Islinger M., Voelkl A., Fahimi H.D., Schrader M. (2018) The peroxisome: an update on mysteries 2.0. Histochem. Cell Biol. 150, 443–471. https://doi.org/10.1007/s00418-018-1722-5
Mayerhofer P.U., Bano-Polo M., Mingarro I., Johnson A.E. (2016) Human peroxin PEX3 is co-translationally integrated into the ER and exits the ER in budding vesicles. Traffic. 17, 117–130. https://doi.org/10.1111/tra.12350
Ast J., Stiebler A.C., Freitag J., Bolker, M. (2013) Dual targeting of peroxisomal proteins. Front. Physiol. 4, 297. https://doi.org/10.3389/fphys.2013.00297
Jungreis I., Chan C.S., Waterhouse R.M., Fields G., Lin M.F., Kellis M. (2016) Evolutionary dynamics of abundant stop codon readthrough. Mol. Biol. Evol. 33, 3108–3132. https://doi.org/10.1093/molbev/msw189
Schueren F., Thoms S. (2016) Functional translational readthrough: a systems biology perspective. PLoS Genet. 12, e1006196. https://doi.org/10.1371/journal.pgen.1006196
Haimovich G., Cohen-Zontag O., Gerst J.E. (2016) A role for mRNA trafficking and localized translation in peroxisome biogenesis and function? Biochim. Biophys. Acta. 1863, 911–921. https://doi.org/10.1016/j.bbamcr.2015.09.007
Zipor G., Haim-Vilmovsky L., Gelin-Licht R., Gadir N., Brocard C., Gerst J.E. (2009) Localization of mRNAs coding for peroxisomal proteins in the yeast, Saccharomyces cerevisiae. Proc. Natl. Acad. Sci. USA. 106, 19848–19853. https://doi.org/10.1073/pnas.0910754106
Yarmishyn A.A., Kremenskoy M., Batagov A.O., Preuss A., Wong J.H., Kurochkin I.V. (2016) Genome-wide analysis of mRNAs associated with mouse peroxisomes. BMC Genomics. 17, 1028. https://doi.org/10.1186/s12864-016-3330-x
Chouaib R., Safieddine A., Pichon X., Imbert A., Kwon O.S., Samacoits A., Traboulsi A.M., Robert M.C., Tsanov N., Coleno E., Poser I., Zimmer C., Hyman A., Le Hir H., Zibara K., Peter M., Mueller F., Walter T., Bertrand E. (2020) A dual protein-mRNA localization screen reveals compartmentalized translation and widespread co-translational RNA targeting. Dev. Cell. 54(6), 773–791.e5. https://doi.org/10.1016/j.devcel.2020.07.010
Baumann S., Konig J., Koepke J., Feldbrugge M. (2014) Endosomal transport of septin mRNA and protein indicates local translation on endosomes and is required for correct septin filamentation. EMBO Rep. 15, 94–102. https://doi.org/10.1002/embr.201338037
Higuchi Y., Ashwin P., Roger Y., Steinberg G. (2014) Early endosome motility spatially organizes polysome distribution. J. Cell Biol. 204, 343–357. https://doi.org/10.1083/jcb.201307164
Cioni J.M., Lin J.Q., Holtermann A.V., Koppers M., Jakobs M.A.H., Azizi A., Turner-Bridger B., Shigeoka T., Franze K., Harris W.A., Holt C.E. (2019) Late endosomes act as mRNA translation platforms and sustain mitochondria in axons. Cell. 176, 56–72.e15. https://doi.org/10.1016/j.cell.2018.11.030
McLeod T., Abdullahi A., Li M., Brogna S. (2014) Recent studies implicate the nucleolus as the major site of nuclear translation. Biochem. Soc. Trans. 42, 1224–1228. https://doi.org/10.1042/BST20140062
Reid D.W., Nicchitta C.V. (2012) The enduring enigma of nuclear translation. J. Cell Biol. 197, 7–9. https://doi.org/10.1083/jcb.201202140
Dahlberg J., Lund E. (2012) Nuclear translation or nuclear peptidyl transferase? Nucleus. 3, 320‒321. https://doi.org/10.4161/nucl.20754
Allfrey V.G. (1954) Amino acid incorporation by isolated thymus nuclei. I. The role of desoxyribonucleic acid in protein synthesis. Proc. Natl. Acad. Sci. USA. 40, 881–885. https://doi.org/10.1073/pnas.40.10.881
Allfrey V.G., Mirsky A.E. (1955) Protein synthesis in isolated cell nuclei. Nature. 176, 1042–1049. https://doi.org/10.1038/1761042a0
Стволинская Н.С., Газарян К.Г. (1976) Проблема внутриядерного синтеза белков. Цитология. 18, 125–138.
Goidl J.A., Canaani D., Boublik M., Weissbach H., Dickerman H. (1975) Polyanion-induced release of polyribosomes from HeLa cell nuclei. J. Biol. Chem. 250, 9198–9205.
Birnstiel M.L., Hyde B.B. (1963) Protein synthesis by isolated pea nucleoli. J. Cell Biol. 18, 41–50. https://doi.org/10.1083/jcb.18.1.41
Pena C., Hurt E., Panse V.G. (2017) Eukaryotic ribosome assembly, transport and quality control. Nat. Struct. Mol. Biol. 24, 689–699. https://doi.org/10.1038/nsmb.3454
Espinar-Marchena F.J., Babiano R., Cruz J. (2017) Placeholder factors in ribosome biogenesis: please, pave my way. Microb. Cell. 4, 144–168. https://doi.org/10.15698/mic2017.05.572
Dahlberg J.E., Lund E., Goodwin E.B. (2003) Nuclear translation: what is the evidence? RNA. 9, 1–8. https://doi.org/10.1261/rna.2121703
Al-Jubran K., Wen J., Abdullahi A., Roy Chaudhury S., Li M., Ramanathan P., Matina A., De S., Piechocki K., Rugjee K.N., Brogna S. (2013) Visualization of the joining of ribosomal subunits reveals the presence of 80S ribosomes in the nucleus. RNA. 19, 1669–1683. https://doi.org/10.1261/rna.038356.113
Baboo S., Cook P.R. (2014) “Dark matter” worlds of unstable RNA and protein. Nucleus. 5, 281–286. https://doi.org/10.4161/nucl.29577
Yewdell J.W., David A. (2013) Nuclear translation for immunosurveillance. Proc. Natl. Acad. Sci. USA. 110, 17612–17613. https://doi.org/10.1073/pnas.1318259110
Apcher S., Millot G., Daskalogianni C., Scherl A., Manoury B., Fahraeus R. (2013) Translation of pre-spliced RNAs in the nuclear compartment generates peptides for the MHC class I pathway. Proc. Natl. Acad. Sci. USA. 110, 17951–17956. https://doi.org/10.1073/pnas.1309956110
Martins R.P., Malbert-Colas L., Lista M.J., Daskalogianni C., Apcher S., Pla M., Findakly S., Blondel M., Fahraeus R. (2019) Nuclear processing of nascent transcripts determines synthesis of full-length proteins and antigenic peptides. Nucleic Acids Res. 47, 3086–3100. https://doi.org/10.1093/nar/gky1296
Iborra F.J., Jackson D.A., Cook P.R. (2001) Coupled transcription and translation within nuclei of mammalian cells. Science. 293, 1139–1142. https://doi.org/10.1126/science.1061216
Kim S.H., Koroleva O.A., Lewandowska D., Pendle A.F., Clark G.P., Simpson C.G., Shaw P.J., Brown J.W. (2009) Aberrant mRNA transcripts and the nonsense-mediated decay proteins UPF2 and UPF3 are enriched in the Arabidopsis nucleolus. Plant Cell. 21, 2045–2057. https://doi.org/10.1105/tpc.109.067736
Baboo S., Bhushan B., Jiang H., Grovenor C.R., Pierre P., Davis B.G., Cook P.R. (2014) Most human proteins made in both nucleus and cytoplasm turn over within minutes. PLoS One. 9, e99346. https://doi.org/10.1371/journal.pone.0099346
Lykke-Andersen J., Shu M.D., Steitz J.A. (2000) Human Upf proteins target an mRNA for nonsense-mediated decay when bound downstream of a termination codon. Cell. 103, 1121–1131. https://doi.org/10.1016/s0092-8674(00)00214-2
Wilkinson M.F., Shyu A.B. (2002) RNA surveillance by nuclear scanning? Nat. Cell Biol. 4, E144–E147. https://doi.org/10.1038/ncb0602-e144
Iborra F.J., Jackson D.A., Cook P.R. (2004) The case for nuclear translation. J. Cell Sci. 117, 5713–5720. https://doi.org/10.1242/jcs.01538
David A., Dolan B.P., Hickman H.D., Knowlton J.J., Clavarino G., Pierre P., Bennink J.R., Yewdell J.W. (2012) Nuclear translation visualized by ribosome-bound nascent chain puromycylation. J. Cell Biol. 197, 45‒57. https://doi.org/10.1083/jcb.201112145
Enam S.U., Zinshteyn B., Goldman D.H., Cassani M., Livingston N.M., Seydoux G., Green R. (2020) Puromycin reactivity does not accurately localize translation at the subcellular level. eLife. 9, e60303. https://doi.org/10.7554/eLife.60303
Hobson B.D., Kong L., Hartwick E.W., Gonzalez R.L., Sims P.A. (2020) Elongation inhibitors do not prevent the release of puromycylated nascent polypeptide chains from ribosomes. eLife. 9, e60048. https://doi.org/10.7554/eLife.60048
Ivanov P., Kedersha N., Anderson P. (2019) Stress granules and processing bodies in translational control. Cold Spring Harb. Perspect. Biol. 11(5), a032813. https://doi.org/10.1101/cshperspect.a032813
Corbet G.A., Parker R. (2019) RNP Granule formation: lessons from P-bodies and stress granules. Cold Spring Harb. Symp. Quant. Biol. 84, 203–215. https://doi.org/10.1101/sqb.2019.84.040329
Banani S.F., Lee H.O., Hyman A.A., Rosen M.K. (2017) Biomolecular condensates: organizers of cellular biochemistry. Nat. Rev. Mol. Cell Biol. 18, 285–298. https://doi.org/10.1038/nrm.2017.7
Zhang H., Elbaum-Garfinkle S., Langdon E.M., Taylor N., Occhipinti P., Bridges A.A., Brangwynne C.P., Gladfelter A.S. (2015) RNA controls polyQ protein phase transitions. Mol. Cell. 60, 220–230. https://doi.org/10.1016/j.molcel.2015.09.017
Langdon E.M., Qiu Y., Ghanbari Niaki A., McLaughlin G.A., Weidmann C.A., Gerbich T.M., Smith J.A., Crutchley J.M., Termini C.M., Weeks K.M., Myong S., Gladfelter A.S. (2018) mRNA structure determines specificity of a polyQ-driven phase separation. Science. 360, 922–927. https://doi.org/10.1126/science.aar7432
Van Treeck B., Parker R. (2018) Emerging roles for intermolecular RNA-RNA interactions in RNP assemblies. Cell. 174, 791–802. https://doi.org/10.1016/j.cell.2018.07.023
Begovich K., Wilhelm J.E. (2020) An in vitro assembly system identifies roles for RNA nucleation and ATP in yeast stress granule formation. Mol. Cell. 79, 991–1007.e4. https://doi.org/10.1016/j.molcel.2020.07.017
Kershaw C.J., Nelson M.G., Lui J., Bates C.P., Jennings M.D., Hubbard S.J., Ashe M.P., Grant C.M. (2020) Integrated multi-omics reveals common pro-perties underlying stress granule and P-body formation. bioRxiv. 2020.05.18.102517. https://doi.org/10.1101/2020.05.18.102517
Davidson A., Parton R.M., Rabouille C., Weil T.T., Davis I. (2016) Localized translation of gurken/TGF-alpha mRNA during axis specification is controlled by access to Orb/CPEB on processing bodies. Cell Rep. 14, 2451–2462. https://doi.org/10.1016/j.celrep.2016.02.038
Weil T.T., Parton R.M., Herpers B., Soetaert J., Veenendaal T., Xanthakis D., Dobbie I.M., Hals-tead J.M., Hayashi R., Rabouille C., Davis I. (2012) Drosophila patterning is established by differential association of mRNAs with P bodies. Nat. Cell Biol. 14, 1305–1313. https://doi.org/10.1038/ncb2627
Cougot N., Molza A.E., Giudice E., Cavalier A., Thomas D., Gillet R. (2013) Structural organization of the polysomes adjacent to mammalian processing bodies (P-bodies). RNA Biol. 10, 314–320. https://doi.org/10.4161/rna.23342
Moon S.L., Morisaki T., Khong A., Lyon K., Parker R., Stasevich T.J. (2019) Multicolour single-molecule tracking of mRNA interactions with RNP granules. Nat. Cell Biol. 21, 162–168. https://doi.org/10.1038/s41556-018-0263-4
Mateju D., Eichenberger B., Voigt F., Eglinger J., Roth G., Chao J.A. (2020) Single-molecule imaging reveals translation of mRNAs localized to stress granules. Cell. 183, 1801–1812.e13. https://doi.org/10.1016/j.cell.2020.11.010
Yasuda K., Zhang H., Loiselle D., Haystead T., Macara I.G., Mili S. (2013) The RNA-binding protein Fus directs translation of localized mRNAs in APC-RNP granules. J. Cell Biol. 203, 737–746. https://doi.org/10.1083/jcb.201306058
Mili S., Moissoglu K., Macara I.G. (2008) Genome-wide screen reveals APC-associated RNAs enriched in cell protrusions. Nature. 453, 115–119. https://doi.org/10.1038/nature06888
Buchan J.R. (2014) mRNP granules. Assembly, function, and connections with disease. RNA Biol. 11, 1019–1030. https://doi.org/10.4161/15476286.2014.972208
Moujaber O., Stochaj U. (2018) Cytoplasmic RNA granules in somatic maintenance. Gerontology. 64, 485–494. https://doi.org/10.1159/000488759
van Leeuwen W., Rabouille C. (2019) Cellular stress leads to the formation of membraneless stress assemblies in eukaryotic cells. Traffic. 20, 623–638. https://doi.org/10.1111/tra.12669
Burke J.M., Lester E.T., Tauber D., Parker R. (2020) RNase L promotes the formation of unique ribonucleoprotein granules distinct from stress granules. J. Biol. Chem. 295, 1426–1438. https://doi.org/10.1074/jbc.RA119.011638
Pimentel J., Boccaccio G.L. (2014) Translation and silencing in RNA granules: a tale of sand grains. Front. Mol. Neurosci. 7, 68. https://doi.org/10.3389/fnmol.2014.00068
Formicola N., Vijayakumar J., Besse F. (2019) Neuronal ribonucleoprotein granules: dynamic sensors of localized signals. Traffic. 20, 639–649. https://doi.org/10.1111/tra.12672
Bramham C.R., Wells D.G. (2007) Dendritic mRNA: transport, translation and function. Nat. Rev. Neurosci. 8, 776–789. https://doi.org/10.1038/nrn2150
Graber T.E., Hebert-Seropian S., Khoutorsky A., David A., Yewdell J.W., Lacaille J.C., Sossin W.S. (2013) Reactivation of stalled polyribosomes in synaptic plasticity. Proc. Natl. Acad. Sci. USA. 110, 16205–16210. https://doi.org/10.1073/pnas.1307747110
Tatavarty V., Ifrim M.F., Levin M., Korza G., Barbarese E., Yu J., Carson J.H. (2012) Single-molecule imaging of translational output from individual RNA granules in neurons. Mol. Biol. Cell. 23, 918–929. https://doi.org/10.1091/mbc.E11-07-0622
Batish M., van den Bogaard P., Kramer F.R., Tyagi S. (2012) Neuronal mRNAs travel singly into dendrites. Proc. Natl. Acad. Sci. USA. 109, 4645–4650. https://doi.org/10.1073/pnas.1111226109
Schisa J.A. (2012) New insights into the regulation of RNP granule assembly in oocytes. Int. Rev. Cell Mol. Biol. 295, 233–289. https://doi.org/10.1016/B978-0-12-394306-4.00013-7
Kotani T., Maehata K., Takei N. (2017) Regulation of translationally repressed mRNAs in zebrafish and mouse oocytes. Results Probl. Cell Differ. 63, 297–324. https://doi.org/10.1007/978-3-319-60855-6_13
Winata C.L., Korzh V. (2018) The translational regulation of maternal mRNAs in time and space. FEBS Lett. 592, 3007–3023. https://doi.org/10.1002/1873-3468.13183
Horie M., Kotani T. (2016) Formation of mos RNA granules in the zebrafish oocyte that differ from cyclin B1 RNA granules in distribution, density and regulation. Eur. J. Cell Biol. 95, 563–573. https://doi.org/10.1016/j.ejcb.2016.10.001
Kloc M., Dougherty M.T., Bilinski S., Chan A.P., Brey E., King M.L., Patrick C.W., Jr., Etkin L.D. (2002) Three-dimensional ultrastructural analysis of RNA distribution within germinal granules of Xenopus. Dev. Biol. 241, 79–93. https://doi.org/10.1006/dbio.2001.0488
Kloc M., Bilinski S., Chan A.P., Etkin L.D. (2001) Mitochondrial ribosomal RNA in the germinal granules in Xenopus embryos revisited. Differentiation. 67, 80–83. https://doi.org/10.1046/j.1432-0436.2001.067003080.x
Kobayashi S., Amikura R., Okada M. (1994) Localization of mitochondrial large rRNA in germinal granules and the consequent segregation of germ line. Int. J. Dev. Biol. 38, 193–199.
Merry T.L., Chan A., Woodhead J.S.T., Reynolds J.C., Kumaga H., Kim S.J., Lee C. (2020) Mitochondrial-derived peptides in energy metabolism. Am. J. Physiol. Endocrinol. Metab. 319, E659–E666. https://doi.org/10.1152/ajpendo.00249.2020
Lui J., Castelli L.M., Pizzinga M., Simpson C.E., Hoyle N.P., Bailey K.L., Campbell S.G., Ashe M.P. (2014) Granules harboring translationally active mRNAs provide a platform for P-body formation following stress. Cell Rep. 9, 944–954. https://doi.org/10.1016/j.celrep.2014.09.040
Morales-Polanco F., Bates C., Lui J., Casson J., Solari C.A., Pizzinga M., Forte G., Griffin C., Garner K.E.L., Burt H.E., Dixon H.L., Hubbard S., Portela P., Ashe M.P. (2021) Core Fermentation (CoFe) granules focus coordinated glycolytic mRNA localization and translation to fuel glucose fermentation. iScience. 24, 102069. https://doi.org/10.1016/j.isci.2021.102069
Pizzinga M., Bates C., Lui J., Forte G., Morales-Polanco F., Linney E., Knotkova B., Wilson B., Solari C.A., Berchowitz L.E., Portela P., Ashe M.P. (2019) Translation factor mRNA granules direct protein synthetic capacity to regions of polarized growth. J. Cell Biol. 218, 1564–1581. https://doi.org/10.1083/jcb.201704019
Pichon X., Bastide A., Safieddine A., Chouaib R., Samacoits A., Basyuk E., Peter M., Mueller F., Bertrand E. (2016) Visualization of single endogenous polysomes reveals the dynamics of translation in live human cells. J. Cell Biol. 214, 769–781. https://doi.org/10.1083/jcb.201605024
Nair R.R., Zabezhinsky D., Gelin-Licht R., Haas B., Dyhr M.C.A., Sperber H.S., Nusbaum C., Gerst J.E. (2020) Multiplexed mRNA assembly into ribonucleo-protein particles plays an operon-like role in the control of yeast cell physiology. bioRxiv. 2020.06.28.175851. https://doi.org/10.1101/2020.06.28.175851
Schwarz A., Beck M. (2019) The benefits of cotranslational assembly: a structural perspective. Trends Cell Biol. 29, 791–803. https://doi.org/10.1016/j.tcb.2019.07.006
Bertolini M., Fenzl K., Kats I., Wruck F., Tippmann F., Schmitt J., Auburger J.J., Tans S., Bukau B., Kramer G. (2021) Interactions between nascent proteins translated by adjacent ribosomes drive homomer assembly. Science. 371, 57–64. https://doi.org/10.1126/science.abc7151
Shiber A., Doring K., Friedrich U., Klann K., Merker D., Zedan M., Tippmann F., Kramer G., Bukau B. (2018) Cotranslational assembly of protein complexes in eukaryotes revealed by ribosome profiling. Nature. 561, 268–272. https://doi.org/10.1038/s41586-018-0462-y
Duncan C.D., Mata J. (2011) Widespread cotranslational formation of protein complexes. PLoS Genet. 7, e1002398. https://doi.org/10.1371/journal.pgen.1002398
Kassem S., Villanyi Z., Collart M.A. (2017) Not5-dependent co-translational assembly of Ada2 and Spt20 is essential for functional integrity of SAGA. Nucleic Acids Res. 45, 1186–1199. https://doi.org/10.1093/nar/gkw1059
Liu F., Jones D.K., de Lange W.J., Robertson G.A. (2016) Cotranslational association of mRNA encoding subunits of heteromeric ion channels. Proc. Natl. Acad. Sci. USA. 113, 4859–4864. https://doi.org/10.1073/pnas.1521577113
Kamenova I., Mukherjee P., Conic S., Mueller F., El-Saafin F., Bardot P., Garnier J.M., Dembele D., Capponi S., Timmers H.T.M., Vincent S.D., Tora L. (2019) Co-translational assembly of mammalian nuclear multisubunit complexes. Nat. Commun. 10, 1740. https://doi.org/10.1038/s41467-019-09749-y
Panasenko O.O., Somasekharan S.P., Villanyi Z., Zagatti M., Bezrukov F., Rashpa R., Cornut J., Iqbal J., Longis M., Carl S.H., Pena C., Panse V.G., Collart M.A. (2019) Co-translational assembly of proteasome subunits in NOT1-containing assemblysomes. Nat. Struct. Mol. Biol. 26, 110–120. https://doi.org/10.1038/s41594-018-0179-5
Moissoglu K., Stueland M., Gasparski A.N., Wang T., Jenkins L.M., Hastings M.L., Mili S. (2020) RNA localization and co-translational interactions control RAB13 GTPase function and cell migration. EMBO J. 39, e104958.https://doi.org/10.15252/embj.2020104958
Wagner S., Herrmannova A., Hronova V., Gunisova S., Sen N.D., Hannan R.D., Hinnebusch A.G., Shirokikh N.E., Preiss T., Valasek L.S. (2020) Selective translation complex profiling reveals staged initiation and co-translational sssembly of initiation factor complexes. Mol. Cell. 79(4), 546–560.e7. https://doi.org/10.1016/j.molcel.2020.06.004
Villanyi Z., Ribaud V., Kassem S., Panasenko O.O., Pahi Z., Gupta I., Steinmetz L., Boros I., Collart M.A. (2014) The Not5 subunit of the ccr4-not complex connects transcription and translation. PLoS Genet. 10, e1004569. https://doi.org/10.1371/journal.pgen.1004569
Willett M., Flint S.A., Morley S.J., Pain V.M. (2006) Compartmentalisation and localisation of the translation initiation factor (eIF) 4F complex in normally growing fibroblasts. Exp. Cell Res. 312, 2942–2953. https://doi.org/10.1016/j.yexcr.2006.05.020
Willett M., Brocard M., Davide A., Morley S.J. (2011) Translation initiation factors and active sites of protein synthesis co-localize at the leading edge of migrating fibroblasts. Biochem. J. 438, 217–227. https://doi.org/10.1042/BJ20110435
Gray N.K., Hrabalkova L., Scanlon J.P., Smith R.W. (2015) Poly(A)-binding proteins and mRNA localization: who rules the roost? Biochem. Soc. Trans. 43, 1277‒1284. https://doi.org/10.1042/BST20150171
Campbell S.G., Hoyle N.P., Ashe M.P. (2005) Dynamic cycling of eIF2 through a large eIF2B-containing cytoplasmic body: implications for translation control. J. Cell Biol. 170, 925–934. https://doi.org/10.1083/jcb.200503162
Koppers M., Cagnetta R., Shigeoka T., Wunderlich L.C., Vallejo-Ramirez P., Qiaojin Lin J., Zhao S., Jakobs M.A., Dwivedy A., Minett M.S., Bellon A., Kaminski C.F., Harris W.A., Flanagan J.G., Holt C.E. (2019) Receptor-specific interactome as a hub for rapid cue-induced selective translation in axons. eLife. 8, e48718. https://doi.org/10.7554/eLife.48718
Tcherkezian J., Brittis P.A., Thomas F., Roux P.P., Flanagan J.G. (2010) Transmembrane receptor DCC associates with protein synthesis machinery and regulates translation. Cell. 141, 632–644. https://doi.org/10.1016/j.cell.2010.04.008
Wang D.O., Kim S.M., Zhao Y., Hwang H., Miura S.K., Sossin W.S., Martin K.C. (2009) Synapse- and stimulus-specific local translation during long-term neuronal plasticity. Science. 324, 1536–1540. https://doi.org/10.1126/science.1173205
Hoyle N.P., Ashe M.P. (2008) Subcellular localization of mRNA and factors involved in translation initiation. Biochem. Soc. Trans. 36, 648–652. https://doi.org/10.1042/BST0360648
Chudinova E.M., Nadezhdina E.S. (2018) Interactions between the translation machinery and microtubules. Biochemistry (Mosc.). 83, S176–S189. https://doi.org/10.1134/S0006297918140146
Lawrence J.B., Singer R.H. (1986) Intracellular localization of messenger RNAs for cytoskeletal proteins. Cell. 45, 407–415. https://doi.org/10.1016/0092-8674(86)90326-0
Mingle L.A., Okuhama N.N., Shi J., Singer R.H., Condeelis J., Liu G. (2005) Localization of all seven messenger RNAs for the actin-polymerization nucleator Arp2/3 complex in the protrusions of fibroblasts. J. Cell Sci. 118, 2425–2433. https://doi.org/10.1242/jcs.02371
Condeelis J., Singer R.H. (2005) How and why does beta-actin mRNA target? Biol. Cell. 97, 97–110. https://doi.org/10.1042/BC20040063
Chaudhuri A., Das S., Das B. (2020) Localization elements and zip codes in the intracellular transport and localization of messenger RNAs in Saccharomyces ce-revisiae. Wiley Interdiscip. Rev. RNA. 11(4), e1591. https://doi.org/10.1002/wrna.1591
Weil T.T. (2014) mRNA localization in the Drosophila germline. RNA Biol. 11, 1010–1018. https://doi.org/10.4161/rna.36097
Lasko P. (2012) mRNA localization and translational control in Drosophila oogenesis. Cold Spring Harb. Perspect. Biol. 4(10), a012294.https://doi.org/10.1101/cshperspect.a012294
Zappulo A., van den Bruck D., Ciolli Mattioli C., Franke V., Imami K., McShane E., Moreno-Estelles M., Calviello L., Filipchyk A., Peguero-Sanchez E., Muller T., Woehler A., Birchmeier C., Merino E., Rajewsky N., Ohler U., Mazzoni E.O., Selbach M., Akalin A., Chekulaeva M. (2017) RNA localization is a key determinant of neurite-enriched proteome. Nat. Commun. 8, 583. https://doi.org/10.1038/s41467-017-00690-6
Shigeoka T., Koppers M., Wong H.H., Lin J.Q., Cagnetta R., Dwivedy A., de Freitas Nascimento J., van Tartwijk F.W., Strohl F., Cioni J.M., Schaeffer J., Carrington M., Kaminski C.F., Jung H., Harris W.A., Holt C.E. (2019) On-site ribosome remodeling by locally synthesized ribosomal proteins in axons. Cell Rep. 29, 3605–3619.e10. https://doi.org/10.1016/j.celrep.2019.11.025
Wilk R., Hu J., Blotsky D., Krause H.M. (2016) Diverse and pervasive subcellular distributions for both coding and long noncoding RNAs. Genes Dev. 30, 594–609. https://doi.org/10.1101/gad.276931.115
Lecuyer E., Yoshida H., Parthasarathy N., Alm C., Babak T., Cerovina T., Hughes T.R., Tomancak P., Krause H.M. (2007) Global analysis of mRNA loca-lization reveals a prominent role in organizing cellular architecture and function. Cell. 131, 174–187. https://doi.org/10.1016/j.cell.2007.08.003
Dermit M., Dodel M., Lee F.C.Y., Azman M.S., Schwenzer H., Jones J.L., Blagden S.P., Ule J., Mardakheh F.K. (2020) Subcellular mRNA localization regulates ribosome biogenesis in migrating cells. Dev. Cell. 55, 298–313.e10. https://doi.org/10.1016/j.devcel.2020.10.006
Kashida S., Wang D.O., Saito H., Gueroui Z. (2019) Nanoparticle-based local translation reveals mRNA as a translation-coupled scaffold with anchoring function. Proc. Natl Acad. Sci. USA. 116, 13346–13351. https://doi.org/10.1073/pnas.1900310116
Chabanon H., Mickleburgh I., Hesketh J. (2004) Zipcodes and postage stamps: mRNA localisation signals and their trans-acting binding proteins. Brief. Funct. Genomic. Proteomic. 3, 240–256. https://doi.org/10.1093/bfgp/3.3.240
Andreassi C., Riccio A. (2009) To localize or not to localize: mRNA fate is in 3'UTR ends. Trends Cell Biol. 19, 465–474. https://doi.org/10.1016/j.tcb.2009.06.001
Bullock S.L. (2011) Messengers, motors and myste-ries: sorting of eukaryotic mRNAs by cytoskeletal transport. Biochem. Soc. Trans. 39, 1161–1165. https://doi.org/10.1042/BST0391161
Marchand V., Gaspar I., Ephrussi A. (2012) An intracellular transmission control protocol: assembly and transport of ribonucleoprotein complexes. Curr. Opin. Cell Biol. 24, 202–210. https://doi.org/10.1016/j.ceb.2011.12.014
Song T., Zheng Y., Wang Y., Katz Z., Liu X., Chen S., Singer R.H., Gu W. (2015) Specific interaction of KIF11 with ZBP1 regulates the transport of β-actin mRNA and cell motility. J. Cell Sci. 128, 1001–1010. https://doi.org/10.1242/jcs.161679
Huttelmaier S., Zenklusen D., Lederer M., Dictenberg J., Lorenz M., Meng X., Bassell G.J., Condeelis J., Singer R.H. (2005) Spatial regulation of β-actin translation by Src-dependent phosphorylation of ZBP1. Nature. 438, 512‒515. https://doi.org/10.1038/nature04115
Jønson L., Vikesaa J., Krogh A., Nielsen L. K., Hansen T., Borup R., Johnsen A.H., Christiansen J., Nielsen F.C. (2007) Molecular composition of IMP1 ribonucleoprotein granules. Mol. Cell. Proteomics. 6, 798‒811. https://doi.org/10.1074/mcp.M600346-MCP200
Abouward R., Schiavo G. (2020) Walking the line: mechanisms underlying directional mRNA transport and localisation in neurons and beyond. Cell. Mol. Life Sci.https://doi.org/10.1007/s00018-020-03724-3
Dalla Costa I., Buchanan C.N., Zdradzinski M.D., Sahoo P.K., Smith T.P., Thames E., Kar A.N., Twiss J.L. (2021) The functional organization of axonal mRNA transport and translation. Nat. Rev. Neurosci. 22, 77‒91. https://doi.org/10.1038/s41583-020-00407-7
Cosker K.E., Fenstermacher S.J., Pazyra-Murphy M.F., Elliott H.L., Segal R.A. (2016) The RNA-binding protein SFPQ orchestrates an RNA regulon to promote axon viability. Nat. Neurosci. 19, 690–696. https://doi.org/10.1038/nn.4280
Kim E., Jung H. (2020) Local mRNA translation in long-term maintenance of axon health and function. Curr. Opin. Neurobiol. 63, 15–22. https://doi.org/10.1016/j.conb.2020.01.006
Baumann S., Komissarov A., Gili M., Ruprecht V., Wieser S., Maurer S.P. (2020) A reconstituted mammalian APC-kinesin complex selectively transports defined packages of axonal mRNAs. Sci. Adv. 6, eaaz1588. https://doi.org/10.1126/sciadv.aaz1588
Bergalet J., Lecuyer E. (2014) The functions and regulatory principles of mRNA intracellular trafficking. Adv. Exp. Med. Biol. 825, 57–96. https://doi.org/10.1007/978-1-4939-1221-6_2
Chin A., Lecuyer E. (2017) RNA localization: making its way to the center stage. Biochim. Biophys. Acta Gen. Subj. 1861, 2956–2970. https://doi.org/10.1016/j.bbagen.2017.06.011
Lazzaretti D., Bono F. (2017) mRNA localization in metazoans: a structural perspective. RNA Biol. 14, 1473–1484. https://doi.org/10.1080/15476286.2017.1338231
Paquin N., Menade M., Poirier G., Donato D., Drouet E., Chartrand P. (2007) Local activation of yeast ASH1 mRNA translation through phosphorylation of Khd1p by the casein kinase Yck1p. Mol. Cell. 26, 795–809. https://doi.org/10.1016/j.molcel.2007.05.016
Andreassi C., Luisier R., Crerar H., Darsinou M., Blokzijl-Franke S., Lenn T., Luscombe N.M., Cuda G., Gaspari M., Saiardi A., Riccio A. (2021) Cytoplasmic cleavage of IMPA1 3' UTR is necessary for maintaining axon integrity. Cell Rep. 34, 108778. https://doi.org/10.1016/j.celrep.2021.108778
Moissoglu K., Yasuda K., Wang T., Chrisafis G., Mili S. (2019) Translational regulation of protrusion-localized RNAs involves silencing and clustering after transport. eLife. 8, e44752. https://doi.org/10.7554/eLife.44752
Forrest K.M., Gavis E.R. (2003) Live imaging of endogenous RNA reveals a diffusion and entrapment mechanism for nanos mRNA localization in Drosophila. Curr. Biol. 13, 1159–1168.
Semotok J.L., Lipshitz H.D. (2007) Regulation and function of maternal mRNA destabilization during early Drosophila development. Differentiation. 75, 482–506. https://doi.org/10.1111/j.1432-0436.2007.00178.x
Blower M.D., Feric E., Weis K., Heald R. (2007) Genome-wide analysis demonstrates conserved localization of messenger RNAs to mitotic microtubules. J. Cell Biol. 179, 1365–1373. https://doi.org/10.1083/jcb.200705163
Alliegro M.C. (2011) The centrosome and spindle as a ribonucleoprotein complex. Chromosome Res. 19, 367–376. https://doi.org/10.1007/s10577-011-9186-7
Blower M.D., Nachury M., Heald R., Weis K. (2005) A Rae1-containing ribonucleoprotein complex is required for mitotic spindle assembly. Cell. 121, 223–234. https://doi.org/10.1016/j.cell.2005.02.016
Rosic S., Kohler F., Erhardt S. (2014) Repetitive centromeric satellite RNA is essential for kinetochore formation and cell division. J. Cell Biol. 207, 335–349. https://doi.org/10.1083/jcb.201404097
Mamon L.A., Ginanova V.R., Kliver S.F., Yakimova A.O., Atsapkina A.A., Golubkova E.V. (2017) RNA-binding proteins of the NXF (nuclear export factor) family and their connection with the cytoskeleton. Cytoskeleton. 74, 161–169. https://doi.org/10.1002/cm.21362
Ito K.K., Watanabe K., Kitagawa D. (2020) The emerging role of ncRNAs and RNA-binding proteins in mitotic apparatus formation. Noncoding RNA. 6(1), 13. https://doi.org/10.3390/ncrna6010013
Groisman I., Huang Y.S., Mendez R., Cao Q., Theurkauf W., Richter J.D. (2000) CPEB, maskin, and cyclin B1 mRNA at the mitotic apparatus: implications for local translational control of cell division. Cell. 103, 435–447.
Fingerhut J.M., Yamashita Y.M. (2020) mRNA localization mediates maturation of cytoplasmic cilia in Drosophila spermatogenesis. J. Cell Biol. 219, e202003084. https://doi.org/10.1083/jcb.202003084
Amato R., Morleo M., Giaquinto L., di Bernardo D., Franco B. (2014) A network-based approach to dissect the cilia/centrosome complex interactome. BMC Genomics. 15, 658. https://doi.org/10.1186/1471-2164-15-658
Muller H., Schmidt D., Steinbrink S., Mirgorods-kaya E., Lehmann V., Habermann K., Dreher F., Gustavsson N., Kessler T., Lehrach H., Herwig R., Gobom J., Ploubidou A., Boutros M., Lange B.M. (2010) Proteomic and functional analysis of the mitotic Drosophila centrosome. EMBO J. 29, 3344–3357. https://doi.org/10.1038/emboj.2010.210
Davies A.H., Barrett I., Pambid M.R., Hu K., Stratford A.L., Freeman S., Berquin I.M., Pelech S., Hieter P., Maxwell C., Dunn S.E. (2011) YB-1 evokes susceptibility to cancer through cytokinesis failure, mitotic dysfunction and HER2 amplification. Oncogene. 30, 3649–3660. https://doi.org/10.1038/onc.2011.82
Iaconis D., Monti M., Renda M., van Koppen A., Tammaro R., Chiaravalli M., Cozzolino F., Pignata P., Crina C., Pucci P., Boletta A., Belcastro V., Giles R.H., Surace E.M., Gallo S., Pende M., Franco B. (2017) The centrosomal OFD1 protein interacts with the translation machinery and regulates the synthesis of specific targets. Sci. Rep. 7, 1224. https://doi.org/10.1038/s41598-017-01156-x
Lambert J.D., Nagy L.M. (2002) Asymmetric inheritance of centrosomally localized mRNAs during embryonic cleavages. Nature. 420, 682–686. https://doi.org/10.1038/nature01241
Ryder P.V., Fang J., Lerit D.A. (2020) centrocortin RNA localization to centrosomes is regulated by FMRP and facilitates error-free mitosis. J. Cell Biol. 219, e202004101. https://doi.org/10.1083/jcb.202004101
Safieddine A., Coleno E., Salloum S., Imbert A., Traboulsi A.M., Kwon O.S., Lionneton F., Georget V., Robert M.C., Gostan T., Lecellier C.H., Chouaib R., Pichon X., Le Hir H., Zibara K., Mueller F., Walter T., Peter M., Bertrand E. A choreography of centrosomal mRNAs reveals a conserved localization mechanism involving active polysome transport. Nat. Commun. 12, 1352. https://doi.org/10.1038/s41467-021-21585-7
Romero-Brey I., Bartenschlager R. (2014) Membranous replication factories induced by plus-strand RNA viruses. Viruses. 6, 2826–2857. https://doi.org/10.3390/v6072826
Paul D., Madan V., Bartenschlager R. (2014) Hepatitis C virus RNA replication and assembly: living on the fat of the land. Cell Host Microbe. 16, 569–579. https://doi.org/10.1016/j.chom.2014.10.008
Reid D.W., Campos R.K., Child J.R., Zheng T., Chan K.W.K., Bradrick S.S., Vasudeva, S.G., Garcia-Blanco M.A., Nicchitta C.V. (2018) Dengue virus selectively annexes endoplasmic reticulum-associated translation machinery as a strategy for co-opting host cell protein synthesi. J. Virol. 92, e01766-17. https://doi.org/10.1128/JVI.01766-17
Ropidi M.I.M., Khazali A.S., Rashid N. N., Yusof R. (2020) Endoplasmic reticulum: a focal point of Zika virus infection. J. Biomed. Sci. 27, 27. https://doi.org/10.1186/s12929-020-0618-6
Knoops K., Kikkert M., Worm, S. H., Zevenhoven-Dobbe J.C., van der Meer Y., Koster A.J., Mommaas A.M., Snijder E.J. (2008) SARS-coronavirus replication is supported by a reticulovesicular network of modified endoplasmic reticulum. PLoS Biol. 6, e226. https://doi.org/10.1371/journal.pbio.0060226
Ulasli M., Verheije M.H., de Haan C.A., Reggiori F. (2010) Qualitative and quantitative ultrastructural analysis of the membrane rearrangements induced by coronavirus. Cell. Microbiol. 12, 844–861. https://doi.org/10.1111/j.1462-5822.2010.01437.x
Haimovich G., Olender T., Baez C., Gerst J.E. (2020) Identification and enrichment of SECReTE cis-acting RNA elements in the Coronaviridae and other (+) single-strand RNA viruses. bioRxiv. 2020.04.20.050088. https://doi.org/10.1101/2020.04.20.050088
Wu K.E., Fazal F.M., Parker K.R., Zou J., Chang H.Y. (2020) RNA-GPS predicts SARS-CoV-2 RNA residency to host mitochondria and nucleolus. Cell Syst. 11, 102–108.e3. https://doi.org/10.1016/j.cels.2020.06.008
Somasundaran M., Zapp M.L., Beattie L.K., Pang L., Byron K.S., Bassell G.J., Sullivan J.L., Singer R.H. (1994) Localization of HIV RNA in mitochondria of infected cells: potential role in cytopathogenicity. J. Cell Biol. 126, 1353–1360. https://doi.org/10.1083/jcb.126.6.1353
Sanz M.A., Castello A., Ventoso I., Berlanga J.J., Carrasco L. (2009) Dual mechanism for the translation of subgenomic mRNA from Sindbis virus in infected and uninfected cells. PLoS One. 4, e4772. https://doi.org/10.1371/journal.pone.0004772
Desmet E.A., Anguish L.J., Parker J.S. (2014) Virus-mediated compartmentalization of the host translational machinery. mBio. 5, e01463–01414. https://doi.org/10.1128/mBio.01463-14
V'Kovski P., Gerber M., Kelly J., Pfaender S., Ebert N., Braga Lagache S., Simillion C., Portmann J., Stalder H., Gaschen V., Bruggmann R., Stoffel M.H., Heller M., Dijkman R., Thiel V. (2019) Determination of host proteins composing the microenvironment of coronavirus replicase complexes by proximity-labeling. eLife. 8, e42037. https://doi.org/10.7554/eLife.42037
Kastan J.P., Dobrikova E.Y., Bryant J.D., Gromeier M. (2020) CReP mediates selective translation initiation at the endoplasmic reticulum. Sci. Adv. 6, eaba0745. https://doi.org/10.1126/sciadv.aba0745
Walsh D., Arias C., Perez C., Halladin D., Escandon M., Ueda T., Watanabe-Fukunaga R., Fukunaga R., Mohr I. (2008) Eukaryotic translation initiation factor 4F architectural alterations accompany translation initiation factor redistribution in poxvirus-infected cells. Mol. Cell. Biol. 28, 2648–2658. https://doi.org/10.1128/MCB.01631-07
Katsafanas G.C., Moss B. (2020) Specific Anchoring and local translation of poxviral ATI mRNA at cytoplasmic inclusion bodies. J. Virol. 94, e01671-19. https://doi.org/10.1128/JVI.01671-19
Ng C.S., Jogi M., Yoo J.S., Onomoto K., Koike S., Iwasaki T., Yoneyama M., Kato H., Fujita T. (2013) Encephalomyocarditis virus disrupts stress granules, the critical platform for triggering antiviral innate immune responses. J. Virol. 87, 9511–9522. https://doi.org/10.1128/JVI.03248-12
Bonenfant G., Williams N., Netzband R., Schwarz M.C., Evans M.J., Pager C.T. (2019) Zika virus subverts stress granules to promote and restrict viral gene expression. J. Virol. 93, e00520-19. https://doi.org/10.1128/JVI.00520-19
Nikolic J., Civas A., Lama Z., Lagaudriere-Gesbert C., Blondel D. (2016) Rabies virus infection induces the formation of stress granules closely connected to the viral factories. PLoS Pathog. 12, e1005942. https://doi.org/10.1371/journal.ppat.1005942
Robinson C.-A., Kleer M., Mulloy R.P., Castle E.L., Boudreau B.Q., Corcoran J.A. (2020) Human coronaviruses disassemble processing bodies. bioRxiv. 2020.11.08.372995. https://doi.org/10.1101/2020.11.08.372995
Luo L., Li Z., Zhao T., Ju X., Ma P., Jin B., Zhou Y., He S., Huang J., Xu X., Zou Y., Li P., Liang A., Liu J., Chi T., Huang X., Ding Q., Jin Z., Huang C., Zhang Y. (2021) SARS-CoV-2 nucleocapsid protein phase separates with G3BPs to disassemble stress granules and facilitate viral production. Sci. Bull. (Beijing). https://doi.org/10.1016/j.scib.2021.01.013
Savastano A., Ibanez de Opakua A., Rankovic M., Zweckstetter M. (2020) Nucleocapsid protein of SARS-CoV-2 phase separates into RNA-rich polymerase-containing condensates. Nat. Commun. 11, 6041. https://doi.org/10.1038/s41467-020-19843-1
Perdikari T.M., Murthy A.C., Ryan V.H., Watters S., Naik M.T., Fawzi N.L. (2020) SARS-CoV-2 nucleocapsid protein phase-separates with RNA and with human hnRNPs. EMBO J. 39(24), e106478. https://doi.org/10.15252/embj.2020106478
Reineke L.C., Lloyd R.E. (2013) Diversion of stress granules and P-bodies during viral infection. Virology. 436, 255–267. https://doi.org/10.1016/j.virol.2012.11.017
McCormick C., Khaperskyy D.A. (2017) Translation inhibition and stress granules in the antiviral immune response. Nat. Rev. Immunol. 17, 647–660. https://doi.org/10.1038/nri.2017.63
Doh J.H., Lutz S., Curcio M.J. (2014) Co-translational localization of an LTR-retrotransposon RNA to the endoplasmic reticulum nucleates virus-like particle assembly sites. PLoS Genet. 10, e1004219. https://doi.org/10.1371/journal.pgen.1004219
Doucet A.J., Hulme A.E., Sahinovic E., Kulpa D.A., Moldovan J.B., Kopera H.C., Athanikar J.N., Hasnaoui M., Bucheton A., Moran J.V., Gilbert N. (2010) Characterization of LINE-1 ribonucleoprotein particles. PLoS Genet. 6, e1001150. https://doi.org/10.1371/journal.pgen.1001150
Goodier J.L., Zhang L., Vetter M.R., Kazazian H.H., Jr. (2007) LINE-1 ORF1 protein localizes in stress granules with other RNA-binding proteins, including components of RNA interference RNA-induced silencing complex. Mol. Cell. Biol. 27, 6469–6483. https://doi.org/10.1128/MCB.00332-07
Mita P., Wudzinska A., Sun X., Andrade J., Nayak S., Kahler D.J., Badri S., LaCava J., Ueberheide B., Yun C.Y., Fenyo D., Boeke J.D. (2018) LINE-1 protein localization and functional dynamics during the cell cycle. eLife. 7, e30058. https://doi.org/10.7554/eLife.30058
Goodier J.L., Mandal P.K., Zhang L., Kazazian H.H., Jr. (2010) Discrete subcellular partitioning of human retrotransposon RNAs despite a common mechanism of genome insertion. Hum. Mol. Genet. 19, 1712–1725. https://doi.org/10.1093/hmg/ddq048
Potter M.D., Nicchitta C.V. (2000) Regulation of ribosome detachment from the mammalian endoplasmic reticulum membrane. J. Biol. Chem. 275, 33828–33835. https://doi.org/10.1074/jbc.M005294200
Nicchitta C.V., Lerner R.S., Stephens S.B., Dodd R.D., Pyhtila B. (2005) Pathways for compartmentalizing protein synthesis in eukaryotic cells: the template-partitioning model. Biochem. Cell Biol. 83, 687–695. https://doi.org/10.1139/o05-147
Jagannathan S., Reid D.W., Cox A.H., Nicchitta C.V. (2014) De novo translation initiation on membrane-bound ribosomes as a mechanism for localization of cytosolic protein mRNAs to the endoplasmic reticulum. RNA. 20, 1489–1498. https://doi.org/10.1261/rna.045526.114
Reid D.W., Nicchitta C.V. (2015) Diversity and selectivity in mRNA translation on the endoplasmic reticulum. Nat. Rev. Mol. Cell Biol. 16, 221–223. https://doi.org/10.1038/nrm3958
Stephens S.B., Dodd R.D., Brewer J.W., Lager P.J., Keene J.D., Nicchitta C.V. (2005) Stable ribosome binding to the endoplasmic reticulum enables compartment-specific regulation of mRNA translation. Mol. Biol. Cell. 16, 5819–5831. https://doi.org/10.1091/mbc.e05-07-0685
Lerner R.S., Nicchitta C.V. (2006) mRNA translation is compartmentalized to the endoplasmic reticulum following physiological inhibition of cap-dependent translation. RNA. 12, 775–789. https://doi.org/10.1261/rna.2318906
Reid D.W., Chen Q., Tay A.S., Shenolikar S., Nicchitta C.V. (2014) The unfolded protein response triggers selective mRNA release from the endoplasmic reticulum. Cell. 158, 1362–1374. https://doi.org/10.1016/j.cell.2014.08.012
Staudacher J.J., Naarmann-de Vries I.S., Ujvari S.J., Klinger B., Kasim M., Benko E., Ostareck-Lederer A., Ostareck D.H., Bondke Persson A., Lorenzen S., Meier J.C., Bluthgen N., Persson P.B., Henrion-Caude A., Mrowka R., Fahling M. (2015) Hypoxia-induced gene expression results from selective mRNA partitioning to the endoplasmic reticulum. Nucleic Acids Res. 43, 3219–3236. https://doi.org/10.1093/nar/gkv167
Reid D.W., Nicchitta C.V. (2015) Comment on “Principles of ER cotranslational translocation revealed by proximity-specific ribosome profiling”. Science. 348, 1217. https://doi.org/10.1126/science.aaa7257
Jan C.H., Williams C.C., Weissman J.S. (2015) Response to Comment on “Principles of ER cotranslational translocation revealed by proximity-specific ribosome profiling”. Science. 348, 1217. https://doi.org/10.1126/science.aaa8299
Unsworth H., Raguz S., Edwards H.J., Higgins C.F., Yague E. (2010) mRNA escape from stress granule sequestration is dictated by localization to the endoplasmic reticulum, FASEB J. 24, 3370–3380. https://doi.org/10.1096/fj.09-151142
Sivan G., Kedersha N., Elroy-Stein O. (2007) Ribosomal slowdown mediates translational arrest during cellular division. Mol. Cell. Biol. 27, 6639–6646. https://doi.org/10.1128/MCB.00798-07
Chartron J.W., Hunt K.C., Frydman J. (2016) Cotranslational signal-independent SRP preloading during membrane targeting. Nature. 536, 224–228. https://doi.org/10.1038/nature19309
del Alamo M., Hogan D.J., Pechmann S., Albanese V., Brown P.O., Frydman J. (2011) Defining the specifi-city of cotranslationally acting chaperones by systematic analysis of mRNAs associated with ribosome-nascent chain complexes. PLoS Biol. 9, e1001100. https://doi.org/10.1371/journal.pbio.1001100
Mauro V.P., Edelman G.M. (2007) The ribosome filter redux. Cell Cycle. 6, 2246–2251. https://doi.org/10.4161/cc.6.18.4739
Shi Z., Fujii K., Kovary K.M., Genuth N.R., Rost H.L., Teruel M.N., Barna M. (2017) Heterogeneous ribosomes preferentially translate distinct subpools of mRNAs genome-wide. Mol. Cell. 67, 71–83.e77. https://doi.org/10.1016/j.molcel.2017.05.021
Genuth N.R., Barna M. (2018) Heterogeneity and specialized functions of translation machinery: from genes to organisms. Nat. Rev. Genet. 19, 431–452. https://doi.org/10.1038/s41576-018-0008-z
Emmott E., Jovanovic M., Slavov N. (2019) Ribosome stoichiometry: from form to function. Trends Biochem. Sci. 44, 95–109. https://doi.org/10.1016/j.tibs.2018.10.009
Simsek D., Tiu G.C., Flynn R.A., Byeon G.W., Leppek K., Xu A.F., Chang H.Y., Barna M. (2017) The mammalian ribo-interactome reveals ribosome functional diversity and heterogeneity. Cell. 169, 1051–1065.e18. https://doi.org/10.1016/j.cell.2017.05.022
Komili S., Farny N.G., Roth F.P., Silver P.A. (2007) Functional specificity among ribosomal proteins regulates gene expression. Cell. 131, 557–571. https://doi.org/10.1016/j.cell.2007.08.037
Gerst J.E. (2018) Pimp my ribosome: ribosomal protein paralogs specify translational control. Trends Genet. 34, 832–845. https://doi.org/10.1016/j.tig.2018.08.004
Segev N., Gerst J.E. (2018) Specialized ribosomes and specific ribosomal protein paralogs control translation of mitochondrial proteins. J. Cell Biol. 217, 117–126. https://doi.org/10.1083/jcb.201706059
Ferretti M.B., Karbstein K. (2019) Does functional specialization of ribosomes really exist? RNA. 25, 521–538. https://doi.org/10.1261/rna.069823.118
Marc P., Margeot A., Devaux F., Blugeon C., Corral-Debrinski M., Jacq C. (2002) Genome-wide analysis of mRNAs targeted to yeast mitochondria. EMBO Rep. 3, 159–164. https://doi.org/10.1093/embo-reports/kvf025
Mercer T.R., Neph S., Dinger M.E., Crawford J., Smith M.A., Shearwood A.M., Haugen E., Bracken C.P., Rackham O., Stamatoyannopoulos J.A., Filipovska A., Mattick J.S. (2011) The human mitochondrial transcriptome. Cell. 146, 645–658. https://doi.org/10.1016/j.cell.2011.06.051
Minis A., Dahary D., Manor O., Leshkowitz D., Pilpel Y., Yaron A. (2014) Subcellular transcriptomics-dissection of the mRNA composition in the axonal compartment of sensory neurons. Dev. Neurobiol. 74, 365–381. https://doi.org/10.1002/dneu.22140
Gumy L.F., Yeo G.S., Tung Y.C., Zivraj K.H., Willis D., Coppola G., Lam B.Y., Twiss J.L., Holt C.E., Fawcett J.W. (2011) Transcriptome analysis of embryonic and adult sensory axons reveals changes in mRNA repertoire localization RNA. 17, 85–98. https://doi.org/10.1261/rna.2386111
Taylor A.M., Berchtold N.C., Perreau V.M., Tu C.H., Li Jeon N., Cotman C.W. (2009) Axonal mRNA in uninjured and regenerating cortical mammalian axons. J. Neurosci. 29, 4697–4707. https://doi.org/10.1523/JNEUROSCI.6130-08.2009
Padron A., Iwasaki S., Ingolia N.T. (2019) Proximity RNA labeling by APEX-Seq reveals the organization of translation initiation complexes and repressive RNA granules. Mol. Cell. 75, 875–887.e5. https://doi.org/10.1016/j.molcel.2019.07.030
Wang P., Tang W., Li Z., Zo, Z., Zhou Y., Li R., Xiong T., Wang J., Zou P. (2019) Mapping spatial transcriptome with light-activated proximity-dependent RNA labeling, Nat. Chem. Biol. 15, 1110–1119. https://doi.org/10.1038/s41589-019-0368-5
Benhalevy D., Anastasakis D.G., Hafner M. (2018) Proximity-CLIP provides a snapshot of protein-occupied RNA elements in subcellular compartments. Nat. Methods. 15, 1074‒1082. https://doi.org/10.1038/s41592-018-0220-y
Lee J.H., Daugharthy E.R., Scheiman J., Kalhor R., Yang J.L., Ferrante T.C., Terry R., Jeanty S.S., Li C., Amamoto R., Peters D.T., Turczyk B.M., Marblestone A.H., Invers S.A., Bernard A., Mali P., Rios X., Aach J., Church G.M. (2014) Highly multiplexed subcellular RNA sequencing in situ. Science. 343, 1360–1363. https://doi.org/10.1126/science.1250212
Wang X., Allen W.E., Wright M.A., Sylwestrak E.L., Samusik N., Vesuna S., Evans K., Liu C., Ramakrishnan C., Liu J., Nolan G.P., Bava F.A., Deisseroth K. (2018) Three-dimensional intact-tissue sequencing of single-cell transcriptional states. Science. 361(6400), eaat5691. https://doi.org/10.1126/science.aat5691
Fürth D., Hatini V., Lee J.H. (2019) In situ transcriptome accessibility sequencing (INSTA-seq). bioRxiv. 722819. https://doi.org/10.1101/722819
Chen K.H., Boettiger A.N., Moffitt J.R., Wang S., Zhuang X. (2015) RNA imaging. Spatially resolved, highly multiplexed RNA profiling in single cells. Science. 348, https://doi.org/10.1126/science.aaa6090
Eng C.L., Lawson M., Zhu Q., Dries R., Koulena N., Takei Y., Yun J., Cronin C., Karp C., Yuan G.C., Cai L. (2019) Transcriptome-scale super-resolved imaging in tissues by RNA seqFISH. Nature. 568, 235–239. https://doi.org/10.1038/s41586-019-1049-y
Xia C., Fan J., Emanuel G., Hao J., Zhuang X. (2019) Spatial transcriptome profiling by MERFISH reveals subcellular RNA compartmentalization and cell cycle-dependent gene expression Proc. Natl. Acad. Sci. USA. 116, 19490–19499. https://doi.org/10.1073/pnas.1912459116
Biswas J., Liu Y., Singer R.H., Wu B. (2019) Fluorescence imaging methods to investigate translation in single cells. Cold Spring Harb. Perspect. Biol. 11, a032722. https://doi.org/10.1101/cshperspect.a032722
Koppel I., Fainzilber M. (2018) Omics approaches for subcellular translation studies. Mol. Omics. 14, 380–388. https://doi.org/10.1039/c8mo00172c
Taliaferro J.M. (2019) Classical and emerging techniques to identify and quantify localized RNAs. Wiley Interdiscip. Rev. RNA. 10, e1542. https://doi.org/10.1002/wrna.1542
Buxbaum A.R., Haimovich G., Singer R.H. (2015) In the right place at the right time: visualizing and understanding mRNA localization. Nat. Rev. Mol. Cell Biol. 16, 95–109. https://doi.org/10.1038/nrm3918
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


