Молекулярная биология, 2021, T. 55, № 6, стр. 944-955
Разработка мультиплексной ОТ-ПЦР с иммобилизованными праймерами для идентификации возбудителей инфекционной пневмонии человека
С. А. Лапа a, *, Р. А. Мифтахов a, Е. С. Клочихина a, Ю. И. Аммур b, С. А. Благодатских c, В. Е. Шершов a, А. С. Заседателев a, А. В. Чудинов a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Научно-исследовательский институт вакцин и сывороток им. И.И. Мечникова
105064 Москва, Россия
c Научный центр прикладной микробиологии и биотехнологии
142279 Оболенск, Россия
* E-mail: lapa@biochip.ru
Поступила в редакцию 12.03.2021
После доработки 12.04.2021
Принята к публикации 13.04.2021
Аннотация
Разработан прототип системы, основанной на мультиплексной ПЦР с обратной транскрипцией (ОТ-ПЦР) в иммобилизованной фазе, для выявления возбудителей инфекционной пневмонии человека. Сконструированы праймеры для идентификации ДНК шести видов бактерий и РНК двух вирусов: гриппа А и SARS-CoV-2. Накопление сигнала удлиняющихся иммобилизованных праймеров обусловлено включением в растущую цепь флуоресцентно меченных нуклеотидов. Детекция сигнала происходит после удаления всех компонентов смеси, что существенно снижает фон и увеличивает чувствительность анализа. Применение специализированного детектора позволяет считывать сигналы удлиненных праймеров непосредственно через прозрачную покровную пленку реакционной камеры. Такое решение призвано предотвратить перекрестную контаминацию и подходит для одновременного тестирования большого количества образцов. Предлагаемая платформа способна выявлять несколько различных возбудителей пневмонии в одном образце; она имеет открытую архитектуру, что позволяет расширять спектр определяемых патогенных бактерий и вирусов.
ВВЕДЕНИЕ
Инфекционная пневмония – острый воспалительный процесс легочной ткани, вызываемый возбудителями бактериальной, вирусной и грибковой природы: стрептококками, стафилококками, вирусами гриппа, коронавирусами и другими (наиболее распространенные возбудители пневмонии подробно рассмотрены в обзорах [1–5]).
Вирусные и бактериальные пневмонии часто имеют сходную клиническую картину. Этиологию пневмоний, вызванных коронавирусами и вирусами гриппа, не всегда удается определить своевременно в условиях клинической лаборатории, что приводит к ошибкам в выборе стратегии лечения пациента. В период эпидемий ситуацию усугубляет распространение внутрибольничных бактериальных инфекций (коинфекций) в условиях массового заражения [6, 7], поэтому ускоренное дифференциальное определение возбудителя представляет важную задачу, от решения которой зависит быстрая и точная постановка диагноза и, следовательно, проведение своевременной и корректной терапии [8].
На сегодняшний день в условиях клинической лаборатории возбудителей инфекций идентифицируют с использованием культуральных методов [9], а также тест-систем, основанных на ПЦР или серологических тестах, которые в большинстве случаев ориентированы на определение одного вида возбудителя [10, 11]. Часто постановка диагноза и назначение терапии основаны только на клинической картине заболевания [12] либо дополняются данными радиологических методов [13, 14].
Мультиплексная ПЦР считается перспективным инструментом молекулярно-биологических исследований и клинической диагностики. Применение мультиплексной ПЦР для одновременного выявления нескольких возбудителей пневмонии человека в одном образце крайне важно, учитывая сложность определения этиологии заболевания классическими клиническими методами, в том числе идентификации возбудителей респираторных инфекций [15–17]. В клинических лабораториях РНК-содержащие вирусы выявляют с использованием обратной транскрипции (ОТ) [18, 19].
ОТ в мультиплексном варианте применяют для обнаружения РНК-содержащих вирусов – возбудителей респираторных инфекций [20–23]. Мультиплексный вариант ОТ-ПЦР используют для одновременного выявления РНК- и ДНК-содержащих вирусов [24]. Описано одновременное обнаружение вироидов (РНК) и эубактерий (ДНК) с помощью методики, сочетающей ОТ и ПЦР [25]. ОТ-ПЦР с последующим гибридизационным анализом на биологическом микрочипе рассмотрена в [26, 27].
Проведение мультиплексной ОТ-ПЦР на биологическом микрочипе способно повысить производительность, чувствительность и надежность множественного выявления нуклеиновых кислот бактериальных и вирусных инфекционных агентов в образце. Этот метод хорошо сочетается с масштабами тестирования, проводимого клиническими лабораториями.
В настоящей работе предложен ускоренный способ определения возбудителя в образце с применением метода мультиплексной ОТ-ПЦР в иммобилизованной фазе. Этот способ обладает высокой скоростью проведения анализа при одновременном обнаружении ряда патогенных агентов. Прототип разработанной диагностической системы устойчив к перекрестной контаминации, совместим со стандартными in situ амплификаторами, а также с детекторами флуоресцентного сигнала, разработанными для биологических микрочипов. Прототип обладает открытой архитектурой, благодаря чему возможно расширение спектра выявляемых патогенных агентов при выполнении условия совместимости многопраймерной системы.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Штаммы. В работе использовали деконтаминированные полногеномные ДНК штаммов бактерий из коллекции ГНЦ прикладной микробиологии и биотехнологии (п. Оболенск) и вирусную РНК из коллекции Института вакцин и сывороток (Москва). Работы с клиническими изолятами и живыми культурами проводили на базе указанных учреждений.
ДНК из культур выделяли с использованием CTAB-метода [28]. Суспензию бактериальной культуры готовили в 1× ТЕ-буфере. Клетки лизировали с использованием раствор лизоцима (10 мг/мл). Для разрушения белков и отделения белков от ДНК использовали протеиназу К (“Thermo Fisher”, США) и раствор CTAB/NaCl. Белки, клеточные элементы, ДНК разделяли с использованием раствора хлороформ/изоамиловый спирт в соотношении 24 : 1. ДНК отделяли от остальной части раствора с помощью изопропилового спирта и отмывали от остатков реактивов 70%-ным раствором этанола (х.ч.). Отмытую и высушенную ДНК растворяли в дистиллированной воде.
Качество и количество ДНК определяли с помощью электрофореза в 1%-ном агарозном геле и спектрофотометрически (GeneQuant Pro RNA/DNA Calculator, “Amersham Pharmacia Biotech”, Великобритания).
Получены деконтаминированные ДНК следующих штаммов: Staphylococcus aureus ATCC 25923, S. aureus ATCC 43300, Haemophilus influenzae ATCC 49247, Legionella pneumophila ATCC 33152, Pseudomonas aeruginosa 10662 NCTC ATCC 25668, Klebsiella pneumoniae 9633 NCTC ATCC 13883, Streptococcus pneumoniae ATCC 49619.
Вирус SARS-CoV-2 получен путем наработки в культуре клеток Vero (АТСС, США) вирусных частиц из клинического образца, полученного от больного COVID-19. Присутствие РНК SARS-CoV-2 в вирусном материале анализировали методом ОТ-ПЦР в реальном времени с праймерами к гену N [29]. Таксономическая принадлежность изолята к виду Severe acute respiratory syndrome-related coronavirus (SARS-CoV-2, клада GH) установлена путем секвенирования гена S (идентификационный номер GenBank MW161041.1) и полного генома (идентификационный номер GenBank MW514307.1) с последующим филогенетическим анализом. Последовательность гена S штамма Dubrovka имела 99.2%-ное сходство со штаммом Wuhan-Hu-1 (NC_045512.20). Особенность штамма Dubrovka – делеция 27 нуклеотидов в гене S (кодируют 9 аминокислотных остатков в положении 68–76 S-белка – YMSLGPMVL), которая объясняет относительно высокий уровень различий (0.8%) между этими штаммами.
Вирусы гриппа А (штамм А/Panama/2007/99 H3N2) и В (В/Leningrad/179/86) получены из Коллекции микроорганизмов III и IV групп патогенности НИИВС им И.И. Мечникова.
Вирусный материал инактивировали в лизирующем растворе (набор для выделения РНК “Магно-Сорб”, Россия), содержащем хаотропный агент – гуанидинизотиоцианат. Инактивацию вируса SARS-CoV-2 проверяли по наличию или отсутствию цитопатического эффекта на чувствительной культуре клеток Vero.
Вирусную геномную РНК выделяли из 140 мкл лизата, используя коммерческий набор QIAamp Viral RNA Mini Kit (“Qiagen”, Германия) согласно инструкции производителя. В первом случае очищенную вирусную РНК элюировали с мембраны колонок QIAamp Mini Spin 60 мкл воды, не содержащей РНКаз, и хранили при температуре –70°C до использования.
Вирусную РНК экстрагировали также с использованием CTAB-метода (в модифицированном варианте), который требуется оптимизировать, чтобы применять одновременно как для РНК-, так и для ДНК-содержащих патогенных агентов.
Праймеры. Нуклеотидные последовательности геномных мишеней выравнивали с помощью алгоритма ClustalW (www.clustal.org). Праймеры конструировали с использованием сетевого ресурса www.idtdna.com. Определяли физико-химические характеристики каждого праймера, включая тестирование на присутствие как внутри- так и межмолекулярных вторичных структур. Специфичность анализировали с помощью алгоритма BLAST (NIH, США).
Мультиплексная ПЦР в объеме. Реакционная смесь (30 мкл) содержала 1.5 ед. Taq-полимеразы (“Thermo Scientific”, США) в буфере той же фирмы, природные дезоксинуклеозидтрифосфаты (dNTP), каждый в концентрации 200 мкМ, праймеры в концентрации 5 мкМ и полногеномную бактериальную матрицу (либо смесь бактериальных ДНК). Реакцию проводили на ДНК-амплификаторе MiniCycler (“MJResearch”, США). Температурно-временной профиль ПЦР состоял из предварительной денатурации при 95°С в течение 5 мин, за которой следовали 30 циклов: 95°С (денатурация ДНК) в течение 20 с; 66°С (отжиг праймеров) в течение 30 с; 72°С (достройка праймеров) в течение 30 с и завершающая инкубация при 72°С в течение 5 мин.
Градиентную ПЦР и определение чувствительности системы с помощью ПЦР в режиме реального времени проводили на амплификаторе IQ5 (“Bio-Rad”).
Горизонтальный электрофорез для контроля продуктов ОТ, и ПЦР. Продукты ПЦР разделяли в 4%-ном агарозном геле (Agarose LE, “Helicon”, Россия), для окрашивания использовали бромистый этидий.
Иммобилизация праймеров, биологические микрочипы. Праймеры после синтеза и очистки растворяли в воде (Milli Q), концентрацию доводили до 8 мМ, смешивали с компонентами геля и наносили на чип, представляющий собой обработанное силикатное стекло либо полимерную подложку. Дальнейшую обработку подложки и процедуры изготовления чипа проводили согласно [30].
Мультиплексная ОТ-ПЦР в иммобилизованной фазе. ОТ-ПЦР проводили с использованием обратной транскриптазы MMLV и других компонентов набора “РЕВЕРТА-L” (ФГУН ЦНИИЭ Роспотребнадзора, Россия) и Hot Start Taq-полимеразы (“Thermo Scientific”) в соответствующем буфере, либо с помощью набора OneStep RT-PCR Kit (“Qiagen”). Смесь содержала природные dNTP (по 400 мкМ каждого) и праймеры – 5–10 мкМ прямого и 0.5–1.0 мкМ обратного (различались для разных пар праймеров в результате оптимизации). В качестве флуоресцентного субстрата полимеразы использовали Cy5-dUTP [31] в концентрации 8 мкМ. Смесь помещали на чип и герметизировали с помощью Frame-Seal 25 mkl (“Bio-Rad”, США). Реакции проводили на ДНК-амплификаторе для in situ ПЦР TGradient Thermocycler (“Biometra”, США). ОТ проводили в течение 30 мин при 42°C, после чего проводили ПЦР в следующих условиях: 95°C в течение 3 мин (начальная денатурация); 36 циклов по 20 с при 95°C, 30 с при 64°C и 40 с при 72°C; завершающая инкубация в течение 5 мин при 72°C.
Чувствительность ОТ-ПЦР на чипе определяли раститровкой ДНК/РНК анализируемых образцов в интервале 101–105 копий на реакционный объем (25 мкл).
Определение удлинения иммобилизованных праймеров, интерпретация результатов анализа. Считывание флуоресцентного сигнала с чипа после удлинения иммобилизованных праймеров осуществляли согласно схеме [32] с применением анализатора “Чип-детектор” (ИМБ, Россия). Интенсивность сигналов определяли с помощью программного обеспечения “ImaGeWare” ver. 3.50 (ИМБ, Россия).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Ранее мы сообщали о разработке мультиплексной ПЦР для определения видов бактерий – возбудителей пневмонии человека [17, 33]. В дальнейшем на основе сконструированных праймеров, специфичных к S. aureus и St. pneumoniae, разработали мультиплексную ПЦР на чипе с включением метки в ДНК в процессе ее удлинения. Флуоресцентная метка, ковалентно встроенная в последовательность иммобилизованного праймера в процессе ПЦР, позволяла проводить “жесткую” отмывку чипа без риска потери сигнала и, соответственно, без снижения чувствительности системы [32].
В нашей работе расширен набор специфичных праймеров. Теперь он охватывает шесть основных видов бактерий, возбудителей пневмонии, а также два РНК-содержащих вируса: вирус гриппа А и коронавирус нового типа, вызывающий COVID-19 (табл. 1), что привело к использованию ОТ. В качестве необходимых условий при конструировании праймеров учитывали специфичность и внутривидовую консервативность выбранных участков генетических мишеней. Для удобства идентификации возбудителя на стадии оптимизации многопраймерной системы “в общем объеме” конструировали пары праймеров, позволяющих получить ПЦР-продукты различной длины. При конструировании мультиплексной ПЦР руководствовались требованием совместимости праймеров, т.е. отсутствия межмолекулярных взаимодействий для всех праймеров системы, а также стандартными требованиями близких температур плавления дуплексов и отсутствия внутримолекулярных взаимодействий (шпилек). BLAST-анализ проводили, выбирая видоспецифичные консервативные участки генов-мишеней, используемых для видовой идентификации патогенных микроорганизмов и вирусов. В табл. 1 указаны последовательности, фланкирующие выбранные регионы, а также длины получаемых ПЦР-продуктов. Для создания микрочипа иммобилизовали либо обратный праймер в каждой паре, либо так называемый “вложенный” праймер, лежащий внутри амплифицируемого региона, фланкируемого прямым и обратным праймерами. Выбор определялся сравнительным тестированием праймеров при оптимизации системы. В табл. 1 приведены результирующие праймеры, полученные после оптимизации системы.
Таблица 1.
Праймеры для видоспецифичного определения бактериальных и вирусных возбудителей пневмонии человека
| Возбудитель | Праймер | Нуклеотидная последовательность | Длина, нуклеотиды |
Длина ПЦР-продукта, п.н. |
|---|---|---|---|---|
| Streptococcus pneumoniae | lytA-f1 | 5'-ATTGACGAATTGCTCTTGTCTCA-3' | 23 | 204 |
| lytA-r1 | 5'-AAGTTTACGCATGGCCTGGCTCA-3' | 23 | ||
| cpsB-f1 | 5'-TTGATGTAGATGACGGTCCCAAG-3' | 23 | 217 | |
| cpsB-r1 | 5'-TATATCTCTGCGCCATAAGCAAT-3' | 23 | ||
| Staphylococcus aureus | ebpS-f1 | 5'-ACTCGACTGAGGATAAAGCGTCT-3' | 23 | 283 |
| ebpS-r1 | 5'-CCTCCAAATATCGCTAATGCACC-3' | 23 | ||
| Haemophilus influenzae | fucK-f1 | 5'-TGCTCACTCAACGCTTAACTGGT-3' | 23 | 193 |
| fucK-r1 | 5'-TTCTGGGCTAATGGTGTACGTAA-3' | 23 | ||
| Pseudomonas aeruginosa | oprL-f1 | 5'-GCGTGCGATCACCACCTTCTACT-3' | 23 | 321 |
| oprL-r1 | 5'-TTCTTCAGCTCGACGCGACGGTT-3' | 23 | ||
| Legionella pneumophila | sidA-f1 | 5'-TTCCACTGGTGGGTGGGTTTTG-3' | 22 | 370 |
| sidA-r1 | 5'-TCATGTTGGAGTTCTATGGCACG-3' | 23 | ||
| Klebsiella pneumoniae | rmpA-f2 | 5'-GGACTACCTCTGTTTCATATTAC-3' | 23 | 373 |
| rmpA-r2 | 5'-CCCCATTTTTCAGTAGGCAT-3' | 20 | ||
| SARS-CoV-2 | E-forw | 5'-TCGTTTCGGAAGAGACAGGTACG-3' | 23 | 143 |
| E-rev | 5'-AAGACTCACGTTAACAATATTGC-3' | 23 | ||
| Вирус гриппа А | M2-forw | 5'-CACGCTCACCGTGCCCAGTGAGCG-3' | 24 | 203 |
| M2-rev | 5'-TATATGAGGCCCATGCAACTGGCA-3' | 24 |
Важное условие видоспецифичной идентификации патогенных агентов – внутривидовая консервативность выбранных мишеней. В идеале это должны быть гены, кодирующие белки, характерные только для исследуемых микроорганизмов или вирусов, либо гены, последовательности которых резко отличаются от последовательностей гомологичных генов родственных видов, непатогенных для человека. При этом предпочтительно использовать гены, кодирующие факторы патогенности. Так, большинство отобранных мишеней определяют вирулентность идентифицируемых патогенных агентов.
В табл. 1 приведены генетические мишени, использованные для идентификации патогенов. Мишени выбирали на основании доказанной возможности их применения для видовой идентификации соответствующего возбудителя.
Ген lytA St. pneumoniae кодирует один из факторов вирулентности – аутолизин, участвующий в ряде клеточных процессов [34]; ген cpsB (тирозин-специфичная протеинфосфатаза B) стрептококка участвует в регуляции биосинтеза капсульных полисахаридов [35]. Ген ebpS (эластинсвязывающий белок S) относится к группе генов S. aureus, которые кодируют белки, участвующие в связывании молекул на поверхности клеток [36]. Ген fucK, применяемый для видовой идентификации H. influenzae [37], относится к генам углеводного обмена. Ген oprL кодирует пептидогликан-ассоциированный белок, который участвует в инвагинации мембраны во время деления клеток псевдомонад, в частности P. aeruginosa. Этот ген важен для поддержания целостности мембраны [38]. Для видовой идентификации L. pneumophila и K. pneumoniae используют детерминанты вирулентности этих бактерий – SidA и RmpA, соответственно, [39, 40].
В случае РНК-содержащих вирусов: SARS-CoV-2 и вируса гриппа A использовали ген Е (кодирует белок оболочки (envelope protein) [41]) и сегмент 7 матриксного белка 2 (M2) [42] соответственно.
Строили множественные выравнивания последовательностей каждого выбранного региона из архива NIH (США) и проводили поиск консервативных участков для конструирования фланкирующих праймеров. В случае РНК-содержащих вирусов, обладающих высокой вариабельностью, использовали вырожденные нуклеотидные позиции. Теоретическую специфичность праймеров проверяли с помощью алгоритма BLAST (NIH, США).
При разработке систем с открытой архитектурой большое значение приобретает совместимость всего пула праймеров. Под совместимостью понимается отсутствие каких-либо комплементарных взаимодействий всего набора праймеров. Расширение системы приводит к необходимости проверки отсутствия такого рода взаимодействий вновь вводимых праймеров с уже оптимизированной системой. Проведение таких процедур возможно даже в ручном режиме.
На стадии оптимизации полезно проводить тестирование праймеров с помощью ПЦР в одном объеме с последующим электрофоретическим разделением продуктов реакции. Кроме того, иммобилизованные в различных ячейках праймеры лишены возможности взаимодействия между собой, что несколько облегчает задачу.
На рис. 1 представлена принципиальная схема выявления одной генетической мишени с применением иммобилизованных праймеров.
Рис. 1.
Схема анализа с применением иммобилизованных праймеров. Литерами f и r обозначены прямой и обратный фланкирующие праймеры, литерами r1 и r2 – вложенные иммобилизованные праймеры (содержат аминогруппы на 5′-конце), dU* – меченный цианиновым красителем дезоксиуридин, встраиваемый в процессе удлинения праймера.
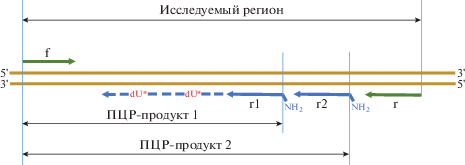
Система сконструирована так, что для каждого возбудителя на чипе присутствует несколько специфичных праймеров. Иногда иммобилизовали обратный фланкирующий праймер “r”, в ряде случаев – вложенные праймеры (на схеме “r1” и “r2”), выбираемые по результатам предварительной оптимизации (тестирования в общем объеме). И так для каждого возбудителя.
На начальном этапе процесса оптимизации мультиплексной системы в общем объеме тестировали несколько специфичных праймеров для каждого региона. Соответствующие электрофореграммы представлены на рис. 2.
Рис. 2.
Экспериментальный выбор специфичных праймеров при оптимизации системы по результатам анализа продуктов ПЦР. а: 1 – маркер длин фрагментов дцДНК DNA Ladder 50 bp (жирным полосам маркера соответствуют длины 250 и 500 п.н. дцДНК); 2 – F1 + R1 (163 п.н.); 3 – F2 + R1 (78 п.н.); 4 – F3 + R1 (180 п.н.); 5 – F1 + R2 (115 п.н.); 6 – F2 + R2 (30 п.н.); 7 – F3 + R2 (132 п.н.). б: 1 – Ladder 50 bp; 2 –F4 + R3 (144 п.н.); 3 – F5 + R3 (131 п.н.); 4 – F6 + R3 (169 п.н.); 5 – F4 + R4 (87 п.н.); 6 – F5 + R4 (74 п.н.); 7 – F6 + R4 (112 п.н.); 8 – F4 + R5 (126 п.н.); 9 – F5 + R5 (113 п.н.); 10 – F6 + R5 (151 п.н.).
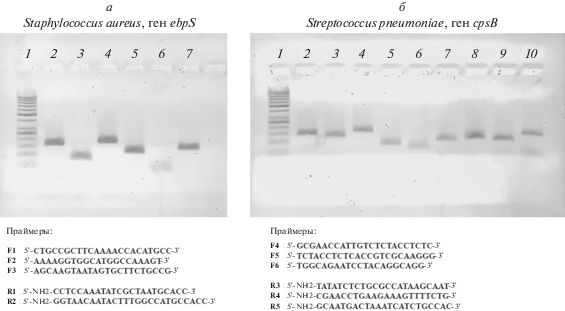
Из риc. 2 видно, что длины ПЦР-продуктов в каждой из лунок геля соответствуют теоретически ожидаемым. Однако видны четкие различия в выходе продукта, его однородности (в ряде случаев видны побочные продукты) либо эффект “primer-dimer”, что наблюдается, например, в лунке 10 (рис. 2б). Эти эффекты устранили в процессе оптимизации системы.
В случае РНК-содержащих вирусов первый этап оптимизации включал стадию ОТ. На рис. 3 показан результат проведения ОТ-ПЦР РНК SARS-CoV-2 и вируса гриппа А.
Рис. 3.
ОТ-ПЦР-анализ РНК коронавируса типа 2 и вируса гриппа А, выделенной из клинического образца. а: 1 – маркер длин фрагментов дцДНК GeneRuler 50 bp (жирным полосам маркера соответствуют длины 250 и 500 п.н. дцДНК); 2 – F7 + R6, количество исходной РНК 103 копий на реакционную пробирку; 3 – F7 + R6, количество исходной РНК 104 копий на реакционную пробирку; 4 – отрицательный контроль (видны праймеры). Использованы праймеры E-f и E-r, теоретическая длина продукта 143 п.н. соответствует наблюдаемой на электрофореграмме. б – Раздельное проведение ОТ и ПЦР при оптимизации (тестирование с помощью ОТ как прямого, так и обратного праймеров). 1 – Маркер длин продуктов ДНК GeneRuler 50 bp; 2 – ОТ F8, ПЦР F8 + R7; 3 – ОТ R7, ПЦР F8 + R7; 4 – ОТ F8, ПЦР F9 + R7; 5 – ОТ R7, ПЦР F9 + R8. Праймер R8 – экспериментальный, модифицированный (с введением вырожденных позиций) на основании предлагаемого WHO [42], остальные праймеры сконструированы специально для создания предлагаемой тест-системы.
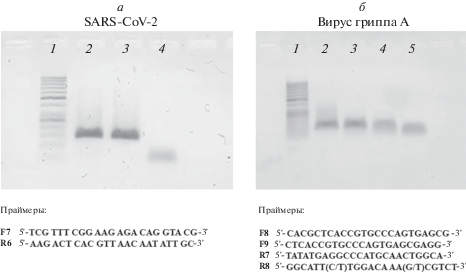
После первой стадии следовала оптимизация системы с применением всех выбранных пар праймеров, в том числе, с применением градиентной ПЦР и варьированием концентрации компонентов системы, включая праймеры.
Определяли чувствительность и специфичность сконструированной тест-системы, в том числе, при попарном использовании анализируемых мишеней на одном биологическом микрочипе. По результатам экспериментов заменяли некоторые иммобилизованные праймеры, а также свободные фланкирующие праймеры в реакционной смеси.
Система показала более высокие отношения сигнал–фон по сравнению с использованием меченого праймера в растворе и определением удлинения праймера гибридизацией после проведения ПЦР. Преимущества предлагаемого подхода и схема анализа приведены в [32].
Температурно-временной профиль реакции оптимизировали с применением градиентной ПЦР; оптимизировали концентрации каждого реагента смеси, за исключением компонентов реакционного буфера, который использовали в соответствии с рекомендациями производителя. Специфичность праймеров к мишеням определяли для каждой пары праймеров и соответствующей им мишени, затем для той же пары праймеров – с другими мишенями (для исключения ложноспецифического взаимодействия), и лишь после этого тестировали мультипраймерную систему. В эксперименте использовали полноразмерные препараты бактериальной геномной ДНК, а также выделенную и очищенную РНК SARS-CoV-2 и вируса гриппа А.
После оптимизации многопраймерной системы в режиме ПЦР проводили тестирование и оптимизацию в режиме ОТ-ПЦР, что потребовало замены некоторых праймеров.
Праймеры для иммобилизации подвергали той же многостадийной процедуре тестирования, после чего осуществляли иммобилизацию и проверку их работы в различных режимах, подобно описанному для фланкирующих праймеров. Тестирование проводили на деконтаминированных образцах выделенной геномной ДНК/РНК.
Результирующие последовательности иммобилизованных праймеров, выбранные после оптимизации системы, приведены в табл. 2.
Таблица 2.
Иммобилизованные праймеры для ОТ-ПЦР на биочипе
| Возбудитель | Праймер | Нуклеотидная последовательность праймера | Длина, нуклеотиды |
Длина ПЦР-продукта, п.н.* |
|---|---|---|---|---|
| Streptococcus pneumoniae | R3 | 5'-NH2-TATATCTCTGCGCCATAAGCAAT-3' | 23 | – |
| R5 | 5'-NH2-GCAATGACTAAATCATCTGCCAC-3' | 23 | – | |
| Staphylococcus aureus | R1 | 5'-NH2-CCTCCAAATATCGCTAATGCACC-3' | 23 | |
| R2 | 5'-NH2-GGTAACAATACTTTGGCCATGCCACC-3' | 26 | – | |
| Haemophilus influenzae | R8 | 5'-NH2-TTCACCTGCATAACGCATAGGAG-3' | 23 | – |
| R9 | 5'-NH2-CATAACGCATAGGAGGGAAATGG-3 | 23 | – | |
| Pseudomonas aeruginosa | R10 | 5'-NH2-GTAGCGACCGGACGCTCTTTAС-3' | 22 | – |
| R11 | 5'-NH2-GCTCTTTACCATAGGAAACCAG-3' | 22 | – | |
| Legionella pneumophila | R6 | 5'-NH2-TCATTATATTTATCATTGTTTGGCTC-3' | 26 | – |
| R7 | 5'-NH2-ACGTTTCGCTACAAGATCTATAA-3' | 23 | – | |
| Klebsiella pneumoniae | R13 | 5'-NH2-GGCGTCAGATACAGGACGGCT-3' | 21 | – |
| R14 | 5'- NH2-CCACTCCACCGGCAGTGCTCAC-3' | 22 | – | |
| SARS-CoV-2 | E-f-n | 5'-NH2-CTTGCTTTCGTGGTATTCTTGCT-3' | 23 | – |
| E-r-n | 5'-NH2-CACACAATCGAAGCGCAGTAAGG-3' | 23 | – | |
| Вирус гриппа А | INF-F1 | 5'-NH2-CTCACCGTGCCCAGTGAGCGAGG-3' | 23 | – |
* Зависит от используемого прямого праймера (см. рис. 1).
Схема специализированного биологического микрочипа и примеры анализа образцов представлены на рис. 4.
Рис. 4.
Специализированный биологический микрочип для видового определения возбудителей пневмонии методом мультиплексной ОТ-ПЦР. а – Схема: M – флуоресцентный маркер для автоматического наложения сетки при программном обеспечении расчета интенсивности сигнала ячеек; Gel – ячейка пустого геля; Sta – Staphylococcus aureus (здесь и во всех последующих случаях цифры “1” и “2” обозначают использование двух различных праймеров внутри последовательности, фланкируемой краевыми праймерами, литера “а” обозначает использование праймера в концентрации в 2 раза меньшей, чем в ячейке чипа без литеры – это необходимо для повышения надежности автоматического анализа); Str – Streptococcus pneumoniae, Leg – Legionella pneumophila, Hae – Haemophilus influenzae, Pse – Pseudomonas aeruginosa, Kle – Klebsiella pneumoniae, Cov – SARS-CoV-2, Inf – вирус гриппа A; Con1 – внутренний контроль обратной транскрипции (резервный; в настоящее время не используется), Con2 – внутренний контроль ПЦР. б–е – результаты определения вида (примеры) соответственно: St. aureus, S. pneumoniae, H. influenzae, SARS-CoV-2, вирус гриппа A. Для удобства приведены фото с инвертированными сигналами (негативы).

В системе предусмотрены два внутренних контроля – для стадии ОТ и стадии ПЦР. Чувствительность системы определяли раститровкой каждого из анализируемых образцов ДНК/РНК. В зависимости от мишени чувствительность системы с иммобилизованными праймерами составила от 102 до 104 копий нуклеиновой кислоты на образец (только для имеющихся в коллекции образцов; статистических данных в настоящее время недостаточно). Увеличению чувствительности способствовало включение метки в растущую цепь иммобилизованного праймера. Такое решение позволяет осуществить ковалентную пришивку метки и удалить все компоненты смеси, которые увеличивают фоновый шум и за счет этого уменьшают чувствительность системы. Предлагаемый подход более подробно описан в недавней работе [32]; он представляет собой усовершенствование классической методологии применения ферментативных реакций на гидрогелевых чипах, где используется меченый праймер, находящийся в растворе.
Система может быть расширена для анализа более широкого спектра возбудителей путем добавления иммобилизованных праймеров при условии контроля их совместимости с разработанной мультиплексной ОТ-ПЦР.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Первая работа, описывающая проведение ПЦР на гидрогелевых биочипах, была опубликована в 2000 г. [43] научной группой ИМБ РАН (Россия). Вскоре ПЦР применили для выявления рифампицин-устойчивых штаммов возбудителя туберкулеза [44, 45], модификации метода ПЦР на чипе посвящена методическая работа [46].
Предложены оригинальные подходы к проведению ферментативных реакций в иммобилизованной фазе для создания компактных специализированных биочипов [47, 48], которые получили развитие в технике изотермической амплификации, “бридж-ПЦР” и, в конечном счете, в секвенировании нового поколения (NGS). Технические сложности проведения ПЦР на чипе по сравнению с гибридизационным анализом привели к тому, что в настоящее время “золотым стандартом” диагностических систем, основанных на технологии специализированных гидрогелевых биочипов, стало применение гибридизационного анализа [49–53]. На основе этого подхода создан ряд сертифицированных систем для клинической диагностики (“ТБ-Биочип”, “СИ-Биочип” и другие).
В большинстве методов, основанных на ОТ, реакцию проводят в жидкой фазе (без использования биочипов) и лишь последующая гибридизация выполняется на чипе [26, 27].
Классическая схема детекции сигнала после проведения ферментативной реакции на биочипе предусматривает гибридизацию флуоресцентно меченного праймера на удлиненный иммобилизованный праймер. Эта схема делает невозможным проведение реакции в режиме реального времени, она весьма чувствительна к условиям детекции, так как представляет собой квазиравновесную систему; при этом повышен риск “смывания” гибридизованного праймера. Другой недостаток – повышенный фон и сравнительно низкое соотношение сигналов совершенных и несовершенных дуплексов из-за необходимости использования флуоресцентно меченных праймеров.
Одно из усовершенствований предлагаемого в настоящей работе подхода состоит во включении меченых нуклеотидов в растущую иммобилизованную цепь ДНК, что позволяет полностью удалить все компоненты реакционной смеси и тем самым резко снизить фоновый сигнал. При сочетании этого подхода с технологией “микрофлюидики” можно заменять растворы после каждого цикла амплификации и проводить реакции в режиме реального времени. Сочетание ОТ с ПЦР позволяет анализировать как РНК-, так и ДНК-содержащие патогенные агенты (вирусы и бактерии).
Различия в химической природе нуклеиновых кислот затрудняют разработку универсального метода экстракции (в частности, для получения максимального выхода выделяемой нуклеиновой кислоты (ДНК или РНК) используют различные соотношения хлороформ–изоамиловый спирт; в случае РНК необходимо использовать буферные растворы, обработанные диэтилпирокарбонатом и т.п.). Для создания универсального метода обнаружения РНК- и ДНК-содержащих патогенных агентов необходима унификация метода выделения нуклеиновых кислот. Мы использовали СТАВ-метод как один из наиболее бережных методов выделения суммарных ДНК и РНК из образцов, содержащих как бактериальные и эукариотические клетки, так и вирусные частицы [54, 55].
Масштабирование предлагаемой системы затрудняют различия в природе тестируемых образцов, присутствие ингибиторов, загрязнений, микроорганизмов-спутников. Присутствие в экстракте тотальной ДНК и РНК может усложнить анализ и требует более тщательного подхода к конструированию видоспецифичных праймеров.
Дальнейшее усовершенствование прототипа предлагаемой системы предполагает проведение статистического анализа, а также определение чувствительности и специфичности анализа с применением широкого спектра образцов.
ВЫВОДЫ
Сконструированы высокоспецифичные праймеры для идентификации шести видов бактерий и двух вирусов, вызывающих пневмонию. Разработана и оптимизирована ОТ-ПЦР в иммобилизованной фазе и биологический микрочип для одновременного выявления нескольких возбудителей в исследуемом образце. Накопление флуоресцентного сигнала осуществляется встраиванием меченых нуклеотидов в растущую цепь ДНК. Разработанный прототип обладает открытой архитектурой, благодаря чему может быть расширен спектр анализируемых патогенных агентов. Система ориентирована на применение в клинических лабораториях, где нужно параллельно тестировать большое количество образцов и важно быстро получить ответ для выработки своевременной и адекватной стратегии лечения пациента.
Исследование поддержано грантом Российского научного фонда (№ 20-14-00287).
Настоящая статья не содержит описания каких-либо исследований с участием людей или животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Henig O., Kaye K.S. (2017) Bacterial pneumonia in older adults. Infect. Dis. Clin. North. Am. 31, 689–713.
Haq I.J., Battersby A.C., Eastham K., McKean M. (2017) Community acquired pneumonia in children. BMJ (Clinical Res. ed.). 356, j686.
Ruuskanen O., Lahti E., Jennings L.C., Murdoch D.R. (2011) Viral pneumonia. Lancet. 9, 1264–1275.
Galván J.M., Rajas O., Aspa J. (2015) Review of non-bacterial infections in respiratory medicine: viral pneumonia. Arch. Bronconeumol. 51, 590–597.
Dandachi D., Rodriguez-Barradas M.C. (2018) Viral pneumonia: etiologies and treatment. J. Invest. Med. 66, 957–965.
Chen X., Liao B., Cheng L., Peng X., Xu X., Li Y., Hu T., Li J. Zhou X., Ren B. (2020). The microbial coinfection in COVID-19. Appl. Microbiol. Biotechnol. 104, 7777–7785.
Prasso J.E., Deng J.C. (2017) Postviral complications: bacterial pneumonia. Clin. Chest. Med. 38, 127–138.
Harris M., Clark J., Coote N., Fletcher P., Harnden A., McKean M., Thomson A. (2011) British thoracic society standards of care committee. British thoracic society guidelines for the management of community acquired pneumonia in children: update 2011. Thorax. 66(Suppl. 2), ii1–ii23.
Budayanti N.S., Suryawan K., Iswari I.S., Sukrama D.M. (2019) The quality of sputum specimens as a predictor of isolated bacteria from patients with lower respiratory tract infections at a tertiary referral hospital, Denpasar, Bali-Indonesia. Front. Med. (Lausanne). 6, 64.
Lee N., Rainer T.H., Ip M., Zee B., Ng M.H., Antonio G.E., Chan E., Lui G., Cockram C.S., Sung J.J., Hui D.S. (2006) Role of laboratory variables in differentiating SARS-coronavirus from other causes of community-acquired pneumonia within the first 72 h of hospitalization. Eur. J. Clin. Microbiol. Infect. Dis. 25, 765–772.
Dorigo-Zetsma J.W., Zaat S.A., Wertheim-van Dillen P.M., Spanjaard L., Rijntjes J., van Waveren G., Jensen J.S., Angulo A.F., Dankert J. (1999) Comparison of PCR, culture, and serological tests for diagnosis of Mycoplasma pneumoniae respiratory tract infection in children. J. Clin. Microbiol. 37, 14–17.
Cunha B.A. (2006) The atypical pneumonias: clinical diagnosis and importance. Clin. Microbiol. Infect. 12(Suppl.), 3, 12–24.
Azoulay E., Russell L., Van de Louw A., Metaxa V., Bauer P., Povoa P., Montero J.G., Loeches I.M., Mehta S., Puxty K., Schellongowski P., Rello J., Mokart D., Lemiale V., Mirouse A. (2020) Diagnosis of severe respiratory infections in immunocompromised patients. Intensive Care Med. 46, 298–314.
Mabie M., Wunderink R.G. (2003) Use and limitations of clinical and radiologic diagnosis of pneumonia. Semin. Respir. Infect. 18, 72–79.
Aydemir Y., Aydemir Ö., Pekcan S., Özdemir M. (2015) Value of multiplex PCR to determine the bacterial and viral aetiology of pneumonia in school-age children. Paediatr. Int. Child Health. 37, 29–34.
Wagner K., Springer B., Imkamp F., Opota O., Greub G., Keller P.M. (2018) Detection of respiratory bacterial pathogens causing atypical pneumonia by multiplex Lightmix® RT-PCR. Int. J. Med. Microbiol. 308, 317–323.
Лапа С.А., Клочихина Е.С., Мифтахов Р.А., Золотов А.М., Заседателев А.С., Чудинов А.В. (2020) Мультиплексная ПЦР для выявления бактериальных возбудителей инфекционной пневмонии. Биоорган. химия. 46, 550–552.
You H.-L., Chang S.-J., Yu H.-R., Li C.-C. Chen, C.-H., Liao W.-T. (2017) Simultaneous detection of respiratory syncytial virus and human metapneumovirus by one-step multiplex real-time RT-PCR in patients with respiratory symptoms. BMC Pediatrics, 17, 89.
van Kasteren P.B., van der Veer B., van den Brink S., Wijsman L., de Jonge J., van den Brandt A., Molenkamp R., Reusken C.B.E.M., Meijer A. (2020) Comparison of seven commercial RT-PCR diagnostic kits for COVID-19. J. Clin. Virol. 128, 104412.
Malhotra B., Swamy M.A., Reddy P.V.J., Kumar N., Tiwari J.K. (2016) Evaluation of custom multiplex real time RT-PCR in comparison to fast track diagnostics respiratory 21 pathogens kit for detection of multiple respiratory viruses. Virol. J. 13, 91.
Zhang H., Wang Y., Porter E., Lu N., Li Y., Yuan F., Bai J. (2019) Development of a multiplex real-time RT-PCR assay for simultaneous detection and differentiation of influenza A, B, C and D viruses. Diagn. Microbiol. Infect. Disease. 95, 59–66.
Chung H.Y., Jian M.J., Chang C.K., Lin J.C., Yeh K.M., Chen C.W., Chiu S.K., Wang Y.H., Liao S.J., Li S.Y., Hsieh S.S., Tsai S.H., Perng C.L., Yang J.R., Liu M.T., Chang F.Y., Shang H.S. (2021) Novel dual multiplex real-time RT-PCR assays for the rapid detection of SARS-CoV-2, influenza A/B, and respiratory syncytial virus using the BD MAX open system. Emerg. Microbes. Infect. 10, 161–166.
Zhou B., Deng Y.-M., Barnes J.R., Sessions O.M., Chou T.-W., Wilson M., Wentworth D. (2017) Multiplex reverse transcription-PCR for simultaneous surveillance of influenza A and B viruses. J. Clin. Microbiol. 55, 3492–3501.
Abarshi M.M., Mohammed I.U., Jeremiah S.C., Legg J.P., Kumar P.L., Hillocks R.J., Maruthi M.N. (2012) Multiplex RT-PCR assays for the simultaneous detection of both RNA and DNA viruses infecting cassava and the common occurrence of mixed infections by two cassava brown streak viruses in East Africa. J. Virol. Methods. 179, 176–184.
Malandraki I., Varveri C., Vassilakos N. (2018) One-step multiplex quantitative RT-PCR for the simultaneous detection of viroids and phytoplasmas. Methods Mol. Biol. 1857, 151–157.
Lung O., Fisher M., Beeston A., Hughes K.B., Clavijo A., Goolia, M., Deregt D. (2011) Multiplex RT-PCR detection and microarray typing of vesicular disease viruses. J. Virol. Methods. 175, 236–245.
Scuderi G., Catara A.F., Licciardello G. (2019) Genotyping citrus tristeza virus isolates by sequential multiplex RT-PCR and microarray hybridization in a Lab-on-Chip device. Methods Mol. Biol. 2015, 127–142.
Wilson K. (2001) Preparation of genomic DNA from bacteria. Curr. Protoc. Molec. Biol. 56, 2.4.1–2.4.5.
Chan J.F., Yip C.C., To K.K., Tang T.H., Wong S.C., Leung K.H., Fung A.Y., Ng A.C., Zou Z., Tsoi H.W., Choi G.K., Tam A.R., Cheng V.C., Chan K.H., Tsang O.T., Yuen K.Y. (2020) Improved molecular diagnosis of COVID-19 by the novel, highly sensitive and specific COVID-19-RdRp/Hel real-time reverse transcription-PCR assay validated in vitro and with clinical specimens. J. Clin. Microbiol. 58(5), e00310–e00320.
Rubina A.Yu., Pan’kov S.V., Dementieva E.I., Pen’kov D.N., Butygin A.V., Vasiliskov V.A., Chudi-nov A.V., Mikheikin A.L., Mikhailovich V.M., Mipzabekov A.D. (2004) Hydrogel drop microchips with immobilized DNA: properties and methods for large-scale production. Anal. Biochem. 325, 92–106.
Shershov V.E., Lapa S.A., Kuznetsova V.E., Spitsyn M.A., Guseinov T.O., Polyakov S.A., Stomahin A.A., Zasedatelev A.S., Chudinov A.V. (2017) Comparative study of novel fluorescent cyanine nucleotides: hybridization analysis of labeled PCR products using a biochip. J. Fluoresc. 27, 2001–2016.
Лапа С.А., Клочихина Е.С., Мифтахов Р.А., Заседателев А.С., Чудинов А.В. (2021) Мультиплексная ПЦР на чипе с прямой детекцией удлинения иммобилизованного праймера. Биоорган. химия.https://doi.org/10.31857/S0132342321050298
Lapa S.A., Klochikhina E.S., Miftakhov R.A., Zasedatelev A.S., Chudinov A.V. (2021) Development of multi-primer PCR system with an open architecture for rapid detection of infectious pneumonia causative agents. AIP Conf. Proc. In Press.
Mellroth P., Daniels R., Eberhardt A., Rönnlund D., Blom H., Widengren J., Normark S., Henriques-Normark B. (2012) LytA, major autolysin of Streptococcus pneumoniae, requires access to nascent peptidoglycan. J. Biol. Chem. 287, 11018–11029.
Morona J.K., Morona R., Miller D.C., Paton J.C. (2002) Streptococcus pneumoniae capsule biosynthesis protein CpsB is a novel manganese-dependent phosphotyrosine-protein phosphatase. J. Bacteriol. 184, 577–583.
Downer R., Roche F., Park P.W., Mecham R.P., Foster T.J. (2002) The elastin-binding protein of Staphylococcus aureus (EbpS) is expressed at the cell surface as an integral membrane protein and not as a cell wall-associated protein J. Biol. Chem. 4, 243–250.
Abdeldaim G.M., Stralin K., Olcén P., Blomberg J., Mölling P., Herrmann B. (2013) Quantitative fucK gene polymerase chain reaction on sputum and nasopharyngeal secretions to detect Haemophilus influenzae pneumonia. Diagn. Microbiol. Infect. Dis. 76, 141–146.
Llamas M.A., Rodríguez-Herva J.J., Hancock R.E., Bitter W., Tommassen J., Ramos J.L. (2003) Role of Pseudomonas putida tol-oprL gene products in uptake of solutes through the cytoplasmic membrane. J. Bacteriol. 185, 4707–4716.
Gilmour M.W., Bernard K., Tracz D.M., Olson A.B., Corbett C.R., Burdz T., Ng B., Wiebe D., Broukhan-ski G., Boleszczuk P., Tang P., Jamieson F., Van Domselaar G., Plummer F.A., Berry J.D. (2007) Molecular typing of a Legionella pneumophila outbreak in Ontario, Canada. J. Med. Microbiol. 56, 336–341.
Li G., Sun S., Zhao Z.Y., Sun Y. (2019) The pathogenicity of rmpA or aerobactin-positive Klebsiella pneumoniae in infected mice. J. Int. Med. Res. 47, 4344–4352.
Ishige T., Murata S., Taniguchi T., Miyabe A., Kitamura K., Kawasaki K., Nishimura M., Igari H., Matsushita K. (2020) Highly sensitive detection of SARS-CoV-2 RNA by multiplex rRT-PCR for molecular diagnosis of COVID-19 by clinical laboratories. Clin. Chim. Acta. 507, 139–142.
Inui K., Nguyen T., Tseng H.J., Tsai C.M., Tsai Y.L., Chung S., Padungtod P., Zhu H., Guan Y., Kalpra-vidh W., Claes F. (2019) A field-deployable insulated isothermal RT-PCR assay for identification of influenza A (H7N9) shows good performance in the laboratory. Influenza Other Respir. Viruses. 13, 610–617.
Strizhkov B.N., Drobyshev A.L., Mikhailovich V.M., Mirzabekov A.D. (2000) PCR amplification on a microarray of gel-immobilized oligonucleotides: detection of bacterial toxin- and drug-resistant genes and their mutations. BioTechniques. 29, 844–857.
Михайлович В.М., Лапа С.А., Грядунов Д.А., Стрижков Б.Н., Соболев А.Ю., Скотникова О.И., Иртуганова О.А., Мороз А.М., Литвинов В.И., Шипина Л.К., Владимирский М.А., Черноусова Л.Н., Ерохин В.В., Мирзабеков А.Д. (2001) Выявление устойчивых к рифампицину штаммов микобактерий туберкулеза методом гибридизации и полимеразной цепной реакции на специализированном туберкулезном микрочипе. Бюлл. эксп. биол. мед. 131, 94–98.
Mikhailovich V., Lapa S., Gryadunov D., Sobolev A., Strizhkov B., Chernyh N., Skotnikova O., Irtuganova O., Moroz A., Litvinov V., Vladimirskii M., Perelman M., Chernousova L., Erokhin V., Zasedatelev A., Mirzabekov A. (2001) Identification of rifampin-resistant Mycobacterium tuberculosis strains by hybridization, PCR, and ligase detection reaction on oligonucleotide microchips. J. Clin. Microbiol. 39, 2531–2540.
Tillib S.V., Strizhkov B.N., Mirzabekov A.D. (2001) Integration of multiple PCR amplifications and DNA mutation analyses by using oligonucleotide microchip. Anal. Biochem. 292, 155–160.
Westin L., Xu X., Miller C., Wang L., Edman C.F., Nerenberg M. (2000) Anchored multiplex amplification on a microelectronic chip array. Nat. Biotechnol. 18, 199–204.
Pemov A., Modi H., Chandler D.P., Bavykin S. (2005) DNA analysis with multiplex microarray-enhanced PCR. Nucl. Acids Res. 33, e11.
Lapa S., Mikheev M., Shchelkunov S., Mikhailovich V., Sobolev A., Blinov V., Babkin I., Guskov A., Sokunova E., Zasedatelev A., Sandakhchiev L., Mirzabekov A. (2002) Species-level identification of orthopoxviruses with an oligonucleotide microchip. J. Clin. Microbiol. 40, 753–757.
Михеев М.В., Лапа С.А., Щелкунов С.Н., Чикова А.К., Михайлович В.М., Соболев А.Ю., Бабкин И.В., Грядунов Д.А., Булавкина М.А., Гуськов А.А., Сокунова Е.Б., Кочнева Г.В., Блинов В.М., Сандахчиев Л.С., Заседателев А.С., Мирзабеков А.Д. (2003) Видовая идентификация ортопоксвирусов на олигонуклеотидных микрочипах. Вопр. вирусол. 48, 4–9.
Bespyatykh J.A., Zimenkov D.V., Shitikov E.A., Kulagina E.V., Lapa S.A., Gryadunov D.A., Ilina E.N., Govorun V.M. (2014) Spoligotyping of Mycobacterium tuberculosis complex isolates using hydrogel oligonucleotide microarrays. Infect. Genet. Evol. 26, 41–46.
Рудинский Н.И., Михайлович В.М., Донников М.С., Лапа С.А., Суханова А.Л., Казеннова Е.В., Бобков А.Ф., Заседателев А.С., Покровский В.В., Мирзабеков А.Д. (2004) Разработка биологических микрочипов для выявления мутаций устойчивости ВИЧ-1 к ингибиторам протеазы и результаты их применения. Вопр. вирусол. 49, 10–15.
Gryadunov D., Mikhailovich V., Lapa S., Roudinskii N., Donnikov M., Pan’kov S., Markova O., Kuz’min A., Chernousova L., Skotnikova O., Moroz A., Zasedatelev A., Mirzabekov A. (2005) Evaluation of hybridisation on oligonucleotide microarrays for analysis of drug-resistant Mycobacterium tuberculosis. Clin. Microbiol. Infect. 11, 531–539.
Inglis P.W., Pappas M.C.R., Resende L.V., Grattapaglia D. (2018) Fast and inexpensive protocols for consistent extraction of high quality DNA and RNA from challenging plant and fungal samples for high-throughput SNP genotyping and sequencing applications. PLoS One. 13, e0206085.
Chiquito-Almanza E., Acosta-Gallegos J.A., García-Álvarez N.C., Garrido-Ramírez E.R., Montero-Tavera V., Guevara-Olvera L., Anaya-López J.L. (2017) Simultaneous detection of both RNA and DNA viruses infecting dry bean and occurrence of mixed infections by BGYMV, BCMV and BCMNV in the Central-West Region of Mexico. Viruses. 9, 63.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


