Молекулярная биология, 2021, T. 55, № 6, стр. 956-964
Риск рассеянного склероза: анализ взаимодействий между вариантами ядерного и митохондриального геномов
М. С. Козин a, b, *, И. С. Киселёв a, b, Н. М. Баулина a, b, Г. В. Павлова a, b, А. Н. Бойко a, О. Г. Кулакова a, О. О. Фаворова a
a Российский национальный исследовательский медицинский университет
им. Н.И. Пирогова Министерства здравоохранения России
117997 Москва, Россия
b Научно-технологический университет “Сириус”
354340 Сочи, Россия
* E-mail: kozinmax1992@gmail.com
Поступила в редакцию 15.02.2021
После доработки 22.04.2021
Принята к публикации 07.05.2021
Аннотация
В последнее время появляется все больше данных о том, что взаимодействие митохондриального и ядерного геномов в значительной степени определяет риск развития различных нейродегенеративных заболеваний. Однако роль мито-ядерных взаимодействий в развитии рассеянного склероза – тяжелого хронического нейродегенеративного заболевания полигенной природы – мало изучена. Нами проведен анализ ассоциации с рассеянным склерозом всех возможных двухкомпонентных мито-ядерных сочетаний, включающих по одному из семи полиморфных вариантов ядерного генома, локализованных в области генов UCP2, KIF1B и в локусе PVT1 (гены MYC, PVT1 и MIR1208), и по одному из 10 полиморфизмов митохондриального генома, а также отдельных генетических вариантов, входящих в состав этих сочетаний. Исследование выполнено на выборке из 507 больных рассеянным склерозом и 321 здорового индивида контрольной группы (все русские по этнической принадлежности). Выявлено два ассоциированных с рассеянным склерозом сочетания, включающих ядерный и митохондриальный компоненты. Сочетание UCP2(rs660339)*A + MT-ATP6(rs193303045)*G характеризовалось значениями p = 0.015; ОШ = 1.39 [95% ДИ 1.05–1.87], сочетание PVT1(rs2114358)*G + + MT-ND1(rs1599988)*C – значениями p = 0.012, ОШ = 1.77 [95% ДИ 1.10–2.84]. Из отдельных компонентов сочетаний только аллель rs660339*A ядерного гена UCP2, кодирующего разобщающий белок 2 семейства митохондриальных переносчиков анионов, был самостоятельно ассоциирован с рассеянным склерозом (p = 0.028; ОШ = 1.36 [95% ДИ 1.01–1.84]). Проведенное исследование расширяет существующие представления о влиянии мито-ядерных взаимодействий и вариативности ядерных генов, продукты которых функционируют в митохондриях, на риск развития РС.
ВВЕДЕНИЕ
Рассеянный склероз (РС) – хроническое заболевание центральной нервной системы (ЦНС), для которого характерны процессы аутоиммунного воспаления, демиелинизации и нейродегенерации, приводящие к повреждению ЦНС и к прогрессирующей неврологической дисфункции [1]. По данным ВОЗ в мире насчитывается около 2.5 млн больных РС.
РС это комплексное полигенное заболевание, которое развивается у генетически предрасположенных лиц при воздействии неблагоприятных факторов внешней среды. В основе полигенного наследования РС лежит существование множества независимых и/или взаимодействующих полиморфных вариантов генов, каждый из которых лишь в незначительной степени влияет на риск развития заболевания [2]. В настоящее время методом широкогеномного анализа ассоциаций (genome-wide association studies, GWAS) выявлено более 200 независимых ядерных локусов, ассоциированных с развитием РС [3]. Два таких исследования выполнены для митохондриального генома [4, 5], однако не обнаружено ни одного варианта мтДНК, ассоциированного с РС с необходимым уровнем значимости. Cовокупная вариабельность всех найденных локусов генома может объяснить только до 48% наблюдаемой наследуемости РС [3]. Это несоответствие получило название “недостающей” наследуемости (missing heritability) [6].
Одним из возможных объяснений феномена “недостающей” наследуемости может быть существование нелинейных (эпистатических) взаимодействий между компонентами генома, при которых эффект носительства генетического варианта одного гена зависит от носительства варианта другого, неаллельного, гена (генов), поскольку такие взаимодействия не выявляются при анализе ассоциации каждого из этих генов с заболеванием (по отдельности, one by one), в том числе при GWAS. Установлена вовлеченность эпистатических взаимодействий между генами ядерного и митохондриального геномов (мито-ядерного взаимодействия) в формировании патологического фенотипа при классических митохондриальных заболеваниях, а также при сахарном диабете типа 2, болезнях Паркинсона и Альцгеймера, шизофрении [7–9].
Хотя достоверно установлена важность дисфункции митохондрий в развитии нейродегенерации при РС [10] и показана связь вариабельности мтДНК с риском РС в различных популяциях (см. обзор [11]), мито-ядерные взаимодействия как фактор риска РС стали объектом изучения только в самое последнее время. Ранее мы выявили два ассоциированных с РС эпистатических сочетания, включающих один и тот же ядерный компонент ‒ однонуклеотидный полиморфизм (SNP) rs4410871 гена длинной некодирующей РНК (днРНК) PVT1 ‒ и SNP rs1599988 (m.4216) или rs28359178 (m.13708) мтДНК [12].
В настоящей работе мы продолжили поиск мито-ядерных взаимодействий, влияющих на риск РС, включив в исследование новые варианты ядерных генов из локуса PVT1 ‒ дополнительные SNP гена PVT1, SNP генов MYC и MIR1208, а также варианты ядерных генов за пределами этого локуса ‒ генов UCP2 и KIF1B, продукты которых влияют на функционирование митохондрий [13, 14]. Анализировали ассоциацию с развитием РС семи полиморфных вариантов перечисленных ядерных генов поодиночке и в составе сочетаний с 10 полиморфизмами мтДНК у этнических русских. Эти полиморфизмы расположены в области восьми генов, пять из которых кодируют митохондриальные белки ‒ компоненты электронно-транспортной цепи (ЭТЦ) митохондрий (MT-ND1, MT-ND2, MT-ND3, MT-ND5 и COX1), остальные гены ‒ субъединицу 6 АТР-синтазы (ATP6), 16S рРНК (MT-RNR2) и лейцин-специфичную тРНК (MT-TL2). Описание этих SNP представлено в табл. 1.
Таблица 1.
Характеристика включенных в исследование полиморфных участков ядерного и митохондриального геномов
| Rs ID | Локус | Ген | Продукт гена* |
| Полиморфизмы ядерного генома | |||
| rs4645948 | PVT1 (8q24.21) | MYC | Протоонкогенный белок Myc |
| rs4733789 | PVT1 (8q24.21) | PVT1 | Длинная некодирующая РНК |
| rs7841504 | PVT1 (8q24.21) | PVT1 | Длинная некодирующая РНК |
| rs2114358 | PVT1 (8q24.21) | PVT1 | Длинная некодирующая РНК |
| rs2648841 | PVT1 (8q24.21) | MIR1208 | микроРНК |
| rs10492972 | 1p36.22 | KIF1B | Транспортный белок, член семейства кинезинов 1B |
| rs660339 | 11q13.4 | UCP2 | Разобщающий белок 2 |
| Полиморфизмы митохондриального генома | |||
| rs3928305 | m.1719 G > A | MT-RNR2 | 16S рРНК |
| rs1599988 | m.4216 T > C | MT-ND1 | Субъединица 1 NADH-дегидрогеназы (комплекс I ЭТЦ) |
| rs28357975 | m.4580 G > A | MT-ND2 | Субъединица 2 NADH-дегидрогеназы (комплекс I ЭТЦ) |
| rs28357980 | m.4917 A > G | MT-ND2 | Субъединица 2 NADH-дегидрогеназы (комплекс I ЭТЦ) |
| rs2015062 | m.7028 C > T | COX1 | Субъединица 1 цитохром-c-оксидазы (комплекс IV ЭТЦ) |
| rs193303045 | m.9055 G > A | ATP6 | Субъединица 6 АТР-синтазы |
| rs2853826 | m.10398 A > G | MT-ND3 | Субъединица 3 NADH-дегидрогеназы (комплекс I ЭТЦ) |
| rs2853498 | m.12308 A > G | MT-TL2 | Лейцин-специфическая тРНК |
| rs3899498 | m.13368 G > A | MT-ND5 | Субъединица 5 NADH-дегидрогеназы (комплекс I ЭТЦ) |
| rs28359178 | m.13708 G > A | MT-ND5 | Субъединица 5 NADH-дегидрогеназы (комплекс I ЭТЦ) |
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Характеристика индивидов, вошедших в исследование. Группа больных РС состояла из 507 индивидов (353 женщины, 154 мужчины), которым диагноз “достоверный РС” был поставлен согласно критериям МакДональда в редакции от 2010 года [15]. Средний возраст индивидов при дебюте заболевания составлял 27.5 ± 9.2 лет, на момент исследования – 39.0 ± 10.6 лет. В контрольную группу вошел 321 здоровый индивид (210 женщин, 111 мужчин) без признаков неврологических заболеваний. Средний возраст в контрольной группе на момент забора крови ‒ 39.6 ± ± 13.5 лет. Все включенные в исследование индивиды были этническими русскими (согласно данным анкетирования русскими были все члены их семей в двух поколениях) и проживали в европейской части России.
Выделение ДНК и генотипирование. ДНК выделяли из цельной крови с использованием коммерческих наборов QIAamp DNA Blood Mini Kit (“Qiagen”, Германия). Генотипирование ядерных генов MYC, PVT1, MIR1208, UCP2 и KIF1B проводили методом полимеразной цепной реакции (ПЦР) в реальном времени на детектирующем амплификаторе StepOnePlus (“Thermo Fisher Scientific”, США); для rs660339, rs4645948, rs2114358 и rs10492972 использовали коммерческие наборы TaqMan Pre-designed SNP Genotyping Assay Kit (“Thermo Fisher Scientific”); для rs2648841, rs4733789 и rs7841504 ‒ коммерческие наборы компании OOO “ДНК-Синтез” (Россия). Полиморфизмы мтДНК были генотипированы ранее [12] методом ПЦР с последующим анализом полиморфизма длин рестрикционных фрагментов (ПЦР–ПДРФ). Полиморфизм PVT1 rs4410871 также типирован ранее [16].
Статистический анализ. Анализ отклонения наблюдаемых частот генотипов исследованных полиморфных вариантов ядерного генома от равновесия Харди–Вайнберга, а также анализ структуры неравновесия по сцеплению (LD) между парами SNP в локусе PVT1 с помощью коэффициента D', предложенного Левонтином, и коэффициента корреляции r2 Пирсона проводили с использованием программного обеспечения Haploview версии 4.2 [17]. При анализе ассоциации аллелей и генотипов SNP ядерного генома с РС использовали точный критерий Фишера; ассоциацию считали статистически значимой, если значение p было менее 0.05, а 95% доверительный интервал (ДИ) для отношения шансов (OШ) не пересекал 1. Поиск ассоциированных с РС сочетаний, включающих аллели/генотипы полиморфных участков яДНК и мтДНК, проводили с помощью программного обеспечения APSampler (http://apsampler.sourceforge.net), использующего метод Монте-Карло марковскими цепями и Байесовскую непараметрическую статистику [18]. Валидацию результатов проводили при помощи стандартных статистических подходов – оценки значения p в точном критерии Фишера, ОШ и его 95% ДИ. Сочетания считали ассоциированными с РС, если отдельные его компоненты характеризовались значением p, большим, чем значение p сочетания. Возможное нелинейное взаимодействие (эпистаз) между аллелями в биаллельных сочетаниях оценивали с использованием подхода, предложенного в [19]. В его основе лежит оценка характера взаимодействия между аллелями (или генотипами) двух локусов при их совместном носительстве с помощью двух ранее описанных статистических критериев: по значению pFLINT в точном трехфакторном тесте, подобном точному критерию Фишера (the exact three-way Fisher-like interaction numeric test, FLINT) [20], и исходя из значений фактора синергии (synergy factor, SF) и его 95% ДИ [21]. Значения pFLINT, SF и его 95% ДИ оценивали с помощью программных средств, входящих в программу APSampler. Взаимодействие между аллелями в сочетании считали эпистатическим при величине pFLINT менее 0.05, а значение 95% ДИ для SF не пересекало 1; если эти условия не соблюдались, взаимодействие считали аддитивным.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Равновесие Харди–Вайнберга соблюдалось в контрольной группе (p > 0.05) для всех генотипированных полиморфных участков яДНК, а также для исследованного ранее rs4410871 гена PVT1.
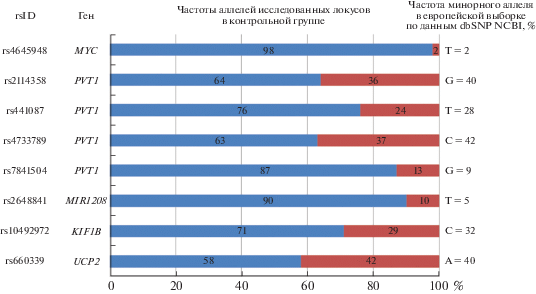
На рис. 1 представлены наблюдавшиеся нами частоты аллелей, включенных в исследование SNP ядерного генома, в контрольной группе в сравнении с частотами их минорных аллелей (MAF) в выборке представителей европейских популяций из базы данных NCBI SNP. Абсолютные отличия частот аллелей всех исследованных полиморфных участков, наблюдаемые у этнических русских, от данных, представленных в базе NCBI, составляют менее 5%.
Рис. 1.
Частоты аллелей исследованных SNP ядерного генома в контрольной группе этнических русских в сравнении с частотами минорных аллелей (MAF) в выборке представителей европейских популяций из базы данных NCBI SNP.
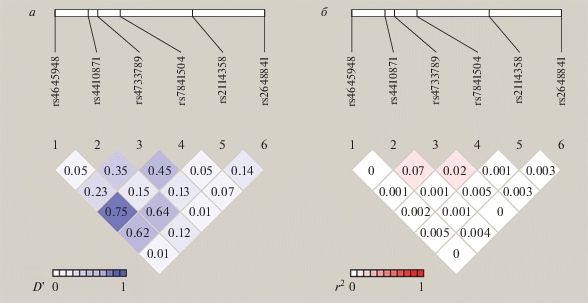
В контрольной группе этнических русских мы проанализировали неравновесное сцепление между всеми SNP локуса PVT1, типированными в настоящем исследовании (rs2648841, rs4733789, rs7841504, rs2114358, rs4645948), а также rs4410871 из этого локуса, генотипированного ранее [16]. Структура неравновесного сцепления SNP представлена на рис. 2. Значение D' для отдельных пар полиморфизмов превышало 0.6, однако значение r2 не превышало 0.1 ни для одной из пар полиморфизмов, что свидетельствует об отсутствии сцепления.
Рис. 2.
Анализ структуры неравновесия по сцеплению (LD) полиморфных вариантов локуса PVT1 в контрольной группе этнических русских с помощью коэффициентов D' (а) и r2 (б).
Проведен анализ ассоциации с РС полиморфных вариантов ядерного генома. Среди семи исследованных полиморфизмов только rs660339 гена UCP2 был значимо ассоциирован с РС. При этом значимое повышение риска РС характерно для носительства аллеля A (p = 0.028; ОШ = 1.36 [95% ДИ 1.01‒1.84]) (табл. 2) и для носительства гетерозиготного генотипа AG (p = 0.011; ОШ = 1.40 [95% ДИ 1.06‒1.85]). Ассоциации всех остальных исследованных полиморфизмов с РС не были статистически значимыми.
Таблица 2.
Ассоциированные с РС биаллельные сочетания, включающие митохондриальный и ядерный компоненты, анализ ассоциации этих компонентов с РС
| Биаллельное сочетание | Носители, n (%) | Значение p | ОШ [95% ДИ] | |
|---|---|---|---|---|
| больные РС (N = 507) |
индивиды
контрольной группы (N = 321) |
|||
| Ассоциированные с РС биаллельные сочетания, включающие ядерный и митохондриальный компоненты | ||||
| PVT1(rs2114358)*G + + m.4216(rs1599988)*C |
66 (13.0) | 25 (7.8) | 0.012 | 1.77 [1.10–2.84] |
| UCP2(rs660339)*A + + m.9055(rs193303045)*G | 339 (66.8) | 190 (59.2) | 0.015 | 1.39 [1.05–1.87] |
| Компоненты биаллельных сочетаний | ||||
| UCP2(rs660339)*A | 365 (72.0) | 210 (65.4) | 0.028 | 1.36 [1.01–1.84] |
| m.9055(rs193303045)*G | 472 (93.0) | 290 (90.3) | 0.10 | 1.42 [0.86–2.38] |
| m.4216(rs1599988)*C | 103 (20.3) | 57 (17.8) | 0.21 | 1.18 [0.83–1.70] |
| PVT1(rs2114358)*G | 305 (60.2) | 186 (58.0) | 0.29 | 1.10 [0.82–1.45] |
Поиск ассоциированных с РС биаллельных сочетаний, включающих полиморфные варианты ядерного и митохондриального геномов, проводили с использованием мультилокусного анализа при помощи APSampler. Ассоциированные с РС сочетания содержали аллель ядерного гена PVT1(rs2114358)*G или UCP2(rs660339)*A, а в качестве второго компонента в состав этих сочетаний входили аллели мтДНК m.4216(rs1599988)*C и m.9055(rs193303045)*G соответственно (табл. 2). Только носительство аллеля UCP2(rs660339)*A, ядерного компонента второго сочетания, было значимо ассоциировано с РС, хотя и с меньшим уровнем значимости, чем у всего сочетания. Ассоциации с РС аллелей PVT1(rs2114358)*G, m.9055(rs193303045)*G и m.4216(rs1599988)*C поодиночке не обнаружено.
Еще два сочетания, MYC(rs4645948)*C + + m.4216(rs1599988)*C и MIR1208(rs2648841)*C + + mt7028(rs2015062)*G, имели значения p меньше 0.05, однако доверительный интервал отношения шансов для этих сочетаний пересекал 1. Эти сочетания не были включены в дальнейшее рассмотрение.
При помощи метода, предложенного в [19], проанализировали природу взаимодействия между компонентами выявленных сочетаний, ассоциированных с РС (табл. 3). Обнаружили аддитивный вклад носительства компонентов ядерного и митохондриального геномов в составе сочетания UCP2(rs660339)*A + m.9055(rs193303045)*G. В сочетании PVT1(rs2114358)*G + m.4216(rs1599988)*C взаимодействие, согласно значениям фактора SF и его 95% ДИ, носит эпистатический характер, однако величина pFLINT составляет 0.054, т.е. превышает выбранный нами уровень <0.05.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
С целью изучения сочетанного влияния ядерного и митохондриального геномов на риск развития РС нами проведен анализ ассоциации с этим заболеванием семи SNP пяти ядерных генов в составе сочетаний с 10 SNP мтДНК. Выявлены два сочетания аллелей ядерной и митохондриальной ДНК ‒ PVT1(rs2114358)*G + m.4216(rs1599988)*C и UCP2(rs660339)*A + m.9055(rs193303045)*G, значимо ассоциированных с РС, тогда как их компоненты или не были ассоциированы с заболеванием поодиночке, или ассоциированы с меньшим уровнем значимости.
Ранее [12] мы обнаружили два ассоциированных с РС сочетания (оба эпистатические), включающих в качестве ядерного компонента SNP rs4410871 гена РНК PVT1, а в качестве митохондриальных компонентов SNP генов первой и пятой субъединиц NADH-дегидрогеназы (первый комплекс ЭТЦ).
Ген PVT1 расположен в локусе 8q24, который иногда называют “генной пустыней” [22], поскольку в нем почти отсутствуют белоккодирующие гены, за исключением гена MYC, продукт которого участвует в прохождении клеточного цикла, апоптозе и метаболизме [23]. Первый интрон гена PVT1 содержит ген микроРНК MIR1204; другие интроны содержат гены MIR1205, MIR1206 и MIR1207, а также псевдогены RNU1-106P, RNU4-25P и еще один ген днРНК TMEM75. Помимо этого, в локусе PVT1 расположен ген MIR1208. Положение исследованных SNP в локусе представлено на рис. 3.
Рис. 3.
Схематическое изображение строения локуса PVT1 на хромосоме 8 (8q24.21). Синим цветом обозначены гены днРНК – PVT1 и TMEM75, зеленым – остальные гены локуса. Показана экзон-интронная структура генов PVT1, MYC и CASC11. Красным обозначены SNP в гене PVT1, входящие в состав выявленных нами сочетаний с митохондриальными генами: rs2114358 (данные настоящего исследования) и rs4410871 (данные [12]). Остальные исследованные нами SNP обозначены зеленым шрифтом.
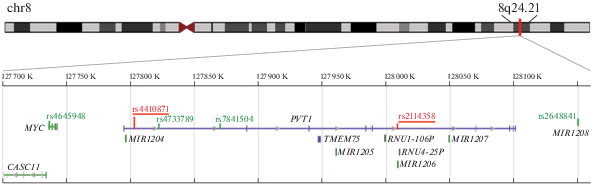
С целью более детального анализа вовлеченности участков локуса PVT1 в развитие РС в исследование включили пять не изученных нами ранее SNP локуса PVT1, потенциально способных влиять на функционирование генов локуса. Полиморфные варианты rs4733789 и rs7841504 расположены в первом интроне гена PVT1. Выбор этих SNP обусловлен данными о присутствии внутри первого интрона гена PVT1 энхансера 822E, способного связываться как с промотором самого PVT1, так и с промотором гена MYC [24]. По данным базы The Genotype-Tissue Expression (GTEx) project, полиморфизм rs4733789 влияет на экспрессию обоих этих генов. Вариант rs4645948 включен в исследование исходя из данных о его влиянии на уровень экспрессии гена MYC. Так, согласно [25], носители генотипов rs4645948*СT и rs4645948*TT характеризуются более высоким уровнем экспрессии MYC, чем носители генотипа СС. На культурах клеток HONE1 и HNE1С назофарингеальной карциномы с использованием репортерного гена люциферазы показано, что носительство аллеля rs4645948*T приводит к значительно более высокой транскрипционной активности MYC по сравнению с носительством аллеля C. Включенный в наше исследование SNP rs2114358 находится в интроне 5 гена PVT1, в котором локализуется ген MIR1206, и, как показано in silico, влияет на структуру зрелой miR-1206 [26]. Наконец, полиморфизм rs2648841, расположенный в локусе PVT1 в области pre-miR-1208, выбран для исследования исходя из его потенциального влияния на эффективность сплайсинга miR-1208.
Нам не удалось показать ассоциацию с РС ни одного из исследованных в настоящей работе SNP локуса PVT1, однако выявлено ассоциированное с РС биаллельное сочетание rs2114358 гена PVT1 с митохондриальным компонентом (rs1599988 гена MT-ND1). Это сочетание можно отнести к эпистатическим согласно одному из используемых нами критериев ‒ значению фактора SF [21], однако значение pFLINT второго применяемого нами критерия ‒ точного трехфакторного теста [20], равное 0.054, превышает выбранный нами уровень значимости <0.05.
Полученные результаты можно расширить данными еще для одного полиморфизма в локусе PVT1: в нашей предыдущей работе [12] выявлено ассоциированное с РС мито-ядерное сочетание с эпистатическим характером взаимодействия, которое включало митохондриальный SNP m.13708 и SNP гена PVT1 rs4410871 в качестве ядерного компонента. Таким образом, ни один из шести исследованных нами SNP в локусе PVT1 не ассоциирован с РС самостоятельно, но два из них, rs4410871 и rs2114358, входят в состав ассоциированных с РС мито-ядерных сочетаний. Полученные нами данные позволяют предположить, что днРНК PVT1 вовлечена в регуляцию функционирования митохондрий. Это предположение находит подтверждение в работе [27], в которой показано, что PVT1 участвует в модуляции митохондриальной сети, а подавление экспрессии гена Pvt1 в культуре мышечных клеток мыши приводит к усилению экспрессии генов, связанных с митохондриями.
Помимо полиморфных участков в локусе PVT1 на хромосоме 8, в настоящее исследование включены полиморфизмы ядерных генов UCP2 (хромосома 11) и KIF1B (хромосома 11). В нашей работе rs660339 гена UCP2 оказался ассоциированным с РС у этнических русских. Ассоциация rs660339 гена UCP2 с РС показана ранее в испанской популяции [28]. Продукт гена UCP2 – белок семейства митохондриальных переносчиков анионов (mitochondrial anion carrier proteins, MACP), участвует в разобщении процесса окислительного фосфорилирования, в результате чего вместо синтеза АТР происходит диссипация энергии в виде тепла. Белок UCP2 осуществляет перенос анионов из матрикса митохондрий в межмебранное пространство и перенос протонов в противоположную сторону. Известно, что белок UCP2 влияет на количество образующихся в митохондриях свободных радикалов, которые относятся к повреждающим факторам и играют существенную роль в патогенезе РС.
Этот полиморфизм вошел также в состав ассоциированного с РС мито-ядерного сочетания, вторым компонентом которого выступал аллель G rs193303045 гена MT-ATP6, кодирующего субъединицу 6 митохондриальной АТР-синтазы. Показано, что взаимодействие между компонентами сочетания имеет аддитивный характер, другими словами, при совместном носительстве UCP2(rs660339)*A и MT-ATP6(rs193303045)*G эффекты каждого из этих аллелей складываются.
Продукт гена KIF1B – моторный белок, отвечающий за антероградный транспорт митохондрий и синаптических везикул в аксонах нейронов. Данные по его ассоциации с РС носят противоречивый характер. Ассоциация аллеля C полиморфизма rs10492972 с РС [29] показана методом GWAS на полногеномном уровне значимости (p = 2.5 × × 10–10), однако участникам International Multiple Sclerosis Genetics Consortium (IMSGC) не удалось воспроизвести этот результат [30]. В недавнем исследовании, выполненном на русской популяции жителей Пермского края, показано влияние аллеля Т полиморфизма rs10492972 на выраженность клинических проявлений по шкалам EDSS и SCRIPPS [31]. В нашем исследовании этот полиморфизм не был ассоциирован с риском РС.
В целом, наше исследование указывает на влияние ядерных генов, функционирующих в митохондриях, и мито-ядерных генетических взаимодействий на риск развития РС. Разработка подходов, облегчающих оценку совокупных эффектов вариантов митохондриальных и ядерных ДНК на риск РС, будет иметь решающее значение для выявления “недостающей” наследуемости, поскольку на сегодняшний день ясно, что анализ эффектов каждого отдельного гена на риск полигенного заболевания не обеспечивает достаточной прогностической силы. Однако настоящее исследование выполнено на относительно небольшом количестве пациентов; полученные результаты не проходят поправку на множественные сравнения и могут быть ложноположительными. Несомненно, полученные результаты нуждаются в воспроизведении на независимой выборке.
Работа подготовлена в рамках Государственного задания АААА-А19-119042590026-5 и при финансовой поддержке Российского фонда фундаментальных исследований в рамках научного проекта №19-315-51003.
Все процедуры, использованные в данной работе, соответствуют этическим стандартам институционального комитета по исследовательской этике и Хельсинкской декларации 1964 года и ее последующим изменениям или сопоставимым нормам этики. От всех участников получено информированное письменное согласие на участие в экспериментах.
Авторы сообщают об отсутствии потенциального конфликта интересов.
Список литературы
Karussis D. (2014) The diagnosis of multiple sclerosis and the various related demyelinating syndromes: a critical review. J. Autoimmun. 48–49, 134–142.
Baranzini S.E., Oksenberg J.R. (2017) The genetics of multiple sclerosis: from 0 to 200 in 50 years. Trends Genet. 33(12), 960–970.
International Multiple Sclerosis Genetics Consortium. (2019) Multiple sclerosis genomic map implicates peripheral immune cells and microglia in susceptibility. Science. 365(6460), eaav7188.
Tranah G.J., Santaniello A., Caillier S.J., D’Alfonso S., Boneschi F.M., Hauser S.L., Oksenberg J.R. (2015) Mitochondrial DNA sequence variation in multiple sclerosis. Neurology. 85(4), 325–330.
Hudson G., Gomez-Duran A., Wilson I.J., Chinnery P.F. (2014) Recent mitochondrial DNA mutations increase the risk of developing common late-onset human diseases. PLoS Genet. 10(5), e1004369.
Maher B. (2008) Personal genomes: The case of the missing heritability. Nature. 456(7218), 18–21.
Morrow E.H., Camus M.F. (2017) Mitonuclear epistasis and mitochondrial disease. Mitochondrion. 35, 119–122.
Andrews S.J., Fulton-Howard B., Patterson C., McFall G.P., Gross A., Michaelis E.K., Goate A., Swerdlow R.H., Pa J., Alzheimer’s disease neuroimaging initiative. (2020) Mitonuclear interactions influence Alzheimer’s disease risk. Neurobiol. Aging. 87, 138.e7–138.e14.
Schulmann A., Ryu E., Goncalves V., Rollins B., Christiansen M., Frye M.A., Biernacka J., Vawter M.P. (2019) Novel complex interactions between mitochondrial and nuclear DNA in schizophrenia and bipolar disorder. Mol. Neuropsychiatry. 5(1), 13–27.
Campbell G., Mahad D.J. (2018) Mitochondrial dysfunction and axon degeneration in progressive multiple sclerosis. FEBS Lett. 592(7), 1113–1121.
Козин М.С., Кулакова О.Г., Фаворова О.О. (2018) Участие митохондрий в развитии нейродегенерации при рассеянном склерозе. Биохимия. 83(7), 1002–1021.
Kozin M., Kulakova O., Kiselev I., Baulina N., Boyko A., Favorova O. (2020) Mitonuclear interactions influence multiple sclerosis risk. Gene. 758, 144962.
Pareyson D., Saveri P., Sagnelli A., Piscosquito G. (2015) Mitochondrial dynamics and inherited peripheral nerve diseases. Neurosci. Lett. 596, 66–77.
Hass D.T., Barnstable C.J. (2021) Uncoupling proteins in the mitochondrial defense against oxidative stress. Prog. Retin. Eye. Res. 8, 100941.
Polman C.H., Reingold S.C., Banwell B., Clanet M., Cohen J.A., Filippi M., Fujihara K., Havrdova E., Hutchinson M., Kappos L., Lublin F.D., Montalban X., O’Connor P., Sandberg-Wollheim M., Thompson A.J., Waubant E., Weinshenker B., Wolinsky J.S. (2011) Diagnostic criteria for multiple sclerosis: 2010 revisions to the McDonald criteria. Ann. Neurol. 69(2), 292–302.
Kiselev I., Bashinskaya V., Baulina N., Kozin M., Popova E., Boyko A., Favorova O., Kulakova O. (2019) Genetic differences between primary progressive and relapsing-remitting multiple sclerosis: The impact of immune-related genes variability. Mult. Scler. Relat. Disord. 29, 130–136.
Barrett J.C., Fry B., Maller J., Daly M.J. (2005) Haploview: analysis and visualization of LD and haplotype maps. Bioinformatics. 21(2), 263–265.
Favorov A.V., Andreewski T.V., Sudomoina M.A., Favorova O.O., Parmigiani G., Ochs M.F. (2005) A Markov chain Monte Carlo technique for identification of combinations of allelic variants underlying complex diseases in humans. Genetics. 171, 2113–2121.
Barsova R.M., Lvovs D., Titov B.V., Matveeva N.A., Shakhnovich R.M., Sukhinina T.S., Kukava N.G., Ruda M.Y., Karamova I.M., Nasibullin T.R., Mustafina O.E., Osmak G.J., Tsareva E.Y., Kulakova O.G., Favorov A.V., Favorova O.O. (2015) Variants of the coagulation and inflammation genes are replicably associated with myocardial infarction and epistatically interact in Russians. PLoS One. 10, 1–16.
White D.R., Pesner R., Reitz K.P. (1983) An exact significance test for three-way interaction effects. Cross-Cultural Res. 18, 103–122.
Cortina-Borja M., Smith A.D., Combarros O., Lehmann D.J. (2009) The synergy factor: a statistic to measure interactions in complex diseases. BMC Res. Notes. 2, 1–7.
Huppi K., Pitt J.J., Wahlberg B.M., Caplen N.J. (2012) The 8q24 gene desert: an oasis of non-coding transcriptional activity. Front. Genet. 3, 1–11.
Stine Z.E., Walton Z.E., Altman B.J., Hsieh A.L., Dang C.V. (2015) MYC, metabolism, and cancer. Cancer Discov. 5(10), 1024–1039.
Cho S.W., Xu J., Sun R., Mumbach M.R., Carter A.C., Chen Y.G., Yost K.E., Kim J., He J., Nevins S.A., Chin S.F., Caldas C., Liu S.J., Horlbeck M.A., Lim D.A., Weissman J.S., Curtis C., Chang H.Y. (2018) Promoter of lncRNA gene PVT1 is a tumor suppressor DNA boundary element. Cell. 173(6), 1398–1412.e22.
Guo Z., Wang Y., Zhao Y., Guo Z., Wang Y., Zhao Y., Jin Y., An L., Xu H., Liu Z., Chen X., Zhou H., Wang H., Zhang W. (2019) A functional 5'-UTR polymorphism of MYC contributes to nasopharyngeal carcinoma susceptibility and chemoradiotherapy induced toxicities. J. Cancer. 10(1), 147–155.
Martin-Guerrero I., Gutierrez-Camino A., Lopez-Lopez E., Bilbao-Aldaiturriaga N., Pombar-Gomez M., Ardanaz M., Garcia-Orad A. (2015) Genetic variants in miRNA processing genes and pre-miRNAs are associated with the risk of chronic lymphocytic leukemia. PLoS One. 10(3), e0118905.
Alessio E., Buson L., Chemello F., Peggion C., Grespi F., Martini P., Massimino M.L., Pacchioni B., Millino C., Romualdi C., Bertoli A., Scorrano L., Lanfranchi G., Cagnin S. (2019) Single cell analysis reveals the involvement of the long non-coding RNA Pvt1 in the modulation of muscle atrophy and mitochondrial network. Nucl. Acids Res. 47(4), 1653–1670.
Otaegui D., Saenz A., Ruiz-Martinez J., Olaskoaga J., López de Munain A. (2007) UCP2 and mitochondrial haplogroups as a multiple sclerosis risk factor. Mult. Scler. 13(4), 454–458.
Aulchenko Y.S., Hoppenbrouwers I.A., Ramagopalan S.V., Broer L., Jafari N., Hillert J., Link J., Lundström W., Greiner E., Sadovnick A.D., Goossens D., Van Broeckhoven C., Del-Favero J., Ebers G.C., Oostra B.A., van Duijn C.M., Hintzen R.Q. (2008) Genetic variation in the KIF1B locus influences susceptibility to multiple sclerosis. Nat. Genet. 40(12), 1402–1403.
International Multiple Sclerosis Genetics Consortium (IMSGC) (2010) Lack of support for association between the KIF1B rs10492972[C] variant and multiple sclerosis. Nat. Genet. 42(6), 469–470.
Арбузова Е.Е., Селянина Н.В., Каракулова Ю.В. (2019) Ассоциации однонуклеотидных полиморфизмов гена KIF1B с выраженностью клинических проявлений рассеянного склероза. Ж. Неврол. Психиатр. им. С.С. Корсакова. 119(10–2), 58–62.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


