Молекулярная биология, 2022, T. 56, № 1, стр. 35-54
МикроРНК как потенциальные регуляторы инфицирования SARS-CоV-2 и модификаторы клинической картины COVID-19
А. Н. Кучер a, Ю. А. Королёва a, А. А. Зарубин a, М. С. Назаренко a, *
a Научно-исследовательский институт медицинской генетики, Томский национальный исследовательский медицинский центр Российской академии наук
634050 Томск, Россия
* E-mail: maria.nazarenko@medgenetics.ru
Поступила в редакцию 09.03.2021
После доработки 14.05.2021
Принята к публикации 25.05.2021
- EDN: IIFEDF
- DOI: 10.31857/S0026898422010049
Аннотация
Пандемия, вызванная вирусом SARS-CoV-2, определила актуальность выявления факторов, способных влиять как на риск инфицирования, так и на тяжесть течения COVID-19. Среди этих факторов особый интерес представляют микроРНК, обладающие широким регуляторным потенциалом. В представленном обзоре обсуждается возможная роль микроРНК человека и генома/микроРНК SARS-CoV-2 в инфицировании и определении клинической картины COVID-19. Обобщена информация о SARS-CoV-2-специфичных микроРНК человека, уровне их экспрессии в различных органах (клетках) в норме и при развитии заболеваний, которые являются факторами риска тяжелого течения COVID-19. Обсуждается возможное участие SARS-CoV-2 в развитии клинической картины COVID-19, в том числе и посредством подавления микроРНК и РНК-связывающих белков человека, изменения уровня экспрессии генов в инфицированных клетках, возможных эпигенетических модификаций генома человека с участием микроРНК коронавирусного происхождения.
ВВЕДЕНИЕ
В 2020 году распространение респираторной инфекции COVID-19, вызванной коронавирусом SARS-CoV-2, привело к возникновению пандемии. COVID-19 поражает людей любого возраста, а клиническая картина данного заболевания варьирует от бессимптомного течения до тяжелого и критического состояния [1, 2]. В систематических обзорах, обобщающих данные о факторах риска, клинических признаках и симптомах COVID-19, отмечено, что тяжелое течение заболевания и повышенный риск летального исхода характерны для пациентов старше 60 лет, а также для лиц с такими сопутствующими заболеваниями, как артериальная гипертензия, сахарный диабет, сердечно-сосудистые и хронические респираторные заболевания [3, 4]. Кроме того, COVID-19 выступает в качестве фактора риска развития ряда патологий – вирусной пневмонии, дыхательной и острой почечной недостаточности, сепсиса [5], воспалительного поражения сосудов, повреждения миокарда, аритмии [6], тромботических осложнений (таких как острая ишемия конечностей, тромбоз брюшной и грудной аорты, инфаркт миокарда, венозная тромбоэмболия, острое нарушение мозгового кровообращения, диссеминированное внутрисосудистое свертывание) [7], неврологических нарушений [8]. В этой связи актуальным представляется поиск эндогенных факторов, способствующих инфицированию и/или неблагоприятному течению заболевания, вызванному SARS-CoV-2. Особый интерес в качестве маркеров функционального состояния организма и молекул с потенциальным терапевтическим эффектом вызывают микроРНК [9, 10].
МикроРНК – это некодирующие РНК длиной около 19–24 н. [11, 12]. В зависимости от того, из какого именно участка двухцепочечного предшественника-шпильки (3' либо 5') происходит зрелая одноцепочечная последовательность, микроРНК обозначают miR-3p, либо miR-5p [11, 13]. Механизм действия микроРНК заключается в комплементарном связывании с мРНК-мишенями, что приводит к торможению трансляции или разрушению мРНК [14].
МикроРНК вовлечены в регуляцию экспрессии более 60% всех белоккодирующих генов млекопитающих, они участвуют во всех основных биологических и патологических процессах, в том числе и в противовирусном ответе [12, 15]. К настоящему времени накоплен большой объем данных, свидетельствующих о том, что спектр и уровень экспрессии микроРНК зависят от функционального состояния организма и изменяется при развитии заболеваний [16, 17]. Имеются данные о том, что микроРНК клеток могут связываться с кодирующими регионами геномов РНК-содержащих вирусов (таких как ВИЧ, вирусы гепатита С, лихорадки денге и гриппа), тем самым оказывая противовирусное действие [2, 12, 14, 18]. Коронавирусы также относятся к РНК-содержащим вирусам, поэтому нельзя исключать, что среди множества факторов, влияющих на риск инфицирования и характер течения инфекции при сопутствующих коморбидных состояниях, определенную роль могут играть такие малые биомолекулы, как микроРНК.
Вирус SARS-CoV-2 – возбудитель COVID-19, принадлежит к роду бета-коронавирусов, включающему также вирусы SARS-CoV (возбудитель тяжелого острого респираторного синдрома) и MERS-CoV (возбудитель ближневосточного респираторного синдрома). Вспышки инфекций, вызванных SARS-CoV и MERS-CoV, произошли в 2002–2003 и 2012 гг. соответственно, но они не приобрели характера пандемии. Филогенетический анализ выявил до 79% сходства между геномами SARS-CoV-2 и SARS-CoV и до 50% – между геномами SARS-CoV-2 и MERS-CoV [18, 19].
Геном SARS-CoV-2 представлен одноцепочечной РНК положительной полярности. Длина референсной последовательности этой РНК, представленной в базе данных NCBI (NC_045512.2), составляет порядка 29.9 т.н. Около двух третей генома с 5'-конца заняты генами полипротеинов (ORF1a и ORF1b). Полипротеины вируса расщепляются протеазами на неструктурные белки (Nsp), которые формируют репликационно-транскрипционный комплекс. В состав оставшейся трети генома SARS-CoV-2 входят гены структурных белков – гликопротеина шиповидных отростков (S-белка, или белка шипа, от spike – шип), белков мембраны (M, membrane), оболочки (E, envelope) и нуклеокапсида (N, nucleocapsid). Также геномная РНК SARS-CoV-2 включает нетранслируемые области: 5'-UTR и 3'-UTR [12, 19‒22].
S-белок способствует проникновению вируса в клетку, связываясь для этого с ангиотензинпревращающим ферментом 2 (ACE2) [21]. Белок E формирует оболочку вируса и участвует в образовании гидрофильных пор в клеточных мембранах, белок М определяет форму оболочки вируса и служит центральным организатором сборки коронавируса [15, 23]. Белок N связан с геномной РНК коронавируса, а также с Nsp3 – неструктурным белком репликационно-транскрипционного комплекса, помогающим упаковке генома в вирионы [20].
Вирус SARS-CoV-2 относится к внутриклеточным патогенам, и для обеспечения своего функционирования и репликации использует клеточную машинерию организма-хозяина. Поэтому успешность инфицирования будет зависеть от функционального состояния клетки-хозяина (соотношения факторов, благоприятных и неблагоприятных для этого процесса) и возможности быстро “перестроить” молекулярную физиологию клетки и организма в целом так, чтобы обеспечить размножение и распространение вируса (например, возможность “уйти” от иммунного контроля организма). Подавлению входа SARS-CoV-2 в клетку и ограничению его репликации может способствовать повышенный уровень микроРНК, мишенью которых служат гены S, M, N, E и ORF1ab, в то время как низкий уровень этих микроРНК создает благоприятные условия для инфицирования клетки и репликации коронавируса [15]. Однако в патогенезе COVID-19 могут участвовать микроРНК не только клеток человека, но и SARS-CoV-2: геном этого коронавируса потенциально способен продуцировать микроРНК, которые могут участвовать в эпигенетических процессах в инфицированных клетках [19]. Кроме того, микроРНК человека могут вызывать разнонаправленные эффекты – как подавлять инфицирование, так и выступать в роли провирусных факторов [19, 24], а инфицирование клеток SARS-CoV-2 приводит к изменению экспрессии микроРНК в клетках человека [11]. Более высокий уровень инфицирования SARS-CoV-2 по сравнению с другими коронавирусами позволяет предположить, что именно различия в структуре специфичных участков генома коронавирусов определяют их влияние как на риск заражения, так и на течение COVID-19. Особенности структуры генома коронавирусов также могут приводить к формированию различных мишеней, связывающих микроРНК человека и, соответственно, определять “сценарии” течения болезни при наличии сопутствующих патологий, вызывающих изменение спектра и уровня экспрессии микроРНК в организме.
В обзоре рассмотрены различные аспекты роли микроРНК в патогенезе COVID-19. В качестве источника информации использованы база PubMed и интернет-ресурсы, содержащие данные об экспрессии микроРНК в тканях и клетках человека, и их значимости в развитии различных заболеваний человека (TissueAtlas [25], FANTOM5 [26] и Human MicroRNA Disease Database – HMDD [16]).
МикроРНК ЧЕЛОВЕКА, СПЕЦИФИЧНЫЕ ДЛЯ ГЕНОМА SARS-CoV-2
На основании поискового запроса с использованием различных комбинаций терминов microRNA, miRNA, SARS-CoV-2 и COVID-19 в базе данных PubMed на конец 2020 года выявлены 16 публикаций, в которых представлена информация о микроРНК, потенциально способных связываться с геномом SARS-CoV-2. Число таких микроРНК (по данным разных исследователей) варьировало от трех до более 1000, что связано с использованием разных баз данных, аналитических инструментов, а также с различиями в числе привлеченных микроРНК и последовательностей генома SARS-CoV-2 (табл. S1 , см. Приложение на сайте http://www.molecbio.ru/downloads/ 2022/1/supp_Kucher_rus.pdf). Некоторые микроРНК, имеющие мишени в геноме SARS-CoV-2, также способны связываться с геномами SARS-CoV и MERS-CoV, а в отдельных случаях и с геномами низкопатогенных коронавирусов, вызывающих легкие формы острой респираторной инфекции. Эти микроРНК были исключены из дальнейшего анализа, чтобы провести поиск микроРНК, которые могут определять высокую патогенность именно SARS-CoV-2. Кроме того, критерием включения SARS-CoV-2-специфичных микроРНК в анализ было их упоминание в качестве специфичных в двух и более публикациях (с целью снижения вероятности ошибочного описания потенциально значимых для инфицирования клеток микроРНК). В итоге отобраны 39 микроРНК, отвечающих указанным критериям (табл. 1).
Таблица 1.
МикроРНК человека, потенциально способные связываться с различными генами коронавируса SARS-CoV-2
| микроРНК | MIMAT, номера miRBase* | Гены-мишени | Источник |
|---|---|---|---|
| hsa-miR-16-2-3p | MIMAT0000069 | Не указаны | [2, 11] |
| hsa-miR-23b-5p | MIMAT0004587 | ORF8 | [15, 19] |
| Не указаны | [27] | ||
| hsa-miR-34c-3p | MIMAT0004677 | Не указаны | [2, 14] |
| hsa-miR-125a-3p | MIMAT0004602 | Ген S-белка | [19] |
| Не указаны | [2, 11] | ||
| hsa-miR-141-3p | MIMAT0000432 | ORF1ab | [28] |
| Не указаны | [2, 11] | ||
| hsa-miR-142-3p | MIMAT0000434 | Не указаны | [9, 11] |
| hsa-miR-193b-5p | MIMAT0004767 | Ген M-белка | [28] |
| Не указаны | [11] | ||
| hsa-miR-195-3p | MIMAT0004615 | ORF1ab, S | [28] |
| Не указаны | [2, 11, 29] | ||
| hsa-miR-197-5p | MIMAT0022691 | Nsp3, ORF1a | [30] |
| ORF1ab | [19] | ||
| Не указаны | [11] | ||
| hsa-miR-203a-5p | MIMAT0031890 | Ген S-белка | [28] |
| Не указаны | [2, 14] | ||
| hsa-miR-208a-5p | MIMAT0026474 | Ген N-белка | [15, 19] |
| ORF1ab | [28] | ||
| Не указаны | [2] | ||
| hsa-miR-208b-5p | MIMAT0026474 | ORF1ab | [28] |
| Не указаны | [2, 14] | ||
| hsa-miR-325 | MIMAT0000771 | Ген M-белка | [15] |
| ORF1ab | [28] | ||
| Не указаны | [2] | ||
| hsa-miR-378c | MIMAT0016847 | ORF1ab | [19] |
| Не указаны | [2, 27, 31] | ||
| hsa-miR-548ag | MIMAT0018969 | ORF1ab | [15, 28] |
| Ген S-белка | [15] | ||
| Не указаны | [2] | ||
| hsa-miR-549a-3p | MIMAT0003333 | ORF1ab | [19, 28] |
| ORF3a | [15] | ||
| Не указаны | [2] | ||
| hsa-miR-605-5p | MIMAT0003273 | ORF1ab | [28] |
| Не указаны | [2, 28] | ||
| hsa-miR-628-3p | MIMAT0004809 | ORF1ab | [19, 28] |
| Не указаны | [2] | ||
| hsa-miR-668-3p | MIMAT0003881 | 5'-UTR | [18] |
| Не указаны | [2, 14] | ||
| hsa-miR-1246 | MIMAT0005898 | ORF3a | [15] |
| Не указаны | [11] | ||
| hsa-miR-1293 | MIMAT0005883 | 3'-UTR | [22] |
| ORF1ab | [28] | ||
| Не указаны | [2, 11] | ||
| hsa-miR-1910-3p | MIMAT0026917 | ORF1ab/ORF7a/ген N белка | [15] |
| Не указаны | [11] | ||
| hsa-miR-2392 | MIMAT0019043 | ORF8 | [15, 19] |
| hsa-miR-3120-5p | MIMAT0019198 | ORF1ab | [19] |
| Не указаны | [31] | ||
| hsa-miR-3132 | MIMAT0014997 | ORF3a | [19] |
| Не указаны | [11] | ||
| hsa-miR-3135b | MIMAT0018985 | ORF7a | [15, 19] |
| hsa-miR-3155a | MIMAT0015029 | Ген N-белка | [15, 19] |
| Не указаны | [2] | ||
| hsa-miR-3190-3p | MIMAT0022839 | ORF1ab | [15, 19] |
| ORF8 | [15] | ||
| Ген S-белка | [28] | ||
| Не указаны | [2] | ||
| hsa-miR-3914 | MIMAT0018188 | ORF1ab | [19, 28] |
| Не указаны | [31] | ||
| hsa-miR-4510 | MIMAT0019047 | ORF3a | [15] |
| Ген S-белка | [19] | ||
| hsa-miR-4684-3p | MIMAT0019770 | ORF7a | [19] |
| Ген N-белка | [28] | ||
| Не указаны | [2] | ||
| hsa-miR-5087 | MIMAT0021079 | ORF1ab | [28] |
| Не указаны | [2, 32] | ||
| hsa-miR-5590-3p | MIMAT0022300 | ORF7a | [15] |
| ORF1ab | [28] | ||
| Не указаны | [2] | ||
| hsa-miR-6736-5p | MIMAT0027373 | ORF1ab | [19] |
| Не указаны | [11] | ||
| hsa-miR-6741-5p | MIMAT0027383 | Ген N-белка | [15] |
| ORF1ab | [19] | ||
| Не указаны | [29] | ||
| hsa-miR-6751-5p | MIMAT0027402 | ORF3a | [15, 19] |
| hsa-miR-6837-3p | MIMAT0027577 | ORF1ab | [19] |
| 5'-UTR | [18] | ||
| Не указаны | [2] | ||
| hsa-miR-8066 | MIMAT0030993 | Ген N-белка | [15, 19] |
| Не указаны | [12, 31] | ||
| hsa-miR-12119 | MIMAT0049013 | ORF1ab | [15] |
| Ген N-белка | [19] |
⃰ * Данные взяты из базы miRBase [33].
Необходимо отметить, что разные последовательности зрелых микроРНК, происходящих из общего предшественника, могут отличаться способностью связываться с геномами разных коронавирусов. Так, hsa-miR-195-3p способна связываться только с геномом SARS-CoV-2, в то время как hsa-miR-195-5p может связываться также с геномами как SARS-CoV, так и других коронавирусов [2, 34]. Поэтому в дальнейшем (во всех возможных случаях) внимание уделяли именно той последовательности микроРНК (-3p или -5p), которую изучали в привлеченных к рассмотрению исследованиях. Тем не менее, работы, в которых последовательность микроРНК не была конкретизирована (-3p или -5p), также включали в рассмотрение (далее микроРНК приведены в том виде, в котором они указаны в оригинальных публикациях).
Участки генома SARS-CoV-2 различаются по числу микроРНК, способных с ними связываться: больше всего микроРНК (22) описано для ORF1ab, а также для генов, кодирующих S- и N-белок (табл. 1), что, в целом, согласуется с размерами соответствующих генов (хотя размер гена, кодирующего S-белок, в несколько раз больше гена, кодирующего белок нуклеокапсида) [35]. В качестве мишеней отдельных микроРНК описаны от одного до трех генов SARS-CoV-2, но в ряде случаев локализация мишеней микроРНК авторами исследований не указана (см. табл. 1). На настоящий момент неизвестно, может ли влиять на ограничение инфицирования то, какой именно ген SAR-CoV-2 служит мишенью для микроРНК клеток организма-хозяина, но можно предположить, что наличие нескольких мишеней для разных микроРНК благоприятствует защите человека от инфекции. Это связано со способностью генома SARS-CoV-2 мутировать, что может приводить как к потере, так и к появлению новых мишеней для одной и той же микроРНК или к возникновению мишеней для других микроРНК, которые ранее не имели мишеней на геноме коронавируса. Такая ситуация наблюдалась, в частности, при сравнении профилей связывания 24 микроРНК человека с 67 различными геномами SARS-CoV-2, выявленными на территории 24 стран [19]. В их число входили и четыре микроРНК, привлеченные к анализу в нашей работе: hsa-miR-23b-5p, -378с, -549а-3р и -12119, мишени которых “потеряны” в 2, 1, 11 и 2 геномах SARS-CoV-2 соответственно. Примечательно, что некоторые кластеры микроРНК, выделенные на основании профилей связывания с геномом SARS-CoV-2, оказались ассоциированными с повышенной смертностью от COVID-19 в странах, где они были выявлены [19], что свидетельствует о значимости как микроРНК хозяина, так и особенностей генома коронавируса для клинической картины COVID-19. С этой точки зрения наиболее уязвимым геном SARS-CoV-2 является ORF1ab, поскольку с ним потенциально может связываться наибольшее число микроРНК, а самый заметный защитный эффект могут оказывать hsa-miR-1910-3p и hsa-miR-3190-3p, имеющие не менее трех генов-мишеней в геноме данного коронавируса (табл. 1).
Способность защищать клетки хозяина зависит не только от наличия мишеней в геноме SARS-CoV-2, но и от уровня экспрессии микроРНК. В клетках органов, через которые вирус проникает в организм, уровень экспрессии микроРНК может влиять на защиту от SARS-CoV-2, в других органах – на риск развития осложнений. Известно, что для экспрессии микроРНК характерны тканевая и клеточная специфичность, чувствительность к воздействию средовых факторов и зависимость от функционального состояния организма. Поэтому мы проанализировали данные из общедоступных баз по экспрессии микроРНК в органах (и клетках) здоровых индивидов (TissueAtlas [25], FANTOM5 [26]), а затем обобщили информацию об изменении уровня экспрессии этих микроРНК при некоторых заболеваниях, значимых для определения клинической картины COVID-19 (PubMed, Human MicroRNA Disease Database [16]).
ЭКСПРЕССИЯ микроРНК, ПОТЕНЦИАЛЬНО СПОСОБНЫХ СВЯЗЫВАТЬСЯ С ГЕНОМОМ ВИРУСА SARS-CoV-2, В ТКАНЯХ И КЛЕТКАХ ЧЕЛОВЕКА
При выборе тканей, органов и типов клеток, в которых анализировали микроРНК, потенциально способные связываться с геномом SARS-CoV-2, учитывали результаты эпидемиологических исследований их чувствительности к воздействию данного коронавируса и/или поражаемость при COVID-19 [3–8]. Всего проанализировали 16 тканей (рис. 1) и 20 типов клеток (рис. 2) различных органов.
Рис. 1.
Теплокарта, отражающая экспрессию микроРНК, потенциально способных связываться с геномом вируса SARS-CoV-2, в тканях различных органов человека (согласно TissueAtlas [25]). Использованы значения логарифма квантильно нормированного уровня экспрессии микроРНК – log2(QNE).
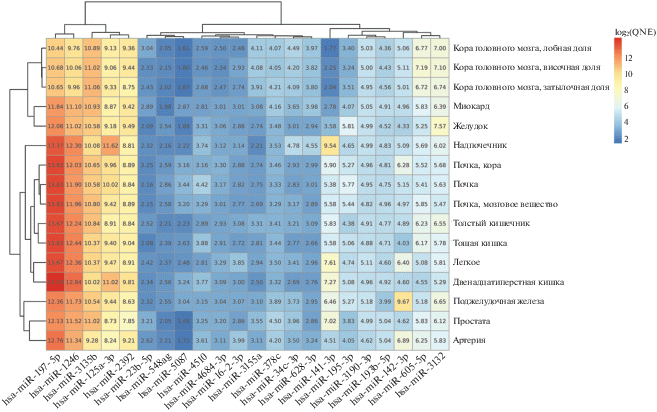
Рис. 2.
Теплокарта, отражающая экспрессию микроРНК, потенциально способных связываться с геномом вируса SARS-CoV-2, в первичных культурах клеток человека (согласно атласу FANTOM5 [26]). Использован логарифм уровня экспрессии микроРНК, нормализованного по количеству прочтений на миллион – или log2(CPM); n – число образцов, информация по которым представлена в базе FANTOM5.

Имеющиеся данные об уровне экспрессии микроРНК в тканях и органах здорового человека весьма немногочисленны: в TissueAtlas [25] представлены результаты анализа образцов, полученных от одного индивида, умершего естественной смертью. Экспрессия 22 из 39 рассмотренных микроРНК выявлена в тканях легкого, различных отделов головного мозга, желудочно-кишечного тракта, сердца, артерий, почек и предстательной железы (всего 16 органов) (рис. 1). Выделены три кластера микроРНК: в первый входят пять микроРНК с относительно высоким уровнем экспрессии в тканях всех органов (hsa-miR-197-5p, -1246,-3135b, -2392 и -125а-3р), во второй – семь микроРНК со средним уровнем экспрессии (hsa-miR-3132, -605-5p, -142-3p, -193b-5p, -3190-3p, -195-3p, ‑141-3p), и в третий – 12 микроРНК с низким уровнем экспрессии. Зарегистрирована экспрессия лишь одной из двух микроРНК, имеющих наибольшее число мишеней на геноме SARS-CoV-2 – hsa-miR-3190-3p (табл. 1), но ее уровень был относительно невысоким (ниже средних значений) (рис. 1). Однако уровень экспрессии микроРНК в пределах кластеров также характеризуется тканевой специфичностью. В зависимости от уровня экспрессии микроРНК ткани органов также группируются в три кластера (рис. 1). В ткани легкого на наиболее высоком уровне экспрессируются hsa-miR-197-5p, -1246, -3135b (>10 log2(квантильно нормализованного уровня экспрессии микроРНК – QNE)), и эти микроРНК могут рассматриваться в качестве потенциально значимых для ограничения инфицирования SARS-CoV-2.
Поскольку ткани имеют гетерогенную клеточную структуру, на втором этапе проанализированы особенности экспрессии 39 отобранных микроРНК в различных типах клеток, информация по которым представлена в базе FANTOM5 [26], в том числе – в эпителиальных клетках разных органов, в клетках крови и др. (рис. 2). Информация об уровне экспрессии в выбранных для рассмотрения клетках была доступна для 36 из 39 микроРНК. Экспрессия 11 из 36 этих микроРНК не выявлена в анализируемых типах клеток (hsa-miR-3135b, ‑8066, -6741-5p, -6736-5p, -5590-3p, -5087, -4684-3p, -3914, -3132, -208a-5p и -208b-5р – данные по этим микроРНК не представлены на рис. 2), экспрессия еще шести микроРНК (hsa-miR-548ag, ‑3120-5p, -4510, -1246, -2392, -6751-5p) регистрировалась на очень низком уровне (<1.0 log2(CPM)) в единичных типах клеток, а оставшиеся 19 микроРНК экспрессируются на более высоком уровне (>1.0 log2(CPM)), который варьировал в разных типах клеток. Лишь hsa-miR-16-2-3p имеет средний (и примерно равный) уровень экспрессии во всех анализируемых типах клеток (5.71–7.91 log2(CPM)). Ни одна из микроРНК, имеющих не менее трех мишеней в геноме SARS-CoV-2 (табл. 1), не относится к категории высоко экспрессирующихся в рассмотренных типах клеток (рис. 2).
Уровень экспрессии некоторых микроРНК был специфичным для разных типов клеток: hsa-miR-142-3p имела высокий уровень экспрессии (>10 log2(CPM)) в клетках крови и невысокий (или нулевой) в других типах клеток; hsa-miR-141-3p – высокий уровень экспрессии в эпителиальных клетках респираторного и пищеварительного тракта, предстательной железы, а также в B‑лимфоцитах, средний уровень – в других типах клеток крови и низкий – во всех остальных типах клеток. Уровень экспрессии hsa-miR-125a-3p был низким в клетках крови и средним в других типах клеток. Уровень экспрессии других микроРНК также зависел от типа клеток, но в целом был невысоким (в большинстве случаев – <4.0 log2(CPM)). Исходя из приведенных данных об уровне экспрессии микроРНК в различных типах клеток в норме (рис. 2), можно заключить, что для участия в определении ответа организма на инфицирование SARS-CoV-2 и в клинической картине COVID-19 наиболее перспективны hsa-miR-16-2-3p, -125a-3p, -141-3p, -142-3p и, в меньшей степени, hsa-miR-378, -628-3p, -1293, -23b-5p, -34c-3p, -193b-5p, -668-3p и -195-3p. Эти микроРНК имеют мишени в геноме SARS-CoV-2, поэтому можно ожидать, что высокий уровень их экспрессии будет снижать риск инфицирования и/или тяжелого течения болезни. Особое место в этом ряду занимает hsa-miR-141-3p, характеризующаяся высоким уровнем экспрессии в эпителиальных клетках нижних дыхательных путей и пищеварительного тракта, которые служат первым барьером на пути инфицирования SARS-CoV-2.
ЭКСПРЕССИЯ микроРНК, ПОТЕНЦИАЛЬНО СПОСОБНЫХ СВЯЗЫВАТЬСЯ С ГЕНОМОМ ВИРУСА SARS-CoV-2, ПРИ РАЗЛИЧНЫХ ЗАБОЛЕВАНИЯХ
Поскольку уровень экспрессии микроРНК зависит от функционального состояния организма и может меняться при развитии патологий [16, 17], а ряд болезней и коморбидных состояний выступают в качестве фактора риска развития тяжелого течения COVID-19 [3, 4, 36], нами проанализированы изменения профилей микроРНК, специфичных для SARS-CoV-2, при различных заболеваниях. Такая информация доступна для 16 микроРНК (табл. 2), среди которых, к сожалению, отсутствовали выделенные на предыдущих этапах такие потенциально значимые для определения риска инфицирования SARS-CoV-2 и характера клинической картины COVID-19 микроРНК, как hsa-miR-1910-3p, hsa-miR-3190-3p (имеют не менее трех мишеней на геноме SARS-CoV-2 (табл. 1)), hsa-miR-3135b (высокий уровень экспрессии в различных органах (рис. 1)).
Таблица 2.
Изменение уровня микроРНК, потенциально способных связываться с геномом вируса SARS-CoV-2, при различных заболеваниях
| Патология | Источник микроРНК | МикроРНК | Изменение уровня микроРНК | Источ-ник |
|---|---|---|---|---|
| Заболевания дыхательной системы | ||||
| ХОБЛ | Легочная артерия | miR-197 | ↓ по сравнению с некурящими | [37] |
| Плазма крови | miR-628-3p | ↓ по сравнению с курящими и с некурящими | [38] | |
| Сыворотка крови | miR-1246 | ↓ у пациентов через 10 лет; у 75% выявлена эмфизема | [39] | |
| ХОБЛ, курильщики | Кровь | miR-203 | ↑ по сравнению с некурящими, но ниже (не значимо), чем у курильщиков | [40] |
| Ткани легких | miR-203 | ↓ по сравнению как с курящими, так и с некурящими | ||
| Легочная гипертензия | Лейкоциты крови | miR-1246 | ↓ | [41] |
| miR-23b | ↑ | |||
| Тяжелая легочная гипертензия | Лейкоциты крови | miR-208b | ↑ по сравнению с контролем и легочной гипертензией средней степени тяжести | |
| Легочная гипертензия у мышей | Гипоксичная легочная ткань | miR-125a | ↑ | [42] |
| Циркулирующие микроРНК | miR-125a | ↓ | ||
| Бронхиальная астма | Бронхиальный эпителий | miR-203 | ↓ | [43] |
| LPS-индуцированная пневмония у мышей | Ткани легких | miR-203 | ↑ повышается и достигает максимума через 5 дней | [44] |
| Заболевания сердечно-сосудистой системы | ||||
| Гипертензия и сердечная недостаточность | Мононуклеарные клетки периферической крови | miR-208b | ↑ по сравнению с гипертензией без сердечной недостаточности | [45] |
| Гипертензия с гипертрофией левого желудочка | Плазма крови | miR-208 | ↑ | [46] |
| Гипертензия, в т.ч. с гипертрофией левого желудочка | Мононуклеары периферической крови | miR-208 | ↑ | [17, 47] |
| Гипертонический нефросклероз | Ткани почек | miR-141 | ↑ | [48] |
| Сердечная недостаточность, стадии С или D | Сыворотка крови | miR-197-5p | ↑ | [49] |
| Сердечная недостаточность | Плазма крови | miR-193b-5p | ↓ | [50] |
| Сердечная недостаточность с уменьшенной фракцией выброса левого желудочка | Плазма крови | miR-193b-5p | ↓ | [50] |
| Ишемия во время хирургического вмешательства | Миокард правого предсердия | miR-195 | ↓ | [51] |
| Ишемическая и неишемическая сердечная недостаточность | Плазма крови | miR-195-3p | ↑ | [52] |
| Ишемическая болезнь сердца | Плазма крови | miR-208a | ↑ | [53] |
| Обструктивная ишемическая болезнь сердца | Плазма крови | miR-3135b | ↑ | [54] |
| Гипертрофия миокарда левого желудочка, мыши | Миокард левого желудочка | miR-23 | ↑ | [55] |
| miR-195 | ↑ | |||
| Гипертрофия миокарда левого желудочка | Миокард левого желудочка | miR-195 | ↑ | [56] |
| Гипертрофическая кардиомиопатия, кошки | Сыворотка крови | miR-1246 | ↑ | [57] |
| Фибрилляция предсердий | Миокард предсердий | miR-208a | ↓ по сравнению с синусовым ритмом | [58] |
| Хроническая фибрилляция предсердий | Миокард | miR-208b | ↑ | [59] |
| Инфаркт миокарда | Плазма крови | miR-195-3p | ↑ через 8 ч (максимальное значение) и 12 ч после инфаркта | [60] |
| Инфаркт миокарда | Миокард | miR-208 | ↑ | [61] |
| Стенокардия | Плазма крови | miR-208a | ↑ по сравнению с контролем и инфарктом миокарда | [62] |
| Диастолическая дисфункция миокарда | Плазма крови | miR-1246 | ↑ | [63] |
| Стабильная компенсированная дилатационная кардиомиопатия (систолическая + диастолическая дисфункция); декомпенсированная застойная сердечная недостаточность, вторичная по отношению к дилатационной кардиомиопатии | Плазма крови | miR-142-3p | ↓ | [63] |
| Аневризма коронарной артерии у детей с болезнью Кавасаки | Экзосомы сыворотки крови | miR-1246 | ↓ по сравнению со здоровыми и инфицированными вирусами Эпштейна–Барр и болезни Ауески | [64] |
| Лица с высоким риском сердечно-сосудистой патологии | Сыворотка крови | miR-23b | ↑ | [65] |
| Сахарный диабет и его осложнения | ||||
| Сахарный диабет типа 2 | Плазма крови | miR-197 | ↓ | [66] |
| Сахарный диабет типа 1 или 2 | Периферическая кровь | miR-23b | ↓ | [67] |
| Преддиабетическое состояние | Сыворотка крови | miR-193b | ↑ | [68] |
| Диабетическая нефропатия | Экзосомы сыворотки крови | miR-1246 | ↑ по сравнению с контролем и с больными диабетом без нефропатии | [69] |
| Ишемическая диабетическая кардиомиопатия | Миокард левого желудочка | miR-23b | ↓ | [70] |
| Пролиферативная диабетическая ретинопатия | Стекловидное тело | miR-142-3p | ↑ | [71] |
| Аутоиммунные заболевания | ||||
| Язвенный колит, активная фаза | Воспаленная толстая кишка | miR-378c | ↑ при проведении анти-TNF-терапии по сравнению с другими видами терапии | [72] |
| Системная красная волчанка | Ткани почек | miR-23b | ↓ | [73] |
| Ревматоидный артрит | Синовиальные ткани суставов | miR-23b | ↓ | [73] |
| Болезнь Крона, язвенный колит, активная фаза; ревматоидный артрит | Сыворотка крови | miR-1246 | ↑ по сравнению с контролем и неактивной формой заболевания (для болезни Крона и язвенного колита) | [74] |
| Кожная волчанка, подострое и дискоидальное поражение | Сыворотка крови | miR-1246 | ↓ | [75] |
| Кожная волчанка, подострое поражение | Сыворотка крови | miR-23b | ↓ | [75] |
| Болезнь Грейвса | Мононуклеары периферической крови | miR-23b-5p | ↑ в стадии ремиссии по сравнению с трудноизлечимыми пациентами | [76] |
Согласно данным, представленным в базе HMDD [16], уровень большинства микроРНК, специфичных для SARS-CoV-2, изменяется в крови или пораженных органах при различных заболеваниях, в том числе при патологиях дыхательной и сердечно-сосудистой системы (особенно при артериальной гипертензии), при сахарном диабете и его осложнениях, аутоиммунных (табл. 2) и ряде других заболеваний. К сожалению, не во всех анализируемых публикациях указано, из какой цепи происходит микроРНК с измененным уровнем экспрессии, а иногда микроРНК рассматриваются лишь на уровне семейств, но они не были исключены из дальнейшего анализа.
Наибольший интерес представляют заболевания дыхательной системы, при которых изменяется уровень экспрессии микроРНК человека, специфичных для связывания с геномом SARS-CoV-2. Зарегистрировано изменение уровня экспрессии семи SARS-CoV-2-специфичных микроРНК человека при ХОБЛ, легочной гипертензии, бронхиальной астме и бактериальной пневмонии (табл. 2). Три из этих микроРНК (hsa-miR-125a-3p, -197-5p и -1246) относятся к категории высоко экспрессирующихся в тканях разных органов, в том числе и в ткани легкого (см. рис. 1).
Уровень специфичных для SARS-CoV-2 микроРНК определен в ткани легкого и/или в крови пациентов, либо модельных объектов (мышей) с разными заболеваниями легочной системы. В большинстве исследований при развитии патологии легких регистрировали снижение уровня различных микроРНК. Так, ни при одном из рассмотренных заболеваний дыхательной системы не выявлено увеличения SARS-CoV-2-специфичных микроРНК в ткани легкого человека (табл. 2).
Снижение уровня hsa-miR-203 зарегистрировано в клетках бронхиального эпителия при бронхиальной астме [43] и в ткани легкого у курящих пациентов с ХОБЛ по сравнению со здоровыми индивидами как курящими, так и не курящими [40]. Однако в крови этих же пациентов с ХОБЛ уровень hsa-miR-203 был выше, чем у некурящих, но несколько меньше (статистически незначимо), чем у курящих здоровых индивидов [40]. Можно предположить, что курение является значимым фактором, влияющим на уровень экспрессии hsa-miR-203, а увеличение у курящих уровня данной микроРНК может объяснять то, что некоторые исследователи не рассматривают курение как фактор риска COVID-19 [77].
Снижение уровня hsa-miR-203 в ткани легкого при ХОБЛ (в норме эта микроРНК экспрессируется в клетках дыхательного эпителия на невысоком уровне (рис. 2)) предположительно может быть неблагоприятным фактором в отношении COVID-19, но высокий уровень hsa-miR-203 в лейкоцитах крови может сдерживать распространение инфекционного процесса за пределы легкого. Благоприятствовать развитию COVID-19 должен также более низкий уровень hsa-miR-203 в эпителии бронхов при астме (табл. 2), но астму не рассматривают как фактор риска COVID-19 [78, 79].
В то же время увеличение экспрессии miR-203 в ткани легких наблюдали на модельных объектах при пневмонии, индуцированной воздействием липополисахарида (LPS) [44]. Повышение уровня hsa-miR-203 зарегистрировано также в образцах слизистой оболочки носа младенцев, инфицированных респираторно-синцитиальным вирусом [80]. Следует отметить, что у абсолютного большинства пациентов с COVID-19 выявляются сопутствующие инфекции бактериальной, вирусной или грибковой природы [81].
Интересными представляются также данные о связи снижения уровня hsa-miR-1246 в сыворотке с увеличением продолжительности заболевания ХОБЛ, причем у 3/4 пациентов с низким уровнем данной микроРНК регистрировалась эмфизема легких [39]. Снижение уровня этой микроРНК наблюдали также в лейкоцитах крови при легочной гипертензии [41]. Нsa-miR-1246 относится к категории микроРНК, высоко экспрессирующихся в различных органах (рис. 1), но ее уровень снижен в легочной ткани курильщиков. При этом has-miR-1246 регулирует экспрессию гена ACE2, кодирующего один из ключевых рецепторов SARS-CoV-2 [21, 82]. Соответственно, низкий уровень hsa-miR-1246 может благоприятствовать инфицированию вирусом SARS-CoV-2 как легких, так и других органов через усиление экспрессии ACE2.
Разнонаправленное влияние курения на SARS-CoV-2-специфичные микроРНК человека (в частности, miR-1246 и miR-203) свидетельствует о сложности установления причины изменения уровня экспрессии разных микроРНК (экзогенный фактор/патология) и, соответственно, выделения условий, определяющих как риск инфицирования, так и характер течения COVID-19. Кроме того, в некоторых случаях при одной и той же патологии наблюдали разнонаправленные изменения экспрессии микроРНК в разных тканях (табл. 2), как это показано и для miR-125a, уровень которой при легочной гипертензии у мышей возрастал в ткани легкого, но снижался в крови [42].
Таким образом, выявлены изменения уровня экспрессии ряда SARS-CоV-2-специфичных микроРНК при заболеваниях дыхательной системы. Эти изменения могут быть разнонаправленными, иногда одна и та же микроРНК может экспрессироваться на разном уровне в разных тканях (легкие, кровь), они могут модифицироваться средовыми воздействиями (в частности, курением и бактериальной инфекцией).
Изменение уровня SARS-CоV-2-специфичных микроРНК человека при других заболеваниях может влиять на риск развития осложнений при COVID-19. Уровень экспрессии данных биомолекул при различных патологиях чаще определяли в клетках или в экзосомах крови, реже – в пораженных органах (табл. 2).
Сердечно-сосудистые заболевания считаются фактором риска осложненного течения COVID-19 [3, 4, 83]. Однако в большинстве исследований отмечено увеличение уровня экспрессии SARS-CоV-2-специфичных микроРНК (табл. 2), что должно защищать организм от развития COVID-19. В данном случае, следует принять во внимание несколько моментов.
Во-первых, увеличение уровня SARS-CoV-2-специфичных микроРНК человека часто отражает тяжесть патологического процесса (табл. 2). Например, при сердечной недостаточности наблюдается повышение уровня hsa-miR-197-5p в сыворотке крови и корреляция уровня данной микроРНК с фиброзом миокарда и неблагоприятными сердечными событиями у пациентов [49]. Иными словами, более высокие уровни ряда микроРНК отражают тяжесть или позднюю стадию течения основного заболевания, и это может быть более значимым прогностическим фактором здоровья и/или исхода сопутствующего заболевания и COVID-19, чем возможность блокировать распространение в организме SARS-CoV-2.
Во-вторых, SARS-CoV-2 попадает в организм через дыхательную систему, и после инфицирования может меняться паттерн экспрессии генов (в том числе и генов микроРНК) как в легких, так и в других органах [9, 84].
В-третьих, лишь три микроРНК, уровень которых изменялся при заболеваниях сердечно-сосудистой системы, относились к категории высоко экспрессирующихся (hsa-miR-197-5p, -3135, -1246), а одна (hsa-miR-141) экспрессировалась на среднем уровне (рис. 1, 2). В то же время уровень экспрессии микроРНК может иметь значение для подавления распространения коронавируса в организме на этапе виремии и, конечно, этот процесс будет зависеть от вирусной нагрузки на организм в целом.
При заболеваниях сердечно-сосудистой системы зарегистрировано снижение уровня экспрессии только пяти микроРНК (табл. 2): hsa-miR-193b-5p в плазме крови при сердечной недостаточности [50]; hsa-miR-195 в миокарде при ишемии во время хирургического вмешательства [51]; hsa-miR-208a в миокарде при фибрилляции предсердий [58]; hsa-miR-142-3p в плазме крови при систолической и диастолической дисфункции, вторичной по отношению к дилатационной кардиомиопатии [63]; и hsa-miR-1246 – в экзосомах сыворотки крови при аневризме коронарной артерии у детей с болезнью Кавасаки [64].
Интересно, что у детей и взрослых, инфицированных SARS-CoV-2, иногда наблюдают мультисистемный воспалительный синдром, по клинической картине сходный с болезнью Кавасаки [85–87]. Не исключено, что снижение уровня hsa-miR-1246 путем связывания с геномом SARS-CoV-2 может не только способствовать проникновению коронавируса в клетку посредством увеличения уровня экспрессии его рецептора (ACE2), но и изменять другие метаболические пути, определяющие патогенез каваски-подобного мультисистемного воспаления и ряда аутоиммунных заболеваний, что все чаще отмечается в эпидемиологических исследованиях [88]. Кроме того, в цельной крови пациентов со средним и тяжелым течением COVID-19 был снижен уровень hsa-miR-142-3p. Эту микроРНК рассматривают в качестве одного из биомаркеров тяжелой формы COVID-19 и потенциальной терапевтической мишени, так как снижение ее уровня способствует развитию воспалительного процесса [9].
Изменения уровня экспрессии SARS-CoV-2-специфичных микроРНК выявлены и при других заболеваниях (табл. 2), факторах риска тяжелого течения COVID-19, таких как сахарный диабет, патология печени и др. [89, 90]. Так, в плазме крови больных сахарным диабетом типа 2 наблюдали снижение уровней hsa-miR-197 [66] и hsa-miR-23b [67]. Снижение уровня последней микроРНК зарегистрировано и в миокарде левого желудочка при ишемической диабетической кардиомиопатии [70] (табл. 2), что можно рассматривать как фактор риска осложненного течения COVID-19 и согласуется с эпидемиологическими наблюдениями [3, 4, 36].
В то же время в сыворотке крови наблюдали увеличение экспрессии hsa-miR-1246 при диабетической нефропатии [69] и hsa-miR-193b у лиц с преддиабетическим состоянием [68]. В первом случае, как уже отмечалось, высокий уровень микроРНК и ее “нейтрализация” путем связывания с геном SARS-CoV-2 не может обладать защитным эффектом, так как низкий уровень этой микроРНК способствует увеличению уровня ACE2 и, тем самым, создает благоприятные условия для проникновения коронавируса в клетку. Что касается hsa-miR-193b, то, согласно [68], увеличение ее экспрессии наблюдали, во-первых, только при преддиабете, но не при развившемся диабете; во-вторых, это увеличение нестабильно, так как уровень данной микроРНК возвращается к исходному у лиц с преддиабетом и у мышей с непереносимостью глюкозы при воздействиях, приводящих к нормализации метаболических параметров (например, при регулярных физических упражнениях). Это еще раз указывает на то, что уровень микроРНК может модифицироваться различными факторами. В целом, можно предположить, что при сахарном диабете типа 2 низкий уровень hsa-miR-197 (высокий уровень экспрессии в разных органах в норме – рис. 1) и hsa-miR-23b (низкий уровень экспрессии – рис. 1) могут способствовать осложненному течению COVID-19.
Анализ изменения уровня микроРНК при аутоиммунных заболеваниях представляет интерес для оценки риска инфицирования SARS-CoV-2, определения клинической картины COVID-19 и развития осложнений после COVID-19 в виде аутоиммунных заболеваний. Патофизиология аутоиммунных заболеваний кишечника (таких, как болезнь Крона, язвенный колит) и наличие SARS-CoV-2 в клетках кишечника создают условия, способствующие инфицированию SARS-CoV-2, но нет доказательств того, что у пациентов с воспалительными заболеваниями кишечника COVID-19 встречается чаще, чем в общей популяции, что может быть связано с особенностями терапии данных заболеваний [91]. Более того, обсуждается возможность использования препаратов, применяемых при аутоиммунных заболеваниях, в терапии COVID-19 [92]. Однако мнения о риске развития осложнений COVID-19 у пациентов с аутоиммунными заболеваниями противоречивы [93].
В пользу низкого риска развития COVID-19 и его осложнений при некоторых формах лечения аутоиммунных заболеваний свидетельствуют данные об увеличении экспрессии SARS-CoV-2-специфической hsa-miR-378с в воспаленных тканях толстого кишечника у лиц с язвенным колитом, принимающих ингибиторы TNF, по сравнению с принимающими другие лекарственные препараты пациентами и здоровыми лицами [72]. Уровень hsa-miR-1246 был увеличен также в сыворотке крови пациентов с активной фазой язвенного колита, болезни Крона и ревматоидного артрита [74], а hsa-miR-23b-5p – в мононуклеарных клетках крови пациентов с болезнью Грейвса в стадии ремиссии, по сравнению с пациентами с тяжелым течением болезни [76]. Возможно, именно тяжесть заболевания или степень ее компенсации будут определять риск тяжелого течения COVID-19 у пациентов с аутоиммунными заболеваниями.
С другой стороны, в сыворотке крови пациентов с различными формами системной красной волчанки выявлено снижение SARS-CoV-2-специфичных микроРНК – hsa-miR-1246 и hsa-miR-23b [75]. Показано, что после COVID-19 у пациентов могут развиться аутоиммунные заболевания, в том числе – системная красная волчанка и синдром Гийена–Барре [93]. Таким образом, разнонаправленные изменения уровня hsa-miR-1246, зарегистрированные при разных аутоиммунных заболеваниях, могут указывать на дифференцированный риск развития и течения коронавирусной инфекции в зависимости от конкретной нозологии.
Приведенные данные свидетельствуют о различиях в изменении экспрессии SARS-CoV-2-специфичных микроРНК при различных заболеваниях человека. При этом следует принимать во внимание тот факт, что инфицирование SARS-CoV-2 также влияет на функциональное состояние клетки, в том числе и посредством изменения транскрипции генов.
SARS-CoV-2 КАК ИСТОЧНИК микроРНК И РЕГУЛЯТОР ЭКСПРЕССИИ микроРНК ОРГАНИЗМА-ХОЗЯИНА
Известно, что инфицирование вирусами может приводить к изменению экспрессии генов, включая гены микроРНК, в клетках организма-хозяина [94–96]. Так, обнаружены различия в уровне экспрессии 20 микроРНК в сыворотке крови здоровых индивидов и больных гепатитом С, в том числе и в уровне hsa-miR-23b, специфично связывающейся с геномом SARS-CoV-2 (ее уровень снижен при гепатите С) [96].
In vitro показано, что энтеровирус человека типа 71 (HEV71), который рассматривается в качестве ведущей причины вирусного энцефалита у детей в большинстве азиатских стран, изменял экспрессию 69 микроРНК в клетках нейробластомы, среди которых – hsa-miR-1246 [94]. В этом же исследовании показано, что индуцируемая HEV71 hsa-miR-1246 регулирует уровень экспрессии гена DLG3, ассоциированного с неврологическими расстройствами [94]. Этот ген экспрессируется в разных тканях, в том числе в лобной коре головного мозга [97], он необходим для процессов обучения [98]. С учетом этого сделан вывод, что hsa-miR-1246 может быть вовлечена в патогенез вирусного энцефалита, вызванного HEV71 [94].
У пациентов с СOVID-19 встречаются неврологические нарушения, такие как головная боль и головокружение, энцефалопатия и делирий, а также нарушение мозгового кровообращения, синдром Гийена–Барре, острый поперечный миелит и острый энцефалит [99]. Поэтому не исключено, что вирус SARS-CoV-2 сам “регулирует уровень” hsa-miR-1246, изменение экспрессии которой часто регистрируется при заболеваниях, в том числе влияющих на характер течения COVID-19 (табл. 2).
Интерес вызывают исследования, посвященные анализу влияния SARS-CoV-2 на экспрессию генов в инфицированных клетках. Показано, что заражение клеток бронхиального эпителия человека вирусом SARS-CoV-2 изменяет экспрессию 327 генов, продукты которых участвуют в воспалительном ответе и регуляции метаболизма [100].
Инфицирование первичных эпителиальных клеток легких человека вирусами MERS-CoV и SARS-CoV-2 вызвало изменение более чем в 2 раза экспрессии 127 и 50 генов (из 44556 проанализированных) соответственно, причем лишь восемь генов оказались общими для двух инфекций, а изменение их экспрессии – однонаправленным [101]. Это дополнительно указывает на специфичность взаимоотношений SARS-CoV-2 с инфицированными клетками человека по сравнению с другими коронавирусами.
Изучение микроРНК в образцах крови пациентов со среднетяжелым и тяжелым течением COVID-19 позволило выделить четыре группы, различающихся уровнями экспрессии, которые, по-видимому, могут служить биомаркерами развития данного заболевания [9]. В первую группу вошли hsa-miR-146a-5p, hsa-miR-21-5p и hsa-miR-142-3p, которые постоянно подавляются; во вторую – микроРНК (включая hsa-miR-3605-3p), которые постоянно активируются; к третьей отнесены микроРНК, которые активируются только при тяжелой форме COVID-19 – в том числе hsa-miR-15b-5p, hsa-miR-486-3p и hsa-miR-486-5p; в четвертую группу вошли микроРНК, подавляемые только при тяжелом течении инфекции – в том числе hsa-miR-181a-2-3p, hsa-miR-31-5p и hsa-miR-99a-5p. Четыре микроРНК – hsa-miR-146a-5p, hsa-miR-21-5p, hsa-miR-142-3p и hsa-miR-15b-5p – авторами процитированного исследования определены в качестве потенциальных участников патогенеза инфекционного заболевания, вызываемого SARS-CoV-2. Некоторые из этих результатов согласуются с наблюдениями других исследователей. Так, низкий уровень hsa-miR-146a-5p выявлен в крови пациентов, не отвечающих на лечение COVID-19 тоцилизумабом, а самый низкий уровень был у лиц с неблагоприятным исходом коронавирусной инфекции [84].
Среди микроРНК, уровень которых изменялся в крови пациентов с COVID-19 [9], одна – hsa-miR-142-3p – отнесена к категории SARS-CoV-2 специфичных (табл. 1). Отмечены разнонаправленные изменения уровня этой микроРНК при заболеваниях (табл. 2). В частности, снижение уровня hsa-miR-142-3p зарегистрировано при некоторых патологиях сердца [63], а увеличение – при осложнениях сахарного диабета [71]. Отсюда следует, что если даже геном SARS-CoV-2 имеет мишени для микроРНК, то для оценки патогенетической значимости данных молекул важно принимать во внимание то, какую функцию выполняет эта микроРНК в организме и насколько создаваемые при ее участии условия благоприятны для инфицирования. Можно предположить, что именно низкий уровень hsa-miR-142-3p благоприятствует инфицированию SARS-CoV-2. Кроме того, нельзя исключать, что значение тех или иных микроРНК для развития COVID-19 (протективное/неблагоприятное/нейтральное) может изменяться в зависимости от стадии и клинической картины болезни.
Коронавирусы, в том числе и SARS-CoV-2, могут модифицировать профили микроРНК клеток организма-хозяина, действуя как губки для микроРНК человека, чтобы таким образом облегчить репликацию вируса и/или избегание иммунных ответов организма-хозяина [29, 102]. Такая возможность обусловлена не только комплементарностью генома SARS-CoV-2 и ряда микроРНК человека, но и высоким содержанием вирусной РНК в клетке (варьирует от 0.1 до 50% от общей клеточной РНК [102], тогда как доля микроРНК хозяина составляет всего около 0.01% (цитируется по [29]).
Помимо микроРНК человека, тем же путем (выступая в качестве своеобразной губки) геном SARS-CoV-2 может блокировать РНК-связывающие белки, играющие важную роль в посттрансляционных регуляторных сетях в клетках человека [100]. Существует также мнение, что путем соединения генома SARS-CoV-2 с РНК-связывающими белками клеток человека вирус уходит от действующих в клетках механизмов разрушения РНК, в том числе и с участием микроРНК человека [22]. Иными словами, геном SARS-CoV-2 посредством исключения из физиологических процессов инфицированной клетки микроРНК, мРНК и/или белковых молекул, потенциально значимых для определения ответа на инфицирование, создает благоприятные условия для размножения и распространения в организме человека. В частности, сверхэкспрессию гена CSF1 в альвеолярных и бронхиальных эпителиальных клетках человека после заражения SARS-CoV-2 объясняли тем, что геном коронавируса (и, в частности, ген S-белка) выступает в качестве РНК, конкурирующей с hsa-miR-1207-5p, имеющей сайты связывания на мРНК гена CSF1 [103]. Предполагается, что высокий уровень экспрессии CSF1 в эпителиальных клетках может способствовать неконтролируемому воспалению в наиболее тяжелых случаях COVID-19 [103].
РНК-содержащие вирусы способны формировать вирусные микроРНК (v-miRNA, обозначают также как малые вирусные РНК – svRNA) и, соответственно, принимать участие в эпигенетической регуляции функционирования генома зараженных клеток [10, 15, 19, 95, 104]. Происходящие из геномной области nsp3 и гена, кодирующего N‑белок, малые вирусные РНК SARS-CoV размером 18–22 н. (svRNA-N) обладали патогенетической значимостью, так как ингибирование in vivo svRNA-N значительно снижало степень поражения легких и экспрессию провоспалительных цитокинов у зараженных SARS-CoV мышей [95]. Примечательно, что биогенез данных вирусных РНК зависел от степени репликации вируса.
Накапливаются данные, полученные с использованием вычислительных подходов, о наличии в геноме SARS-CoV-2 участков, способных продуцировать микроРНК, потенциально значимых для эпигенетической регуляции генома человека. Так, по данным Khan M.A. и соавт. [19], геномы SARS-CoV и SARS-CoV-2 могут продуцировать 126 и 170 зрелых вирусных микроРНК соответственно. Мишенями этих микроРНК вирусов SARS-CoV и SARS-CoV-2 служат 5292 и 6369 генов человека соответственно, из которых только 2992 общие. МикроРНК SARS-CoV-2 имели мишени не только на генах, способствующих избеганию иммунного ответа хозяина, но и на генах, продукты которых участвуют в развитии сердца и головного мозга, в сигнальном пути инсулина и ряде других [19]. Предполагается, что это может определять особенности клинической картины у пациентов с коморбидными заболеваниями сердечно-сосудистой, эндокринной системы и т.д.
В другом исследовании [15] в геноме SARS-CoV-2 определены 29 последовательностей, потенциально выступающих в качестве предшественников вирусных микроРНК, мишенями которых могут быть 1367 генов человека. Среди генов-мишеней этих вирусных микроРНК оказались мРНК белков, участвующих в важнейших клеточных процессах, таких как транскрипция, метаболизм, система защиты, сигнальные пути Wnt и EGFR. Например, 96 генов-мишеней вирусных микроРНК участвуют в регуляции транскрипции генов в клетках человека. В число этих генов входят гены, кодирующие компоненты медиаторного комплекса (MED1, MED9, MED12L, MED19), основные факторы транскрипции (TAF4, TAF5, TAF7L) и сайт-специфические факторы транскрипции (STAT1) и др.
Только шесть предполагаемых вирусных микроРНК, имеющих мишени на генах, связанных с пролиферацией, дифференцировкой, передачей сигналов, старением клеток, а также вовлеченных в регуляцию иммунного ответа (TNF-сигнальный путь, путь передачи сигналов хемокинов), рассматриваются в качестве ключевых в развитии цитокинового шторма [10]. Высокий консерватизм, характерный для этих вирусных микроРНК, позволяет отнести их к числу перспективных для разработки вакцин.
Опубликованы экспериментальные исследования, свидетельствующие о возможности продукции зрелых микроРНК-подобных последовательностей SARS-CoV-2, способных влиять на экспрессию генов зараженных клеток. Так, Merino G.A. и соавт. [105] экспериментально (с помощью РНК-секвенирования) подтверждена экспрессия восьми таких последовательностей в культуре клеток рака легкого человека (линия Calu-3), инфицированных SARS-CoV-2. Примечательно, что эти вирусные микроРНК отличались по структуре от известных микроРНК человека, но потенциально имели мишени на 109 генах, дифференциально экспрессирующихся после заражения SARS-CoV-2, 28 из которых подавлялись в клетках, инфицированных вирусами.
Таким образом, несмотря на непродолжительное время исследований, накапливаются данные об участии (как потенциальном, так и доказанном) микроРНК человека и генома/микроРНК SARS-CoV-2 в инфицировании и развитии клинической картины COVID-19. Эти данные свидетельствуют о сложности выявления микроРНК, критичных для данных процессов. Установлены десятки микроРНК, которые потенциально могут связываться с геномом SARS-CоV-2. Разнонаправленные изменения экспрессии этих микроРНК регистрируются при разных заболеваниях, в том числе и при патологиях, определяющих риск инфицирования SARS-CoV-2 и тяжесть течения COVID-19.
В то же время изменения экспрессии микроРНК при патологиях не всегда могут однозначно трактоваться как благоприятный или неблагоприятный фактор риска развития и определения характера течения COVID-19. В данном случае следует учитывать функциональную роль каждой отдельной микроРНК (в том числе, и с точки зрения создания благоприятных условий для инфицирования SARS-CoV-2), а также то, какой процесс они маркируют (нормальное физиологическое состояние, стадию патологического процесса, тяжесть течения, ответ на лекарственные препараты и т.д.). Важно принимать во внимание и то, что SARS-CoV-2 также может влиять на функционирование зараженных клеток и организма в целом, изменять уровень экспрессии генов в клетках, а также продуцировать вирусные микроРНК, которые могут участвовать в эпигенетической регуляции генома зараженных клеток.
Написание обзора не потребовало специального финансирования.
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Ejaz H., Alsrhani A., Zafar A., Javed H., Junaid K., Abdalla A.E., Abosalif K.O.A., Ahmed Z., Younas S. (2020) COVID-19 and comorbidities: deleterious impact on infected patients. J. Infect. Publ. Hlth. 13(12), 1833–1839.
Fulzele S., Sahay B., Yusufu I., Lee T.J., Sharma A., Kolhe R., Isales C.M. (2020) COVID-19 virulence in aged patients might be impacted by the host cellular microRNAs abundance/profile. Aging Dis. 11(3), 509–522.
Callender L.A., Curran M., Bates S.M., Mairesse M., Weigandt J., Betts C.J. (2020) The impact of pre-existing comorbidities and therapeutic interventions on COVID-19. Front. Immunol. 11, 1991.
Jutzeler C.R., Bourguignon L., Weis C.V., Tong B., Wong C., Rieck B., Pargger H., Tschudin-Sutter S., Egli A., Borgwardt K., Walter M. (2020) Comorbidities, clinical signs and symptoms, laboratory findings, imaging features, treatment strategies, and outcomes in adult and pediatric patients with COVID-19: A systematic review and meta-analysis. Travel. Med. Infect. Dis. 37, 101825.
Murk W., Gierada M., Fralick M., Weckstein A., Klesh R., Rassen J.A. (2021) Diagnosis-wide analysis of COVID-19 complications: an exposure-crossover study. CMAJ. 193(1), E10–E18.
Chang W.T., Toh H.S., Liao C.T., Yu W.L. (2021) Cardiac Involvement of COVID-19: a comprehensive review. Am. J. Med. Sci. 361(1), 14–22.
Avila J., Long B., Holladay D., Gottlieb M. (2021) Thrombotic complications of COVID-19. Am. J. Emerg. Med. 39, 213–218.
Collantes M., Espiritu A.I., Sy M., Anlacan V., Jamora R. (2021). Neurological manifestations in COVID-19 infection: a systematic review and meta-analysis. Can. J. Neurol. Sci. 48(1), 66–76.
Tang H., Gao Y., Li Z., Miao Y., Huang Z., Liu X., Xie L., Li H., Wen W., Zheng Y., Su W. (2020) The noncoding and coding transcriptional landscape of the peripheral immune response in patients with COVID-19. Clin. Transl. Med. 10(6), e200.
Satyam R., Bhardwaj T., Goel S., Jha N.K., Jha S.K., Nand P., Ruokolainen J., Kamal M.A., Kesari K.K. (2021) miRNAs in SARS-CoV-2: a spoke in the wheel of pathogenesis. Curr. Pharm. Des. 27(13), 1628–1641.
Chow J.T., Salmena L. (2020) Prediction and analysis of SARS-CoV2-targeting microRNA in human lung epithelium. Genes. 11(9), 1002.
Jafarinejad-Farsangi S., Jazi M.M., Rostamzadeh F., Hadizadeh M. (2020) High affinity of host human microRNAs to SARS-CoV-2 genome: an in silico analysis. Noncoding RNA Res. 5(4), 222–231.
Guo L., Yu J., Yu H., Zhao Y., Chen S., Xu C., Chen F. (2015) Evolutionary and expression analysis of miR-#-5p and miR-#-3p at the miRNAs/isomiRs levels. Biomed. Res. Int. 2015, 168358.
Guterres A., de Azeredo Lima C.H., Miranda R.L., Gadelha M.R. (2020) What is the potential function of microRNAs as biomarkers and therapeutic targets in COVID-19. Infect. Genet. Evol. 85, 104417.
Saçar Demirci M.D., Adan A. (2020) Computational analysis of microRNA-mediated interactions in SARS-CoV-2 infection. Peer. J. 8, e9369.
Huang Z., Shi J., Gao Y., Cui C., Zhang S., Li J., Zhou Y., Cui Q. (2019). HMDD v3.0: a database for experimentally supported human microRNA-disease associations. Nucl. Acids Res. 47(D1), D1013–D1017.
Zhao X., Wang Y., Sun X. (2020) The functions of microRNA-208 in the heart. Diabetes Res. Clin. Pract. 160, 108004.
Chan A.P., Choi Y., Schork N.J. (2020) Conserved genomic terminals of SARS-CoV-2 as coevolving functional elements and potential therapeutic targets. mSphere. 5(6), e00754-20.
Khan M.A., Sany M., Islam M.S., Islam A. (2020) Epigenetic regulator miRNA pattern differences among SARS-CoV, SARS-CoV-2, and SARS-CoV-2 world-wide isolates delineated the mystery behind the epic pathogenicity and distinct clinical characteristics of pandemic COVID-19. Front. Genet. 11, 765.
Abu-Izneid T., AlHajri N., Mohammed Ibrahim A., Noushad Javed M., Mustafa Salem K., Hyder Pottoo F., Amjad Kamal M. (2021) Micro-RNAs in the regulation of immune response against SARS-CoV-2 and other viral infections. J. Adv. Res. 30, 133–145.
Aydemir M.N., Aydemir H.B., Korkmaz E.M., Budak M., Cekin N., Pinarbasi E. (2021) Computationally predicted SARS-COV-2 encoded microRNAs target NFKB, JAK/STAT and TGFB signaling pathways. Gene Rep. 22, 101012.
Mukherjee M., Goswami S. (2020) Global cataloguing of variations in untranslated regions of viral genome and prediction of key host RNA binding protein-microRNA interactions modulating genome stability in SARS-CoV-2. PLoS One. 15(8), e0237559.
Farshbaf A., Mohtasham N., Zare R., Mohajertehran F., Rezaee S.A. (2021) Potential therapeutic approaches of microRNAs for COVID-19: challenges and opportunities. J. Oral. Biol. Craniofac. Res. 11(2), 132–137.
Girardi E., López P., Pfeffer S. (2018) On the importance of host microRNAs during viral infection. Front. Genet. 9, 439.
Ludwig N., Leidinger P., Becker K., Backes C., Fehlmann T., Pallasch C., Rheinheimer S., Meder B., Stähler C., Meese E., Keller A. (2016) Distribution of miRNA expression across human tissues. Nucl. Acids Res. 44(8), 3865–3877.
de Rie D., Abugessaisa I., Alam T., Arner E., Arner P., Ashoor H., Åström G., Babina M., Bertin N., Burroughs A.M., Carlisle A.J., Daub C.O., Detmar M., Deviatiiarov R., Fort A., Gebhard C., Goldowitz D., Guhl S., Ha T.J., Harshbarger J., Hasegawa A., Hashimoto K., Herlyn M., Heutink P., Hitchens K.J., Hon C.C., Huang E., Ishizu Y., Kai C., Kasukawa T., Klinken P., Lassmann T., Lecellier C.H., Lee W., Lizio M., Makeev V., Mathelier A., Medvedeva Y.A., Mejhert N., Mungall C.J., Noma S., Ohshima M., Okada-Hatakeyama M., Persson H., Rizzu P., Roudnicky F., Sætrom P., Sato H., Severin J., Shin J.W., Swoboda R.K., Tarui H., Toyoda H., Vitting-Seerup K., Winteringham L., Yamaguchi Y., Yasuzawa K., Yoneda M., Yumoto N., Zabierowski S., Zhang P.G., Wells C.A., Summers K.M., Kawaji H., Sandelin A., Rehli M., FANTOM Consortium, Hayashizaki Y., Carninci P., Forrest A.R.R., de Hoon M.J.L. (2017). An integrated expression atlas of miRNAs and their promoters in human and mouse. Nat. Biotechnol. 35(9), 872–878.
Sardar R., Satish D., Birla S., Gupta, D. (2020) Integrative analyses of SARS-CoV-2 genomes from different geographical locations reveal unique features potentially consequential to host-virus interaction, pathogenesis and clues for novel therapies. Heliyon. 6(9), e04658.
Pierce J.B., Simion V., Icli B., Pérez-Cremades D., Cheng H.S., Feinberg M.W. (2020) Computational analysis of targeting SARS-CoV-2, viral entry proteins ACE2 and TMPRSS2, and interferon genes by host microRNAs. Genes. 11(11), 1354.
Bartoszewski R., Dabrowski M., Jakiela B., Matalon S., Harrod K.S., Sanak M., Collawn J.F. (2020) SARS-CoV-2 may regulate cellular responses through depletion of specific host miRNAs. Am. J. Physiol. Lung Cell Mol. Physiol. 319(3), L444–L455.
Hosseini Rad Sm A., McLellan A.D. (2020) Implications of SARS-CoV-2 mutations for genomic RNA structure and host microRNA targeting. Int. J. Mol. Sci. 21(13), 4807.
Arisan E.D., Dart A., Grant G.H., Arisan S., Cuhadaroglu S., Lange S., Uysal-Onganer P. (2020) The prediction of miRNAs in SARS-CoV-2 genomes: hsa-miR databases identify 7 key miRs linked to host responses and virus pathogenicity-related KEGG pathways significant for comorbidities. Viruses. 12(6), 614.
Balmeh N., Mahmoudi S., Mohammadi N., Karabedianhajiabadi A. (2020) Predicted therapeutic targets for COVID-19 disease by inhibiting SARS-CoV-2 and its related receptors. Inform. Med. Unlocked. 20, 100407.
Kozomara A., Birgaoanu M., Griffiths-Jones S. (2019) miRBase: from microRNA sequences to function. Nucl. Acids Res. 47(D1), D155–D162.
Nersisyan S., Engibaryan N., Gorbonos A., Kirdey K., Makhonin A., Tonevitsky A. (2020) Potential role of cellular miRNAs in coronavirus-host interplay. Peer J. 8, e9994.
Naqvi A., Fatima K., Mohammad T., Fatima U., Singh I.K., Singh A., Atif S.M., Hariprasad G., Hasan G.M., Hassan M.I. (2020). Insights into SARS-CoV-2 genome, structure, evolution, pathogenesis and therapies: structural genomics approach. Biochim. Biophys. Acta Mol. Basis Dis. 1866(10), 165878.
Li J., Huang D.Q., Zou B., Yang H., Hui W.Z., Rui F., Yee N., Liu C., Nerurkar S.N., Kai J., Teng M., Li X., Zeng H., Borghi J.A., Henry L., Cheung R., Nguyen M.H. (2021). Epidemiology of COVID-19: a systematic review and meta-analysis of clinical characteristics, risk factors, and outcomes. J. Med. Virol. 93(3), 1449–1458.
Musri M.M., Coll-Bonfill N., Maron B.A., Peinado V.I., Wang R.S., Altirriba J., Blanco I., Oldham W.M., Tura-Ceide O., García-Lucio J., de la Cruz-Thea B., Meister G., Loscalzo J., Barberà J.A. (2018) MicroRNA dysregulation in pulmonary arteries from chronic obstructive pulmonary disease. relationships with vascular remodeling. Am. J. Respir. Cell Mol. Biol. 59(4), 490–499.
Sundar I.K., Li D., Rahman I. (2019) Small RNA-sequence analysis of plasma-derived extracellular vesicle miRNAs in smokers and patients with chronic obstructive pulmonary disease as circulating biomarkers. J. Extracell. Vesicles. 8(1), 1684816.
Cazorla-Rivero S., Mura-Escorche G., Gonzalvo-Hernández F., Mayato D., Córdoba-Lanús E., Casanova C. (2020) Circulating miR-1246 in the progression of chronic obstructive pulmonary disease (COPD) in patients from the BODE cohort. Int. J. Chron. Obstruct. Pulmon. Dis. 15, 2727–2737.
Shi L., Xin Q., Chai R., Liu L., Ma Z. (2015) Ectopic expressed miR-203 contributes to chronic obstructive pulmonary disease via targeting TAK1 and PIK3CA. Int. J. Clin. Exp. Pathol. 8(9), 10662–10670.
Wei C., Henderson H., Spradley C., Li L., Kim I.K., Kumar S., Hong N., Arroliga A.C., Gupta S. (2013) Circulating miRNAs as potential marker for pulmonary hypertension. PLoS One. 8(5), e64396.
Huber L.C., Ulrich S., Leuenberger C., Gassmann M., Vogel J., von Blotzheim L.G., Speich R., Kohler M., Brock M. (2015) Featured article: microRNA-125a in pulmonary hypertension: regulator of a proliferative phenotype of endothelial cells. Ex. Biol. Med. (Maywood). 240(12), 1580–1589.
Jardim M.J., Dailey L., Silbajoris R., Diaz-Sanchez D. (2012) Distinct microRNA expression in human airway cells of asthmatic donors identifies a novel asthma-associated gene. Am. J. Respir. Cell. Mol. Biol. 47(4), 536–542.
Ke X.F., Fang J., Wu X.N., Yu C.H. (2014) MicroRNA-203 accelerates apoptosis in LPS-stimulated alveolar epithelial cells by targeting PIK3CA. Biochem. Biophys. Res. Commun. 450(4), 1297–1303.
Marketou M.E., Kontaraki J.E., Maragkoudakis S., Patrianakos A., Konstantinou J., Nakou H., Vougia D., Logakis J., Chlouverakis G., Vardas P.E., Parthenakis F.I. (2018) MicroRNAs in peripheral mononuclear cells as potential biomarkers in hypertensive patients with heart failure with preserved ejection fraction. Am. J. Hypertens. 31(6), 651–657.
Huang X., Li Z., Bai B., Li X., Li Z. (2015) High expression of microRNA-208 is associated with cardiac hypertrophy via the negative regulation of the sex-determining region Y-box 6 protein. Exp., Ther. Med. 10(3), 921–926.
Kontaraki J.E., Marketou M.E., Parthenakis F.I., Maragkoudakis S., Zacharis E.A., Petousis S., Kochiadakis G.E., Vardas P.E. (2015) Hypertrophic and antihypertrophic microRNA levels in peripheral blood mononuclear cells and their relationship to left ventricular hypertrophy in patients with essential hypertension. J. Am. Soc. Hypertens. 9(10), 802–810.
Wang G., Kwan B.C., Lai F.M., Choi P.C., Chow K.M., Li P.K., Szeto C.C. (2010) Intrarenal expression of miRNAs in patients with hypertensive nephrosclerosis. Am. J. Hypertens. 23(1), 78–84.
Liu W., Zheng J., Dong J., Bai R., Song D., Ma X., Zhao L., Yao Y., Zhang H., Liu T. (2018) Association of miR-197-5p, a circulating biomarker for heart failure, with myocardial fibrosis and adverse cardiovascular events among patients with stage C or D heart failure. Cardiology. 141(4), 212–225.
Wong L.L., Armugam A., Sepramaniam S., Karolina D.S., Lim K.Y., Lim J.Y., Chong J.P., Ng J.Y., Chen Y.T., Chan M.M., Chen Z., Yeo P.S., Ng T.P., Ling L.H., Sim D., Leong K.T., Ong H.Y., Jaufeerally F., Wong R., Chai P., Low A.F., Lam C.S., Jeyaseelan K., Richards A.M. (2015) Circulating microRNAs in heart failure with reduced and preserved left ventricular ejection fraction. Eur. J. Heart Fail. 17(4), 393–404.
Hu Q., Luo W., Huang L., Huang R., Chen R. (2016) Apoptosis-related microRNA changes in the right atrium induced by remote ischemic perconditioning during valve replacement surgery. Sci. Rep. 6, 18959.
He X., Ji J., Wang T., Wang M.B., Chen X.L. (2017) Upregulation of circulating miR-195-3p in heart failure. Cardiology. 138(2), 107–114.
Liu H., Yang N., Fei Z., Qiu J., Ma D., Liu X., Cai G., Li S. (2016) Analysis of plasma miR-208a and miR-370 expression levels for early diagnosis of coronary artery disease. Biomed. Rep. 5(3), 332–336.
Liu W., Ling S., Sun W., Liu T., Li Y., Zhong G., Zhao D., Zhang P., Song J., Jin X., Xu Z., Song H., Li Q., Liu S., Chai M., Dai Q., He Y., Fan Z., Zhou Y.J., Li Y. (2015) Circulating microRNAs correlated with the level of coronary artery calcification in symptomatic patients. Sci. Rep. 5, 16099.
van Rooij E., Sutherland L.B., Liu N., Williams A.H., McAnally J., Gerard R.D., Richardson J.A., Olson E.N. (2006) A signature pattern of stress-responsive microRNAs that can evoke cardiac hypertrophy and heart failure. Proc. Natl. Acad. Sci. USA. 103(48), 18255–18260.
Zhang X., Ji R., Liao X., Castillero E., Kennel P.J., Brunjes D.L., Franz M., Möbius-Winkler S., Drosatos K., George I., Chen E.I., Colombo P.C., Schulze P.C. (2018) MicroRNA-195 regulates metabolism in failing myocardium via alterations in sirtuin 3 expression and mitochondrial protein acetylation. Circulation. 137(19), 2052–2067.
Weber K., Rostert N., Bauersachs S., Wess G. (2015) Serum microRNA profiles in cats with hypertrophic cardiomyopathy. Mol. Cell. Biochem. 402(1–2), 171–180.
Slagsvold K.H., Johnsen A.B., Rognmo O., Høydal M.A., Wisløff U., Wahba A. (2014) Mitochondrial respiration and microRNA expression in right and left atrium of patients with atrial fibrillation. Physiol. Genomics. 46(14), 505–511.
Cañón S., Caballero R., Herraiz-Martínez A., Pérez-Hernández M., López B., Atienza F., Jalife J., Hove-Madsen L., Delpón E., Bernad A. (2016) miR-208b upregulation interferes with calcium handling in HL-1 atrial myocytes: Implications in human chronic atrial fibrillation. J. Mol. Cell. Cardiol. 99, 162–173.
Long G., Wang F., Duan Q., Yang S., Chen F., Gong W., Yang X., Wang Y., Chen C., Wang D.W. (2012) Circulating miR-30a, miR-195 and let-7b associated with acute myocardial infarction. PLoS One. 7(12), e50926.
Boštjančič E., Brandner T., Zidar N., Glavač D., Štajer D. (2018) Down-regulation of miR-133a/b in patients with myocardial infarction correlates with the presence of ventricular fibrillation. Biomed. Pharmacother. 99, 65–71.
Li C., Fang Z., Jiang T., Zhang Q., Liu C., Zhang C., Xiang Y. (2013) Serum microRNAs profile from genome-wide serves as a fingerprint for diagnosis of acute myocardial infarction and angina pectoris. BMC Med. Genomics. 6, 16.
Nair N., Kumar S., Gongora E., Gupta S. (2013) Circulating miRNA as novel markers for diastolic dysfunction. Mol. Cell. Biochem. 376(1–2), 33–40.
Wang Y.F., Lian X.L., Zhong J.Y., Su S.X., Xu Y.F., Xie X.F., Wang Z.P., Li W., Zhang L., Che D., Yu L., Huang P., Jia H.L., Gu X.Q. (2019) Serum exosomal microRNA let-7i-3p as candidate diagnostic biomarker for Kawasaki disease patients with coronary artery aneurysm. IUBMB Life. 71(7), 891–900.
He L.P., Zhao X.S., He L.P. (2018) Abnormally expressed miR-23b in Chinese Mongolian at high cardiovascular risk may contribute to monocyte/macrophage inflammatory reaction in atherosclerosis. Biosci. Rep. 38(6), BSR20180673.
Zampetaki A., Kiechl S., Drozdov I., Willeit P., Mayr U., Prokopi M., Mayr A., Weger S., Oberhollenzer F., Bonora E., Shah A., Willeit J., Mayr M. (2010) Plasma microRNA profiling reveals loss of endothelial miR-126 and other microRNAs in type 2 diabetes. Circ. Res. 107(6), 810–817.
Zhao B., Li H., Liu J., Han P., Zhang C., Bai H., Yuan X., Wang X., Li L., Ma H., Jin X., Chu Y. (2016) MicroRNA-23b targets Ras GTPase-activating protein SH3 domain-binding protein 2 to alleviate fibrosis and albuminuria in diabetic nephropathy. J. Am. Soc. Nephrol. 27(9), 2597–2608.
Párrizas M., Brugnara L., Esteban Y., González-Franquesa A., Canivell S., Murillo S., Gordillo-Bastidas E., Cussó R., Cadefau J.A., García-Roves P.M., Servitja J.M., Novials A. (2015) Circulating miR-192 and miR-193b are markers of prediabetes and are modulated by an exercise intervention. J. Clin. Endocrinol. Metab. 100(3), E407–E415.
Kim H., Bae Y.U., Jeon J.S., Noh H., Park H.K., Byun D.W., Han D.C., Ryu S., Kwon S.H. (2019) The circulating exosomal microRNAs related to albuminuria in patients with diabetic nephropathy. J. Transl. Med. 17(1), 236.
Torella D., Ellison G.M., Torella M., Vicinanza C., Aquila I., Iaconetti C., Scalise M., Marino F., Henning B.J., Lewis F.C., Gareri C., Lascar N., Cuda G., Salvatore T., Nappi G., Indolfi C., Torella R., Cozzolino D., Sasso F.C. (2014) Carbonic anhydrase activation is associated with worsened pathological remodeling in human ischemic diabetic cardiomyopathy. J. Am. Heart Assoc. 3(2), e000434.
Friedrich J., Steel D.H.W., Schlingemann R.O., Koss M.J., Hammes H.P., Krenning G., Klaassen I. (2020) microRNA expression profile in the vitreous of proliferative diabetic retinopathy patients and differences from patients treated with anti-VEGF therapy. Transl. Vis. Sci. Technol. 9(6), 16.
Dubois-Camacho K., Diaz-Jimenez D., De la Fuente M., Quera R., Simian D., Martínez M., Landskron G., Olivares-Morales M., Cidlowski J.A., Xu X., Gao G., Xie J., Chnaiderman J., Soto-Rifo R., González M.J., Calixto A., Hermoso M.A. (2019) Inhibition of miR-378a-3p by inflammation enhances IL-33 levels: a novel mechanism of alarmin modulation in ulcerative colitis. Front. Immunol. 10, 2449.
Zhu S., Pan W., Song X., Liu Y., Shao X., Tang Y., Liang D., He D., Wang H., Liu W., Shi Y., Harley J.B., Shen N., Qian Y. (2012) The microRNA miR-23b suppresses IL-17-associated autoimmune inflammation by targeting TAB2, TAB3 and IKK-α. Nat. Med. 18(7), 1077–1086.
Krissansen G.W., Yang Y., McQueen F.M., Leung E., Peek D., Chan Y.C., Print C., Dalbeth N., Williams M., Fraser A.G. (2015) Overexpression of miR-595 and miR-1246 in the sera of patients with active forms of inflammatory bowel disease. Inflamm. Bowel Dis. 21(3), 520–530.
Méndez-Flores S., Furuzawa-Carballeda J., Hernández-Molina G., Ramírez-Martinez G., Regino-Zamarripa N.E., Ortiz-Quintero B., Jiménez-Alvarez L., Cruz-Lagunas A., Zúñiga J. (2019) MicroRNA expression in cutaneous lupus: a new window to understand its pathogenesis. Mediators Inflamm. 2019, 5049245.
Hiratsuka I., Yamada H., Munetsuna E., Hashi-moto S., Itoh M. (2016) Circulating microRNAs in Graves’ disease in relation to clinical activity. Thyroid. 26(10), 1431–1440.
Tsigaris P., Teixeira da Silva J.A. (2020) Smoking prevalence and COVID-19 in Europe. Nicotine Tob. Res. 22(9), 1646–1649.
Liu S., Cao Y., Du T., Zhi Y. (2020) Prevalence of comorbid asthma and related outcomes in COVID-19: a systematic review and meta-analysis. J. Allergy Clin. Immunol. Pract. 9(2), 693–701.
Wakabayashi M., Pawankar R., Narazaki H., Ueda T., Itabashi T. (2021) Coronavirus disease 2019 and asthma, allergic rhinitis: molecular mechanisms and host-environmental interactions. Curr. Opin. Allergy Clin. Immunol. 21(1), 1–7.
Inchley C.S., Sonerud T., Fjærli H.O., Nakstad B. (2015) Nasal mucosal microRNA expression in children with respiratory syncytial virus infection. BMC Infect. Dis. 15, 150.
Zhu X., Ge Y., Wu T., Zhao K., Chen Y., Wu B., Zhu F., Zhu B., Cui L. (2020) Co-infection with respiratory pathogens among COVID-2019 cases. Virus Res. 285, 198005.
Zhang H., Rostami M.R., Leopold P.L., Mezey J.G., O’Beirne S.L., Strulovici-Barel Y., Crystal R.G. (2020) Expression of the SARS-CoV-2 ACE2 receptor in the human airway epithelium. Am. J. Respir. Crit. Care Med. 202(2), 219–229.
Azevedo R.B., Botelho B.G., Hollanda J., Ferreira L., Junqueira de Andrade L.Z., Oei S., Mello T.S., Muxfeldt E.S. (2021) COVID-19 and the cardiovascular system: a comprehensive review. J. Hum. Hypertens. 35(1), 4–11.
Sabbatinelli J., Giuliani A., Matacchione G., Latini S., Laprovitera N., Pomponio G., Ferrarini A., Baroni S.S., Pavani M., Moretti M., Gabrielli A., Procopio A.D., Ferracin M., Bonafè M., Olivieri F. (2020) Decreased serum levels of the inflammaging marker miR-146a are associated with clinical response to tocilizumab in COVID-19 patients. Mech. Ageing Dev. 193, 111413.
Kabeerdoss J., Pilania R.K., Karkhele R., Kumar T.S., Danda D., Singh S. (2021) Severe COVID-19, multisystem inflammatory syndrome in children, and Kawasaki disease: immunological mechanisms, clinical manifestations and management. Rheumatol. Int. 41(1), 19–32.
Sokolovsky S., Soni P., Hoffman T., Kahn P., Scheers-Masters J. (2021) COVID-19 associated Kawasaki-like multisystem inflammatory disease in an adult. Am. J. Emerg. Med. 39, 253.e1–253.e2.
Alsaied T., Tremoulet A.H., Burns J.C., Saidi A., Dionne A., Lang S.M., Newburger J.W., de Ferranti S., Friedman K.G. (2021) Review of cardiac involvement in multisystem inflammatory syndrome in children. Circulation. 143(1), 78–88.
Galeotti C., Bayry J. (2020) Autoimmune and inflammatory diseases following COVID-19. Nat. Rev. Rheumatol. 16(8), 413–414.
Wolff D., Nee S., Hickey N.S., Marschollek M. (2021) Risk factors for Covid-19 severity and fatality: a structured literature review. Infection. 49 (1), 15–28.
Zhou Y., Chi J., Lv W., Wang Y. (2021) Obesity and diabetes as high-risk factors for severe coronavirus disease 2019 (COVID-19). Diabetes Metab. Res. Rev. 37(2), e3377.
Monteleone G., Ardizzone S. (2020) Are patients with inflammatory bowel disease at increased risk for COVID-19 infection? J. Crohns Colitis. 14(9), 1334–1336.
Favalli E.G., Ingegnoli F., De Lucia O., Cincinelli G., Cimaz R., Caporali R. (2020) COVID-19 infection and rheumatoid arthritis: faraway, so close! Autoimmun. Rev. 19(5), 102523.
Liu Y., Sawalha A.H., Lu Q. (2021) COVID-19 and autoimmune diseases. Curr. Opin. Rheumatol. 33(2), 155–162.
Xu L.J., Jiang T., Zhao W., Han J.F., Liu J., Deng Y.Q., Zhu S.Y., Li Y.X., Nian Q.G., Zhang Y., Wu X.Y., Qin E.D., Qin C.F. (2014) Parallel mRNA and microRNA profiling of HEV71-infected human neuroblastoma cells reveal the up-regulation of miR-1246 in association with DLG3 repression. PLoS One. 9(4), e95272.
Morales L., Oliveros J.C., Fernandez-Delgado R., tenOever B.R., Enjuanes L., Sola I. (2017) SARS-CoV-encoded small RNAs contribute to infection-associated lung pathology. Cell Host Microbe. 21, 344–355.
El-Hefny M., Fouad S., Hussein T., Abdel-Hameed R., Effat H., Mohamed H., Abdel Wahab A.H. (2019) Circulating microRNAs as predictive biomarkers for liver disease progression of chronic hepatitis C (genotype-4) Egyptian patients. J. Med. Virol. 91(1), 93–101.
Bastian F.B., Roux J., Niknejad A., Comte A., Fonseca Costa S.S., de Farias T.M., Moretti S., Parmentier G., de Laval V.R., Rosikiewicz M., Wollbrett J., Echchiki A., Escoriza A., Gharib W.H., Gonzales-Porta M., Jarosz Y., Laurenczy B., Moret P., Person E., Roelli P., Sanjeev K., Seppey M., Robinson-Rechavi M. (2021) The Bgee suite: integrated curated expression atlas and comparative transcriptomics in animals. Nucl. Acids Res. 49(D1), D831–D847.
UniProt Consortium (2021) UniProt: the universal protein knowledgebase in 2021. Nucl. Acids Res. 49(D1), D480–D489.
Ahmad I., Rathore F.A. (2020) Neurological manifestations and complications of COVID-19: a literature review. J. Clin. Neurosci. 77, 8–12.
Srivastava R., Daulatabad S.V., Srivastava M., Janga S.C. (2020) Role of SARS-CoV-2 in altering the RNA-binding protein and miRNA-directed post-transcriptional regulatory networks in humans. Int. J. Mol. Sci. 21(19), 7090.
Jang Y., Seo S.H. (2020) Gene expression pattern differences in primary human pulmonary epithelial cells infected with MERS-CoV or SARS-CoV-2. Arch. Virol. 165(10), 2205–2211.
Blanco-Melo D., Nilsson-Payant B.E., Liu W.C., Uhl S., Hoagland D., Møller R., Jordan T.X., Oishi K., Panis M., Sachs D., Wang T.T., Schwartz R.E., Lim J.K., Albrecht R.A., tenOever B.R. (2020) Imbalanced host response to SARS-CoV-2 drives development of COVID-19. Cell. 181(5), 1036–1045.e9.
Bertolazzi G., Cipollina C., Benos P.V., Tumminello M., Coronnello C. (2020) miR-1207-5p can contribute to dysregulation of inflammatory response in COVID-19 via targeting SARS-CoV-2 RNA. Front Cell Infect. Microbiol. 10, 586592.
Mishra R., Kumar A., Ingle H., Kumar H. (2020) The interplay between viral-derived miRNAs and host immunity during infection. Front. Immunol. 10, 3079.
Merino G.A., Raad J., Bugnon L.A., Yones C., Kamenetzky L., Claus J., Ariel F., Milone D.H., Stegmayer G. (2021) Novel SARS-CoV-2 encoded small RNAs in the passage to humans. Bioinformatics. 36(24), 5571–5581.
Дополнительные материалы
- скачать ESM.docx
- Таблица S1. Методические подходы, использованные в исследованиях, посвященных поиску микроРНК, потенциально способных связываться с геномами SARS-CoV-2 и других коронавирусов
Список литературы.
Инструменты
Молекулярная биология


