Молекулярная биология, 2022, T. 56, № 1, стр. 126-134
Минорный T-аллель однонуклеотидного полиморфизма rs13360222 снижает активность энхансера гена HAVCR2 в клеточной модели макрофагов человека
А. Н. Уварова a, b, А. С. Устюгова a, Н. А. Митькин a, А. М. Шварц a, К. В. Корнеев a, Д. В. Купраш a, b, *
a Центр высокоточного редактирования и генетических технологий для биомедицины, Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Биологический факультет Московского государственного университета им. М.В. Ломоносова
119234 Москва, Россия
* E-mail: kuprash@gmail.com
Поступила в редакцию 16.08.2021
После доработки 08.09.2021
Принята к публикации 08.09.2021
- EDN: CDJJUH
- DOI: 10.31857/S0026898422010116
Аннотация
Рецептор TIM-3, кодируемый геном HAVCR2 (Hepatitis A Virus Cellular Receptor 2), относится к группе иммунологических “контрольных точек” (checkpoints) и выполняет важную роль в предотвращении развития аутоиммунных реакций. Этот рецептор экспрессируется на поверхности различных иммуноцитов. С одной стороны, роль TIM-3, представленного на Т-клетках, активно изучается в контексте поиска перспективных терапевтических мишеней при иммунотерапии рака, с другой стороны, его функции в клетках миелоидного ряда остаются малоизученными. Нами проведен делеционный анализ промоторной области гена HAVCR2, функционально охарактеризован его энхансер и изучено влияние ряда однонуклеотидных полиформизмов (SNP) на активность этих регуляторных элементов в релевантной модели макрофагоподобных клеток человека – активированной моноцитарной линии U937. Показано, что ассоциированные с развитием ряда патологий SNP rs10515746(А) и rs4704853(А), находящиеся в промоторе гена HAVCR2, не влияют на активность этого регуляторного элемента в активированных моноцитах. Однако в энхансере, в третьем интроне гена, находится SNP rs13360222, минорный T-вариант которого значимо снижает способность энхансера активировать промотор HAVCR2, предположительно вследствие ослабления связывания ядерного рецептора ESR2.
ВВЕДЕНИЕ
Иммунологическая контрольная точка (immunological checkpoint) TIM-3 ‒ белок, который изначально был охарактеризован как маркер T-лимфоцитов, продуцирующих интерферон-γ (IFNγ). TIM-3 играет ключевую роль в ингибировании Th1 иммунного ответа и продукции провоспалительных цитокинов, таких как фактор некроза опухолей (TNF) и IFNγ, поэтому дисрегуляция его экспрессии может приводить к развитию аутоиммунных патологий [1]. TIM-3 представлен на Т-лимфоцитах, а также на клетках врожденного иммунитета, таких как макрофаги, дендритные клетки, естественные киллерные (NK) клетки и натуральные Т-киллеры (NKT) [2]. T-клеточный TIM-3 активно исследуют в качестве мишени при иммунотерапии онкологических заболеваний, однако его функции в других иммуноцитах изучены хуже. Появляется все больше данных по ингибиторной роли TIM-3 и в клетках миелоидного ряда дифференцировки. Например показано, что TIM-3, при участии своего лиганда фосфатидилсерина, опосредует фагоцитоз апоптотических клеток и стимулирует кросс-презентацию антигена CD8+ дендритными клетками in vitro и in vivo [3]. Другой лиганд TIM-3 – HMGB1 – играет важную роль в осуществлении транспорта нуклеиновых кислот в эндосомы для дальнейшего их распознавания рецепторами врожденного иммунитета в дендритных клетках. Так, инфильтрирующие опухоль дендритные клетки экспрессируют повышенный уровень TIM-3, который, связываясь с HMGB1, блокирует его функции и таким образом супрессирует опосредованный паттернраспознающими рецепторами иммунный ответ против опухолевых нуклеиновых кислот [4].
Yang с соавт. [5] показали, что TIM-3 ‒ негативный регулятор опосредованного Toll-подобными рецепторами (TLR) иммунного ответа макрофагов. Подавление экспрессии TIM-3 приводит к повышенной активации макрофагов в экспериментальной мышиной модели сепсиса, что способствует формированию системного воспалительного ответа, тогда как сверхэкспрессия TIM-3 в макрофагах подавляет TLR-зависимую продукцию провоспалительных цитокинов. В NK-клетках TIM-3 служит маркером их активации или созревания и может подавлять NK-зависимую цитотоксичность [6]. У пациентов с тяжелой формой меланомы подавление TIM-3 способствует восстановлению активности NK-клеток [7]. В связи c широким спектром функций TIM-3 на разных типах клеток детальное изучение его роли в биологии не только Т-лимфоцитов, но и других популяций иммуноцитов, в частности макрофагов, вовлеченных в противоопухолевый иммунный надзор, относится к важной задаче современной онкоиммунологии.
Нами исследовано два однонуклеотидных полиморфизма (SNP) промоторной области гена HAVCR2, ассоциированных с риском развития различных патологий: rs10515746 и rs4704853. Высокая частота минорного аллеля rs10515746(T) наблюдается в группе пациентов с раком желудка, эссенциальной тромбоцитемией, ревматоидным артритом и болезнью Крона [8‒11]. Частота минорного варианта rs4704853(А) бывает повышена у пациентов с раком молочной железы по сравнению с представителями контрольной группы [12]. В результате проведенного анализа регуляции экспрессии репортерного гена мы выяснили, что эти SNP не оказывают влияния на активность промотора HAVCR2 в клеточной модели активированных моноцитов человека. Кроме того, мы функционально охарактеризовали энхансерную регуляторную область, находящуюся в третьем интроне гена HAVCR2, и выявили влияние минорного варианта SNP rs13360222(Т) в этой области на способность энхансера активировать промотор TIM-3.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Биоинформатическое определение границ регуляторных последовательностей и выбор SNP. Для определения границ промоторов и энхансеров использовали сервис USCS Genome Browser, сборка GRCh37/hg19 (http://genome.ucsc.edu), содержащий большой набор эпигеномных данных ENCODE [13]. Основными параметрами для выбора границ регуляторных последовательностей было наличие пиков ацетилирования Lys27 гистона H3 (H3K27Ac), Lys9 гистона H3 (H3K9Ac), моно- и триметилирования Lys4 гистона H3 (H3K4me1, H3K4me3), а также данные по чувствительности к ДНКазе I и наличию сайтов посадки транскрипционных факторов (ТФ), определенных ранее методом иммунопреципитации (ChIP-Seq) [14]. Расположение полиморфизмов в сайтах посадки ТФ оценивали с помощью ресурса GTRD [15] на основе данных ChIP-Seq. Влияние альтернативных вариантов полиморфизмов на вероятность связывания релевантных ТФ проводили с помощью сервиса PERFECTOS-APE [16] с использованием баз данных HOCOMOCO11 [17], JASPAR [18] и SELEX [19]. Учитывали факторы, для которых различие в аллеле SNP давало наименьшее p-значение для мотива связывания (p-value < 0.0005) и наибольшее изменение силы связывания ТФ (fold change > 5.0).
Создание репортерных конструкций. Для создания репортерных конструкций разные варианты промотора (1 992, 1 505, 453, 199, 97, 32 п.н.) и предполагаемый энхансер (1 025 п.н.) гена HAVCR2, а также контрольный фрагмент схожего с энхансером размера (1 080 п.н.) из первого интрона гена STAT3 (не проявляет энхансерной активности) [20] амплифицировали методом полимеразной цепной реакции (ПЦР) с использованием очищенной геномной ДНК человека (“Евроген”, Россия) и пар праймеров, указанных в табл. 1. Контрольную последовательность использовали, чтобы исключить влияние размера плазмиды на уровень сигнала в люциферазных тестах. Последовательности промотора были клонированы в вектор pGL3-basic (“Promega”, США) по сайтам HindIII/NcoI перед геном люциферазы. Последовательности энхансера и контрольного участка клонировали после гена люциферазы по сайтам BamHI/SalI. Растворы плазмид получали с помощью набора Plasmid Midiprep (“Евроген”). Для получения последовательностей промоторов и энхансеров, содержащих альтернативные аллели полиморфизмов, использовали праймеры с точечной мутацией на месте SNP по методу, описанному ранее [21, 22]. Все полученные конструкции верифицировали секвенированием по Сэнгеру.
Таблица 1.
Олигонуклеотиды, использованные при получении репортерных плазмид, содержащих исследуемые промоторы и энхансеры
| Название олигонуклеотида | Последовательность (5' → 3') |
|---|---|
| Праймеры для клонирования регуляторных элементов гена HAVCR2 | |
| promHAVCR2-nested-F | GAAATGAGCAGAAAACAAAGTGGT |
| promHAVCR2-nested-R | CATATTCCTGCTCCCCGACA |
| promHAVCR2-BsaI(NcoI)-R | TTAAGGTCTCCCATGGAGCTTGCAGAAGAAAAG |
| promHAVCR2-F-BsaI(HindIII)-1992 | TTTTGGTCTCAAGCTTGAACACAGGAGGCGGA |
| promHAVCR2-F-BsaI(HindIII)-1505 | TTTTGGTCTCAAGCTTAACAGCCTGACCAACATGGAG |
| promHAVCR2-F-BsaI(HindIII)-453 | TTTTGGTCTCAAGCTTTGATTTGAGTGAATGAATCCATG |
| promHAVCR2-F-BsaI(HindIII)-199 | TTTTGGTCTCAAGCTTAATGTGACTGTAGACCTGGCA |
| promHAVCR2-F-BsaI(HindIII)-97 | TTTTGGTCTCAAGCTTATTGTGGAGTAGACAGTTGGA |
| promHAVCR2-F-BsaI(HindIII)-32 | TTTTGGTCTCAAGCTTAGGTGTCCTCTGACTTTTCTTC |
| enhHAVCR2-F-BamHI | TTTTGGATCCCAATGGTGTGATCTCGGCTCA |
| enhHAVCR2-R-SalI | TTTTGTCGACGAGAAGGGAGACAGGGTTGC |
| ContrEnh-F-BamHI | AGGATCCGGATTACAGGTGTATTTCACCAT |
| ContrEnh-R-SalI | TATGTCGACGTTGATGTAATTCCTTTAAATCTAT |
| Праймеры для введения минорных вариантов полиморфизмов | |
| Overlap_ rs10515746_promHAVCR2_T_F | GGGAGTTGCTATGGTCTGTAAATGTGAG |
| Overlap_rs10515746_promHAVCR2_A_R | ACAGACCATAGCAACTCCCAGCATAAGC |
| Overlap_ rs4704853_promHAVCR2_T_F | GCCTCTTGGGGTAGGGGAGAGG |
| Overlap_rs4704853_promHAVCR2_A_R | CTACCCCAAGAGGCTTTGGCCATGA |
| Overlap_rs13360222_enhHAVCR2_A_F | GCTGGGCAACATGACCCTCTTCATA |
| Overlap_rs13360222_enhHAVCR2_T_R | AGGGTCATGTTGCCCAGCCACC |
| Overlap_rs73308313_enhHAVCR2_T_F | CTTGGGTTAGTCATTTCACTTCCCTAAT |
| Overlap_rs73308313_enhHAVCR2_A_R | GGAAGTGAAATGACTAACCCAAGGTC |
Клеточные линии. Моноцитарную клеточную линию U937 [23] культивировали в среде RPMI-1640 с аланил-глутамином (“ПанЭко”, Россия) с добавлением 10% эмбриональной телячьей сыворотки (FBS; “Biological Industries”, Израиль), смеси антибиотиков (100 U/мл пенициллин и 100 мкг/мл стрептомицин), 10 мМ Na-соли HEPES (“ПанЭко”) и 1% раствора незаменимых аминокислот MEM NEAA (“Gibco”, США) при 37°C и 5% CO2. Активацию моноцитарной линии U937 проводили в течение 24 ч при добавлении форбол-12-миристат-13-ацетата (PMA), как описано ранее [24].
Люциферазный тест. Трансфекцию клеток исследуемыми репортерными плазмидами проводили методом электропорации с использованием Neon™ Transfection System (“Thermo Fisher Scientific”, США). В качестве внутреннего контроля использовали плазмиду, содержащую ген люциферазы Renilla pRL-CMV (“Promega”, США). Через 24 ч после трансфекции клетки лизировали с использованием набора Dual-Luciferase Reporter Assay System (“Promega”) и с помощью люминометра 20/20n (“Turner BioSystems”, США) измеряли сигнал от люцифераз Lampyridae и Renilla, как описано ранее [25]. Сигнал от люциферазы Renilla использовали для нормализации на внешние вариабельные параметры: эффективность трансфекции и лизиса клеток и т.п.
Статистическая обработка результатов. Статистический анализ данных проводили с помощью программного обеспечения GraphPad Prism (версия 6.01 для Windows, GraphPad Software, США; www.graphpad.com). Для определения степени достоверности использовали двусторонний непарный t-критерий Стьюдента. Данные были получены не менее чем в трех независимых экспериментах и представлены как среднее значение ± стандартная ошибка среднего (SEM). Значимое различие идентифицировали при значении P < 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Альтернативные варианты SNP rs10515746 и rs4704853 не влияют на активность промотора HAVCR2 в активированных моноцитах U937
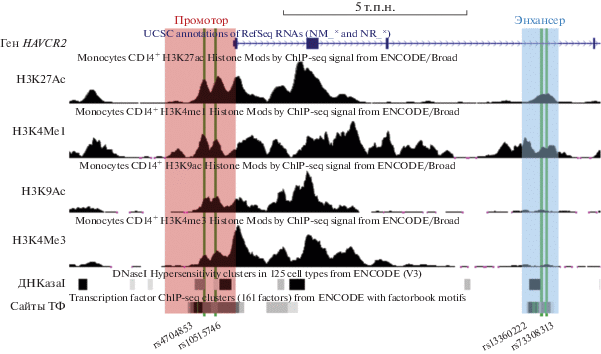
Границы регуляторных элементов гена HAVCR2 определяли с помощью геномного браузера UCSC Genome Browser, предоставляющего доступ к аннотациям геномов большого числа видов и позволяющего выявлять регуляторные последовательности на основании эпигенетических признаков. В качестве промоторной области гена HAVCR2 был выбран участок генома размером 1 992 п.н. (chr5: 156535992‒156537986, сборка GRCh37/hg19; рис. 1) с соответствующими эпигенетическими метками: повышенная чувствительность к ДНКазе I, высокая плотность сайтов связывания ТФ, а также модификации гистонов H3K4me3, H3K27Ac и H3K9Ac. В этой области были отобраны два некодирующих SNP: rs10515746 и rs4704853, ‒ ассоциированных с риском развития различных патологий, ‒ с целью изучить их влияние на активность промотора гена HAVCR2. Для этого несколько вариантов промотора гена HAVCR2, содержащих минорные либо мажорные аллели этих SNP, клонировали в репортерный вектор pGL3-basic перед кодирующей последовательностью гена люциферазы.
Рис. 1.
Схема расположения изучаемых SNP и предполагаемых регуляторных элементов локуса гена HAVCR2. Промоторная область HAVCR2 обозначена красным, предполагаемый энхансер – синим. Вертикальными линиями указано расположение изучаемых SNP. Гистограммы отражают уровни модификаций гистонов, ассоциированных с активными регуляторными участками (монометилирование H3K4, ацетилирование H3K27, H3K9) и активными промоторами (триметилирование H3K4) в CD14+ моноцитах. Прямоугольниками отмечены кластеры гиперчувствительности к ДНКазе I и сайты связывания факторов транскрипции (ТФ) по данным ENCODE ChIP-Seq. Схема построена на основе визуализации в UCSC Genome Browser (GRCh37/hg19).
Способность выбранных SNP влиять на активность соответствующего промотора анализировали с использованием люциферазного теста в модели активированных моноцитов – стимулированных PMA клеток линии U937 [26]. Оказалось, что альтернативные варианты исследуемых полиморфизмов rs10515746 и rs4704853 не оказывают влияния на активность промотора HAVCR2 в активированных моноцитах (рис. 2а).
Рис. 2.
Альтернативные варианты SNP rs10515746 и rs4704853 не влияют на активность промотора HAVCR2 в активированных моноцитах U937 (а) потенциально из-за их дистального расположения от функционально активной части промотора (б). Уровень активности репортерной люциферазы в клетках, трансфицированных плазмидами на основе репортерного вектора pGL3-basic, содержащих промотор гена HAVCR2 с мажорными вариантами исследуемых полиморфизмов (WT) и их минорными аллелями (а), либо содержащих различающиеся по длине варианты промотора HAVCR2 (б). Гистограммы отражают уровень активности репортерной люциферазы, нормализованный на активность люциферазы внутреннего контроля в активированных моноцитах U937. На виде (а) представлены результаты 5 экспериментов в виде средних значений ± SEM; на виде (б) ‒ не менее 3 экспериментов в виде средних значений ± SEM. *P < 0.05 ‒ достоверное различие от промотора размером 1 992 п.н. (непарный t-критерий Стьюдента); n.s. ‒ статистически недостоверное различие.
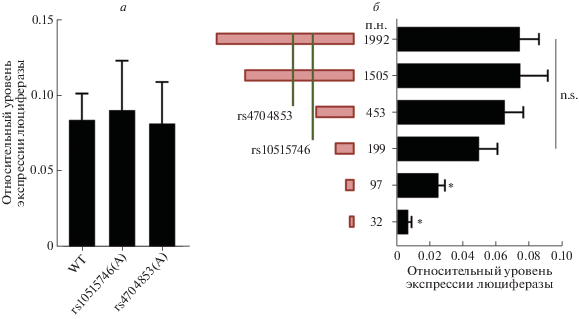
На следующем этапе был проведен делеционный анализ промотора, содержащего более распространенные аллели исследуемых SNP. Для этого в плазмиду pGL3-basic перед геном люциферазы клонировали пять вариантов промоторных последовательностей длиной 1 505, 453, 199, 97 и 32 п.н. до старта трансляции гена HAVCR2 (рис. 2б). Промоторная активность достоверно не различалась для делеционных вариантов промотора длиной ≥ 199 п.н., что свидетельствует о том, что в активированных моноцитах наиболее активная область промотора располагается в пределах 200 п.н. до старт-кодона гена HAVCR2. Результаты делеционного анализа объясняют, почему расположенные вне функционально активной области промотора гена HAVCR2 полиморфизмы rs10515746 и rs4704853 не влияли на его активность в активированных моноцитах.
Минорный аллель полиморфизма rs13360222(T) снижает активность энхансерной области, расположенной в интроне 3 гена HAVCR2
В качестве потенциального энхансера была выбрана область в интроне 3 гена HAVCR2 размером 1 025 п.н. (chr5: 156527046‒156528070, сборка GRCh37/hg19; рис. 1), содержащая эпигенетические метки H3K4me1, H3K9Ac и H3K27Ac, а также кластеры повышенной чувствительности хроматина к ДНКазе I и потенциальные участки связывания ТФ, определенные методом ChIP-Seq. Последовательность предполагаемого энхансера клонировали в плазмиду pGL3-basic, в которую предварительно был внесен промотор гена HAVCR2 (1 992 п.н.). В качестве контроля была создана аналогичная конструкция, где вместо потенциального энхансера использована последовательность идентичного размера из интрона 1 гена STAT3 (chr17: 40508494‒40509573, сборка GRCh37/hg19), не проявляющая энхансерную активность (иррелевантный контроль). В используемой репортерной системе энхансерная область в пять раз повышала активность промотора гена HAVCR2 в активированных PMA моноцитах U937 (рис. 3).
Рис. 3.
Минорный аллель SNP rs13360222(T) снижает активность энхансера гена HAVCR2 в активированных моноцитах U937. В левой части рисунка изображена схема плазмид на основе вектора pGL3-basic, содержащих репортерный ген люциферазы под промотором гена HAVCR2, а также энхансеры с альтернативными аллелями: rs13360222 и rs73308313. Вертикальными красными линиями обозначено расположение соответствующих SNP в энхансерной области. Справа представлен уровень активности репортерной люциферазы в клетках, трансфицированных плазмидами: с промотором HAVCR2; с промотором HAVCR2 и иррелевантной последовательностью, не обладающей энхансерными свойствами (контроль); с промотором и разными вариантами предполагаемого энхансера HAVCR2. На графике представлены результаты шести экспериментов в виде средних значений ± SEM. *P < 0.05 ‒ достоверное различие между активностью энхансеров с мажорными вариантами SNP (WT) и минорным вариантом rs13360222(T); ***P < 0.001 ‒ достоверное различие от контрольной конструкции (непарный t-критерий Стьюдента); n.s. ‒ статистически недостоверное различие.
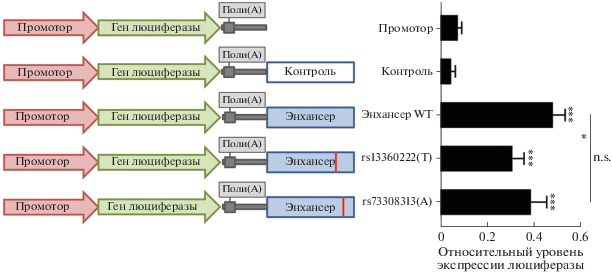
На следующем этапе сконструированы репортерные плазмиды, содержащие минорные варианты полиморфизмов rs73308313 и rs13360222 в энхансере гена HAVCR2. Для этих SNP нет информации об ассоциациях с заболеваниями, однако, по данным ChIP-Seq биоинформатического ресурса GTRD, rs73308313 располагается в области потенциального участка связывания ТФ SPI1, а rs13360222 – ESR2. Кроме того, по данным ресурса PERFECTOS-APE, минорные варианты rs73308313(А) и rs13360222(Т) понижают расчетную аффинность сайтов этих ТФ, что может указывать на их потенциальную роль в изменении регуляторных свойств энхансера в зависимости от представленного аллеля. Нами показано, что альтернативные аллели SNP rs73308313 не оказывали достоверного влияния на способность энхансера активировать промотор гена HAVCR2, тогда как введение в последовательность энхансера минорного варианта rs13360222(Т) приводило к значимому снижению активности промотора HAVCR2 (рис. 3).
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Накопление больших объемов данных, полученных на образцах от пациентов, в сочетании с полногеномным поиском ассоциаций (genome-wide association studies, GWAS) позволяет связать геномные характеристики с предрасположенностью к прогрессии определенных заболеваний. Однако информация об ассоциациях не объясняет механизмов влияния генетической составляющей и носит только диагностический характер [27]. В частности, SNP, выявляемые в ходе GWAS, требуют дальнейшей функциональной аннотации. С целью изучить особенности регуляции гена HAVCR2 в активированных моноцитах мы проанализировали влияние SNP rs10515746 и rs4704853, ассоциированных с онкологическими и/или аутоиммунными заболеваниями, на активность промотора этого гена. Согласно полученным результатам, альтернативные варианты исследуемых полиморфизмов не оказывают влияния на активность промотора гена HAVCR2 в клеточной модели активированных моноцитов U937. Мы также провели делеционный анализ промотора и выяснили, что для относительно высокой активности промотора в использованной экспериментальной системе достаточно области размером около 200 п.н. до старт-кодона TIM-3. Следовательно, отсутствие влияния разных аллелей исследуемых полиморфизмов промотора rs10515746 и rs4704853 на его активность может быть связано с их расположением в дистальной области, не оказывающей принципиального влияния на активность промотора.
Кроме того, мы функционально охарактеризовали энхансер, располагающийся в интроне 3 гена HAVCR2. Присутствие в составе репортерных конструкций энхансерного элемента приводило к пятикратному повышению активности промотора HAVCR2 в клеточной модели активированных человеческих моноцитов. Эти результаты служат дополнительным подтверждением универсальности вышеописанных эпигенетических подходов для выбора потенциальных регуляторных элементов в геноме [28]. Мы проанализировали полиморфизм rs13360222, расположенный в анализируемом энхансере, и, по нашим данным, его минорный вариант Т значимо снижает активность промотора гена HAVCR2 в активированных моноцитах. По данным ChIP-Seq этот SNP располагается в области связывания ТФ ESR2, кроме того, согласно данным ресурса PERFECTOS-APE, при наличии минорного варианта rs13360222(Т) в энхансерной последовательности снижается расчетная аффинность существующего сайта связывания ESR2. Согласно данным ресурса The Tissue Atlas, ESR2 экспрессируется в клеточной линии U937 [29], кроме того, была показана экспрессия ESR2 в активированных моноцитах [30], что подтверждает возможное участие этого ТФ в регулировании экспрессии HAVCR2 в использованной нами клеточной модели. Ранее показано, что активация эстрогенового рецептора ESR2 может приводить к многократной стимуляции экспрессии гена HAVCR2 в клетках остеосаркомы [31]. Также известно, что эстроген стимулирует экспрессию TIM-3 на иммунных клетках, участвующих в воспалительных процессах тканей сердца, что может быть связано с различным уровнем смертности от сердечных заболеваний у мужчин и женщин [32]. Кроме того, влияние эстрогена на экспрессию TIM-3 может быть связано с важной ролью данного рецептора в формировании иммунологической толерантности при беременности, а именно в предотвращении активации макрофагов в ответ на антигены плода [33]. Таким образом, полиморфизм rs13360222 может быть ассоциирован с некоторыми патологиями, ассоциированными с хроническим воспалением, а также с нарушением течения беременности, посредством регуляции транскрипции гена HAVCR2. Дальнейшее изучение молекулярных механизмов влияния rs13360222(Т) на регуляцию экспрессии TIM-3, в том числе и в других популяциях иммунных клеток, представляется интересной задачей.
Исследование выполнено при финансовой поддержке Российского научного фонда (проект № 19-14-00341).
Настоящая статья не содержит каких-либо исследований с участием людей или животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Zhu C., Anderson A., Schubart A., Xiong H., Imitola J., Khoury S., Zheng X., Strom T., Kuchroo V. (2005) The Tim-3 ligand galectin-9 negatively regulates T helper type 1 immunity. Nat. Immunol. 6(12), 1245–1252.
Das M., Zhu S., Kuchroo V. (2017) Tim-3 and its role in regulating anti-tumor immunity. Immunol. Rev. 276(1), 97–111.
Nakayama M., Akiba H., Takeda K., Kojima Y., Hashiguchi M., Azuma M., Yagita H., Okumura K. (2009) Tim-3 mediates phagocytosis of apoptotic cells and cross-presentation. Blood. 113(16), 3821–3830.
Chiba S., Baghdadi M., Akiba H., Yoshiyama H., Kinoshita I., Dosaka-Akita H., Fujioka Y., Ohba Y., Gorman J., Colgan J., Hirashima M., Uede T., Takaoka A., Yagita H., Jinushi M. (2012) Tumor-infiltrating DCs suppress nucleic acid-mediated innate immune responses through interactions between the receptor TIM-3 and the alarmin HMGB1. Nat. Immunol. 13(9), 832–842.
Yang X., Jiang X., Chen G., Xiao Y., Geng S., Kang C., Zhou T., Li Y., Guo X., Xiao H., Hou C., Wang R., Lin Z., Li X., Feng J., Ma Y., Shen B., Li Y., Han G. (2013) T cell Ig mucin-3 promotes homeostasis of sepsis by negatively regulating the TLR response. J. Immunol. 190(5), 2068–2079.
Ndhlovu L.C., Lopez-Vergès S., Barbour J.D., Brad Jones R., Jha A.R., Long B.R., Schoeffler E.C., Fujita T., Nixon D.F., Lanier L.L. (2012) Tim-3 marks human natural killer cell maturation and suppresses cell-mediated cytotoxicity. Blood. 119(16), 3734–3743.
da Silva I.P., Gallois A., Jimenez-Baranda S., Khan S., Anderson A.C., Kuchroo V.K., Osman I., Bhardwaj N. (2014) Reversal of NK cell exhaustion in advanced melanoma by Tim-3 blockade. Cancer Immunol. Res. 2(5), 410–422.
Cao B., Zhu L., Zhu S., Li D., Zhang C., Xu C., Zhang S. (2010) Genetic variations and haplotypes in TIM-3 gene and the risk of gastric cancer. Cancer Immunol. Immunother. 59(12), 1851–1857.
Han F., Wang G., Li Y., Tian W., Dong Z., Cheng S., Liu Y., Qu T., Wang X., Wang Y., Zhang B., Ju Y. (2017) Investigation of T-cell immunoglobulin- and mucin-domain-containing molecule-3 (TIM-3) polymorphisms in essential thrombocythaemia (ET). Hematology. 22(6), 361–367.
Sun L., Wu H., Cao S.G., Xia X.P., Lin X.Q., Jin J., Ding R., Jiang Y. (2017) [Association of Crohn’s disease with T cell immunoglobulin and mucin domain 3 gene polymorphisms in Chinese patients]. Zhonghua nei ke za zhi. 56(9), 667–672.
Chae S.C., Park Y.R., Shim S.C., Yoon K.S., Chung H.T. (2004) The polymorphisms of Th1 cell surface gene Tim-3 are associated in a Korean population with rheumatoid arthritis. Immunol. Lett. 95(1), 91–95.
Wang Z., Liu X., Wang X., Chong T., Lin S., Wang M., Ma X., Liu K., Xu P., Feng Y., Dai Z., Wang Z., Liu X., Wang X., Chong T., Lin S., Wang M., Ma X., Liu K., Xu P., Feng Y., Dai Z. (2016) Polymorphisms in TIM-3 and breast cancer susceptibility in Chinese women: a case-control study. Oncotarget. 7(28), 43703–43712.
Dunham I., Kundaje A., Aldred S.F., Collins P.J., Davis C.A., Doyle F., Epstein C.B., Frietze S., Harrow J., Kaul R., Khatun J., Lajoie B.R., Landt S.G., Lee B.K., Pauli F., Rosenbloom K.R., Sabo P., Safi A., Sanyal A., Shoresh N., Simon J.M., Song L., Trinklein N.D., Altshuler R.C., Birney E., Brown J.B., Cheng C., Djebali S., Dong X., Ernst J., Furey T.S., Gerstein M., Giardine B., Greven M., Hardison R.C., Harris R.S., Herrero J., Hoffman M.M., Iyer S., Kellis M., Kheradpour P., Lassmann T., Li Q., Lin X., Marinov G.K., Merkel A., Mortazavi A., Parker S.C.J., Reddy T.E., Rozowsky J., Schlesinger F., Thurman R.E., Wang J., Ward L.D., Whitfield T.W., Wilder S.P., Wu W., Xi H.S., Yip K.Y., Zhuang J., Bernstein B.E., Green E.D., Gunter C., Snyder M., Pazin M.J., Lowdon R.F., Dillon L.A.L., Adams L.B., Kelly C.J., Zhang J., Wexler J.R., Good P.J., Feingold E.A., Crawford G.E., Dekker J., Elnitski L., Farnham P.J., Giddings M.C., Gingeras T.R., Guigó R., Hubbard T.J., Kent W.J., Lieb J.D., Margulies E.H., Myers R.M., Stamatoyannopoulos J.A., Tenenbaum S.A., Weng Z., White K.P., Wold B., Yu Y., Wrobel J., Risk B.A., Gunawardena H.P., Kuiper H.C., Maier C.W., Xie L., Chen X., Mikkelsen T.S., Gillespie S., Goren A., Ram O., Zhang X., Wang L., Issner R., Coyne M.J., Durham T., Ku M., Truong T., Eaton M.L., Dobin A., Tanzer A., Lagarde J., Lin W., Xue C., Williams B.A., Zaleski C., Röder M., Kokocinski F., Abdelhamid R.F., Alioto T., Antoshechkin I., Baer M.T., Batut P., Bell I., Bell K., Chakrabortty S., Chrast J., Curado J., Derrien T., Drenkow J., Dumais E., Dumais J., Duttagupta R., Fastuca M., Fejes-Toth K., Ferreira P., Foissac S., Fullwood M.J., Gao H., Gonzalez D., Gordon A., Howald C., Jha S., Johnson R., Kapranov P., King B., Kingswood C., Li G., Luo O.J., Park E., Preall J.B., Presaud K., Ribeca P., Robyr D., Ruan X., Sammeth M., Sandhu K.S., Schaeffer L., See L.H., Shahab A., Skancke J., Suzuki A.M., Takahashi H., Tilgner H., Trout D., Walters N., Wang H., Hayashizaki Y., Reymond A., Antonarakis S.E., Hannon G.J., Ruan Y., Carninci P., Sloan C.A., Learned K., Malladi V.S., Wong M.C., Barber G.P., Cline M.S., Dreszer T.R., Heitner S.G., Karolchik D., Kirkup V.M., Meyer L.R., Long J.C., Maddren M., Raney B.J., Grasfeder L.L., Giresi P.G., Battenhouse A., Sheffield N.C., Showers K.A., London D., Bhinge A.A., Shestak C., Schaner M.R., Kim S.K., Zhang Z.Z., Mieczkowski P.A., Mieczkowska J.O., Liu Z., McDaniell R.M., Ni Y., Rashid N.U., Kim M.J., Adar S., Zhang Z., Wang T., Winter D., Keefe D., Iyer V.R., Zheng M., Wang P., Gertz J., Vielmetter J., Partridge E.C., Varley K.E., Gasper C., Bansal A., Pepke S., Jain P., Amrhein H., Bowling K.M., Anaya M., Cross M.K., Muratet M.A., Newberry K.M., McCue K., Nesmith A.S., Fisher-Aylor K.I., Pusey B., DeSalvo G., Parker S.L., Balasubramanian S., Davis N.S., Meadows S.K., Eggleston T., Newberry J.S., Levy S.E., Absher D.M., Wong W.H., Blow M.J., Visel A., Pennachio L.A., Petrykowska H.M., Abyzov A., Aken B., Barrell D., Barson G., Berry A., Bignell A., Boychenko V., Bussotti G., Davidson C., Despacio-Reyes G., Diekhans M., Ezkurdia I., Fran-kish A., Gilbert J., Gonzalez J.M., Griffiths E., Harte R., Hendrix D.A., Hunt T., Jungreis I., Kay M., Khurana E., Leng J., Lin M.F., Loveland J., Lu Z., Manthravadi D., Mariotti M., Mudge J., Mukherjee G., Notredame C., Pei B., Rodriguez J.M., Saunders G., Sboner A., Searle S., Sisu C., Snow C., Steward C., Tapanari E., Tress M.L., Van Baren M.J., Washietl S., Wilming L., Zadissa A., Zhang Z., Brent M., Haussler D., Valencia A., Addleman N., Alexander R.P., Auerbach R.K., Balasubramanian S., Bettinger K., Bhardwaj N., Boyle A.P., Cao A.R., Cayting P., Charos A., Cheng Y., Eastman C., Euskirchen G., Fleming J.D., Grubert F., Habegger L., Hariharan M., Harmanci A., Iyengar S., Jin V.X., Karczewski K.J., Kasowski M., Lacroute P., Lam H., Lamarre-Vincent N., Lian J., Lindahl-Allen M., Min R., Miotto B., Monahan H., Moqtaderi Z., Mu X.J., O’Geen H., Ouyang Z., Patacsil D., Raha D., Ramirez L., Reed B., Shi M., Slifer T., Witt H., Wu L., Xu X., Yan K.K., Yang X., Struhl K., Weissman S.M., Penalva L.O., Karmakar S., Bhanvadia R.R., Choudhury A., Domanus M., Ma L., Moran J., Victorsen A., Auer T., Centanin L., Eichenlaub M., Gruhl F., Heermann S., Hoeckendorf B., Inoue D., Kellner T., Kirchmaier S., Mueller C., Reinhardt R., Schertel L., Schneider S., Sinn R., Wittbrodt B., Wittbrodt J., Jain G., Balasundaram G., Bates D.L., Byron R., Canfield T.K., Diegel M.J., Dunn D., Ebersol A.K., Frum T., Garg K., Gist E., Hansen R.S., Boatman L., Haugen E., Humbert R., Johnson A.K., Johnson E.M., Kutyavin T.V., Lee K., Lotakis D., Maurano M.T., Neph S.J., Neri F. V., Nguyen E.D., Qu H., Reynolds A.P., Roach V., Ry-nes E., Sanchez M.E., Sandstrom R.S., Shafer A.O., Stergachis A.B., Thomas S., Vernot B., Vierstra J., Vong S., Wang H., Weaver M.A., Yan Y., Zhang M., Akey J.M., Bender M., Dorschner M.O., Groudine M., MacCoss M.J., Navas P., Stamatoyannopoulos G., Beal K., Brazma A., Flicek P., Johnson N., Lukk M., Luscombe N.M., Sobral D., Vaquerizas J.M., Batzoglou S., Sidow A., Hussami N., Kyriazopoulou-Panagiotopoulou S., Libbrecht M.W., Schaub M.A., Miller W., Bickel P.J., Banfai B., Boley N.P., Huang H., Li J.J., Noble W.S., Bilmes J.A., Buske O.J., Sahu A.D., Kharchenko P.V., Park P.J., Baker D., Taylor J., Lochovsky L. (2012) An integrated encyclopedia of DNA elements in the human genome. Nature. 489(7414), 57–74.
Ernst J., Kellis M. (2017) Chromatin-state discovery and genome annotation with ChromHMM. Nat. Protoc. 12(12), 2478–2492.
Kolmykov S., Yevshin I., Kulyashov M., Sharipov R., Kondrakhin Y., Makeev V.J., Kulakovskiy I.V., Kel A., Kolpakov F. (2021) GTRD: an integrated view of transcription regulation. Nucleic Acids Res. 49(D1), D104–D111.
Vorontsov I.E., Kulakovskiy I.V., Khimulya G., Nikolaeva D.D., Makeev V.J. (2015) PERFECTOS-APE: Predicting regulatory functional effect of SNPs by approximate p-value estimation. In: Proceedings of the International Conference on Bioinformatics Models, Methods and Algorithms (BIOINFORMATICS-2015), pp. 102–108. https://doi.org/10.5220/0005189301020108
Kulakovskiy I.V., Vorontsov I.E., Yevshin I.S., Sharipov R.N., Fedorova A.D., Rumynskiy E.I., Medvedeva Y.A., Magana-Mora A., Bajic V.B., Papatsenko D.A., Kolpakov F.A., Makeev V.J. (2018) HOCOMOCO: towards a complete collection of transcription factor binding models for human and mouse via large-scale ChIP-Seq analysis. Nucleic Acids Res. 46(D1), D252–D259.
Fornes O., Castro-Mondragon J.A., Khan A., Van Der Lee R., Zhang X., Richmond P.A., Modi B.P., Correard S., Gheorghe M., Baranašić D., Santana-Garcia W., Tan G., Chèneby J., Ballester B., Parcy F., Sandelin A., Lenhard B., Wasserman W.W., Mathelier A. (2020) JASPAR 2020: Update of the open-access database of transcription factor binding profiles. Nucleic Acids Res. 48(D1), D87–D92.
Jolma A., Kivioja T., Toivonen J., Cheng L., Wei G., Enge M., Taipale M., Vaquerizas J.M., Yan J., Sillanpää M.J., Bonke M., Palin K., Talukder S., Hughes T.R., Luscombe N.M., Ukkonen E., Taipale J. (2010) Multiplexed massively parallel SELEX for cha-racterization of human transcription factor binding specificities. Genome Res. 20(6), 861–873.
Schwartz A.M., Demin D.E., Vorontsov I.E., Kasyanov A.S., Putlyaeva L.V., Tatosyan K.A., Kulakovskiy I.V., Kuprash D.V. (2017) Multiple single nucleotide polymorphisms in the first intron of the IL2RA gene affect transcription factor binding and enhancer activity. Gene. 602, 50–56.
Afanasyeva M.A., Putlyaeva L.V., Demin D.E., Kulakovskiy I.V., Vorontsov I.E., Fridman M.V., Makeev V.J., Kuprash D.V., Schwartz A.M. (2017) The single nucleotide variant rs12722489 determines differential estrogen receptor binding and enhancer properties of an IL2RA intronic region. PLoS One. 12(2), e0172681.
Ustiugova A.S., Korneev K.V., Kuprash D.V., Afanasyeva M.A. (2019) Functional SNPs in the human autoimmunity-associated locus 17q12-21. Genes (Basel). 10(2), 77.
Sundström C., Nilsson K. (1976) Establishment and characterization of a human histiocytic lymphoma cell line (U-937). Int. J. Cancer. 17(5), 565–577.
Korneev K. V., Sviriaeva E.N., Mitkin N.A., Gorbacheva A.M., Uvarova A.N., Ustiugova A.S., Polanovsky O.L., Kulakovskiy I.V., Afanasyeva M.A., Schwartz A.M., Kuprash D.V. (2020) Minor C allele of the SNP rs7873784 associated with rheumatoid arthritis and type-2 diabetes mellitus binds PU.1 and enhances TLR4 expression. Biochim. Biophys. Acta Mol. Basis Dis. 1866(3), 165626.
Gorbacheva A.M., Uvarova A.N., Ustiugova A.S., Bhattacharyya A., Korneev K.V., Kuprash D.V., Mitkin N.A. (2021) EGR1 and RXRA transcription factors link TGF-β pathway and CCL2 expression in triple negative breast cancer cells. Sci. Rep. 11(1), 114120. https://doi.org/10.1038/s41598-021-93561-6
Minafra L., Di Cara G., Albanese N.N., Cancemi P. (2011) Proteomic differentiation pattern in the U937 cell line. Leuk. Res. 35(2), 226–236.
Visscher P.M., Wray N.R., Zhang Q., Sklar P., McCarthy M.I., Brown M.A., Yang J. (2017) 10 years of GWAS discovery: biology, function, and translation. Am. J. Hum. Genet. 101(1), 5.
Mora A., Sandve G.K., Gabrielsen O.S., Eskeland R. (2016) In the loop: promoter–enhancer interactions and bioinformatics. Brief. Bioinform. 17(6), 980.
Uhlén M., Fagerberg L., Hallström B.M., Lindskog C., Oksvold P., Mardinoglu A., Sivertsson Å., Kampf C., Sjöstedt E., Asplund A., Olsson I.M., Edlund K., Lundberg E., Navani S., Szigyarto C.A.K., Odeberg J., Djureinovic D., Takanen J.O., Hober S., Alm T., Edqvist P.H., Berling H., Tegel H., Mulder J., Rockberg J., Nilsson P., Schwenk J.M., Hamsten M., Von Feilitzen K., Forsberg M., Persson L., Johansson F., Zwahlen M., Von Heijne G., Nielsen J., Pontén F. (2015) Tissue-based map of the human proteome. Science. 347(6220), 1260419.
Campesi I., Marino M., Montella A., Pais S., Franconi F. (2017) Sex differences in estrogen receptor α and β levels and activation status in LPS-stimulated human macrophages. J. Cell. Physiol. 232(2), 340–345.
Vivar O.I., Zhao X., Saunier E.F., Griffin C., Mayba O.S., Tagliaferri M., Cohen I., Speed T.P., Leitman D.C. (2010) Estrogen receptor β binds to and regulates three distinct classes of target genes. J. Biol. Chem. 285(29), 22059–22066.
Fish E.N. (2008) The X-files in immunity: sex-based differences predispose immune responses. Nat. Rev. Immunol. 8(9), 737–744.
Zhang Y.H., He M., Wang Y., Liao A.H. (2017) Modulators of the balance between M1 and M2 macrophages during pregnancy. Front. Immunol. 8, 120.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


