Молекулярная биология, 2022, T. 56, № 3, стр. 439-450
HEMK-подобные метилтрансферазы в регуляции клеточных процессов
Н. С. Бизяев a, А. В. Шувалов a, b, Е. З. Алкалаева a, b, *
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
b Центр высокоточного редактирования и генетических технологий для биомедицины
119991 Москва, Россия
* E-mail: alkalaeva@eimb.ru
Поступила в редакцию 26.11.2021
После доработки 26.11.2021
Принята к публикации 02.12.2021
- EDN: NHGHIP
- DOI: 10.31857/S0026898422030028
Аннотация
Трансляционная метилтрансфераза (метилаза) HEMK2 человека, ортологи которой обнаружены у множества прокариот и эукариот, метилирует такие разнообразные субстраты, как остатки глутамина и лизина в составе белков, дезоксиаденозин в ДНК, а также соединения мышьяка. Важным субстратом метилазы HEMK2 является остаток глутамина в инвариантном GGQ-мотиве эукариотического фактора терминации трансляции 1 (eRF1). Метилирование этого остатка ортологами HEMK2 консервативно у эукариот, архей и бактерий, хотя факторы терминации бактерий имеют другие аминокислотные последовательности и структуру. В данном обзоре рассмотрены особенности функционирования метилазы HEMK2 человека и ее ортологов как многофункциональных ферментов, регулирующих клеточные процессы, в частности, биосинтез белка.
ВВЕДЕНИЕ
Метилаза (метилтрансфераза) HEMK2 человека (известная также, как N6AMT1, или KMT9, или C21orf127) и ее ортологи в других эукариотах и археях, участвует в метилировании широкого спектра субстратов, используя в качестве донора метильной группы S-аденозилметионин (SAM). Предполагается, что этот фермент способен метилировать остатки глутамина в факторе терминации трансляции (eRF1), а также в факторах ремоделирования хроматина CHD5 и NUT [1–6], остатки лизина в гистоне H4 [2, 7], токсичные соединения мышьяка [8], а также, возможно, остатки дезоксиаденозина в ДНК [9].
Таблица 1.
Функции HEMK2 человека и ее ортологов
| Функция | Организм | Комментарий | Ссылка |
|---|---|---|---|
| Метилирование остатка глутамина (Q) в факторах терминации трансляции I класса | Бактерии, археи, эукариоты | [4–7, 10–12, 43, 45, 46] | |
| Метилирование остатка глутамина (Q) в факторах ремоделирования хроматина CHD5, NUT | Человек | [20, 45] | |
| Метилирование остатка глутамина (Q) около 50 иных белков-кандидатов, в том числе участвующих в процессах трансляции (RRP1) и сигналинга (TGFB3, ARHGEF10) | Человек | Только биоинформатическое предсказание по аминокислотной последовательности оптимального сайта метилирования [45] | [45] |
| Метилирование остатка лизина (K) в гистоне H4 | Человек | [2, 20] | |
| Метилирование монометиларсенита | Человек, дрожжи | Физиологическое значение не установлено | [8, 27] |
| Метилирование неорганического трехвалентного арсенита | Дрожжи | [8, 27] | |
| Метилирование остатка дезоксиаденозина | Человек | Подвергается сомнению [7, 22, 31] | [9] |
| Связывание хроматина в промоторных областях | Человек | [2] | |
| Связывание и влияние на созревание 60S субъединицы | Дрожжи | Не ясна связь с процессом метилирования eRF1, которую обсуждают в [21] | [21] |
| Влияние на созревание 5.8S и 25S рРНК | Дрожжи | Механизм не показан, только констатация фенотипа мутантов [21] | [21] |
Остатки глутамина в факторах терминации трансляции Escherichia coli также подвергаются метилированию метилазой HEMK (Protein release factor methylation C, PrmC) и в митохондриях человека метилазой HEMK1. Обе эти метилазы гомологичны HEMK2 [4, 5, 10–13]. Ген HEMK E. coli входит в состав одного оперона с генами, кодирующими фактор терминации трансляции RF1 и глутамил-тРНК-редуктазу, которая участвует в синтезе гема. Из-за этого некоторое время считалось, что и HEMK необходима для синтеза гема, что обусловило название фермента (heme – гем) [10, 14]. Однако в дальнейшем участия HEMK в синтезе гема не обнаружили [12].
Все указанные метилазы мы будем далее называть HEMK-подобными метилазами. Необходимо отметить, что метилирование факторов терминации трансляции не является уникальной модификацией компонентов трансляции. Как способ регуляции трансляции описано также метилирование рибосомных белков, трансляционных факторов, а также рРНК [5, 15].
СТРУКТУРА, ОРТОЛОГИ И ФЕРМЕНТАТИВНАЯ АКТИВНОСТЬ HEMK-ПОДОБНЫХ МЕТИЛАЗ
Функциональный домен всех HEMK-подобных метилаз устроен сходным образом (рис. 1). Он образован Россмановской укладкой, состоящей из семи β-тяжей, которые расположены приблизительно в одной плоскости и окружены с обеих сторон группами α-спиралей. Такая структура функционального домена позволяет относить HEMK-подобные метилазы к самому крупному первому (I) классу метилаз. Функциональный участок содержит консервативные глицин-богатые и NPPY-мотивы, связывающие, соответственно, SAM и метилируемый субстрат (у арсенита и его производных его значение неизвестно). Все они имеют также консервативные остатки пролина (P), участвующего в стабилизации структуры активного центра, и аспартата (D), участвующего в связывании SAM (рис. 1) [9, 10, 13, 16–20]. Точечный мутант метилазы человека N122A с нефункциональным мотивом NPPY не может метилировать ни остаток глутамина eRF1, ни остаток лизина гистона H4 [2]. Точно также дрожжевые точечные мутанты D77A по консервативному остатку аспартата, а также N122A по NPPY-мотиву не способны эффективно метилировать eRF1 [21].
Рис. 1.
Структуры HEMK-подобных метилаз. На структуре и в последовательностях желтой линией выделен консервативный остаток пролина, зеленым показан консервативный глицин-богатый участок, фиолетовой линией – консервативный остаток аспартата кислой петли, красным – NPPY-мотив. а – Обобщенная схема функционального домена HEMK-подобных метилаз, образованного семью β-тяжами (обозначены оранжевым цветом), которые расположены в одной плоскости и окружены с двух сторон α-спиралями (выделены синим), по данным [16, 19]. б – Структуры функционального домена HEMK бактерии E. coli [22] (PDB: 1T43) и HEMK2 человека [2] (PDB: 6H1E). Структуры визуализированы с помощью BioRender.com. в – Сравнение аминокислотных последовательностей HEMK-подобных метилаз. Идентичные, а также сильно и слабо сходные по свойствам остатки выделены темно-серым, серым и светло-серым цветами соответственно. г – Дерево сходства последовательностей HEMK-подобных метилаз. Голубым выделены бактериальные и митохондриальные HEMK-подобные метилазы, красным – архейные, черным эукариотические.

Метилирование белковых субстратов происходит по SN2-механизму за счет оптимального позиционирования субстрата в активном центре с помощью водородных связей, а также дестабилизации SAM, что катализирует нуклеофильную атаку неподеленной пары электронов азота на метильную группу SAM [16, 22].
Несмотря на сходство третичной структуры функционального домена и нескольких консервативных мотивов и остатков, бактериальные HEMK-подобные метилазы довольно сильно отличаются от эукариотических/архейных по первичной структуре. При выравнивании и сравнении последовательностей HEMK-подобных метилаз эукариотические и архейные метилазы кластеризуются в одну группу, что указывает на их близкое родство, тогда как митохондриальные метилазы кластеризуются с метилазой HEMK E. coli (рис. 1г). Более того, бактериальные HEMK-подобные метилазы имеют в своем составе дополнительный N-домен, отсутствующий у эукариотических и архейных ортологов HEMK2 [10, 16, 17] (рис. 1в). Интересно, что N-домен бактериальной HEMK охарактеризован как один из примеров пептидов, фолдинг которых начинается непосредственно внутри выходного туннеля рибосомы [23–25].
HEMK2 и его ортологам у архей и эукариот, в отличие от бактериальной HEMK, для эффективного осуществления метилирования необходимо образовать комплекс с белком TRMT112 [1–3, 8, 17, 26, 27]. По данным изотермической титрационной калориметрии (ITC) формирование функционального комплекса HEMK2-TRMT112, среди прочего, необходимо для того, чтобы метилаза связала SAM [2]. Однако у некоторых архей (Methanobacteriales и Thermococcales) обнаружен только ортолог HEMK2, но не TRMT112 [28]. Таким образом, не до конца ясно, насколько эффективно у них функционирует метилаза.
Связанный с метилазой TRMT112 стабилизирует конформацию ее β3–β4-тяжей, необходимую для связывания SAM, а также маскирует гидрофобный регион фермента, что значительно повышает растворимость гетерокомплекса [17, 20, 26]. В отсутствие гомолога TRMT112 метилаза дрожжей MTQ2 (от MethylTransferase glutamine(Q), ортолог HEMK2) образует нерастворимые агрегаты [26]. В культурах клеток млекопитающих нокдаун TRMT112 не ведет к резкому уменьшению количества HEMK2, как в случае другого ее партнера – WBSCR22 [29]. Однако в клетках дрожжей нокдаун гомолога TRMT112 уменьшает количество MTQ2, снижая, вероятно, ее стабильность [30].
Ген HEMK2 человека, расположенный на хромосоме 21, кодирует как функциональную полноразмерную изоформу HEMK2α, так и нефункциональную изоформу HEMK2β, в которой отсутствует NPPY-мотив. Обе эти изоформы транскрибируются [1, 29, 31]. Однако нефункциональная изоформа HEMK2β не способна взаимодействовать с TRMT112 [29]. Клетки мыши также содержат PRED28α и PRED28β (ортологи HEMK2α и HEMK2β соответственно) [31]. Локализация гена стала причиной появления альтернативного названия HEMK2 – C21orf127 (Chromosome 21 Open Reading Frame 127).
Данные о внутриклеточной локализации белка противоречивы. Методом иммуноокрашивания показана ядерная локализация PRED28α и PRED28β мыши [31]. На основании этих данных в базе Uniprot (идентификационный номер Q9Y5N5, дата обращения 23.11.2021) по аналогии также указана ядерная локализация HEMK2 человека. Однако с помощью измерения флуоресценции слитого с GFP белка NRF1 арабидопсиса (ортолог HEMK2) удалось обнаружить лишь в цитоплазме, но не в ядрах клеток [32]. Можно предположить, что добавление GFP влияет на способность метилазы оставаться в ядре. Возможно, что локализация метилазы изменяется под действием неких факторов, которые не детектировались в исследованиях. Возможно, что внутриклеточная локализация ортологов в различных организмах не консервативна. Кроме того, предполагается, что малая молекулярная масса метилазы не должна затруднять ее перемещение между ядром и цитоплазмой, и фермент может локализоваться и в ядре, и в цитоплазме [31]. При этом HEMK1 человека, сходная с бактериальным ферментом, ожидаемо локализуется в митохондриях [13].
ФИЗИОЛОГИЧЕСКОЕ ЗНАЧЕНИЕ HEMK-ПОДОБНЫХ МЕТИЛАЗ
HEMK-подобные метилазы бактерий
Скрининг профиля экспрессии мутантов E. coli с нокаутом HEMK предсказывает влияние метилазы на экспрессию 3275 генов. В частности, нокаут HEMK приводит к подавлению экспрессии генов, связанных с аэробным дыханием, но активирует экспрессию генов анаэробного дыхания. Однако нокаут HEMK не влияет на уровень экспрессии фактора терминации RF1 [12] несмотря на то, что кодирующие их гены находятся в одном опероне [10, 14]. Но сверхэкспрессия HEMK супрессирует токсичность, вызванную сверхэкспрессией гена, кодирующего фактор терминации трансляции RF2, что можно рассматривать как доказательство влияния HEMK на RF2 [10]. Кроме того, у бактерий с нокаутом HEMK повышена частота сквозного прочтения на стоп-кодонах UAG и UGA [12, 33].Такие бактерии плохо растут на богатой среде и совсем не растут на бедной среде [10, 33]. Мутации и делеции гена HEMK в штамме EC958 E. coli (подгруппа штамма ST121) вызывают повышение экспрессии генов, связанных с бактериальным жгутиком (флагеллой) и увеличение количества жгутиков, что ведет к увеличению ее подвижности. Комплементация мутаций и делеций этого гена приводят к восстановлению дикого типа клеток [34].
HEMK-подобные метилазы эукариот и архей
Обнаружено, что ген HEMK2 человека, а также его ортологи у эукариот и архей связаны со множеством физиологических процессов, а его присутствие необходимо для нормального роста и развития клеток различных эукариот и архей и устойчивости в различных стрессовых условиях.
Так, у археи Haloferax volcanii нокаут по гену, кодирующему ортолог HEMK2, увеличивает в 2 раза время, необходимое для одного раунда деления клеток, и приводит к уменьшению размера колоний [3].
Нокаут гена MTQ2, кодирующего цитозольный/ядерный дрожжевой ортолог HEMK2, а равно внесение в него точечных мутаций, инактивирующих метилирующую активность, приводит к замедлению роста на стандартных средах, повышению чувствительности к падению температуры, высоким концентрациям солей, кальция, кофеина, а также к антибиотикам паромомицину, генетицину (регулируют трансляцию) и полимиксин-β-сульфату (влияет на синтез триглицеридов) [4, 21]. Показано, что MTQ2 ассоциирован с 60S субъединицами рибосом и, вероятно, является фактором их экспорта из ядра. Нокаут гена MTQ2 или его мутация, вызывающие потерю связывания белка с SAM или белковым субстратом, приводит к нарушению биогенеза 60S рибосом и “застреванию” предшественников 60S субъединиц в ядре. Также метилаза стимулирует созревание 5.8S и 25S рРНК у дрожжей [21] (рис. 2). Более того, отдельно показано, что дрожжевой гомолог TRMT112 необходим для биогенеза обеих субъединиц рибосом [30].
Рис. 2.
Схема работы многофункциональной метилазы HEMK2 человека и ее эукариотических ортологов (а) и субстраты HEMK2 (б). Розовым выделена цитоплазма, фиолетовым – ядро. Черные стрелки показывают экспорт метилазы HEMK2 или ее комплекса с TRMT112 (HEMK2-TRMT112) через ядерную мембрану. Зеленые стрелки указывают на субстраты метилирования HEMK2 или HEMK2-TRMT112, красные – на связывание и влияние на функции. Зеленым цветом выделены метилируемые атомы. Рисунок нарисован с использованием BioRender.com.
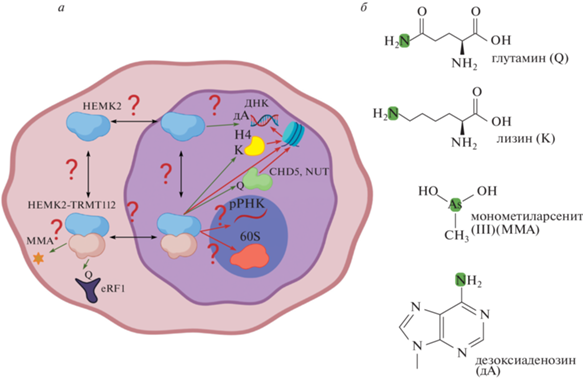
В клетках дрожжей нокаут митохондриального гена MTQ1, кодирующего ортолог HEMK1, не приводит к замедлению роста на стандартных средах, однако замедляет его при росте на средах с неферментируемыми источниками углерода, спиртами, а также повышает эффективность сквозного прочтения стоп-кодонов [4].
Ген, кодирующий NRF1 (ортолог HEMK2), экспрессируется во всех тканях цветкового растения Arabidopsis thaliana. Нокаут этого гена приводит к замедлению роста, уменьшению размеров и гиперчувствительности к дефициту железа. Последнее согласуется с тем, что функциональный белок NRF1 необходим для экспрессии большинства генов, индуцируемых недостатком железа, с чем и связано его название (Non-response To Fe-Deficiency 1). Более того, по данным Ribo-Seq нокаут метилазы NRF1 приводит к “зависанию” рибосом около стоп-кодона [32].
Показано, что ортолог HEMK2 активно экспрессируется в нервной системе круглого червя Caenorhabditis elegans [35]. Этот ген необходим для высвобождения ацетилхолина холинергическими нейронами. У мутантных по данному гену червей нарушены паттерны поведения, кроме того, они менее чувствительны к ингибиторам холинэстераз [35].
Нокдаун ортологов генов HEMK1 и HEMK2 дрозофилы подавляет деление стволовых клеток кишечника, но не энтероцитов [36].
Ген HEMK2 человека и его мышиный ортолог экспрессируются во всех тканях, но данные по относительному уровню экспрессии в разных органах противоречивы [8, 31, 37]. Также показано, что сверхэкспрессия HEMK2 и его ортологов обеспечивает устойчивость клеток человека и дрожжей к некоторым соединениям мышьяка [8]. Число копий гена HEMK2 и уровень его экспрессии понижены в клетках рака желудка и печени, что стимулирует их рост, формирование колоний, миграцию и инвазию. И, наоборот, сверхэкспрессия гена подавляет эти процессы [9]. В другом исследовании нокаут и нокдаун HEMK2, напротив, понижали скорость деления клеток рака простаты, но не влияли на такие неопухолевые клетки, как HEK293 или C2C12 [2]. Нокаут HEMK2 в клетках K562 приводит к нарушению окислительного фосфорилирования [38]. Нокаут HEMK2 вызывает эмбриональную летальность у мышей, обусловленную нарушением раннего эмбрионального развития, а нокдаун HEMK2 в клеточной линии HEK-293T человека на 50–60% снижает пролиферацию клеток и несколько повышает уровень экспрессии белка-ингибитора циклинзависимой киназы p21, отвечающей за клеточный ответ на повреждения ДНК [39]. HEMK2 считается также биомаркером некоторых заболеваний. Его экспрессия снижается при немелкоклеточном раке легкого (NSCLC) [40], а уровень белка HEMK2 повышен при миодистрофии Дюшенна [41]. С помощью коиммунопреципитации выявлена значительная представленность HEMK2 в области промоторов генов, что указывает на ее участие в регуляции транскрипции (табл. 1, рис. 2). Делеция HEMK2 ведет к нарушению экспрессии генов, связанных с клеточным циклом [2].
Показано, что митохондриальный HEMK1 крупного рогатого скота связан с развитием иммунной системы [42]. Однако нокаут HEMK1 в клеточной линии HeLa человека не влияет значимо на рост клеток, число копий мтДНК, а также на мембранный потенциал митохондрий [13].
МЕТИЛИРУЕМЫЕ СУБСТРАТЫ HEMK-ПОДОБНЫХ МЕТИЛАЗ
Остаток глутамина в факторах терминации трансляции и в факторах ремоделирования хроматина
Показано, что как эукариотические/архейные, так и бактериальные HEMK-подобные метилазы метилируют остатки глутамина в составе белков в N5 положении [4–7, 10–13, 43–46]. Все остатки глутамина (Q), метилируемые HEMK2 человека, объединяет общий сайт узнавания: GQ0хххR, где позиция метилируемого Q обозначена 0. Определена относительная эффективность метилирования пептидов, в которых присутствовал каждый возможный аминокислотный остаток в каждом положении от –6 до +7, по сравнению с последовательностью дикого типа. Так, в положении +1 наблюдается предпочтение остатков серина (S), аргинина (R) и глицина (G), а в +7 – остатка аргинина (R). Эти результаты получены на коротких пептидах [20, 45]. Предполагается, что такое метилирование глутамина необратимо [46].
Наиболее изученным и универсальным субстратом всех HEMK-подобных метилаз является остаток глутамина в составе ультраконсервативного GGQ-мотива факторов терминации трансляции класса I (RF1, RF2 бактерий, aRF1 архей, митохондриальных mRF (mtRF) и цитоплазматического eRF1 эукариот). Показано, что этот остаток метилирован почти в 100% RF1 и RF2 E. coli [12, 43], в 50% митохондриального mRF1 дрожжей [4] и 50–75% цитоплазматических eRF1 дрожжей и человека [6, 39]. Однако в митохондриях человека митохондриальные факторы терминации трансляции и их производные метилированы на уровне всего нескольких процентов [13] (табл. 1, рис. 2).
HEMK 2 метилирует также CHD5 и NUT, участвующие в ремоделировании хроматина и развитии рака [20, 45] (табл. 1, рис. 2). Анализ доменов белков выявил еще девять потенциальных белков-субстратов HEMK2, в том числе участвующих в процессах трансляции (RRP1) и сигналинга (TGFB3, ARHGEF10). На возможность метилирования еще более 50 белков указывает анализ биоинформатических данных [45] (табл. 1, рис. 2).
Метилирование бактериальных RF1 и RF2 повышает константу гидролиза пептидил-тРНК (kcat) на всех трех стоп-кодонах в 1.1–450 раз [43, 47–50], особенно сильно при гидролизе пептидил-тРНК, оканчивающихся остатками пролина (P) или глицина (G). У E. coli найдено 119 и 279 таких белков соответственно; их трансляция может быть наиболее чувствительной к метилированию [47]. Разница в значениях kcat метилированной и неметилированной форм максимальна при кислых значениях pH (6–7) и минимальна при щелочных (7.0–8.5) [50]. Метилирование обуславливает также увеличение коэффициента специфичности гидролиза пептидил-тРНК (kcat/KM) с участием факторов терминации в 2–14 раз [43, 48, 49]. Интересно, что различие в значениях kcat и kcat/KМ у неметилированных и метилированных форм снижается при увеличении длины пептида в in vitro системе [43]. Метилирование RF2 повышает также уровень гидролиза пептидил-тРНК при независимой от стоп-кодона терминации с участием ArfA/RF2 в 20 раз [48, 49]. Однако отсутствие функциональной митохондриальной HEMK1 не оказывает явного влияния на трансляцию белка в митохондриях [13], однако и уровень метилирования митохондриальных факторов значительно меньше, чем у бактерий (несколько процентов против почти 100%) [12, 13, 43].
Рентгеноструктурный анализ как бактериальных [49], так и эукариотических [51] комплексов рибосомы и факторов терминации, а также докинг бактериальных комплексов in silico [52] показывают, что метилирование остатка глутамина фактора терминации стабилизирует положение этого остатка и особенно его боковой группы, которая иначе не имеет фиксированного положения, что приводит к облегчению атаки на молекулу воды [49]. Показано также, что метилирование эукариотического eRF1 не влияет на конформацию свободной молекулы белка [53]. Для эффективного метилирования эукариотический eRF1 и архейный aRF1 должны находиться в составе тройного комплекса с GTP и eRF3 (или aRF3=aEF1α), а сама метилаза – в гетерокомплексе с TRMT112 [1, 3, 6, 21, 26].
Предполагается, что процесс метилирования eRF1 в ядре с помощью дрожжевого MTQ2, ассоциированного с созревающими 60S субъединицами, может служить частью системы контроля качества рибосом [21].
На молекулярном уровне влияние метилирования eRF1 на его функционирование не показано. Известно лишь, что при нокауте NRF1 арабидопсиса происходит зависание рибосом в районе стоп-кодона, что может указывать на неэффективную терминацию трансляции в отсутствие метилирования eRF1 [32]. Обсуждается также возможность того, что отсутствие холодоустойчивости у клеток дрожжей с нокаутом MTQ2 может быть связано с нарушениями аппарата трансляции [4].
Не обнаружено кросс-реактивности между бактериальными и эукариотическими/архейными HEMK-подобными метилазами. Показано, что HEMK E. coli не способна метилировать eRF1, в том числе в присутствии eRF3 и GTP [6]. Однако различные эукариотические ортологи HEMK2 частично кросс-реактивны: в частности, HEMK2 человека эффективно метилирует eRF1 дрожжей и, наоборот, MTQ2 дрожжей эффективно метилирует eRF1человека [1, 6].
Остаток лизина в гистоне H4
Комплекс HEMK2–TRMT112 осуществляет монометилирование остатка лизина 12 в гистоне H4 (H4K12), что подтверждено как in vitro, так и in vivo. Это привело к появлению альтернативного названия HEMK2 – KMT9 (от Lysine (K) N-methyltransferase 9). В культурах клеток человека доля монометилированного гистона H4 составляет 0.04–0.12%, что сравнимо с долей иных модификаций гистоновых белков in vivo [2]. Метилирование остатков лизина и глутамина происходит в одном и том же активном центре, более того, метилируемый атом азота в обоих случаях расположен практически в одном и том же месте. Однако различаются некоторые аминокислотные остатки метилазы, важные для связывания, а также позиционирование белковой цепи субстрата. К примеру, HEMK2 с мутацией Y125A в NPPY-мотиве способна метилировать гистон H4, но не eRF1 [2, 20]. При этом, в отличие от eRF1, связывание гистона H4 происходит лишь в присутствии второго субстрата – SAM [2]. При pH 8.0 HEMK2–TRMT112 метилирует гистон Н4 in vitro значительно хуже, чем eRF1. Эффективность метилирования остатка лизина повышается при защелачивании реакционной смеси вследствие депротонирования аминогруппы [7].
Этой модификацией остатка лизина гистона H4 обогащены промоторы генов, связанных с регуляцией клеточного цикла. Считается, что именно нарушение монометилирования H4K12 приводит к развитию рака предстательной железы при нарушении нормальной экспрессии HEMK2 [2].
Соединения мышьяка
Соединения мышьяка токсичны для человека, они вызывают развитие рака, болезней нервной, эндокринной, сердечно-сосудистой, дыхательной, пищеварительной, половой и выделительной систем. Предполагается, что они участвуют в повреждении молекул ДНК, изменении профиля репарации и метилирования, хромосомных перестройках, нарушении клеточного цикла. В организме неорганические арсенаты восстанавливаются до трехвалентного состояния (арсениты) и подвергаются моно- и диметилированию. При этом физиологическая роль данного процесса окончательно не ясна и остается дискуссионной, так как метилированные арсениты считаются более токсичными, чем неметилированные [54, 55].
В клетках человека метилирование трехвалентного неорганического арсенита до моно- и диметильных производных осуществляется специализированной метилазой AS3MT [54], HEMK2 же участвует лишь в метилировании наиболее токсичного монометиларсенита до диметиларсенита (табл. 1, рис. 2). Уровень экспрессии AS3MT в клетках человека значительно выше, чем HEMK2 [8]. Показано, что сверхэкспрессия HEMK2 обеспечивает устойчивость клеточной культуры UROtsa человека к неорганическому арсениту и монометиларсениту [8]. На подобную функцию in vivo указывает корреляция между различными полиморфизмами HEMK2 и содержанием монометиларсенита в выделениях жительниц Аргентинских Анд [56]. Предполагается, что HEMK2 может метилировать монометиларсенит параллельно с AS3MT, что функционально значимо только при определенных условиях или в определенных тканях [8].
В отличие от HEMK2 человека, MTQ2 дрожжей метилирует не только монометиларсенит, но и неорганический трехвалентный арсенит с образованием монометиларсенита, что ведет к активации Hog1 в HOG-сигнальном пути (табл. 1). Это, по-видимому, блокирует возможность дальнейшего проникновения соединений мышьяка в клетку. Нокаут гена данного фермента повышает устойчивость дрожжей к неорганическому арсениту, но не к монометиларсениту. Для нормального функционирования в данном процессе метилаза должна находиться в комплексе с TRMT112 [8, 27].
Остаток дезоксиаденозина в ДНК
N6-метил-2'-дезоксиаденозин (6mA) – распространенная модификация остатка дезоксиаденозина в ДНК бактерий, связанная с функционированием систем рестрикции-модификации, репарации, репликации и сегрегации хромосом. В ДНК различных групп эукариот в последнее время также выявляют 6mA, уровень которого значительно ниже, чем у прокариот. Показано, что эта модификация может участвовать в регуляции хроматина, активации и подавлении экспрессии генов, в процессах эмбрионального развития. При этом физиологическая роль 6mA и его доля в различных группах эукариот отличаются. Наименьшая доля (порядка 10–2–10–4%) характерна для позвоночных [9, 57–59]. Стоит отметить, что остатки аденозина в РНК бактерий и эукариот также подвергаются этой модификации, что важно для функционирования многих тРНК и рРНК, но данный процесс осуществляют иные ферменты [60].
N6-метилирование дезоксиаденозина в ДНК эукариот катализируют разные ферменты. Согласно биоинформатическим данным, метилазы N6-аденозина и N6-дезоксиаденозина независимо появлялись в разных группах эукариот не менее 13 раз [58].
Предполагается, что в метилировании остатка дезоксиаденозина до 6mA в ДНК инфузорий, дрозофил, зеленых водорослей участвуют MTA70-подобные метилазы. У зеленых водорослей в этом могут участвовать и Dams-подобные метилазы [58]. Найдена и описана MTA70-подобная метилаза DAMT-1, катализирующая эту реакцию у C. elegans [61]. Однако у человека фермент, метилирующий остаток дезоксиаденозина до 6mA, не обнаружен. Предполагается, что эту реакцию может осуществлять HEMK2 (табл. 1, рис. 2). Во-первых, HEMK-подобные метилазы по структуре своего функционального домена близки к группе γ N6-аденин-ДНК-метилаз [62]. Во-вторых, по некоторым данным, HEMK2, содержащая NPPY-мотив, необходима для появления 6mA в ДНК in vivo и in vitro [9]. Доля 6mA в геномной ДНК человека положительно коррелирует с уровнем экспрессии HEMK2, а их уровень понижен в опухолевых клетках. Обратные эффекты вызывает деметилаза ALKBH1. Доля 6mA в образцах крови человека составляет около 0.056%. Большая абсолютная доля метилирования приходится на межгенные участки, а относительная – на экзоны [9]. Исходя из этого, HEMK2 имеет еще одно (альтернативное) название – N6AMT1 (от N6 Adenine-Specific DNA Methyltransferase 1).
Однако эти данные не подтверждены результатами других исследователей. Так обнаружено, что идентичный натуральному (свободный от тагов и дополнительных аминокислот) рекомбинантный HEMK2–TRMT112, полученный в клетках E. coli, способен метилировать как остаток глутамина в eRF1, так и остаток лизина в гистоне H4, но не мог метилировать остаток дезоксиаденозина в ДНК [7]. Это согласуется с данными, также не подтверждающими возможность HEMK2 катализировать реакцию метилирования остатка дезоксиаденозина [22, 31]. Кроме того, известен и другой потенциальный кандидат на роль метилазы остатка дезоксиаденозина– METL-4 – гомолог метилазы DAMT-1 C. elegans [58]. Таким образом, функция метилирования остатка дезоксиаденозина в ДНК с помощью HEMK2 остается спорной.
ЗАКЛЮЧЕНИЕ
Суммируя все имеющиеся на сегодняшний момент данные, можно заключить, что HEMK2 человека и ее ортологи у других эукариот и архей представляет собой необычный пример многофункциональной метилазы, катализирующей несколько различных реакций. Обычно многофункциональность фермента достигается за счет нескольких структурно обособленных доменов, каждый из которых катализирует собственную реакцию, аллостерически контролирует другой домен или взаимодействует с клеточным окружением, как это происходит у белков, обладающих дополнительными (moonlighting) функциями [63–66]. Однако известны и многофункциональные ферменты, в которых один и тот же домен (и даже один активный сайт) в зависимости от условий может катализировать несколько различных реакций, как это наблюдается у неразборчивых (promiscuous) ферментов. Часть этих реакций, вероятно, побочные, но другая часть может иметь физиологическое значение [65, 67–69]. HEMK2 можно отнести как раз к неразборчивым ферментам. Несмотря на то, что не ясно, насколько физиологически равнозначны реакции метилирования ее многочисленных субстратов, делаются попытки разработать цельную модель физиологической функции HEMK2 и ее эукариотических ортологов. Предложена модель, в которой SAM, в том числе через HEMK1 и HEMK2, служит основным регулятором трансляции в стволовых клетках кишечника, что влияет на активность их деления [36].
Не до конца понятен точный спектр субстратов HEMK2 и ее ортологов. Так, спорным выглядит метилирование ими остатка дезоксиаденозина [7, 22, 31]. Напротив, можно ожидать, что HEMK2 способна метилировать аспарагин так же, как глутамин. Обнаружено N-4-метилирование аспарагина в C-фикоцианине Anabaena variabilis. При этом остаток аспарагина находится после двух остатков глицина (GGN), что напоминает метилируемый мотив GGQ в факторах терминации. Замена аспарагина на глутамин приводит к in vivo метилированию глутамина, поэтому было выдвинуто предположение, что либо метилазы, осуществляющие данную реакцию, способны использовать в качестве субстрата оба аминокислотных остатка, либо за это ответственны два разных фермента [70, 71]. Однако показано, что HEMK2 не способна метилировать пептид, содержащий такую замену [45]. Таким образом, метилирование аспарагина в GGN-мотиве осуществляют, вероятно, близкие гомологи HEMK2.
HEMK2 это многофункциональный фермент, поэтому нельзя однозначно связать физиологические эффекты, вызываемые их нокаутом и нокдауном, с метилированием конкретных молекул, так как механистическую роль одновременного метилирования всех субстратов HEMK2 и ее ортологов до настоящего времени не изучали. Для того, чтобы научиться адресно контролировать процессы, зависимые от метилируемых молекул, требуется тщательное изучение механизмов модификации каждого субстрата на молекулярном уровне.
Помимо влияния на эффективность трансляции посредством метилирования eRF1, MTQ2 (дрожжевой ортолог HEMK2) участвует в биогенезе и экспорте созревших 60S субъединиц из ядра, а также в созревании 5.8S и 25S рРНК. Однако не очень понятно, почему такая функция ортолога связана с присутствием в нем отдельных аминокислот, участвующих в метилировании. Предполагается, что либо для правильного биогенеза 60S субъединиц и рРНК необходима проверка качества собранных рибосом с участием метилированного eRF1, либо в этот процесс вовлечены иные субстраты метилазы [21]. Также можно предположить, что эти аминокислотные остатки важны для поддержания правильной структуры метилазы, необходимой для ее функционирования в процессах созревания 60S субъединиц рибосомы. Остается непонятным, насколько этот процесс консервативен и касается других групп живых существ, включая человека.
Известно также, что некоторые факторы трансляции выполняют многочисленные функции вне трансляции. К примеру, eEF1α слизевика Dictyostelium может связывать и модулировать актин [72, 73], eEF1α человека и лягушки – связывать и участвовать в разрывании микротрубочек [74], растительный eEF1α – активировать фосфатидилинозитол-4-киназу [75]. А изучение дрожжей, мутантных по факторам терминации трансляции eRF1 и eRF3, указывает на то, что их нормальное функционирование связано не только непосредственно с осуществлением терминации, но и с многочисленными клеточными процессами [76]. Все это поднимает вопрос о значении взаимного влияния процесса терминации и иных клеточных процессов, в том числе и у рассматриваемых HEMK-подобных метилаз.
Мы предполагаем, что комплекс HEMK2–TRMT112 активен как в цитоплазме, где он метилирует монометиларсенит и eRF1, так и в ядре, где метилирует гистон H4, регуляторы хроматина CHD5 и NUT. В ядре этот комплекс может регулировать созревание рРНК и 60S субъединиц по аналогии с дрожжами. Более того, HEMK2 может быть вовлечена в метилирование ДНК. Однако множество аспектов этого процесса остаются не раскрытыми. В частности, не ясно, каким образом происходит и как регулируется транспорт HEMK2, TRMT112 или их комплекса через ядерную мембрану (табл. 1, рис. 2).
Работа по анализу влияния HEMK-подобных метилаз на терминацию трансляции проведена при поддержке гранта Российского научного фонда (№ 14-14-00487), физиологическое значение HEMK-подобных метилаз изучали при поддержке гранта Российского научного фонда (19-14-00349).
В настоящей работе не использовали людей или животных в качестве объектов исследования
Авторы сообщают об отсутствии конфликта интересов.
Список литературы
Figaro S., Scrima N., Buckingham R.H., Heurgué-Hamard V. (2008) HemK2 protein, encoded on human chromosome 21, methylates translation termination factor eRF1. FEBS Lett. 582, 2352–2356.
Metzger E., Wang S., Urban S., Willmann D., Schmidt A., Offermann A., Allen A., Sum M., Obier N., Cottard F., Ulferts S., Preca B.-T., Hermann B., Maurer J., Greschik H., Hornung V., Einsle O., Perner S., Imhof A., Jung M., Schüle R. (2019) KMT9 monomethylates histone H4 lysine 12 and controls proliferation of prostate cancer cells. Nat. Struct. Mol. Biol. 26, 361–371.
van Tran N., Muller L., Ross R.L., Lestini R., Létoquart J., Ulryck N., Limbach P.A., de Crécy-Lagard V., Cianférani S., Graille M. (2018) Evolutionary insights into Trm112-methyltransferase holoenzymes involved in translation between archaea and eukaryotes. Nucl. Acids Res. 46, 8483–8499.
Polevoda B., Span L., Sherman F. (2006) The yeast translation release factors Mrf1p and Sup45p (eRF1) are methylated, respectively, by the methyltransferases Mtq1p and Mtq2p. J. Biol. Chem. 281, 2562–2571.
Polevoda B., Sherman F. (2007) Methylation of proteins involved in translation. Mol. Microbiol. 65, 590–606.
Heurgué-Hamard V., Champ S., Mora L., Merkoulova-Rainon T., Kisselev L.L., Buckingham R.H. (2005) The glutamine residue of the conserved GGQ motif in Saccharomyces cerevisiae release factor eRF1 is methylated by the product of the YDR140w gene. J. Biol. Chem. 280, 2439–2445.
Woodcock C.B., Yu D., Zhang X., Cheng X. (2019) Human HemK2/KMT9/N6AMT1 is an active protein methyltransferase, but does not act on DNA in vitro, in the presence of Trm112. Cell Discov. 5, 50.
Ren X., Aleshin M., Jo W.J., Dills R., Kalman D.A., Vulpe C.D., Smith M.T., Zhang L. (2011) Involvement of N-6 adenine-specific DNA methyltransferase 1 (N6AMT1) in arsenic biomethylation and its role in arsenic-induced toxicity. Environ. Hlth Perspect. 119, 771–777.
Xiao C.-L., Zhu S., He M., Chen D., Zhang Q., Chen Y., Yu G., Liu J., Xie S.-Q., Luo F., Liang Z., Wang D.-P., Bo X.-C., Gu X.-F., Wang K., Yan G.-R. (2018) N6-methyladenine DNA modification in the human genome. Mol. Cell. 71, 306–318.e7.
Heurgue-Hamard V. (2002) The hemK gene in Escherichia coli encodes the N5-glutamine methyltransferase that modifies peptide release factors. EMBO J. 21, 769–778.
Ishizawa T., Nozaki Y., Ueda T., Takeuchi N. (2008) The human mitochondrial translation release factor HMRF1L is methylated in the GGQ motif by the methyltransferase HMPrmC. Biochem. Biophys. Res. Commun. 373, 99–103.
Nakahigashi K., Kubo N., Narita S., Shimaoka T., Goto S., Oshima T., Mori H., Maeda M., Wada C., Inokuchi H. (2002) HemK, a class of protein methyl transferase with similarity to DNA methyl transferases, methylates polypeptide chain release factors, and hemK knockout induces defects in translational termination. Proc. Natl. Acad. Sci. 99, 1473–1478.
Fang Q., Kimura Y. (2021) Mammalian HEMK1 methylates glutamine residue of the GGQ motif of mitochondrial release factors. Research Square. 1–21. https://doi.org/10.21203/rs.3.rs-1049003/v1
Nakayashiki T., Nishimura K., Inokuchi H. (1995) Cloning and sequencing of a previously unidentified gene that is involved in the biosynthesis of heme in Escherichia coli. Gene. 153, 67–70.
Sergiev P.V., Aleksashin N.A., Chugunova A.A., Polikanov Y.S., Dontsova O.A. (2018) Structural and evolutionary insights into ribosomal RNA methylation. Nat. Chem. Biol., 14, 226–235.
Schubert H.L., Phillips J.D., Hill C.P. (2003) Structures along the catalytic pathway of PrmC/HemK, an N5-glutamine AdoMet-dependent methyltransferase. Biochemistry. 42, 5592–5599.
Liger D., Mora L., Lazar N., Figaro S., Henri J., Scrima N., Buckingham R.H., van Tilbeurgh H., Heurgué-Hamard V., Graille M. (2011) Mechanism of activation of methyltransferases involved in translation by the Trm112 ‘hub’ protein. Nucl. Acids Res. 39, 6249–6259.
Martin J.L., McMillan F.M. (2002) SAM (dependent) I AM: the S-adenosylmethionine-dependent methyltransferase fold. Curr. Opin. Struct. Biol. 12, 783–793.
Schubert H.L., Blumenthal R.M., Cheng X. (2003) Many paths to methyltransfer: a chronicle of convergence. Trends Biochem. Sci. 28, 329–335.
Gao J., Wang B., Yu H., Wu G., Wan C., Liu W., Liao S., Cheng L., Zhu Z. (2020) Structural insight into HEMK2–TRMT112-mediated glutamine methylation. Biochem. J. 477, 3833–3838.
Lacoux C., Wacheul L., Saraf K., Pythoud N., Huvelle E., Figaro S., Graille M., Carapito C., Lafontaine D.L.J., Heurgué-Hamard V. (2020) The catalytic activity of the translation termination factor methyltransferase Mtq2-Trm112 complex is required for large ribosomal subunit biogenesis. Nucl. Acids Res. 48, 12310–12325.
Yang Z., Shipman L., Zhang M., Anton B.P., Roberts R.J., Cheng X. (2004) Structural characterization and comparative phylogenetic analysis of Escherichia coli HemK, a protein (N5)-glutamine methyltransferase. J. Mol. Biol. 340, 695–706.
Holtkamp W., Kokic G., Jager M., Mittelstaet J., Komar A.A., Rodnina M.V. (2015) Cotranslational protein folding on the ribosome monitored in real time. Science. 350, 1104–1107.
Mercier E., Rodnina M.V. (2018) Co-translational folding trajectory of the HemK helical domain. Biochemistry. 57, 3460–3464.
Liutkute M., Maiti M., Samatova E., Enderlein J., Rodnina M.V. (2020) Gradual compaction of the nascent peptide during cotranslational folding on the ribosome. ELife. 9, e60895.
Heurgué-Hamard V., Graille M., Scrima N., Ulryck N., Champ S., van Tilbeurgh H., Buckingham R.H. (2006) The zinc finger protein Ynr046w is plurifunctional and a component of the eRF1 methyltransferase in yeast. J. Biol. Chem. 281, 36140–36148.
Lee J., Levin D.E. (2018) Intracellular mechanism by which arsenite activates the yeast stress MAPK Hog1. Mol. Biol. Cell. 29, 1904–1915.
Bourgeois G., Létoquart J., van Tran N., Graille M. (2017) Trm112, a protein activator of methyltransferases modifying actors of the eukaryotic translational apparatus. Biomolecules. 7, 7.
Leetsi L., Õunap K., Abroi A., Kurg R. (2019) The common partner of several methyltransferases TRMT112 regulates the expression of N6AMT1 isoforms in mammalian cells. Biomolecules. 9, 422.
Sardana R., Johnson A.W. (2012) The methyltransferase adaptor protein Trm112 is involved in biogenesis of both ribosomal subunits. Mol. Biol. Cell. 23, 4313–4322.
Ratel D., Ravanat J.-L., Charles M.-P., Platet N., Breuillaud L., Lunardi J., Berger F., Wion D. (2006) Undetectable levels of N6-methyl adenine in mouse DNA: cloning and analysis of PRED28, a gene coding for a putative mammalian DNA adenine methyltransferase. FEBS Lett. 580, 3179–3184.
Kailasam S., Singh S., Liu M., Lin C., Yeh K. (2020) A HemK class glutamine-methyltransferase is involved in the termination of translation and essential for iron homeostasis in Arabidopsis. New Phytol. 226, 1361–1374.
Mora L., Heurgué-Hamard V., de Zamaroczy M., Kervestin S., Buckingham R.H. (2007) Methylation of bacterial release factors RF1 and RF2 is required for normal translation termination in vivo. J. Biol. Chem. 282, 35638–35645.
Kakkanat A., Phan M.-D., Lo A.W., Beatson S.A., Schembri M.A. (2017) Novel genes associated with enhanced motility of Escherichia coli ST131. PLoS One. 12, e0176290.
Nordquist S.K., Smith S.R., Pierce J.T. (2018) Systematic functional characterization of human 21st chromosome orthologs in Caenorhabditis elegans. G3 Genes Genomes Genet. 8, 967–979.
Obata F., Tsuda-Sakurai K., Yamazaki T., Nishio R., Nishimura K., Kimura M., Funakoshi M., Miura M. (2018) Nutritional control of stem cell division through S-adenosylmethionine in Drosophila intestine. Dev. Cell. 44, 741–751.e3.
Fagerberg L., Hallström B.M., Oksvold P., Kampf C., Djureinovic D., Odeberg J., Habuka M., Tahmasebpoor S., Danielsson A., Edlund K., Asplund A., Sjöstedt E., Lundberg E., Szigyarto C.A.-K., Skogs M., Takanen J.O., Berling H., Tegel H., Mulder J., Nilsson P., Schwenk J., Lindskog C., Danielsson F., Mardinoglu A., Sivertsson A., von Feilitzen K., Forsberg M., Zwahlen M., Olsson I., Navani S., Huss M., Nielsen J., Ponten F., Uhlén M. (2014) Analysis of the human tissue-specific expression by genome-wide integration of transcriptomics and antibody-based proteomics. Mol. Cell. Proteom. 13, 397–406.
Arroyo J.D., Jourdain A.A., Calvo S.E., Ballarano C.A., Doench J.G., Root D.E., Mootha V.K. (2016) A genome-wide CRISPR death screen identifies genes essential for oxidative phosphorylation. Cell Metabolism. 24, 875–885.
Liu P., Nie S., Li B., Yang Z.-Q., Xu Z.-M., Fei J., Lin C., Zeng R., Xu G.-L. (2010) Deficiency in a glutamine-specific methyltransferase for release factor causes mouse embryonic lethality. Mol. Cell. Biol. 30, 4245–4253.
Liang B., Shao Y., Long F., Jiang S.-J. (2016) Predicting diagnostic gene biomarkers for non-small-cell lung cancer. BioMed Res. Internat. 2016, 1–8.
Spitali P., Hettne K., Tsonaka R., Charrout M., van den Bergen J., Koeks Z., Kan H.E., Hooijmans M.T., Roos A., Straub V., Muntoni F., Al-Khalili-Szigyarto C., Koel-Simmelink M.J.A., Teunissen C.E., Lochmül-ler H., Niks E.H., Aartsma-Rus A. (2018) Tracking disease progression non-invasively in Duchenne and Becker muscular dystrophies. J. Cachexia, Sarcopenia Muscle. 9, 715–726.
Wagner P., Yin T., Brügemann K., Engel P., Weimann C., Schlez K., König S. (2021) Genome-wide associations for microscopic differential somatic cell count and specific mastitis pathogens in Holstein cows in compost-bedded pack and cubicle farming systems. Animals. 11, 1839.
Dincbas-Renqvist V. (2000) A post-translational modification in the GGQ motif of RF2 from Escherichia coli stimulates termination of translation. EMBO J. 19, 6900–6907.
Desai N., Yang H., Chandrasekaran V., Kazi R., Min-czuk M., Ramakrishnan V. (2020) Elongational stalling activates mitoribosome-associated quality control. Science. 370, 1105–1110.
Kusevic D., Kudithipudi S., Jeltsch A. (2016) Substrate specificity of the HEMK2 protein glutamine methyltransferase and identification of novel substrates. J. Biol. Chem. 291, 6124–6133.
Schubert H.L. (2006) 14 modification of glutamine residues in proteins involved in translation. Enzymes. 24, 437–453.
Pierson W.E., Hoffer E.D., Keedy H.E., Simms C.L., Dunham C.M., Zaher H.S. (2016) Uniformity of peptide release is maintained by methylation of release factors. Cell Rep. 17, 11–18.
Zeng F., Jin H. (2016) Peptide release promoted by methylated RF2 and ArfA in nonstop translation is achieved by an induced-fit mechanism. RNA. 22, 49–60.
Zeng F., Jin H. (2018) Conformation of methylated GGQ in the peptidyl transferase center during translation termination. Sci. Rep. 8, 2349.
Indrisiunaite G., Pavlov M.Y., Heurgué-Hamard V., Ehrenberg M. (2015) On the pH dependence of class-1 RF-dependent termination of mRNA translation. J. Mol. Biol. 427, 1848–1860.
Li W., Chang S.T.-L., Ward F.R., Cate J.H.D. (2020) Selective inhibition of human translation termination by a drug-like compound. Nat. Commun. 11, 4941.
Trobro S., Åqvist J. (2007) A model for how ribosomal release factors induce peptidyl-tRNA cleavage in termination of protein synthesis. Mol. Cell. 27, 758–766.
Andér M., Åqvist J. (2009) Does glutamine methylation affect the intrinsic conformation of the universally conserved GGQ motif in ribosomal release factors? Biochemistry. 48, 3483–3489.
Thomas D.J., Li J., Waters S.B., Xing W., Adair B.M., Drobna Z., Devesa V., Styblo M. (2007) Arsenic (+3 oxidation state) methyltransferase and the methylation of arsenicals. Exp. Biol. Med. 232, 3–13.
Kapaj S., Peterson H., Liber K., Bhattacharya P. (2006) Human health effects from chronic arsenic poisoning – a review. J. Env. Sci. Hlth. Part A. 41, 2399–2428.
Harari F., Engström K., Concha G., Colque G., Vah-ter M., Broberg K. (2013) N6-adenine-specific DNA methyltransferase 1 (N6AMT1) polymorphisms and arsenic methylation in Andean women. Environ. Health Perspect. 121, 797–803.
Parashar N.C., Parashar G., Nayyar H., Sandhir R. (2018) N6-adenine DNA methylation demystified in eukaryotic genome: from biology to pathology. Biochimie. 144, 56–62.
Iyer L.M., Zhang D., Aravind L. (2016) Adenine methylation in eukaryotes: apprehending the complex evolutionary history and functional potential of an epigenetic modification. BioEssays. 38, 27–40.
Ratel D., Ravanat J.-L., Berger F., Wion D. (2006) N6-methyladenine: the other methylated base of DNA. BioEssays. 28, 309–315.
Sergiev P.V., Golovina A.Y., Osterman I.A., Nesterchuk M.V., Sergeeva O.V., Chugunova A.A., Evfratov S.A., Andreianova E.S., Pletnev P.I., Laptev I.G., Petriukov K.S., Navalayeu T.I., Koteliansky V.E., Bogdanov A.A., Dontsova O.A. (2016) N6-methylated adenosine in RNA: from bacteria to humans. J. Mol. Biol. 428, 2134–2145.
Greer E.L., Blanco M.A., Gu L., Sendinc E., Liu J., Aristizábal-Corrales D., Hsu C.-H., Aravind L., He C., Shi Y. (2015) DNA methylation on N6-adenine in C. elegans. Cell. 161, 868–878.
Bujnicki J.M., Radlinska M. (1999) Is the HemK family of putative S-adenosylmethionine-dependent methyltransferases a “missing” zeta subfamily of adenine methyltransferases? A hypothesis. Internat. Union Biochem. Mol. Biol. Life. 48, 247–249.
Kirschner K., Bisswanger H. (1976) Multifunctional proteins. Annu. Rev. Biochem. 45, 143–166.
Jeffery C.J. (1999) Moonlighting proteins. Trends Biochem. Sci. 24, 8–11.
Copley S. (2003) Enzymes with extra talents: moonlighting functions and catalytic promiscuity. Curr. Opin. Chem. Biol. 7, 265–272.
Moore B.D. (2004) Bifunctional and moonlighting enzymes: lighting the way to regulatory control. Trends Plant Sci. 9, 221–228.
O’Brien P.J., Herschlag D. (1999) Catalytic promiscuity and the evolution of new enzymatic activities. Chem. Biol. 6, R91–R105.
Hult K., Berglund P. (2007) Enzyme promiscuity: mechanism and applications. Trends Biotechnol. 25, 231–238.
Seelig B. (2017) Multifunctional enzymes from reduced genomes – model proteins for simple primordial metabolism? Mol. Microbiol. 105, 505–507.
Thomas B.A., McMahon L.P., Klotz A.V. (1995) N5-methylasparagine and energy-transfer efficiency in C‑phycocyanin. Biochemistry. 34, 3758–3770.
Clarke S. (2002) The methylator meets the terminator. Proc. Natl. Acad. Sci. USA. 99, 1104–1106.
Demma M., Warren V., Hock R., Dharmawardhane S., Condeelis J. (1990) Isolation of an abundant 50 000-dalton actin filament bundling protein from Dictyostelium amoebae. J. Biol. Chem. 265, 2286–2291.
Edmonds B.T., Murray J., Condeelis J. (1995) pH regulation of the F-actin binding properties of Dictyostelium elongation factor 1α. J. Biol. Chem. 270, 15222–15230.
Shiina N., Gotoh Y., Kubomura N., Iwamatsu A., Nishida E. (1994) Microtubule severing by elongation factor 1α. Science. 266, 282–285.
Yang W., Burkhart W., Cavallius J., Merrick W.C., Boss W.F. (1993) Purification and characterization of a phosphatidylinositol 4-kinase activator in carrot cells. J. Biol. Chem. 268, 392–398.
Inge-Vechtomov S., Zhouravleva G., Philippe M. (2003) Eukaryotic release factors (eRFs) history. Biol. Cell. 95, 195–209.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


