Молекулярная биология, 2022, T. 56, № 3, стр. 355-390
Эффект положения гена: роль локального окружения в регуляции активности генов
Л. В. Болдырева a, *, Е. Н. Андреева a, А. В. Пиндюрин a
a Институт молекулярной и клеточной биологии Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
* E-mail: asd@mcb.nsc.ru
Поступила в редакцию 14.07.2021
После доработки 25.08.2021
Принята к публикации 25.08.2021
- EDN: BIYHRE
- DOI: 10.31857/S0026898422030041
Аннотация
При перемещении гена в результате хромосомных перестроек или при встраивании идентичных генетических конструкций в разные районы генома наблюдается так называемый эффект положения гена – явление, при котором уровень экспрессии одного и того же гена существенно зависит от его положения в геноме. Геном эукариот имеет доменную организацию, а активность генов в пределах таких доменов определяется не столько нуклеотидной последовательностью гена, сколько структурой окружающего хроматина, т.е. эпигенетически. Хроматин представляет собой сложный комплекс ДНК, РНК и ассоциированных с ними структурных и регуляторных белков. Эпигенетический статус хроматина определяется целым рядом факторов: временем репликации данного участка генома, регуляторными мотивами ДНК, контактами с внутренней ядерной оболочкой (ламиной) и другими районами хромосом (топологически ассоциированные домены). Эффект положения гена заключается в изменении его эпигенетического состояния и является уникальным инструментом для исследования молекулярных и биохимических процессов. Понимание молекулярных механизмов эффекта положения гена у человека имеет важное значение в клинической сфере, в частности, для выявления и лечения ретровирусных инфекций, поскольку локальный состав хроматина может определять, например, переход в латентное/активное состояние такой инфекции, как ВИЧ. Кроме того, большое число нейродегенеративных заболеваний человека обусловлено эпигенетической инактивацией генов в результате экспансии коротких повторов. Наконец, для полноценного применения методов генной терапии важно владеть знаниями и подходами, которые с достаточной точностью могут обеспечивать необходимый уровень экспрессии внедряемых трансгенов.
ИСТОРИЯ ОТКРЫТИЯ ЭФФЕКТА ПОЛОЖЕНИЯ, ЕГО ТИПЫ
Феномен эффекта положения (ЭП) гена был описан у дрозофилы уже вскоре после того, как генетики пришли к определению понятия гена как дискретной единицы наследственной информации, расположенной и функционирующей в определенном участке хромосомы. Наблюдения феномена ЭП свидетельствовали об обратимом изменении проявления признака в результате изменения только лишь положения гена, определяющего этот признак, в геноме. Сегодня термин ЭП охватывает все случаи изменения экспрессии гена за счет каких-либо отклонений от его нормального хромосомного окружения (контекста), но не связанного с мутациями или делециями самого гена.
Еще в 1925 году А. Стертевант показал, что фенотипическое проявление двух (и более) копий гена Bar у дрозофилы существенно различается, когда они находятся в одной хромосоме или в разных гомологичных хромосомах [1] (рис. 1а). Явление, обнаруженное A. Стертевантом, отличается от других типов эффекта положения: мутантный фенотип устанавливается стабильно, поэтому позже Э. Льюис назвал этот феномен стабильным эффектом положения [2]. В 1930 году Г. Дж. Меллер обнаружил еще одно удивительное явление – мозаичное проявление фенотипа гена white у дрозофилы, обусловленное хромосомными перестройками, переносящими этот ген в непосредственную близость к прицентромерному гетерохроматину (ГХ) [3], представляющему собой неактивную часть генома (подробнее см. раздел “Состояния хроматина, эпигенетические метки и гистоновый код”). При этом ген инактивируется лишь в части клеток (рис. 1б). Такой вариант ЭП, названный эффектом положения мозаичного типа, дополнительно свидетельствовал о том, что сам ген остается неизмененным, а инактивация происходит в результате некоего изменения его состояния [2, 4–6]. Эксперименты по реверсии хромосомных перестроек показали восстановление нормального уровня активности гена, кроме тех случаев, когда рядом с ним оставался обширный блок ГХ [7–9]. Из этих наблюдений родилось понятие спрединга, или гетерохроматинизации – распространения свойств ГХ на прилежащие гены [10]. Показано, что степень инактивации гена, подверженного ЭП, напрямую зависит от расстояния до границы с ГХ [2, 11]. Более поздние исследования, выполненные также на дрозофиле, показали, что для индукции ЭП важна внутренняя организация блока ГХ. В частности, внутренняя перестройка ГХ без изменения его количества может приводить к релаксации ЭП [12, 13]. Изучение ЭП, возникающего в результате гетерохроматинизации тандемных вставок трансгенов, созданных на основе P-элемента (ДНК-транспозона дрозофилы) также подтвердило важность внутренней организации ГХ блока для ЭП [14]. Однако до сих пор не вполне ясно, что играет главную роль в процессе гетерохроматинизации при ЭП – количество ГХ или именно присутствие, количество и расположение неких инактивирующих элементов. Например, в случае вставки Р-элемента вблизи прицентромерного ГХ хромосомы 2 не обнаружено корреляции между степенью инактивации гена white в транспозоне и размером и организацией прилежащего ГХ блока [15]. С другой стороны, при инверсии In(1LR)pn2a в хромосоме X дрозофилы степень инактивации генов постепенно снижается при уменьшении количества прилежащего ГХ [13]. Вместе эти факты могут объясняться сложностью общей организации ГХ доменов, и значением для ЭП как комбинации каких-то инактивирующих мотивов ДНК, так и общего размера ГХ блока. Явление гетерохроматинизации наглядно показано и изучено также в политенных хромосомах слюнных желез личинок дрозофилы, в которых с помощью световой микроскопии можно видеть домены репрессированного и активного хроматина, выявляемые как диски и междиски соответственно [16–18]. Цитогенетические исследования политенных хромосом в разных случаях ЭП дали возможность детально изучить морфологию хроматина, в частности выявить как непрерывную, так и прерывистую гетерохроматинизацию в результате ЭП [19, 20].
Рис. 1.
Типы и характерные свойства эффекта положения (ЭП) гена у D. melanogaster. а – Стабильный ЭП гена Bar. б – Мозаичный ЭП генов white и yellow, вызванных инверсией In(1)wm и свободной дупликацией Dp(1;f)1187 хромосомы Х соответственно. в – Схема участков генома, способных вызывать ЭП встроек P-элементов. г – Электронно-микроскопические снимки ультратонких срезов района 65AB политенных хромосом слюнных желез личинок (междиск в норме, а также при 1 и 13 копиях P-элемента, формирующих новый тонкий (отмечен стрелкой) и крупный (отмечен стрелкой и скобкой) диск соответственно) (Любезно предоставлены В.Ф. Семешиным и В.В. Шломой и частично опубликованы в свободном доступе [39]). д – Саузерн-блот-анализ недорепликации ДНК гена white в политенных хромосомах слюнных желез (СЖ) относительно диплоидных тканей (Д) личинок дрозофилы в результате сдвига репликации в более позднюю временную стадию, как следствие гетерохроматинизации в результате ЭП, вызванного инверсией In(1)wm, и восстановление политенизации при мутации гена-модификатора ЭП SuUR. е – Непрямое флуоресцентное иммуноокрашивание политенных хромосом слюнных желез личинок, показывающее связывание белка гетерохроматина SUUR c районами 2B и 2E, которые подвергаются гетерохроматинизации в результате ЭП при дупликации части хромосомы Х (Dp(1;1)pn2B).
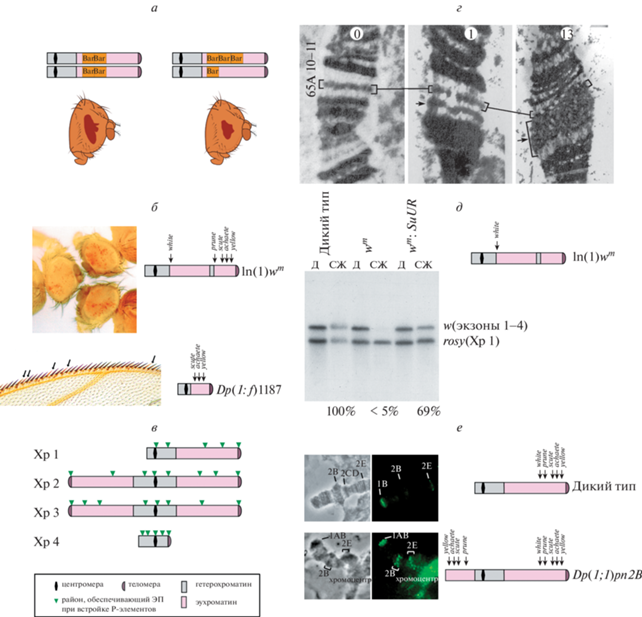
Помимо инактивации генов в результате гетерохроматинизации, у дрозофилы описан и реципрокный эффект: ген cubitus interruptus, в норме расположенный в окружении ГХ хромосомы 4, инактивировался при его перемещении в активную часть генома – эухроматин. Это явление, названное эффектом Дубинина, впоследствии наблюдали и у других генов ГХ [21–25]. Был сделан вывод, что, по всей вероятности, любой ген может быть подвержен ЭП – изменению уровня экспрессии вследствие влияния локального окружения хроматина. При этом далеко не любой локальный контекст способен приводить к ЭП гена [26].
В 1980-е годы благодаря разработке метода встраивания ДНК-конструкций в геном (P-элемент опосредованная трансформация) [27, 28] стали находить многочисленные примеры существенных различий в активности генов rosy и white, маркирующих трансген по окраске глаз дрозофилы, в зависимости от района встройки в геном в составе искусственных P-элементов, в том числе и случаи ЭП [29, 30] (рис. 1в). Несколько масштабных скрининговых исследований активности вставок P-элемента в случайные локусы генома, показали, что вызывать ЭП способны районы прицентромерного ГХ всех хромосом дрозофилы. При этом был обнаружен ЭП трансгенов, встроенных в теломерные районы хромосом, так называемый теломерный ЭП. Самыми удивительными стали примеры инактивации трансгенов, картированных в некоторых участках эухроматиновой части хромосом, до этого момента считавшейся полностью активной частью генома [31–33]. В этот период сформировалось понятие интеркалярного ГХ – локальных участков генома, имеющих репрессированный статус (по множеству черт сходный с прицентромерным ГХ), диффузно разбросанных во всей эухроматиновой части генома [31, 34–36]. Для ЭП вставок Р-элемента, как и для реверсий хромосомных перестроек, показано восстановление экспрессии репортерного гена при перемещении трансгена из инактивирующего хромосомного контекста [31, 32, 37]. При помощи просвечивающей электронной микроскопии ультратонких срезов политенных хромосом дрозофилы обнаружили, что P-элементные вставки активного или неактивного генетического материала создают соответствующие морфологические ультраструктуры [38, 39] (рис. 1г). По данным световой микроскопии неоднократно показано, что в политенных хромосомах дрозофилы район, подверженный ЭП, претерпевает морфологические изменения, подвергаясь видимой гетерохроматинизации, визуально компактизуясь и приобретая ряд свойств ГХ, в том числе позднюю репликацию, приводящую к недорепликации ДНК [19, 40–43] (рис. 1д). Методом иммуноокрашивания установлено, что в районах, подверженных гетерохроматинизации в результате ЭП, локализуются белки хроматина, в норме отсутствующие в этих районах [44, 45] (рис. 1е).
Еще один вариант ЭП – феномен, названный транс-инактивацией. В отличие от описанной выше гетерохроматинизации, распространяющейся вдоль хромосомы от расположенных на ней в цис-положении блоков ГХ, при транс-инактивации сайленсинг генов происходит в другой хромосоме. Так, у дрозофилы при ЭП гена brown аллель bwD, подверженный ЭП, доминирует над нормальной копией гена, находящейся в гомологичной хромосоме. Инактивация в этом случае вызвана большим фрагментом (1.6 млн.п.н.) прицентромерного ГХ хромосомы 2, помещенного в локус bwD посредством сложной транспозиции. Оказалось, что такая инактивация нормального аллеля bw происходит вследствие его гомологичной конъюгации с подверженным ЭП аллелем bwD, приводящей к попаданию нормального аллеля под влияние прицентромерного ГХ в интерфазном ядре [46–49]. Затем обнаружили еще несколько случаев ЭП, возникающих аналогичным путем [50–55]. Это свидетельствует о транс-инактивации при ЭП как об одном из механизмов эпигенетической инактивации [56–61].
В 1990-е годы начались активные поиски и изучение ЭП и у других организмов, помимо дрозофилы. У дрожжей обнаружен как прицентромерный, так и теломерный ЭП [31, 62]. При этом у двух видов дрожжей ГХ-районы генома формируются разными молекулярными механизмами [63, 64]. Получены убедительные доказательства того, что у дрожжей Schizosaccharomyces pombe ЭП возникает при перемещении генов в район центромер [65], а у Saccharomyces cerevisiae – при гетерохроматинизации в теломерных районах [66]. Благодаря такой межвидовой дифференциации, дрожжи стали уникальной моделью, которая заложила основы понимания различных механизмов формирования ГХ, вызывающего ЭП гена в прицентромерных и теломерных районах. Теломерный ЭП, обнаруженный как у дрожжей, так и у дрозофилы, имеет лишь часть свойств, характерных для ЭП, вызванного другими типами репрессированного хроматина (прицентромерным и интеркалярным) [67].
Изучение ЭП у млекопитающих (по большей части у мыши и человека) стало возможным лишь в начале 21 века, когда появились технические условия и необходимый инструментарий молекулярной генетики. Однако то, что существенная часть генов-модификаторов ЭП, обнаруженных у дрозофилы, высоко консервативна и имеет ряд гомологов и ортологов как у млекопитающих, так и у человека, дает основание предполагать, что и молекулярные механизмы репрессии, приводящие к ЭП, эволюционно консервативны [68–71]. Феномен ЭП у млекопитающих показан путем анализа тысяч параллельных репортерных встроек TRIP (Thousands of Reporters Integrated in Parallel) в геноме культивируемых стволовых клеток мыши [72, 73]. Проанализировав экспрессию тысяч встроек в различные локации, обнаружили, что уровень экспрессии одного и того же репортерного гена может различаться в ~1000 раз в зависимости от конкретного сайта встройки в геном. Другой крупномасштабный генетический скрининг проведен у мыши при помощи тандемной встройки GFP-экспрессирующего трансгена, имеющего мозаичный фенотип, чтобы выявить генетические факторы, способные модифицировать ЭП у млекопитающих [74, 75]. В результате были охарактеризованы около 40 генов-модификаторов ЭП мыши, большинство из которых оказались гомологами соответствующих генов дрозофилы [76] (см. раздел “Гены-модификаторы ЭП”).
Подверженность генов человека ЭП показана в ряде экспериментов, в которых репортерные конструкции встраивали в различные участки генома культивируемых клеток [77–80]. Методом 3D-FISH выявлены активные и репрессированные домены генома человека, обеспечивающие значительное повышение или снижение уровня экспрессии интегрированной в них репортерной конструкции [77]. Сформулировано также представление о транскрипционных территориях в геноме млекопитающих, впервые обнаруженных для совместно регулируемых кластеров генов, таких как Hox. Нарушения таких кластеров приводят к развитию заболеваний, в частности, лейкоза [81]. С помощью серийного анализа экспрессии генов SAGE (Serial Analysis of Gene Expression) построена карта транскриптома человека [82]. По уровню транскрипционной активности были выделены домены двух классов. Высоко экспрессирующиеся домены обогащены основаниями C и G, имеют более высокую плотность генов и повышенный индекс коротких диспергированных повторов (Short Interspersed Nuclear Elements, SINE), а сами гены содержат преимущественно короткие интроны. Cлабо экспрессирующиеся домены имеют противоположные свойства [83, 84]. Домены этих типов описаны и в геноме мыши [85], более того, их границы оказались достаточно консервативными и воспроизводимыми в сравнении с геномом человека [86]. Большинство генов, принадлежащих высоко экспрессирующимся доменам, относятся к генам домашнего хозяйства или экспрессируются в большинстве тканей, хотя в этих доменах встречается и небольшой процент узкоспецифичных генов [84, 87]. Вставки трансгенов в высоко и слабо экспрессирующиеся домены часто характеризуются соответствующими изменениями уровня экспрессии [77–80].
Один из самых распространенных молекулярных механизмов эпигенетической репрессии, обнаруженных у млекопитающих, – метилирование ДНК, у дрозофилы практически полностью отсутствует [88]. Метилирование ДНК играет важную роль в репрессии инородных встроек в геном млекопитающих, в том числе ретровирусных [89, 90]. Нарушение метилирования ДНК в геноме человека обнаружено при разных видах рака [91, 92]. И, безусловно, метилирование CpG-островков играет решающую роль в репрессии генов при ЭП, возникающем в результате экспансии тандемных повторов, что приводит к развитию различных нарушений и синдромов у человека [93, 94] (подробнее см. раздел “Клинические аспекты ЭП”). С одной стороны, отсутствие этого эпигенетического механизма у дрозофилы на десятилетия задержало понимание его роли при ЭП. С другой же, подобное различие позволяет изолированно изучать молекулярные механизмы эпигенетической репрессии как с участием метилирования ДНК, так и без него [95].
Представления о молекулярных механизмах, лежащих в основе ЭП гена, значительно расшились за последние десятилетия и начинают использоваться как в фундаментальных исследованиях, так и в клинической практике. Во-первых, это относится к выявлению и лечению ретровирусных инфекций. Локальный состав хроматина может определять переход ВИЧ в латентное (вплоть до недетектируемого) или активное состояние. При заражении ВИЧ не только активно реплицируется в Т-лимфоцитах, но также может переходить в латентную фазу. Таким образом вирус сохраняется в клетках человека и может активироваться уже после окончания антиретровирусной терапии [96, 97]. Во-вторых, с ЭП генов связано большое количество генетически обусловленных заболеваний и отклонений в развития человека. Такие заболевания часто связаны с ЭП, возникающим в результате экспансии (увеличения числа копий) тандемных повторов вблизи генов [98, 99] (см. раздел “Клинические аспекты эффекта положения”).
Все приведенные примеры ЭП гена иллюстрируют существенную зависимость уровня экспрессии гена от его локального окружения. Нужно отметить, что до сих пор еще нет точного понимания молекулярных механизмов активации и инактивации гена при ЭП, особенно в случае мозаичных вариантов ЭП (когда стохастически в части клеток одной ткани (органа) ген инактивирован, а в других активен). И до сих пор открытыми остаются вопросы о сходстве или принципиальных различиях молекулярных механизмов инактивации/реактивации генов при разных типах ЭП. Ниже мы последовательно рассмотрим функционирование уровней эпигенетической инактивации, которые могут обеспечивать репрессию генов при ЭП. Исчерпывающее описание разнообразных случаев ЭП и их обсуждение представлено в обзорах [2, 13, 17, 18, 26, 37, 69–71, 100–104].
СОСТОЯНИЯ ХРОМАТИНА, ЭПИГЕНЕТИЧЕСКИЕ МЕТКИ И ГИСТОНОВЫЙ КОД
На протяжении большей части клеточного цикла ДНК в ядре эукариотических клеток связана с разнообразными белками, формируя сложную многоуровневую структуру – хроматин. Именно хроматин обеспечивает строгую иерархию уровней упаковки и неслучайную пространственную конфигурацию геномной ДНК, имеющей большую линейную протяженность (около 2 м в клетках человека), в пределах клеточного ядра, размеры которого измеряются микронами. При этом координируется своевременный доступ к генетической информации множества факторов, регулирующих каждую функцию генома: точную пространственно-временную экспрессию генов, правильный порядок репликации ДНК и верную передачу эпигенетических меток через клеточные деления, выявление и репарацию повреждений и ошибок в ДНК. В ходе развития организма и дифференцировки клеток различные районы хромосом подвергаются эпигенетической репрессии, механизмы которой имеют много общего с репрессией при ЭП: внесение модификаций в гистоны, связывание белков-репрессоров [105–109].
Уже в первых цитологических исследованиях хромосом были выделены два типа хроматина – эухроматин и ГХ. В целом эти понятия остаются актуальными и на сегодняшний день. К эухроматину относится часть генома, насыщенная белоккодирующими генами и регуляторными элементами. Эухроматин имеет низкую степень компактизации и характерный набор гистоновых модификаций. В состав ГХ входит значительная доля геномной ДНК (у многих эукариотических организмов это около трети общей протяженности генома [110]), однако эти области бедны генами, обогащены средне- и высокоповторяющимися последовательностями, сохраняют компактное состояние в течение практически всего клеточного цикла. Кроме того, эти области реплицируются в поздней S-фазе, отличаются от эухроматина набором модификаций гистонов и негистоновых белков, а также обладают выраженной способностью к пространственным контактам между собой и низким уровнем мейотической рекомбинации [17, 111, 112]. Изначально к ГХ относили лишь прицентромерные районы хромосом, которые содержат протяженные участки с регулярным расположением нуклеосом, а также эпигенетически наследуемые модификации гистонов и специфические наборы негистоновых белков-репрессоров [113–116]. С развитием представлений о молекулярной организации хроматина под ГХ стали подразумевать все районы хромосом, находящиеся в состоянии эпигенетической репрессии, поскольку многие районы так называемого интеркалярного ГХ имеют практически такие же свойства, как и прицентромерный ГХ [35, 117, 118]. Эпигенетическое наследование состояния ГХ тесно связано с репликацией ДНК в S-фазе клеточного цикла. Ферменты, вносящие модификации гистонов, характерные для “молчащих” районов, обнаруживаются только в вилках репликации, работающих в поздней S-фазе [35, 119]. Воспроизведение модификаций гистонов определяет дальнейшую сборку всех соответствующих компонентов хроматина после репликации (подробнее далее по тексту). При ЭП ген попадает в другой хромосомный контекст и, как следствие, происходит изменение временного паттерна репликации, что приводит к перепрограммированию его эпигенетического состояния [119–121]. Домены прицентромерного ГХ формируются уже на ранних этапах эмбриогенеза [122, 123]. Считается, что их сборка начинается на определенных цис-элементах, сайленсерах, от которых линейно распространяются модификации гистонов, а следом негистоновые белки ГХ до тех пор, пока не дойдут до “барьера” (подробнее см. раздел “Регуляторные элементы генома”) или до конкурирующего состояния хроматина [71, 124, 125]. Таким образом, ГХ состояние может захватить любой район, если он окажется физически близко к ГХ домену, что часто и происходит при ЭП [18] (последовательные этапы установления эпигенетического состояния хроматина подробно рассмотрены далее).
Первый уровень упаковки хроматина – нуклеосомная укладка. Нуклеосомы представляют собой регулярно расположенные белковые глобулы, вокруг которых ДНК делает 1¾ оборота [126–128]. В состав одной глобулы входят гистоны H2A, H2B, H3 и H4. Молекулы H2A, H2B, H3 и H4 формируют кóровый октамер, имеющий клиновидную форму, узкую часть которого образует тетрамер (H3–H4)2, а широкая часть состоит из двух димеров H2A–H2B [129, 130]. C-концевые глобулярные участки гистонов заключены в кóровой части октамера, а их подвижные N-хвосты свободно расходятся в стороны [131, 132]. Одна молекула линкерного гистона Н1 связывается с внешней стороной нуклеосомы в районе тетрамера (H3–H4)2, фиксируя на ней нить ДНК [133, 134]. Длина фрагмента ДНК, приходящегося на одну нуклеосому, варьирует и составляет до 200 п.н., из которых непосредственно с гистоновым октамером связаны 145–147 п.н. [132, 135, 136]. В клетках одного организма гистоны каждого типа (кроме H4) представлены набором вариантов. Известно до четырех вариантов гистона H3, восьми – H2A, 11 – H1 и не менее трех вариантов H2B [134, 137, 138]. Молекулы гистонов могут содержать различные ковалентные модификации определенных аминокислотных остатков их свободных N-хвостов и, в меньшей степени, глобулярной С-концевой части. Эти модификации вносятся в молекулы гистонов уже по завершении их синтеза, т.е. это посттрансляционные модификации, которые могут изменяться в течение клеточного цикла.
Многочисленные исследования показали, что конкретные комбинации гистоновых модификаций однозначно соответствуют определенному статусу хроматиновых доменов и даже отдельных частей генов [121, 139, 140] (рис. 2). Это явление получило название гистонового кода. Согласно современным представлениям, гистоновый код реализуется двумя путями. Во-первых, за счет изменения заряда и/или конформации молекулы разные варианты гистонов по-разному взаимодействуют с другими белками хроматина и с ДНК, изменяя архитектуру нуклеосом и доменов хроматина в целом [134, 141, 142]. Во-вторых, N-хвосты гистонов, в зависимости от комбинации их ковалентных модификаций служат сайтами для посадки разных семейств негистоновых структурных белков хроматина и ремоделирующих факторов [140, 143, 144]. Таким образом, реализуется эпигенетический метаболизм генома – установление, наследование и своевременное переключение состояний хроматина, что позволяет точно координировать во времени и пространстве молекулярные машины клеточных процессов [145–149] (рис. 2).
Рис. 2.
Условная схема организации репрессированного состояния хроматина – гетерохроматина, и активного состояния хроматина – эухроматина (а), схема нуклеосомы и относительного расположения ковалентных модификаций в N-хвостах гистонов (б). В рамках (а) перечислены известные модификации гистонов, свойственные каждому из этих состояний хроматина. bio – биотинилирование; ac – ацетилирование; me/me2/me3 – моно, ди- и триметилирование аминокислот в составе указанного гистона. В рамках (б) приведены известные гены-модификаторы ЭП дрожжей, дрозофилы и человека, связанные с моно- ди- и триметилированием N-хвостов гистоновых белков. Красными овалами условно обозначен линкерный гистон Н1 (на панелях а и б).
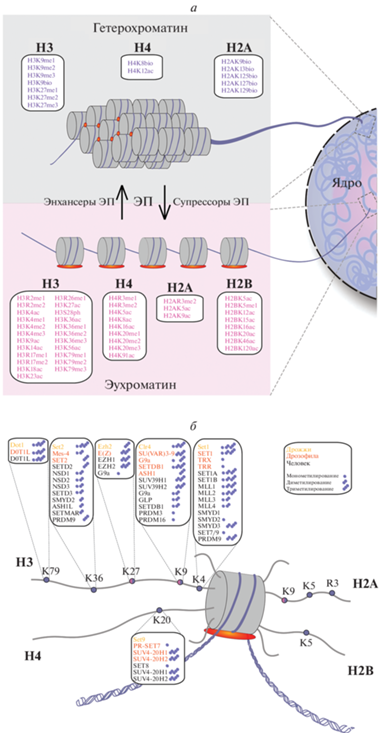
Гистоновый код – начальный уровень создания эпигенетического контекста, служит динамической платформой, интегрирующей различные эпигенетические сигнальные пути, он определяет уровни упаковки хроматина более высокого порядка [150–152]. Гиперацетилирование гистонов H3 и H4 и метилирование лизина в положении 4 гистона Н3 коррелирует с установлением активного статуса хроматина у большинства видов, от дрожжей до дрозофилы и человека [153–155]. Ацетильные группы уменьшают общий заряд остатков лизина; считается, что это ослабляет взаимодействие молекул гистонов с фосфатными группами в ДНК и облегчает доступ машины транскрипции [156]. Помимо изменения биофизических свойств хроматина, ацетилированный лизин служит мишенью для бромодомен-содержащих белков, в число которых входят различные активаторы транскрипции и хроматин-ремоделирующие факторы [157, 158]. При этом связывание конкретных белков с N-хвостами гистонов зависит от комбинации их модификаций; и таким образом реализуется комбинаторная пластичность гистонового кода [143, 144, 159]. Хотя гиперацетилирование гистонов обычно связано с активным состоянием хроматина, существуют и исключения – например, ацетилирование H4K12 (H4K12ac) характерно именно для районов ГХ [160].
Различные варианты метилирования N-концов гистонов играют ключевую роль в разделении активных и репрессированных доменов хромосом (рис. 2а). Обобщая, можно сказать, что к активирующим модификациям гистонов относится метилирование H3K4, H3K36 и H3K79, в то время как для репрессированных районов хроматина характерно метилирование H3K9, H3K27 и H4K20 [161–164] (рис. 2а). По существующим представлениям, формирование ГХ и, в результате, репрессию генов при ЭП обеспечивает определенный набор модификаций гистонов, что влечет связывание соответствующих комбинаций негистоновых белков-репрессоров. Поэтому для понимания принципов формирования репрессированных доменов хроматина, в частности при ЭП, необходима последовательная идентификация клеточных ферментов, осуществляющих модификации гистонов и выяснение молекулярных механизмов, лежащих в основе функционирования гистонового кода.
ГЕНЫ-МОДИФИКАТОРЫ ЭФФЕКТА ПОЛОЖЕНИЯ КОДИРУЮТ СТРУКТУРНЫЕ И РЕГУЛЯТОРНЫЕ БЕЛКИ ХРОМАТИНА
ЭП гена представляет собой изменение его эпигенетического состояния за счет изменений его нормального хромосомного окружения, не связанного с мутациями или делециями самого гена. Именно поэтому ЭП исторически стал стартовой площадкой, с которой начали изучать молекулярные факторы разных эпигенетических состояний хроматина – активного и репрессированного. Первые исследования механизмов формирования хроматина были основаны на тех наблюдениях, согласно которым фенотипы ЭП изменяются супрессорными (ослабляющими) и энхансерными (усиливающими) мутациями различных генов – модификаторов ЭП [110, 165, 166]. Продукты генов-супрессоров ЭП являются структурными компонентами ГХ, ферментами, которые модифицируют гистоновые белки, или же компонентами ядерной оболочки (ламины) [125, 167–171]. Гены-энхансеры ЭП, наоборот, кодируют продукты, участвующие в установлении активного статуса эухроматина, который противостоит гетерохроматинизации [71, 89, 125, 172] (рис. 2б, 3а).
Рис. 3.
Схемы скрининговых экспериментов по поиску генов-модификаторов ЭП у дрозофилы (а), мыши (б) и в культивируемых клетках человека (в). GFPdim – клетки с ЭП репортерного гена GFP; GFPbright – клетки с супрессией ЭП (усиление экспрессии) репортерного гена GFP. Наиболее эффективный способ выявления генов-модификаторов в указанных подходах – инсерционный мутагенез при помощи специальных трансгенных конструкций, позволяющих легко идентифицировать поврежденные гены. Модифицировано по [70] с разрешения издателя.
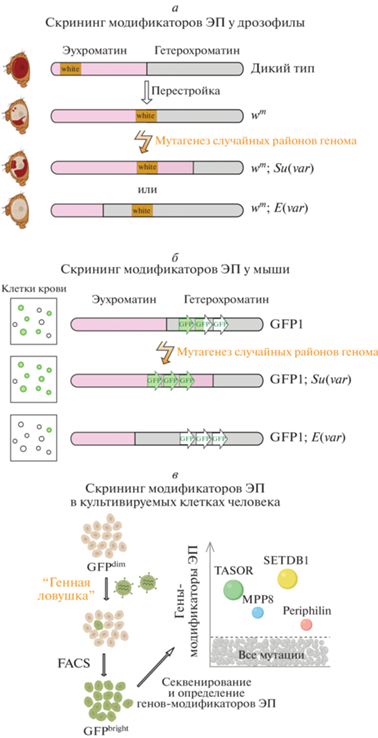
Значительную часть генов-модификаторов ЭП выявили еще в ранних скрининговых исследованиях, выполненных на дрозофиле [32, 173, 174] (рис. 3а). На сегодняшний день известно около 150 генов-супрессоров (Su(var)), и более 200 генов-энхансеров (E(var)) ЭП, однако молекулярная функция установлена лишь примерно у каждого десятого из них [70, 71, 166] (Табл. S1 , см. Приложение на сайте http://www.molecbio.ru/downloads/2022/3/supp_Boldyreva_rus). Одним из первых был охарактеризован ген Su(var)2-5 дрозофилы, открытие которого стало фундаментом для формирования представлений о процессах генетической репрессии. Этот ген кодирует обнаруженный в ГХ белок HP1a, содержащий N-концевой хромодомен и C‑концевой хромотеневой домен, обеспечивающие его способность взаимодействовать с ди- и триметилированным H3K9 (H3K9me2/3) и с другими белками хроматина [175–177]. Этот белок обнаружен в районах хромосом, подверженных гетерохроматинизации при ЭП [44, 178], как и белок SU(VAR)3-9, продукт одноименного гена-супрессора ЭП [166], который содержит N-концевой хромодомен и C-концевой домен SET [179]. Белок SU(VAR)3-9 высоко консервативен, он обладает активностью гистон-метилтрансферазы, которая специфически метилирует H3K9 [180, 181]. Молекулярный анализ функций белков HP1a и SU(VAR)3-9 предполагает механизм “чтения–записи”, который лежит в основе распространения гетерохроматинизации при ЭП. SU(VAR)3-9, устанавливает модификацию H3K9me2/3, образующую сайт, специфически распознаваемый хромодоменом HP1a, а N-концевой участок SU(VAR)3-9 связывает хромотеневой домен HP1a, стабилизируя тем самым взаимодействие между HP1a и H3K9me2/3 и обеспечивая дальнейшее распространение H3K9me2/3. Этот процесс считается основным механизмом репрессии в прицентромерном ГХ, он эволюционно консервативен, но при этом вовлекает еще множество других факторов, уже менее консервативных [176, 182]. Кроме того, роль HP1a и SU(VAR) 3–9 не существенна для других типов ЭП (теломерного) и для репрессии вставок P-элементов в ГХ хромосоме 4 дрозофилы, в которой существенно большее значение имеют другие гистон-метилтрансферазы [183–186]. Гомологи значительной части модификаторов ЭП, обнаруженных у дрозофилы, найдены и у других эукариот – S. cerevisiae [187], Sh. pombe [188], растений [189] и млекопитающих [75, 76] (рис. 2б), хотя иногда функции этих белков у различных организмов в процессе эволюции изменяются [69–71]. Современные исследования ЭП сосредоточены на определении и анализе довольно сложных паттернов активности генов-модификаторов ЭП и последовательности событий, которые поддерживают в равновесии динамический баланс между ГХ и эухроматином в эукариотическом ядре. Помимо генов-модификаторов ЭП, влияние на степень инактивации генов, подверженных ЭП, оказывают и другие факторы – общее количество ГХ в клетке, скорость развития и температура (у дрозофилы она важна на ранних стадиях развития, когда происходит установление эпигенетически наследуемого репрессированного состояния ГХ). Так, отсутствие хромосомы Y (почти полностью гетерохроматиновой) у самцов Х0 дрозофилы приводит к значительному усилению ЭП генов [190]. Этот факт дополнительно свидетельствует о значении баланса между компонентами активного и репрессированного хроматина в клетке и факторов, обеспечивающих их разграничение во времени и пространстве.
С целью выявления генов мыши, способных быть модификаторами ЭП, проведен крупномасштабный генетический скрининг на основе тандемной вставки GFP-экспрессирующего трансгена, показывающей фенотип мозаичного ЭП [74, 75] (рис. 3б). Выявлены и охарактеризованы примерно 40 генов-модификаторов ЭП мыши, значительная часть которых оказалась ортологичной модификаторам ЭП у дрозофилы [76]. Выявленный с помощью этого скрининга белковый комплекс модификаторов ЭП мыши назван MommeD [74]. В его состав входят гистон-метилтрансферазы SUVAR39H1 и SETDB1 [191–193], гистон-деацетилаза HDAC1 [194], хроматин-ремоделирующие факторы SMARCA5, SMARCC1, PBRM1 и BAZ1B [195], регулятор транскрипции TRIM28 [196], фактор транскрипции KLF1 [197], ДНК-метилтрансферазы DNMT1 и DNMT3B [198, 199]. Выявлено также несколько новых генов-модификаторов ЭП мыши: Smchd1, Rlf и D14Abb1e (последний ортологичен гену TASOR человека (см. ниже)) [200]. Только три белка из этого списка не имеют ортологов у дрозофилы, это ДНК-метилтрансферазы DNMT1, DNMT3B и SMCHD1, которые участвуют в поддержании инактивации хромосомы Х самок, опосредованной метилированием CpG-островков [201].
Подобный скрининг по выявлению генов-модификаторов ЭП человека проведен в 2015 году с использованием культивируемых клеток, содержащих условно гаплоидный набор хромосом, и метода генной ловушки (gene trap) [202, 203]. С целью выявления новых доминантных модификаторов ЭП в клетках человека получен набор встроек GFP-трансгенов, демонстрирующих ЭП [200] (рис. 3б). В результате этого скрининга выявлены четыре гена-модификатора ЭП, один из которых, SETDB1, кодирует гистон-метилтрансферазу и является гомологом такого же гена мыши, функция которого – установление репрессирующей гистоновой метки H3K9me3 [204]. Функция модификаторов ЭП у остальных трех генов выявлена впервые. Подтверждено, что белковые продукты этих генов человека входят в один репрессорный комплекс, названный HUSH (HUman Silencing Hub). В состав этого комплекса входят белки TASOR (Transgene Activation SuppressOR), хромодомен-содержащий фосфопротеин MPP8 и Periphilin. Фосфопротеин MPP8 связывается с гистон-метилтрансферазой SETDB1 [205], а его хромодомен распознает и связывает гистоновую метку H3K9me3 [206]. Это позволяет предположить, что комплекс HUSH человека может осуществлять гетерохроматинизацию с использованием механизма чтения–записи, работающего у дрозофилы, одновременно обеспечивая функциональную связь этого процесса с механизмами репрессии, опосредованной метилированием ДНК [204].
Таким образом, говоря о формировании репрессированных доменов хроматина, следует рассуждать категориями взаимосвязанных молекулярных механизмов установления, распознавания и поддержания эпигенетического состояния, координации этих процессов с другими жизненно важными процессами клетки. К настоящему времени известно более 10 основных эволюционно стабильных сигнальных путей, через которые осуществляются оперативные и долговременные ответы эукариотической клетки на внешние и внутренние сигналы (Pathway Interaction Database http://pid.nci.nih.gov, http://www.ndexbio.org/). Их анализ показал, что многие белки-модификаторы ЭП вовлечены в эти сигнальные пути, а нарушения в соответствующих генах связаны с различными заболеваниями [70] (табл. S1 Приложения).
В целом, результаты изучения молекулярной функции генов-модификаторов ЭП и механизмов репрессии предполагают, что в клетках эукариот существует ряд альтернативных механизмов, способных устанавливать эпигенетически репрессированное состояние хроматина. В различных типах ГХ репрессия обусловлена разными наборами белков, однако некоторые из них могут быть взаимозаменяемыми. Сам феномен ЭП до сих пор остается уникальной моделью, позволяющей изучать как отдельные функциональные звенья, так и процессы установления эпигенетического статуса хроматина.
РЕГУЛЯТОРНЫЕ ЭЛЕМЕНТЫ ГЕНОМА
Считается, что последовательности ДНК, регулирующие работу генома, занимают значимую часть генома эукариот [207–210]. К основным регуляторным элементам относятся: промоторы, инсуляторы, энхансеры и сайленсеры. Эти элементы способны существенно влиять на уровень экспрессии гена: нуклеотидный состав и архитектура промоторной области определяют уровень, время и тканевую специфичность активности гена. Энхансерные элементы генома способны усиливать экспрессию гена (при сохранении той же промоторной области) и обладают тканеспецифичностью. Инсуляторы, расположенные между энхансером и промотором, блокируют взаимодействие этих элементов. Сайленсерами называют те участки ДНК, с которых начинается сборка и распространение репрессированного состояния хроматина. Одно из главных свойств регуляторных элементов – способность изолированно осуществлять свою функцию. Эти элементы узнаются ДНК-связывающими и хроматин-модифицирующими белками, они участвуют также в поддержании доменной организации генома [208, 209, 211]. С разработкой методики фиксации пространственной конформации хромосом (Hi-C) [212] стало ясно, что даже линейно удаленные цис-регуляторные элементы генома пространственно сближены с контролируемыми ими мишенями [213–216]. Так, взаимодействие линейно удаленных друг от друга промоторов и энхансеров обусловлено, по всей видимости, формированием крупномасштабных петель ДНК [217, 218].
Инсуляторы были открыты в геноме дрозофилы как последовательности ДНК, способные устанавливать границы между разными состояниями хроматина [219–222]. В настоящий момент инсуляторные мотивы и специфически связывающиеся с ними инсуляторные белки найдены у разных организмов [223, 224], но наибольшее их разнообразие обнаружено у дрозофилы [225]. Инсуляторные белки дрозофилы различаются по своим физическим и биохимическим свойствам. Белки Su(Hw), CTCF, BEAF-32, Ibf1, Ibf2, Pita, ZIPIC, Zw5/Dwg, GAF, Opbp и Clamp связываются со специфическими последовательностями ДНК [226, 227]. Так, удаленно расположенные инсуляторы gypsy образуют кластеры, локализованные на периферии ядра, а связывающийся с ними Su(Hw)-зависимый комплекс осуществляет взаимодействие с ядерной ламиной [211, 228]. Часть из этих белков (CTCF, Zw5/Dwg, Pita, ZIPIC, Opbp) способна образовывать гомодимеры [226, 229–231]. Белок CP190 и продукты гена mod(mdg4) не способны связываться со специфическими ДНК, но могут гомодимеризоваться при помощи доменов BTB/POZ [232, 233]. Кроме того, многочисленные изоформы MOD(MDG4) образуют гетеромультимерные комплексы, в том числе, с другими BTB/POZ-белками [225, 234]. Инсуляторные белки входят в состав мультисубъединичных комплексов [210, 225]. Полногеномный анализ сайтов связывания отдельных инсуляторных белков выявил их комбинации, характерные для конкретных локусов генома [211, 235, 236]. Описанные инсуляторы и инсуляторные белки изучены только у дрозофилы, в клетках же млекопитающих пока обнаружен только СTCF и его паралоги – BORIS (Brother Of Regulator of Imprinted Sites)/CTCFL и СTCF2 [237, 238]. Молекулы белка СTCF, встречающегося у многих эукариот, могут взаимодействовать друг с другом, формируя кластеры и способствуя образованию закрытых хроматиновых доменов [239, 240]. Показано, что направление дальних взаимодействий внутри топологически ассоциированных доменов (ТАДов) человека и млекопитающих определяется ориентацией мотивов ДНК, связывающих белок CTCF [217, 241].
Формирование и динамика архитектуры ядра в большой степени зависит от процесса экструзии петель хроматина [242, 243]. Один из основных факторов, участвующих в этом процессе в клетках млекопитающих – белковый комплекс когезин, способный физически выталкивать петли ДНК вплоть до сайтов локализации СTCF [244–246]. Показано, что когезин необходим для правильного формирования ТАДов и поддержания границ между активными и репрессированными компартментами генома [247]. Однако и в отсутствие когезина в клетках млекопитающих сохраняются отдельные крупные ТАДы [247]. В то же время, у дрозофилы до сих пор не найдено механизма экструзии петель хроматина, подобного когезинзависимому механизму у млекопитающих [248]. Существуют данные, свидетельствующие о том, что инсуляторы не только участвуют в формировании границ крупных консервативных ТАДов (см. раздел “Геномная компартментализация”), но и вовлечены в организацию динамических и тканеспецифичных суб-ТАДов [249, 250]. Сравнительно недавно показана также связь инсуляторов и инсуляторных белков с процессом репликации. Например, инсуляторный белок дрозофилы Su(Hw) взаимодействует in vivo с субъединицами комплекса ORC и необходим для его рекрутирования на Su(Hw)-связующие элементы [251, 252]. За последние годы установлено, что помимо канонических функций (архитектура ядра и регуляция транскрипции), инсуляторные комплексы выполняют дополнительные функции, они участвуют в паузировании РНК-полимеразы II, сплайсинге, экспорте мРНК и репарации ДНК [209, 210, 253, 254].
Сайленсерами названы отдельные элементы генома, способные, в отличие от энхансеров, подавлять транскрипцию. Впервые такие элементы были найдены у дрожжей [255, 256]. Функция сайленсеров, по-видимому, реализуется с участием гораздо более сложных, чем у энхансеров, многоступенчатых механизмов, детали которых во многом еще остаются неизученными [257, 258]. Классический и наиболее исследованный пример сайленсеров – PRE (Polycomb Response Elements), инициирующие сборку репрессирующих белковых комплексов Polycomb, обеспечивая эпигенетическое наследование репрессированного состояния [259]. Ключевой ролью комплексов Polycomb считается поддержание наследуемого эпигенетического статуса районов генома, экспрессия генов в которых определяет специализацию клеток при дифференцировке, либо их плюрипотентное состояние [260–262]. У млекопитающих обнаружено разрушение доменов, образованных комплексом Polycomb, при когезинзависимой экструзии петель, однако этот процесс не зависел от СTCF. При этом контакты, обусловленные взаимодействием суперэнхансеров, устойчивы к когезинзависимой экструзии хроматина [247]. Известны мотивы ДНК, которые способны опосредованно привлекать корепрессоры, не приводя при этом к долговременной репрессии и эпигенетической памяти, как это происходит в случае PRE. Например, транскрипционный фактор KRAB, специфично связывающийся посредством своих доменов цинковых пальцев с определенными мотивами ДНК в промоторных областях генов, способен привлекать корепрессор KAP1 и индуцировать формирование неактивного хроматина при помощи HP1/SU(VAR)3-9-зависимого механизма [263]. Известен особый класс лигандзависимых корепрессоров ядерных рецепторов. Эти корепрессоры способны выступать в роли как репрессоров, так и активаторов, в зависимости от конформации ядерного рецептора и локального контекста хроматина [264, 265]. Ключевую роль в этих процессах играют гистон-деацетилазы, которые рекрутируются корепрессорами [265, 266]. Таким образом, через соответствующие транскрипционные факторы корепрессоры специфично привлекаются к местам активной транскрипции, где, по-видимому, осуществляют тонкую регуляцию уровня транскрипции, обеспечивая механизмы обратной либо сигналзависимой регуляции [267].
В недавних масштабных исследованиях в геномах дрозофилы, нематоды, мыши и человека выявлены десятки тысяч регуляторных элементов [208, 216, 268–271]. В частности, функциональный скрининг, выполненный на дрозофиле, показал, что в зависимости от локального окружения хроматина, ткани и стадии развития значительная часть регуляторных элементов может выступать и как сайленсер, и как энхансер. Делеция таких элементов зачастую вызывает нарушение функции соответствующих генов-мишеней [216]. С нарушением цис-регуляторных элементов связан широкий ряд заболеваний, при которых, в частности, наблюдается нарушение функции генов вследствие ЭП [272, 273].
Все сказанное хорошо укладывается в современную парадигму, согласно которой метаболизм генома представляет собой сложную иерархически организованную саморегулирующуюся машину, все уровни которой уравновешены и взаимосвязаны [274, 275]. Регуляторные элементы генома, по-видимому, играют важнейшую роль в этих процессах, однако для детального понимания функций регуляторных элементов в метаболизме генома необходим еще существенный пласт исследований.
ГЕНОМНАЯ КОМПАРТМЕНТАЛИЗАЦИЯ
Начало эры высокопроизводительного секвенирования ДНК и биоинформатических методов анализа открыло возможности полногеномного картирования с высоким разрешением различных компонентов хроматина (в частности, описанных выше) и их устойчивых комбинаций, ассоциированных с конкретными участками генома [249, 276–278]. В результате выполнения проектов ENCODE и modENCODE получены данные о профилях связывания различных белков и распределениях посттрансляционных модификаций гистонов в различных типах клеток дрозофилы, нематоды, мыши и человека (https://www.encodeproject.org/) [268, 269]. Подтвердились предположения о том, что профили связывания многих белков хроматина с геномной ДНК и ковалентные модификации самих молекул ДНК являются тканеспецифическими и в значительной мере определяют активность генов при развитии организма, формировании тканей и дифференцировке клеток. Кроме того, эти данные позволили выделить в изученных геномах функциональные хроматиновые домены – как стабильно эпигенетически наследующиеся и даже эволюционно консервативные, так и претерпевающие оперативные модификации при дифференцировке, нарушении развития и заболеваниях [270, 279–281].
Доменная организация хроматина – один из уровней регуляции экспрессии генов, важный механизм которого состоит в ограничении перекрестного влияния регуляторных элементов генома, находящихся в контрастных состояниях хроматина [282, 283] (см. раздел “Регуляторные элементы генома”). Неслучайное расположение материала хромосом внутри интерфазного клеточного ядра обнаружили еще в 1970-е годы. Методами радиоактивного мечения, а также FISH-гибридизации в 1980-е годы показано, что каждая хромосома занимает определенный пространственный компартмент в объеме ядра; эти компартменты названы хромосомными территориями [284–286]. Затем обнаружили, что обогащенные генами хромосомы, либо отдельные их участки, как правило, располагаются ближе к центру ядра, а обедненные – ближе к его периферии, и эта особенность эволюционно консервативна [287–291]. В дальнейшем выявили связь между удаленностью того или иного участка хромосомы от центра ядра и его GC-составом, временем репликации и активностью генов в данном участке [292–296]. Поскольку набор этих свойств характерен для большинства участков хромосом, располагающихся на периферии ядра и взаимодействующих с ядерной ламиной, эту зону выделили как отдельный ядерный компартмент, а связанные с ним участки хромосом назвали доменами, ассоциированными с ламиной (ЛАДами) [297, 298]. В составе ЛАДов обнаруживаются прицентромерные районы ГХ и часть теломерных районов хромосом [299–301]; показано специфичное обогащение ЛАДов гистоновыми метками репрессированного хроматина – H3K9me2/3 и H3K27me3 [299, 302]. Выявлены также участки ДНК размером 4–6 т.п.н., которые достаточны для связывания хроматина с ядерной ламиной и способствуют репрессии генов. Показано, что с такими последовательностями связывается деацетилаза гистонов HDAC3 в комплексе с репрессором транскрипции cKrox и белком внутреннего слоя ядерной оболочки Lap2β, что, вероятно, инициирует установление и распространение репрессированного статуса в ЛАДах [303]. Другой белок внутреннего слоя ядерной оболочки, эмерин, связывает и каталитически активирует HDAC3 [304, 305]. Таким образом, в репрессивных механизмах в ЛАДах, по-видимому, участвует также гистоновый код. Существует целый ряд работ, подтверждающий подавление экспрессии генов, входящих в ЛАДы, в различных модельных системах. Так, показано подавление транскрипции в теломерном локусе дрожжей при его контактах с ядерной мембраной, а также отдаление от ядерной мембраны Ig-локуса B-лимфоцитов человека перед его активацией [295, 296]. С другой стороны, активация транскрипции неактивных генов, расположенных в ЛАДах, сопровождается деконденсацией хроматина и перемещением соответствующих локусов от периферии ядра в его центр [306–308]. При ЭП транскрипция гена в каждой конкретной клетке коррелирует с удалением гена от места локализации сателлитной ДНК в ГХ, расположенной на периферии ядра [309].
При том, что контакты отдельных участков хроматина с ядерной ламиной динамичны и взаимосвязаны с экспрессией генов, изучение процесса дифференцировки эмбриональных стволовых клеток мыши выявило конститутивные ЛАДы и интер-ЛАДы – районы генома, которые стабильно контактируют или не контактируют с ядерной ламиной, а также факультативные ткане- и стадиеспецифичные ЛАДы [306, 310]. Конститутивные ЛАДы оказались районами ГХ, наиболее обедненными генами; их характеризует также AT-богатый состав и насыщенность длинными диспергированными повторами (Long Interspersed Nuclear Element, LINE) и консервативность их протяженности и расположения в геномах мыши и человека [310]. Исходя из этого предполагается, что конститутивные ЛАДы выполняют роль некоего якорного каркаса для пространственной организации ДНК генома внутри эукариотического ядра [300].
Помимо ЛАДов, весомая доля районов ГХ найдена еще в одном ядерном компартменте, названном ядрышко-ассоциированными доменами (Nucleolus-Associated Domains, NAD) [311, 312]. Кроме локусов генов рРНК, которые, как долго считалось, образуют ядрышко, этот компартмент также содержит участки ГХ практически всех хромосом [313]. Обнаружено большое перекрытие между ЛАДами и NAD [311, 313]. NAD также обогащены репрессирующими метками гистонового кода, большинство генов в них неактивны. Предполагают, что в ЛАДах и NAD хроматин находится в одном и том же, репрессированном, состоянии, а распределение конкретных неактивных районов генома между этими компартментами происходит случайным образом. Так, показано, что некоторые участки хромосом, ассоциированные с ядрышком в материнской клетке, могут после митотического деления оказываться на периферии ядра в дочерних клетках [311, 313]. Ряд данных говорит о том, что локализация определенных участков генома в NAD тесно связана с возрастом и старением [314, 315].
Разработка методов фиксации конформации хромосомы – от 3С до Hi-C – открыла возможность сопоставить данные о составе хроматиновых доменов с их взаимным расположением в ядре на уровне всего генома [218, 316, 317]. Выявляемые этими методами контактные домены – ТАДы – часто содержат в своем составе топологические структуры более низкого порядка, называемые суб-ТАДами [318]. Так, построена карта топологических взаимодействий хроматина с высоким разрешением (до 4 т.п.н.) в эмбриональных стволовых клетках и клетках-предшественниках нейронов человека [319]. Установлено, что контакты чаще происходят между областями хроматина внутри ТАДов, чем между областями генома, входящими в разные ТАДы [320]. ТАДы устойчиво сохраняют свои границы в процессе развития организма и дифференцировки клеток [318]. Более того, принципы организации генома в контактные домены консервативны у разных видов – от дрозофилы до млекопитающих [321]. ТАДы колокализуются с временными доменами репликации [322], определяют уровень транскрипции генов, входящих в них, и служат барьером, разграничивающим активность генов, принадлежащих другим доменам [250]. Оказалось, что каждый ТАД содержит характерный преобладающий набор эпигенетических маркеров, соответствующий определенному состоянию хроматина [211]. С использованием метода, совместившего Hi-C и DamID, недавно показали, что ЛАДы соответствуют неактивной фракции ТАДов (доменам типа В), а снижение количества ламина приводит к перемещению части ЛАДов в активную фракцию ТАДов (домены типа А) [323]. Предполагают, что миграция районов генома в ЛАДы из активного домена типа А способствует ограничению доступа транскрипционных факторов к промоторам генов, расположенных в ЛАДах [324]. Нарушения ядерной компартментализации и границ ТАДов выявляют при различных заболеваниях человека и разных видах рака [250, 325–327]. Все эти данные указывают на важность изучения не только нарушений функции отдельных генов, но и отслеживания эпигенетических эффектов, в частности, пространственной организации генома внутри ядра.
Недавно обнаружили существенную роль биофизических свойств хроматина в его пространственной организации внутри ядра [328]. Выявлены характеристики ГХ доменов, сходные со свойствами капель жидкости, которые обусловлены высокой склонностью белка HP1 к олигомеризации и физическими свойствами таких конгломератов [329–331]. На данный момент эти свойства объясняют динамическую устойчивость районов ГХ и присущий им эффект “спрединга”. Очищенные белки HP1a дрозофилы и HP1α человека in vitro способны к разделению жидких фаз (Liquid-Liquid Phase Separation, LLPS) и самопроизвольному формированию безмембранных обособленных капель [330]. В ядре, по-видимому, за счет фазового разделения транскрипционные факторы задерживаются на поверхности таких ГХ капель, в то время как связанная с нуклеосомами ДНК проникает внутрь капель беспрепятственно [330, 332, 333]. Предполагают, что ГХ капли могут сливаться, даже если они сформировались на отдаленных участках генома [334, 335]. Домены активного хроматина также формируют компартменты с жидкостными свойствами, которые заключают в себе машину транскрипции и склонны к слиянию, сближая, например, промоторы с энхансерами [336, 337]. В этой связи стоит отметить, что у млекопитающих обнаружены суперэнхансеры – регуляторные элементы очень большой протяженности (с медианным размером в несколько десятков т.п.н.), которые состоят из наборов отдельных энхансерных элементов, способных взаимодействовать друг с другом в пространстве и интегрально усиливать транскрипционную активность множественных генов-мишеней [338]. Количество суперэнхансеров в клетке на 1–2 порядка меньше обычных энхансеров, при этом их мишенями часто становятся гены, определяющие идентичность клетки [338, 339]. Функционирование суперэнхансеров определяется способностью транскрипционных мастер-факторов (master transcription factors) привлекать хроматин-ремоделирующие факторы, коактиваторы, в том числе способные ковалентно модифицировать гистоны, а также другие факторы транскрипции [338, 340]. Высокая локальная концентрация по крайней мере некоторых из перечисленных факторов обеспечивает разделение жидких фаз и пространственную компартментализацию суперэнхансеров и сближенных с ними промоторов генов-мишеней [338–340]. Комбинация методик фазового разделения хроматина и Hi-C открыла возможность сравнительного анализа всех типов пространственных взаимодействий внутри ядра. Оказалось, что как домены активного и репрессированного хроматина, так и ТАДы, ранжируются по стабильности и длительности существования [341].
Таким образом, процессы, которые регулируются пространственной структурой генома, взаимосвязаны с процессами установления пространственной структуры хроматина в ядре, по-видимому, в обоих направлениях. Развитие современных методов исследований сформировало базу для разработки новых методов масштабного изучения и сравнительного анализа как роли локального состояния хроматина, так и отдельных функциональных элементов генома в регуляции экспрессии генов. Эта база может широко использоваться для изучения эпигенетических эффектов, в частности генов ЭП, у различных организмов.
КЛИНИЧЕСКИЕ АСПЕКТЫ ЭФФЕКТА ПОЛОЖЕНИЯ ГЕНА
При заболеваниях человека, связанных с ЭП генов, наблюдается перемещение (путем перестроек) регуляторных элементов либо их удаление (в результате делеции), а также перемещение гена посредством перестроек и делеций в контрастное состояние хроматина. Кроме того, выявлены различные отклонения в формировании ГХ: нарушение экспрессии генов, обусловленное их гетерохроматинизацией (при перестройках либо в результате локальной экспансии (увеличения числа) повторенных последовательностей); сверхэкспрессия генов, связанная с уменьшением количества ГХ [342, 343].
В число заболеваний человека, связанных с ЭП генов, входят некоторые виды рака, патологии развития, чаще всего вызванные перестройками хромосом (транслокациями, инверсиями и делециями), в том числе соматических, и широкий ряд нейродегенеративных заболеваний, обусловленных экспансией коротких повторов в промоторных областях определенных генов. Злокачественные гемопатии, как правило, возникают вследствие приобретенных соматических перестроек, чаще всего, транслокаций. При этих заболеваниях вовлеченный в перестройку ген зачастую подпадает под влияние энхансерной либо же промоторной области другого гена, что приводит к изменению уровня и паттерна экспрессии первого гена [344]. В качестве типичного примера можно привести неходжкинскую лимфому Беркитта, при которой ген c-MYC (Хр. 8) в результате транслокации попадает под влияние энхансера гена тяжелой цепи иммуноглобулина в хромосоме 14, что приводит к сверхэкспрессии c-MYC в B-лимфоцитах [345]. Подобные случаи ЭП, связанные с влиянием чужеродных регуляторных элементов, выявлены в случае генов BCL2 при фолликулярной лимфоме и BCL6 при диффузной В-крупноклеточной лимфоме [346, 347]. Еще один пример заболеваний, обусловленных перемещением гена в другое состояние хроматина – медуллобластомы 3 и 4 групп, наиболее частая причина детской смертности от рака. При медуллобластомах выявлена сверхэкспрессия генов GFI1 и GFI1B, которые переносятся соматическими перестройками из репрессированных районов генома в области, обогащенные ацетилированными H3K9 и H3K27, ассоциированные с суперэнхансерами [348].
Ряд заболеваний, входящих в класс заболеваний со сбалансированными хромосомными аберрациями (BCA), связан с перестройками, точки разрыва которых расположены на расстоянии до 400 т.п.н. как выше, так и ниже гена, подверженного ЭП в результате этих перестроек [349, 350]. В отдельных случаях ЭП может, по-видимому, распространяться на 1 млн.п.н. и даже более от точки разрыва перестройки (табл. S2, см. Приложение на сайте http://www.molecbio.ru/downloads/ 2022/3/supp_Boldyreva_rus). Что интересно, на расстоянии от 400 до 800 т.п.н. от точек разрыва хромосомных перестроек не находили. Это может объясняться присутствием критических регуляторных элементов в этих областях (вероятно, суперэнхансеров), поскольку их удаление приводит к летальным нарушениям. Вторым объяснением может быть роль пространственной структуры хроматина, а не цис-элементов, нарушение которой может существенно влиять на ядерную компартментализацию. На текущий момент известно около 40 синдромов и заболеваний человека, которые развиваются при нарушении экспрессии генов в результате ЭП, для большинства из этих генов найдены мутации со сходными фенотипическими проявлениями [343].
Еще одна распространенная причина заболеваний, связанных с ЭП генов, – изменение числа тандемных повторов в промоторных или 5ʹ-нетранслируемых районах генов, что приводит к нарушению их экспрессии [98, 351]. В настоящее время известно около 50 таких заболеваний человека (чаще всего дегенеративных), которые характеризуются тяжелым течением, тогда как эффективных методов их лечения и предотвращения пока не существует. Повторяющиеся последовательности составляют по различным оценкам от 50 до 70% генома человека [352, 353]. К тандемным повторам относят такие участки генома, в которых две и более копии повторенной последовательности ДНК ориентированы голова-к-хвосту. По длине повторенного мотива (единицы повтора) эти последовательности делят на микросателлиты (повтор 1–6 п.н.), минисателлиты (7–100 п.н.) и макросателлиты (100 п.н. и более) [351, 354, 355]. Самые короткие тандемные повторы (тринуклеотиды) составляют 1–3% протяженности генома человека [352, 355]. Микросателлитами часто обогащены промоторные районы генов [356, 357], включая гены, существенные для нейрогенеза, дифференцировки нейронов, развития нервной системы и адгезии клеток [358]. Наиболее распространенные в геноме человека микросателлиты (более трети всех микросателлитов, 36.4%) представлены A/T-богатыми мотивами, а в промоторных областях генов человека часто локализуется CCG/CGG (74% случаев этого сателлита в геноме), хотя его представленность в геноме в целом очень невелика (менее 1% от всех обнаруженных микросателлитов) [357]. Похожий мотив CCCG/CGGG обнаруживается преимущественно (62%) также в промоторных областях генов человека. Микросаттелиты CCG/CGG высоко консервативны у млекопитающих, тогда как другие G/C-богатые мотивы не относятся к консервативным [359]. Экспансия именно CCG/CGG часто приводит к развитию заболеваний, связанных с гетерохроматинизацией генов в результате ЭП (табл. S3). По числу копий все тандемные повторы в одних и тех же геномных локусах сильно варьируют у разных лиц и могут влиять на уровень активности генов [94, 356–358, 360–364]. Механизм репрессии генов, подверженных гетерохроматинизации в результате экспансии сателлитов, по-видимому, аналогичен механизмам инактивации тандемных вставок трансгенов, которая осуществляется эпигенетически и приводит к гетерохроматинизации как самих повторенных последовательностей, так и прилежащих участков генома (генов) [365, 366]. Ключевую роль в этом процессе у млекопитающих играет метилирование ДНК GC-богатых повторов [94, 367, 368]. Считается, что число копий тандемных повторов может легко уменьшаться или увеличиваться за счет ошибок в ходе репликации или репарации ДНК, и неравной рекомбинации [98, 342, 369, 370]. Важно отметить, что частота других мутаций, например, однонуклеотидных замен (SNP) намного ниже, чем частота изменения числа копий коротких повторов в геноме [371–375]. Вклад различных тандемных повторов в развитие заболеваний, связанных с расстройствами аутистического спектра (ASD, Autistic Spectrum Disorders) оценен в недавних работах [376, 377]. Анализ 17231 геномов индивидов с вновь диагностированными ASD, членов семей с больными ASD, а также здоровых людей (популяционный контроль) с использованием алгоритма ExpansionHunter Denovo, детектирующего повторы с длиной мотива от 2 до 20 п.н., выявил 37865 тандемно повторенных мотивов в 31793 разных локусах генома человека. В богатых генами участках генома тандемные повторы наиболее часто обнаруживались в областях, прилежащих (на расстоянии около 1 т.п.н.) к точкам инициации транскрипции либо в 5'-нетранслируемых областях. Интересно отметить, что 2588 участков генома с экспансией тандемных повторов, выявленных в этой работе у индивидов с ASD, находятся в генах, вовлеченных в развитие нервной и сердечно-сосудистой систем, а также мышечной ткани [99]. С помощью биоинформатического метода MonSTR показано возникновение 35 новых полиморфных тандемных повторов, из которых 25 найдены в геномах больных ASD. Большинство таких повторов локализовано вблизи генов PDCD1, KCNB1, AGO1, CACNA2D3, FOXP1, RFX3 и MED13L, мутации в которых, как показано ранее, ассоциированы с заболеваниями ASD [376]. Результаты еще одного сравнительного анализа 544 геномов подтвердили, что значительная часть уникальных локусов CCG/CGG-микросателлитов (всего в геноме человека их чуть более 6000) расположена в 5'‑нетранслируемых областях генов (1836), экзонах (1529) или в непосредственной близости от промоторных областей (849). Такие повторы идентифицированы либо внутри генов, либо рядом с ними (всего 4370 генов), 410 из этих генов, как показано ранее, связаны с заболеваниями нервной системы человека [377].
Молекулярные механизмы патогенеза заболеваний, связанных с ЭП, нередко имеют комплексную природу, в них может участвовать не только ЭП, но и другие нарушения, что осложняет выявление роли ЭП. Так, недавно обнаружили дополнительные механизмы лице-плече-лопаточной миодистрофии Ландузи–Дежерина – считающегося классическом примером ЭП. Снижение числа копий теломерных повторов, наблюдаемое при этом заболевании, приводит к уменьшению количества ГХ и сверхэкспрессии генов в норме репрессированных теломерным ГХ. У здоровых людей в прителомерном районе хромосомы 4 находится около 100 копий макросателлита D4Z4 (единица повтора длиной 3.3 т.п.н.), в то время как у больных таких копий лишь 1–10 [378–381], и в клетках мышц повышена экспрессия генов FRG1, FRG2 и ANT1, расположенных рядом с областью повторов D4Z4 [382]. Исходя из данных, полученных на мышиных моделях, предположили, что наиболее вероятной причиной появления симптомов миодистрофии Ландузи–Дежерина можно считать сверхэкспрессию гена FMRG1 [383], которая сопровождается также нарушением сплайсинга генов, специфичных для мышц [384].
Экспансия микросателлитных повторов приводит к развитию ряда моногенных заболеваний человека (табл. S3 Приложения). Заболевание проявляется, когда экспансия превышает некий критический пороговый уровень, который сильно варьирует и специфичен для конкретной патологии. Одним из первых таких заболеваний стал синдром Мартина–Белл (синдром ломкой X хромосомы), при котором умственная отсталость сопровождается также нарушениями аутистического спектра [385]. В норме число тринуклеотидов CGG в 5'-нетранслируемой области гена FMR1 варьирует от 5 до 44, тогда как у больных находят более 200 копий CGG [386, 387]. Репрессия гена FMR1 обусловлена гиперметилированием промотора и 5'-нетранслируемой области этого гена [388]. Ген FMR1 кодирует РНК-связывающий белок FMRP, участвующий в метаболизме (трансляции, транспорте и стабильности) мРНК [389]. Недавно в модельной системе показали возможность участия микроРНК в механизме репрессии гена FMR1 [390], что также свидетельствует о сложных молекулярных механизмах ЭП, связанных с экспансией микросателлитов.
Понимание молекулярных механизмов ЭП имеет большое значение и для выявления и лечения ретровирусных инфекций, в первую очередь, ВИЧ. ВИЧ не только активно реплицируется в инфицированных Т-лимфоцитах, он может переходить в латентную фазу вплоть до недетектируемого уровня, в результате чего сохраняется в клетках человека и способен проявить себя по окончании антиретровирусной терапии (АРТ) [96, 391]. ВИЧ может переходить в латентное состояние на двух этапах: пре- и постинтегративном. Прединтегративное (эписомное) латентное состояние возникает, когда цикл репликации ВИЧ прерывается до события интеграции вирусной кДНК в геном клеток человека [392]. Эписомная кДНК ВИЧ может быть линейной или кольцевой, но обычно она обнаруживается в течение недолгого времени, хотя такие формы провируса способны и дольше сохраняться в макрофагах или в клетках головного мозга, тем не менее, клинически эти формы менее значимы, чем интегративная латентность ВИЧ [393–395]. Интегративная латентность ВИЧ возникает в результате блокировки ядерного экспорта вирусной РНК и трансляции белков вирусной оболочки. В значительной степени ВИЧ подавляется еще на уровне транскрипции, что делает провирус трудно детектируемым [396–398]. Для элиминации клеток с латентными провирусами применяют соединения-реактиваторы, которые запускают процессы репликации и наработки вирусных частиц в клетке, делая ее уязвимой для АРТ. Однако оказалось, что вследствие влияния локального окружения хроматина и регуляторных элементов генома такие препараты активируют только определенные группы провирусов, а значительная их часть сохраняется в латентном состоянии [399]. На сегодняшний день это является главной причиной, затрудняющей полное удаление ВИЧ. Поэтому изучение эпигенетических механизмов инактивации провируса ВИЧ служит целям поиска и разработки новых подходов к диагностике и лечению ВИЧ-инфекции. Значительный прогресс обеспечило использование штрихкодированных провирусов ВИЧ, основанное на методе TRIP (см. раздел “Новые методы изучения эффекта положения”). Этот метод позволяет не только определить места встраивания ретровируса в геном и детектировать уровень его экспрессии в зависимости от локального окружения хроматина, но также выявить предрасположенность к выходу из латентного состояния при применении того или иного реактивирующего соединения [391].
Транскрипция ВИЧ представляет собой сложный механизм, в котором участвуют как вирусные, так и клеточные факторы. Провирус ВИЧ нарабатывает короткие зрелые транскрипты, кодирующие трансактиватор (TAT) и регулятор экспрессии вириона (REV). Когда количество трансактиватора TAT достигнет определенного порога, он связывается с вирусной РНК, инициируя транскрипцию на промоторе провируса путем рекрутирования комплекса факторов элонгации (P-TEFb), состоящего из циклинзависимой киназы 9 (CDK9) и циклина T1 [400, 401]. Для активации РНК-полимеразы II должно произойти высвобождение CDK9 и связывание NELF (Negative Elongation Factor) и DSIF (DRB Sensitivity Inducing Factor) [402]. ТАТ также связывает белок PBAF (Polybromo-associated BRG-Associated Factor), ремоделирующий хроматин, который, по-видимому, обеспечивает открытую конформацию хроматина в месте встраивания провируса [403]. Нарушения этого механизма активации провируса приводят к переходу ВИЧ в латентное состояние. Так, фосфорилирование TAT киназой CDK2 приводит к задержке инициации транскрипции провируса [404]. Транскрипция провируса также зависит от наличия и соотношения транскрипционных факторов и транскрипционных репрессоров клетки-хозяина. Ядерный фактор NF-κB, участвующий в активации Т-клеток, может активировать транскрипцию ВИЧ даже в отсутствие белка TAT [405]. В таких клетках включается механизм обратной регуляции, опосредованный NFAT (Nuclear Factor of Activated T-cells), и накапливается фосфорилированная форма TAT [406, 407].
В инактивацию провируса ВИЧ вовлечен ряд белков, участвующих в процессах эпигенетической репрессии. В их число входят YY1 и LSF [408], которые действуют как модификаторы ЭП. Фактор CBF способен связываться с промотором провируса ВИЧ и эпигенетически подавлять транскрипцию, рекрутируя гистон-деацетилазы [409, 410]. Показана существенная зависимость активности провирусов от эпигенетического состояния хроматина в районах встроек и влияние метилирования CpG в промоторе провируса ВИЧ, которое препятствует доступу транскрипционных факторов [90, 411]. Показано, что вставки провируса ВИЧ чаще происходят в районы открытого хроматина [412–414]. Недавно обнаружили, что эндогенные ретровирусы (остатки копий ретровирусов, внедрявшихся в геномную ДНК когда-либо ранее) человека могут взаимодействовать с экзогенными вирусами, включая ВИЧ. Возможна как сборка химерных вирусных частиц из белков и РНК эндогенных и экзогенных вирусов, так и реактивация латентного провируса [415–417]. Кроме того, эти события могут приводить к интеграции в геном кДНК невирусного происхождения [418]. Для группы ретровирусов человека hsERV показано, что остатки ретровирусов в геноме млекопитающих могут играть роль энхансеров эндогенных генов [419]. Предполагается, что в эволюции белоккодирующих генов [419] они играют роль, сходную с ролью тандемных повторов [363]. В связи с этим разрабатываются альтернативные терапевтические стратегии, направленные на эпигенетическую инактивацию провирусов в геноме инфицированных клеток [397].
НОВЫЕ МЕТОДЫ ИЗУЧЕНИЯ ЭФФЕКТА ПОЛОЖЕНИЯ
В последние десятилетия кардинально расширились возможности изучения и анализа влияния локального окружения хроматина на экспрессию генов. Методы, основанные на анализе корреляций экспрессии генов и полногеномных профилей распределения белков хроматина, позволяют выявлять закономерности на уровне всего генома (см. раздел “Геномная компартментализация”). C другой стороны, совершенствуются методы создания и внедрения генно-инженерных конструкций, а также культивирования клеточных линий различного происхождения и генотипа. Революционными стали методы мультиплексного анализа TRIP и MPRA (Massive Parallel Reporter Assay), основанные на использовании ДНК-штрихкодов и позволяющие одновременно анализировать экспрессию одного и того же репортерного гена во множестве разных локальных хроматиновых контекстов и функционирование множества разных вариантов регуляторных последовательностей [72, 216, 271] (рис. 4а). С помощью этих подходов определена способность различных цис-регуляторных мотивов ДНК влиять на активность близлежащих и удаленных генов, которая в большой степени варьирует в зависимости от типа клеток и значительно зависит от локальной конформации хроматина [420].
Рис. 4.
Принцип и возможности экспериментальных подходов TRIP и MPRA. а – Штрихкодированные репортерные конструкции, которые содержат ограниченный набор разных промоторных элементов, интегрируются в случайные локусы генома клеток при помощи векторов на основе ДНК-транспозонов или лентивирусов. В дальнейшем картируют сайты вставок репортерных конструкций и определяют уровень их транскрипционной активности. б – Штрихкодированные конструкции, несущие огромное разнообразие последовательностей промоторных элементов, интегрируются в один и тот же локус генома посредством гомологичной или сайт-специфической рекомбинации. Последующие измерения степени доступности промоторной ДНК могут быть выполнены на единичных клетках. Штрихкоды служат универсальными метками как последовательностей ДНК (а и б), так и сайтов их локализации в геноме (а). ПР – промотор, ШК – штрихкод, ИП – исследуемая последовательность ДНК, ОРС – открытая рамка считывания.
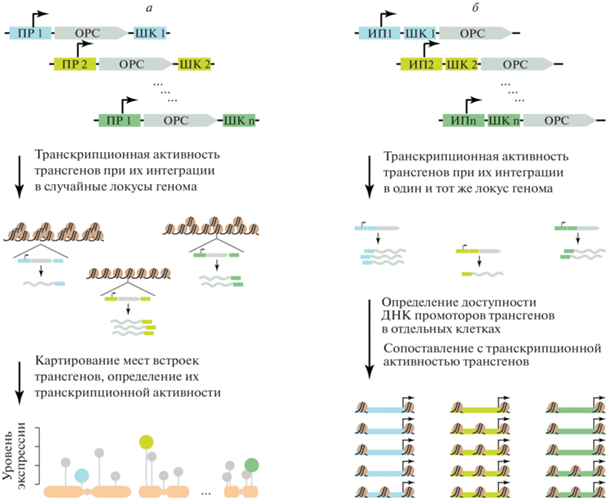
На основе MPRA получил новый виток развития такой классический метод изучения состояний хроматина, как определение плотности упаковки нуклеосом [421, 422] (рис. 4б). В частности, показана зависимость между посадкой различных факторов транскрипции и формированием закрытого либо открытого состояния хроматина на промоторах разного типа [423]. А один из новых методов, MIAA (Multiplexed Integrated Accessibility Assay), позволил массово анализировать способность различных мотивов ДНК формировать открытую конформацию хроматина, которая, как правило, связана с активацией экспрессии генов [424]. В этой методике химеры исследуемых белков с ДНК-аденин-метилтрансферазой Dam Escherichia coli метилируют GATC-мотивы в геномной ДНК. Усовершенствованный вариант этого метода – MadID, основан на использовании неспецифичной ДНК-аденин-метилтрансферазы M.EcoGII E. coli, что позволяет идентифицировать контакты исследуемого белка с геномной ДНК практически в любом хромосомном контексте, в частности, в участках ГХ, обедненных мотивами GATC [425].
Современный арсенал методов молекулярной генетики позволяет использовать различные подходы и их комбинации для функциональной диссекции механизмов ЭП, и выявлять роль отдельных генов и их продуктов в этих механизмах. Так, проведенные за последнее десятилетие крупномасштабные скрининговые исследования с использованием генных ловушек позволили выявить и охарактеризовать несколько десятков генов-модификаторов ЭП у млекопитающих, включая человека [74–76, 195, 200] (рис. 3б, 3в). Вставки репортерных конструкций различного дизайна в геномы культивируемых клеточных линий используются для изучения способности конкретных участков генома влиять на уровень экспрессии генов и вызывать их ЭП [77–80]. Системы регулируемой индукции экспрессии репортерного гена позволяют точно определить ту интенсивность экспрессии трансгена, которая может противостоять гетерохроматинизации при ЭП. Так, с использованием системы на основе дрожжевых белков GAL4 и термочувствительного варианта белка GAL80ts (ингибитор GAL4) показано, что увеличение экспрессии репортерного трансгена в 100 и более раз от фонового уровня препятствует ГХ репрессии в случае транс-инактивации, вызываемой инверсией In(2)A4 у дрозофилы [426].
В качестве удобной модели для функциональной диссекции механизмов генной репрессии у человека используют культивируемые клетки, содержащие условно гаплоидный набор хромосом [427]. Для поиска генов, вовлеченных в изучаемый молекулярный механизм, разработаны несколько вариаций метода генной ловушки. Этот метод заключается в массовом повреждении генов лентивирусными вставками с последующим функциональным скринингом и идентификацией сайтов вставок провирусов (поврежденных генов) [202, 203]. Большие возможности для изучения ЭП у человека открывает молекулярная характеристика сбалансированных хромосомных аномалий (Balanced Chromosomal Abnormalities, BCA). Анализ геномов пациентов с ВСА позволяет, в частности, обнаружить скомпенсированные случаи ЭП и определить хромосомные перестройки, а также возможное образование химерных транскриптов в результате хромосомных перестроек, с точностью до нуклеотида [326, 428, 429].
Третье поколение технологий секвенирования ДНК (TGS) открыло возможности непрерывного прочтения длинных последовательностей, в том числе, высоко повторенных фрагментов ДНК (Pacific Biosciences (PacBio) и Oxford Nanopore Technologies (ONT)), обеспечив, тем самым, прорыв в изучении роли тандемных повторов в развитии заболеваний человека. Так, сравнительный анализ более 500 индивидуальных геномов выявил более 6000 уникальных локусов повторов, значительная доля которых расположена в непосредственной близости к промоторным областям генов, а также сильно варьирует по длине. Такие повторы идентифицированы либо внутри, либо рядом более чем с 4000 генов, лишь десятая часть которых, как показано ранее, связана с заболеваниями нервной системы человека [377]. TGS открывает новые горизонты исследований распространения тандемных повторов в геноме человека и их роли в развитии различных заболеваний.
Получение и накопление больших массивов данных секвенирования геномов и необходимость сравнительного анализа данных, полученных от большого числа индивидов, влечет за собой активную разработку новых методов обработки и биоинформатического получения мета-данных. Например, с помощью метода MonSTR можно сравнивать данные полногеномного секвенирования выборки геномов, полученных от пациентов с подтвержденным диагнозом ASD и их родителей. Этот подход позволил также идентифицировать и охарактеризовать полиморфизмы коротких тандемных повторов, возникающих de novo, и связанных прежде всего с изменением числа таких повторов в конкретном локусе. Таким образом выявлено возникновение 35 новых полиморфизмов тандемных повторов, из которых 25 найдены в геномах больных ASD. Большинство таких полиморфизмов располагаются вблизи генов, мутации которых ассоциированы с заболеваниями класса ASD [376].
ЗАКЛЮЧЕНИЕ
История исследований ЭП гена насчитывает без малого 100 лет. Этот феномен демонстрирует фундаментальные различия между активным и репрессированным хроматином. ЭП генов представляет собой изменение лишь эпигенетического состояния, не связанного с мутациями или делециями самого гена. Различные виды ЭП удобно использовать для исследования и характеристики молекулярных процессов, лежащих в основе установления и переключения эпигенетического состояния. Современный арсенал методов полногеномных исследований и новые возможности массового молекулярного моделирования вместе с инструментами биоинформатического анализа открывают новые перспективы изучения эпигенетических механизмов генной репрессии и регуляции, в частности, с использованием ЭП гена как уникальной модельной системы.
Учитывая перспективы широкого внедрения генной инженерии и геномного редактирования как методов терапии генетических нарушений, необходимо иметь максимально точные представления о путях взаимного влияния генетического и эпигенетического уровня функционирования генома. Вводя трансгенные конструкции в ту или иную геномную локацию, нужно быть уверенным, во-первых, в безопасности и нейтральности такого внедрения для сохранения локального состояния хроматина, а во-вторых, обеспечить правильный уровень и паттерн экспрессии трансгена в условиях эпигенетического влияния локального геномного контекста. С этой, практической, точки зрения безусловно важно досконально понимать законы и механизмы эпигенетической регуляции и геномного метаболизма эукариотической клетки.
Авторы выражают благодарность А.А. Огиенко за помощь в подготовке и оформлении рисунков, М.Н. Егоровой за подготовку отдельных материалов. Первый автор выражает большую благодарность Е.С. Беляевой и И.Ф. Жимулеву за многолетнее научное руководство и мощную научную базу, а также Д.Е. Корякову за мудрые конструктивные замечания и Т.Д. Колесниковой за ценные советы по подготовке отдельных материалов обзора.
Подготовка данного обзора выполнена при финансовой поддержке Российского фонда фундаментальных исследований в рамках научного проекта №20-14-50398 (Funding: The reported study was funded by RFBR, project number 20-14-50398).
Настоящая статья не содержит каких-либо исследований с использованием животных в качестве объектов.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Sturtevant A.H. (1925) The effects of unequal crossing over at the bar locus in Drosophila. Genetics. 10, 117–147.
Lewis E.B. (1950) The phenomenon of position effect. Adv. Genet. 3, 73–115.
Muller H.J. (1930) Types of visible variations induced by X-rays in Drosophila. J. Genet. 22, 299–334.
Dobzhansky T. (1936) Position effects on genes. Biol. Rev. 11, 364–384.
Demerec M. (1940) Genetic behavior of euchromatic segments inserted into heterochromatin. Genetics. 25, 618–627.
Muller H.J. (1932) Further studies on the nature and causes of gene mutations. Proc. Sixth Int. Congr. Genet. 1, 213–255.
Паншин И.Б. (1938) Цитогенетические аспекты эффекта положения генов white (mottled) и cubitus interruptus. Биол. журн. 7, 838–868.
Tartof K.D., Hobbs C., Jones M. (1984) A structural basis for variegating position effects. Cell. 37, 869–878.
Reuter G., Wolff I., Friede B. (1985) Functional properties of the heterochromatic sequences inducing wm4 position-effect variegation in Drosophila melanogaster. Chromosoma. 93, 132–139.
Прокофьева-Бельговская А.А. (1945) Гетерохроматизация как изменение цикла хромосомы. Журн. общей биологии. 4(2), 93–124.
Schultz J. (1939) The function of heterochromatin. Proc. Natl. Acad. Sci. USA. 22, 27–33.
Tolchkov E.V., Kramerova I.A., Lavrov S.A., Rasheva V.I., Bonaccorsi S., Alatortsev V.E., Gvozdev V.A. (1997) Position-effect variegation in Drosophila melanogaster X chromosome inversion with a breakpoint in a satellite block and its suppression in a secondary rearrangement. Chromosoma. 106, 520–525.
Гвоздев В.А., Алаторцев В.Е., Аравин А.А., Калмыкова А.И., Коган Г.Л., Лавров С.А., Наумова Н.М., Нурминский Д.И., Рашева В.И., Толчков Е.В., Тулин А.В., Шевелев Ю.Я. (1999) Гетерохроматин: молекулярная эволюция и эффекты положения генов у Drosophila melanogaster. Молекуляр. биология. 33, 14–25.
Sabl J.F., Henikoff S. (1996) Copy number and orientation determine the susceptibility of a gene to silencing by nearby heterochromatin in Drosophila. Genetics. 142, 447–458.
Howe M., Dimitri P., Berloco M., Wakimoto B.T. (1995) Cis-effects of heterochromatin on heterochromatic and euchromatic gene activity in Drosophila melanogaster. Genetics. 140, 1033–1045.
Zhimulev I.F., Belyaeva E.S., Fomina O.V., Protopopov M.O., Bolshakov V.N. (1986) Cytogenetic and molecular aspects of position effect variegation in Drosophila melanogaster. Chromosoma. 94, 492–504.
Жимулев И.Ф. (1993) Гетерохроматин и эффект положения гена. Новосибирск: Наука, 490 с.
Жимулев И.Ф., Беляева Е.С. (2003) Гетерохроматин, эффект положения гена и генетический сайленсинг. Генетика. 39, 187–201.
Belyaeva E.S., Zhimulev I.F. (1991) Cytogenetic and molecular aspects of position effect variegation in Drosophila. III. Continuous and discontinuous compaction of chromosomal material as a result of position effect variegation. Chromosoma. 100, 453–466.
Talbert P.B., Henikoff S. (2000) A reexamination of spreading of position-effect variegation in the white-roughest region of Drosophila melanogaster. Genetics. 154, 259–272.
Дубинин Н.П., Сидоров Б.Н. (1935) Эффект положения гена hairy. Биол. журн. 4(3), 555–568.
Хвостова В.В. (1939) Роль инертных частей хромосом в эффекте положения гена cubitus interruptus у Drosophila melanogaster. Изв АН СССР, Сер. биол. наук. 4, 541–574.
Hessler A.Y. (1958) V-type position effect at the light locus in Drosophila melanogaster. Genetics. 43, 395–403.
Schultz J. (1936) Variegation in Drosophila and the inert chromosome regions. Proc. Natl. Acad. Sci. USA. 22, 27–33.
Baker W.K. (1953) V-type position effects on a gene normally located in heterochromatin. Genetics. 38, 328–344.
Spofford J.B. (1976) Position-effect variegation in Drosophila. In: The Genetics and Biology of Drosophila. Eds Ashburner M., Novitski E. London: Acad. Press. 1, 955–1018.
Rubin G.M., Spradling A.C. (1982) Genetic transformation of Drosophila with transposable element vectors. Science. 218, 348–353.
Spradling A.C., Rubin G.M. (1982) Transposition of cloned P elements into Drosophila germ line chromosomes. Science. 218, 341–347.
Hazelrigg T., Levis R., Rubin G.M. (1984) Transformation of white locus DNA in Drosophila: dosage compensation, zeste interaction, and position effects. Cell. 36, 469–481.
Steller H., Pirrotta V. (1985) Expression of the Drosophila white gene under the control of the hsp70 heat shock promoter. EMBO J. 4, 3765–3772.
Ahmad K., Golic K.G. (1996) Somatic reversion of chromosomal position effects in Drosophila melanogaster. Genetics. 144, 657–670.
Wallrath L.L., Elgin S.C. (1995) Position effect variegation in Drosophila is associated with an altered chromatin structure. Genes Dev. 9, 1263–1277.
Balasov M.L., Belyaeva E.S., Shestopal S.A., Makunin I.V., Zhimulev I.F. (2000) The transposon A(R)4-24P[white, rosy] in Drosophila melanogaster is subject to position-effect variegation at a non-centromeric insertion site. Mol. Gen. Genet. 264, 105–111.
Zhimulev I.F., Semeshin V.F., Kulichkov V.A., Be-lyaeva E.S. (1982) Intercalary heterochromatin in Drosophila. 1. Localization and general characteristics. Chromosoma. 87, 197–122.
Zhimulev I.F., Belyaeva E.S. (2003) Intercalary heterochromatin and genetic silencing. Bioessays. 25, 1040–1051.
Belyaeva E.S., Demakov S.A., Pokholkova G.V., Alekseyenko A.A., Kolesnikova T.D., Zhimulev I.F. (2006) DNA underreplication in intercalary heterochromatin regions in polytene chromosomes of Drosophila melanogaster correlates with the formation of partial chromosomal aberrations and ectopic pairing. Chromosoma. 115, 355–366.
Weiler K.S., Wakimoto B.T. (1995) Heterochromatin and gene expression in Drosophila. Annu. Rev. Genet. 29, 577–605.
Semeshin V.F., Demakov S.A., Perez Alonso M., Be-lyaeva E.S., Bonner J.J., Zhimulev I.F. (1989) Electron microscopical analysis of Drosophila polytene chromosomes. V. Characteristics of structures formed by transposed DNA segments of mobile elements. Chromosoma. 97, 396–412.
Semeshin V.F., Shloma V.V., Zhimulev I.F. (2001) Formation and morphology of dark puffs in Drosophila melanogaster polytene chromosomes. Hereditas. 134, 15–22.
Hartmann-Goldstein I.J. (1967) On the relationship between heterochromatization and variegation in Drosophila, with special reference to temperature-sensitive periods. Genet. Res. 10, 143–159.
Kornher J.S., Kauffman S.A. (1986) Variegated expression of the Sgs-4 locus in Drosophila melanogaster. Chromosoma. 94, 205–216.
Karpen G.H., Spradling A.C. (1990) Reduced DNA polytenization of a minichromosome region undergoing position-effect variegation in Drosophila. Cell. 63, 97–107.
Umbetova G.H., Belyaeva E.S., Baricheva E.M., Zhimulev I.F. (1991) Cytogenetic and molecular aspects of position effect variegation in Drosophila melanogaster. IV. Underreplication of chromosomal material as a result of gene inactivation. Chromosoma. 101, 55–61.
Belyaeva E.S., Demakova O.V., Umbetova G.H., Zhimulev I.F. (1993) Cytogenetic and molecular aspects of position-effect variegation in Drosophila melanogaster. V. Heterochromatin-associated protein HP1 appears in euchromatic chromosomal regions that are inactivated as a result of position-effect variegation. Chromosoma. 102, 583–590.
Belyaeva E.S., Boldyreva L.V., Volkova E.I., Na-nayev R.A., Alekseyenko A.A., Zhimulev I.F. (2003) Effect of the Suppressor of Underreplication (SuUR) gene on position-effect variegation silencing in Drosophila melanogaster. Genetics. 165, 1209–1220.
Slatis H.M. (1955) Position effects at the brown locus in Drosophila melanogaster. Genetics. 40, 5–23.
Henikoff S., Dreesen T.D. (1989) Trans-inactivation of the Drosophila brown gene: evidence for transcriptional repression and somatic pairing dependence. Proc. Natl. Acad. Sci. USA. 86, 6704–6708.
Dernburg A.F., Broman K.W., Fung J.C., Marshall W.F., Philips J., Agard D.A., Sedat J.W. (1996) Perturbation of nuclear architecture by long-distance chromosome interactions. Cell. 85, 745–759.
Беляева Е.С., Похолкова Г.В., Жимулев И.Ф. (2006) Эффект положения мозаичного типа brownD и его связь с ассоциациями гетерохроматиновых районов у Drosophila melanogaster. ДАН. 409 (4), 559–561.
Hinton T. (1950) A correlation of phenotypic changes and chromosomal rearrangements at the two ends of an inversion. Genetics. 35, 188–205.
Dorer D.R., Henikoff S. (1997) Transgene repeat arrays interact with distant heterochromatin and cause silencing in cis and trans. Genetics. 147, 1181–1190.
Martin-Morris L.E., Csink A.K., Dorer D.R., Talbert P.B., Henikoff S. (1997) Heterochromatic trans-inactivation of Drosophila white transgenes. Genetics. 147, 671–677.
Sass G.L., Henikoff S. (1999) Pairing-dependent mislocalization of a Drosophila brown gene reporter to a heterochromatic environment. Genetics. 152, 595–604.
Csink A.K., Bounoutas A., Griffith M.L., Sabl J.F., Sage B.T. (2002) Differential gene silencing by trans-heterochromatin in Drosophila melanogaster. Genetics. 160, 257–269.
Абрамов Ю.А., Кибанов М.В., Гвоздев В.А., Лавров С.А. (2011) Генетический и молекулярный анализы явления транс-инактивации генов у Drosophila melanogaster, обусловленного эу-гетерохроматиновой перестройкой гомологичной хромосомы. ДАН. 437(2), 261–265.
Sage B.T., Csink A.K. (2003) Heterochromatic self-association, a determinant of nuclear organization, does not require sequence homology in Drosophila. Genetics. 165, 1183–1193.
Thakar R., Csink A.K. (2005) Changing chromatin dynamics and nuclear organization during differentiation in Drosophila larval tissue. J. Cell. Sci. 118, 951–960.
Шацких А.С., Гвоздев В.А. (2013) Формирование гетерохроматина и транскрипция в связи с транс-инактивацией генов и их пространственной организацией в ядре. Биохимия. 78, 784–794.
Nisha P., Plank J.L., Csink A.K. (2008) Analysis of chromatin structure of genes silenced by heterochromatin in trans. Genetics. 179(1), 359–373.)
Abramov Y.A., Shatskikh A.S., Maksimenko O.G., Bonaccorsi S., Gvozdev V.A., Lavrov S.A. (2016) The differences between cis- and trans-gene inactivation caused by heterochromatin in Drosophila. Genetics. 202, 93–106.
Shatskikh A.S., Abramov Y.A., Lavrov S.A. (2017) Trans-inactivation: repression in a wrong place. Fly (Austin). 11, 96–103.
Tartof K.D. (1994) Position effect variegation in yeast. Bioessays. 16, 713–714.
Grunstein M., Gasser S.M. (2013) Epigenetics in Saccharomyces cerevisiae. Cold Spring Harb. Perspect. Biol. 5 (7), a017491.
Allshire R.C., Ekwall K. (2015) Epigenetic regulation of chromatin states in Schizosaccharomyces pombe. Cold Spring Harb. Perspect. Biol. 7, a018770.
Allshire R.C., Javerzat J.P., Redhead N.J., Cranston G. (1994) Position effect variegation at fission yeast centromeres. Cell. 76, 157–169.
Gottschling D.E., Aparicio O.M., Billington B.L., Zakian V.A. (1990) Position effect at S. cerevisiae telomeres: reversible repression of Pol II transcription. Cell. 63, 751–762.
Ottaviani A., Gilson E., Magdinier F. (2008) Telomeric position effect: from the yeast paradigm to human pathologies? Biochimie. 90, 93–107.
Corradini N., Rossi F., Giordano E., Caizzi R., Verni F., Dimitri P. (2007) Drosophila melanogaster as a model for studying protein-encoding genes that are resident in constitutive heterochromatin. Heredity (Edinb). 98, 3–12.
Girton J.R., Johansen K.M. (2008) Chromatin structure and the regulation of gene expression: the lessons of PEV in Drosophila. Adv. Genet. 61, 1–43.
Fodor B.D., Shukeir N., Reuter G., Jenuwein T. (2010) Mammalian Su(var) genes in chromatin control. Annu. Rev. Cell. Dev. Biol. 26, 471–501.
Elgin S.C., Reuter G. (2013) Position-effect variegation, heterochromatin formation, and gene silencing in Drosophila. Cold Spring Harb. Perspect. Biol. 5, a017780.
Akhtar W., de Jong J., Pindyurin A.V., Pagie L., Meuleman W., de Ridder J., Berns A., Wessels L.F., van Lohuizen M., van Steensel B. (2013) Chromatin position effects assayed by thousands of reporters integrated in parallel. Cell. 154, 914–927.
Akhtar W., Pindyurin A.V., de Jong J., Pagie L., Ten Hoeve J., Berns A., Wessels L.F., van Steensel B., van Lohuizen M. (2014) Using TRIP for genome-wide position effect analysis in cultured cells. Nat. Protoc. 9, 1255–1281.
Blewitt M.E., Vickaryous N.K., Hemley S.J., Ashe A., Bruxner T.J., Preis J.I., Arkell R., Whitelaw E. (2005) An N-ethyl-N-nitrosourea screen for genes involved in variegation in the mouse. Proc. Natl. Acad. Sci. USA. 102, 7629–-7634.
Ashe A., Morgan D.K., Whitelaw N.C., Bruxner T.J., Vickaryous N.K., Cox L.L., Butterfield N.C., Wicking C., Blewitt M.E., Wilkins S.J., Anderson G.J., Cox T.C., Whitelaw E. (2008) A genome-wide screen for modifiers of transgene variegation identifies genes with critical roles in development. Genome Biol. 9, R182.
Blewitt M., Whitelaw E. (2013) The use of mouse models to study epigenetics. Cold Spring Harb. Perspect. Biol. 5, a017939.
Gierman H.J., Indemans M.H., Koster J., Goetze S., Seppen J., Geerts D., van Driel R., Versteeg R. (2007) Domain-wide regulation of gene expression in the human genome. Genome Res. 17, 1286–1295.
Liu N., Lee C.H., Swigut T., Grow E., Gu B., Bassik M.C., Wysocka J. (2018) Selective silencing of euchromatic L1s revealed by genome-wide screens for L1 regulators. Nature. 553, 228–232.
Maricque B.B., Chaudhari H.G., Cohen B.A. (2018) A massively parallel reporter assay dissects the influence of chromatin structure on cis-regulatory activity. Nat. Biotechnol. 10, 1038/nbt.4285.
Leemans C., van der Zwalm M.C.H., Brueckner L., Comoglio F., van Schaik T., Pagie L., van Arensbergen J., van Steensel B. (2019) Promoter-intrinsic and local chromatin features determine gene repression in LADs. Cell. 177, 852–864. e814.
Gould A. (1997) Functions of mammalian Polycomb group and trithorax group related genes. Curr. Opin. Genet. Dev. 7, 488–494.
Caron H., van Schaik B., van der Mee M., Baas F., Riggins G., van Sluis P., Hermus M.C., van Asperen R., Boon K., Voute P.A., Heisterkamp S., van Kampen A., Versteeg R. (2001) The human transcriptome map: clustering of highly expressed genes in chromosomal domains. Science. 291, 1289–1292.
Lercher M.J., Urrutia A.O., Pavlicek A., Hurst L.D. (2003) A unification of mosaic structures in the human genome. Hum. Mol. Genet. 12, 2411–2415.
Versteeg R., van Schaik B.D., van Batenburg M.F., Roos M., Monajemi R., Caron H., Bussemaker H.J., van Kampen A.H. (2003) The human transcriptome map reveals extremes in gene density, intron length, GC content, and repeat pattern for domains of highly and weakly expressed genes. Genome Res. 13, 1998–2004.
Mijalski T., Harder A., Halder T., Kersten M., Horsch M., Strom T.M., Liebscher H.V., Lotts-peich F., de Angelis M.H., Beckers J. (2005) Identification of coexpressed gene clusters in a comparative analysis of transcriptome and proteome in mouse tissues. Proc. Natl. Acad. Sci. USA. 102, 8621–8626.
Singer G.A., Lloyd A.T., Huminiecki L.B., Wolfe K.H. (2005) Clusters of co-expressed genes in mammalian genomes are conserved by natural selection. Mol. Biol. Evol. 22, 767–775.
Lercher M.J., Urrutia A.O., Hurst L.D. (2002) Clustering of housekeeping genes provides a unified model of gene order in the human genome. Nat. Genet. 31, 180–183.
Lyko F., Beisel C., Marhold J., Paro R. (2006) Epigenetic regulation in Drosophila. Curr. Top. Microbiol. Immunol. 310, 23–44.
Nishibuchi G., Dejardin J. (2017) The molecular basis of the organization of repetitive DNA-containing constitutive heterochromatin in mammals. Chromosome Res. 25, 77–87.
Pearson R., Kim Y.K., Hokello J., Lassen K., Friedman J., Tyagi M., Karn J. (2008) Epigenetic silencing of human immunodeficiency virus (HIV) transcription by formation of restrictive chromatin structures at the viral long terminal repeat drives the progressive entry of HIV into latency. J. Virol. 82, 12291–12303.
Serman A., Vlahovic M., Serman L., Bulic-Jakus F. (2006) DNA methylation as a regulatory mechanism for gene expression in mammals. Coll. Antropol. 30, 665–671.
Dawson M.A., Kouzarides T. (2012) Cancer epigenetics: from mechanism to therapy. Cell. 150, 12–27.
LaCroix A.J., Stabley D., Sahraoui R., Adam M.P., Mehaffey M., Kernan K., Myers C.T., Fagerstrom C., Anadiotis G., Akkari Y.M., Robbins K.M., Gripp K.W., Baratela W.A.R., Bober M.B., Duker A.L., Doherty D., Dempsey J.C., Miller D.G., Kircher M., Bamshad M.J., Nickerson D.A., Mefford H.C., Sol-Church K. (2019) GGC repeat expansion and exon 1 methylation of XYLT1 is a common pathogenic variant in Baratela-Scott syndrome. Am. J. Hum. Genet. 104, 35–44.
Quilez J., Guilmatre A., Garg P., Highnam G., Gymrek M., Erlich Y., Joshi R.S., Mittelman D., Sharp A.J. (2016) Polymorphic tandem repeats within gene promoters act as modifiers of gene expression and DNA methylation in humans. Nucl. Acids Res. 44, 3750–3762.
Cedar H., Bergman Y. (2009) Linking DNA methylation and histone modification: patterns and paradigms. Nat. Rev. Genet. 10, 295–304.
Turner A.W., Margolis D.M. (2017) Chromatin regulation and the histone code in HIV latency. Yale J. Biol. Med. 90, 229–243.
Lange U.C., Verdikt R., Ait-Ammar A., Van Lint C. (2020) Epigenetic crosstalk in chronic infection with HIV-1. Semin. Immunopathol. 42, 187–200.
Khristich A.N., Mirkin S.M. (2020) On the wrong DNA track: molecular mechanisms of repeat-mediated genome instability. J. Biol. Chem. 295, 4134–4170.
Trost B., Engchuan W., Nguyen C.M., Thiruvahindrapuram B., Dolzhenko E., Backstrom I., Mirceta M., Mojarad B.A., Yin Y., Dov A., Chandrakumar I., Prasolava T., Shum N., Hamdan O., Pellecchia G., Howe J.L., Whitney J., Klee E.W., Baheti S., Amaral D.G., Anagnostou E., Elsabbagh M., Fernandez B.A., Hoang N., Lewis M.E.S., Liu X., Sjaarda C., Smith I.M., Szatmari P., Zwaigenbaum L., Glazer D., Hartley D., Stewart A.K., Eberle M.A., Sato N., Pearson C.E., Scherer S.W., Yuen R.K.C. (2020) Genome-wide detection of tandem DNA repeats that are expanded in autism. Nature. 586, 80–86.
Tartof K.D., Bishop C., Jones M., Hobbs C.A., Locke J. (1989) Towards an understanding of position effect variegation. Dev. Genet. 10, 162–176.
Spradling A.C., Karpen G.H. (1990) Sixty years of mystery. Genetics. 126, 779–784.
Zhimulev I.F. (1998) Polytene chromosomes, heterochromatin, and position effect variegation. Adv. Genet. 37, 1–555.
Parnell T.J., Grade S.K., Geyer P.K., Wallrath L.L. (2006) Position effect variegation in human genetic disease. eLS, Wiley Online Library. https://doi.org/10.1038/npg.els.0006040
Schulze S.R., Wallrath L.L. (2007) Gene regulation by chromatin structure: paradigms established in Drosophila melanogaster. Annu. Rev. Entomol. 52, 171–192.
Kramer J.A., McCarrey J.R., Djakiew D., Krawetz S.A. (1998) Differentiation: the selective potentiation of chromatin domains. Development. 125, 4749–4755.
Rasmussen T.P. (2003) Embryonic stem cell differentiation: a chromatin perspective. Reprod. Biol. Endocrinol. 1, 100.
Szutorisz H., Canzonetta C., Georgiou A., Chow C.M., Tora L., Dillon N. (2005) Formation of an active tissue-specific chromatin domain initiated by epigenetic marking at the embryonic stem cell stage. Mol. Cell. Biol. 25, 1804–1820.
Шевченко A.И., Павлова С.В., Дементьева Е.В., Голубева Д.В., Закиян С.М. (2006) Модификации хроматина в процессе инактивации Х-хромосомы у самок млекопитающих. Генетика. 42(9), 1225–1234.
Ball A.R., Jr., Yokomori K. (2011) Damage site chromatin: open or closed? Curr. Opin. Cell Biol. 23, 277–283.
Eissenberg J.C., Reuter G. (2009) Cellular mechanism for targeting heterochromatin formation in Drosophila. Int. Rev. Cell. Mol. Biol. 273, 1–47.
Peng J.C., Karpen G.H. (2008) Epigenetic regulation of heterochromatic DNA stability. Curr. Opin. Genet. Dev. 18, 204–211.
Janssen A., Colmenares S.U., Karpen G.H. (2018) Heterochromatin: guardian of the genome. Annu. Rev. Cell. Dev. Biol. 34, 265–288.
Richards E.J., Elgin S.C. (2002) Epigenetic codes for heterochromatin formation and silencing: rounding up the usual suspects. Cell. 108, 489–500.
Dillon N., Festenstein R. (2002) Unravelling heterochromatin: competition between positive and negative factors regulates accessibility. Trends Genet. 18, 252–258.
Dimitri P., Corradini N., Rossi F., Verni F. (2005) The paradox of functional heterochromatin. Bioessays. 27, 29–41.
Huisinga K.L., Brower-Toland B., Elgin S.C. (2006) The contradictory definitions of heterochromatin: transcription and silencing. Chromosoma. 115, 110–122.
Колесникова Т.Д., Андреева Е.Н., Пиндюрин А.В., Ананько Н.Г., Белякин С.Н., Шлома В.В., Юрлова А.А., Макунин И.В., Похолкова Г.В., Волкова Е.И., Заруцкая Е.А., Кокоза Е.Б., Семешин В.Ф., Беляева Е.С., Жимулев И.Ф. (2006) Ген SuUR и его участие в организации эпигенетически репрессированных районов хромосом Drosophila melanogaster. Генетика. 42(8), 1013–1028.
Belyaeva E.S., Goncharov F.P., Demakova O.V., Kolesnikova T.D., Boldyreva L.V., Semeshin V.F., Zhimulev I.F. (2012) Late replication domains in polytene and non-polytene cells of Drosophila melanogaster. PLoS One. 7, e30035.
McNairn A.J., Gilbert D.M. (2003) Epigenomic replication: linking epigenetics to DNA replication. Bioessays. 25, 647–656.
Allshire R., Bickmore W. (2000) Pausing for thought on the boundaries of imprinting. Cell. 102, 705–708.
Turner B.M. (2002) Cellular memory and the histone code. Cell. 111, 285–291.
Vlassova I.E., Graphodatsky A.S., Belyaeva E.S., Zhimulev I.F. (1991) Constitutive heterochromatin in early embryogenesis of Drosophila melanogaster. Mol. Gen. Genet. 229, 316–318.
Bogolyubova I., Bogolyubov D. (2020) Heterochromatin morphodynamics in late oogenesis and early embryogenesis of mammals. Cells. 9(6), e1497.
Labrador M., Corces V.G. (2002) Setting the boundaries of chromatin domains and nuclear organization. Cell. 111, 151–154.
Ebert A., Schotta G., Lein S., Kubicek S., Krauss V., Jenuwein T., Reuter G. (2004) Su(var) genes regulate the balance between euchromatin and heterochromatin in Drosophila. Genes Dev. 18, 2973–2983.
Kornberg R.D. (1974) Chromatin structure: a repeating unit of histones and DNA. Science. 184, 868–871.
Oudet P., Gross-Bellard M., Chambon P. (1975) Electron microscopic and biochemical evidence that chromatin structure is a repeating unit. Cell. 4, 281–300.
Finch J.T., Lutter L.C., Rhodes D., Brown R.S., Rushton B., Levitt M., Klug A. (1977) Structure of nucleosome core particles of chromatin. Nature. 269, 29–36.
Thomas J.O., Kornberg R.D. (1975) An octamer of histones in chromatin and free in solution. Proc. Natl. Acad. Sci. USA. 72, 2626–2630.
Arents G., Burlingame R.W., Wang B.C., Love W.E., Moudrianakis E.N. (1991) The nucleosomal core histone octamer at 3.1 A resolution: a tripartite protein assembly and a left-handed superhelix. Proc. Natl. Acad. Sci. USA. 88, 10148–10152.
Lilley D.M., Pardon J.F., Richards B.M. (1977) Structural investigations of chromatin core protein by nuclear magnetic resonance. Biochemistry. 16, 2853–2860.
Luger K., Mader A.W., Richmond R.K., Sargent D.F., Richmond T.J. (1997) Crystal structure of the nucleosome core particle at 2.8 A resolution. Nature. 389, 251–260.
Varshavsky A.J., Bakayev V.V., Georgiev G.P. (1976) Heterogeneity of chromatin subunits in vitro and location of histone H1. Nucl. Acids Res. 3, 477–492.
Fyodorov D.V., Zhou B.R., Skoultchi A.I., Bai Y. (2018) Emerging roles of linker histones in regulating chromatin structure and function. Nat. Rev. Mol. Cell. Biol. 19, 192–206.
Allan J., Hartman P.G., Crane-Robinson C., Aviles F.X. (1980) The structure of histone H1 and its location in chromatin. Nature. 288, 675–679.
Mirzabekov A.D., Shick V.V., Belyavsky A.V., Ba-vykin S.G. (1978) Primary organization of nucleosome core particle of chromatin: sequence of histone arrangement along DNA. Proc. Natl. Acad. Sci. USA. 75, 4184–4188.
Talbert P.B., Henikoff S. (2010) Histone variants – ancient wrap artists of the epigenome. Nat. Rev. Mol. Cell. Biol. 11, 264–275.
Martire S., Banaszynski L.A. (2020) The roles of histone variants in fine-tuning chromatin organization and function. Nat. Rev. Mol. Cell. Biol. 21, 522–541.
Strahl B.D., Allis C.D. (2000) The language of covalent histone modifications. Nature. 403, 41–45.
Jenuwein T., Allis C.D. (2001) Translating the histone code. Science. 293, 1074–1080.
Tessarz P., Kouzarides T. (2014) Histone core modifications regulating nucleosome structure and dynamics. Nat. Rev. Mol. Cell. Biol. 15, 703–708.
Zentner G.E., Henikoff S. (2013) Regulation of nucleosome dynamics by histone modifications. Nat. Struct. Mol. Biol. 20, 259–266.
Taverna S.D., Li H., Ruthenburg A.J., Allis C.D., Patel D.J. (2007) How chromatin-binding modules interpret histone modifications: lessons from professional pocket pickers. Nat. Struct. Mol. Biol. 14, 1025–1040.
Lee J.S., Smith E., Shilatifard A. (2010) The language of histone crosstalk. Cell. 142, 682–685.
Коряков Д.Е. (2006) Модификации гистонов и регуляция работы хроматина. Генетика. 42(9), 1170–1185.
Kouzarides T. (2007) Chromatin modifications and their function. Cell. 128, 693–705.
Bannister A.J., Kouzarides T. (2011) Regulation of chromatin by histone modifications. Cell Res. 21, 381–395.
Zhang T., Cooper S., Brockdorff N. (2015) The interplay of histone modifications – writers that read. EMBO Rep. 16, 1467–1481.
Lawrence M., Daujat S., Schneider R. (2016) Lateral thinking: how histone modifications regulate gene expression. Trends Genet. 32, 42–56.
Cheung P., Allis C.D., Sassone-Corsi P. (2000) Signaling to chromatin through histone modifications. Cell. 103, 263–271.
Fischle W., Wang Y., Allis C.D. (2003) Histone and chromatin cross-talk. Curr. Opin. Cell. Biol. 15, 172–183.
Gelato K.A., Fischle W. (2008) Role of histone modifications in defining chromatin structure and function. Biol. Chem. 389, 353–363.
Bernstein B.E., Kamal M., Lindblad-Toh K., Beki-ranov S., Bailey D.K., Huebert D.J., McMahon S., Karlsson E.K., Kulbokas E.J., 3rd, Gingeras T.R., Schreiber S.L., Lander E.S. (2005) Genomic maps and comparative analysis of histone modifications in human and mouse. Cell. 120, 169–181.
Pokholok D.K., Harbison C.T., Levine S., Cole M., Hannett N.M., Lee T.I., Bell G.W., Walker K., Rolfe P.A., Herbolsheimer E., Zeitlinger J., Lewitter F., Gifford D.K., Young R.A. (2005) Genome-wide map of nucleosome acetylation and methylation in yeast. Cell. 122, 517–527.
Schubeler D., MacAlpine D.M., Scalzo D., Wirbelauer C., Kooperberg C., van Leeuwen F., Gottschling D.E., O’Neill L.P., Turner B.M., Delrow J., Bell S.P., Groudine M. (2004) The histone modification pattern of active genes revealed through genome-wide chromatin analysis of a higher eukaryote. Genes Dev. 18, 1263–1271.
Wade P.A., Pruss D., Wolffe A.P. (1997) Histone acetylation: chromatin in action. Trends Biochem. Sci. 22, 128–132.
Marmorstein R., Zhou M.M. (2014) Writers and readers of histone acetylation: structure, mechanism, and inhibition. Cold Spring Harb. Perspect. Biol. 6, a018762.
Lloyd J.T., Glass K.C. (2018) Biological function and histone recognition of family IV bromodomain-containing proteins. J. Cell. Physiol. 233, 1877–1886.
Murr R. (2010) Interplay between different epigenetic modifications and mechanisms. Adv. Genet. 70, 101–141.
Turner B.M., Birley A.J., Lavender J. (1992) Histone H4 isoforms acetylated at specific lysine residues define individual chromosomes and chromatin domains in Drosophila polytene nuclei. Cell. 69, 375–384.
Kouzarides T. (2002) Histone methylation in transcriptional control. Curr. Opin. Genet. Dev. 12, 198–209.
Lachner M., O’Sullivan R.J., Jenuwein T. (2003) An epigenetic road map for histone lysine methylation. J. Cell. Sci. 116, 2117–2124.
Black J.C., Van Rechem C., Whetstine J.R. (2012) Histone lysine methylation dynamics: establishment, regulation, and biological impact. Mol. Cell. 48, 491–507.
Rando O.J. (2012) Combinatorial complexity in chromatin structure and function: revisiting the histone code. Curr. Opin. Genet. Dev. 22, 148–155.
Locke J., Kotarski M.A., Tartof K.D. (1988) Dosage-dependent modifiers of position effect variegation in Drosophila and a mass action model that explains their effect. Genetics. 120, 181–198.
Schotta G., Ebert A., Dorn R., Reuter G. (2003) Position-effect variegation and the genetic dissection of chromatin regulation in Drosophila. Semin. Cell. Dev. Biol. 14, 67–75.
James T.C., Elgin S.C. (1986) Identification of a nonhistone chromosomal protein associated with heterochromatin in Drosophila melanogaster and its gene. Mol. Cell. Biol. 6, 3862–3872.
Wustmann G., Szidonya J., Taubert H., Reuter G. (1989) The genetics of position-effect variegation modifying loci in Drosophila melanogaster. Mol. Gen. Genet. 217, 520–527.
Csink A.K., Henikoff S. (1996) Genetic modification of heterochromatic association and nuclear organization in Drosophila. Nature. 381, 529–531.
Pindyurin A.V., Ilyin A.A., Ivankin A.V., Tselebrovsky M.V., Nenasheva V.V., Mikhaleva E.A., Pagie L., van Steensel B., Shevelyov Y.Y. (2018) The large fraction of heterochromatin in Drosophila neurons is bound by both B-type lamin and HP1a. Epigenetics Chromatin. 11, 65.
Guthmann M., Burton A., Torres-Padilla M.E. (2019) Expression and phase separation potential of heterochromatin proteins during early mouse development. EMBO Rep. 20, e47952.
Meyer-Nava S., Nieto-Caballero V.E., Zurita M., Valadez-Graham V. (2020) Insights into HP1a-chromatin interactions. Cells. 9(8), e1866.
Elgin S.C. (1996) Heterochromatin and gene regulation in Drosophila. Curr. Opin. Genet. Dev. 6, 193–202.
Brown E.J., Nguyen A.H., Bachtrog D. (2020) The Drosophila Y chromosome affects heterochromatin integrity genome-wide. Mol. Biol. Evol. 37, 2808–2824.
Eissenberg J.C., James T.C., Foster-Hartnett D.M., Hartnett T., Ngan V., Elgin S.C. (1990) Mutation in a heterochromatin-specific chromosomal protein is associated with suppression of position-effect variegation in Drosophila melanogaster. Proc. Natl. Acad. Sci. USA. 87, 9923–9927.
Jacobs S.A., Taverna S.D., Zhang Y., Briggs S.D., Li J., Eissenberg J.C., Allis C.D., Khorasanizadeh S. (2001) Specificity of the HP1 chromo domain for the methylated N-terminus of histone H3. EMBO J. 20, 5232–5241.
Nielsen P.R., Nietlispach D., Mott H.R., Callaghan J., Bannister A., Kouzarides T., Murzin A.G., Murzina N.V., Laue E.D. (2002) Structure of the HP1 chromodomain bound to histone H3 methylated at lysine 9. Nature. 416, 103–107.
Vogel M.J., Pagie L., Talhout W., Nieuwland M., Kerkhoven R.M., van Steensel B. (2009) High-resolution mapping of heterochromatin redistribution in a Drosophila position-effect variegation model. Epigenetics Chromatin. 2, 1.
Reuter G., Gausz J., Gyurkovics H., Friede B., Bang R., Spierer A., Hall L.M., Spierer P. (1987) Modifiers of position-effect variegation in the region from 86C to 88B of the Drosophila melanogaster third chromosome. Mol. Gen. Genet. 210, 429–436.
Rea S., Eisenhaber F., O’Carroll D., Strahl B.D., Sun Z.W., Schmid M., Opravil S., Mechtler K., Ponting C.P., Allis C.D., Jenuwein T. (2000) Regulation of chromatin structure by site-specific histone H3 methyltransferases. Nature. 406, 593–599.
Schotta G., Ebert A., Krauss V., Fischer A., Hoffmann J., Rea S., Jenuwein T., Dorn R., Reuter G. (2002) Central role of Drosophila SU(VAR)3-9 in histone H3-K9 methylation and heterochromatic gene silencing. EMBO J. 21, 1121–1131.
Eskeland R., Eberharter A., Imhof A. (2007) HP1 binding to chromatin methylated at H3K9 is enhanced by auxiliary factors. Mol. Cell Biol. 27, 453–465.
Fanti L., Giovinazzo G., Berloco M., Pimpinelli S. (1998) The heterochromatin protein 1 prevents telomere fusions in Drosophila. Mol. Cell. 2, 527–538.
Seum C., Reo E., Peng H., Rauscher F.J., 3rd, Spierer P., Bontron S. (2007) Drosophila SETDB1 is required for chromosome 4 silencing. PLoS Genet. 3, e76.
Brower-Toland B., Riddle N.C., Jiang H., Huisinga K.L., Elgin S.C. (2009) Multiple SET methyltransferases are required to maintain normal heterochromatin domains in the genome of Drosophila melanogaster. Genetics. 181, 1303–1319.
Robin J.D., Ludlow A.T., Batten K., Magdinier F., Stadler G., Wagner K.R., Shay J.W., Wright W.E. (2014) Telomere position effect: regulation of gene expression with progressive telomere shortening over long distances. Genes Dev. 28, 2464–2476.
Aparicio O.M., Billington B.L., Gottschling D.E. (1991) Modifiers of position effect are shared between telomeric and silent mating-type loci in S. cerevisiae. Cell. 66, 1279–1287.
Grewal S.I., Jia S. (2007) Heterochromatin revisited. Nat. Rev. Genet. 8, 35–46.
Henderson I.R., Jacobsen S.E. (2007) Epigenetic inheritance in plants. Nature. 447, 418–424.
Berloco M., Palumbo G., Piacentini L., Pimpinelli S., Fanti L. (2014) Position effect variegation and viability are both sensitive to dosage of constitutive heterochromatin in Drosophila. G3 (Bethesda). 4, 1709–1716.
Aagaard L., Laible G., Selenko P., Schmid M., Dorn R., Schotta G., Kuhfittig S., Wolf A., Lebersorger A., Singh P.B., Reuter G., Jenuwein T. (1999) Functional mammalian homologues of the Drosophila PEV-modifier Su(var)3-9 encode centromere-associated proteins which complex with the heterochromatin component M31. EMBO J. 18, 1923–1938.
Hendrich B., Bird A. (1998) Identification and characterization of a family of mammalian methyl-CpG binding proteins. Mol. Cell. Biol. 18, 6538–6547.
Ayyanathan K., Lechner M.S., Bell P., Maul G.G., Schultz D.C., Yamada Y., Tanaka K., Torigoe K., Rauscher F.J., 3rd (2003) Regulated recruitment of HP1 to a euchromatic gene induces mitotically heritable, epigenetic gene silencing: a mammalian cell culture model of gene variegation. Genes Dev. 17, 1855–1869.
Hassig C.A., Tong J.K., Fleischer T.C., Owa T., Grable P.G., Ayer D.E., Schreiber S.L. (1998) A role for histone deacetylase activity in HDAC1-mediated transcriptional repression. Proc. Natl. Acad. Sci. USA. 95, 3519–3524.
Timms R.T., Tchasovnikarova I.A., Lehner P.J. (2016) Position-effect variegation revisited: HUSHing up heterochromatin in human cells. Bioessays. 38, 333–343.
Whitelaw N.C., Chong S., Morgan D.K., Nestor C., Bruxner T.J., Ashe A., Lambley E., Meehan R., Whitelaw E. (2010) Reduced levels of two modifiers of epigenetic gene silencing, Dnmt3a and Trim28, cause increased phenotypic noise. Genome Biol. 11, R111.
Sorolla A., Tallack M.R., Oey H., Harten S.K., Daxinger L.C., Magor G.W., Combes A.N., Ilsley M., Whitelaw E., Perkins A.C. (2015) Identification of novel hypomorphic and null mutations in Klf1 derived from a genetic screen for modifiers of alpha-globin transgene variegation. Genomics. 105, 116–122.
Rountree M.R., Bachman K.E., Baylin S.B. (2000) DNMT1 binds HDAC2 and a new co-repressor, DMAP1, to form a complex at replication foci. Nat. Genet. 25, 269–277.
Bachman K.E., Rountree M.R., Baylin S.B. (2001) Dnmt3a and Dnmt3b are transcriptional repressors that exhibit unique localization properties to heterochromatin. J. Biol. Chem. 276, 32282–32287.
Tchasovnikarova I.A., Timms R.T., Matheson N.J., Wals K., Antrobus R., Gottgens B., Dougan G., Dawson M.A., Lehner P.J. (2015) Gene silencing. Epigenetic silencing by the HUSH complex mediates position-effect variegation in human cells. Science. 348, 1481–1485.
Blewitt M.E., Gendrel A.V., Pang Z., Sparrow D.B., Whitelaw N., Craig J.M., Apedaile A., Hilton D.J., Dunwoodie S.L., Brockdorff N., Kay G.F., White-law E. (2008) SmcHD1, containing a structural-maintenance-of-chromosomes hinge domain, has a critical role in X inactivation. Nat. Genet. 40, 663–669.
Carette J.E., Guimaraes C.P., Wuethrich I., Blo-men V.A., Varadarajan M., Sun C., Bell G., Yuan B., Muellner M.K., Nijman S.M., Ploegh H.L., Brummelkamp T.R. (2011) Global gene disruption in human cells to assign genes to phenotypes by deep sequencing. Nat. Biotechnol. 29, 542–546.
Duncan L.M., Timms R.T., Zavodszky E., Cano F., Dougan G., Randow F., Lehner P.J. (2012) Fluorescence-based phenotypic selection allows forward genetic screens in haploid human cells. PLoS One. 7, e39651.
Schultz D.C., Ayyanathan K., Negorev D., Maul G.G., Rauscher F.J., 3rd (2002) SETDB1: a novel KAP-1-associated histone H3, lysine 9-specific methyltransferase that contributes to HP1-mediated silencing of euchromatic genes by KRAB zinc-finger proteins. Genes Dev. 16, 919–932.
Kokura K., Sun L., Bedford M.T., Fang J. (2010) Methyl-H3K9-binding protein MPP8 mediates E-cadherin gene silencing and promotes tumour cell motility and invasion. EMBO J. 29, 3673–3687.
Li J., Li Z., Ruan J., Xu C., Tong Y., Pan P.W., Tempel W., Crombet L., Min J., Zang J. (2011) Structural basis for specific binding of human MPP8 chromodomain to histone H3 methylated at lysine 9. PLoS One. 6, e25104.
Riethoven J.J. (2010) Regulatory regions in DNA: promoters, enhancers, silencers, and insulators. Methods Mol. Biol. 674, 33–42.
Negre N., Brown C.D., Ma L., Bristow C.A., Miller S.W., Wagner U., Kheradpour P., Eaton M.L., Loriaux P., Sealfon R., Li Z., Ishii H., Spokony R.F., Chen J., Hwang L., Cheng C., Auburn R.P., Davis M.B., Domanus M., Shah P.K., Morrison C.A., Zieba J., Suchy S., Senderowicz L., Victorsen A., Bild N.A., Grundstad A.J., Hanley D., MacAlpine D.M., Mannervik M., Venken K., Bellen H., White R., Gerstein M., Russell S., Grossman R.L., Ren B., Posakony J.W., Kellis M., White K.P. (2011) A cis-regulatory map of the Drosophila genome. Nature. 471, 527–531.
Разин С.В., Гаврилов А.А., Ульянов С.В. (2015) Регуляторные элементы эукариотического генома, контролирующие транскрипцию. Молекуляр. биология. 49(2), 212–223.
Мельникова Л.С., Георгиев П.Г., Головнин А.К. (2020) Функции и механизмы действия инсуляторов в геномах высших эукариот. Acta Naturae. 12(4), 15–33.
Schwartz Y.B., Cavalli G. (2017) Three-dimensional genome organization and function in Drosophila. Genetics. 205, 5–24.
Lieberman-Aiden E., van Berkum N.L., Williams L., Imakaev M., Ragoczy T., Telling A., Amit I., Lajoie B.R., Sabo P.J., Dorschner M.O., Sandstrom R., Bernstein B., Bender M.A., Groudine M., Gnirke A., Stamatoyannopoulos J., Mirny L.A., Lander E.S., Dekker J. (2009) Comprehensive mapping of long-range interactions reveals folding principles of the human genome. Science. 326, 289–293.
de Laat W., Klous P., Kooren J., Noordermeer D., Palstra R.J., Simonis M., Splinter E., Grosveld F. (2008) Three-dimensional organization of gene expression in erythroid cells. Curr. Top. Dev. Biol. 82, 117–139.
Salem T., Gomard T., Court F., Moquet-Torcy G., Brockly F., Forne T., Piechaczyk M. (2013) Chromatin loop organization of the junb locus in mouse dendritic cells. Nucl. Acids Res. 41, 8908–8925.
Gheldof N., Smith E.M., Tabuchi T.M., Koch C.M., Dunham I., Stamatoyannopoulos J.A., Dekker J. (2010) Cell-type-specific long-range looping interactions identify distant regulatory elements of the CFTR gene. Nucl. Acids Res. 38, 4325–4336.
Gisselbrecht S.S., Palagi A., Kurland J.V., Rogers J.M., Ozadam H., Zhan Y., Dekker J., Bulyk M.L. (2020) Transcriptional silencers in Drosophila serve a dual role as transcriptional enhancers in alternate cellular contexts. Mol. Cell. 77, 324–337 e328.
Rao S.S., Huntley M.H., Durand N.C., Stamenova E.K., Bochkov I.D., Robinson J.T., Sanborn A.L., Machol I., Omer A.D., Lander E.S., Aiden E.L. (2014) A 3D map of the human genome at kilobase resolution reveals principles of chromatin looping. Cell. 159, 1665–1680.
Cope N.F., Fraser P., Eskiw C.H. (2010) The yin and yang of chromatin spatial organization. Genome Biol. 11, 204.
Udvardy A., Maine E., Schedl P. (1985) The 87A7 chromomere. Identification of novel chromatin structures flanking the heat shock locus that may define the boundaries of higher order domains. J. Mol. Biol. 185, 341–358.
Kellum R., Schedl P. (1991) A position-effect assay for boundaries of higher order chromosomal domains. Cell. 64, 941–950.
Holdridge C., Dorsett D. (1991) Repression of hsp70 heat shock gene transcription by the suppressor of hairy-wing protein of Drosophila melanogaster. Mol. Cell. Biol. 11, 1894–1900.
Geyer P.K., Corces V.G. (1992) DNA position-specific repression of transcription by a Drosophila zinc finger protein. Genes Dev. 6, 1865–1873.
Valenzuela L., Kamakaka R.T. (2006) Chromatin insulators. Annu. Rev. Genet. 40, 107–138.
Bushey A.M., Dorman E.R., Corces V.G. (2008) Chromatin insulators: regulatory mechanisms and epigenetic inheritance. Mol. Cell. 32, 1–9.
Kyrchanova O., Georgiev P. (2014) Chromatin insulators and long-distance interactions in Drosophila. FEBS Lett. 588, 8–14.
Zolotarev N., Maksimenko O., Kyrchanova O., Sokolinskaya E., Osadchiy I., Girardot C., Bonchuk A., Ciglar L., Furlong E.E.M., Georgiev P. (2017) Opbp is a new architectural/insulator protein required for ribosomal gene expression. Nucl. Acids Res. 45, 12285–12300.
Bag I., Dale R.K., Palmer C., Lei E.P. (2019) The zinc-finger protein CLAMP promotes gypsy chromatin insulator function in Drosophila. J. Cell. Sci. 132(5), e-jcs226092.
Gerasimova T.I., Byrd K., Corces V.G. (2000) A chromatin insulator determines the nuclear localization of DNA. Mol. Cell. 6, 1025–1035.
Zolotarev N., Fedotova A., Kyrchanova O., Bonchuk A., Penin A.A., Lando A.S., Eliseeva I.A., Kulakovskiy I.V., Maksimenko O., Georgiev P. (2016) Architectural proteins Pita, Zw5, and ZIPIC contain homodimerization domain and support specific long-range interactions in Drosophila. Nucl. Acids Res. 44, 7228–7241.
Bonchuk A., Kamalyan S., Mariasina S., Boyko K., Popov V., Maksimenko O., Georgiev P. (2020) N-terminal domain of the architectural protein CTCF has similar structural organization and ability to self-association in bilaterian organisms. Sci. Rep. 10, 2677.
Bonchuk A., Boyko K., Fedotova A., Nikolaeva A., Lushchekina S., Khrustaleva A., Popov V., Georgiev P. (2021) Structural basis of diversity and homodimerization specificity of zinc-finger-associated domains in Drosophila. Nucl. Acids Res. 49, 2375–2389.
Golovnin A., Mazur A., Kopantseva M., Kurshakova M., Gulak P.V., Gilmore B., Whitfield W.G., Geyer P., Pirrotta V., Georgiev P. (2007) Integrity of the Mod(mdg4)-67.2 BTB domain is critical to insulator function in Drosophila melanogaster. Mol. Cell. Biol. 27, 963–974.
Bonchuk A., Maksimenko O., Kyrchanova O., Ivlieva T., Mogila V., Deshpande G., Wolle D., Schedl P., Georgiev P. (2015) Functional role of dimerization and CP190 interacting domains of CTCF protein in Drosophila melanogaster. BMC Biol. 13, 63.
Bonchuk A., Denisov S., Georgiev P., Maksimenko O. (2011) Drosophila BTB/POZ domains of “ttk group” can form multimers and selectively interact with each other. J. Mol. Biol. 412, 423–436.
Van Bortle K., Ramos E., Takenaka N., Yang J., Wahi J.E., Corces V.G. (2012) Drosophila CTCF tandemly aligns with other insulator proteins at the borders of H3K27me3 domains. Genome Res. 22, 2176–2187.
Schwartz Y.B., Linder-Basso D., Kharchenko P.V., Tolstorukov M.Y., Kim M., Li H.B., Gorchakov A.A., Minoda A., Shanower G., Alekseyenko A.A., Riddle N.C., Jung Y.L., Gu T., Plachetka A., Elgin S.C., Kuroda M.I., Park P.J., Savitsky M., Karpen G.H., Pirrotta V. (2012) Nature and function of insulator protein binding sites in the Drosophila genome. Genome Res. 22, 2188–2198.
Hore T.A., Deakin J.E., Marshall Graves J.A. (2008) The evolution of epigenetic regulators CTCF and BORIS/CTCFL in amniotes. PLoS Genet. 4, e1000169.
Kadota M., Hara Y., Tanaka K., Takagi W., Tanegashima C., Nishimura O., Kuraku S. (2017) CTCF binding landscape in jawless fish with reference to Hox cluster evolution. Sci. Rep. 7, 4957.
Yusufzai T.M., Tagami H., Nakatani Y., Felsenfeld G. (2004) CTCF tethers an insulator to subnuclear sites, suggesting shared insulator mechanisms across species. Mol. Cell. 13, 291–298.
Kyrchanova O., Ivlieva T., Toshchakov S., Parshikov A., Maksimenko O., Georgiev P. (2011) Selective interactions of boundaries with upstream region of Abd-B promoter in Drosophila bithorax complex and role of dCTCF in this process. Nucl. Acids Res. 39, 3042–3052.
Sanborn A.L., Rao S.S., Huang S.C., Durand N.C., Huntley M.H., Jewett A.I., Bochkov I.D., Chinnappan D., Cutkosky A., Li J., Geeting K.P., Gnirke A., Melnikov A., McKenna D., Stamenova E.K., Lan-der E.S., Aiden E.L. (2015) Chromatin extrusion explains key features of loop and domain formation in wild-type and engineered genomes. Proc. Natl. Acad. Sci. USA. 112, E6456–6465.
Merkenschlager M., Nora E.P. (2016) CTCF and cohesin in genome folding and transcriptional gene regulation. Annu. Rev. Genomics Hum. Genet. 17, 17–43.
Rowley M.J., Corces V.G. (2018) Organizational principles of 3D genome architecture. Nat. Rev. Genet. 19, 789–800.
Fudenberg G., Imakaev M., Lu C., Goloborodko A., Abdennur N., Mirny L.A. (2016) Formation of chromosomal domains by loop extrusion. Cell Rep. 15, 2038–2049.
Hansen A.S. (2020) CTCF as a boundary factor for cohesin-mediated loop extrusion: evidence for a multi-step mechanism. Nucleus. 11, 132–148.
Banigan E.J., Mirny L.A. (2020) Loop extrusion: theory meets single-molecule experiments. Curr. Opin. Cell. Biol. 64, 124–138.
Rhodes J.D.P., Feldmann A., Hernandez-Rodriguez B., Diaz N., Brown J.M., Fursova N.A., Blackledge N.P., Prathapan P., Dobrinic P., Huseyin M.K., Szczurek A., Kruse K., Nasmyth K.A., Buckle V.J., Vaquerizas J.M., Klose R.J. (2020) Cohesin disrupts polycomb-dependent chromosome interactions in embryonic stem cells. Cell. Rep. 30, 820–835. e810.
Matthews N.E., White R. (2019) Chromatin architecture in the fly: living without CTCF/cohesin loop extrusion?: alternating chromatin states provide a basis for domain architecture in Drosophila. Bioessays. 41, e1900048.
Ernst J., Kheradpour P., Mikkelsen T.S., Shoresh N., Ward L.D., Epstein C.B., Zhang X., Wang L., Issner R., Coyne M., Ku M., Durham T., Kellis M., Bernstein B.E. (2011) Mapping and analysis of chromatin state dynamics in nine human cell types. Nature. 473, 43–49.
Lupianez D.G., Kraft K., Heinrich V., Krawitz P., Brancati F., Klopocki E., Horn D., Kayserili H., Opitz J.M., Laxova R., Santos-Simarro F., Gilbert-Dussardier B., Wittler L., Borschiwer M., Haas S.A., Osterwalder M., Franke M., Timmermann B., Hecht J., Spielmann M., Visel A., Mundlos S. (2015) Disruptions of topological chromatin domains cause pathogenic rewiring of gene-enhancer interactions. Cell. 161, 1012–1025.
Vorobyeva N.E., Mazina M.U., Golovnin A.K., Kopytova D.V., Gurskiy D.Y., Nabirochkina E.N., Georgieva S.G., Georgiev P.G., Krasnov A.N. (2013) Insulator protein Su(Hw) recruits SAGA and Brahma complexes and constitutes part of origin recognition complex-binding sites in the Drosophila genome. Nucl. Acids Res. 41, 5717–5730.
Мазина М.Ю., Воробьева Н.Е., Краснов А.Н. (2013) Способность Su(Hw) создавать платформу для формирования ориджинов репликации не зависит от типа окружающего хроматина. Цитология. 55, 218–224.
Воробьева Н.Е., Мазина М.Ю. (2019) Функции инсуляторов в контексте современных полногеномных исследований. Генетика. 55(2), 146–155.
Gurudatta B.V., Yang J., Van Bortle K., Donlin-Asp P.G., Corces V.G. (2013) Dynamic changes in the genomic localization of DNA replication-related element binding factor during the cell cycle. Cell Cycle. 12, 1605–1615.
Brand A.H., Breeden L., Abraham J., Sternglanz R., Nasmyth K. (1985) Characterization of a “silencer” in yeast: a DNA sequence with properties opposite to those of a transcriptional enhancer. Cell. 41, 41–48.
Laurenson P., Rine J. (1992) Silencers, silencing, and heritable transcriptional states. Microbiol. Rev. 56, 543–560.
Kamakaka R.T. (1997) Silencers and locus control regions: opposite sides of the same coin. Trends Biochem. Sci. 22, 124–128.
Pirrotta V., Gross D.S. (2005) Epigenetic silencing mechanisms in budding yeast and fruit fly: different paths, same destinations. Mol. Cell. 18, 395–398.
Pirrotta V. (2017) Of giraffes’ necks and the inheritance of chromatin states. Nat. Genet. 49, 821–823.
Schwartz Y.B., Pirrotta V. (2007) Polycomb silencing mechanisms and the management of genomic programmes. Nat. Rev. Genet. 8, 9–22.
Ringrose L., Paro R. (2007) Polycomb/Trithorax response elements and epigenetic memory of cell identity. Development. 134, 223–232.
Bracken A.P., Helin K. (2009) Polycomb group proteins: navigators of lineage pathways led astray in cancer. Nat. Rev. Cancer. 9, 773–784.
Groner A.C., Meylan S., Ciuffi A., Zangger N., Ambrosini G., Denervaud N., Bucher P., Trono D. (2010) KRAB-zinc finger proteins and KAP1 can mediate long-range transcriptional repression through heterochromatin spreading. PLoS Genet. 6, e1000869.
White J.H., Fernandes I., Mader S., Yang X.J. (2004) Corepressor recruitment by agonist-bound nuclear receptors. Vitam. Horm. 68, 123–143.
Emmett M.J., Lazar M.A. (2019) Integrative regulation of physiology by histone deacetylase 3. Nat. Rev. Mol. Cell. Biol. 20, 102–115.
Chanda D., Xie Y.B., Choi H.S. (2010) Transcriptional corepressor SHP recruits SIRT1 histone deacetylase to inhibit LRH-1 transactivation. Nucl. Acids Res. 38, 4607–4619.
Reynolds N., O’Shaughnessy A., Hendrich B. (2013) Transcriptional repressors: multifaceted regulators of gene expression. Development. 140, 505–512.
Celniker S.E., Dillon L.A., Gerstein M.B., Gunsalus K.C., Henikoff S., Karpen G.H., Kellis M., Lai E.C., Lieb J.D., MacAlpine D.M., Micklem G., Piano F., Snyder M., Stein L., White K.P., Waterston R.H. (2009) Unlocking the secrets of the genome. Nature. 459, 927–930.
modENCODE_Consortium, Roy S., Ernst J., Kharchenko P.V., Kheradpour P., Negre N., Eaton M.L., Landolin J.M., Bristow C.A., Ma L., Lin M.F., Washietl S., Arshinoff B.I., Ay F., Meyer P.E., Robine N., Washington N.L., Di Stefano L., Berezikov E., Brown C.D., Candeias R., Carlson J.W., Carr A., Jungreis I., Marbach D., Sealfon R., Tolstorukov M.Y., Will S., Alekseyenko A.A., Artieri C., Booth B.W., Brooks A.N., Dai Q., Davis C.A., Duff M.O., Feng X., Gorchakov A.A., Gu T., Henikoff J.G., Kapranov P., Li R., MacAlpine H.K., Malone J., Minoda A., Nordman J., Okamura K., Perry M., Powell S.K., Riddle N.C., Sakai A., Samsonova A., Sandler J.E., Schwartz Y.B., Sher N., Spokony R., Sturgill D., van Baren M., Wan K.H., Yang L., Yu C., Feingold E., Good P., Guyer M., Lowdon R., Ahmad K., Andrews J., Berger B., Brenner S.E., Brent M.R., Cherbas L., Elgin S.C., Gingeras T.R., Grossman R., Hoskins R.A., Kauf-man T.C., Kent W., Kuroda M.I., Orr-Weaver T., Perrimon N., Pirrotta V., Posakony J.W., Ren B., Russell S., Cherbas P., Graveley B.R., Lewis S., Micklem G., Oliver B., Park P.J., Celniker S.E., Henikoff S., Karpen G.H., Lai E.C., MacAlpine D.M., Stein L.D., White K.P., Kellis M. (2010) Identification of functional elements and regulatory circuits by Drosophila modENCODE. Science. 330, 1787–1797.
Hoffman M.M., Ernst J., Wilder S.P., Kundaje A., Harris R.S., Libbrecht M., Giardine B., Ellenbogen P.M., Bilmes J.A., Birney E., Hardison R.C., Dunham I., Kellis M., Noble W.S. (2013) Integrative annotation of chromatin elements from ENCODE data. Nucl. Acids Res. 41, 827–841.
White M.A. (2015) Understanding how cis-regulatory function is encoded in DNA sequence using massively parallel reporter assays and designed sequences. Genomics. 106, 165–170.
Kleinjan D.J., Coutinho P. (2009) Cis-ruption mechanisms: disruption of cis-regulatory control as a cause of human genetic disease. Brief Funct. Genomic Proteomic. 8, 317–332.
Diederichs S., Bartsch L., Berkmann J.C., Frose K., Heitmann J., Hoppe C., Iggena D., Jazmati D., Karschnia P., Linsenmeier M., Maulhardt T., Mohrmann L., Morstein J., Paffenholz S.V., Ropenack P., Ruckert T., Sandig L., Schell M., Steinmann A., Voss G., Wasmuth J., Weinberger M.E., Wullenkord R. (2016) The dark matter of the cancer genome: aberrations in regulatory elements, untranslated regions, splice sites, non-coding RNA and synonymous mutations. EMBO Mol. Med. 8, 442–457.
West A.G., Fraser P. (2005) Remote control of gene transcription. Hum. Mol. Genet. 14(1), R101–111.
Meier K., Brehm A. (2014) Chromatin regulation: how complex does it get? Epigenetics. 9, 1485–1495.
Ernst J., Kellis M. (2010) Discovery and characterization of chromatin states for systematic annotation of the human genome. Nat. Biotechnol. 28, 817–825.
Filion G.J., van Bemmel J.G., Braunschweig U., Talhout W., Kind J., Ward L.D., Brugman W., de Castro I.J., Kerkhoven R.M., Bussemaker H.J., van Steensel B. (2010) Systematic protein location mapping reveals five principal chromatin types in Drosophila cells. Cell. 143, 212–224.
Kharchenko P.V., Alekseyenko A.A., Schwartz Y.B., Minoda A., Riddle N.C., Ernst J., Sabo P.J., Larschan E., Gorchakov A.A., Gu T., Linder-Basso D., Plachetka A., Shanower G., Tolstorukov M.Y., Luquette L.J., Xi R., Jung Y.L., Park R.W., Bishop E.P., Canfield T.K., Sandstrom R., Thurman R.E., MacAlpine D.M., Stamatoyannopoulos J.A., Kellis M., Elgin S.C., Kuroda M.I., Pirrotta V., Karpen G.H., Park P.J. (2011) Comprehensive analysis of the chromatin landscape in Drosophila melanogaster. Nature. 471, 480–485.
Ho J.W., Jung Y.L., Liu T., Alver B.H., Lee S., Ikegami K., Sohn K.A., Minoda A., Tolstorukov M.Y., Appert A., Parker S.C., Gu T., Kundaje A., Riddle N.C., Bishop E., Egelhofer T.A., Hu S.S., Alekseyenko A.A., Rechtsteiner A., Asker D., Belsky J.A., Bowman S.K., Chen Q.B., Chen R.A., Day D.S., Dong Y., Dose A.C., Duan X., Epstein C.B., Ercan S., Feingold E.A., Ferrari F., Garrigues J.M., Gehlenborg N., Good P.J., Haseley P., He D., Herrmann M., Hoffman M.M., Jeffers T.E., Kharchenko P.V., Kolasinska-Zwierz P., Kotwaliwale C.V., Kumar N., Langley S.A., Larschan E.N., Latorre I., Libbrecht M.W., Lin X., Park R., Pazin M.J., Pham H.N., Plachetka A., Qin B., Schwartz Y.B., Shoresh N., Stempor P., Vielle A., Wang C., Whittle C.M., Xue H., Kingston R.E., Kim J.H., Bernstein B.E., Dernburg A.F., Pirrotta V., Kuroda M.I., Noble W.S., Tullius T.D., Kellis M., MacAlpine D.M., Strome S., Elgin S.C., Liu X.S., Lieb J.D., Ahringer J., Karpen G.H., Park P.J. (2014) Comparative analysis of metazoan chromatin organization. Nature. 512, 449–452.
Yue F., Cheng Y., Breschi A., Vierstra J., Wu W., Ryba T., Sandstrom R., Ma Z., Davis C., Pope B.D., Shen Y., Pervouchine D.D., Djebali S., Thurman R.E., Kaul R., Rynes E., Kirilusha A., Marinov G.K., Williams B.A., Trout D., Amrhein H., Fisher-Aylor K., Antoshechkin I., DeSalvo G., See L.H., Fastuca M., Drenkow J., Zaleski C., Dobin A., Prieto P., Lagarde J., Bussotti G., Tanzer A., Denas O., Li K., Bender M.A., Zhang M., Byron R., Groudine M.T., McCleary D., Pham L., Ye Z., Kuan S., Edsall L., Wu Y.C., Rasmussen M.D., Bansal M.S., Kellis M., Keller C.A., Morrissey C.S., Mishra T., Jain D., Dogan N., Harris R.S., Cayting P., Kawli T., Boyle A.P., Euskirchen G., Kundaje A., Lin S., Lin Y., Jansen C., Malladi V.S., Cline M.S., Erickson D.T., Kirkup V.M., Learned K., Sloan C.A., Rosenbloom K.R., Lacerda de Sousa B., Beal K., Pignatelli M., Flicek P., Lian J., Kahveci T., Lee D., Kent W.J., Ramalho Santos M., Herrero J., Notredame C., Johnson A., Vong S., Lee K., Bates D., Neri F., Diegel M., Canfield T., Sabo P.J., Wilken M.S., Reh T.A., Giste E., Shafer A., Kutyavin T., Haugen E., Dunn D., Reynolds A.P., Neph S., Humbert R., Hansen R.S., De Bruijn M., Selleri L., Rudensky A., Josefowicz S., Samstein R., Eichler E.E., Orkin S.H., Levasseur D., Papayannopoulou T., Chang K.H., Skoultchi A., Gosh S., Disteche C., Treuting P., Wang Y., Weiss M.J., Blobel G.A., Cao X., Zhong S., Wang T., Good P.J., Lowdon R.F., Adams L.B., Zhou X.Q., Pazin M.J., Feingold E.A., Wold B., Taylor J., Mortazavi A., Weissman S.M., Stamatoyannopoulos J.A., Snyder M.P., Guigo R., Gingeras T.R., Gilbert D.M., Hardison R.C., Beer M.A., Ren B. (2014) A comparative encyclopedia of DNA elements in the mouse genome. Nature. 515, 355–364.
Kundaje A., Meuleman W., Ernst J., Bilenky M., Yen A., Heravi-Moussavi A., Kheradpour P., Zhang Z., Wang J., Ziller M.J., Amin V., Whitaker J.W., Schultz M.D., Ward L.D., Sarkar A., Quon G., Sandstrom R.S., Eaton M.L., Wu Y.C., Pfenning A.R., Wang X., Claussnitzer M., Liu Y., Coarfa C., Harris R.A., Shoresh N., Epstein C.B., Gjoneska E., Leung D., Xie W., Hawkins R.D., Lister R., Hong C., Gascard P., Mungall A.J., Moore R., Chuah E., Tam A., Canfield T.K., Hansen R.S., Kaul R., Sabo P.J., Bansal M.S., Carles A., Dixon J.R., Farh K.H., Feizi S., Karlic R., Kim A.R., Kulkarni A., Li D., Lowdon R., Elliott G., Mercer T.R., Neph S.J., Onuchic V., Polak P., Rajagopal N., Ray P., Sallari R.C., Siebenthall K.T., Sinnott-Armstrong N.A., Stevens M., Thurman R.E., Wu J., Zhang B., Zhou X., Beaudet A.E., Boyer L.A., De Jager P.L., Farnham P.J., Fisher S.J., Haussler D., Jones S.J., Li W., Marra M.A., McManus M.T., Sunyaev S., Thomson J.A., Tlsty T.D., Tsai L.H., Wang W., Waterland R.A., Zhang M.Q., Chadwick L.H., Bernstein B.E., Costello J.F., Ecker J.R., Hirst M., Meissner A., Milosavljevic A., Ren B., Stamatoyannopoulos J.A., Wang T., Kellis M. (2015) Integrative analysis of 111 reference human epigenomes. Nature. 518, 317–330.
Стегний B.Н. (2006) Эволюционное значение архитектоники хромосом как формы эпигенетического контроля онто- и филогенеза эукариот. Генетика. 42(9), 1215–1224.
Гаврилов А.А., Разин С.В. (2015) Компартментализация клеточного ядра и пространственная организация генома. Молекуляр. биология. 49(1), 21–39.
Zorn C., Cremer C., Cremer T., Zimmer J. (1979) Unscheduled DNA synthesis after partial UV irradiation of the cell nucleus. Distribution in interphase and metaphase. Exp. Cell. Res. 124, 111–119.
Cremer T., Cremer C., Schneider T., Baumann H., Hens L., Kirsch-Volders M. (1982) Analysis of chromosome positions in the interphase nucleus of Chinese hamster cells by laser-UV-microirradiation experiments. Hum. Genet. 62, 201–209.
Manuelidis L. (1985) Individual interphase chromosome domains revealed by in situ hybridization. Hum. Genet. 71, 288–293.
Croft J.A., Bridger J.M., Boyle S., Perry P., Teague P., Bickmore W.A. (1999) Differences in the localization and morphology of chromosomes in the human nucleus. J. Cell. Biol. 145, 1119–1131.
Cremer T., Cremer C. (2001) Chromosome territories, nuclear architecture and gene regulation in mammalian cells. Nat. Rev. Genet. 2, 292–301.
Cremer M., Kupper K., Wagler B., Wizelman L., von Hase J., Weiland Y., Kreja L., Diebold J., Speicher M.R., Cremer T. (2003) Inheritance of gene density-related higher order chromatin arrangements in normal and tumor cell nuclei. J. Cell. Biol. 162, 809–820.
Kupper K., Kolbl A., Biener D., Dittrich S., von Hase J., Thormeyer T., Fiegler H., Carter N.P., Speicher M.R., Cremer T., Cremer M. (2007) Radial chromatin positioning is shaped by local gene density, not by gene expression. Chromosoma. 116, 285–306.
Tanabe H., Muller S., Neusser M., von Hase J., Calcagno E., Cremer M., Solovei I., Cremer C., Cremer T. (2002) Evolutionary conservation of chromosome territory arrangements in cell nuclei from higher primates. Proc. Natl. Acad. Sci. USA. 99, 4424–4429.
Mayer R., Brero A., von Hase J., Schroeder T., Cremer T., Dietzel S. (2005) Common themes and cell type specific variations of higher order chromatin arrangements in the mouse. BMC Cell Biol. 6, 44.
Goetze S., Mateos-Langerak J., Gierman H.J., de Leeuw W., Giromus O., Indemans M.H., Koster J., Ondrej V., Versteeg R., van Driel R. (2007) The three-dimensional structure of human interphase chromosomes is related to the transcriptome map. Mol. Cell. Biol. 27, 4475–4487.
Grasser F., Neusser M., Fiegler H., Thormeyer T., Cremer M., Carter N.P., Cremer T., Muller S. (2008) Replication-timing-correlated spatial chromatin arrangements in cancer and in primate interphase nuclei. J. Cell. Sci. 121, 1876–1886.
Andrulis E.D., Neiman A.M., Zappulla D.C., Sternglanz R. (1998) Perinuclear localization of chromatin facilitates transcriptional silencing. Nature. 394, 592–595.
Kosak S.T., Skok J.A., Medina K.L., Riblet R., Le Beau M.M., Fisher A.G., Singh H. (2002) Subnuclear compartmentalization of immunoglobulin loci during lymphocyte development. Science. 296, 158–162.
Kind J., van Steensel B. (2010) Genome-nuclear lamina interactions and gene regulation. Curr. Opin. Cell. Biol. 22, 320–325.
Briand N., Collas P. (2020) Lamina-associated domains: peripheral matters and internal affairs. Genome Biol. 21, 85.
Guelen L., Pagie L., Brasset E., Meuleman W., Faza M.B., Talhout W., Eussen B.H., de Klein A., Wessels L., de Laat W., van Steensel B. (2008) Domain organization of human chromosomes revealed by mapping of nuclear lamina interactions. Nature. 453, 948–951.
van Steensel B., Belmont A.S. (2017) Lamina-associated domains: links with chromosome architecture, heterochromatin, and gene repression. Cell. 169, 780–791.
Lochs S.J.A., Kefalopoulou S., Kind J. (2019) Lamina associated domains and gene regulation in development and cancer. Cells. 8(3), e271.
Harr J.C., Luperchio T.R., Wong X., Cohen E., Wheelan S.J., Reddy K.L. (2015) Directed targeting of chromatin to the nuclear lamina is mediated by chromatin state and A-type lamins. J. Cell. Biol. 208, 33–52.
Zullo J.M., Demarco I.A., Pique-Regi R., Gaffney D.J., Epstein C.B., Spooner C.J., Luperchio T.R., Bernstein B.E., Pritchard J.K., Reddy K.L., Singh H. (2012) DNA sequence-dependent compartmentalization and silencing of chromatin at the nuclear lamina. Cell. 149, 1474–1487.
Boyle S., Gilchrist S., Bridger J.M., Mahy N.L., Ellis J.A., Bickmore W.A. (2001) The spatial organization of human chromosomes within the nuclei of normal and emerin-mutant cells. Hum. Mol. Genet. 10, 211–219.
Demmerle J., Koch A.J., Holaska J.M. (2012) The nuclear envelope protein emerin binds directly to histone deacetylase 3 (HDAC3) and activates HDAC3 activity. J. Biol. Chem. 287, 22080–22088.
Peric-Hupkes D., Meuleman W., Pagie L., Bruggeman S.W., Solovei I., Brugman W., Graf S., Flicek P., Kerkhoven R.M., van Lohuizen M., Reinders M., Wessels L., van Steensel B. (2010) Molecular maps of the reorganization of genome-nuclear lamina interactions during differentiation. Mol. Cell. 38, 603–613.
Therizols P., Illingworth R.S., Courilleau C., Boyle S., Wood A.J., Bickmore W.A. (2014) Chromatin decondensation is sufficient to alter nuclear organization in embryonic stem cells. Science. 346, 1238–1242.
Brueckner L., Zhao P.A., van Schaik T., Leemans C., Sima J., Peric-Hupkes D., Gilbert D.M., van Steensel B. (2020) Local rewiring of genome-nuclear lamina interactions by transcription. EMBO J. 39, e103159.
Лавров С.А., Шацких А.С., Кибанов М.В., Гвоздев В.А. (2013) Транскрипционная инактивация генов при эффекте положения у D. melanogaster коррелирует на уровне отдельных клеток с их перемещением в гетерохроматиновый компартмент ядра. Молекуляр. биология. 47(2), 1–6.
Meuleman W., Peric-Hupkes D., Kind J., Beaudry J.B., Pagie L., Kellis M., Reinders M., Wessels L., van Steensel B. (2013) Constitutive nuclear lamina-genome interactions are highly conserved and associated with A/T-rich sequence. Genome Res. 23, 270–280.
Nemeth A., Conesa A., Santoyo-Lopez J., Medina I., Montaner D., Peterfia B., Solovei I., Cremer T., Dopazo J., Langst G. (2010) Initial genomics of the human nucleolus. PLoS Genet. 6, e1000889.
Bizhanova A., Kaufman P.D. (2021) Close to the edge: heterochromatin at the nucleolar and nuclear peripheries. Biochim. Biophys. Acta Gene Regul. Mech. 1864, 194666.
van Koningsbruggen S., Gierlinski M., Schofield P., Martin D., Barton G.J., Ariyurek Y., den Dunnen J.T., Lamond A.I. (2010) High-resolution whole-genome sequencing reveals that specific chromatin domains from most human chromosomes associate with nucleoli. Mol. Biol. Cell. 21, 3735–3748.
Dillinger S., Straub T., Nemeth A. (2017) Nucleolus association of chromosomal domains is largely maintained in cellular senescence despite massive nuclear reorganisation. PLoS One. 12, e0178821.
Wang M., Lemos B. (2019) Ribosomal DNA harbors an evolutionarily conserved clock of biological aging. Genome Res. 29, 325–333.
de Wit E., de Laat W. (2012) A decade of 3C technologies: insights into nuclear organization. Genes Dev. 26, 11–24.
Sati S., Cavalli G. (2017) Chromosome conformation capture technologies and their impact in understanding genome function. Chromosoma. 126, 33–44.
Dixon J.R., Selvaraj S., Yue F., Kim A., Li Y., Shen Y., Hu M., Liu J.S., Ren B. (2012) Topological domains in mammalian genomes identified by analysis of chromatin interactions. Nature. 485, 376–380.
Phillips-Cremins J.E., Sauria M.E., Sanyal A., Gerasimova T.I., Lajoie B.R., Bell J.S., Ong C.T., Hookway T.A., Guo C., Sun Y., Bland M.J., Wagstaff W., Dalton S., McDevitt T.C., Sen R., Dekker J., Taylor J., Corces V.G. (2013) Architectural protein subclasses shape 3D organization of genomes during lineage commitment. Cell. 153, 1281–1295.
Sexton T., Yaffe E., Kenigsberg E., Bantignies F., Leblanc B., Hoichman M., Parrinello H., Tanay A., Cavalli G. (2012) Three-dimensional folding and functional organization principles of the Drosophila genome. Cell. 148, 458–472.
Wang Q., Sun Q., Czajkowsky D.M., Shao Z. (2018) Sub-kb Hi-C in D. melanogaster reveals conserved characteristics of TADs between insect and mammalian cells. Nat. Commun. 9, 188.
Pope B.D., Ryba T., Dileep V., Yue F., Wu W., Denas O., Vera D.L., Wang Y., Hansen R.S., Canfield T.K., Thurman R.E., Cheng Y., Gulsoy G., Dennis J.H., Snyder M.P., Stamatoyannopoulos J.A., Taylor J., Hardison R.C., Kahveci T., Ren B., Gilbert D.M. (2014) Topologically associating domains are stable units of replication-timing regulation. Nature. 515, 402–405.
Zheng X., Hu J., Yue S., Kristiani L., Kim M., Sauria M., Taylor J., Kim Y., Zheng Y. (2018) Lamins organize the global three-dimensional genome from the nuclear periphery. Mol. Cell. 71(5), 802–815. e7.
Buchwalter A., Kaneshiro J.M., Hetzer M.W. (2019) Coaching from the sidelines: the nuclear periphery in genome regulation. Nat. Rev. Genet. 20, 39–50.
Hnisz D., Weintraub A.S., Day D.S., Valton A.L., Bak R.O., Li C.H., Goldmann J., Lajoie B.R., Fan Z.P., Sigova A.A., Reddy J., Borges-Rivera D., Lee T.I., Jaenisch R., Porteus M.H., Dekker J., Young R.A. (2016) Activation of proto-oncogenes by disruption of chromosome neighborhoods. Science. 351, 1454–1458.
Redin C., Brand H., Collins R.L., Kammin T., Mitchell E., Hodge J.C., Hanscom C., Pillalamarri V., Seabra C.M., Abbott M.A., Abdul-Rahman O.A., Aberg E., Adley R., Alcaraz-Estrada S.L., Alkuraya F.S., An Y., Anderson M.A., Antolik C., Anyane-Yeboa K., Atkin J.F., Bartell T., Bernstein J.A., Beyer E., Blumenthal I., Bongers E.M., Brilstra E.H., Brown C.W., Bruggenwirth H.T., Callewaert B., Chiang C., Corning K., Cox H., Cuppen E., Currall B.B., Cushing T., David D., Deardorff M.A., Dheedene A., D’Hoog-he M., de Vries B.B., Earl D.L., Ferguson H.L., Fisher H., FitzPatrick D.R., Gerrol P., Giachino D., Glessner J.T., Gliem T., Grady M., Graham B.H., Griffis C., Gripp K.W., Gropman A.L., Hanson-Kahn A., Harris D.J., Hayden M.A., Hill R., Hochstenbach R., Hoffman J.D., Hopkin R.J., Hubshman M.W., Innes A.M., Irons M., Irving M., Jacobsen J.C., Janssens S., Jewett T., Johnson J.P., Jongmans M.C., Kahler S.G., Koolen D.A., Korzelius J., Kroisel P.M., Lacassie Y., Lawless W., Lemyre E., Leppig K., Levin A.V., Li H., Liao E.C., Lim C., Lose E.J., Lucente D., Macera M.J., Manavalan P., Mandrile G., Marcelis C.L., Margolin L., Mason T., Masser-Frye D., McClellan M.W., Mendoza C.J., Menten B., Middelkamp S., Mikami L.R., Moe E., Mohammed S., Mononen T., Mortenson M.E., Moya G., Nieuwint A.W., Ordulu Z., Parkash S., Pauker S.P., Pereira S., Perrin D., Phelan K., Aguilar R.E., Poddighe P.J., Pregno G., Raskin S., Reis L., Rhead W., Rita D., Renkens I., Roelens F., Ruliera J., Rump P., Schilit S.L., Shaheen R., Sparkes R., Spiegel E., Stevens B., Stone M.R., Tagoe J., Thakuria J.V., van Bon B.W., van de Kamp J., van Der Burgt I., van Essen T., van Ravenswaaij-Arts C.M., van Roosmalen M.J., Vergult S., Volker-Touw C.M., Warburton D.P., Waterman M.J., Wiley S., Wilson A., Yerena-de Vega M.C., Zori R.T., Levy B., Brunner H.G., de Leeuw N., Kloosterman W.P., Thorland E.C., Morton C.C., Gusella J.F., Talkowski M.E. (2017) The genomic landscape of balanced cytogenetic abnormalities associated with human congenital anomalies. Nat. Genet. 49, 36–45.
Kraft K., Magg A., Heinrich V., Riemenschneider C., Schopflin R., Markowski J., Ibrahim D.M., Acuna-Hidalgo R., Despang A., Andrey G., Wittler L., Timmermann B., Vingron M., Mundlos S. (2019) Serial genomic inversions induce tissue-specific architectural stripes, gene misexpression and congenital malformations. Nat. Cell. Biol. 21, 305–310.
Erdel F., Rippe K. (2018) Formation of chromatin subcompartments by phase separation. Biophys. J. 114, 2262–2270.
Strom A.R., Emelyanov A.V., Mir M., Fyodorov D.V., Darzacq X., Karpen G.H. (2017) Phase separation drives heterochromatin domain formation. Nature. 547, 241–245.
Larson A.G., Elnatan D., Keenen M.M., Trnka M.J., Johnston J.B., Burlingame A.L., Agard D.A., Redding S., Narlikar G.J. (2017) Liquid droplet formation by HP1alpha suggests a role for phase separation in heterochromatin. Nature. 547, 236–240.
Razin S.V., Ulianov S.V. (2020) Divide and rule: phase separation in eukaryotic genome functioning. Cells. 9(11), e2480.
Hyman A.A., Weber C.A., Julicher F. (2014) Liquid-liquid phase separation in biology. Annu. Rev. Cell. Dev. Biol. 30, 39–58.
Babokhov M., Hibino K., Itoh Y., Maeshima K. (2020) Local chromatin motion and transcription. J Mol Biol. 432, 694–700.
Tatarakis A., Behrouzi R., Moazed D. (2017) Evolving models of heterochromatin: from foci to liquid droplets. Mol. Cell. 67, 725–727.
Machida S., Takizawa Y., Ishimaru M., Sugita Y., Sekine S., Nakayama J.I., Wolf M., Kurumizaka H. (2018) Structural basis of heterochromatin formation by human HP1. Mol. Cell. 69, 385–397 e388.
Hnisz D., Shrinivas K., Young R.A., Chakraborty A.K., Sharp P.A. (2017) A phase separation model for transcriptional control. Cell. 169, 13–23.
Silveira M.A.D., Bilodeau S. (2018) Defining the transcriptional ecosystem. Mol. Cell. 72, 920–924.
Wang X., Cairns M.J., Yan J. (2019) Super-enhancers in transcriptional regulation and genome organization. Nucl. Acids Res. 47, 11481–11496.
Grosveld F., van Staalduinen J., Stadhouders R. (2021) Transcriptional regulation by (super)enhancers: from discovery to mechanisms. Annu. Rev. Genomics Hum. Genet. 22, 127–146.
Zhang J., Yue W., Zhou Y., Liao M., Chen X., Hua J. (2021) Super enhancers-functional cores under the 3D genome. Cell Prolif. 54, e12970.
Belaghzal H., Borrman T., Stephens A.D., Lafontaine D.L., Venev S.V., Weng Z., Marko J.F., Dekker J. (2021) Liquid chromatin Hi-C characterizes compartment-dependent chromatin interaction dynamics. Nat. Genet. 53, 367–378.
Hahn M., Dambacher S., Schotta G. (2010) Heterochromatin dysregulation in human diseases. J. Appl. Physiol. 109, 232–242.
Robin J.D., Magdinier F. (2017) Higher-order chromatin organization in diseases: from chromosomal position effect to phenotype variegation. Handbook Epigenet. 2nd ed. Acad. Press. Chapter 6, 73–92.
Rabbitts T.H. (1994) Chromosomal translocations in human cancer. Nature. 372, 143–149.
Rabbitts T.H. (1991) Translocations, master genes, and differences between the origins of acute and chronic leukemias. Cell. 67, 641–644.
Korsmeyer S.J. (1992) Chromosomal translocations in lymphoid malignancies reveal novel proto-oncogenes. Annu. Rev. Immunol. 10, 785–807.
Lossos I.S., Akasaka T., Martinez-Climent J.A., Siebert R., Levy R. (2003) The BCL6 gene in B-cell lymphomas with 3q27 translocations is expressed mainly from the rearranged allele irrespective of the partner gene. Leukemia. 17, 1390–1397.
Northcott P.A., Lee C., Zichner T., Stutz A.M., Erkek S., Kawauchi D., Shih D.J., Hovestadt V., Zapatka M., Sturm D., Jones D.T., Kool M., Remke M., Cavalli F.M., Zuyderduyn S., Bader G.D., VandenBerg S., Esparza L.A., Ryzhova M., Wang W., Wittmann A., Stark S., Sieber L., Seker-Cin H., Linke L., Kratochwil F., Jager N., Buchhalter I., Imbusch C.D., Zipprich G., Raeder B., Schmidt S., Diessl N., Wolf S., Wiemann S., Brors B., Lawerenz C., Eils J., Warnatz H.J., Risch T., Yaspo M.L., Weber U.D., Bartholomae C.C., von Kalle C., Turanyi E., Hauser P., Sanden E., Darabi A., Siesjo P., Sterba J., Zitterbart K., Sumerauer D., van Sluis P., Versteeg R., Volckmann R., Koster J., Schuhmann M.U., Ebinger M., Grimes H.L., Robinson G.W., Gajjar A., Mynarek M., von Hoff K., Rutkowski S., Pietsch T., Scheurlen W., Felsberg J., Reifenberger G., Kulozik A.E., von Deimling A., Witt O., Eils R., Gilbertson R.J., Korshunov A., Taylor M.D., Lichter P., Korbel J.O., Wechsler-Reya R.J., Pfister S.M. (2014) Enhancer hijacking activates GFI1 family oncogenes in medulloblastoma. Nature. 511, 428–434.
Kleinjan D.J., van Heyningen V. (1998) Position effect in human genetic disease. Hum. Mol. Genet. 7, 1611–1618.
Kleinjan D.A., van Heyningen V. (2005) Long-range control of gene expression: emerging mechanisms and disruption in disease. Am. J. Hum. Genet. 76, 8–32.
Hannan A.J. (2018) Tandem repeats mediating genetic plasticity in health and disease. Nat. Rev. Genet. 19, 286–298.
Lander E.S., Linton L.M., Birren B., Nusbaum C., Zody M.C., Baldwin J., Devon K., Dewar K., Doyle M., FitzHugh W., Funke R., Gage D., Harris K., Heaford A., Howland J., Kann L., Lehoczky J., LeVine R., McEwan P., McKernan K., Meldrim J., Mesirov J.P., Miranda C., Morris W., Naylor J., Raymond C., Rosetti M., Santos R., Sheridan A., Sougnez C., Stange-Thomann Y., Stojanovic N., Subramanian A., Wyman D., Rogers J., Sulston J., Ainscough R., Beck S., Bentley D., Burton J., Clee C., Carter N., Coulson A., Deadman R., Deloukas P., Dunham A., Dunham I., Durbin R., French L., Grafham D., Gregory S., Hubbard T., Humphray S., Hunt A., Jones M., Lloyd C., McMurray A., Matthews L., Mercer S., Milne S., Mullikin J.C., Mungall A., Plumb R., Ross M., Shownkeen R., Sims S., Waterston R.H., Wilson R.K., Hillier L.W., McPherson J.D., Marra M.A., Mardis E.R., Fulton L.A., Chinwalla A.T., Pepin K.H., Gish W.R., Chissoe S.L., Wendl M.C., Delehaunty K.D., Miner T.L., Delehaunty A., Kramer J.B., Cook L.L., Fulton R.S., Johnson D.L., Minx P.J., Clifton S.W., Hawkins T., Branscomb E., Predki P., Richardson P., Wenning S., Slezak T., Doggett N., Cheng J.F., Olsen A., Lucas S., Elkin C., Uberbacher E., Frazier M., Gibbs R.A., Muzny D.M., Scherer S.E., Bouck J.B., Sodergren E.J., Worley K.C., Rives C.M., Gorrell J.H., Metzker M.L., Naylor S.L., Kucherlapati R.S., Nelson D.L., Weinstock G.M., Sakaki Y., Fujiyama A., Hattori M., Yada T., Toyoda A., Itoh T., Kawagoe C., Watanabe H., Totoki Y., Taylor T., Weissenbach J., Heilig R., Saurin W., Artiguenave F., Brottier P., Bruls T., Pelletier E., Robert C., Wincker P., Smith D.R., Doucette-Stamm L., Rubenfield M., Weinstock K., Lee H.M., Dubois J., Rosenthal A., Platzer M., Nyakatura G., Taudien S., Rump A., Yang H., Yu J., Wang J., Huang G., Gu J., Hood L., Rowen L., Madan A., Qin S., Davis R.W., Federspiel N.A., Abola A.P., Proctor M.J., Myers R.M., Schmutz J., Dickson M., Grimwood J., Cox D.R., Olson M.V., Kaul R., Shimizu N., Kawasaki K., Minoshima S., Evans G.A., Athanasiou M., Schultz R., Roe B.A., Chen F., Pan H., Ramser J., Lehrach H., Reinhardt R., McCombie W.R., de la Bastide M., Dedhia N., Blocker H., Hornischer K., Nordsiek G., Agarwala R., Aravind L., Bailey J.A., Bateman A., Batzoglou S., Birney E., Bork P., Brown D.G., Burge C.B., Cerutti L., Chen H.C., Church D., Clamp M., Copley R.R., Doerks T., Eddy S.R., Eichler E.E., Furey T.S., Galagan J., Gilbert J.G., Harmon C., Hayashizaki Y., Haussler D., Hermjakob H., Hokamp K., Jang W., Johnson L.S., Jones T.A., Kasif S., Kaspryzk A., Kennedy S., Kent W.J., Kitts P., Koonin E.V., Korf I., Kulp D., Lancet D., Lowe T.M., McLysaght A., Mikkelsen T., Moran J.V., Mulder N., Pollara V.J., Ponting C.P., Schuler G., Schultz J., Slater G., Smit A.F., Stupka E., Szustakowki J., Thierry-Mieg D., Thierry-Mieg J., Wagner L., Wallis J., Wheeler R., Williams A., Wolf Y.I., Wolfe K.H., Yang S.P., Yeh R.F., Collins F., Guyer M.S., Peterson J., Felsenfeld A., Wetterstrand K.A., Patrinos A., Morgan M.J., de Jong P., Catanese J.J., Osoegawa K., Shizuya H., Choi S., Chen Y.J. (2001) Initial sequencing and analysis of the human genome. Nature. 409, 860–921.
de Koning A.P., Gu W., Castoe T.A., Batzer M.A., Pollock D.D. (2011) Repetitive elements may comprise over two-thirds of the human genome. PLoS Genet. 7, e1002384.
Schaap M., Lemmers R.J., Maassen R., van der Vliet P.J., Hoogerheide L.F., van Dijk H.K., Basturk N., de Knijff P., van der Maarel S.M. (2013) Genome-wide analysis of macrosatellite repeat copy number variation in worldwide populations: evidence for differences and commonalities in size distributions and size restrictions. BMC Genomics. 14, 143.
Richard G.F., Kerrest A., Dujon B. (2008) Comparative genomics and molecular dynamics of DNA repeats in eukaryotes. Microbiol. Mol. Biol. Rev. 72, 686–727.
Vinces M.D., Legendre M., Caldara M., Hagihara M., Verstrepen K.J. (2009) Unstable tandem repeats in promoters confer transcriptional evolvability. Science. 324, 1213–1216.
Sawaya S., Bagshaw A., Buschiazzo E., Kumar P., Chowdhury S., Black M.A., Gemmell N. (2013) Microsatellite tandem repeats are abundant in human promoters and are associated with regulatory elements. PLoS One. 8, e54710.
Bilgin Sonay T., Carvalho T., Robinson M.D., Greminger M.P., Krutzen M., Comas D., Highnam G., Mittelman D., Sharp A., Marques-Bonet T., Wagner A. (2015) Tandem repeat variation in human and great ape populations and its impact on gene expression divergence. Genome Res. 25, 1591–1599.
Sawaya S.M., Lennon D., Buschiazzo E., Gemmell N., Minin V.N. (2012) Measuring microsatellite conservation in mammalian evolution with a phylogenetic birth-death model. Genome Biol. Evol. 4, 636–647.
Sawaya S.M., Bagshaw A.T., Buschiazzo E., Gemmell N.J. (2012) Promoter microsatellites as modulators of human gene expression. Adv. Exp. Med. Biol. 769, 41–54.
Gymrek M., Willems T., Guilmatre A., Zeng H., Markus B., Georgiev S., Daly M.J., Price A.L., Pritchard J.K., Sharp A.J., Erlich Y. (2016) Abundant contribution of short tandem repeats to gene expression variation in humans. Nat. Genet. 48, 22–29.
Rockman M.V., Wray G.A. (2002) Abundant raw material for cis-regulatory evolution in humans. Mol. Biol. Evol. 19, 1991–2004.
Gemayel R., Vinces M.D., Legendre M., Verstrepen K.J. (2010) Variable tandem repeats accelerate evolution of coding and regulatory sequences. Annu. Rev. Genet. 44, 445–477.
Jansen A., Gemayel R., Verstrepen K.J. (2012) Unstable microsatellite repeats facilitate rapid evolution of coding and regulatory sequences. Genome Dyn. 7, 108–125.
Dorer D.R., Henikoff S. (1994) Expansions of transgene repeats cause heterochromatin formation and gene silencing in Drosophila. Cell. 77, 993–1002.
Garrick D., Fiering S., Martin D.I., Whitelaw E. (1998) Repeat-induced gene silencing in mammals. Nat. Genet. 18, 56–59.
Brahmachary M., Guilmatre A., Quilez J., Hasson D., Borel C., Warburton P., Sharp A.J. (2014) Digital genotyping of macrosatellites and multicopy genes reveals novel biological functions associated with copy number variation of large tandem repeats. PLoS Genet. 10, e1004418.
Poeta L., Drongitis D., Verrillo L., Miano M.G. (2020) DNA hypermethylation and unstable repeat diseases: a paradigm of transcriptional silencing to decipher the basis of pathogenic mechanisms. Genes (Basel). 11(6), e684.
Willems T., Gymrek M., Highnam G., Mittelman D., Erlich Y. (2014) The landscape of human STR variation. Genome Res. 24, 1894–1904.
Li Y.C., Korol A.B., Fahima T., Beiles A., Nevo E. (2002) Microsatellites: genomic distribution, putative functions and mutational mechanisms: a review. Mol. Ecol. 11, 2453–2465.
Campbell C.D., Chong J.X., Malig M., Ko A., Dumont B.L., Han L., Vives L., O’Roak B.J., Sudmant P.H., Shendure J., Abney M., Ober C., Eich-ler E.E. (2012) Estimating the human mutation rate using autozygosity in a founder population. Nat. Genet. 44, 1277–1281.
Kondrashov A.S. (2003) Direct estimates of human per nucleotide mutation rates at 20 loci causing Mendelian diseases. Hum. Mutat. 21, 12–27.
Sun J.X., Helgason A., Masson G., Ebenesersdottir S.S., Li H., Mallick S., Gnerre S., Patterson N., Kong A., Reich D., Stefansson K. (2012) A direct characterization of human mutation based on microsatellites. Nat. Genet. 44, 1161–1165.
Weber J.L., Wong C. (1993) Mutation of human short tandem repeats. Hum. Mol. Genet. 2, 1123–1128.
Brinkmann B., Klintschar M., Neuhuber F., Huhne J., Rolf B. (1998) Mutation rate in human microsatellites: influence of the structure and length of the tandem repeat. Am. J. Hum. Genet. 62, 1408–1415.
Mitra I., Huang B., Mousavi N., Ma N., Lamkin M., Yanicky R., Shleizer-Burko S., Lohmueller K.E., Gymrek M. (2021) Patterns of de novo tandem repeat mutations and their role in autism. Nature. 589, 246–250.
Annear D.J., Vandeweyer G., Elinck E., Sanchis-Juan A., French C.E., Raymond L., Kooy R.F. (2021) Abundancy of polymorphic CGG repeats in the human genome suggest a broad involvement in neurological disease. Sci. Rep. 11, 2515.
Rijkers T., Deidda G., van Koningsbruggen S., van Geel M., Lemmers R.J., van Deutekom J.C., Figlewicz D., Hewitt J.E., Padberg G.W., Frants R.R., van der Maarel S.M. (2004) FRG2, an FSHD candidate gene, is transcriptionally upregulated in differentiating primary myoblast cultures of FSHD patients. J. Med. Genet. 41, 826–836.
Wijmenga C., Hewitt J.E., Sandkuijl L.A., Clark L.N., Wright T.J., Dauwerse H.G., Gruter A.M., Hofker M.H., Moerer P., Williamson R., van Ommen G.B., Padberg G.W., Frants R.R. (1992) Chromosome 4q DNA rearrangements associated with facioscapulohumeral muscular dystrophy. Nat. Genet. 2, 26–30.
van Deutekom J.C., Wijmenga C., van Tienhoven E.A., Gruter A.M., Hewitt J.E., Padberg G.W., van Ommen G.J., Hofker M.H., Frants R.R. (1993) FSHD associated DNA rearrangements are due to deletions of integral copies of a 3.2 kb tandemly repeated unit. Hum. Mol. Genet. 2, 2037–2042.
Hewitt J.E., Lyle R., Clark L.N., Valleley E.M., Wright T.J., Wijmenga C., van Deutekom J.C., Francis F., Sharpe P.T., Hofker M., Frants R.R., Williamson R. (1994) Analysis of the tandem repeat locus D4Z4 associated with facioscapulohumeral muscular dystrophy. Hum. Mol. Genet. 3, 1287–1295.
Gabellini D., Green M.R., Tupler R. (2002) Inappropriate gene activation in FSHD: a repressor complex binds a chromosomal repeat deleted in dystrophic muscle. Cell. 110, 339–348.
Gabellini D., D’Antona G., Moggio M., Prelle A., Zecca C., Adami R., Angeletti B., Ciscato P., Pellegrino M.A., Bottinelli R., Green M.R., Tupler R. (2006) Facioscapulohumeral muscular dystrophy in mice overexpressing FRG1. Nature. 439, 973–977.
Xynos A., Neguembor M.V., Caccia R., Licastro D., Nonis A., Di Serio C., Stupka E., Gabellini D. (2013) Overexpression of facioscapulohumeral muscular dystrophy region gene 1 causes primary defects in myogenic stem cells. J. Cell. Sci. 126, 2236–2245.
Hagerman R., Hagerman P. (2013) Advances in clinical and molecular understanding of the FMR1 premutation and fragile X-associated tremor/ataxia syndrome. Lancet Neurol. 12, 786–798.
Kremer E.J., Pritchard M., Lynch M., Yu S., Holman K., Baker E., Warren S.T., Schlessinger D., Sutherland G.R., Richards R.I. (1991) Mapping of DNA instability at the fragile X to a trinucleotide repeat sequence p(CCG)n. Science. 252, 1711–1714.
Verkerk A.J., Pieretti M., Sutcliffe J.S., Fu Y.H., Kuhl D.P., Pizzuti A., Reiner O., Richards S., Victoria M.F., Zhang F.P., Eussen B.E., van Ommen G.B., Blonden L.A.J., Riggins G.J., Chastain J.L., Kunst C.B., Galjaard H., Caskey C.T., Nelson D.L., Oostra B.A., Warren S.T. (1991) Identification of a gene (FMR-1) containing a CGG repeat coincident with a breakpoint cluster region exhibiting length variation in fragile X syndrome. Cell. 65, 905–914.
Hansen R.S., Gartler S.M., Scott C.R., Chen S.H., Laird C.D. (1992) Methylation analysis of CGG sites in the CpG island of the human FMR1 gene. Hum. Mol. Genet. 1, 571–578.
Maurin T., Zongaro S., Bardoni B. (2014) Fragile X syndrome: from molecular pathology to therapy. Neurosci. Biobehav. Rev. 46, Pt 2, 242–255.
Dolskiy A.A., Yarushkin A.A., Grishchenko I.V., Lemskaya N.A., Pindyurin A.V., Boldyreva L.V., Pustylnyak V.O., Yudkin D.V. (2021) miRNA expression and interaction with the 3'UTR of FMR1 in FRAXopathy pathogenesis. Noncoding RNA Res. 6, 1–7.
Chen H.C., Martinez J.P., Zorita E., Meyerhans A., Filion G.J. (2017) Position effects influence HIV latency reversal. Nat. Struct. Mol. Biol. 24, 47–54.
Pierson T.C., Zhou Y., Kieffer T.L., Ruff C.T., Buck C., Siliciano R.F. (2002) Molecular characterization of preintegration latency in human immunodeficiency virus type 1 infection. J. Virol. 76, 8518–8531.
Pang S., Koyanagi Y., Miles S., Wiley C., Vinters H.V., Chen I.S. (1990) High levels of unintegrated HIV-1 DNA in brain tissue of AIDS dementia patients. Nature. 343, 85–89.
Pierson T.C., Kieffer T.L., Ruff C.T., Buck C., Gange S.J., Siliciano R.F. (2002) Intrinsic stability of episomal circles formed during human immunodeficiency virus type 1 replication. J. Virol. 76, 4138–4144.
Kelly J., Beddall M.H., Yu D., Iyer S.R., Marsh J.W., Wu Y. (2008) Human macrophages support persistent transcription from unintegrated HIV-1 DNA. Virology. 372, 300–312.
Mbonye U., Karn J. (2017) The molecular basis for Human Immunodeficiency Virus latency. Annu. Rev. Virol. 4, 261–285.
Vansant G., Bruggemans A., Janssens J., Debyser Z. (2020) Block-and-lock strategies to cure HIV infection. Viruses. 12(1), e84.
Shukla A., Ramirez N.P., D’Orso I. (2020) HIV-1 proviral transcription and latency in the New Era. Viruses. 2(5), e555.
Lindqvist B., Svensson Akusjarvi S., Sonnerborg A., Dimitriou M., Svensson J.P. (2020) Chromatin maturation of the HIV-1 provirus in primary resting CD4+ T cells. PLoS Pathog. 16, e1008264.
Roy S., Delling U., Chen C.H., Rosen C.A., Sonenberg N. (1990) A bulge structure in HIV-1 TAR RNA is required for Tat binding and Tat-mediated trans-activation. Genes Dev. 4, 1365–1373.
Zhu Y., Pe’ery T., Peng J., Ramanathan Y., Marshall N., Marshall T., Amendt B., Mathews M.B., Price D.H. (1997) Transcription elongation factor P-TEFb is required for HIV-1 Tat transactivation in vitro. Genes Dev. 11, 2622–2632.
Ping Y.H., Rana T.M. (2001) DSIF and NELF interact with RNA polymerase II elongation complex and HIV-1 Tat stimulates P-TEFb-mediated phosphorylation of RNA polymerase II and DSIF during transcription elongation. J. Biol. Chem. 276, 12951–12958.
Easley R., Carpio L., Dannenberg L., Choi S., Alani D., Van Duyne R., Guendel I., Klase Z., Agbottah E., Kehn-Hall K., Kashanchi F. (2010) Transcription through the HIV-1 nucleosomes: effects of the PBAF complex in Tat activated transcription. Virology. 405, 322–333.
Ammosova T., Berro R., Jerebtsova M., Jackson A., Charles S., Klase Z., Southerland W., Gordeuk V.R., Kashanchi F., Nekhai S. (2006) Phosphorylation of HIV-1 Tat by CDK2 in HIV-1 transcription. Retrovirology. 3, 78.
West M.J., Lowe A.D., Karn J. (2001) Activation of human immunodeficiency virus transcription in T cells revisited: NF-kappaB p65 stimulates transcriptional elongation. J. Virol. 75, 8524–8537.
Hogan P.G., Chen L., Nardone J., Rao A. (2003) Transcriptional regulation by calcium, calcineurin, and NFAT. Genes Dev. 17, 2205–2232.
Kinoshita S., Su L., Amano M., Timmerman L.A., Kaneshima H., Nolan G.P. (1997) The T cell activation factor NF-ATc positively regulates HIV-1 replication and gene expression in T cells. Immunity. 6, 235–244.
Coull J.J., Romerio F., Sun J.M., Volker J.L., Galvin K.M., Davie J.R., Shi Y., Hansen U., Margolis D.M. (2000) The human factors YY1 and LSF repress the human immunodeficiency virus type 1 long terminal repeat via recruitment of histone deacetylase 1. J. Virol. 74, 6790–6799.
Tyagi M., Karn J. (2007) CBF-1 promotes transcriptional silencing during the establishment of HIV-1 latency. EMBO J. 26, 4985–4995.
He G., Margolis D.M. (2002) Counterregulation of chromatin deacetylation and histone deacetylase occupancy at the integrated promoter of human immunodeficiency virus type 1 (HIV-1) by the HIV-1 repressor YY1 and HIV-1 activator Tat. Mol. Cell. Biol. 22, 2965–2973.
Tyagi M., Pearson R.J., Karn J. (2010) Establishment of HIV latency in primary CD4+ cells is due to epigenetic transcriptional silencing and P-TEFb restriction. J. Virol. 84, 6425–6437.
Debyser Z., Christ F., De Rijck J., Gijsbers R. (2015) Host factors for retroviral integration site selection. Trends Biochem. Sci. 40, 108–116.
Marini B., Kertesz-Farkas A., Ali H., Lucic B., Lisek K., Manganaro L., Pongor S., Luzzati R., Recchia A., Mavilio F., Giacca M., Lusic M. (2015) Nuclear architecture dictates HIV-1 integration site selection. Nature. 521, 227–231.
Lusic M., Siliciano R.F. (2017) Nuclear landscape of HIV-1 infection and integration. Nat. Rev. Microbiol. 15, 69–82.
Gonzalez-Hernandez M.J., Swanson M.D., Contreras-Galindo R., Cookinham S., King S.R., Noel R.J., Jr., Kaplan M.H., Markovitz D.M. (2012) Expression of human endogenous retrovirus type K (HML-2) is activated by the Tat protein of HIV-1. J. Virol. 86, 7790–7805.
Contreras-Galindo R., Kaplan M.H., Contreras-Galindo A.C., Gonzalez-Hernandez M.J., Ferlenghi I., Giusti F., Lorenzo E., Gitlin S.D., Dosik M.H., Yamamura Y., Markovitz D.M. (2012) Characterization of human endogenous retroviral elements in the blood of HIV-1-infected individuals. J. Virol. 86, 262–276.
Contreras-Galindo R., Kaplan M.H., Dube D., Gonzalez-Hernandez M.J., Chan S., Meng F., Dai M., Omenn G.S., Gitlin S.D., Markovitz D.M. (2015) Human endogenous retrovirus type K (HERV-K) particles package and transmit HERV-K-related sequences. J. Virol. 89, 7187–7201.
Geuking M.B., Weber J., Dewannieux M., Gorelik E., Heidmann T., Hengartner H., Zinkernagel R.M., Hangartner L. (2009) Recombination of retrotransposon and exogenous RNA virus results in nonretroviral cDNA integration. Science. 323, 393–396.
Буздин А.А. (2010) Функциональный анализ вставок эндогенных ретровирусов в контексте эволюции генома человека. Биоорган. химия. 36, 38–46.
Birnbaum R.Y., Patwardhan R.P., Kim M.J., Findlay G.M., Martin B., Zhao J., Bell R.J., Smith R.P., Ku A.A., Shendure J., Ahituv N. (2014) Systematic dissection of coding exons at single nucleotide resolution supports an additional role in cell-specific transcriptional regulation. PLoS Genet. 10, e1004592.
Yan C., Chen H., Bai L. (2018) Systematic study of nucleosome-displacing factors in budding yeast. Mol. Cell. 71, 294–305. e294.
Ramani V., Qiu R., Shendure J. (2019) High sensitivity profiling of chromatin structure by MNase-SSP. Cell. Rep. 26, 2465–2476. e2464.
Levo M., Avnit-Sagi T., Lotan-Pompan M., Kalma Y., Weinberger A., Yakhini Z., Segal E. (2017) Systematic investigation of transcription factor activity in the context of chromatin using massively parallel binding and expression assays. Mol. Cell. 65, 604–617. e606.
Hammelman J., Krismer K., Banerjee B., Gifford D.K., Sherwood R.I. (2020) Identification of determinants of differential chromatin accessibility through a massively parallel genome-integrated reporter assay. Genome Res. 30, 1468–1480.
Sobecki M., Souaid C., Boulay J., Guerineau V., Noordermeer D., Crabbe L. (2018) MadID, a versatile approach to map protein-DNA interactions, highlights telomere-nuclear envelope contact sites in human cells. Cell Rep. 25, 2891–2903. e2895.
Шацких А.С., Оленкина О.М., Солодовников А.A., Лавров С.А. (2018) Системы регулируемой экспрессии генов как инструмент исследования гетерохроматинового эффекта положения у Drosophila melanogaster. Биохимия. 83(5), 712–723.
Kotecki M., Reddy P.S., Cochran B.H. (1999) Isolation and characterization of a near-haploid human cell line. Exp. Cell. Res. 252, 273–280.
Higgins A.W., Alkuraya F.S., Bosco A.F., Brown K.K., Bruns G.A., Donovan D.J., Eisenman R., Fan Y., Farra C.G., Ferguson H.L., Gusella J.F., Harris D.J., Herrick S.R., Kelly C., Kim H.G., Kishikawa S., Korf B.R., Kulkarni S., Lally E., Leach N.T., Lemyre E., Lewis J., Ligon A.H., Lu W., Maas R.L., MacDonald M.E., Moore S.D., Peters R.E., Quade B.J., Quintero-Rivera F., Saadi I., Shen Y., Shendure J., Williamson R.E., Morton C.C. (2008) Characterization of apparently balanced chromosomal rearrangements from the developmental genome anatomy project. Am. J. Hum. Genet. 82, 712–722.
Chiang C., Jacobsen J.C., Ernst C., Hanscom C., Heilbut A., Blumenthal I., Mills R.E., Kirby A., Lindgren A.M., Rudiger S.R., McLaughlan C.J., Baw-den C.S., Reid S.J., Faull R.L., Snell R.G., Hall I.M., Shen Y., Ohsumi T.K., Borowsky M.L., Daly M.J., Lee C., Morton C.C., MacDonald M.E., Gusella J.F., Talkowski M.E. (2012) Complex reorganization and predominant non-homologous repair following chromosomal breakage in karyotypically balanced germline rearrangements and transgenic integration. Nat. Genet. 44, 390–397. S391.
Дополнительные материалы
- скачать ESM.docx
- Таблица 1. Модификаторы ЭП у дрозофилы, человека и мыши, связанные с ними сигнальные пути и заболевания человека.
Таблица 2. Заболевания, связанные с ЭП генов человека, возникающего в результате хромосомных перестроек.
Таблица 3. Заболевания человека, развивающиеся в результате ЭП, обусловленном экспансией микросателлитов (согласно [82] с модификациями).
СПИСОК ЛИТЕРАТУРЫ ПРИЛОЖЕНИЯ
Инструменты
Молекулярная биология


