Молекулярная биология, 2022, T. 56, № 3, стр. 521-527
Новый метод оценки влияния модифицированных трифосфатов дезоксинуклеозидов на изменение состава комбинаторных библиотек для проведения SELEX
С. А. Лапа a, *, О. С. Антипова a, А. В. Чудинов a
a Институт молекулярной биологии им. В.А. Энгельгардта Российской академии наук
119991 Москва, Россия
* E-mail: lapa@biochip.ru
Поступила в редакцию 21.12.2021
После доработки 13.01.2022
Принята к публикации 13.01.2022
- EDN: CZQYHQ
- DOI: 10.31857/S0026898422030090
Аннотация
Предложен новый способ оценки субстратной эффективности дезоксинуклеозидтрифосфатов (dNTP), содержащих функциональные группы, для селекции модифицированных аптамеров (mod-SELEX). Метод предполагает проведение трех последовательных раундов ПЦР с комбинаторной библиотекой и модифицированным dNTP – кандидатом для проведения mod-SELEX. Вывод о применимости конкретного производного dNTP основан на характере изменения амплификационной кривой в течение трех раундов ПЦР в режиме реального времени и не требует проведения раундов селекции аптамеров. Если в процессе амплификации библиотека вырождается (становится менее представительной), значит, конкретная модификация dNTP не может использоваться с выбранной полимеразой и другими выбранными условиями амплификации библиотеки, поскольку приводит к конкурентной амплификации. В случае, когда характер амплификационной кривой не меняется, делается вывод, что используемый модифицированный трифосфат дезоксинуклеозида не влияет на распределение олигонуклеотидов с различными последовательностями в составе библиотеки, т.е. не приводит к изменению ее состава с точки зрения применяемого метода детекции. Именно такие производные могут использоваться в выбранных условиях проведения селекции аптамеров. Метод применим для быстрой оценки субстратной пригодности модифицированных dNTP для проведения mod-SELEX и будет полезен при отборе аптамеров для клинической диагностики и научных исследований.
ВВЕДЕНИЕ
Аптамеры – нуклеиновые аналоги моноклональных антител, обладающие рядом важных преимуществ. В настоящий момент аптамеры применяются как самостоятельные диагностические инструменты для выявления заболеваний различной этиологии, так и в качестве средств терапии. В последовательность аптамеров для увеличения их сродства к мишеням вводят модификации. Отбор модифицированных аптамеров к белковым мишеням (mod-SELEX) – сложный и трудоемкий процесс [1, 2]. Из-за низкой совместимости модифицированных dNTP с используемой ДНК-полимеразой ПЦР часто либо ингибируется, либо происходит конкурентная амплификация с обогащением исходной ДНК-библиотеки не аптамерами с высоким сродством к мишени, а легко амплифицируемыми последовательностями (конкурентная амплификация) [3, 4].
В настоящее время для контроля обогащения библиотек применяется ряд методических подходов: секвенирование методом NGS комбинаторных библиотек, начиная с определенного раунда селекции [5, 6], анализ кривых плавления комплексов библиотека–лиганд [7], ЯМР [8, 9], ВЭЖХ [10] и другие [11]. В случае cell-SELEX предложено секвенирование после каждого шага селекции [12], что весьма трудозатратно и дорого.
Все описанные методы основаны на контроле библиотек в процессе проведения SELEX. Однако при применении модификаций более рациональным подходом может стать предварительная проверка совместимости модифицированных субстратов (dNTP) с используемой ДНК-полимеразой и оценка “нецелевого” обогащения модифицированной библиотеки, обусловленного конкурентной амплификацией, до проведения селекции аптамеров. Несмотря на ряд работ по субстратной совместимости модифицированных dNTP с ДНК-полимеразами [1, 2, 13, 14], до настоящего времени не разработан экспресс-метод оценки применимости различных модифицированных dNTP в селекции аптамеров без проведения раундов SELEX. Субстратная несовместимость в случае комбинаторных библиотек может не детектироваться методом ПЦР в режиме реального времени, поскольку быстрая кинетика накопления сигнала при амплификации может быть обусловлена конкурентной амплификацией “в обход” сложных последовательностей. Так происходит накопление сигнала за счет “нецелевого” обогащения библиотеки (конкурентная амплификация). Это приводит к необходимости использования сложных процедур проверки, осуществляемых не до, а уже в процессе mod-SELEX. Учитывая многостадийность и трудозатратность процедуры селекции с модифицированными dNTP, особенно в случае cell-SELEX [12, 15, 16], где требуется проведение трудоемких дополнительных процедур (наращивание культур чистых клеточных линий, мягкие методы создания комплексов для предотвращения разрушения клеточных стенок, повышенные требования к специфичности амплификации библиотек из-за присутствия нецелевых ДНК и РНК), разработка такого метода весьма актуальна.
Нами представлен новый метод определения субстратной эффективности модифицированных дезоксинуклеозидов в применении к селекции аптамеров, осуществляемый до начала процедуры SELEX.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
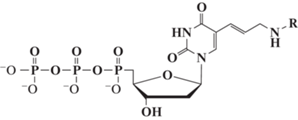
Модифицированные аналоги трифосфатов дезоксиуридина. Все использованные в работе соединения синтезированы в ИМБ РАН. Синтез модифицированных dUTP подробно описан в [17]. Химические структуры соединений приведены в табл. 1.
Таблица 1.
Эффективность амплификации комбинаторной ДНК-библиотеки, выход целевого продукта и характер изменения амплификационных кривых в течение трех последовательных раундов амплификации для различных mod-dUTP при полном замещении природного dTTP
 |
|||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| mod-dUTP [2] | R | Полимераза* | Полимераза** | ||||||||
| Taq | Vent (exo-) | Taq | Vent (exo-) | ||||||||
| Е | η | Е | η | 1 | 2 | 3 | 1 | 2 | 3 | ||
| dU2 | 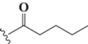 |
1.76 | 0.79 | 1.94 | 1.03 |  |
 |
 |
 |
 |
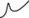 |
| dU8 | 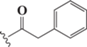 |
1.75 | 0.68 | 1.91 | 0.77 |  |
 |
 |
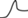 |
 |
 |
| dU9 | 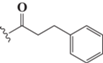 |
1.62 | 0.19 | 1.79 | 0.68 | 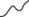 |
 |
 |
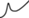 |
 |
 |
| dU10 | 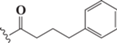 |
1.61 | 0.54 | 1.82 | 0.68 |  |
 |
 |
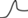 |
 |
 |
| dU13 | 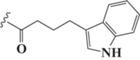 |
1.67 | 0.32 | 1.62 | 0.55 |  |
 |
 |
 |
 |
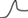 |
| dT (К+) | -H | 1.76 | 1.00 | 1.85 | 1.00 |  |
 |
 |
 |
 |
 |
* Эффективность амплификации и выход продукта (по данным [2]). В качестве матрицы использована комбинаторная составная ДНК-библиотека (длина 86 н.). Е – эффективность амплификации (Е = 10 tgα, где Е – эффективность амплификации, tgα – тангенс угла наклона прямого участка сигмоидной кривой накопления флуоресцентного сигнала, представленной в логарифмическом масштабе [19]), η – выход продукта, нормированный на выход немодифицированной ДНК (ПЦР с использованием природного dTTP). К+ ‒ положительный контроль (природный dTTP). Нумерация модифицированных dUTP по [2]. ** Характер амплификационных кривых (по данным настоящего исследования). 1, 2, 3 – последовательные раунды ПЦР. Приведены схематичные кривые накопления сигнала амплификации для каждого раунда ПЦР.
Комбинаторная ДНК-библиотека и праймеры для ее амплификации. В качестве матрицы использовали комбинаторную библиотеку Matrix_1f 5′-CTGTCAGCTCCATACTGGTAGCC-(N)40-GCGTTCGAATCTAGACGGTACGA-3′ длиной 86 н. и праймеры для ее амплификации: Forw_А CTGTCAGCTCCATACTGGTAGCC и Rev_A TCGTACCGTCTAGATTCGAACGC, описанные впервые в [18].
ПЦР в режиме реального времени. Реакционная смесь содержала природные dATP, dCTP и dGTP в концентрации 0.2 мМ каждого, а также различные модифицированные dUTP, структуры которых указаны в табл. 1 (при полной замене природного dTTP на один из модифицированных аналогов), также в концентрации 0.2 мМ; 1.5 U Taq- либо 0.5 U Vent (exo-) ДНК-полимеразы (реакционный буфер соответствовал примененной полимеразе); праймеры; комбинаторную библиотеку в начальной концентрации 105 копий на реакционный объем (20 мкл). Для мониторинга амплификации в реакционную смесь добавляли краситель EvaGreen (“Biotium”, Россия). Реакцию проводили на ДНК-амплификаторе IQ5 (“Bio-Rad”, США) по следующей программе: предварительная денатурация при 95°С в течение 3 мин, за которой следовали 50 циклов (95°С, 30 с; 65°С, 30 с; 72°С, 40 с). После 50-го цикла проводили инкубацию при 72°С в течение 5 мин.
После первого раунда амплификации отбирали 1 мкл реакционного объема и использовали его в качестве матрицы для проведения второго раунда ПЦР. После второго раунда также отбирали 1 мкл реакционного объема для проведения третьего раунда амплификации.
Контроль продуктов реакции. Контроль продуктов реакции осуществляли с помощью ПЦР указанного выше состава (проводили отбор из того же мастер-микса) по тому же температурно-временному профилю за исключением того, что число циклов ПЦР сокращали до 32 во избежание появления артефактов и побочных продуктов амплификации ДНК. Реакцию проводили на ДНК-амплификаторе MiniCycler (“MJ Research”, США).
Проводили электрофорез ПЦР-продуктов в денатурирующих условиях. Образцы прогревали в течение 1 мин при 90°C в присутствии 75%-ого формамида и наносили на полиакриламидный гель (T = 15%, C = 7%), содержащий 20 М формамида. Электрофорез проводили в TBE-буфере. Гели окрашивали красителем SYBR Green. Контроль полноразмерных целевых продуктов осуществляли визуально после электрофореза.
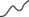
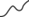
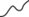
Интерпретация результатов ПЦР в режиме реального времени. При анализе процесса амплификации комбинаторных библиотек (рис. 1) визуально разделяли несколько типов характеристических амплификационных кривых: классическую сигмоиду, кривую с одним максимумом, кривую с двумя максимумами, кривую “затухающей” амплификации (характеризуется низким выходом продукта и невыраженной формой, стремящейся к прямой линии с низкими значениями тангенса угла наклона). Характеристические типы кривых схематично представлены в табл. 1.
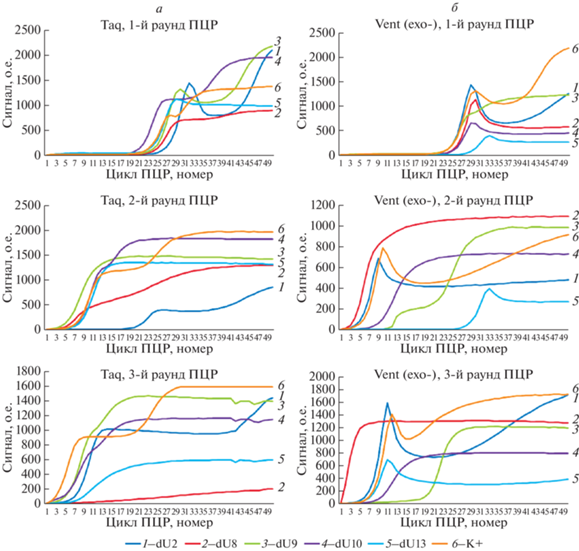
Рис. 1.
Кривые амплификации с модифицированными dUTP и ДНК-полимеразами. a – Taq; б – Vent (exo-). Основные характеристики процесса, а также характеристические типы амплификационных кривых приведены в табл. 1.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Влияние субстрата (модифицированного dNTP) на поведение кривой амплификации комбинаторной ДНК-библиотеки оценивали, проводя три последовательных раунда ПЦР в режиме реального времени (без стадий селекции аптамеров). В качестве матрицы для каждого следующего этапа брали 1 мкл реакционной смеси из пробирки предыдущего этапа.
Наблюдали несколько типов амплификационных кривых, форма которых зависела от степени влияния конкретного модифицированного dNTP на процесс амплификации библиотеки. На рис. 1 приведены результаты проведения трех раундов ПЦР с различными производными дезоксиуридинтрифосфата с полимеразами Taq и Vent (exo-). Из рис. 1 видно, что кривая амплификации может сохранять свою форму на протяжении всех трех раундов ПЦР, может постепенно приходить к каноническому виду сигмоиды, характерной для фиксированных нуклеотидных последовательностей матрицы либо вырожденных (обогащенных) комбинаторных библиотек, также может наблюдаться ингибирование ПЦР с постепенным затуханием амплификации. В качестве контроля использовали образец, не содержащий модифицированных дезоксинуклеозидов.
В табл. 1 приведены структуры использованных в работе дезоксиуридинтрифосфатов с различными функциональными группами-заместителями и показатели их субстратной эффективности. В правой части таблицы схематично показаны основные характеристические формы кривых амплификации, наблюдаемые в зависимости от использованного модифицированного дезоксиуридинтрифосфата и ДНК-полимеразы.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Для успешного отбора модифицированных аптамеров (mod-SELEX) требуется субстратная совместимость модифицированных dNTP с выбранной полимеразой. При их недостаточной совместимости возможна конкурентная амплификация последовательностей исходной ДНК-библиотеки, содержащих меньшее количество нуклеотидов или их повторов, комплементарных модифицированному dNTP. Часто низкая эффективность встраивания модификаций остается незамеченной в процессе проведения mod-SELEX. В результате библиотека будет обеднена модифицированными аптамерами.
Кривые амплификации исходных комбинаторных библиотек отличаются от кривых, характерных для амплификации матриц с фиксированными последовательностями. Различие заключается в том, что кривая накопления сигнала при амплификации фиксированной последовательности имеет сигмоидную форму, а в случае комбинаторной библиотеки проходит через один или более максимумов. По всей видимости, это связано с тем, что после определенного цикла ПЦР и распределения амплифицированных продуктов по реакционному объему образуются локальные очаги, в которых велика концентрация дуплексов с некомплементарной внутренней частью. Это приводит к временному замедлению амплификации и снижению наблюдаемого накопления сигнала. Однако по мере образования новых комплементарных дуплексов – продуктов амплификации – кривая проходит через максимум. По-видимому, этот процесс может приводить к появлению более чем одного максимума, что и наблюдается при амплификации комбинаторных библиотек (рис. 1).
Ранее было показано, что в процессе обогащения ДНК-библиотеки кривая ее амплификации в режиме реального времени с каждым следующим раундом приближается по форме к канонической сигмоидной кривой, характерной для фиксированной последовательности ДНК [7, 20]. Это служит показателем обогащения библиотеки специфичными к мишени олигонуклеотидами (рис. 2), т.е., в процессе обогащения постепенно ослабевают факторы, указанные выше (образование дуплексов с некомплементарными внутренними частями олигонуклеотидов библиотеки).
Рис. 2.
Амплификационные кривые, получаемые при использовании в ПЦР различных матриц. а –Матрица с фиксированной последовательностью; кривая имеет канонический сигмоидный вид. б – Вырожденная комбинаторная библиотека (1–6 – стадии обогащения библиотеки, в процессе которых амплификационная кривая постепенно приобретает канонический сигмоидный вид). По данным [20].
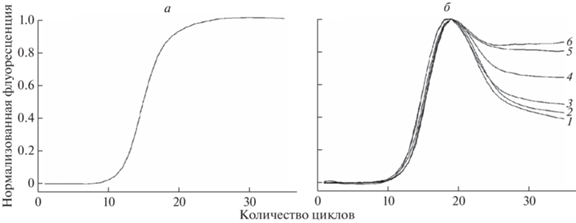
Однако библиотека может обогащаться не специфичными, а наиболее легко амплифицируемыми последовательностями [3]. Наиболее актуальной эта проблема становится при использовании модифицированных dNTP, которые служат, как правило, плохими субстратами для полимераз, характеризуются медленной кинетикой встраивания и/или в той или иной степени ингибируют ПЦР.
Из табл. 1 видно, что характер амплификационных кривых зависит как от использованного модифицированного dNTP, так и от полимеразы. Taq-полимераза оказалась способной амплифицировать библиотеки с применением модифицированного dUTP с линейным алкановым заместителем (dU2). Это подтверждается как показателями эффективности ПЦР “Е” и значением выхода целевого продукта “η”, так и относительно неизменным характером кривой амплификации. При этом гетероциклические производные оказываются для нее сложными субстратами, что приводит к изменению формы кривой амплификации – за три цикла ПЦР она либо становится сигмоидной, либо ингибируется (как в случае dU8). При этом может наблюдаться как быстрая кинетика накопления сигнала, так и высокий выход полноразмерного продукта, т.е. эти показатели не подходят для оценки влияния модифицированных dNTP на поведение комбинаторных библиотек в процессе mod-SELEX.
В случае производных с бензильным заместителем наблюдали независимое от длины используемого линкера вырождение библиотек и сигмоидный характер кривой амплификации после третьего раунда ПЦР. Причем это было характерно и при применении Vent (exo-)-полимеразы. Интересно, но производное dU13 с конденсированным гетероциклом незначительно влияло на характер кривой амплификации при использовании Vent (exo-)-полимеразы, хотя показатели эффективности амплификации и выхода были низкими.
При изменении характера кривой амплификации делали вывод о неприменимости конкретного модифицированного dNTP в выбранных условиях, которые могут содержать несколько значимых факторов (используемая полимераза, условия амплификации, комбинаторная библиотека и праймеры для ее амплификации и др.).
Из табл. 1 видно, что при использовании “плохих” модифицированных субстратов характер кривой менялся на протяжении трех циклов амплификации, при отсутствии детектируемого влияния модификаций он оставался неизменным. Если характер кривой амплификации не менялся на протяжении трех раундов ПЦР, делали вывод о том, что влияние модификации dUTP на распределение последовательностей внутри комбинаторной библиотеки минимально (либо отсутствует), т.е. такое производное не приводит к вырождению библиотеки при амплификации и может быть применено для проведения mod-SELEX.
ВЫВОДЫ
Нами разработан метод быстрой оценки применимости модифицированных dNTP в mod-SELEX.
Метод позволяет за три загрузки амплификатора проверить весь необходимый набор трифосфатов – кандидатов для планируемого mod-SELEX.
Следует заметить, что предлагаемый подход не исключает контроля обогащения библиотек в процессе селекции, но в случае mod-SELEX позволяет предсказать влияние модификаций на обогащение библиотек и избежать неудач в последующем процессе отбора модифицированных аптамеров.
Исследование поддержано грантом Российского фонда фундаментальных исследований (№ 18-29-09151).
Настоящая статья не содержит каких-либо исследований с участием людей и животных в качестве объектов исследований.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Rohloff J.C., Gelinas A.D., Jarvis T.C., Ochsner U.A., Schneider D.J., Gold L., Janjic N. (2014) Nucleic acid ligands with protein-like side chains: modified aptamers and their use as diagnostic and therapeutic agents. Mol. Ther. Nucl. Acids. 3, e201.
Лапа С.А., Павлов А.С., Кузнецова В.Е., Шершов В.Е., Спицын М.А., Гусейнов Т.О., Радько С.П., Заседателев А.С., Лисица А.В., Чудинов А.В. (2019) Ферментативное получение модифицированных ДНК: изучение кинетики ПЦР в режиме реального времени. Молекуляр. биология. 53, 513–523.
Tolle F., Wilke J., Wengel J., Mayer G. (2014) By-product formation in repetitive PCR amplification of DNA libraries during SELEX. PLoS One. 9, e114693.
Shao K., Shi X., Zhu X., Cui L., Shao Q., Ma D. (2017) Construction and optimization of an efficient amplification method of a random ssDNA library by asymmetric emulsion PCR. Biotechnol. Appl. Biochem. 64, 239–243.
Schütze T., Wilhelm B., Greiner N., Braun H., Peter F., Mörl M., Erdmann V.A., Lehrach H., Konthur Z., Menger M., Arndt P.F., Glökler J. (2011) Probing the SELEX process with next-generation sequencing. PLoS One. 6, e29604.
Kolm C., Cervenka I., Aschl U.J., Baumann N., Jakwerth S., Krska R., Mach R.L., Sommer R., DeRosa M.C., Kirschner A.K.T., Farnleitner A.H., Reischer G.H. (2020) DNA aptamers against bacterial cells can be efficiently selected by a SELEX process using state-of-the art qPCR and ultra-deep sequencing. Sci. Rept. 10, 20917.
Luo Z., He L., Wang J., Fang X., Zhang L. (2017) Developing a combined strategy for monitoring the progress of aptamer selection. Analyst. 142, 3136–3139.
Sanchez J.C., Zhang L., Evoli S., Schnicker N.J., Nunez-Hernandez M., Yu L., Wereszczynski J., Pufall M.A., Musselman C.A. (2020) The molecular basis of selective DNA binding by the BRG1 AT-hook and bromodomain. Biochim. Biophys. Acta Gene Regul. Mech. 1863, 194566.
Amano R., Aoki K., Miyakawa S., Nakamura Y., Kozu T., Kawai G., Sakamoto T. (2017) NMR monitoring of the SELEX process to confirm enrichment of structured RNA. Sci. Rep. 7, 283.
Müller J., El-Maarri O., Oldenburg J., Pötzsch B., Mayer G. (2008) Monitoring the progression of the in vitro selection of nucleic acid aptamers by denaturing high-performance liquid chromatography. Anal. Bioanal. Chem. 390, 1033–1037.
Thevendran R., Citartan M. (2022) Assays to estimate the binding affinity of aptamers. Talanta. 238, 122971.
Gooch J., Tungsirisurp S., Costanzo H., Napier R., Frascione N. (2021) Generating aptamers towards human sperm cells using massively parallel sequencing. Anal. Bioanal. Chem. 413, 5821–5834.
Чудинов А.В., Шершов В.Е., Павлов А.С., Волкова О.С., Кузнецова В.Е., Заседателев А.С., Лапа С.А. (2020) Одновременное встраивание модифицированных производных dU и dC в растущую цепь ДНК в реакции удлинения праймера и ПЦР. Биоорг. химия. 46, 546–549.
Лапа С.А., Волкова О.С., Кузнецова В.Е., Заседателев А.С., Чудинов А.В. (2022) Изучение множественного ферментативного встраивания модифицированных нуклеотидов пуриновой и пиримидиновой природы в растущую цепь ДНК. Молекуляр. биология. 56, 157–167.
Rahimizadeh K., AlShamaileh H., Fratini M., Chakravarthy M., Stephen M., Shigdar S., Veedu R.N. (2017) Development of cell-specific aptamers: recent advances and insight into the selection procedures. Molecules. 22, 2070.
Sefah K., Shangguan D., Xiong X., O’Donoghue M.B., Tan W. (2010) Development of DNA aptamers using cell-SELEX. Nat. Protoc. 5, 1169–1185.
Чудинов А.В., Киселева Я.Ю., Кузнецова В.Е., Шершов В.Е., Спицын М.А., Гусейнов Т.О., Лапа С.А., Тимофеев Э.Н., Арчаков А.И., Лисица А.В., Радько С.П., Заседателев А.С. (2017) Ферментативный синтез ДНК с высокой степенью модификации. Молекуляр. биология. 51, 534–544.
Лапа С.А., Ромашова К.С., Спицын М.А., Шершов В.Е., Кузнецова В.Е., Гусейнов Т.О., Заседателева О.А., Радько С.П., Тимофеев Э.Н., Лисица А.В., Чудинов А.В. (2018) Получение модифицированных комбинаторных ДНК-библиотек методом ПЦР в обратной эмульсии с последующим разделением цепей. Молекуляр. биология. 52, 984–996.
Ramakers C., Ruijter J.M., Deprez R.H., Moorman A.F. (2003) Assumption-free analysis of quantitative realtime polymerase chain reaction (PCR) data. Neurosci. Lett. 339, 62–66.
Радько С.П., Лапа С.А., Чудинов А.В., Хмелёва С.А., Маннанова М.М., Курбатов Л.К., Киселёва Я.Ю., Заседателев А.С., Лисица А.В. (2019) Оценка разнообразия комбинаторных ДНК-библиотек на основе анализа формы амплификационных кривых для мониторинга эффективности селекции аптамеров. Биомед. хим. 65, 477–484.
Дополнительные материалы отсутствуют.
Инструменты
Молекулярная биология


