Молекулярная биология, 2022, T. 56, № 4, стр. 574-584
HECTD2 cупрессирует пролиферацию клеток колоректального рака, направляя LPCAT1 на убиквитинирование и деградацию
L. Ma a, D. H. Li a, Z. Xu a, *
a Department of General Surgery, Qingdao Municipal Hospital, Qingdao University
266000 Qingdao, China
* E-mail: xuzhixuzhi1981@163.com
Поступила в редакцию 09.11.2021
После доработки 29.12.2021
Принята к публикации 29.12.2021
- EDN: AEVLMU
- DOI: 10.31857/S0026898422040073
Аннотация
Колоректальный рак (КРР) представляет собой злокачественное новообразование, характеризующееся плохой общей выживаемостью и высокой частотой рецидивов, в то время как биомаркеры КРР еще плохо изучены. Нами обнаружено, что лизофосфатидилхолинацилтрансфераза-1 (LPCAT1) высоко экспрессирована при КРР, а сверхэкспрессия LPCAT1 значительно облегчала пролиферацию клеток КРР, в то время как дефицит LPCAT1 приводил к обратному эффекту. Кроме того, содержащий HECT-домен белок-2 (HECT domain-containing 2 protein; HECTD2) идентифицирован как пост-трансляционный медиатор LPCAT1, так как при иммунопреципитации анти-LPCAT1-антителами находился в комплексе с высокоубиквитинированным LPCAT1. Кроме того, повышенная экспрессия LPCAT1 восстанавливала пролиферацию клеток КРР, нарушенную сверхэкспрессией HECTD2. На основании полученных данных ось HECTD2/LPCAT1 можно считать потенциальным прогностическим биомаркером КРР.
Колоректальный рак (КРР) ‒ распространенное злокачественное заболевание, которое занимает второе место по смертности от онкологических заболеваний во всем мире: в 2020 году от него скончалось около 935 000 человек [1]. Считается, что хирургическая резекция, лучевая терапия и химиотерапия могут быть использованы для лечения КРР, однако прогноз остается неблагоприятным из-за высокой частоты рецидивов [2]. В связи с этим необходимо выяснить механизмы, участвующие в прогрессировании КРР, и биомаркеры, предсказывающие рецидив заболевания.
Лизофосфатидилхолинацилтрансфераза-1 (LPCAT1) относится к важным ферментам, которые участвуют в процессе метаболизма липидов [3], обладает ацетилтрансферазной активностью и находится в аппарате Гольджи, эндоплазматическом ретикулуме и других органеллах клетки [4]. На основании накопленных данных по структуре, функциям и механизмам регуляции LPCAT1 доказана функциональная роль LPCAT1 в синтезе альвеолярного сурфактанта [5], дегенерации сетчатки [6] и заболеваниях нервной системой [7]. Недавно обнаружено, что уровень LPCAT1 повышен при различных видах злокачественных опухолей, таких как рак эндометрия [8], рак простаты [9] и аденокарцинома легких [10]. Кроме того, F. Mansilla и соавт. [11] пришли к выводу, что LPCAT1 может способствовать накоплению метаболитов холина, изменяя липидный профиль КРР. Однако другие механизмы, опосредованные LPCAT1 при КРР, и функциональная роль LPCAT1 в прогрессировании КРР пока не выяснены.
Убиквитинирование ‒ распространенная посттрансляционная модификация [12]. В ходе этого процесса ряд ферментов, включая активирующие убиквитин, конъюгирующие убиквитин, лигирующие и деградирующие, выступают в качестве важнейших участников, которые сортируют внутриклеточные белки, выбирают и специфически модифицируют убиквитином белки-мишени [13, 14]. Показано, что убиквитинирование играет решающую роль в регуляции локализации, метаболизма и деградации белков [15]. Кроме того, доказано, что убиквитинирование участвует в регуляции практически всех видов жизнедеятельности, таких как клеточный цикл [16], пролиферация [17], апоптоз и аутофагия [18], дифференциация [19], метастазирование [20], воспаление и иммунитет [21]. Функционально значимый, грандиозный механизм убиквитинирования уже исследован достаточно детально при различных видах злокачественных опухолей, таких как рак молочной железы [20], немелкоклеточный рак легкого [17] и внутрипеченочная холангиокарцинома [22]. Кроме того, E3-убиквитинлигаза, содержащий HECT-домен белок-2 (HECTD2), участвует в прогрессировании различных заболеваний, таких как бактериальное воспаление легких [23], экспериментальное повреждение легких [24] и рак простаты [25]. Однако вклад HECTD2-опосредованного убиквитинирования в развитие КРР еще предстоит изучить.
Учитывая решающую роль LPCAT1 в росте и метастазировании злокачественных опухолей, мы оценили уровни LPCAT1 и исследовали биологическую функцию этого белка при КРР. Кроме того, выясняли, может ли HECTD2 опосредовать убиквитинирование LPCAT1. В результате проведенного исследования выявлено функциональное влияние оси HECTD2/LPCAT1 на прогрессирование КРР, что позволяет считать ее потенциальной мишенью для разработки терапевтической стратегии КРР.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Биоинформационный анализ. Экспрессия LPCAT1 в тканях КРР и нормальных тканях толстой кишки была извлечена из базы данных ENCORI [26] и базы данных Human Protein Atlas [27].
Коллекция тканей и культура клеток. Четыре фиксированных в формалине и парафинированных ткани КРР и соответствующие нормальные ткани были получены из Муниципальной больницы Циндао, Университет Циндао (Qingdao Municipal Hospital, Qingdao University, Китай).
От каждого пациента было получено информированное согласие. Экспериментальная стратегия проводилась в соответствии с Хельсинкской декларацией и была одобрена этическим комитетом Муниципальной больницы Циндао, Циндаоского университета.
Клеточные линии КРР: LOVO и AGS ‒ были приобретены в Американской коллекции типовых культур (American Type Culture Collection, США). Клетки культивировали в среде RPMI 1640, содержащей 10% эмбриональной сыворотки крупного рогатого скота (FBS; “Invitrogen”, США) в увлажненной атмосфере 5% CO2 при 37°C.
Иммуноблотинг. Суммарный белок выделяли из клеток или тканей КРР с помощью буфера для лизиса RIPA, белки разделяли электрофорезом в SDS-ПААГ и переносили на PVDF-мембрану. Мембрану блокировали 5%-ным обезжиренным молоком в течение 1 ч при комнатной температуре и инкубировали с моноклональными кроличьими антителами против LPCAT1 (1/2000; ab214034, “Abcam”, Великобритания), HECTD2 (1/3000; ab173572, “Abcam”) и GAPDH (1/1000; ab181602, “Abcam”) в течение ночи при 4°С. После этого мембраны обрабатывали вторичными антителами ‒ конъюгированными с пероксидазой хрена (HRP) козьими антителами против кроличьего IgG (1/1000; ab97051, “Abcam”). Полосы визуализировали с использованием хемилюминесцентного субстрата ‒ ChemistarTM High-sig ECL Western Blotting Substrate (“Tanon Science & Technology Co., Ltd.”, Китай).
Трансфекция клеток. Конструкты pcDNA-LPCAT1 (pLPCAT1), pcDNA-HECTD2 (pHECTD2), sh-LPCAT1-1 и sh-LPCAT1-2 и соответствующие пустые плазмиды и векторы, экспрессирующие скремблированную shРНК (NC), были разработаны компанией “Genechem” (Китай) (табл. S1 Приложения, см. Дополнительные материалы на сайте http://www.molecbio.ru/ downloads/2022/4/supp_Ma_rus.pdf). С помощью реагента для трансфекции Lipofectamine 2000 (11668-019; “Invitrogen”, США) плазмиды и лентивирусы, несущие shРНК, рансфицировали в клетки КРР в соответствии с инструкциями производителя.
Количественная ПЦР с обратной транскрипцией (ОТ-ПЦР). Для выделения суммарной РНК из клеток или тканей КРР использовали реагент Trizol (“Absin”, Китай), а для обратной транскрипции и амплификации кДНК ‒ набор PrimeScript RT Reagent Kit (DRR037A; “Takara”, Япония) и SYBR Master Mix (“Takara”) соответственно. Относительная экспрессия HECTD2 и LPCAT1 была нормализована на GAPDH и рассчитана методом 2‒ΔΔCt. Последовательности праймеров представлены в табл. S2 Приложения.
Анализ образования колоний. Клетки КРР вносили в 6-луночный планшет (2 × 103 клеток/лунка) на 12 суток. После фиксации с помощью параформальдегида клетки окрашивали 0.5%-ным раствором кристаллического фиолетового. Клонированные клетки подсчитывали и визуализировали под световым микроскопом.
Подсчет клеток. Для подсчета клеток использовали набор Cell Counting kit-8 (CCK-8; #C0038, “Beyotime”, Китай). В лунки 96-луночного планшета вносили по 4 × 103 клеток КРР в 100 мкл клеточной суспензии и инкубировали с реагентом CCK-8 (10 мкл/лунка; “Beyotime”) в течение 24, 48, 72 и 96 ч. Через 1 ч инкубации при 37°C измеряли оптическую плотность при 450 нм (OD450) на микропланшетном ридере.
Окрашивание 5-этинил-2'-дезоксиуридином (EdU). При оценке жизнеспособности клеток КРР анализировали включение в них EdU, используя EdU kit (“Roche”, Швейцария) в соответствии с инструкциями производителя. EdU-позитивные клетки визуализировали с помощью флуоресцентного микроскопа Zeiss (“Carl Zeiss”, Германия) на пяти случайных полях и подсчитывали с помощью программного обеспечения Image-Pro Plus 6.0 (“Media Cybernetics”, США).
Анализ коиммунопреципитации (Co-IP). Для выделения суммарного белка из клеток КРР использовали буфер для лизиса Co-IP (”Beyotime”). Dynabeads Protein G (1004D, “Life Technologies”, США) инкубировали в течение 2 ч с кроличьими моноклональными антителами против HA-tag (#3724; “Cell Signaling Technology”, США) или поликлональными против DDDK-tag (связывают Flag) (ab1162; “Abcam”). Затем белковый лизат инкубировали с комплексом антитело‒магнитные бусины при 4°C в течение ночи. Бусины промывали элюирующим буфером, а иммунопреципитат и входящие в него белки анализировали методом иммуноблотинга.
Масс-спектрометрия. После процедуры Co-IP очищенный HECTD2 и его иммунопреципитаты анализировали методом жидкостной хроматографии с тандемной масс-спектрометрией (LC-MS/MS) на системах Expert TM nanoLC (“Thermo Fisher Scientific”, США) и AB Sciex TripleTOF® 5600+ (“Sciex”, США) по протоколам производителей.
Анализ убиквитинирования. Клетки КРР трансфицировали плазмидами, несущими гены, кодирующие HA-HECTD2, Flag-LPCAT1, His-ub (убиквитин с гистидиновой меткой) или His-ub-K63R (His-меченый убиквитин с заменой Lys63 на Arg), и инкубировали с протеасомным ингибитором MG132 (10 мкМ; “Sigma”, США) в течение 6 ч. Flag-LPCAT1 осаждали анти-Flag-антителами и проводили иммуноблотинг, в котором использовали антитела против убиквитина (#3936, “Cell Signaling Technology”).
Анализ полураспада. Для оценки стабильности белка LPCAT1 клетки КРР трансфицировали pHECTD2 и обрабатывали циклогексимидом (“Sigma”) в концентрации 40 мМ в течение 0, 2, 4 и 6 ч. Белки лизатов, полученных в результате лизиса клеток, разделяли электрофорезом в SDS-ПААГ и затем анализировали иммуноблотингом.
Статистический анализ. Данные анализировали с помощью программы GraphPad Prism 5.01 и представляли как среднее ± стандартное отклонение (SD). Для анализа различий между двумя или несколькими группами использовали t-тест Стьюдента и дисперсионный анализ (ANOVA). Различия считали значимыми при p-value < 0.05.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Экспрессия LPCAT1 активируется при колоректальном раке
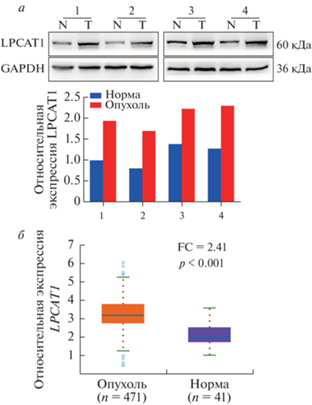
Уровни белка LPCAT1 оценили в тканях КРР и соответствующих нормальных тканях и обнаружили, что LPCAT1 высоко экспрессирован в опухолевых тканях КРР (рис. 1а). Для проверки этого результата мы проанализировали базы данных ENCORI (http://www.sysu.edu.cn/) и выявили гораздо более высокие уровни мРНК LPCAT1 в опухолевых тканях по сравнению с нормальными тканями в образцах КРР, представленными в базе The Cancer Genome Atlas (TCGA; https://www.cancer.gov/about-nci/organization/ccg/research/structural-genomics/tcga) (рис. 1б). В совокупности эти данные свидетельствуют о высокой экспрессии LPCAT1 при КРР.
Рис. 1.
Повышенная экспрессия LPCAT1 при КРР. а ‒ Экспрессия LPCAT1 в клинических образцах опухолевых тканей КРР и соответствующих нормальных тканей, полученных от 4 пациентов (образцы 1‒4). Иммуноблотинг проведен с антителами против LPCAT1. б ‒ Результаты анализа базы данных ENCORI по экспрессии LPCAT1 при КРР. FC ‒ кратность изменения; p ‒ логарифмический ранговый критерий.
При повышенной экспрессии LPCAT1 ускоряется пролиферация клеток колоректального рака
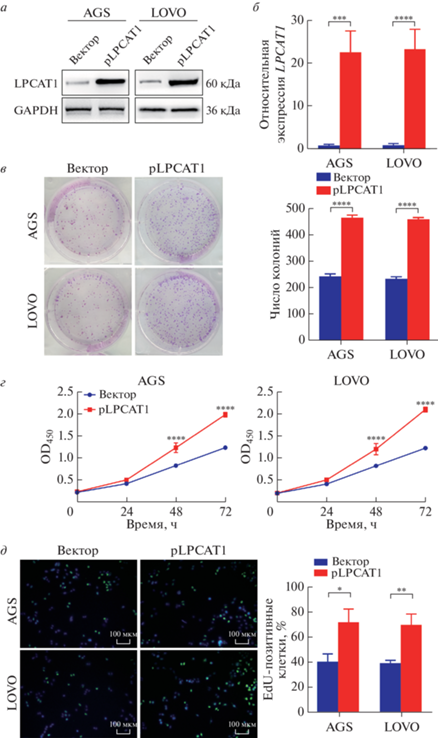
Для изучения функциональной роли LPCAT1 при КРР мы создали клетки AGS и LOVO, сверхэкспрессирующие LPCAT1, и исследовали эффективность трансфекции (рис. 2a и 2б). По результатам анализов колониеобразования (рис. 2в) и CCK-8 (рис. 2г) при сверхэкспрессии LPCAT1 повышается скорость пролиферации клеток КРР, а также значительно увеличивается число клеток, инкорпорировавших EdU (рис. 2д). В целом, полученные данные подтвердили канцерогенную роль LPCAT1 при КРР.
Рис. 2.
Повышенный уровень LPCAT1 способствует пролиферации клеток КРР. Клетки КРР трансфицировали плазмидами, содержащими ген LPCAT1, и анализировали эффективность экспрессии целевого гена методом иммуноблотинга (а) и количественной ОТ-ПЦР (б). Для оценки скорости пролиферации клеток КРР использовали тесты колониеобразования (в), CCK-8 (г) и EdU (д). Все эксперименты выполнены в трех повторах, данные представлены как среднее ± SD. *p < 0.05, **p < 0.01, ***p < 0.001 и ****p < 0.0001.
При дефиците LPCAT1 скорость пролиферации клеток колоректального рака снижена
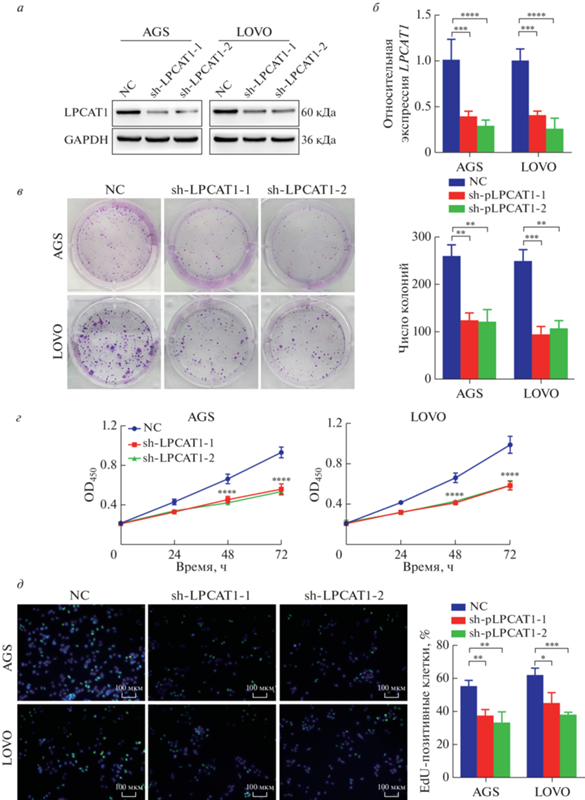
Учитывая, что повышенный уровень LPCAT1 способствовал пролиферации клеток КРР, мы проверили биологический эффект истощения LPCAT1 на эти клетки. Для функциональных экспериментов LPCAT1-истощенные клетки КРР получили с использованием коротких интерферирующих РНК (siРНК), нацеленных на мРНК LPCAT1 (рис. 3a и 3б). По результатам колониеобразования (рис. 3в) и CCK-8 (рис. 3г) истощение по LPCAT1 приводило к снижению скорости пролиферации клеток КРР. Кроме того, EdU-анализ показал, что при дефиците LPCAT1 явно снижалось содержание EdU-позитивных клеток (рис. 3д). Таким образом, на основании полученных результатов можно говорить о том, что дефицит LPCAT1 индуцирует замедление пролиферации клеток КРР.
Рис. 3.
При дефиците LPCAT1 замедляется пролиферация клеток КРР. Клетки КРР трансдуцировали лентивирусами, несущими shРНК к гену LPCAT1 (sh-LPCAT1) или скремблированную shРНК (NC). Экспрессию LPCAT1 оценивали методом иммуноблотинга (a) и количественной ОТ-ПЦР (б). Скорость пролиферации клеток КРР оценивали методами колониеобразования (в), CCK-8 (г) и EdU (д). Все эксперименты выполнены в трех повторах, данные представлены как среднее ± SD. *p < 0.05, **p < 0.01, ***p < 0.001 и ****p < 0.0001.
HECTD2 убиквитинирует LPCAT1 в клетках колоректального рака
После сверхэкспрессии Flag-HECTD2 в клетках LOVO, последующей иммунопреципитации анти-Flag-антителами проведен масс-спектрометрический анализ осажденных белков. Вместе с белком HECTD2 соосаждался белок LPCAT1, из чего можно строить гипотезу о их взамодействии (рис. 4а). Кроме того, уровень HECTD2 в тканях КРР был ниже, чем в нормальных тканях (рис. 4б и 4в).
Рис. 4.
В клетках КРР HECTD2 убиквитинирует LPCAT1. Для идентификации белков, взаимодействующих с LPCAT1, использована масс-спектрометрия (а). Уровень экспрессии HECTD2 в тканях КРР (Т) и нормальных тканях (N) оценивали методом количественной ОТ-ПЦР (б) и иммуноблотинга (в). Взаимодействие между HECTD2 и LPCAT1 оценивали с использованием Co-IP и убиквитинирования. В клетках LOVO сверхэкспрессировали Flag-LPCAT1 или HA-HECTD2, и белок Flag-LPCAT1 осаждали анти-Flag-антителами (IP:Flag), после чего проводили иммуноблотинг (IB) с анти-Flag- и анти-HA-антителами (г). Анализ на убиквитинирование проводили в клетках LOVO, трансфицированных His-ub или His-ub-K63R (д). Уровень белка LPCAT1 исследовали через 0, 2, 4 и 6 ч после обработки клеток циклогексимидом (CHX) (е). Все эксперименты проведены в трех повторах, данные представлены как среднее значение ± SD. *p < 0.05, **p < 0.01, ***p < 0.001 и ****p < 0.0001.
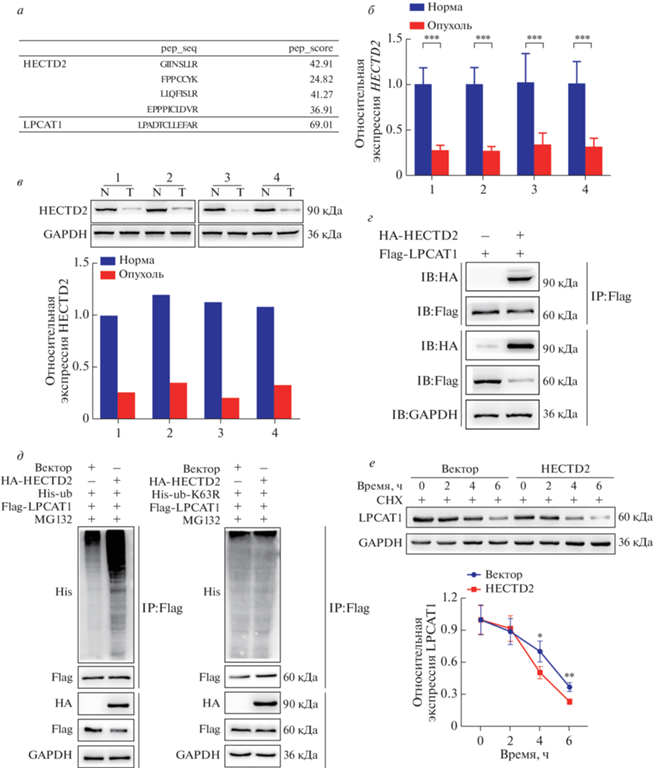
С целью исследовать взаимодействие между HECTD2 и LPCAT1 мы проведен анализ Co-IP и обнаружили, что HECTD2 c Flag-тегом интенсивно соосаждается с LPCAT1 в клетках КРР (рис. 4г). Кроме того, по данным анализа убиквитинирования сверхэкспрессия HECTD2 повышает уровень убиквитинирования LPCAT1 (рис. 4д). Для проверки этих результатов оценили период полужизни белка LPCAT1 и обнаружили, что он сокращается при сверхэкспрессия HECTD2, то есть HECTD2 снижает стабильность белка LPCAT1 (рис. 4е). В результате проведенного исследования показано, что HECTD2 функционирует как E3-убиквитинлигаза, “руководящая” убиквитинированием и деградацией белка LPCAT1 при КРР.
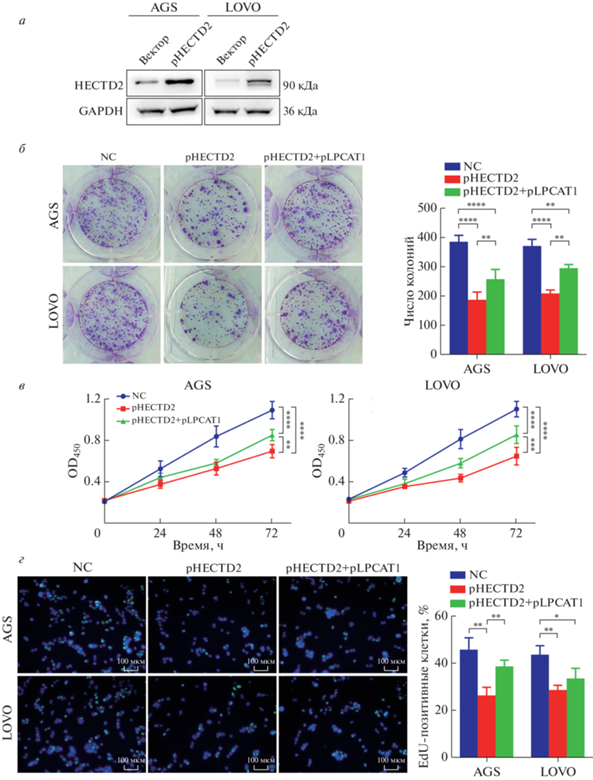
HECTD2 супрессирует пролиферацию клеток колоректального рака через убиквитинирование LPCAT1
С целью определить, действует ли HECTD2 как канцерогенный драйвер прогрессирования КРР LPCAT1-зависимым образом, плазмиды со сверхэкспрессией HECTD2 и LPCAT1 совместно трансфицировали в клетки КРР (рис. 5a). Обнаружено, что сверхэкспрессия HECTD2 приводила к значительному снижению пролиферативной активности клеток КРР, которая восстанавливалась при сверхэкспрессии LPCAT1 (рис. 5б и 5в). Кроме того, сверхэкспрессия LPCAT1 играла критическую роль в восстановлении числа EdU-позитивных клеток, потеря которых была вызвана сверхэкспрессией HECTD2 (рис. 5г). В совокупности эти результаты показали, что HECTD2 подавляет прогрессирование КРР посредством убиквитинирования LPCAT1.
Рис. 5.
HECTD2 супрессирует пролиферацию клеток КРР через убиквитинирование LPCAT1. Клетки КРР котрансфицировали плазмидами, экспрессирующими HECTD2 (pHECTD2) и LPCAT1 (pLPCAT1), и оценивали эффективность трансфекции методом иммуноблотинга (а). Пролиферацию клеток КРР оценивали методами колониеобразования (б), CCK-8 (в) и включения EdU (г). Все эксперименты проведены в трех повторах, данные представлены как среднее ± SD. *p < 0.05, **p < 0.01, ***p < 0.001 и ****p < 0.0001. NC ‒ отрицательный контроль.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
Нами показано, что LPCAT1 действует как канцерогенный фактор при КРР, в то время как HECTD2 идентифицирован как белок, способствующий убиквитинированию и деградации белка LPCAT1. Таким образом, определена функциональная роль оси HECTD2/LPCAT1 в прогрессировании КРР.
Ранее сообщалось об онкогенной роли LPCAT1 при различных видах злокачественных образований, таких как рак желудка [28], плоскоклеточный рак полости рта [29] и рак предстательной железы [30]. В частности, F. Liu и др. [31] продемонстрировали, что LPCAT1 необходим для пролиферации клеток при многих онкологических заболеваниях, а повышенная экспрессия LPCAT1 тесно связана с плохим прогнозом. P. Lebok и др. [32] выявили, что сверхэкспрессия LPCAT1 способствовует росту опухоли при раке молочной железы и ассоциируется с плохим прогнозом. Кроме того, Y. Du и соавт. [33] пришли к выводу, что позитивная регуляция LPCAT1 индуцирует пролиферацию и инвазию клеток при светлоклеточной почечно-клеточной карциноме. Соответственно нами показано, что экспрессия LPCAT1 повышена в тканях КРР. Трансфекция клеток КРР плазмидами, обеспечивающими сверхэкспрессию LPCAT1 или подавляющими ее под действием siРНК к LPCAT1, приводила соответственно к ускоренной пролиферации клеток или наоборот ее замедлению при истощении по LPCAT1.
Уже показано, что убиквитинирование играет важную роль в регуляцию прогрессии злокачественных новообразований, в том числе при КРР. Так, J. Hu и соавт. [34] сообщали, что TRIM39 стимулирует активность Rab7 через супрессию его убиквитинирования, тем самым сдерживая аутофагический поток и метастазирование опухоли при КРР, а B. Li и др. [35] показали, что USP10 повышает уровень деубиквитинирования NLRP7, способствуя росту опухоли и коррелируемой злокачественной опухолью поляризации макрофагов при КРР. В проведенном нами исследовании HECTD2 интенсивно соосаждался с LPCAT1 в экспериментах по коиммунопреципитации и идентифицирован как возможный партнер LPCAT1 на основании данных масс-спектрометрии. Более того, сверхэкспрессия HECTD2 приводила к повышению уровня убиквитинирования белка LPCAT1 и индуцировала его деградацию. Ранее в моделях пневмонии показано, что HECTD2 усиливает воспаление через убиквитинирование белка PIAS1 и его деградацию [24]. Однако нами выявлено, что сверхэкспрессия HECTD2 подавляла пролиферацию клеток КРР, которая восстанавливалась при повышении уровня LPCAT1. В некоторых исследованиях сообщалось о повышенной экспрессии 19S-регулятора при КРР [36, 37]. Этот регулятор распознает убиквитинированные белки и направляет их в протеасомы для деградации. Интересно, что, несмотря на повышенное количество 19S-регулятора, экспрессия HECTD2 при КРР снижена, что позволяет опухоли поддерживать уровень LPCAT1.
Таким образом, на основании полученных результатов можно сделать вывод, что LPCAT1, по-видимому, способствует прогрессированию КРР, а HECTD2 ‒ убиквитинированию и деградации белка LPCAT1, тем самым ограничивая пролиферацию клеток КРР. Таким образом, ось HECTD2/ LPCAT1 можно рассматривать в качестве потенциального прогностического биомаркера при колоректальном раке.
Это исследование не получило какого-либо специального гранта от финансирующих агентств в государственном, коммерческом или некоммерческом секторах.
Все эксперименты проведены в соответствии с Хельсинкской декларацией и были одобрены Комитетом по этике Муниципальной больницы Циндао Университета Циндао (Qingdao Municipal Hospital, Qingdao University, Китай).
Все испытуемые согласились участвовать в данном исследовании и подписали полную форму информированного согласия.
Вклад авторов: Lei Ma и Zhi Xu разработали экспериментальную стратегию. Lei Ma и Dehui Li провели эксперименты и проанализировали результаты. Рукопись подготовлена Lei Ma и отредактирована Zhi Xu. Все авторы одобрили окончательный вариант рукописи.
Список литературы
(2020) Erratum: Global cancer statistics 2018: GLOBOCAN estimates of incidence and mortality worldwide for 36 cancers in 185 countries. CA Cancer J. Clin. 70, 313.
Feng M., Zhao Z., Yang M., Ji J., Zhu D. (2021) T-cell-based immunotherapy in colorectal cancer. Cancer Lett. 498, 201‒209.
Barbosa A., Savage D., Siniossoglou S. (2015) Lipid droplet-organelle interactions: emerging roles in lipid metabolism. Curr. Opin. Cell Biol. 35, 91‒97.
Shindou H., Hishikawa D., Harayama T., Yuki K., Shimizu T. (2009) Recent progress on acyl CoA: lysophospholipid acyltransferase research. J. Lipid Res. 50(Suppl.), S46‒S51.
Lin S., Ikegami M., Moon C., Naren A. Shannon J. (2015) Lysophosphatidylcholine acyltransferase 1 (LPCAT1) specifically interacts with phospholipid transfer protein StarD10 to facilitate surfactant phospholipid trafficking in alveolar type II cells. J. Biol. Chem. 290, 18559‒18574.
Dai X., Zhang H., Han J., He Y., Zhang Y., Qi Y., Pang J.J. (2016) Effects of subretinal gene transfer at different time points in a mouse model of retinal dege-neration. PLoS One. 11, e0156542.
Okubo M., Yamanaka H., Kobayashi K., Kanda H., Dai Y., Noguchi K. (2012) Up-regulation of platelet-activating factor synthases and its receptor in spinal cord contribute to development of neuropathic pain following peripheral nerve injury. Mol. Pain. 8, 8.
Zhao T., Zhang Y., Ma X., Wei L., Hou Y., Sun R., Jiang J. (2021) Elevated expression of LPCAT1 predicts a poor prognosis and is correlated with the tumour microenvironment in endometrial cancer. Cancer Cell Int. 21, 269.
Han C., Yu G., Mao Y., Song S., Li L., Zhou L., Wang Z., Liu Y., Li M., Xu B. (2020) LPCAT1 enhances castration resistant prostate cancer progression via increased mRNA synthesis and PAF production. PLoS One. 15, e0240801.
Wei C., Dong X., Lu H., Tong F., Chen L., Zhang R., Dong J., Hu Y., Wu G., Dong X. (2019) LPCAT1 promotes brain metastasis of lung adenocarcinoma by up-regulating PI3K/AKT/MYC pathway. J. Exp. Clin. Cancer Res. 38, 95.
Mansilla F., da Costa K., Wang S., Kruhøffer M., Lewin T., Orntoft T., Coleman R., Birkenkamp-Demtröder K. (2009) Lysophosphatidylcholine acyltransferase 1 (LPCAT1) overexpression in human colorectal cancer. J. Mol. Med. (Berl.) 87, 85‒97.
Deribe Y.L., Pawson T. Dikic I. (2010) Post-translational modifications in signal integration. Nat. Struct. Mol. Biol. 17, 666.
Schwartz A. Ciechanover A. (2009) Targeting proteins for destruction by the ubiquitin system: implications for human pathobiology. Annu. Rev. Pharmacol. Toxicol. 49, 73‒96.
Jones D., Crowe E., Stevens T.A. Candido E.P.M. (2001) Functional and phylogenetic analysis of the ubiquitylation system in Caenorhabditis elegans: ubiquitin-conjugating enzymes, ubiquitin-activating enzymes, and ubiquitin-like proteins. Genome Biol. 3(1), RESEARCH0002. https://doi.org/10.1186/gb-2001-3-1-research0002
Wójcik C., DeMartino G.N. (2003) Intracellular loca-lization of proteasomes. Int. J. Biochem. Cell Biol. 35, 579‒589.
Song Y., Wu C., Wu K., Han Q., Miao X., Ma D., Leung C. (2021) Ubiquitination regulators discovered by virtual screening for the treatment of cancer. Front. Cell Dev. Biol. 9, 665646.
Xu Y., Wang C., Jiang X., Zhang Y., Su H., Jiang J., Ren H., Qiu X. (2021) KLHL38 involvement in non-small cell lung cancer progression via activation of the Akt signaling pathway. Cell Death Dis. 12, 556.
Castro-Gonzalez S., Simpson S., Shi Y., Chen Y., Benjamin J., Serra-Moreno R. (2021) HIV Nef-mediated ubiquitination of BCL2: implications in autophagy and apoptosis. Front. Immunol. 12, 682624.
Liu J., Li X., Zhou G., Zhang Y., Sang Y., Wang J., Li Y., Ge W., Sun Z., Zhou X. (2021) Silica nanoparticles inhibiting the differentiation of round spermatid and chromatin remodeling of haploid period via MIWI in mice. Environ. Pollut. 284, 117446.
Gong Z., Li A., Ding J., Li Q., Zhang L., Li Y., Meng Z., Chen F., Huang J., Zhou D., Hu R., Ye J., Liu W., You H. (2021) OTUD7B deubiquitinates LSD1 to go-vern its binding partner specificity, homeostasis, and breast cancer metastasis. Adv. Sci. (Weinh.). 8(15), e2004504.
Shmuel-Galia L., Humphries F., Lei X., Ceglia S., Wilson R., Jiang Z., Ketelut-Carneiro N., Foley S., Pechhold S., Houghton J., Muneeruddin K., Shaffer S., McCormick B., Reboldi A., Ward D., Marshak-Rothstein A., Fitzgerald K. (2021) Dysbiosis exacerbates colitis by promoting ubiquitination and accumulation of the innate immune adaptor STING in myeloid cells. Immunity. 54, 1137‒1153.e8.
Lepore A., Choy P., Lee N., Carella M., Favicchio R., Briones-Orta M., Glaser S., Alpini G., D’Santos C., Tooze R., Lorger M., Syn W., Papakyriakou A., Giamas G., Bubici C., Papa S. (2021) Phosphorylation and stabilization of PIN1 by JNK promote intrahepatic cho-langiocarcinoma growth. Hepatology. 74(5), 2561‒2579.
Kapur R., Semple J. (2016) Alleviation of gram-negative bacterial lung inflammation by targeting HECTD2. Ann. Trans. Med. 4, 488.
Coon T., McKelvey A., Lear T., Rajbhandari S., Dunn S., Connelly W., Zhao J., Han S., Liu Y., Weathington N., McVerry B., Zhang Y., Chen B. (2015) The proinflammatory role of HECTD2 in innate immunity and experimental lung injury. Sci. Trans. Med. 7, 295ra109.
Sun T., Wang X., He H., Sweeney C., Liu S., Brown M., Balk S., Lee G., Kantoff P. (2014) MiR-221 promotes the development of androgen independence in prostate cancer cells via downregulation of HECTD2 and RAB1A. Oncogene. 33, 2790‒2800.
Li J.H., Liu S., Zhou H., Qu L.H. Yang J.H. (2014) starBase v2.0: decoding miRNA-ceRNA, miRNA-ncRNA and protein-RNA interaction networks from large-scale CLIP-Seq data. Nucleic Acids Res. 42, D92–D97.
Uhlen M., Oksvold P., Fagerberg L., Lundberg E., Jonasson K., Forsberg M., Zwahlen M., Kampf C., Wes-ter K., Hober S., Wernerus H., Bjorling L., Ponten F. (2010) Towards a knowledge-based Human Protein Atlas. Nat. Biotechnol. 28, 1248‒1250.
Uehara T., Kikuchi H., Miyazaki S., Iino I., Setoguchi T., Hiramatsu Y., Ohta M., Kamiya K., Morita Y., Tanaka H., Baba S., Hayasaka T., Setou M., Konno H. (2016) Overexpression of lysophosphatidylcholine acyltransferase 1 and concomitant lipid alterations in gastric cancer. Ann. Surg. Oncol. 23(Suppl. 2), S206‒S213.
Shida-Sakazume T., Endo-Sakamoto Y., Unozawa M., Fukumoto C., Shimada K., Kasamatsu A., Ogawara K., Yokoe H., Shiiba M., Tanzawa H., Uzawa K. (2015) Lysophosphatidylcholine acyltransferase1 overexpression promotes oral squamous cell carcinoma progression via enhanced biosynthesis of platelet-activating factor. PLoS One. 10, e0120143.
Grupp K., Sanader S., Sirma H., Simon R., Koop C., Prien K., Hube-Magg C., Salomon G., Graefen M., Heinzer H., Minner S., Izbicki J., Sauter G., Schlomm T., Tsourlakis M. (2013) High lysophosphatidylcholine acyltransferase 1 expression independently predicts high risk for biochemical recurrence in prostate cancers. Mol. Oncol. 7, 1001‒1011.
Liu F., Wu Y., Liu J., Ni R.J., Yang A.G., Bian K., Zhang R. (2020) A miR-205‒LPCAT1 axis contributes to proliferation and progression in multiple cancers. Biochem. Biophys. Res. Commun. 527, 474‒480.
Lebok P., von Hassel A., Meiners J., Hube-Magg C., Simon R., Hoflmayer D., Hinsch A., Dum D., Fraune C., Gobel C., Moller K., Sauter G., Jacobsen F., Buscheck F., Prien K., Krech T., Krech R.H., von der Assen A., Wolber L., Witzel I., Schmalfeldt B., Geist S., Paluchoswski P., Wilke C., Heilenkotter U., Terracciano L., Muller V., Wilczak W., Burandt E.C. (2019) Up-regulation of lysophosphatidylcholine acyltransferase 1 (LPCAT1) is linked to poor prognosis in breast cancer. Aging. 11, 7796‒7804.
Du Y., Wang Q., Zhang X., Wang X., Qin C., Sheng Z., Yin H., Jiang C., Li J., Xu T. (2017) Lysophosphatidylcholine acyltransferase 1 upregulation and concomitant phospholipid alterations in clear cell renal cell carcinoma. J. Exp. Clin. Cancer Res. 36, 66.
Hu J., Ding X., Tian S., Chu Y., Liu Z., Li Y., Li X., Wang G., Wang L., Wang Z. (2021) TRIM39 deficiency inhibits tumor progression and autophagic flux in colorectal cancer via suppressing the activity of Rab7. Cell Death Dis. 12, 391.
Li B., Qi Z.P., He D.L., Chen Z.H., Liu J.Y., Wong M.W., Zhang J.W., Xu E.P., Shi Q., Cai S.L., Sun D., Yao L.Q., Zhou P.H., Zhong Y.S. (2021) NLRP7 deubiquitination by USP10 promotes tumor progression and tumor-associated macrophage polarization in colorectal cancer. J. Exp. Clin. Cancer Res. 40, 126.
Erokhov P.A., Kulikov A.M., Karpova Y.D., Rodoman G.V., Sumedi I.R., Goncharov A.L., Razbirin D.V., Gorelova V.S., Sharova N.P., Astakhova T.M. (2021) Proteasomes in patient rectal cancer and diffe-rent intestine locations: where does proteasome pool change? Cancers (Basel). 13, 1108.
Boland K., Flanagan L., McCawley N., Pabari R., Kay E.W., McNamara D.A., Murray F., Byrne A.T., Ramtoola Z., Concannon C.G., Prehn J.H. (2016) Targe-ting the 19S proteasomal subunit, Rpt4, for the treatment of colon cancer. Eur. J. Pharmacol. 780, 53‒64.
Дополнительные материалы
- скачать ESM_1.doc
- Supplementary Table 1. Sequences of siRNA Against Specific Targets
Supplementary Table 2. Sequences of PCR primers used in this study
Инструменты
Молекулярная биология


