Молекулярная биология, 2023, T. 57, № 1, стр. 109-123
Инсуляторный белок Cp190 регулирует экспрессию генов дифференцировки сперматоцитов в клетках мужского зародышевого пути Drosophila melanogaster
С. Е. Романов a, b, В. В. Шлома a, Д. Е. Коряков a, С. Н. Белякин a, П. П. Лактионов a, b, *
a Институт молекулярной и клеточной биологии Сибирского отделения Российской академии наук
630090 Новосибирск, Россия
b Новосибирский национальный исследовательский государственный университет
630090 Новосибирск, Россия
* E-mail: laktionov@mcb.nsc.ru
Поступила в редакцию 23.06.2022
После доработки 04.08.2022
Принята к публикации 15.08.2022
- EDN: AWRYNA
- DOI: 10.31857/S0026898423010147
Аннотация
Белок CP190 – один из ключевых компонентов инсуляторных комплексов дрозофилы, изучение которого важно для понимания механизмов регуляции генов в процессе дифференцировки клеток. Однако мутанты по гену Cp190 погибают, не доживая до взрослого состояния, что существенно усложняет изучение функций этого гена в клетках взрослого организма. Чтобы преодолеть эту проблему и исследовать регуляторные эффекты CP190 в развитии тканей имаго, мы разработали систему условного спасения мутантов Cp190. Спасающая конструкция, содержащая кодирующую последовательность гена Cp190, направленно удаляется при помощи Cre/loxP-опосредованной рекомбинации только в половых клетках семенников, что позволяет изучать эффекты мутации в клетках мужского зародышевого пути. С использованием высокопроизводительного транскриптомного анализа установлено, что инактивации CP190 влияет на экспрессию генов в клетках зародышевого пути. Обнаружено, что мутация Cp190 противоположным образом влияет на тканеспецифичные гены, экспрессия которых подавляется CP190, и гены домашнего хозяйства, активатором которых выступает CP190. Мутация Cp190 также приводила к активации экспрессии субпопуляции генов дифференцировки сперматоцитов, которые регулируются транскрипционным комплексом tMAC. Полученные результаты указывают на то, что основной функцией CP190 в процессе сперматогенеза является координация взаимодействий между генами дифференцировки и специфичными для них транскрипционными активаторами.
ВВЕДЕНИЕ
Специализация клеток многоклеточного организма сопровождается активацией “генов развития” под воздействием энхансеров [1], а ключевую роль в регуляции промотор–энхансерных взаимодействий играют инсуляторные белки [2]. Они образуют физические контакты между специальными участками ДНК-инсуляторами, и тем самым обеспечивают пространственные взаимодействия промоторов и цис-регуляторных элементов. Сходным образом инсуляторные белки блокируют неспецифичные промотор–энхансерные взаимодействия и устанавливают барьеры между соседними доменами на хромосомах.
У Drosophila melanogaster обнаружена почти дюжина специализированных белков, которые в различных комбинациях связываются с инсуляторами [3–6]. Один из ключевых – белок CP190, взаимодействует с большинством известных инсуляторов. Белок CP190 участвует в формировании физических ДНК-контактов, регуляции трехмерной архитектуры ядра, а также в блокировке промотор-энхансерных взаимодействий [7–9]. В ряде случаев CP190 выступает в роли транскрипционного фактора и привлекает к генам-мишеням регуляторные белки и эпигенетические модификаторы [6, 10–12].
Потеря функции CP190 сопровождается нарушениями транскрипции, которые, в частности, затрагивают гомеозисные гены, кодирующие ключевые регуляторы развития [8, 13, 14]. Нуль-мутации гена Cp190 являются рецессивными леталями, приводящими к гибели D. melanogaster на стадии куколки [15, 16], что затрудняет исследование регуляторных эффектов CP190 в клетках имаго. По этой причине влияние этого белка на экспрессию генов и морфологию ядра изучали либо на культурах клеток, либо на ранних стадиях развития [8, 13], что накладывает ряд ограничений при интерпретации результатов.
В нашей работе в качестве модельной системы клеточной дифференцировки мы использовали сперматогенез D. melanogaster. В ходе дифференцировки клеток мужского зародышевого пути под воздействием специализированных транскрипционных факторов tMAC и tTAF активируются более 1000 специфичных генов [17, 18]. При этом лишь небольшая доля геномных сайтов связывания tMAC и tTAF находится в непосредственной близости от генов-мишеней [18]. Это может предполагать регуляцию генов посредством сети пространственных взаимодействий между регуляторными элементами генома при участии инсуляторных белков. Чтобы изучить влияние CP190 на регуляцию генов в процессе клеточной дифференцировки, разработана генетическая система, которая позволяет избирательно исследовать проявления летальных мутаций Cp190 в клетках зародышевого пути взрослых самцов. С использованием этого подхода мы оценили влияние белка CP190 на экспрессию генов в семенниках и проанализировали его влияние на регуляцию генов дифференцировки сперматоцитов.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Молекулярное клонирование и трансформация дрозофилы. Спасающая конструкция (GenBank ON783212), содержащая кодирующую последовательность гена Cp190 под контролем промотора гена Ubi63E, создана на основе вектора pUAST-attB, как описано в разделе “Результаты исследования”. С использованием этой конструкции проведен phiC31-опосредованный трансгенез линии y–w–;P{y[+t7.7] = CaryP}attP40;M{vas-int.B}ZH-102D, несущей сайт интеграции attP40 во второй хромосоме, в результате чего получили линию y1w67;CP190-mCD8-GFP [19].
Линии мух. Все линии D. melanogaster содержали при температуре 25°С на стандартном корме. В работе использовали следующие линии: y1w67;CP190-mCD8-GFP, несущую спасающую конструкцию; y1w67;nanos-Cre, содержащую трансген nanos-Cre, интегрированный в сайт attP40 [18, 20]; y1w67;+/+;Cp1902,e1/TM6 [15] и y1w67;+/+;Cp1903,e1/TM6 [16], содержащие мутантные аллели Cp190. Стандартными генетическими скрещиваниями получены линии y1w67;CDSCP190;Cp1902,e1/TM6 и y1w67;nanos-Cre;Cp1903,e1/TM6. Для проведения Cre/loxP-опосредованной рекомбинации скрещивали самок, несущих спасающую конструкцию, с самцами, несущими конструкцию nanos-Cre. При проведении контрольных экспериментов в рамках транскриптомного анализа использовали линию y1w67;{Ubi>stop>mCD8-GFP}, несущую трансген {Ubi>stop>mCD8-GFP} в сайте attP40 [20].
Выделение РНК и обратная транскрипция. Для анализа экспрессии в целых семенниках диссектировали 25 пар семенников и выделяли РНК с использованием реагента TRIZOL (“Invitrogen”, CША, 15596018). кДНК нарабатывали, используя 1 мкг суммарной РНК, обратную транскрипцию проводили с помощью Superscript II Reverse Transcriptase (“Invitrogen”, 18080093) c использованием dT-олигонуклеотидов в качестве затравки.
Количественная ПЦР. Количественную ПЦР проводили с использованием реакционной смеси БиоМастер HS-qPCR SYBR Blue (“Биолабмикс”, Россия, MHC030). Последовательности использованных праймеров приведены в табл. S1 (см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/1/supp_ Romanov_rus.pdf). Эффективность удаления спасающей кассеты оценивали при помощи метода ΔΔCt, экспрессию CP190 измеряли методом стандартной кривой. Для нормирования использовали ген Actin42A. Стандартную кривую строили, используя серийные разведения образцов геномной ДНК личиночных имагинальных дисков самок у1w67;CP190-mCD8-GFP.
Микроскопия. Для анализа флуоресценции GFP in vivo семенники переносили на предметное стекло в каплю PBS, накрывали покровным стеклом и сразу анализировали на флуоресцентном микроскопе. Семенники для иммуноокрашивания фиксировали стандартным методом (2%-ный раствор формальдегида в PBST) [21]. В работе использовали первичные антитела крысы к CP190 (любезно предоставлены А.К. Головниным) [22] и вторичные антитела (anti-Rat Cross-Adsorbed A568, “Thermo Fisher Scientific”, США, A-11077). Препараты анализировали на конфокальном микроскопе Сarl Zeiss LSM 710 (Германия).
Анализ транскриптома методом РНК-seq. В эксперименте использовали семенники самцов генотипов y1w67;CP190-mCD8-GFP/nanos-Cre и y1w67;CP190-mCD8-GFP/nanos-Cre;Cp1902/Cp1903. Контролем служили семенники самцов y1w67;Ubi>stop>mCD8-GFP/nanos-Cre с репортерной конструкцией Ubi>stop>mCD8-GFP, интегрированной в сайт attP40 во второй хромосоме [20]. Суммарную РНК выделяли из 25 пар семенников с использованием реагента TRIZOL. Пробоподготовку библиотек проводили с использованием набора TruSeq RNA Sample Preparation v2 Kit (“Illumina”, США, RS-121-2002). Секвенирование библиотек проводили на платформе Illumina MiSeq в режиме парных прочтений 2 × 75 п.н. Получено от 3.7 до 6.5 млн парных прочтений на образец.
Геномное выравнивание осуществляли с помощью инструмента HiSat2 [23] (сборка генома dm6, Release 6 plus ISO1 MT, Aug. 2014) со стандартными параметрами. Количество прочтений для каждого гена суммировано при помощи FeatureCounts [24] с использованием геномной аннотации UCSC refGene (https://hgdownload.soe.ucsc.edu/goldenPath/dm6/bigZips/genes/dm6.refGene.gtf.gz). Использовали от двух до трех биологических повторов эксперимента с каждым генотипом; профили экспрессии генов в образцах разных генотипов показали высокую корреляцию (рис. S1 , см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/1/supp_Romanov_rus.pdf). Дифференциальную экспрессию генов анализировали при помощи алгоритма DESeq2 [25] с параметрами “ashr” [26] и “ihv” [27]. Данные по экспрессии генов в целых семенниках мутантов can, comr, mip40, bam получены ранее и доступны в базе данных Gene Expression Omnibus (GSE97182) [18]. Данные по экспрессии генов в тканях дрозофилы получены из базы modENCODE [28] и кластеризованы при помощи программы Cluster 3.0 [29]. Обогащение тканеспецифичными генами анализировали при помощи пакета TissueEnrich [30]. Данные секвенирования транскриптома единичных клеток семенников получены из базы Fly Cell Atlas и проанализированы с использованием программного пакета Seurat [31, 32]. Результаты секвенирования доступны в базе данных Gene Expression Omnibus (PRJNA847720).
Связывание Can, Comr и Mip40 c CP190-зависимыми генами. Данные по связыванию белков Can, Comr и Mip40 с хромосомами клеток мужского зародышевого пути получены ранее при помощи метода DamID-seq (GSE97182) [18]. Сайты связывания белков картировали с использованием набора программ DamID-Seq [33]. Все гены в геноме были разделены на заданное число фрагментов. Обогащение связывания белков в области генов исследуемой выборки оценивали, сопоставляя частоту встречаемости центров GATC-фрагментов, связанных белком (согласно данным DamID-seq), в каждом фрагменте гена, со средним значением по всем генам D. melanogaster. Статистическую значимость обогащения оценивали с помощью точного теста Фишера.
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ
Система условного спасения обеспечивает жизнеспособность мутантов Cp190
Мутанты по гену, кодирующему инсуляторный белок CP190, не доживают до взрослой стадии [15, 16], поэтому исследование функции этого гена в тканях имаго D. melanogaster сопряжено с экспериментальными трудностями. Чтобы преодолеть эту проблему и установить регуляторную функцию CP190 в клетках зародышевого пути взрослых самцов, мы разработали генетическую систему для тканеспецифичного условного спасения мутации Cp190 (рис. 1а).
Рис. 1.
Генетическая система для условного спасения мутантов с потерей функции Cp190. а – Схема спасающей конструкции Cp190-mCD8-GFP и конструкции nanos-Cre для экспрессии рекомбиназы Cre в клетках зародышевого пути дрозофилы. б – Принципиальная схема условного спасения мутантов Cp190 2/Cp1903 (см. пояснения в тексте). в – Снимки семенников самцов, не несущих спасающую конструкцию, с одной дозой спасающей конструкции и с условным спасением мутации Cp190. Режим фазового контраста (верхний ряд) и детекция флуоресценции GFP (нижний ряд). Желтым пунктиром обозначены границы семенников. Условные обозначения: Ст.т – хвосты сперматид; Ст – головы сперматид; Тр – дыхательные трубочки; Сг – область семенника, содержащая сперматогонии (непрерывная линия); Сц – область семенника, содержащая сперматоциты (пунктирная линия).
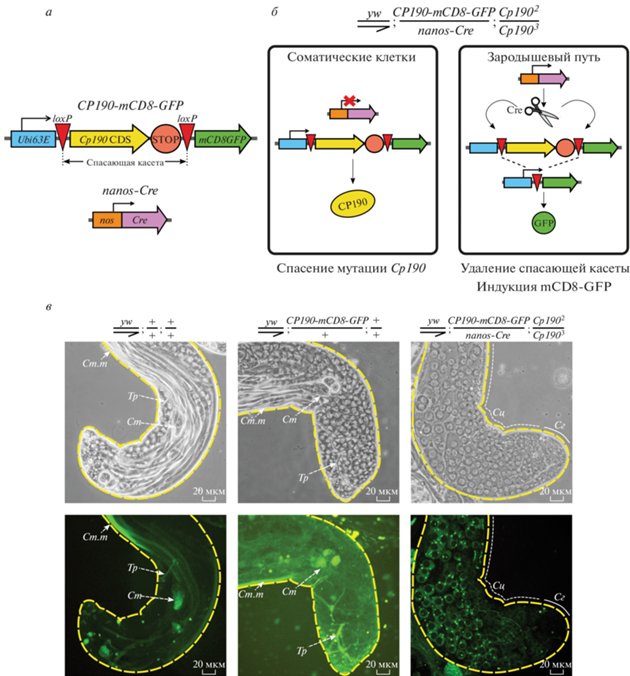
Система состоит из двух генетических конструкций. Первая – спасающая конструкция CP190-mCD8-GFP – содержит кодирующую последовательность гена Cp190 под контролем конститутивного промотора гена Ubi63E, который успешно использовали ранее для спасения летальной мутации Cp190 [34, 35]. Вслед за кодирующей последовательностью Cp190 помещен гибридный терминатор транскрипции HIS3-SV40 и кодирующая последовательность гена химерного репортерного белка mCD8-GFP [36, 37]. Кодирующая последовательность Cp190 и терминатор транскрипции фланкированы loxP-сайтами узнавания рекомбиназы Cre и образуют спасающую генную кассету (рис. 1а). Вторая конструкция содержит ген рекомбиназы Cre под управлением промотора гена nanos, что обеспечивает наработку белка исключительно в клетках зародышевого пути (рис. 1а) [18, 20]. При наличии обеих конструкций в организме мутантов Cp190 спасающая кассета будет специфически удалена из клеток зародышевого пути рекомбиназой Cre. В соматических клетках спасающая кассета не вырезается, обеспечивая жизнеспособность организма. При удалении кассеты в клетках зародышевого пути кодирующая последовательность гена химерного репортерного белка mCD8-GFP сближается с промотором гена Ubi63E, что позволяет получить клетки с дефицитом Cp190, помеченные GFP (рис. 1б).
С использованием системы phiC31-опосредованного трансгенеза генетическая конструкция CP190-mCD8-GFP была встроена в сайт attP40 во второй хромосоме [19]. Трансгенные мухи как с одной, так и с двумя копиями конструкции жизнеспособны и фертильны (данные не показаны). При этом одной копии оказалось достаточно для спасения летального фенотипа компаунд-гетерозигот Cp190 2/Cp1903, у которых не образуется функциональный белок CP190 [15, 16]. Одновременно с этим экспрессия одной или двух копий спасающей конструкции не вызывала аномалий развития как в комбинации с Cp1902/Cp1903, так и на фоне дикого типа. Это свидетельствует об умеренном уровне эктопической экспрессии CP190, поскольку сильное повышение экспрессии этого гена вызывает серьезные дефекты развития [34].
В серии генетических скрещиваний получены самцы, несущие конструкции CP190-mCD8-GFP и nanos-Cre на фоне мутации Cp190 (y1w67; CP190-mCD8-GFP/nanos-Сre; Cp1902/Cp1903). В клетках зародышевого пути таких самцов выявлена активация репортерного белка mCD8-GFP, начиная со стадии ранних сперматоцитов, что свидетельствовало об удалении спасающей генной кассеты (рис. 1в). Такие самцы обладали нормальной жизнеспособностью и не имели видимых нарушений сперматогенеза. GFP-негативные сперматоциты и GFP-позитивные соматические клетки оболочки визуально не обнаружены, что указывает на высокую эффективность и специфичность удаления спасающей конструкции.
В контрольных образцах семенников y1w67, а также семенников самцов, несущих спасающую конструкцию в отсутствие Cre-рекомбиназы, наблюдалась только фоновая автофлуоресценция, характерная для дыхательных трубочек и фрагментов сперматид (рис. 1в). Это подтверждает надежное блокирование экспрессии репортерного белка во всех типах клеток семенника.
Дополнительная проверка эффективности удаления спасающей кассеты из генома клеток зародышевого пути при помощи количественной ПЦР показала, что спасающая конструкция сохраняется в 49.6% клеток целого семенника (рис. 2а). Это значение соответствует доле соматических клеток в семеннике (50.4%), оцененной исходя из данных секвенирования транскриптома единичных клеток семенника [31]. Этот результат свидетельствует о том, что спасающая кассета удаляется в подавляющем большинстве половых клеток.
Рис. 2.
Cre-опосредованная рекомбинация приводит к удалению спасающей кассеты и утрате CP190 в клетках мужского зародышевого пути. а – Количество копий спасающей кассеты в семенниках c условным спасением сравнивали с количеством в имагинальных дисках личинок, несущих двойную дозу конструкции CP190-mCD8-GFP. Число копий спасающей кассеты нормировали на дозу конструкции. Пунктирной линией обозначена доля соматических клеток в семенниках дрозофилы по данным секвенирования транскриптома единичных клеток [31]. б – Расположение мишеней для ОТ-кПЦР в гене Cp190. Соответствующие праймеры использованы для измерения экспрессии эндогенного и трансгенного Cp190. в – Нормированная экспрессия Cp190 в семенниках трехдневных имаго с конструкциями CP190-mCD8-GFP и nanos-Cre. Экспрессию измеряли при помощи праймеров к локусам, обозначенным на диаграмме б – Экспрессию нормировали на активность гена домашнего хозяйства Actin42A. Величина P-value получена с помощью теста Стьюдента с двусторонним распределением. г – Конфокальные изображения семенников с иммуноокрашиванием антителами анти-CP190. В верхнем ряду – семенник дикого типа (y w). В нижнем ряду – семенник с условным спасением. ДНК окрашивали красителем Hoechst. Непрерывной желтой линией подчеркнуты границы семенников. Звездочкой отмечен апикальный конец семенника. Длинной стрелкой показаны соматические клетки оболочки. Пунктирной линией показано скопление CP190-негативных сперматоцитов. Короткой стрелкой показан CP190-позитивный сперматоцит. Масштаб 10 мкм.
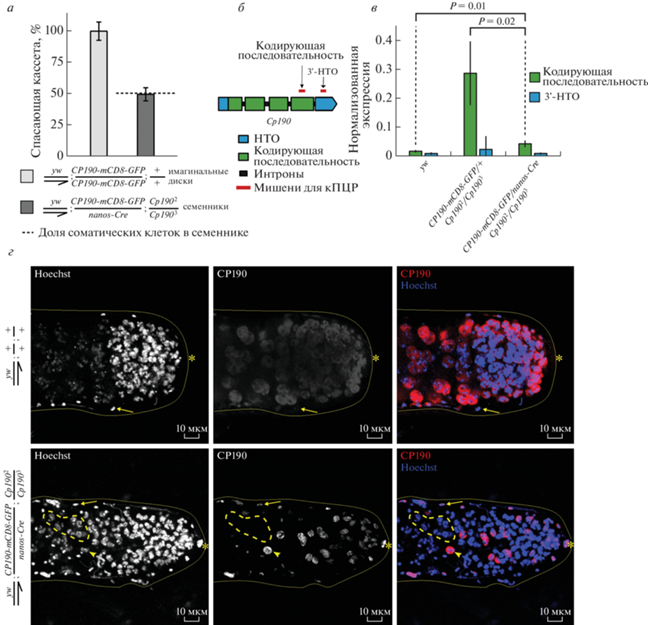
Система условного спасения значительно снижает уровень эктопической экспрессии Cp190 в клетках мужского зародышевого пути имаго
Мы проанализировали уровни экспрессии мРНК эндогенного и трансгенного гена Cp190 методом количественной ПЦР с праймерами на кодирующую область гена Cp190 и 3′-нетранслируемую область эндогенного Cp190 (рис. 2б). Первая пара праймеров позволяет измерить уровень экспрессии и эндогенного, и трансгенного Cp190, а вторая пара – только эндогенной мРНК Cp190. Установлено, что удаление спасающей кассеты под воздействием nanos-Cre приводило к снижению экспрессии кодирующей последовательности Сp190 в целых семенниках в 3.8 раза (рис. 2в). Вместе с тем, уровень экспрессии кодирующей последовательности Cp190 оставался в 2.4 раза выше по сравнению с контролем дикого типа, что, по всей видимости, можно объяснить повышенным (в сравнении с эндогенным) уровнем экспрессии трансгенного Cp190 в соматических клетках, а также сохранением спасающей конструкции в единичных сперматоцитах (рис. 2г).
Получение мутантных по Cp190 клеток зародышевого пути подтверждено иммуноокрашиванием (рис. 2г). В семенниках y1w67;CP190-mCD8-GFP/nanos-Cre;Cp1902/Cp1903 белок CP190 обнаружен в ядрах клеток на апикальном конце семенника, где располагаются недифференцированные сперматогонии, а также в ядрах соматических клеток оболочки. Вместе с тем, экспрессия CP190 наблюдалась лишь в единичных ядрах сперматоцитов. В семенниках дикого типа стабильный уровень экспрессии CP190 наблюдался как в соматических, так и в половых клетках. Таким образом, разработанная система обеспечивает спасение мутантного фенотипа Cp190 в соматических клетках, а также достаточно эффективное удаление спасающей кассеты в клетках мужского зародышевого пути, что на фоне мутации позволяет изолированно воспроизвести мутантный фенотип Cp190 в половых клетках взрослых самцов.
Мутация Cp190 влияет на экспрессию генов дифференцировки в клетках мужского зародышевого пути
Влияние CP190 на экспрессию генов в семенниках имаго оценивали с помощью высокопроизводительного секвенирования транскриптома (РНК-seq) и последующего анализа дифференциальной экспрессии (табл. S2 , см. Дополнительные материалы на сайте http://www.molecbio.ru/downloads/2023/1/supp_Romanov_rus.zip). В экспериментах использовали трехнедельных самцов, так как в этом возрасте семенники y1w67;CP190-mCD8-GFP/nanos-Cre и y1w67;CP190-mCD8-GFP/nanos-Cre;Cp1902/Cp1903 имеют эквивалентный уровень экспрессии Cp190 (рис. S2 , см. Дополнительные материалы на сайте http:// www.molecbio.ru/downloads/2023/1/supp_Romanov_ rus.zip). Важно отметить, что активность спасающей кассеты контролируется промотором гена Ubi63E, из-за чего уровень транскрипции Cp190 превышает эндогенный (рис. 2в), что может влиять на экспрессию генов. Чтобы аккуратно учесть этот эффект в нашем эксперименте, мы оценили влияние повышенной экспрессии Cp190 в соматических клетках на транскриптом целых семенников.
С этой целью мы измерили экспрессию генов в семенниках самцов y1w67;CP190-mCD8-GFP/nanos-Cre;+/+, в которых экспрессируется эндогенный функциональный ген Cp190, а спасающая кассета активна только в соматических клетках. Для сравнения использовали экспрессию генов в семенниках самцов контрольной линии. Присутствие спасающей конструкции привело к умеренным изменениям транскрипции: экспрессия 12 генов изменялась более чем в 8 раз (рис. 3а). Анализ семенников с условным спасением мутации Cp190 в соматических клетках (y1w67;CP190-mCD8-GFP/nanos-cre;Cp1902/Cp1903) выявил 89 генов, чья экспрессия изменялась более чем в 8 раз в сравнении с контролем (рис. 3а).
Рис. 3.
Истощение CP190 в клетках мужского зародышевого пути сопровождается активацией семенник-специфичных генов и инактивацией генов домашнего хозяйства. а – Дифференциальная экспрессия генов в семенниках с повышенной экспрессией CP190 в соматических клетках (диаграмма слева) или в семенниках с условным спасением (диаграмма справа) по результатам РНК-seq. По вертикальной оси отложен логарифмированный диапазон изменения экспрессии (Log2FoldChange, LFC) трансгенных семенников по сравнению с контролем. По горизонтальной оси отложена нормированная средняя экспрессия в семенниках контрольной линии. Гены со статистически значимым изменением экспрессии (|LFC| > 1, P < 0.05) помечены красным цветом. Значения LFC генов с незначимым изменением экспрессии (серые точки, N.S.) были для наглядности усечены при помощи эмпирического байесовского подхода. Пунктирными линиями указаны значения LFC ± 3 и 0. Красным показано число красных точек со значением LFC в интервале между ближайшими пунктирными линиями. б – Пересечение между множествами генов, экспрессия которых значимо повысилась и понизилась в семенниках с условным спасением или в семенниках с повышенной экспрессией CP190 в соматических клетках (красные точки на диаграмме а. Гены, экспрессия которых изменилась только на фоне условного спасения, обозначены как Набор № 1 и Набор № 2. в – Нормированная экспрессия генов из Наборов № 1 и № 2 в разных тканях дрозофилы по данным modENCODE [28]. г – Анализ тканеспецифичной экспрессии CP190-зависимых генов в клетках мужского зародышевого пути.
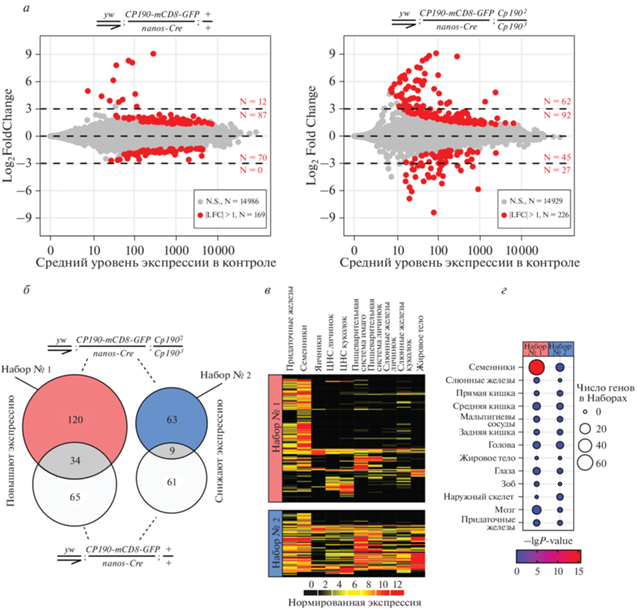
Кроме того, из 226 генов, дифференциально экспрессирующихся на фоне условного спасения мутации Cp190 (экспрессия 154 генов повысилась, 72 – понизилась), транскрипция 43 изменялась и на фоне повышенной экспрессии Сp190 (у 34 генов повысилась, у 9 снизилась) (рис. 3б). Эти 43 гена были исключены из дальнейшего анализа, направленного на выделение специфического эффекта мутации в гене Cp190 на экспрессию генов в клетках зародышевого пути.
После этой поправки обнаружили 120 генов, чья экспрессия усиливалась на фоне мутации Cp190 (Набор № 1), и 63 гена, уровень экспрессии которых снижался (Набор № 2) (рис. 3б). Чтобы охарактеризовать эти выборки, мы проанализировали профили экспрессии входящих в них генов в разных тканях с использованием транскриптомных данных базы modENCODE [28]. Оказалось, что в Наборе №1 можно выделить три основных кластера, представленных генами, активными в мужской репродуктивной, нервной и пищеварительной системах (рис. 3в). Гены же Набора № 2 экспрессируются повсеместно и лишь 14 из них можно причислить к семенник-специфичным. Анализ паттернов экспрессии генов в различных тканях дрозофилы с использованием программы TissueEnrich [30] подтвердил преобладание семенник-специфичных генов в Наборе № 1, но не в Наборе № 2 (рис. 3г). Таким образом, белок CP190 может участвовать как в поддержании экспрессии повсеместно активных генов в семенниках, так и в репрессии (или модуляции) активности тканеспецифичных генов, в том числе и генов, участвующих в сперматогенезе.
Вместе с тем, необходимо учитывать, что клеточный состав семенника достаточно разнообразен, он представлен несколькими типами соматических клеток, а также клетками зародышевого пути, находящимися на разных стадиях дифференцировки. Различные клеточные типы имеют характерные отличия в профиле экспрессии, что подтверждается данными секвенирования транскриптома единичных клеток семенников. Охарактеризованные профили экспрессии отдельных клеточных популяций позволяют выделить специфичные гены-маркеры, а также установить, в каких клетках активен тот или иной ген в норме.
Чтобы определить, в каких конкретно клетках семенника в норме активны Cp190-зависимые гены, мы использовали гены-маркеры различных клеточных популяций семенников дрозофилы из базы данных DRscDB [31, 38]. В Наборе № 1 обнаружены лишь единичные гены-маркеры соматических клеток, тогда как маркеры сперматоцитов в сумме составляли 10% (рис. 4а). В Наборе № 2 маркерами жировых и пигментных клеток были 22% генов, маркеры же клеток зародышевого пути были единичными (рис. 4а). Кластеризация выборки генов Набора № 1 по уровню экспрессии в разных клеточных популяциях семенника с использованием базы данных транскриптома единичных клеток Fly Cell Atlas [31] позволила установить, что транскрипция 66% генов характерна для сперматоцитов (рис. 4б). Не менее 60% экспрессии таких генов обеспечивается клетками зародышевого пути, что позволяет отнести большинство генов Набора № 1 к генам дифференцировки сперматоцитов (рис. 4б). Напротив, лишь пятую часть генов Набора № 2 можно идентифицировать как гены дифференцировки сперматоцитов, а их общее количество в 5.7 раза меньше, чем в Наборе № 1. Все вместе это позволяет предположить, что CP190 оказывает преимущественно репрессорное воздействие по отношению к генам дифференцировки сперматоцитов.
Рис. 4.
Утрата CP190 в клетках мужского зародышевого пути приводит к нарушению активности генов дифференцировки сперматоцитов. а – Обогащение генов-маркеров клеточных типов в Наборах № 1 и № 2 по данным DRscDB [38]. Клеточные типы аннотированы в эксперименте по секвенированию транскриптома единичных клеток [31], в некоторых случаях они включают несколько субпопуляций с собственным набором генов-маркеров. Принадлежность субпопуляций клеточным типам обозначена цветными прямоугольниками. Размер кругов характеризует представленность маркеров во всем наборе. Статистическая значимость обогащения вычислена в точном тесте Фишера. Маркеры со значимым обогащением обведены красным. б – Экспрессия генов из Наборов № 1 и № 2 в клеточных популяциях семенника по данным Fly Cell Atlas [31]. Принадлежность популяций клеточным типам и анатомическим структурам в семеннике закодирована цветными прямоугольниками. Цвет точек характеризует уровень экспрессии гена в сравнении со средней экспрессией в клетках семенника. Размер точек описывает долю клеток в популяции, в которых обнаружены транскрипты данного гена. На столбчатой диаграмме показан вклад клеток зародышевого пути и соматических клеток в общую транскрипцию каждого гена в целом семеннике. При помощи иерархической кластеризации по уровням экспрессии в популяциях клеток гены в каждом наборе разделены на два кластера. В Наборе № 1 отчетливо выделяются гены с высокой активностью в сперматоцитах (гены дифференцировки сперматоцитов). В Наборе № 2 кластеры можно охарактеризовать как гены соматической специализации и гены дифференцировки сперматоцитов. в – Сравнение оценок изменения экспрессии в двух кластерах из Набора 2. Оценки диапазона изменений экспрессии (Log2FoldChange) получены отдельно для генов дифференцировки сперматоцитов (красный цвет) и генов соматической специализации (синий цвет) из предположения, что в семенниках с условным спасением инактивация произошла только в клетках зародышевого пути (темно-серые прямоугольники) или только в соматических клетках (светло-серые прямоугольники). Наблюдаемые значения Log2FoldChange показаны белыми прямоугольниками. Условные обозначения: ***P < 5 × 10–8 в тесте Вилкоксона, N.S. – P > 0.05.
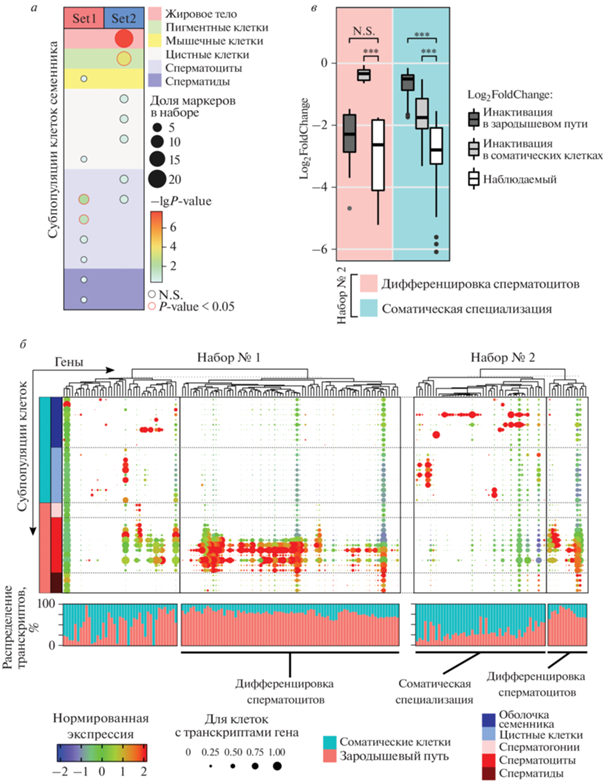
Основная часть генов из Набора № 2 в норме активна преимущественно в соматических клетках семенников (рис. 4б). Вместе с тем, оставался открытым вопрос, изменяется ли уровень экспрессии этих генов в клетках зародышевого пути на фоне истощения Cp190. С использованием данных о транскриптомах единичных клеток [31], позволяющих оценить вклад отдельной клеточной популяции семенников в уровень экспрессии генов, мы смоделировали два профиля экспрессии, соответствующих образцам целых семенников, в одном профиле исследуемые гены были инактивированы в клетках зародышевого пути, а в другом – в соматических клетках. На следующем этапе мы сопоставили реальные изменения экспрессии генов Набора № 2 с моделями инактивации генов в отдельных клеточных популяциях. Инактивация генов, активных преимущественно в соматических клетках, только в соматических или только в половых клетках не позволяет смоделировать такое же сильное падение транскрипции, какое мы наблюдали в семенниках с условным спасением (P < 5 × 10–8 в тесте Вилкоксона) (рис. 4в). Напротив, оценка генов дифференцировки сперматоцитов, полученная на основе гипотезы об инактивации в зародышевом пути, не отличается от реальных значений (P = 0.23 в тесте Вилкоксона) (рис. 4в). Такое наблюдение может указывать на то, что падение транскрипции генов Набора № 2, характерных для соматических клеток, происходит как в половых, так и в соматических клетках.
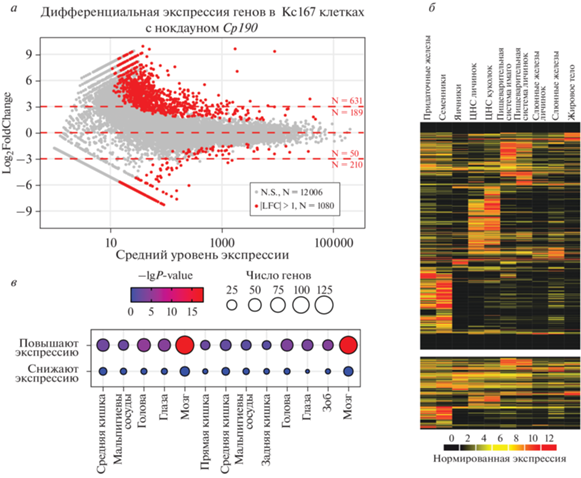
Интересно отметить, что схожие регуляторные эффекты CP190 обнаружены и в культуре соматических клеток дрозофилы. С использованием полученных ранее данных транскриптомного анализа клеток Kc167 на фоне инактивации Cp190 посредством РНК-интерференции мы обнаружили повышение экспрессии 820 генов и снижение только 260 (рис. 5а) [13]. Активирующиеся гены также представлены преимущественно генами, специфичными для семенников, нервной ткани и пищеварительной системы (рис. 5б, в). Гены же, экспрессия которых снижалась на фоне истощения Cp190, имели неспецифичные профили экспрессии (рис. 5б, в).
Рис. 5.
CP190 подавляет активность тканеспецифичных генов и способствует активации генов домашнего хозяйства в клетках Кс167. а – Эффект инактивации Cp190 в клетках Kc167 согласно данным эксперимента [13]. Дифференциальную экспрессию анализировали таким же способом, как на рис. 3а. б – Экспрессия CP190-зависимых генов (красные точки на диаграмме а) в тканях дрозофилы согласно данным modENCODE [28]. в – Анализ тканеспецифичной экспрессии CP190-зависимых генов клеток Kc167.
Мутация Cp190 нарушает экспрессию генов-мишеней tMAC
Активация генов дифференцировки происходит скоординировано в фазе роста сперматоцитов первого порядка. Описано несколько мутаций, которые нарушают этот процесс [17]. Так, мутация гена bam вызывает остановку сперматогенеза на стадии сперматогониев, в результате чего семенники обогащаются слабо дифференцированными клетками зародышевого пути [39]. Гены mip40, comr и can являются генами задержки мейоза, они кодируют транскрипционные факторы, а их мутации приводят к остановке сперматогенеза на стадии первичных сперматоцитов [17].
Нарушения в работе Mip40 и Comr, которые входят в состав семенник-специфичного комплекса задержки мейоза tMAC, вызывают драматичные изменения экспрессии генов в семенниках [17, 18]. Нарушения же в работе белков из группы семенник-специфичных факторов транскрипции tTAF, включая Can, вызывают менее выраженные изменения в транскриптоме семенников. Вместе tTAF и tMAC обеспечивают активацию программы дифференцировки сперматоцитов, причем только часть генов регулируется этими факторами напрямую [18].
Учитывая связь CP190 с генами дифференцировки, мы предположили функциональное взаимодействие между CP190, tTAF и tMAC. Ранее мы получили профили экспрессии генов в семенниках мутантов bam, mip40, comr и can, а также картировали сайты связывания Mip40, Comr и Can с хромосомами в клетках зародышевого пути [18]. Мы проанализировали экспрессию генов, которые активируются в семенниках с условным спасением (Набор № 1), в семенниках дикого типа и мутантов bam, mip40, comr и can (рис. 6а).
Рис. 6.
Делеция CP190 в клетках мужского зародышевого пути приводит к нарушению активности tMAC-зависимых генов дифференцировки сперматоцитов. а – Экспрессия генов из двух кластеров Набора № 1 в семенниках дикого типа и мутантов с нарушением дифференцировки клеток мужского зародышевого пути. Цветными прямоугольниками показано на каких стадиях сперматогенеза останавливается дифференцировка клеток в семенниках каждого генотипа. Гены mip40 и comr кодируют компоненты семенник-специфичного транскрипционного активатора tMAC. Ген can кодирует один из белков tTAF. Статистическая значимость отличий экспрессии генов в семенниках мутантов и дикого типа установлена в тесте Вилкоксона. б – Обогащение Набора № 1 генами, вокруг которых в хромосомах клеток зародышевого пути самцов в разных участках связываются транскрипционные факторы Can, Comr и Mip40 [18]. Статистическая значимость обогащения измерена с помощью точного теста Фишера. Обозначения: TSS – сайт начала транскрипции; TES – сайт завершения транскрипции. в – То же, что в а, для генов из двух кластеров Набора № 2. г – То же, что в б, для генов из Набора № 2.
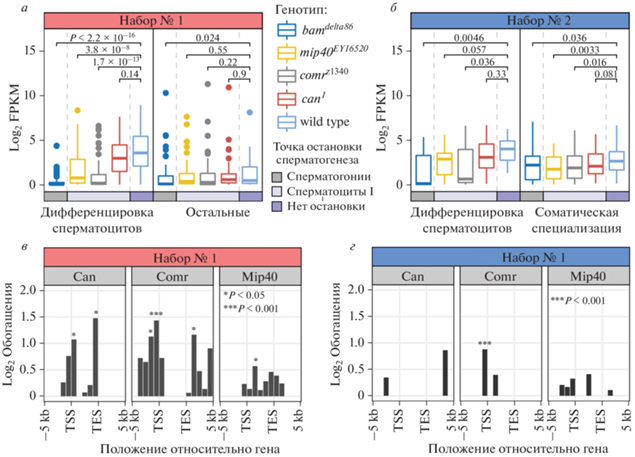
Активность генов дифференцировки сперматоцитов из Набора № 1 (рис. 4б) значительно снижена у мутантов bam, mip40 и comr, но не у мутантов can (рис. 6а). Остальные гены из Набора № 1 почти не реагируют на мутации mip40, comr и can, только мутация bam приводит к незначительному снижению их экспрессии, что можно объяснить присутствием среди них генов с высокой активностью в сперматоцитах (рис. 4б). Таким образом, CP190 ограничивает активность tMAC-зависимых генов дифференцировки сперматоцитов.
Профили распределения Comr, Mip40 и Can на хромосомах показывают, что эти белки чаще ожидаемого связываются с генами из Набора № 1, причем наиболее интенсивно связывается транскрипционный фактор Comr (рис. 6б). Таким образом, мутация Cp190 затрагивает активность генов, непосредственно регулируемых комплексом tMAC.
Для экспрессии генов Набора № 2 характерны менее выраженные различия между мутантами bam, mip40, comr и can, и диким типом (рис. 6в), хотя и у мутантов с нарушениями в tMAC, и у мутантов bam снижается экспрессия генов дифференцировки сперматоцитов и генов, характерных для соматических клеток. Гены из Набора № 2 имеют некоторую связь с транскрипционным фактором Comr (рис. 6г), что указывает на возможность прямой активации их экспрессии комплексом tMAC, и белок CP190 участвует в этом процессе.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
В этой работе мы подробно изучили влияние белка CP190 на активность генов в клетках зародышевого пути D. melanogaster. Как оказалось, основное влияние мутация Cp190 оказывает на субпопуляцию генов дифференцировки сперматоцитов, которые регулируются транскрипционным комплексом tMAC. Интересно, что в отсутствие белка CP190 активность этих генов усиливается в 2 раза и более, что позволяет сделать вывод о модуляции их активности этим белком в норме, ограничивающим уровень их экспрессии.
В целом, на культурах клеток дрозофилы показаны умеренные изменения транскриптома на фоне истощения инсуляторного белка CP190 [8, 13, 40]. Нами также выявлены лишь 183 гена, экспрессия которых зависит от CP190 в сперматоцитах. Экспрессия 120 из этих генов повышалась на фоне мутации Cp190, а 63 – снижалась, что говорит в пользу превалирующей репрессорной функции CP190. На культурах клеток показано также, что CP190, как и другие инсуляторные белки, в большей степени склонен к подавлению транскрипции, несмотря на одновременную активаторную функцию [13, 41].
Мы обнаружили, что CP190 противоположным образом влияет на активность тканеспецифичных генов и генов домашнего хозяйства, что указывает на возможность существования нескольких механизмов, с помощью которых CP190 контролирует активность этих генов. Можно предположить, что CP190 способен выступать в качестве транскрипционного фактора для генов, активация которых зависит от CP190. Это косвенно подтверждается тем, что для активации CP190-связанных генов требуется кооперативное действие CP190 и фактора M1BP – регулятора генов домашнего хозяйства [13]. Причем промоторы генов, экспрессия которых снижается в клетках с нокдауном, обогащены CP190, в то время как репрессорный эффект CP190, по всей вероятности, реализуется не напрямую [13].
Важно отметить, что активация генетической программы дифференцировки клеток мужского зародышевого пути сопровождается значительной перестройкой архитектуры хроматина [42]. Так, на стадии сперматоцитов уменьшается количество дистальных контактов, а промоторы активных генов дифференцировки оказываются в изолированном окружении [42]. Показано участие CP190, наряду с белком Chro, в формировании и поддержании топологически ассоциированных доменов (ТАД) [41]. Причем обнаружено, что нарушение экспрессии генов в результате удаления CP190 коррелирует с изменением границ окружающего их ТАД [41]. Более того, ТАДы, содержащие гены домашнего хозяйства, имеют тенденцию к более высокой стабильности [41]. Это позволяет предположить, что наблюдаемое дифференциальное влияние CP190 на экспрессию тканеспецифичных генов и генов домашнего хозяйства может быть обусловлено различиями в механизмах регуляции на уровне структуры хроматина, в результате чего гены домашнего хозяйства активируются под действием CP190, тогда как тканеспецифичные гены подавляются.
Учитывая отсутствие влияния на дифференцировку клеток мужского зародышевого пути, нормальную фертильность мутантных самцов, а также умеренный эффект на изменение паттерна экспрессии генов, инсуляторный белок CP190 вряд ли можно рассматривать в качестве ключевого регуляторного фактора сперматогенеза. Для сравнения, мутация сomr (tMAC) затрагивает экспрессию более 2500 генов и вызывает задержку мейоза [43]. Однако с учетом ключевой роли CP190 в регуляции архитектуры ядра можно предположить, что недостаточность CP190 приводит к сбоям в коммуникации специфичных факторов транскрипции и их регуляторных элементов, а также к более общим нарушениям структуры хроматина, что вызывает нарушения в механизмах тонкой регуляции экспрессии.
В ходе исследования мы обнаружили, что нарушение экспрессии CP190 приводит к инактивации tMAC-зависимых генов дифференцировки сперматоцитов. Примечательно, что в соматических клетках дрозофилы функционирует гомологичный tMAC репрессорный комплекс dREAM – партнер CP190, основная функция которого состоит в регуляции генов дифференцировки и клеточного цикла [44–47]. Взаимодействие CP190 и dREAM в культурах клеток играет важную роль в управлении парами дивергентных генов – близлежащих генов с противоположно направленными промоторами [46]. Привлечение dREAM к дивергентным генам дрозофилы происходит за счет CP190 и позволяет добиваться снижения экспрессии только одного гена в паре. Наличие у dREAM и tMAC общих субъединиц (например, Mip40 и Caf1), а также связь между CP190 и tMAC-зависимыми генами позволяет предположить, что CP190 участвует в привлечении tMAC к хромосомам. Дальнейшие исследования позволят объяснить, почему основной эффект истощения CP190 в клетках мужского зародышевого пути заключается в дерегуляции генов дифференцировки сперматоцитов.
Авторы работы выражают благодарность А.К. Головнину (ИБГ РАН) за предоставленные антитела, А.В. Пиндюрину (ИМКБ СР РАН) за помощь в клонировании, а также ЦКП “Молекулярная и клеточная биология” за предоставленное оборудование.
Исследование выполнено при финансовой поддержке Российского фонда фундаментальных исследований в рамках научных проектов № 19-34-90108, 17-00-00181, 20-34-70141 и 19-04-00872, а также при поддержке Программы фундаментальных научных исследований FWGZ-2021-0017 (122011900429-5).
Условия содержания Drosophila melanogaster соответствовали нормативам, указанным в Приказе Минздрава России № 267 от 19.06.2003 “Об утверждении правил лабораторной практики в Российской Федерации”.
Авторы заявляют об отсутствии конфликта интересов.
Список литературы
Long H.K., Prescott S.L., Wysocka J. (2016) Ever-changing landscapes: transcriptional enhancers in development and evolution. Cell. 167, 1170–1187.
Kyrchanova O., Georgiev P. (2014) Chromatin insulators and long-distance interactions in Drosophila. FEBS Lett. 588, 8–14.
Yang J., Corces V.G. (2011) Chromatin insulators: a role in nuclear organization and gene expression. In: Advances in Cancer Res. Ed. Woude G.W. 110. London: Acad. Press, 43–76.
Nègre N., Brown C.D., Shah P.K., Kheradpour P., Morrison C.A., Henikoff J.G., Feng X., Ahmad K., Russell S., White R.A.H., Stein L., Henikoff S., Kellis M., White K.P. (2010) A comprehensive map of insulator elements for the Drosophila genome. PLoS Genet. 6, e1000814.
Maksimenko O., Bartkuhn M., Stakhov V., Herold M., Zolotarev N., Jox T., Buxa M.K., Kirsch R., Bonchuk A., Fedotova A., Kyrchanova O., Renkawitz R., Georgiev P. (2015) Two new insulator proteins, Pita and ZIPIC, target CP190 to chromatin. Genome Res. 25, 89–99.
Zolotarev N., Maksimenko O., Kyrchanova O., Sokolinskaya E., Osadchiy I., Girardot C., Bonchuk A., Ciglar L., Furlong E.E.M., Georgiev P. (2017) Opbp is a new architectural/insulator protein required for ribosomal gene expression. Nucl. Acids Res. 45, 12285–12300.
Cuartero S., Fresán U., Reina O., Planet E., Espinàs M.L. (2014) Ibf1 and Ibf2 are novel CP190-interacting proteins required for insulator function. EMBO J. 33, 637–647.
Schwartz Y.B., Linder-Basso D., Kharchenko P.V., Tolstorukov M.Y., Kim M., Li H.-B., Gorchakov A.A., Minoda A., Shanower G., Alekseyenko A.A., Riddle N.C., Jung Y.L., Gu T., Plachetka A., Elgin S.C.R., Kuroda M.I., Park P.J., Savitsky M., Karpen G.H., Pirrotta V. (2012) Nature and function of insulator protein binding sites in the Drosophila genome. Genome Res. 22, 2188–2198.
Kaushal A., Dorier J., Wang B., Mohana G., Taschner M., Cousin P., Waridel P., Iseli C., Semenova A., Restrepo S., Guex N., Aiden E. L., Gambetta M.C. 2022. Essential role of Cp190 in physical and regulatory boundary formation. Sci. Adv. 8, eabl8834.
Sabirov M., Kyrchanova O., Pokholkova G.V., Bonchuk A., Klimenko N., Belova E., Zhimulev I.F., Maksimenko O., Georgiev P. (2021) Mechanism and functional role of the interaction between CP190 and the architectural protein Pita in Drosophila melanogaster. Epigenet. Chromatin. 14, 16.
Bohla D., Herold M., Panzer I., Buxa M.K., Ali T., Demmers J., Krüger M., Scharfe M., Jarek M., Bartkuhn M., Renkawitz R. (2014) A functional insulator screen identifies NURF and dREAM components to be required for enhancer-blocking. PLoS One. 9, e107765.
Ali T., Krüger M., Bhuju S., Jarek M., Bartkuhn M., Renkawitz R. (2017) Chromatin binding of Gcn5 in Drosophila is largely mediated by CP190. Nucl. Acids Res. 45, 2384–2395.
Bag I., Chen S., Rosin L.F., Chen Y., Liu C.-Y., Yu G.-Y., Lei E.P. (2021) M1BP cooperates with CP190 to activate transcription at TAD borders and promote chromatin insulator activity. Nat. Commun. 12, 4170.
Savitsky M., Kim M., Kravchuk O., Schwartz Y.B. (2016) Distinct roles of chromatin insulator proteins in control of the Drosophila bithorax complex. Genetics. 202, 601–617.
Butcher R.D.J., Chodagam S., Basto R., Wakefield J.G., Henderson D.S., Raff J.W., Whitfield W.G.F. (2004) The Drosophila centrosome-associated protein CP190 is essential for viability but not for cell division. J. Cell Sci. 117, 1191–1199.
Oliver D., Sheehan B., South H., Akbari O., Pai C.Y. (2010) The chromosomal association/dissociation of the chromatin insulator protein Cp190 of Drosophila melanogaster is mediated by the BTB/POZ domain and two acidic regions. BMC Cell Biol. 11, 101.
White-Cooper H. (2010) Molecular mechanisms of gene regulation during Drosophila spermatogenesis. Reproduction. 139, 11–21.
Laktionov P.P., Maksimov D.A., Romanov S.E., Antoshina P.A., Posukh O.V., White-Cooper H., Koryakov D.E., Belyakin S.N. (2018) Genome-wide analysis of gene regulation mechanisms during Drosophila spermatogenesis. Epigenetics Chromatin. 11, 14.
Markstein M., Pitsouli C., Villalta C., Celniker S.E., Perrimon N. (2008) Exploiting position effects and the gypsy retrovirus insulator to engineer precisely expressed transgenes. Nat. Genet. 40, 476–483.
Лактионов П.П., Максимов Д.А., Андреева Е.Н., Шлома В.В., Белякин С.Н. (2013) Генетическая система для мечения соматических и герминальных клеточных линий в гонадах Drosophila melanogaster. Цитология. 55, 185–189.
Solovei I., Cremer M. (2010) 3D-FISH on cultured cells combined with immunostaining. In: Methods Mol. Biol. 659. Eds Bridger J.M., Volpi E.M. New York: Humana Press, 117–126.
Golovnin A., Volkov I., Georgiev P. (2012) SUMO conjugation is required for the assembly of Drosophila Su(Hw) and Mod(mdg4) into insulator bodies that facilitate insulator complex formation. J. Cell Sci. 125, 2064–2074.
Kim D., Paggi J.M., Park C., Bennett C., Salzberg S.L. (2019) Graph-based genome alignment and genotyping with HISAT2 and HISAT-genotype. Nat. Biotechnol. 37, 907–915.
Liao Y., Smyth G.K., Shi W. (2014) FeatureCounts: an efficient general purpose program for assigning sequence reads to genomic features. Bioinformatics. 30, 923–930.
Love M.I., Huber W., Anders S. (2014) Moderated estimation of fold change and dispersion for RNA-seq data with DESeq2. Genome Biol. 15, 550.
Stephens M. (2016) False discovery rates: a new deal. Biostatistics. 18, 275–294.
Ignatiadis N., Klaus B., Zaugg J.B., Huber W. (2016) Data-driven hypothesis weighting increases detection power in genome-scale multiple testing. Nat. Meth. 13, 577–580.
Brown J.B., Boley N., Eisman R., May G.E., Stoiber M.H., Duff M.O., Booth B.W., Wen J., Park S., Suzuki A.M., Wan K.H., Yu C., Zhang D., Carlson J.W., Cherbas L., Eads B.D., Miller D., Mockaitis K., Roberts J., Davis C.A., Frise E., Hammonds A.S., Olson S., Shenker S., Sturgill D., Samsonova A.A., Weiszmann R., Robinson G., Hernandez J., Andrews J., Bickel P.J., Carninci P., Cherbas P., Gingeras T.R., Hoskins R.A., Kaufman T.C., Lai E.C., Oliver B., Perrimon N., Graveley B.R., Celniker S.E. (2014) Diversity and dynamics of the Drosophila transcriptome. Nature. 512, 393–399.
de Hoon M.J.L., Imoto S., Nolan J., Miyano S. (2004) Open source clustering software. Bioinformatics. 20, 1453–1454.
Jain A., Tuteja G. (2019) TissueEnrich: tissue-specific gene enrichment analysis. Bioinformatics. 35, 1966–1967.
Li H., Janssens J., De Waegeneer M., Kolluru S.S., Davie K., Gardeux V., Saelens W., David F.P.A., Brbić M., Spanier K., Leskovec J., McLaughlin C.N., Xie Q., Jones R.C., Brueckner K., Shim J., Tattikota S.G., Schnorrer F., Rust K., Nystul T.G., Carvalho-Santos Z., Ribeiro C., Pal S., Mahadevaraju S., Przytycka T.M., Allen A.M., Goodwin S.F., Berry C.W., Fuller M.T., White-Cooper H., Matunis E.L., DiNardo S., Galenza A., O’Brien L.E., Dow J.A.T.; FCA Consortium, Jasper H., Oliver B., Perrimon N., Deplancke B., Quake S.R., Luo L., Aerts S., Agarwal D., Ahmed-Braimah Y., Arbeitman M., Ariss M.M., Augsburger J., Ayush K., Baker C.C., Banisch T., Birker K., Bodmer R., Bolival B., Brantley S.E., Brill J.A., Brown N.C., Buehner N.A., Cai X.T., Cardoso-Figueiredo R., Casares F., Chang A., Clandinin T.R., Crasta S., Desplan C., Detweiler A.M., Dhakan D.B., Donà E., Engert S., Floc’hlay S., George N., González-Segarra A.J., Groves A.K., Gumbin S., Guo Y., Harris D.E., Heifetz Y., Holtz S.L., Horns F., Hudry B., Hung R.J., Jan Y.N., Jaszczak J.S., Jefferis G.S.X.E., Karkanias J., Karr T.L., Katheder N.S., Kezos J., Kim A.A., Kim S.K., Kockel L., Konstantinides N., Kornberg T.B., Krause H.M., Labott A.T., Laturney M., Lehmann R., Leinwand S., Li J., Li J.S.S., Li K., Li K., Li L., Li T., Litovchenko M., Liu H.H., Liu Y., Lu T.C., Manning J., Mase A., Matera-Vatnick M., Matias N.R., McDonough-Goldstein C.E., McGeever A., McLachlan A.D., Moreno-Roman P., Neff N., Neville M., Ngo S., Nielsen T., O’Brien C.E., Osumi-Sutherland D., Özel M.N., Papatheodorou I., Petkovic M., Pilgrim C., Pisco A.O., Reisenman C., Sanders E.N., Dos Santos G., Scott K., Sherlekar A., Shiu P., Sims D., Sit R.V., Slaidina M., Smith H.E., Sterne G., Su Y.H., Sutton D., Tamayo M., Tan M., Tastekin I., Treiber C., Vacek D., Vogler G., Waddell S., Wang W., Wilson R.I., Wolfner M.F., Wong Y.E., Xie A., Xu J., Yamamoto S., Yan J., Yao Z., Yoda K., Zhu R., Zinzen R.P. (2022) Fly Cell Atlas: a single-nucleus transcriptomic atlas of the adult fruit fly. Science. 375, eabk2432.
Hao Y., Hao S., Andersen-Nissen E., Mauck W.M., Zheng S., Butler A., Lee M.J., Wilk A.J., Darby C., Zager M., Hoffman P., Stoeckius M., Papalexi E., Mimitou E.P., Jain J., Srivastava A., Stuart T., Fleming L.M., Yeung B., Rogers A.J., McElrath J.M., Blish C.A., Gottardo R., Smibert P., Satija R. (2021) Integrated analysis of multimodal single-cell data. Cell. 184, 3573–3587. e29.
Maksimov D.A., Laktionov P.P., Belyakin S.N. (2016) Data analysis algorithm for DamID-seq profiling of chromatin proteins in Drosophila melanogaster. Chromosome Res. 24(4), 481‒494.
Akbari O.S., Oliver D., Eyer K., Pai C.-Y.Y. (2009) An entry/gateway cloning system for general expression of genes with molecular tags in Drosophila melanogaster. BMC Cell Biol. 10, 8.
Evans C.J., Olson J.M., Ngo K.T., Kim E., Lee N.E., Kuoy E., Patananan A.N., Sitz D., Tran P., Do M.-T., Yackle K., Cespedes A., Hartenstein V., Call G.B., Banerjee U. (2009) G-TRACE: rapid Gal4-based cell lineage analysis in Drosophila. Nat. Methods. 6, 603–605.
Pindyurin A.V., Pagie L., Kozhevnikova E.N., van Arensbergen J., van Steensel B. (2016) Inducible DamID systems for genomic mapping of chromatin proteins in Drosophila. Nucl. Acids Res. 44, 5646–5657.
Lee T., Luo L. (2001) Mosaic analysis with a repressible cell marker (MARCM) for Drosophila neural development. Trends Neurosci. 24, 251–254.
Hu Y., Tattikota S.G., Liu Y., Comjean A., Gao Y., Forman C., Kim G., Rodiger J., Papatheodorou I., Santos G.D., Mohr S.E., Perrimon N. (2021) DRscDB: A single-cell RNA-seq resource for data mining and data comparison across species. Comput. Struct. Biotechnol. J. 19, 2018–2026.
Gönczy P., Matunis E., DiNardo S. (1997) bag-of-marbles and benign gonial cell neoplasm act in the germline to restrict proliferation during Drosophila spermatogenesis. Development. (Cambridge, England). 124, 4361–4371.
Bartkuhn M., Straub T., Herold M., Herrmann M., Rathke C., Saumweber H., Gilfillan G.D., Becker P.B., Renkawitz R. (2009) Active promoters and insulators are marked by the centrosomal protein 190. EMBO J. 28, 877–888.
Chathoth K.T., Mikheeva L.A., Crevel G., Wolfe J.C., Hunter I., Beckett-Doyle S., Cotterill S., Dai H., Harrison A., Zabet N.R. (2022) The role of insulators and transcription in 3D chromatin organization of flies. Genome Res. 32, 682–698.
Ilyin A.A., Kononkova A.D., Golova A.V., Shloma V.V., Olenkina O.M., Nenasheva V.V., Abramov Y.A., Kotov A.A., Maksimov D.A., Laktionov P.P., Pindyurin A.V., Galitsyna A.A., Ulianov S.V., Khrameeva E.E., Gelfand M.S., Belyakin S.N., Razin S.V., Shevelyov Y.Y. (2022) Comparison of genome architecture at two stages of male germline cell differentiation in Drosophila. Nucl. Acids Res. 50, 3203–3225.
Лактионов П.П., White-Cooper H., Максимов Д.А., Белянкин С.Н. (2014) Транскрипционный фактор Comr играет роль прямого активатора в генетической программе сперматогенеза у D. melanogaster. Молекуляр. биология. 48, 153‒165.https://doi.org/10.1134/S0026893314010087
Lee H., Ohno K., Voskoboynik Y., Ragusano L., Martinez A., Dimova D.K. (2010) Drosophila RB proteins repress differentiation-specific genes via two different mechanisms. Mol. Cell. Biol. 30, 2563–2577.
DeBruhl H., Wen H., Lipsick J.S. (2013) The complex containing Drosophila Myb and RB/E2F2 regulates cytokinesis in a histone H2Av-dependent manner. Mol. Cell. Biol. 33, 1809–1818.
Korenjak M., Kwon E., Morris R.T., Anderssen E., Amzallag A., Ramaswamy S., Dyson N.J. (2014) dREAM co-operates with insulator-binding proteins and regulates expression at divergently paired genes. Nucl. Acids Res. 42, 8939–8953.
Beall E.L., Lewis P.W., Bell M., Rocha M., Jones D.L., Botchan M.R. (2007) Discovery of tMAC: a Drosophila testis-specific meiotic arrest complex paralogous to Myb-Muv B. Genes Dev. 21, 904–919.
Дополнительные материалы
- скачать ESM_1.xlsx
- Приложение 1. Таблица S1. Список использованных праймеров.
- скачать ESM_2.xlsx
- Приложение 2. Таблица S2. Результат анализа дифференциальной экспрессии при помощи инструмента DESeq2.
- скачать ESM_3.tiff
- Приложение 3. Рисунок S1. Тепловая карта с попарными корреляциями Пирсона между образцами RNA-seq.
- скачать ESM_4.tiff
- Приложение 4. Рисунок S2. Нормализованная экспрессия фрагментов Cp190 в семенниках трехнедельных имаго с конструкциями CP190-mCD8-GFP и nanos-Cre.
Инструменты
Молекулярная биология


