Российские нанотехнологии, 2020, T. 15, № 1, стр. 5-16
БИОКОМПЬЮТЕРЫ: РЕШАЕМЫЕ ЗАДАЧИ, СОВРЕМЕННОЕ СОСТОЯНИЕ И ПЕРСПЕКТИВЫ
П. М. Готовцев 1, *, Д. А. Кириллова 1, Р. Г. Василов 1
1 Национальный исследовательский центр “Курчатовский институт”
Москва, Россия
* E-mail: gotovtsevpm@gmail.com
Поступила в редакцию 29.04.2020
После доработки 29.04.2020
Принята к публикации 13.05.2020
Аннотация
Представлен обзор и проведен анализ подходов, направленных на создание вычислительных систем на основе биологических компонентов – биокомпьютеров. Рассмотрены подходы к вычислениям как с использованием молекул ДНК, так и с использованием живых клеток. Проведен анализ каждого из описанных подходов, отмечены основные преимущества и недостатки. На сегодня существует ряд факторов, существенным образом ограничивающих данное направление. В первую очередь, это отсутствие четко выделенных вычислительных задач, на которых было бы показано, что какой-либо из биокомпьютеров обладал бы значительным преимуществом перед современными вычислительными системами. Во-вторых, фундаментальным ограничением является низкая скорость самого процесса вычислений, ограниченная скоростью биохимических реакций. Отмечено, что ДНК-компьютеры обладают рядом интересных особенностей, которые могут стать причиной для их дальнейших исследований и разработок.
ОГЛАВЛЕНИЕ
Введение
1. Вычисления с использованием белковых молекул
2. Компьютеры на основе нуклеиновых кислот
3. Вычисления в отдельных клетках
4. Многоклеточные биокомпьютеры
5. Решаемые задачи
6. Обсуждение
Заключение
ВВЕДЕНИЕ
Термин “биокомпьютер” гораздо чаще ассоциируется с научной фантастикой, чем с наукой. Это связано как с отсутствием законченного устройства со всеми характерными атрибутами современного компьютера, так и с отсутствием единого подхода для создания такой вычислительной машины. Существует значительный объем научных работ, направленных на реализацию вычислений с использованием различных биологических объектов – отдельных ферментов [1, 2], ДНК [3–5] или РНК [6] и даже целых клеток [7–9]. Причинами такого разнообразия подходов являются высокая сложность биологических систем [10, 11] и их способность самостоятельно хранить и перерабатывать информацию [12, 13]. Все эти факторы дают возможность активно искать в живых системах подходы к созданию вычислительных систем на самых разных уровнях организации. Отметим, что существуют многоклеточные системы, способные выступать в роли вычислительных, самой эффективной из которых является мозг [14]. В связи с чем обсуждается использование распределенных вычислений между клетками с различной функциональной нагрузкой [15]. На рис. 1 показано разнообразие подходов в плане размера используемых компонентов, которое изучается и анализируется для создания биологических вычислительных систем.
Рис. 1.
Разные уровни организации живых систем, которые исследуются с целью реализации биовычислений.
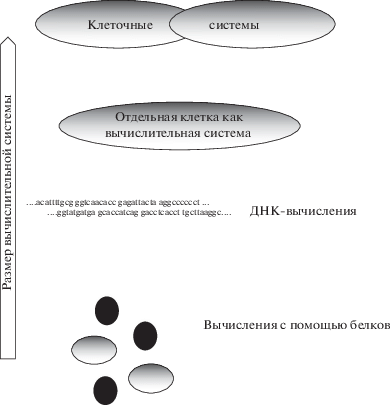
Современный компьютер отличается рядом важнейших особенностей, таких как наличие способов ввода и вывода информации, редактируемой памятью, и самое важное – способностью реализовывать широкий спектр различных алгоритмов [16]. Если говорить о биокомпьютере, то необходимо учитывать все указанные выше элементы, чтобы считать, что речь идет именно о компьютере. В то же время существует большое количество подходов и наработок, демонстрирующих отдельные компоненты, на основе которых в перспективе возможно попробовать создать биокомпьютер. Далее рассмотрим решения, о которых можно говорить как о компьютере, и перспективные подходы, которые могут стать компонентом компьютера. Рассмотрение начнем в соответствии со схемой, представленной на рис. 1, начиная с наименьших по размеру компонентов.
1. ВЫЧИСЛЕНИЯ С ИСПОЛЬЗОВАНИЕМ БЕЛКОВЫХ МОЛЕКУЛ
Белки являются наиболее интересным классом биомолекул для создания вычислительных систем. Множество вариантов структуры, каталитические свойства, способность участвовать в электрохимических процессах дают широкие возможности для поиска и создания компонентов для биокомпьютеров. Однако сложности с предсказанием структуры белков по аминокислотной последовательности [17] в значительной степени тормозили проектирование белковых вычислительных систем. Тем не менее в последнее время появился ряд работ, направленных на создание биологических компонентов, которые могли бы выступать в роли компонентов вычислительных систем и в первую очередь транзисторов.
Возможность использования металлопротеинов в роли транзисторов – одно из перспективных направлений, которое активно обсуждалось в начале 2000-х годов [18, 19]. Так, в [18] показано, что за счет возможности участия в окислительно-восстановительных реакциях азурин может выступать в роли своего рода биотранзистора. Однако эксплуатация такого транзистора возможна лишь в водной среде определенного химического состава, и частота его работы сильно лимитируется скоростью окислительно-восстановительных процессов. Тем не менее такое устройство могло бы обсуждаться для аналоговых цепей, которые вызывают серьезный интерес со стороны синтетической биологии [20].
Антитело, соединенное с двумя наночастицами золота, также демонстрирует свойства, характерные для транзистора [21]. Более того, добавив в систему квантовую точку, авторы продемонстрировали возможность закрытия/открытия разработанного ими биотранзистора по оптическому сигналу.
Каскады ферментативных реакций можно использовать в качестве логических элементов и выстраивать на этом системы логического вывода [2, 5]. На рис. 2 представлены некоторые простейшие варианты реализации логических элементов с помощью ферментативных реакций. Отметим, что таких вариантов много для каждого из звеньев, и целью таблицы является продемонстрировать теоретическую реализуемость данного подхода.
Рис. 2.
Варианты логических элементов на основе ферментативных реакций. E, E1, E2 – ферменты; A, B, C, D, F – интермедиаты.

Элемент “И” может быть представлен в виде простейшей ферментативной реакции биосинтеза либо АТФ-зависимой ферментативной реакцией. Во втором случае фермент получает из вещества А вещество С, затратив аденозинтрифосфат (АТФ), за который можно принять вещество В. Элемент “ИЛИ” можно реализовать с помощью двух реакций, делающих из близких веществ тот же продукт. Элемент И-НЕ можно реализовать так же, как И, только считать продукт D нулем. Либо можно использовать двухферментную систему, где наличие вещества С является выходом, равным единице, а вещества F, равным нулю. Чтобы появилось вещество F, должно образоваться вещество D, которое участвует в ферментативной реакции с C. Для образования вещества D нужны А и В. Как видно из сказанного выше, теоретически такие схемы разрабатываются просто, однако подбор всех компонентов будущей логической цепи является сложной задачей, особенно с учетом обязательного условия по детектируемости выходных значений [2].
Значительным шагом в преодолении указанных сложностей с системами на основе белков стало создание синтетических белковых логических цепей [1]. В данной работе авторы предложили de novo синтезированные небольшие белки, с помощью которых реализовали основные логические элементы. Очевидно, что уже в ближайшем будущем стоит ждать работ с различными логическими схемами на основе данного подхода.
Тем не менее, несмотря на достигнутые успехи и кажущуюся простоту создания логических элементов, полноценного вычислительного устройства на базе белков создано не было.
2. КОМПЬЮТЕРЫ НА ОСНОВЕ НУКЛЕИНОВЫХ КИСЛОТ
Молекула ДНК вызывает значительный интерес в качестве основы для создания вычислительной системы. Во-первых, молекула кодирует в своей первичной структуре информацию и этот код легко считываем. Во-вторых, возможно считывать информацию с одной молекулы сразу в нескольких местах. В-третьих, возможно разрезать молекулу ДНК с помощью рестриктаз по определенным сайтам с образованием липких концов [22]. Таким образом, появляется теоретическая возможность получить гигантскую параллельность вычислений, использовав множество молекул [23, 22 ]. В связи с этим наиболее нашумевшими демонстрациями биовычислительных систем стали ДНК-компьютеры.
Первые работы в данной области опубликованы в 90-х годах, сразу была показана возможность реализации параллельных вычислений теоретических задач, относящихся к NP-полным [24, 25]. Предложенные в ранних работах подходы основаны на активном использовании полимеразно-цепной реакции (ПЦР) и лигаз. Легирование проводилось по липким концам. При этом кодирование выбиралось авторами непосредственно под выбранное ими решение. В данных работах кодирование базировалось на размерах цепочек ДНК различных добавляемых в реакционную смесь молекул. Такой подход был обусловлен единственным доступным способом прочитать итоги вычислений – путем примерного определения количества пар оснований с помощью электрофореза в геле. Добавление рестриктазы FokI или аналогов, способных разрезать ДНК с образованием липких концов, существенно повышало вариативность образующихся молекул и дало возможность вводить данные с использованием более длинных молекул ДНК [26, 27]. Важно отметить, что все вычисления проводятся за счет реакций с молекулами ДНК без проведения процессов транскрипции и трансляции. Таким образом, работа первых ДНК-компьютеров включала в себя следующие стадии:
– разработка системы кодирования и синтез молекул ДНК;
– выбор биохимических подходов к проведению реакций, подбор лигаз и рестриктаз;
– проведение множества параллельных реакций;
– ПЦР и электрофорез для фиксации результата вычислений.
Выбор такой схемы во многом обусловлен тем, что процесс секвенирования, несмотря на появление нанопоровых секвенаторов, до сих пор остается достаточно длительным и трудоемким [28, 29], даже по сравнению с описанными выше стадиями. Кроме того, значительные сложности вызывало направленное редактирование молекул ДНК, комбинация рестрикция с образованием липких концов/лигирование оказалась наиболее простой и доступной большому количеству лабораторий.
В данном подходе молекулы ДНК выступают в роли и памяти, и системы кодирования. Исходными молекулами и/или подбором ферментов задается алгоритм. При этом уже показана возможность реализации ряда классических задач вроде задачи коммивояжера с использованием ДНК-компьютеров [6, 24, 26, 30]. Также показана возможность вычисления квадратного корня, используя бинарные операции с молекулами ДНК [31].
Расширить возможности описанного выше подхода возможно не только за счет модификации системы кодирования, но и за счет использования новых молекулярно-биологических подходов. Так, в [32] комбинируют ПЦР и сайт-направленный мутагенез, при этом авторы выбрали весьма не тривиальный подход к кодированию на основе языка Thue [33]. Данный язык относится к так называемым эзотерическим языкам программирования и по сути представляет собой нулевой тип в иерархии Хомского [34]. Используя подобные нестандартные подходы, авторам удалось добиться возможности проводить процессы во множестве реакционных пробирок, также продемонстрировав возможность решать NP-полные задачи. Кроме того, авторы доказывают, что разработанная ими система может быть отнесена к недетерминированным машинам Тьюринга, категории машин, которые теоретически способны решать NP-полные задачи [35, 36].
Рассмотрим техническую сторону обсуждаемых выше подходов. Серьезным недостатком всех этих систем является значительное время между вводом информации (сбором всех нужных молекул в пробирки) и прочтением результата (проведение гель-электрофореза). Если прибавить к этому синтез необходимых молекул ДНК, т.е. подготовку информации к вводу, то получится, что это время может составлять порядка десяти часов. Отметим, что речь идет об одном цикле вычисления. На сегодня возможны автоматизация и роботизация всех требуемых процессов [37] настолько, что человеку потребуется задать программу для работы всех систем по приготовлению реагентов и проведению реакций, установить емкости с расходными веществами и материалами. Однако это не приведет к существенному сокращению времени работы, так как лимитирующими факторами является само время протекания необходимых реакций. Упрощенно весь процесс вычислений представлен на рис. 3а.
Рис. 3.
Схематичное представление работы ДНК-компьютера: а – при обычном кодировании; б – при кодировании, позволяющем распределить процесс на различные емкости.
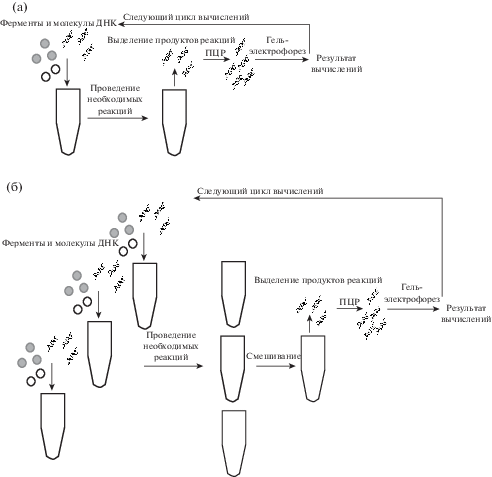
Важной особенностью работы [32] является то, что благодаря выбранной системе кодирования и подходу к проведению ПЦР и сайт-направленного мутагенеза авторам удалось добиться возможности разбивать процесс вычисления на параллельные, не дублирующие друг друга реакции (рис. 3б). Таким образом, процесс вычислений возможно проводить в многолуночных планшетах, проводя параллельно сотни различных реакций. Это приводит к интересной особенности ДНК-компьютера – усложнение задачи приводит не к увеличению времени вычислений, а к увеличению занимаемого для вычислений пространства.
Перспективы развития ДНК-вычислений авторы [32] связывают в первую очередь с адаптацией системы редактирования CRISPR/Cas9 для in vitro-редактирования молекул ДНК для замены или в дополнение к сайт-направленному мутагенезу. Со времени своего появления данная система претерпела ряд усовершенствований, которые существенно повысили ее точность, эффективность применения в различных организмах и расширили возможности по редактированию ДНК [38–40]. В связи с чем можно ожидать развития работ в области создания ДНК-компьютеров, но уже с использованием системы CRISPR/Cas9.
Очевидно, что серьезнейшим недостатком всех описанных выше ДНК-компьютеров является долгое время работы при расчете по одному алгоритму. К этому стоит прибавить постоянный расход реагентов, высокую стоимость автоматизации и роботизации этого процесса. Кроме того, на выходе ответ содержится в молекулах ДНК, следовательно, для его анализа требуется провести ряд действий, чтобы перевести его в электронный вид.
Если говорить о ДНК-компьютерах со всеми атрибутами именно компьютера, о которых было сказано, то на сегодня описанный выше подход является единственным. В то же время существует ряд работ, в которых используются вычислительные особенности молекул ДНК. Так, в [41] показана возможность создания логических элементов с использованием молекул ДНК. В данной работе молекулы иммобилизуются на подложке и проводимые с этими молекулами реакции с использованием АТФ и дополнительных ДНК-сенсоров позволяют реализовывать логический вывод. Важное преимущество этого подхода – выходом является электрический сигнал, который в дальнейшем легко обрабатывать.
Еще одним перспективным направлением является ДНК-нанотехнология – создание различных нанобъектов (оригами, молекулярных машин и т.д.) с использованием молекул ДНК [42]. Теоретически изменяя морфологию ДНК-структур под действием тех или иных внешних факторов и различных ферментов, можно открывать/закрывать доступ к функциональным участкам молекулы и выстраивать на этом вычислительный процесс.
Таким образом, существуют научные заделы в области молекулярной биологии, нанотехнологии и биохимии, которые позволили бы в значительной степени продвинуться в разработке более совершенных ДНК-компьютеров.
3. ВЫЧИСЛЕНИЯ В ОТДЕЛЬНЫХ КЛЕТКАХ
В живых клетках происходит множество процессов, которые можно использовать как базу для проведения вычислений [7, 43, 44]. Хотя в литературе отдельные клетки и особенно нейроны называют компьютерами, но если рассматривать термин компьютер исходя из тех предпосылок, о которых говорилось выше, то очевидно, что говорить о клетке-компьютере преждевременно. Рассмотрим подходы, направленные на создание синтетических систем в клетках, которые могут позволить проводить отдельные вычисления, а возможности использования данных подходов для создания перспективных вычислительных устройств с различными клетками рассмотрим в следующем разделе.
Регулирование работы генов представляет собой прекрасную природную систему, с помощью которой можно проводить различные вычисления [45, 46]. Развитие методов генетической инженерии дало возможность создавать подобные регулируемые системы и генерировать ответ, чаще всего, в виде биосинтеза флуоресцентных белков [47, 49].
Наиболее распространенным подходом является разработка генетических логических цепей [48, 50, 51], которая является важной частью современной синтетической биологии [52]. В основе этого метода лежит возможность регулирования процесса транскрипции за счет использования репрессоров и активаторов, а также процесса трансляции, используя РНК-переключатели [48, 53]. При этом можно последовательно выстраивать каскады реакций, с помощью которых реализовывать стандартные логические операторы [50]. Таким образом, можно реализовать генетическую логическую сеть, благодаря которой клетка сможет делать логическую обработку нескольких входных сигналов (химические вещества, добавленные в среду) и генерировать выходной сигнал за счет биосинтеза [54, 55].
Данный подход широко используется для разработки биосенсоров [15], создания перспективных штаммов-продуцентов [48] и при разработке функциональных материалов, содержащих бактерии [56, 57]. Его главным преимуществом является относительная простота в разработке и реализации за счет использования программного обеспечения для проектирования генетических логических цепей и накопленных больших библиотек биочастей, компонентов для этих цепей [50, 58–60]. Отметим, что в качестве базы для программного обеспечения для разработки генетических логических цепей использовалась та же среда Verilog, которая активно используется в электронике [50].
Процессы в живых организмах носят аналоговый характер, но задача их математического моделирования для последующего создания является более сложной в силу многообразия протекающих в клетках взаимосвязанных процессов [61]. Тем не менее в этой области ведутся многочисленные исследования, а также работы по созданию аналоговых синтетических цепей в клетках [20, 62]. Для моделирования процессов активно используются методы теории автоматического управления и теории аналоговых электрических цепей [20, 63–66].
Все перечисленные в данном разделе подходы являются важной областью синтетической биологии, так как нацелены на создание новых для организма генетических цепей, которые придадут организму новые функции [20, 50]. Все эти подходы основываются на таких инженерных подходах, как теоретические основы электроники и теория автоматического управления.
Отметим, что возможности по интеграции в клетку большого количества новых генетических цепей ограничены. Работа цепи – это постоянный биосинтез, требующий ресурсов и энергии [67, 60 ]. Очевидно, что клетка получает ресурсы из окружающей среды, однако ее способности к их потреблению не бесконечны. Таким образом, даже если клетка находится в богатой питательной среде, то часть потребляемых ею ресурсов будет расходоваться на работу цепи и, следовательно, меньше ресурсов будет использоваться на все остальные внутриклеточные процессы. Вопрос, сколько новых, не связанных с жизнедеятельностью клетки конкретного микроорганизма, генов можно в эту клетку вставить, остается открытым. Сомнительно, что клетку бактерий или дрожжей получится превратить в вычислительную машину с тысячами логических элементов. Более перспективным подходом является создание синтетической клетки, в которой процессы поддержания жизнедеятельности будут связаны с вычислительными и оптимизированы функции транспорта питательных веществ в клетку. При этом специальным образом должна быть подобрана среда, существенно обогащенная аминокислотами. В [68] создана синтетическая бактериальная клетка и продемонстрирована принципиальная возможность синтеза минимального генома de novo и замены в бактерии всей ДНК на синтетическую. Данный эксперимент сопровождался разработкой математической модели, которая описывала внутриклеточные процессы [61]. Таким образом, в перспективе разработка синтетических клеток с использованием стандартных базовых микроорганизмов может быть реализована в соответствии с базовым инженерным подходом: разработка математической модели, ее исследование и только потом работа с клетками.
Другим подходом к использованию модифицированных клеток для вычислений является распределение вычислительных задач между клетками, что приводит к клеточным системам (рис. 2).
4. МНОГОКЛЕТОЧНЫЕ БИОКОМПЬЮТЕРЫ
Высокая эффективность работы мозга вызывает значительный интерес исследователей к поиску биологических энергоэффективных вычислительных систем [14]. Однако при сегодняшнем уровне исследований можно обсуждать только системы на основе различных одноклеточных микроорганизмов.
В природе микроорганизмы образуют биопленки, причем они могут включать в себя множество различных организмов с разными свойствами. В одной биопленке можно обнаружить как фототрофные микроорганизмы вроде микроводорослей, так и различные бактерии и дрожжи, между которыми существуют активные взаимодействия [69–71]. Микроорганизмы синтезируют биополимеры для матрикса и обмениваются химическими сигналами, которые регулируют существование всей биопленки [69, 70]. Свойства биопленок не могли не привлечь внимание биотехнологов. Сегодня как естественно выращенные биопленки, так и системы с иммобилизованными микроорганизмами активно используются в различных областях, от очистки сточных вод до биотехнологического производства [72, 73]. Именно способность микроорганизмов в биопленках обмениваться химическими сигналами, кворум-сенсинг био-пленок, может быть основой для создания клеточных вычислительных систем [15]. В настоящее время активно развиваются подходы в части синтетической биологии для управления биопленками [74].
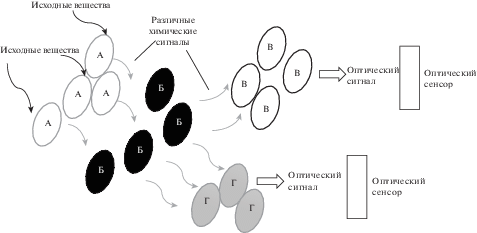
Рисунок 4 схематично показывает общий подход к созданию подобной вычислительной системы. Основа данного подхода – это распределение вычислений между клетками и передача между ними сигнала за счет небольших молекул. Каждая из разновидностей клеток А, Б, В и Г содержит собственные генетические логические цепи, для запуска которых необходимо получение химического сигнала. Все вычисления завершаются в клетках В и Г, выходным сигналом из которых является синтез флюоресцентных белков, что фиксируется оптическим сенсором. В целом возможно реализовать выходной сигнал в виде легко детектируемых химических соединений или, что более перспективно, в виде электрохимического сигнала за счет ферментативных окислительно-восстановительных процессов. Значительный опыт накоплен в области микробных биоэлектрохимических систем, включая биотопливные элементы, с иммобилизацией микроорганизмов на различных электропроводящих материалах [75–79]. Данный опыт позволяет говорить о том, что существуют значительные заделы для регистрации такого рода сигналов.
Рис. 4.
Вариант создания вычислительной системы на основе клеток микроорганизмов с различными генетическими логическими цепями.
Биоэлектрохимические сигналы проще детектировать, они также являются аналоговыми, следовательно, можно перейти к аналоговым вычислениям в клетках. Существует ряд работ, посвященных математическому моделированию микробных биотопливных элементов и получению корреляции между концентрацией субстрата или субстратов и электрическим сигналом [80–82].
5. РЕШАЕМЫЕ ЗАДАЧИ
В [24, 25, 32] было показано, что ДНК-компьютеры теоретически способны решать NP-полные задачи. Многие задачи из этой категории успешно решаются и с помощью существующих вычислительных систем с использованием различных алгоритмических подходов [83–85]. Однако главным преимуществом ДНК-вычислений является то, что с увеличением сложности задачи возможно не увеличивать время вычислений, а увеличивать пространство для вычислений, т.е. количество вовлеченных в вычисление молекул ДНК. Тем не менее анализа теоретических вычислительных ограничений высоко автоматизированного и роботизированного ДНК-компьютера при решении каких-либо задач указанного выше класса не проведено.
Еще одно направление, которое стоит проанализировать с точки зрения использования ДНК-компьютеров, – это эволюционные вычисления и в особенности генетические алгоритмы. Данный вид вычислений становится очень популярным в последние десятилетия для решения целого спектра задач, включая задачи оптимизации [86, 87]. Для работы генетических алгоритмов при решении некоторых задач важную роль играет размер исходной популяции возможных решений [88]. Однако рост популяции повышает требования к аппаратной базе и увеличивает время вычислений даже с учетом подходов, позволяющих проводить параллельные вычисления [89]. При этом необходимо решать многие задачи, опираясь на большие объемы данных [90, 91], что повышает вариативность исходной популяции и ее размер при использовании генетических алгоритмов. Более того, именно анализ больших данных становится одним из самых востребованных направлений в информационных технологиях сегодня. Как уже говорилось выше, повышение сложности задачи при ДНК-вычислениях решается не за счет использования времени, а за счет использования пространства. При реализации на ДНК-компьютере генетического алгоритма первичная популяция решений может быть закодирована в исходных молекулах ДНК, таким образом может быть использована та возможность параллельных вычислений, которая является еще одним важным преимуществом ДНК-компьютеров. Главной сложностью является синтез большого количества различных молекул ДНК, из которых формируется первичная популяция.
В данном обзоре кроме ДНК-компьютеров рассмотрены подходы к созданию логических цепей с использованием реакций различных белков и пептидов или с использованием подходов синтетической биологии. Все перечисленные решения базируются на существующей и активно используемой вычислительной парадигме, поэтому с точки зрения решаемых вычислительных задач никаких новых перспектив они не привносят. Однако могут найти применение в ряде практических областей, о чем будет сказано далее.
6. ОБСУЖДЕНИЕ
Существует множество подходов к созданию биокомпьютеров, которые могут быть реализованы. Однако ни один из них не доведен до создания полноценной вычислительной машины, что объясняется следующими факторами:
– большое время, затрачиваемое на вычисления. Биологические процессы, которые обсуждались выше, гораздо медленнее в сравнении с современными вычислительными машинам. Причем существует нижний предел времени вычислений, преодолеть который невозможно, так как это время, необходимое на проведение базовых биохимических реакций;
– сложность технической реализации, особенно в случае ДНК-компьютеров. Чтобы какой-то из описанных биокомпьютеров работал так, чтобы участие человека ограничивалось вводом данных и получением результатов, необходима высокая степень автоматизации и роботизации для работы с микролитровыми объемами жидкостей;
– высокая стоимость вычислений, так как они требуют либо синтеза ДНК, либо использования чистых химических веществ. Кроме того, ДНК-компьютеры в случае их автоматизации и роботизации требуют значительных энергозатрат;
– отсутствие востребованности решения каких-либо задач, которые не решались бы существующими вычислительными средствами;
– сильнейшая конкуренция со стороны как быстро развивающихся традиционных компьютеров, так и перспективных квантовых компьютеров [92, 93].
Таким образом, развитие биокомпьютеров именно в том понимании термина компьютер, который был отражен во введении, крайне заторможено. Важным стимулом к их развитию может стать формулировка вычислительной задачи или спектра задач, которые были бы трудноразрешимыми или требовали бы значительного количества ресурсов от традиционных компьютеров.
Технические трудности в виде большой и сложной роботизации процессов вычислений для ДНК-компьютеров могут быть преодолены в будущем за счет использования микрофлюидных решений. Данная область последние десятилетия активно развивается, включая создание микрофлюидных ПЦР-систем и устройств ДНК-анализа [94–96]. Таким образом, можно ожидать, что накопленный опыт позволит создать решения и для ДНК-компьютеров, в которых одной из ключевых стадий является проведение ПЦР.
Описанные подходы находят широкое применение в самых разных областях. Так, именно вычислительные свойства генетических логических цепей могут быть использованы в системах распределенных клеточных биосенсоров или биоинтерфейсов [15, 97], в качестве компонента умных материалов, демонстрируя обратную связь в виде цветовой индикации [56, 57], в биомедицине как для создания умных лекарств, так и для создания умных материалов [98–101].
ЗАКЛЮЧЕНИЕ
Биокомпьютеры по-прежнему остаются чем-то редко обсуждаемым даже в научных лабораториях. Основным сдерживающим фактором является отсутствие каких-то узко специфичных вычислительных задач, при решении которых какой-либо из биологических подходов оказался бы более эффективен в сравнении с традиционными вычислительными системами. Однако фундаментальное ограничение по времени вычислений сильно снижает перспективную универсальность биокомпьютеров как средства, которое может быстро дать решение. В то же время исследования в данной области в целом оказали позитивное влияние как на изучение физико-химических свойств ДНК, так и на создание новых подходов в синтетической биологии.
Работа выполнена при поддержке НИЦ “Курчатовский институт” в рамках тематического плана “Разработка технологических решений по созданию бионических имплантируемых сенсорных устройств и метаболических преобразователей энергии”.
Список литературы
Chen Z., Kibler R.D., Hunt A. et al. // Science. 2020. V. 84. P. 78.https://doi.org/10.1126/science.aay2790
Katz E. // Chem. Phys. Chem. 2019. V. 20. № 1. P. 9.https://doi.org/10.1002/cphc.201800900
Qian L., Winfree E. // Science. 2011. V. 332 (6034). P. 1196.https://doi.org/10.1126/science.1200520
Regev A., Shapiro E. // Nature. 2002. V. 419. P. 343.
Katz E. // Curr. Opin. Biotechnol. 2015. V. 34. P. 202.
Faulhammer D., Cukras A.R., Lipton R.J., Landweber L.F. // Proc. National Academy Sciences. 2000. V. 97 (4). P. 1385.https://doi.org/10.1073/pnas.97.4.1385
Castelli L., Pesenti R., Segr D. // IEEE Life Sci. Lett. 2016. V. XX(99). P. 0–3.https://doi.org/10.1109/LLS.2016.2644648
Simpson M.L., Sayler G.S., Fleming J.T., Applegate B. // Trends Biotechnol. 2001. V. 19. P. 317.
Yehl K., Lu T. // Curr. Opin. Biomed. Eng. 2017. V. 4. P. 143.
Oltvai Z.N., Barabási A-L. // Science. 2002. V. 298 (5594). P. 763. https://doi.org/10.1126/science.1078563
Kitano H. // Science. 2002. V. 295. P. 1662.
Westerhoff H.V., Winder C., Messiha H. // FEBS Lett. 2009. V. 583 (24). P. 3882. https://doi.org/10.1016/j.febslet.2009.11.018
Hartwell L.H., Hopfield J.J., Leibler S., Murray A.W. // Nature. 1999. V. 402 (6761). P. S47. https://doi.org/10.1038/35011540
Kovalchuk M. // Nanotechnologies in Russia. 2011. V. 6. № 1–2. P. 1. https://doi.org/10.1134/S1995078011010149
Gotovtsev P.M., Konova I.A. // 2019 International Conference on Sensing and Instrumentation in IoT Era (ISSI). IEEE. 2019. P. 1. https://doi.org/10.1109/ISSI47111.2019.9043737
Turing A.M. // Mind. 1950. V. 59 (236). P. 433.
Senior A.W., Evans R., Jumper J. et al. // Nature. 2020. V. 577 (7792). P. 706. https://doi.org/10.1038/s41586-019-1923-7
Alessandrini A., Salerno M., Frabboni S., Facci P. // Appl. Phys. Lett. 2005. V. 86 (13). P. 1. https://doi.org/10.1063/1.1896087
De Silva A.P., Uchiyama S. // Nature Nanotechnol. 2007. V. 2. P. 399.
Sarpeshkar R. // Philos. Trans. A. 2014. V. 372 (2012). P. 20130110. https://doi.org/10.1098/rsta.2013.0110
Chen Y.S., Hong M.Y., Huang G.S. // Nature Nanotechnology. 2012. V. 7 (3). P. 197. https://doi.org/10.1038/nnano.2012.7
Benenson Y., Paz-Elizur T., Adar R. et al. // Nature. 2001. V. 414 (6862). P. 430. https://doi.org/10.1038/35106533
Livstone M.S., Van Noort D., Landweber L.F. // Trends Biotechnol. 2003. V. 21. P. 98.
Lipton R.J. // Science. 1995. V. 268 (5210). P. 542. https://doi.org/10.1126/science.7725098
Adleman L.M. // Science. 1994. V. 266 (5187). P. 1021. https://doi.org/10.1126/science.7973651
Benenson Y., Adar R., Paz-Elizur T. et al. // Proc. National Academy Sciences. 2003. V. 100 (5). P. 2191. https://doi.org/10.1073/pnas.0535624100
Adar R., Benenson Y., Linshiz G. et al. // Proc. National Academy Sciences. 2004. V. 101 (27). P. 9960. https://doi.org/10.1073/pnas.0400731101
Shendure J., Balasubramanian S., Church G.M. et al. // Nature. 2017. V. 550 (7676). P. 345. https://doi.org/10.1038/nature24286
Kono N., Arakawa K. // Development, Growth Differentiation. 2019. V. 61 (5). P. 316. https://doi.org/10.1111/dgd.12608
Cull P. // Bio Systems 2013. V. 112 (3). P. 196. https://doi.org/10.1016/j.biosystems.2012.12.005
Zhou C., Geng H., Wang P., Guo C. // Small. 2019. V. 15 (49). P. 1903489. https://doi.org/10.1002/smll.201903489
Currin A., Korovin K., Ababi M. et al. // J. Royal Society. Interface. 2017. V. 14 (128). P. 20160990. https://doi.org/10.1098/rsif.2016.0990
McNaughton R., Narendran P., Otto F. // J. ACM. 1988. V. 35 (2). P. 324. https://doi.org/10.1145/42282.42284
Chomsky N. // IRE Trans. Information Theory. 1956. V. 2 (3). P. 113. https://doi.org/10.1109/TIT.1956.1056813
Maass W. // Trans. Am. Mathematical Soc. 1985. V. 292 (2). P. 675. https://doi.org/10.1090/s0002-9947-1985-0808746-4
Galil Z., Kannan R., Szemeredi E. // J. ACM. 1986. P. 39.
Boles K.S., Kannan K., Gill J. et al. // Nature Biotechnol. 2017. V. 35 (7). P. 672. https://doi.org/10.1038/nbt.3859
Jakočiūnas T., Jensen M.K., Keasling J.D. // Metab. Eng. 2015. V. 34. P. 44. https://doi.org/10.1016/j.ymben.2015.12.003
Anzalone A.V., Randolph P.B., Davis J.R. et al. // Nature. 2019. V. 576 (7785). P. 149. https://doi.org/10.1038/s41586-019-1711-4
Simon A.J., D’Oelsnitz S., Ellington A.D. // Nature Biotechnol. 2019. V. 37 (7). P. 730. https://doi.org/10.1038/s41587-019-0157-4
Wei T., Li M., Zhang Y.Y. et al. // Nucl. Sci. Techniq. 2017. V. 28 (3). P. 35. https://doi.org/10.1007/s41365-017-0191-1
Seeman N.C., Sleiman H.F. // Nature Rev. Mater. 2017. V. 3 P. 1. https://doi.org/10.1038/natrevmats.2017.68
Daniel R., Rubens J.R., Sarpeshkar R., Lu T.K. // Nature. 2013. V. 497 (7451). P. 619. https://doi.org/10.1038/nature12148
Atay O., Doncic A., Skotheim J.M. // Cell Systems. 2016. V. 3(2). P. 121. https://doi.org/10.1016/j.cels.2016.06.010
McAdams H.H., Shapiro L. // Science. 1995. V. 269 (5224). P. 650. https://doi.org/10.1126/science.7624793
Lauffenburger D.A. // Proc. National Academy Sciences. 2000. V. 97 (10). P. 5031. https://doi.org/10.1073/pnas.97.10.5031
Collins J.J., Gardner T.S., Cantor C.R. // Nature. 2000. V. 403 (6767). P. 339. https://doi.org/10.1038/35002131
Vaidyanathan P., Der B.S., Bhatia S. et al. // Proc. IEEE. 2015. V. 103 (11). P. 2196. https://doi.org/10.1109/JPROC.2015.2443832
Shin J., Zhang S., Der B.S. et al. // Molecular Systems Biology. 2020. 16 (3). 1. https://doi.org/10.15252/msb.20199401
Nielsen A.K., Der B.S., Shin J. et al. // Science. 2016. V. 352 (6281). P. 53. https://doi.org/10.1126/science.aac7341
Andrews L.B., Nielsen A.K., Voigt C.A. // Science. 2018. V. 361 (6408). eaap8987. https://doi.org/10.1126/science.aap8987
Hanczyc M.M. // Artif. Life. 2020. P. 1. https://doi.org/10.1162/artl_a_00318
Green A.A., Kim J., Ma D. et al. // Nature. 2017. V. 548 (7665). P. 117. https://doi.org/10.1038/nature23271
Tamsir A., Tabor J.J., Voigt C.A. // Nature. 2011. V. 469 (7329). P. 212. https://doi.org/10.1038/nature09565
Clancy K., Voigt C.A. // Curr. Opin. Biotechnol. 2010. V. 21 (4). P. 572. https://doi.org/10.1016/j.copbio.2010.07.005
Smith R.S.H., Bader C., Sharma S. et al. // Adv. Funct. Mater. 2020. V. 30 (7). P. 1907401. https://doi.org/10.1002/adfm.201907401
Moser F., Tham E., González L.M. et al. // Adv. Funct. Mater. 2019. P. 1901788. https://doi.org/10.1002/adfm.201901788
Shetty R.P., Endy D., Knight T.F. // J. Biol. Eng. 2008. V. 2 (1). P. 5. https://doi.org/10.1186/1754-1611-2-5
Shetty R., Lizarazo M., Rettberg R., Knight T.F. // Methods Enzymol. 2011. V. 498. P. 311. https://doi.org/10.1016/B978-0-12-385120-8.00013-9
Konig H., Frank D., Heil R., Coenen C. // Curr. Genomics. 2013. V. 14 (1). P. 11. https://doi.org/10.2174/1389202911314010003
Karr J.R., Sanghvi J.C., MacKlin D.N. et al. // Cell. 2012. V. 150 (2). P. 389. https://doi.org/10.1016/j.cell.2012.05.044
Mishra D., Rivera P.M., Lin A. et al. // Nature Biotechnol. 2014. (November). https://doi.org/10.1038/nbt.3044
Del Vecchio D. // Trends Biotechnol. 2015. V. 33 (2). P. 111. https://doi.org/10.1016/j.tibtech.2014.11.009
Bonnet J., Yin P., Ortiz M.E., Subsoontorn P., Endy D. // Science. 2013. V. 340 (6132). P. 599. https://doi.org/10.1126/science.1232758
Wellstead P., Bullinger E., Kalamatianos D. et al. // Ann. Rev. Control. 2008. V. 32 (1). P. 33. https://doi.org/10.1016/j.arcontrol.2008.02.001
Hori Y., Kim T.-H., Hara S. // Automatica. 2011. V. 47 (6). P. 1203. https://doi.org/10.1016/j.automatica.2011.02.042
Purnick P.E.M., Weiss R. // Nat. Rev. Mol. Cell. Biology. 2009. V. 10 (6). P. 410. https://doi.org/10.1038/nrm2698
Hutchison C.A., Chuang R.-Y., Noskov V.N. et al. // Science. 2016. V. 351 (6280). aad6253–aad6253. https://doi.org/10.1126/science.aad6253
Klapper I., Dockery J. // SIAM Rev. 2010. V. 52 (2). P. 21. https://doi.org/10.1137/080739720
Selvarajoo K. // Front. Microbiol. 2018. V. 9. P. 1721. https://doi.org/10.3389/fmicb.2018.01721
Niu B., Wang H. // Discrete Dynamics Nature Society. 2012. P. 1. https://doi.org/10.1155/2012/698057
Zhang H., Wang H., Jie M. et al. // Bioresour. Technol. 2020. V. 295. P. 122302. https://doi.org/10.1016/j.biortech.2019.122302
Gotovtsev P.M., Yuzbasheva E.Y., Gorin K.V. et al. // Appl. Biochem. Microbiol. 2015. V. 51 (8). P. 792. https://doi.org/10.1134/S0003683815080025
Fang K., Park O.J., Hong S.H. // Biotechnol. Adv. 2020. V. 40. P. 107518.
Jiang Y., Liang P., Liu P. et al. // Biosens. Bioelectron. 2017. V. 94. P. 344. https://doi.org/10.1016/j.bios.2017.02.052
Logan B.E. // Nat. Rev. Microbiol. 2009. V. 7(5). P. 375. https://doi.org/10.1038/nrmicro2113
Santoro C., Arbizzani C., Erable B., Ieropoulos I. // J. Power Sources. 2017. V. 356. P. 225. https://doi.org/10.1016/j.jpowsour.2017.03.109
Antipova K., Parunova Y., Vishnevskaya M. et al. // 2019 12th International Conference on Developments in eSystems Engineering (DeSE). 2019. P. 513. https://doi.org/10.1109/DeSE.2019.00099
Vishnevskaya M., Gazizova D., Victorenko A., Konova I. // IOP Conference Series: Earth and Environmental Science. Institute of Physics Publishing. 2019. https://doi.org/10.1088/1755-1315/337/1/012002
Reshetilov A.N., Plekhanova Y.V., Tarasov S.E. et al. // Appl. Biochem. Microbiol. 2017. V. 53 (1). P. 123. https://doi.org/10.1134/S0003683817010161
Do T.Q.N., Varničić M., Flassig R.J. et al. // Bioelectrochem. 2015. V. 106. Pt. A. P. 3. https://doi.org/10.1016/j.bioelechem.2015.07.007
Su L., Jia W., Hou C., Lei Y. // Biosens. Bioelectron. 2011. V. 26 (5). P. 1788. https://doi.org/10.1016/J.BIOS.2010.09.005
Arabi B.H. // Proceedings – 2016 UKSim-AMSS 18th International Conference on Computer Modelling and Simulation, UKSim. 2016. Institute of Electrical and Electronics Engineers Inc. 2016. P. 43.
Prates M., Avelar P.H.C., Lemos H. et al. // Proc. AAAI Conf. Artificial Intelligence. 2019. V. 33 (01). P. 4731. https://doi.org/10.1609/aaai.v33i01.33014731
Manjunath T.D., Samarth S., Prafulla N., Nayak J.S. // Hopfield Network Based Approximation Engine for NP Complete Problems. Springer, Cham. 2020. P. 319.
Kar A.K. // Expert Systems With Applications. 2016. V. 59. P. 20.
Lee C.K.H. // Eng. Applications Artificial Intelligence. 2018. V. 76. P. 1. https://doi.org/10.1016/j.engappai.2018.08.011
Piotrowski A.P. // Swarm Evolutionary Comput. 2017. V. 32. P. 1. https://doi.org/10.1016/j.swevo.2016.05.003
Alba E., Tomassini M. // IEEE Trans. Evolutionary Comput. 2002. V. 6 (5). P. 443. https://doi.org/10.1109/TEVC.2002.800880
Michael K., Miller K.W. // Computer. 2013. V. 46 (6). P. 22. https://doi.org/10.1109/mc.2013.196
Tannahill B.K., Jamshidi M. // Computers Electrical Eng. 2014. V. 40 (1). P. 2. https://doi.org/10.1016/j.compeleceng.2013.11.016
Harrow A.W., Montanaro A. // Nature. 2017. V. 549. P. 203.
Arute F., Arya K., Babbush R. et al. // Nature. 2019. V. 574 (7779). P. 505. https://doi.org/10.1038/s41586-019-1666-5
Boyd-Moss M., Baratchi S., Di Venere M., Khoshmanesh K. // Lab Chip. 2016. V. 16. P. 3177.
Zhang Y., Jiang H.R. // Anal. Chim. Acta. 2016. V. 914. P. 7.
Bruijns B., van Asten A., Tiggelaar R., Gardeniers H. // Biosensors. 2016. V. 6 (3). P. 41. https://doi.org/10.3390/bios6030041
Gotovtsev P.M., Dyakov A.V. // 2016 IEEE 3rd World Forum on Internet of Things (WF-IoT). IEEE. 2016. P. 542. https://doi.org/10.1109/WF-IoT.2016.7845476
Wagner H.J., Sprenger A., Rebmann B., Weber W. // Adv. Drug Delivery Rev. 2016. V. 105. P. 77.
Chen B., Dai Z. // Quantitative Biology. 2020. https://doi.org/10.1007/s40484-020-0197-2
Scheller L., Fussenegger M. // Curr. Opin. Biotechnol. 2019. V. 58. P. 108.
Hoffman T., Antovski P., Tebon P. et al. // Adv. Funct. Mater. 2020. https://doi.org/10.1002/adfm.201909882
Дополнительные материалы отсутствуют.
Инструменты
Российские нанотехнологии


