Поверхность. Рентгеновские, синхротронные и нейтронные исследования, 2022, № 12, стр. 3-7
Исследование липоксигеназы человека методом молекулярной и квантовой механики
А. А. Юрченко a, *, П. Д. Короткова b, **, В. И. Тимофеев c, d, А. Б. Шумм b, e, Ю. А. Владимиров a, b, c
a Российский национальный исследовательский медицинский университет
им. Н.И. Пирогова Министерства здравоохранения РФ
117997 Москва, Россия
b Московский государственный университет им. М.В. Ломоносова
119991 Москва, Россия
c Институт кристаллографии им. А.В. Шубникова ФНИЦ “Кристаллография и фотоника” РАН
119333 Москва, Россия
d Национальный исследовательский центр “Курчатовский институт”
123182 Москва, Россия
e Физический институт им. П.Н. Лебедева РАН
119991 Москва, Россия
* E-mail: yurchekoanastasiaaa@yandex.ru
** E-mail: korotkovapol@gmail.com
Поступила в редакцию 18.01.2022
После доработки 28.03.2022
Принята к публикации 28.03.2022
- EDN: IVCJWH
- DOI: 10.31857/S1028096022120330
Аннотация
Липоксигеназы являются классом ферментов, которые катализируют реакцию диоксигенации полиненасыщенных жирных кислот. Запуская перикисное окисление липидов, липоксигеназы могут принимать участие в процессе ферроптоза, который ассоциирован с рядом патологических состояний, в частности с некоторыми заболеваниями нервной системы. Липоксигеназы представляют собой железосодержащие белки, в которых ион железа расположен в активном центре фермента и необходим для катализа. С использованием комбинированного метода квантовой и молекулярной механики получена модель металлсодержащего сайта липоксигеназы LOX-15. При помощи метода молекулярной динамики и анализа координационных связей иона железа активного центра фермента показано, что такая модель более стабильна по сравнению со стандартной молекулярно-механической моделью и больше подходит для изучения механизма функционирования фермента.
ВВЕДЕНИЕ
Липоксигеназы – это класс ферментов, катализирующих реакцию диоксигенации полиненасыщенных жирных кислот. Липоксигеназы являются железосодержащими белками, в которых ион железа расположен в активном центре фермента и необходим для катализа [1]. Липоксигеназы обнаружены у животных, растений и грибов [1]. Несмотря на такую широкую распространенность, функции липоксигеназ в живой клетке и механизм липоксигеназной реакции изучены не до конца [2, 3]. Тем не менее считают, что липоксигеназы человека принимают участие в процессе ферроптоза – программируемой гибели клеток, когда происходит зависимое от железа перекисное окисление липидов [4, 5]. Также считают, что с ферроптозом ассоциирован ряд патологических состояний, например, некоторые болезни мозга, в том числе болезни Паркинсона и Альцгеймера [6–8]. Таким образом, изучение процесса ферроптоза является значимой научной задачей. Известна пространственная структура липоксигеназы человека LOX-15 [9]. Она состоит из двух доменов – малого b-домена и большого а-спирального каталитического домена. Липоксигеназа человека – это мембранно-ассоциированный белок. Предположительно, он выполняет свою роль в комплексе с белком-партнером [9]. Исследование функций такой системы экспериментальными методами представляет собой чрезвычайно сложную задачу. Однако возможно использовать для исследования свойств и функций такого комплекса методы вычислительной биологии. Например, применяя метод молекулярного докинга и молекулярной динамики, можно построить модель исследуемой системы и изучить ее структуру и динамические свойства. С помощью комбинированного метода квантовой и молекулярной механики можно промоделировать катализируемую ферментом реакцию, детально изучив таким образом механизм катализа [10]. Однако для металлсодержащих ферментов обычно требуется предварительное построение модели металлсодержащего сайта [11]. Такая модель может быть построена с использованием методов квантовой химии [11]. В настоящей работе была получена модель квантовой и молекулярной механики (КМ/ММ) активного сайта липоксигеназы LOX-15. Методом молекулярной динамики показано, что такая модель более стабильна и больше подходит для изучения механизма функционирования фермента.
МАТЕРИАЛЫ И МЕТОДЫ
Построение модели
Для построения модели КМ/ММ металлсодержащего сайта человеческой липоксигеназы LOX-15 (PDBID:4NRE) применен подход, реализованный в MCPB.py (4-(4-chloro-2-methylphenoxy) butanoic acid) [12]. На первом этапе были подготовлены файлы для дальнейшей квантово-механической оптимизации. Для этого был использован интернет-сервис H++ [13]. Для подготовки иона железа применяли программу metalpdb2mol2.py из программного пакета AmberTools19 [14]. После подготовки файлы объединяли и затем с помощью программы pdb4amber [14] получали файл необходимого формата. На следующем этапе была проведена подготовка входных параметров для квантово-механической оптимизации с помощью MCPB.py [12]. Квантово-механическую оптимизацию модели проводили с использованием программного пакета GAMESS-US [15]. Расчет осуществляли в приближении B3LYP [16] подхода Кона–Шэма [17], основанном на гибридном функционале, в рамках теории функционала плотности [18]. Параметры силового поля для Amber [19] генерировали с помощью MCPB.py [12] и tleap [14].
Молекулярная динамика
Моделирование методом молекулярной динамики проводили с помощью программы Amber 18 [19]. В качестве силового поля было выбрано поле ff14SB [20]. В качестве модели воды брали модель TIP3P как наиболее подходящую для использования с силовым полем ff14SB. В систему было добавлено 0.15 М KCl, а также несколько ионов K+ и Cl– для нейтрализации заряда системы. Для релаксации структуры и во избежание стерических клэшей была минимизирована потенциальная энергия. Давление и температура в системе были уравновешены до 1 атм и 310 К путем запуска моделирования с ограничениями в ансамблях NVT и NPT. Давление и температуру в системе контролировали с помощью термостата Берендсена [21] и баростата Паринелло–Рахмана [22]. Продуктивную симуляцию молекулярной динамики в течение 5 нс для каждой из систем проводили в изотермо-изобарическом ансамбле с шагом 2 фс. Ван-дер-ваальсовы и кулоновские взаимодействия были усечены до 1.4 нм, что оптимально для используемого силового поля [20].
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
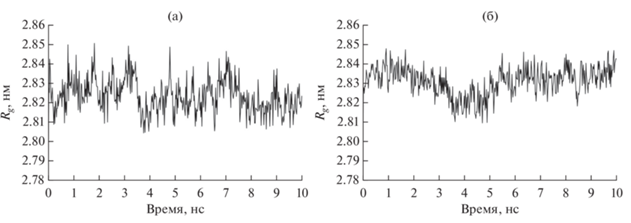
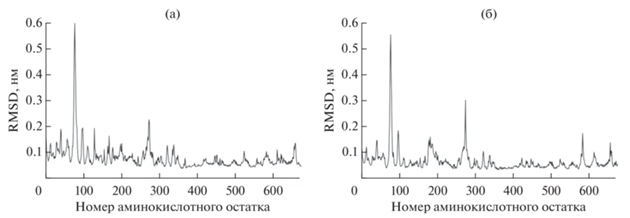
В качестве начального анализа траекторий были рассчитаны среднеквадратичное отклонение RMSD (root-mean-square deviation), радиус гирации Rg и среднеквадратичные флуктуации RMSF (root-mean-square fluctuations) для каждой из моделируемых систем. На рис. 1 показаны графики среднеквадратичных отклонений для классической и гибридной системы. Видно, что отклонения находятся в диапазоне 0.1–0.18 нм, что с учетом их размера говорит о стабильности моделируемых систем. На рис. 2 представлены графики радиуса инерции для каждой из систем. Из рисунка видно, что компактность систем существенно не меняется в процессе моделирования. На рис. 3 показаны среднеквадратичные флуктуации для атомов C-α каждой из систем. Из рисунка видно, что в районе 80–90 остатков имеется подвижный участок.
Известно, что ион железа в молекуле липоксигеназы LOX-15 координируют три остатка гистидина: His378, His373 и His553 [9] (рис. 4). Поэтому для квантово-механической оптимизации были выбрани эти три аминокислотных остатка и ион железа. Примененный подход Кона–Шэма (B3LYP) представляет собой гибридный функционал, основанный на сочетании метода Хартри–Фока и теории функционала плотности с использованием обменно-корреляционного потенциала Беке–Ли–Янга–Парра [23, 24]. Данный подход наиболее распространен при решении задач в области квантовой химии применительно к макромолекулам, он дает высокую точность расчетов и предъявляет относительно низкие требования к вычислительным мощностям [25]. В результате расчетов были получены параметры силового поля для иона железа и координирующих его остатков. Для проверки полученной модели с помощью программы Amber [19] и силового поля ff14SB [20] была промоделирована молекулярная динамика липоксигеназы LOX-15 в течение 10 нс с использованием стандартной модели металлсодержащего сайта и модели КМ/ММ. Для оценки качества описания металлсодержащего сайта анализировали динамику расстояний от иона железа до координирующих его атомов азота гистидинов (рис. 5, табл. 1). Из таблицы видна тенденция к увеличению расстояния, а также к увеличению стандартного отклонения при использовании молекулярно-механической модели. Из этого следует, что координационная связь в случае стандартной молекулярно-механической модели присутствует не всегда, а также то, что металлсодержащий сайт относительно нестабилен. Рис. 5 подтверждает эти выводы. Например, видно, что для 553-го гистидина расстояние от координирующего атома азота до атома железа достигает в некоторых случаях 0.9 нм, что однозначно говорит о разрыве координационной связи. Для 373-го гистидина расстояние между ионом железа и координирующим атомом азота стабилизируется вблизи значения 0.411 нм со стандартным отклонением 0.034. Кроме того, из графиков можно видеть, что в случае применения молекулярно-механической модели наблюдается существенное отклонение расстояния между координирующими атомами азота и ионом железа в процессе моделирования от данных рентгеноструктурного анализа, что также говорит о том, что квантово-механическая модель более пригодна для исследования динамики LOX-15.
Рис. 4.
Ион железа и координирующие его аминокислотные остатки в активном центре липоксигеназы LOX-15.

Рис. 5.
Зависимость расстояния между атомом азота NE2 553-го (а, г), 373-го (б, д) и 378-го (в, е) гистидина и железом от времени в молекулярно-механической (а–в) и квантово-механической (г–е) моделях.

Таблица 1.
Средние расстояния и стандартные отклонения расстояний между атомом азота NE2 553-го, 373-го и 378-го гистидина и ионом железа
| № аминокислотного остатка | Молекулярная механика | Квантовая механика | ||
|---|---|---|---|---|
| расстояние, нм | стандартное отклонение | расстояние, нм | стандартное отклонение | |
| 553 | 0.619 | 0.082 | 0.405 | 0.018 |
| 373 | 0.411 | 0.034 | 0.214 | 0.021 |
| 378 | 0.231 | 0.018 | 0.208 | 0.009 |
Список литературы
Brash A.R. // J. Biol. Chem. 1999. V. 274. № 34. P. 23679. https://doi.org/10.1074/jbc.274.34.23679
Nelson M.J., Seitz S.P. // Current Opinion Struct. Biol. 1994. V. 4. № 6. P. 878. https://doi.org/10.1016/0959-440x(94)90270-4
Куракин Г.Ф., Самоукина А.М., Потапова Н.А. // Биохимия. 2020. Т. 85. № 9. С. 1256. https://doi.org/10.31857/S0320972520090067
Dixon S.J., Stockwell B.R. // Nature Chem. Biol. 2014. V. 10. № 1. P. 9. https://doi.org/10.1038/nchembio.1416
Stoyanovsky D.A., Tyurina Y.Y., Shrivastava I., Bahar I., Tyurin V.A., Protchenko O., Jadhav S., Bolevich S.B., Kozlov A.V., Vladimirov Y.A., Shvedova A.A., Philpott C.C., Bayir H., Kagan V.E. // Free Radical Biol. Med. 2019. V. 133. P. 153. https://doi.org/10.1016/j.freeradbiomed.2018.09.008
Weiland A., Wan, Y., Wu W., Lan X., Han X., Li Q., Wang J. // Mol. Neurobiol. 2019. V. 56. № 7. P. 4880. https://doi.org/10.1007/s12035-018-1403-3
Mahoney-Sánchez L., Bouchaoui H., Ayton S., Devos D., Duce J.A., Devedjian J.C. // Progress Neurobiol. 2021. V. 196. P. 101890. https://doi.org/10.1016/j.pneurobio.2020.101890
Zhang G., Zhang Y., Shen Y., Wang Y., Zhao M., Sun L. // J. Alzheimer’s Disease. 2021. V. 80. № 3. P. 907. https://doi.org/10.3233/JAD-201369
Kobe M.J., Neau D.B., Mitchell C.E., Bartlett S.G., Newcomer M.E. // J. Biol. Chem. 2014. V. 289. № 12. P. 8562. https://doi.org/10.1074/jbc.M113.543777
Senn H.M., Thiel W. // Ang. Chem. Int. Ed. 2009. V. 48. № 7. P. 1198. https://doi.org/10.1002/anie.200802019
Li P., Merz Jr K.M. // Chem. Rev. 2017. V. 117. № 3. P. 1564. https://doi.org/10.1021/acs.chemrev.6b00440
Li P., Merz Jr K.M. // J. Chem. Inf. Modeling. 2016. V. 56. № 4. P. 599. https://doi.org/10.1021/acs.jcim.5b00674
Anandakrishnan R., Aguilar B., Onufriev A.V. // Nucl. Acids Res. 2012. V. 40. № W1. P. W537. https://doi.org/10.1093/nar/gks375
Salomon-Ferrer R., Case D.A., Walker R.C. // WIRs: Comput. Mol. Sci. 2013. V. 3. № 2. P. 198. https://doi.org/10.1002/wcms.1121
Barca G.M.J, Bertoni C., Carrington L. et al. // J. Chem. Phys. 2020. V. 152. № 15. P. 154102. https://doi.org/10.1063/5.0005188
Becke A.D. // J Chem. Phys. 1993. V. 98. P. 5648. https://doi.org/10.1063/1.464913
Kohn W., Sham L.J. // Phys. Rev. A. 1965. V. 140. № 4. P. A1133. https://doi.org/10.1103/PhysRev.140.A1133
Hohenberg P., Kohn W. // Phys. Rev. B. 1964. V. 136. 3. P. B864. https://doi.org/10.1103/PhysRev.136.B864
Case D.A., Cheatham T.E. III, Darden T., Gohlke H., Luo R, Merz K.M. Jr, Onufriev A., Simmerling C., Wang B., Woods R.J. // J. Comput. Chem. 2005. V. 26. № 16. P. 1668. https://doi.org/10.1002/jcc.20290
Maier J.A., Martinez C., Kasavajhala K., Wickstrom L., Hauser K.E., Simmerling C. // J. Chem. Theory Comput. 2015. V. 11. № 8. P. 3696. https://doi.org/10.1021/acs.jctc.5b00255
Berendsen H.J.C., Postma J.V., van Gunsteren W.F., DiNola A.R.H.J., Haak J.R. // J. Chem. Phys. 1984. V. 81. № 8. P. 3684. https://doi.org/10.1063/1.448118
Parrinello M., Rahman A. // J. Chem. Phys. 1982. V. 76. № 5. P. 2662. https://doi.org/10.1063/1.443248
Becke A.D. // Phys. Rev. A. 1988. V. 38. № 6. P. 3098. https://doi.org/10.1103/PhysRevA.38.3098
Ernzerhof M., Scuseria G.E. // J. Chem. Phys. 1999. V. 110. № 11. P. 5029. https://doi.org/10.1063/1.478401
Goerigk L., Hansen A., Bauer C., Ehrlich S., Najibi A., Grimme S. // Phys. Chem. Chem. Phys. 2017. V. 19. № 48. P. 32184. https://doi.org/10.1039/C7CP04913G
Дополнительные материалы отсутствуют.
Инструменты
Поверхность. Рентгеновские, синхротронные и нейтронные исследования