Прикладная биохимия и микробиология, 2021, T. 57, № 5, стр. 496-503
Выделение и идентификация липопептидов штамма Bacillus subtilis 26Д
Е. А. Черепанова 1, И. В. Галяутдинов 1, Г. Ф. Бурханова 1, И. В. Максимов 1, *
1 Институт биохимии и генетики – обособленное структурное подразделение Федерального государственного бюджетного научного учреждения Уфимский федеральный исследовательский центр РАН
450054 Уфа, Россия
* E-mail: igor.mak2011@yandex.ru
Поступила в редакцию 09.04.2021
После доработки 15.04.2021
Принята к публикации 22.04.2021
Аннотация
Бактерии рода Bacillus занимают особое место среди микроорганизмов, стимулирующих рост растений, поскольку способствуют защите растений от патогенов и вредителей. Показано, что защитные свойства бактериям придают вырабатываемые ими биологически активные вещества, секретируемые во внеклеточное пространство. Среди них большой интерес вызывают липопептиды. Из среды культивирования бактерий эндофитного штамма Bacillus subtilis 26Д, являющегося основой биопрепарата Фитоспорин-М, методом высокоэффективной жидкостной хроматографии и электрораспылительного масс-спектрометрического детектирования выделена липопептидная фракция, в которой идентифицировано два сурфактина (сурфактин C13 и сурфактин C15). Проведенный анализ генов липопептид-синтетаз штамма бактерии B. subtilis 26Д выявил наличие гена сурфактин-синтетазы (Bssfp), отвечающего за синтез сурфактинов, но генов итурин- (BsituD) и фенгицин- (BsfenD) синтетаз не было обнаружено.
Бактерии рода Bacillus (Cohn) являются одними из самых распространённых микроорганизмов, занимающих разные экологические ниши (вода, воздух, почва, ткани растений, животных и человека). На сегодняшний день наиболее известный и изученный вид этого рода – Bacillus subtilis Cohn. Препараты, содержащие в качестве действующего начала различные штаммы этого вида, активно используют в различных отраслях сельского хозяйства (растениеводстве, животноводстве, птицеводстве, рыбоводстве и ветеринарии), а также в медицине в качестве пробиотиков для нормализации микробиома кишечника, укрепления иммунитета и защиты от различных болезнетворных микроорганизмов.
Среди значимых для растениеводства штаммов B. subtilis особо следует остановиться на стимулирующих рост растений эндофитных штаммах, способных длительное время обитать в тканях растений, не причиняя им повреждений. Такие штаммы, образующие тесные мутуалистические взаимоотношения со своими хозяевами и антагонистичные к широкому спектру фитопатогенов, можно представить, как уникальный компонент микрофитобиома, позволяющий им, эффективно колонизируя и присутствуя в тканях растения, конкурировать с патогенами за нишу трофности.
Исследователи обращают внимание на способность эндофитных бактерий регулировать численность патогенной микрофлоры путем синтеза антибиотических веществ и антистрессовых соединений [1, 2]. Среди таких метаболитов бактерий привлекают внимание липопептиды (ЛП), особенностью которых является нерибосомальный синтез с помощью многофункциональных белковых комплексов, называемых нерибосомальными пептидными синтетазами (НРПС) или ЛП-синтетазами [3]. В зависимости от аминокислотной последовательности и состава жирных кислот, ЛП принято подразделять на три основных класса: сурфактины, итурины и фенгицины. Имеются сведения, что внутри данных групп ЛП могут различаться по длине и разветвленности жирнокислотной цепи, обеспечивая разнообразие изоформ [4].
В российском сегменте растениеводства широко применяется коммерческий фунгицидный биопрепарат Фитоспорин-М (“Башинком”, Россия), содержащий в качестве основы бактерии штамма B. subtilis 26Д, обладает как рост-стимулирующими, так и фунгицидными свойствами. Хотя известно, что ЛП-богатая фракция, в том числе и у штамма B. subtilis 26Д, обладает высокой фунгицидной и рострегулирующей активностью, играет определенную роль в процессах защиты растений от различных патогенов [5, 6], до сих пор спектр ЛП у штамма B. subtilis 26Д и их физико-химические особенности не исследованы.
Основной целью этой работы является получение липопептид-богатой фракции метаболитов эндофитной бактерий B. subtilis 26Д и идентификация основных липопептидов, вырабатываемых этим штаммом.
Методика. Объектом исследования являлся штамм эндофитных бактерий B. subtilis 26Д (ВНИИСХМ, № 128), входящий в состав препарата Фитоспорин-М (НВП “Башинком”, Россия) и хранящийся в лаборатории биохимии иммунитета растений ИБГ УФИЦ РАН. Для культивирования B. subtilis 26Д использовали питательную среду Luria Bertani (LB) [4]. После приготовления питательную среду стерилизовали в автоклаве 2540 ML (“Tuttnauer”, Израиль) 25 мин при давлении 1.5 атм., t = 121°С. Для приготовления суспензии бактерий в стерильных условиях в колбы с питательной средой LB вносили клетки B. subtilis 26Д и инкубировали 12 ч на шейкере Orbital Shaker-Incubator ES-20 (“BioSan”, Латвия) при 170 об./мин и t = 37°С.
Отбор проб производили каждые 12 ч в течение 7 сут. В пробах гравиметрическим методом оценивались микробная биомасса и концентрация ЛП.
Получение фракции ЛП из среды культивирования B. subtilis 26Д. Выделение фракции ЛП проводили методом осаждения из культуральной жидкости бактериальной культуры, как описано [7]. Суспензию бактериальных клеток центрифугировали 45 мин при 1600 g на центрифуге Hettich Universal 320R (“Hettich”, Германия), осадок использовали для определения сухой биомассы, а из супернатанта извлекали фракцию, богатую ЛП. Надосадочную жидкость подкисляли 2 М HCl до рН 2.0 (рН-метр, “HANNA Instruments”, Германия) и оставляли на ночь при 4°С. Сформировавшийся осадок отделяли центрифугированием при 4000 g 30 мин, отмывали дистиллированной водой, подкисленной HCl до рН 2.0 и снова центрифугировали в тех же условиях. Полученный осадок суспензировали в небольшом количестве дистиллированной воды, доводя рН до 7.0 слабым раствором КОН, добавляя этанол до 80%. Через 1.5 ч периодического взбалтывания центрифугированием отделяли осадок, надосадочную жидкость пропускали через фильтр Amicon Ultracel – 3K (“Merck”, Германия). Фракцию, прошедшую через фильтр, с массой менее 3 кДа, собирали и высушивали в вакууме в вакуум-концентраторе Concentrator 5301 (“Eppendorf”, Германия) при 30°С.
Гравиметрическая оценка сухой биомассы клеток. Осадок, полученный после центрифугирования бактериальной суспензии, для удаления солей питательной среды осторожно промывали 2 мл стерилизованной воды MilliQ. Образец снова центрифугировали (1600 g, 15 мин), супернатант удаляли и осадок суспензировали в 1.5 мл стерильной воды и переносили в предварительно взвешенные 1.5 мл пробирки Эппендорфа. Снова центрифугировали при тех же условиях, супернатант отбрасывали, а пробирки сушили при 30°C на Concentrator 5301 (“Eppendorf”, Германия) до постоянного веса. Для оценки высушенной биомассы пробирки взвешивали на весах СЕ224-С (“Сартогосм”, Россия) с последующим пересчётом на объём отобранного образца.
Гравиметрическая оценка концентрации ЛП. ЛП, синтезируемые бактериями B. subtilis 26Д, измеряли в ЛП-богатой фракции. Для этого 1.5 мл пластиковые пробирки взвешивали до внесения в них очищенной ЛП-фракции, растворенной в 80% этанола, и после ее высушивания при 30°C на Concentrator 5301 (“Eppendorf”, Германия) до постоянного веса. Пробирки взвешивали на весах СЕ224-С и вес пересчитывали на объем культуральной жидкости, из которого была получена ЛП-фракция.
Тонкослойную хроматографию (ТСХ) ЛП-фракции проводили на пластинах с силикагелем Sorbfil (Imid ltd., Россия) [8]. В качестве подвижной фазы использовали элюент хлороформ:метанол:вода в соотношении 65 : 25 : 4 [9]. Пластины после элюирования проявляли тремя способами: 3%-ным раствором ванилина в этиловом спирте, подкисленным раствором серной кислоты, 0.1%-ным раствором нингидрина и парами йода.
Высокоэффективная жидкостная хроматография (ВЭЖХ). ЛП, выделенные из культурального фильтрата бактерий B. subtilis, анализировали на хроматографе Agilent 1260 (фирма Agilent Technologies Inc., США) с масс-спектрометрическим детектированием (масс-спектрометр с ионизацией электрораспылением AmazonX, “Bruker Daltonik GmbH”, Германия). Перед проведением ВЭЖХ высушенную фракцию ЛП растворяли в 20%-ном метаноле. В качестве элюента ЛП в колонке ReproSil-Pur 120 C18-AQ (125 × 4 мм, 5 µм) (“Dr. Maisch Phases”, Германия) в использовали раствор метанол:вода (70 : 30, об./об.) со скоростью потока 0.5 мл/мин. Объем пробы 20 мкл. Регистрация масс-спектров проводилась в режиме положительных ионов в диапазоне m/z от 70 до 2700. Напряжение на капилляре распылителя составляло 3500 В. Для управления и сбора данных использовалось программное обеспечение TrapControl 7.0 (“Bruker Daltonik” GmbH, Германия).
Выделение геномной ДНК из клеток штамма B. subtilis 26Д. Часть бактериальной биомассы с агаризованной среды переносили в пластиковые пробирки типа Eppendorf (“SSI”, США) и добавляли 250 мкл лизирующего буфера следующего состава (%): тритон Х100 – 1.0, твин 20 – 1.0, смола Сhelex 100 (“BioRad”, США) – 1.0, крезоловый красный – 0.005. Содержимое пробирок перемешивали, прогревали в твердотельном термостате Гном (ДНК-Технология, Россия) при температуре 95°С в течении 15 мин, после чего еще раз перемешивали и центрифугировали на препаративной центрифуге типа 5415R (“Eppendorf”, Германия) 3 мин при 12 000 g. Для амплификации использовали 2 мкл супернатанта на одну реакцию. Подбор праймеров к ЛП-синтетазам осуществлялся на основе литературных данных (табл. 1).
Таблица 1.
Нуклеотидные последовательности подобранных праймеров к генам B. subtilis, кодирующим липопептид-синтетазы
| Название гена | NCBI | Праймеры | Последовательность, 5'-3' | Размер ампликона, п.н. | Ссылка |
|---|---|---|---|---|---|
| Bssfp | EU882341 | SfpF SfpR |
atgaagatttacggaattta ttataaaagctcttcgtacg |
675 | [10] |
| BsituD | D21876 | ituDF ituDR |
atgaaaatttacggagtatatat ttataacagctcttcatacgtt |
674 | [11] |
| BsfenD | AJ011849 | FenDF FenDR |
tttggcagcaggagaagttt gctgtccgttctgctttttc |
964 | [10] |
| BsBac | NR102783 | BacF BacR |
accagaaagccacggctaaac ggcggaaaccccctaacact |
356 | [12] |
Амплификацию полноразмерных генов sfp, ituD и fenD из генома штамма бактерий B. subtilis 26Д проводили в амплификаторе типа ТП4-ПЦР-01-Терцик (“ДНК-Технологии”, Россия) с использованием соответствующих праймеров (табл. 1).
Выделение тотальной РНК. Из культуры B. subtilis 26Д через 1 и 3 сут роста бактерий была выделена тотальная РНК с использованием реагента “Trizol” согласно протоколу фирмы-поставщика (“Sigma”, Германия). Концентрацию нуклеиновых кислот измеряли на спектрофотометре Smart Spec Plus (“Bio-Rad”, США). Для синтеза кДНК проводили реакцию обратной транскрипции с использованием M-MuLV обратной транскриптазы (“Синтол”, Россия).
Анализ экспрессии генов. Анализ ЛП-синтетаз проводили методом количественной ПЦР в режиме реального времени на приборе “iCycler CFX Connect Real-Time PCR Detection System” (“BioRad”, США) с использованием интеркалирующего красителя SYBR Green I (“Синтол”, Россия). Изменения в экспрессии интересующего гена определяли на основании вычисления уровня нормализованной экспрессии генов c помощью программного обеспечения поставщика прибора (“BioRad”, США).
Аналитический гель-электрофорез ДНК проводили в 1%-ном агарозном геле. Маркер – MassRuler DNA Loading Dye (“Thermo Scientific”, США).
Все эксперименты проведены в 3 биологических и не менее 3 аналитических повторностях. При обработке результатов использовались программы Statistica 12.0 фирмы “Stat Soft”. На рисунках приведены средние значения биологических повторов и их стандартные отклонения.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Результаты изучения динамики накопления сухой массы бактерий и ЛП в зависимости от времени культивирования представлены на рис. 1. Как видно, концентрация ЛП достигала максимума через 60 ч культивирования. ТСХ обогащенной ЛП фракции показала, что в культуральном фильтрате штамма B. subtilis 26Д присутствовал ЛП с подвижностью Rf 0.65, окрашивающийся при проявлении раствором ванилина, нингидрином и парами йода (рис. 2). ЛП из культурального фильтрата бактерии штамма B. subtilis 26Д совпадал по подвижности с коммерческим сурфактином (“Sigma”, США) по значению Rf. Таким образом, использование ТСХ позволило обнаружить присутствие сурфактина в культуральном фильтрате штамма B. subtilis 26Д.
Рис. 1.
Динамика накопления сухой массы бактерий (г/л, (1)) и липопептидов (ЛП, г/л, (2)) в зависимости от времени культивирования.
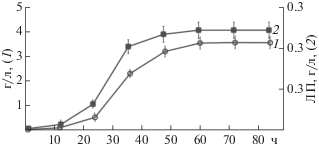
Рис. 2.
Результаты окрашивания пластинок растворами ванилина (а), нингидрина (б) и парами йода (в) после ТСХ коммерческого сурфактина (1) и фракции метаболитов B. subtilis 26Д (2), богатой липопептидами.

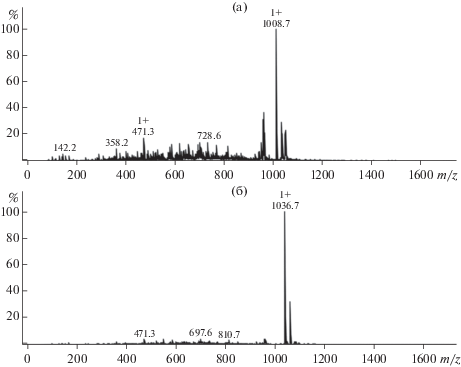
В обогащенной ЛП фракции B. subtilis 26Д при проведении ВЭЖХ (рис. 3) с последующим масс-спектрометрическим детектированием идентифицировано два сурфактина с ацильной цепью C13 (3.3 мин) с m/z 1008.7 [M + H] и C15 с m/z 1036.7 [M + H] (5.5 мин) (рис. 4). Наряду с идентифицированными сурфактинами с ацильной цепью C13 и C15, масс-спектрометрический анализ выявил соединения с m/z 268.0, 358.2, 453.2, 471.3, 697.6, 794.2 и 810.7. Соединения с m/z 453.2 и 471.3 близки по молекулярным массам к поликетидам, описанным в литературе [13].
Рис. 3.
ВЭЖХ–хроматограмма на колонке ReproSil-Pur 120 C18-AQ липопептидной фракции, выделенной из культурального фильтрата Bacillus subtilis 26Д.
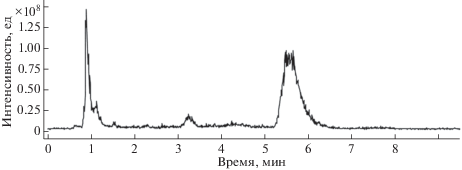
Рис. 4.
Масс-спектр липопептидной фракции, продуцируемой Bacillus subtilis 26Д, отвечающий сурфактину С13 (а) и сурфактину С15 (б).
Для определения наличия или отсутствия генов, кодирующих ЛП-синтетазы применяется метод ПЦР с ДНК бактерий. Способность бактерий к синтезу ЛП можно оценить по экспрессии генов ЛП-синтетаз с использованием праймеров, нацеленных на консервативные участки [2, 10, 14]. В результате ПЦР-анализа в геноме штамма B. subtilis 26Д обнаружен ген Bssfp, кодирующий сурфактин-синтетазу (рис. 5а). Праймеры BsituD и BsfenD, использованные для детекции генов, кодирующих итурин- и фенгицин-синтетазы, соответственно, не формировали транскриптов на базе кДНК штамма B. subtilis 26Д. Соответственно, проведённый анализ наличия генов ЛП-синтетаз обнаружил в геноме штамма B. subtilis 26Д ген сурфактин-синтетазы, отвечающий за синтез сурфактина, и показал отсутствие генов итурин-синтетазы и фенгицин-синтетазы.
Рис. 5.
Результаты ПЦР ДНК бактерий B. subtilis 26Д с праймерами к генам Bssfp (1), BsituD (2) и BsfenD (3) (а) и транскрипционной активности гена Bssfp (б) через 0 (1), 1 (2) и 3 (3) сут роста бактерий B. subtilis 26Д в культуре.
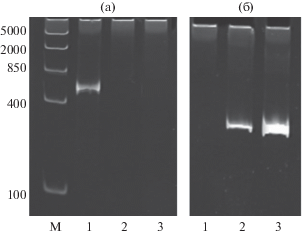
Для определения способности продуцировать сурфактин бактериями B. subtilis 26Д изучена экспрессия гена Bssfp в культуральной среде штамма B. subtilis 26Д. Показано (рис. 5б), что на 1 и 3 сут роста бактерий в культуре активно накапливается мРНК Bssfp B. subtilis 26Д. Таким образом, нами показано, что эндофитный штамм бактерии B. subtilis 26Д эффективно экспрессирует ген сурфактин-синтетазы в культуре.
В современной литературе встречается много работ, посвященных продукции ЛП различными видами бактерий, в особенности различными штаммами Bacillus spp. Для выявления способности бактерий к синтезу ЛП часто идентифицируют у них гены НРПС [2, 3, 10]. Среди генов, кодирующих НРПС, наиболее изучены семейства синтетаз сурфактина, фенгицина и итурина. Так, у штамма Bacillus tequilensis обнаружены гены, ответственные за синтез сурфактина, фенгицина, бациллибацина и бацилина [15]. В геноме штамма B. velezensis обнаружены нуклеотидные последовательности, кодирующие ЛП: фенгицин-б, итурин, сурфактин-а, бутирозин, плантазолицин, киянимицин и др. [16]. В геноме штамма B. amyloliquefaciens subsp. plantarum из здоровых тканей винограда Vitis vinifera определены гены, кодирующие белки, ответственные за синтез бациллаена, диацидина, микролактина, сурфактина и фенгицина [17]. Проведeнный анализ нуклеотидных последовательностей ЛП-синтетаз штамма B. subtilis 26Д показал наличие только гена сурфактин-синтетазы Bssfp, отвечающего за синтез сурфактина, но не фенгицина и итурина. Также нами была впервые в клетках B. subtilis 26Д показана экспрессия этого гена.
Известно, что бактерии Bacillus spp. могут одновременно вырабатывать различные ЛП и их структурные аналоги (изоформы). Семейство сурфактина включает в себе молекулы различной структуры, но все они – гептапептиды, связанные с β-гидроксикислотой, образуют циклическую структуру лактонного кольца [18]. Так, среди метаболитов бактерий Bacillus clausii DTM1 обнаруживается до 12 сурфактиновых аналогов, различающихся характером пептидных остатков и/или длиной и разветвлением цепи жирных кислот [19]. Методом ТСХ в культуральном фильтате штамма B. subtilis 26Д обнаружен ЛП с Rf 0.65, совпадающий по подвижности с коммерческим сурфактином. Ранее Клюге с соавт. [9] описали наличие сурфактина с Rf 0.65 у штамма B. subtilis ATCC 21332, а Джой с соавт. [20] – с Rf 0.72 и 0.55. у бактерий штамма Bacillus licheniformis SB2.
С использованием ВЭЖХ с масс-спектрометрическим детектированием позволило идентифицировать в ЛП-богатой фракции метаболитов B. subtilis 26Д две разновидности сурфактина – с длинами жирнокислотных цепей C13 и C15. Подобные изомеры сурфактина из культурального фильтрата штамма B. licheniformis HSN221 ранее были выделены и описаны [21]. Фариа с соавт. [22] получили масс-спектры ЛП штамма Bacillus subtilis LSFM-05 с m/z 1022, 1036, 1044 и 1058 с длинами жирнокислотных цепей C13 и C14. Чен с соавт. [16] сообщили о присутствии у B. licheniformis MB01 изоформ сурфактина С12, С13, С14 и С15. Сходные с полученными в настоящей работе результатами были опубликованы в Фульпото с соавт. [23], где штамм B. subtilis 41651 A1 синтезировал изоформы сурфактина с жирнокислотными цепями С13 и С15.
Бактериальные ЛП имеют широкое практическое применение, которое связано в первую очередь с их биологической и избирательной биоцидной активностями, определяющими специфическими различиями в их действии в зависимости от их природы [24, 25]. Кроме того, интерес у исследователей вызывает синергический защитный эффект ЛП [26, 27]. Обнаружено, что совместно сурфактин и фенгицин из B. subtilis GM5 подавляли развитие гриба Fusarium oxysporum [2]. Есть сведения об усилении антифунгальной активности при совместном воздействии фенгицина и микосубтилина [28].
Сурфактины, продуцируемые бактериями рода Bacillus, известны не только своей биосурфактантной активностью, а также проявлением гемолитических, противовирусных [29–31] антибактериальных и инсектицидных [19, 25, 32, 33] свойств.
В настоящее время наиболее актуальны исследования, связанные с созданием микроорганизмов с измененным для определенных целей геномом. Штамм, синтезирующий единственный вид ЛП с известным числом конкретных изомеров может быть удобен для создания генно-инженерных конструкций, регуляции синтеза данного ЛП и изменения некоторых его свойств. Так, у некоторых модифицированных сурфактинов наблюдалась пониженная гемолитическая активность, следовательно, они имели более низкую токсичность по отношению к эритроцитам, но при этом не теряли антибактериальнуюактивность [34].
Проведенные исследования показали, что B. subtilis 26Д является источником двух изоформ сурфактина и может быть использован для создания генно-инженерных конструкций на основе данного штамма. Разработка методов очистки вторичных метаболитов из культурального фильтрата B. subtilis 26Д в дальнейшем может способствовать созданию технологий выделения сурфактинов, которые могут быть востребованы в различных отраслях промышленности – от фармацевтики до производства поверхностно активных сурфактантов.
Работа выполнена при финансовой поддержке гранта РФФИ офи_м 17-29-08014, по темам государственного задания AAAA-A21-121011990120-7 и № АААА-А16-116020350033-8, с использованием оборудования ЦКП “Агидель” и уникальной научной установки “КОДИНК”. Идентификация ЛП проведена в ЦКП-САЦ ФИЦ КазНЦ РАН.
Список литературы
Pusenkova L.I., Il’yasova E.Yu., Lastochkina O.V., Maksimov I.V., Leonova S.A. // Eurasian Soil Science. 2016. V. 49. № 10. P. 1136–1144. https://doi.org/10.1134/S1064229316100112
Mardanova A.M., Hadieva G.F., Lutfullin M.T., Khilyas I.V., Minnullina L.F., Gilyazeva A.G., Bogomolnaya L.M., Sharipova M.R. // Agricultural Sci. 2017. V. 8. № 1. P. 1–20. https://doi.org/10.4236/as.2017.81001
Roongsawang N., Washio K., Morikawa M. // Int. J. Mol. Sci. 2011. V. 12. P. 141–172. https://doi.org/10.3390/ijms12010141
Paraszkiewicz K., Bernat P., Kuśmierska A., Chojniak J., Płaza G. // J. Environ. Manage. 2018. V. 209. Art. 65. https://doi.org/10.1016/j.jenvman.2017.12.033
Нафикова А.Р., Сурина О.Б., Хайруллин Р.М., Максимов И.В. // Агрохимия. 2018. № 5. С. 39–44. (rus).https://doi.org/10.7868/S000218811805006X
Черепанова Е.А., Благова Д.К., Бурханова Г.Ф., Сарварова Е.С., Максимов И.В. // Экобиотех. 2019. Т. 2. № 3. С. 339–346. https://doi.org/10.31163/2618-964X-2019-2-3-339-346
Vater J., Kablitz B., Wilde C., Franke P., Mehta N., Cameotra S.S. // Appl. Environ. Microbiol. 2002. V. 68. № 12. P. 6210–6219.
Alajlani M., Shiekh A., Hasnain S., Brantner A. // Chromatographia. 2016. V. 79. № 21. P. 1527–1532. https://doi.org/10.1007/s10337-016-3164-3
Kluge B., Vater J., Salnikow J., Eckart K. // FEBS Lett. 1988. V. 231. P. 107–110. https://doi.org/10.1016/0014-5793(88)80712-9
Gond S.K., Bergen M.S., Torres M.S., White J.F.Jr. // Microbiol. Res. 2015. V. 172. № 1. P. 79–87. https://doi.org/10.1016/j.micres.2014.11.004
Zhao X., Zhou Zh., Han Y, Wang Zh., Fan J., Xiao H. // Microbiol. Res. 2013. V. 168. № 9. P. 598–606. https://doi.org/10.1016/j.micres.2013.03.001
Kunst F., Ogasawara N., Moszer I., Albertini A.M. et al. // Nature. 1997. V. 390. № 6657. P. 249–256. https://doi.org/10.1038/36786
Wang T., Liang Y., Wu M., Chen Z., Lin J., Yang L. // Chin. J. Chem. Eng., 2015. V. 23. P. 744–754. https://doi.org/10.1016/j.cjche.2014.05.020
Afsharmanesh H., Perez-Garcia A., Zeriouh H., Ahmadzadeh M., Romero D. // Food Control. 2018. V. 94. № 1. P. 48–55. https://doi.org/10.1016/j.foodcont.2018.03.002
Xu W.F., Ren H.S., Ou T., Lei T., Wei J.H., Huang C.S., Li T., Strobel G., Zhou Z.Y., Xie J. // Microb. Ecol. 2019. V. 77. P. 651–663. https://doi.org/10.1007/s00248-018-1247-4
Chen L., Heng J., Qin S., Bian K. // PLoS One. 2018. V. 13. № 6. Art. e0198560. https://doi.org/10.1371/journal.pone.0198560
Pinto C., Sousa S., Froufe H., Egas C., Clément C., Fontaine F., Gomes A.C. // Stand. Genomic Sci. 2018. V. 13. Art. 30. https://doi.org/10.1186/s40793-018-0327-x
Peypoux F., Bonmatin J.M., Wallach J. Recent trends in the biochemistry of surfactin. Appl Microbiol Biotechnol. 1999. V. 51. № 5. P. 553–563. https://doi.org/10.1007/s002530051432
Guo D.L., Wan B., Xiao S.J., Allen S., Gu Y.C., Ding L.S., Zhou Y. // Natural Product Com. 2015. V. 10. № 12. P. 2151–2153. https://doi.org/10.1177/1934578X1501001235
Joy S., Rahman P.K., Sharma S. // Chem Eng J. 2017. V. 317. P. 232–241. https://doi.org/10.1016/j.cej.2017.02.054
Lee D.W., Kim B.S. // Plant Pathol J. 2015. V. 31. № 1. P. 1–11. https://doi.org/10.5423/PPJ.RW.08.2014.0074
Faria A.F., Teodoro-Martinez D.S., de Oliveira Barbosa G.N., Vaz B.G., Silva Í.S., Garcia J.S., Tótola M.R., Eberlin M.N., Grossman M., Alves O.L. // Process Biochem. 2011. V. 46. P. 1951–1957. https://doi.org/10.1016/J.PROCBIO.2011.07.001
Phulpoto I.A., Yu Z., Hu B., Wang Y., Ndayisenga F., Li J., Liang H., Qazi M.A. // Microb. Cell. Fact. 2020. V. 19. № 1. Art. 145. https://doi.org/10.1186/s12934-020-01402-4
Grady E.N., MacDonald J., Ho M.T., Weselowski B., McDowell T., Solomon O., Renaud J., Yuan Z.C. // BMC Microbiol. 2019. V. 19. № 1. Art. 5. https://doi.org/10.1186/s12866-018-1380-8
Maksimov I.V., Blagova D.K., Veselova S.V., Sorokan A.V., Burkhanova G.F., Cherepanova E.A., Sarvarova E.R., Rumyantsev S.D., Alekseev V.Yu., Khayrullin R.M. // Biological Control. 2020. V. 144. Art. 104242. https://doi.org/10.1016/j.biocontrol.2020.104242
Falardeau J., Wise C., Novitsky L., Avis T.J. // J. Chem. Ecol. 2013. V. 39. P. 869–878. https://doi.org/10.1007/s10886-013-0319-7
Khong N.G., Randoux B., Tayeh Ch., Coutte F., Bourdon N., Tisserant B., Laruelle F., Jacques P., Reignault P. // Commun. Agric. Appl. Biol. Sci. 2012. V. 77. № 3. P. 39–51.
Mihalache G., Balaes T., Gostin I., Stefan M., Coutte F., Krier F. // Environ. Sci. Pollut. Res. Int. 2018. V. 25. P. 29784–29793. https://doi.org/10.1007/s11356-017-9162-7
Yuan L., Zhang S., Wang Y., Li Y., Wang X., Yang Q. // J. Virol. 2018. V. 92. № 21. Art. e00809-18. https://doi.org/10.1128/JVI.00809-18
Johnson B.A., Hage A., Kalveram B., Mears M., Plante J.A., Rodriguez S.E., Ding Z., Luo X., Bente D., Bradrick S.S., Freiberg A.N., Popov V., Rajsbaum R., Rossi S., Russell W.K., Menachery V.D. // J. Virol. 2019. V. 93. № 22. Art. e01282-19. https://doi.org/10.1128/JVI.01282-19
Ibrahim A., Odon V., Kormelink R. // Front Plant Sci. 2019. V. 10. Art. 803. https://doi.org/10.3389/fpls.2019.00803
Rodríguez M., Marín A., Torres M., Béjar V., Campos M., Sampedro I. // Front Microbiol. 2018. V. 9. Art. 3114. https://doi.org/10.3389/fmicb.2018.03114
Sorokan A., Benkovskaya G., Blagova D., Maksimov I. // AIP Conference Proceedings. 2063, 2019. Art. 020001. https://doi.org/10.1063/1.5087308
Théatre A., Cano-Prieto C., Bartolini M., Laurin Y., Deleu M., Niehren J., Fida T., Gerbinet S., Alanjary M., Medema M.H., Léonard A., Lins L., Arabolaza A., Gramajo H., Gross H., Jacques P. // Front. Bioeng. Biotechnol. 2021. V. 9. Art. 623701. https://doi.org/10.3389/fbioe.2021.623701
Дополнительные материалы отсутствуют.
Инструменты
Прикладная биохимия и микробиология


