Программирование, 2022, № 3, стр. 22-29
МУЛЬТИКЛАССОВАЯ U-NET СЕГМЕНТАЦИЯ ЭЛЕКТРОННОЙ МИКРОСКОПИИ МОЗГА, ИСПОЛЬЗУЯ ОРИГИНАЛЬНЫЙ И ПОЛУСИНТЕТИЧЕСКИЙ ОБУЧАЮЩИЕ ДАТАСЕТЫ
А. А. Гетманская a, *, Н. А. Соколов a, **, В. Е. Турлапов a, ***
a Нижегородский государственный университет Н.И. Лобачевского
603950 Нижний Новгород, пр. Гагарина, д. 23, Россия
* E-mail: getmanskaya.alexandra@gmail.com
** E-mail: nikcsokolof@mail.ru
*** E-mail: vadim.turlapov@itmm.unn.ru
Поступила в редакцию 20.12.2021
После доработки 15.01.2022
Принята к публикации 20.01.2022
- EDN: NBLNHZ
- DOI: 10.31857/S0132347422030050
Аннотация
Подготовлена ручная многоклассовая разметка 20 слоев известного открытого датасета EPFL, для 6 классов: 1) митохондрии, включая границы; 2) границы митохондрий; 3) мембраны клеток; 4) постсинаптические уплотнения (PSD); 5) оболочки аксона; 6) везикулы. Создано программное обеспечение для генерации синтетических размеченных датасетов и сам синтетический датасет, уравновешивающий представленность классов. Исследованы результаты мультиклассовой сегментации данных электронной микроскопии (ЭМ) мозга для каждого класса, при бинарной классификации, и на 5 и 6 классов, с использованием модифицированной модели U-Net. Модель обучалась на фрагментах размером 256 × 256 исходного разрешения ЭМ. При 6-классовой сегментации митохондрии были сегментированы с индексом Дайса 0,908, что чуть ниже бинарной (0.911), и 5-классовой сегментации (0.910). Расширение датасета синтетическими изображениями в эксперименте улучшило результаты классификации отдельных классов. Расширение датасета размеченного вручную (860 изображений 256 × 256) синтетическим датасетом (100 изображений 256 × 256, содержащих наименее представленные классы: аксоны и PSD) дало существенный прирост точности выделения оболочки аксона и PSD 6-классовой моделью U-Net, соответственно: с 0.228 до 0.790 и с 0.553 до 0.745
1. ВВЕДЕНИЕ
Рассматривая развитие применения методов искусственного интеллекта, и особенно глубоких нейросетей (DNN), в реконструкции данных электронной микроскопии (EM) мозга за последние 10 с небольшим лет, можно начать с публикации 2010 г. [1]. Она фактически провозгласила начало использования серийной блочной сканирующей электронной микроскопии в качестве источника трехмерной наногистологии высокого разрешения для клеток и тканей. Далее действительно последовала серия работ, направленных на создание датасетов для тренировки сетей глубокого обучения, методов и моделей DNN сегментации данных EM, ориентированных на бинарную классификацию органелл клеток мозга: нейронных мембран [2]; супервоксельную сегментацию митохондрий (2012) [3]. Одновременно, на основе сегментации органелл нейронов и соединений между нейронами (синапсов), ставится задача 3D-реконструкции сети нейронов мозга, задача коннектомики мозга (2012) [4]. В этой задаче особенную важность приобретает сегментация таких органелл как: постсинаптические уплотнения (PSD); везикулы; аксоны.
В публикации 2015 г. [5], команда из 24 авторов, причастных к организации первого международного соревнования по 2D-сегментации EM изображений мозга, утверждает, что уже на конференции по коннектомике 2014 г., организованной Медицинским институтом Говарда Хьюза и Обществом Макса Планка, стало очевидно, что сверточные сети стали доминирующим подходом к обнаружению границ клеток на серийных EM-изображениях. Авторы также предлагают переходить к 3D-обработке EM и коллективным усилиям в коннектомике, отмечая, тем не менее, что лучшие современные алгоритмы 3D-реконструкции по-прежнему требуют серьезной ручной корректуры, реально доступной только краудсорсингу. Этот взгляд подтверждает вышедшая несколькими месяцами ранее статья [6], 21 автора из ведущих университетов USA, сообщающая о создании их общими усилиями насыщенной 3D-реконструкции малого (0.13 мм3) участка EM неокортекса мыши и базы данных на 1700 синапсов из этого участка.
Появление в 2015 г. сети U-Net [7] открыло целую серию новых моделей и адаптаций для сегментации данных EM мозга. Источник успеха U-Net в вовлечении контекстной информации входного изображения на всех уровнях обработки. Практически сразу публикация [8] экспериментально подтвердила, что skip-соединение U-Net архитектуры эффективно для решения задач сегментации в биомедицине. На базе U-Net строились также модели с параллельными входами, позволяющие использовать корреляцию между входами, в том числе, между слоями EM в трехмерном пространстве [9, 10]. Затем последовали попытки использовать возможности трехмерных сверток для кратного увеличения объема контекста в U-Net и U-Net-подобных сетях: 3DU-Net [11] (2016), V-Net [12] (2016), DeepMedic [13] (2017), HighRes3DNet [14] (2017). Это также принесло заметный эффект, так как для 3D-окрестности вокселя, радиусом в 1 воксель, объем контекстных данных возрастает в 3 раза, а для радиуса в 2 вокселя – в 5 раз.
Интересна линия развития семантической сегментации, реализованная на полностью сверточных нейронных сетях (Fully Convolutional Networks), представленная публикациями 2015 [15] и 2016 г. [16]. Особенно интересна и перспективна последняя. Для реконструкции 3D взаимосвязей системы нейронов предлагается оригинальная глубокая контекстная сеть, с трехкратной редукцией разрешения, анализирующая многомасштабную контекстную информацию в иерархической структуре разрешений. В архитектуру сети включены вспомогательные классификаторы, анализирующие семантическое значение иерархии изображений, удовлетворяясь низкоуровневыми контекстными признаками. В качестве датасета задачи сегментации использован ISBI 2012 г. Метод ориентирован на минимизацию вмешательства человека и демонстрирует дрейф к востребованному сегодня объяснимому искусственному интеллекту (XAI).
Преимущества использования 3D-анализа данных несомненны, однако, использование 3D сверточных нейронных сетей (CNN), с ядром 3D- свертки, значительно увеличивает количество параметров обучения, вычислительные затраты и потребление памяти, что особенно чувствительно для применения GPU. Поэтому архитектуры, использующие 3D-свертки, все же постепенно заменяются архитектурами, которые уменьшают количество параметров обучения, необходимый объем памяти, увеличивают скорость обучения, сохраняя качество, регулируя баланс между сетями с 3D-свертками и 2D-свертками. В этом процессе как правило используются разнообразные методы предобработки, часто дающие эффект порядка 5% и более [17–19]. Например, как в публикации 2020 г. [20] – использование усиления контраста на основе адаптивной гамма-коррекции с распределением веса (AGCWD) [21]. Другая тенденция – факторизация сверточных ядер низкого ранга [22–25].
Публикация 2019 г. [26] сообщает о создании системы UNI-EM, с интерфейсом удобным для профильных специалистов. После разметки небольшого числа тренировочных данных система применяет 2D- и 3D-сети глубокого обучения и возвращает сегментацию EM мозга для коррекции разметки и параметров обучения. UNI-EM поставляется с набором 2D DNN: U-Net, ResNet, HighwayNet и DenseNet.
Публикация конца 2019 г. [27] определяет лучший вариант U-Net на примере детектирования везикул на данных EM Transmission Electron Microscopy (TEM) разрешения – 1.56 нм (в 2–3 раза выше обычного), сравнивая U-Net и Fully Residual U-Net (FRU-Net) архитектуру. Установлено, что последняя дает повышение точности на 4–5%. При бинарной классификации на 3 разных датасетах TEM, для FRU-Net ошибка не превысила 10%. Для U-Net ошибки составили: 17, 27, 17%.
Публикация 2021 г. [28] исследует возможности фактически Fully Residual U-Net (FRU-Net) с 4 уровнями понижения разрешения (исходное разрешение 640 × 640 понижается четыре раза вдвое до 40 × 40) на примере бинарной 2D-классификации клеточных мембран. Построена аугментация, увеличившая датасет в 8 раз за счет поворотов и отражений. На данных Drosophila EM dataset (ISBI 2012 EM segmentation challenge leaderboard, June 2020) удалось достигнуть точности сегментации мембран порядка 98–99%. Публикация также 2021 г. [29] предлагает более сложную структуру сети, названную иерархическая свертка (HVEC), в качестве альтернативы простой трехмерной свертке. Эта структура наследует идею упомянутой выше публикации 2016 г. [16] с тремя уровнями понижения разрешения и дополнительными выходами для каждого уровня, дополняя далее архитектуру понижения разрешения веткой повышения разрешения типичной для U-Net.
Применение методов искусственного интеллекта в обработке данных EM в значительной мере тормозится небольшим объемом размеченных данных для тренировки и тестирования DNN. Открытые данные ЕМ в целом представлены всего несколькими размеченными объемами, как из-за трудоемкости подготовки препарата для электронного микроскопа, так и из-за дефицита специалистов для ручной разметки. В ходе наших исследований найдены четыре общедоступных данных электронной микроскопии, самые ранние и популярные из которых размечены только 1 классом (митохондрии или мембраны). И еще в двух наборах выделено несколько классов. Как следствие, подавляющее большинство нейронных сетей в обработке EM обучаются только бинарной классификации.
В связи с изложенным выше, коллектив авторов видел в качестве своих главных задач: 1) создание датасета с ручной мультиклассовой разметкой для списка классов, обеспечивающего решение основных современных задач сегментации данных EM; 2) создание алгоритмов автоматической генерации датасета синтетических объектов заданных основных классов и создание датасета синтетических объектов, прежде всего для дефицитно представленных в традиционном датасете; 3) исследование возможностей мультиклассовой сегментации U-Net-образных архитектур, начиная с U-Net (в данной работе), использующих датасеты с ручной и дополняющей ее синтетической разметкой.
2. МАТЕРИАЛЫ И МЕТОДЫ
В этом разделе представлена информация об общедоступных наборах данных. Наиболее широко используемые наборы данных для оценки митохондриальной сегментации были предоставлены Lucchi et al. [3].
Можно видеть, что в трех из четырех размеченных открытых наборов данных размечен только 1 класс. И только в одном наборе выделено несколько классов. Поэтому подавляющее большинство нейронных сетей в электронной микроскопии обучаются только на два класса (объект и фон).
В качестве исходных данных для разметки мы взяли “набор данных EPFL” или “набор данных митохондриальной сегментации Lucchi” с сайта https://www.epfl.ch/labs/cvlab/data/data-em/. Эти данные изначально содержат маски только для митохондрий. Поэтому для анализа и оценки алгоритмов мультиклассовой сегментации мы вручную разметили 20 слоев из тренировочной выборки (1024 × 768) и 3 слоя тестовой выборки для следующих классов: 1) Митохондрии вместе с границей; 2) Границы митохондрий; 3) Мембраны; 4) Постсинаптические уплотнения (PSD); 5) Оболочки аксонов; 6) Везикулы.
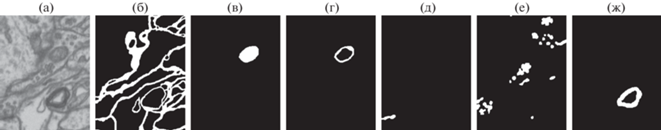
Точная разметка 1 слоя вручную занимает 5–8 ч. Наша разметка набора данных EPFL доступна по адресу https://github.com/GraphLabEMproj/unet. Мы планируем продолжить работу над разметкой и разметим оба имеющихся набора. Вы можете увидеть пример разметки фрагмента слоя на рис. 1.
Рис. 1.
Пример нашей разметки изображения: а) исходное изображение, б) мембраны, в) митохондрия с границами, г) границы митохондрий, д) PSD, е) везикулы, ж) оболочка аксона.
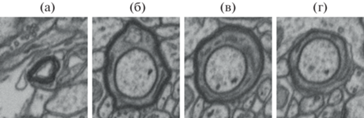
Так уж вышло, что оболочка аксона в тренировочном наборе данных представлена только на первых 36 слоях и выглядит совершенно не похоже на оболочку аксона в тестовом наборе данных (рис. 2). В тестовом наборе данных аксон представлен на первых 70 слоях и меняет свою форму с вытянутой на более округлую, а также обладает более темной внутренностью и внутренним кольцом.
Рис. 2.
Оболочки аксонов в тренировочном и тестовом наборах EPFL: а) оболочка аксона в тренировочном наборе, б) оболочка аксона в тестовом наборе 1-й слой, в) оболочка аксона в тестовом наборе 35-й слой, г) оболочка аксона в тестовом наборе 70-й слой.
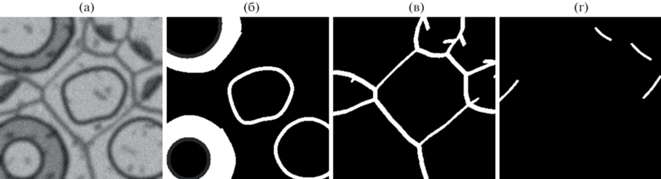
Для синтетического набора данных было сгенерировано 100 изображений размером 256 × 256 пикселей, содержащих наименее представленные классы: постсинаптическое уплотнение и оболочки аксона. Пример данных представлен на рис. 3. Программа для генерации данных была написана на языке C#. Форма, размер и уровни серого компартментов были подобраны таким образом, чтобы быть похожими на форму, размер и уровни серого тестового набора данных EPFL. Для придания большей похожести изображение было размыто фильтром Гаусса с радиусом ядра 7 и добавлен Гауссов шум силой 20. Преимущество синтетического набора заключается в том, что можно получить любое необходимое количество изображений вместе с разметкой автоматически.
Рис. 3.
Пример синтетических данных (на рисунке только ненулевые маски): а) слой; б) маска оболочек аксона; в) маска мембран; г) маска PSD.
2.1. Архитектура сети
U-Net считается одной из стандартных архитектур сверточной нейронной сети для задач сегментации изображений. Архитектура состоит из сжимающего пути для захвата глобального контекста и симметричного разжимающего пути, обеспечивающего точную локализацию. За основу нейросети был взят проект U-Net https://github.com/zhixuhao/unet. В исходном проекте U-Net использовался для бинарной классификации мембран. В нашем исследовании мы используем U-Net для многоклассовой сегментации. Мы скопировали оригинальный репозиторий, все изменения в коде и наша разметка данных Lucci доступны по адресу https://github.com/GraphLabEMproj/ unet.
Следуя автору кода https://github.com/ zhixuhao/unet в реализации U-Net есть отличия от классической сети U-Net [7]:
• Вход сети представляет собой изображение размером 256 × 256 × 1.
• Вывод сети: 256 × 256 × $num\_classes$, где $num\_classes$ – количество классов.
• Функция активации сигмоида гарантирует, что маска находится в диапазоне [0, 1].
Кроме того, мы добавили пакетную нормализацию после каждой свертки и слоев активации ReLU.
3. ЭКСПЕРИМЕНТЫ И РЕЗУЛЬТАТЫ
3.1. Критерии оценки
Для оценки мы используем коэффициент сходства Дайса (DSC, коэффициент Серенсена) и индекс Жаккара (JAC), которые обычно используются в области сегментации медицинских изображений. Если мы определим true positive (TP), количество пикселей, правильно идентифицированных как целевой класс; true negative (TN), количество пикселей, правильно обнаруженных в качестве фона; false positive (FP), количество пикселей, ложно идентифицированных как целевой класс; false negative (FN), количество пикселей, ложно обнаруженных в качестве фона, мы можем определить метрики следующим образом:
Значения коэффициента Дайса, как и Жаккара, варьируются от нуля до единицы. В отличие от Жаккара, соответствующая разностная функция не является корректной метрикой расстояния, поскольку не удовлетворяет неравенству треугольника. Жаккар и Дайс эквивалентны в том смысле, что они могут быть выражены друг через друга:
Поскольку в нашем исследовании мы рассматриваем мультиклассовую сегментацию, нас интересуют мультиклассовые метрики. Поскольку расстояние Жаккара (или Дайса) сравнивает 2 множества, в случае многоклассовой классификации результатом будет вектор расстояний Жаккара (или Дайса) для каждого класса. При обучении нейронной сети для вычисления ошибки используется функция, возвращающая скалярное значение. Следовательно, для мультиклассовой сегментации необходимо свернуть вектор расстояния. Чтобы свернуть вектор в скаляр, мы используем линейную свертку:
В данной работе для функции ошибок используются коэффициенты линейной свертки Дайса с весовыми коэффициентами ${{\lambda }_{i}}$, равными $1{\text{/}}N$.
3.2. Эксперименты
Для получения оригинального тренировочного набора двадцать изображений тренировочного набора в высоком разрешении были нарезаны на фрагменты размером 256 × 256 с перекрытием в четверть размера фрагмента. Всего было получено 860 фрагментов. Для того чтобы дополнительно увеличить обучающую выборку были проведены следующие операции: случайные повороты изображений, случайные смещения и случайные изменения масштаба в небольшом диапазоне (5%).
Для получения смешанного тренировочного набора мы добавляем к оригинальным 860 фрагментам 100 фрагментов синтетического набора, получаем 960 фрагментов.
Из тренировочной выборки выделяется 20% изображений в проверочный набор, размер батча равнялся 7. Тестируем модель на 3 слоях (129 фрагментов). Мы используем оптимизатор Adam со скоростью обучения $2 \times {{10}^{{ - 5}}}$. Кривые обучения эксперимента вы можете увидеть на рис. 4 и 5.
Рис. 4.
Кривые обучения для оригинального набора данных слева направо: кривая обучения 6-классовой сегментации, кривая обучения 5-классовой сегментации, кривая обучения бинарной сегментации митохондрий.
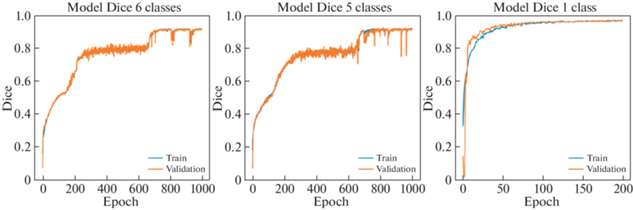
Рис. 5.
Кривые обучения для набора данных с добавлением синтетики слева направо: кривая обучения 6-классовой сегментации, кривая обучения 5-классовой сегментации, кривая обучения бинарной сегментации митохондрий.
Эксперимент 1. Классы сегментации (5): митохондрия вместе с границей, мембраны, PSD, оболочки аксонов, везикулы. Количество эпох: 200
Таблица 1.
Публичные размеченные наборы данных электронной микроскопии
| № | Название | Объем данных | Размеченный объем | Классы разметки | Разрешение нм/воксель |
|---|---|---|---|---|---|
| 1 | AC4, ISBI 2013 [5] | 4096 × 4096 × 1850 | 1024 × 1024 × 00 | мембраны | 6 × 6 × 30 |
| 2 | EPFLb, Lucchi [3] | 1065 × 2048 × 1536 | 2 набора 1024 × 768 × 165 | митохондрии | 5 × 5 × 5 |
| 3 | Kasthuri et al. [6] | 2 набора 1463 × 1613 × 85, 1334 × 1553 × 75 | митохондрии | 3 × 3 × 30 | |
| 4 | UroCella [20] | 1366 × 1180 × 1056 | 5 наборов 256 × 256 × 256 | митохондрии, эндолизосомы, веретеновидные везикулы | 16 × 16 × 15 |
0a Данные расположены на GitHub: https://github.com/MancaZerovnikMekuc/UroCell.
b Данные расположены по адресу: https://www.epfl.ch/labs/cvlab/data/data-em/
Эксперимент 2. Классы сегментации (6): митохондрия вместе с границами, граница митохондрии, мембраны, PSD, аксон, везикулы. Количество эпох: 1000.
Добавлен еще 1 класс митохондриальных границ.
Эксперимент 3. Класс сегментации: Митохондрия вместе с границей. Используется сегментация на митохондрии класса 1. Количество эпох: 1000.
Как видно из сравнительной таблицы (см. табл. 3), мультиклассовая сегментация митохондрий по качеству ненамного уступает бинарной сегментации.
Класс “границы митохондрий” является подклассом “митохондрии вместе с границами”, и это дополнительное выделение границ улучшает результаты сегментации объединяющего класса. Сеть обучалась на несбалансированных классах, так как размеры компартментов и их встречаемость в слое различаются в десятки раз.
4. ОБСУЖДЕНИЕ
В разделе обсуждения мы представляем таблицу: Таблица 3: “Сравнение результатов сегментации митохондрий”, в которой мы разместили наиболее показательные результаты по сегментации мембран с использованием бинарных и мультиклассовых моделей.
Мы тестируем наши модели на полном тестовом объеме EPFL и используем эти значения вместо результатов табл. 2. Мы не можем напрямую сравнивать результаты из табл. 3, потому что наши модели были обучены на значительно сокращенной версии набора данных EPFL. Но мы можем выдвинуть несколько гипотез, которые необходимо проверить. Наихудшие результаты были получены в слоях, содержащих аксон, участки митохондрий, митохондрии с более темными границами и включениями, чем на размеченных слоях, обрезанные границей изображения, нечеткие мембраны. Мы предполагаем, что разметка большего количества слоёв или генерация синтетических данных с надлежащими характеристиками улучшат результаты.
Таблица 2.
Результаты сегментации электронной микроскопии на оригинальном наборе данных (ORG) и на смешанном с синтетическими наборе данных (SYN), метрика: коэффициент Дайса
| Класс | Количество классов | |||||
|---|---|---|---|---|---|---|
| 6 | 5 | 2 | ||||
| ORG | SYN | ORG | SYN | ORG | SYN | |
| Митохондрии вместе с границей | 0.932 | 0.938 | 0.928 | 0.924 | 0.952 | 0.950 |
| Границы митохондрий | 0.742 | 0.748 | – | – | – | – |
| Мембраны | 0.743 | 0.750 | 0.741 | 0.701 | – | – |
| Постсинаптические уплотнения (PSD) | 0.553 | 0.745 | 0.728 | 0.633 | – | – |
| Везикулы | 0.709 | 0.711 | 0.671 | 0.688 | – | – |
| Оболочки аксонов | 0.228 | 0.790 | 0.098 | 0.716 | – | – |
Таблица 3.
Сравнение методов сегментации митохондрий
| Метод | Кол-во классов | Публикация | Набор данных | JAC | Dice |
|---|---|---|---|---|---|
| Cheng et al. (3D) [25] | 1 | M. Yuan et al. [29] | EPFL | 0.889 | 0.941 |
| 3D U-Net [11] | 1 | M. Yuan et al. [29] | EPFL | 0.878 | 0.935 |
| Cheng et al. (2D) [25] | 1 | M. Yuan et al. [29] | EPFL | 0.865 | 0.928 |
| U-Net [7] | 1 | M. Yuan et al. [29] | EPFL | 0.844 | 0.915 |
| U-Net | 1 | ours | EPFL | 0.840 | 0.911 |
| U-Net | 5 | ours | EPFL+syn | 0.840 | 0.910 |
| U-Net | 1 | ours | EPFL | 0.838 | 0.909 |
| U-Net | 6 | ours | EPFL | 0.833 | 0.908 |
| U-Net | 6 | ours | EPFL+syn | 0.833 | 0.908 |
| V-Net | 2 | M. Žerovnik Mekuč et al. [20] | UroCell | – | 0.898 |
| HighRes3DNet | 2 | M. Žerovnik Mekuč et al. [20] | UroCell | – | 0.883 |
| U-Net | 5 | ours | EPFL+syn | 0.793 | 0.882 |
| HighRes3DNet | 1 | M. Žerovnik Mekuč et al. [20] | UroCell | – | 0.862 |
| Lucchi et al. [3] | 1 | M. Yuan et al. [29] | EPFL | 0.755 | 0.86 |
| U-Net | 2 | M. Žerovnik Mekuč et al. [20] | UroCell | – | 0.855 |
5. ЗАКЛЮЧЕНИЕ
В ходе подготовки статьи выполнена ручная многоклассовая разметка 20 слоев тренировочного набора и 3 слоев тестового набора для известного датасета EPFL, который включает такие классы, как: 1) митохондрии, включая границы; 2) границы митохондрий; 3) мембраны; 4) постсинаптические уплотнения (PSD); 5) оболочки аксона; 6) везикулы. Разработано программное обеспечение для создания синтетических размеченных датасетов с теми же классами. Создан синтетический размеченный набор данных, включающий аксоны, PSD и мембраны.
Представлены результаты мультиклассовой сегментации данных электронной микроскопии головного мозга с использованием модифицированной U-Net с разбиением слоев данных на фрагменты размером 256 × 256 при сохранении исходного разрешения.
Исследование показало, что результаты бинарной, пятиклассовой и шестиклассовой сегментации близки по качеству: 0.911, 0.910 и 0.908 соответственно. На качество сегментации влияет наличие достаточного количества специфических признаков, отличающих выделенные классы, и представленность этих признаков в обучающей выборке.
Расширение набора данных синтетическими изображениями улучшает результаты классификации. Расширение датасета размеченного вручную (860 изображений 256 × 256) синтетическим датасетом (100 изображений 256 × 256, содержащих наименее представленные классы: аксоны, PSD, мембраны) дало существенный прирост точности шестиклассовой модели (см. табл. 2): с 0.228 до 0.790; с 0.553 до 0.745; с 0.743 до 0.750 соответственно, пропорционально ликвидированному дефициту. Исследование выполнено при поддержке гранта программы стратегического академического лидерства “Приоритет 2030” (проект Н-483-99_2021-2022).
Список литературы
Deerinck T. et al. Enhancing serial block-face scanning electron microscopy to enable high resolution 3D nanohistology of cells and tissues // Microscopy and Microanalysis. 2010. V. 16, S2. P. 1138–1139. https://doi.org/10.1017/S1431927610055170
Ciresan D.C. et al. Deep neural networks segment neuronal membranes in electron microscopy images // IN NIPS. 2012. P. 2852–2860.
Lucchi A. et al. Supervoxel-based segmentation of mitochondria in EM image stacks with learned shape features // IEEE Transactions on Medical Imaging. 2012. V. 31. № 2. P. 474–486. https://doi.org/10.1109/TMI.2011.2171705
Helmstaedter M., Mitra P.P. Computational methods and challenges for large-scale circuit mapping // Current Opinion in Neurobiology. 2012. V. 22. № 1. P. 162–169. ISSN 0959-4388. https://doi.org/10.1016/j.conb.2011.11.010. URL: http: //www.sciencedirect.com/science/article/pii/S0959438811002133; Neurotechnology.
Arganda-Carreras I. et al. Crowdsourcing the creation of image segmentation algorithms for connectomics // Frontiers in Neuroanatomy. 2015. V. 9. P. 1–13. ISSN 1662-5129. https://doi.org/10.3389/fnana.2015.00142. URL: https://www.frontiersin.org/article/10.3389/fnana.2015.00142.
Kasthuri N. et al. Saturated reconstruction of a volume of neocortex // Cell. 2015. V. 162. P. 648–661.
Ronneberger O., Fischer P., Brox T. U-Net: Convolutional Networks for Biomedical Image Segmentation. 2015. arXiv: 1505.04597 [cs.CV].
Drozdzal M. et al. The Importance of Skip Connections in Biomedical Image Segmentation. 2016. arXiv: 1608.04117 [cs.CV].
Fakhry A.E., Zeng T., Ji S. Residual deconvolutional networks for brain electron microscopy image segmentation // IEEE Transactions on Medical Imaging. 2017. V. 36. P. 447–456.
Xiao C. et al. Deep contextual residual network for electron microscopy image segmentation in connectomics // 2018 IEEE 15th International Symposium on Biomedical Imaging (ISBI 2018). 2018. P. 378–381. https://doi.org/10.1109/ISBI.2018.8363597.
Çiçek Ö. et al. 3D U-Net: Learning Dense Volumetric Segmentation from Sparse Annotation. 2016. arXiv: 1606.06650 [cs.CV].
Milletari F., Navab N., Ahmadi S.-A. V-Net: Fully Convolutional Neural Networks for Volumetric Medical Image Segmentation // 2016 Fourth International Conference on 3D Vision (3DV). 2016. P. 565–571.
Kamnitsas K. et al. Efficient multi-scale 3D CNN with fully connected CRF for accurate brain lesion segmentation // Medical Image Analysis. 2017. V. 36. P. 61–78.
Li W. et al. On the compactness, efficiency, and representation of 3D convolutional networks: Brain parcellation as a pretext task // Information Processing in Medical Imaging / Ed. by M. Niethammer Cham Springer International Publishing, 2017. P. 348–360.
Long J., Shelhamer E., Darrell T. Fully Convolutional Networks for Semantic Segmentation. 2015. arXiv: 1411.4038 [cs.CV].
Chen H. et al. Deep Contextual Networks for Neuronal Structure Segmentation // Proceedings of the Thirtieth AAAI Conference on Artificial Intelligence (AAAI-16). 2016. P. 1167–1173. URL: https://ojs.aaai.org/index.php/AAAI/article/view/10141/10000.
Liu T. et al. A modular hierarchical approach to 3D electron microscopy image segmentation // Journal of Neuroscience Methods. 2014. V. 226. P. 88–102.
Liu J. et al. Automatic detection and segmentation of mitochondria from SEM images using deep neural network // 2018 40th Annual International Conference of the IEEE Engineering in Medicine and Biology Society (EMBC). 2018. P. 628–631.
Oztel I. et al. Mitochondria segmentation in electron microscopy volumes using deep convolutional neural network // 2017 IEEE International Conference on Bioinformatics and Biomedicine (BIBM). 2017. P. 1195–1200. https://doi.org/10.1109/BIBM.2017.8217827.
Žerovnik Mekuč M. et al. Automatic segmentation of mitochondria and endolysosomes in volumetric electron microscopy data // Computers in Biology and Medicine. 2020. V. 119. P. 103693. ISSN 0010-4825. https://doi.org/10.1016/j.compbiomed. 2020. 103693. URL: https://www.sciencedirect.com/science/article/pii/S0010482520300792.
Huang S.-C., Cheng F., Chiu Y. Efficient contrast enhancement using adaptive gamma correction with weighting distribution // IEEE Transactions on Image Processing. 2013. V. 22. P. 1032–1041.
Szegedy C. et al. Rethinking the Inception Architecture for Computer Vision // 06/2016. https://doi.org/10.1109/CVPR.2016.308
Chollet F. Xception: Deep Learning with Depthwise Separable Convolutions // 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). 2017. P. 1800–1807.
Xie S. et al. Aggregated Residual Transformations for Deep Neural Networks // 2017 IEEE Conference on Computer Vision and Pattern Recognition (CVPR). 2017. P. 5987–5995.
Cheng H.-C., Varshney A. Volume Segmentation Using Convolutional Neural Networks with Limited Training Data // 2017 IEEE International Conference on Image Processing (ICIP). 2017. P. 590–594. https://doi.org/10.1109/ICIP.2017.8296349.
Urakubo H. et al. UNI-EM: An environment for deep neural network-based automated segmentation of neuronal electron microscopic images // Scientific Reports. 2019. V. 9. P. 19413. DOI: . URL: https://www.biorxiv.org/content/biorxiv/early/2019/04/12/607366.full.pdf.https://doi.org/10.1038/s41598-019-55431-0
Gómez-de-Mariscal E. et al. Deep-learning-based segmentation of small extracellular vesicles in transmission electron microscopy images // Scientific Reports. 2019. V. 9. URL: https://doi.org/10.1038/s41598-019-49431-3
Quan T.M., Hildebrand D.G.C., Jeong W.-K. FusionNet: A deep fully residual convolutional neural network for image segmentation in connectomics // Frontiers in Computer Science. 2021. V. 3. P. 34. ISSN 2624-9898. https://doi.org/10.3389/fcomp.2021.613981. URL: https://www.frontiersin.org/article/10.3389/fcomp.2021. 613981.
Yuan Z. et al. HIVE-Net: Centerline-aware HIerarchical view-ensemble convolutional network for mitochondria segmentation in EM images // Computer Methods and Programs in Biomedicine. 2021. V. 200. P. 105925.
Дополнительные материалы отсутствуют.
Инструменты
Программирование


