Успехи современной биологии, 2019, T. 139, № 2, стр. 107-113
Эпигенные сети: теория, модели, эксперимент
А. В. Галимзянов 1, *, Е. Э. Ступак 1, Р. Н. Чураев 1
1 Уфимский Институт биологии Уфимского федерального исследовательского центра РАН
Уфа, Россия
* E-mail: galim@anrb.ru
Поступила в редакцию 21.11.2018
После доработки 21.11.2018
Принята к публикации 25.12.2018
Аннотация
Работа посвящена описанию концепции эпигенных сетей, направленной на понимание феномена наследственности. Приведены результаты многолетних исследований управляющих генных и эпигенных сетей, полученные авторами благодаря интегративному подходу с использованием аппарата математической биологии, кибернетики, генной инженерии и биоинформатики. Эпигенные сети – управляющие генные сети, обладающие эпигенетическими свойствами или содержащие эпигены. Под эпигеном понимаются особые наследственные единицы, имеющие не менее двух режимов функционирования подчиненных им генов, способные сохранять каждый из режимов в последовательном ряду поколений. Эпигены являются ячейками функциональной наследственной памяти, выполняя особую роль в онто- и филогенезе. Эпигенетические системы можно проектировать и синтезировать экспериментально. Одной из таких систем является двухкомпонентный стационарный эпиген с заданными и управляемыми наследуемыми динамическими свойствами, функционирующий in vivo. Искусственные эпигенетические конструкции могут служить моделями механизмов дифференцировки и клеточной памяти, а также использоваться в качестве бактериальных биосенсоров. Обобщенные пороговые модели природных и синтетических молекулярно-генетических систем управления экспрессией генов обладают как аналитической, так и прогностической ценностью.
ВВЕДЕНИЕ
С открытием в 1953 году Уотсоном и Криком пространственной структуры молекулы ДНК в генетике начало формироваться направление, связанное с изучением надгенных систем, осуществляющих контроль за функциональными состояниями генов, причем сразу с опорой на теоретические изыскания. Уже в 1961 году Жакоб и Моно описали лактозный оперон у Escherichia coli (Jacob, Monod, 1961), а также представили гипотетическую модель альтернативной биохимической регуляции белкового синтеза – молекулярный триггер (Monod, Jacob, 1961), математическое исследование которого провели Саймон (Simon, 1965) и Д.С. Чернавский с коллегами (Григоров и др., 1967). В это время В.А. Ратнер, основываясь на приложении кибернетического подхода к молекулярной генетике (Ляпунов, 1963), сформулировал концепцию молекулярно-генетических систем управления (МГСУ) (Ратнер, 1966), а Кауфман ввел в поле зрения науки новый объект – генные сети (ГС) (Kauffman, 1969). С позиций теории МГСУ В.А. Ратнером и Р.Н. Чураевым была исследована генетическая система, контролирующая онтогенез у фага лямбда (Ратнер, 1970; Чураев, Ратнер, 1975; Ratner, Tchuraev, 1978), обосновано существование двух-оперонного триггера (Ратнер, Чураев, 1971). Вскоре Р.Н. Чураев выдвинул гипотезу об эпигене (Чураев, 1975а), поставил задачу синтеза эпигенов и предложил новаторскую методологию ее решения (Чураев, 1981), параллельно создавая динамическую теорию молекулярно-генетических систем управления (Чураев, 1987). В серии работ Яблонки и Ламб (Jablonka, Lamb, 1989, 1995; Jablonka et al., 1992) изучались системы, обеспечивающие стабильное наследование функциональных состояний в клеточных линиях, названные авторами эпигенетическими наследственными системами. Начиная с 2000-х гг., в проблематику теоретического подхода входят также вопросы интеграции и анализа больших данных по структурно-функциональной организации молекулярно-генетических систем (D’Souza et al., 2017), построения их комплексных, масштабируемых моделей (Glont et al., 2018; Khatamian et al., 2018), определяя содержание биоинформатики и компьютерной системной биологии. В этом направлении в России действует научная школа под руководством акад. Н.А. Колчанова (Системная…, 2008; Orlov et al., 2017).
Настоящая статья описывает основные положения, этапы и достижения теории эпигенов и эпигенных сетей, построенной на обширном материале классической и современной молекулярной генетики, с использованием теории управления, математических методов, компьютерного моделирования и оригинальных экспериментальных доказательств in vivo.
ЭПИГЕНЫ
Ядром теории является новый класс наследственных единиц – эпигены, в которых часть наследственной информации хранится, кодируется и передается потомству вне первичной структуры молекул ДНК генома. Гипотеза об эпигене была выдвинута в 1975 году (Чураев, 1975а). Согласно этой гипотезе, эпигены – особые наследственные единицы (циклические генные системы), имеющие не менее двух режимов функционирования подчиненных им генов, способные сохранять каждый из режимов в последовательном ряду поколений. Введен термин “эпигенотип” – как перечень генов, входящих в эпигены, с указанием их функционального состояния (включен/выключен). Проанализированы схемы скрещиваний особей с различными эпигенотипами и обнаружены события, выходящие за рамки законов Менделя, в частности поглощение и образование эпигетерозигот.
Основным свойством эпигена является способность сохранять функциональное состояние после прекращения вызвавшего его внешнего воздействия. Для сохранения измененного состояния эпигена изменение первичной последовательности входящих в него генов не является необходимым. Строго показано отличие эпигенов от генов: они различаются по способу кодирования генетической информации; содержат разную “информацию по смыслу”, которая записана в разных алфавитах разными кодами; переход из одного функционального состояния в альтернативные детерминирован и обратим. Эти свойства эпигенов инвариантны относительно конкретных молекулярных механизмов, их реализующих. Эпигены – надгенные системы, то есть состоят из генов или их частей. Эпигены и гены – наследственные единицы разных рангов.
Согласно структурно-функциональной классификации эпигенов (Чураев, 1982), выделяются эпигены стационарные и нестационарные, сцепленные и несцепленные, элементарные и сложные; введена операция декомпозиции сложных эпигенов. В той же работе была поставлена задача синтеза из оперонов сложных эпигенов, способных хранить заданные режимы функционирования подчиненных им генов. Все вместе привело к идее эпигенной инженерии – нового подхода к управлению частью наследственной информации, основанного на синтезе эпигенов с заданными управляемыми наследуемыми режимами функционирования.
В развитие идеи эпигенной инженерии и в целях экспериментального доказательства существования эпигенов в лаборатории математической и молекулярной генетики Уфимского Института биологии для клеток E. coli была сконструирована рекомбинантная плазмида pCEPI, содержащая циклическую дигенную систему с отрицательными обратными связями (ЦДС(–)) (Чураев и др. 2001; Tchuraev et al., 2000; Stupak Е., Stupak I., 2006). Структурно-функциональная организация ее такова: один ген кодирует репрессор для другого гена, кодирующего репрессор для первого гена. Эксперименты in vivo показали, что эта система способна поддерживать в ряду последовательных клеточных делений два альтернативных уровня экспрессии генов и, соответственно, два фенотипа клетки, то есть является эпигеном. Функционально ЦДС(−) могут действовать в клетках как ячейки клеточной памяти или генетические переключатели. На основе ЦДС(–) создана экспериментальная молекулярная модель самодифференцировки клеток. В данной модели особое – метастабильное состояние управляющей генной (эпигенной) сети позволяет клеточной популяции после ряда генераций разделиться на две субпопуляции с копиями эпигенов, находящихся в альтернативных функциональных состояниях (Чураев и др., 2006). Также экспериментально показана возможность использования ЦДС(−) в качестве управляющего элемента бактериального биосенсора (Ступак Е., Ступак И., 2012), обладающего памятью на генотоксическое воздействие. Биосенсоры такого класса можно применять для мониторинга временного присутствия генотоксикантов в среде и тестирования последствий кратковременного воздействия токсических веществ.
В рамках теории доказано существование общей наследственной памяти (ОНП), обеспечивающей хранение наследственной информации в некоторых кодах в онтогенезе и при передаче ее от одного поколения к другому (Чураев, 2005, 2010). Общая наследственная память состоит из двух подпамятей – структурной и функциональной. Ячейками первой являются гены, кодирующие информацию структурным способом. Материальным воплощением ячеек второй являются эпигены, кодирующие функциональную информацию, в частности динамическим способом (то есть путем циркулирования сигналов в циклической системе элементов). Функциональная наследственная память как категория – это множество функционально эквивалентных молекулярных механизмов, обеспечивающих кодирование, хранение и передачу в ряду генераций функциональной информации. В работах (Чураев, 1997, 2005) рассмотрены примеры трех классов реально существующих эпигенетических механизмов: динамический – хранение информации в циклических системах генов динамическим способом; транспозиционный, основанный на сайт-специфической транспозиции, и модификационный, когда модифицированные состояния ДНК или хромосомных белков носят обратимый характер. Динамический способ хранения и реализации наследственной информации нашел свое отражение в концепции генных и эпигенных сетей.
ГЕННЫЕ И ЭПИГЕННЫЕ СЕТИ
Согласно одному из постулатов теории, в каждой клетке любого организма можно выделить управляющую систему, функция которой – управление быстрыми метаболическими и медленными онтогенетическими процессами. В клеточной управляющей подсистеме в качестве элементов рассматриваются генетические блоки (G-блоки). Генетический блок – это ген, взятый в совокупности с механизмами транскрипции, процессинга, транспорта и депо конечного продукта. Передача сигналов от одного G-блока к другому осуществляется посредством регуляторных молекул разной степени специфичности. Гены (генетические блоки) и функциональные информационные связи между ними (положительные и отрицательные) образуют клеточную генную сеть. Структура генных продуктов и регуляторных фрагментов геномной ДНК однозначно определяет структуру клеточной генной сети.
Динамика генной сети характеризуется значениями концентраций ее макромолекулярных компонентов (ДНК, РНК, белков) и уровнями активности промоторов генов во времени. Для моделирования динамики генных сетей, управляющих метаболическими, онтогенетическими и наследственными процессами, приводящими к самовоспроизведению, разработан формализм обобщенных пороговых моделей (ОПМ) (Чураев, 1986, 1993; Tchuraev, 1991; Tchuraev, Galimzyanov, 2009).
В работе (Tchuraev, 2006) выявлены пять принципов строения внутриклеточных генных сетей: подчиненность каждого генетического блока управляющему контуру, модульность (элементы трех видов – генетические триггеры, осцилляторы и логические комбинаторы с задержкой), иерархия по времени, гетерархичность, соответствие структуры и онтогенетической функции. Сформулированы также общие законы функционирования эукариотических управляющих клеточных генных сетей.
На теоретических математических моделях доказано существование в произвольных клеточных генных сетях контуров циклических систем, управляющих экспрессией генов. Также доказано, что некоторые из этих контуров обладают свойствами ячеек динамической памяти, способных запоминать (сохранять) более одного режима функционирования и наследовать эти режимы в последовательном ряду генераций. Такие циклические системы относятся к классу динамических эпигенов.
В работе (Tchuraev, 2006) предложена процедура трансформации внутриклеточной управляющей генной сети в сеть следующего уровня иерархии, элементами которой будут генетические триггеры (бистабильные ячейки памяти), осцилляторы и логические комбинаторы. Первый и второй из них представляют собой стационарные и нестационарные эпигены соответственно. Трансформированные сети целесообразно представлять как эпигенные сети, элементы которых – эпигены.
Рассматривая эпигенные сети в онтогенетическом аспекте, их можно приравнять к механизмам клеточной памяти – системам поддержания детерминированных состояний в клональных производных родительских клеток. В этом направлении решена задача синтеза простейшей эпигенной сети, способной обеспечить три основных свойства любого эукариотического организма: дивергентную детерминацию, сохранение детерминированных состояний и воспроизведение исходного “зиготического” функционального состояния (Tchuraev, 1980). Стоит подчеркнуть, что такая система не требует функциональной генетической гетерогенности для клеточной дифференцировки, в частности ооплазматической сегрегации и межклеточных взаимодействий, то есть весьма корректно объясняет причину того, каким образом в отдельных исходно идентичных клетках активируются различные группы генов.
Большую роль эпигены и эпигенные системы могут играть и в филогенезе. В работе (Чураев, 1987) предполагается существование дополнительного эволюционного механизма формирования наследственных программ онтогенеза, основанного на проявлениях функциональной наследственной памяти. Эпигенные сети могут служить основой недарвиновских стратегий эволюции посредством “запоминания относительно неудачных ходов эволюции” и сохранения запасных вариантов онтогенеза. Представленная теория также объясняет отдельные феномены наследования приобретенных признаков и молекулярные механизмы стресс-индуцируемой эволюции.
Таковы, в целом, контуры оригинальной теории. Необходимо отметить, что концепция эпигенов и эпигенных сетей согласуется с экспериментальным материалом эпигенетики (опероны, метилирование ДНК, гистоновый код), обладает прогностической ценностью, являясь дедуктивной теорией. В свою очередь, сетевая парадигма (многоуровневые сети, “сети сетей”) (De Domenico, 2018) переместилась в центр современной генетики и служит основой для перспективных исследований в области системной и синтетической биологии.
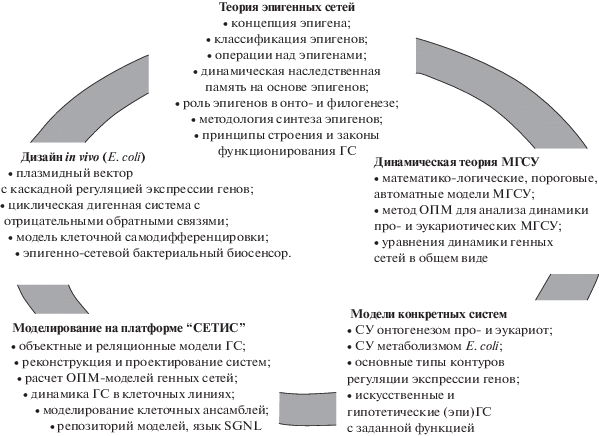
Основное содержание теоретического, экспериментального и биоинформационного направлений в комплексном исследовании генных и эпигенных сетей отражено на рисунке.
МОДЕЛИ МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИХ СИСТЕМ
В настоящее время накапливаются большие массивы экспериментальных данных по структуре и функциям молекулярно-генетических систем (Игнатьева и др., 2017). Их чрезвычайная сложность, в частности, многокомпонентность, разнородность и взаимосвязанность элементов, требует развития специальных инструментов для освоения фактического материала – математических методов и компьютерных программ.
Развитие математических методов анализа природных МГСУ осуществляется в рамках динамической теории молекулярно-генетических систем управления, сопутствующей теории эпигенов. Начиная с 1970-х гг. были последовательно разработаны автоматные (Чураев, Ратнер, 1972а, б), математико-логические (Чураев, 1975б) и пороговые (Ratner, Tchuraev, 1978) модели молекулярных систем управления, метод обобщенных пороговых моделей для исследования прокариотических (Чураев, 1986; Tchuraev, 1991) и эукариотических МГСУ (Чураев, 1993), ОПМ с многоуровневой экспрессией генов (Tchuraev, Galimzyanov, 2009). Отличительная особенность ОПМ-формализма заключается в том, что он объединяет в себе преимущества дискретных и непрерывных методов. В моделях учитываются динамические характеристики основных молекулярно-генетических процессов, модульная организации регуляторных районов, многоуровневая экспрессия и мультикопийность генов. Среди решаемых задач: расчет кинетических кривых для концентраций генных продуктов (РНК, белков), поиск стационарных состояний – выявление паттернов активности генов во времени, оценка параметрической устойчивости.
Одновременно с расширением, совершенствованием методической базы происходило ее апробирование на природных МГСУ про- и эукариот. Первая в мире портретная модель молекулярно-генетической системы управления развитием фага лямбда (и одна из первых моделей генных сетей) была опубликована вместе с гипотезой об эпигене (Чураев, Ратнер, 1975) и потом расширялась (Kananyan et al., 1981). Геном фага содержит 48 генов (для сравнения, геном E. coli насчитывает более 4.5 тыс. генов), но имеет сложную управляющую систему. Полученные в модели результаты показали, что надежность функционирования фага как раз и обеспечивается через параметрическую устойчивость его управляющей системы. По сути, впервые на фактическом экспериментальном материале, на примере конкретного организма, демонстрировалось важнейшее свойство живых систем: как через неустойчивые взаимодействия неустойчивых элементов в неустойчивых условиях получается устойчивый результат.
В разное время на языке формализма обобщенных пороговых моделей были исследованы особенности функционирования природных прокариотических генных сетей – двухоперонного триггера (Ратнер, Чураев, 1971), фосфатного регулона (Прокудина, Чураев, 1989), молекулярно-генетических систем управления биосинтезом триптофана (Чураев, Прокудина, 1989) и катаболизмом L-арабинозы у E. coli (Prokudina et al., 1991), транспозонов семейства Tn3 и кворум-чувствительной системы индукции свечения у Vibrio fischeri. Также изучены на качественном и количественном уровнях циклические моногенные системы с одним и двумя уровнями перекодирования (Чураев, 1986), искусственный генетический переключатель с метастабильным состоянием, реализующий молекулярную модель самодифференцировки клеток (бифуркатор) (Чураев и др., 2006; Galimzyanov, 2006), гипотетическая пятиоперонная система, способная к дифференцировке и воспроизведению исходного состояния, то есть обладающая функционалом стволовых клеток с единичным дифференцирующим потенциалом (Tchuraev, 1980). Исследования эукариотических систем управления экспрессией генов проведены на примере генных сетей, управляющих онтогенетическими процессами у Drosophila melanogaster и Arabidopsis thalianа (Чураев, Галимзянов, 2001, 2003; Tchuraev, Galimzyanov, 2003; Galimzyanov, Tchuraev, 2004). Все эти модели обладают как аналитической, так и прогностической ценностью.
В направлении компьютерного моделирования генных и эпигенных сетей созданы программный комплекс и Java-платформа “СЕТИС”, базы данных “Генные сети” и “Клеточные ансамбли”, репозиторий моделей, а также язык SETIES Standard Gene Network Language (SGNL) для их комплексной репрезентации (Галимзянов, Чураев, 2007; Галимзянов, 2015, 2016a,б). Семейство прог-рамм “СЕТИС” объединяет компоненты для реконструкции и визуализации природных и синтетических (эпи)генных сетей, автоматического построения и анализа их обобщенных пороговых моделей, прогнозирования поведения реальных систем, моделирования динамики генных сетей в клеточных линиях и в клеточных ансамблях. Вместе они позволяют исследовать особенности функционирования молекулярно-генетических систем в норме, с учетом мутагенеза и трансгенеза, дискриминировать гипотезы о возможных механизмах взаимодействия между генами-регуляторами и генами-мишенями, тестировать генные сети на эпигенетические свойства, моделировать эпимутации, изучать динамическую память клеточной популяции, учитывать модуляцию шумовых эффектов, исследовать работу генов онтогенеза и в целом изучать особенности сетевого механизма реализации наследственной информации.
ЗАКЛЮЧЕНИЕ
Подводя итог, можно сказать, что представленный подход способствует пониманию функционирования управляющих механизмов в генных и эпигенных сетях. Теория эпигенов и сопутствующая ей динамическая теория молекулярно-генетических систем управления экспрессией генов ориентированы на получение новых знаний о принципах структурной организации и законах функционирования генных и эпигенных сетей и в целом о феномене наследственности. Развитие экспериментальных in vivo и компьютерных моделей клеточных сетевых структур в сопряжении с фенотипическими свойствами живых систем обогащает инструментарий для решения фундаментальных и прикладных задач системной биологии, биомедицины и экологии.
Список литературы
Галимзянов А.В. Java-платформа (программное ядро) “SETIES 5.0 J-Core” для исследования молекулярно-генетических систем // Офиц. бюл. Роспатента “Программы для ЭВМ. Базы данных. Топологии интегральных микросхем”. 2015. № 10(108) 2015, 20.10.2015. Гос. регистрация прогр. для ЭВМ № 2 015 660 238.
Галимзянов А.В. База данных “Генные сети” программного комплекса “СЕТИС” // Офиц. бюл. Роспатента “Программы для ЭВМ. Базы данных. Топологии интегральных микросхем”. 2016a. № 1(111) 2016, 20.01.2016. Гос. регистрация базы данных № 2 015 621 810.
Галимзянов А.В. База данных “Клеточные ансамбли” программного комплекса “СЕТИС” // Офиц. бюл. Роспатента “Программы для ЭВМ. Базы данных. Топологии интегральных микросхем”. 2016б. № 1(111) 2016, 20.01.2016. Гос. регистрация базы данных № 2 015 621 809.
Галимзянов А.В., Чураев Р.Н. Программный комплекс для исследования генных сетей, динамических эпигенов и клеточных ансамблей “СЕТИС” // Офиц. бюл. Роспатента “Программы для ЭВМ. Базы данных. Топологии интегральных микросхем”. 2007. № 4. Офиц. регистрация прогр. для ЭВМ № 2 007 611 213.
Григоров Л.Н., Полякова М.С., Чернавский Д.С. Модельное исследование триггерных схем и процесса дифференциации // Мол. биол. 1967. Т. 1. № 3. С. 410–418.
Игнатьева Е.В., Афонников Д.А., Колчанов Н.А. Интернет-доступные информационные ресурсы по генным сетям, включающие данные по человеку и животным // Вавил. журн. ген. и сел. 2017. Т. 21. № 8. P. 895–902. DOI: https://doi.org/10.18699/VJ17.310
Ляпунов А.А. Об управляющих системах живой природы и общем понимании жизненных процессов // Проблемы кибернетики. 1963. Вып. 10. С. 179–193.
Прокудина Е.И., Чураев Р.Н. Моделирование бактериального фосфатного регулона методом обобщенных пороговых моделей: Препринт. Уфа: БНЦ УрО АН СССР, 1989. 26 с.
Ратнер В.А. Генетические управляющие системы. Новосибирск: Наука, 1966. 181 с.
Ратнер В.А. О некоторых особенностях управления онтогенезом у фагов λ и Т4 // Онтогенез. 1970. Т. 1. № 2. С. 166–175.
Ратнер В.А., Чураев Р.Н. Существует ли двухоперонная система управления (триггер)? Некоторые факты и эвристическое значение триггера // Генетика. 1971. Т. 7. № 9. С. 175–179.
Системная компьютерная биология / Ред. Н.А. Колчанов, С.С. Гончаров, В.А. Лихошвай, В.А. Иванисенко; Рос. акад. наук, Сиб. отд-ние, Ин-т цитологии и генетики (и др.). Новосибирск: Изд-во СО РАН, 2008. 769 с. (Интеграционные проекты СО РАН; вып. 14).
Ступак Е.Э., Ступак И.В. Циклическая дигенная система как управляющий элемент бактериального биосенсора // Прикл. биохим. микробиол. 2012. Т. 48. № 1. С. 18–22.
Чураев Р.Н. Гипотеза о эпигене // Исследования по математической генетике / Ред. В.А. Ратнер. Новосибирск: ИЦиГ СО АН СССР, 1975a. С. 77–94.
Чураев Р.Н. Математико-логические модели молекулярных систем управления // Исследования по математической генетике / Ред. В.А. Ратнер. Новосибирск: ИЦиГ СО АН СССР, 1975б. С. 67–76.
Чураев Р.Н. О синтезе эпигенов: Препринт. Новосибирск: ИЦиГ СО АН СССР, 1981. 35 с.
Чураев Р.Н. Прикладные аспекты концепции эпигенов // Журн. общ. биол. 1982. Т. 43. № 1. С. 79–87.
Чураев Р.Н. Метод обобщенных пороговых моделей для анализа динамики молекулярно-генетических систем управления: Препринт. Уфа: БФАН СССР, 1986. 21 с.
Чураев Р.Н. Моделирование молекулярно-генетических систем управления: Дис. … д-ра биол. наук. Уфа: БФАН СССР, 1987. 414 с.
Чураев Р.Н. Метод обобщенных пороговых моделей для анализа динамики эукариотических молекулярно-генетических систем управления: Препринт. Уфа: УНЦ РАН, 1993. 32 с.
Чураев Р.Н. Элементы неканонической теории наследственности: Препринт. Уфа: УНЦ РАН, 1997. 54 с.
Чураев Р.Н. Контуры неканонической теории наследственности: от генов к эпигенам // Журн. общ. биол. 2005. Т. 66. № 2. С. 99–122.
Чураев Р.Н. Эпигены – наследственные единицы надгенного уровня // Экол. генетика. 2010. Т. VIII. № 4. С. 17–24.
Чураев Р.Н., Галимзянов А.В. Моделирование реальных эукариотических управляющих генных подсетей на основе метода обобщенных пороговых моделей // Мол. биол. 2001. Т. 35. № 6. С. 1088–1094.
Чураев Р.Н., Галимзянов А.В. О параметрической устойчивости генных сетей, управляющих онтогенетическими процессами // Мол. биол. 2003. Т. 37. № 1. С. 8–96.
Чураев Р.Н., Прокудина Е.И. Моделирование системы управления биосинтезом триптофана методом обобщенных пороговых моделей // Генетика. 1989. Т. 25. № 3. С. 535–543.
Чураев Р.Н., Ратнер В.А. Моделирование молекулярно-генетических систем управления на языке теории автоматов. Сообщение 1. Опероны и оперонные системы // Исследования по теоретической генетике / Ред. В.А. Ратнер. Новосибирск: ИЦиГ СО АН СССР, 1972a. С. 210–227.
Чураев Р.Н., Ратнер В.А. Моделирование молекулярно-генетических систем управления на языке теории автоматов. Сообщение 2. Ферменты и полиферментные системы // Исследования по теоретической генетике / Ред. В.А. Ратнер. Новосибирск: ИЦиГ СО АН СССР, 1972б. С. 228–239.
Чураев Р.Н., Ратнер В.А. Моделирование динамики системы управления развитием λ-фага. // Исследования по математической генетике / Ред. В.А. Ратнер. Новосибирск: ИЦиГ СО АН СССР, 1975. С. 5–66.
Чураев Р.Н., Ступак И.В., Тропынина Т.С., Ступак Е.Э. Сконструирован двухкомпонентный эпиген с наперед заданными свойствами // ДАН. 2001. Т. 378. № 6. С. 837–840.
Чураев Р.Н., Ступак И.В., Ступак Е.Э., Галимзянов А.В. Новое свойство эпигенов: метастабильные эпигенотипы // ДАН. 2006. Т. 406. № 4. С. 570–573.
Galimzyanov A.V. “GREENCE” technology for in silico analysis of gene network dynamics in individual cells of a clonal population // Biophysics (Biofizika). 2006. V. 51. Suppl. 1. P. S66-S69. DOI: https://doi.org/10.1134/S0006350906070141
Galimzyanov A.V., Tchuraev R.N. An investigation of structural stability of Drosophila control gene subnetwork in computer experiments // Bioinformatics of genome regulation and structure / Eds N. Kolchanov, R. Hofestaedt. Boston / Dordrecht / London: Kluwer Acad. Publishers, 2004. P. 301–309.
D’Souza M., Sulakhe D., Wang S., Xie B. et al. Strategic integration of multiple bioinformatics resources for system level analysis of biological networks // Methods Mol. Biol. 2017. V. 1613. P. 85–99. DOI: https://doi.org/10.1007/978-1-4939-7027-8_5
De Domenico M. Multilayer network modeling of integrated biological systems: Comment on “Network science of biological systems at different scales: A review” by Gosak et al. // Phys. Life Rev. 2018. V. 24. P. 149–152. DOI: https://doi.org/10.1016/j.plrev.2017.12.006
Glont M., Nguyen T.V.N., Graesslin M. et al. BioModels: expanding horizons to include more modelling approaches and formats // Nucleic Acids Res. 2018. V. 46. Database issue. P. D1248–D1253. DOI: https://doi.org/10.1093/nar/gkx1023
Jablonka E., Lamb M. The inheritance of acquired epigenetic variations // J. Theor. Biol. 1989. V. 139. № 1. P. 69–83. DOI: https://doi.org/10.1016/S0022-5193(89)80058-X
Jablonka E., Lamb M. Epigenetic inheritance and evolution: the Lamarckian dimension. Oxford: Oxford Univ. Press, 1995. 346 p.
Jablonka E., Lachmann M., Lamb M. Evidence, mechanisms and models for the inheritance of acquired characters // J. Theor. Biol. 1992. V. 158. № 2. P. 245–268. DOI: https://doi.org/10.1016/S0022-5193(05)80722-2
Jacob F., Monod J. Genetic regulatory mechanisms in the synthesis of proteins // J. Mol. Biol. 1961. V. 3. P. 318–356. DOI: https://doi.org/10.1016/S0022-2836(61)80072-7
Kananyan G.Kh., Ratner V.A, Tchuraev R.N. Enlarged model of lambda phage ontogenesis // J. Theor Biol. 1981. V. 88. № 3. P. 393–407. DOI: https://doi.org/10.1016/0022-5193(81)90273-3
Kauffman S.A. Metabolic stability and epigenesis in randomly constructed genetic nets // J. Theor. Biol. 1969. V. 22. № 3. P. 437–467. DOI: https://doi.org/10.1016/0022-5193(69)90015-0
Khatamian A., Paull E.O., Califano A., Yu J. SJARACNe: a scalable software tool for gene network reverse engineering from big data // Bioinformatics. 2018. DOI: https://doi.org/10.1093/bioinformatics/bty907
Monod J., Jacob F. General conclusions: teleonomic mechanisms in cellular metabolism, growth, and differentiation // Cold Spring Harbor Symposia on Quantitative Biology. 1961. V. 26. P. 389–401. DOI: https://doi.org/10.1101/sqb.1961.026.01.048
Orlov Y.L., Kolchanov N.A., Hofestädt R., Wong L. Editorial – Bioinformatics development at the BGRS/SB conference series: 10th anniversary // J. Bioinform. Comput. Biol. 2017. V. 15. № 2. P. 1 702 001. DOI: https://doi.org/10.1142/S0219720017020012
Prokudina E.I., Valeev R.Y., Tchuraev R.N. A new method for the analysis of the dynamics of the molecular genetic control systems. II. Application of the method of generalized threshold models in the investigation of concrete genetic systems // J. Theor. Biol. 1991. V. 151. № 1. P. 89–110. DOI: https://doi.org/10.1016/S0022-5193(05)80145-6
Ratner V.A., Tchuraev R.N. Simplest genetic systems controlling ontogenesis: organization principles and models of their function // Progress in Theoretical Biology. N.Y.: Acad. Press, 1978. V. 5. P. 82–127.
Simon Z. Multi-steady-state model for cell differentiation // J. Theor. Biol. 1965. V. 8. № 2. P. 258–263. DOI: https://doi.org/10.1016/0022-5193(65)90076-7
Stupak E.E., Stupak I.V. Inheritance and state switching of genetic toggle switch in different culture growth phases // FEMS Microbiol Lett. 2006. V. 258. № 1. P. 37–42. DOI: https://doi.org/10.1111/j.1574-6968.2006.00194.x
Tchuraev R.N. On a stochastic model of a molecular genetic system capable of differentiation and reproduction of the initial state // Biom. J. 1980. V. 22. № 2. P. 189–194.
Tchuraev R.N. A new method for the analysis of the dynamics of the molecular genetic control systems. I. Description of the method of generalized threshold models // J. Theor. Biol. 1991. V. 151. № 1. P. 71–87. DOI: https://doi.org/10.1016/S0022-5193(05)80144-4
Tchuraev R.N. General principles of organization and laws of functioning in governing gene networks // Bioinformatics of genome regulation and structure II / Eds N. Kolchanov, R. Hofestaedt. N.Y.: Springer Science Media, Inc. 2006. P. 367–377.
Tchuraev R.N., Galimzyanov A.V. Parametric stability evaluation in computer experiments on the mathematical model of Drosophila control gene subnetwork // In Silico Biol. 2003. V. 3. P. 101–115.
Tchuraev R.N., Galimzyanov A.V. Gene and epigene networks: two levels in organizing the hereditary system // J. Theor. Biol. 2009. V. 259. № 4. P. 659–669. DOI: https://doi.org/10.1016/j.jtbi.2009.03.034
Tchuraev R.N., Stupak I.V., Tropynina T.S., Stupak E.E. Epigenes: design and construction of new hereditary units // FEBS Lett. 2000. V. 486. P. 200-202. DOI: https://doi.org/10.1016/S0014-5793(00)02300-0
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии