Успехи современной биологии, 2020, T. 140, № 3, стр. 211-224
Гены аминогликозидфосфотрансфераз у почвенных бактерий рода Streptomyces
Н. Н. Рудакова 1, *, М. Г. Алексеева 1, В. Н. Даниленко 1
1 Институт общей генетики им. Н.И. Вавилова РАН
Москва, Россия
* E-mail: natachka92@mail.ru
Поступила в редакцию 14.01.2020
После доработки 14.01.2020
Принята к публикации 14.01.2020
Аннотация
Устойчивость к антибиотикам является одной из самых больших проблем в современной медицине и глобальной угрозой для здравоохранения. Серьезную угрозу для антимикробной терапии в настоящее время представляют аминогликозидфосфотрансферазы (Aph), поэтому изучение функций и получение 3D-структур аминогликозидфосфотрансфераз является важной и актуальной задачей, позволяющей развивать подходы для преодоления резистентности к аминогликозидным антибиотикам. Почвенные актинобактерии рода Streptomyces содержат наибольшее количество генов aph, данные гены могли передаться им из штаммов-продуцентов антибиотиков. В обзоре проведен анализ современных сведений по актинобактериям рода Streptomyces как резервуара генов лекарственной устойчивости, а также подходов к выявлению генов aph, связанных с устойчивостью к аминогликозидным антибиотикам, на примере модельного штамма S. rimosus АТСС 10970 (продуцента окситетрациклина). Обсуждаются данные по созданию тест-систем для скрининга ингибиторов – потенциальных лекарственных препаратов. Ингибирование белков, обеспечивающих природный уровень устойчивости бактерий к ряду аминогликозидных антибиотиков, могло бы способствовать преодолению множественной лекарственной устойчивости патогенных актинобактерий и расширению спектра применяемых препаратов за счет синергичного эффекта антибиотика с соединением-ингибитором Aph.
ВВЕДЕНИЕ
Устойчивость к антибиотикам является одной из самых больших проблем в современной медицине и глобальной угрозой для здравоохранения. В соответствии с информацией Центров по контролю и профилактике заболеваний минимум 2 млн человек инфицированы бактериями, устойчивыми к антибиотикам, и, по меньшей мере, 7.23 млн человек ежегодно умирают от заболеваний, вызванных устойчивыми к антибиотикам микроорганизмами (Hossion, Sasaki, 2013). Всемирная организация здравоохранения (ВОЗ) обратилась в ООН с просьбой обсудить данную проблему и принять соответствующие рекомендации. Проблема обусловлена не только катастрофическим распространением клинических штаммов бактерий с множественной лекарственной устойчивостью (МЛУ), но и распространением МЛУ-бактерий в продуктах питания, среди сельскохозяйственных животных и в растениях, а также в почве и водных источниках (Link et al., 2007). В связи с этим становится актуальным исследование почвенных бактерий как основного резервуара и возможного источника генов лекарственной устойчивости (Gibson et al., 2015).
Аминогликозиды представляют собой большую группу биологически активных вторичных метаболитов (Davies, Wright, 1997). С момента открытия данной группы антибактериальных препаратов их широко применяют в качестве терапевтического средства при лечении различных тяжелых инфекционных заболеваний, вызванных грамотрицательными микроорганизмами (Hermann, 2007). Однако спектр применения аминогликозидных антибиотиков сужается из-за появления бактерий, обладающих устойчивостью к ним (Wright et al., 1998). Тем не менее, на сегодня аминогликозиды являются одними из широко применяемых в клинической практике антибиотиков благодаря их высокой эффективности и низкой себестоимости (Forge, Schacht, 2000; Block, Blanchard, 2019).
Существует несколько основных механизмов устойчивости к аминогликозидам: модификация мишени действия аминогликозидов – рибосом; уменьшение проницаемости мембраны и активный транспорт из клетки; ферментативная модификация антибиотика; образование биопленок. Однако среди всех известных механизмов устойчивости к аминогликозидам модификация ферментами является наиболее распространенным (Wright, Thompson, 1999).
Ферменты, модифицирующие аминогликозиды, катализируют реакции модификации различных -OH и -NH2 групп 2-диоксистрептаминового ядра или остатков сахаров. К ним относятся нуклеотидилтрансферазы (аденилилтрансферазы), фосфотрансферазы и ацетилтрансферазы (Wright, 2011). Ацетилтрансферазы модифицируют аминогруппу молекулы аминогликозида, фосфотрансферазы и аденилилтрансферазы действуют на гидроксильную группу. Действие аминогликозидмодифицирующих ферментов (соответственно ацетилирование, фосфорилирование и аденилирование) приводит к такому изменению структуры молекулы антибиотика, которое не позволяет ему связываться с бактериальной рибосомой, в результате чего синтез белка не ингибируется, и бактериальная клетка сохраняет жизнеспособность (Zarate et al., 2018).
Распространенным механизмом устойчивости к аминогликозидам у штаммов бактерий является ферментативная модификация молекулы антибиотика фосфотрансферазами (Frase et al., 2012). Именно поэтому изучение структуры и функций аминогликозидфосфотрансфераз является важной и актуальной задачей, позволяющей развивать подходы, применяемые для преодоления резистентности к аминогликозидным антибиотикам.
АМИНОГЛИКОЗИДНЫЕ АНТИБИОТИКИ
Аминогликозиды (аминоциклитолы) представляют собой большую группу водорастворимых антибиотиков, обладающих широким антимикробным спектром активности (Wright, Thompson, 1999; Chandrika, Garneau-Tsodikova, 2018). Молекула аминогликозидного антибиотика содержит два или более аминосахара, которые связаны гликозидными связями с аминоциклитольным кольцом (Busscher et al., 2005). Также к аминогликозидам относят спектиномицин, в молекуле которого между аминоциклитольным кольцом и аминосахаром нет связей (Bryskier, 2005; Veyssier, Bryskier, 2005).
Аминогликозидные антибиотики обладают бактерицидным действием, механизм которого заключается в связывании аминогликозида с декодирующим сайтом рибосомы и нарушением синтеза белков (Sutcliffe, 2005; Hermann, 2005, 2007).
К аминогликозидным антибиотикам относятся: стрептомицин, канамицин, неомицин, гентамицин, тобрамицин, амикацин и др. Выделяют три поколения (табл. 1) аминогликозидных антибиотиков (Решедько, 1999; Hotta, Kondo, 2018).
Таблица 1.
Поколения аминогликозидных антибиотиков
| I поколение | II поколение | III поколение |
|---|---|---|
| Стрептомицин | Гентамицин | Амикацин |
| Канамицин | Тобрамицин | Изепамицин |
| Неомицин | Сизомицин | |
| Мономицин | Нетилмицин | |
| Паромомицин |
На данный момент новейшим аминогликозидным антибиотиком является плазомицин – полусинтетическое производное сизомицина. В настоящее время препарат на основе плазомицина находится на стадии клинических испытаний (Serio et al., 2017; Shaeer et al., 2019).
Антибактериальное действие аминогликозидов опосредовано их химической структурой. Поскольку аминогликозидные антибиотики содержат значительное количество положительно заряженных группировок, они обладают высоким сродством к отрицательно заряженным молекулам таким, как нуклеиновые кислоты (Jana, Deb, 2006).
Рибосомы – одна из основных мишеней действия антибиотиков в бактериальной клетке. Большая часть используемых в клинической практике антибиотиков ингибирует стадию элонгации в процессе синтеза белка: аминогликозиды, хлорамфеникол, линкозамиды, макролиды, оксазолидиноны, стрептограмины и тетрациклины. Несмотря на большой размер рибосом, антибиотики взаимодействуют только с несколькими сайтами. Антибиотики, взаимодействующие с 30S субъединицей, связываются либо с P-сайтом, либо с A-сайтом (Ogle et al., 2003; Vicens, Westhof, 2003; Ogle, Ramakrishnan, 2005; Zaher, Green, 2009; Wilson, 2014).
Основная мишень аминогликозидных антибиотиков – малая субъединица бактериальной рибосомы (30S), в состав которой входит 21 белок и 16S рРНК. Аминогликозиды связываются с 16S рРНК, а точнее с А-сайтом, что приводит к нарушению трансляции при синтезе белка (Fourmy et al., 1996; Carter et al., 2000; Ramirez, Tolmasky, 2010). Модель молекулярного взаимодействия аминогликозидных антибиотиков с А-сайтом была подробно изучена методом рентгеноструктурного анализа (Magnet, Blanchard, 2005).
ПРОДУЦЕНТЫ АМИНОГЛИКОЗИДОВ – ПОЧВЕННЫЕ БАКТЕРИИ РОДА Streptomyces
Наибольшее число антибиотиков (не менее 70%), широко применяемых на практике, относится к веществам, образуемым почвенными актинобактериями (порядок Actinomycetales). Продуцентами наиболее значимых аминогликозидных антибиотиков являются почвенные бактерии, относящиеся к роду Streptomyces (Block, Blanchard, 2019). Продуценты различных аминогликозидов представлены в табл. 2 (Wright et al., 1998).
Таблица 2.
Продуценты аминогликозидных антибиотиков
| Название антибиотика | Штамм-продуцент | Источник |
|---|---|---|
| Канамицин | Streptomyces kanamyceticus ATCC 12853 | Nepal et al., 2009 |
| Стрептомицин | Streptomyces griseus С-96 | Takahashi, Nakashima, 2018 |
| Гентамицин | Micromonospora purpurea ATCC 31164 | Li et al., 2013 |
| Спектиномицин | Streptomyces spectabilis ATCC 27741 | Thapa et al., 2008 |
| Бутирозин | Bacillus circulans ATCC 21557 | Ota et al., 2000 |
| Тобрамицин | Streptomyces tenebrarius, Streptomyces cremeus subsp. nebramycini |
Motkova et al., 1984 |
| Неомицин | Streptomyces fradiae NCIM 2418 | Vastrad, Neelagund, 2014 |
| Амикацин | Полусинтетическое производное канамицина | |
| Изепамицин | Полусинтетическое производное гентамицина В |
Актинобактерии представляют собой одну из крупнейших бактериальных фил и являются широко распространенными в водных и наземных экосистемах. Большинство представителей этой группы бактерий – сапрофиты, обитающие в почве микроорганизмы, но они встречаются и в пресной, и в соленой воде, а также в воздухе. Они обычно присутствуют в почве при плотностях 106–109 клеток бактерий на грамм почвы, причем стрептомицеты составляют более 95% всех штаммов актиномицетов, выделенных из почвы (Barka et al., 2015). Некоторые актинобактерии являются возбудителями заболеваний человека и животных: Actinomyces israelii — возбудитель актиномикоза; Actinomyces meyeri, Actinomyces neuii, Actinomyces turicensis — возбудители заболеваний, локализующихся в различных частях тела, ротовой полости, кожных покровов, слизистых (Kononen, Wade, 2015).
Streptomyces – самый большой род актиномицетов. К нему относятся грамположительные аэробные бактерии, образующие сеть разветвленных нитей, субстратный и воздушный мицелий. На настоящий момент насчитывается около 843 видов и 38 подвидов рода Streptomyces (LPSN, http://www.bacterio.net/streptomyces.html). Клеточная стенка стрептомицетов содержит аланин, глутаминовую кислоту, глицин и LL-2,6-диаминопимелиновые кислоты (LL-DAP). Их ДНК отличается высоким содержанием гуанина и цитозина, причем в наиболее широко изученных видах, например Streptomyces coelicor, содержание G + C составляет до 72.1% (Bentley et al., 2002). Основные среды их обитания – почва и морская вода (Siti et al., 2017). Род Streptomyces является самым крупным родом, способным синтезировать антибиотики, и используется с 1940–1950-х гг. в промышленном производстве антибиотиков (Newman, Cragg, 2007).
Хотя стрептомицеты рассматриваются преимущественно как свободно живущие наземные почвообразующие бактерии, некоторые виды являются симбионтами с грибами, насекомыми, растениями и животными (Seipke et al., 2012), некоторые штаммы обитают в морских почвах (Fiedler et al., 2005). Несколько видов стрептомицетов являются растительными патогенами и вызывают болезни, поражая корни и клубни (Zhang, Loria, 2017). Наиболее экономически важное заболевание, вызванное представителями рода Streptomyces, – картофельная парша, характеризующаяся поверхностными поражениями клубней картофеля (Loria et al., 2006). Еще один патогенный вид, S. ipomoeae, вызывает почвенную гниль на сладком картофеле (Tomihama et al., 2016). К другим патогенным видам стрептомицетов относятся: S. europaeiscabiei, S. stelliscabiei, S. luridiscabiei, S. puniciscabiei, S. niveiscabiei, S. reticuliscabiei и S. caviscabies (Goyer et al., 1996; Bouchek-Mechiche et al., 2000; Park et al., 2003). Многочисленные исследования представителей данного рода показали, что их можно встретить почти во всех экосистемах нашей планеты, при этом они успешно конкурируют с представителя других филогенетических групп за счет их необычного метаболизма и вторичных метаболитов, доминируя в микробной популяции (Berdy, 2012).
Многие представители рода Streptomyces хорошо изучены, поскольку являются коммерчески значимыми продуцентами вторичных метаболитов и гидролитических ферментов. Одним из таких штаммов является Streptomyces lividans, классический объект молекулярно-генетических исследований, также используемый для получения гомологичных и гетерологичных гидролитических ферментов для промышленного применения. В связи с коммерческой ценностью штамма, его метаболитические пути и механизмы секреции некоторых веществ изучены очень подробно (Gullón, Mellado, 2018).
Развитие технологий, связанных с секвенированием геномов и анализом библиотек данных, а также вычислительных ресурсов компьютерной техники, ускоряет и упрощает идентификацию многочисленных кластеров генов биосинтеза (BGCs, biosynthetic gene clusters) природных лекарственных соединений в геномах стрептомицетов. Однако большинство этих BGC-кластеров являются молчащими или экспрессируются в исходных штаммах на невысоком уровне, что делает актуальными их исследования при помощи редактирования генома. Разрабатываются многочисленные стратегии, в том числе, с применением CRISPR/CRISPR-Cas-технологий. Данный метод редактирования геномов обладает более высокой точностью по сравнению с другими методиками и более высокой эффективностью для редактирования генома в различных модельных организмах, включая стрептомицетов (Tao et al., 2014; Cobb et al., 2015).
Кроме того, штаммы стрептомицетов являются перспективными источниками новых антибиотиков против штаммов с множественной/широкой лекарственной устойчивостью, в том числе таких, как метициллин-устойчивый Staphylococcus aureus. Ранее стрептомицеты уже были источниками таких антибиотиков, в частности ванкомицина. За прошедшие годы количество новых антибиотических соединений значительно уменьшилось, в результате чего меньшее количество лекарственных соединений дошло до этапа клинических испытаний. Хотя за время исследований, начиная с обнаружения стрептомицина в 1944 г., было выявлено более 10 400 биологически активных веществ у Streptomyces. При этом поиск новых антибиотиков замедляется, а темп образования устойчивости к ним возрастает, что делает актуальным поиск новых антибиотиков и их продуцентов из необычных мест обитания (Berdy, 2012). Для этой цели в первую очередь рассматриваются штаммы из малоизученных регионов: мангровые заросли, пустыни, морские и пресноводные резервуары, а также эндофитные формы. Микроорганизмы, населяющие данные регионы, сталкиваются со сложными условиями окружающей среды, высокой соленостью, повышенными температурами, низкой влажностью. Данные штаммы стрептомицетов обладают уникальным метаболизмом и образуют уникальные биологически активные соединения (Kemung et al., 2018).
КЛАССИФИКАЦИЯ АМИНОГЛИКОЗИДФОСФОТРАНСФЕРАЗ
Аминогликозидфосфотрансферазы – это ферменты, модифицирующие аминогликозидные антибиотики путем фосфорилирования их гидро-ксильных групп в присутствии АТФ в качестве кофактора (Smith, Baker, 2002). Ранее считалось, что донором фосфатной группировки является только АТФ, но недавно было установлено, что несколько фосфотрансфераз используют ГТФ вместо АТФ (Shakya, Wright, 2010; Shi, Berghuis, 2012; Smith et al., 2012). Идентифицировано более 30 фосфотрансфераз из клинических штаммов и штаммов-продуцентов аминогликозидных антибиотиков, идентичность по аминокислотным последовательностям которых варьирует от 20 до 40%.
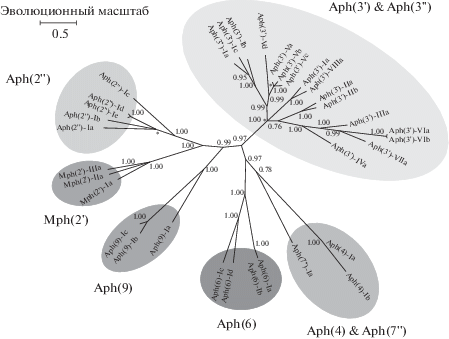
Установлено, что ферменты семейства аминогликозидфосфотрансфераз схожи по структуре и функциям с серин-треониновыми протеинкиназами эукариот и по современной классификации отнесены к киназам, модулирующим устойчивость бактерий к антибиотикам (Kim, Mobashery, 2005; Morar, Wright, 2010). Киназы, модифицирующие антибиотики, включают аминогликозидфосфотрансферазы (Аph) и макролидфосфотрансферазы (Мph). На рис. 1 показан филогенетический анализ киназ, модифицирующих аминогликозидные антибиотики (Shakya et al., 2011).
В зависимости от положения гидроксильной группы антибиотика, модифицируемой ферментом, различают 7 подсемейств аминогликозидфосфотрансфераз: Aph(2"), Aph(3'), Aph(3"), Aph(4), Aph(6), Aph(9) и Aph(7") (Vakulenko, Mobashery, 2003; Ramirez, Tolmasky, 2010; Shakya et al., 2011). Информация о данных подсемействах представлена в виде табл. 3 (Vakulenko, Mobashery, 2003; Ramirez, Tolmasky, 2010; Shi et al., 2013).
Таблица 3.
Распространение аминогликозидфосфотрансфераз различного типа у штаммов-продуцентов аминогликозидных антибиотиков
| Aph | Название гена | Расположение в геноме | Выявлен у штамма |
Субстрат |
|---|---|---|---|---|
| Aph(3')-Va | aph(3')-Va, aphA-5a | Хромосома | Streptomyces fradiae | Неомицин, паромомицин |
| Aph(3')-Vb | aph(3')-Vb, aphA-5b, rph | Хромосома | Streptomyces ribosidificus | |
| Aph(3')-Vc | aph(3')-Vc, aphA-5c | Хромосома | Micromonospora chalcea | |
| Aph(3")-Ia | aph(3")-Ia, aphE, aphD2 | Хромосома | Streptomyces griseus | Стрептомицин |
| Aph(4)-Ib | aph(4)-Ib, hyg | Хромосома | Streptomyces hygroscopicus | Гигромицин |
| Aph(6)-Ia | aph(6)-Ia, aphD, strA | Хромосома | Streptomyces griseus | Стрептомицин |
| Aph(6)-Ib | aph(6)-Ib, sph | Хромосома | Streptomyces griseus | |
| Aph(7")-Ia | aph(7")-Ia, aph7" | Хромосома | Streptomyces hygroscopicus | Гигромицин |
| Aph(9)-Ib | aph(9)-Ib, spcN | Хромосома | Streptomyces flavopersicus | Спектиномицин |
Подсемейство аминогликозид-3'-фосфотрансфераз является наиболее распространенным и изученным, в состав данного подсемейства входят восемь изоферментов. У актинобактерий были обнаружены ферменты Aph(3')-V: у штамма-продуцента неомицина Streptomyces fradiae (Va), у Streptomyces ribosidificus (Vb) и у Micromonospora chalcea (Vc) (Thompson, Gray, 1983; Hoshiko et al., 1988; Salauze et al., 1991; Wright, Thompson, 1999).
У штамма-продуцента стрептомицина Streptomyces griseus были обнаружены две стрептомицинфосфотрансферазы Aph(3")-Ia и Aph(6)-Ia (Heinzel et al., 1988; Collins et al., 2007).
В последние годы было проведено секвенирование большого количества геномов актинобактерий. При аннотации геномов у актинобактерий рода Streptomyces было выявлено от 4 до 14 генов аминогликозидфосфотрансфераз. Они определяют исходный (природный) уровень устойчивости к аминогликозидным антибиотикам. Функции генов, аннотированных как aph в секвенированных геномах, в настоящее время недостаточно изучены и требуют дальнейшей проверки, являются ли эти гипотетические ферменты истинными aph-генами устойчивости (Anderson et al., 2002).
При изучении спектра устойчивости к антибиотикам у 110 штаммов рода Streptomyces обнаружено, что штамм S. rimosus ATCC 10970 (продуцент окситетрациклина) – единственный среди изученных – обладает устойчивостью к большинству аминогликозидных антибиотиков в концентрации 10–20 мкг/мл (Даниленко и др., 1977).
АМИНОГЛИКОЗИДФОСФОТРАНСФЕРАЗЫ Streptomyces rimosus
Аминогликозидфосфотрансфераза Aph(3')-VIII
При многоступенчатой селекции клеток S. rimosus ATCC 10970, устойчивых к возрастающим дозам канамицина (от 10 до 4 × 104 мкг/мл), удалось получить штамм, обладающий активностью аминогликозид-3'-фосфотрансферазы и содержащий перестройки в хромосомной ДНК. Был обнаружен детерминант антибиотикоустойчивости штамма S. rimosus ATCC 10970, обозначенный как Kmr, амплифицирующийся в составе фрагмента ДНК размером 10.3 × 106 п.н. и характеризующийся генетической нестабильностью, частота перехода Kmr ↔ Kms составляет 1 × 10–3. Этот тип нестабильности широко распространен среди актиномицетов и характерен для большинства признаков, не связанных с первичным метаболизмом (Потехин, Даниленко, 1985).
Детерминант Kmr был клонирован в штамм Streptomyces lividans 66 в составе векторной плазмиды SLP1.2, подробно изучалось явление амплификации данного детерминанта в составе сконструированных гибридных плазмид pSU1–pSU13. Было показано, что субклонирование данного фрагмента хромосомной ДНК в составе описанных плазмид обеспечивает устойчивость клеток Streptomyces lividans к канамицину, неомицину и паромомицину. Грубые бесклеточные экстракты таких трансформантов имеют активность аминогликозидфосфотрансферазы, субстратная специфичность которой соответствует спектру антибиотикоустойчивости. Нуклеотидная последовательность фрагмента ДНК, содержащая детерминанту Kmr, определена, найдена открытая рамка считывания для гена аминогликозид-3'-фосфотрансферазы. На основании нуклеотидной последовательности и субстратной специфичности кодируемого фермента, ген aph из S. rimosus ATCC 10970 отнесен к новому классу генов и обозначен как aph(3')-VIII, а его продукт обозначен как Aph(3')-VIII (Потехин, Даниленко, 1985; Стародубцева и др., 1985; Даниленко и др., 1997).
В ходе дальнейших исследований, ген aph(3')-VIII был клонирован в одноклеточную зеленую водоросль Chlamydomonas reinhardtii в составе плазмидного вектора pSU973. Было продемонстрировано, что ядерные трансформанты этой водоросли, несущие вектор pSU973:aph(3')-VIII обладают высокой устойчивостью к паромомицину (300–500 мкг/мл). Анализ суммарных белков трансформантов с использованием ПААГ-электрофореза в неденатурирующих условиях показал, что аминогликозидфосфотрансферазную активность проявляет белок с молекулярной массой около 30 кДа (Sizova et al., 1996, 2001).
Нуклеотидная последовательность гена aph(3')-VIII была уточнена и задепонирована в GenBank под номером AAG11411. Данная последовательность имеет длину 804 п.н. и кодирует белок AphVIII протяженностью 267 аминокислот с молекулярной массой 29.1 кДа и изоэлектрической точкой 4.59. Сравнение аминокислотных последовательностей Aph(3')-VIII и аминогликозидфосфотрансфераз из штаммов-продуцентов аминогликозидных антибиотиков позволило выявить значительные отличия и отнести данный фермент в отдельную группу (Сизова и др., 2002).
Для выяснения роли гена aph(3')-VIII штамма S. rimosus ATCC 10970 было также проведено его клонирование в E. coli BL21(DE3) в составе плазмидного вектора pSI10. Ранее у S. lividans были обнаружены хромосомные мутации, влияющие на уровень устойчивости клеток к аминогликозидам, возможно, локализованные в гене, кодирующем активатор белка Aph(3')-VIII (Потехин, Даниленко, 1985). При этом роль активаторов белковой активности у Streptomyces могут играть серин-треониновые протеинкиназы эукариотического типа. Для проверки гипотезы о том, что AphVIII активируется путем фосфорилирования протеинкиназами, изучали гетерологичную экспрессию гена aph(3')-VIII в трансформированных прокариотических клетках, не содержащих протеинкиназы эукариотического типа (E. coli) и эукариотических клетках (Chlamydomonas reinhardtii). При этом было продемонстрировано, что трансформанты Chlamydomonas обладают более высоким уровнем устойчивости к паромомицину при более низком содержании белка Aph(3')-VIII. Данное явление может быть связано с активацией белка посредством его фосфорилирования серин-треониновыми протеинкиназами. Сравнение аминокислотных остатков Aph(3')-VIII и серин-треониновых протеинкиназ актиномицетов выявило локальное подобие протяженностью 38 аминокислот в консервативной области, участвующей в связывании АТФ. При биоинформатическом поиске потенциальных областей фосфорилирования было выявлено четыре области для серин-треониновых протеинкиназ, в том числе, Ca2+-зависимых (Сизова и др., 2002).
Аминогликозидфосфотрансферазную активность определяли радиохимическим методом в грубом бесклеточном экстракте штамма S. lividans ТК64, содержащем плазмиду pSU951:aph(3')-VIII. В качестве отрицательного контроля использовали бесплазмидный штамм S. lividans ТК64, а в качестве положительного – экстракт клеток E. coli, трансформированных плазмидой pSV2neо и продуцирующих стрептомицин-трансферазу. После инкубирования аминогликозидных антибиотиков с 32Р-γАТФ с бесклеточным экстрактом радиомеченные фосфорилированные антибиотики выделяли методом тонкослойной хроматографии с полиэтиленимин-целлюлозой и измеряли уровень радиоактивности полученных образцов. Было показано, что энзим Aph(3')-VIII обладает фосфотрансферазной активностью в отношении аминогликозидных антибиотиков канамицина, неомицина и паромомицина (Даниленко и др., 1997).
Также изучали аминогликозидфосфотрансферазную активность Aph(3')-VIII в грубых клеточных экстрактах Chlamydomonas reinhardtii методом авторадиографии, в результате чего была выявлена активность фермента в отношении паромомицина и неомицина (Sizova et al., 1996).
Определение субстратной специфичности Aph(3')-VIII проводили, выделяя водорастворимую фракцию белков E. coli, трансформированных вектором pET16b:aph(3')-VIII. Посредством метода гель-электрофореза в денатурирующих условиях выявили фракцию белка с молекулярной массой около 30 кДа. Трансформанты были устойчивы к паромомицину в концентрации до 60 мкг/мл (Сизова и др., 2002).
В дальнейших работах выделение и очистку Aph(3')-VIII, содержащую His10, из экстрактов E. coli проводили с использованием колонок с Ni-NTA-агарозой в нативных условиях, после чего проводили SDS-PAGE. Очистку фракций, содержащих 31.5 кДа-белок, проводили хроматографически (Елизаров и др., 2005). Анализ канамицинкиназной активности фермента проводили в реакционной смеси, содержавшей 0.3 мкг Aph(3')-VIII, 1.2 мг/мл канамицина, 7.5 мМ 32Р-γАТФ, 4 мМ NaHCO3, 20 мМ Трис-HCl pH 7.8, 10 мМ MgCl2, 60 мМ KCl и 3мМ ДТТ. При этом с использованием метода авторадиографии была продемонстрирована высокая активность фермента в отношении аминогликозидного антибиотика канамицина (Елизаров и др., 2012).
Актиномицеты представляют собой перспективную модель для изучения взаимодействия между протеинкиназами эукариотического типа и аминогликозидфосфотрансферазами в регуляции клеточных процессов и лекарственной устойчивости. При помощи методов иммунопреципитации с антителами к Aph(3')-VIII и мечения in vitro было выявлено, что эндогенные протеинкиназы в экстрактах S. rimosus активно фосфорилируют Aph(3')-VIII по двум остаткам серина. При этом количество фосфата, включенного в Aph(3')-VIII, в 1.84 раза выше в присутствии Ca2+. Дальнейший анализ показал, что фосфорилирование Aph(3')-VIII катализируется серин-треониновыми протеинкиназами (СТПК) с молекулярной массой 55 и 74 кДа. Причем активность 55 кДа-киназы зависит от кальция и кальмодулина. Установлено, что канамицинфосфотрансферазная активность фосфорилированного белка Aph(3')-VIII в 3.72 раза выше, по сравнению с немодифицированным энзимом. Исследуемые протеинкиназы участвуют в регуляции устойчивости к канамицину в клетках S. rimosus, которая может модулироваться через изменение активности специфических лиганд-зависимых СТПК (Елизаров и др., 2005).
Методами биоинформатического анализа и молекулярного моделирования было выявлено 4 потенциальных сайта фосфорилирования Aph(3')-VIII: S95, S146, S160, S215. Для дальнейшей идентификации сайтов фосфорилирования получали мутантные варианты гена aph(3')-VIII методом точковых мутаций, при этом заменяли в выявленных позициях Ser → Ala. Сравнительный анализ канамицинкиназной активности нефосфорилированной и фосфорилированной форм исходного и мутантного вариантов белка Aph(3')-VIII показал, что Ca2+-зависимое фосфорилирование Ser146 в Aph(3')-VIII приводит к 6–7-кратному увеличению канамицинкиназной активности Aph(3')-VIII. Таким образом, Ser146, расположенный в активационной петле фермента, критичен для его активности. Также показано, что Aph(3')-VIII является ферментом, активируемым протеинкиназами (Елизаров и др., 2012).
Полноатомная модель структуры Aph(3')-VIII была создана с использованием сервера для моделирования Swiss-Model на основе доступных в базе PDB структур ферментов Aph(3')-II (идентификатор 1ND4) и Aph(3')-III (идентификатор 1L8T). Молекулярное моделирование позволило выявить (рис. 2) сайт фосфорилирования Ser146 в активационной петле фермента (Елизаров и др., 2012).
Рис. 2.
Модель третичной структуры Aph(3')-VIII. Положения S95, S146, S160, S215 – вероятные сайты фосфорилирования Aph(3')-VIII (Елизаров и др., 2012).

Анализ молекулярной динамики комплекса нефосфорилированного AphVIII с канамицином, АТФ и двумя ионами Mg2+ выявил изменения структуры фермента за счет ослабления контакта между С- и N-концевыми доменами. Подвижность доменов обусловливает высвобождение АТФ от субстрата, связанного с С-концевой долей, и взаимодействие фосфата с N-концевой долей, что приводит к каталитически неактивной форме Aph(3')-VIII (Елизаров и др., 2012).
Методом рентгеноструктурного анализа получена 3D-структура Aph(3')-VIII с разрешением 2.15 Å, идентификационный номер в PDB – 4H05 (рис. 3). В результате анализа полученной структуры и сравнения с уже имеющимися структурами аминогликозид-3'-фосфотрансфераз была выявлена область Ser146 в активационной петле фермента. Показано изменение конформации в данной области, происходящее при взаимодействии с субстратом (Boyko et al., 2016).
Рис. 3.
Аph(3')-VIII и ее сравнение со структурами других аминогликозид-(3')-фосфотрансфераз (Boyko et al., 2016). (а) – мономер AphVIII; (б) – сравнение AphVIII и Aph-II в комплексе с канамицином А (ID PDB: 1ND4); (в) – сравнение AphVIII и Aph-III в комплексе с АДФ и канамицином A (ID PDB: 1L8T); (г) – увеличенный вид области связывания с субстратом, регион гибкой петли NPL в области 21–27 аминокислотных остатков (нумерация по AphVIII) обозначен стрелкой, также указана петля между β4- и β5-складчатыми слоями. Aph-I в комплексе с АДФ (ID PDB: 4EJ7).

Таким образом, в штамме S. rimosus АТСС 10 970 была идентифицирована и охарактеризована аминогликозид-3'-фосфотрансфераза нового типа – AphVIII (Потехин, Даниленко, 1985; Сизова и др., 2002), установлена 3D-структура фермента, код PDB 4H05 (Boyko et al., 2016).
Модуляция активности Aph(3')-VIII серин-треониновыми протеинкиназами используется на практике с целью создания высокоэффективных тест-систем для отбора ингибиторов СТПК с использованием конструкта белка Aph(3')-VIII и целевых СТПК человека и бактерий. Принцип работы тест-систем подобного типа построен на том, что фосфорилирование протеинкиназами фермента Aph(3')-VIII, инактивирующего аминогликозидные антибиотики, увеличивает устойчивость клеток бактерий к канамицину. Ингибиторы протеинкиназ, напротив, делают клетки бактерий более чувствительными к канамицину. Эти свойства тест-системы позволяют проводить биомишень-направленный поиск ингибиторов протеинкиназ как потенциальных лекарственных препаратов нового поколения. Первой тест-системой подобного типа была система, разработанная на основе штамма Streptomyces lividans ТК24 (66) Aph+ (Danilenko et al., 2008). Одной из таких систем является тест-система E. coli/aphVIII/pk25, используемая для прескрининга ингибиторов Pk25 штамма Streptomyces lividans и ее структурных гомологов. В частности, данная тест-система может быть использована для прескрининга ингибиторов СТПК ряда патогенных микроорганизмов, таких как PknA и PknJ штаммов M. tuberculosis, StkP S. pneumonia и некоторых СТПК человека, в том числе PKA, CaMKI, Pac2 (Беккер и др., 2010).
Другой тест-системой подобного типа является cистема E. coli/aphVIII/pim-1. Принцип и особенности работы данной системы представлены на рис. 4. Сайт-направленный мутагенез в области Ser146 позволил оптимизировать этот сайт для наиболее эффективного фосфорилирования протеинкиназой Pim-1. СТПК семейства Pim положительно регулируют клеточный цикл и играют важную роль в патогенезе опухолей системы крови, поддерживая пролиферацию клеток. Ингибиторы СТПК семейства Pim являются потенциальными лекарственными препаратами для терапии лейкозов и лимфом (Zhukova et al., 2011).
Рис. 4.
Принцип работы тест-системы: фосфорилирование Aph(3')-VIII по Ser146 посредством СТПК повышает устойчивость E. coli к канамицину; добавление ингибитора препятствует фосфорилированию AphVIII, снижает устойчивость к канамицину (Zhukova et al., 2011).

Еще одной системой подобного типа является недавно разработанная тест-система E. coli/aphVIII/gsk3β. Данная система включает в себя ген каталитического домена протеинкиназы GSK3β и ген Aph(3')-VIII субстрата для фосфорилирования. Для оптимизации работы тест-системы получены две модификации Aph(3')-VIII в области сайта фосфорилирования Ser146. Целенаправленный отбор ингибиторов протеинкиназы GSK3β является перспективным направлением в лекарственной терапии диабета 2-го типа, болезни Альцгеймера и хронических воспалительных заболеваний (Алексеева и др., 2018).
Аминогликозидфосфотрансфераза Aph(3'')-Id
Анализ генома S. rimosus ATCC 10970 (Pethick et al., 2013) позволил идентифицировать 14 генов, аннотированных как aph, включая aphVIII. Эти 14 aph-генов были обозначены как aphSR1–14. Исходя из филогенетического сходства, только три из 14 генов, aphSR5 (aphVIII), aphSR3 и aphSR2, были отнесены к известным подсемействам Aph(3'), Aph(3'') и Aph(7'') соответственно.
Впервые для Streptomyces – не продуцентов аминогликозидных антибиотиков у штамма S. rimosus ATCC 10970 идентифицирован ген aph(3'')-Id, кодирующий стрептомицин-фосфотрансферазу, биохимически охарактеризован фермент Aph(3")-Id (Alekseeva et al., 2019). Гены aph, принадлежащие к подсемейству Aph(3''), являются стрептомицинфосфотрансферазами. Известно, что устойчивость к стрептомицину, опосредованная аминогликозидфосфотрансферазами, является результатом действия двух классов ферментов – Aph(3') и Aph(6) (Wright, Thompson, 1999). Филогенетический анализ фосфотрансфераз из клинических штаммов бактерий и продуцентов аминогликозидных антибиотиков (Shakya et al., 2011) и 14 Aph S. rimosus ATCC10970 показал, что AphSR3 принадлежит к подсемейству Aph(3''). Ранее были описаны гены aph(3''): aph(3'')-Ia – у продуцента стрептомицина Streptomyces griseus (Heinzel et al., 1988), aph(3'')-Ib – на плазмиде RSF1010 Escherichia coli (Scholz et al., 1989) и aph(3'')-Ic – у Mycobacterium fortuitum (Ramón-García et al., 2006). Многократное выравнивание последовательностей выявило, что AphSR3 имеет все консервативные домены, характерные для подсемейства Aph(3'') (Heinzel et al., 1988; Wright, Thompson, 1999; Ramón-García et al., 2006). Ген aphSR3 был обозначен как aph(3'')-Id. Анализ устойчивости к аминогликозидным антибиотикам показал, что ген aph(3'')-Id обусловливает устойчивость клеток E. coli к стрептомицину. Белок Aph(3'')-Id (AphSR3) впервые выделен в нативных условиях. Оценка фосфотрансферазной активности белка Aph(3'')-Id in vitro с помощью двух методов показала, что белок Aph(3'')-Id является ферментативно активным и способен катализировать фосфорилирование субстрата – аминогликозидного антибиотика стрептомицина. Способность новой идентифицированной стрептомицин-фосфотрансферазы подвергаться аутофосфорилированию in vitro является уникальным свойством для этого класса ферментов. Это свойство хорошо известно для эукариотических серин-треониновых и тирозин протеинкиназ (Hashimoto et al., 2008), а также для бактериальных серин-треониновых протеинкиназ эукариотического типа (Damle, Mohanty, 2014). Автофосфорилирование является важным механизмом регуляции биологической активности протеинкиназ. Однако в литературе отсутствуют данные об автофосфорилировании аминогликозидфосфотрансфераз. Следует отметить, что автофосфорилирование серина/треонина и тирозина является уникальным свойством для данной группы ферментов (Alekseeva et al., 2019).
Аминогликозидфосфотрансфераза AphSR2
Описано исследование влияния серин-треониновых протеинкиназ на повышение устойчивости к аминогликозидным антибиотикам при совместном клонировании в E. coli гена aphSR2 и генов СТПК pkSR1 и pkSR2, локализованных в одном кластере генома Streptomyces rimosus АТСС 10 970. При совместном клонировании данных генов в E. coli было выявлено, что aphSR2 обусловливает устойчивость к неомицину, которая моделируется pkSR1. Показано, что клонирование в E. coli гена pkSR1 придает устойчивость к неомицину и гигромицину. E. coli, содержащая pkSR1, обладает повышенной устойчивостью к неомицину, совместная экспрессия генов aphSR2 и pkSR1 приводит к повышению уровня устойчивости в два раза. Представленные данные являются вторым примером влияния СТПК на модуляцию уровня устойчивости к аминогликозидным антибиотикам у бактерий рода Streptomyces. Показано, что AphSR2 — вторая Aph стрептомицетов, в частности S. rimosus, и что уровень устойчивости повышается СТПК и является аккумулятивным результатом их совместной экспрессии. В практическом плане полученные данные могут быть использованы для изучения распространения и особенностей функций генов, определяющих природную устойчивость к аминогликозидным антибиотикам у актинобактерии рода Streptomyces (Рудакова и др., 2020).
ЗАКЛЮЧЕНИЕ
Результаты многолетних собственных исследований и анализ литературы позволяют, как нам представляется, утверждать, что аминогликозидфосфотрансферазы в настоящее время представляют серьезную угрозу для антимикробной терапии (Shakya, Wright, 2010; Ribeiro da Cunha et al., 2019). Классические представители Aph, распространенные у клинических штаммов бактерий, обусловливают устойчивость к антибиотикам, и по субстратной специфичности их можно разделить на несколько групп: канамицин-фосфотрансферазы, стрептомицин-фосфотрансферазы, гентамицин-фосфотрансферазы, гигромицин-фосфотрансферазы, остальные Aph могут выполнять другие функции (Shakya et al., 2011; Wright, 2011). Для преодоления проблемы устойчивости к антибиотикам комбинируют достижения в области секвенирования генома нового поколения, биоинформатики и аналитической химии.
Аминогликозидфосфотрансферазы хорошо изучены у актинобактерий – продуцентов аминогликозидных антибиотиков, но недостаточно изучены у других актинобактерий рода Streptomyces.
Современные подходы к идентификации генов, ответственных за устойчивость к антибиотикам, включают секвенирование геномной ДНК с последующей аннотацией генов. Актинобактерии рода Streptomyces содержат 4–16 генов aph. Функции генов, аннотированных как aph в секвенированных геномах, в настоящее время недостаточно изучены и требуют дальнейшего исследования для проверки, являются ли эти гипотетические ферменты истинными aph-генами устойчивости (Wright, 2019).
Гены, которые кодируют аминогликозидфосфотрансферазы бактерий, имеют общего эволюционного предка. Классификация исследуемых генов aph бактерий рода Streptomyces на основании филогенетического сходства с известными ранее aph-генами, относящимися к 7 подсемействам аминогликозидфосфотрансфераз, позволяет выявить гены, связанные с устойчивостью к аминогликозидным антибиотикам, и теоретически предсказать спектр устойчивости, кодируемый данным aph-геном.
Так, у модельного штамма S. rimosus АТСС 10 970 с высоким уровнем устойчивости к аминогликозидам на основе филогенетического сходства только три гена aphSR5 (aph(3')-VIII), aphSR3 (aph(3'')-Id) и aphSR2 относятся к известным подсемействам Aph(3'), Aph(3'') и Aph(7''). Аминогликозидфосфотрансферазы Aph(3') модифицируют 3'-OH-группу у широкого спектра аминогликозидов, включая канамицин, неомицин, паромомицин, ливидомицин, рибостамицин, бутирозин, амикацин и изепамицин, Aph(3'') модифицируют 3''‑ОН-группы стрептомицина, а Aph(7'') обеспечивают устойчивость бактерий к гигромицину.
Появление лекарственно-устойчивых бактерий создает необходимость разработки новых лекарственных средств. Получение кристаллических структур исследуемых ферментов позволяет проводить докинг in silico ингибиторов Aph-белков, относящихся к разным подсемействам. Ингибирование белков, обеспечивающих природный уровень устойчивости бактерий к ряду аминогликозидных антибиотиков, могло бы способствовать преодолению множественной лекарственной устойчивости патогенных актинобактерий и расширению спектра применяемых препаратов за счет синергичного эффекта антибиотика с соединением-ингибитором Aph.
Список литературы
Алексеева М.Г., Мавлетова Д.А., Даниленко В.Н. Тест-система Escherichia coli/aphVIII/gsk3β для селективного скрининга ингибиторов серин-треониновой протеинкиназы GSK3β // Генетика. 2018. Т. 54. № 13. С. 14–17.
Беккер О., Алексеева М., Осолодкин Д. и др. Новая тест-система для скрининга ингибиторов серин-треониновых протеинкиназ: Escherichia coli APHVIII/Pk25 // Acta Nat. 2010. Т. 2. № 3. С. 126–139.
Даниленко В.Н., Пузынина Г.Г., Ломовская Н.Д. Множественная устойчивость к антибиотикам у актиномицетов // Генетика. 1977. Т. 13. № 10. С. 1831–1841.
Даниленко В.Н., Акопянц К.Э., Сизова И.А., Мичурина Т.А. Определение нуклеотидной последовательности и характеристика нового аминогликозидфосфотрансферазного гена aphVIII из штамма Streptomyces rimosus // Генетика. 1997. Т. 33. № 11. С. 1478–1486.
Елизаров С., Сергиенко О., Сизова И., Даниленко В. Зависимость активности аминогликозид-3'-фосфотрансферазы типа VIII от серин-треонин-протеинкиназ у Streptomyces rimosus // Мол. биол. 2005. Т. 39. № 2. С. 255–263.
Елизаров С., Алексеева М., Новиков Ф. и др. Идентификация сайтов фосфорилирования аминогликозидфосфотрансферазы VIII Streptomyces rimosus // Биохимия. 2012. Т. 77. № 11. С. 1504–1512.
Потехин Я.А., Даниленко В.Н. Детерминант устойчивости к канамицину Streptomyces rimosus: амплификация в составе хромосомы и обратимая генетическая нестабильность // Мол. биол. 1985. Т. 19. № 3. С. 805–811.
Решедько Г. Значение ферментативной модификации аминогликозидов в развитии резистентности у бактерий // Клин. микробиол. антимикроб. химиотер. 1999. Т. 1. № 1. С. 40–50.
Рудакова Н.Н., Алексеева М.Г., Захаревич Н.В. и др. Аминогликозидфосфотрансфераза AphSR2 Streptomyces rimosus ATCC 10970: зависимость устойчивости к антибиотикам от серин-треониновых протеинкиназ PkSR1 и PkSR2 // Генетика. 2020. Т. 56. № 1. С. 1–7.
Сизова И., Хегеманн П., Фурманн М., Даниленко В. Аминогликозид-3'-фосфотрансфераза из Streptomyces rimosus: сравнение с аминогликозид-3'-фосфотрансферазами из штаммов-продуцентов аминогликозидов и с эукариотическими протеинкиназами // Мол. биол. 2002. Т. 36. № 1. С. 1–10.
Стародубцева Л.И., Таисова А.С., Даниленко В.Н. Изучение явления амплификации детерминанта устойчивости к канамицину Kmr в составе сконструированных гибридных плазмид в штамме Streptomyces lividans // Антибиот. мед. биотехнол. 1985. № 5. С. 565–572.
Alekseeva M.G., Boyko K.M., Nikolaeva A.Y. et al. Identification, functional and structural characterization of novel aminoglycoside phosphotransferase APH(3'')-Id from Streptomyces rimosus subsp. rimosus ATCC 10970 // Arch. Biochem. Biophys. 2019. V. 671. № 4. P. 111–122.
Anderson A.S., Clark D.J., Gibbons P.H., Sigmund J.M. The detection of diverse aminoglycoside phosphotransferases within natural populations of actinomycetes // J. Ind. Microbiol. Biotechnol. 2002. V. 29. № 2. P. 60–69.
Barka E.A., Vatsa P., Sanchez L. et al. Taxonomy, physiology, and natural products of Actinobacteria // Mol. Biol. Rev. 2015. V. 80. № 1. P. 1–43.
Bentley S.D., Chater K.F., Cerdeno-Tarraga A.M. et al. Complete genome sequence of the model actinomycete Streptomyces coelicolor A3 // Nature. 2002. V. 417. P. 141–147.
Berdy J. Thought and facts about antibiotics: where we are now and where we are heading // J. Antibiot. 2012. V. 65. № 8. P. 385–395.
Block M., Blanchard D.L. Aminoglycosides // StatPearls Publishing. 2019. 621 p.
Bouchek-Mechiche K., Gardan L., Normand P., Jouan B. DNA relatedness among strains of Streptomyces pathogenic to potato in France: description of three new species, S. europaeiscabiei sp. nov. and S. stelliscabiei sp. nov. associated with common scab, and S. reticuliscabiei sp. nov. associated with netted scab // Int. J. Syst. Evol. Microbiol. 2000. № 50. P. 91–99.
Boyko K.M., Gorbacheva M.A., Korzhenevskiy D.A. et al. Structural characterization of the novel aminoglycoside phosphotransferase AphVIII from Streptomyces rimosus with enzymatic activity modulated by phosphorylation // Biochem. Biophys. Res. Comm. 2016. V. 477. P. 595–601.
Bryskier A. Antibiotics and antibacterial agents: classifications and structure-activity relationships // Antimicrobial agents: antibacterials and antifungals agents. N.Y.: Wiley, 2005. P. 13–38.
Busscher G., Rutjes F., Delft F. 2-Deoxystreptamine: central scaffold of aminoglycoside antibiotics // Chem. Rev. 2005. № 105. P. 775–791.
Carter A., Clemons W., Brodersen D. et al. Functional insights from the structure of the 30S ribosomal subunit and its interactions with antibiotics // Nature. 2000. № 407. P. 340–348.
Chandrika T.N., Garneau-Tsodikova S. Comprehensive review of chemical strategies for the preparation of new aminoglycosides and their biological activities // Chem. Soc. Rev. 2018. V. 19. № 47 (4). P. 1189–1249.
Cobb R.E., Wang Y., Zhao H. High-efficiency multiplex genome editing of Streptomyces species using an engineered CRISPR/Cas system // ACS Synth. Biol. 2015. V. 4. P. 723−728.
Collins A.C., Ashenafi M., Saunders A.A., Byrnes W.M. Cloning and expression of streptomycin inactivating enzymes APH(6)-Ia and APH(6)-Id // Cell. Mol. Biol. 2007. V. 15. № 53 (3). P. 74–79.
Damle N.P., Mohanty D. Mechanism of autophosphorylation of mycobacterial PknB explored by molecular dynamics simulations // Biochemistry. 2014. V. 53. P. 4715–4726.
Danilenko V.N., Simonov A.Y., Lakatosh S.A. et al. Search for inhibitors of bacterial and human protein kinases among derivatives of diazepines[1,4] annelated with maleimide and indole cycles // J. Med. Chem. 2008. V. 51. № 24. P. 7731–7736.
Davies J., Wright G. Bacterial resistance to aminoglycoside antibiotics // Tr. Microbiol. 1997. V. 5. № 6. P. 234–240.
Fiedler H.P., Bruntner C., Bull A.T. et al. Marine actinomycetes as a source of novel secondary metabolites // Antonie van Leeuwenhoek. 2005. V. 87. P. 37–42.
Forge A., Schacht J. Aminoglycoside antibiotics // Audiol. Neurootol. 2000. V. 5. P. 3–22.
Fourmy D., Recht M., Blanchard S., Puglisi J. Structure of the A site of Escherichia 16S ribosomal RNA complexed with an aminoglycoside antibiotic // Science. 1996. V. 27. P. 1367–1374.
Frase H., Toth M., Vakulenko S. Revisiting the nucleotide and aminoglycoside substrate specificity of the bifunctional aminoglycoside acetyltransferase(6')-Ie/aminoglycoside phosphotransferase(2'')-Ia enzyme // J. Biol. Chem. 2012. V. 287. P. 43262–43269.
Gibson M.K., Crofts T.S., Dantas G. Antibiotics and the developing infant gut microbiota and resistome // Curr. Opin. Microbiol. 2015. V. 27. P. 51–6.
Goyer C., Faucher E., Beaulieu C. Streptomyces caviscabies sp. nov., from deep-pitted lesions in potatoes in Quebec, Canada // Int. J. Syst. Evol. Microbiol. 1996. V. 46. P. 635–639.
Gullón S., Mellado R.P. The cellular mechanisms that ensure an efficient secretion in Streptomyces // Antibiotics. 2018. V. 7. № 33. P. 1–13.
Hashimoto Y.K., Satoh T., Okamoto M., Takemori H.J. Importance of autophosphorylation at Ser186 in the A-loop of salt inducible kinase 1 for its sustained kinase activity // Cell. Biochem. 2008. V. 104. P. 1724–1739.
Heinzel P., Werbitzky O., Distler J., Piepersberg W. Second streptomycin resistance gene from Streptomyces griseus codes for streptomycin-3"-phosphotransferase. Relationships between antibiotic and protein kinases // Arch. Microbiol. 1988. V. 150. № 2. P. 184–192.
Hermann T. Drugs targeting the ribosome // Curr. Opin. Struct. Biol. 2005. V. 15. P. 355–366.
Hermann T. Aminoglycoside antibiotics: old drugs and new therapeutic approaches // Cell. Mol. Life Sci. 2007. V. 64. P. 1841–1852.
Hoshiko S., Nojiri C., Matsunaga K. et al. Nucleotide sequence of the ribostamycin phosphotransferase gene and of its control region in Streptomyces ribosidificus // Gene. 1988. V. 68. P. 285–296.
Hossion A.M., Sasaki K. Novel quercetin glycosides as potent anti-MRSA and anti-VRE agents // Rec. Pat. Antiinfect. Drug Discov. 2013. V. 8. № 3. P. 198–205.
Hotta K., Kondo S. Kanamycin and its derivative, arbekacin: significance and impact // J. Antibiot. 2018. V. 71. P. 417–424.
Jana S., Deb J. Molecular understanding of aminoglycoside action and resistance // Microbiol. Biotechnol. 2006. V. 70. P. 140–150.
Kemung H., Tan L., Khan T. et al. Streptomyces as a prominent resource of future anti-MRSA drugs // Front. Microbiol. 2018. V. 9. P. 2221–2247.
Kim C., Mobashery S. Phosphoryl transfer by aminoglycoside 3'-phosphotransferases and manifestation of antibiotic resistance // Bioorg. Chem. 2005. V. 33. P. 149–158.
Kononen E., Wade W.G. Actinomyces and related organisms in human infections // Clin. Microbiol. Rev. 2015. V. 28. № 2. P. 419–442.
Krause K.M., Serio A.W., Kane T. R., Connolly L.E. Aminoglycosides: an overview // Cold Spring Harb Perspect Med. 2016. V. 6. № 6. P. 1–19.
Li D., Li H., Ni X. et al. Construction of a gentamicin C1a-overproducing strain of Micromonospora purpurea by inactivation of the gacD gene // Microbiol. Res. 2013. V. 168. № 5. P. 263–267.
Link N.A., Chen P., Lu W.J. et al. Collective form of cell death requires homeodomain interacting protein kinase // J. Cell Biol. 2007. V. 178. № 4. P. 567–574.
Loria R., Kers J., Joshi M. Evolution of plant pathogenicity in Streptomyces // Ann. Rev. Phytopathol. 2006. V. 44. P. 469–487.
Magnet S., Blanchard J. Molecular insights into aminoglycoside action and resistance // Chem. Rev. 2005. V. 105. P. 477–497.
Morar M., Wright G. The genomic enzymology of antibiotic resistance // Ann. Rev. Genet. 2010. V. 44. P. 25–51.
Motkova M.O., Gladkikh E.G., Korobkova T.P. Stability of the antibiotic formation trait of the tobramycin producer Streptomyces cremeus subsp. Tobramycini // Antibiotiki. 1984. V. 29. № 2. P. 83–85.
Nepal K.K., Oh T.J., Sohng J.K. Heterologous production of paromamine in Streptomyces lividans TK24 using kanamycin biosynthetic genes from Streptomyces kanamyceticus ATCC 12853 // Mol. Cells. 2009. V. 27. № 5. P. 601–608.
Newman D.J., Cragg G.M. Natural products as sources of new drugs over the last 25 years // J. Nat. Prod. 2007. V. 70. № 3. P. 461–477.
Ogle J.M., Ramakrishnan V. Structural insights into translational fidelity // Ann. Rev. Biochem. 2005. V. 74. P. 129–177.
Ogle J.M., Carter A.P., Ramakrishnan V. Insights into the decoding mechanism from recent ribosome structures // Tr. Biochem. Sci. 2003. V. 28. P. 259–266.
Ota Y., Tamegai H., Kudo F. et al. Butirosin-biosynthetic gene cluster from Bacillus circulans // J. Antibiot. 2000. V. 53. № 10. P. 1158–1167.
Park D.H., Yu Y.M., Kim J.S. et al. Characterization of Streptomycetes causing potato common scab in Korea // Plant Dis. 2003. V. 87. P. 1290–1299.
Pethick F.E., MacFadyen A.C., Tang Z. et al. Draft genome sequence of the oxytetracycline-producing bacterium Streptomyces rimosus ATCC 10970 // Genome Announcem. 2013. V. 1. № 2. P. 1–2.
Ramirez M., Tolmasky M. Aminoglycoside modifying enzymes // Drug Resist. Updates. 2010. V. 13. P. 151–171.
Ramón-García S., Otal I., Martín C. et al. Novel streptomycin resistance gene from Mycobacterium fortuitum // Antimicrob. Agents Chemother. 2006. V. 50. № 11. P. 3920–3922.
Ribeiro da Cunha B., Fonseca L.P., Calado C.R. Antibiotic discovery: where have we come from, where do we go? // Antibiotics. 2019. V. 8. № 2. P. e45.
Salauze D., Perez-Gonzalez J.A., Piepersberg W., Davies J. Characterisation of aminoglycoside acetyltransferase-encoding genes of neomycin-producing Micromonospora chalcea and Streptomyces fradiae // Gene. 1991. V. 101. P. 143–148.
Scholz P., Haring V., Wittmann-Liebold B. et al. Complete nucleotide sequence and gene organization of the broad-host-range plasmid RSF1010 // Gene. 1989. V. 75. № 2. P. 271–288.
Seipke R.F., Kaltenpoth M., Hutchings M.I. Streptomyces as symbionts: an emerging and widespread theme? // FEMS Microbiol. Rev. 2012. V. 36. P. 862–876.
Serio A., Magalhães M., Blanchard J.S., Connolly L. Aminoglycosides: mechanisms of action and resistance // Antimicrobial drug resistance. Luxembourg: Springer, 2017. P. 213–229.
Shaeer K.M., Zmarlicka M.T., Chahine E.B., Piccicacco N. Plazomicin: a next-generation aminoglycoside // Pharmacotherapy. 2019. V. 39. № 1. P. 77–93.
Shakya T., Wright G. Nucleotide selectivity of antibiotic kinases // Antimicrob. Agents Chemother. 2010. V. 54. P. 1909–1913.
Shakya T.A., Stogios P., Waglechner N. et al. Small molecule discrimination map of the antibiotic resistance kinome // Chem. Biol. 2011. V. 18. P. 1591–1601.
Shi K., Berghuis A. Structural basis for dual nucleotide selectivity of aminoglycoside 2''-phosphotransferase IVa provides insight on determinants of nucleotide specificity of aminoglycoside kinases // Biol. Chem. 2012. V. 287. P. 13094–13102.
Shi K., Caldwell S., Fong D., Berghuis A. Prospects for circumventing aminoglycoside kinase mediated antibiotic resistance // Front. Cell. Infect. Microbiol. 2013. V. 3. № 22. P. 1–17.
Siti J.A., Mohd B., Syarul N.B. et al. Discovery of antimalarial drugs from Streptomycetes metabolites using a metabolomic approach // J. Tropic. Med. 2017. V. 2017. P. 1–7.
Sizova I.A., Lapina T.V., Frolova O.N. et al. Stable nuclear transformation of Chlamydomonas reinhardtii with a Streptomyces rimosus gene as the selective marker // Gene. 1996. V. 181. P. 13–18.
Sizova I., Fuhrmann M., Hegemann P.A. Streptomyces rimosus aphVIII gene coding for a new type phosphotransferase provides stable antibiotic resistance to Chlamydomonas reinhardtii // Gene. 2001. V. 277. P. 221–229.
Smith C.A. Baker E.N. Aminoglycoside antibiotic resistance by enzymatic deactivation // Curr. Drug Targ. Infect. Disord. 2002. V. 2. P. 143–160.
Smith C., Toth M., Frase H. et al. Aminoglycoside- 2''phosphotransferase-IIIa (APH(2'')-IIIa) prefers GTP over ATP: structural templates for nucleotide recognition in the bacterial aminoglycoside-2'' kinases // Biol. Chem. 2012. V. 287. P. 12893–12903.
Sutcliffe J. Improving on nature: antibiotics that target the ribosome // Curr. Opin. Microbiol. 2005. V. 8. P. 534–542.
Takahashi Y., Nakashima T. Actinomycetes, an inexhaustible source of naturally occurring antibiotics // Antibiotics. 2018. V. 7. № 45. P. 45.
Tao W., Yang A., Deng Z., Sun Y. CRISPR/Cas9-based editing of Streptomyces for discovery, characterization, and production of natural products // Front. Microbiol. 2014. V. 9. P. 1660–1668.
Thapa L.P., Oh T.J., Liou K., Sohng J.K. Biosynthesis of spectinomycin: heterologous production of spectinomycin and spectinamine in an aminoglycoside-deficient host, Streptomyces venezuelae YJ003 // J. Appl. Microbiol. 2008. V. 105. № 1. P. 300–308.
Thompson C.J., Gray G.S. Nucleotide sequence of a streptomycete aminoglycoside phosphotransferase gene and its relationship to phosphotransferases encoded by resistance plasmids // PNAS USA. 1983. V. 80. P. 5190–5194.
Tomihama T., Nishi Y., Sakai M., Ikenaga M. Draft genome sequences of Streptomyces scabiei S58, Streptomyces turgidiscabies T45, and Streptomyces acidiscabies a10, the pathogens of potato common scab, isolated in Japan // Genome Announcem. 2016. V. 4. P. 1–2.
Vakulenko S., Mobashery S. Versatility of aminoglycosides and prospects for their future // Clin. Microbiol. Rev. 2003. V. 16. № 3. P. 430–450.
Vastrad B.M., Neelagund S.E. Optimization of medium composition for the production of neomycin by Streptomyces fradiae NCIM 2418 in solid state fermentation // Biotechnol. Res. Int. 2014. V. 2014. P. 1–11.
Veyssier P., Bryskier A. Aminocyclitol aminoglycosides // Antimicrobial agents: antibacterials and antifungals agents. N.Y.: Wiley, 2005. P. 453–469.
Vicens Q., Westhof E. Molecular recognition of aminoglycoside antibiotics by ribosomal RNA and resistance enzymes: an analysis of X-ray crystal structures // Biopolymers. 2003. V. 70. P. 42–57.
Wilson D. Ribosome-targeting antibiotics and mechanisms of bacterial resistance // Nat. Rev. Microbiol. 2014. V. 12. P. 35–48.
Wright G.D. Molecular mechanisms of antibiotic resistance // Chem. Comm. 2011. V. 47. № 14. P. 4055–4061.
Wright G.D. Environmental and clinical antibiotic resistomes, same only different // Curr. Opin. Microbiol. 2019. V. 51. P. 57–63.
Wright G.D., Thompson P.R. Aminoglycoside phosphotransferases: proteins, structure, and mechanism // Front. Biosci. 1999. V. 4. P. D9–21.
Wright G., Berghuis A., Mobashery S. Aminoglycoside antibiotics: structures, functions, and resistance // Resolving the antibiotic paradox: progress in drug design and resistance / Eds B.P. Rosen, S. Mobashery. N.Y.: Plenum Press, 1998. P. 27–69.
Zaher H.S., Green R. Fidelity at the molecular level: lessons from protein synthesis // Cell. 2009. V. 136. P. 746–762.
Zarate S.G., De la Cruz Claure M.L., Benito-Arenas R. et al. Overcoming aminoglycoside enzymatic resistance: design of novel antibiotics and inhibitors // Molecules. 2018. V. 23. № 2. P. e284.
Zhang Y., Loria R. Emergence of novel pathogenic Streptomyces species by site-specific accretion and cis-mobilization of pathogenicity islands // Mol. Plant-Microbe Interact. 2017. V. 30. № 1. P. 72–82.
Zhukova Yu., Alekseeva M., Zakharevich N. et al. Pim family of protein kinases: structure, functions, and roles in hematopoietic malignancies // Mol. Biol. 2011. V. 45. № 5. P. 695–703.
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии