Успехи физиологических наук, 2019, T. 50, № 3, стр. 45-64
Роль мобильных генетических элементов в возникновении жизни
a Башкирский государственный медицинский университет
450008 Уфа, Россия
b Башкирский государственный университет
450076 Уфа, Россия
* E-mail: ruji79@mail.ru
Поступила в редакцию 04.10.2018
После доработки 08.12.2018
Принята к публикации 04.02.2019
Аннотация
Мобильные генетические элементы обладают универсальным свойством саморегуляции, необходимым для непрерывного самовоспроизведения биополимеров. Данное свойство отражается на протяжении всей эволюции живых организмов с сохранением транспозонов и происходящих от них структур у всех видов. У бактерий и архей ретроэлементы необходимы для взаимообмена базового и вспомогательного наборов генов, а также для горизонтального переноса адаптивных генов. У эукариот мобильные генетические элементы занимают основную долю геномов и служат ключевыми источниками для эволюционных преобразований, возникновения белок-кодирующих генов и регуляторных структур. Кардинальные отличия структуры и особенностей функционирования геномов эукариот возникли благодаря транспозонам, от которых произошли центромеры и теломеры, центромерный белок и теломераза, транскрипционные факторы и сайты связывания с ними, интроны и регуляторы сплайсинга, некодирующие РНК и комплементарные им последовательности в составе белок-кодирующих генов. Это отражает универсальную способность к саморегуляции на протяжение всей эволюции живого мобильных генетических элементов и входящих в их состав повторяющихся нуклеотидных последовательностей, которые могли быть первоначальными структурами биополимеров, давшими начало всем живым организмам. Консервативность свойств транспозонов на протяжении всей эволюции, а также эффективное возникновение новых механизмов саморегуляции биополимеров, ставших основой для появления эукариот и многоклеточности, позволяет предположить, что вирусоподобные элементы, обладающие свойствами транспозонов, стали основой возникновения жизни на Земле. Подтверждением служат многочисленные свидетельства взаимопревращений вирусов и транспозонов, способность мобильных генетических элементов к горизонтальному переносу и паразитизму в геномах вирусов, а также обнаружение полинтонов и вирусов PLV, обладающих свойствами и вирусов и транспозонов.
ВВЕДЕНИЕ
Повторяющиеся нуклеотидные последовательности (ПНП) являются избыточными, порою сложными и зачастую линие-специфичными элементами. Они представляют собой важнейшие “строительные” материалы для генов и геномов. Согласно их происхождению, характеристикам нуклеотидных последовательностей (НП) и путям распространения, ПНП в геномах подразделяются на мобильные генетические элементы (МГЭ) и сателлитные последовательности (СП), а также иерархически связанные с ними подсемейства и подгруппы [120]. Эффективность исследования биологических объектов зависит от систематизации составляющих их ключевых компонентов с определением структурно-функциональной роли каждой единицы. Для компьютерного моделирования генетических процессов оптимальным является использование универсальных кодировок, составляющих основу биополимеров последовательностей. Наиболее удобными объектами для этого являются ПНП, фланкирующие последовательности МГЭ, ключевых игроков эволюционных преобразований геномов всех живых организмов. Особенности структурной организации МГЭ и их способность к амплификации собственных последовательностей стали основой универсальных свойств живого, необходимых для непрерывного эволюционного прогресса. Появляется все больше данных о том, что МГЭ являются ключевыми источниками возникновения регуляторных структур геномов [34, 44, 76, 85, 93], экзонов [70, 73, 114] и интронов белок-кодирующих генов (БКГ) [6, 120]. МГЭ ответственны за структурные преобразования геномов, так как способствуют дупликации БКГ [73, 133], образованию межгенных областей геномов и теломер на концах хромосом [49, 69]. В эволюции МГЭ служат источниками как транскрипционных факторов (ТФ) [37, 44], так и сайтов связывания с ТФ [34, 59, 61, 85], источниками микроРНК [23, 51, 78, 84, 103] и мишенями данных молекул, сайтов альтернативного сплайсинга [70, 107, 128] и регуляторов сплайсинга [96]. Это говорит об универсальности МГЭ в качестве саморегуляторных элементов, являющихся наиболее подходящими вариантами для объяснения механизмов возникновения жизни. Их активное и ключевое участие в эволюции эукариот [7, 9] и управлении онтогенезом [8] подтверждает данное предположение.
Было обнаружено, что у эукариот МГЭ служат основой для формирования длинных некодирующих РНК (днРНК) [62, 65, 86], которые, помимо собственной функциональной активности самостоятельно или в составе РНП (рибонуклеопротеинов), являются важными источниками возникновения новых БКГ и пептидов [104, 126]. Данный механизм образования БКГ мог использоваться при зарождении жизни, при этом ключевую роль играли ПНП в составе МГЭ в качестве основы для образования активных доменов молекул РНК [62] и шпилечных структур при образовании микроРНК [23, 51, 78, 84, 103]. Функциональный дуализм МГЭ связан со способностью их транскриптов подвергаться процессингу разными ферментативными системами. При взаимодействии со сплайсосомой образуются мРНК, транслируемые в белки, необходимые для транспозиции. Процессинг же системой РНК-интерференции (РНКи) приводит к образованию некодирующих РНК (нкРНК), так как РНКи первоначально возникла в качестве универсального механизма защиты от инсерций [109]. МГЭ служат основой возникновения БКГ [37, 38, 77, 80], для которых также может быть характерна обработка транскриптов разными системами процессинга. Для генов днРНК и микроРНК данное свойство подтверждено. Показана способность при-микроРНК [32, 75, 87] и днРНК транслироваться в пептиды [18, 87, 92]. Поскольку обязательными составляющими МГЭ являются ПНП, фланкирующие их и необходимые для интеграции в геном, использование современных цифровых технологий с расшифровкой “кодировки повторов” при помощи математического моделирования может стать основой для анализа биологических процессов и прогнозирования молекулярных реакций. Примером эффективности применения “кодировки повторов” в естественных условиях в онтогенезе и эволюционных преобразований является способность МГЭ к локус-специфическим транспозициям благодаря узнаванию определенных ПНП в структуре ДНК [42, 48, 54]. Так как МГЭ высокочувствительны к стрессу [52, 60, 88, 116] и способны регулировать гены in cis и in trans [34, 59, 61, 85], локус-специфические транспозиции позволяют использовать их в качестве активных модуляторов работы генома при развитии организмов их хозяев.
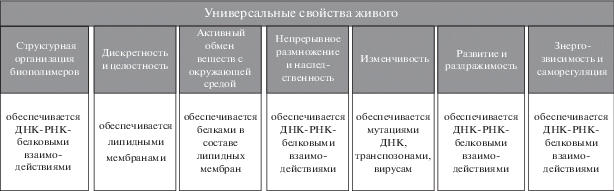
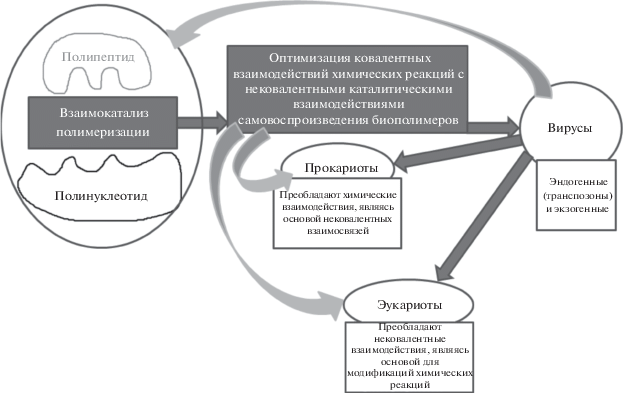
ПНП, составляющие основу МГЭ, долгое время считались второстепенными, “мусорными” компонентами геномов [35, 95]. Некоторые исследователи остаются приверженцами подобной концепции, однако с момента публикации Барбарой Мак Клинток в 1984 году статьи [89] о роли МГЭ в качестве важных регуляторов работы геномов, в литературе появилось множество работ, подтверждающих и дополняющих ее представления [34, 44, 59, 61, 76, 93]. Более того, накопленные научные данные позволяют предположить, что МГЭ и фланкирующие их ПНП служили основой для возникновения жизни и всех последующих этапов эволюции. Живое – это открытая высокодинамичная система, которая характеризуется универсальными свойствами (рис. 1) [16] и принципом комплементарности биомолекул в стремлении их к стабилизации с использованием ПНП как консервативных единиц [122].
Живое – мир биополимеров, обладающих способностью к преемственности за счет матричного синтеза, что обеспечивает его относительное бессмертие при условии постоянной изменчивости и смерти отдельных дискретных единиц. Ключевым принципом для возникновения данных процессов послужил отбор и использование наиболее универсальных ПНП, способных обеспечивать взаимокатализ молекул ДНК, РНК и белков. При этом отсеивались множества неподходящих путей, оставляя наиболее стабильные и успешные для эволюции. У бактерий они были связаны с обменом информаций между отдельными особями при помощи плазмид, инсерционных последовательностей (IS-элементы), транспозонов, интегронов с их генными кассетами, сложно организованных модульных структур (интегрирующие конъюгативные (ICE) и мобилизуемые элементы (IME), геномные острова) [1] и ретроэлементов (РЭ), обеспечивающих взаимопревращения базового набора генов (в составе генома) со вспомогательным [10]. У эукариот стал преобладать способ эндогенного паразитизма МГЭ в их геномах с использованием ДНК-транспозонов и ретроэлементов [7]. Однако у всех живых организмов основой для обмена информацией являлись выработанные в эволюции универсальные ПНП и способы их использования. Систематизация и подробный анализ всех известных ПНП с определением их функций может стать основой для программирования биологических процессов.
ОСОБЕННОСТИ СОСТАВА ПОВТОРОВ В ГЕНОМАХ ПРОКАРИОТ И ЭУКАРИОТ
У бактерий и архей ПНП в составе плазмид, интегронов и фагов служат ключевыми источниками приобретения новых признаков изменчивости и эволюции. У бактерий и архей также большое значение имеют ПНП в составе МГЭ, так как они обеспечивают переход генов вспомогательных категорий в базовые [10]. Несмотря на преобладание ДНК-транспозонов, ретроэлементы у бактерий подразделяются на 17 групп: ретроны, интроны группы II, DGR (diversity-generating retroelements), Abi-подобные, ассоциированные с CRISPR-Cas, подобные группе II ретроэлементы и 11 других мало изученных групп [117].
DGR представляют собой генетические кассеты. Они могут предоставлять огромное разнообразие в короткую, определенную область генома. Они достигают гипермутабельности путем замены вариабельной области сильно мутированной кДНК копией, генерированной при помощи обратной транскриптазы данного МГЭ. DGR дают преимущество хозяину, увеличивая его адаптивный потенциал. Они перемещаются между геномами путем взаимосвязей с различными МГЭ, фагами и плазмидами. При этом они демонстрируют высокую гибкость в размещении их компонентов и легко приобретают дополнительные паралоговые гены-мишени [106].
CRISPR используется, как адаптивная иммунная система, против инвазий вирусов и плазмид, с использованием определенных ПНП [118]. Выявлены сходства между CRISPR-Cas9 и РНКи, что предполагает их преемственность в эволюции. Например, в обоих механизмах используются малые РНК с целевой специфичностью 18–20 нуклеотидов [19]. Обзор геномов бактерий и архей показывает, что многие Tn7-подобные транспозоны содержат минимальные системы типа I-F CRISPR-Cas, которые состоят из скомбинированных генов cas8f и cas5f, cas7f и cas6f с коротким массивом CRISPR. Несколько небольших групп Tn7-подобных транспозонов сходны с усеченным типом I-B CRISPR-Cas. Филогенетический анализ показал, что эволюция содержащих CRISPR-Cas транспозонов включала единичный захват предкового локуса I-F-типа и два независимых захвата локусов I-B-типов. То есть транспозоны способны использовать компоненты системы защиты хозяина и перепрофилировать их на разных этапах жизненного цикла элемента [100]. Взаимосвязь МГЭ с CRISPR обусловлена общностью ферментов с РЭ. Так, обратная транскриптаза оказалась широко распространенной у бактерий, составляя наибольшее разнообразие у зеленых серных бактерий и цианобактерий [117].
В 2008 г. Roberts et al. предложили обновленную версию исходной системы номенклатуры для всех типов автономных МГЭ прокариот (табл. 1), за исключением инсерционных последовательностей (IS), для которых уже существует номенклатурная система, представленная на сайте www-IS.biotoul.fr [102]. У бактерий и архей РЭ оказались важными источниками разнообразия белков, потенциально связанных с их защитой, регуляцией и внедрением [98]. Помимо плазмид, интегронов, профагов и CRISPR-локусов, которые обеспечивают изменчивость, транспозоны прокариот могут содержать полезные для клетки гены, например, устойчивости к антибиотикам и тяжелым металлам. Часто такие гены локализованы на конъюгативных МГЭ (в них находятся гены, обеспечивающие образование конъюгативного мостика между бактериями и перенос ДНК в реципиентную клетку). У бактерий и архей встречаются инсерционные элементы одних и тех же семейств, что говорит о возможности их передачи друг другу [10]. У эукариот подобный адаптивный обмен невозможен, однако в геноме содержится множество неактивных в норме МГЭ, чувствительных к стрессу. Например, высокие концентрации железа в почве вызывают у риса Oryza sativa мобилизацию многих LTR, что вызывает адаптивную активацию генов, участвующих в гомеостазе железа [45]. МГЭ обнаруживаются в геномах почти всех видов прокариот, занимая у некоторых из них значительную долю генома, например, 11% всей ДНК у Clostridium difficile, 25% – у Enterococcus faecalis. У бактерии Orientia tsutsugamushi 46.7% генома составляют интегративные и конъюгативные элементы, а также 10 типов МГЭ. Прокариотические МГЭ характеризуются значительными различиями в их генетической организации, в ответственной за их инсерции генах, а также в их дополнительных и пассажирских генах. МГЭ способны также взаимодействовать путем рекомбинации между собой, формируя новые химерные элементы [102].
Таблица 1.
Типы мобильных генетических элементов прокариот (по данным [102])
| Тип МГЭ | Описание | Пример МГЭ |
|---|---|---|
| Композитные транспозоны | Фланкированы IS-элементами, транспозаза которых отвечает за катализ вырезания и вставки | Tn5 |
| Групповые транспозоны | Кодируют ферменты, вовлеченные в вырезание и интеграцию в основном в сайт специфической рекомбиназы или резольвазы, а также несколько добавочных генов в одной генетической группе | Tn3; Tn554; Tn4430 |
| (Интегративные)/ Конъюгативные транспозоны (ICEs)/(CTns) | Несут гены для вырезания, конъюгативного переноса и интеграции в новый геном хозяина. Несут широкий спектр добавочных генов, включая устойчивость к антибиотикам | Tn916; pSAM2; Tn5397 |
| (Интегративные)/Мобилизуемые транспозоны (IMEs)/(MTns) | Могут передвигаться между бактериальными клетками через поры и мосты спаривания, образуемые другими “вспомогательными” элементами | Tn4451; SGI1; Tn4555 |
| Мобильные геномные острова (MGI) | Могут кодировать тирозин- или серин-специфические рекомбиназы, катализирующие их вырезание и интеграцию, но не содержат вовлеченные в перенос гены. Название MGI отражает фенотип, который он кодирует (например, MGI токсинов, адгезинов и т.п.) | HPIYPS; SCCmec |
| Интегративный или мобильный профаг | Фаговый геном, инсертированный как часть линейной структуры хромосомы бактерии, которая способна к вырезанию и вставке | Lambda; Mu; phiC31 |
| Интегративный сателлитный профаг | Инсертированный геном фага требует генных продуктов “хелперных” фагов для завершения его цикла репликации | P4 |
| Интроны группы I | Малый посттранскрипционный сплайсинговый, кодирующий эндонуклеазу, элемент | Unnamed |
| Интроны группы II | Малый посттранскрипционный сплайсинговый, кодирующий рестрикционную эндонуклеазу элемент | Обратная транскриптаза |
| ISтроны | Химерный рибозим, состоящий из интрона группы I, связанного с IS605 | CdlSt1 |
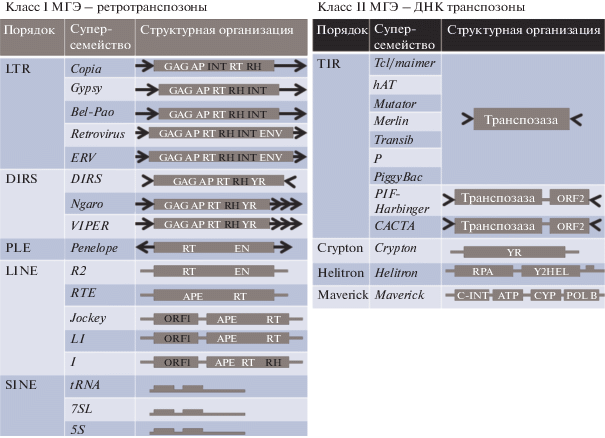
У эукариот МГЭ классифицируют по механизму перемещения и структуре на классы, подклассы (для ДНК-транспозонов), порядки, надсемейства, семейства, подсемейства. Класс I, ретроэлементы, характеризуются транспозициями по механизму “копирование–вставка”. Класс II, ДНК-транспозоны, перемещаются двумя способами: по механизму “вырезание–вставка” (порядки TIR и Crypton) и “катящегося кольца” (порядки Helitron и Maverick) [123]. МГЭ класса I транспозируют через промежуточное звено РНК. При этом исходная последовательность остается на прежнем месте [113]. Все ретроэлементы имеют два общих гена: gag и pol. Гены gag кодируют белки GAG (Group-specific Antigen), которые формируют вирусоподобные частицы VLPs (Virus-Like Particles). Гены pol кодируют ферменты обратную транскриптазу (RT – reverse transcriptase), интегразу (IN) и протеазу (PR) [17]. Оба класса МГЭ встречаются как у эукариот, так и у бактерий и архей, но у эукариот ретроэлементы оказались наиболее распространенными, занимая от 3 до 91% всей геномной ДНК [113]. Классификация МГЭ эукариот на порядки основана на их общей организации, особенностях ферментов перемещения и механизмов встраивания в геном (рис. 2) [123]. Наиболее полная классификация МГЭ представлена в Repbase (http://www.girinst.org/repbase/).
Рис. 2.
Классификация МГЭ эукариот по Wicker. LTR – long terminal repeats (длинные концевые повторы). GAG – капсидный белок, AP – aspartic proteinase (аспартатная протеиназа), INT – integrase (интеграза), RT – reverse transcriptase (обратная транскриптаза), RH – RNase H (РНКаза-Н), TIR – terminal inverted repeats (инвертированные концевые повторы), ENV – envelope protein (оболочечный белок), YR – тирозиновая рекомбиназа, EN – endonucleases (эндонуклеаза), APE – apurinic endonucleases (апуриновая эндонуклеаза), ORF – open reading frame (открытая рамка считывания), DFNR – diagnostic feature in non-coding region (диагностические признаки некодирующей области), RPA – replication protein A (репликативный белок А), HEL – helicase (хеликаза), ATP – упаковывающая АТФаза, CYP – cysteine protease (цистеиновая протеаза), POLB – DNA polymerase B (ДНК-полимераза В).
ВЗАМОСВЯЗЬ МОБИЛЬНЫХ ГЕНЕТИЧЕСКИХ ЭЛЕМЕНТОВ И САТЕЛЛИТОВ
В геномах эукариот МГЭ и СП классифицируются как основные компоненты ПНП геномов [90]. Значительная фракция эукариотических геномов представлена СП, составляющей тандемные ПНП. Эти элементы главным образом расположены в центромерах, перицентромерах и теломерах, являясь основным компонентом конститутивного гетерохроматина. Хотя первоначально СП считалась инертной, появляется множество доказательств ее транскрипционной активности. Оказалось, что нкРНК сателлитов играют роль в создании гетерохроматина в центромерах и теломерах [21], а МГЭ служат источниками создания тандемных повторов в эволюции [55, 83, 90]. Учитывая важную роль СП в ключевых структурно-функциональных преобразованиях геномов, отличающих эукариот от бактерий и архей, можно предположить, что ПНП МГЭ являлись основной движущей силой возникновения эукариот и сложнейших путей регуляции их геномов. Комбинированные изменения, связанные с повторами, нарушают структуры генов и геномов, например, путем экспансии экзонных, интронных и межгенных последовательностей, внося значительный вклад во все спектры мутаций. Так, при изучении 12 геномов млекопитающих с классификацией вариаций размеров интронов по МГЭ-доминантным и СП-доминантным экспансиям интронов, были определены некоторые закономерности. Например, выявлена доминантность по РЭ у млекопитающих и доминантность по ДНК-транспозонам у низших позвоночных, амфибий и рыб. Модели паттернов повторов генов мышей и крыс оказались наиболее яркими – была определена двумерная дивергенция: гены с доминантностью по СП были обогащены тканеспецифическим развитием и транскрипционной регуляцией во всех линиях млекопитающих. Кроме того, обнаружена тенденция большей роли МГЭ, по сравнению с СП, в увеличении размера интронов [120].
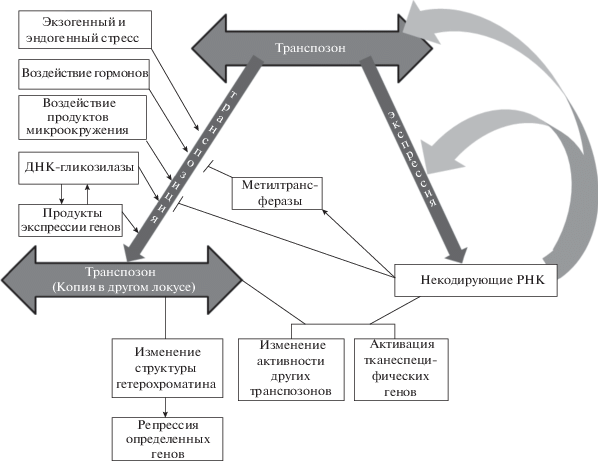
СП являются тандемно повторяющимися элементами, организованными в длинных массивах. Хотя МГЭ и СП различаются по структуре, геномной организации, механизмам распространения и эволюционной динамике, они могут разделять сходство последовательностей и шаблоны их организации. Это указывает на то, что сложные взаимоотношения могут определять их эволюцию и роль, которую они могут оказывать на архитектуру и функцию генома. Гомология НП между СП и МГЭ у некоторых видов говорит о происхождении СП от МГЭ, что обычно основано на простой амплификации частей МГЭ в виде тандемных массивов [90]. Так, обнаружено, что СП у двустворчатых моллюсков были образованы из древних МГЭ. Более того, данные МГЭ использованы как таксономический маркер у разных видов моллюсков [83]. Получены многочисленные подтверждения роли МГЭ в возникновении тандемных повторов, формировании сателлитной ДНК и центромер хромосом [53, 79, 90, 119, 124]. Для центромер растений характерно распределение с сателлитами специфических МГЭ, которые взаимодействуют с белком CenH3, принимая активное участие в функции центромер [90]. Выявлено, что МГЭ в составе центромер играют ключевую роль в формировании кинетохора [79, 124]. Было обнаружено происхождение тандемных повторов от МГЭ у морских беспозвоночных [119], морских китообразных [125], дрозофилы [56], у человека и шимпанзе (от Alu и ERVK9) [74], кенгуру [43], кукурузы [110], арабидопсиса [63] и эгилопса [29]. В то же время, оказалось, что консервативный для эукариот центромер-специфический белок CENP-B, который связывается с сателлитами центромер, произошел от транспозонов TC1/mariner [66]. Таким образом, саморегуляция МГЭ заключается в том, что в эволюции они стали источниками как центромер [53, 79, 90, 119, 124], так и взаимодействующего с ними белка CENP-B [66]. Подобные свойства саморегуляции стали причинами ключевых эволюционных преобразований живого. ПНП МГЭ стали основой как теломер [49, 69], так и гена теломеразы, взаимодействующего с ними [25]. МГЭ в эволюции стали ключевыми источниками как ТФ [37, 38, 44, 77, 80], так и сайтов связывания с ними [44, 76, 93]. МГЭ оказались важными источниками как генов нкРНК [23, 51, 78, 84, 103], так и их мишенями, что наряду со способностью МГЭ регулировать экспрессию БКГ in cis и in trans [34, 59, 61, 85] стало основой для глобального эпигенетического контроля работы генома. Консервативность свойства саморегуляции, амплификации собственных последовательностей с обретением новых адаптивных свойств вследствие мутаций МГЭ говорит о том, что данные элементы могли служить основой для возникновения жизни на Земле.
ЗНАЧЕНИЕ ПОВТОРОВ В ЭВОЛЮЦИИ ЖИВОГО
Универсальная способность МГЭ к саморегуляции используется благодаря ПНП в их составе, которые выполняют ключевые функции в эволюционных преобразованиях (табл. 2). Сами МГЭ представляют собой диспергированные ПНП, так как образуют множество копий внутри генома. Накопленные данные о роли ПНП в изменчивости и обретении универсальных свойств биологических объектов, говорят о том, что ПНП служили первичным необходимым признаком жизни (табл. 3). Функции МГЭ разнообразны и включают в себя регуляцию активности генов и сплайсинга РНК, использование их последовательностей в качестве альтернативных промоторов [3]. Инсерции МГЭ вблизи генов способны изменять паттерн их экспрессии. Встраивание в интрон может привести к формированию белка de novo за счет конкурентного отбора альтернативных сплайсинговых вариантов [107, 128]. Кроме того, некоторые МГЭ, например, SINE, накоплены в интронах геномов некоторых генов в большом количестве, образуя структуры стволовых петель, регулирующих экспорт, локализацию, стабильность, транскрипцию и процессинг РНК [70]. При зарождении жизни на Земле среди множества биополимеров, происходил отбор МГЭ, способных к воспроизведению собственных последовательностей благодаря ряду универсальных свойств, среди которых можно особо выделить взаимопаразитизм вирусоподобных структур, сходных по структуре и функции с МГЭ. Это означает, что одни МГЭ могут инсертировать в последовательности, образованные в результате амплификации других МГЭ. Новые структуры проходят отбор на пригодность и обретение новых адаптивных свойств. Естественно, что при этом одним из первых универсальных свойств, в ходе отбора возникли системы защиты от взаимопаразитизма, которые стали консервативными на протяжении всех последующих этапов эволюции, что стало основой как для возникновения эукариот [7], так и для межклеточной кооперации, необходимой для возникновения многоклеточности [9].
Таблица 2.
Роль повторов в составе МГЭ в эволюции
| Механизм использования повторов МГЭ | Роль в эволюции и онтогенезе | Источник литературы |
|---|---|---|
| Кодирование сайтов связывания с ТФ | Регуляция экспрессии генов | Feschotte, 2008; Lei, 2005; Nishihara, 2006 |
| Кодирование генетической информации | Трансляция в структурные домены белков | Kralovicova, 2015; Kubiak, 2017 |
| Переход вспомогательных генов в категорию базовых | Появление новых адаптационных признаков у бактерий и архей | Равин, 2013 |
| Сайты альтернативного сплайсинга и UTR | Появление новых изоформ белков | Schmitz, 2011; Elkon, 2013; Irima, 2014 |
| Защита теломерных концов хромосом | Обеспечение и регуляция количества делений клеток | Kopera, 2011; Garavis, 2013 |
| Создание центромер | Обеспечение митоза | Guo, 2016; Hara, 2012; Ferreri, 2011 |
| Обеспечение локус-специфических интеграций МГЭ | Программируемая активация определенных генов | Feng, 2013; Fujiwara, 2015; Han, 2014 |
| Возникновение сателлитных последовательностей | Структурно-функциональная регуляция работы генома | Lopez-Flores, 2004; Mestrovic, 2015; |
Таблица 3.
Функциональная роль МГЭ в эволюции и онтогенезе
| Механизм влияния | Роль в эволюции и онтогенезе | Источник литературы |
|---|---|---|
| Источники микроРНК, siРНК, piРНК | Эпигенетическая регуляция | Borchert, 2011; Gim, 2014; Li, 2011; Lorenzetti, 2016; Roberts, 2013 |
| Основа для построения генов днРНК | Johnson, 2014; Kapusta, 2014; Lu, 2014 |
|
| Дупликация БКГ при помощи обратной транскриптазы МГЭ | Возникновение новых генов, содержащих повторы МГЭ в своем составе | Kubiak, 2017; Huang, 2009 |
| Одомашнивание генов МГЭ | Duan, 2017; Dupressoir, 2012; Lescale, 2016; Lin, 2007 | |
| Активация МГЭ в ответ на стресс | Регуляция генов в ответ на стресс | Grandbastien, 2015; Jardim, 2015; Makarevitch, 2015; Todeschini, 2005 |
| Транспозиция в области, содержащие специфические повторы | Изменение активности определенных генов как адаптационный ответ | Feng, 2013; Fujiwara, 2015; Han, 2014 |
| Источники интронов БКГ | Возникновение альтернативных сплайсинговых вариантов и экзонизация | Le, 2015; Wang, 2012; Novikova, 2017; Schmitz, 2011 |
| Источники экзонов БКГ | Использование повторов МГЭ для возникновения новых функциональных доменов | Kralovicova, 2015; Kubiak, 2017 |
| Создание тандемных повторов | Структурные перестройки геномов, необходимые для новых регуляторных путей | Lopez-Flores, 2004; Mestrovic, 2015 |
| Возникновение регуляторных элементов | Регуляция работы генов в пространстве и времени | Feschotte, 2008; Lei, 2005; Nishihara, 2006 |
| Регуляция сплайсинга | Тканеспецифическая и стадиеспецифическая экспрессия альтернативных сплайсинговых вариантов | Lei, 2005; Pastor 2009; Kralovicova, 2015 |
| Процессинг транскриптов системой РНКи | Изменения метилирования ДНК и модификаций гистонов | Samantarrai, 2013; Xu, 2013; Zhang, 2016 |
| Влияние на экспрессию генов in cis и in trans | Управление работой генов | Lowe, 2012; de Souza, 2013; Jacques, 2013; Jjingo, 2014 |
| Создание генов центромер-связывающих белков | Возникновение митоза | Zdobnov, 2005; Feschotte, 2008 |
| Возникновение теломеразы | Структурно-функциональная регуляция работы генома, возникновение мейоза | Casacuberta, 2017 |
Существует множество подтверждений того, что эукариоты в эволюции возникли благодаря повторам МГЭ. Во-первых, в их геномах, в отличие от бактерий и архей, МГЭ составляют значительную долю. Например, у растения Asparagus officinalis только LTR-содержащие ретроэлементы занимают 91% всей ДНК [73]. За счет распространения МГЭ, размеры геномов эукариот могут отличаться в 70 000 раз [40]. Во-вторых, ПНП теломер и теломераза произошли от МГЭ в эволюции [49, 69], что стало универсальным признаком всех эукариот и дало возможность отличного от прокариот способа управления работы геномом. В-третьих, от МГЭ произошли сплайсосомные интроны, благодаря чему БКГ эукариот имеют мозаичную структуру, что важно для их регуляции [57] с участием МГЭ и произошедших от них функциональных структур генома [6]. МГЭ служат также источниками сайленсеров и энхансеров сплайсинга, связанных с малыми ядерными РНП и SR-белками [76]. В-четвертых, система РНКи, первоначально возникла для защиты от МГЭ [109]. Однако глобальное распространение МГЭ и их участие в возникновении новых БКГ [24, 26, 38, 91, 130] и регуляторных структур [41, 114] позволило использовать РНКи для эпигенетической регуляции экспрессии генов в онтогенезе. В-пятых, система модификации гистонов, характерная для эукариот, и направленная на регуляцию экспрессии генов стала универсальным признаком благодаря МГЭ. Это обусловлено ролью МГЭ в возникновении нкРНК [23, 51, 78, 84, 103], которые обладают способностью управлять модификациями гистонов в составе ферментативных комплексов [31, 37, 105]. Показано, что нкРНК, произошедшие от МГЭ, могут влиять на модификации определенных участков генома путем воздействия, как на гистоны, так и на метилирование ДНК [127, 132].
Эволюционные пути с использованием новых ПНП, возникших в результате интеграции в геном, при возникновении новых генов у эукариот могут отражать сходные процессы при зарождении жизни на Земле. ПНП составляют основу МГЭ, которые служат важными источниками функциональных доменов днРНК. Было показано, что 83% функциональных доменов днРНК содержат повторы МГЭ [62]. В то же время, помимо использования пространственных доменов собственной РНК-молекулы в различных биологических процессах [82], выявлена способность днРНК связываться с рибосомами и транслироваться в функциональные пептиды [18, 87, 92, 131]. Филогенетические исследования показали, что ряд БКГ произошли в эволюции от генов днРНК [104, 126]. МГЭ служат важными источниками генов микроРНК [23, 51, 78, 84, 103], транскрипты которых, в свою очередь, помимо процессинга в функциональные микроРНК, способны транслироваться в пептиды [32, 75, 87]. Интересно, что днРНК также могут служить прекурсорами малых нкРНК [64], а некоторые гены ретроэлементов используются в качестве генов днРНК [86]. Учитывая непосредственную роль МГЭ в возникновении БКГ [37, 38, 77, 80], и изменении их путем инсерций в интроны [120] и экзоны [70, 73], а также происхождение большей части регуляторных ПНП геномов эукариот от МГЭ [44, 76, 93], можно говорить о ключевой роли МГЭ и фланкирующих их ПНП в эволюции. В связи с этим МГЭ могут рассматриваться как первопричины возникновения и эволюции всех биологических процессов. Ключевым звеном в данных процессах служат ПНП, отбор и сохранение которых является формой кодирования генетической информации.
Геном хозяина контролирует мобильность МГЭ механизмами косупрессии, опосредуемой малыми интерферирующими РНК (siРНК) [15], микроРНК, piwi РНК [27]. Однако МГЭ сами играют важную роль в качестве источника некодирующих РНК [23, 51, 78, 84, 103], что говорит об использовании хозяевами способности к саморегуляции МГЭ в эволюции и онтогенезе [7, 8] (рис. 3). Повторы являются повсеместно распространенными элементами в белках и играют важную роль в клеточных функциях и в эволюционных процессах. Были проведены методы детекции повторов и анализ большого набора данных, содержащих информацию о сотнях геномов многоклеточных животных. Анализ структурных данных показал, что повторы с открытой структурой и независимо складывающиеся единицы более непостоянны и склонны к внутренней неупорядоченности. Такие неупорядоченные повторы также значительно обогащены сайтами с высоким функциональным потенциалом, таким как линейные мотивы. Более того, наиболее непостоянные повторы характеризуются высоким сходством последовательностей между их единицами. Многие из непостоянных повторов проявляют признаки рекомбинации и часто формируются путем консервативной эволюции. Многие из этих консервативных повторов участвуют во взаимодействиях хозяина с патогеном, способствуя быстрой, но тонкой адаптации в биологических гонках вооружений [108]. Повторяющиеся сотни и тысячи раз в геноме повторы составляют основную долю всей ядерной ДНК большинства эукариотических геномов. Однако большинство биоинформационных исследований сфокусированы на низкокопийные ДНК, включая гены. Поэтому анализируются лишь повторы, представленные одной или несколькими копиями, что часто маскирует или игнорирует данные считывания ДНК или РНК. Одновременно остаются открытыми множество вопросов о происхождении, эволюции и функциях ПНП в геномах. В работе Biscotti et al. проведена детальная характеристика природы всех повторов, в частности, их организация, распространенность, хромосомная локализация, вариации последовательностей внутри и между хромосомами, их транскрипционная и экспрессионная активности. Сравнение природы и локализации ПНП между близкими видами дает обширную информацию об их эволюции и амплификации. Некоторые ПНП чрезвычайно хорошо сохраняются между видами, тогда как другие более вариабельны и определяют существенные различия. Эти данные свидетельствуют о контрастных способах эволюции [22].
РОЛЬ ПОВТОРОВ В СТРЕМЛЕНИИ К СТАБИЛИЗАЦИИ И ПРИНЦИПЕ КОМПЛЕМЕНТАРНОСТИ
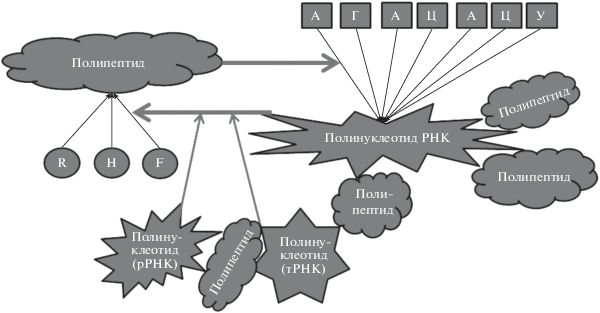
С современных позиций в зарождении жизни имел решающее значение автокаталитический цикл неклеточных РНК-организмов с образованием собственных копий [4]. При возникновении живой материи под действием ультрафиолетовых лучей образовывались аминокислоты, липиды и другие органические вещества, образовались коацерваты – многомолекулярные комплексы биополимеров способные к самосборке [101]. В первичном бульоне возникало большое количество возможных комбинаций органических веществ, каждое из которых могло взаимодействовать с другими молекулами как путем химических реакций, так и нековалентных взаимосвязей. При этом шел отбор наиболее стабильных веществ, способных к химическим и нековалентным взаимодействиям с другими молекулами – в наиболее высокой гармонии между РНК и белками благодаря принципу комплементарности. В связи с этим первые формы жизни, способные к непрерывному самовоспроизведению, представляли собой вирусоподобные структуры. Дальнейшие химические и нековалентные взаимодействия позволили гармонизировать систему непрерывных и преемственных взаимосвязей липидов, углеводов, пептидов и нуклеиновых кислот, эволюция которых продолжается и в настоящее время, проявляясь биохимическими и структурными особенностями во всех имеющихся формах жизни. Несмотря на то, что (с современных позиций) в зарождении жизни имел решающее значение автокаталитический цикл РНК-организмов образования собственных копий [4], главную роль играло взаимодействие РНК с белками в виде взаимокатализа (использование специфических белков для образования полимеров РНК и активное участие молекул РНК для образования новых белков, которые, в свою очередь, служат катализаторами образования новых РНК). Для зарождения жизни ключевую роль играло образование собственных копий белков и полинуклеотидов как оптимальных, эволюционно сложившихся взаимокатализаторов. Действительно, из аминокислот образуются полипептиды при катализе и контроле полинуклеотидов (РНК, ДНК), в то время как для образования полинуклеотидов обязателен катализ и контроль полипептидов (РНК-полимераза, ДНК-полимераза). Данная динамическая взаиморегуляция, в которой пространственная структура РНК позволила им образовывать оптимальный набор молекул для стабильного взаимодействия с белками как для собственного воспроизведения, так и для катализа образования полипептидов, стало основой для жизни с непрерывным воспроизведением биополимеров во времени и пространстве (рис. 4). При этом решающее значение имело образование оптимальных пространственных структур транспортных и рибосомальных РНК, где используется особый вид кодировки нековалентных взаимодействий с аминокислотами и белками, обеспечивающих самовоспроизведение, отобранных среди миллиарда возможных вариантов. Для главного принципа всего живого – непрерывного преумножения собственных копий во времени и пространстве биомолекул полипептидов и полинуклеотидов, используются закономерности взаимокаталитических функций данных биомолекул. Дальнейшие биохимические реакции с использованием большого разнообразия новых биомолекул, обеспечивали наибольшую надежность данного принципа, основанного на комплементарности структур со стремлением к стабилизации.
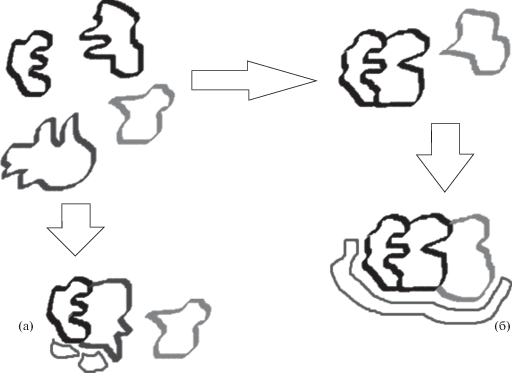
Возникновение жизни было обусловлено конденсацией органических молекул, их химическими и нековалентными взаимодействиями с отбором наиболее стабильных вариантов, способных к катализу друг друга. При этом формировались различные способы кодирования информации, основанные на использовании и отборе наиболее универсальных повторов, необходимых для комплементарности при нековалентном взаимодействии молекул. В динамичной системе постоянное стремление к хаосу заканчивается только при приобретении относительно стабилизированного состояния, например, в реакциях атомных ядер при достижении равновесия между силами электростатического отталкивания протонов и ядерными силами притяжения [13]. Живое является неотъемлемой частью материального мира и подчиняется тем же законам. Однако возникновение жизни стало возможным благодаря универсальным особенностям около 4 миллиардов лет назад на Земле в оптимальных условиях на момент времени. Универсальные особенности биополимеров возникли в связи со стремлением к стабилизации и появлением принципа комплементарности, основанном на приобретении органической молекулой более стабильного состояния при присоединении к ее активному центру вещества “как ключ к замку”, с образованием слабых, нековалентных связей, которых достаточно для стабилизации. Нековалентные связи обычно образуются во временных взаимодействиях за счет комбинации слабых связей: ионных, водородных, ван-дер-ваальсовых или гидрофобных [122] (рис. 5). При этом взаимодействие биомолекул оказалось высокоспецифичным. Так, узнавание аминоацил-тРНК-синтетазой строго определенных аминокислот и антикодонов, узнавание ТАТА-бокса ДНК ТАТА-связывающим белком, являются консервативными свойствами живого [20].
МОДИФИКАЦИИ УНИВЕРСАЛЬНЫХ СВОЙСТВ САМОРЕГУЛЯЦИИ В ЭВОЛЮЦИИ
Существуют две основные гипотезы происхождения жизни. Сторонники химической эволюции считают, что жизнь возникла непосредственно на Земле из косной материи в результате абиогенного синтеза органических соединений. Сторонники гипотезы панспермии склоняются к версии, что бактерии занесены на Землю из космоса под давлением солнечных лучей, с астероидами и, в первую очередь, углистыми метеоритами, богатыми различными органическими веществами [33]. Исследование метеоритов, упавших на Землю в разное время, дает основание для развития данной гипотезы [2]. В 1924 г. биохимик Опарин предположил, что при массивных электрических разрядах в земной атмосфере, которая около 4 млрд лет назад состояла из аммиака, метана, углекислого газа и паров воды, могли появиться органические соединения, достаточные для возникновения жизни. В 1955 г. данное предположение было подтверждено Миллером С., который пропускал электрические заряды через смесь газов и паров воды, воспроизводя условия первобытной Земли, при этом получив простые жирные кислоты, аминокислоты, мочевину, органические кислоты. В начале 1980-х гг. была установлена способность РНК к саморепродукции в отсутствие белковых ферментов. Таким образом, сегодня считается, что протобионт представлял собой молекулы РНК, которые совмещали в себе черты фенотипа и генотипа – то есть РНК могла быть подвержена генетическим преобразованиям и естественному отбору. Процесс эволюции шел от РНК к белку, а далее к образованию молекулы ДНК, у которой С–Н связи более прочны, чем С–ОН связи РНК [11].
Считается, что первые прокариоты возникли более 4 млрд лет назад [11]. Эукариоты появились значительно позднее, что стало основой значительного возрастания как количества живых организмов, так и их разнообразия. Если размеры геномов бактерий находятся в диапазоне от 0.58 млн пар нуклеотидов (п. н.) у Mycoplasma genitalium до 9.2 млн п. н. у Bradhyrhizobium japonicum (в 16 раз), архей – от 0.49 млн п. н. у Nanoarchaeum equitans до 5.75 млн п. н. у Methanosarcina acetivorans (в 12 раз) [10], то размеры геномов эукариот составляют от 2.3 млн п. н. у Encephalitozoon itestinalis до 148 852 млн п. н. у Paris japonica (в 70 000 раз). При этом разнообразие размеров геномов между таксонами эукариот связано не с количеством белок-кодирующих генов (как у прокариотов), а с различным содержанием некодирующих ДНК, доминирующее значение в происхождении которых занимают МГЭ [40].
У прокариот, в геномах которых основную часть составляют белок-кодирующие последовательности (80–90%), изменчивость в адаптации происходит за счет необходимости формирования биохимических реакций, обеспечивающих нековалентные взаимодействия биополимеров для непрерывной преемственности структурной организации. При этом важным фактором изменчивости, обеспечивающей адаптивное изменение биохимических реакций, служит паразитизм чужеродных НП – обмен информацией при помощи вирусов (фагов) и плазмид. У эукариот, большинство которых представлены многоклеточными организмами, обмен информацией значительно изменился – белок-кодирующие области геномов составляют незначительную часть. При этом главными факторами изменчивости служат МГЭ, обеспечивающие большое количество различных нековалентных взаимодействий биополимеров, лежащих в основе возможных биохимических реакций для обеспечения жизнедеятельности (рис. 6).
Предполагается, что первыми на Земле появились архебактерии, имевшие различную морфологию и способные восстанавливать углекислый газ и серу, окислять водород, выделять сероводород и метан и создавать органические вещества из неорганических. Использование в качестве источника энергии солнечного света позволило фотосинтезирующим бактериям образовывать из воды и углекислоты органические вещества с выделением кислорода, в результате чего сформировалась кислородная атмосфера с появлением оксибактерий. Наиболее убедительные палеонтологические останки эукариот обнаружены в породах возрастом около 1.4 млрд. лет [2]. Существенным фактором, способствовавшим формированию и эволюции эукариот, стал также симбиоз вирусами, которые, подобно предкам митохондрий и пластид, потеряли способность к самостоятельности, функционируя исключительно с целью лучшей адаптации клеток и приобретения ими новых адаптивных свойств, необходимых для оптимальной саморепродукции. Предполагается, что в ядро эукариот возникло из древнего вируса группы NCLDV (Nucleo Cytoplasmic Large DNA Viruses) [47]. Прокариотам свойственны жесткие способы защиты от внедрения вирусов в геном, так как это может нарушить биосинтетические пути, обеспечивающие взаимодействие и преемственность биополимеров. Появление эукариот и резкий скачок эволюции с образованием многоклеточных организмов связан с оптимизацией симбиоза геномов и МГЭ, которые обеспечивали адаптацию к стрессорам, что способствовало приобретению новых признаков и выживанию в меняющихся условиях обитания (рис. 7) [52, 60, 88, 116]. Роль чувствительных к стрессу структур, в возникновении эукариот, объясняет их происхождение от архей, характерной чертой которых, является обитание в агрессивных условиях среды [10, 14]. Эндогенные вирусы играют роль в адаптации к стрессу также у прокариот. Например, при исследовании культур E. coli выявлено, что реверсия мутации лактозного гена происходит с участием как плазмиды F ', так и инсерционного элемента [5].
С позиции принципа комплементарности жизнь могла зародиться в виде вирусоподобных структур, обладающих как свойствами МГЭ, так и вирусов, основу которых составляли ПНП, которые с вероятностным отбором комбинировались в различных модификациях для взаимодействий полинуклеотидов, полипептидов, полисахаридов и липидов, с формированием относительно стабильных органических молекул. Наиболее удачные пути взаимодействий, приводящие к увеличению количества биополимеров, в свою очередь, становились основой для дальнейших усложнений, неотъемлемым свойством которых являлась изменчивость. Изменчивость обеспечивалась за счет паразитизма одних МГЭ с другими, как универсального способа, обеспечивающего возможность выживания биомассы на всех этапах эволюции, начиная с самых ранних и заканчивая настоящим временем. Вирусные свойства взаимопаразитизма менялись в различных эволюционных звеньях, давая основу разветвлениям филогенетического древа. Эффективный симбиотический паразитизм первых вирусоподобных форм жизни стал основой усложняющихся взаимосвязей между различными биополимерами во всех возможных модификациях, часть из которых оказалась относительно стабильной, рождая все новые структуры. Это объясняет ключевую роль обмена информации у прокариот при помощи горизонтального переноса (ГП) плазмидами, фагами, интегронами в качестве источника эволюционной изменчивости, что является отражением первоначальных свойств при зарождении жизни. Кроме того, имеется ряд данных о взаимопревращениях МГЭ и вирусов в эволюции, а так как МГЭ составляют большую часть структур геномов эукариот, можно предположить, что все этапы эволюции отражают универсальные принципы использования ПНП, возникшие при зарождении жизни. Так, состав МГЭ соответствует распределению вирусов для разных доменов жизни. У прокариот наиболее распространены ДНК-транспозоны, а для вирусов характерно преобладание ДНК-содержащих видов. Среди вирусов эукариот преобладают РНК-содержащие, тогда как характерно также превалирование ретроэлементов над ДНК-транспозонами [68]. Имеются данные о том, что экзогенные РНК-содержащие ретровирусы произошли от Ty3/Gypsy LTR-содержащих ретроэлементов [111]. Предполагается также, что ДНК-вирусы возникли в эволюции от ДНК-транспозонов, а РНК-вирусы – от ретроэлементов, способных к ГП и утративших свойства интеграции наряду с приобретением признаков вирулентности. Доказательством служат обнаруженные в ряде исследований промежуточные формы элементов – способные к ГП МГЭ [36], а также способность эндогенных ретроэлементов проявлять свойства вирусов при обмене информации между нейронами в головном мозге. Произошедшие от Ty3/Gypsy LTR-МГЭ при развитии нервной системы способны образовывать частицы, покрытые капсидами из эндогенных белков Arc и переносить информацию между отдельными клетками одного организма [97]. Тропизм вирусов, не содержащих в своем составе кодирующих необходимых для интеграции белков, и способность к интеграции в геномы строго определенных тканей [112, 115] также говорит об их общем происхождении с МГЭ. Это обусловлено использованием активации специфических МГЭ геномами хозяев для тканеспецифической регуляции генной экспрессии [58, 94, 99]. Например, при заражении, герпесвирусы и аденовирусы интегрируют часть своего генома, онковирусы [112] и вирус гепатита В могут встраивать в ДНК хозяина весь свой геном [115].
Было показано, что вирусы насекомых могут активировать ГП сходных с гелитронами МГЭ [30]. Выявлена способность МГЭ к ГП между различными видами организмов. Так, описан ГП четырех семейств МГЭ между клопами Rhodnius prolixus и беличьими обезьянами, на которых они паразитируют [50]. Доказан факт ГП P, mariner, copia между представителями разных видов животных. Описан ГП Ty1/copia и Ty3/gypsy между видами рода Drosophila, а также ретроэлементов семейства RIRE1 между видами рода Oryza. Определенные LTR-содержащие РЭ способны формировать вирусоподобные частицы с инфекционной активностью. Некоторые РЭ растений содержат открытые рамки считывания, кодирующие белки, подобные белку оболочки Env ретровирусов, что также не исключает их возможный горизонтальный перенос между растениями. Более того, филогенетический анализ показал, что ретровирусы, вероятнее всего, возникли из LTR-ретроэлементов в результате приобретения дополнительного гена env, продукт трансляции которого позволил проторетровирусу сформировать вирусную частицу, которая стала обладать инфекционными свойствами [12]. Как и МГЭ, вирусы также способны к межвидовым трансмиссиям, что говорит об их общем происхождении. Так, ретровирус HIV семейства Lentiviridae возник у людей после множества независимых зоонозных событий, включающих межвидовые трансмиссии вируса обезьяньего иммунодефицита (SIV) среди приматов, не относящихся к человеку [121].
Доказана способность взаимопревращений МГЭ и вирусов в эволюции. Например, у растений вирусы семейства Caulimoviridae произошли от LTR-содержащих ретроэлементов [81]. Вирофаги, по генетической гомологии представляют собой эволюционное звено между двуцепочечными ДНК-содержащими вирусами и эукариотическими ДНК-транспозонами Maverick/Polinton [46]. Элементы семейства МГЭ Polinton представляют собой альтернативную форму существования между вирусами и МГЭ, так как они кодируют собственные белки капсидов, протеиназу созревания капсидов, ретровирусоподобную интегразу, ДНК-полимеразу и упаковывающую ДНК АТФазу [72]. Полинтоны могут распространяться при помощи ГП [39], а сравнительный геномный анализ с другими вирусами показал, что от них произошли многие вирусы эукариот, включая аденовирусы и мегавирусы [71]. Имеются также данные о взаимопревращениях плазмид, вирусов и МГЭ, что говорит об универсальности данных структур в консервативном свойстве обмена генетической информацией, возникшем при зарождении жизни на Земле. Последующие этапы эволюции усложнили формы существования биологических объектов с использованием мембранных структур. Так, показано общее происхождение линейных цитоплазматических плазмид бактерий с полинтонами, вирофагами и полинтон-подобными вирусами [72]. У галоархей в плазмиде pHRDV1 содержится 6 открытых рамок считывания, сходных с таковыми у плеоморфных вирусов Halorubrum pleomprphic virus. При этом характерно сходное расположения генов, а также наличие идентичных белков VP3, VP4, VP8 у вирусов и плазмиды [28]. Обнаружена также эволюционная взаимосвязь между линейными митохондриальными плазмидами, полинтонами и бактериальными тективирусами [71]. В эволюции non-LTR ретроэлементы считаются предками LTR-содержащих РЭ и ретровирусов, а их механизм трансляции обусловлен бицистронной РНК [67].
В ряде исследований выявлено множество эволюционных взаимосвязей между линейными плазмидами, бактериофагами, МГЭ и двухцепочечными ДНК-вирусами эукариот [68]. Это свидетельствует об универсальных свойствах данных структур и о наличии их общего предка, возникшего при появлении первых признаков жизни на Земле. Данные структуры являются первичным свойством живого, основанным на отборе и консервации оптимальных ПНП, необходимых для взаимокатализа самовоспроизведения биополимеров. Открытие элементов, характеризующихся свойствами вирусов и МГЭ [71], а также взаимопревращения в эволюции МГЭ и вирусов [3, 36, 81, 134] говорит о том, что данные элементы являются необходимым свойством, без которого должный уровень изменчивости, обеспечивающий поддержание жизни был бы невозможен. Наиболее вероятно, что они были также основой возникновения биологической материи и всех ее последующих этапов развития, о чем свидетельствуют данные роли МГЭ и ПНП в эволюции и онтогенезе. Обнаружены вирусы PLV, которые сходны с полинтонами и вирофагами по размеру генома и консервативным морфогенетическим модулям. PLV могут кодировать тирозиновую рекомбиназу-интегразу, сходную с таковой у бактерий и фагов. Кроме того, PLV характеризуются формой существования как в виде вирусов, так и МГЭ и способны, подобно полинтонам, интегрировать в геномы хозяев [129].
ЗАКЛЮЧЕНИЕ
На основании анализа литературных данных получены закономерности ключевой роли МГЭ и входящих в их состав ПНП в эволюции всех живых организмов, что позволяет предположить, что МГЭ являлись первоначальными элементами, которые стали основой возникновения всех живых организмов:
1. Ретроэлементы являются обязательной составной частью CRISPR бактерий.
2. Ретроэлементы жизненно необходимы для жизнедеятельности бактерий и архей, так как обеспечивают взаимопревращения базового и вспомогательного наборов генов в ходе эволюции.
3. Доказана возможность возникновения новых клеточных генов у прокариот из последовательностей МГЭ.
4. Доказана регуляторная роль МГЭ в обеспечении жизнедеятельности бактерий.
5. Получены многочисленные доказательства взаимопревращений вирусов и МГЭ в эволюции всех живых организмов. Кроме того, выявлены элементы, способные существовать как в виде вирусов, так и в виде МГЭ, обеспечивая в ряде случаев передачу адаптивных признаков хозяевам.
6. Накоплен достаточный материал, доказывающий происхождения БКГ и ТФ эукариот от МГЭ в эволюции. При этом большинство сайтов связывания с ТФ также происходят от МГЭ, что свидетельствует о наличии у МГЭ универсальных свойств, необходимых для самовоспроизведения, что могло быть приобретено при возникновении жизни на Земле в ходе длительного отбора биополимеров.
7. Доказано возникновение большей части регуляторных элементов в геномах эукариот от МГЭ.
8. Все сплайсосомные интроны произошли от МГЭ, их сохранение в геномах эукариот обусловлено наличием в составе МГЭ повторов, использующихся для саморегуляции процессированными продуктами собственной транскрипции.
9. МГЭ служат важнейшими источниками альтернативных сплайсинговых вариантов, что необходимо для возникновения новых белков в эволюции.
10. Получены доказательства сходства МГЭ и вирусов по ряду свойств, при этом МГЭ могут паразитировать в геномах наиболее крупных вирусов, участвуя в адаптивных процессах их жизненного цикла.
11. МГЭ способствовали возникновению эукариот, при этом эндогенный паразитизм стал основой для консервативных свойств геномов их хозяев. От МГЭ произошли тандемные повторы и центромеры, в то же время от МГЭ произошел консервативный для эукариот центромер-связывающий белок, что говорит о значимости и универсальности свойства саморегуляции МГЭ в эволюции и позволяет предположить, что МГЭ служили первоначальными элементами, от которых произошли все последующие формы жизни на Земле. От МГЭ произошли теломеры и теломераза. Таким образом, благодаря МГЭ эукариоты приобрели способность к митозу и мейозу.
12. В ряде работ доказано возникновение от МГЭ микроРНК, siРНК, piРНК и днРНК благодаря процессингу системами, направленными для защиты против паразитизма МГЭ хозяевами. Образуемые нкРНК используются при этом для регуляции экспрессии не только МГЭ, но и БКГ, так как многие из них в эволюции либо произошли непосредственно от МГЭ, либо содержат их НП благодаря экзонизации инсертированных элементов. Данные обстоятельства подтверждают универсальность свойств саморегуляции МГЭ, направленных на непрерывное самовоспроизведение биополимеров, что было ключевым обстоятельством, необходимым для возникновения жизни на Земле.
Список литературы
Ильина Т.С. Мобильные ISCR-элементы: структура, функции и роль в создании, наращивании и распространении блоков бактериальных генов множественной резистентности к антибиотикам // Молекулярная генетика, микробиология и вирусология. 2012. Т. 4. С. 3–13.
Катола В.М. Древние прокариоты: происхождение, эволюционный путь и роль земли // Бюллетень физиологии и патологии дыхания. 2014. № 52. С. 129–135.
Киселев О.И. Эндогенные ретровирусы: структура и функция в геноме человека // Вопросы вирусологии. 2013. № 1. С. 102–115.
Крицкий М.С., Телегина Т.А. Коферменты и эволюция мира РНК // Успехи биологической химии. 2004. Т. 44. С. 341–464.
Маркель А.Л. Стресс и эволюция // Вестник ВОГиС. 2008. Т. 12. № 1/2. С. 206–215.
Мустафин Р.Н., Хуснутдинова Э.К. Роль интронов в эволюции // Успехи физиологических наук. 2015. Т. 46. № 1. С. 93–102.
Мустафин Р.Н. Роль вирусов в эволюции геномов // Успехи физиологических наук. 2016. Т. 47. № 2. С. 77–97.
Мустафин Р.Н., Хуснутдинова Э.К. Роль мобильных элементов генома в онтогенезе // Успехи физиологических наук. 2016. Т. 47. № 3. С. 70–96.
Мустафин Р.Н., Хуснутдинова Э.К. Общие универсальные генетические закономерности в основе возникновения и эволюции многоклеточных эукариот // Успехи физиологических наук. 2018. Т. 49. № 3. С. 52–81.
Равин Н.В., Шестаков С.В. Геном прокариот // Вавиловский журнал генетики и селекции. 2013. Т. 17. № 4/2. С. 972–984.
Садохин А.П. Концепции современного естествознания. М. 2006. 447 с.
Сормачева И.Д., Блинов А.Г. LTR-ретротранспозоны растений // Вавиловский журнал генетики и селекции. 2011. Т. 15. № 2. С. 351–381.
Сулейманов Е.В., Фаддеев М.А. Состав, строение и свойства атомных ядер. Учебно-методическое пособие. Нижний Новгород. 2005. 39 с.
Шестаков С.В. Роль архей в происхождении эукариот // Экологическая генетика. 2017. Т. 15. № 4. С. 52–59.
Юрченко Н.Н., Коваленко Л.В., Захаров И.К. Мобильные генетические элементы: нестабильность генов и геномов // Вавиловский журнал генетики и селекции. 2011. Т. 15. № 2. С. 261–270.
Albert B., Johnson A., Lewis J. et al. Molecular Biology of the Cell. 2015. 1465 p.
Alzohairy A.M., Gyulai G., Jansen R.K., Bahieldin A. Transposable elements domesticated and neofunctionalized by eukaryotic genomes // Plasmid. 2013. V. 69. № 1. P. 1-15.
Anderson D.M., Anderson K.M., Chang C.L. et al. A micropeptide encoded by a putative long noncoding RNA regulates muscle performance // Cell. 2015. V. 160. № 4. P. 595–606.
Barrangou R., Birmingham A., Wiemann S. et al. Advances in CRISPR-Cas9 genome engineering: lessons learned from RNA interference // Nucleic Acids Res. 2015. V. 43. № 7. P. 3407–3419.
Barrett L.W., Fletcher S., Wilton S.D. Regulation of eukaryotic gene expression by the untranslated gene regions and other non-coding elements // Cell Mol. Life Sci. 2012. V. 69. № 21. P. 3613–3634.
Biscotti M.A., Canapa A., Forconi M. et al. Transcription of tandemly repetitive DNA: functional roles // Chromosome Res. 2015. V. 23. P. 463–477.
Biscotti M.A., Olmo E., Heslop-Harrison J.S. Repetitive DNA in eukaryotic genomes // Chromosome Res. 2015. V. 23. P. 415–420.
Borchert G.M., Holton N.W., Williams J.D. et al. Comprehensive analysis of microRNA genomic loci identifies pervasive repetitive-element origins // Mobile Genetic Elements. 2011. V. 1. P. 8–17.
Campillos M., Doerks T., Shah P.K., Bork P. Computational characterization of multiple Gag-like human protein // Trends Genet. 2006. V. 22. № 11. P. 585–589.
Casacuberta E. Drosophila: Retrotransposons Making up Telomeres // Viruses. 2017. V. 9. № 7. pii:E192. doi https://doi.org/10.3390/v9070192
Casola C., Lawing A.M., Betran E., Feschotte C. PIF-like transposons are common in drosophila and have been repeatedly domesticated to generate new host genes // Mol. Biol. Evol. 2007. V. 24. № 8. P. 1872–1888.
Castellano L., Rizzi E., Krell J. et al. The germline of the malaria mosquito produces abundant miRNAs, endo-siRNAs and 29-nt small RNAs // BMC Genomics. 2015. V. 16. № 1. P. 100–106.
Chen S., Wang C., Xu J.P., Yang Z.L. Molecular characterization of pHRDV1, a new virus-like mobile genetic element closely related to pleomorphic viruses in haloarchaea // Extremophiles. 2014. V. 18. P. 195–206.
Cheng Z.J., Murata M. A centromeric tandem repeat family originating from a part of Ty3/gypsy-retroelement in wheat and its relatives // Genetics. 2003. V. 164. P. 665–72.
Coates B.S. Horizontal transfer of a non-autonomus Helitron among insects and viral genome // BMC Genomics. 2015. V. 16. № 1. P. 137–140.
Cordaux R., Udit S., Batzer M.A., Feschotte C. Birth of a chimeric primate gene by capture of the transposase gene from a mobile element // Proc. Natl. Acad. Sci. USA. 2006. V. 103. № 21. P. 8101–8106.
Couzigou J.M., Lauressergues D., Becard G., Comier J.P. miRNA-encoded peptides (miPEPs): A new tool to analyze the role of miRNAs in plant biology // RNA Biol. 2015. V. 12. P. 1178–1180.
de Duve C. Life as a cosmic imperative // Philos. Trans. A Math. Phys. Eng. Sci. 2011. V. 369. № 1936. P. 620–623.
de Souza F.S., Franchini L.F., Rubinstein M. Exaptation of transposable elements into novel cis-regulatory elements: is the evidence always strong // Mol. Biol. Evol. 2013. V. 30. № 6. P. 1239–1251.
Doolittle W.F., Sapienza C. Selfish genes, the phenotype paradigm and genome evolution // Nature. 1980. V. 284. № 5757. P. 601–603.
Dotto B.R., Carvalho E.L., da Silva A.F. et al. HTT-DB: new features and updates // Database (Oxford). 2018. V. 1. https://doi.org/10.1093/database/bax102
Duan C.G., Wang X., Xie S. et al. A pair of transposon-derived proteins function in a histone acetyltransferase complex for active DNA demethylation // Cell. Res. 2017. V. 27. № 2. P. 226–240.
Dupressoir A., Lavialle C., Heidmann T. From ancestral infectious retroviruses to bona fide cellular genes: role of the captured syncytins in placentation // Placenta. 2012. V. 33. № 9. P. 663–671.
Dupuy C., Periquet G., Serbielle C. et al. Transfer of a chromosomal Maverick to endogenous bracovirus in a parasitoid wasp // Genetiva. 2011. V. 139. № 4. P. 489–496.
Elliott T.A., Gregory T.R. Do larger genomes contain more diverse transposable elements // BMC Evol. Biol. 2015. V. 15. № 1. P. 69–81.
Elkon R., Ugalde A.P., Agami R. Alternative cleavage and polyadenylation: extent, regulation and function // Nat. Rev. Genet. 2013. V. 14. № 7. P. 496–506.
Feng G., Leem Y.E., Levin H.L. Transposon integration expression of stress response genes // Nucleic Acids Res. 2013. V. 41. № 2. P. 775–789.
Ferreri G.C., Brown J.D., Obergfell C. et al. Recent amplification of the kangaroo endogenous retrovirus, KERV, limited to the centromere // J. Virol. 2011. V. 85. № 10. P. 4761–4771.
Feschotte C. Transposable elements and the evolution of regulatory networks // Nat. Rev. Genet. 2008. V. 9. № 5. P. 397–405.
Finatto T., de Oliveira A., Chaparro C. et al. Abiotic stress and genome dynamics: specific genes and transposable elements response to iron excess in rice // Rice. 2015. V. 8. P. 13.
Fischer M.G., Suttle C.A. A virophage at the origin of large DNA transposons // Science. 2011. V. 332. P. 231–234.
Forterre P., Gala M. Giant viruses and the origin of modern eukaryotes // Curr. Opin. Microbiol. 2016. V. 31. P. 44–46.
Fujiwara H. Site-specific non-LTR retrotransposons // Microbiol. Spectr. 2015. V. 3. № 2. MDNA3-0001-2014. https://doi.org/10.1128/microbiolspec.MDNA3-0001-2014
Garavis M., Gonzalez C., Villasante A. On the origin of the eukaryotic chromosome: the role of noncanonical DNA structures in telomere evolution // Genome Biol. Evol. 2013. V. 5. P. 1142–1150.
Gilbert C., Schaack S., Pace J.K. 2nd et al. A role for host-parasite interactions in the horizontal transfer of transposons across phyla // Nature. 2010. V. 464. P. 1347–1350.
Gim J., Ha H., Ahn K. et al. Genome-Wide Identification and Classification of microRNAs derived from repetitive elements // Genomic Inform. 2014. V. 12. P. 261–267.
Grandbastien M.A. LTR retrotransposons, handy hitchhikers of plant regulation and stress response // Biochim. Biophys. Acta. 2015. V. 1849. № 4. P. 403–416.
Guo X., Su H., Shi Q. et al. De Novo Centromere Formation and Centromeric Sequence Expansion in Wheat and its Wide Hybrids // PLoS Genet. 2016. V. 12. № 4. e1005997.
Han M.J., Xu H.E., Zhang H.H. et al. Spy: a new group of eukaryotic DNA transposons without target site duplications // Genome Biol. Evol. 2014. V. 6. № 7. P. 1748–1757.
Hara T., Hirai Y., Jahan I. et al. Tandem repeat sequences evolutionarily related to SVA-type retrotransposons are expanded in the centromere region of the western hoolock gibbon, a small ape // J. Hum. Genet. 2012. V. 57. № 12. P. 760–765.
Heikkinen E., Launonen V., Muller E., Bachmann L. The pvB370 BamHI satellite DNA family of the Drosophila virilis group and its evolutionary relation to mobile dispersed genetic pDv elements // J. Mol. Evol. 1995. V. 41. № 5. P. 604–614.
Irimia M., Roy S.W. Origin of spliceosomal introns and alternative splicing // Cold. Spring Harb. Perspect. Biol. 2014. V. 6. № 6. pii: a016 071.
Ito J., Suqimoto R., Nakaoka H. et al. Systematic identification and characterization of regulatory elements derived from human endogenous retroviruses // PLoS Genet. 2017. V. 13. № 7. e1 006 883.
Jacques P.E., Jeyakani J., Bourgue G. The majority of primate-specific regulatory sequences are derived from transposable elements // PLoS Genet. 2013. V. 9. № 5. e1 003 504.
Jardim S.S., Schuch A.P., Pereira C.M., Loreto E.L. Effects of heat and UV radiation on the mobilization of transposon mariner-Mos1 // Cell. Stress Chaperones. 2015. V. 20. № 5. P. 843–851.
Jjingo D., Conley A.B., Wang J. et al. Mammalian-wide interspersed repeat (MIR)-derived enhancers and the regulation of human gene expression // Mob. DNA. 2014. V. 5. P. 5–14.
Johnson R., Guigo R. The RIDL hypothesis: transposable elements as functional domains of long noncoding RNAs // RNA. 2014. V. 20. № 7. P. 959–976.
Kapitonov V.V., Jurka J. Molecular paleontology of transposable elements from Arabidopsis thaliana // Genetica. 1999. V. 107. № 1–3. P. 27–37.
Kapusta A., Kronenberg Z., Lynch V.J. et al. Transposable elements are major contributors to the origin, diversification, and regulation of vertebrate long noncoding RNAs // PLoS Genet. 2013. V. 9. № 4. e1 003 470.
Kapusta A., Feschotte C. Volatile evolution of long noncoding RNA repertoires: mechanisms and biological implications // Trends Genet. 2014. V. 30. № 10. P. 439–452.
Kipling D., Warburton P.E. Centromeres, CENP-B and Tigger too // Trends Genet. 1997. V. 13. № 4. P. 141–145.
Kojima K.K., Matsumoto T., Fujiwara H. Eukaryotic translational coupling in UAAUG stop-start codons for the bicistronic RNA translation of the non-long terminal repeat retrotranspson SART1 // Mol. Cell. Biol. 2005. V. 25. P. 7675–7686.
Koonin E.V., Dolja V.V., Krupovic M. Origins and evolution of viruses of eukaryotes: The ultimate modularity // Virology. 2015. V. 479–489. P. 2–25.
Kopera H.C., Moldovan J.B., Morrish T.A. et al. Similarities between long interspersed element-1 (LINE-1) reverse transcriptase and telomerase // Proc. Natl. Acad. Sci. USA. 2011. V. 108. P. 20 345–20 350.
Kralovicova J., Patel A., Searle M., Vorechovsky I. The role of short RNA loops in recognition of a single-hairpin exon derived from a mammalian-wide interspersed repeat // RNA Biology. 2015. V. 12. № 1. P. 54–69.
Krupovic M., Koonin E.V. Polintons: a hotbed of eukaryotic virus, transposon and plasmid evolution // Nat. Rev. Microbiol. 2015. V. 13. № 2. P. 105–115.
Krupovic M., Koonin E.V. Self-synthesizing transposons: unexpected key players in the evolution of viruses and defense systems // Curr. Opin. Microbiol. 2016. V. 31. P. 25–33.
Kubiak M.R., Makalowska I. Protein-Coding Gene’s Retrocopies and Their Functions // Viruses. 2017. V. 9. pii: E80.
Kulski J.K., Anzai T., Inoko H. ERVK9, transposons and the evolution of MHC class I duplicons within the alpha-block of the human and chimpanzee // Cytogenet. Genome Res. 2005. V. 110. P. 181–192.
Lauressergues D., Couzigou J.M., Clemente H.S. et al. Primary transcripts of microRNAs encode regulatory peptides // Nature. 2015. V. 520. № 7545. P. 90–93.
Lei H., Vorechovsky I. Identification of splicing silencers and enhancers in sense Alus: a role for pseudoacceptors in splice site repression // Mol. Cell. Biol. 2005. V. 25. P. 6912–20.
Lescale C., Deriano L. The RAG recombinase: Beyond breaking // Mich. Ageing. Dev. 2016. № 16. P. 30 263–9. https://doi.org/10.1016/j.mad.2016.11.003
Li Y., Li C., Xia J., Jin Y. Domestication of transposable elements into microRNA genes in plants // PLoS One. 2011. V. 6. e19 212.
Li B., Choulet F., Heng Y. et al. Wheat centromeric retrotransposons: the new ones take a major role in centromeric structure // Plant J. 2013. V. 73. P. 952–965.
Lin R., Ding L., Casola C. et al. Transposase-derived transcription factors regulate light signaling in Arabidopsis // Science. 2007. V. 318. P. 1302–1305.
Llorens C., Munoz-Pomer A., Bernad L. et al. Network dynamics of eukaryotic LTR retroelements beyond phylogenetic trees // Biology Direct. 2009. V. 4. P. 41–72.
Long Y., Wang X., Youmans D.T., Cech T.R. How do lncRNAs regulate transcription // Sci. Adv. 2017. V. 3. № 9. eaao2110.
Lopez-Flores I., de la Herran R., Garrido-Ramos M.A. et al. The molecular phylogeny of oysters based on a satellite DNA related to transposons // Gene. 2004. V. 339. P. 181–188.
Lorenzetti A.P., Antonio G.Y., Paschoal A.R., Domingues D.S. Plant TE-MIR DB: a database for transposable element-related microRNAs in plant genomes // Funct. Integr. Genomics. 2016. V. 16. P. 235–242.
Lowe C.B., Haussler D. 29 mammalian genomes reveal novel exaptations of mobile elements for likely regulatory functions in the human genome // PLoS One. 2012. V. 7. № 8. e43 128.
Lu X., Sachs F., Ramsay L. et al. The retrovirus HERVH is a long noncoding RNA required for human embryonic stem cell identity // Nat. Struct. Mol. Biol. 2014. V. 21. P. 423–425.
Lv S., Pan L., Wang G. Commentary: Primary Transcripts of microRNAs Encode Regulatory Peptides // Front Plant Sci. 2016. V. 7. P. 1436.
Makarevitch I., Waters A.J., West P.T. et al. Transposable elements contribute to activation of maize genes in response to abiotic stress // PLoS Genet. 2015. V. 11. № 1. e1004915.
McClintock B. The significance of responses of the genome to challenge // Science. 1984. V. 226. № 4676. P. 792–801.
Mestrovic N., Mravinac B., Pavlek M. et al. Structural and functional liaisons between transposable elements and satellite DNAs // Chromosome Res. 2015. V. 23. P. 583–596.
Muehlbauer G.J., Bhau B.S., Syed N.H. et al. A hAT superfamily transposase recruited by the cereal grass genome // Mol. Genet. Genomics. 2006. V. 274. № 6. P. 553–63.
Nelson B.R., Makarewich C.A., Anderson D.M. et al. A peptide encoded by a transcript annotated as long noncoding RNA enhances SERCA activity in muscle // Science. 2016. V. 351. № 6270. P. 271–275.
Nishihara H., Smit A., Okada N. Functional noncoding sequences derived from SINEs in the mammalian genome // Genome Res. 2006. V. 16. № 7. P. 864–874.
Notwell J.H., Chung T., Heavner W., Bejerano G. A family of transposable elements co-opted into developmental enhancers in the mouse neocortex // Nat. Commun. 2015. V. 6. P. 6644.
Orgel L.E., Crick F.H. Selfish DNA: the ultimate parasite // Nature. 1980. V. 284. № 5757. P. 604–607.
Pastor T., Talotti G., Lewandowska M.A., Pagani F. An Alu-derived intronic splicing enhancer facilitates intronic processing and molulates aberrant splicing in ATM // Nucleic Acids Res. 2009. V. 37. P. 7258–7267.
Pastuzyn E.D., Day C.E., Kearns R.B. et al. The Neuronal Gene Arc Encodes a Repurposed Retrotransposon Gag Protein that Mediates Intercellular RNA Transfer // Cell. 2018. V. 172. № 1–2. P. 275–288.
Paul B.G., Burstein D., Castelle C.J. et al. Retroelement-guided protein diversification abounds in vast lineages of Bacteria and Archaea // Nat. Microbiol. 2017. V. 2. P. 17045.
Pavlicev M., Hiratsuka K., Swaggart K.A. et al. Detecting endogenous retrovirus-driven tissue-specific gene transcription // Genome Biol. Evol. 2015. V. 7. № 4. 1082–1097.
Peters J.E., Makarova K.S., Shmakov S., Koonin E.V. Recrutment of CRISPR-Cas systems by Tn7-like transpsosons // Proc. Natl. Acad. Sci. USA. 2017. V. 114. E7358–E7366.
Rauchfuss H. Chemical Evolution and the Origin of Life. Springer. 2008. 354 p.
Roberts A.P., Chandler M., Courvalin P. et al. Revised Nomenclature for Transposable Genetic Elements // Plasmid. 2008. V. 60. P. 167–173.
Roberts J.T., Cooper E.A., Favreau C.J. Formation from transposable element insertions and noncoding RNA mutations // Mobile Genetic Elements. 2013. V. 1. № 6. e27755.
Ruiz-Orera J., Messeguer X., Subirana J.A., Alba M.M. Long-non-coding RNAs as a source of new peptides // Elife. 2014. V. 3. e03523. https://doi.org/10.7554/eLife.03523
Samantarrai D., Dash S., Chhetri B., Mallick B. Genomic and epigenomic cross-talks in the regulatory landscape of miRNAs in breast cancer // Mol. Cancer Res. 2013. V. 11. P. 315–328.
Schillinger T., Lisfi M., Chi J. et al. Analysis of a comprehensive dataset of diversity generating retroelements generated by the program DiGReF // BMC Genomics. 2012. V. 13. P. 430.
Schmitz J., Brosius J. Exonization of transposed elements: A challenge and opportunity for evolution // Biochimie. 2011. V. 93. № 11. P. 1928–1934.
Schuler A., Bornberg-Bauer E. Evolution of Protein Domain Repeats in Metazoa // Mol. Biol. Evol. 2016. V. 33. P. 3170–3182.
Shabalina S.A., Koonin E.V. Origins and evolution of eukaryotic RNA interference // Trends Ecol. Evol. 2008. V. 23. P. 578–587.
Sharma A., Wolfgruber T.K., Presting G.G. Tandem repeats derived from centromeric retrotransposons // BMC Genomics. 2013. V. 14. P. 142.
Skalka A.M. Retroviral DNA Transposition: Themes and Variations // Microbiol. Spectr. 2014. V. 2. № 5. MDNA300 052 014.
Speiseder T., Nevels M., Dobner T. Determination of the transforming activities of adenovirus oncogenes // Methods Mol. Biol. 2014. V. 1089. P. 105–115.
Startek M.P., Nogly J., Gromadka A. et al. Inferring transposons activity chronology by TRANAcendence-TEs database and de-novo mining tool // BMC Bioinformatics. 2017. V. 18. P. 422.
Tajnik M., Vigilante A., Braun S. et al. Inergenic Alu exonisation facilitates the evolution of tissue-specific transcript ends // Nucleic Acids Res. 2015. V. 43. № 21. P. 10492–10505.
Tarocchi M., Polvani S., Marroncini G., Galli A. Molecular mechanism of hepatitis B virus-induced hepatocarcinogenesis // World. J. Gastroenterol. 2014. V. 20. P. 11630–11640.
Todeschini A.L., Morillon A., Springer M., Lesage P. Severe adenine starvation activates Ty1 transcription and retrotransposition in Saccharomyces cerevisiae // Mol. Cell. Biol. 2005. V. 25. P. 7459–7472.
Toro N., Nisa-Martinez R. Comprehensive phylogenetic analysis of bacterial reverse transcriptases // PLoS One. 2014. V. 9. e114083.
Unniyampurath U., Pilankatta R., Krishnan M.N. RNA interference in the age of CRISPR: wil CRISPR interfere with RNAi // Int. J. Mol. Sci. 2016. V. 17. № 3. P. 291.
Wang S., Zhang L., Meyer E., Matz M.V. Characterization of a group of MITEs with unusual features from two coral genomes // PLoS ONE. 2010. V. 5. № 5. e10700.
Wang D., Su Y., Wang X. et al. Transposon-derived and satellite-derived repetitive sequences play distinct functional roles in Mammalian intron size // Evol. Bioinform. Online. 2012. V. 8. P. 301–319.
Wayengera M. On the general theory of the origins of retroviruses // Theoretical Biology and Medical Modeling. 2010. V. 7. P. 5–18.
Westermarck J., Ivaska J., Corthals G.L. Identification of protein interactions involved in cellular signaling // Molecular and cellular proteomics. 2013. V. 12. № 7. P. 1752–1763.
Wicker T., Sabot F., Hua-Van A. et al. A unified classification system for eukaryotic transposable elements // Nat. Rev. Genet. 2007. V. 8. P. 973–982.
Wolfgruber T.K., Sharma A., Schneider K.L. et al. Maize centromere structure and evolution: Sequence analysis of centromeres 2 and 5 reveals dynamic loci shaped primarily by retrotransposons // PLoS Genet. 2009. V. 5. P. 13–16.
Wong L.H., Choo K.H. Evolutionary dynamics of transposable elements at the centromere // Trends. Genet. 2004. V. 20. P. 611–616.
Xie C., Zhang Y.E., Chen J.Y. et al. Hominoid-specific de novo protein-coding genes originating from long non-coding RNAs // PLoS Genet. 2012. V. 8: e1 002 942.
Xu C., Tian J.M.B. SiRNA-mediated DNA methylation and H3K9 dimethylation in plants // Protein Cell. 2013. V. 4. P. 656–663.
Yuan Z., Sun X., Liu H., Xie J. MicroRNA genes derived from repetitive elements and expanded by segmental duplication events in mammalian genomes // PLoS One. 2011. V. 6. Iss. 3. e17666.
Yutin N., Shevchenko S., Kapitonov V. et al. A novel group of diverse Polinton-like viruses discovered by metagenome analysis // BMC Biol. 2015. V. 13. P. 95.
Zdobnov E.M., Campillos M., Harrington E.D. et al. Protein coding potential of retroviruses and other transposable elements in vertebrate genomes // Nucleic Acids Res. 2005. V. 33. P. 946–954.
Zhang J., Mujahid H., Hou Y. et al. Plant Long ncRNAs: A New Frontier for Gene Regulatory Control // American J. Plant Sciences. 2013. V. 4. № 5. ID:32139.
Zhang G., Esteve P., Chin H.G. et al. Small RNA-mediated DNA (cytosine-5) methyltransferase 1 inhibition leads to aberrant DNA methylation // Nucleic Acids Research. 2015. V. 43. № 12. P. 6112–6124.
Zhu Z., Tan S., Zhang Y., Zhang Y.E. LINE-1-like retrotransposons contribute to RNA-based gene duplication in dicots // Sci. Rep. 2016. V. 6. P. 24755.
Zhuo X., Feschotte C. Cross-Species Transmission and Differential Fate of an Endogenous Retrovirus in Three Mammal Lineages // PLoS Pathg. 2015. V. 11. e1 005 279.
Дополнительные материалы отсутствуют.
Инструменты
Успехи физиологических наук