Вестник Военного инновационного технополиса «ЭРА», 2022, T. 3, № 2, стр. 145-150
ДНК-МИКРОЧИПЫ И ИХ ПРИМЕНЕНИЕ В МЕДИЦИНЕ И БИОЛОГИИ
А. В. Морозов 1, А. Н. Молодченко 1, *, А. Д. Рябчикова 1, И. А. Чуркин 2
1 Военный инновационный технополис “ЭРА”
Анапа, Россия
2 48 Центральный научно-исследовательский институт
Сергиев-Посад, Россия
* E-mail: era_1@mil.ru
Поступила в редакцию 09.03.2022
После доработки 19.03.2022
Принята к публикации 19.03.2022
Аннотация
Технология ДНК-микрочипов все больше находит свое применение в генетических исследованиях и идентификации патогенных микроорганизмов. Представлен обзор основных технологий конструирования ДНК-микрочипов, способов иммобилизации молекул нуклеиновых кислот на их поверхности и применения чипов в биомедицинских исследованиях.
ОГЛАВЛЕНИЕ
Введение
1. Типы микрочипов
1.1. С иммобилизованными зондами
1.2. In situ-синтезированные микрочипы
1.3. Самосборные чипы
1.4. Технология гидрогелевых микрочипов
1.5. Способы мечения олигонуклеотидов
2. Применение биочипов
2.1. Анализ экспрессии генов
2.2. Обнаружение патогенов
2.3. Генотипирование
Заключение
ВВЕДЕНИЕ
Одними из важнейших веществ во всех живых организмах являются нуклеиновые кислоты, обеспечивающие хранение и передачу генетической информации. В связи с этим крайне важным является определение состава и содержания нуклеиновых кислот в биологических образцах. В настоящее время для детектирования целевых нуклеиновых кислот широко применяются ДНК-зонды. ДНК-зонд (англ. DNA probe) – одноцепочечный фрагмент ДНК, способный к гибридизации со специфичным участком молекулы исследуемой ДНК. Принцип гибридизации основан на ассоциации цепочек ДНК между собой при комплементарности их нуклеотидных последовательностей, что позволяет идентифицировать нужную последовательность. Для идентификации проводят мечение зонда или анализируемых молекул. Для этого применяют радиоактивные изотопы, хромофоры (флуоресцентное мечение), или группы, делающие возможным детектирование в ходе последующей ферментативной реакции (например, биотиновое мечение). ДНК-зонды применяют в таких методах, как нозерн-блот, дот-блот, саузерн-блот, а также в ДНК-микрочипах [1].
ДНК-микрочипы (микрочипы, биочипы) – это технология, в которой 1000 ДНК-зондов иммобилизованы на поверхности и используются для измерения относительной концентрации последовательностей нуклеиновых кислот в смеси путем гибридизации и последующего обнаружения объектов гибридизации.
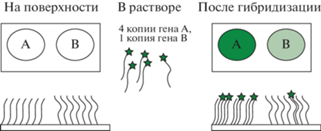
ДНК-микрочипы представляют собой группу технологий, в которых определенные последовательности ДНК либо вводятся, либо синтезируются на поверхности таким образом, что ДНК ковалентно или нековалентно прикрепляется к ней. На рис. 1 показана упрощенная схема микрочипа, где прямоугольниками обозначена подложка с двумя ячейками, на которых иммобилизованы ДНК-зонды двух типов (последовательности “А” и “В”) до и после гибридизации.
В основе диагностики с помощью микрочипов лежит принцип установления фактом лиганд-рецепторного взаимодействия. Один из взаимодействующих партеров – лиганд или рецептор – иммобилизуется на небольшой, но строго определенной поверхности микрочипа, которую называют спот (поверхность микрочипа в ряде случаев может быть структурирована, например, микроячейками (нанопланшетами) или микроэлектродами).
Обычно ДНК-чипы используются для зондирования раствора смеси меченых нуклеиновых кислот, а связывание (путем гибридизации) этих “мишеней” с “зондами” на матрице используется для измерения относительных концентраций нуклеиновой кислоты в растворе. Существуют и другие способы количественного определения различных последовательностей нуклеиновых кислот в образце, включая количественную ПЦР, “цифровую ПЦР” и гибридизацию с оптически маркированными “последовательностями зондов”.
Бурное развитие технологии ДНК-матриц началось в конце 90-х–начале 2000-х годов, когда были адаптированы как новые методы производства, так и флуоресцентное обнаружение. Отметим, что за это время произошел постепенный переход к производству чипов с использованием небольших олигонуклеотидных последовательностей с 25–60 парами нуклеотидов. Переход к олигонуклеотидным зондам стал возможен благодаря увеличению количества общедоступной информации о последовательности ДНК. Использование олигонуклеотидов (в отличие от более длинных последовательностей) привело к увеличению специфичности гибридизации ДНК-зонда с целевой последовательностью (ДНК-мишенью), так как олигонуклеотиды могли быть сконструированы таким образом, чтобы связываться с участками генов, которые больше всего отличались бы от других участков ДНК. Рассмотрим четыре основных типа микрочипов: с иммобилизованными зондами, синтезированные in situ, самосборные и гидрогелевые биочипы [2].
1. ТИПЫ МИКРОЧИПОВ
1.1. С иммобилизованными зондами
Иммобилизация ДНК-зондов выдвигает ряд требований к выбору материала подложки, способам иммобилизации олигонуклеотидов, их химической модификации. Выделим наиболее предпочтительные [3]:
− высокая функциональность поверхности подложки;
− ковалентные связи между ДНК и поверхностью;
− доступность иммобилизованного ДНК для взаимодействующих с ней молекул;
− низкий уровень неспецифических взаимодействий.
Прикрепление олигонуклеотидов осуществляется различными способами. Наиболее распространенным является непосредственное нанесение раствора модифицированного олигонуклеотида на подготовленную поверхность подложки. В результате протекающей реакции конденсации полинуклеотидная цепь оказывается зафиксированной на ней.
В качестве подложек для иммобилизации могут выступать различные материалы: кремний, стекло, золото, полимеры. Наиболее часто используют стекло в силу ряда его характерных свойств [3]:
− нерастворимость в органических и неорганических растворителях;
− относительная химическая инертность;
− низкая фоновая флуоресценция;
− отсутствие неспецифических взаимодействий;
− достаточная прочность;
− доступность и простота в обращении.
В 1996 г. впервые опубликован метод, позволяющий изготавливать чипы ДНК с высокой плотностью размещения на стеклянных подложках. С помощью сверхточных инструментов раствор ДНК наносят на стекла размером 1 × 3 дюйма. Обычно поверхность стекла покрывают специальным веществом, например полилизином, что помогает сохранить ДНК на подложке. Споты на стекле позволяют флуоресцентно маркировать образец. Флуоресцентное детектирование обеспечивает несколько преимуществ по сравнению с радиоактивными или хемилюминесцентными метками. Во-первых, флуоресцентное детектирование довольно чувствительно и имеет большой динамический диапазон. Во-вторых, флуоресцентная маркировка менее дорогая и более простая, чем радиоактивная или хемилюминесцентная. В-третьих, флуоресцентная маркировка позволяет помечать два (или более) образца разными цветами.
1.2. In situ-синтезированные микрочипы
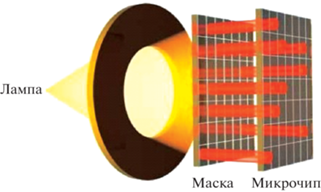
Данный метод основан на адресном синтезе олигонуклеотидов на поверхности чипа. Синтез проводят путем поэтапного добавления к растущей цепочке нуклеотидов, содержащих на 5´-конце лабильную защитную группу, снятие которой возможно под действием светового излучения, электрического напряжения или в ходе кислотного гидролиза [3]. В варианте, предложенном фирмой Affimetrix, олигонуклеотиды синтезируются непосредственно на подложке методом фотолитографии с использованием специальных микромасок, заимствованных из производства микросхем. Применение данного метода позволило добиться поразительных результатов: на одном чипе фирмы Affimetrix могут находиться миллионы различных спотов (участков, содержащих одинаковые молекулы-зонды) размером всего лишь несколько микрон [3]. На рис. 2 представлена последовательность этапов синтеза олигонуклеотидных ДНК-зондов с применением литографической маски.
Рис. 2.
Последовательность этапов синтеза олигонуклеотидных ДНК-зондов с применением литографической маски [13].
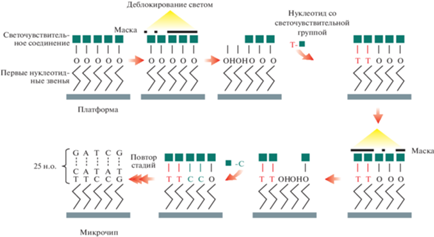
Согласно рис. 2 исходная стеклянная платформа покрыта слоем светочувствительного соединения, предотвращающего ее взаимодействие с первым нуклеотидным звеном, ковалентно связанным с платформой. С помощью литографической маски (рис. 3) свет направляется на участки микрочипа, с которыми должен связываться нуклеотид. После разрушения защитного слоя микрочип обрабатывают реакционноспособным производным нуклеотида, который связывается с поверхностью микрочипа в заранее запланированных местах. Нуклеотид также несет в своем составе светочувствительную группу, что позволяет повторять цикл наращивания нуклеотидной цепи. В результате получают микрочип, содержащий олигозонды определенной длины [3].
Важным преимуществом технологии Affymetrix является то, что, поскольку последовательности ДНК непосредственно синтезируются на поверхности, требуется только небольшая коллекция реагентов (четыре модифицированных нуклеотида плюс небольшая часть реагентов, необходимых для этапов деблокирования и связывания) для построения произвольно сложного массива зондов. Однако исходная технология Affymetrix была ограниченной в универсальности, поскольку каждая модель массива олигонуклеотидов требовала построения уникального набора фотолитографических масок, чтобы направлять свет на массив на каждом этапе процесса синтеза. В 2002 г. был опубликован метод [3] (NimblegenSystemsInc.), в котором стадия фотодепротекции выполняется с использованием микрозеркал (аналогичных тем, что находятся в видеопроекторах) для прямого освещения пикселях на матрице [4]. Это позволяет создавать произвольные массивы в небольших объемах с гораздо меньшими затратами, чем при использовании фотолитографических методов с использованием масок для прямого света. Одним из ограничений этого метода является то, что общее количество адресных пикселей (например, блок олигонуклеотидов, который может быть синтезирован) лимитировано количеством адресных позиций в устройстве микрозеркала.
1.3. Самосборные чипы
Альтернативный подход к построению массивов зондов был создан группой Дэвида Уолта в Университете Тафтса [5–7] и лицензирован для компании Illumina. Их способ включал в себя синтез ДНК на небольших шариках из полистирола и осаждение этих гранул на конце волоконно-оптической решетки, в которой были вытравлены концы волокон, чтобы образовать углубление размером немного больше одного шарика. Различные типы ДНК будут синтезированы на разных шариках, и нанесение этих шариков на волоконно-оптический кабель приведет к случайной сборке массива олигонуклеотидов. В ранних версиях этих массивов гранулы оптически кодировались различными комбинациями флуорофора, чтобы можно было определить, какой олигонуклеотид в каком положении находился на матрице (“декодирование матрицы”) [5–7]. Оптическое декодирование флуоресцентной маркировкой ограничило общее количество уникальных шариков, которые можно было различить. Таким образом, более поздние и современные способы декодирования гранул включают в себя гибридизацию и обнаружение ряда коротких флуоресцентно меченных олигонуклеотидов в последовательной серии этапов. Это не только позволяет использовать огромное количество различных типов шариков в одном чипе, но и функционально тестирует чип до его использования в биологическом анализе. В более поздних версиях чипов Illumina использовалась прозрачная стеклянная поверхность для размещения гранул вместо волоконно-оптической решетки.
1.4. Технология гидрогелевых микрочипов
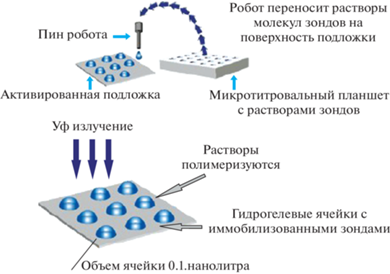
Основным этапом диагностирования на ДНК‑чипах является гибридизация. От того как будет происходить гибридизация, зависят результаты исследований. Часто низкая эффективность и специфичность гибридизации объясняются взаимодействием анализируемой последовательности ДНК и иммобилизованного зонда в тонком слое поверхности чипа. Так как объем данного слоя в сравнении с остальным объемом ячейки очень мал, то всего лишь небольшая часть молекул анализируемой ДНК оказывается в ней каждый момент времени. Для увеличения поверхности взаимодействия иммобилизованного зонда с исследуемым образцом и повышения эффективности гибридизации применяют технологию гидрогелевых чипов. Согласно данной технологии ДНК-зонды иммобилизуют в полиакриламидном геле. Для этого растворы зондов смешивают с гелеобразующими мономерами, а затем наносят их с помощью игольчатых растров (пинов) механического робота на модифицированную пластиковую или стеклянную подложку биочипа. После чего проводят совместную полимеризацию зондов с компонентами геля под действием УФ-излучения, в результате чего иммобилизованные молекулы ковалентно присоединяются к мономерам растущей полимерной цепи и равномерно распределяются во всем объеме каждой гелевой ячейки (рис. 4). Данная технология была изобретена в Институте молекулярной биологии им. Энгельгардта РАН (Москва).
1.5. Способы мечения олигонуклеотидов
Для установления факта гибридизации проводят мечение зондов или анализируемых молекул. Применяют радиоактивные и нерадиоактивные метки.
В качестве радиоактивной метки чаще всего используют фосфор-32 (32P), также применяются сера-35 (35S) и тритий (3H). Детекция радиоактивно меченых молекул осуществляется посредством радиографии. Главным преимуществом радиоактивных меток является высокая чувствительность (10–19 М). Несмотря на это, использование радиоактивно меченых зондов ограничено в связи с возможностью радиолиза как самого зонда, так и мишени коротким (8–14 дней) временем полураспада большинства радионуклидов, а также связанной с ними опасностью для здоровья персонала. Также необходимо учитывать психологический и экологический аспекты, возникающие при работе с радионуклидами и при утилизации отходов. Поэтому в настоящее время особое внимание уделяется нерадиоактивному мечению.
В случае нерадиоактивного мечения зонд соединяют с какой-либо репортерной группой: флуорохромом, гаптеном, с которым связывается меченое антитело, ферментом, дающим окрашенный или люминесцирующий продукт. Распространение получили три основные способа мечения: удлинение затравки, ник-трансляция или мечение с помощью ПЦР. Обнаружение сигнала о связывании осуществляется под люминесцентным микроскопом.
2. ПРИМЕНЕНИЕ БИОЧИПОВ
2.1. Анализ экспрессии генов
Процессами внутреннего метаболизма клеток, их делением и дифференцировкой управляет сложная сеть молекулярных взаимодействий, которая определяет уровень экспрессии (функционирования) различных генов. Измерение уровня экспрессии генов является преобладающим применением ДНК-микрочипов. Показателем последней служит концентрация соответствующей матричной РНК (мРНК), которая синтезируется на ДНК – носителе наследственной информации. Динамика изменения концентрации определенной мРНК указывает на включение или выключение конкретного гена, а значительные отклонения этого показателя от нормы могут свидетельствовать о наличии различных заболеваний. Технология микрочипов ДНК позволяет одновременно отслеживать экспрессию десятков тысяч генов, создавая таким образом своеобразный молекулярный “портрет” клетки. Изучая профили генной экспрессии в клетке, можно ставить диагноз, определять группу риска и давать прогноз развития многих видов раковых заболеваний.
Процедура подготовки образца для анализа на микрочипах включает в себя выделение суммарной РНК, на основе которой посредством реакции обратной транскрипции синтезируют цепи кодирующей ДНК (кДНК). Из каждой молекулы такой кДНК с помощью фермента Т7 РНК-полимеразы можно получить множество копий РНК. Как правило, образующиеся молекулы РНК при этом одновременно получают флуоресцентную метку (для этого при синтезе используются меченые нуклеотиды). В работе с олигонуклеотидными ДНК-зондами для мечения образца получаемой РНК часто употребляют флуоресцентную метку одного типа, а уровни экспрессии генов определяют, сравнивая получаемые флуоресцентные сигналы с сигналами внутренних контрольных точек микрочипа.
При работе с микрочипами на основе кДНК, как правило, необходимы две метки: контрольный образец (взятый из нормальных клеток) метят одним флуоресцентным красителем, исследуемый образец (взятый, например, из раковой опухоли) – другим; далее их смешивают и гибридизуют с одним микрочипом. По соотношению сигналов от двух разных меток в каждой ячейке микрочипа судят о повышении или понижении уровня экспрессии данного гена при заболевании.
2.2. Обнаружение патогенов
Еще одна область применения технологии биочипов – обнаружение мутаций в геноме патогенных агентов, что позволяет безошибочно определить вид вируса или бактерии. Для этого протяженные фрагменты геномной ДНК или РНК патогена гибридизируют с микрочипом, содержащим специальные олигонуклеотидные зонды.
В [7] представлен микрочип, способный диагностировать опасные для человека виды ортопоксвирусов – возбудителей оспы. Кроме того, данный микрочип способен отличить зараженных ортопоксвирусами от тех, кто поражен вирусами герпеса. Это крайне важно, поскольку клиническая картина больных ортопоксвирусами очень сходна с клинической картиной больных, инфицированных вирусами герпеса 1, 2 и 3 типов, что позволяет правильно выбрать способ лечения.
Эффективно применяются биологические ДНК-микрочипы для этиологической верификации острых кишечных инфекций бактериальной природы [8]. Удалось выявить и идентифицировать ДНК патогенных и условно-патогенных возбудителей острых кишечных инфекций: Salmonella spp., Shigella spp. + EIEC, Campylobacter jejuni, Proteus mirabilis, Klebsiella pneumoniae [8].
Разработан микрочип, позволяющий определять 15 вариантов гемагглютинина (Н1–Н15) и два варианта нейраминидазы (N1, N2) вируса гриппа А. Процедура идентификации вирусного подтипа не требует выполнения предварительных стадий культивирования вируса, а результаты анализа могут быть получены в течение 10 ч. Показано, что предложенный подход обладает высокой специфичностью и может использоваться для разработки ДНК микрочипов, нацеленных на генотипирование вирусов, характеризующихся высокой изменчивостью.
2.3. Генотипирование
ДНК-микрочипы широко используются для выявления однонуклеотидных полиморфизмов (single-nucleotide-polymorphism, SNP). Для обнаружения SNP использовалось несколько альтернативных подходов.
Первый подход представляет собой аллельную дискриминацию путем гибридизации. Олигонуклеотиды, которые являются комплементарными к каждой аллели, помещаются на подложку, а меченые геномные ДНК гибридизуются на микрочипе. Необходимо, чтобы все варианты SNP находились в центре олигонуклеотида (обычно 25 последовательностей нуклеотидов на чипах Affymetrix), поскольку это положение оказывает наибольшее влияние на гибридизацию.
Второй метод называется “GoldenGateAssay”. В его основе лежит полимеразная цепная реакция. В раствор геномной ДНК помещаются молекулы, комплементарные геномной ДНК и на 3'‑конце содержащие различные модификации SNP, а на 5'-конце – различные праймеры для последующего проведения ПЦР, кроме того, добавляется комплементарная (другой цепи геномной ДНК) молекула с другой стороны от SNP, содержащая на 5'-конце еще один праймер для ПЦР. Полимераза будет осуществлять синтез только с того праймера, 3'-конец которого соответствует SNP. В зависимости от того, с какого праймера с видоизмененным 3'-концом будет идти ПЦР, такой SNP и наблюдается в образце.
ЗАКЛЮЧЕНИЕ
Возможности технологии ДНК-биочипов чрезвычайно велики. Проведение многопараметрического анализа биологического образца позволяет охватить многие области современной медицины. В настоящее время исследователи пытаются интегрировать в единую схему все стадии проведения анализа, включая обработку биологического образца, выделение нуклеиновых кислот, амплификацию специфических мишеней непосредственно на биочипе с количественной идентификацией в режиме реального времени. Подобные “лаборатории на чипе” дадут возможность повысить качество лабораторной диагностики, уменьшить вероятность заражения медперсонала потенциальным инфекционным материалом и снизить количество ложноположительных и ложноотрицательных результатов.
Список литературы
Савченко П.А. Дисс. “Особенности распространения вирусов гриппа А на территории юга Центральной Сибири” канд. биол. наук. Сибирский федеральный университет, Красноярск. 2017.
Bolón-Canedo V. et al. Challenges and future trends for microarray analysis // Microarray bioinformatics. New York: Humana, 2019. P. 283.
Сойбанов В.Д. и др. Патент № 2715333 C1, РФ. Набор реагентов для выявления и идентификации ДНК возбудителей бруцеллеза методом полимеразной цепной реакции в реальном времени “ом-скрин-бруцеллез-рв”: заявл. 12.11.2018, опубл. 26.02.2020.
Pal A. DNA Microarray // Protocols in Advanced Genomics and Allied Techniques. New York: Springer, 2022. P. 221.
Li Z., Askim J.R., Suslick K.S. // Chem. Rev. 2018. V. 119. № 1. P. 231.
Sun J. et al. // Biosens. Bioelectron. 2019. V. 139. P. 111.
Галета А.А. // Инновационные аспекты развития науки и техники. 2021. № 2. С. 12.
Сухарева А.С., Кулуев Б.Р. // Биомика. 2018. Т. 10. № 1. С. 69.
Daems D. et al. // ACS Appl. Mat. Inter. 2018. V. 10. № 28. P. 23539.
Lietard J. et al. // Nucleic Acids Res. 2021.
Organick L. et al. // Nature Biotechnol. 2018. V. 36. № 3. P. 242.
Szymczak L.C., Kuo H.Y., Mrksich M. // Anal. Chem. 2018. V. 90. № 1. P. 266.
Грядунов Д.А. Дисс. “Гидрогелевые биочипы – инструменты многопараметрического анализа маркеров бактериальных, вирусных и растительных геномов” канд. биол. наук. ФНИЦ эпидемиологии и микробиологии им. почетного акад. Н.Ф. Гамалеи, Москва. 2018.
Сахин У. и др. Патент № 2644686 C2, РФ. Идентификация опухолеассоциированных антигенов для диагностики и терапии: заявл. 26.03.2013, опубл. 13.02.2018.
Грядунов Д.А., Заседателев А.С. // Наука из первых рук. 2017. № 4. С. 50.
Дополнительные материалы отсутствуют.
Инструменты
Вестник Военного инновационного технополиса «ЭРА»