Зоологический журнал, 2019, T. 98, № 12, стр. 1432-1442
Изменчивость контрольного региона мтДНК у гусей Anser fabalis Latham 1787 на Дальнем востоке России
Д. В. Волковский a, *, П. В. Фисенко b, **, Ю. Н. Журавлев a
a Федеральный научный центр биоразнообразия наземной биоты Восточной Азии ДВО РАН
690022 Владивосток, Россия
b Федеральный научный центр агробиотехнологий Дальнего Востока
им. А.К. Чайки ДВО РАН
692539 Приморский край, п. Тимирязевский, Россия
* E-mail: volkovskydv@mail.ru
** E-mail: phisenko@bk.ru
Поступила в редакцию 26.12.2018
После доработки 30.04.2019
Принята к публикации 19.06.2019
Аннотация
Изучена изменчивость нуклеотидных последовательностей первого домена контрольного региона мтДНК гуменников (Anser fabalis Latham 1787) у 65 образцов. Подтверждено устойчивое разделение между A. f. serrirostris Swinhoe 1871 и A. f. middendorffii Severtzov 1873 на филогенетическом дереве и сети гаплотипов. При филогенетическом сравнении последовательностей A. fabalis, изученных нами ранее, с последовательностями гусей из Японии, определенных до подвида, лишь одна группировалась с последовательностями A. f. middendorffii. Сделано предположение о наиболее вероятных путях весенней миграции гусей, зимующих в Японии. Обнаружено сходство некоторых гусей из Амурской области с Anser cygnoides Linnaeus 1758.
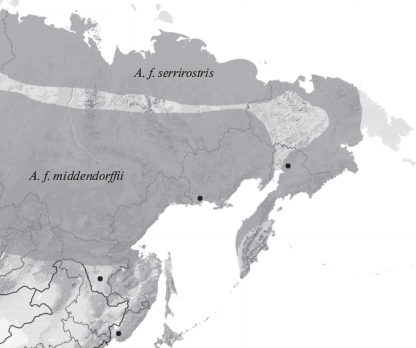
Гусь гуменник (Anser fabalis Latham 1787) распространен почти на всей территории Северо-Востока России (рис. 1), где представлен двумя подвидами: A. f. serrirostris Swinhoe 1871 – восточный тундровый гуменник и A. f. middendorffii Severtzov 1873 – таежный гуменник (Черешнев, 1996). Гуси этого вида, как и Anser albifrons Scopoli 1769, являются представителями дальневосточной фауны, объектами эволюционных исследований, возможными переносчиками заболеваний. Кроме того, вид имеет хозяйственную ценность. За последние 20 лет было проведено много генетических исследований рода Anser, в том числе и интересующего нас вида A. fabalis. Так, в ряде работ (Ruokonen et al., 2000; Slacka et al., 2003; Ruokonen et al., 2007; Ruokonen et al., 2008; Поярков и др., 2010; Ruokonen, Aarvak, 2011; Liu et al., 2013) изучены филогения и филогеография гусей. Определен уровень разнообразия контрольного региона мтДНК у семи видов гусеобразных рода Anser (Ruokonen et al., 2000). Изучены филогенетические отношения A. fabalis и Anser brachyrhynchus Baillon 1834 – в Палеарктике и Восточной Неарктике, обнаружены три митохондриальные клады (Ruokonen et al., 2008). Проведен поиск морфологических и генетических маркеров пяти исторических таксонов рода Anser в том числе и A. fabalis (Ruokonen, Aarvak, 2011). Cеквенирован полный митохондриальный геном A. fabalis (Liu et al., 2013). Подобные исследования позволяют определить межвидовой и внутривидовой уровень генетического разнообразия, характеризуют изучаемый вид гусей в местах сбора образцов. В работе Руоконен (Ruokonen et al., 2008) характеризуется изменчивость контрольного региона у A. fabalis на всем протяжении ареала.
Рис. 1.
Схема гнездовых ареалов подвидов A. fabalis на Дальнем Востоке [Черешнев, 1996]. Черными точками отмечены места сделанных нами выборок.
В одной из наших предыдущих работ (Волковский и др., 2013) мы исследовали генетическое разнообразие двух видов гусей: A. albifrons и A. fabalis. Было обнаружено устойчивое деление гаплотипов A. fabalis на кластеры, но нам не удалось выяснить причину этой кластеризации. В этой работе мы оцениваем изменчивость первого домена контрольного региона мтДНК гусей вида – A. fabalis, добытых охотниками в России на территории Амурской области. Сравниваем уровень изменчивости амурских гусей с изменчивостью гусей из других географических точек.
Целью данной работы является изучение изменчивости первого домена контрольного региона мтДНК у A. fabalis на Дальнем Востоке России. Сравнив последовательности контрольного региона гусей, мы определили однородность каждой выборки из разных регионов Дальнего Востока России и Японии, уровень различий между выборками. Знание о генетическом разнообразии гусей в разных регионах России позволит оценить риски для генофонда исследуемого вида и сделать заключение о том, какой из регионов является наиболее ценным для сохранения генофонда вида.
МАТЕРИАЛ И МЕТОДИКА
Секвенированные ранее (Волковский и др., 2013) 34 образца A. fabalis из Приморского, Камчатского краев и Магаданской обл., а также добытые охотниками в Амурской обл. в 2012 и 2016 годах (26 шт.) и не вошедшие в предыдущую работу четыре образца из Камчатского и один из Приморского краев послужили материалом для этого исследования (номера ГенБанка: JX649122– JX649145, KY864345–KY864365). Размер полученных фрагментов составил около 334 пн без тандемных повторов. Для получения сравнительных данных также были взяты доступные последовательности, по данным других авторов, из ГенБанка (AB551526–AB551534, EU186805– EU186825), в том числе Anser cygnoides Linnaeus 1758 из работы Пояркова с соавторами (2010) и последовательности из неопубликованной работы китайских коллег – D. D. Guo с соавторами (FJ711443–FJ711453, AY552166–AY552169).
ДНК выделяли по стандартной методике (Aljanabi, Martinez, 1997). Условия секвенирования описаны в нашей предыдущей работе (Волковский и др., 2013). Полученные последовательности выравнивались вручную в программе SeaView (Gouy et al., 2010). Уровень молекулярной изменчивости (Фst и Fst) между выборками при уровне значимости P = 0.01, а также нуклеотидное и гаплотипическое разнообразие рассчитывались в программе Arlequin (Excoffier et al., 2005). Неукорененную сеть гаплотипов строили в программе NetWork v5 методом MJ (Fluxus Technology Ltd., 2010). Филогенетический анализ нуклеотидных последовательностей методом Байеса MCMC проводили в MrBayes v3.1.2 (Ronquist, Huelsenbeck, 2003) при использовании GTR. Анализ включал 5 × 106 циклов, с отбором каждого 1000-го сгенерированного дерева. Укореняли с помощью внешней группы A. cygnoides из работы Пояркова с соавторами (2010). Генетические дистанции расчитывались в MEGA v7 (Kumar et al., 2016).
РЕЗУЛЬТАТЫ
Сравнение последовательностей мтДНК гусей вида A. fabalis, добытых в Приморском, Хабаровском, Камчатском краях, Магаданской, Томской и Амурской областях, с последовательностями гусей из Японии, по совпадающему участку мтДНК в 219 пн, дало 50 гаплотипов (табл. 1), объединяющих в себе последовательности из четырех разных исследований (Ruokonen et al., 2008; Eda et al., 2011; Волковский и др., 2013). Поскольку в исследовании Руоконен с соавторами (Ruokonen et al., 2008) не было приведено таблицы гаплотипов с указанием количества входящих в них образцов, мы считали эти гаплотипы уникальными. Таким образом, из 50 гаплотипов уникальными были 39, а оставшиеся 11 разделились на 9 малых и два крупных гаплотипа, объединяющих в себе значительное количество образцов. Из 219 сайтов вариабельными были 45 (табл. 2), уникальными 10, а парсимониально-информативными 34. Нуклеотидное разнообразие для исследованных выборок приведено в табл. 3. Гаплотипическое разнообразие выборок из Японии и Магаданской обл. (и их стандартное отклонение) равно 0.5367 (0.1154) и 0.7253 (0.104), соответственно. Наличие преобладающего количества уникальных гаплотипов в выборках из Приморского края и Амурской обл. определило более высокие значения их гаплотипического разнообразия – 0.95, а абсолютная уникальность всех гаплотипов выборки из Камчатского края дало значение, равное 1.
Таблица 1.
Список гаплотипов
| Количество образцов | Номер последовательности в ГенБанке | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| А | К | М | П | Т | Х | Ч | Ям | Я | ||
| S1 | 1 | 7 | 2 | 2 | 3 | JX649122-123, AB551532-533, EU186810-811, EU186822 | ||||
| S2 | 3 | JX649124 | ||||||||
| S3 | 2 | JX649125 | ||||||||
| S4 | 1 | JX649126 | ||||||||
| S5 | 1 | JX649127 | ||||||||
| S6 | 1 | JX649128 | ||||||||
| S7 | 1 | JX649129 | ||||||||
| S8 | 1 | JX649130 | ||||||||
| S9 | 1 | JX649131 | ||||||||
| S10 | 1 | JX649132 | ||||||||
| S11 | 1 | JX649134 | ||||||||
| S12 | 1 | JX649133 | ||||||||
| S13 | 2 | JX649135, EU186825 | ||||||||
| S14 | 1 | JX649136 | ||||||||
| S15 | 1 | JX649137 | ||||||||
| S16 | 1 | JX649138 | ||||||||
| S17 | 1 | JX649139 | ||||||||
| S18 | 1 | JX649140 | ||||||||
| S19 | 1 | JX649141 | ||||||||
| S20 | 1 | JX649142 | ||||||||
| S21 | 1 | JX649143 | ||||||||
| S22 | 1 | JX649144 | ||||||||
| S23 | 1 | JX649145 | ||||||||
| S24 | 1 | 1 | 1 | KY864345, AB551534 | ||||||
| S25 | 1 | KY864346 | ||||||||
| S26 | 1 | KY864347 | ||||||||
| S27 | 1 | KY864348 | ||||||||
| S28 | 1 | KY864351 | ||||||||
| S29 | 1 | KY864353 | ||||||||
| S30 | 3 | KY864354, KY864364, AY552167* | ||||||||
| S31 | 1 | KY864355 | ||||||||
| S32 | 1 | 1 | KY864356, EU186824 | |||||||
| S33 | 1 | KY864357 | ||||||||
| S34 | 1 | KY864358 | ||||||||
| S35 | 1 | KY864359 | ||||||||
| S36 | 1 | KY864360 | ||||||||
| S37 | 1 | KY864361 | ||||||||
| S38 | 1 | KY864362 | ||||||||
| S39 | 1 | KY864363 | ||||||||
| S40 | 1 | KY864365 | ||||||||
| S41 | 1 | EU186823 | ||||||||
| S42 | 1 | KY864352 | ||||||||
| M1 | 5 | 2 | 1 | 17 | KY864349, AB551526, AB551527, EU186805, EU186813 | |||||
| M2 | 3 | 1 | KY864350, EU186818 | |||||||
| M3 | 1 | 1 | AB551528, EU186815 | |||||||
| M4 | 2 | 1 | AB551529, EU186806, EU186817 | |||||||
| M5 | 1 | AB551530 | ||||||||
| M6 | 1 | AB551531 | ||||||||
| M7 | 1 | EU186814 | ||||||||
| M8 | 1 | EU186816 | ||||||||
| C1 | AY552169* | |||||||||
| C2 | AY552168* | |||||||||
| C3 | AY552166* | |||||||||
| C4 | 15 | FJ711443, FJ711445 | ||||||||
| C5 | 22 | FJ711444, FJ711447, FJ711448 | ||||||||
| C6 | 1 | FJ711446 | ||||||||
| C7 | 2 | FJ711449, FJ711450 | ||||||||
| C8 | 7 | FJ711451, FJ711452 | ||||||||
| C9 | 1 | FJ711453 | ||||||||
Таблица 2.
Нуклеотидные замены в последовательностях мтДНК A. fabalis
| 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 1 | 2 | 2 | ||||||||||||
| 2 | 3 | 4 | 4 | 5 | 5 | 5 | 5 | 7 | 9 | 9 | 0 | 0 | 1 | 1 | 1 | 2 | 2 | 2 | 3 | 4 | 4 | 5 | 5 | 5 | 5 | 6 | 6 | 6 | 6 | 6 | 7 | 7 | 7 | 7 | 7 | 8 | 8 | 8 | 9 | 9 | 9 | 9 | 0 | 1 | |
| 4 | 3 | 4 | 5 | 1 | 2 | 6 | 7 | 5 | 3 | 8 | 8 | 9 | 0 | 1 | 6 | 1 | 3 | 4 | 3 | 2 | 4 | 1 | 3 | 4 | 9 | 1 | 3 | 6 | 7 | 9 | 0 | 2 | 3 | 6 | 9 | 0 | 3 | 5 | 4 | 7 | 8 | 9 | 9 | 0 | |
| S1 | G | T | A | G | T | T | G | A | C | A | C | T | C | C | G | G | T | A | A | C | C | C | C | A | C | C | C | C | C | C | – | T | G | T | C | C | A | C | A | C | C | C | T | T | T |
| S2 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S3 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S4 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | C | . | . |
| S5 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | C | . | . | . | . | . | G | . | . | . | . | . | . | . | . |
| S6 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S7 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | C | . |
| S8 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | C |
| S9 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | – | . | . | . | . | . | G | . | . | . | . | . | . | . | . |
| S10 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | T | . | . | . | . | T | . | . | . | C | . | . | . | . | . | G | T | . | . | . | . | . | . | . |
| S11 | A | . | . | . | . | . | A | G | . | . | . | . | . | . | . | . | . | . | . | T | . | T | . | . | . | A | T | . | . | . | C | – | . | . | . | . | G | T | . | . | T | T | . | . | C |
| S12 | A | . | . | . | . | . | A | G | . | . | . | . | . | . | . | . | . | . | . | T | . | T | . | . | . | A | T | . | . | . | C | . | . | C | . | . | G | T | . | . | T | T | . | . | C |
| S13 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S14 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S15 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | C | – | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S16 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | C | . | . | C | . | . | . | . | . | . | . | T | . | . | C |
| S17 | A | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | C | – | . | C | . | . | . | . | . | . | . | T | . | . | C |
| S18 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | – | . | . | C | . | . | . | . | . | . | T | . | . | . | C |
| S19 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | C | . | . | . | . | . | G | . | . | . | . | . | . | . | C |
| S20 | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | T | . | . | . | C | – | . | . | . | . | . | . | . | . | . | T | . | . | C |
| S21 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | A | . | . | . | . | C | . | . | . | . | . | G | . | . | . | . | . | . | . | C |
| S22 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | T | . | . | . | A | . | . | . | . | C | . | . | . | . | . | . | . | . | . | . | . | . | . | C |
| S23 | A | . | . | . | . | . | A | G | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | – | . | . | C | . | . | G | . | . | . | T | T | . | . | C |
| S24 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | T | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S25 | A | . | . | A | C | . | . | G | . | . | . | . | . | . | . | . | . | . | . | . | T | T | . | . | . | . | . | . | A | . | C | . | A | C | A | T | G | . | . | . | . | T | . | . | C |
| S26 | . | . | . | . | . | . | . | . | . | G | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | T | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S27 | A | . | . | A | C | C | . | G | T | . | . | . | . | . | . | . | . | . | . | . | T | T | . | . | . | . | . | . | . | . | C | – | A | C | A | T | . | . | . | T | . | T | . | . | C |
| S28 | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | T | . | . | – | . | . | . | . | . | . | . | C | . | T | . | . | . | . |
| S29 | . | . | . | . | . | . | . | . | . | . | . | C | . | . | . | . | . | . | . | . | . | T | . | . | . | A | T | T | . | T | C | . | . | . | A | G | G | T | . | . | T | T | . | . | C |
| S30 | A | C | G | A | . | . | A | G | . | . | T | C | . | T | A | A | C | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | C | . | A | . | G | T | . | . | T | T | . | . | C |
| S31 | . | . | . | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S32 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | T | . | . | . | . | T | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S33 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | T | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S34 | . | . | . | . | . | . | . | . | . | . | . | C | . | T | A | A | C | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | . | . | . | . | G | . | . | . | . | . | . | . | . |
| S35 | . | . | . | . | . | . | . | . | . | . | T | C | . | T | A | A | C | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | . | . | . | . | G | T | . | . | . | . | . | . | C |
| S36 | A | C | . | A | . | . | . | . | . | . | . | . | . | T | A | A | . | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | . | . | . | . | . | . | . | . | T | T | . | . | C |
| S37 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | T | . | . | . | . | T | . | . | – | . | . | . | . | . | . | T | . | . | . | . | . | . | . |
| S38 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | G | . | . | T | T | . | . | . | . | T | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| S39 | A | C | G | A | . | . | A | G | . | . | T | C | . | T | A | A | C | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | . | . | A | . | . | T | . | . | T | T | . | . | C |
| S40 | A | C | G | A | . | . | . | . | . | . | T | C | . | T | A | A | C | G | G | T | . | T | . | . | . | A | T | . | . | T | C | – | C | . | A | . | G | T | . | . | T | T | . | . | C |
| S41 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | T | . | . | . |
| S42 | A | . | . | A | C | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | T | . | . | . | G | . | . | . | . | C | A | C | . | A | T | . | . | . | . | . | . | . | . | C |
| M1 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| M2 | A | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| M3 | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| M4 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | – | . | . |
| M5 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | G | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| M6 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | A | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | . | . | . | . | . |
| M7 | A | . | . | . | . | . | . | . | . | . | . | . | T | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | . | . | . | . | . | . | . | . | T | . | . | . | . |
| M8 | A | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | . | T | . | . | T | G | . | . | . | . | – | . | A | . | . | . | . | . | . | . | . | . | . | . | . |
Таблица 3.
Анализ молекулярной изменчивости между выборками A. fabalis
| Япония (25) | Камчатка (14) | Магадан (14) | Приморье (14) | Амур (26) | |
|---|---|---|---|---|---|
| Япония | 0.00658 (0.005) | 0.25251*** | 0.34106*** | 0.21017*** | 0.1373*** |
| Камчатка | 0.45121*** | 0.03497 (0.019) | 0.13736*** | 0.01099 | 0.02292 |
| Магадан | 0.61698*** | 0.32162*** | 0.00517 (0.004) | 0.08284*** | 0.13486*** |
| Приморье | 0.33107*** | 0.09994** | 0.2622*** | 0.02529 (0.015) | 0.01723 |
| Амур | 0.23816*** | 0.11899** | 0.22074*** | 0.08773* | 0.06352 (0.033) |
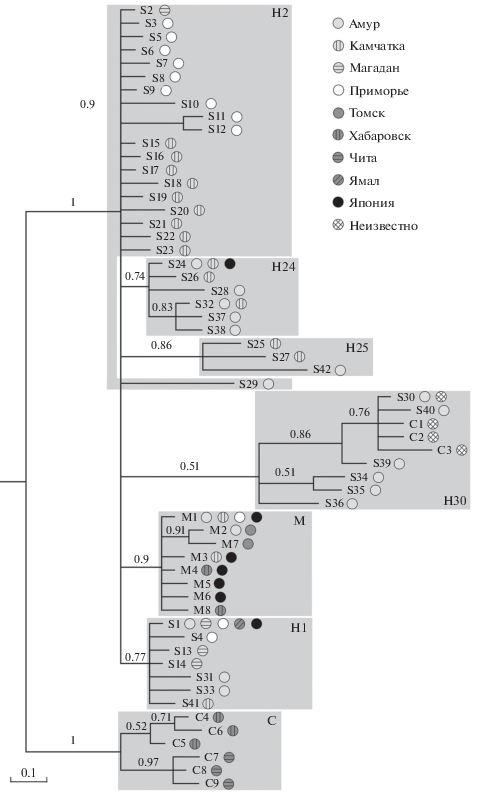
По результатам Байесова анализа (рис. 2) мы выявили гаплогруппы и ветви гаплотипов, положение которых на дереве не было определено, и присвоили каждой группе свое название. Гаплогруппа М устойчиво отделилась от других гаплотипов, отмеченных буквой H, как на Байесовом дереве, так и на деревьях, построенных другими методами (NJ, UPGMA, ME, MP, ML). В гаплогруппу M вошли последовательности гусей из исследований Руоконен и Эда (2008; 2011), определенных как A. f. middendorffii, а также часть последовательностей, полученных нами при секвенировании мтДНК образцов из Амурской обл. и Приморского края. Гаплогруппы H в основном включали последовательности, не определенные до подвида, A. fabalis из Приморского, Камчатско краев, Амурской, Магаданской областей и небольшого числа особей, определенных M. Руоконен и Эда (2008; 2011) как A. f. serrirostris. Также стоит отметить, что часть гаплотипов из Амурской обл. образовали гаплогруппу H30 с образцами A. cygnoides (Guo et al., неопубликованные данные).
Рис. 2.
Филогенетическая реконструкция гаплотипов мтДНК A. fabalis, построенная основным методом Байеса по модели GTR. В узлах ветвления указана апостериорная вероятность. Названия и условные обозначения выборок приведены в правом верхнем углу рисунка. A. f. middendorffii входят в состав гаплотипов под буквой М, A. f. serrirostris – S, а A. cygnoides – C. Названия гаплогрупп указаны в углах прямоугольников.
На неукорененной сети гаплотипов (рис. 3) гаплогруппы выделились более четко, чем на дереве. Сеть наглядно показала, что различие между гаплотипами М внутри одноименной гаплогруппы не превышает одну замену. В то же время различия между гаплотипами в других гаплогруппах составили от одной до девяти замен. На сети хорошо видно, что в гаплогруппах H больше уникальных гаплотипов, чем в M. Наглядность сети позволяет определить качественный и количественный состав каждого гаплотипа. Качественный состав не уникальных гаплотипов A. fabalis в основном был смешанным. Некоторые гаплотипы объединили в себе последовательности гусей, которые были добыты не только в разных географических точках, но и принадлежали к разным видам. Таким гаплотипом стал S30, найденный у двух особей A. fabalis из Амурской обл. и у одного гуся, последовательность которого (AY552167) получена из ГенБанка, где она присутствует под названием A. cygnoides. Генетические дистанции между гаплогруппами приведены в табл. 4.
Рис. 3.
Сеть гаплотипов A. fabalis. Размер круга пропорционален частоте встречаемости данного гаплотипа. Названия и условные обозначения выборок приведены в левом нижнем углу рисунка. Черными точками указаны нуклеотидные замены, белыми точками – гипотетические гаплотипы. A. f. middendorffii входят в состав гаплотипов под буквой М, A. f. serrirostris – S, а A. cygnoides – C. Гаплогруппы выделены многоугольниками, а названия гаплогрупп указаны в их углах.
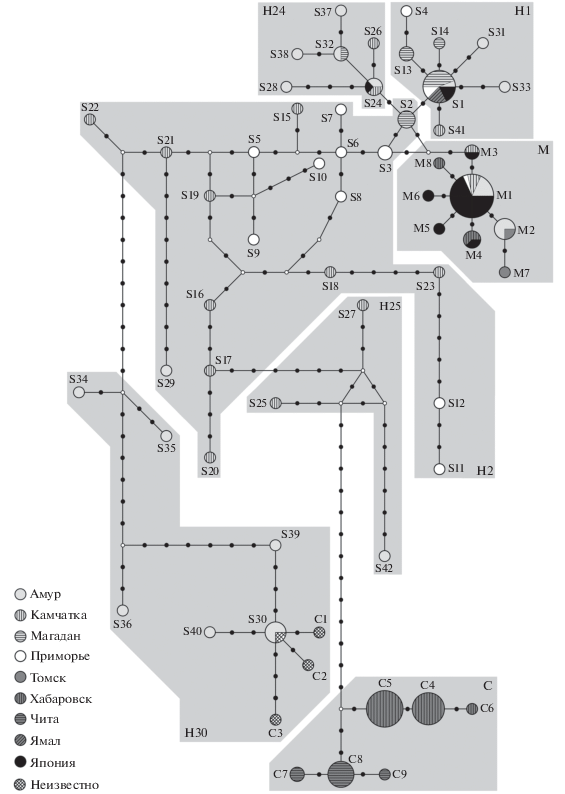
Таблица 4.
Средние значения генетических дистанций между гаплогруппами (количество образцов) A. fabalis, рассчитанные по 2-параметрической модели Кимуры
| Гаплогруппы (количество образцов) |
H1 | H2 | H24 | H25 | H30 | M | C |
|---|---|---|---|---|---|---|---|
| H1 (23) | 0.008 | 0.007 | 0.016 | 0.023 | 0.008 | 0.022 | |
| H2 (23) | 0.029 | 0.008 | 0.014 | 0.02 | 0.009 | 0.019 | |
| H24 (9) | 0.021 | 0.032 | 0.017 | 0.024 | 0.01 | 0.023 | |
| H25 (3) | 0.064 | 0.06 | 0.072 | 0.024 | 0.015 | 0.018 | |
| H30 (12) | 0.115 | 0.098 | 0.119 | 0.124 | 0.024 | 0.018 | |
| M (38) | 0.023 | 0.03 | 0.029 | 0.064 | 0.115 | 0.022 | |
| C (48) | 0.101 | 0.092 | 0.109 | 0.082 | 0.084 | 0.103 |
Для определения точного значения величины отличий выборок друг от друга был проведен анализ молекулярной изменчивости (AMOVA) (табл. 3). Индекс фиксации составил Фst = 0.23223 (Р = 0), Fst = 0.13813 (Р = 0). При парном сравнении выборок минимальное значение Фst = 0.08773 (P = = 0.02703) между выборками из Приморского края и Амурской обл., а минимальное Fst = = 0.01099 (P = 0.27027) между выборками из Приморского и Камчатского краев. Разница в значениях Фst и Fst этих же пар выборок не значительна. Уровень изменчивости между камчатской и амурской выборками немного выше Фst = 0.11899 (P = 0.00901) при Fst = 0.02292 (P = 0.07207). Максимально отличающимися друг от друга были выборки из Японии и Магаданской области, где Фst = 0.61698 (P = 0), а Fst = 0.34106 (P = 0). Наименее отличалась от гусей из Японии выборка из Амурской области, где Фst = 0.23816 (P = 0), а Fst = 0.1373 (P = 0).
ОБСУЖДЕНИЕ
Сравнив наши последовательности контрольного региона мтДНК образцов A. fabalis из Приморского и Камчатского краев, а также Магаданской и Амурской областей с последовательностями A. fabalis из ГенБанка, мы обнаружили, что часть наших образцов имеет высокий уровень сходства с образцами, определенными, как A. f. serrirostris. Исключением стали восемь образцов из Амурской обл. и один образец из Приморского края, которые имели общие гаплотипы с образцами A. f. middendorffii.
На Байесовом дереве A. f. serrirostris и A. f. middendorffii устойчиво разделились на гаплогруппы. Это говорит о большем уровне отличий между двумя подвидами, нежели у исследованных ранее A. albifrons (Волковский и др., 2016), где подвиды не группировались отдельно друг от друга.
Сеть гаплотипов так же как и Байесово дерево разделила A. f. serrirostris и A. f. middendorffii на гаплогруппы. Внутри гаплогруппы М минимальное количество замен между гаплотипами не превышало одной. Количество замен между гаплотипами в гаплогруппах H варьировало от одной до девяти. Такое уменьшение разнообразия гаплотипов М можно связать с тем, что численность A. f. middendorffii значительно снизилась в конце 20 века (Черешнев, 1996) в результате неконтролируемой охоты. Вследствие этого снизилось и генетическое разнообразие этой формы. Так, в некоторых субъектах Российской Федерации данный подвид внесен в Красные книги как подвид, находящийся под угрозой исчезновения.
Анализ молекулярной изменчивости показал (табл. 3), что гуси, зимующие в Японии, имеют наибольшее сходство среди изученных выборок с выборкой из Амурской обл. В то же время выборка гусей из Приморского края занимала второе место по уровню сходства с выборкой из Японии. Так как выборка из Приморского края с большой долей вероятности включает в себя образцы гусей, добытых в период весенней миграции с разных мест зимовок, то можно предположить, что гуси из Японии мигрируют весной через Приморский край и далее через Амурскую обл. Минимальное значение Fst между выборками из Приморского и Камчатского краев также может свидетельствовать о том, что через Приморский край часть гусей мигрирует в направлении Камчатского края.
Присутствие гаплотипов A. cygnoides у части A. fabalis из Амурской обл. может говорить о наличии гибридизации между ними. Видовую принадлежность гаплотипов к A. cygnoides (Guo et al., неопубликованные данные) можно поставить и под сомнение, так как согласно информации о них в ГенБанке, эти последовательности принадлежат выведенным породам домашних гусей Zi, Rhine, Landish и Huoyan.
Исходя из того, что последовательность, образующая один гаплотип с A. fabalis принадлежит породе Zi, а A. cygnoides, попадающие в один кластер с A. fabalis, принадлежат породам Rhine, Landish и Huoyan, можно предположить, что для выведения этих пород использовались A. fabalis, зимующие в Китае.
Таким образом, мы сравнили последовательности первого домена контрольного региона мтДНК гусей из нашего предыдущего исследования, в котором мы изучали изменчивость двух видов гусей (Волковский и др., 2013), с гаплотипами гусей, определенными до подвида. Сравнение позволило узнать, что A. fabalis, мигрирующие над территорией Приморского края, имеют более высокий уровень сходства с A. f. serrirostris и обнаруженная нами ранее кластеризация не является подвидовой. Также мы предположили, что гуси из Японии (табл. 3) мигрируют к местам гнездования через Приморский край. Гуси из Камчатского края, скорее всего, зимуют не в Японии. Мы убедились в том, что, несмотря на более обширный ареал, гуси подвида A. f. middendorffii генетически более однородны, чем A. f. serrirostris. Было обнаружено сходство части гусей из Амурской обл. с A. cygnoides. Дальнейшее изучение изменчивости гусей, добытых в Амурской обл., может дать ответы на появившиеся вопросы.
Список литературы
Волковский Д.В., Куликова И.В., Герасимов Ю.Н., Журавлев Ю.Н., 2013. Особенности генетического разнообразия Anser albifrons Scopoli, 1769 и Anser fabalis Latham, 1787 на Дальнем Востоке России // Генетика. Т. 49. № 4. С. 491–504. [Volkovsky D.V., Kulikova I.V., Gerasimov Yu.N., Zhuravlev Yu.N., 2013. Genetic diversity of Anser albifrons Scopoli, 1769 and Anser fabalis Latham, 1787 in the Russian Far East // Russian Journal of Genetics. V. 49. № 4. P. 428–440]
Волковский Д.В., Фисенко П.В., Герасимов Ю.Н., Журавлев Ю.Н., 2016. Изменчивость контрольного региона мтДНК у гусей Anser albifrons Scopoli, 1769 // Генетика. Т. 52. № 3. С. 357–363. [Volkovsky D.V., Fisenko P.V., Gerasimov Yu.N., and Zhuravlev Yu.N., 2016. Variability of the mtDNA Control Region in Goose Anser albifrons Scopoli, 1769 // Russian Journal of Genetics. V. 52. № 3. P. 314–319.]
Поярков Н.Д., Кленова А.В., Холодова М.В., 2010. Генетическое разнообразие гуся сухоноса (Anser cygnoides L.) на территории России: анализ полиморфизма контрольного региона митохондриальной ДНК // Генетика. Т. 46. № 4. С. 558–562. [Poiarkov N.D., Klenova A.V., Kholodova M.V., 2010. Genetic diversity of Swan goose (Anser cygnoides l.) in Russia: analysis of the mitochondrial DNA control region polymorphism // Russian Journal of Genetics. V. 46. № 4. P. 493–496]
Черешнев И.А., 1996. Позвоночные животные Северо-Востока России. Владивосток: Дальнаука. 308 с.
Aljanabi S.M., Martinez I., 1997. Universal and rapid salt-extraction of high quality genomic DNA for PCR-based techniques // Nucleic Acids Research. V. 25. № 22. P. 4692–4693.
Eda M., Shimada T., Mizota C., Koike H., 2011. The identification of Bean Goose Anser fabalis subspecies wintering in Japan, based on studies of the mitochondrial DNA control region: a preliminary study // Japanese Journal of Ornithology. V. 60. V. 1. P. 100–104.
Excoffier L., Laval G., Schneider S., 2005. Arlequin ver. 3.0: an integrated software package for population genetics data analysis // Evolutionary bioinformatics online. V. 1. P. 47–50.
Fluxus Technology Ltd., 2010. Network 4.6.0.0. User Guide. Online: http://www.fluxus-engineering.com/ sharenet.htm. 54 p.
Gouy M., Guindon S., Gascuel O., 2010. SeaView version 4: a multiplatform graphical user interface for sequence alignment and phylogenetic tree building // Molecular Biology and Evolution. V. 27. № 2. P. 221–224.
Kumar S., Stecher G., and Tamura K., 2016. MEGA7: Molecular Evolutionary Genetics Analysis version 7.0 for bigger datasets // Molecular Biology and Evolution. V. 33. P. 1870–1874.
Liu G., Zhou L., Zhang L. et al., 2013. The complete mitochondrial genome of bean goose (Anser fabalis) and implications for anseriformes taxonomy [PLoS One]. Online: https://doi.org/. V. 8. № 5. P. e63334.https://doi.org/10.1371/journal.pone.0063334
Ronquist F., Huelsenbeck J.P., 2003. MrBayes 3: Bayesian phylogenetic inference under mixed models // Bioinformatics. V. 19. P. 1572–1574.
Ruokonen M., Aarvak T., 2011. Typology revisited: historical taxa of the bean goose – pink-footed goose complex // Ardea. V. 99. P. 103–112.
Ruokonen M., Andersson A.-C., Tegelstrom H., 2007. Using historical captive stocks in conservation. The case of the lesser white-fronted goose // Conservation Genetics. V. 8. P. 197–207.
Ruokonen M., Kvist L., Lumme J., 2000. Close relatedness between mitochondrial DNA from seven Anser goose species // Journal of Evolutionary Biology. V. 13. P. 532–540.
Ruokonen M., Litvin K., Aarvak T., 2008. Taxonomy of the bean goose – pink-footed goose // Molecular Phylogenetics and Evolution. V. 48. P. 554–562.
Slacka K.E., Jankea A., Pennyb D., Arnasona U., 2003. Two new avian mitochondrial genomes (penguin and goose) and a summary of bird and reptile mitogenomic features // Gene. V. 302. P. 43–52.
Дополнительные материалы отсутствуют.
Инструменты
Зоологический журнал


