Успехи современной биологии, 2023, T. 143, № 1, стр. 68-76
О биологической роли ацетилирования/деацетилирования гистонов в процессе прорастания зародышей растений
Г. Х. Вафина 1, *, Е. Е. Ступак 1
1 Уфимский Институт биологии – обособленное структурное подразделение Уфимского федерального исследовательского центра Российской академии наук
Уфа, Россия
* E-mail: vafinagh@mail.ru
Поступила в редакцию 06.06.2022
После доработки 06.07.2022
Принята к публикации 08.07.2022
- EDN: HLPYRO
- DOI: 10.31857/S0042132423010106
Аннотация
Переход зародышей из состояния покоя в воздушно-сухих семенах в состояние с высокоактивным метаболизмом в процессе прорастания сопровождается значительными изменениями как пространственных, так и временных паттернов экспрессии генов и контролируется многоуровневыми регуляторными сетями. Ацетилирование гистонов – один из наиболее динамичных эпигенетических механизмов, играющий важную роль на этапах быстрых преобразований функциональной активности клеток. Характер и степень ацетилирования белков хроматина зависят от транскрипционной активности хроматина, а также связаны с репликацией ДНК и клеточным циклом. Получение в перспективе полной картины участия модификации гистонов в процессах прорастания семян будет полезно для повышения урожайности сельскохозяйственных культур, как способ оценки качества и жизнеспособности семян перед посевом, и позволит разработать методы управления реализации генетического потенциала растений.
ВВЕДЕНИЕ
Важнейшим этапом жизненного цикла растения является период прорастания, который начинается с набухания семян и заканчивается появлением корешка (Nonogaki et al., 2010; Nonogaki, 2014). Переход зародыша из состояния покоя в воздушно-сухих семенах в состояние с высокоактивным метаболизмом требует значительных изменений как пространственных, так и временных паттернов экспрессии генов и контролируется многоуровневыми регуляторными сетями (van Zanten et al., 2011; Luján-Soto, Dinkova, 2021). Одними из регуляторных механизмов, имеющих решающее значение для многих биологических процессов, являются эпигенетические модификации хроматина, включающие в себя метилирование ДНК, модификации гистонов и ремоделирование хроматина (Zhang, Ogas, 2009).
Комплекс ядерной ДНК с гистонами образует первый уровень организации хромосом. Несмотря на высокую консервативность последовательности аминокислот, гистоны могут быть представлены как гистоновыми вариантами, так и подвергаться посттрансляционным модификациям, тем самым осуществляя вклад в генную регуляцию и эпигенетический сайленсинг (Mariño-Ramírez et al., 2005; Liu et al., 2010). Нуклеосома, являющаяся основным повторяющимся звеном хроматина, состоит из восьми молекул гистонов, образующих нуклеосомный кор (по два H2A, H2B, H3 и H4), на которую в свою очередь закручена ДНК, насчитывающая 145–147 п.н. С точки зрения своего расположения в гистоновом октамере, гистоны Н3 и Н4 играют ключевую роль, оказывая влияние на структуру и динамику хроматина, а также на эффективность транскрипции (Azad et al., 2018). Гистон H1 связывается с ненуклеосомной ДНК, стабилизируя нуклеосому, тем самым способствуя компактизации хроматиновой фибриллы (Davie, 2003). ДНК связана с гистоновым кором ионными взаимодействиями между отрицательно заряженным сахарофосфатным остовом и гистонами, богатыми положительно заряженными аминокислотами аргинином (R) и лизином (K) (Davie, 2003). Комплекс достаточно динамичен и взаимодействие ДНК с белками может усиливаться или ослабляться под действием различных факторов. N-концы аминокислотных остатков гистонов, свободно выходящие на поверхность нуклеосом, подвержены многочисленным модификациям.
Одной из наиболее изученных посттрансляционных модификаций является ацетилирование определенных лизинов, расположеных в N-концевых хвостовых доменах основных гистонов. Ацетильная группа нейтрализует положительный заряд гистонов, усиливает гидрофобность, увеличвает размеры аминогруппы, что влияет на упаковку хроматина (Loidl, 1988, 1994; Hong et al., 1993; Garcia-Ramirez et al., 1995) и доступность регуляторных областей ДНК (Vettese-Dadey et al., 1996). Также ацетильная группа является сайтом связывания ряда белков, участвующих в ремоделировании хроматина, транскрипции, репликации, рекомбинации и репарации ДНК (Lee et al., 1993; Musselman et al., 2012; Gong et al., 2016; Hu et al., 2019). Например, у риса (Oryza sativa) нуклеосома потенциально имеет 38 сайтов ацетилирования лизина, из которых экспериментально доказано ацетилирование в положениях 9, 18, 23, 27, 36, 79 и 122 гистона H3 (H3K9, H3K18, H3K23, H3K27, H3K36, H3K79, H3K122) и в положениях 5, 8, 12, 16, 91 гистона H4 (H4K5, H4K8, H4K12, H4K16 и H4K91), что обеспечивает значительные возможности для контроля взаимодействия ДНК с транскрипционными факторами и регуляторными белками (Du et al., 2013; Lu et al., 2015; Mahrez et al., 2016; Xue et al., 2018).
Известно, что ацетилирование гистонов деконденсирует хроматин и активирует транскрипционную активность, а также связано с процессами репликации ДНК и клеточным циклом (Spencer, Davie, 1999; Jasencakova et al., 2000). Например, ацетилирование по сайтам H4K5, H4K8, H4K12, H4K16 и H3K9, H3K14 H3K18, H3K23 влечет деконденсирование структуры нуклеосом (Loidl, 1988, 1994; Garcia-Ramirez et al., 1995), изменяет взаимодействие гистонов с ДНК (Hong et al., 1993), облегчает доступ и связывание факторов транскрипции с генами, транскрибируемыми РНК-полимеразами II или III (Lee et al., 1993; Vettese-Dadey et al., 1996; Fuchs et al., 2006). В свою очередь, паттерн и степень ацетилирования белков хроматина зависит от транскрипционной активности хроматина, что формирует петли положительной обратной связи, поддерживающей транскрипцию (Martin et al., 2021).
Большая часть сайтов ацетилирования консервативна для разных видов растений. Так, сайт H3K36ac был обнаружен как у голосемянных, так и у многих видов покрытосемянных растений (Mahrez et al., 2016). В то же время распределение ацетилированных лизинов в гистонах H3 и H4 по генам видоспецифично. Было показано, что у Arabidopsis thaliana и Oryza sativa области хроматина с ацетилированными лизинами по сайтам H4K16ac и H3K23ac демонстрировали высокий уровень экспрессии генов, хотя гены, обогащенные H4K16ac, у этих растений связаны с различными биологическими процессами (Lu et al., 2015).
АЦЕТИЛТРАНСФЕРАЗЫ И ДЕАЦЕТИЛАЗЫ ГИСТОНОВ РАСТЕНИЙ
Ключевыми ферментами, регулирующими ацетилирование гистонов, являются гистоновые ацетилтрансферазы (HAT, histone acetyltransferase) и гистоновые деацетилазы (HDAC, histone deacetylase) (Kouzarides, 2006; Hartl et al., 2017).
HATs переносят ацетильную группу (CH3COO–), донором которой является ацетил-КоА, на ε-аминогруппу (${\text{NH}}_{3}^{ + }$) специфических остатков лизина на N-концах гистоновых белков (в основном H3 и H4) (Kumar et al., 2021). Следовательно, ацетилирование гистонов может регулироваться наличием ацетил-КоА в растительных клетках. Показано, что мутации в генах ферментов, участвующих в биосинтезе и катаболизме ацетил-КоА снижают или соответственно повышают уровни ацетилирования гистонов по всему геному (Hu et al., 2019; Xu et al., 2021).
Гистоновые ацетилтрансферазы делятся на два класса: HAT-A и HAT-B. По доменному составу HAT-A группируются в четыре семейства: 1) HAG – ацетилтрансферазы семейства GNAT (GCN5-related N-terminal acetyltransferase); 2) HAM – ацетилтрансферазы семейства MYST (MOZ, Ybf2/Sas3, Sas2 и Tip60); 3) HAC – ацетилтрансферазы, относящиеся к белкам р300/СВР (cAMP-responsive element-binding protein); 4) HAF – ацетилтрансферазы семейства ТАТА-связывающих белков (TAFII250) (Liu et al., 2016; Fina et al., 2017; Gan et al., 2021). У арабидопсиса обнаружены три белка типа GNAT: GCN5 (HAG1), ELP3 (HAG3) и HAG2; два типа MYST: HAM1 (HAG4) и HAM2 (HAG5); пять типов p300/CBP белков: HAC1, HAC2, HAC4, HAC5 и HAC12 и два типа TAF1: HAF1 и HAF2 (Pandey et al., 2002; Servet et al., 2010; Boycheva et al., 2014).
Помимо HAT-домена, гистоновые ацетилтрансферазы семейств GNAT, p300 и TAFII250 содержат бромодомен, который распознает и связывается с ацетилированными остатками лизина. Предполагают, что взаимодействие с определенными ацетилированными остатками может способствовать активации HATs, облегчая ацетилирование других мишеней (Gong et al., 2016). Показано, что ацетилтрансфераза GCN5, специфично ацетилирующая H3K14ac, повышает уровень ацетилирования H3K9ac, способствуя привлечению других HATs, осуществляющих ацетилирование H3K9 или предотвращающих активность HDACs, таких как HDA19, которые выполняют удаление этой гистоновой метки (Kim et al., 2020). Ацетилтрансферазы MYST содержат хромодомен, который может связываться с РНК (Kumar et al., 2021).
HAT-A локализуются в ядре клетки и ацетилируют коровые гистоны нуклеосом (Brownell, Allis, 1996; Roth et al., 2001). Кроме того, HAT-A действуют как коактиваторы и регуляторы транскрипции, а также способствуют взаимодействию между компонентами транскрипционного аппарата и могут поддерживать процесс транскрипции (Boycheva et al., 2014). HAT-B находятся как в ядре, так и в цитоплазме и катализируют ацетилирование вновь синтезированных гистонов перед их сборкой в нуклеосомы; обладают высокой специфичностью в отношении ацетилирования гистона H4 (Parthun et al., 1996; Kolle et al., 1998; Haigney et al., 2015).
Гистоновые деацетилазы удаляют ацетильную группу с аминогрупп, причем по крайней мере некоторые из них деацетилируют как гистоновые, так и негистоновые белки (Yruela et al., 2021). У A. thaliana описано 18 генов HDAC, у O. sativa идентифицировано так же 18 генов HDAC, у пшеницы (Triticum aestivum L.) – 49 генов HDAC, у винограда (Vitis vinifera L.) – 13 генов HDAC (Hollender, Liu, 2008; Xu et al., 2021; Martínez et al., 2021).
У растений HDACs делятся на три подсемейства: RPD3/HDA1 (Reduced Potassium Dependency 3); SIR2 (Silent Information Regulator 2) и специфичное для растений семейство HD2 (Pandey et al., 2002; Alinsug et al., 2012; Hu et al., 2019). По гомологии с дрожжевыми деацетилазами семейства дополнительно подразделяются на классы. У A. thaliana 12 деацетилаз относятся к RPD3/HDA1: из них шесть являются HDAC класса I (HDA6, HDA7, HDA9, HDA10, HDA17 и HDA19), пять – класса II (HDA5, HDA8, HDA14, HDA15 и HDA18) и один класс IV (HDA2). Две (SRT1 и SRT2) принадлежат к семейству SIR2-подобных (класс III); и четыре относятся к типу HD2 (HD-туины): HD2A, HD2B, HD2C, HD2D, по другой номенклатуре HDT 1–4, соответственно (Pandey et al., 2002; Hollender, Liu, 2008).
Активность HDACs, по-видимому, частично регулируется белок-белковыми взаимодействиями. Кроме того, HDACs регулируются экспрессией генов, субклеточной локализацией и посттрансляционными модификациями, такими как фосфорилирование, сумоилирование, протеолиз, и доступностью метаболических кофакторов (Dokmanovic, Marks, 2005). Например, HATs и HDACs могут участвовать в регуляции деградации белков и скорость деградации зависит от статуса ацетилирования белка (Sadoul et al., 2008). Было высказано предположение, что восприимчивость гистонов к протеолизу в хроматине может регулироваться добавлением или удалением ацетильных или метильных групп на лизиновых остатках вблизи мест расщепления и что посттрансляционные модификации могут регулировать скорость протеолиза (Mandal et al., 2014), тем самым действуя как нуклеосомный контроль экспрессии генов (Vafina, Ivanov, 2016; Vafina et al., 2017, 2018).
АЦЕТИЛИРОВАНИЕ/ДЕАЦЕТИЛИРОВАНИЕ ГИСТОНОВ В ПРОЦЕССЕ ПРОРАСТАНИЯ ЗАРОДЫШЕЙ РАСТЕНИЙ
Переход зародыша от воздушно-сухого состояния в состояние с высокоактивным метаболизмом является критическим этапом в жизненном цикле семенных растений (Nonogaki, 2014). Прорастание включает серию последовательных событий, начиная с поглощения воды воздушно-сухими семенами и заканчивая удлинением оси зародыша и проклевыванием корешка (рис. 1). Как известно, запуск ростовых процессов осуществляется за счет растяжения клеток. Растяжение хотя и происходит сначала относительно медленно, однако оно обеспечивает более быстрое соприкосновение корешка с почвенной влагой, а затем проникновение его в почву и продвижение за фронтом воды, чем если бы процесс роста начался с деления клеток (Данович и др., 1982). В сухих зрелых семенах клетки зародыша могут находиться в состояниях G1 и G2, однако преобладают клетки в состоянии G1, по-видимому, наименее уязвимом состоянии меристематической клетки (Данович и др., 1982).
Рис. 1.
Физиолого-морфологические и молекулярно-генетические процессы и схемы ацетилирования/деацетилирования, связанные с соответствующими стадиями прорастания зародышей растений. В квадратах обозначены названия видов растений (Bd – Brachypodium distachyon; Zm – Zea mayse; At – Arabidopsis thaliana). Гистоновые ацетилтрансферазы (НАТ) показаны серым шестиугольником, гистоновые деацетилазы (HDA) – серым овалом, корегуляторы – бесцветные овалы, сайты ацетилирования гистонов – прямоугольники. АБК – абсцизовая кислота, ГБ – гибберелины.
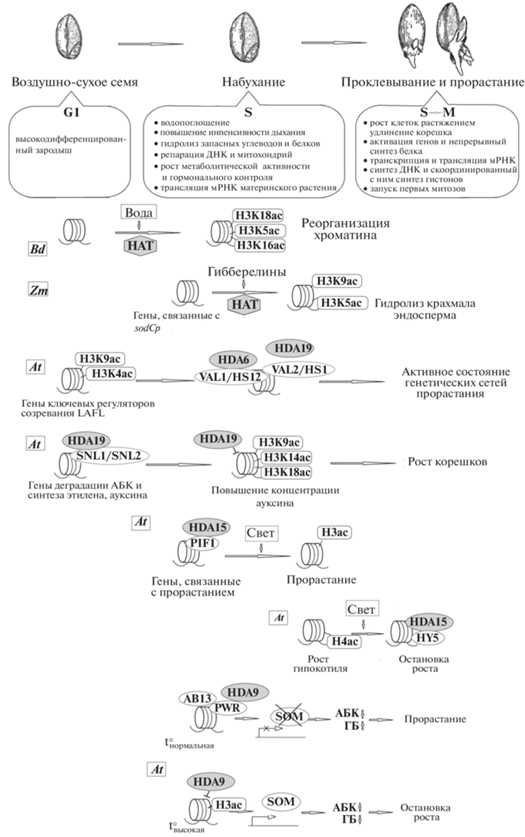
В прорастающих зародышах пшеницы зарегистрировано пять пиков митозов (Yadav, Das, 1974) сначала в корешке, потом в листочках и только после достижения корнем 1.5 см – в стеблевой меристеме (см. Данович и др., 1982). Наличие нескольких частых пиков митозов в начале прорастания объясняется, по-видимому, переходом клетки от состояния G1 к синтезу ДНК порциями, тем самым обеспечивая десинхронизацию клеточных делений, что характерно для последующего роста органа (Ivanov et al., 2002). Во время созревания семян ядра сжимаются, а хроматин становится очень компактным (van Zanten et al., 2011). Уменьшение уплотнения хроматина и увеличение размера ядер при набухании семян связаны с дисперсией гетерохроматических повторяющихся последовательностей (van Zanten et al., 2011). Наиболее сильная деконденсация хроматина наблюдается непосредственно перед прорастанием (van Zanten et al., 2011).
При прорастании происходят динамические пространственно-временные изменения уровня ацетилирования гистонов в процессе растяжения, деления и дифференцировки клеток (рис. 1). Так у Brachypodium distachyon обнаружено, что при набухании семян повышается уровень ацетилирования H3K18, H4K16 и H4K5, причем в последующем при прорастании уровень H4K16ac и H4K5ac снижается (Wolny et al., 2017). H4K16ac и H4K5ac обычно ассоциированы с репликацией ДНК, но поскольку репликация начинается на более поздних этапах прорастания, авторы предполагают, что в данном случае эти модификации отражают процесс реорганизации хроматина при переходе клетки в метаболически активное состояние (Wolny et al., 2017).
Эпигенетические механизмы способны модулировать экспрессию генов, связанных с состоянием покоя и прорастанием, путем активации или репрессии специфических подмножеств генов (Carrera-Castaño et al., 2020). При этом ацетилирование/деацетилирование гистонов оказывает как положительное, так и отрицательное влияние на состояние покоя, в зависимости от генов, регулируемых этой меткой. Например, показано, что гистондеацетилаза HDA9 принимает участие в поддержании состояния покоя в сухих семенах, деацетилируя гены, связанные с ростом проростков (Zanten et al., 2014). Рост синтеза деацетилаз HDA6 и HDA19 при прорастании обеспечивает снижение ацетилирования H3K9 и H3K4 в 5'-областях генов транскрипционных факторов LAFL (LEC1, ABI3, FUS3, LEC2), являющихся ключевыми регуляторами созревания, что запускает генетические сети, связанные с прорастанием (Tanaka et al., 2008; Luján-Soto, Dinkova, 2021). Взаимодействие гистондеацетилаз с генами обеспечивается регуляторными белками, в данном случае транскрипционными репрессорами VAL (VIVIPAROUS/ABI3-LIKE) (Zhou et al., 2013; Chhun et al., 2016; Smolikova et al., 2021). HDA6 направляется регуляторным белком VAL1, HDA19 – VAL2, по другой номенклатуре HSI2 (HIGH-LEVEL EXPRESSION OF SUGAR-INDUCIBLE GENE 2) и HSL1 (HSI2-like 1) соответственно (Chhun et al., 2016). У арабидопсиса при набухании семян в темноте регуляторный белок PIF1 (phytochrome-interacting factor) взаимодействует с HDA15, что приводит к деацетилированию гистонов H3 и, как следствие, подавлению экспрессии генов, необходимых для прорастания, в том числе генов гормональных систем (Kumar et al., 2021). На свету PIF1 расщепляется, комплекс HDA15–PIF1 сходит с хроматина, что запускает процесс прорастания семян (Kumar et al., 2021). По такому же пути происходит активация светом генов биосинтеза хлорофилла у проростков с участием другого светочувствительного фактора транскрипции PIF3 (Liu et al., 2013). Взаимодействие HDA15 с фактором морфогенеза HY5 (ELONGATED HYPOCOTYL5), напротив, происходит под воздействием света и приводит к подавлению экспрессии генов, связанных с удлинением гипокотиля в результате деацетилирования гистона H4 (Zhao et al., 2019).
Комплекс регуляторных белков SNL1 (SWI-INDEPENDENT3 (SIN3)-LIKE1) и SNL2 с HDA19 участвует в поддержании состояния покоя и в регуляции прорaстания путем деацетилирования гистонов Н3K9, H3K18, H3K14 в целевых генах, связанных с синтезом, деградацией абсцизовой кислоты (АБК), этилена, и ауксинов, а также сигнальными путями растительных гормонов (Luján-Soto, Dinkova, 2021). В состоянии покоя в клетке повышается уровень белков SNL1 и SNL2, которые направляют деацетилазу к гистонам, ассоциированным с генами гидролиза АБК и некоторыми генами биосинтеза этилена, и связанных с ним регуляторных систем, в результате чего растет уровень АБК и подавляется стимуляция прорастания этиленом (Wang et al., 2013). При набухании и прорастании семян уровень белков SNL1 и SNL2 снижается, при этом основную роль в стимуляции прорастания играет не ослабление механизма контроля прорастания, описанного выше, а повышение уровня ацетилирования генов ауксинового пути (преимущественно AUX1) и, соответственно, повышение уровня их экспрессии, что стимулирует удлинение, а в последствии и деление клеток корешка (Wang et al., 2016). Также показано участие гистоновых деацетилаз в росте корня путем регуляции уровня переносчика ауксина PIN1 (Nguyen et al., 2013). При набухании семян HDA9 в комплексе с белком PWR (POWERDRESS), обладающим высоким сродством к ацетилированному гистону H3, принимает участие в регуляции экспрессии белка SOM (регуляторный белок, содержащий структурный мотив “цинковые пальцы” типа СССН), который подавляет прорастание при повышенной температуре (Yang et al., 2019). Гистоновые деацетилазы семейства НВ2 отрицательно регулируют переход транзитно-амплифицирующихся клеток к растяжению путем регуляции метаболизма гиббереллинов в корневой меристеме и зоне элонгации (Li et al., 2017). В свою очередь, уровень гормонов влияет на степень ацетилирования гистонов, например, в алейроновом слое набухших семян кукурузы (Zea mays L.) под действием гиббереллина повышается содержание H3K9ac и H4K5ac в гистонах, ассоциированных с геном sodCp, кодирующим медь/цинк-зависимую супероксиддисмутазу, что приводит к накоплению пероксида водорода в клетке, запускающего регуляторную систему, обеспечивающую гидролиз крахмала эндосперма, что необходимо для питания проростка (Hou et al., 2015).
Показано участие ацетилирования гистонов в дифференцировке клеток проростка. Так у арабидопсиса в регуляторных сетях, отвечающих за дифференцировку волосковых и безволосковых клеток эпидермиса корней проростков, участвуют две гистоновые ацетилтрансферазы (GCN5 и HAF2) и три гистоновые деацетилазы (HDA6, HDA18 и HDA19) (Chen et al., 2016). Кроме того, ацетилирование принимает участие и в процессах поддержания разнообразия популяции проростков. Например, показано участие варьирования уровня экспрессии гена HD2B в неоднородности семян одной популяции Arabidopsis thaliana по скорости прорастания, при этом низкий уровень экспрессии наблюдался у семян, не вышедших из состояния покоя (Yano et al., 2013).
Запуск ростовых процессов при прорастании осуществляется за счет растяжения клеток, в то время как митотический цикл инициируется в меристеме побегов и корня на заключительных стадиях прорастания семян (Nieuwland et al., 2016). При этом синтез ДНК в S-фазе скоординирован с синтезом гистонов, а также с изменением характера их ацетилирования и деацетилирования (Chen, Tian, 2007). Первоначально гистоны ацетилируются HAT-B в цитоплазме, а затем транспортируются в ядро. Значимость ацетилирования гистона H4, в частности сайтов H4K5 и H4K12, при сборке хроматина во время S-фазы подтверждается совпадением повышения активности гистон-ацетилтрансферазы HAT-B с репликацией ДНК, что обнаружено в меристематических клетках кукурузы (Lechner et al., 2000). После сборки гистоны вновь подвергаются модификации, при этом формируется видоспецифичный паттерн ацетилирования/деацетилирования (Jasencakova et al., 2000, 2001; Wako et al., 2002). В эухроматине ацетилированные гистоны в значительном количестве присутствуют в ранней и средней S-фазе, деацетилировaние происходит в поздней S-фазе, и фазах М и G1; в гетерохроматиновых доменах гистоны сильно ацетилированы на поздних стадиях S-фазы и G2, в то время как деацетилирование происходит непосредственно перед митозом (Chen, Tian, 2007). В профазе митоза у ряда растений наблюдается корреляция характера распределения H4K5ас с размером генома. Так у Allium cepa, Callisia repens, Araucaria angustifolia и Nothрoscordum pulchellum, обладающих большими геномами, наблюдается равномерное рапределение H4K5ac в профазных/прометафазных хромосомах, в то время как у ряда растений с малыми геномами, Arachis pusilla, Bixa orellana, Costus spiralis, Eleutherine bulbosa, Indigofera campestris, Phaseolus lunatus, Phaseolus vulgaris, Poncirus trifoliata и Solanum lycopersicum, концевые области хроматина обогащены H4K5ac, а в проксимальных – гистон Н4 содержит в этом положении деацетилированный лизин (Feitoza et al., 2017). Однако равномерное рапределение H4K5ac у растений с небольшими геномами также встречается, например, у Eleocharis geniculata, Rhynchospora pubera и Rhynchospora tenuis. Исследования с использованием ингибиторов гистоновых ацетилаз (трихостатин А, бутират натрия) показали, что нарушение фазоспецифического деацетилирования гистонов H4 и H3, в частности по сайтам H3K9 и H4K5, изменяет укладку хроматина, вызывая задержку клеточного цикла (Li et al., 2005; Zhang et al., 2017; Vafina et al., 2020). Также было показано, что в условиях гиперацетилированного хроматина нарушается конденсация хромосом, что препятствует разделению сестринских хроматид и нарушает ход митоза (Cimini et al., 2003).
ЗАКЛЮЧЕНИЕ
Таким образом, эпигенетические механизмы являются важной составляющей многоуровневых регуляторных сетей клетки. Ацетилирование гистонов — один из наиболее динамичных эпигенетических механизмов, играющий важную роль на этапах быстрых преобразований функциональной активности клеток. Получение в перспективе полной картины участия модификации гистонов в процессах прорастания семян будет полезно для повышения урожайности сельскохозяйственных культур, как способ оценки качества и жизнеспособности семян перед посевом, и позволит разработать методы управления реализации генетического потенциала растений.
Список литературы
Данович К.Н., Соболев А.М., Жданова Л.П. и др. Физиология семян. М.: Наука, 1982. 318 с.
Alinsug M.V., Chen F.F., Luo M. et al. Subcellular localization of class II HDAs in Arabidopsis thaliana: nucleocytoplasmic shuttling of HDA15 is driven by light // PLoS One. 2012. V. 7 (2). P. e30846.
Azad G.K., Swagatika S., Kumawat M. et al. Modifying chromatin by histone tail clipping // J. Mol. Biol. 2018. V. 430 (18). P. 3051–3067.
Boycheva I., Vassileva V., Iantcheva A. Histone acetyltransferases in plant development and plasticity // Curr. Genom. 2014. V. 15 (1). P. 28–37.
Brownell J.E., Allis C.D. Special HATs for special occasions: linking histone acetylation to chromatin assembly and gene activation // Curr. Opin. Genet. Dev. 1996. V. 6 (2). P. 176–184.
Carrera-Castaño G., Calleja-Cabrera J., Pernas M. et al. An updated overview on the regulation of seed germination // Plants. 2020. V. 9 (6). P. 703.
Chen Z.J., Tian L. Roles of dynamic and reversible histone acetylation in plant development and polyploidy // Biochim. Biophys. Acta. 2007. V. 1769 (5–6). P. 295–307.
Chen W. Q., Li D. X., Zhao F. et al. One additional histone deacetylase and 2 histone acetyltransferases are involved in cellular patterning of Arabidopsis root epidermis // Plant Signal. Behav. 2016. V. 11. P. e1131373.
Chhun T., Chong S.Y., Park B.S. et al. HSI2 repressor recruits MED13 and HDA6 to down-regulate seed maturation gene expression directly during Arabidopsis early seedling growth // Plant Cell Physiol. 2016. V. 57. P. 1689–1706.
Cimini D., Mattiuzzo M., Torosantucci L., Degrassi F. Histone hyperacetylation in mitosis prevents sister chromatid separation and produces chromosome segregation defects // Mol. Biol. Cell. 2003. V. 14 (9). P. 3821–3833.
Davie J.R. Inhibition of histone deacetylase activity by butyrate // J. Nutr. 2003. V. 133 (7). P. 2485S–2493S.
Dokmanovic M., Marks P.A. Prospects: histone deacetylase inhibitors // J. Cell. Biochem. 2005. V. 96 (2). P. 293–304.
Du Z., Li H., Wei Q. et al. Genome-wide analysis of histone modifications: H3K4me2, H3K4me3, H3K9ac, and H3K27ac in Oryza sativa L. Japonica // Mol. Plant. 2013. V. 6 (5). P. 1463–1472.
Feitoza L., Costa L., Guerra M. Condensation patterns of prophase/prometaphase chromosome are correlated with H4K5 histone acetylation and genomic DNA contents in plants // PLoS One. 2017. V. 12 (8). P. e0183341.
Fina J.P., Masotti F., Rius S.P. et al. HAC1 and HAF1 histone acetyltransferases have different roles in UV-B responses in Arabidopsis // Front. Plant Sci. 2017. V. 8. P. 1179.
Fuchs J., Demidov D., Houben A., Schubert I. Chromosomal histone modification patterns – from conservation to diversity // Trends Plant Sci. 2006. V. 11. P. 199–208.
Gan L., Wei Z., Yang Z. et al. Updated mechanisms of GCN5 – the monkey king of the plant kingdom in plant development and resistance to abiotic stresses // Cells. 2021. V. 10 (5). P. 979.
Garcia-Ramirez M., Rocchini C., Ausio J. Modulation of chromatin folding by histone acetylation // J. Biol. Chem. 1995. V. 270 (30). P. 17923–17928.
Gong F., Chiu L.Y., Miller K.M. Signaling to genome maintenance and cancer // PLoS Genet. 2016. V. 12 (9). P. e1006272.
Haigney A., Ricketts M.D., Marmorstein R. Dissecting the molecular roles of histone chaperones in histone acetylation by type B histone acetyltransferases (HAT–B) // J. Biol. Chem. 2015. V. 290 (51). P. 30648–30657.
Hartl M., Fussl M., Boersema P.J. et al. Lysine acetylome profiling uncovers novel histone deacetylase substrate proteins in Arabidopsis // Mol. Syst. Biol. 2017. V. 13 (10). P. 949.
Hollender C., Liu Z. Histone deacetylase genes in Arabidopsis development // J. Integr. Plant Biol. 2008. V. 50. P. 875–885.
Hong L., Schroth G.P., Matthews H.P. et al. Studies of the DNA binding properties of histone H4 amino terminus // J. Biol. Chem. 1993. V. 268 (1). P. 305–314.
Hou H., Wang P., Zhang H. et al. Histone acetylation is involved in gibberellin-regulated sodCp gene expression in maize aleurone layers // Plant Cell Phys. 2015. V. 56 (11). P. 2139–2149.
Hu Y., Lu Y., Zhao Y., Zhou D.X. Histone acetylation dynamics integrates metabolic activity to regulate plant response to stress // Front. Plant Sci. 2019. V. 10. P. 1236.
Ivanov V.B., Dobrochaev A.E., Baskin T.I. What the distribution of cell lengths in the root meristem does and does not reveal about cell division // J. Plant Growth Reg. 2002. V. 21 (1). P. 60–67.
Jasencakova Z., Meister A., Walter J. et al. Histone H4 acetylation of euchromatin and heterochromatin is cell cycle dependent and correlated with replication rather than with transcription // Plant Cell. 2000. V. 12. P. 2087–2100.
Jasencakova Z., Meister A., Schubert I. Chromatin organization and its relation to replication and histone acetylation during the cell cycle in barley // Chromosoma. 2001. V. 110 (2). P. 83–92.
Kim S., Sophie J.M., Piquerez J.S. et al. GCN5 modulates salicylic acid homeostasis by regulating H3K14ac levels at the 5' and 3' ends of its target genes // Nucl. Acids Res. 2020. V. 48 (11). P. 5953–5966.
Kolle D., Sarg B., Lindner H., Loidl P. Substrate and sequential site specificity of cytoplasmic histone acetyltransferases of maize and rat liver // FEBS Lett. 1998. V. 421 (2). P. 109–114.
Kouzarides T. Chromatin modifications and their function // Cell. 2007. V. 128 (4). P. 693–705.
Kumar V., Thakur J.K., Prasad M. Histone acetylation dynamics regulating plant development and stress responses // Cell. Mol. Life Sci. 2021. V. 78 (10). P. 4467–4486.
Lechner T., Lusser A., Pipal A. et al. RPD3-type histone deacetylases in maize embryos // Biochemistry. 2000. V. 39 (7). P. 1683–1692.
Lee D.Y., Hayes J.J., Pruss D., Wolffe A.P. et al. A positive role for histone acetylation in transcription factor access to nucleosomal DNA // Cell. 1993. V. 72 (1). P. 73–84.
Li H., Torres-Garcia J., Latrasse D. et al. Plant-specific histone deacetylases HDT1/2 regulate GIBBERELLIN 2–OXIDASE2 expression to control Arabidopsis root meristem cell number // Plant Cell. 2017. V. 29 (9). P. 2183–2196.
Li Y., Butenko Y., Grafi G. Histone deacetylation is required for progression through mitosis in tobacco cells // Plant J. 2005. V. 41 (3). P. 346–352.
Liu C., Lu F., Cui X., Cao X. Histone methylation in higher plants // Annu. Rev. Plant Biol. 2010. V. 61. P. 395–420.
Liu X., Chen C.-Y., Wang K.-C. et al. PHYTOCHROME INTERACTING FACTOR3 associates with the histone deacetylase HDA15 in repression of chlorophyll biosynthesis and photosynthesis in etiolated Arabidopsis seedlings // Plant Cell. 2013. V. 25 (4). P. 1258–1273.
Liu X., Yang S., Yu C.W. et al. Histone acetylation and plant development // Enzymes. V. 40. / Eds C. Lin, Sh. Luan. Burlington: Academic Press, 2016. P. 173–199.
Loidl P. Towards an understanding of the biological function of histone acetylation // FEBS Lett. 1988. V. 227 (2). P. 91–95.
Loidl P. Histone acetylation: facts and questions // Chromosoma. 1994. V. 103 (7). P. 441–449.
Lu L., Chen X., Sanders D. et al. High-resolution mapping of H4K16 and H3K23 acetylation reveals conserved and unique distribution patterns in Arabidopsis and rice // Epigenetics. 2015. V. 10 (11). P. 1044–1053.
Luján-Soto E.V., Dinkova T.D. Time to wake up: epigenetic and small-RNA-mediated regulation during seed germination // Plants. 2021. V. 10 (2). P. 236.
Mahrez W., Arellano M.S., Moreno-Romero J. et al. H3K36ac is an evolutionary conserved plant histone modification that marks active genes // Plant Physiol. 2016. V. 170 (3). P. 1566–1577.
Mandal P., Verma N., Azad G.K. et al. Epigenetics: role of histone proteases in cellular functions and diseases // Molecular mechanisms and physiology of disease: implications for epigenetics and health / Eds N. Maulik, T. Karagiannis. N.Y.: Springer Science+Business Media, 2014. P. 113–125.
Mariño-Ramírez L., Kann M.G., Shoemaker B.A., Landsman D. Histone structure and nucleosome stability // Exp. Rev. Proteom. 2005. V. 2 (5). P. 719–729.
Martin B.J.E., Brind’Amour J., Kuzmin A. et al. Transcription shapes genome-wide histone acetylation patterns // Nat. Commun. 2021. V. 12 (1). P. 210.
Martínez Ó., Arjones V., González M.V. Histone deacetylase inhibitors increase the embryogenic potential and alter the expression of embryogenesis-related and HDAC-encoding genes in grapevine (Vitis vinifera L., cv. Mencía) // Plants. 2021. V. 10 (6). P. 1164.
Musselman C.A., Lalonde M.E., Côté J., Kutateladze T.G. Perceiving the epigenetic landscape through histone readers // Nat. Struct. Mol. Biol. 2012. V. 19 (12). P. 1218–1227.
Nguyen H.N., Kim J.H., Jeong C.Y. et al. Inhibition of histone deacetylation alters Arabidopsis root growth in response to auxin via PIN1 degradation // Plant Cell Rep. 2013. V. 32 (10). P. 1625–1636.
Nieuwland J., Stamm P., Wen B. et al. Re-induction of the cell cycle in the Arabidopsis post-embryonic root meristem is ABA-insensitive, GA-dependent and repressed by KRP6 // Sci. Rep. 2016. V. 6. P. 23586.
Nonogaki H. Seed dormancy and germination-emerging mechanisms and new hypotheses // Front. Plant Sci. 2014. V. 5. P. 233.
Nonogaki H., Bassel G.W., Bewley J.D. Germination – still a mystery // Plant Sci. 2010. V. 179 (6). P. 574–581.
Pandey R., Müller A., Napoli C.A. et al. Analysis of histone acetyltransferase and histone deacetylase families of Arabidopsis thaliana suggests functional diversification of chromatin modification among multicellular eukaryotes // Nucl. Acids Res. 2002. V. 30 (23). P. 5036–5055.
Parthun M.R., Widom J., Gottschling D.E. The major cytoplasmic histone acetyltransferase in yeast: links to chromatin replication and histone metabolism // Cell. 1996. V. 87 (1). P. 85–94.
Roth S.Y., Denu J.M., Allis C.D. Histone acetyltransferases // Annu. Rev. Biochem. 2001. V. 70. P. 81–120.
Sadoul K., Boyault C., Pabion M., Khochbin S. Regulation of protein turnover by acetyltransferases and deacetylases // Biochimie. 2008. V. 90. P. 306–312.
Servet C., Conde e Silva N., Zhou D.-X. Histone acetyltransferase AtGCN5/HAG1 is a versatile regulator of developmental and inducible gene expression in Arabidopsis // Mol. Plant. 2010. V. 3 (4). P. 670–677.
Smolikova G., Strygina K., Krylova E. et al. Transition from seeds to seedlings: hormonal and epigenetic aspects // Plants. 2021. V. 10 (9). P. 1884.
Spencer V.A., Davie J.R. Role of covalent modifications of histones on regulating gene expression // Gene. 1999. V. 240 (1). P. 1–12.
Tanaka M., Kikuchi A., Kamada H. The Arabidopsis histone deacetylases HDA6 and HDA19 contribute to the repression of embryonic properties after germination // Plant Phys. 2008. V. 146 (1). P. 149–161.
Vafina G.H., Ivanov R.S. Localization of Arg-X proteolysis in the supramolecular structures of cell nuclei during the induction of growth in mature wheat germs // Indian J. Plant Physiol. 2016. V. 21 (3). P. 370–373.
Vafina G.H., Ivanov R.S., Ivanova E.A. Analysis of Arg-X proteolytic activity in the supramolecular structures of cell nuclei influenced by inhibitor deacetylation of proteins during the germination of wheat // Indian J. Plant Physiol. 2017. V. (3). P. 358–364.
Vafina G.H., Ivanov R.S., Ivanova E.A. Changes of Arg-X proteolysis localization under conditions of deacetylation inhibition of nuclear proteins in spring and winter wheat seedlings // Acta Physiol. Plant. 2018. V. 40. P. 78.
Vafina G.H., Ivanov R.S., Kalashnik N. Features of the formation of Arg-X proteolytic system of cellular nuclei during germination of wheat seeds // Bulg. J. Agric. Sci. 2020. V. 26 (6). P. 1158–1165. https://www.agrojournal.org/26/06–08.html.
Vettese-Dadey M., Grant P.A., Hebbes T.R. et al. Acetylation of histone H4 plays a primary role in enhancing transcription factor binding to nucleosomal DNA in vitro // EMBO J. 1996. V. 15 (10). P. 2508–2518.
Wako T., Fukuda M., Furushima-Shimogawara R. et al. Cell cycle-dependent and lysine residue-specific dynamic changes of histone H4 acetylation in barley // Plant Mol. Biol. 2002. V. 49 (6). P. 645–653.
Wang Z., Cao H., Sun Y. et al. Arabidopsis paired amphipathic helix proteins SNL1 and SNL2 redundantly regulate primary seed dormancy via abscisic acid-ethylene antagonism mediated by histone deacetylation // Plant Cell. 2013. V. 25 (1). P. 149–166.
Wang Z., Chen F., Li X. et al. Arabidopsis seed germination speed is controlled by SNL histone deacetylase-binding factor-mediated regulation of AUX1 // Nat. Commun. 2016. V. 7. P. 13412.
Wolny E., Braszewska-Zalewska A., Kroczek D., Hasterok R. Histone H3 and H4 acetylation patterns are more dynamic than those of DNA methylation in Brachypodium distachyon embryos during seed maturation and germination // Protoplasma. 2017. V. 254. P. 2045–2052.
Xu Q., Liu Q., Chen Z. et al. Histone deacetylases control lysine acetylation of ribosomal proteins in rice // Nucl. Acids Res. 2021. V. 49 (8). P. 4613–4628.
Xue C., Liu S., Chen C. et al. Global proteome analysis links lysine acetylation to diverse functions in Oryza sativa // Proteomics. 2018. V. 18 (1). P. 1700036.
Yadav S.P., Das H.K. Discontinuous incorporation of amino acids in embryo proteins of wheat during germination // Dev. Biol. 1974. V. 36 (1). P. 183–186.
Yang W., Chen Z., Huang Y. et al. Powerdress as the novel regulator enhances Arabidopsis seeds germination tolerance to high temperature stress by histone modification of SOM locus // Plant Sci. 2019. V. 284. P. 91–98.
Yano R., Takebayashi Y., Nambara E. et al. Combining association mapping and transcriptomics identify HD2B histone deacetylase as a genetic factor associated with seed dormancy in Arabidopsis thaliana // Plant J. 2013. V. 74 (5). P. 815–828.
Yruela I., Moreno-Yruela C., Olsen C.A. Zn2+-dependent histone deacetylases in plants: structure and evolution // Trends Plant Sci. 2021. V. 26 (7). P. 741–757.
van Zanten M., Koini M.A., Geyer R. et al. Seed maturation in Arabidopsis thaliana is characterized by nuclear size reduction and increased chromatin condensation // PNAS USA. 2011. V. 108 (50). P. 20219–20224.
van Zanten M., Zöll C., Wang Z. et al. Histone deacetylase 9 represses seedling traits in Arabidopsis thaliana dry seeds // Plant J. 2014. V. 80 (3). P. 475–488.
Zhang H., Ogas J. An epigenetic perspective on developmental regulation of seed genes // Mol. Plant. 2009. V. 2 (4). P. 610–627.
Zhang Q., Wang P., Hou H. et al. Histone acetylation and reactive oxygen species are involved in the preprophase arrest induced by sodium butyrate in maize roots // Protoplasma. 2017. V. 254. P. 167–179.
Zhao L., Peng T., Chen C.-Y. et al. HY5 interacts with the histone deacetylase HDA15 to repress hypocotyl cell elongation in photomorphogenesis // Plant Physiol. 2019 V. 180 (3). P. 1450–1466.
Zhou Y., Tan B., Luo M. et al. HISTONE DEACETYLASE 19 interacts with HSL1 and participates in the repression of seed maturation genes in Arabidopsis seedlings // Plant Cell. 2013. V. 25 (1). P. 134–148.
Дополнительные материалы отсутствуют.
Инструменты
Успехи современной биологии


