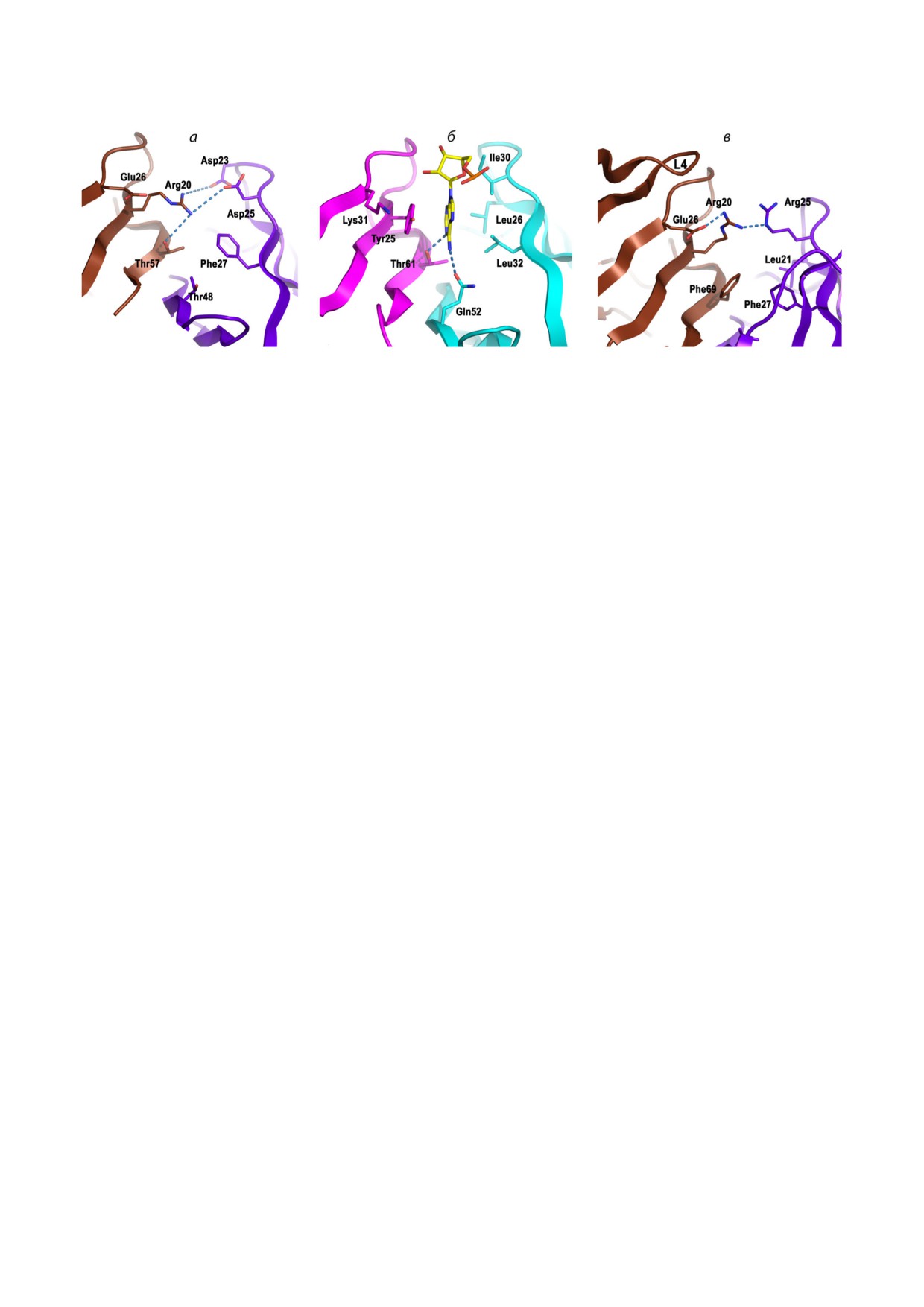БИОХИМИЯ, 2021, том 86, вып. 7, с. 985 - 995
УДК 577.21
ПРОСТРАНСТВЕННАЯ СТРУКТУРА И РНК СВЯЗЫВАЮЩИЕ
СВОЙСТВА Lsm БЕЛКА ИЗ Halobacterium salinarum
© 2021
М.С. Фандо, А.О. Михайлина, Н.В. Леконцева,
С.В. Тищенко, А.Д. Никулин*
Институт белка Российской академии наук, 142290 Пущино,
Московская обл., Россия; электронная почта: nikulin@vega.protres.ru
Поступила в редакцию 09.03.2021
После доработки 13.04.2021
Принята к публикации 13.04.2021
В настоящей работе определена структура и исследованы РНК связывающие свойства Lsm белка археи
Halobacterium salinarum, отличительной особенностью которого является короткая петля L4, соединяющая
тяжи β3 и β4. Поскольку бактериальные белки семейства Lsm, называемые Hfq, имеют короткую петлю L4
и образуют гексамеры, тогда как Lsm белки архей, SmAP, имеют длинную петлю L4 и образуют гептамеры,
было высказано предположение, что длина петли L4 может влиять на четвертичную структуру белков. Бо
лее того, петля L4 прикрывает область SmAP, соответствующую одному из сайтов связывания РНК в белках
Hfq и, таким образом, может влиять на РНК связывающие свойства белка. Наши результаты показывают,
что SmAP из H. salinarum формирует гептамеры и обладает теми же РНК связывающими свойствами, что и
гомологичные белки с длинной петлей L4. Следовательно, длина L4 не влияет на количество мономеров в
Lsm белках и на их РНК связывающие свойства.
КЛЮЧЕВЫЕ СЛОВА: Lsm белки, SmAP, четвертичная структура белков, РНК связывающие свойства бел
ков, малые регуляторные РНК, мрРНК, Halobacterium salinarum.
DOI: 10.31857/S0320972521070034
ВВЕДЕНИЕ
присутствии специфически связываемых малых
ядерных РНК и выполняют роль каркасных бел
Белки архей SmAP (Sm Archaeal Protein) от
ков малых ядерных рибонуклеопротеинов
носятся к семейству Lsm (Sm like) белков,
(мяРНП) [2, 9, 10]. Архейные белки семейства
представители которого имеют характерную
Lsm имеют от одного до трех паралогов (SmAP1,
пространственную структуру пятитяжевого
SmAP2, SmAP3) в зависимости от организма и
β листа с N концевой α спиралью [1-3]. Тяжи
существуют в виде гомологичных гептамеров,
β1, β2 и β3 составляют структурный мотив Sm1,
независимо от присутствия РНК [2]. Тем не ме
а тяжи β4 и β5 - мотив Sm2. Эти участки белка
нее существуют примеры архейных белков,
соединены петлей L4, длина которой варьирует
которые имеют другую степень олигомериза
в Lsm белках из разных организмов
[4].
ции, например, SmAP белок из Methanococcus
Lsm белки формируют четвертичную структуру
(Methanocaldococcus) jannaschii формирует гомо
в виде тороидальных колец, как правило, из 6
гексамер и описывается как Hfq подобный бе
или 7 мономеров. Бактериальные Lsm белки,
лок [11, 12], а SmAP2 из Archaeoglobus fulgidus
называемые Hfq, образуют стабильные в раство
(AF Sm2) может формировать как гексамеры,
ре гомогексамеры [5]. Эукариотические Lsm
так и гептамеры в зависимости от присутствия
белки характеризуются наличием ряда парало
РНК и рН среды [13]. Полагают, что зависи
гов в различных клетках организма; они форми
мость структуры от кислотности среды обуслов
руют гетерологичные гептамеры (Lsm1-7 и
лена изменением зарядов контактирующих в
Lsm2-8) [6-8]. В то же время Sm белки эукари
межмономерном пространстве аминокислот
от организовывают гетерогептамеры только в
ных остатков глутаминовой кислоты (Glu) [14].
Несмотря на значительный объем накопленных
Принятые сокращения: ДСН - додецилсульфат экспериментальных данных о природе Lsm бел
натрия; ДТТ - дитиотреитол; ПААГ - полиакриаламид
ков, механизм формирования четвертичной
ный гель; Mant AMP - 2′/3′ (N метил антранилоил) аде
нозин 5′ монофосфат; Mant GMP - 2′/3′ (N метил антра
структуры с различным числом мономеров до
нилоил) гуанозин 5′ монофосфат.
сих пор не установлен. На основании сравнения
* Адресат для корреспонденции.
первичных и четвертичных структур Lsm бел
985
986
ФАНДО и др.
ков из бактерий и архей/эукариот была выдви
ция об аминокислотной последовательности
нута гипотеза о взаимосвязи длины петли L4 и
белка SmAP из H. salinarum длиной 60 а.о.
степени олигомеризации Lsm белков [4], одна
ко ограниченность структурных данных каса
а в БД UniProt - протяженностью 69 а.о. (https://
тельно белков с различной длиной петли не поз
воляла подтвердить или опровергнуть её. Уста
пользовались оба варианта белка SmAP. Далее
новление гексамерной структуры Hfq подобно
вариант длиной 60 а.о. будет обозначаться как
го SmAP белка из археи M. jannaschii с короткой
HsaSmAP60, а длиной 69 а.о. - HsaSmAP69. Ге
петлей L4 вновь заставило исследователей заду
ны обоих вариантов нарабатывали с помощью
маться о влиянии этой петли на олигомериза
метода полимеразной цепной реакции (ПЦР) с
цию белка. Следует, однако, учитывать, что ами
использованием хромосомной ДНК археи
нокислотная последовательность этого белка
H. salinarum R1 в качестве матрицы. ПЦР прово
ближе к гексамерным белкам Hfq, поэтому ука
дили с использованием следующих олигонуклео
занный пример не может служить основанием
тидных праймеров:
для оценки выдвинутой гипотезы. Для проясне
ния этого вопроса в представленной работе на
HsaFor 5′ CATCACCATGGGCGGCCGACC 3′ и
ми определена и проанализирована структура
HsaRev 5′ GGGATAAGCTTTCATGGTTTGATG
SmAP белка из галофильной археи Halobacterium
GTGACG 3′ для HsaSmAP60;
salinarum, в котором петля L4 является короткой
и составляет всего 3 аминокислотных остатка
HsaFor 5′ CCATGGATGCCACCACCGG 3′ и
(а.о.).
HsaRev 5′ GGGATAAGCTTTCATGGTTTGATG
Недавно нами была продемонстрирована
GTGACG 3′ для HsaSmAP69.
высокая специфичность ряда SmAP белков к
уридинмонофосфату (UMP) и олиго(U) РНК
Полученные ПЦР фрагменты клонировали
при низком сродстве этих белков к другим рибо
в экспрессионный вектор pProExHtb («Invitro
нуклеотидам [15]. Сравнение структур белков
gen», США) с использованием эндонуклеаз ре
SmAP и их бактериальных гомологов Hfq в ком
стрикции Bsp19I и HindIII («СибЭнзим», Рос
бинации с биохимическими данными позволи
сия). Полученные конструкции проверяли сек
ло предположить, что белки SmAP имеют толь
венированием («Синтол», Россия) и трансфор
ко один, универсально консервативный ури
мировали ими клетки E. coli BL21(DE3), допол
дин специфический сайт, характерный для все
нительно содержащими плазмиду pLacIRARE
го семейства Lsm. Это значительно отличает
(«Novagen», Индия), кодирующую пять тРНК,
белки SmAP от бактериальных белков Hfq, у ко
узнающих редкие для E. coli кодоны соответ
торых существенную роль в функционировании
ствующих аминокислот.
играет второй, аденин специфический РНК
Выделение и очистка белков. Клетки культи
связывающий сайт. В SmAP белках этот регион
вировали в колбах емкостью 2 л при температу
прикрыт от внешних контактов протяжённой
ре
37
°С и интенсивном перемешивании
петлёй L4, к тому же, структура соответствую
(170 об./мин) на среде LB (Lysogeny broth), со
щего участка в архейных белках значительно от
держащей 100 мкг/мл ампициллина и 10 мкг/мл
личается от бактериальных. В белке SmAP из
хлорамфеникола до достижения значения опти
H. salinarum петля L4 имеет минимальную длину
ческой плотности ОП600 ≈ 0,7 о.е. Для активации
и не мешает доступу к участку, соответствующе
Т7 РНК полимеразы в среду добавляли изопро
му аденин связывающему сайту белка Hfq. Из
пил β D 1 тиогалактопиранозид (ИПТГ) до
мерение сродства такого SmAP белка к оли
конечной концентрации 0,5 мМ. После добав
го(А) РНК позволит оценить влияние длины
ления индуктора клетки инкубировали при
петли L4 на функциональность этого участка
20 °С в течение 12 ч для предотвращения агрега
белка. Таким образом, определение структуры и
ции нарабатываемых белков. Клетки собирали
измерение РНК связывающих свойств белка
центрифугированием при 8 000 g в течение
SmAP из H. salinarum позволит оценить влияние
20 мин при 4 °C, затем клеточную массу ресус
петли L4 на структуру и функцию архейных бел
пендировали в
30
мл лизирующего буфера
ков семейства Lsm.
(20 мМ Tris HCl, рН 8,0, 0,5 М NaCl, 1 мМ фе
нилметилсульфонил фторид (PMSF), 1 мМ ди
тиотреитол (ДТТ), 0,1% Triton X 100) и разруша
МАТЕРИАЛЫ И МЕТОДЫ
ли на проточном прессе EmulsiFlex C3 («Avestin»,
Канада). Клеточный дебрис осаждали центри
Клонирование генов белка SmAP из H. sali
фугированием в течение 30 мин при 14 000 g. Су
narum. В базе данных NCBI имеется информа
пернатант прогревали при 70 °C в течение
БИОХИМИЯ том 86 вып. 7 2021
РОЛЬ ПЕТЛИ L4 В Lsm БЕЛКАХ
987
10 мин. Денатурировавшие термолабильные
хранили в буфере PBS с 50% глицерина при тем
белки E. coli осаждали низкоскоростным цент
пературе -20 °С.
рифугированием при 14 000 g и температуре
Анализ вестерн блот. Фракции белков разде
+4 °С в течение 30 мин. Супернатант наносили
ляли при помощи электрофореза с 14% ным
на колонку с агарозой, модифицированной
ПААГ ДСН, после чего переносили на
Ni NTA («Qiagen», Германия), уравновешенную
PVDF мембрану («Bio Rad», США) с использо
буфером, содержащим 20 мМ Tris HCl, рН 8,0,
ванием Trans Blot Turbo Blotting System того же
0,5 М NaCl, и промывали тем же буфером с до
производителя (1 А, 25 В, 30 мин). Блокировали
бавлением 1 М LiCl. Для элюции белка исполь
мембрану в буфере TBS (50 мМ Tris НСl, pH 7,5,
зовали линейный градиент имидазола от 0 до
150 мМ NaCl). Далее обрабатывали мембрану
300 мМ. Удаление олигогистидиновой последо
буфером TBSTM (TBS pH 7,5, 0,05% Tween 20,
вательности осуществляли инкубацией препа
5% ное обезжиренное сухое молоко) в течение
рата белка с TEV протеазой в соотношении
1 ч при комнатной температуре. Затем мембрану
10 : 1 соответственно, переводя диализом в бу
инкубировали с антисывороткой кролика, раз
фер, содержащий 0,3 M NaCl, 20 мМ Tris HCl,
веденной буфером TBST (TBS pH 7,5, 0,05%
рН 8,0, 1 мМ ДТТ, 0,5 мМ ЭДТА. После протео
Tween 20) (1 : 2000) в течение 1 ч при комнатной
лиза раствор прогревали в течение 10 мин при
температуре. После трехкратного промывания
70 °C, денатурировавшую протеазу осаждали
(по 10 мин каждое) в TBST мембрану инкубиро
центрифугированием при 14 000 g в течение
вали в течение 1 ч при комнатной температуре с
30 мин при температуре +4 °C. Разделение бел
козьими антителами к IgG кролика, конъюгиро
ков по наличию олигогистидиновой концевой
ванными с пероксидазой хрена (Goat Anti
последовательности осуществляли путём нане
Rabbit IgG H&L (HRP) (ab205718); «Abcam», Ве
сения смеси белков на колонку с агарозой, мо
ликобритания) (1 : 20 000). Промывали мембра
дифицированной Ni NTA («Invitrogen»). На фи
ну в буфере TBST 2 раза по 5 мин, затем 1 раз в
нальной стадии целевые белки очищали гель
буфере TBS в течение 2 мин. Инкубировали
фильтрацией на колонке Superdex 75 («Cytiva»,
мембрану с хемилюминесцентным субстратом
США), уравновешенной буфером, содержащим
(Clarity™ Western ECL Substrate, «Bio Rad») в
20 мМ Tris HCl, pH25°С 8,0, 300 мМ NaCl. Полу
темноте в течение 5 мин. Анализ проводили с
ченные препараты белков хранили при -20 °С.
использованием системы детекции ChemiDoc
Выделение антител, специфичных к белку
MP Imaging System («Bio Rad»).
SmAP из H. salinarum. Сыворотка крови кроли
Анализ сродства белков к РНК по изменению
ка, содержащая антитела, специфичные к белку
подвижности в ПААГ при электрофорезе в недена
SmAP из H. salinarum (ATSmAP), была получена в
турирующих условиях. РНК олигонуклеотиды
Институте биофизики клетки РАН (Пущино,
синтезированы химически и предоставлены
Россия). Белки сыворотки кролика осаждали
фирмой «Синтол» (Россия). В работе использо
2 М сульфатом аммония и отделяли центрифу
вали РНК олигонуклеотиды с 18 уридинами
гированием при 20 000 g и 4 °С в течение 15 мин.
или аденинами на 3′ конце. На 5′ конце нахо
Далее сыворотку очищали с помощью двух по
дится 16 нуклеотидов, не имеющих специфи
следовательных хроматографий. На первом эта
ческого сродства к белкам SmAP/Hfq [15]; они
пе использовали аффинную хроматографию на
служат спейсерами при иммобилизации олиго
Protein A Sepharose («Pharmacia LKB», США).
нуклеотидов на чипы в экспериментах по ана
Сыворотку наносили на смолу в буфере PBS
лизу сродства исследуемых белков к РНК мето
(137 мМ NaCl, 2,7 мМ KCl, 10 мМ Na2HPО4,
дом поверхностного плазмонного резонанса
1,8 мМ KH2PО4, pH 7,4) и промывали тем же бу
(ППР, см. ниже):
фером. Элюцию суммарных антител проводили
глициновым буфером с концентрацией 250 мМ,
U18 РНК: 5′ GUGGUCAGUCGAGUGG (U)18 3′
pH 3,0. Затем антитела, специфичные к белку
SmAP из H. salinarum, очищали на смоле из ага
А18 РНК: 5′ GUGGUCAGUCGAGUGG (A)18 3′.
розы, модифицированной Ni NTA, с иммоби
лизованным на ней рекомбинантным белком
Для оценки сродства белков к РНК по изме
SmAP, содержащим 6 остатков гистидина на
нению подвижности олигонуклеотидов в ПААГ
N конце. Суммарные очищенные антитела на
в неденатурирующих условиях образец РНК
носили на Ni NTA SmAP в буфере PBS, промы
(3 мкг) прогревали в течение 10 мин при 60 °С,
вали буфером PBS c 2 M LiCl. Элюцию ATSmAP
добавляли белок в различных молярных соотно
проводили градиентом буфера PBS с содержа
шениях, затем полученную смесь инкубировали
нием MgCl2 от 1 до 4 M. Фракции, содержащие
при 37 °С в течение 10 мин. После этого прово
чистые ATSmAP, не загрязненные белком SmAP,
дили электрофоретический анализ в 10% ном
БИОХИМИЯ том 86 вып. 7 2021
988
ФАНДО и др.
ПААГ (акриламид : метилен бис акриламид =
зотропию флуоресценции измеряли на длине
= 19 : 1) в 90 мМ трис ацетатном буфере (ТАЕ) с
волны 448 нм при возбуждении УФ на длине
рН 7,8, содержащем 2 мМ MgCl2. В образцы пе
волны 335 нм. Для каждой точки проводили 5
ред нанесением добавляли глицерин и бромфе
независимых измерений величины интенсив
ноловый синий до конечных концентраций 5 и
ности флуоресценции и затем автоматически
0,1% соответственно. Электродный буфер имел
рассчитывали среднюю величину и погреш
следующий состав: 90 мМ ТАЕ (рН 7,8), 10 мМ
ность в программе QtiPlot («Iondev Srl», Румы
MgCl2. После окончания электрофореза РНК в
ния). Результат отображали графически с по
геле окрашивали раствором, содержащим
мощью программы QtiPlot.
0,25% ный толуидиновый синий, 5% ную ук
Кристаллизация белка SmAP из H. salinarum.
сусную кислоту, 10% ный этанол.
Для экспериментов по кристаллизации исполь
Анализ сродства исследуемых белков к РНК
зовали метод диффузии паров в висящей капле
методом поверхностного плазмонного резонанса.
на 24 луночных плашках (VDX greased plates,
Анализ осуществляли с использованием олиго
«Hampton Researсh», США). Кристаллизацию
нуклеотидов А18 РНК и U18 РНК, биотинили
проводили в каплях объемом 2-3 мкл, поме
рованных по 5′ концу. Связывание белка с целе
щенных на силиконизированные стеклянные
вой РНК проводили при 25 °C с использованием
пластины («Hampton Researсh») при 24 °С. Объ
системы ProteOn XPR36 («Bio Rad»). Биотини
ем противораствора составлял 400 мкл.
лированные РНК прогревали 5 мин при 95 °C и
Кристаллы белка HsaSmAP69, пригодные
наносили со скоростью потока 30 мкл/мин на
для рентгеноструктурного анализа, получены не
сенсорный чип ProteOn NLC с иммобилизован
были.
ным авидином до заполнения поверхности чи
Кристаллы белка HsaSmAP60 были получе
па, характеризующегося значением 500-700
ны в условиях D9 (15% w/v ПЭГ 8000; 100 мМ
единиц ответа (response unit, RU). Поверхность
MES, рН 6,5; 200 мМ ацетат кальция) набора
чипа дважды промывали восстанавливающим
Nuс Pro 2 («Jena Bioscience») при соотношении
раствором, содержащим 0,05% w/v додецил
белок : противораствор = 2 : 1 и концентрации
сульфат натрия для удаления не связавшейся
белка в образце 9,8 мг/мл. Кристаллы вырастали
РНК.
в течение недели до размера 100-150 мкм в дли
Для каждого набора сенсограмм были при
ну и 20-30 мкм в ширину. Кристаллы комплек
готовлены серии разведений белков в буфере,
са HsaSmAP60 с UMP были получены инкуба
содержащем 50 мМ Tris HCl (pH 7,5), 10 мМ
цией кристаллов белка в криопротекторном
MgCl2, 350 мМ NaCl, 2 мМ ДТТ с 0,05% v/v
растворе (0,17 М ацетат натрия, 0,085 М какоди
Tween 20 буфером (TMN). Нанесение образца
лат натрия, рН 6,5, 25,5% ПЭГ 8000, 15% глице
включает фазу ассоциации длиной 300-500 сек
рин) с добавлением рибонуклеотида в концент
и фазу диссоциации 700-3000 сек. В конце каж
рации 20 мМ.
дого цикла поверхность чипа промывали
Получение наборов дифракционных данных. С
0,1-0,2% ным ДСН, чтобы добиться полной
кристаллов белка HsaSmAP60 в комплексе с
диссоциации белка с поверхности PНК. Кине
UMP были собраны дифракционные данные на
тический анализ проводили при использовании
экспериментальной станции ID23 1 источника
набора из 4-5 сенсограмм с применением моде
синхротронного излучения ESRF (Гренобль,
ли для одностадийной реакции (по Ленгмюру) с
Франция) (таблица). Первичную обработку дан
коррекцией на масс транспорт. Были определе
ных проводили с помощью программы XDS
ны константы скоростей ассоциации (ka) и дис
(Институт медицинских исследований им. Макса
социации (kd) комплексов и рассчитана равно
Планка, Гейдельберг, Германия) [18], затем набор
весная константа диссоциации (КD = kd/ka).
структурных факторов интегрировали и приво
Анализ сродства исследуемых белков к флуо
дили к единой шкале в программе Aimless комп
ресцентно меченым рибонуклеотидам по анизо
лекса ССР4 (Лаборатория молекулярной биоло
тропии флуоресценции. Эксперименты проводили
гии MRC, Кэмбридж, Великобритания) [19].
на флуоресцентном спектрометре Cary Eclipse
Определение и уточнение структуры белка.
(«Agilent», США) при температуре 22 °C в кюве
Структуру белка HsaSmAP60 в комплексе с
те 0,3 × 0,3 см согласно описанной ранее методи
UMP определяли методом молекулярного заме
ке [16, 17]. Для проведения измерений раствор
щения с использованием программы MoRDa
Mant AMP или Mant GMP («Jena Bioscience»,
Германия) с концентрацией 1 мМ в буфере со
ac.uk/ccp4online/). Автоматический поиск наи
става 0,2 M NaCl, 10 мМ MgCl2, 50 мМ Tris HCl
более подходящей модели для решения структу
(pH 8,0) титровали раствором белка с увеличе
ры показал наилучший результат для мономера
нием его концентрации от 10-6 до 10-4 М. Ани
белка AF Sm2 из Archaeoglobus fulgidus (PDB
БИОХИМИЯ том 86 вып. 7 2021
РОЛЬ ПЕТЛИ L4 В Lsm БЕЛКАХ
989
ID: 1ljo). Уточнение модели проводили в прог
Статистика набора дифракционных данных и уточнения
структуры белка SmAP из H. salinarum (HsaSmAP60) в
рамме phenix.refine [21] комплекса Phenix (Лабо
комплексе с UMP
ратория молекулярной биологии MRC) [22] с
последовательным использованием протоколов
Характеристика
Значение
rigid body refinement (уточнение положения каж
дого мономера белка как отдельного твердого
Код PDB
6tfl
тела) и simulated annealing - Cartesian (уточнение
Длина волны (Å)
0.9724
координат индивидуальных атомов и индивиду
Область разрешения (Å)
50.0 2.4 (2.5 2.4)
альных В факторов с использованием симуля
Пространственная группа
P 41 21 2
ции молекулярной динамики). После первого
Параметры ячейки (Å, °)
156.45; 156.45; 106.63; 90; 90; 90
цикла уточнения использовали возможность ав
Общее число рефлексов
1318697 (82803)
томатического дополнения модели связанными
Число уникальных рефлексов
52360 (5118)
молекулами воды с помощью программы
Избыточность
25.2
(16.0)
phenix.refine. На этом шаге также дополняли не
Полнота данных (%)
99.8
(98.4)
заполненные участки электронной плотности
Среднее значение I/Σ(I)
7.3
(2.3)
аминокислотными остатками согласно амино
B фактор Вильсона
42.7
кислотной последовательности. В электронной
R merge
0.348
(0.908)
плотности, не принадлежащей полипептидным
R meas
0.355
(0.937)
цепям белка, было идентифицировано 14 моле
R pim
0.069
(0.232)
кул UMP, 1 молекула глицерина и 28 ионов
кальция. Для них проведено уточнение индиви
CC1/2
0.99
(0.89)
дуальных координат, В факторов, а также вели
CC
1.00
(0.97)
чин замещения (occupancy). После каждого эта
Число рефлексов, использо
52247 (5101)
па уточнения соответствие структуры и расчет
ванных для уточнения
ной карты электронной плотности проверяли в
Число рефлексов, использо
2700 (279)
программе Coot (Лаборатория молекулярной би
ванных для расчета R free
ологии MRC) [23]. На финальных стадиях уточ
R work
0.178
(0.270)
нения использовали протокол без применения
R free
0.230
(0.346)
симуляции отжига с проверкой соответствия па
CC(work)
0.94
(0.72)
раметров геометрии полипептидной цепи и ли
CC(free)
0.91
(0.65)
гандов идеальным значениям, что позволило
Число атомов общее
7187
получить структуру высокого качества с мини
В молекулах белка
6841
мальными отклонениями по геометрии и низ
и нуклеотидах
кими значениями R фактора (таблица).
В лигандах
34
В молекулах воды
312
СКО длин связей
0.007
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
СКО валентных углов
0.818
СКО двугранных углов
24.76
Идентификация варианта белка SmAP археи
СКО хиральных углов
0.059
H. salinarum. В ходе поиска последовательности
СКО планарных углов
0.004
белка SmAP из H. salinarum, а также кодирующе
Число остатков в областях
го его гена, в базах данных NCBI и UniProt бы
карты Рамачандрана
ли обнаружены две записи с отличающейся дли
Предпочтительной (%)
98.57
ной N концевой части белка на 9 аминокислот
Допустимой (%)
1.43
ных остатков (рис. 1). Оба варианта гена были
Запрещенной (%)
0.00
клонированы, затем белки наработаны в
Число нежелательных
0.00
E. coli - HsaSmAP60 (60 а.о.) и HsaSmAP69
ротамеров (%)
(69 а.о.). Отличие в первичной структуре белков
Оценка нежелательных
5.61
не сказалось на их свойствах при выделении и
контактов (clashscore)
очистке. С помощью анализа вестерн блот нами
Средний B фактор атомов
47.50
было показано, что в клетках H. salinarum присут
(общий)
ствует белок длиной 69, а не 60 а.о. (рис. 2).
Белка
47.40
РНК связывающие свойства SmAP из H. sali
Лигандов
73.52
narum. Сродство белков к РНК олигонуклеоти
Воды
49.21
дам оценивали двумя методами: по изменению
подвижности РНК-белкового комплекса при
Примечание. Данные в скобках соответствуют слою высо
электрофорезе в ПААГ в неденатурирующих ус
кого разрешения 2,5-2,4 Å.
БИОХИМИЯ том 86 вып. 7 2021
990
ФАНДО и др.
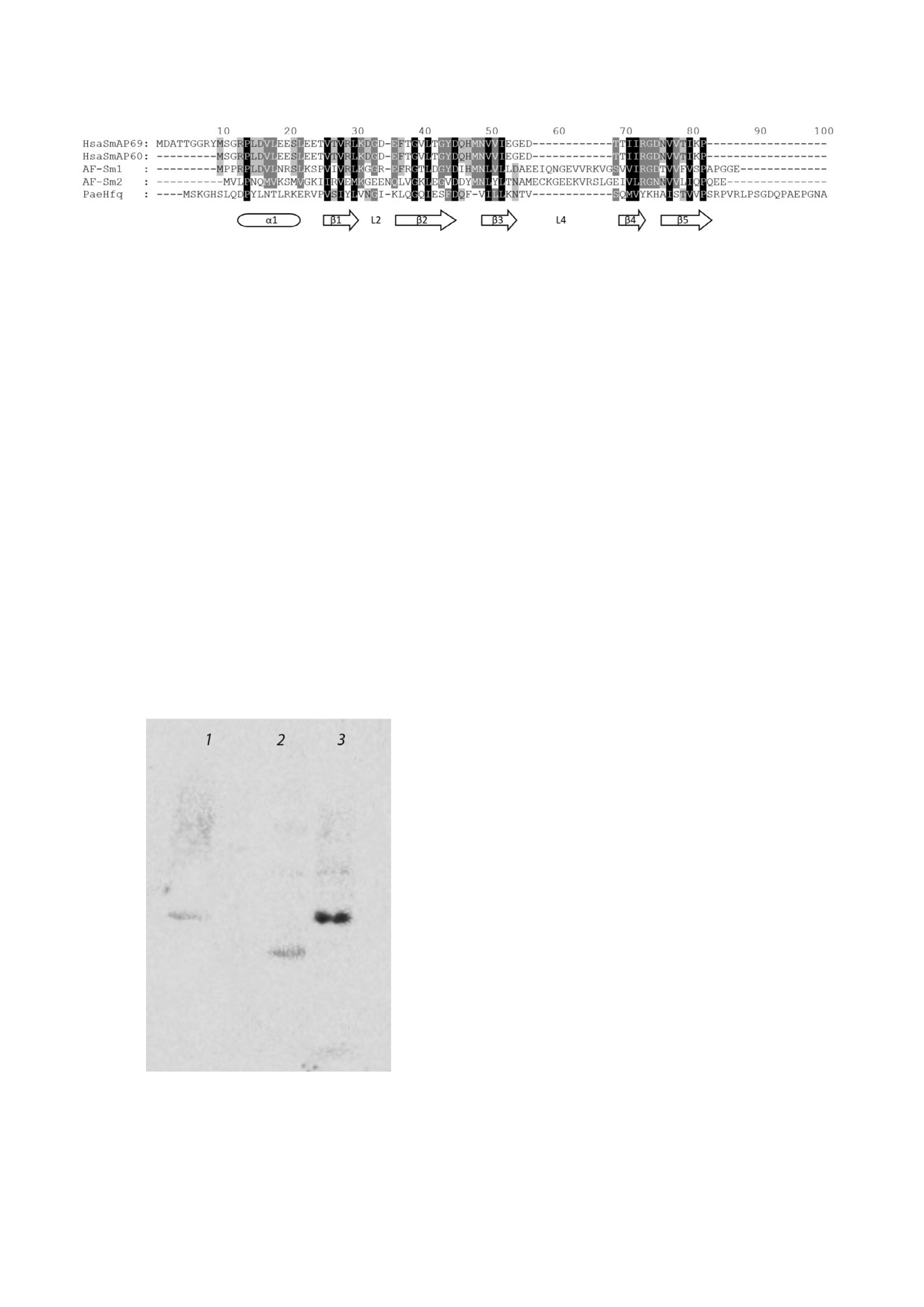
Рис. 1. Сравнение последовательностей мономеров белков HsaSmAP69 (SmAP из H. salinarum длиной 69 а.о. согласно за
писи UniProt Q9HPS2), HsaSmAP60 (SmAP из H. salinarum длиной 60 а.о., UniProt B0R5R2), AF Sm1 (SmAP1 из A. fulgidus,
UniProt O29386), AF Sm2 (SmAP2 из A. fulgidus, UniProt O29885), PaeHfq (Hfq из Pseudomonas aeruginosa, UniProt
Q9HUM0). Черным цветом выделены аминокислотные остатки со степенью гомологии 100%, темно серым - от 80 до
100%, светло серым - от 60 до 80%. Внизу приведена соответствующая вторичная структура белков. Выравнивание по
следовательностей проводили в программе ClustalX с учетом пространственной структуры белков. Анализ гомологии про
водили в программе GeneDoc
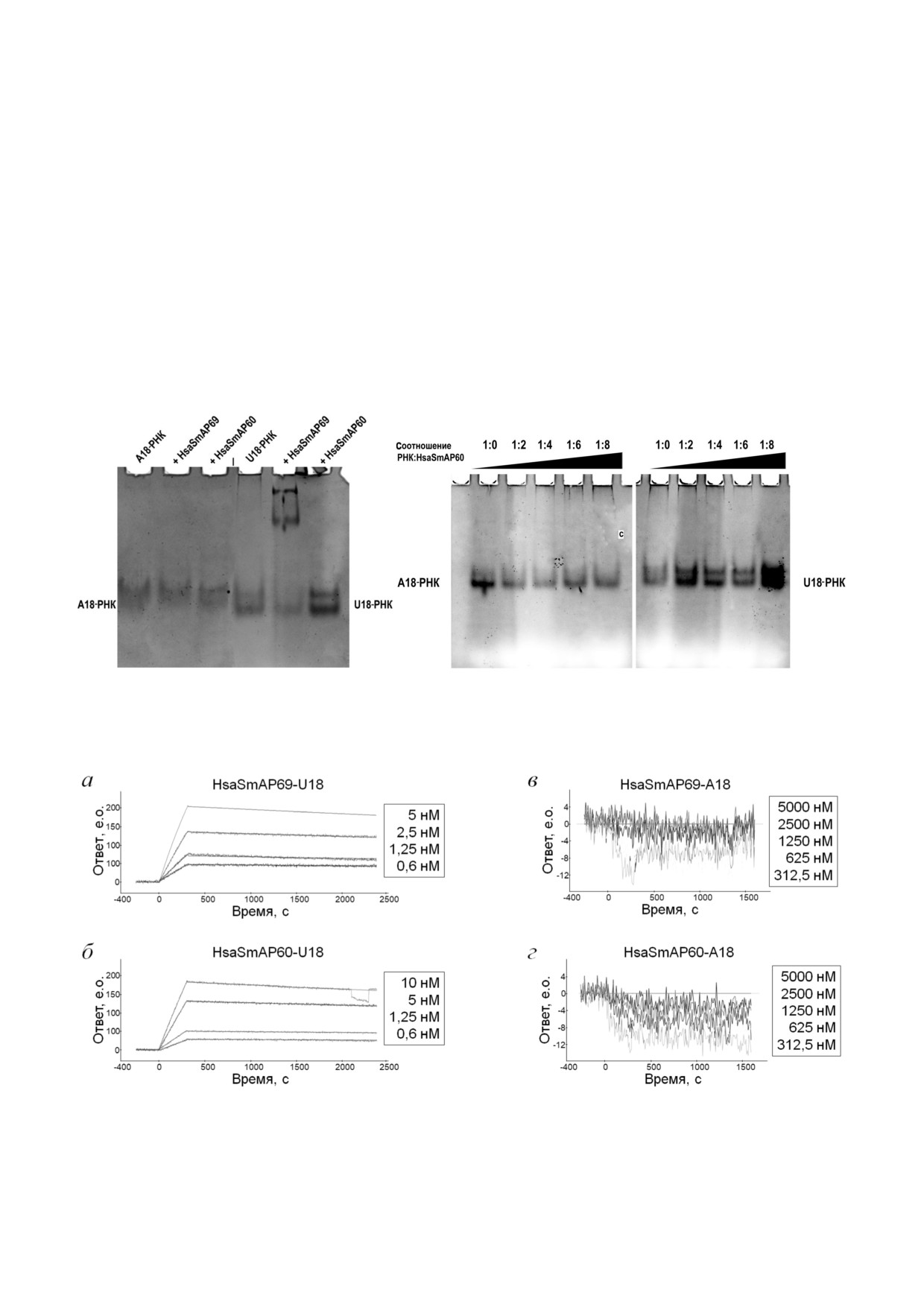
ловиях («гель шифт») и методом поверхностно
данным для комплексов гомологичных белков с
го плазмонного резонанса. На рис. 3 представ
такой же РНК [15]. Как указано выше, вариант
лены результаты «гель шифта»: показано, что
HsaSmAP69, в отличие от HsaSmAP60, имеет до
оба варианта белка HsaSmAP не связываются с
полнительную N концевую последователь
А18 РНК. Комплекс с U18 РНК формировался
ность, содержащую преимущественно положи
только с удлинённым вариантом белка,
тельно заряженные а.о. (рис. 1), которые, по
HsaSmAP69 (рис. 3).
всей видимости, способствуют связыванию бел
Использование более чувствительного по
ка с РНК. Аналогичный эффект N концевой
сравнению с «гель шифтом» метода ППР пока
последовательности ранее наблюдался нами для
зало, что с U18 РНК способны взаимодейство
Hfq подобного SmAP белка из археи M. jan$
вать оба белка (рис. 4, а, б), причём константы
naschii: при удалении этого участка KD комплек
диссоциации комплексов отличаются менее,
са белка с олиго(U) также увеличивалась при
чем на порядок: KD(HsaSmAP69 - U18 РНК) =
мерно на порядок [12].
= 0,12 ± 0,05 нМ, KD(HsaSmAP60 - U18 РНК) =
Данные, полученные методом ППР, под
= 0,93 ± 0,11 нМ. Сродство белков к U18 РНК
твердили отсутствие связывания белков с
оказалось близким к имеющимся в литературе
А18 РНК (рис. 4, в, г).
Проведенный ранее структурно кинетичес
кий анализ РНК белковых взаимодействий
SmAP из других архей показал, что их сродство к
олиго(А) РНК является незначительным [15];
согласно нашим данным, для HsaSmAP связы
вание не детектируется даже при высоких кон
центрациях белков (рис. 4).
Нуклеотид связывающие свойства SmAP из
H. salinarum. Ранее мы успешно использовали
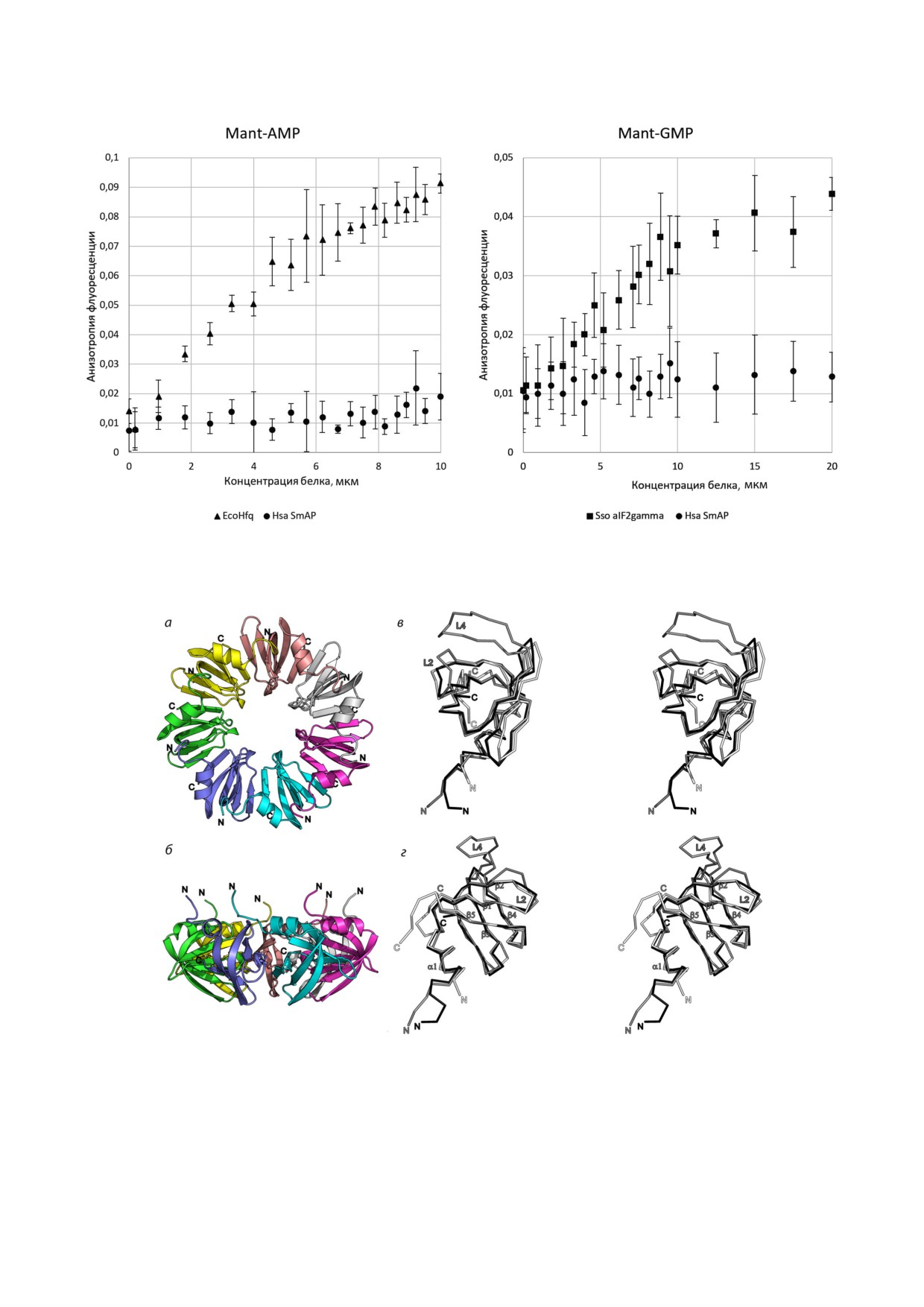
анализ нуклеотид связывающих свойств белков
Hfq и SmAP для оценки их сродства к РНК по
изменению флуоресценции [15-17]. Аналогич
ные эксперименты для HsaSmAP69 показали,
что этот белок не связывает нуклеотиды AMP и
GMP (рис. 5). Эти данные подтверждают, что
белок HsaSmAP не имеет участков специфичес
кого связывания аденинов и гуанинов.
Пространственная структура белка SmAP из
H. salinarum. Структура белка HsaSmAP опреде
лена методом молекулярного замещения с ис
пользованием сервиса MoRDa («BioMEX», Ве
ликобритания) [20]. Этот сервис использует на
бор инструкций для решения задачи молекуляр
Рис. 2. Вестерн блот: 1 - лизат клеток H. salinarum; 2 - ре
ного замещения с автоматическим перебором
комбинантный SmAP из H. salinarum длиной 60 а.о.
(HsaSmAP60); 3 - рекомбинантный SmAP из H. salinarum
пространственных структур из базы данных с
длиной 69 а.о. (HsaSmAP69)
наивысшей степенью гомологии относительно
БИОХИМИЯ том 86 вып. 7 2021
РОЛЬ ПЕТЛИ L4 В Lsm БЕЛКАХ
991
заданной последовательности белка. Каждая
мера белка HsaSmAP (рис. 6, а и б). Структура
выбранная структура используется для незави
мономера полностью соответствует структурам
симого решения задачи молекулярного замеще
других белков этого класса и содержит N конце
ния, что позволяет оптимизировать поиск реше
вую α спираль и β бочонок из пяти тяжей, при
ния и улучшить его точность. На выходе выдает
чём тяж β2 имеет большую длину и излом посе
ся наилучшее решение из возможных для всего
редине. Сравнение со структурами белков Hfq
пула первоначальных моделей. Для нашего слу
из E. coli и AF Sm2 из A. fulgidus показало не
чая наилучшей моделью оказалась структура
большое отклонение между координатами
мономера SmAP белка из A. fulgidus AF Sm2
Сα цепи консервативной части белков - 1,05 Å
(PDB ID: 1ljo) [13, 24].
и 0,84 Å соответственно (рис. 6, в и г). Интерес
В асимметричной части ячейки кристалла
но отметить, что третичная структура мономера
найдено 14 мономеров, образующие два гепта HsaSmAP несколько ближе к структуре бактери
а
б
Рис. 3. Проверка способности SmAP из H. salinarum образовывать комплексы с А18 РНК и U18 РНК по изменению под
вижности в ПААГ при электрофорезе в неденатурирующих условиях: а - при молярном соотношении РНК : белок = 1 : 2;
б - для HsaSmAP60 при различных молярных соотношениях РНК : белок
Рис. 4. Сенсограммы, отображающие кинетический анализ взаимодействия HsaSmAP69 (а, в) и HsaSmAP60 (б, г) с
U18 РНК (а, б) и А18 РНК (в, г). Для каждого набора сенсограмм указаны используемые концентрации белков. Ровные
линии соответствуют теоретической аппроксимации полученных экспериментальных данных
БИОХИМИЯ том 86 вып. 7 2021
992
ФАНДО и др.
Рис. 5. Кривые титрования белка HsaSmAP69 флуоресцентно мечеными нуклеотидами Mant AMP и Mant GMP. В каче
стве контроля (специфически связывающихся с нуклеотидами белков) использованы белок Hfq из E. coli (EcoHfq) для
Mant AMP и гамма субъединица aIF2 из Sulfolobus solfataricus (Sso aIF2gamma) для Mant GMP
Рис. 6. Структура белка SmAP из H. salinarum: а - общий вид структуры гептамера со стороны α спиралей мономеров
белка (проксимальная сторона). Обозначены N и С концы мономеров; б - общий вид структуры с боковой стороны
гептамера; в и г - наложение Cα цепей мономеров SmAP из H. salinarum (PDB ID: 6tfl) (черная), AF Sm2 из A. fulgidus
(PDB ID: 1ljo) (серая) и Hfq из E. coli (PDB ID: 1hk9) (белая). Стереопары: мономер на панели г повернут относительно
панели в на 90° по вертикальной оси. Обозначены N и С концы белков, элементы вторичной структуры, петли L2 и L4.
journal/biokhsm/)
БИОХИМИЯ том 86 вып. 7 2021
РОЛЬ ПЕТЛИ L4 В Lsm БЕЛКАХ
993
Рис. 7. Сравнение уридин специфического РНК связывающего участка белков: а - SmAP из H. salinarum (PDB ID: 6tfl);
б - Hfq из P. aeruginosa (PDB ID: 4mml). Водородные связи показаны пунктирными линиями. в - Изменение ориентации
связанных со SmAP и Hfq нуклеотидов относительно друг друга. Соседние мономеры белка Hfq показаны малиновым и
голубым, SmAP - коричневым и фиолетовым. Наложение структур произведено в программе Сооt
ального белка Hfq, чем к структуре AF Sm2: оба
риальных белках Hfq. Сравнение структуры
белка имеют короткую последовательность пет
комплексов белков Hfq и SmAP показывает, что
ли L4, что сказывается на ориентации петли L2,
модификация области контактов с рибонуклео
соединяющей тяжи β1 и β2. Тем не менее белок
тидом вызывает изменение положения основа
Hfq имеет больше витков спирали α1 по сравне
ния относительно межмономерного интерфейса
нию с архейными белками, что влияет на кон
белков (рис. 7, в). UMP в SmAP белках развер
формацию петли L1, соединяющую спираль и
нут примерно на 90° по сравнению с его ориен
тяж β1 (рис. 6, г). Несмотря на короткую после
тацией в комплексах с белками Hfq при мини
довательность петли L4, белок HsaSmAP фор
мальном изменении локальной структуры бел
мирует гептамер, а не гексамер, что указывает
ков. Возможно, такое изменение положения
на независимость четвертичной структуры
уридина на поверхности SmAP белков связано с
Lsm белков от длины петли L4.
изменением роли белка в клетках архей по срав
В каждом гептамере белка HsaSmAP найдено
нению с бактериальными гомологами.
7 молекул UMP (рис. 7). Они связаны в стандарт
Сравнение структуры области белка
ном для Lsm белков уридин специфическом
HsaSmAP, соответствующей аденин специфи
РНК связывающем участке белка на прокси
ческому сайту связывания белка Hfq, показало
мальной стороне мультимера. Дополнительных
её полную непригодность для взаимодействия с
мест связывания UMP, как и в случае белка Hfq
рибонуклеотидами, несмотря на то что эта часть
из P. aeruginosa [25], зафиксировано не было.
дистальной поверхности гептамера HsaSmAP не
Основание нуклеотида расположено между бо
прикрыта петлей L4, как в большинстве SmAP
ковыми цепями аминокислотных остатков
белков (рис. 8). В бактериальных белках Hfq
Arg51 и His37 одного мономера белка, причём,
аденин связывающий сайт сформирован не
как и в аналогичных комплексах белков SmAP с
сколькими гидрофобными остатками, что соз
уридинами, боковая цепь аргинина находится в
дает благоприятные условия для фиксации ос
стэкинге с основанием. Атомы Arg51 не форми
нования аденина в правильном положении бла
руют водородные связи с атомами основания -
годаря стэкингу с консервативным остатком
их образуют атомы боковой цепи Asn39 с N4 и
Phe/Tyr (в Hfq из E. coli - Tyr25). При этом ато
О4 уридина, а также атомы кислорода главной
мы боковых цепей консервативных Thr и Gln
цепи Asp53 с О2 и Gln36 соседнего мономера с
формируют водородные связи с атомами осно
О4 (рис. 7, а). Такая система водородных связей
вания (рис. 8, б). В аналогичном участке белка
характерна для известных структур SmAP бел
HsaSmAP не наблюдается такого кармана из
ков в комплексах с олиго(U) РНК и UMP [15,
гидрофобных остатков, а доступ к нему закрыт
24, 26-28]. Важную роль в дискриминации ри
Arg20, положение боковой цепи которого за
бонуклеотидов от дезоксирибонуклеотидов иг
фиксировано тремя водородными связями
рает контакт боковой цепи His37 с O2 атомом
(рис. 8, а). Аналогичная картина наблюдается
UMP, аналогичный контакту O2 с His57 в бакте
для архейного белка AF Sm1 (рис. 8, в) и для
4 БИОХИМИЯ том 86 вып. 7 2021
994
ФАНДО и др.
Рис. 8. Сравнение областей белков, соответствующих аденин специфическому РНК связывающему участку Hfq:
а - SmAP из H. salinarum (PDB ID: 6tfl); б - Hfq из E. coli (PDB ID: 3gib); в - AF Sm1 из A. fulgidus (PDB ID: 1i5l). Сосед
ние мономеры белка Hfq показаны малиновым и голубым, SmAP белков - коричневым и фиолетовым
других SmAP белков [15]. Такое расположение
Финансирование. Работа выполнена при фи
аминокислотных остатков, по видимому, не
нансовой поддержке Российского фонда фунда
позволяет этому участку SmAP белков выпол
ментальных исследований (грант № 18 04
нять роль РНК связывающего сайта независи
00222).
мо от длины петли L4, что также подтверждает
Благодарности. Авторы выражают благодар
ся нашими данными по измерению сродства
ность В. Балобанову (ИБ РАН) за помощь в из
HsaSmAP к А18 РНК и Mant AMP.
мерениях, В.В. Врублевской (ИБК РАН ФИЦ
Суммируя полученные нами данные по
ПНЦБИ РАН) за предоставление антител и со
структуре белка HsaSmAP и его сродству к оли
трудникам ИБП РАН ФИЦ ПНЦБИ РАН за пре
гоРНК и отдельным нуклеотидам, можно утверж
доставление возможности проведения измерений
дать, что этот белок имеет только один, уридин
методом поверхностного плазмонного резонанса.
специфический, РНК связывающий сайт. Нес
Конфликт интересов. Авторы заявляют об от
мотря на то что короткая петля L4 открывает
сутствии конфликта интересов.
доступ к дистальной поверхности гептамера, бе
Соблюдение этических норм. Настоящая
лок HsaSmAP не обладает сродством к оли
статья не содержит описания каких либо иссле
го(А) РНК, поскольку такой сайт в их структуре
дований с участием людей или животных в каче
не формируется.
стве объектов.
СПИСОК ЛИТЕРАТУРЫ
1.
Murina, V. N., and Nikulin, A. D. (2011) RNA binding
7. Zhou, L., Hang, J., Zhou, Y., Wan, R., Lu, G., et al. (2013)
Sm like proteins of bacteria and archaea. Similarity and dif
Crystal structures of the Lsm complex bound to the 3′ end
ference in structure and function, Biochemistry (Moscow),
sequence of U6 small nuclear RNA, Nature, 506, 116 120,
76, 1434 1449, doi: 10.1134/S0006297911130050.
doi: 10.1038/nature12803.
2.
Mura, C., Randolph, P. S., Patterson, J., and Cozen, A. E.
8. Roth, A. J., Shuman, S., and Schwer, B. (2018) Defining
(2013) Archaeal and eukaryotic homologs of Hfq, RNA
essential elements and genetic interactions of the yeast
Biol., 10, 636 651, doi: 10.4161/rna.24538.
Lsm2 8 ring and demonstration that essentiality of Lsm2
3.
Lekontseva, N. V., Stolboushkina, E. A., and Nikulin,
8 is bypassed via overexpression of U6 snRNA or the U6
A. D. (2021) Diversity of LSM Family proteins: similarities
snRNP subunit Prp24, RNA, 24, 853 864, doi: 10.1261/
and differences, Biochemistry (Moscow), 86, S38 S49,
rna.066175.118.
doi: 10.1134/S0006297921140042.
9. Pomeranz Krummel, D. A., Oubridge, C., Leung,
4.
Sauter, C., Basquin, J., and Suck, D. (2003) Sm like pro
A. K. W., Li, J., and Nagai, K. (2009) Crystal structure of
teins in Eubacteria: the crystal structure of the Hfq protein
human spliceosomal U1 snRNP at 5.5 Å resolution,
from Escherichia coli, Nucleic Acids Res., 31, 4091 4098,
Nature, 458, 475 480, doi: 10.1038/nature07851.
doi: 10.1093/nar/gkg480.
10. Wilusz, C. J., and Wilusz, J. (2005) Eukaryotic Lsm pro
5.
Sauer, E. (2013) Structure and RNA binding properties of
teins: lessons from bacteria, Nat. Struct. Mol. Biol., 12,
the bacterial LSm protein Hfq, RNA Biol., 10, 610 618,
1031 1036, doi: 10.1038/nsmb1037.
doi: 10.4161/rna.24201.
11. Nielsen, J. S., Bøggild, A., Andersen, C. B. F. F.,
6.
Sharif, H., and Conti, E. (2013) Architecture of the Lsm1
Nielsen, G., Boysen, A., et al. (2007) An Hfq like protein
7 Pat1 complex: a conserved assembly in eukaryotic
in archaea: crystal structure and functional characteriza
mRNA turnover, Cell Rep., 5, 283 291, doi: 10.1016/j.celrep.
tion of the Sm protein from Methanococcus jannaschii,
2013.10.004.
RNA, 13, 2213 2223, doi: 10.1261/rna.689007.
БИОХИМИЯ том 86 вып. 7 2021
РОЛЬ ПЕТЛИ L4 В Lsm БЕЛКАХ
995
12.
Nikulin, A., Mikhailina, A., Lekontseva, N.,
20. Vagin, A., and Lebedev, A. (2015) MoRDa, an automatic
Balobanov, V., Nikonova, E., and Tishchenko, S. (2017)
molecular replacement pipeline, Acta Crystallogr., A71,
Characterization of RNA binding properties of the
s19 s19, doi: 10.1107/S2053273315099672.
archaeal Hfq like protein from Methanococcus jannaschii,
21. Afonine, P. V, Grosse Kunstleve, R. W., Echols, N.,
J. Biomol. Struct. Dyn., 35, 1615 1628, doi: 10.1080/
Headd, J. J., Moriarty, N. W., et al. (2012) Towards auto
07391102.2016.1189849.
mated crystallographic structure refinement with
13.
Törö, I., Basquin, J., Teo Dreher, H., and Suck, D. (2002)
phenix.refine, Acta Crystallogr., D68, 352 367, doi: 10.1107/
Archaeal Sm proteins form heptameric and hexameric
S0907444912001308.
complexes: crystal structures of the sm1 and sm2 proteins
22. Adams, P. D., Afonine, P. V., Bunkóczi, G., Chen, V. B.,
from the hyperthermophile Archaeoglobus fulgidus, J. Mol.
Davis, I. W., et al. (2010) PHENIX: a comprehensive
Biol., 320, 129 142, doi: 10.1016/j.biochi.2020.05.001.
Python based system for macromolecular structure solu
14.
Kilic, T., Sanglier, S., Van Dorsselaer, A., and Suck, D.
tion, Acta Crystallogr., D66, 213221, doi: 10.1107/
(2006) Oligomerization behavior of the archaeal Sm2 type
S0907444909052925.
protein from Archaeoglobus fulgidus, Protein Sci., 15, 2310
23. Emsley, P., and Cowtan, K. (2004) Coot: model building
2317, doi: 10.1016/j.biochi.2020.05.001.
tools for molecular graphics, Acta Crystallogr., D60, 2126
15.
Lekontseva, N., Mikhailina, A., Fando, M.,
2132, doi: 10.1107/S0907444904019158.
Kravchenko, O., Balobanov, V., et al. (2020) Crystal
24. Törö, I., Thore, S., Mayer, C., Basquin, J., Séraphin, B.,
structures and RNA binding properties of Lsm proteins
and Suck, D. (2001) RNA binding in an Sm core domain:
from archaea Sulfolobus acidocaldarius and Methanococ$
X ray structure and functional analysis of an archaeal Sm
cus vannielii: similarity and difference of the U binding
protein complex, EMBO J.,
20,
22932303,
mode, Biochimie, 175, 1 12, doi: 10.1016/j.biochi.2020.
doi: 10.1093/emboj/20.9.2293.
05.001.
25. Murina, V., Lekontseva, N., and Nikulin, A. (2013) Hfq
16.
Balobanov, V., Lekontseva, N., Mikhaylina, A., and
binds ribonucleotides in three different RNA binding sites,
Nikulin, A. (2020) Use of fluorescent nucleotides to map
Acta Crystallogr., D69,
15041513, doi:
10.1107/
RNA binding sites on protein surface, Methods Mol. Biol.,
S090744491301010X.
2113, 251 262, doi: 10.1007/978 1 0716 0278 2_17.
26. Thore, S., Mayer, C., Sauter, C., Weeks, S., and Suck, D.
17.
Nemchinova, M., Balobanov, V., Nikonova, E.,
(2003) Crystal structures of the Pyrococcus abyssi Sm core
Lekontseva, N., Mikhaylina, A., et al. (2017) An experi
and its complex with RNA. Common features of RNA
mental tool to estimate the probability of a nucleotide pres
binding in archaea and eukarya, J. Biol. Chem., 278, 1239
ence in the crystal structures of the nucleotide-protein
1247, doi: 10.1074/jbc.M207685200.
complexes, Protein J., 36, 157 165, doi: 10.1016/j.biochi.
27. Mura, C., Phillips, M., Kozhukhovsky, A., and
2020.05.001.
Eisenberg, D. (2003) Structure and assembly of an aug
18.
Kabsch, W. (2010) XDS, Acta Crystallogr., D66, 125 132,
mented Sm like archaeal protein 14 mer, Proc. Natl. Acad.
doi: 10.1107/S0907444909047337.
Sci. USA, 100, 4539 4544, doi: 10.1073/pnas.0538042100.
19.
Winn, M. D., Ballard, C. C., Cowtan, K. D., Dodson,
28. Mura, C., Kozhukhovsky, A., Gingery, M., and
E. J., Emsley, P., et al. (2011) Overview of the CCP4 suite
Phillips, M. (2003) The oligomerization and ligand bind
and current developments, Acta Crystallogr., D67, 235 242,
ing properties of Sm like archaeal proteins (SmAPs),
doi: 10.1107/S0907444910045749.
Protein Sci., 12, 832 847, doi: 10.1110/ps.0224703.ensure.
STRUCTURE AND RNA BINDING PROPERTIES OF LSM PROTEIN
FROM Halobacterium salinarum
M. S. Fando, A. O. Mikhaylina, N. V. Lekontseva, S. V. Tishchenko, and A. D. Nikulin*
Institute of Protein Research of the Russian Academy of Sciences, 142290 Pushchino,
Moscow Region, Russia; e$mail: nikulin@vega.protres.ru
The structure and the RNA binding properties of the Lsm protein from Halobacterium salinarum have been deter
mined. A distinctive feature of this protein is the presence of a short L4 loop connecting the β3 and β4 strands. Since
bacterial Lsm proteins (also called Hfq proteins) have a short L4 loop and form hexamers, whereas archaeal Lsm pro
teins (SmAP) have a long L4 loop and form heptamers, it has been suggested that the length of the L4 loop may affect
the quaternary structure of Lsm proteins. Moreover, the L4 loop covers the region of SmAP corresponding to one of
the RNA binding sites in Hfq, and thus can affect the RNA binding properties of the protein. Our results show that
the SmAP from H. salinarum forms heptamers and possesses the same RNA binding properties as homologous pro
teins with the long L4 loop. Therefore, the length of the L4 does not govern the number of monomers in the protein
particles and does not affect the RNA binding properties of Lsm proteins.
Keywords: Lsm proteins, SmAP, protein quaternary structure, protein RNA binding properties, small regulatory
RNA, sRNA, Halobacterium salinarum
БИОХИМИЯ том 86 вып. 7 2021
4*