БИОХИМИЯ, 2022, том 87, вып. 12, с. 2046 - 2064
УДК 578.834.1;616.9
БИОЛОГИЯ КОРОНАВИРУСА SARS-CoV-2
Обзор
© 2022 Р.Н. Мингалеева1*, Н.А. Нигматулина2, Л.М. Шарафетдинова1, А.М. Ромозанова1,
А.Г. Габдулхакова1, Ю.В. Филина1, Р.Ф. Шавалиев2, А.А. Ризванов1, Р.Р. Мифтахова1
1 Казанский (Приволжский) федеральный университет,
420008 Казань, Россия; электронная почта: rimma.mingaleeva@gmail.com
2 Республиканская клиническая больница Министерства здравоохранения Республики Татарстан,
420064 Казань, Россия
Поступила в редакцию 13.07.2022
После доработки 05.10.2022
Принята к публикации 17.10.2022
Новая коронавирусная инфекция Covid-19, возникшая в конце 2019 г. в Китае, вызвала сильнейший
социальный и экономический кризис, затронувший весь мир. Высокая частота ошибок в реплика-
ции РНК-вирусов, зоонозный характер передачи, высокая трансмиссивность позволили бета-коро-
навирусам вызвать уже третью пандемию в мире с начала 2003 г.: SARS-CoV - в 2003 г, MERS-CoV -
в 2012 г и SARS-CoV-2 - в 2019 г. Последняя пандемия объединила научное сообщество и послужила
мощным толчком в изучении биологии коронавирусов: были выявлены новые пути проникновения
вируса в организм человека, изучены особенности репликативного цикла, выявлены новые функции
белков коронавируса. Стоит учитывать, что пандемия сопровождалась необходимостью в получе-
нии и публикации результатов в короткие сроки, что привело к появлению массива противоречивых
данных и низкой воспроизводимости результатов исследований. Нами была систематизирована и
проанализирована научная литература, проведена фильтрация результатов по достоверности приме-
няемых методов анализа и подготовлен обзор, описывающий молекулярные механизмы функциони-
рования коронавируса SARS-CoV-2. В работе рассмотрена организация генома вируса SARS-CoV-2,
механизмы экспрессии его генов и входа вируса в клетку, приведена информация по ключевым мута-
циям, характеризующим разные варианты вируса, и их вклад в патогенез заболевания.
КЛЮЧЕВЫЕ СЛОВА: SARS-CoV-2, Covid-19, S-белок, мутация, VOC.
DOI: 10.31857/S032097252212020X, EDN: NIWMWX
ВВЕДЕНИЕ
тяжёлых инфекций нижних дыхательных пу-
тей, включая бронхит, пневмонию и атипичную
Коронавирусы определяют как группу род-
пневмонию - тяжёлый острый респираторный
ственных РНК-содержащих вирусов, пора-
синдром (SARS). Вирус птичьего инфекцион-
жающих млекопитающих и птиц. Инфицируя
ного бронхита (IBV) стал первым обнаружен-
человека, эти вирусы вызывают заболевания
ным коронавирусом [1]. Коронавирусы, пора-
дыхательных путей различной степени тяже-
жающие человека, HCoV-229E и HCoV-OC43,
сти: от инфекций верхних дыхательных путей,
впервые были обнаружены в 1966 и 1967 гг.
сходных с симптомами сезонных простуд, до В 2003 г. в Китае был обнаружен зоонозный
Принятые сокращения: ACE2 - ангиотензин-превращающий фермент 2 (angiotensin-converting enzyme 2); DMV -
двухмембранные везикулы, специальные органеллы репликации (double-membrane vesicle); E - белок оболочки; HCoV -
коронавирус человека (Human coronavirus); HE - гемагглютинин-эстераза; HR - гептадный повтор (heptad repeat);
IBV - вирус птичьего инфекционного бронхита (infectious bronchitis virus); M - мембранный белок; MERS - ближнево-
сточный респираторный синдром (Middle East respiratory syndrome); Mpro - 3CL-подобная протеаза; N - нуклеокапсид-
ный белок; nsp - неструктурные белки (non-structural proteins); ORF - открытая рамка считывания; pp - полипротеин;
RBD - рецептор-связывающий домен (receptor-binding domain); S - Spike-белок; S1 и S2 - субъединицы S-белка; SARS -
тяжёлый острый респираторный синдром (severe acute respiratory syndrome); TRS - последовательности, регулирующие
транскрипцию (transcription-regulating sequence); VOC - варианты, вызывающие озабоченность (variant of concern).
* Адресат для корреспонденции.
2046
БИОЛОГИЯ SARS-CoV-2
2047
коронавирус SARS-CoV, распространение ко-
рус, поражающий собак (СCoV), кошек (FCoV),
торого привело к эпидемии с охватом 8000 за-
и коронавирус, вызывающий трансмиссив-
документированных случаев и коэффициен-
ный гастроэнтерит у свиней (TGEV), име-
том летальности 10%. Это породило всплеск
ют один прототип - Alphacoronavirus 1. Пока-
острого интереса к коронавирусам, в результа-
зано, что вспышка синдрома острой диареи
те которого впоследствии было выявлено ещё
свиней (SADS) была вызвана коронавирусом
два вируса: HCoV-NL63 (Нидерланды, 2004 г.)
SADS-CoV - потомком коронавируса лету-
и HCoV-HKU1 (Гонконг, 2004 г.), которые еже-
чих мышей BatCoV-HKU2, принадлежащего
годно циркулируют по всему миру. В 2012 г. в
к роду Alphacoronavirus. Сходство коронавиру-
Саудовской Аравии был выявлен второй вы-
са альпак (ACoV), относящегося также к роду
сокопатогенный коронавирус зоонозного про-
Alphacoronavirus, с вирусом человека HCoV-
исхождения, MERS-CoV, который до сих пор
229E наводит на мысль о зоонозном происхож-
периодически детектируется у людей. Согласно
дении последнего. Считается, что HCoV-OC43
данным на июль 2022 г, MERS-CoV был под-
произошёл от бычьего коронавируса (BCoV)
тверждён у 2591 человека. Он поражает лёгкие и
примерно в 1890-х гг. [4].
вызывает тяжёлые клинические проявления, ко-
Кошки, хорьки, собаки и другие млекопи-
эффициент летальности заболевания - до 35%
тающие заражаются не только коронавируса-
ми, характерными для их видов, но также ин-
Седьмым обнаруженным коронавирусом,
фицируются SARS-CoV-2 [4]. Предполагают,
поражающим людей, стал SARS-CoV-2, вы-
что SARS-CoV и SARS-CoV-2, попали в попу-
зывающий заболевание Covid-19. Согласно
ляцию людей от летучих мышей через проме-
данным Университета Джона Хопкинса (Johns
жуточных хозяев. Высокая степень гомологии
Hopkins University Coronavirus Resource Center,
генома коронавирусов пальмовых циветт и
SARS-CoV указывает на большую вероятность
болевших Covid-19, по состоянию на сен-
перехода вируса к человеку именно от них [5].
тябрь 2022 г., составило 612 млн человек, сре-
Происхождение SARS-CoV-2 более спорно.
ди которых умерло 6,5 млн. В России, по дан-
Промежуточными хозяевами для этого виру-
ным на март 2022 г., Covid-19 зарегистрирован
са могли служить панголины или другие виды
у 20 млн человек, 378 тыс. человек скончалось
животных, но, возможно, вирус перешёл к че-
(коэффициент летальности - 1,9%). Масшта-
ловеку непосредственно от летучих мышей [6].
бы распространения SARS-CoV-2, лёгкость
Опубликованы работы, согласно которым весь
передачи вируса от человека к человеку, пред-
рецептор-связывающий мотив вируса SARS-
положительное существование промежуточ-
CoV-2 мог быть введён путём рекомбинации с
ных межвидовых форм новых коронавирусов
коронавирусами панголинов [7]. В недавних ис-
диктуют потребность в разработке методов де-
следованиях показано, что SARS-CoV-2 может
текции и борьбы с ними [2].
представлять собой мозаичный геном, который
объединяет геномы трёх коронавирусов лету-
чих мышей (RmYN02, RpYN06 и RaTG13), об-
СЕМЕЙСТВО КОРОНАВИРУСОВ
наруженных в китайской провинции Ухань [8].
В целом, летучие мыши имеют свыше 1300 ви-
Коронавирусы относятся к порядку
дов, образуют огромные стаи (до нескольких
Nidovirales, семейству Coronaviridae, подсемей-
млн особей в группе), перемещаются на боль-
ству Orthocoronavirineae, внутри которого вы-
шие расстояния и присутствуют на всех конти-
деляют 4 рода коронавирусов: Alphacoronavirus,
нентах, приобретая и распространяя множество
Betacoronavirus, Gammacoronavirus и Deltacorona-
вирусов. Среди идентифицированных вирусов
virus. Представители первых двух родов инфи-
летучих мышей как минимум 60 могут быть па-
цируют исключительно млекопитающих, тогда
тогенными для человека [9]. Предполагают, что
как представители двух последних имеют бо-
историю происхождения большинства корона-
лее широкий спектр хозяев, включая птиц [3].
вирусов человека можно проследить до вирусов
К роду Alphacoronavirus относятся HCoV-229E и
летучих мышей. Это предположение основано
HCoV-NL63; HCoV-HKU1, SARS-CoV, MERS-
на двух фактах: (1) разнообразие и количество
CoV, HCoV-OC43 и SARS-CoV-2 являются пред-
коронавирусов летучих мышей намного превы-
ставителями Betacoronavirus.
шает разнообразие этих вирусов у других мле-
Для вирусов, имеющих ветеринарное зна-
копитающих; (2) были выявлены коронавирусы
чение, описано множество случаев межвидово-
летучих мышей, очень похожие на вирусы чело-
го переноса коронавирусной инфекции между
века (Bat-229E-подобный, Bat-NL63-подобный
млекопитающими. Считается, что коронави-
и Bat-SARS-подобный вирусы). Вероятность
БИОХИМИЯ том 87 вып. 12 2022
2048
МИНГАЛЕЕВА и др.
перескока этих вирусов от вида к виду опреде-
АРХИТЕКТУРА КОРОНАВИРУСОВ
ляется, во-первых, совместимостью вируса с
рецепторами, расположенными на клетках ор-
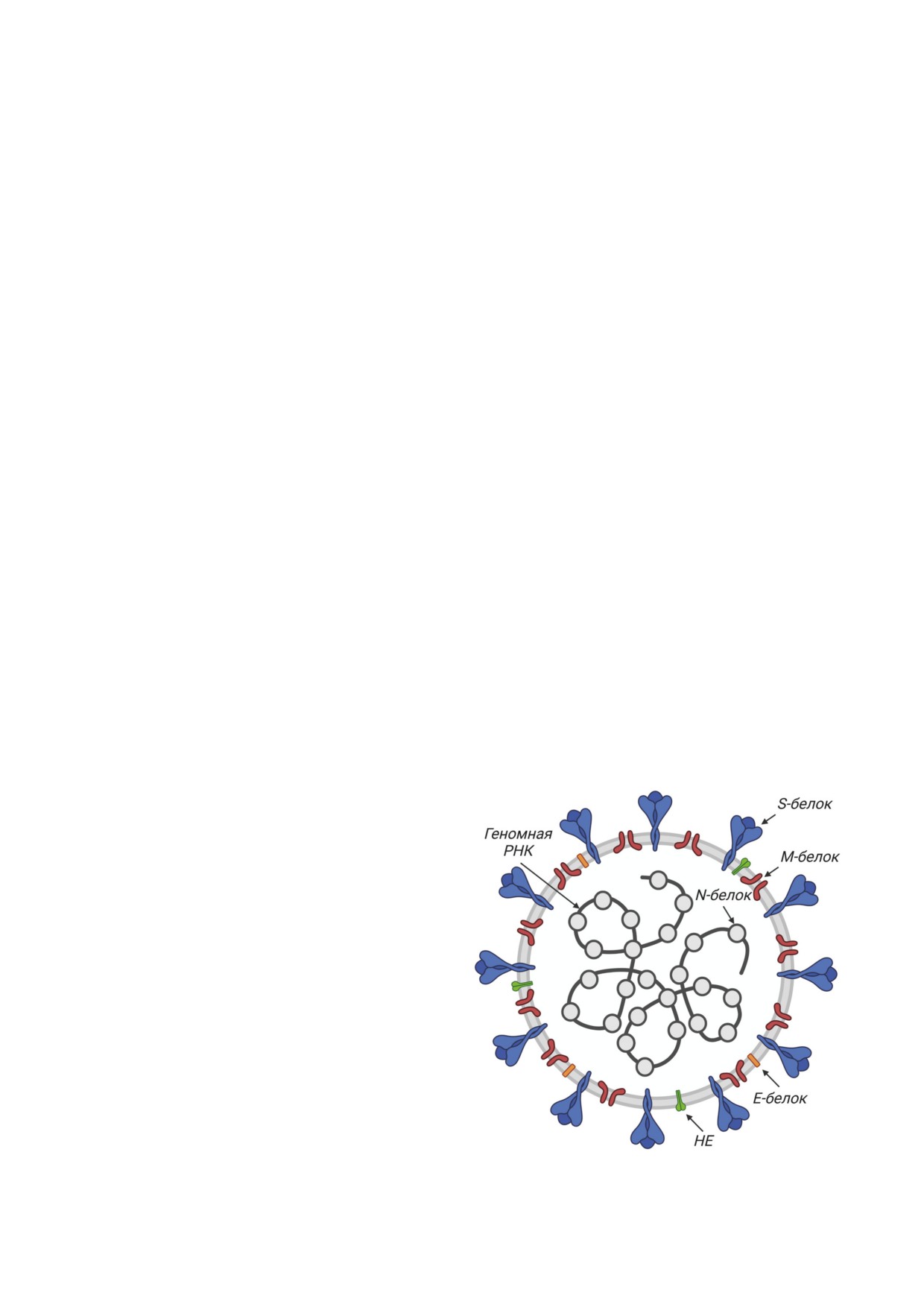
Коронавирусы имеют сферическую фор-
ганизма-хозяина (восприимчивостью клеток);
му вириона диаметром 80-120 нм, обрамлён-
во-вторых, способностью вируса реплициро-
ную так называемыми «шипами» - тримерами
ваться в клетках организма-хозяина (пермис-
S-белка (рис. 1). У коронавирусов HCoV-OC43
сивностью клеток); в-третьих, доступностью
и HCoV-HKU1 в формировании так называ-
клеток, восприимчивых к вирусу; в-четвёртых,
емой «короны» участвует также гемагглюти-
способностью уклоняться от иммунного ответа
нин-эстераза (HE). Вирусная оболочка поддер-
хозяина. Центральную роль в процессе пере-
живается мембранным белком (М) и содержит
скока коронавирусов к человеку играет вирус-
небольшие вкрапления белка оболочки (E).
ный белок S (Spike) [10].
Под оболочкой вириона находится спираль-
SARS-CoV-2 впервые был обнаружен в де-
но-симметричный нуклеокапсид, образован-
кабре 2019 г. Отсутствие иммунитета к новой
ный одноцепочечной геномной РНК, покры-
инфекции позволило вирусу беспрепятствен-
той нуклеокапсидным белком (N) [12].
но размножаться и, как следствие, мутировать.
S и HE - трансмембранные белки, отве-
В марте 2020 г. Европа превратилась в эпи-
чающие за проникновение вируса в клетку.
центр пандемии нового коронавируса - по-
S-Белок (128-160 кДа) определяет тропизм ви-
явился вариант вируса, имеющий преимуще-
руса, он связывается с рецепторами, локали-
ство перед вирусом дикого типа: единственная
зованными на поверхности клетки-мишени.
мутация D614G в S-белке позволила сделать
У вирусов HCoV-OC43 и HCoV-HKU1 белок
вирус более трансмиссивным. В апреле 2020 г.
HE (48-67 кДа) облегчает прикрепление виру-
был задокументирован 1 млн случаев забо-
са к клетке [13]. Для некоторых коронавирусов
левания Covid-19. Вариант вируса с D614G
показано, что присутствие S-белка на поверх-
продолжил накапливать мутации, направлен-
ности инфицированной клетки способствует её
ные на быстрое изменением антигена (S-бел-
слиянию с соседней неинфицированной клет-
ка) и повышение трансмиссивности вируса.
кой. Благодаря такой стратегии образуются ги-
С декабря 2020 г. стали появляться новые ва-
гантские многоядерные клетки, или синцитий,
рианты, которые были классифицированы
функцией которого является облегчение рас-
Всемирной Организацией Здравоохранения
пространения вируса между клетками [14].
как варианты, вызывающие озабоченность
N-Белок определяет архитектуру генома ви-
(variant of concern, VOC). Вариант B.1.1.7 (Аль-
руса, образуя нуклеокапсид с геномной РНК.
фа) впервые был обнаружен 14 декабря 2020 г.
Локализуясь в области эндоплазматического
в Великобритании. Следующим вариантом
ретикулума и аппарата Гольджи, он участвует
стал B.1.351 (Бета) - он был обнаружен 18 дека-
в сборке и отпочковывании вирусных частиц.
бря 2020 г. в Южной Африке. Третий вариант,
P.1 (B.1.1.28.1, Гамма), выявили 6 января 2021 г.
в Токио у туриста из Бразилии. В декабре 2020 г.
появился вариант B.1.617.2 (Дельта), который
заместил собой все остальные варианты вируса.
На смену варианту Дельта в ноябре 2021 г. при-
шёл, обнаруженный в Южной Африке, вариант
Омикрон (B.1.1.529) [11]. Подварианты Омикро-
на BA.1, BA.2 и BA.3 запустили четвёртую волну
Covid-19, следом за которой пришла уже пятая,
бушующая и сейчас, волна, сформированная
подвариантами BA.4 и BA.5, среди которых
BA.5 пока занимает лидирующее положение.
По данным Европейского центра предотвраще-
ния и контроля над заболеваемостью (ECDC),
к сентябрю 2022 г. в категории VOC остались
подварианты Омикрона BA.2, BA.4 и BA.5, а
пристальное внимание уделяется подвариантам
BA.4, BA.5, BA.2.75 и BA 2.12.1. На сайте https://
covariants.org читатель сможет ознакомиться с
филогенетическим древом возникновения ос-
Рис. 1. Строение коронавируса: указаны структурные бел-
новных вариантов SARS-CoV-2.
ки N, S, M, E, HE и геномная РНК; объяснения в тексте
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2049
Считается, что N-белок также участвует в регуля-
сборки вируса. Взаимодействия M и E достаточ-
ции и модулировании репликации и транскрип-
но для производства и высвобождения вирус-
ции. Было показано, что он способен не только
ных частиц [2, 13].
неспецифически связывать РНК, но и специфи-
E-Белок является небольшим интеграль-
чески взаимодействовать с некоторыми после-
ным белком (8-12 кДа), он заякорен в мембра-
довательностями, регулирующими транскрип-
ну трансмембранным доменом, его эктодомен
цию (TRS). Нуклеокапсидные белки различных
гликозилирован, а эндодомен пальмитирован.
коронавирусов взаимодействуют с множеством
Для вирусов SARS-CoV и IBV показано, что
других белков, в том числе с nsp 3 (неструктур-
E-белок образует гомопентамеры, действующие
ный белок 3) и РНК-геликазой DDX1 клетки-
как ионные каналы. Такие структуры модули-
хозяина. Предполагается, что комплекс, образо-
руют процесс высвобождения вирионов, при-
ванный DDX1 и фосфорилированным нуклео-
нимая активное участие в заражении клетки.
капсидным белком, контролирует баланс между
Интересно, что E-белок в избытке образуется
репликацией и транскрипцией путём модули-
внутри инфицированной клетки, и только часть
рования уровня переключения матрицы на по-
его включается в оболочку вириона, а большая
следовательности TRS-B. N-Белок SARS-CoV-2
часть находится там, где собираются и отпочко-
способствует ассоциации РНК с комплексом
вываются вирусные частицы [14].
nsp 7-nsp 8-nsp 12 и, вероятно, инициации ре-
пликации и транскрипции [15].
М-Белок является коровым мембранным
СТРОЕНИЕ ГЕНОМА КОРОНАВИРУСОВ
белком. Он встроен в липидный бислой тре-
И МЕХАНИЗМЫ ЕГО ЭКСПРЕССИИ
мя трансмембранными доменами, гликозили-
рованный эктодомен белка выступает наружу.
Коронавирусы имеют самые длинные не-
М-Белок поддерживает вирусную оболочку,
сегментированные геномы среди РНК-содер-
определяет форму и размер вирусного капсида,
жащих вирусов [16]. Так, геном SARS CoV-2
поскольку именно он взаимодействует с други-
размером от 26 до 32 т.н. кодирует информа-
ми структурными белками. Взаимодействие M
цию о 16 неструктурных (nsp 1-16), 4 структур-
и S необходимо для удержания S-белка в про-
ных (S, M, N и E) и 11 дополнительных белках
межуточном компартменте между ЭПР и аппа-
(ORF3a, ORF3b, ORF3c, ORF3d, ORF6, ORF7a,
ратом Гольджи (ERGIC) и включения его в но-
ORF7b, ORF8, ORF9b, ORF9c и ORF10) [12,
вые вирионы. Связывание M и N стабилизирует
17]. Носителем генетической информации ко-
нуклеокапсид, а также внутренний кор вириона
ронавирусов является смысловая одноцепо-
и в конечном счёте способствует завершению
чечная РНК. Каждый вирусный транскрипт,
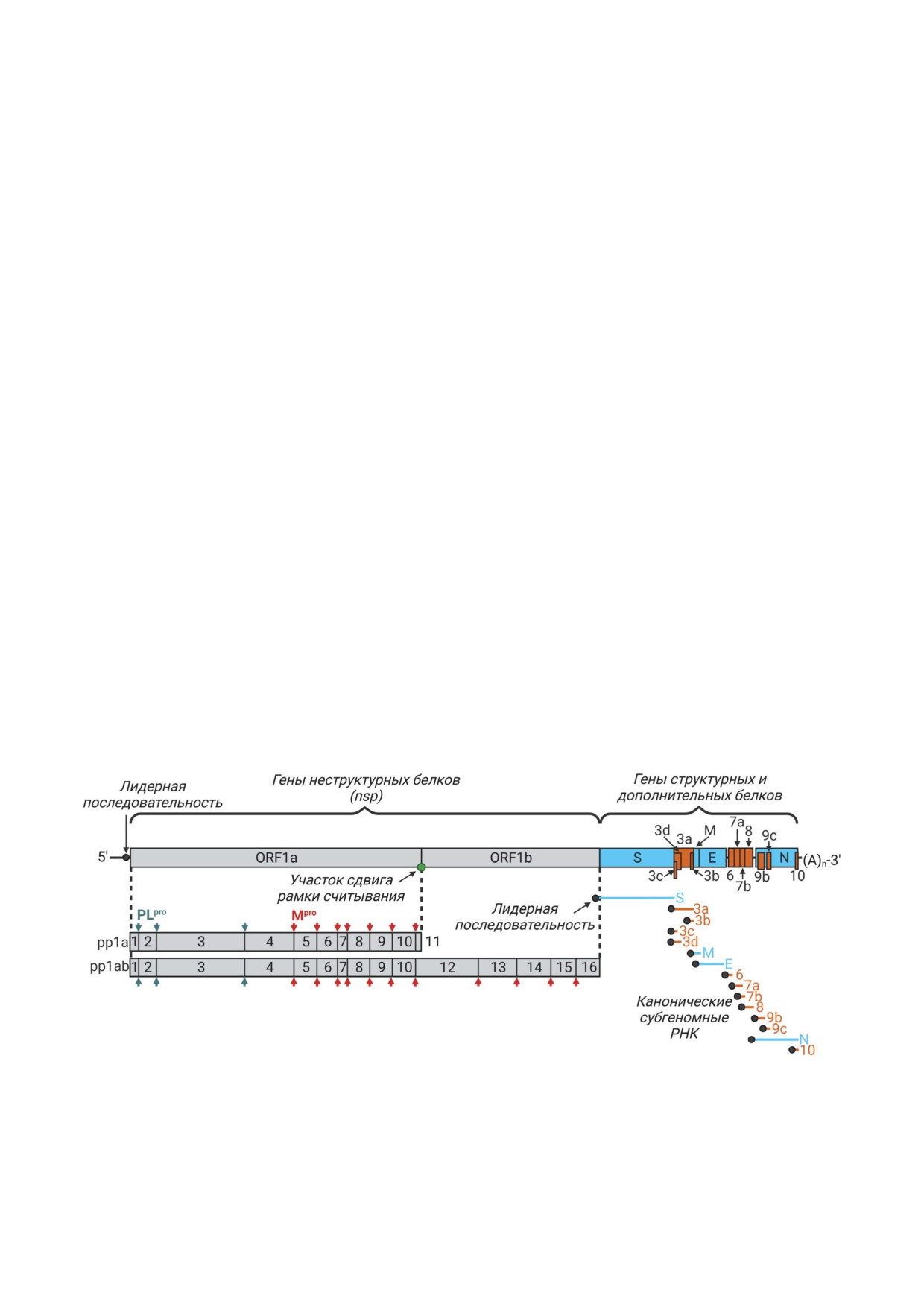
Рис. 2. Организация генома коронавируса на примере SARS-CoV-2. Наличие 5′-кэп и 3′-поли(А)-хвоста на концах ге-
номной РНК позволяет сразу транслировать в клетке неструктурные белки с рамок считывания ORF1a и ORF1b. Рам-
ки разделены между собой участком сдвига рамки считывания. Результатом трансляции являются две полипротеино-
вых цепи pp1a и pp1ab. Отдельные неструктурные белки образуются в результате протеолитического процессинга pp1a
и pp1ab папаин-подобной протеазой PLpro и 3CL-подобной протеиназой Mpro. Гены структурных и дополнительных
белков транскрибируются в набор субгеномных мРНК. Геномная РНК и все субгеномные мРНК содержат одинако-
вую лидерную последовательность на своих 5′-концах. Неструктурные белки и их гены обозначены серым цветом, гены
структурных и дополнительных белков и их транскрипты обозначены голубым и оранжевым цветом соответственно
19
БИОХИМИЯ том 87 вып. 12 2022
2050
МИНГАЛЕЕВА и др.
как и геномная РНК, кэпированы с 5′-конца и
своего генома. Так, неструктурные белки об-
полиаденилированы - с 3′-конца. Такое стро-
разуются сразу после проникновения вируса
ение генома позволяет рибосоме клетки рас-
в клетку в виде двух крупных полипротеинов,
познавать геномную РНК как мРНК и сразу
которые в дальнейшем подвергаются процес-
включать её в процесс трансляции вирусных
сингу. Для транскрипции структурных и до-
белков без необходимости упаковки в вирион
полнительных белков коронавирусы образуют
транскрипционного комплекса. Кроме того,
специальные органеллы репликации - DMV,
все вирусные РНК имеют специальную ли-
которые представляют собой ретикулове-
дерную последовательность на своём 5′-конце,
зикулярную сеть двухмембранных везикул
благодаря которой происходит разграничение
с взаимосвязанными внешними мембрана-
вирусных и клеточных РНК (рис. 2) [18].
ми (рис. 3) [19]. Вирусы производят DMV из
При адаптации под процессы, происходя-
мембран ЭПР клетки-хозяина путём их по-
щие в эукариотической клетке, коронавирусы
следовательных перестроек под регуляцией
выработали разные механизмы трансляции
неструктурных белков nsp 3, nsp 4 и nsp 6, а
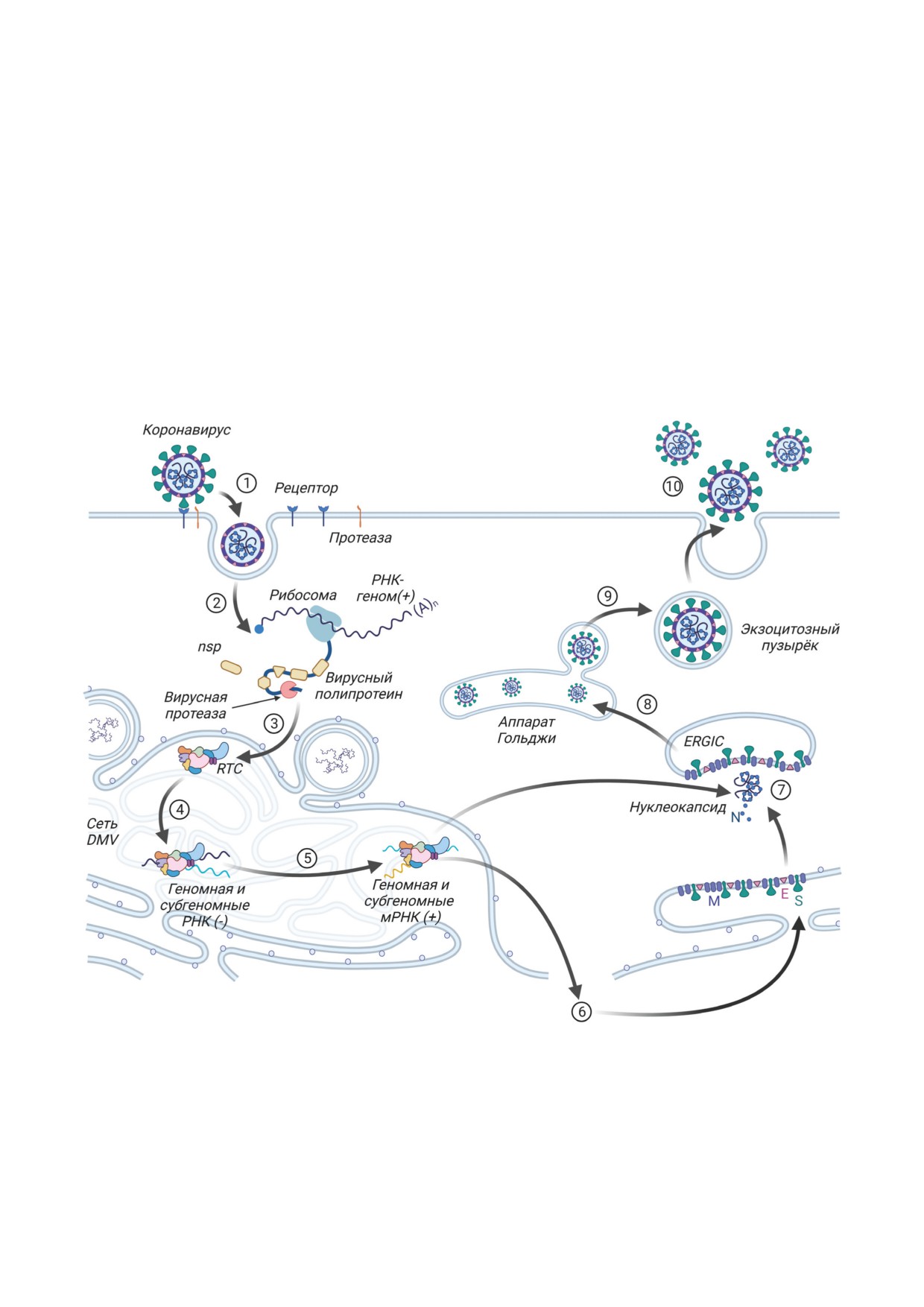
Рис. 3. Жизненный цикл коронавирусов. Поверхность вируса покрыта S-белком, который взаимодействует с рецеп-
тором и активирует слияние вируса с мембраной клетки после своего расщепления протеазой, находящейся на по-
верхности клетки (1). Геномная РНК, попадая внутрь клетки, сразу распознаётся рибосомой, и происходит трансля-
ция полипротеинов и их процессинг до отдельных неструктурных белков (2). В мембранах эндоплазматической сети
происходит формирование двухмембранных везикул (DMV), внутри которых происходит сборка репликазно-тран-
скрипционного комплекса (RTC) (3). Геномная смысловая РНК сначала переводится в антисмысловую форму с об-
разованием геномной и субгеномных РНК (4), а затем в смысловую форму геномной РНК и субгеномных мРНК (5).
В эндоплазматической сети с субгеномных мРНК происходит трансляция структурных и дополнительных белков (6).
Геномная РНК взаимодействует с N-белком, образуя нуклеокапсид (7), который, объединяясь со структурными бел-
ками, формирует вирион (8). Зрелый вирион (9) выводится из клетки путём экзоцитоза (10)
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2051
также белков клетки-хозяина [17]. В реплика-
НЕСТРУКТУРНЫЕ БЕЛКИ:
тивно-транскрипционных комплексах DMV
ВНУТРИКЛЕТОЧНЫЙ СИНТЕЗ И
проходят процессы репликации и транскрип-
ФУНКЦИИ
ции субгеномных РНК коронавирусов [20].
Образование органелл репликации в целом
Две трети генома с 5′-конца занимают
свойственно РНК-вирусам со смысловой
рамки считывания ORF1a и ORF1b. В них за-
цепью генома. Оно оправдано с нескольких
кодирована информация обо всех неструк-
позиций: (1) происходит скопление в одном
турных белках вируса (для SARS CoV-2 - это
месте всех факторов вируса и клетки-хозяина,
nsp 1-16), гены этих белков слиты между со-
необходимых для репликации и транскрип-
бой и не имеют стоп-кодонов, за исключением
ции; (2) промежуточные продукты репликации
участка на стыке рамок считывания. Неструк-
и транскрипции не выходят за пределы DMV и
турные белки определяют репликацию вируса
не распознаются системами клеточного про-
и сборку его транскрипционного комплекса, в
тивовирусного ответа; (3) идёт более строгая
котором проходит транскрипция, процессинг,
координация процессов репликации и тран-
модификация РНК, а также коррекция непра-
скрипции [21]. Наряду с трансляцией неструк-
вильно встроенных нуклеотидов.
турных белков, в клетке сразу происходит на-
Трансляция ORF1a и ORF1b приводит к
копление полноразмерных антисмысловых
образованию двух полипептидных цепей (pp1a
геномных копий, которые используются как
и pp1ab). Стоп-кодон, расположенный на сты-
матрицы для образования новых смысловых
ке рамок считывания ORF1a и ORF1b, позво-
геномных РНК. Синтезированные смысловые
ляет рибосоме закончить синтез в этом месте
геномы используются либо для трансляции
с образованием pp1a (440-500 кДа). Иногда
большего количества неструктурных белков и
рибосома, дойдя до специальной регулятор-
репликативно-транскрипционных комплек-
ной последовательности (slippery sequence)
сов, либо упаковываются в новые вирионы.
X XXY YYZ (X - три одинаковых нуклеотида;
Кроме того, смысловая РНК используется для
Y - A/U; Z - A/С/G), расположенной непо-
транскрипции субгеномных РНК путём пре-
средственно около стоп-кодона ORF1a, стре-
рывистой транскрипции. Образуются сначала
мится выскользнуть из рамки считывания и
промежуточные антисмысловые субгеномные
перескочить на 1 нуклеотид назад. У корона-
РНК, которые
«переводятся» в смысловые
вирусов этот процесс усилен, поскольку сразу
субгеномные мРНК. Структурные и допол-
следом за регуляторной последовательностью
нительные белки синтезируются и переме-
находится стабильная РНК-структура (псевдо-
щаются сначала в ERGIC, а затем мигрируют
узел), натыкаясь на которую рибосома делает
в аппарат Гольджи, где происходит образо-
паузу, и вероятность перескока увеличивает-
вание зрелого вириона. Вирион отщепляется
ся (рис. 4). Если сдвиг рамки считывания про-
от клетки путём конститутивного экзоцитоза
изошёл, то образуется pp1ab (740-810 кДа), ко-
(рис. 3) [22, 23].
торый своим N-концом сходен с pp1a.
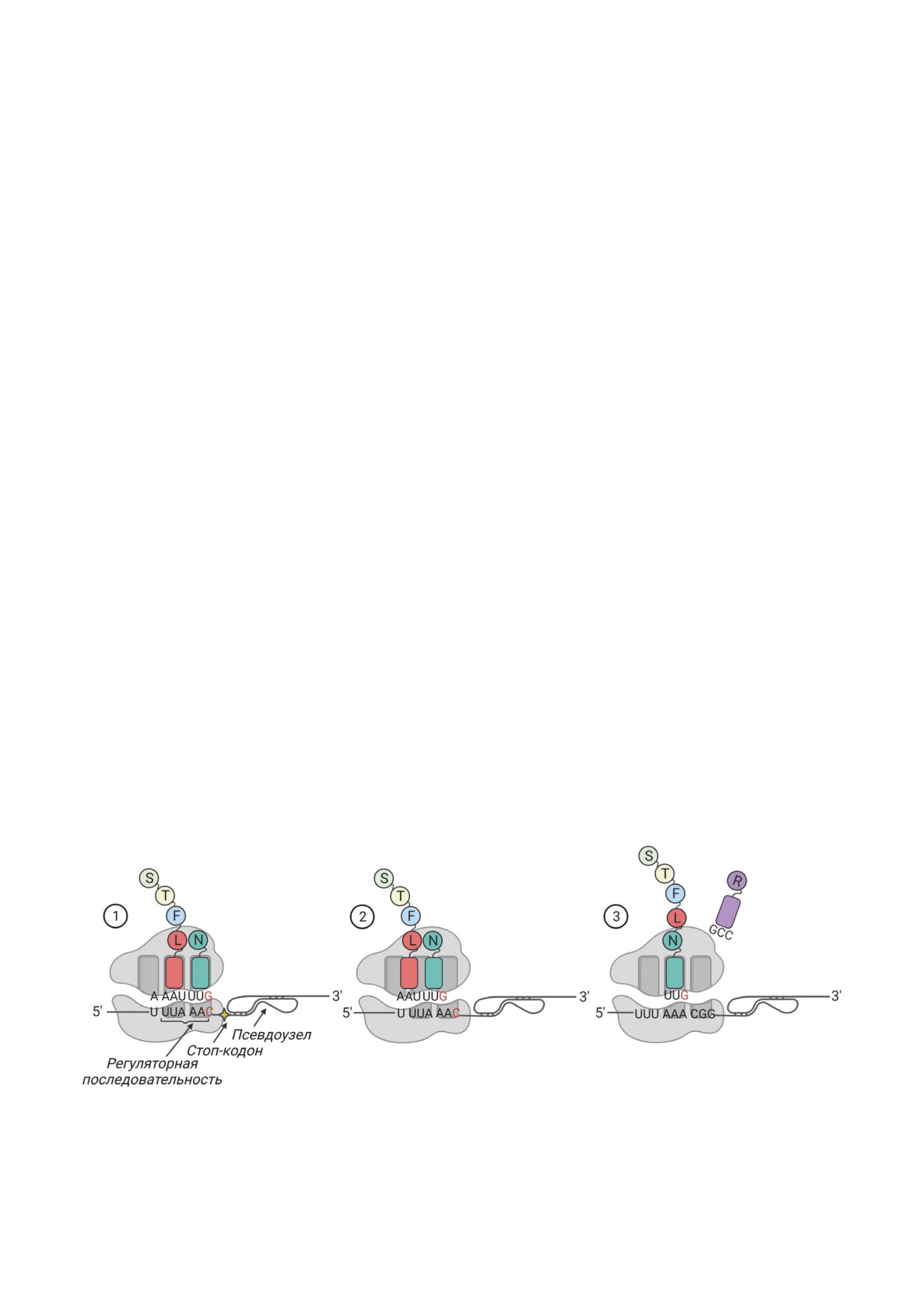
Рис. 4. Сдвиг рамки считывания при синтезе pp1ab SARS-CoV-2. Недалеко от регуляторной последовательности (slippery
sequence) располагается стоп-кодон, а следом за ним - стабильная РНК-структура (псевдоузел) (1). Когда рибосома
подходит к псевдоузлу, скорость трансляции замедляется, благодаря чему становится возможным перескок рибосомы
на 1 нуклеотид назад. Это приводит к изменению кодонного состава, что связано с исчезновением стоп-кодона (2).
Высвобожденный третий нуклеотид триплета становится первым в новом триплете. К нему подходит соответствующая
тРНК, и трансляция белка продолжается (3)
БИОХИМИЯ том 87 вып. 12 2022
19*
2052
МИНГАЛЕЕВА и др.
Расположенные внутри белки высвобожда-
ки-хозяина начинает деградировать с 5′-конца.
ются после протеолитического расщепления
При этом трансляция вирусных РНК продол-
двумя цистеиновыми протеазами - папаин-по-
жает идти, поскольку их 5′-концы защищены
добной протеазой PLpro и 3CL-подобной проте-
лидерными последовательностями [18].
иназой Mpro (major protease, 3CLpro), первая из
Nsp 2-16 составляют вирусный реплика-
которых находится внутри гена nsp 3, а вторая -
тивно-транскрипционный комплекс (RTC),
nsp 5. Папаин-подобная протеаза процессирует
они нацелены на определённые субклеточные
nsp 1, nsp 2, nsp 3, а также высвобождает N-ко-
участки, где происходит их взаимодействие с
нец nsp 4, тогда как Mpro отвечает за процессинг
факторами клетки-хозяина, что определяет ход
большинства неструктурных белков: C-конца
цикла репликации. Nsp 2-11 помогают накоп-
nsp 4 и белков nsp 5-16 (рис. 2) [23]. Но, пре-
лению вирусных RTC, моделируя внутрикле-
жде чем начать выполнять свои функции, эти
точные мембраны, участвуют в уклонении от
протеазы подвергаются аутолитическому про-
иммунной системы и обеспечивают кофактора-
цессингу [24, 25]. Полипротеин pp1a процесси-
ми для репликации. Процесс кэпирования РНК
руется до 11 белков, а pp1ab - до 15 белков [18].
протекает при участии nsp 10 (он выступает как
Образование нескольких белков с одной рамки
кофактор), nsp 13 (обладает 5′-трифосфатной
считывания позволяет ускорить выработку бел-
активностью), nsp 14 (является N7-метилтранс-
ков вируса при использовании моноцистрон-
феразой) и nsp
16 (имеет
2′-O-метилтранс-
ного принципа трансляции в эукариотической
феразную активность). В
3′-нетраслируемой
клетке.
области коронавирусов находится последо-
Расположение неструктурных белков и их
вательность AAUAAA, которая служит сигна-
процессинг, по-видимому, отражает необхо-
лом полиаденилирования для белка nsp 8 [26].
димость этих белков в процессе трансляции
Nsp 12-16 берут на себя основные фермен-
генома. Попав в клетку, вирус начинает её
тативные функции: участвуют в синтезе, мо-
«захват» - синтез большого числа pp1a. Когда
дификации и коррекции РНК (табл. 1) [27].
произошла модуляция работы клетки под ви-
Nsp 15, уникальная уридилат-специфичная эн-
русную систему, необходим синтез фермен-
дорибонуклеаза, укорачивает поли(U)-участки,
тов вируса - идёт сдвиг рамки считывания на
которые присутствуют на 5′-конце вирусной
стыке ORF1a и ORF1ab, синтезируется pp1ab.
антисмысловой РНК, что способствует уклоне-
Далее происходит сборка репликазно-тран-
нию от системы врождённого иммунитета [15].
скрипционного комплекса, транскрипция суб-
РНК-зависимая РНК-полимераза коро-
геномных РНК, их трансляция в структурные
навирусов представлена белком nsp 12. Сам
и дополнительные белки. Появляется возмож-
по себе он обладает минимальной полиме-
ность собирать вирионы и выходить из клетки.
разной активностью, которая существенно
В жизненном цикле коронавирусов очень
возрастает при взаимодействии с факторами
быстро появляется неструктурный белок nsp 1.
процессивности nsp 7 и nsp 8. Считается, что
Он отвечает за подавление экспрессии генов
связка nsp 12-nsp 7-nsp 8 представляет собой
клетки-хозяина. Связываясь с 40S-субъедини-
холофермент РНК-полимеразы, минималь-
цей рибосомы, он блокирует трансляцию не-
ный комплекс, необходимый для полимериза-
вирусных белков, в результате чего мРНК клет-
ции нуклеотидов [28]. Полимераза nsp 12 имеет
Таблица 1. Функции белков, принимающих участие в формировании комплекса РНК-зависимой РНК-полимеразы
Название
Участие в репликазно-транскрипционном комплексе
белка
nsp 7, nsp 8
субъединицы холофермента РНК-полимеразы
nsp 9
связывание с РНК и с репликазным комплексом (предположительно)
активаторная субъединица экзонуклеазной активности nsp 14 и метилтрансферазной активности nsp 16;
nsp 10
регуляция сдвига рамки считывания в рибосоме
nsp 12
РНК-зависимая РНК-полимераза, нуклеотидилтрансфераза
nsp 13
геликаза, РНК 5′-фосфатаза
nsp 14
3′→5′ экзорибонуклеаза, N7-метилтрансфераза
nsp 16
образование кэпа РНК, рибозо 2′-O-метилтрансфераза
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2053
высокую степень консервативности - более
95% гомологии среди коронавирусов.
С холоферментом РНК-полимеразы коро-
навирусов также взаимодействует белок nsp 14,
благодаря которому происходит кэпирова-
ние РНК за счёт его метилтрансферазной ак-
тивности. Другой очень важной его функцией
является способность исправлять неправиль-
но встроенные нуклеотиды за счёт своей 3′→5′
экзонуклеазной активности, не свойственной
другим РНК-содержащим вирусам. Способ-
ность к коррекции неправильно встроенных
нуклеотидов позволяет поддерживать целост-
ность огромного генома за счёт снижения ча-
стоты мутаций склонной к ошибкам РНК-
зависимой РНК-полимеразы.
Экзонуклеазная активность обеспечива-
ется при взаимодействии двух белков: nsp 14
и nsp 10, где каталитическую функцию выпол-
няет nsp 14, а активирует его nsp 10 (рис. 5).
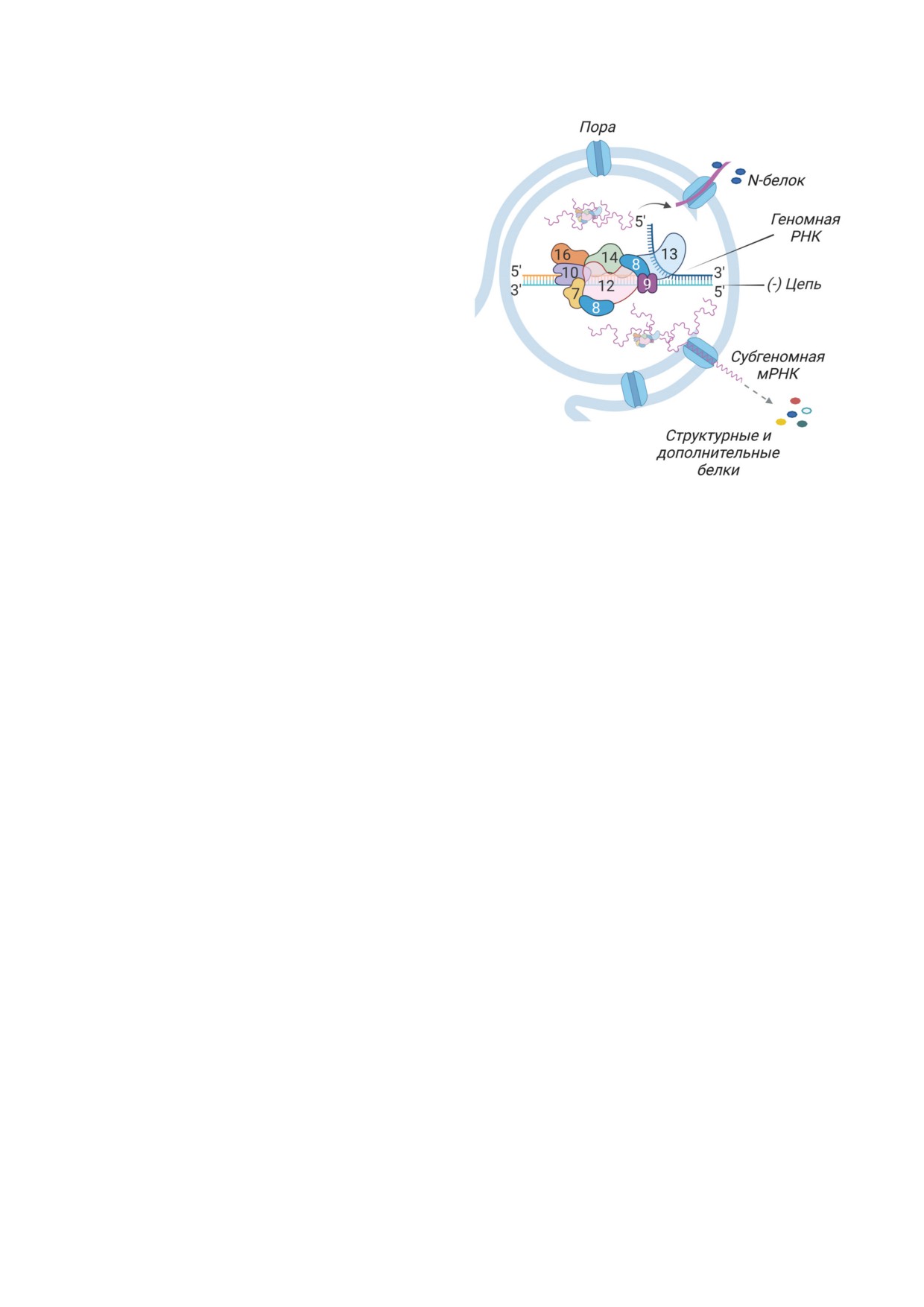
Рис. 5. Внутри двухмембранной везикулы DMV проходит
Последовательность и структура экзонуклеаз-
репликация геномной РНК и транскрипция субгеном-
ного домена nsp 14 сильно отличается от из-
ных РНК. Полимеразный комплекс составляют неструк-
вестных клеточных экзонуклеаз, что делает
турные белки nsp 7, nsp 8, nsp 9, nsp 10, nsp 12, nsp 13,
nsp 14 и nsp 16 (обозначены цифрами). РНК, полученные
этот фермент привлекательной мишенью для
внутри DMV, выходят наружу через поры в мембране.
разработки противовирусных препаратов [29].
Субгеномные РНК транслируются в неструктурные и
дополнительные белки, N-белок встречается с геномной
РНК и образует нуклеокапсид
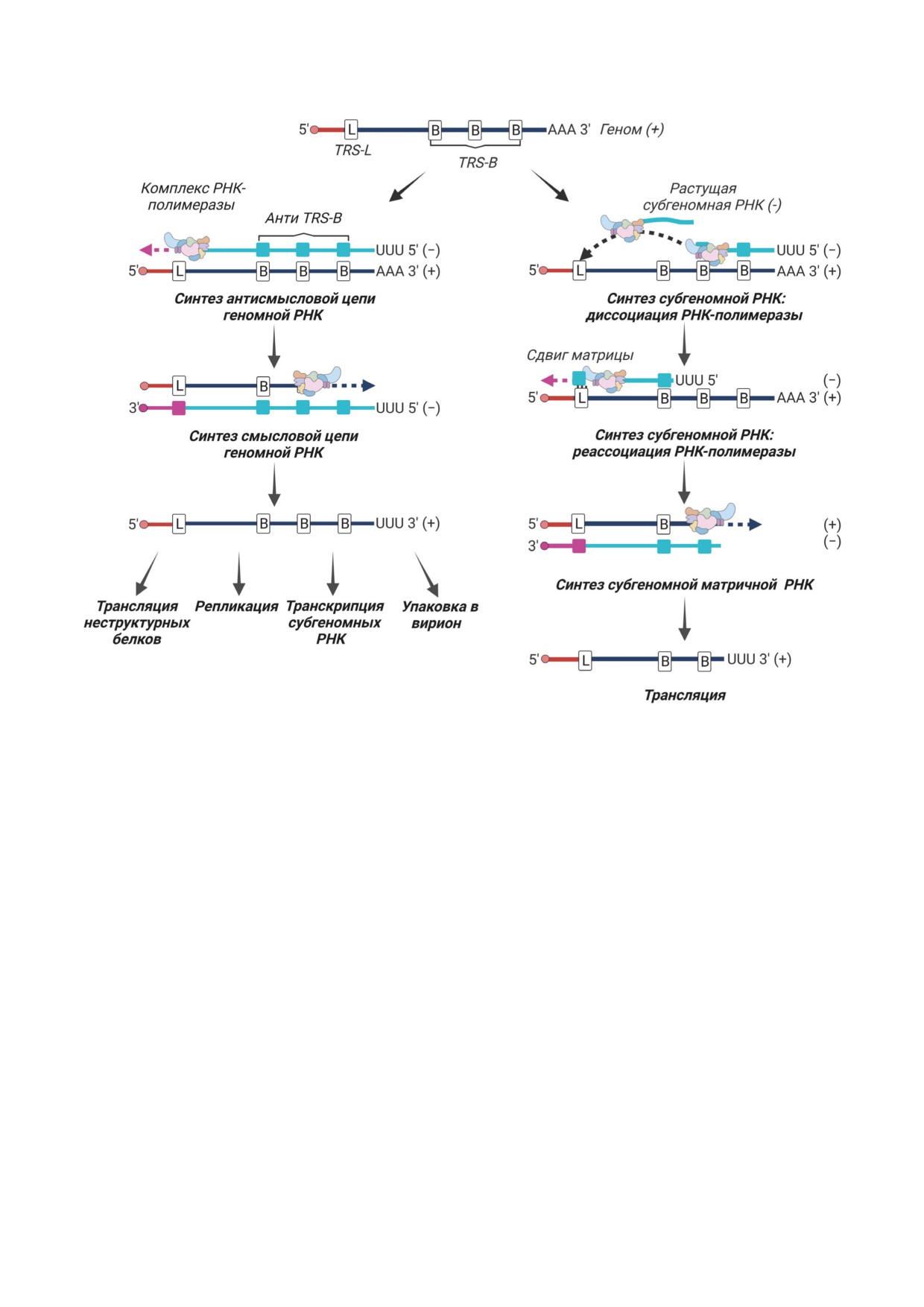
СИНТЕЗ СТРУКТУРНЫХ И
ДОПОЛНИТЕЛЬНЫХ БЕЛКОВ
на начало геномной РНК (на участок TRS-L)
для повторной инициации синтеза. Проис-
В последней трети генома коронавиру-
ходит взаимодействие между комплементар-
сов кодируются структурные и дополнитель-
ными TRS: один расположен на зарождаю-
ные белки. Для их трансляции в инфици-
щейся антисмысловой цепи РНК (TRS-B), а
рованных клетках образуются субгеномные
второй - на матричной смысловой геномной
матричные РНК. Эти молекулы вирус полу-
РНК (TRS-L). Взаимодействие TRS происхо-
чает особым способом - путём «прерыви-
дит по консервативной последовательности
стой» транскрипции, которая встречается у
(5′-ACGAAC-3′ для SARS-CoV и SARS-CoV-2),
коронавирусов и большинства членов поряд-
окружённой вариабельными последователь-
ка Nidovirales, но не детектирована у других
ностями переменной длины, которые также
РНК-вирусов [30].
могут способствовать взаимодействию TRS-B
Вирусные смысловые РНК на
5′-конце
и TRS-L [15]. После реиницииации синтеза
имеют одинаковую лидерную последователь-
РНК на TRS-L копия лидерной последователь-
ность - TRS-L (TRS-lider), варьирующую у
ности, расположенной на матричной цепи, до-
разных коронавирусов от 55 до 92 нуклеоти-
бавляется к зарождающейся РНК, и синтез ан-
дов [31]. В субгеномных мРНК эта последова-
тисмысловых субгеномных РНК завершается.
тельность «слита» с другой регуляторной по-
Эти промежуточные РНК затем используются
следовательностью - TRS-B (или TRS-body).
для транскрипции в субгеномные мРНК, ко-
В геноме коронавирусов последовательность
торые имеют смысловую цепь и используются
TRS-B находится непосредственно перед каж-
как мРНК для трансляции структурных и до-
дой открытой рамкой считывания в районе
полнительных белков (рис. 6) [18]. Субгеном-
структурных и дополнительных генов (за неко-
ные мРНК совпадают своими 3′- и 5′-конца-
торым исключением), а TRS-L - только в од-
ми, но варьируют по внутренним участкам.
ном месте (на 5′-конце).
Такие РНК считаются полицистронными,
В процессе синтеза антисмысловой цепи
однако функционально они моноцистронны,
РНК-зависимая РНК-полимераза доходит
поскольку трансляция ограничивается одной
до TRS-B, после копирования которой она
открытой рамкой считывания, ближайшей к
останавливает свою работу и перескакивает
5′-лидерной последовательности [15].
БИОХИМИЯ том 87 вып. 12 2022
2054
МИНГАЛЕЕВА и др.
Рис. 6. Репликация и «прерывистая» транскрипция. Геномная РНК вируса служит матрицей для синтеза новых ко-
пий геномной РНК (слева) и для транскрипции в субгеномные мРНК по «прерывистому» принципу (справа). Копии
геномных РНК используются для синтеза неструктурных белков, репликации, транскрипции в субгеномные РНК и
упаковки в вирион. Субгеномные мРНК транслируются в структурные и дополнительные белки
Количество отдельных субгеномных РНК,
полученные в результате делеций структурных
синтезируемых в инфицированных клетках,
и дополнительных генов) [15]. Неканониче-
зависит от локализации и эффективности счи-
ские субгеномные РНК могут составлять до 1/3
тывания каждого TRS-B [26]. Показано, что
от общего количества субгеномных РНК [32].
плотность посадки рибосомы определяет ко-
Leary et al. [33] исследовали вариант SARS-
личество транскриптов с данного TRS [15].
CoV-2, содержащий мутацию D614G и 3 со-
Для большинства представителей корона-
седних нуклеотидных замены, охватывающих
вирусов идентифицировано от 5 до 8 субгеном-
два остатка нуклеокапсидного белка (R203K/
ных РНК, 4 из которых кодируют обязатель-
G204R; вариант B.1.1). Анализ последователь-
ные структурные белки вириона (S, E, M и N),
ности предполагает, что эти замены являются
а остальные относятся к дополнительным бел-
результатом гомологичной рекомбинации ос-
кам. В клетках, инфицированных SARS-CoV-2
новной последовательности TRS-L. Благодаря
и SARS-CoV, идентифицировано 11 и 10 субге-
этому появилась новая TRS между доменами
номных мРНК соответственно [17].
связывания РНК и доменами димеризации
Кроме канонических субгеномных РНК,
нуклеокапсида, что привело к появлению но-
были обнаружены другие, неканонические РНК-
вого транскрипта субгеномной РНК. Вирусы с
продукты прерывистой транскрипции
[31].
мутацией K203/R204 могут также иметь повы-
И здесь много интригующего: есть те, в кото-
шенную экспрессию субгеномной РНК из дру-
рых 5′-лидерные последовательности присо-
гих открытых рамок считывания. Способность
единены к неожиданным 3′-сайтам (TRS-L-
SARS-CoV-2 вводить новые мотивы TRS в свой
независимые длинные гибриды и продукты,
геном с потенциалом для новых транскриптов
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2055
субгеномных РНК предполагает, что это сред-
Они не являются важными участниками про-
ство повышения эффективности и адаптации.
цесса репликации, однако, по-видимому,
В данном исследовании также говорится о
играют важную роль в патогенезе вируса, то
том, что сам SARS-CoV-2 может выступать как
есть при взаимодействии вируса с организмом
источник гомологичной рекомбинации [33].
хозяина. Большинство функций, приписыва-
Многие неканонические субгеномные
емых дополнительным белкам, связаны с ме-
РНК обладают кодирующим потенциалом, и
ханизмами уклонения от действия иммунной
их продукты могут представлять собой уко-
системы организма хозяина. К таким функци-
роченные версии дополнительных белков
ям относится, например, подавление секреции
или сами по себе быть не исследованными
цитокинов при участии ORF9c или антаго-
белками. На примере SARS-CoV-2 показана
низм интерферона первого типа при участии
субгеномная РНК, которая способна транс-
ORF3b, ORF6, ORF7a, ORF8 или ORF9b. Кро-
лироваться в укороченный с N-конца (де-
ме того, эти вспомогательные белки (напри-
леция в 143 а.о.) S-белок. Подобный белок
мер, ORF3a) влияют на такие важные клеточ-
был идентифицирован в респираторном ко-
ные процессы, как аутофагия или апоптоз.
ронавирусе свиней [31]. Davidson et al. [34]
ORF3b влияет на функционирование мито-
представили доказательства картирования
хондрий, а ORF9b активирует воспаление [17].
пептидов с помощью тандемной масс-спек-
Многие функции этих белков остаются неиз-
трометрии, указывающие на обнаружение
вестными ввиду недостаточной гомологии с
ранее неизвестных вирусных белков, часть
известными белками [23].
которых может иметь происхождение от не-
канонических субгеномных РНК [34]. Так-
же неканонические субгеномные РНК могут
МЕХАНИЗМ ПРОНИКНОВЕНИЯ
функционировать как дефектные интерфери-
ВИРУСА В КЛЕТКУ
рующие РНК [35].
Наличие неканонических РНК показа-
Коронавирусы проникают в клетку по
но для разных коронавирусов (например, для
завершению двух последовательных собы-
MHV, HCoV-229E, SARS-CoV-2). Не ясно,
тий: (1) вирусный S-белок взаимодействует
синтезируются ли эти неканонические РНК
со своим рецептором на поверхности клетки
в результате необычной прерывистой тран-
и (2) S-белок расщепляется клеточной про-
скрипции (хотя и показано использование
теазой до своей активной формы, способной
TRS-подобных последовательностей для син-
стимулировать слияние вирусной и клеточной
теза субгеномных транскриптов [15]) или они
мембран.
представляют собой продукты рекомбинации.
Описано 4 рецептора, которые исполь-
Считается, что TRS представляют собой
зуются коронавирусами для связывания с
«горячие точки» рекомбинации, а вторичные
клеткой организма-хозяина. S-белки корона-
РНК-структуры способствуют переключению
вируса HCoV-229E, вируса трансмиссивного
матрицы TRS-независимым способом. В экс-
гастроэнтерита свиней (TGEV) и вируса ин-
периментах по транскриптомике и профили-
фекционного перитонита кошек (FIPV) взаи-
рованию рибосом был продемонстрирован
модействуют с аминопептидазой N (APN).
сложный ландшафт РНК и потенциальных
Причём для превращения аминопептидазы
белков SARS-CoV-2, который выходит далеко
свиньи в рецептор вируса человека HCoV-229E
за рамки «канонической» программы экспрес-
достаточно изменить участок всего из 8 а.о. в
сии генов коронавирусов. Аналогичные на-
гипервариабельной области рецептор-связы-
блюдения были сделаны и в отношении других
вающего домена (RBD) [36]. Молекула адге-
коронавирусов [15]. Детальное изучение этих
зии CEACAM1 является рецептором для входа
процессов ещё впереди.
вируса гепатита мышей (MHV), относящего-
На данный момент чётко не установлено,
ся к роду Betacoronavirus. RBD S-белка этого
идентичен ли состав репликазно-транскрип-
вируса расположен в необычном для коро-
ционных комплексов, выполняющих синтез
навирусов месте, в его N-концевом домене,
антисмысловых геномных РНК и субгеном-
где он функционирует как лектин, связывая
ных РНК. Вероятно, баланс между реплика-
остатки углеводов. Когда-то это позволило
цией и транскрипцией определяется взаимо-
древнему коронавирусу расширить свой тро-
действием со специфическими белковыми
пизм и повысить инфекционность. SARS-CoV,
факторами.
SARS-CoV-2 и некоторые SARS-родственные
Дополнительные белки являются самыми
вирусы летучих мышей используют для вхо-
неизученными элементами коронавирусов.
да в клетку ангиотензин-превращающий
БИОХИМИЯ том 87 вып. 12 2022
2056
МИНГАЛЕЕВА и др.
фермент ACE2. MERS-CoV, верблюжий MERS-
стояние недоступности к взаимодействию с
CoV и BatCoV-HKU4 используют белковый ре-
рецептором) [37]. В субъединице S2 выделяют
цептор DPP4 [10].
пептид слияния FP, два домена, содержащих
Три из указанных выше рецепторов (APN,
гептадный повтор (HR1 и HR2), трансмем-
ACE2 и DPP4) обладают пептидазной актив-
бранный (TMD) и цитоплазматический (CTD)
ностью. Однако показано, что протеолити-
домены. На примере ACE2 показано, что с од-
ческая активность этих молекул не важна для
ним димером рецептора связывается 2 триме-
успешного связывания и проникновения ко-
ра S-белка, что ускоряет процесс проникнове-
ронавирусов. Связывание RBD и рецептора
ния вируса.
происходит на внешней стороне рецептора, не
В нативной конформации субъединица S1
затрагивая участок его каталитического цен-
«обёртывает» субъединицу S2, которая обра-
тра. Но присутствие трансмембранных про-
зует центр белка. После отщепления субъеди-
теаз, например, TMPRSS2 (трансмембранная
ницы S1 внутри субъединицы S2 происходят
сериновая протеаза 2), критически важно [10].
масштабные перестройки, в том числе рефол-
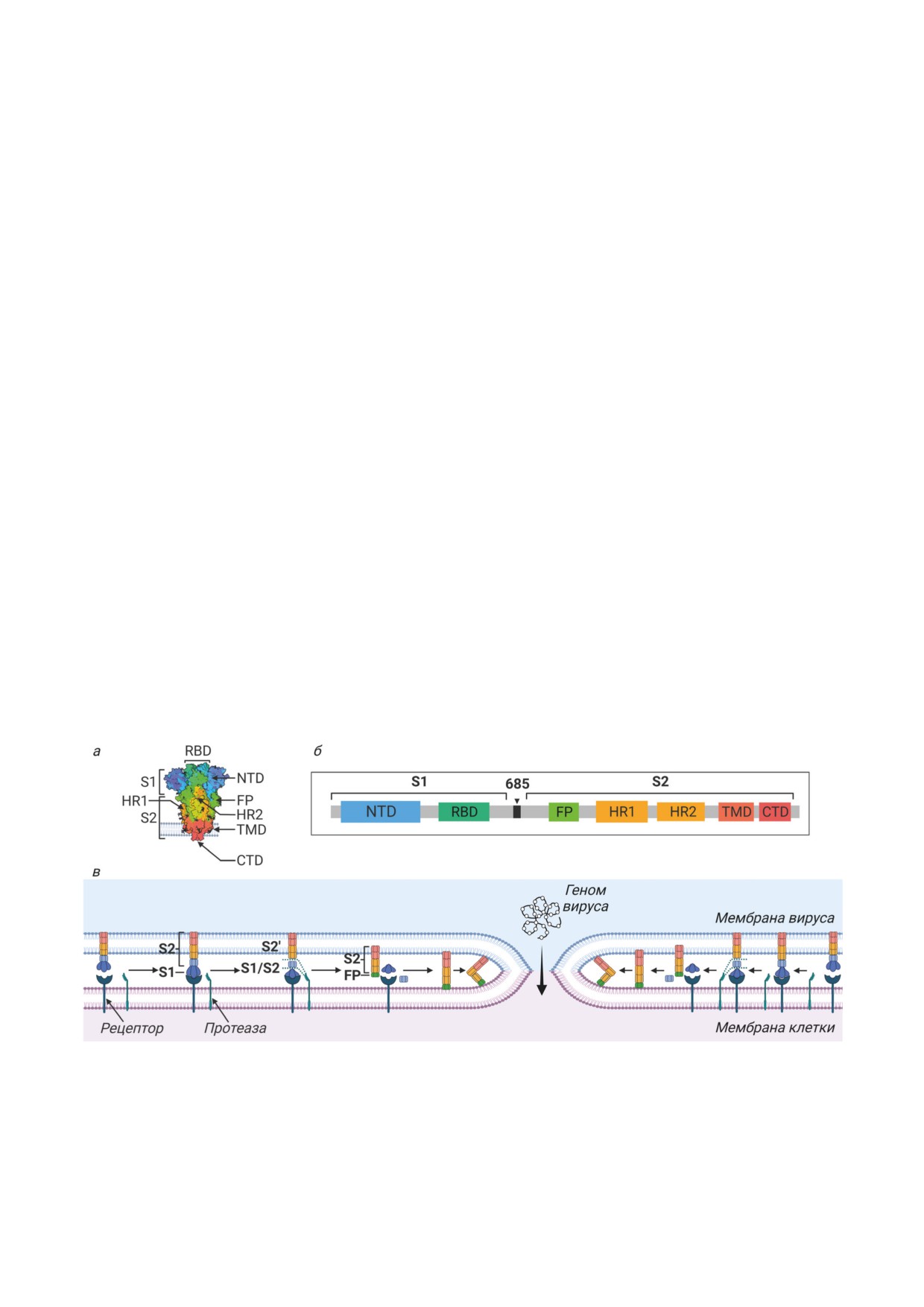
S-Белок (180-200 кДа, 1273 а.о.) распола-
динг домена HR1, благодаря чему пептид слия-
гается на поверхности вириона в виде триме-
ния FP высвобождается и встраивается в мем-
ра, формируя выпуклую «головку» и «стебель».
брану клетки-хозяина [37].
Каждый тример покрыт 66 гликанами, маски-
S-Белок содержит 2 участка, последова-
рующими вирус от узнавания иммунной си-
тельное расщепление которых приводит к его
стемой хозяина и помогающими вирусу при-
активации на клеточной поверхности. Пер-
крепляться. Выделяют 2 субъединицы S-белка:
вый участок, S1/S2, расположен на стыке
S1 отвечает за связывание с рецептором, а S2 -
двух субъединиц. У некоторых, но далеко не
за слияние вируса с мембраной клетки-хозя-
у всех коронавирусов, он узнаётся протеаза-
ина. Основными доменами субъединицы S1
ми, расположенными на поверхности клетки,
являются N-концевой домен (NTD) и RBD.
и катепсинами. MERS-CoV, IBV, SARS-CoV
Внутри домена RBD выделяют рецептор-свя-
и SARS-CoV-2 являются примерами вирусов,
зывающий мотив (RBM), который непосред-
у которых происходит расщепление по S1/
ственно связывается с рецептором. RBD-до-
S2
[38]. Относительно других коронавиру-
мены тримера S-белка могут существовать
сов SARS-CoV-2 имеет эволюционное пре-
в двух различных конформациях: открытой
имущество
- вставку полиосновного сайта
(up-конформация, состояние доступности ре-
расщепления в этом участке, а он является
цептору) и закрытой (down-конформация, со-
минимальным сайтом узнавания фуринами -
Рис. 7. Доменная организация S-белка и его участие в процессе слияния вирусной частицы с клеткой. Кристалло-
графическая (а) и первичная (б) структура S-белка SARS-CoV-2. Внутри субъединиц S1 и S2 выделяют следующие
домены: NTD - N-концевой домен, RBD - рецептор-связывающий домен, пептид слияния FP, домены HR1 и HR2,
трансмембранный (ТМD) и цитоплазматический (СТD) домены. в - Схема взаимодействия клетки и коронавирус-
ной частицы. S-Белок, расположенный на поверхности вирусной мембраны, находит на клеточной мембране соответ-
ствующий рецептор. После их взаимодействия друг с другом клеточная протеаза расщепляет S-белок по сайтам S1/S2
и S2′. Это приводит к высвобождению активированной субъединицы S2 с торчащим по направлению к клетке пепти-
дом слияния FP. Пептид встраивается в мембрану клетки, домены HR1 и HR2 взаимодействуют друг с другом, «под-
тягивая» обе мембраны друг к другу
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2057
пептидазами, широко представленными на
S-тримере D614, вклинивается между домена-
эпителиальных клетках. Это позволило SARS-
ми внутри протомера в шипе G614. Это допол-
CoV-2 использовать широкий спектр разных
нительное взаимодействие, по-видимому, пре-
протеаз для активации процесса проникно-
дотвращает преждевременную диссоциацию
вения в клетку. Второй участок находится
тримера G614, в результате которой S-белок
внутри субъединицы S2 и называется S2′. Его
становится более стабильным, чем в исход-
расщепление может проходить во внеклеточ-
ном варианте вируса. Наличие данной мута-
ном пространстве или на поверхности клет-
ции связано с высокой вирусной нагрузкой у
ки при участии сериновых протеаз, таких как
инфицированных пациентов и высокой ин-
трипсин, TMPRSS2 или нейтрофильная элас-
фекционностью на модели псевдотипирован-
таза. При отсутствии расщепления S1/S2 ак-
ных по системе VSVΔ*G/GFP вирусов in vitro.
тивация S2′ все ещё возможна катепсинами
При этом наличие мутации D614G не коррели-
(катепсином L) в момент созревании вириона
рует с тяжестью заболевания [54].
при прохождении эндоцитозного пути. То есть
Для варианта Дельта описано 4 критиче-
коронавирусы могут сливаться с плазматиче-
ские мутации, влияющие на характеристики
ской мембраной клетки или эндосомами, ис-
вируса: D614G и G142D повышают аффин-
пользуя ранний и поздний пути. Ранний путь
ность к ACE2, а L452R и T478K обеспечивают
предполагает расщепление сначала по S1/S2,
высокую аффинность и неспособность антител
а затем - по S2′ внеклеточными и клеточны-
нейтрализовать вирус (табл. 2). Эти мутации не
ми трансмембранными протеазами (трипсин,
уникальны и встречаются в других вариантах,
нейтрофильная эластаза, TMPRSS2), тогда как
но их сочетание в варианте Дельта может опре-
в позднем пути процессом руководят эндосо-
делять длительное и тяжёлое течение заболева-
мальные протеазы, такие как катепсин L [39].
ния. В работе Zhang et al. [55], опубликованной
После расщепления на участке S2′ пептид
в журнале Science, были описаны 2 характери-
слияния FP экспонируется наружу и про-
стики варианта Дельта, объясняющие его бо-
исходит запуск слияния с мембраной клет-
лее высокую трансмиссивность. Во-первых,
ки-хозяина. Затем возникают гидрофобные
даже при низких уровнях ACE2 слияние S-бел-
взаимодействия между доменами HR1 и HR2
ка вируса варианта Дельта с клеткой происхо-
субъединицы S2, в результате чего образуется
дит эффективно. Во-вторых, инфицирование
структура, состоящая из 6 цепей, которая за-
клеток-мишеней вирусами этого варианта
ставляет мембраны вируса и клетки сближать-
происходит значительно быстрее, чем виру-
ся друг с другом, образуя пору. Пора увеличи-
сами других вариантов. Таким образом, при
вается до тех пор, пока внутрь клетки не зайдёт
относительно коротком воздействии Дельта
генетический материал вируса (рис. 7).
может быстро заражать гораздо большее коли-
чество клеток, что приводит к короткому ин-
кубационному периоду и большей вирусной
МУТАЦИИ SARS-CoV-2
нагрузке во время инфекции [55].
Planas et al. [56] исследовали чувствитель-
Эволюция вируса происходит постоянно.
ность вируса варианта Дельта к моноклональ-
Считается, что особенно интенсивно мутации
ным антителам и антителам, присутствую-
возникают и закрепляются при длительной
щим в сыворотке крови людей, переболевших
персистенции вируса в организме человека с
Covid-19 или получивших вакцину от Covid-19.
ослабленным иммунитетом [40]. Мутации и их
Было показано, что некоторые монокло-
сочетание модулируют вирулентность или кон-
нальные антитела против N-концевого доме-
тагиозность, влияют на клиническую картину
на (NTD) и против RBD, включая бамлани-
и тяжесть последствий Covid-19 (табл. 2).
вимаб, утратили способность связываться с
На заре образования вариантов вируса
S-белком и больше не были способны нейтра-
SARS-CoV-2 мутация D614G в гене S-бел-
лизовать вирус варианта Дельта. Сыворотки,
ка [52] способствовала быстрому распростра-
собранные у людей, переболевших Covid-19
нению вируса и стала первой мутацией, со-
в течение 12 мес. после появления симпто-
хранившейся во всех вариантах, появившихся
мов, были в 4 раза менее эффективны против
после Уханьского [53]. D614G характеризуется
варианта Дельта по сравнению с вариантом
заменой аспарагиновой кислоты на глицин
Альфа (B.1.1.7). Сыворотки людей, получив-
в положении 614 в S-белке. Остаток 614 нахо-
ших одну дозу вакцины Pfizer или AstraZeneca,
дится на границе раздела субъединиц S1 и S2
показали едва заметное ингибирующее дей-
и может влиять на их взаимодействие и стаби-
ствие на вирус варианта Дельта. Сыворотки
лизацию S-белка. Петля, разупорядоченная в
людей, получивших оба компонента вакцины,
БИОХИМИЯ том 87 вып. 12 2022
2058
МИНГАЛЕЕВА и др.
Таблица 2. Мутации S-белка, характерные для VOC SARS-CoV-2 [41, 42] и влияющие на иммунный ответ и аффинность
связывания с рецептором ACE2
Обозначение вариантов
Изменение
Уклонение от
аффинности
Мутации
Альфа1
Бета
Гамма
Дельта
Омикрон
нейтрализую-
связывания
щих антител3
с ACE23
B.1.1.72
B.1.351
P.1
B.1.617.2
BA.1
BA.2
BA.3
BA.4
BA.5
L18F
[43]
G142D
[44]
del143/145
[44]
del144/144
[45]
R214_insEPE
[44]
del242/244
G339D
[44]
S371L
[44]
↑[40]
S373P
[44]
↑[40]
S375F
↑[40] ↓[44]
D405N
↓[46]
R408S
[44]
K417N
↓[45]
K417T
[44]
↓[45]
N440K
[44]
↓[46]
G446S
[47]
L452R
[45, 64]
↑[40]
S477N
[45, 48]
↑[45], ↓[46]
T478K
[44, 45]
↑[40]
E484A
[44, 45]
↓[46]
E484K
↓[46]
F486L
[64]
↑[64]
Q493R
[49]
↑[40]
G496S
↑[46]
Q498R
↑[40]
N501Y
[49]
↑[40, 44, 45]
D614G
↑[50]
N764K
[44]
V1176F
↑[51]
Примечание. Ячейки с серым фоном - наличие у варианта мутации, которая изменяет ответ на нейтрализующие
антитела или аффинность связывания с ACE2; ячейки с бесцветным фоном - отсутствие мутации. 1 - Обозначение
ВОЗ; 2 - обозначение Pango, 3 - в столбце приведены ссылки на литературные источники, в которых описано влияние
мутаций на способность вируса уклоняться от нейтрализующих антител или изменять аффинность к ACE2: повы-
шать (↑) или понижать (↓) её.
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2059
показали нейтрализующий эффект в отноше-
вируса [42]. Мутация T9I E-белка, наблюдае-
нии этого варианта, но были примерно в 3-5 раз
мая во всех подвариантах Омикрона и распо-
менее эффективны, чем против варианта Аль-
ложенная в трансмембранном домене, может
фа [56]. В другом исследовании было показано,
влиять на конфигурацию этого белка, обеспе-
что введение двух доз вакцины даёт лучшую за-
чивая более сильное заякоривание вирусной
щиту, однако это также зависит от варианта ви-
мембраны [61]. У вариантов Бета и Омикрон
руса. Так, эффективность вакцины Pfizer против
было выявлено несколько критических сайтов
Дельта снижается до 88%, тогда как для Альфа
связывания RBD с рецептором ACE2: K417N,
её эффективность составляет 93,4%. Вакцина-
E484K, Q493H, N501Y - для варианта Бета и
ция препаратом AstraZeneca была эффективна
Q493R, Q498R, N501Y - для варианта Оми-
на уровне 66,1% при Альфа-варианте и 59,8% -
крон. Предполагают, что мутации в этих сай-
при варианте Дельта [57].
тах могут привести к расширению тропизма
Повсеместное и быстрое распростране-
этого вируса [62, 63].
ние варианта Омикрон привело к значитель-
S-Белок доминантных, на сентябрь 2022 г.,
ной эволюции вируса в пределах этого VOC.
подвариантов BA.4 и BA.5 идентичен и очень
Для мутаций этого варианта собрано особенно
похож на спайковый белок BA.2. Отличие со-
много сведений о возможности уклонения от
стоит в том, что эти два подварианта имеют
нейтрализующих антител и изменениях аффин-
делецию 69-70, кроме того, характерную для
ности к рецептору (табл. 2). В нем мы можем
Дельта мутацию L452R, новую мутацию F486V,
наблюдать значительное количество мутаций,
а также реверсную мутацию R493Q. В других ча-
расположенных в различных участках генома.
стях генома имеются дополнительные мутации.
В S-белке Омикрона число мутации составля-
Например, для подварианта BA.4 характерны
ет более 30 [58], из них более 10 расположены
мутации L11F - в ORF7b, P151S - в гене N-бел-
в RBD. По некоторым данным, существование
ка, делеция 141-143 - в nsp 1, тогда как BA.5
большого количества мутаций в вирусе этого
имеет мутацию D3N в гене M-белка [64].
варианта указывает на его происхождение от
Предполагают, что замены в S-белке в по-
людей с ослабленным иммунитетом [40].
ложениях 452, 486 и 493 изменяют связывание
Мутации с изменением аффинности внут-
с ACE2 и влияют на взаимодействие с антите-
ри одного варианта/подварианта Омикрон за-
лами. Мутации в положении L452 с заменами
частую имеют противоположный эффект и мо-
на R/M/Q встречались в вариантах Дельта,
гут нейтрализовать друг друга, что может быть
Каппа и Эпсилон, но также независимо воз-
причиной довольно лёгкого течения Covid-19,
никали в нескольких подвариантах BA.2 в раз-
вызванного этим вариантом. В то же время спо-
ных географических точках (например, L452Q
собность к уклонению от нейтрализующих ан-
в BA.2.12.1). Мутация в этом положении ос-
тител могла привести к широкому распростра-
лабляет способность антител нейтрализовать
нению Омикрона: вариант B.1.1.529 в 3,3 раза
вирус [64]. Фенилаланин в положении
486
более трансмиссивен, чем Дельта [59]. Мно-
участвует в связывании вируса с рецепто-
гие мутации варианта Омикрон встречаются в
ром ACE2. Мутация в этом положении приво-
ранее обнаруженных вариантах SARS-CoV-2.
дит к снижению нейтрализующей активности
Уникальной мутацией подварианта BA.1 явля-
антител классов I и II и поликлональной сы-
ется инсерция R214_insEPE. Данная инсерция,
воротки. Предполагают, что именно F486 даёт
по-видимому, появилась в результате реком-
большое преимущество вирусу при уклонении
бинации у людей, инфицированных одновре-
от действия нейтрализующих антител, вклю-
менно несколькими коронавирусами. Была
чая антитела, нейтрализующие BA.1. То есть
установлена идентичная последовательность в
BA.4 и BA.5 приобрели механизм ещё более
S-белке HCoV-229E, которую можно использо-
эффективного уклонения от действия анти-
вать для переключения матрицы [60].
тел, чем предыдущие подварианты [64]. Кроме
Мутациями, встречающимися во всех под-
того, BA.5 обладает наибольшей трансмиссив-
вариантах Омикрона, являются G142D (кроме
ностью, согласно еженедельным сводкам ВОЗ
BA.1, у которого G142), G339D, S371L, S373P,
по состоянию на сентябрь 2022 г., BA.2.12.1
S375F, K417N, N440K, S477N, T478K, E484A,
и BA.2.75 также являются производными BA.2.
Q498R, N501Y, Y505H, D614G, H655Y, N679K,
В районе S-белка BA.2.12.1 присутствуют за-
P681H, N764K, D796Y, Q954H и N969K. Среди
мены L452Q и S704F, а для BA.2.75 - K147E,
них мутации N501Y и Q498R, как предполага-
W152R, F157L, I210V, G257S, D338H, G446S,
ют, усиливают связывание с рецептором ACE2,
N460K и реверсная мутация Q493R.
а мутации H655Y, N679K и P681H увеличивают
Спустя несколько месяцев после начала
расщепление S-белка и облегчают передачу
пандемии Covid-19 в инфицированных образ-
БИОХИМИЯ том 87 вып. 12 2022
2060
МИНГАЛЕЕВА и др.
цах начали выявляться рекобинантные геномы.
форм; в связи с лёгких течением Омикрона
В целом это было предсказуемо - у коронави-
время репликации вируса внутри организма
русов довольно часто происходит гомологич-
ограничено, делая минимальной вероятность
ная и негомологичная рекомбинация. Гомоло-
коинфекции или суперинфекции.
гичная рекомбинация, как предполагают, идёт
по общей схеме с отщеплением РНК-зависи-
мой РНК-полимеразы от матрицы в процессе
ЗАКЛЮЧЕНИЕ
синтеза РНК и её присоединением к гомоло-
гичному участку матрицы другого генома с по-
Механизмы репликации и транскрипции
следующей элонгацией. Негомологичная ре-
генов, взаимодействие белков коронавиру-
комбинация может возникать между геномной
сов с рецепторами человека ранее были изу-
РНК и субгеномными мРНК в результате кол-
чены на примерах SARS-CoV и MERS-CoV.
лапса транскрипционного комплекса во время
Но отсутствие препаратов для эффективной
прерывистой транскрипции. Для того, чтобы
терапии коронавирусных инфекций позво-
рекомбинационное событие имело место, не-
ляет нам заключить, что выявлены не все мо-
обходима одновременная циркуляция в попу-
лекулярные механизмы функционирования
ляции двух и более подвариантов и появление
коронавирусов. Исследование SARS-CoV-2
внутри одной клетки разных геномов.
позволило улучшить наше понимание меха-
Впервые геном, производный двух раз-
низмов внедрения вируса в клетку, опреде-
ных вариантов SARS CoV-2 (20A и 20B по
лить структуру и функции белков коронавиру-
классификации NextStrain), был зафиксиро-
са, выявить ключевые мутации, влияющие на
ван в феврале 2021 г. [65]. Впоследствии сре-
контагиозность и трансмиссивность вируса,
ди секвенированных последовательностей
на клинические проявления заболевания. Для
часто появлялись разные гибридные геномы.
SARS-CoV-2 была детально описана уникаль-
В исследовании Focosi и Maggi [66] можно оз-
ная способность к сдвигу рамки считывания в
накомиться с подробным описанием 40 вари-
процессе транскрипции своей геномной РНК,
антов зафиксированных гибридных геномов,
в результате чего увеличилась вариабельность
полученных в результате рекомбинационных
синтезируемых белков. Данные белки способ-
событий (гибриды Альфа-Дельта, Бета-Дель-
ны «строить» внутриклеточные везикулы, где
та, Дельта-Омикрон, разных подвариантов
происходит репликация и транскрипция ви-
Омикрона). Рекомбинатный вариант XD пред-
русной РНК, а также последующая сборка ви-
ставляет собой геном Дельта AY.4, в который
русных частиц. Формирование синцития ранее
«вклинилась» последовательность гена S-бел-
было показано для некоторых вирусов, напри-
ка BA.1 (нуклеотиды в положениях с 21643
мер, для ВИЧ, вируса простого герпеса. Одна-
по 25581); XD содержит уникальную мутацию
ко механизмы возникновения такой структу-
nsp 2: E172D [67]; гибрид XF также представ-
ры во многом были не изучены до появления
ляет собой рекомбинант варианта Дельта AY.4
SARS-CoV-2. К примеру, было показано, что
и BA.1 с точкой разрыва вблизи конца nsp 3
способность клеток к слиянию при формиро-
(нуклеотид 5386), в нём фрагменты двух раз-
вании синцития определяется наличием уни-
ных геномов расположены последователь-
кальной для S-белка SARS-CoV-2 вставкой
но друг за другом; XE-рекомбинант объеди-
четырёх аминокислот (PRRA) на стыке субъ-
няет геномы BA.1 и BA.2, при этом большая
единиц S1 и S2.
часть генома, включая ген S-белка, принад-
Критическое и многоролевое участие
лежит BA.2. Появились гибриды и последних
S-белка в патогенезе SARS-CoV-2 само по себе
на сегодняшний день вариантов SARS-CoV-2,
очень интересно. Он участвует в слиянии с
например, BA.2.12.1-BA.5; BA.2.76-BA.5.2;
клеткой, образовании синцития, уклонении
BA.5-BA.2-BA.5.1 [66].
от иммунного ответа, гликаны на его поверх-
В настоящее время считается, что эти ва-
ности защищают вирус от быстрого распоз-
рианты не представляют собой угрозу более
навания иммунной системой. Нуклеотидные
высокой трансмиссивности или ухудшения
последовательности S-белка вирусов SARS-
течения заболевания (Global Virus Network,
CoV, MERS-CoV и SARS-CoV-2 во многом схо-
gvn.org/covid-19). Это связано с тем, что волны
жи. Тем не менее ответ организма человека на
пандемии последних вариантов вируса стано-
каждый из этих вирусов отличается. Пандемия
вятся все короче, минимизируя время совмест-
Covid-19 дала нам возможность наблюдать во-
ной циркуляции разных вариантов; высокий
очию эволюцию вируса. Мы стали свидетеля-
индекс репродукции последних вариантов
ми того, как сильно различается исход забо-
ставит высокую планку для рекомбинантных
левания от количества и сочетания мутаций
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2061
в вирусных вариантах. Аминокислотный со-
борьбы с вирусными инфекциями. Стала оче-
став S-белка определяет эффективность про-
видна необходимость отслеживания и кон-
никновения вируса в клетку, его патогенность,
троля форм коронавирусов в живой природе,
трансмиссивность и эволюцию.
необходимость реформы в процессах реги-
На данный момент невозможно опреде-
страции и внедрения медицинских изделий
лить реальный объём мировых последствий,
для инфекционных заболеваний, но самое
вызванных пандемией вируса SARS-CoV-2
главное - создания динамичной среды с до-
как для системы здравоохранения, так и для
ступом к новейшему научному инструмента-
социальной и экономической сфер в целом.
рию и высокоспециализированным научным и
Однозначно можно сказать, что ближайшее
медицинским кадрам для быстрого реагирова-
десятилетие мы будем наблюдать и исследо-
ния на биологические вызовы в будущем.
вать последствия коронавирусной инфекции,
оценивать изменения в медицинской стати-
Вклад авторов. Мифтахова Р.Р., Ризванов А.А.,
стике, изучать риски отложенных осложнений,
Шавалиев Р.Ф. - концепция и структура работы и
таких как ожидаемое увеличение аутоимунных
редактирование текста; Мингалеева Р.Н., Нигмату-
и нейродегенеративных заболеваний; анали-
лина Н.А., Шарафетдинова Л.М., Ромозанова А.М.,
зировать тенденции и риски для пациентов с
Габдулхакова А.Г., Филина Ю.В. - анализ литерату-
хроническими и онкологическими заболева-
ры, написание текста; подготовка иллюстраций.
ниями, исследовать состояние здоровья роже-
Финансирование. Работа выполнена в рам-
ниц и детей, инфицированных SARS-CoV-2 в
ках Программы стратегического академиче-
гестационный период. Особую группу иссле-
ского лидерства Казанского (Приволжского)
дований займут работы по оценке неврологи-
федерального университета.
ческих последствий, связанных с социальным
Конфликт интересов. Авторы заявляют об
дистанцированием, в том числе у детей и под-
отсутствии конфликта интересов.
ростков. Кроме того, актуальным вопросом
Соблюдение этических норм. Настоящая
остаётся вероятность эволюции вируса SARS-
статья не содержит описания каких-либо ис-
CoV-2. Теоретическая возможность эволю-
следований с участием людей или животных в
ции вируса и комбинации транcмиссивности
качестве объектов.
SARS-CoV-2 и летальности MERS-CoV ставят
Дополнительная информация. Рисунки,
перед мировым научным сообществом вопрос
представленные в обзоре, выполнены с ис-
о глобализации и разработке новых методов
СПИСОК ЛИТЕРАТУРЫ
1. Reagan, R. L., Yancey, F., and Brueckner, A. L.
in southern China, Science, 302, 276-278, doi: 10.1126/
(1955) Studies of the avian infectious bronchitis virus
science.1087139.
(wachtel strain) in the cynomolgus monkey, Poultry
6. Zhu, Z., Lian, X., Su, X., Wu, W., Marraro, et al.
Sci., 34, 1448, doi: 10.3382/ps.0341448.
(2020) From SARS and MERS to COVID-19: a brief
2. Alharbi, S. N., and Alrefaei, A. F. (2021) Comparison
summary and comparison of severe acute respiratory
of the SARS-CoV-2 (2019-nCoV) M protein with its
infections caused by three highly pathogenic human
counterparts of SARS-CoV and MERS-CoV species,
coronaviruses, Respiratory Res., 21, 224, doi: 10.1186/
J. King Saud. Univ. Sci., 33, 101335, doi: 10.1016/
s12931-020-01479-w.
j.jksus.2020.101335.
7. Li, X., Giorgi, E. E., Marichannegowda, M. H., Foley, B.,
3. Paul, D., Kolar, P., and Hall, S. G. (2021) A review
Xiao, C., et al. (2020) Emergence of SARS-CoV-2
of the impact of environmental factors on the fate and
through recombination and strong purifying selection,
transport of coronaviruses in aqueous environments, npj
Sci. Adv., 6, doi: 10.1126/sciadv.abb9153.
Clean Water, 4, 7, doi: 10.1038/s41545-020-00096-w.
8. Hassanin, A., Rambaud, O., and Klein, D. (2022)
4. Chan, T., Klaus, J., Meli, M. L., and Hofmann-
Genomic Bootstrap Barcodes and Their Application
Lehmann, R. (2021) SARS-CoV-2 infections in cats,
to Study the Evolution of Sarbecoviruses, 14, 440,
dogs, and other animal species: Findings on infection
doi: 10.3390/v14020440.
and data from Switzerland, Schweiz Arch. Tierheilkd,
9. He, B., Li, Z., Yang, F., Zheng, J., Feng, Y., et al.
163, 821-835, doi: 10.17236/sat00329.
(2013) Virome profiling of bats from Myanmar by
5. Guan, Y., Zheng, B. J., He, Y. Q., Liu, X. L., Zhuang,
metagenomic analysis of tissue samples reveals more
Z. X., et al. (2003) Isolation and characterization of
novel mammalian viruses, PLoS One, 8, e61950,
viruses related to the SARS coronavirus from animals
doi: 10.1371/journal.pone.0061950.
БИОХИМИЯ том 87 вып. 12 2022
2062
МИНГАЛЕЕВА и др.
10.
Millet, J. K., Jaimes, J. A., and Whittaker, G. R.
24.
Xia, B., and Kang, X. (2011) Activation and maturation
(2021) Molecular diversity of coronavirus host cell
of SARS-CoV main protease, Protein Cell, 2, 282-290,
entry receptors, FEMS Microbiol. Rev., 45, fuaa057,
doi: 10.1007/s13238-011-1034-1.
doi: 10.1093/femsre/fuaa057.
25.
Yang, Y., Hussain, S., Wang, H., Ke, M., and Guo, D.
11.
Gómez, C. E., Perdiguero, B., and Esteban, M. (2021)
(2009) Translational control of the subgenomic RNAs
Emerging SARS-CoV-2 variants and impact in global
of severe acute respiratory syndrome coronavirus, Virus
vaccination programs against SARS-CoV-2/COVID-19,
Genes, 39, 10-18, doi: 10.1007/s11262-009-0357-y.
Vaccines (Basel), 9, 243, doi: 10.3390/vaccines9030243.
26.
Brant, A. C., Tian, W., Majerciak, V., Yang, W., and
12.
Bai, Z., Cao, Y., Liu, W., and Li, J. (2021) The
Zheng, Z.-M. (2021) SARS-CoV-2: from its discovery
SARS-CoV-2 nucleocapsid protein and its role in
to genome structure, transcription, and replication,
viral structure, biological functions, and a potential
Cell Biosci., 11, 136, doi: 10.1186/s13578-021-00643-z.
target for drug or vaccine mitigation, Viruses, 13, 1115,
27.
Denison, M. R., Graham, R. L., Donaldson, E. F.,
doi: 10.3390/v13061115.
Eckerle, L. D., and Baric, R. S. (2011) Coronaviruses:
13.
Liu, D. X., Liang, J. Q., and Fung, T. S. (2021) Human
an RNA proofreading machine regulates replication
coronavirus-229E,
-OC43,
-NL63, and
-HKU1
fidelity and diversity, RNA Biol.,
8,
270-279,
(Coronaviridae), Encyclopedia of Virology, 428-440,
doi: 10.4161/rna.8.2.15013.
doi: 10.1016/B978-0-12-809633-8.21501-X.
28.
Kirchdoerfer, R. N., and Ward, A. B. (2019) Structure
14.
Schoeman, D., and Fielding, B. C. (2019) Coronavirus
of the SARS-CoV nsp12 polymerase bound to nsp7
envelope protein: current knowledge, Virol. J., 16, 69,
and nsp8 co-factors, Nat. Commun.,
10,
2342,
doi: 10.1186/s12985-019-1182-0.
doi: 10.1038/s41467-019-10280-3.
15.
Malone, B., Urakova, N., Snijder, E.J. et al. . (2022)
29.
Rona, G., Zeke, A., Miwatani-Minter, B., de Vries, M.,
Structures and functions of coronavirus replication-
Kaur, R., et al. (2021) The NSP14/NSP10 RNA
transcription complexes and their relevance for SARS-
repair complex as a Pan-coronavirus therapeutic
CoV-2 drug design., Nat. Rev. Mol. Cell Biol., 23, 21-
target, Cell Death Differ., 29, 285-292, doi: 10.1038/
39, doi: 10.1038/s41580-021-00432-z.
s41418-021-00900-1.
16.
Maclachlan, N. J., and Dubovi, E. J. (2016) Fenner’s
30.
Sola, I., Almazán, F., Zúñiga, S., and Enjuanes, L.
Veterinary Virology, Academic press.
(2015) Continuous and discontinuous RNA synthesis
17.
Redondo, N., Zaldívar-López, S., Garrido, J. J., and
in coronaviruses, Annu. Rev. Virol.,
2,
265-288,
Montoya, M. (2021) SARS-CoV-2 accessory proteins
doi: 10.1146/annurev-virology-100114-055218.
in viral pathogenesis: knowns and unknowns, Frontiers
31.
Hussain, S., Pan, J., Chen, Y., Yang, Y., Xu, J., et al.
in Immunology, 12, doi: 10.3389/fimmu.2021.708264.
(2005) Identification of novel subgenomic RNAs and
18.
Kim, D., Lee, J.-Y., Yang, J.-S., Kim, J. W., Kim, V. N.,
noncanonical transcription initiation signals of severe
et al. (2020) The architecture of SARS-CoV-2 tran-
acute respiratory syndrome coronavirus, J. Virol., 79,
scriptome, Cell,
181,
914-921.e910, doi:
10.1016/
5288-5295, doi: 10.1128/JVI.79.9.5288-5295.2005.
j.cell.2020.04.011.
32.
Nomburg, J., Meyerson, M., and DeCaprio, J. A.
19.
Báez-Santos, Y. M., St John, S. E., and Mesecar, A. D.
(2020) Pervasive generation of non-canonical
(2015) The SARS-coronavirus papain-like protease:
subgenomic RNAs by SARS-CoV-2, Genome Med.,
structure, function and inhibition by designed antiviral
12, 108, doi: 10.1186/s13073-020-00802-w.
compounds, Antiviral Res., 115, 21-38, doi: 10.1016/j.
33.
Leary, S., Gaudieri, S., Parker, M. D., Chopra, A.,
antiviral.2014.12.015.
James, I., et al. (2021) Generation of a novel SARS-
20.
Angelini, M. M., Akhlaghpour, M., Neuman, B. W.,
CoV-2 sub-genomic RNA due to the R203K/G204R
and Buchmeier, M. J. (2013) Severe acute respiratory
variant in nucleocapsid: homologous recombination has
syndrome coronavirus nonstructural proteins 3, 4, and 6
potential to change SARS-CoV-2 at both protein and
induce double-membrane vesicles, mBio, 4, e00524-13,
RNA level, bioRxiv, doi: 10.1101/2020.04.10.029454.
doi: 10.1128/mBio.00524-13.
34.
Davidson, A. D., Williamson, M. K., Lewis, S.,
21.
Wolff, G., Melia, C. E., Snijder, E. J., and Bárcena, M.
Shoemark, D., Carroll, M. W., et al. (2020) Char-
(2020) Double-membrane vesicles as platforms for
acterisation of the transcriptome and proteome of
viral replication, Trends Microbiol., 28, 1022-1033,
SARS-CoV-2 reveals a cell passage induced in-frame
doi: 10.1016/j.tim.2020.05.009.
deletion of the furin-like cleavage site from the spike
22.
Medina-Enríquez, M. M., Lopez-León, S., Carlos-
glycoprotein, Genome Med., 12, 68, doi: 10.1186/
Escalante, J. A., Aponte-Torres, Z., Cuapio, A., et al.
s13073-020-00763-0.
(2020) ACE2: the molecular doorway to SARS-CoV-2,
35.
Wang, D., Jiang, A., Feng, J., Li, G., Guo, D., et al.
Cell Biosci., 10, 148, doi: 10.1186/s13578-020-00519-8.
(2021) The SARS-CoV-2 subgenome landscape and its
23.
V’kovski, P., Kratzel, A., Steiner, S., Stalder, H., and
novel regulatory features, Mol. Cell, 81, 2135-2147.e5,
Thiel, V. (2021) Coronavirus biology and replication:
doi: 10.1016/j.molcel.2021.02.036.
implications for SARS-CoV-2, Nat. Rev. Microbiol.,
36.
Kolb, A. F., Hegyi, A., and Siddell, S. G. (1997)
19, 155-170, doi: 10.1038/s41579-020-00468-6.
Identification of residues critical for the human
БИОХИМИЯ том 87 вып. 12 2022
БИОЛОГИЯ SARS-CoV-2
2063
coronavirus
229E receptor function of human
49.
Wright, D. W., Harvey, W. T., Hughes, J., Cox, M.,
aminopeptidase N, J. Gen. Virol., 78 (Pt 11), 2795-
Peacock, T. P., et al. (2022) Tracking SARS-CoV-2
2802, doi: 10.1099/0022-1317-78-11-2795.
mutations and variants through the COG-UK-muta-
37.
Jackson, C. B., Farzan, M., Chen, B., and Choe, H.
tion explorer, Virus Evol., 8, veac023, doi: 10.1093/ve/
(2022) Mechanisms of SARS-CoV-2 entry into cells,
veac023.
Nat. Rev. Mol. Cell Biol., 23, 3-20, doi: 10.1038/
50.
Ozono, S., Zhang, Y., Ode, H., Sano, K., Tan, T. S.,
s41580-021-00418-x.
et al. (2021) SARS-CoV-2 D614G spike mutation
38.
Chan, Y. A. Z., Shing Hei (2022) The emergence of
increases entry efficiency with enhanced ACE2-
the spike furin cleavage site in SARS-CoV-2, Mol. Biol.
binding affinity, Nat. Commun., 12, 848, doi: 10.1038/
Evol., 39, msab327, doi: 10.1093/molbev/msab327.
s41467-021-21118-2.
39.
Stevens, C. S. O., Kasopefoluwa Y., and Lee, B. (2021)
51.
Majumdar, P., and Niyogi, S. (2021) SARS-CoV-2
Proteases and variants: context matters for SARS-
mutations: The biological trackway towards viral
CoV-2 entry assays, Curr. Opin. Virol., 50, 49-58,
fitness, Epidemiol. Infect., 149, e110, doi: 10.1017/
doi: 10.1016/j.coviro.2021.07.004.
S0950268821001060.
40.
Kumar, S., Thambiraja, T. S., Karuppanan, K., and
52.
Morais, I. J., Polveiro, R. C., Souza, G. M., Bortolin,
Subramaniam, G. (2022) Omicron and Delta variant
D. I., Sassaki, F. T., et al. (2020) The global population
of SARS-CoV-2: a comparative computational study
of SARS-CoV-2 is composed of six major subtypes, Sci.
of spike protein, J. Med. Virol., 94, 1641-1649, doi:
Rep., 10, 18289, doi: 10.1038/s41598-020-74050-8.
10.1002/jmv.27526.
53.
Zhang, J., Cai, Y., Xiao, T., Lu, J., Peng, H., et al.
41.
Gangavarapu, K., Latif, A. A., Mullen, J. L.,
(2021) Structural impact on SARS-CoV-2 spike
Alkuzweny, M., Hufbauer, E., et al. (2022) Out-
protein by D614G substitution, Science, 372, 525-530,
break.info genomic reports: scalable and dynamic
doi: 10.1126/science.abf2303.
surveillance of SARS-CoV-2 variants and mutations,
54.
Korber, B., Fischer, W. M., Gnanakaran, S., Yoon, H.,
medRxiv, doi: 10.1101/2022.01.27.22269965.
Theiler, J., et al. (2020) Tracking changes in SARS-
42.
Desingu, P. A., Nagarajan, K., and Dhama, K.
CoV-2 spike: evidence that D614G increases infectivity
(2022) Emergence of Omicron third lineage BA. 3
of the COVID-19 virus, Cell,
182,
812-827.e19,
and its importance, J. Med. Virol., 94, 1808-1810,
doi: 10.1016/j.cell.2020.06.043.
doi: 10.1002/jmv.27601.
55.
Zhang, J., Xiao, T., Cai, Y., Lavine, C. L., Peng, H.,
43.
McCallum, M., De Marco, A., Lempp, F. A.,
et al. (2021) Membrane fusion and immune evasion by
Tortorici, M. A., Pinto, D., et al. (2021) N-terminal
the spike protein of SARS-CoV-2 Delta variant, Science,
domain antigenic mapping reveals a site of vulnerability
374, 1353-1360, doi: 10.1126/science.eabl9463.
for SARS-CoV-2, Cell,
184,
2332-2347.e16, doi:
56.
Planas, D., Veyer, D., Baidaliuk, A., Staropoli, I.,
10.1016/j.cell.2021.03.028.
Guivel-Benhassine, F., et al.
(2021) Reduced
44.
Pastorio, C., Zech, F., Noettger, S., Jung, C., Jacob, T.,
sensitivity of SARS-CoV-2 variant Delta to antibody
et al. (2022) Determinants of spike infectivity, processing
neutralization, Nature, 596, 276-280, doi: 10.1038/
and neutralization in SARS-CoV-2 Omicron subvariants
s41586-021-03777-9.
BA.1 and BA.2, bioRxiv, doi: 10.1101/2022.04.13.488221.
57.
Lopez Bernal, J., Andrews, N., Gower, C., Gallagher, E.,
45.
Harvey, W. T., Carabelli, A. M., Jackson, B., Gupta, R.
Simmons, R., et al. (2021) Effectiveness of Covid-19
K., Thomson, E. C., et al. (2021) SARS-CoV-2 variants,
vaccines against the B.1.617.2 (Delta) variant, N. Engl.
spike mutations and immune escape, Nat. Rev. Microbi-
J. Med., 385, 585-594, doi: 10.1056/NEJMoa2108891.
ol., 19, 409-424, doi: 10.1038/s41579-021-00573-0.
58.
Shen, X. (2022) Boosting immunity to Omicron, Nat.
46.
Kumar, S., Karuppanan, K., and Subramaniam, G.
Med., 28, 445-446, doi: 10.1038/s41591-022-01727-0.
(2022) Omicron (BA. 1) and sub-variants (BA.1,
59.
Nishiura, H., Ito, K., Anzai, A., Kobayashi, T.,
BA.2 and BA.3) of SARS-CoV-2 Spike infectivi-
Piantham, C., et al. (2021) Relative reproduction
ty and pathogenicity: a comparative sequence and
number of SARS-CoV-2 Omicron (B.1.1.529) com-
structural-based computational assessment, bioRxiv,
pared with Delta variant in South Africa, J. Clin. Med.,
doi: 10.1101/2022.02.11.480029.
11, 30, doi: 10.3390/jcm11010030.
47.
Starr, T. N., Greaney, A. J., Addetia, A., Hannon,
60.
Venkatakrishnan, A., Anand, P., Lenehan, P. J.,
W. W., Choudhary, M. C., et al. (2021) Prospective
Suratekar, R., Raghunathan, B., et al. (2021) Omicron
mapping of viral mutations that escape antibodies
variant of SARS-CoV-2 harbors a unique insertion
used to treat COVID-19, Science, 371, 850-854, doi:
mutation of putative viral or human genomic origin,
10.1126/science.abf9302.
OSF Preprints, doi: 10.31219/osf.io/f7txy.
48.
Liu, Z., VanBlargan, L. A., Bloyet, L.-M., Rothlauf, P. W.,
61.
Abbas, Q., Kusakin, A., Sharrouf, K., Jyakhwo, S.,
Chen, R. E., et al. (2021) Identification of SARS-
and Komissarov, A. S. (2022) Follow-up investigation
CoV-2 spike mutations that attenuate monoclonal and
and detailed mutational characterization of the SARS-
serum antibody neutralization, Cell Host Microbe, 29,
CoV-2 Omicron variant lineages (BA.1, BA.2, BA.3
477-488.e4, doi: 10.1016/j.chom.2021.01.014.
and BA.1.1), bioRxiv, doi: 10.1101/2022.02.25.481941.
БИОХИМИЯ том 87 вып. 12 2022
2064
МИНГАЛЕЕВА и др.
62. Pan, T., Chen, R., He, X., Yuan, Y., Deng, X., et al.
65. Pedro, N., Silva, C. N., Magalhães, A. C., Cavadas, B.,
(2021) Infection of wild-type mice by SARS-CoV-2
Rocha, A. M., et al. (2021) Dynamics of a dual
B.1.351 variant indicates a possible novel cross-species
SARS-CoV-2 lineage co-infection on a prolonged
transmission route, Signal Transduct. Target. Ther., 6,
viral shedding COVID-19 case: insights into clinical
420, doi: 10.1038/s41392-021-00848-1.
severity and disease duration, Microorganisms, 9, 300,
63. Xu, Z., Liu, K., and Gao, G. F. (2022) Omicron
doi: 10.3390/microorganisms9020300.
variant of SARS-CoV-2 imposes a new challenge for
66. Focosi, D., and Maggi, F. (2022) Recombination in
the global public health, Biosaf. Health, 4, 147-149,
coronaviruses with a focus on SARS-CoV-2, Viruses,
doi: 10.1016/j.bsheal.2022.01.002.
14, 1239, doi: 10.3390/v14061239.
64. Tegally, H., Moir, M., Everatt, J., Giovanetti, M.,
67. Yamasoba, D., Kimura, I., Nasser, H., Morioka, Y.,
Scheepers, C., et al. (2022) Emergence of SARS-CoV-2
Nao, N., et al. (2022) Virological characteristics of
Omicron lineages BA.4 and BA.5 in South Africa, Nat.
SARS-CoV-2 Omicron BA.2 spike, Cell, 185, 2103-
Med., 28, 1785-1790, doi: 10.1038/s41591-022-01911-2.
2115.e19, doi: 10.1016/j.cell.2022.04.035.
BIOLOGY OF THE SARS-CoV-2 CORONAVIRUS
Review
R. N. Mingaleeva1*, N. A. Nigmatulina2, L. M. Sharafetdinova1, A. M. Romozanova1,
A. G. Gabdulkhakova1, Yu. V. Filina1, R. F. Shavaliyev2, A. A. Rizvanov1, and R. R. Miftakhova1
1 Kazan (Volga Region) Federal University, 420008 Kazan, Russia; E-mail: rimma.mingaleeva@gmail.com
2 Republican Clinical Hospital of the Ministry of Health of the Republic of Tatarstan, 420064 Kazan, Russia
The new coronavirus infection Covid-19, which emerged in late 2019 in China, caused a severe social and
economic crisis that affected the whole world. The high frequency of errors in the replication of RNA viruses,
the zoonotic nature of transmission, and high transmissibility allowed betacoronaviruses to cause the third
pandemic in the world since the beginning of 2003: SARS-CoV in 2003, MERS-CoV in 2012 and SARS-
CoV-2 in 2019. The latest pandemic brought the scientific community together and provided a powerful
impetus to the study of the biology of coronaviruses: new ways of penetration of the virus into the human body
have been identified, the features of the replication cycle have been studied, and new functions of coronavirus
proteins have been identified. It should be taken into account that the pandemic was accompanied by the need
to obtain and publish results in a short time, which led to the emergence of an array of conflicting data and low
reproducibility of research results. We systematized and analyzed the scientific literature, filtered the results
according to the reliability of the methods of analysis used, and prepared a review describing the molecular
mechanisms of the functioning of the SARS-CoV-2 coronavirus. The paper considers the organization of
the genome of the SARS-CoV-2 virus, the mechanisms of its gene expression and the entry of the virus into
the cell, provides information on key mutations that characterize different variants of the virus, and their
contribution to the pathogenesis of the disease.
Keywords: SARS-CoV-2, Covid-19, S-protein, mutation, VOC
БИОХИМИЯ том 87 вып. 12 2022