БИОХИМИЯ, 2023, том 88, вып. 10, с. 2014 - 2031
УДК 577.122
ПРОТЕОМНЫЙ ПОДХОД ДЛЯ ИССЛЕДОВАНИЯ
ЭКСПРЕССИИ, ЛОКАЛИЗАЦИИ И ФУНКЦИЙ БЕЛКОВОГО
ПРОДУКТА ГЕНA SOWAHD В ПРОЦЕССЕ
ГРАНУЛОЦИТАРНОЙ ДИФФЕРЕНЦИРОВКИ
© 2023 С.Е. Новикова*, Т.В. Толстова, Н.А. Соловьева*, Т.Е. Фарафонова,
О.В. Тихонова, Л.К. Курбатов, А.Л. Русанов, В.Г. Згода*
НИИ биомедицинской химии имени В.Н. Ореховича,
119121 Москва, Россия; электронная почта: novikova.s.e3101@gmail.com, victor.zgoda@gmail.com
Поступила в редакцию 22.06.2023
После доработки 23.08.2023
Принята к публикации 26.08.2023
Каталогизация белков человека, определение уровня их содержания, клеточной локализации,
выполняемой функции и потенциальной медицинской значимости являются важными задачами,
стоящими перед мировым протеомным сообществом. В настоящее время локализация и функ-
ции белковых продуктов для почти половины белок-кодирующих генов неизвестны или слабо
изучены. Исследование протеома органелл является многообещающим подходом для выявления
локализации и функций белков человека. Протеом клеточного ядра представляет особый интерес,
поскольку многие белки с ядерной локализацией, например, транскрипционные факторы, выпол-
няют регуляторные функции, определяющие судьбу клеток. По результатам метаанализа ядерного
протеома, или нуклеома, клеток линии HL-60 под действием полностью-транс-ретиноевой кис-
лоты (ATRA) выявили, что функция и локализация белкового продукта гена SOWAHD слабо изуче-
на, кроме того, отсутствует исчерпывающая информация об экспрессии SOWAHD на уровне белка.
В клетках линии HL-60 для белок-кодирующего гена SOWAHD определили экспрессию мРНК
на уровне 6,4 ± 0,7 транскриптов на миллион молекул. С помощью направленной масс-спектромет-
рии измерили содержание белка SOWAHD (белок 58, содержащий домен анкириновых повторов)
в диапазоне 0,27-1,25 фмоль/мкг общего белка. С применением пульсового мечения стабильными
изотопами определили, что время полураспада для белкового продукта гена SOWAHD составляет
приблизительно 19 ч. Протеомное профилирование ядерной фракции клеток линии HL-60 пока-
зало, что содержание белка SOWAHD увеличивалось в процессе ATRA-индуцированной грануло-
цитарной дифференцировки, с пиком в 9 ч после добавления индуктора и с последующим сниже-
нием в более поздние временные точки. Результаты исследования впервые указывают на ядерную
локализацию и вовлечённость белкового продукта гена SOWAHD в индуцированную гранулоци-
тарную дифференцировку.
КЛЮЧЕВЫЕ СЛОВА: белковый продукт гена SOWAHD, транскриптом, протеом, ATRA, ядро клетки,
масс-спектрометрия, пульсовое мечение стабильными изотопами, динамика синтеза и деградации белка.
DOI: 10.31857/S0320972523100196, EDN: OYBYYU
ВВЕДЕНИЕ
клеточной локализации и выполняемой функ-
ции, а также выявление потенциальной био-
Белки представляют собой конечные про-
логической и медицинской значимости белков
дукты экспрессии генов. Именно они выпол-
являются основными задачами международ-
няют различные функции в живых клетках,
ного проекта «Протеом человека», запущенно-
опосредуя их жизнедеятельность. Каталогиза-
го в 2010 г. В рамках проекта для регистрации
ция, оценка уровня экспрессии, определение экспрессии белков и определения её уровня,
Принятые сокращения: клетки линии HL-60 - клетки острого миелоидного лейкоза; ATRA - полностью-транс-
ретиноевая кислота; SILAC - стабильное изотопное мечение клеток аминокислотами в культуре; SIS - меченные
стабильными изотопами пептидные стандарты; SOWAHD - белок 58, содержащий домен анкириновых повторов;
SRM - мониторинг выбранных реакций; TMT - изобарные тандемные метки.
* Адресат для корреспонденции.
2014
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2015
т.е. для каталогизации, применяют масс-
упущенных» белков покрытие протеома уве-
спектрометрию и используют моноклональные
личилось с 69,8 до 90,4% от числа всех белок-
антитела, а одним из практических резуль-
кодирующих генов [5].
татов является создание баз знаний, таких
Масс-спектрометрический анализ благо-
как Uniprot [1], NextProt [2], Human Protein
даря высокой производительности и возмож-
Atlas [3] и т.д., содержащих исчерпывающую
ности своего рода «секвенирования» участков
информацию о протеоме человека [4, 5].
аминокислотной последовательности хорошо
По разным источникам, у человека на се-
зарекомендовал себя для обнаружения «недо-
годняшний день насчитывается от 19 379 до
стающих/упущенных» белков [2, 7, 8]. Успеш-
21 306 белок-кодирующих генов [6]. С точки
ное детектирование таких белков было, с
зрения экспрессии, согласно критериям меж-
одной стороны, продиктовано развитием масс-
дународной организации
«Протеом челове-
спектрометрического метода, в том числе со-
ка» (Human Proteome Organization (HUPO)),
вершенствованием масс-анализаторов, приме-
продукты потенциальных белок-кодирующих
нением улучшенных способов пробоподго-
генов в зависимости от доказанности их су-
товки, использованием фракционирования и
ществования на уровне протеома (protein
изобарных меток. С другой стороны, привле-
existence, PE) можно разделить на 5 групп
чение специфических типов биологического
(PE1-PE5) [5]. Наиболее надёжно подтвер-
материала, таких как плаценты, репродуктив-
ждённые белковые продукты получают статус
ные и эмбриональные ткани, позволило заре-
PE1, в то время как остальные (PE 2, 3, 4, 5)
гистрировать «недостающие/упущенные» бел-
представляют собой так называемые «недо-
ки [9]. Исследование белкового состава от-
стающие/упущенные» (missing) белки. Для их
дельных органелл в качестве потенциального
белок-кодирующих генов ранее могли быть
источника «недостающих/упущенных» белков
обнаружены транскрипты у человека (PE2)
также заслуживает внимания как один из под-
или могли быть предсказаны ортологи бел-
ходов для углубления знания о протеоме чело-
ков, подтверждённых у других биологических
века. Особый интерес представляет протеом-
видов (PE3), или белковый продукт был пред-
ный состав клеточного ядра.
сказан с помощью биоинформатических моде-
Подтверждение экспрессии белковых про-
лей (PE4), при этом подтверждённые данные
дуктов или каталогизация, выполненная на
об экспрессии белка у человека отсутствуют.
данный момент времени для более чем 90%
Согласно промежуточным итогам проекта
белок-кодирующих генов [5], является лишь
«Протеом человека», подведённым в 2020 г.,
одной из задач, поставленных перед про-
именно за счёт обнаружения «недостающих/
ектом «Протеом человека». Более сложным
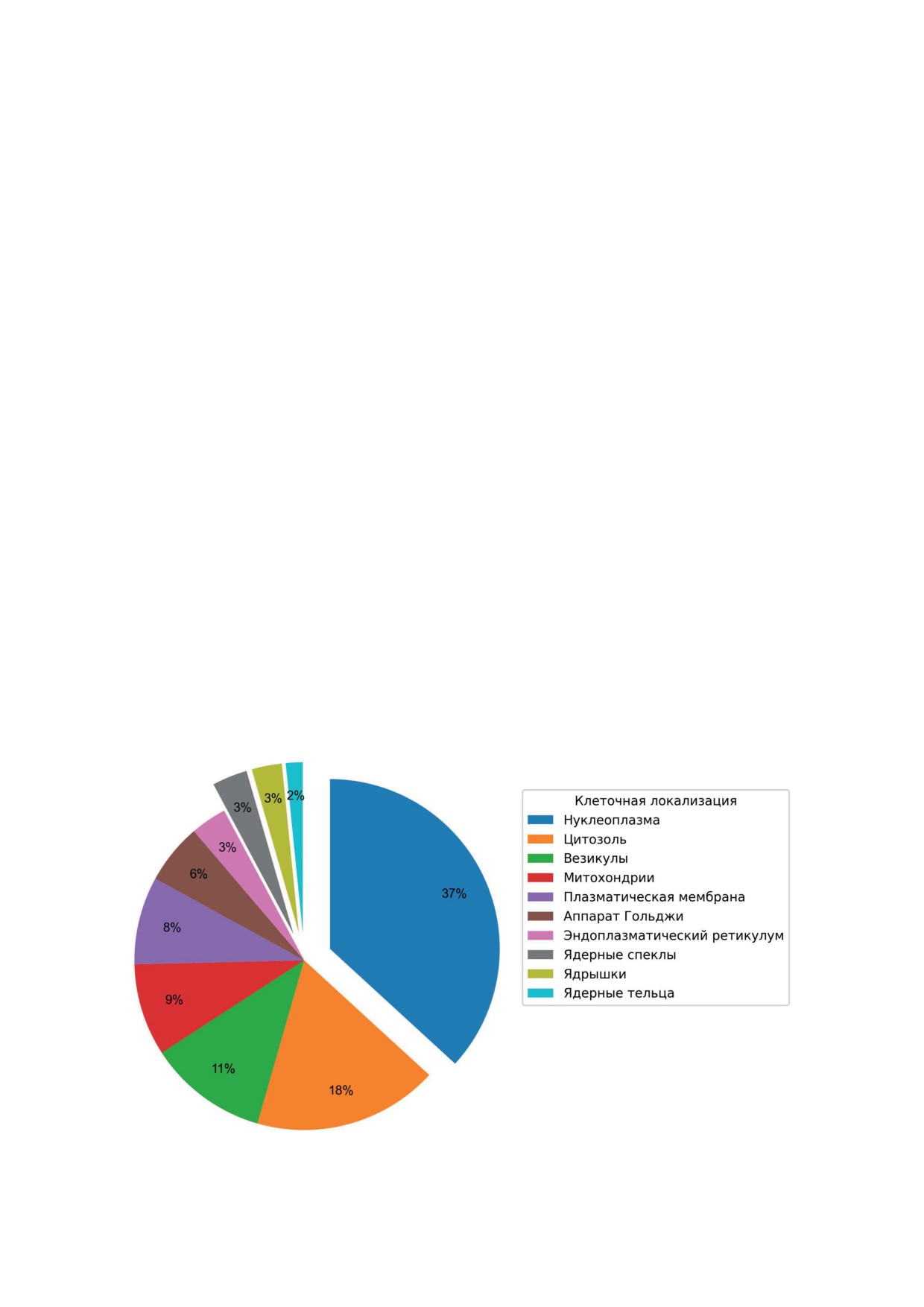
Рис. 1. Круговая диаграмма, демонстрирующая распределение белков с известной клеточной локализацией соглас-
но данным Human Proteome Atlas (версия 22.0). Показаны первые десять групп с наибольшим количеством белков.
Стоит отметить, что среди белков с известной клеточной локализацией примерно треть обнаруживается в ядре
БИОХИМИЯ том 88 вып. 10 2023
2016
НОВИКОВА и др.
представляется определение локализации, функ-
об экспрессии продукта гена SOWAHD только
ции и медицинской значимости белков с уже
на уровне транскрипта (PE2), в то же время,
установленной экспрессией. Для многих бел-
по материалам базы данных NextProt, белок
ков такая информация отсутствует в основных
SOWAHD (белок 58, содержащий домен анки-
протеомных базах данных.
риновых повторов) был зарегистрирован масс-
Клеточную локализацию белков можно
спектрометрическим методом в ряде про-
определить с помощью визуализации их ком-
теомных экспериментов (PE1), в основном в
плексов с флюоресцентно мечеными монокло-
клетках крови, ткани плаценты и тестикулах.
нальными антителами методом микроскопии.
При этом, согласно основным протеомным
Основные результаты, полученные с помо-
базам данных, включая наиболее детальную
щью такого подхода, обобщены в базе дан-
базу знаний NextProt, локализация, функция
ных Human Proteome Atlas [10]. Версия этого
и медицинская значимость белка SOWAHD
ресурса, выпущенная в 2022 г. (версия 22.0),
неизвестны. В результате для белкового про-
содержит данные для 17 200 белок-кодирующих
дукта гена SOWAHD осуществили всесторон-
генов человека, что составляет примерно 86%
ний транскриптомный и протеомный анализ
от их общего числа. При этом только для 13 105
для разъяснения его локализации и вероят-
(около 61%) известна информация о клеточной
ной биологической функции, а также времени
локализации (рис. 1).
полураспада белкового продукта в процессе
Определение функции белка является наи-
гранулоцитарной дифференцировки.
более сложной задачей. Зачастую роль белка
изучают в контексте определённых заболева-
ний, чаще всего наиболее социально значимых,
МАТЕРИАЛЫ И МЕТОДЫ
таких как онкологические и кардиоваскуляр-
ные заболевания, а также болезни центральной
Для количественной оценки белка SOWAHD
нервной системы. В ходе выполнения проекта
были использованы данные протеомного про-
«Протеом человека» активно используется так
филирования с использованием изобарных
называемый хромосомо-центричный подход,
тандемных меток (TMT, Tandem Mass Tag),
когда стоит задача в первую очередь доказать
опубликованные ранее [13]. Эксперименты по
экспрессию всех белков, кодируемых каждой
направленной масс-спектрометрии, пульс-ме-
хромосомой. В таком случае накопление ин-
чению SILAC, транскриптомному анализу про-
формации об экспрессии белкового продукта
ведены непосредственно для данного исследо-
может значительно опережать поступление дан-
вания.
ных о функциональных особенностях белков и
Метаанализ данных по протеомике ядра.
их медицинской значимости.
Для белков, ранее идентифицированных масс-
Белки, входящие в состав ядра, представля-
спектрометрическим методом в ядре клеток
ют особый интерес, поскольку к ним относят-
линии HL-60 под действием ATRA, получили
ся важные регуляторы - транскрипционные
информацию об аннотационном статусе, био-
факторы, коактиваторы и корепрессоры тран-
логической функции, клеточной локализации,
скрипции, ферменты, изменяющие эпигене-
основных публикациях и об аннотации по ка-
тический статус клетки, белки, принимающие
тегориям базы знаний GeneOntology (GO) из
участие в репликации и репарации ДНК [11].
базы данных Uniprot (версия 2023_01).
Исследование протеома ядра в модели инду-
Культивирование клеток линий HL-60, NB4,
цированной гранулоцитарной дифференциров-
K562, HepG2, HEK293 и HeLa. Культуры кле-
ки позволило выявить динамическую пертур-
ток линий HL-60 и K562 были получены из
бацию регуляторных молекул, в том числе и
криобанка «Коллекция культур клеток позво-
транскрипционных факторов [12, 13].
ночных» Института цитологии РАН (Санкт-
Мы провели поиск
«недостающих/упу-
Петербург, Россия). Линия NB4 была закупле-
щенных» белков, а также белков, функция и
на в компании «CLS Cell Lines Service GmbH»
локализация которых плохо исследованы, ис-
(Германия). Культуры клеток линий HepG2,
пользуя масс-спектрометрические данные для
HEK293 и HeLa были взяты из криобанка
ядер клеток линии острого миелоидного лей-
НИИ биомедицинской химии им. В.Н. Орехо-
коза (HL-60) под действием полностью-транс-
вича (ИБМХ, Москва, Россия). Идентичность
ретиноевой кислоты (ATRA), опубликованные
всех клеточных линий была подтверждена
нами ранее [13]. Для белкового продукта гена
ДНК-профилем (анализ коротких тандемных
SOWAHD информация об экспрессии в основ-
повторов, STR).
ных базах данных оказалась противоречивой.
После разморозки клетки 3 раза отмы-
В базе данных Uniprot указана информация
вали от DMSO в ростовой среде RPMI-1640
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2017
(«Gibco», США) с добавлением фетальной
стабильного изотопного мечения клеток ами-
сыворотки крупного рогатого скота (ФБС)
нокислотами в культуре (SILAC, Stable Isotope
до конечной концентрации 10% (v/v) с помо-
Labeling by/with Amino acids in Cell culture).
щью центрифугирования. Культуры культи-
При использовании данного подхода клетки
вировали в ростовой среде RPMI-1640 с до-
культивируют в присутствии так называемых
бавлением ФБС до конечной концентрации
«тяжёлых» аминокислот, содержащих атомы
10% (v/v) в присутствии 100 ед./мл пеницил-
стабильных изотопов 13C и 15N. В клетках «тя-
лина, 100 ед./мл стрептомицина и 2 мМ L-глу-
жёлые» аминокислоты используются при син-
тамина (все реактивы - «Gibco») в СО2-инку-
тезе белка вместо нативных, или «лёгких».
баторе в стандартных условиях (37 °С, 5% СО2,
Масс-спектрометрический анализ позволяет
80% влажности) в течение 7 суток. При дости-
оценить соотношение «лёгких» и «тяжёлых»
жении концентрации 1 млн кл./мл культуры
пептидов в различные моменты времени куль-
рассеивали в соотношении 1 : 3. Подсчёт кле-
тивирования. Данное соотношение исполь-
ток осуществляли в камере Горяева.
зуют для построения математической модели,
Индуцированная гранулоцитарная диффе-
описывающей кинетику деградации белка.
ренцировка клеток линии HL-60. Для диффе-
Такой моделью является кинетическое уравне-
ренцировки клетки линии HL-60 помещали в
ние реакции первого порядка [19, 20]. В дан-
15 мл ростовой среды в концентрации 0,3 млн
ном исследовании пульсовое мечение SILAC
кл./мл и добавляли ATRA («Sigma-Aldrich»,
комбинировали с направленным масс-спек-
США) до конечной концентрации 10 мкM.
трометрическим анализом в режиме монито-
Изучение динамики протеома в процессе
ринга выбранных реакций (Selected Reaction
индуцированной гранулоцитарной диффе-
Monitoring, SRM) вместо панорамной масс-
ренцировки ранее проводилось по различ-
спектрометрии. Такой подход позволяет про-
ным временным схемам: 0, 12 и 48 ч [14]; 0,
водить анализ низкокопийных белков, а также
24, 48, 72 и 96 ч [15]; 0, 3, 24 и 96 ч [12]; 0, 3,
обеспечивает более точное определение вре-
24, 48 и 96 ч [16]. Поверхностный маркер гра-
мени полураспада белка [21]. Время полу-
нулоцитарной дифференцировки CD11b в
распада белка SOWAHD (t1/2) вычисляли по
разы увеличивает свою экспрессию уже к 24 ч
уравнению аппроксимирующей функции для
после обработки ATRA [17], а к 72 ч клетки
зависимости деградации белка SOWAHD от
приобретают фенотип зрелых гранулоцитов
времени с учётом времени удвоения клеток
(нейтрофилов). При этом изменения в про-
линии HL-60 (48 ч).
теоме, которые в том числе ответственны за
На основании аппроксимирующей функ-
экспрессию специфических белков нейтрофи-
ции зависимости деградации белка SOWAHD
лов, могут происходить на начальных стадиях
от времени определяли общее время полурас-
гранулоцитарной дифференцировки. Чтобы
пада, не учитывающее деление клеток, по сле-
выявить ранние пертурбации протеома, мы
дующей формуле:
взяли временные точки 0, 3, 6, 9 и 12 ч, а также
точку 72 ч как ту, в которой клетки приобрета-
t1/2общ = e(L0-b)/a,
ют фенотип зрелых гранулоцитов под воздей-
ствием индуктора.
где L0 - ½ от значения интенсивности дочер-
Для протеомного анализа ядерных фрак-
него иона (MS2, фрагмент y10+, -GGPSGP
ций клетки осаждали с помощью центрифуги-
GSSR) пептида AEELGGPSGPGSSR во вре-
рования и 2 раза промывали осадок фосфат-
менной точке 0 ч, a и b - коэффициенты ап-
но-солевым буфером (PBS) («Sigma-Aldrich»).
проксимирующей функции.
В качестве контроля использовали интактные
Время полураспада белка корректирова-
клетки линии HL-60. Для протеомного экспе-
ли с учётом деления клеток: вычисляли об-
римента клетки линии HL-60 были индуци-
щую константу деградации (kобщ) по формуле
рованы ATRA в трёх биологических повторах.
kобщ = ln2/t1/2общ и константу деления с учётом
Эксперименты по дифференцировке были
времени удвоения (tудв) клеток линии HL-60
проведены в пределах 15-20 пассажей от вы-
(48 ч) по формуле kдел = ln2/tудв, вычисляли
вода клеток из заморозки.
время полураспада белка (t1/2) по формуле t1/2 =
Культивирование интактных клеток линии
= ln2/(kобщ - kдел).
HL-60 для введения изотопно-меченных амино-
Время полураспада белкового продукта
кислот лизина и аргинина. Динамику синтеза
SOWAHD (t1/2) было расcчитано отдельно для
и деградации белка SOWAHD определяли с
каждого их трёх технических повторов с ис-
помощью пульсового мечения стабильными
пользованием аппроксимирующей функции
изотопами [18]. В основе лежит технология
для зависимости деградации белка SOWAHD
20
БИОХИМИЯ том 88 вып. 10 2023
2018
НОВИКОВА и др.
от времени, с последующим вычислением сред-
ток (приблизительно 5 млн клеток) добавляли
него значения. Результаты приведены в табли-
200 мкл буфера, содержащего 10 мМ HEPES-
це 1П Приложения 1 (лист «SRM SILAC»).
NaOH (pH 7,9),
1,5 мМ MgCl2,
10 мМ KCl,
После выхода из заморозки клетки линии
0,1 мМ ЭДТА, 0,32 М сахарозу, а также кок-
HL-60 культивировались в условиях, описан-
тейль ингибиторов протеаз (все реагенты -
ных выше. Затем интактные клетки линии
«Sigma Aldrich»), и 3-5 раз перемешивали пи-
HL-60 были помещены в среду культивиро-
петированием. Затем добавляли ещё 200 мкл
вания из набора SILAC Protein Quantitation
вышеописанного буфера, содержащего, поми-
Kit (Trypsin) RPMI 1640 («Thermo Fisher Sci-
мо прочих компонентов, 0,5%-ный (m/v) NP-40
entific», США), в которую были добавлены «тя-
(«Pierce», США), 3-5 раз перемешивали и ин-
жёлые» аминокислоты 13C6 15N2 L-лизин-2
кубировали 10 мин во льду. Полученную су-
HCl и 13C6 15N4 L-аргинин HCl («Thermo
спензию наслаивали на 800 мкл буфера, содер-
Fisher Scientific») в количестве, рекомендован-
жащего 10 мМ HEPES-NaOH (pH 7,9), 1,5 мМ
ном производителем, а также диализованная
MgCl2, 10 мМ KCl, 0,1 мМ ЭДТА, 1,2 М саха-
ФБС («Gibco») до конечной концентрации
розу, а также коктейль ингибиторов протеаз, и
10% (v/v). Начальная концентрация клеток в
затем центрифугировали в течение
20 мин
среде составляла 0,15 млн кл./мл. Далее клетки
со скоростью 16 000 g при температуре 4 °С.
культивировали в СО2-инкубаторе в стандарт-
К осадку, представляющему собой ядерную
ных условиях (37 °С, 5% СО2, 80% влажности).
фракцию, добавляли 100 мкл лизирующего бу-
Для протеомного анализа клетки отбирали
фера, содержащего
1%-ный (m/v) SDS и
через 0, 1, 6, 9, 24, 72 и 120 ч, после чего осаж-
100 мМ Tris-HCl (pH 8,5) (все реагенты -
дали с помощью центрифугирования и 2 раза
«Sigma-Aldrich»), после чего подвергали воз-
промывали PBS («Sigma-Aldrich», США).
действию ультразвука с помощью дезинтегра-
Выделение общей РНК и полногеномное
тора со щупом («Bandelin Sonopuls», Герма-
транскриптомное секвенирование линий HL-60,
ния) с мощностью 50% в течение 2 мин во
NB4, K562, HepG2, HEK293 и HeLa. Общую
льду. Затем смесь центрифугировали в тече-
РНК выделяли при помощи фирменного на-
ние 10 мин со скоростью 16 000 g при темпе-
бора RNeasy Mini Kit («QIAGEN», США) со-
ратуре 4 °С. Супернатант, содержащий белки
гласно рекомендациям производителя. Каче-
ядерной фракции, отбирали и сохраняли для
ство выделенной РНК определяли на приборе
дальнейшего анализа. В полученных образ-
Agilent 2100 Bioanalyser («Agilent Technologies»,
цах определяли концентрацию общего бел-
США) с использованием чипов RNA 6000
ка колориметрическим методом с помощью
Nano LabChip («Agilent Technologies»). Концен-
коммерческого набора Pierce™ BCA Protein
трацию общей РНК определяли спектрофото-
Assay Kit («Pierce») в соответствии с рекомен-
метрически по поглощению при длине волны
дациями производителя. При выделении ядер-
260 нм на микроспектрофотометре ND-1000
ной фракции с помощью центрифугирования
(«Thermo Fisher Scientific»). Секвенирование
целевые белки могут быть контаминированы
было выполнено на платформе NovaSeq 6000
белками других органелл, например, мито-
Illumina с длиной прочтений 100 пар основа-
хондрий. Аннотация нуклеома клеток линии
ний в трёх технических повторах. Исходные
HL-60, идентифицированного в ходе грануло-
данные РНК-секвенирования в процессе об-
цитарной дифференцировки по категориям
работки претерпевают нормализацию на ос-
«Внутриклеточная локализация» базы данных
новании длины гена и глубины секвенирова-
«GeneOntology», показала контаминацию бел-
ния. В результате экспрессия генов выражена в
ками митохондрий на уровне 11,5%.
количестве транскриптов на миллион (ТРМ).
Гидролитическое расщепление белков. Гид-
Сумма ТРМ экспрессии для всех генов оказы-
ролитическое расщепление белков осущест-
вается одинаковой для различных образцов,
вляли согласно протоколу FASP (Filter-Aided
что предоставляет возможность сравнивать
Sample Preparation) [24] с некоторыми измене-
между собой экспрессионные данные
[22].
ниями. Для восстановления и алкилирования
Данные экспрессии генов семейства SOWAH
дисульфидных связей цистеина к образцам
приведены в таблице 1П Приложения 1 (лист
лизата клеток, содержащим 100 мкг общего
«Транскриптомные данные»).
белка, добавляли трис(2-карбоксиэтил)фос-
Пробоподготовка клеток линии HL-60 к про-
фин(TCEP) и 2-хлорацетамид (САА) («Sigma-
теомному профилированию ядерных фракций.
Aldrich») до конечных концентраций 20 мМ
Для выделения ядерной фракции мы использо-
и 32 мМ соответственно. Затем пробы инку-
вали протокол с участием градиента сахарозы,
бировали при температуре 80 °С в течение
применённый нами ранее [23]. К осадку кле-
40 мин, после чего образцы наносили на кон-
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2019
центрирующие фильтры c отсечением 30 кДа
вляли нормализацию по суммарному сигналу
и
3 раза промывали буфером, содержащим
всех белков в каждом ТМТi-канале. Данные
8 М мочевины в 0,1 М Tris-HCl (pH 8,5), путём
панорамного анализа доступны через репо-
центрифугирования при скорости 11 000 g в
зиторий PRIDE (идентификатор PXD035281)
течение 15 мин при температуре 20 °С. Затем
и репозиторий MassIVE (идентификатор
образцы 3 раза промывали буфером, содер-
MSV000092670).
жащим 0,1 М Tris-HCl (pH 8,5), и 2 раза -
C помощью направленной масс-спектро-
буфером для трипсинолиза
(50 мМ ТЭАБ,
метрии в режиме SRM с использованием SIS-
pH 8,5), путём центрифугирования при ско-
пептидов (SRM/SIS) было измерено содержа-
рости 11 000 g в течение 15 мин и температу-
ние протеотипических пептидов AAPAGWL-
ре 20 °С. Затем к каждому образцу добавляли
SEER и AEELGGPSGPGSSR, картируемых на
50 мкл 50 мМ ТЭАБ (pH 8,5), а также раствор
белок SOWAHD. Критериями выбора пептидов
трипсина («Promega», США) с концентрацией
для синтеза стандартов были: длина 9-20 ами-
0,5 мкг/мкл в соотношении массы трипсина
нокислотных остатков; отсутствие в последо-
к массе общего белка 1 : 50, после чего пробу
вательности цистеина (C), метионина (M),
инкубировали в течение ночи при темпера-
N-концевых глутаминовой кислоты (E) и глу-
туре 37 °С. По окончании инкубации пептиды
тамина (Q), а также пропущенных сайтов гид-
элюировали путём центрифугирования при
ролиза; уникальность была проверена с помо-
скорости 11 000 g в течение 15 мин при тем-
щью ресурса Peptide unicity checker - neXtProt.
пературе 20 °С и промывали фильтр 100 мкл
Выбранные для таргетного анализа пеп-
5%-ной (v/v) муравьиной кислоты. Общее со-
тиды также представлены в учётной записи
держание пептидов определяли с помощью на-
бора Pierce Quantitative Peptide Assays («Pierce»)
entry/NX_A6NJG2/peptides). Твердофазный пеп-
в соответствии с рекомендациями производи-
тидный синтез SIS-стандартов осуществляли
теля. Половину от общего количества пепти-
на автоматическом пептидном синтезаторе
дов замораживали и хранили на -80 °С, затем
Overture («Protein Technologies», Великобри-
данную аликвоту использовали для SRM-из-
тания). При синтезе изотопно-меченных пеп-
мерений, проведённых в данной работе. Дру-
тидов вместо обычного лизина или аргинина
гая половина была использована в экспери-
использовали изотопно-меченные аминокис-
ментах c TMT-метками, описанных ранее [13].
лоты Fmoc-Lys-OH-13C6,15N2 или Fmoc-Arg-
Панорамный и направленный масс-спектро-
OH-13C6,15N2 («Cambridge Isotope Laboratories»,
метрический анализ в режиме SRM с использо-
США).
ванием меченных стабильными изотопами пеп-
Направленный масс-спектрометрический
тидных стандартов (SIS). Панорамный масс-
анализ в режиме SRM проводили по следую-
спектрометрический анализ был выполнен, как
щему протоколу. Каждый экспериментальный
описано ранее [13]. Масс-спектрометрические
образец анализировали в трёх технических
данные анализировали с помощью программ-
повторах. Анализ SRM проводили для тех же
ного обеспечения MaxQuant 2.0.3.0 со встро-
образцов, которые использовались в панорам-
енным поисковым алгоритмом Andromeda.
ном масс-спектрометрическом эксперименте.
Идентификацию проводили относительно
Хроматографическое разделение осуществля-
FASTA-файла, содержащего аминокислотные
лось с помощью системы Agilent 1200 series
последовательности белков человека (2022/02)
(«Agilent Technologies»), соединённой с трой-
и его инвертированного аналога для вычисле-
ным квадрупольным масс-анализатором TSQ
ния частоты ложноположительных идентифи-
Quantiva («Thermo Fisher Scientific»). Образец
каций (FDR). Для белков и пептидов порого-
объёмом 3,5 мкл, содержащий 15 мкг натив-
вое значение FDR = 0,01.
ных пептидов и стандарты SIS, разделяли
Количественный анализ осуществляли на
c использованием аналитической колонки
основании интенсивности репортерных ионов,
ZORBAX SB-C18 (150 × 0,5 мм, диаметр частиц
образующихся при фрагментации изобарных
5 мкм, «Agilent Technologies») в градиенте аце-
ТМТ-меток, с помощью встроенного в Max-
тонитрила со скоростью потока 20 мкл/мин.
Quant алгоритма. Применяли нормализацию
В начале колонку уравновешивали 5%-ным
по взвешенной медиане с использованием
(v/v) раствором B
(80%-ный (v/v) ацетони-
каналов для всех ТМТi (i = 10) репортерных
трил в 0,1%-ной (v/v) муравьиной кислоте) и
ионов в качестве референсных. Применяли
95%-ным (v/v) раствором А (0,1%-ная (v/v)
функцию сопоставления пептидных ионов ме-
муравьиная кислота) в течение 5 мин, затем
жду ЖХ-MC/MC-экспериментами (Matching
концентрацию раствора В линейно увели-
between runs, MBR). Дополнительно осущест-
чивали до 60% (v/v) за 30 мин, после чего
БИОХИМИЯ том 88 вып. 10 2023
20*
2020
НОВИКОВА и др.
концентрацию раствора В увеличивали до
вые для стандартных пептидов (STD/SIS)
99% (v/v) за 1 мин и в течение 5 мин промы-
AEELGGPSGPGSSR и AAPAGWLSEER, кар-
вали колонку
99%-ным (v/v) раствором В,
тируемых на белок SOWAHD, приведены на
затем в течение 1 мин концентрация воз-
рис. 1П Приложения 2.
вращалась к начальным условиям, в которых
колонка уравновешивалась в течение 9 мин.
Масс-спектрометрический анализ осущест-
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЙ
вляли в режиме динамического мониторинга
выбранных реакций (dSRM), используя сле-
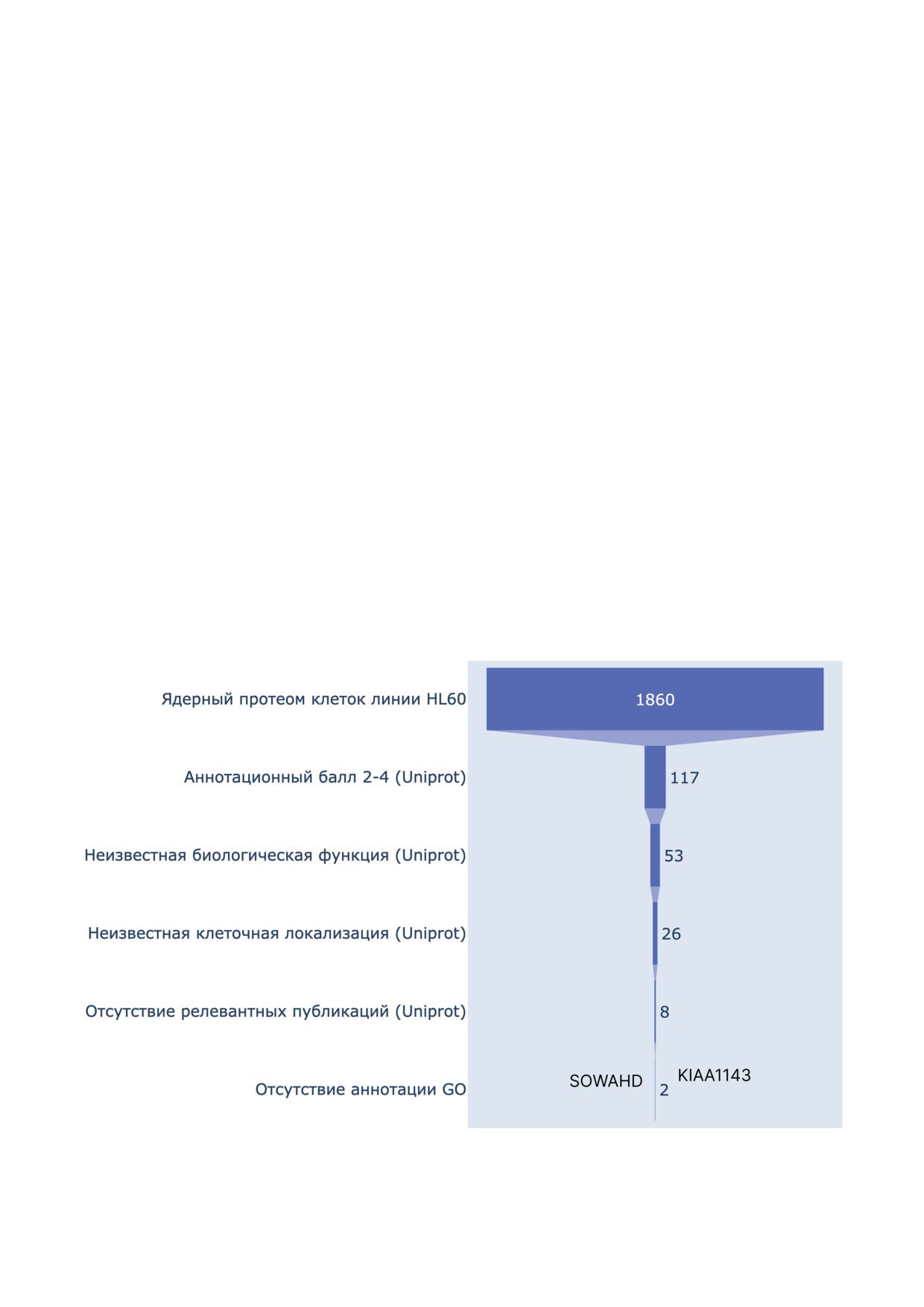
Метаанализ нуклеома клеток линии HL-60.
дующие настройки МС-детектора: напряже-
На рис. 2 показаны результаты метаанализа
ние на капилляре составляло 4000 В, скорость
для белков, ранее идентифицированных масс-
осушающего газа (азот) составляла 7 л/мин,
спектрометрическим методом в ядре клеток
скорость аксилярного газа (азот) составляла
линии HL-60 (1860 белков) (нуклеом). Исполь-
5 л/мин, температура капилляра
- 350 °С,
зуя базу знаний Uniprot (версия 2023_01), про-
окно изоляции для первого и третьего квадру-
теомные данные фильтровали на основании
поля - 0,7 Да, время цикла сканирования -
аннотационного балла Uniprot < 5 (117 бел-
1,2 с, давление газа (аргон) в ячейке соударе-
ков), затем на каждой последующей стадии
ния - 1,5 мТорр. Окно времени удерживания
оставляли белки с неизвестной биологической
на колонке обращённой фазы составило 3 мин
функцией (53 белка), с неизвестной клеточной
для каждого прекурсорного иона. Для количе-
локализацией (26 белков), белки, для которых
ственной оценки в ПО Skyline (версия 4.1.0)
в Uniprot не представлено релевантных публи-
использовались все транзиции, сигнал от ко-
каций, и, наконец, белки, для которых отсут-
торых регистрировался. Для каждой пробы со-
ствовала аннотация GO (2 белка).
держание белка определялось как содержание
По результатам фильтрации удалось вы-
соответствующего ему уникального протеоти-
явить 2 белка, удовлетворяющих всем усло-
пического пептида, измеренного в трёх био-
виям: ANKRD58 (ankyrin repeat domain 58),
логических повторах. Калибровочные кри-
кодируемый геном SOWAHD, и неохарактери-
Рис. 2. Схема фильтрации масс-спектрометрических данных, полученных для ядерного протеома клеток линии HL-60
(1860 белков), на основании аннотационного статуса, биологической функции, клеточной локализации, информации
об основных публикациях и аннотации по категориям базы знаний GO
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2021
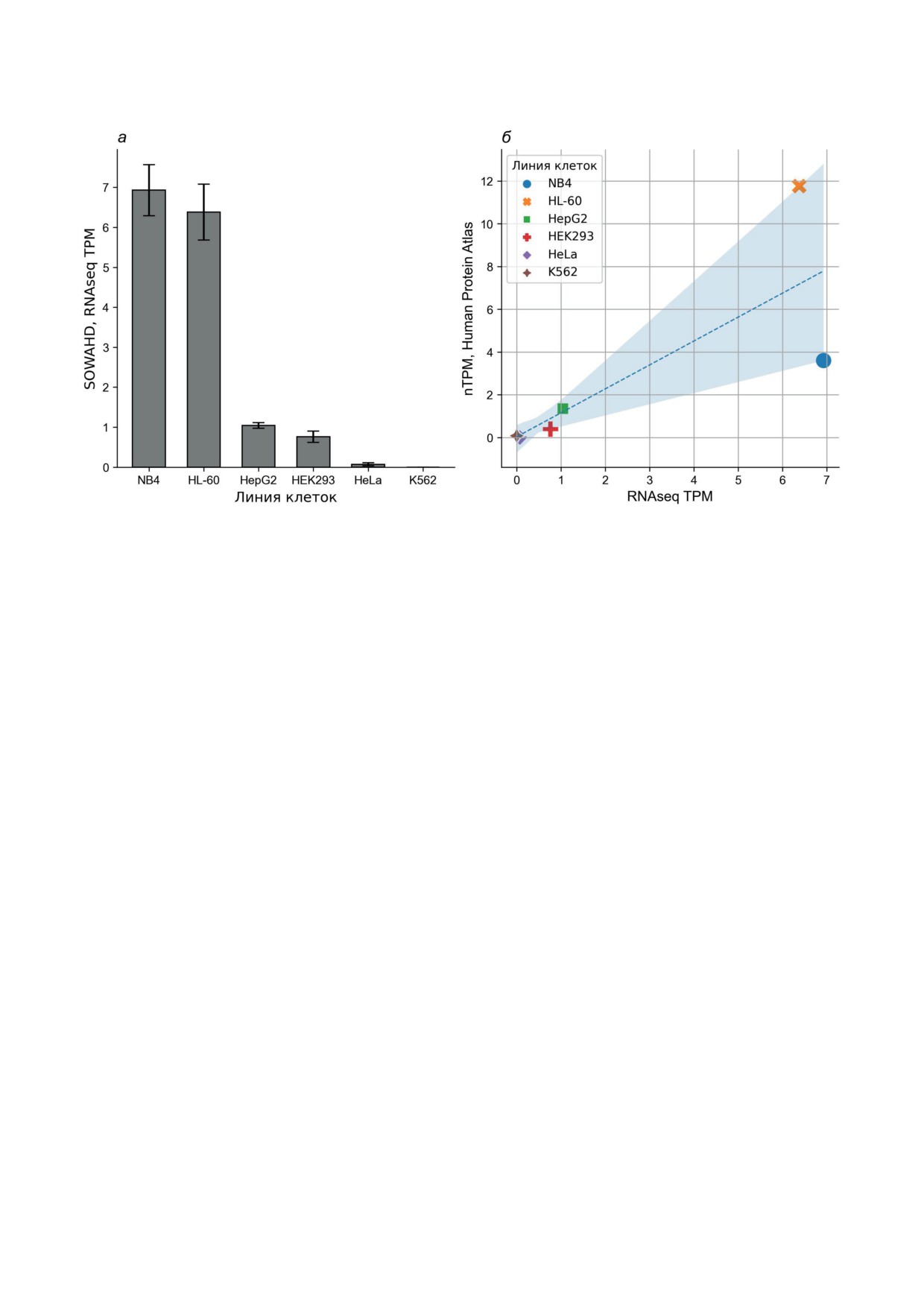
Рис. 3. Результаты транскриптомного анализа. а - Уровень экспрессии мРНК SOWAHD, измеренный с помощью пол-
ногеномного РНК-секвенирования (RNAseq) в клетках линий NB4, HL-60, HepG2, HEK293, HeLa и K562. По оси y
отложено количество транскриптов на миллион, TPM (transcripts per million). б - Корреляция наших эксперименталь-
ных данных и материалов базы знаний Protein Atlas (версия 22.0) для уровня экспрессии мРНК SOWAHD в клетках
линий NB4, HL-60, HepG2, HEK293, HeLa и K562. По оси y отложено нормализованное количество транскриптов
на миллион, nTPM, материалы ProteinAtlas (версия 22.0); по оси x - количество TPM, определённое в нашем экспе-
рименте
зованный белок KIAA1143 (ген KIAA1143, код
ответственно. В клетках хронического миело-
Uniprot Q96AT1). Для белка SOWAHD в базе
генного лейкоза линии K562 экспрессия
данных Uniprot экспрессия указана только на
мРНК SOWAHD оказалась ниже порога детек-
уровне мРНК (PE2). В то же время для гена
ции. Умеренная экспрессия мРНК SOWAHD
KIAA1143 экспрессия на белковом уровне
на уровне 1,1 ± 0,1 и 0,8 ± 0,1 TPM была заре-
подтверждена во всех основных протеом-
гистрирована в клетках эпителиального про-
ных базах данных (PE1). В структуре белка
исхождения линий HepG2 (ткань печени) и
SOWAHD имеются анкириновые повторы, за
HEK293 (эмбриональные почки). Экспрессию
счёт которых осуществляются белок-белковые
мРНК SOWAHD на уровне 0,07 ± 0,03 TPM
взаимодействия, что может указывать на регу-
наблюдали в клетках эпителиального происхо-
ляторную роль SOWAHD. Дальнейшие иссле-
ждения линии HeLa (ткань шейки матки).
дования проводили для данного белка.
Рисунок 3, б показывает корреляцию (R2 =
Экспрессия транскрипта SOWAHD в линиях
= 0,63) наших экспериментальных результатов
клеток гемопоэтического и эпителиального про-
с данными базы знаний Human Protein Atlas
исхождения. Для оценки экспрессии мРНК
(версия 22.0) для экспрессии мРНК SOWAHD
SOWAHD мы использовали эксперименталь-
в линиях клеток NB4, HL-60, HepG2, HEK293,
ные данные полногеномного РНК-секвениро-
HeLa и K562.
вания (RNAseq), полученные для гемопоэти-
Согласно транскриптомным данным, мож-
ческих клеток линий HL-60, NB4 и K562,
но предположить, что мРНК SOWAHD экс-
а также линий клеток эпителиального про-
прессируется преимущественно в клетках мие-
исхождения HeLa, HepG2 и HEK293. Кроме
лоидного происхождения. Экспрессия наблю-
того, сравнивали результаты нашего тран-
дается в клетках острого миелоидного лейкоза,
скриптомного эксперимента с данными базы
но отсутствует в клетках хронического миело-
знаний ProteinAtlas (версия 22.0). Результаты
генного лейкоза. Однако с учётом ограниче-
показаны на рис. 3.
ний для количественной оценки экспрес-
Как видно из рис. 3, а, наибольшую экс-
сии транскриптов на основании величин
прессию мРНК SOWAHD на уровне 6,9 ± 0,6
TPM [25], а также известных на сегодняшний
и 6,4 ± 0,7 TPM наблюдали в клетках острого
день фактов вариабельности омиксных данных
миелоидного лейкоза линий NB4 и HL-60 со-
даже в пределах одной клеточной линии [26],
БИОХИМИЯ том 88 вып. 10 2023
2022
НОВИКОВА и др.
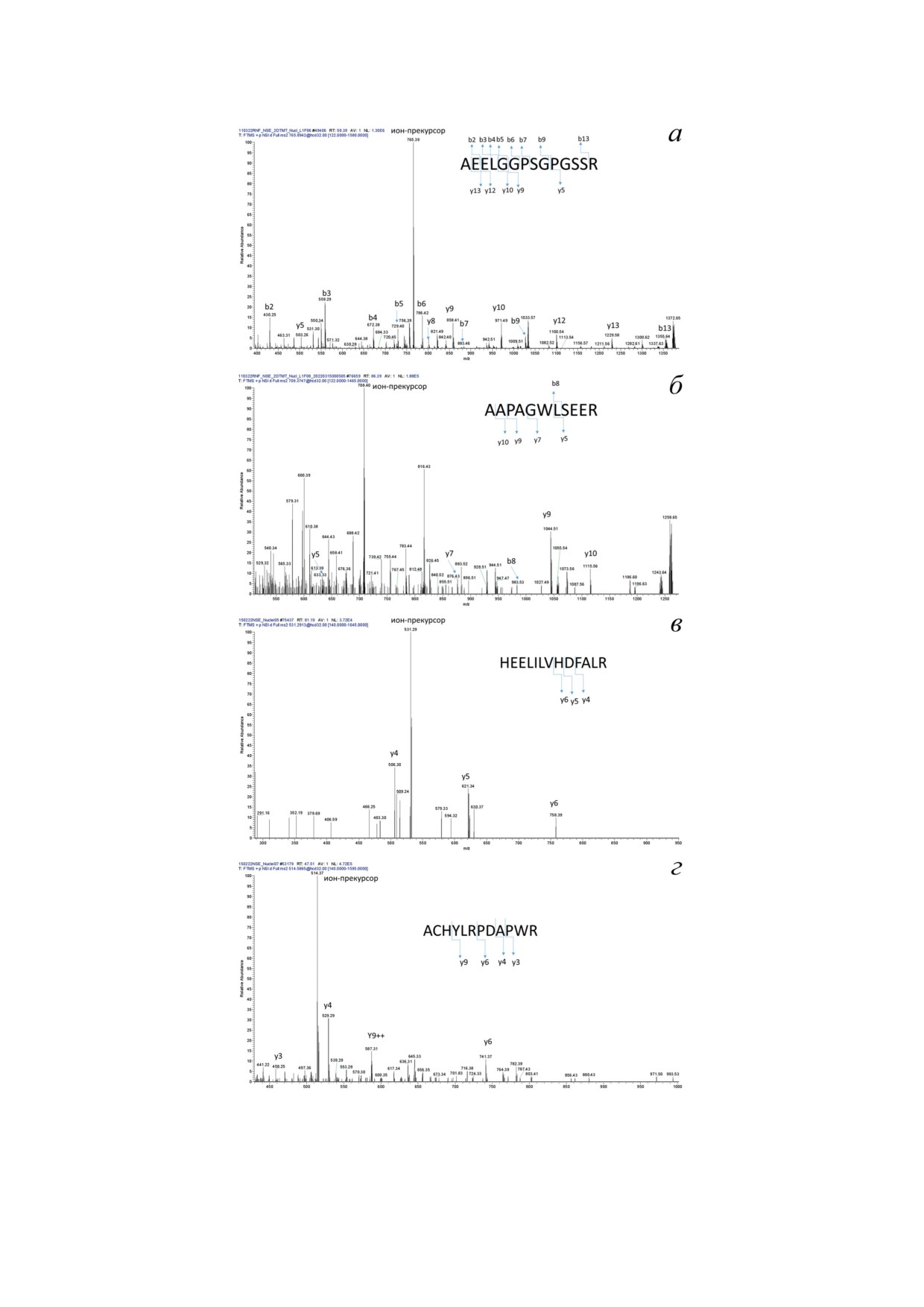
Рис. 4. MS2-cпектры высокого разрешения, полученные для уникальных пептидов AEELGGPSGPGSSR (а),
AAPAGWLSEER (б), HEELILVHDFALR (в) и ACHYLRPDAPWR (г), картируемых на белок SOWAHD. Спектры были
визуализированы в программном обеспечении Thermo Xcalibur Qual Browser (Thermo Xcalibur 3.0.63), экспортированы
в виде рисунка, после чего y- и b-фрагментарные ионы были аннотированы в ручном режиме. Пептиды были иден-
тифицированы в автоматическом режиме в ПО MaxQuant. Номер MS2-cпектра высокого разрешения с наилучшим
качеством и соответствующий исходный файл с данными были указаны в файле «evidence.txt» выдачи MaxQuant
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2023
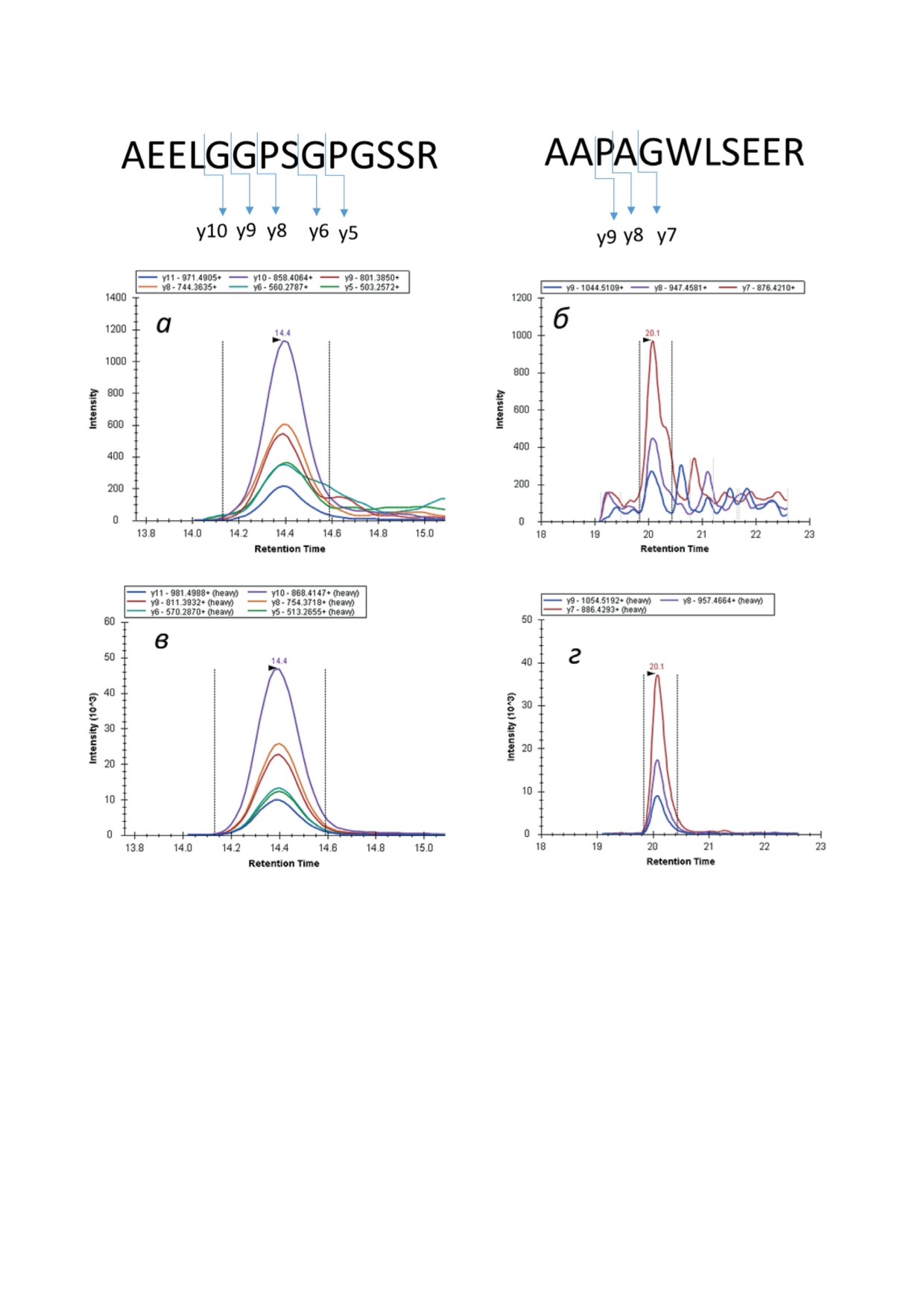
Рис.
5. Результат направленного масс-спектрометрического анализа в режиме SRM с использованием SIS
AEELGGPSGPGSSR и AAPAGWLSEER. Приведены хроматограммы экстрагированного ионного тока для фрагмен-
тарных ионов нативных пептидов (а и б) и их синтетических аналогов (в и г)
тканеспецифичность экспрессии SOWAHD тре-
ческого анализа высокого разрешения ядер
бует дальнейшей валидации с помощью орто-
клеток линии HL-60 под действием ATRA
гональных методов.
белок SOWAHD был идентифицирован по 4-м
В данной работе фокус был направлен
уникальным пептидам (ACHYLRPDAPWR,
на исследование белкового продукта гена
HEELILVHDFALR, AAPAGWLSEER и AEELG-
SОWAHD, а транскриптомные данные высту-
GPSGPGSSR). Спектры высокого разрешения
пали как дополнительное доказательство экс-
показаны на рис. 4.
прессии гена.
Рис. 4 показывает, что масс-спектроме-
Динамическая экспрессия белка SOWAHD
трический анализ высокого разрешения позво-
в ядрах клеток линии HL-60 в ходе ATRA-ин-
лил идентифицировать уникальные пептиды
дуцированной гранулоцитарной дифференци-
(FDR < 0,01), соответствующие 15,6% амино-
ровки. По результатам масс-спектрометри-
кислотной последовательности белка SOWAHD.
БИОХИМИЯ том 88 вып. 10 2023
2024
НОВИКОВА и др.
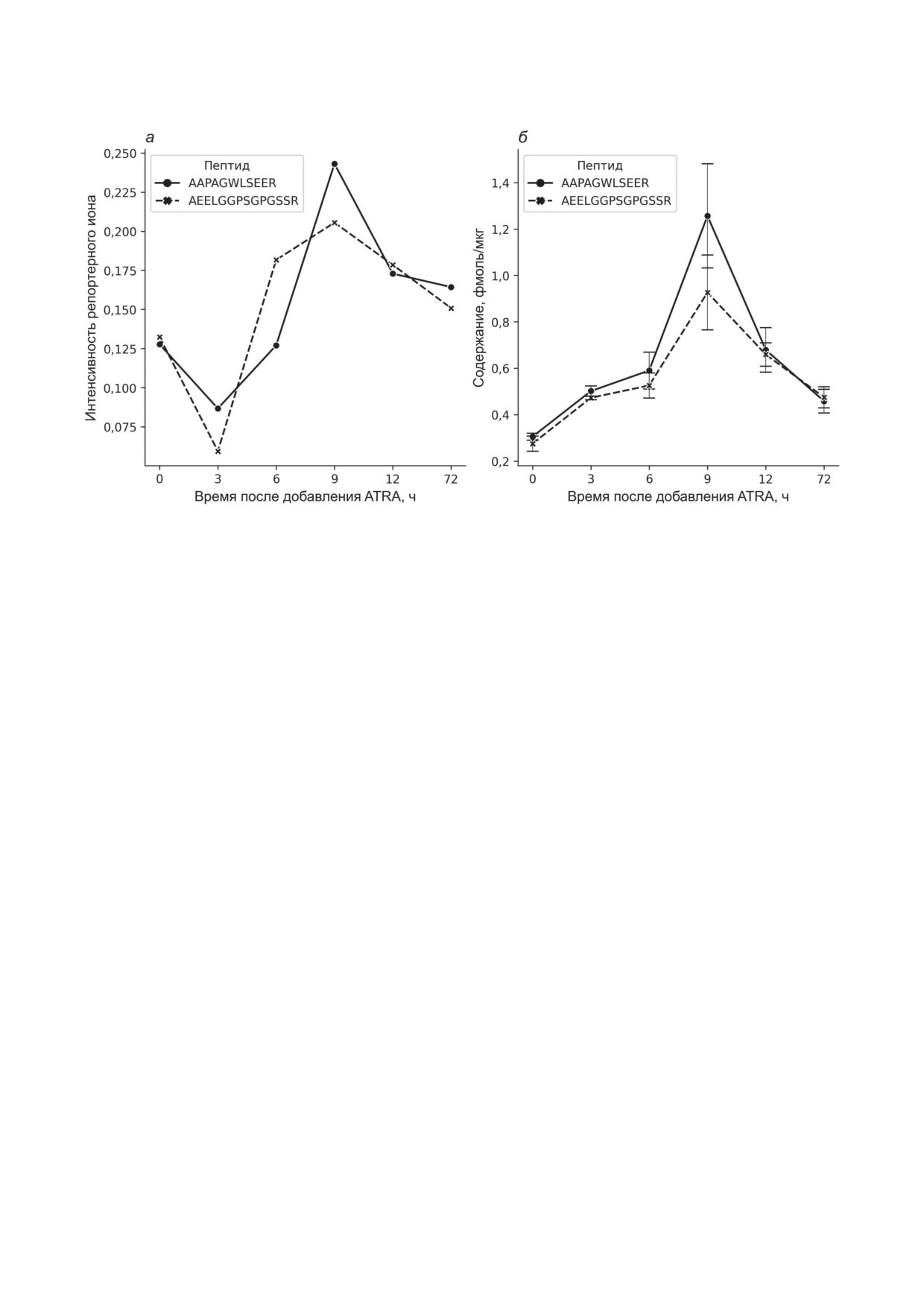
Рис. 6. Динамика экспрессии белка SOWAHD в ядерной фракции клеток линии HL-60 через 0, 3, 6, 9, 12 и 24 ч
после добавления ATRA, индуцирующей гранулоцитарную дифференцировку. а - Результаты относительного ко-
личественного масс-спектрометрического анализа высокого разрешения с использованием TMT-меток, по оси х
отложены временные точки после добавления ATRA, по оси y показана интенсивность репортерного ТМТ-иона.
Интенсивность репортерного ТМТ-иона (MS2-спектр, спектр ионов-фрагментов) для пептидов AAPAGWLSEER
и AEELGGPSGPGSSR была рассчитана в одном техническом повторе. б - Результаты количественного направленного
масс-спектрометрического анализа с использованием изотопно-меченных пептидных стандартов (SRM/SIS), по оси х
отложены временные точки после добавления ATRA, по оси y показано содержание протеотипических пептидов белка
SOWAHD в фмоль/мкг общего белка. Данные получены для уникальных протеотипических пептидов AAPAGWLSEER
и AEELGGPSGPGSSR. SRM/SIS-измерения были осуществлены в 3-х технических повторах в образцах ядер клеток
HL60, индуцированных к дифференцировке ATRA в 3-х биологических повторах; всего на каждую временную точку
приходится 9 LC/SRM-измерений
Для подтверждения идентификации белка
Уникальные пептиды AAPAGWLSEER и
SOWAHD были синтезированы SIS с последо-
AEELGGPSGPGSSR позволили осуществить
вательностями AAPAGWLSEER и AEELGGP-
количественный анализ белка SOWAHD с ис-
SGPGSSR. Изотопно-меченные и соответствую-
пользованием TMT-меток (рис. 6, а), а также
щие нативные пептиды были зарегистриро-
с использованием направленного масс-спек-
ваны методом направленного масс-спектро-
трометрического метода SRM/SIS (рис. 6, б) в
метрического анализа в режиме мониторинга
ядерной фракции клеток линии HL-60 через
выбранных реакций в клетках линии HL-60
3, 6, 9, 12 и 72 ч после добавления ATRA.
(рис. 5).
Масс-спектрометрический анализ высоко-
Рис. 5, б показывает более высокий уро-
го разрешения с TMT-метками показал увели-
вень шума для сигнала от пептида AAPA-
чение содержания белка SOWAHD в ядерной
GWLSEER по сравнению с пептидом AEEL-
фракции в 1,9 раз через 9 ч после добавления
GGPSGPGSSR. Низкая концентрация целево-
ATRA в среду культивирования клеток. По ре-
го белка, а также тот факт, что в этой области
зультатам SRM/SIS-анализа содержание белка
градиента может элюировать больше интерфе-
SOWAHD было определено на уровне 0,3 ±
рирующих пептидов, может объяснять высо-
± 0,06, 0,5 ± 0,06, 0,59 ± 0,2, 1,25 ± 0,63, 0,67 ±
кий уровень шума. С другой стороны, время
± 0,28 и
0,45 ± 0,15 фмоль/мкг по пептиду
элюирования и паттерн фрагментации полно-
AAPAGWLSEER, а также на уровне 0,27 ±
стью совпадают для нативного пептида и его
± 0,07, 0,47 ± 0,02, 0,52 ± 0,16, 0,92 ± 0,45, 0,63 ±
синтетического аналога, за счёт чего мы счи-
± 0,17 и
0,47 ± 0,13 фмоль/мкг по пептиду
таем его измерение достоверным. Кроме того,
AEELGGPSGPGSSR через 0, 3, 6, 9, 12 и 72 ч
профили экспрессии, а также абсолютное со-
после добавления ATRA соответственно. Со-
держание в различные временные точки после
держание белка SOWAHD в ядерной фрак-
добавления ATRA сходны для двух целевых
ции значимо (p-value < 0,01) увеличивалось в
пептидов, картируемых на белок SOWAHD.
1,6, 1,9, 3,7, 2,3 и 1,5 раза через 3, 6, 9, 12 и 72 ч
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2025
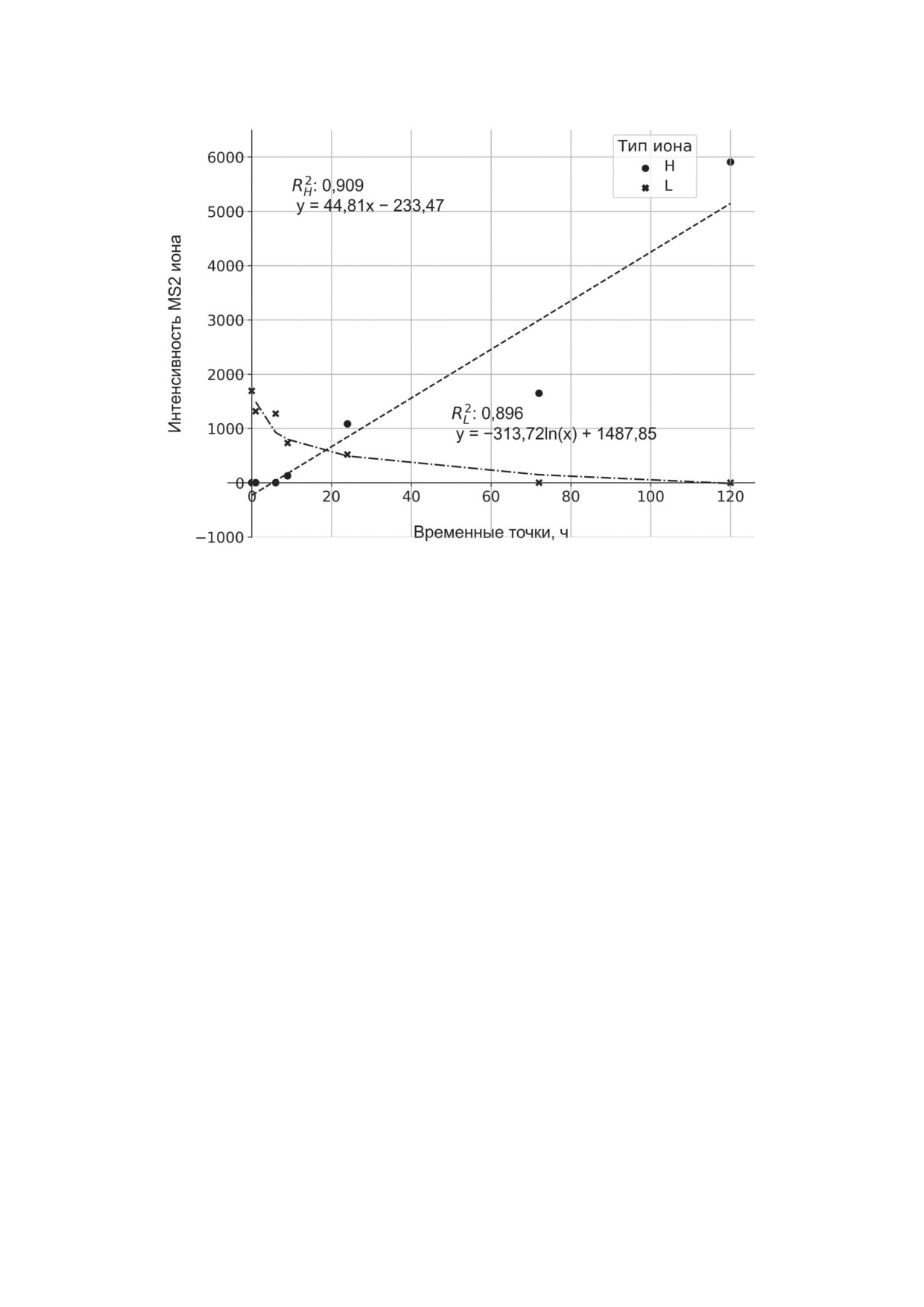
Рис. 7. Динамика синтеза и деградации белкового продукта гена SOWAHD, определённая с использованием пульсо-
вого SILAC-мечения в комбинации с направленным масс-спектрометрическим анализом SRM. Графики зависимости
площади под пиком от времени культивирования в среде SILAC для «лёгкой» (L) и «тяжёлой» (H) формы пептида
SOWAHD. По оси х отложены временные точки (0, 1, 6, 9, 24, 72 и 120 ч), по оси y - площадь под пиком для наиболее
интенсивного дочернего иона (MS2, фрагмент y10+, -GGPSGPGSSR) «лёгкой» (L) и «тяжёлой» (H) формы пептида
SOWAHD (AEELGGPSGPGSSR). Графики аппроксимирующих функций показаны штриховой (- -) линией (линей-
ная зависимость) и штрихпунктирной (-.) линией (логарифмическая зависимость) для «тяжёлой» (H) и «легкой» (L)
формы пептида SOWAHD соответственно. Представлены результаты для первого технического повтора, для второго
и третьего технического повтора графики приведены на рис. 2-3 П Приложения 2
после добавления индуктора гранулоцитарной
SILAC-мечения и направленного масс-спек-
дифференцировки соответственно. Рис. 6 по-
трометрического анализа SRM. Принцип пуль-
казывает, что профили экспрессии, получен-
сового мечения SILAC описан в разделе «Ма-
ные для пептидов AAPAGWLSEER и AEEL-
териалы и методы». Динамику встраивания
GGPSGPGSSR, оказались похожими между
13С-меченного лизина или аргинина из среды
собой.
культивирования в белок SOWAHD определяли
В силу низкого содержания белка SOWAHD,
по результатам SRM-измерений «лёгкой» (L)
а также вероятностного характера фрагмен-
и «тяжёлой» (H) формы пептида SOWAHD
тации в ходе панорамного масс-спектромет-
(AEELGGPSGPGSSR), как показано на рис. 7.
рического анализа спектры фрагментации
Как показывает рис. 7, направленный масс-
(MS2) в качестве, достаточном для вычисле-
спектрометрический анализ позволил опреде-
ния интенсивности репортерных ТМТ-ионов,
лить аппроксимирующие функции, описываю-
были получены только в одном техническом
щие динамику синтеза и деградации SOWAHD,
повторе для пептидов AAPAGWLSEER и
на основании площади под пиком для наи-
AEELGGPSGPGSSR. Тем не менее наблюда-
более интенсивного дочернего иона пептида
лась сходимость результатов, полученных с
AEELGGPSGPGSSR (MS2, фрагмент y10+,
помощью различных методов количествен-
-GGPSGPGSSR). Измерения осуществлялись
ной оценки - масс-спектрометрии высокого
для пептида AEELGGPSGPGSSR, поскольку
разрешения с TMT-метками и направленной
для него было зарегистрировано больше фраг-
масс-спектрометрии в режиме SRM.
ментов, как по данным масс-спектрометрии
Динамика синтеза и деградации белкового
высокого разрешения (рис. 4, а), так и по ре-
продукта SOWAHD. Время полураспада белково-
зультатам направленного анализа (рис. 5). Вре-
го продукта SOWAHD. Для определения дина-
мя полураспада белкового продукта SOWAHD
мики синтеза и деградации белкового продукта
(t1/2) было рассчитано в трёх технических по-
SOWAHD применили комбинацию пульсового
вторах и составило 19 ± 7 ч.
БИОХИМИЯ том 88 вып. 10 2023
2026
НОВИКОВА и др.
ОБСУЖДЕНИЕ РЕЗУЛЬТАТОВ
клеточной карциномой [28]. Экспрессия гена
SOWAHA представляет собой благоприятный
Ген SOWAHD относится к семейству
прогностический фактор в случае рака подже-
SOWAH, в которое входят ещё 3 гена: SOWAHA,
лудочной железы [29]. Исследования были вы-
SOWAHВ и SOWAHС. Согласно нашим пано-
полнены на основе биоинформатического ме-
рамным протеомным данным, только продукт
таанализа данных, полученных из ресурса The
гена SOWAHD был зарегистрирован на уровне
Cancer Genome Atlas, и только в случае с геном
белка в клетках линии HL-60. В то же время
SOWAHA была проведена валидация результа-
наши экспериментальные транскриптомные
тов с помощью транскриптомного анализа.
данные показали, что в линиях эпителиаль-
Как показали наши протеомные данные,
ного происхождения (HeLa, HepG2 и HEK293)
экспрессия белка SOWAHD, измеренная в
наибольшая экспрессия мРНК, на порядок
ядре клеток HL-60, увеличивалась в процессе
превышающая таковую для остальных генов
ATRA-индуцированной гранулоцитарной диф-
семейства, наблюдалась для гена SOWAHС,
ференцировки, что позволяет предположить
в то время как в миелоидных линиях (HL-60
ядерную локализацию белка и его значимость
и NB4) превалировала экспрессия SOWAHD
для созревания нейтрофилов. Пик увеличе-
на фоне пониженной экспрессии других генов
ния экспрессии белка приходился на 9 ч после
семейства.
добавления индуктора с последующим сниже-
Данные об экспрессии белкового продукта
нием в более поздние временные точки, что
SOWAHD отличаются в основных протеом-
может указывать на функциональную значи-
ных базах знаний Uniprot, NextProt и Human
мость белка SOWAHD для ранних стадий ин-
Protein Atlas. Так, в базе знаний Uniprot ука-
дуцированной гранулоцитарной дифференци-
зана информация об экспрессии продукта
ровки.
гена SOWAHD на уровне транскрипта (https://
Ранее с помощью направленной масс-спек-
трометрии в ядрах клеток линии HL-60 были
В базе знаний Human Protein Atlas, опираю-
зарегистрированы транскрипционные фак-
щейся на анализ белковых продуктов с помо-
торы (ТФ) HIC1, STAT1 и RBPJ на уровне
щью моноклональных антител, указано, что
~0,5-1 фмоль/мкг общего белка [13]. Количе-
экспрессия гена SOWAHD на уровне белка под-
ство белка SOWAHD, измеренное в ядерной
тверждена (PE1), но внутриклеточная локали-
фракции в данной работе, схоже с содержани-
зация неизвестна, а информация об уровне экс-
ем вышеупомянутых ТФ.
прессии продукта этого гена приведена только
В данной работе, исследуя динамику бел-
ка SOWAHD в процессе ATRA-индуциро-
ENSG00000187808-SOWAHD). Наконец, база
ванной гранулоцитарной дифференцировки,
знаний NextProt, компилирующая масс-спек-
выявили дугообразный профиль экспрессии
трометрические данные, категоризирует про-
с увеличением содержания в ранние времен-
дукт гена SOWAHD как экспрессирующийся
ные точки и возвращением уровня экспрессии
на уровне белка (PE1), однако информация о
к базовой линии через 72 ч после обработки
его внутриклеточной локализации, биологи-
ATRA. На основании данных направленной
ческой функции и медицинской значимости
масс-спектрометрии похожий профиль был
отсутствует.
получен для белков, вовлечённых в регуляцию
По материалам базы данных NextProt, в
апоптоза и репарации ДНК (STAT1, CASP3,
семействе SOWAH наиболее исследованным
PARP1, PRKDC и UBE2I) [13]. Анализ данных
является белок SOWAHC, для которого была
панорамной масс-спектрометрии, полученных
установлена цитозольная локализация, а так-
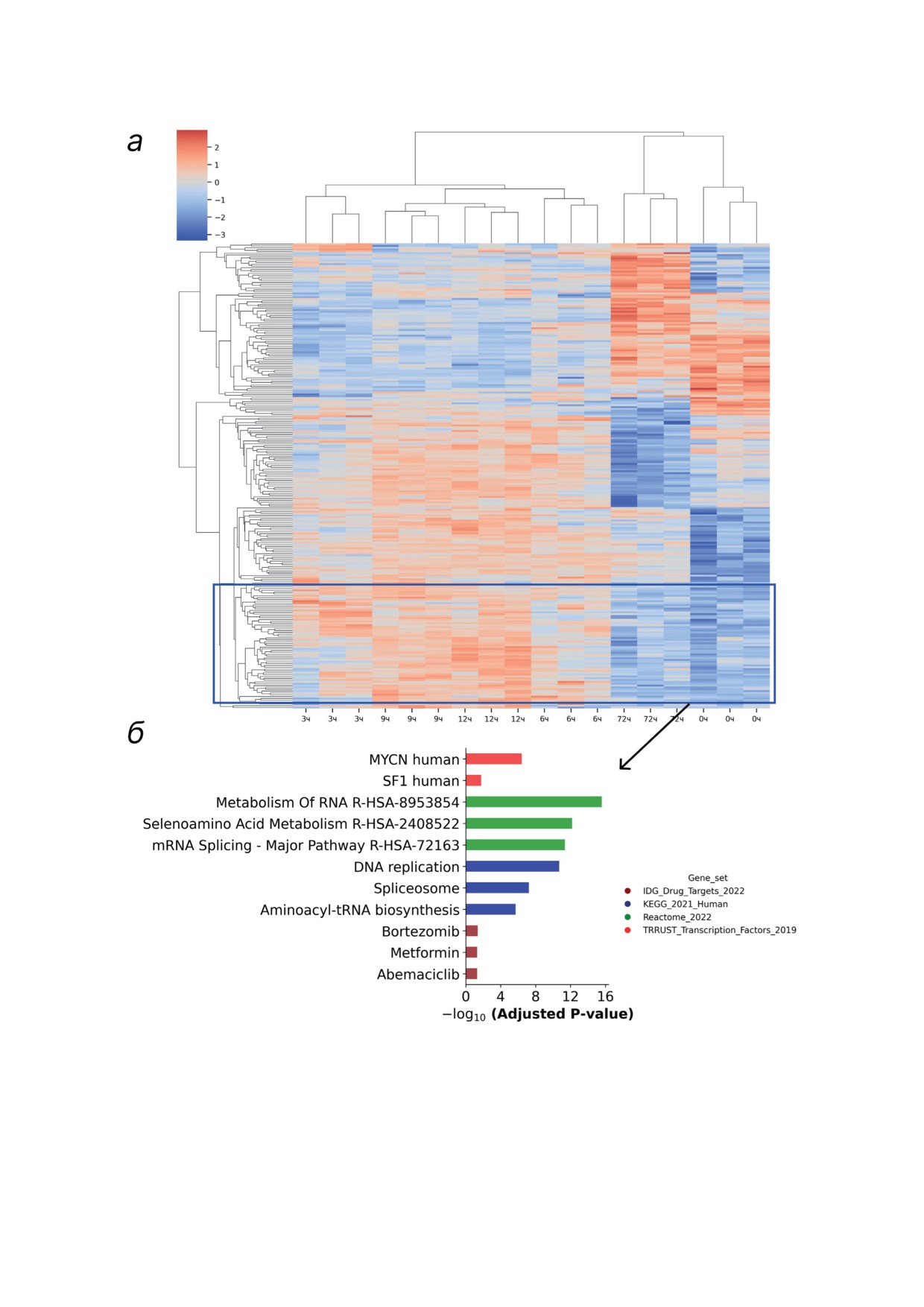
нами ранее [13], также выявил «дугообразный»
же функциональная вовлечённость в сигналь-
профиль экспрессии для белков, вовлечён-
ный путь цикла RHOB GTPазы. Для всех генов
ных в альтернативный сплайсинг и реплика-
семейства, кроме SOWAHD, также приведены
цию ДНК (рис. 8). Кроме того, данные белки,
значимые мутации, которые могут опреде-
согласно анализу относительно базы данных
лять медицинскую значимость. В контексте
TRRUST, могут регулироваться транскрипци-
онкологических заболеваний было показано,
онными факторами MYCN и SF1 (рис. 8).
что гипометилирование гена SOWAHC ассо-
Скорость полураспада белка SOWAHD
циировано с неблагоприятным прогнозом
(~19 ч) сходна с таковой для сигнальных и ре-
для пациентов с раком лёгких [27]. Высокая
гуляторных белков [34]. Нужно отметить, что
экспрессия гена SOWAHB на уровне мРНК
в данном исследовании пульсовое мечение
является благоприятным прогностическим
SILAC применяли совместно с направленным
фактором для пациентов с почечной светло-
масс-спектрометрическим анализом в режиме
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2027
Рис. 8. Результаты анализа данных панорамной масс-спектрометрии. а - «Тепловая» карта, демонстрирующая
профиль экспрессии 254 белков, содержание которых в ядерной фракции клеток линии HL-60 значимо (S0 = 0,1,
FDR = 0,01) изменялось в процессе ATRA-индуцированной гранулоцитарной дифференцировки. Для анализа были
использованы масс-спектрометрические данные, полученные ранее [13]. Для оценки дифференциальной экспрес-
сии применили дисперсионный анализ ANOVA с поправкой на множественность сравнений с помощью пермутаци-
онного теста. б - Для кластера, компоненты которого демонстрировали сходную с SOWAHD экспрессию (выделен
синим), выполнена аннотация относительно сигнальных путей (Reactome [30] и KEGG [31]), наличия потенциаль-
ной транскрипционной регуляции (TRRUST [32]), возможной чувствительности к фармакологическим препаратам
(IDG [33]). Можно предположить, что белок SOWAHD может быть вовлечён в один или несколько из вышеупомянутых
биологических процессов, однако эта гипотеза нуждается в дальнейшей экспериментальной проверке
БИОХИМИЯ том 88 вып. 10 2023
2028
НОВИКОВА и др.
ка SOWAHD, при этом белковый продукт
гена KIAA1143 также может быть потенци-
альным регулятором гранулоцитарной диф-
ференцировки. В будущем планируется про-
вести протеомный анализ белка KIAA1143 для
углублённого представления о процессах ин-
дуцированного созревания лейкозных клеток.
ЗАКЛЮЧЕНИЕ
Данные протеомного анализа указывают,
что SOWAHD вовлечён в процесс ATRA-инду-
цированной гранулоцитарной дифференци-
ровки. Планируемые эксперименты по нок-
дауну SOWAHD будут призваны проверить эту
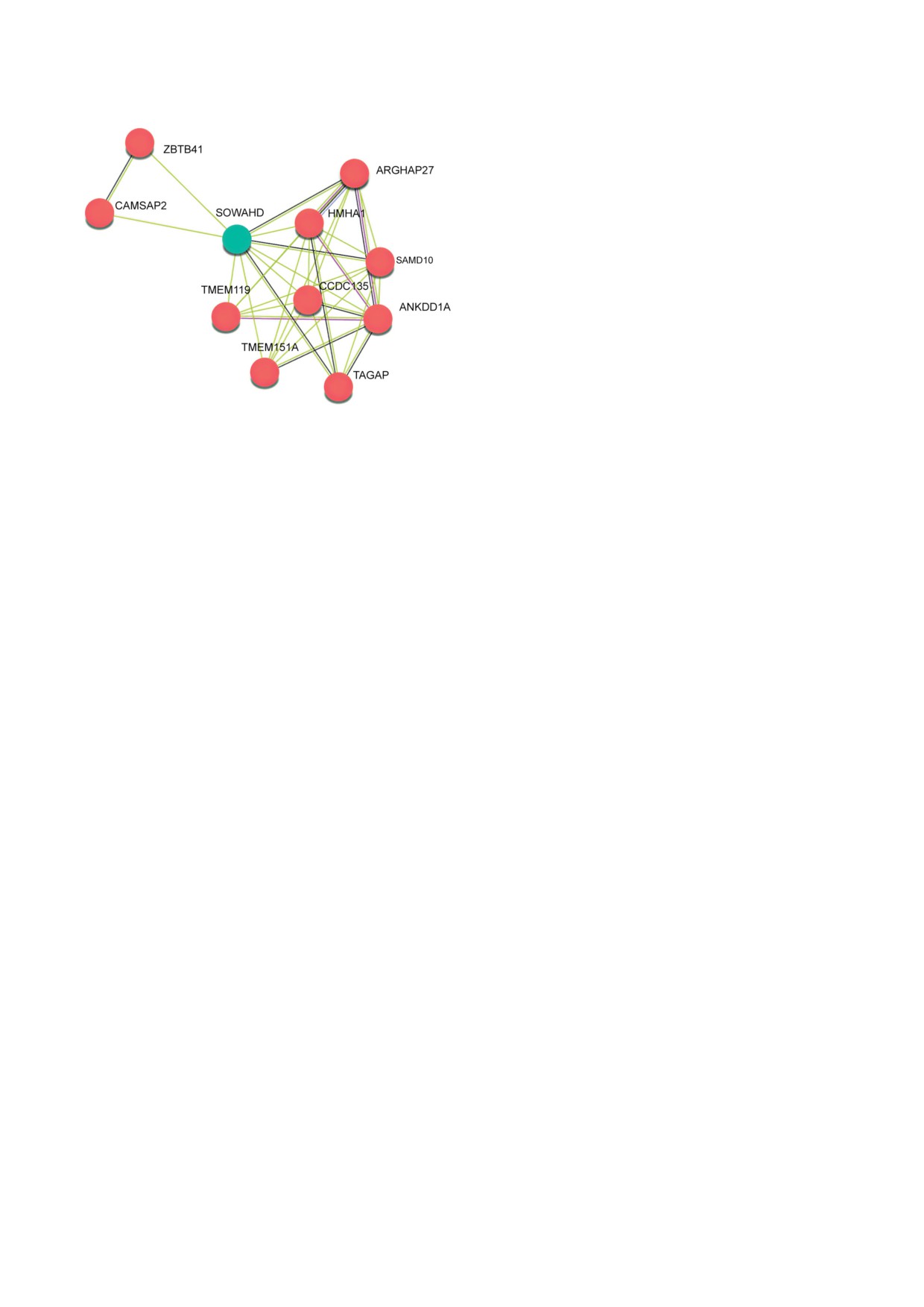
Рис. 9. Схема белок-белковых взаимодействий, вовле-
чённых в васкуляризацию и гемопоэз у рыб вида Danio
теорию. В протеомной базе данных NextProt
rerio (по материалам базы данных STRING [36]), вклю-
хранится информация об экспрессии белка
чающая белок-ортолог SOWAHD
SOWAHD, собранная на основании множе-
ства экспериментов, однако систематическое
SRM. Панорамный анализ, обычно применяе-
исследование экспрессии продукта белок-ко-
мый в пульсовом мечении SILAC, не всегда
дирующего гена SOWAHD на уровне мРНК и
позволяет вычислить величину площади под
белка на одном и том же материале проводи-
пиком для прекурсорных ионов, что приводит
лось впервые. Также уникальными являются
к наличию пропущенных значений (missing
данные по времени полураспада (t1/2) белко-
value) в количественных SILAC-данных. Осо-
вого продукта SOWAHD. Для определения
бенно остро эта проблема стоит для низкоко-
величины t1/2 был применён направленный
пийных белков. Использование направленной
масс-спектрометрический метод, который по-
масс-спектрометрии позволяет улучшить точ-
зволяет анализировать белки с низким содер-
ность и чувствительность определения скоро-
жанием.
сти полураспада [21].
Существуют гипотезы о выполняемой
Вклад авторов. С.Е. Новикова, А.Л. Руса-
функции для ортологов гена SOWAHD у дру-
нов, В.Г. Згода - концепция и руководство ра-
гих видов позвоночных. В экспериментах на
ботой; С.Е. Новикова, Т.В. Толстова, Н.А. Со-
рыбах вида Danio rerio было показано, что ор-
ловьева, Т.Е. Фарафонова, О.В. Тихонова,
толог гена SOWAHD (ankrd58) вовлечён в про-
Л.К. Курбатов - проведение экспериментов;
цесс васкуляризации и гемопоэз [35] (рис. 9).
С.Е. Новикова, О.В. Тихонова, Л.К. Курбатов,
С учётом экспрессии мРНК и белка
А.Л. Русанов, В.Г. Згода - обсуждение резуль-
SOWAHD в миелоидных клетках он может так-
татов исследования; С.Е. Новикова - написа-
же быть задействован в гемопоэзе у человека.
ние текста; В.Г. Згода - редактирование текста
На основании сходства белковой после-
статьи.
довательности можно заключить, что в струк-
Финансирование. Работа выполнена при
туре белка SOWAHD должны присутствовать
финансовой поддержке Российского научного
фонда (грант № 21-74-20122).
uniprotkb/A6NJG2/entry). Анкириновые повто-
Благодарности. Исследование выполнено
ры являются наиболее распространёнными
c использованием оборудования Центра кол-
мотивами, вовлечёнными в белок-белковые
лективного пользования «Протеом человека»
взаимодействия. Белки, содержащие анкири-
в ИБМХ.
новые повторы, такие как опухолевый супрес-
Конфликт интересов. Авторы заявляют об
сор p16, рецептор Nank сигнального пути
отсутствии конфликта интересов.
Notch, ингибитор IκBα ядерного фактора кап-
Соблюдение этических норм. Настоящая
па В и т. д., вовлечены в регуляцию клеточного
статья не содержит описания каких-либо ис-
цикла, активацию транскрипции, апоптоза
следований с участием людей или животных
и пролиферации у различных биологических
в качестве объектов.
видов, в том числе и у человека [37, 38].
Дополнительные материалы. Приложение к
Настоящее исследование было посвяще-
статье опубликовано на сайте журнала «Био-
но всестороннему протеомному анализу бел-
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2029
СПИСОК ЛИТЕРАТУРЫ
1.
Consortium UniProt (2023) UniProt: The Universal
11.
Van Bortle, K., and Corces, V. G. (2012) Nuclear
Protein Knowledgebase in 2023, Nucleic Acids Res.,
organization and genome function, Annu. Rev.
51, D523-D531, doi: 10.1093/nar/gkac1052.
Cell Dev. Biol., 28, 163-187, doi: 10.1146/annurev-
2.
Lane, L., Argoud-Puy, G., Britan, A., Cusin, I.,
cellbio-101011-155824.
Duek, P. D., Evalet, O., Gateau, A., Gaudet, P.,
12.
Vakhrushev, I. V., Novikova, S. E., Tsvetkova, A. V.,
Gleizes, A., Masselot, A., et al. (2012) NeXtProt: a
Karalkin, P. A., Pyatnitskii, M. A., Zgoda, V. G., and
knowledge platform for human proteins, Nucleic Acids
Yarygin, K. N. (2018) Proteomic profiling of HL-60
Res., 40, D76-83, doi: 10.1093/nar/gkr1179.
cells during ATRA-induced differentiation, Bull.
3.
Uhlén, M., Fagerberg, L., Hallström, B. M., Linds-
Exp. Biol. Med., 165, 530-543, doi: 10.1007/s10517-
kog, C., Oksvold, P., Mardinoglu, A., Sivertsson, Å.,
018-4210-y.
Kampf, C., Sjöstedt, E., Asplund, A., et al. (2015) Pro-
13.
Novikova, S., Tolstova, T., Kurbatov, L., Farafonova, T.,
teomics. Tissue-based map of the human proteome,
Tikhonova, O., Soloveva, N., Rusanov, A., Archakov, A.,
Science, 347, 1260419, doi: 10.1126/science.1260419.
and Zgoda, V. (2022) Nuclear proteomics of induced
4.
Kim, M. S., Pinto, S. M., Getnet, D., Nirujogi, R. S.,
leukemia cell differentiation, Cells,
11,
3221,
Manda, S. S., Chaerkady, R., Madugundu, A. K.,
doi: 10.3390/cells11203221.
Kelkar, D. S., Isserlin, R., Jain, S., et al. (2014) A draft
14.
Zheng, P. Z., Wang, K. K., Zhang, Q. Y., Huang,
map of the human proteome, Nature, 509, 575-581,
Q. H., Du, Y. Z., Zhang, Q. H., Xiao, D. K., Shen,
doi: 10.1038/nature13302.
S. H., Imbeaud, S., Eveno, E., et al. (2005) Systems
5.
Adhikari, S., Nice, E. C., Deutsch, E. W., Lane, L.,
analysis of transcriptome and proteome in retinoic
Omenn, G. S., Pennington, S. R., Paik, Y. K., Overall,
acid/arsenic trioxide-induced cell differentiation
C. M., Corrales, F. J., Cristea, I. M., et al. (2020)
apoptosis of promyelocytic leukemia, Proc. Natl.
A high-stringency blueprint of the human proteome,
Acad. Sci. USA,
102,
7653-7658, doi:
10.1073/
Nat. Commun.,
11,
5301, doi:
10.1038/s41467-
pnas.0502825102.
020-19045-9.
15.
Wang, W.-J., Tang, W., and Qiu, Z.-Y.
(2009)
6.
Salzberg, S. L. (2018) Open questions: how many
Comparative proteomics analysis on differentiation
genes do we have? BMC Biol., 16, 94, doi: 10.1186/
of human promyelocytic leukemia HL-60 cells into
s12915-018-0564-x.
granulocyte and monocyte lineages, Chinese J. Cancer,
7.
Kopylov, A. T., Ponomarenko, E. A., Ilgisonis, E. V.,
28, 117-121.
Pyatnitskiy, M. A., Lisitsa, A. V., Poverennaya, E. V.,
16.
Novikova, S., Tikhonova, O., Kurbatov, L., Fara-
Kiseleva, O. I., Farafonova, T. E., Tikhonova, O. V.,
fonova, T., Vakhrushev, I., Lupatov, A., Yarygin, K.,
Zavialova, M. G., et al. (2019) 200+ protein con-
and Zgoda, V. (2021) Omics technologies to deci-
centrations in healthy human blood plasma: targeted
pher regulatory networks in granulocytic cell dif-
quantitative SRM SIS screening of chromosomes 18,
ferentiation, Biomolecules,
11,
907, doi:
10.3390/
13, Y, and the mitochondrial chromosome encoded
biom11060907.
proteome, J. Proteome Res., 18, 120-129, doi: 10.1021/
17.
Jian, P., Li, Z. W., Fang, T. Y., Jian, W., Zhuan, Z.,
acs.jproteome.8b00391.
Mei, L. X., Yan, W. S., and Jian, N. (2011) Retinoic
8.
Poverennaya, E., Kiseleva, O., Ilgisonis, E., Novi-
acid induces HL-60 cell differentiation via the
kova, S., Kopylov, A., Ivanov, Y., Kononikhin, A.,
upregulation of MiR-663, J. Hematol. Oncol., 4, 20,
Gorshkov, M., Kushlinskii, N., Archakov, A.,
doi: 10.1186/1756-8722-4-20.
et al. (2020) Is it possible to find needles in a hay-
18.
Schwanhäusser, B., Gossen, M., Dittmar, G., and
stack? Meta-analysis of 1000+ MS/MS files pro-
Selbach, M. (2009) Global analysis of cellular protein
vided by the Russian proteomic consortium for min-
translation by pulsed SILAC, Proteomics, 9, 205-209,
ing missing proteins, Proteomes, 8, 12, doi: 10.3390/
doi: 10.1002/pmic.200800275.
PROTEOMES8020012.
19.
Claydon, A. J., and Beynon, R. (2012) Proteome
9.
Paik, Y. K., Overall, C. M., Corrales, F., Deutsch,
dynamics: revisiting turnover with a global perspective,
E. W., Lane, L., and Omenn, G. S. (2018) toward
Mol. Cell. Proteomics, 11, 1551-1565, doi: 10.1074/
completion of the human proteome parts list: prog-
mcp.O112.022186.
ress uncovering proteins that are missing or have
20.
Ross, A. B., Langer, J. D., and Jovanovic, M. (2021)
unknown function and developing analytical meth-
Proteome turnover in the spotlight: approaches,
ods, J. Proteome Res., 17, 4023-4030, doi: 10.1021/
applications, and perspectives, Mol. Cell. Proteomics,
acs.jproteome.8b00885.
20, doi: 10.1074/mcp.R120.002190.
10.
Thul, P. J., and Lindskog, C. (2018) The human
21.
Holman, S. W., Hammond, D. E., Simpson, D. M.,
protein atlas: a spatial map of the human pro-
Waters, J., Hurst, J. L., and Beynon, R. J. (2016)
teome, Protein Sci.,
27,
233-244, doi:
10.1002/
Protein turnover measurement using selected reaction
pro.3307.
monitoring-mass spectrometry (SRM-MS), Philos.
БИОХИМИЯ том 88 вып. 10 2023
2030
НОВИКОВА и др.
Trans. R. Soc. A Math. Phys. Eng. Sci., 374, 20150362,
30.
Gillespie, M., Jassal, B., Stephan, R., Milacic, M.,
doi: 10.1098/rsta.2015.0362.
Rothfels, K., Senff-Ribeiro, A., Griss, J., Sevilla, C.,
22.
Patro, R., Duggal, G., Love, M. I., Irizarry, R. A., and
Matthews, L., Gong, C., et al. (2022) The reactome
Kingsford, C. (2017) Salmon provides fast and bias-
pathway knowledgebase, Nucleic Acids Res.,
50,
aware quantification of transcript expression, Nat.
D687-D692, doi: 10.1093/nar/gkab1028.
Methods, 14, 417-419, doi: 10.1038/nmeth.4197.
31.
Kanehisa, M., Sato, Y., Kawashima, M., Furumichi, M.,
23.
Novikova, S. E., Vakhrushev, I. V., Tsvetkova, A. V.,
and Tanabe, M. (2016) KEGG as a reference resource
Shushkova, N. A., Farafonova, T. E., Yarygin, K. N.,
for gene and protein annotation, Nucleic Acids Res.,
and Zgoda, V. G. (2019) Proteomics of transcription
44, D457-62, doi: 10.1093/nar/gkv1070.
factors: identification of pool of HL-60 cell line-
32.
Han, H., Shim, H., Shin, D., Shim, J. E., Ko, Y.,
specific regulatory proteins [in Russian], Biomed.
Shin, J., Kim, H., Cho, A., Kim, E., Lee, T., et al.
Khim., 65, doi: 10.18097/PBMC20196504294.
(2015) TRRUST: a reference database of human
24.
Wiśniewski, J. R., Zougman, A., Nagaraj, N., and
transcriptional regulatory interactions, Sci. Rep., 5,
Mann, M.
(2009) Universal sample preparation
11432, doi: 10.1038/srep11432.
method for proteome analysis, Nat. Methods, 6, 359-
33.
Lin, Y., Mehta, S., Küçük-McGinty, H., Turner,
362, doi: 10.1038/nmeth.1322.
J. P., Vidovic, D., Forlin, M., Koleti, A., Nguyen,
25.
Mohammad, N. S., Nazli, R., Zafar, H., and Fatima, S.
D.-T., Jensen, L. J., Guha, R., et al. (2017) Drug
(2022) Effects of lipid based multiple micronutrients
target ontology to classify and integrate drug discovery
supplement on the birth outcome of underweight pre-
data, J. Biomed. Semantics, 8, 50, doi: 10.1186/s13326-
eclamptic women: a randomized clinical trial, Pak. J.
017-0161-x.
Med. Sci., 38, 219-226, doi: 10.12669/pjms.38.1.4396.
34.
Mathieson, T., Franken, H., Kosinski, J., Kurzawa, N.,
26.
Liu, Y., Mi, Y., Mueller, T., Kreibich, S., Williams,
Zinn, N., Sweetman, G., Poeckel, D., Ratnu, V. S.,
E. G., Van Drogen, A., Borel, C., Frank, M.,
Schramm, M., Becher, I., et al. (2018) Systematic
Germain, P.-L., Bludau, I., et al. (2019) Multi-om-
analysis of protein turnover in primary cells, Nat.
ic measurements of heterogeneity in HeLa cells
Commun., 9, 689, doi: 10.1038/s41467-018-03106-1.
across laboratories, Nat. Biotechnol., 37, 314-322,
35.
Gomez, G., Lee, J. H., Veldman, M. B., Lu, J.,
doi: 10.1038/s41587-019-0037-y.
Xiao, X., and Lin, S. (2012) Identification of vascular
27.
Zhu, Q., Wang, J., Zhang, Q., Wang, F., Fang, L.,
and hematopoietic genes downstream of etsrp by
Song, B., Xie, C., and Liu, J. (2020) Methylation-
deep sequencing in zebrafish, PLoS One, 7, e31658,
driven Genes PMPCAP1, SOWAHC and ZNF454
doi: 10.1371/journal.pone.0031658.
as potential prognostic biomarkers in lung squamous
36.
Szklarczyk, D., Kirsch, R., Koutrouli, M., Nastou, K.,
cell carcinoma, Mol. Med. Rep.,
21,
1285-1295,
Mehryary, F., Hachilif, R., Gable, A. L., Fang, T.,
doi: 10.3892/mmr.2020.10933.
Doncheva, N. T., Pyysalo, S., et al. (2023) The
28.
Ruan, B., Feng, X., Chen, X., Dong, Z., Wang, Q.,
STRING database in 2023: protein-protein association
Xu, K., Tian, J., Liu, J., Chen, Z., Shi, W., et al. (2020)
networks and functional Enrichment analyses for any
Identification of a set of genes improving survival
sequenced genome of interest, Nucleic Acids Res., 51,
prediction in kidney renal clear cell carcinoma through
D638-D646, doi: 10.1093/nar/gkac1000.
integrative reanalysis of transcriptomic data, Dis.
37.
Kang, Y., Xie, H., and Zhao, C. (2019) Ankrd45
Markers, 2020, 8824717, doi: 10.1155/2020/8824717.
is a novel ankyrin repeat protein required for cell
29.
Mori, Y., Yokota, H., Hoshino, I., Iwatate, Y.,
proliferation, Genes (Basel), 10, 462, doi: 10.3390/
Wakamatsu, K., Uno, T., and Suyari, H. (2021) Deep
genes10060462.
learning-based gene selection in comprehensive gene
38.
Kumar, A., and Balbach, J. (2021) Folding and stability
analysis in pancreatic cancer, Sci. Rep., 11, 16521,
of ankyrin repeats control biological protein function,
doi: 10.1038/s41598-021-95969-6.
Biomolecules, 11, 840, doi: 10.3390/biom11060840.
PROTEOMIC APPROACH TO INVESTIGATE THE EXPRESSION,
LOCALIZATION, AND FUNCTIONS OF THE PROTEIN PRODUCT
OF THE SOWAHD GENE DURING GRANULOCYTIC DIFFERENTIATION
S. E. Novikova*, T. V. Tolstova, N. A. Soloveva*, T. E. Farafonova, O. V. Tikhonova,
L. K. Kurbatov, A. L. Rusanov, and V. G. Zgoda*
Institute of Biomedical Chemistry,
119121 Moscow, Russia; e-mail: novikova.s.e3101@gmail.com, victor.zgoda@gmail.com
БИОХИМИЯ том 88 вып. 10 2023
ПРОТЕОМНОЕ ИССЛЕДОВАНИЕ БЕЛКОВОГО ПРОДУКТА ГЕНА SOWAHD
2031
Cataloging human proteins, determining their level of content, cellular localization, function performed,
and potential medical significance are important tasks facing the global proteomic community. At present,
the localization and functions of protein products for almost half of the protein-coding genes are unknown
or poorly understood. The study of the organelle proteome is a promising approach to reveal the localiza-
tion and functions of human proteins. The nuclear proteome is of particular interest because many nucle-
ar-localized proteins, such as transcription factors, perform regulatory functions that determine cell fate.
According to the results of a meta-analysis of the nuclear proteome, or nucleome, of HL-60 cells treated
by all-trans-retinoic acid (ATRA), it was revealed that the function and localization of the protein product
of the SOWAHD gene is poorly understood, in addition, there is no comprehensive information on the
expression of SOWAHD at the protein level. In HL-60 cells for the protein-coding gene SOWAHD, mRNA
expression was determined at the level of 6.4 ± 0.7 transcripts per million molecules. Using targeted mass
spectrometry, the content of SOWAHD protein was measured in the range of 0.27-1.25 fmol/μg of total
protein. Using stable isotope pulse-chase labeling, the half-life for the protein product of the SOWAHD
gene was determined to be approximately 19 h. Proteomic profiling of the nuclear fraction of HL-60 cells
showed that the SOWAHD protein content increased during ATRA-induced granulocytic differentiation,
peaking at 9 h after the addition of the inductor and followed by a decrease at later time points. The results
of the study indicate for the first time the nuclear localization and involvement of the protein product
of the SOWAHD gene in induced granulocytic differentiation.
Keywords: protein product of the SOWAHD gene, transcriptome, proteome, ATRA, cell nucleus, mass spectrometry,
pulse labeling with stable isotopes, dynamics of protein synthesis and degradation
БИОХИМИЯ том 88 вып. 10 2023