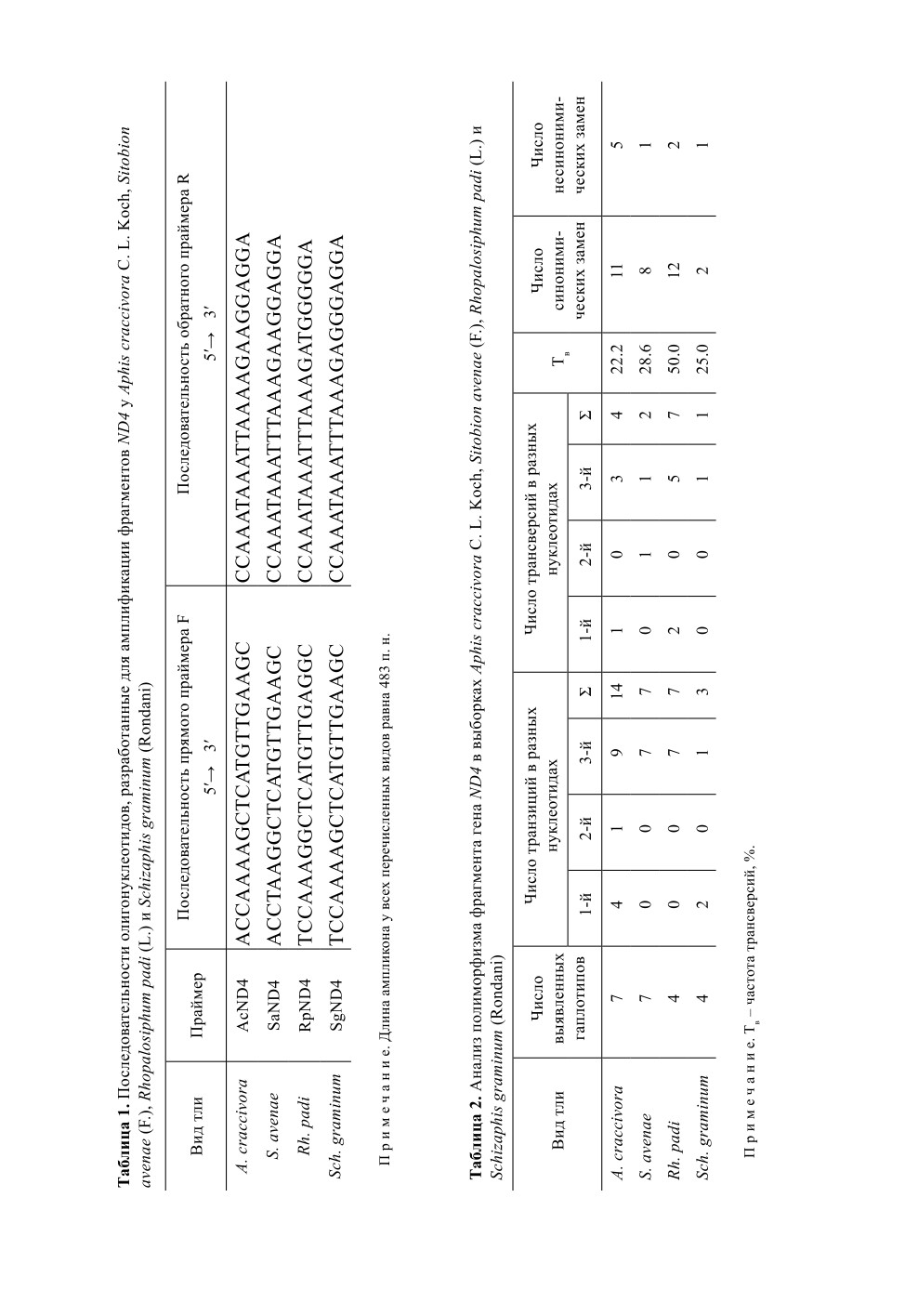
ЭНТОМОЛОГИЧЕСКОЕ ОБОЗРЕНИЕ, 101, 3, 2022
УДК 575.2:595.752.2
МИТОХОНДРИАЛЬНЫЙ ГЕН ND4 КАК МАРКЕР ДЛЯ ИЗУЧЕНИЯ
СТРУКТУРЫ И ДИНАМИКИ ПОПУЛЯЦИЙ ТЛЕЙ
(HEMIPTERA, APHIDIDAE)
© 2022 г. Н. В. Алпатьева,* И. Н. Анисимова,** Е. Е. Радченко***
Всероссийский институт генетических ресурсов растений им. Н. И. Вавилова
ул. Большая Морская, 42, 44, С.-Петербург, 190000 Россия
*e-mail: alpatievanatalia@mail.ru, **e-mail: irina_anisimova@inbox.ru,
***e-mail: eugene_radchenko@rambler.ru (автор для переписки)
Поступила в редакцию 18.05.2021 г.
После доработки 31.03.2022 г.
Принята к публикации 1.07.2022 г.
С использованием технологии высокопродуктивного секвенирования нового поколения (NGS)
изучали полиморфизм фрагмента гена ND4, кодирующего субъединицу 4 НАДН-дегидрогеназы.
Оценивали спектр точечных замен в образцах из популяций люцерновой (Aphis craccivora
C. L. Koch), большой злаковой (Sitobion avenae F.), черемухово-злаковой (Rhopalosiphum padi
L.) и обыкновенной злаковой (Schizaphis graminum Rondani) тлей. Насекомых собирали в
Краснодарском крае и на Северо-Западе России. Выявлены митохондриальные гаплотипы, ну-
клеотидные последовательности которых совпадали с референсными на 96-100 %. Уровень
внутривидового полиморфизма гена ND4 у A. craccivora составил 0.2-4.0, S. avenae - 0.2-1.9,
Rh. padi - 0.9-3.3, и у Sch. graminum - 0.2-0.9 %. Доля полиморфных сайтов в последователь-
ности гена ND4 у разных видов тлей оказалась выше, чем в пределах одного вида. Анализ со-
отношения митохондриальных гаплотипов позволяет исследовать влияние различных факторов
(например, устойчивость растения-хозяина) на структуру популяций насекомых. Выявлено
существенное различие между образцами, собранными одновременно на различающихся по
устойчивости к Sch. graminum сортах сорго, по соотношению гаплотипов Sg1-Sg3, а на умерен-
но устойчивом образце Ефремовское белое обнаружен уникальный гаплотип Sg4.
Ключевые слова: Aphididae, популяции, ген ND4, нуклеотидные замены, транзиции, транс-
версии.
DOI: 10.31857/S0367144522030029, EDN: HNRAUP
В последнее время на многих сельскохозяйственных культурах существенно увели-
чилась вредоносность тлей. Так, в результате интродукции ячменной (русской пше-
ничной) тли Diuraphis noxia (Kurdjumov) в США потери урожая пшеницы и ячменя
только на западе страны с 1986 г. (первое обнаружение вредителя) за 20 лет превысили
1 миллиард долларов (Mittal et al., 2008). Генетическая однородность агроценозов спо-
собствует ускорению микроэволюции вредных организмов (Жученко, 2012). В ряде
случаев идентифицировано весьма значительное число генов устойчивости растений
к тлям, однако далеко не все аллели эффективны. Способность тлей приспосабли-
503
ваться к устойчивому растению-хозяину обусловливает необходимость изучения из-
менчивости генетической структуры популяции, источником которой могут быть им-
миграция вирулентных клонов в локальную популяцию, мутационный процесс и
комбинативная изменчивость внутри локальной популяции. Знание структуры попу-
ляций и эколого-генетических механизмов микроэволюции насекомых необходимо
для разработки принципов регулирования экосистем и эффективного управления по-
пуляциями вредителей.
В последнее время для идентификации видов и изучения популяционной структуры
растительноядных насекомых используют однонуклеотидный полиморфизм (SNP)
митохондриальных и ядерных генов. Наиболее популярны в подобного рода исследо-
ваниях гены, кодирующие субъединицы 1, 4 и 5 фермента НАДН-дегидрогеназы (ND1,
ND4, ND5), субъединицы 1 и 2 цитохромоксидазы (СО1, СО2), а также гены рибосо-
мальной РНК (5.8S rRNA и 28S rRNA); применяют и транскриптомный анализ мито-
хондриального генома в целом (Song et al., 2016; Toju, Baba, 2018; Marquina et al., 2019;
Ponce et al., 2021). Ген, кодирующий субъединицу 1 цитохромоксидазы (СО1), чаще
других используют при исследовании популяций насекомых, в том числе различных
видов тлей (Song et al., 2016; Wongsa et al., 2017). Кроме того, значительный нуклео-
тидной полиморфизм в последовательности этого гена позволяет использовать его как
штрих-код для идентификации видов животного царства (Ratnasingham, Hebert, 2007).
Значительно меньше изучен полиморфизм ND4. Субъединица, кодируемая этим
геном, входит в состав гидрофобного комплекса НАДН-убихинон-оксидоредуктазы -
первого мультибелкового комплекса дыхательной цепи переноса электронов. В анно-
тированной последовательности митохондриальной ДНК люцерновой тли Aphis
craccivora C. L. Koch (КX447142.10) ген ND4 расположен между 6791-й и 8048-й ну-
клеотидными позициями. Аикхионбаре и Майо (Aikhionbare, Mayo, 2000) анализиро-
вали популяции обыкновенной злаковой тли Schizaphis graminum Rondani в США и
охарактеризовали фрагмент этого гена как высокополиморфный по сравнению с фраг-
ментами цитохрома b (cytb) и субъединицей 1 НАДН-дегидрогеназы (ND1). Анало-
гичные результаты получены и с помощью транскриптомного анализа 37 митохондри-
альных генов: в популяции люцерновой тли в Китае обнаружили около
4
%
полиморфных сайтов в последовательности гена ND4 (Song et al., 2016). Ранее нами
при сравнении образцов из популяций обыкновенной злаковой тли, собранных в Даге-
стане и Краснодарском крае на разных культурах и в разное время года, был выявлен
значительный полиморфизм фрагмента этого гена (Radchenko et al., 2019).
Цель настоящей работы - с помощью высокопродуктивного секвенирования нового
поколения (NGS) оценить диагностическую ценность однонуклеотидного полимор-
физма фрагмента гена ND4 для исследования популяций видов тлей, различающихся
по особенностям биологии и специфичности к растениям-хозяевам.
МАТЕРИАЛ И МЕТОДИКА
Материалом для исследования послужили 5 выборок из популяций тлей, питавшихся на
различных растениях-хозяевах в 2019 г. Большая злаковая Sitobion avenae (F.) и черемухово-зла-
ковая Rhopalosiphum padi (L.) тли заселяли посевы ячменя (Hordeum vulgare L.) научно-
производственной базы
«Пушкинские и Павловские лаборатории ВИР» Всероссийского
института генетических ресурсов растений имени Н. И. Вавилова (С.-Петербург, Пушкин), лю-
церновая тля Aphis craccivora C. L. Koch заселяла посевы гуара (Cyamopsis tetragonoloba (L.)
504
Taub.) в Краснодарском крае (Гулькевичский р-н, Кубанская опытная станция ВИР - КОС ВИР).
На КОС ВИР была собрана и обыкновенная злаковая тля Schizaphis graminum (Rondani), питав-
шаяся на сортах зернового сорго, которые различались по устойчивости к вредителю: Ефремов-
ском белом (умеренно устойчивом) и СЛВ-2 (восприимчивом). В каждом случае было собрано
не менее 300 насекомых в нескольких (5-20) точках. Насекомых помещали в пробирки с завин-
чивающимися крышками с 70%-ным этанолом и хранили при температуре -20 °C. Таксономи-
ческую принадлежность насекомых определяли в лаборатории с помощью определителя
Г. Х. Шапошникова (Шапошников, 1964).
Изучали полиморфизм фрагмента гена ND4 длиной 438 пар нуклеотидов (п. н.), ко-
торый расположен между 7048-й и 7485-й позициями референсной последователь-
ности KX447142.1 в информационной базе нуклеотидных последовательностей NCBI
сушили при комнатной температуре. Суммарную ДНК выделяли SDS-буфером
(100 мМ трис-HCl, pH 9.0; 100 мМ ЭДТА; 1.0 % SDS) по разработанному нами прото-
колу (Алпатьева и др., 2019). Секвенирование фрагментов митохондриального гена
ND4 проводили на приборе Illumina MiSeq (Illumina, США) c помощью набора реак-
тивов MiSeq® ReagentKit v3 (600 cycle) с двусторонним чтением (2 × 300 п. н.). Работа
выполнялась с использованием оборудования ЦКП «Геномные технологии, протео-
мика и клеточная биология» Всероссийского научно-исследовательского института
сельскохозяйственной микробиологии. Праймеры для амплификации фрагмента ука-
заны в табл. 1. Данные, полученные в результате секвенирования образцов, обрабаты-
вали с использованием пакетов программного обеспечения «Trimmomatic» (Bolger
et al., 2014) и «QIIME» (Caporaso et al., 2010). На первом этапе проводили первичный
анализ качества прочтения, отбор последовательностей на основе качества чтения от-
дельных оснований (basepairquality), объединение парно-концевых последовательно-
стей с участком перекрытия не менее 35 оснований, а также удаление последователь-
ностей, длина которых составляет менее 180 п. н. На втором этапе обработки из
библиотек удаляли все служебные участки (праймеры), а также последовательности,
содержащие протяженные гомополимерные повторы. Для получения таблицы частот
уникальных последовательностей (ASV, Amplicon Sequence Variant) проводились про-
цедуры денойзинга и удаления химер с помощью программного пакета dada2 (Callahan
et al., 2016).
Последовательности идентифицировали с помощью международной базы нуклео-
тидных последовательностей NCBI GenBank и поисковой системы BLAST (https://
blast.ncbi.nlm.nih.gov/Blast.cgi). Варианты, которые встречались с низкой (менее 1 %)
частотой, исключались из анализа. Выравнивание и кластерный анализ последова-
тельностей проводили с помощью программы MEGA7 (Kumar et al., 2016). В качестве
референсных использовали последовательности гена ND4 из аннотированных мито-
хондриальных геномов насекомых: KX447142.1 (A. craccivora), KJ742384.1 (S. avenae),
KT447631.1 (Rh. padi) и AY531391.1 (Sch. graminum). Для трансляции использовали
код 5.
Попарное сравнение последовательностей по числу нуклеотидных замен на каждый
сайт проводили с помощью метода максимального комбинированного правдоподобия
(maximum composite likelihood method) (Tamura et al., 2004) в программе MEGA7
(Kumar et al., 2016).
505
РЕЗУЛЬТАТЫ
С помощью высокопроизводительного секвенирования (NGS) в пяти суммарных
пробах ДНК A. craccivora, S. avenae, Rh. padi и Sch. graminum было получено более
50 000 последовательностей. После удаления праймеров, используемых для ПЦР,
длина «полезного» фрагмента ND4 составила 438 п. н. Однонуклеотидный (SNP)
полиморфизм, обнаруженный в изученных образцах, позволил идентифицировать га-
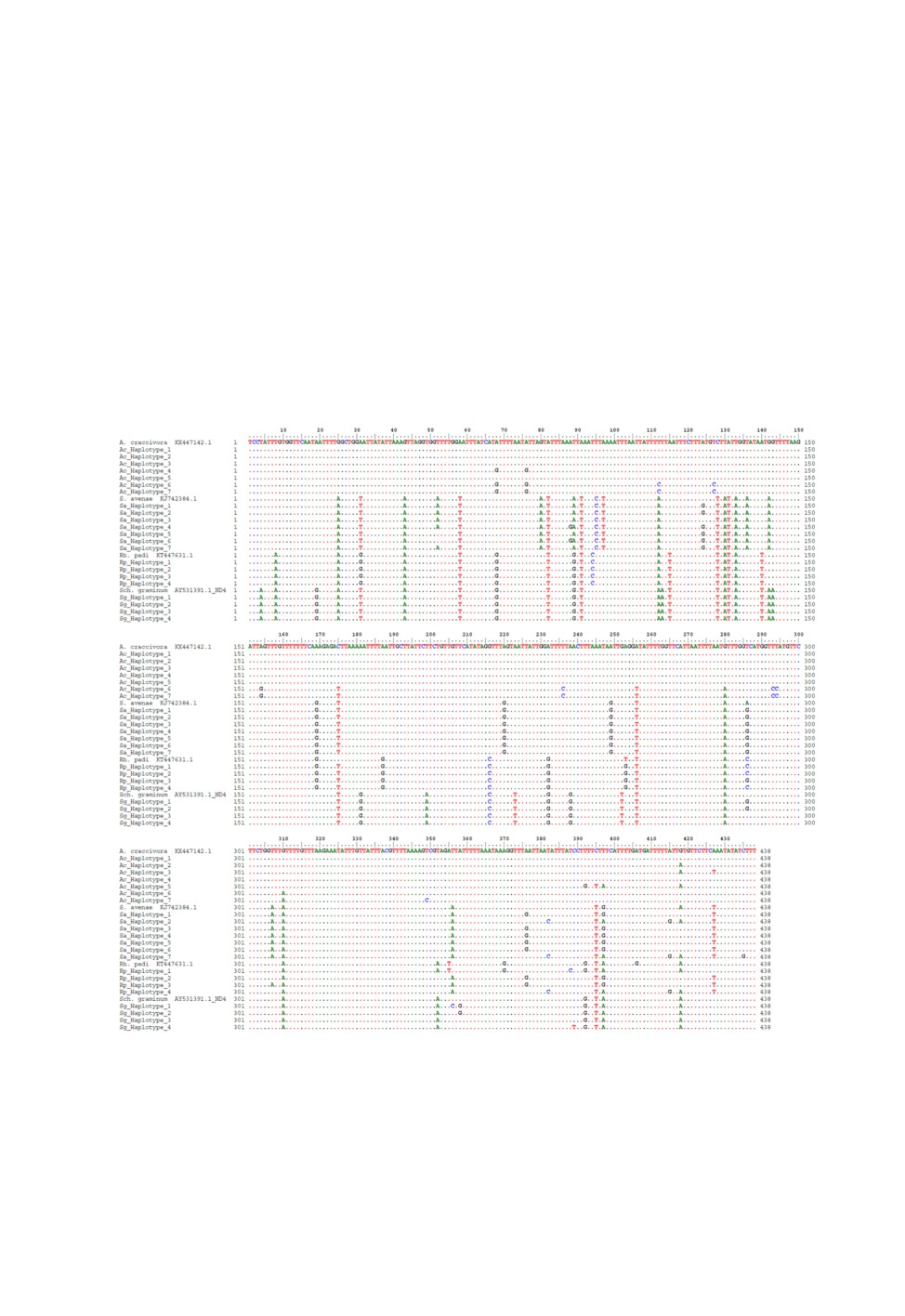
плотипы по гену ND4. Так, в анализируемых последовательностях A. craccivora най-
дено 18 полиморфных сайтов, которые позволили идентифицировать 7 гаплотипов
ND4, в выборках S. avenae - 9 полиморфных сайтов и 7 гаплотипов, Rh. padi - 14 поли-
морфных сайтов и 4 гаплотипа, в двух образцах Sch. graminum - 4 полиморфных сайта
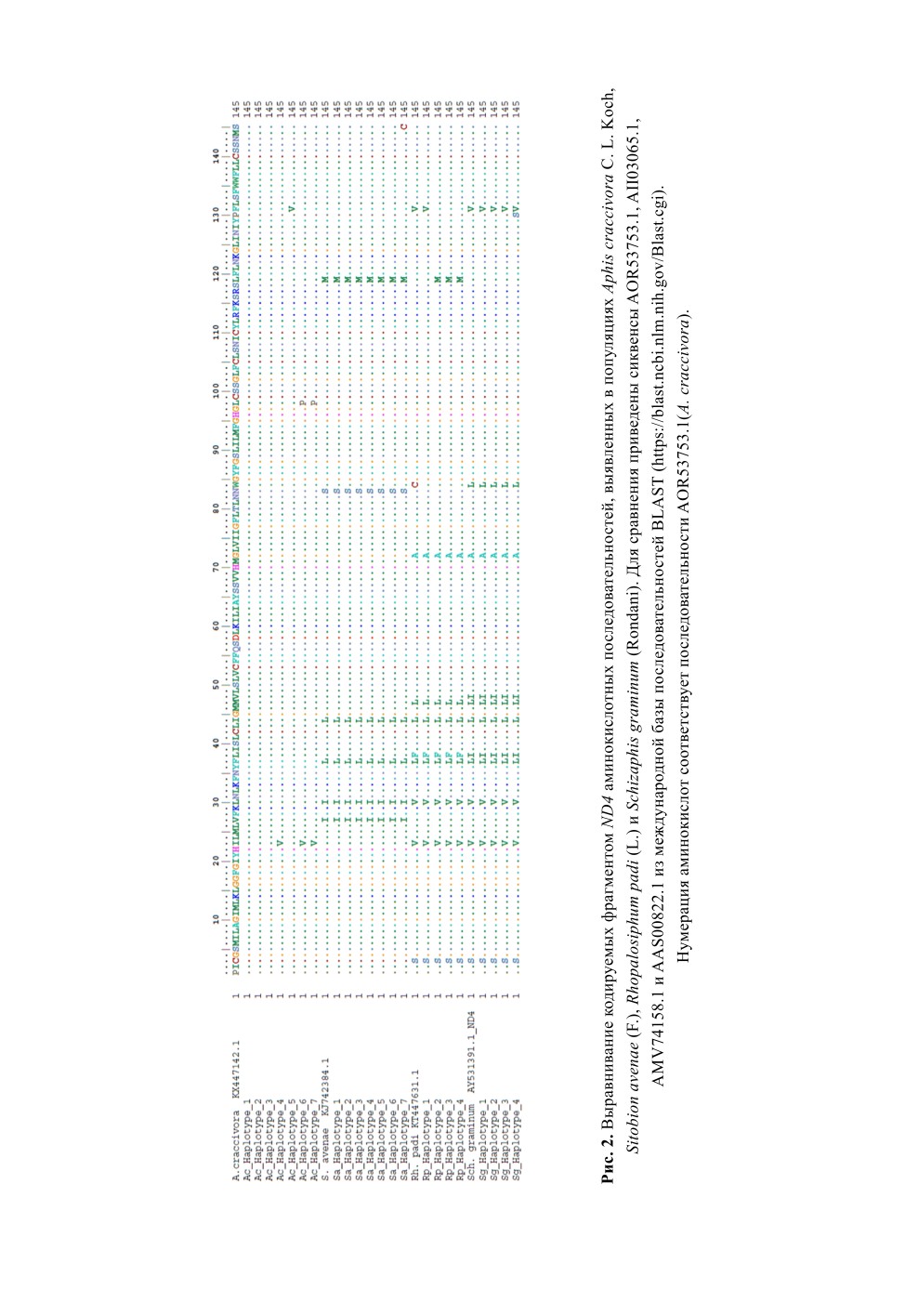
и 4 гаплотипа (рис. 1). В кодируемых белковых последовательностях были найдены
точечные замены (рис. 2). Две субпопуляции Sch. graminum, сформировавшиеся на
разных сортах сорго, различались по соотношению гаплотипов Sg1-Sg3, а на уме-
Рис. 1. Выравнивание нуклеотидных последовательностей фрагмента ND4, выявленных в
популяциях Aphis craccivora C. L. Koch, Sitobion avenae (F.), Rhopalosiphum padi (L.)
и Schizaphis graminum (Rondani). Для сравнения приведены сиквенсы KX447142.1,
KJ742384.1, KT447631.1 и AY531391.1 из международной базы последовательностей
507
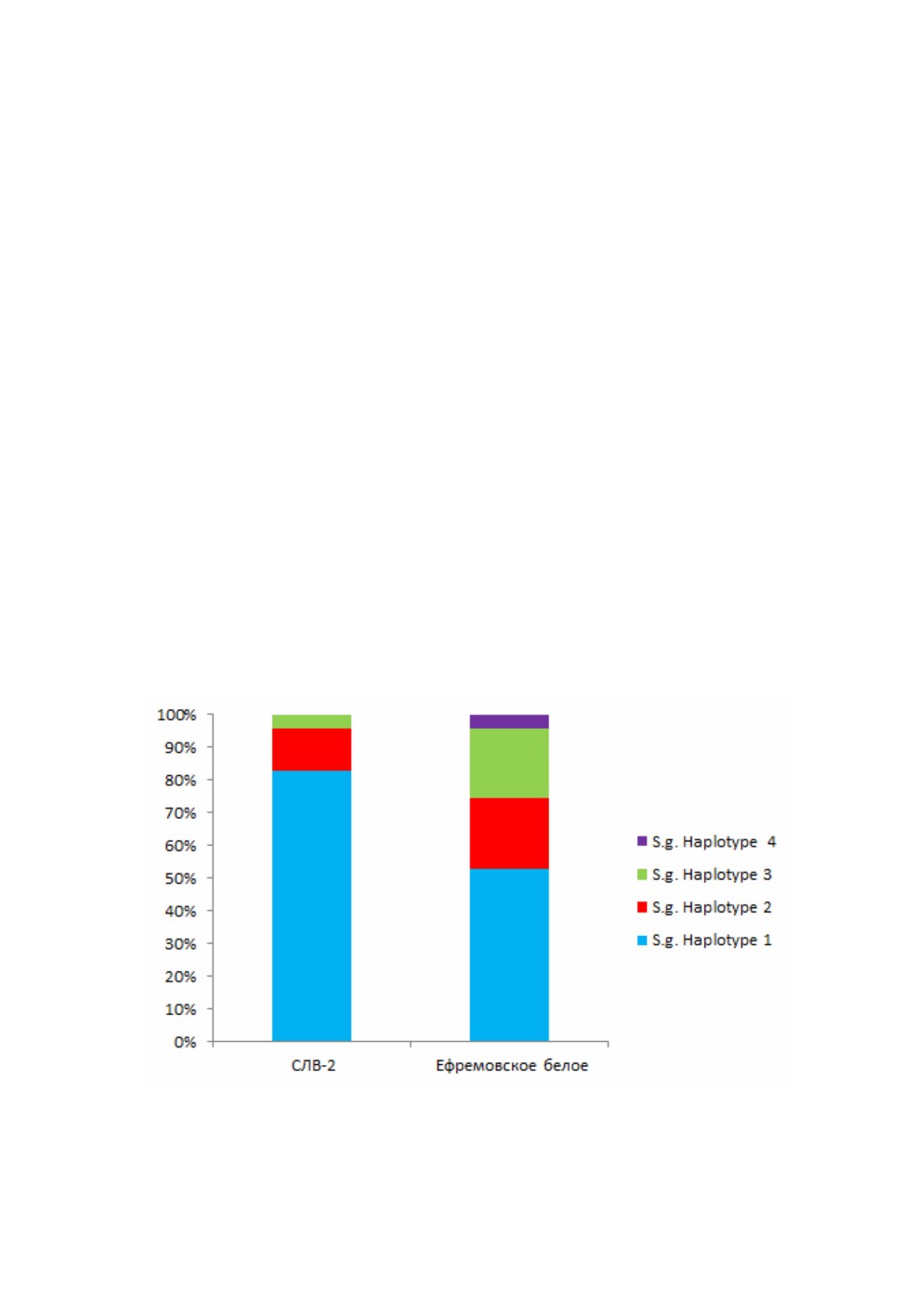
ренно устойчивом образце Ефремовское белое выявлен уникальный гаплотип Sg4. Со-
отношение идентифицированных в субпопуляциях Sch. graminum гаплотипов пред-
ставлено на рис. 3. Все полученные последовательности сходны с референсными
KX447142.1 (A. craccivora), KJ742384.1 (S. avenae), KT447631.1 (Rh. padi) и
AY531391.1 (Sch. graminum) на 96-100 %.
В пределах изученных выборок изменчивость фрагментов (рассчитанная по числу
замен на каждый сайт последовательности и выраженная в процентах) варьировала в
диапазоне 0.2-4.0 % у A. craccivora, 0.2-1.9 % - у S. avenae, 0.9-3.3 % - у Rh. padi,
и 0.2-0.9 % - у Sch. graminum. Идентифицированные варианты различались точеч-
ными мутациями разных типов (транзиции и трансверсии). Транзиции - замены од-
ного пуринового (аденин или гуанин) (A↔G) либо пиримидинового (цитозин или
тимин) (C↔T) основания на другое. Трансверсии - это замены между пуринами и пи-
римидинами (A↔T, A↔C, G↔T, G↔C). Следовательно, ожидаемым отношением
числа транзиций к числу трансверсий является 1 : 2 (если предположить, что все за-
мены одинаково вероятны). Однако показано, что скорость таких мутаций варьирует у
разных организмов (Keller et al., 2006). Оба типа мутаций могут приводить к заменам
аминокислот (смысловые замены) или, напротив, вследствие вырожденности генети-
ческого кода могут быть синонимическими (не приводить к аминокислотным за-
менам). В каждой изученной нами выборке насекомых были обнаружены как смыс-
ловые, так и синонимические замены в кодирующих триплетах. В табл. 2 приведена
характеристика обнаруженных в выборках замен.
У A. craccivora выявлено 18 полиморфных сайтов, среди которых 14 транзиций:
пять G↔A и четыре Т↔С в третьем положении кодонов, одна транзиция G↔A в
первом положении, три Т↔С - в первом и одна Т↔С - во втором, а также 4 транс-
Рис. 3. Соотношение гаплотипов фрагмента гена ND4 (%) в субпопуляциях Schizaphis
graminum (Rondani), собранных одновременно на образцах сорго СЛВ-2
и Ефремовское белое.
509
версии: три A↔T находятся в третьем положении, а одна T↔G - в первом. В пяти
случаях нуклеотидные замены приводят к аминокислотным, т. е. являются смысло-
выми. Все они относятся к консервативным заменам и обусловливают функционально
эквивалентную замену аминокислот. Например, в позиции 211 предполагаемой ами-
нокислотной последовательности белка ND4 замена изолейцина на валин происходит
благодаря транзиции A↔G первого нуклеотида в соответствующем кодоне, а для ами-
нокислоты в позиции 286 T↔C замены нуклеотидов в первом и втором положениях
кодона приводят к замещению лейцин/пролин (см. рис. 2). Все смысловые замены в
вариантах ND4, выявленных у A. craccivora, связаны с взаимным обменом незаря-
женных гидрофобных аминокислот, что не приводит к существенным изменениям
физико-химических свойств белка.
В выборке из популяции S. avenae обнаружено 9 полиморфных сайтов. Среди них -
7 транзиций (шесть G↔A и одна Т↔С в третьем положении кодонов), а также две
трансверсии (A↔T находится в третьем положении кодонов и C↔G - во втором). По-
следняя замена является смысловой и приводит к смене полярных незаряженных ами-
нокислот: серин ↔ цистеин (позиция 333) (см. рис. 2).
В выборке из популяции Rh. padi найдено 14 полиморфных сайтов: 7 транзиций
(пять G↔A и две Т↔С в третьем положении кодонов) и 7 трансверсий (четыре A↔T и
одна С↔G находятся в третьем положении кодонов, а одна A↔T и одна T↔G - в
первом). Две транзиции в первом кодоне привели к смене неполярных гидрофобных
лейцина и метионина в позиции 307, а также валина и фенилаланина - в позиции
319 предполагаемой белковой последовательности фрагмента гена ND4 (см. рис. 2).
В субпопуляциях Sch. graminum обнаружено 4 полиморфных сайта, среди которых
3 транзиции (G↔A в третьем положении кодонов и две T↔C - в первом) и одна A↔T
трансверсия в третьем положении кодона. У варианта Sg4 одна из T↔C трансверсий в
первом кодоне привела к кардинальной замене полярной незаряженной аминокислоты
серин на неполярный, гидрофобный фенилаланин в позиции 318 (см. рис. 2). Такой
вариант замены с частотой 4.1 % обнаружен только в субпопуляции Sch. graminum,
питавшейся на сорте Ефремовское белое (см. рис. 3).
ОБСУЖДЕНИЕ
Доля нуклеотидных замен в исследованных выборках тлей составила 0.2-3.3 %, что
позволило идентифицировать у разных видов от 4 до 7 митохондриальных гапло-
типов. По литературным данным, сходные результаты были получены и для фраг-
мента митохондриального гена СО1, уровень полиморфизма которого варьировал от
0.2 до 2.7 % при исследовании люцерновой тли в Таиланде и достигал 1.04 % в попу-
ляциях A. craccivora, Aphis fabae Scopoli, A. gossypii Glover, Acyrthosiphon pisum
(Harris), Brevicoryne brassicae (L.), Lipaphis pseudobrassicae (Davis) и Myzus persicae
(Sulzer) в Кении (Kinyanjui et al., 2016; Wongsa et al., 2017). Большинство выявленных
нами замен - синонимические (не приводят к смене аминокислот), что также согласу-
ется с результатами предыдущих исследований (Song et al., 2016).
Соотношение числа транзиций и трансверсий - важный показатель, который харак-
теризует изменчивость популяций, в нашем исследовании оказался вариабельным (см.
табл. 2). Считается, что полиморфизм кодирующих участков генома определяется пре-
имущественно транзициями. Так, у трех видов рода Drosophila Fallén - D. melanogaster
510
Meigen (изучен полиморфизм 24 локусов), D. simulans Sturtevant (12 локусов) и
D. pseudoobscura Frolova (5 локусов) - число транзиций превышало число тран-
сверсий вдвое (Moriyama, Powell, 1996). Широко изученный в популяциях насекомых
полиморфизм гена СО1 определялся преимущественно трансверсиями (Karthika et al.,
2016; Wongsa et al., 2017). При исследовании выборки Rh. padi частоты двух типов
мутаций были тождественны, в то время как в выборках A. craccivora, Sch. graminum и
S. avenae преобладали транзиции.
Нами выявлено существенное различие между образцами, собранными одновре-
менно на различающихся по устойчивости к Sch. graminum сортах сорго, по соотно-
шению гаплотипов Sg1-Sg3, а на умеренно устойчивом образце Ефремовское белое
обнаружен уникальный гаплотип Sg4 (см. рис. 3). Ранее при изучении северокавказ-
ских (краснодарской и дагестанской) популяций Sch. graminum мы выявили 16 вари-
антов последовательностей гена ND4, причем подавляющее число замен относилось к
транзициям. В краснодарской популяции по соотношению гаплотипов различались
выборки клонов тли, собранные как одновременно на восприимчивом и умеренно
устойчивом к фитофагу генотипах сорго, так и в разные периоды вегетации на одном
образце растения-хозяина (Radchenko et al., 2019). Выявленный нами значительный
однонуклеотидный полиморфизм фрагмента гена ND4 может быть использован при
изучении структуры популяций тлей.
Доля полиморфных сайтов в последовательности гена ND4 у разных видов тлей ока-
залась выше (4.5-10.4 %), чем у образцов одного вида (до 4.0 %).
Некоторые сайты, вариабельные у одного вида, оказались мономорфными у другого.
Например, только у A. craccivora встречались замены, которые вызывали синонимиче-
ские и смысловые замены аминокислот в позициях 211, 213, 225, 230, 239, 267, 273,
281, 286, 291, 304, у S. avenae - в позициях 205 и 333, у Rh. padi - 390, 305, 306, 311,
317 и 323, у S. graminum - 318. В исследованных выборках сочетание 38 замен в 30 ко-
дирующих триплетах было уникально для изученных видов, что согласуется с резуль-
татами кластерного анализа. На кладограмме (рис. 4) все найденные нуклеотидные
последовательности образуют 4 четко различимые группы, соответствующие изу-
ченным видам A. craccivora, S. avenae, Rh. padi и Sch. graminum. Около 30 % замен
являются смысловыми и обусловливают 10 видоспецифичных замен аминокислот в
последовательности белка. Таким образом, как нуклеотидные, так и аминокислотные
последовательности ND4 позволяют определить видовую принадлежность любого
гаплотипа. При попарном сравнении видов соотношение типов замен (транзиций
и трансверсий) было примерно одинаковым. Лишь A. craccivora отличался от
Sch. graminum большим числом трансверсий по сравнению с транзициями. Возможно,
не только SNP, но и тип замены в последовательности гена ND4 является видоспеци-
фичным признаком.
Значительный полиморфизм и особенности нуклеотидных замен характеризуют
фрагмент митохондриального гена ND4 не только как эффективный маркер для ви-
довой идентификации, но также и как инструмент для изучения внутрипопуляци-
онной изменчивости разных видов тлей при воздействии биотических и абиотических
факторов среды.
511
Рис. 4. Cтепень сходства нуклеотидной последовательности фрагмента ND4 длиной 438 п. н.
у Aphis craccivora C. L. Koch, Sitobion avenae (F.), Rhopalosiphum padi (L.) и Schizaphis
graminum (Rondani). Кладограмма построена в программе MEGA7 (Kumar et al., 2016)
с помощью алгоритма UPGMA (метод невзвешенной попарной группировки
с усреднением - unweighted pair group method using arithmetic averages)
(Sneath, Sokal, 1973).
512
ФИНАНСИРОВАНИЕ
Работа выполнена в рамках государственного задания согласно тематическому
плану ВИР по проекту № 0481-2022-0001 «Структурирование и раскрытие потенциала
наследственной изменчивости мировой коллекции зерновых и крупяных культур ВИР
для развития оптимизированного генбанка и рационального использования в селекции
и растениеводстве» и при финансовой поддержке Российского фонда фундамен-
тальных исследований (проект № 20-016-00048).
СПИСОК ЛИТЕРАТУРЫ
Алпатьева Н. В., Антонова О. Ю., Радченко Е. Е., Абдуллаев Р. А., Карабицина Ю. И., Анисимова И. Н. 2019.
ПЦР-диагностика вредных организмов гуара. Методические указания. СПб.: ВИР,
36 с.
Жученко А. А. 2012. Роль мобилизации генетических ресурсов цветковых растений, их идентификации и
систематизации в формировании адаптивно-интегрированной системы защиты агроценозов, агроэко-
систем и агроландшафтов. Саратов: ГНУ НИИ сельского хозяйства Юго-Востока, 527 с.
Шапошников Г. Х. 1964. Подотряд Aphidinea - тли. В кн.: Г. Я. Бей-Биенко (ред.). Определитель насекомых
европейской части СССР в 5 томах. Т. 1. Низшие, древнекрылые, с неполным превращением. М.; Л.:
Наука, с. 489-616.
Aikhionbare F. O., Mayo Z. B. 2000. Mitochondrial DNA sequences of greenbug (Homoptera: Aphididae) biotypes.
Biomolecular Engineering 16 (6): 199-205.
Bolger A. M., Lohse M., Usadel B. 2014. Trimmomatic: a flexible trimmer for Illumina sequence data. Bioinformatics
30 (15): 2114-2120.
Callahan B. J., McMurdie P. J., Rosen M. J., Han A. W., Johnson A. J. A., Holmes S. P. 2016. DADA2: High-
resolution sample inference from Illumina amplicon data. Nature Methods
13
(7):
581-583.
Caporaso J. G., Kuczynski J., Stombaugh J., Bittinger K., Bushman F. D., Costello E. K., Fierer N., Peña A. G.,
Goodrich J.K., Gordon J. I., Huttley G. A., Kelley S. T., Knights D., Koenig J. E., Ley R. E., Lozupone C. A.,
McDonald D., Muegge B. D., Pirrung M., Reeder J., Sevinsky J. R., Turnbaugh P. J., Walters W. A., Widmann
J., Yatsunenko T., Zaneveld J., Knight R. 2010. QIIME allows analysis of high throughput community
sequencing data. Nature Methods 7 (5): 335-336.
Karthika P., Krishnaveni N., Vadivalagan C., Murugan K., Nicoletti M., Benelli G. 2016. DNA barcoding and
evolutionary lineage of 15 insect pests of horticultural crops in South India. Karbala International Journal of
Keller I., Bensasson D., Nichols R. A. 2007. Transition-transversion bias is not universal: a counter example from
Kinyanjui G., Khamis F. M., Mohamed S., Ombura L. O., Warigia M., Ekesi S. 2016. Identification of aphid
(Hemiptera: Aphididae) species of economic importance in Kenya using DNA barcodes and PCR-RFLP-
based approach. Bulletin of Entomological Research 106 (1): 63-72.
Kumar S., Stecher G., Tamura K. 2016. MEGA7: Molecular evolutionary genetics analysis version 7.0 for bigger
Marquina D., Andersson A. F., Ronquist F. 2019. New mitochondrial primers for metabarcoding of insects, designed
and evaluated using in silico methods. Molecular Ecology Resources 19 (1): 90-104.
Martinez-Torres D., Moya A., Hebert P. D. N., Simon J.-C. 1997. Geographic distribution and seasonal variation of
mitochondrial DNA haplotypes in the aphid Rhopalosiphum padi (Hemiptera: Aphididae). Bulletin of
Mittal S., Dahleen L. S., Mornhinweg D. 2008. Locations of quantitative trait loci conferring Russian wheat aphid
resistance in barley germplasm STARS-9301B. Crop Science 48 (4): 1452-1458.
Moriyama E. N., Powell J. R. 1996. Intraspecific nuclear DNA variation in Drosophila. Molecular Biology and
513
Ponce P., Muñoz-Tobar S., Carrazco-Montalvo A., Villota S. D., Coloma J., Wang C., Holechek S., Cevallos V. 2021.
Two haplotypes of Aedes aegypti detected by ND4 mitochondrial marker in three regions of Ecuador. Insects
12: 200.
Radchenko E. E., Alpatieva N. V., Chumakov M. A., Abdullaev R. A. 2019. Variability of the North Caucasian
populations of the greenbug for host virulence and discovered by molecular markers. Russian Journal of
Radchenko E. E., Kuznetsova T. L., Zubov A. A. 2012. Longterm seasonal polymorphism of the Krasnodar greenbug
population for virulence to sorghum varieties carrying different resistance genes. Russian Journal of Ecology
Ratnasingham S., Hebert D. N. 2007. BOLD: The Barcode of Life Data System (www.barcodinglife.org). Molecular
Sneath P. H. A., Sokal R. R. 1973. Numerical Taxonomy: the Principles and Practice of Numerical Classification.
San Francisco: Freeman, 573 p.
Song N., Zhang H., Li H., Cai W. 2016. All 37 Mitochondrial genes of aphid Aphis craccivora obtained from
transcriptome sequencing: implications for the evolution of aphids. PLoS One
11
(6): e0157857.
Tamura K., Nei M., Kumar S. 2004. Prospects for inferring very large phylogenies by using the neighbor-joining
method. Proceedings of the National Academy of Sciences of the United States of America 101 (30):
11030-11035.
Toju H., Baba Y. G. 2018. DNA metabarcoding of spiders, insects, and springtails for exploring potential linkage
between above- and below-ground food webs. Zoological Letters 4 (4): 1-12.
Wongsa K., Duangphakdee O., Rattanawannee A. 2017. Genetic structure of the Aphis craccivora (Hemiptera:
Aphididae) from Thailand inferred from mitochondrial COI gene sequence. Journal of Insect Science
17 (4): 84.
MITOCHONDRIAL ND4 GENE AS A MARKER TRAIT FOR STUDYING
STRUCTURE AND DYNAMICS OF APHID
(HEMIPTERA, APHIDIDAE) POPULATIONS
N. V. Alpatieva, I. N. Anisimova, E. E. Radchenko
Key words: Aphididae, populations, gene ND4, nucleotide substitutions, transitions,
transversions.
SUMMARY
The polymorphism in a fragment of the gene ND4 encoding NADH dehydrogenase subunit 4 was
investigated using next-generation sequencing (NGS) technology. The spectrum of point substitutions
was evaluated in the populations of alfalfa (Aphis craccivora C. L. Koch), English grain (Sitobion
avenae Fabricius), bird cherry-oat (Rhopalosiphum padi L.) aphids and greenbug (Schizaphis
graminum Rondani) collected in Krasnodar Territory and in the Russian North-West. The mitochondrial
haplotypes have been identified, whose nucleotide sequences coincided with the reference ones by
96-100%. The level of intraspecific polymorphism of the gene ND4 was 0.2-4.0% in A. craccivora,
0.2-1.9% in S. avenae, 0.9-3.3% in Rh. padi, and 0.2-0.9% in Sch. graminum. The proportion of
polymorphic sites in the ND4 gene sequences among different aphid species was higher than within
one species. The analysis of the ratio of mitochondrial haplotypes makes it possible investigating the
influence of various factors (for example, the resistance of the host plant) on the structure of insect
populations. A significant difference in the ratio of Sg1-Sg3 haplotypes was revealed between the
samples collected simultaneously on sorghum cultivars differing in resistance to Sch. graminum, and
a unique Sg4 haplotype was found on a moderately resistant sample of Efremovskoe white.
514