ЖУРНАЛ ОБЩЕЙ ХИМИИ, 2022, том 92, № 3, с. 415-431
УДК 547.773:547.779.1
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
5-АМИНО-3-(ЦИАНОМЕТИЛ)-1Н-ПИРАЗОЛ-
4-КАРБОНИТРИЛА С НЕКОТОРЫМИ
1,3-ДИЭЛЕКТРОФИЛЬНЫМИ АГЕНТАМИ
© 2022 г. А. М. Семеноваa, Я. Р. Гаджиахмедоваa, А. В. Беспаловa, В. В. Доценкоa,b,*,
Н. А. Аксеновb, И. В. Аксеноваb
a Кубанский государственный университет, ул. Ставропольская 149, Краснодар, 350040 Россия
b Северо-Кавказский федеральный университет, Ставрополь, 355009 Россия
*e-mail: victor_dotsenko_@mail.ru
Поступило в Редакцию 7 декабря 2021 г.
После доработки 27 января 2022 г.
Принято к печати 28 января 2022 г.
5-Амино-3-(цианометил)-1Н-пиразол-4-карбонитрил вступает в реакции конденсации с β-дикетонами и
дибензальацетоном с образованием производных 2-(цианометил)пиразоло[1,5-a]пиримидин-3-карбони-
трила. Реакция с цианогуанидином в кислой среде приводит к образованию 2,4-диамино-7-(цианометил)-
пиразоло[1,5-a][1,3,5]триазин-8-карбонитрила. Строение полученных соединений подтверждено спек-
тральными данными, а также результатами квантово-химического исследования возможных реакционных
маршрутов для взаимодействия 5-амино-3-(цианометил)-1Н-пиразол-4-карбонитрила с бензоилтрифто-
рацетоном. Для полученных продуктов проведено предсказание параметров биодоступности in silico,
методом протеин-лигандного докинга спрогнозированы возможные белковые мишени.
Ключевые слова: 3(5)-аминопиразолы, дициандиамид, пиразоло[1,5-a]пиримидины, расчетная биоло-
гическая активность, квантово-химические исследования
DOI: 10.31857/S0044460X22030052
3(5)-Аминопиразолы представляют значитель-
пиридазинов 2 [12], пиразоло[4,3-с]пиридинов 3
ный интерес ввиду их биологического действия;
[13, 14], пиразоло[1,5-a]пиримидинов 4 [15-17],
также они широко используются в качестве легко-
пиразоло[1,5-a]хиназолинов
5
[18], пиразо-
доступных реагентов при разработке и создании
ло[1,5-a]пиридинов 6 [19-21], пиразоло[3,4-d]пи-
разнообразных функциональных производных
римидинов 7 [22], пиразоло-1,3-тиазинов 8 [23],
пиразола {обзорные работы по химии 3(5)-амино-
3,4-диамино-1H-тиено[3,4-c]пиразол-6-карбони-
пиразолов см.
[1-9]}.
5-Амино-3-(цианоме-
трила 9 [24] и др. (схема 1).
тил)-1Н-пиразол-4-карбонитрил
1, получаемый
Полученные гетероциклические соединения, в
взаимодействием малононитрила [10] или димера
первую очередь производные пиразоло[1,5-a]пи-
малононитрила [10, 11] с гидразином, вследствие
римидина 4, привлекают внимание химиков благо-
высокой функционализации может вступать в ре-
даря доступности и интересному профилю биоло-
акции конденсации с образованием различных
гической активности (недавние обзорные работы
полигетероциклических производных пиразола.
по химии и применению пиразоло[1,5-a]пирими-
Так, соединение 1 широко используется в синте-
динов см. [25-29]). Производные пиразоло[1,5-a]-
зе для получения замещенных пиразоло[3,4-d]-
пиримидина широко представлены на рынке ле-
415
416
СЕМЕНОВА и др.
Схема 1.
H
N
NC CNNH2NH2
N
NH2NH2
NH2
NC CN
NC
NC
−NH3
-NH3
NH
2
CN
1
1
R
O
O
N
NH2
NC
N
N N
CH3
HN
N
R
NC
N
NC
N
2
N
O
4
R
NC
CH3
3
5
NH2
Ac
H
O HN
N
N
EWG
Ar
N
NH2
N
N
NC
H2N
N
N
N
Ar
CN
H
1
NC
CN
CN
2
6
H
HS N
H2N
NH2
N
N
HS
N HN
N
S
N
N
N
S
NH2
CN
H
NC
9
NH
CN
7
8
EWG - электроноакцепторная группа (CN, CO2R).
карственных средств; среди последних стоит от-
анаглиптин (Anagliptin, Suiny®), антивирусный
метить агонисты бензодиазепиновых рецепторов
препарат пресатовир (Presatovir) (схема 2). Следу-
ГАМКА - снотворные средства залеплон (Zaleplon,
ет также упомянуть используемый против мучни-
Sonata®), индиплон (Indiplon) и лоредиплон
стой росы фунгицид пиразофос (Pyrazophos).
(Lorediplon), анксиолитик оцинаплон (Ocinaplon),
Нужно отметить, что несмотря на доступность
ингибитор циклин-зависимых киназ с противоо-
пиразола 1 как исходного реагента, в литературе
пухолевым действием динациклиб (Dinaciclib), ин-
описано ограниченное число циклизаций в произ-
гибитор костных морфогенетических сигнальных
водные пиразоло[1,5-a]пиримидина с его участи-
белков (bone morphogenetic proteins, BMPs) дорсо-
ем [15-18, 30-34]. В продолжение исследований
морфин (Dorsomorphin), ингибиторы нейротроф-
в области реакций циклизации 3(5)-аминопиразо-
ных рецепторных тирозинкиназ (NTRK) ларотре-
лов [35-37], мы решили изучить взаимодействие
ктиниб (Larotrectinib, Vitrakvi®), селитректиниб
5-амино-3-(цианометил)-1Н-пиразол-4-карбони-
(Selitrectinib) и репотректиниб (Repotrectinib),
трила 1 с рядом 1,3-диэлектрофильных агентов.
ингибитор протекина MRP1 (multidrug resistance-
Установлено, что аминопиразол 1 легко реаги-
associated protein 1) и Р-гликопротеина (P-gp) ре-
рует с 1,3-дикетонами (дибензоилметаном и бен-
версан (Reversan), антидиабетический препарат
зоилтрифторацетоном) в кипящей АсОН с образо-
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
417
Схема 2.
H3C
H
3C
O
O
H3C
O
O
CN
N
N
N
N
N
H3C
N
N
N
N
S
N
N
N
N
Залеплон
Индиплон
Оцинаплон
O
H3C
N
NH
N
O
O
N
N
N
N
H3C
N
O
N
N
S
N
N
F
N
N
N
H3C
Лоредиплон
Дорсоморфин
OH
Динациклиб
F
H3
C H
N
N
F
N
N
N
N
F
N
F
H
OH
N N
O
H
N
N
N
N
N
N
H
HN
N
H3C
O
O
Ларотректиниб
O
Репотректиниб
CH3
Селитректиниб
O
H3C
O CN
H
O
N
N
N
N
N
H
N
N
H3C
N
N
NH
N
H3C
O
Реверсан
Анаглиптин
H3C
H2N
N N
O
O
O
N
P
CH3
N
H3C
O
N
S
N
N
H3C
O
O
H3C
N
HN
Cl
H3C
Пиразофос
S
O
O
Пресатовир
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
418
СЕМЕНОВА и др.
Схема 3.
Ph
Ph
Ph
N
N
O
NC
N Ph
AcOH, ∆
H
NC
12
Ph
N
PhC(O)CH2C(O)Ph
NC
N
NC
H
N
N
Ph
N
AcOH, ∆
10
NH2
NC
F
F
F
CN
PhC(O)CH2C(O)CF3
N
1
NC
N
NH
NC
N
N
водн. HCl, ∆
NH2
Ph
H
N
N
2N
H
NC
11
N
N
NC
N
NH2
13
ванием производных пиразоло[1,5-a]пиримидина
пуриновых оснований, пиразоло[1,5-a][1,3,5]три-
10 и 11 (выходы 63-66%). Следует указать, что по-
азины представляют особой интерес и обнаружи-
лучение соединения 10 было описано в недавней
вают широкий спектр биологической активности
работе [30] путем взаимодействия халкона (либо
(см. обзорные работы [38, 39]). Соединения 11-13
дибензоилметана) с аминопиразолом 1 в кипя-
являются новыми и в литературе не описаны;
щем ДМФА в присутствии основания (выход 70%,
близкие структурные аналоги соединения 11 и 13
спектральные характеристики идентичны). Реак-
представлены в патентных работах [40, 41] соот-
ция дибензилиденацетона с пиразолом 1 приводит
ветственно.
к образованию с умеренным выходом (39%) про-
Cтроение полученных соединений подтверж-
дукта конденсации 1:1, которому, по данным ЯМР,
дено данными ИК, ЯМР 1H и 13С DEPTQ спек-
соответствует структура дигидропиразолопири-
троскопии. В ИК спектрах всех соединений об-
мидина 12. При использовании цианогуанидина
наруживаются характерные полосы поглощения
(дициандиамида) как диэлектрофильного реагента
сопряженной (2208-2241 см-1) и несопряженной
был получен азааналог соединений 10 и 11, пи-
(2260-2264 см-1) цианогрупп, что указывает на
разоло[1,5-a][1,3,5]триазин 13 с низким выходом
неучастие этих функциональных фрагментов в
17% (схема 3). Низкий выход связан, вероятно, с
реакции. В ИК спектре пиразоло[1,5-a][1,3,5]три-
частичным гидролизом цианогрупп в условиях
азина 13 обнаруживаются интенсивные полосы
реакции и хорошей растворимостью продуктов в
поглощения, соответствующие наличию первич-
водно-кислотном растворе. Будучи биоизостерами
ных NH2-групп. В спектрах ЯМР всех соединений
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
419
по аналогичным структурам и по реакционной
Схема 4.
способности несимметричных фторированных ке-
F
F
Ph
тонов в аналогичных реакциях с 3(5)-аминопира-
F
N
N
NC
1
7
золами, а также на основании детального анализа
2
N
NC
1
7
2
N
6
спектральной картины. Кроме того, мы провели
6
3
4
3
4
NC
5
квантово-химические исследования строения ис-
NC
5
N
N
F
ходных реагентов (пиразола 1 и бензоилтрифто-
Ph
F F
рацетона) в контексте возможных реакционных
11A
11Б
путей взаимодействия.
Анализ литературных данных о взаимодей-
ствии несимметричных кетонов общего строения
обнаруживаются характерные сигналы протонов
CF3C(O)CH2C(O)R с различными 3(5)-аминопира-
СН2CN, атомов углерода цианогрупп и пиразоль-
золами показывает, что в подавляющем большин-
ного цикла. В ИК спектре дигидропиразолопири-
стве случаев реакция протекает региоселективно
мидина 12 обнаруживаются полосы поглощения,
с образованием 7-CF3-изомера [42-51]. При этом
соответствующие валентным колебаниям N-H, в
экзоциклическая аминогруппа взаимодействует с
спектре ЯМР 1Н отсутствует характерный сигнал
карбонильной группой С(O)R, а эндоциклический
ароматического протона Н6, проявляющийся для
NH пиразола атакует карбонильный атом углеро-
соединений 10 и 11 при 8.0-8.5 м. д., но присут-
да С(O)CF3. В то же время, описан ряд примеров
ствует мультиплетный сигнал при 6.92-6.93 м. д.
образования 5-CF3-изомеров в аналогичных усло-
(H5). Выбор между изомерными 4,5-дигидро- и
виях [52-54]. В работе [55] проведен детальный
4,7-дигидроструктурами сделан на основании от-
сравнительный анализ картины в спектрах ЯМР
сутствия в области сильного поля сигнала Н7; кро-
13С для различных региоизомеров на примере
ме того, алкеновые протоны стирильного фрагмен-
2-фенилпиразоло[1,5-a]пиримидинов. Показано,
та -СН=СH- обнаруживаются как пара дублетов
что сигнал углеродов С5 на ~10-13 м. д. сдви-
без дополнительного расщепления, что указывает
нут в область слабого поля относительно сигна-
на связь стирильного фрагмента с четвертичным
лов углеродов С7 при одинаковых заместителях
атомом углерода. Стирильный фрагмент находит-
(схема 5); углерод С5СF3 резонирует при ~146 м. д.,
ся в транс-конфигурации (исходя из значения
тогда как углерод С7СF3 - при ~133-135 м. д.
КССВ 3J 16.1 Гц).
Таким образом, квартет при 134.3 м. д. с констан-
В спектрах ЯМР 1Н и 13С DEPTQ соединений
той спин-спинового взаимодействия 2JCF 38.0 Гц в
спектре соединения 11 следует отнести к углероду
10 и 11 наблюдаются характерные сигналы анне-
лированного пиримидинового цикла - синглет
С7 (региоизомер 11А).
ароматического протона Н6 и сигналы углеродных
Квантово-химические расчеты осуществляли в
атомов С5-С7 (С6 при 108.0-108.2 м. д., С5 и С7 в
программном пакете ORCA 5.0 [56, 57] с исполь-
области слабого поля 134-160 м. д.). Отдельный
зованием гибридного функционала B3LYP [58, 59]
интерес представляет проблема региоселектив-
с дисперсионной поправкой D3BJ [60] в базисном
ности, наблюдаемой при взаимодействии 5-ами-
наборе def2-TZVPP [61]. Для визуализации мо-
но-3-(цианометил)-1Н-пиразол-4-карбонитрила
1
лекулярной геометрии использовали программу
с несимметричным 1,3-дикетоном - бензоилтриф-
ChemCraft 1.8. Для генерации Input-файлов приме-
торацетоном: из реакционной массы был выделен
няли программу Gabedit 2.5 [62]. Все расчеты про-
только один продукт, тогда как, в принципе, воз-
водили с учетом неспецифической сольватации в
можно образование двух региоизомерных пиразо-
рамках континуумной модели CPCM (раствори-
лопиримидинов - 7-CF3-изомера 11А и 5-CF3-изо-
тель - уксусная кислота) [63].
мера 11Б (схема 4).
Молекула бензоилтрифторацетона (4,4,4-три-
Выбор в пользу 7-CF3-региоизомера 11А сде-
фтор-1-фенилбутан-1,3-диона) может существо-
лан на основании анализа литературных данных
вать в виде трех таутомерных форм: дикетонной
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
420
СЕМЕНОВА и др.
Схема 5.
Ph
Ph
N
N
160.0
NC
159.5
NC
80.6
108.2
82.2
108.0
N
N
NC
147.9
NC
134.3
N
N
17.2
Ph
17.2
CF3
10
11A
119.1
По данным работы [53]:
N
Ph
N
CH3
N
CF3
156.2
146.1
157.7
Ph
Ph
Ph
N N
N N
133.7
N N
133.1
147.5
CF
3
CH3
CF3
N
CF3
N
Ph
N
CH3
146.1
155.7
158.3
Ph
Ph
Ph
N N
N N
N N
135.1
146.0
145.2
CF3
Ph
CH3
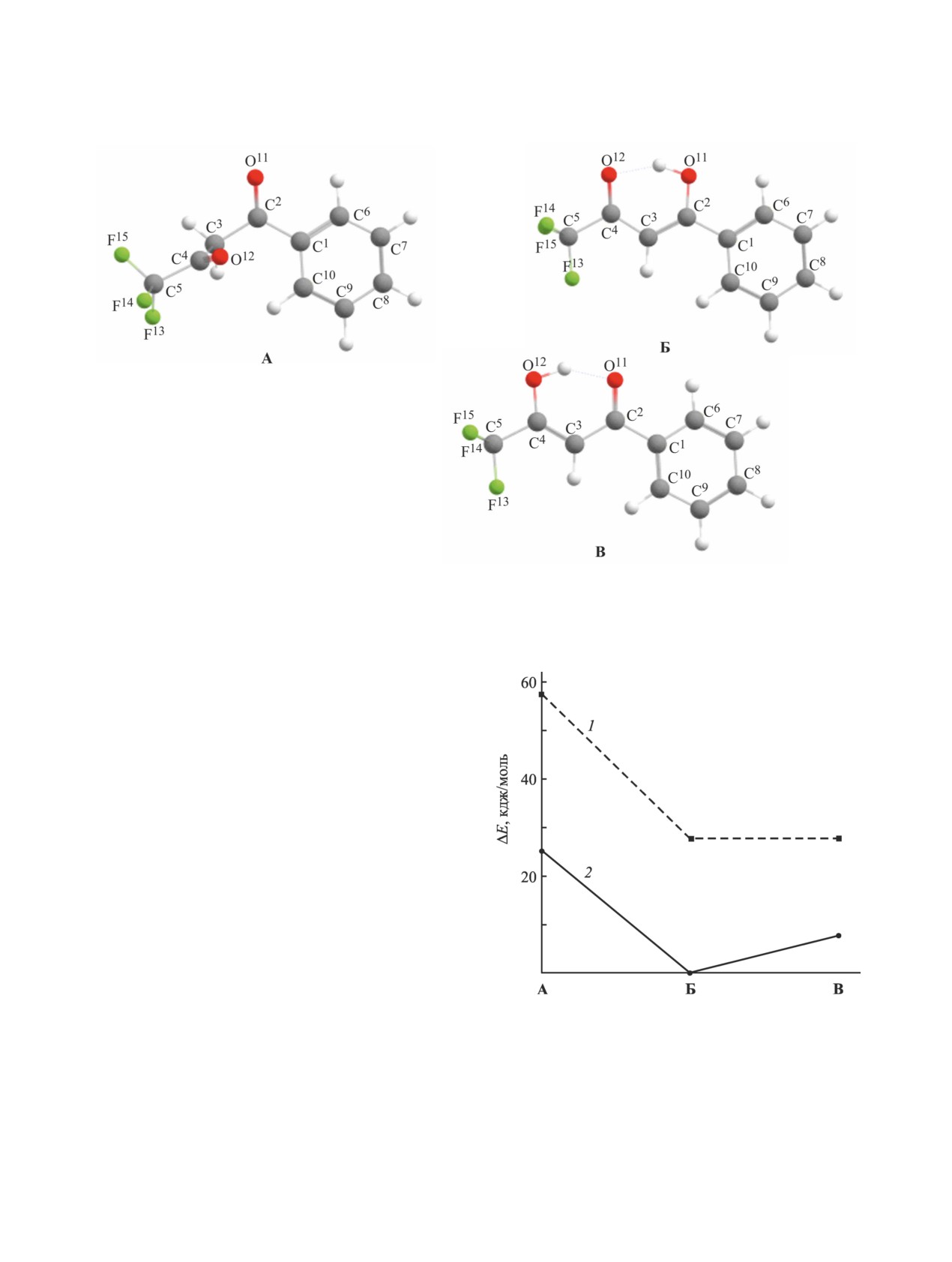
(A) и двух енольных (Б и В). Для оценки устой-
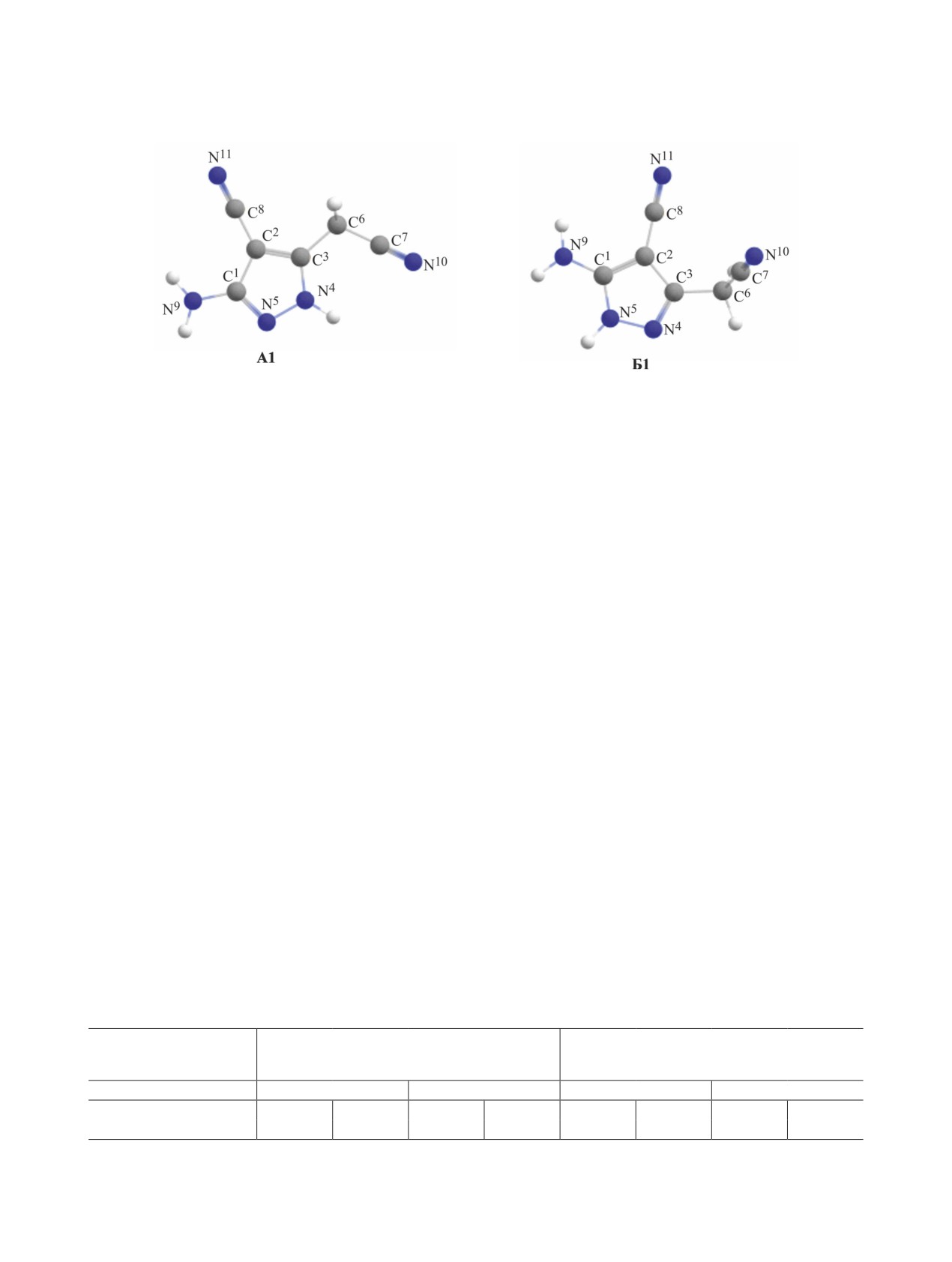
вость таутомерных форм A1 и Б1 5-амино-3-(ци-
чивости данных форм 4,4,4-трифтор-1-фенилбу-
анометил)-1Н-пиразол-4-карбонитрила 1 (рис. 3).
тан-1,3-диона был произведен квантово-хими-
Расчет без учета сольватации показал несколько
ческий DFT-расчет энергии таутомеров как в
большую устойчивость таутомерной формы A1
вакууме, так и в среде уксусной кислоты (учет рас-
(разница в энергии с таутомером Б1 составляет 5.1
творителя производили с помощью модели про-
кДж/моль), в то время как в среде уксусной кис-
водникового поляризуемого континуума CPCM).
лоты более устойчивым оказывается уже таутомер
Оптимизированные молекулярные структуры
Б1 (разница в энергии с оставляет 8.9 кДж/моль).
таутомеров представлены на рис. 1.
Учитывая незначительную разницу в энергии меж-
Результаты расчета энергии таутомеров
ду этими таутомерными формами, можно сделать
4,4,4-трифтор-1-фенилбутан-1,3-диона приведе-
вывод об их совместном присутствии в растворе.
ны на рис. 2. Как можно заметить, по расчетным
Для пиразола 1 теоретически возможно существо-
данным дикетонная форма А является наименее
вание и других таутомеров, однако их разница в
устойчивой как в вакууме, так и в среде АсОН
энергии с вышеуказанными таутомерными форма-
(разница в энергии с наиболее устойчивой еноль-
ми A1 и Б1 составляет более 90 кДж/моль как в ва-
ной формой составляет более 25 кДж/моль). Из
кууме, так и с учетом сольватации, поэтому далее
енольных форм несколько более устойчивой явля-
они не рассматриваются.
ется Б, однако разница в энергии с формой В неве-
С целью определения наиболее вероятных ре-
лика и составляет всего 0.2 кДж/моль в вакууме и
акционных путей были рассчитаны частичные
7.7 кДж/моль в уксусной кислоте, что указывает на
атомные заряды по Малликену для таутомерных
возможное существование таутомерного равнове-
форм как 4,4,4-трифтор-1-фенилбутан-1,3-диона,
сия между енольными формами Б и В.
так и пиразола 1. Результаты проведенных расче-
Аналогичным образом была оптимизирована
тов представлены в табл. 1. Результаты проведен-
молекулярная геометрия и определена устойчи-
ных расчетов показали, что в двух енольных тау-
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
421
Рис. 1. Оптимизированные на уровне B3LYP-D3BJ/def2-TZVPP молекулярные структуры таутомерных форм 4,4,4-триф-
тор-1-фенилбутан-1,3-диона: A - дикетонная форма, Б, В - енольные формы.
томерах
4,4,4-трифтор-1-фенилбутан-1,3-диона
(Б и В) атом углерода С2 обладает более высоким
частичным положительным зарядом по сравнению
с атомом С4, а в таутомерных пиразолах A1 и Б1
атом азота экзоциклической аминогруппы N9 об-
ладает более высоким частичным отрицательным
зарядом, чем эндоциклический атом пиразола N5.
Это позволяет сделать вывод о том, что на первом
этапе реакции наиболее вероятно реализуется ну-
клеофильная атака атома N9 аминогруппы пиразо-
ла 1 на углеродный атом С2 енольной формы бен-
зоилтрифторацетона, что в итоге и обусловливает
региоселективное образование С7-CF3 изомера 11.
Многие производные пиразоло[1,5-a]пири-
мидина [25-29] и пиразоло[1,5-a][1,3,5]триазина
[38, 39, 41] обладают биологической активностью.
В контексте перспективности изучения активности
Рис. 2. Энергии таутомеров 4,4,4-трифтор-1-
синтезированных соединений нами был проведен
фенилбутан-1,3-диона, рассчитанные без учета
расчет параметров биодоступности для структур
влияния растворителя (1) и с учетом неспецифической
10-13 in silico. Первичный анализ структур на со-
сольватации в среде уксусной кислоты (2),
ответствие «правилу пяти» К. Липински [сLogP ≤
относительно минимального значения - энергии
енольной формы Б в среде уксусной кислоты.
5.0, молекулярная масса (MW) ≤ 500, TPSA ≤ 140 Å2,
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
422
СЕМЕНОВА и др.
Рис. 3. Оптимизированные на уровне B3LYP-D3BJ/def2-TZVPP молекулярные структуры таутомерных форм A1 и Б1
пиразола 1.
число акцепторов водородных связей ≤ 10, до-
брану или гематоэнцефалический барьер. В то же
норов ≤ 5] [64-66] сделан с использованием рас-
время, значение logS < -4.0 для соединений 10-12
четного сервиса OSIRIS Property Explorer [67].
указывает на невысокую растворимость (менее
Были оценены параметры сLogP [вычисленный
1×10-4 моль/л). Для прогнозирования параметров
логарифм коэффициента распределения между
ADMET (Absorption, Distribution, Metabolism,
н-октанолом и водой log(coctanol/cwater)], растворимо-
Excretion, Toxicity) использовали программные
сти (logS), площади топологической полярной по-
пакеты SwissADME [68], admetSAR [69], GUSAR
верхности (Topological Polar Surface Area, TPSA),
[70, 71]. Оценка острой токсичности позволяет от-
токсикологических параметров - рисков побочных
нести все соединения к IV (300 мг/кг < LD50 < 2000
эффектов (мутагенные, онкогенные, репродуктив-
мг/кг) и V (2000 мг/кг < LD50 < 5000 мг/кг) классам
ные эффекты), сходства с известными лекарствен-
опасности согласно критериям OECD [72]. Для
ными препаратами (drug-likeness), а также общей
всех соединений прогнозируется высокая гастро-
оценки фармакологического потенциала соедине-
энтеральная абсорбция и возможность прохожде-
ния (drug score). Полученные данные суммирова-
ния через гематоэнцефалический барьер (табл. 3).
ны в табл. 2. В целом, все соединения показывают
Оценка возможного мутагенного действия в тесте
отсутствие рисков токсичности, достаточно благо-
Эймса указывает на вероятное отсутствие подоб-
приятное сочетание параметров биодоступности и
ных эффектов (кроме соединения 12).
высокие значения drug score (0.26-0.43). Значения
Молекулярный докинг с целью поиска воз-
показателей сLogP << 5.0, TPSA (< 140 Å2, кроме
можных протеиновых мишеней для полученных
соединения 13) указывают на вероятную способ-
соединений
10-13 проведен с использовани-
ность к проникновению через клеточную мем-
ем нового протокола протеин-лигандного до-
Таблица 1. Малликеновские заряды на атомах C2 и C4 в енольных таутомерах бензоилтрифторацетона (4,4,4-трифтор-
1-фенилбутан-1,3-диона) и на атомах N5 и N9 в таутомерах 5-амино-3-(цианометил)-1Н-пиразол-4-карбонитрила 1,
рассчитанные на уровне B3LYP-D3BJ/def2-TZVPP
5-Амино-3-(цианометил)-1Н-пиразол-4-
Параметр
4,4,4-Трифтор-1-фенилбутан-1,3-дион
карбонитрил 1
Таутомер
Б
В
A1
Б1
Атом
C2
C4
C2
C4
N5
N9
N5
N9
Заряд по Малликену
0.299
0.139
0.250
0.089
-0.251
-0.298
-0.017
-0.288
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
423
Таблица 2. Риски токсичности и физико-химические параметры соединений 10-13, спрогнозированные с помощью
сервиса OSIRIS Property Explorer
Риск токсичностиа
Физико-химические параметры
Соединение
А
В
С
D
сLogP
logS
MW
TPSA
drug-likeness
drug score
NC
N
Ph
NC
N N
-
-
-
-
2.96
-6.44
335
77.77
-2.78
0.28
Ph
10
NC
N
Ph
-
-
-
-
2.11
-5.47
327
77.77
-10.14
0.32
NC
N N
CF3
11
Ph
HN
NC
N
-
-
-
-
3.72
-6.12
363
77.43
-4.63
0.26
N
Ph
CN
12
NC
N
NH2
N
NC
N N
-
-
-
-
-1.28
-3.96
214
142.7
-4.18
0.43
NH2
13
a Знаком «+» показан высокий риск токсичности, «-» - отсутствие токсичности. A - Мутагенность, B - канцерогенность, С -
раздражающее действие, D - репродуктивные эффекты.
кинга GalaxySagittarius [73] на базе веб-сервера
ин-лигандного взаимодействия. Прогнозируемые
GalaxyWeb [74, 75]. Предварительно 3D-струк-
протеиновые мишени указаны с помощью ID-
туры соединений были оптимизированы сред-
идентификаторов в Protein Data Bank (PDB) и в базе
ствами молекулярной механики в силовом поле
данных UniProt. Как можно заметить из табл. 4,
ММ2 для оптимизации геометрии и минимизации
общими протеиновыми мишенями со значениями
энергии. Докинг проводили в режимах Binding
скоринг-функций ΔGbind = -18÷-22 ккал/моль для
compatability prediction и Re-ranking using docking.
соединений 10 и 11 являются янус-киназа JAK2
В табл. 4 представлены результаты докинга (луч-
(PDB ID 4hge, участвует в передаче сигналов цито-
шие 20 результатов) по каждому из соединений
киновыми рецепторами типа II - рецепторами ин-
10-13 с указанием комплексов мишень-лиганд
терферона), бактериальная гликозил-гидролаза -
с минимальной свободной энергией связывания
α-N-ацетилгалактозаминидаза NagBb (PDB ID
ΔGbind (ккал/моль) и наилучшей оценкой проте-
5wzr), протеинкиназа VEGFR-2 (PDB ID 3wze,
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
424
СЕМЕНОВА и др.
аблица 3. Расчетные параметры ADMETа для соединений 10-13
Острая токсичность (крысы),
Ингибирование цитохромов Р450
LD50, log
10
(ммоль/кг) мг/кгб
+
+
+
+
+
-
+
-
0.002
-0.383
0.692
0.352
10
0.9564
1.0000
0.8965
0.6937
0.7532
0.9185
0.5000
0.6253
337.2
138.8
1652.0
754.5
+
+
+
+
+
-
-
-
0.059
-0.307
0.105
0.220
11
0.9621
1.0000
0.9036
0.8214
0.7772
0.7953
0.6795
0.5997
374.9
161.2
416.8
543.3
+
+
+
+
+
-
+
+
0.081
-0.574
0.346
0.452
12
0.9132
1.0000
0.8810
0.7140
0.7954
0.8449
0.7088
0.5640
437.5
96.87
805.3
1029.0
+
+
+
-
-
-
-
-
0.136
-0.399
0.714
0.344
13
0.8762
0.9974
0.8144
0.6805
0.8119
0.8557
0.7622
0.5606
293.0
85.44
1108.0
473.1
а Знаком «+» или «-» показано наличие или отсутствие эффекта.
б IP - внутрибрюшинный способ введения; IV - внутривенный путь введения; Oral - пероральный путь введения; SC - подкожный
путь введения.
регулирует ангиогенез и развитие эндотелиаль-
бензоилтрифторацетоном протекает региоселек-
ных клеток). Для соединения 12 приоритетны-
тивно с образованием только одного региоизомера,
ми мишенями являются протоонкогенная серин/
для которого данными спектроскопии ЯМР в сово-
треониновая протеинкиназа Pim-1 (PDB ID 3bgq,
купности со сравнительным анализом литературы
ΔGbind = -21.4 ккал/моль), p38альфа митоген-ак-
и квантово-химическими расчетами подтверждено
тивируемая протеинкиназа (MAPK, PDB ID 3itz,
строение
7-(трифторметил)-5-фенил-2-(цианоме-
ΔGbind = -22.9 ккал/моль), для пиразоло-1,3,5-три-
тил)пиразоло[1,5-a]пиримидин-3-карбонитрила.
азина 13 - сериновая протеаза PLAU (PDB ID
Результаты молекулярного докинга и эксперимен-
1gj7, ΔGbind = -15.2 ккал/моль, активатор фибри-
тов по оценке параметров ADMET и биодоступно-
нолитической ферментной системы), циклофи-
сти in silico позволяют рассматривать полученные
лин А (пептидилпролил-цис-транс-изомераза A,
производные пиразола как перспективные объек-
PDB ID 5nor, ΔGbind = -14.2 ккал/моль, участвует
ты для дальнейшего скрининга с целью поиска ан-
в патогенезе аутоиммунных, сердечно-сосудистых
титромботических и противоопухолевых агентов.
и онкологических заболеваний) и др. Трехмер-
Взаимодействие
5-амино-3-(цианометил)-1Н-пи-
ная визуализация результатов докинга реализо-
разол-4-карбонитрила с другими 1,3-диэлектро-
вана средствами программного комплекса UCSF
фильными реагентами и оптимизация описанных
Chimera [76, 77] и представлена на рис. 4.
способов получения конденсированных пиразолов
составят предмет дальнейших исследований.
Таким образом, взаимодействием 5-амино-3-
(цианометил)-1Н-пиразол-4-карбонитрила с рядом
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
1,3-диэлектрофильных реагентов были получены
новые производные пиразоло[1,5-a]пиримидина
ИК спектры получали на спектрофотометре
и пиразоло[1,5-a][1,3,5]триазина. Реакция 5-ами-
Bruker Vertex 70 с приставкой НПВО методом
но-3-(цианометил)-1Н-пиразол-4-карбонитрила с
нарушенного полного внутреннего отражения на
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
425
Таблица 4. Результаты прогнозирования протеин-лигандного взаимодействия для соединений 10-13 с использова-
нием протокола молекулярного докинга GalaxyWeb Sagittarius
Свободная
Пре-докинговая
Общая оценка
Иденти-
энергия
Идентификатор
оценка протеин-
протеин-
фикатор
связывания
Структура
протеина
лигандного
лигандного
протеина
ΔGbind,
UniProt ID
взаимодействия
взаимо-
PDB ID
ккал/моль
(predock score)
действия
(docking score)
4hge
O60674
0.199
-21.184
0.358
2c3k
O14757
0.197
-20.155
0.348
4mbl
P11309
0.185
-21.397
0.345
4gj3
P29597
0.191
-20.303
0.343
4hvh
P52333
0.173
-20.981
0.330
5up3
Q99683
0.157
-21.116
0.315
5wrz
P09874
0.148
-21.794
0.312
NC
3wze
P35968
0.162
-19.627
0.309
N
Ph
3zbx
P08581
0.154
-20.590
0.308
N
NC
N
6cmm
Q99986
0.138
-22.133
0.304
Ph
5gjf
O43318
0.139
-21.926
0.304
10
3itz
Q16539
0.147
-20.725
0.302
4o7c
O60885
0.150
-20.229
0.302
6hzu
P23458
0.144
-20.856
0.301
6mom
Q9NWZ3
0.142
-21.150
0.300
4kwp
P68400
0.150
-19.824
0.299
6npu
P00519, P00519
0.110
-24.626
0.295
1unl
Q00535
0.140
-19.873
0.289
2c5n
P20248, P24941
0.132
-20.879
0.289
3tku
Q9Y5S2
0.119
-21.596
0.281
4hge
O60674
0.117
-17.821
0.251
5wrz
P09874
0.106
-18.881
0.247
4kao
Q05397
0.110
-17.450
0.241
3zc5
P08581
0.097
-18.925
0.239
4li7
O95271, O95271
0.078
-21.168
0.237
2ao6
P10275, Q15596
0.074
-20.670
0.229
6npu
P00519, P00519
0.075
-20.351
0.227
NC
N
Ph
4ft7
O14757
0.096
-17.553
0.227
3q4u
Q04771
0.081
-19.343
0.226
N
NC
N
4idv
Q99558
0.078
-19.499
0.224
CF3
6glb
P52333
0.081
-18.848
0.222
11
5hcl
O60885
0.094
-16.997
0.222
5c87
P55201
0.081
-18.770
0.222
3sxs
P51813
0.067
-20.636
0.222
3piy
Q06187
0.079
-18.944
0.221
4l7o
Q9Y6F1
0.080
-18.791
0.221
5n4v
P11309
0.089
-17.388
0.219
3wze
P35968
0.076
-19.098
0.219
1q5k
P49841, P49841
0.085
-17.803
0.219
5f1z
P29597
0.081
-18.190
0.217
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
426
СЕМЕНОВА и др.
Таблица 4. (Продолжение)
Свободная
Пре-докинговая
Общая оценка
Иденти-
энергия
Идентификатор
оценка протеин-
протеин-
фикатор
связывания
Структура
протеина
лигандного
лигандного
протеина
ΔGbind,
UniProt ID
взаимодействия
взаимо-
PDB ID
ккал/моль
(predock score)
действия
(docking score)
3bgq
P11309
0.301
-21.402
0.461
3itz
Q16539
0.217
-22.891
0.388
4ft7
O14757
0.228
-20.194
0.380
4hvh
P52333
0.208
-21.860
0.371
4c61
O60674
0.205
-20.899
0.361
Ph
4twp
P00519
0.211
-19.459
0.357
HN
NC
4gii
P29597
0.195
-20.985
0.352
N
4o7c
O60885
0.171
-23.919
0.350
N
5gjf
O43318
0.172
-23.679
0.349
Ph
CN
6gqo
P35968
0.192
-20.664
0.347
12
6mom
Q9NWZ3
0.172
-22.486
0.341
5hcx
P00533
0.181
-21.201
0.340
6e2n
Q99683, Q99683
0.171
-20.908
0.328
5jzn
O15075, O15075
0.172
-20.608
0.326
3piy
Q06187
0.147
-23.196
0.321
5d7v
Q13882
0.174
-19.462
0.320
1uwj
P15056, P15056
0.146
-23.061
0.319
3f88
P49841
0.149
-22.289
0.316
3tku
Q9Y5S2
0.153
-21.424
0.314
4fr4
Q8WU08
0.144
-22.466
0.313
1gj7
P00749
0.160
-15.147
0.274
5nor
P62937
0.137
-14.208
0.244
4ear
P00491, P00491
0.111
-14.180
0.217
2j0f
P19971, P19971
0.081
-15.592
0.198
4mtl
Q5VZV1, Q5VZV1
0.066
-16.812
0.192
NC
N
NH2
2obf
P11086
0.074
-15.713
0.192
6o2y
O75874, O75874
0.084
-14.351
0.191
N
NC
N N
3fvo
P39086, P39086
0.079
-14.211
0.185
NH2
3bn9
P01764, Q9Y5Y6
0.063
-15.526
0.179
13
2bzn
Q9P2T1, Q9P2T1
0.067
-14.962
0.179
4n7m
P55212, P55212
0.069
-14.536
0.178
4a6l
P20231
0.068
-14.708
0.178
5i9j
Q14849
0.080
-13.005
0.178
3euf
Q16831, Q16831
0.073
-13.977
0.178
3hme
Q9H8M2
0.072
-14.026
0.178
5w5o
O43353, O43353
0.066
-14.565
0.175
5qqz
P22557, P22557
0.073
-13.562
0.175
6qh9
Q92876, Q92876
0.057
-15.602
0.174
5ou0
P15121
0.063
-14.736
0.173
2a1u
P13804, P38117
0.075
-13.087
0.173
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
427
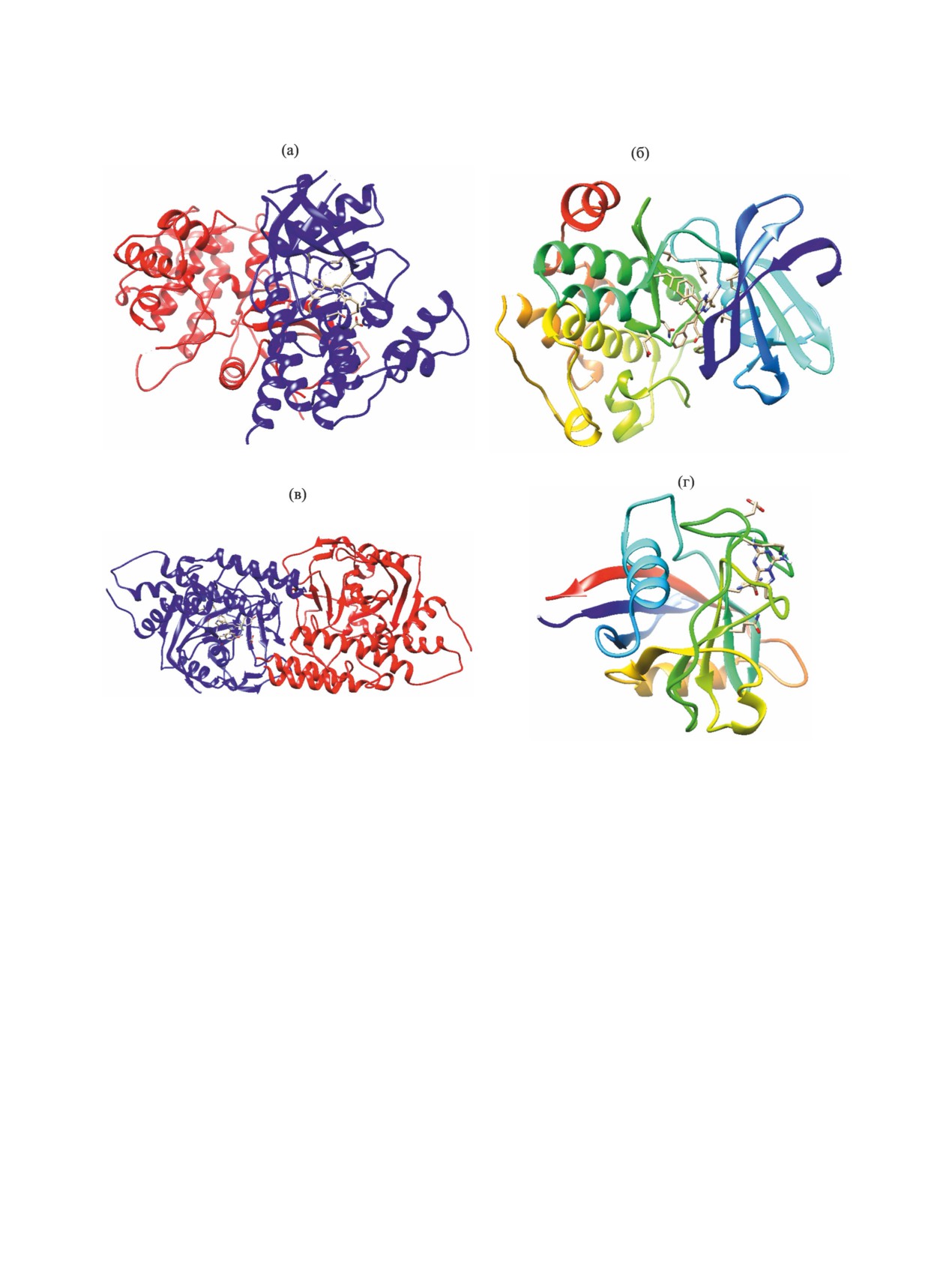
Рис. 4. Прогнозируемые структуры протеин-лигандных комплексов соединения 10 и киназы JAK2 (PDB ID 4hge) (а),
соединения 12 и протеинкиназы Pim-1 (PDB ID 3bgq) (б), соединения 11 и бактериальной α-N-ацетилгалактозаминидазы
(PDB ID 5wzr) (в), соединения 13 и пептидил-пролил-цис-транс-изомеразы A (PDB ID 5nor) (г).
кристалле алмаза, погрешность ±4 см-1. Спек-
5,7-Дифенил-2-(цианометил)пиразоло[1,5-a]-
тры ЯМР регистрировали на приборе Bruker
пиримидин-3-карбонитрил
(10). Смесь
0.5 г
Avance III HD 400MHz (400.17 MГц на ядрах 1Н,
(3.4 ммоль) пиразола 1 и 0.76 г (3.4 ммоль) ди-
100.63 МГц - 13С) в растворе ДМСО-d6, в качестве
бензоилметана в 4 мл ледяной АсОН кипятили
стандарта использовали остаточные сигналы рас-
6 ч, затем охлаждали. Реакционную массу выдер-
творителя. Элементный анализ проводили на при-
живали 72 ч при 20°С, осадок отфильтровывали,
боре Elementar Vario Microcube. Индивидуальность
промывали EtOH, петролейным эфиром и сушили
полученных образцов контролировали методом
при 60°С. Выход 63%, светло-коричневый мелко-
ТСХ на пластинах Сорбфил-А (ООО Имид, Красно-
кристаллический порошок. Соединение 10 рас-
дар), элюент - ацетон или этилацетат, проявитель -
творимо в ДМФА, ДМСО и умеренно - в горячем
пары иода, УФ детектор.
хлороформе. ИК спектр, ν, см-1: 2260 сл, 2222 с
5-Амино-3-(цианометил)-1Н-пиразол-4-карбо-
(2C≡N), 1614 с, 1599 с, 1551 с (C=С, С=N). Спектр
нитрил 1 получали реакцией димера малонони-
ЯМР 1Н, δ, м. д.: 4.55 с (2Н, CH2CN), 7.59-7.66 м
трила с гидразингидратом [10].
(6Н, H-Ph), 8.11 с (1Н, Н6), 8.15-8.17 м (2Н, H-Ph),
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
428
СЕМЕНОВА и др.
8.38-8.40 м (2Н, H-Ph). Спектр ЯМР 13С DEPTQ,
7.72 д (1Н, Ph-CH=CH, 3J 16.1 Гц). Спектр ЯМР
δC, м. д.: 17.2 (CH2), 80.6 (С3), 108.2* (С6Н), 112.7
13С DEPTQ, δC, м. д.: 23.2 (CH2), 71.9 (C5), 80.1
(С≡N), 116.2 (C≡N), 127.8* (2С, Ph), 128.6* (2С,
(С3), 112.3* (С6Н), 113.0 (С≡N), 117.1 (C≡N), 127.9*
Ph), 129.2* (2С, Ph), 129.6 (C1, Ph), 130.1* (2С, Ph),
(2С, Ph), 128.8* (С4Н Ph), 129.0* (2С, Ph), 129.1*
131.75* (C4, Ph), 131.83* (C4, Ph), 135.4 (C1, Ph),
(2С, Ph), 129.4* (2С, Ph), 129.5* (С4Н Ph), 129.6*
147.9 (C7), 150.8 (C2 или С3а), 151.8 (C3а или С2),
(CH=), 129.9* (CH=), 133.7 (C1 Ph), 134.2 (C1 Ph),
159.5 (С5). Здесь и далее звездочкой отмечены сиг-
136.4 (C7), 149.4 (C2 или С3а), 154.3 (C3а или С2).
налы в противофазе. Найдено, %: C 75.15; H 4.08;
Найдено, %: C 76.06; H 4.90; N 19.15. C23H17N5.
N 20.76. C21H13N5. Вычислено, %: C 75.21; H 3.91;
Вычислено, %: C 76.01; H 4.71; N 19.27. M 363.41.
N 20.88. M 335.36.
2,4-Диамино-7-(цианометил)пиразоло[1,5-a]-
7-(Трифторметил)-5-фенил-2-(цианометил)-
[1,3,5]триазин-8-карбонитрил (13). Смесь 0.5 г
пиразоло[1,5-a]пиримидин-3-карбонитрил
(3.4 ммоль) пиразола 1 и 0.29 г (3.4 ммоль) дициан-
(11). Смесь 0.6 г (4.1 ммоль) пиразола 1 и 0.89 г
диамида в 5 мл воды кипятили 8.5 ч в присутствии
(4.1 ммоль) бензоилтрифторацетона в 5 мл ледяной
0.1 мл конц. раствора HCl. Смесь охлаждали, через
АсОН кипятили 10 ч, затем охлаждали. Реакцион-
48 ч осадок отфильтровывали, промывали водой,
ную массу выдерживали 72 ч при 20°С, осадок от-
перемешивали с водным раствором соды, отфиль-
фильтровывали, промывали EtOH, петролейным
тровывали, промывали водой и сушили при 60°С.
эфиром и сушили при 60°С. Получали соедине-
Выход 17%, коричневый порошок. ИК спектр, ν,
ние 11 в аналитически чистом виде, коричневый
см-1: 3435, 3331, 3146 уш. с (N-H), 2264 сл, 2226
мелкокристаллический порошок, растворимый в
с (2 C≡N), 1602 уш. с, 1573 уш. с (C=С, С=N).
ДМФА. Выход 66%. ИК спектр, ν, см-1: 2262 сл,
Спектр ЯМР 1Н, δ, м. д.: 4.25 с (2Н, CH2CN), 7.12
2241 с (2 C≡N), 1632 с, 1576 с (C=С, С=N). Спектр
и 7.29 уш. с (2Н, NH2), 8.05 и 8.43 уш. с (2Н, NH2).
ЯМР 1Н, δ, м. д.: 4.66 с (2Н, CH2CN), 7.61-7.67 м
Спектр ЯМР 13С DEPTQ, δC, м. д.: 16.8 (CH2), 74.1
(3Н, H3-Н5, Ph), 8.40 д. д (1Н, H2, Н6, Ph, 3J 7.8, 4J
(C3), 113.4 (С≡N), 116.3 (C≡N), 149.1 (C5), 150.0
1.7 Гц), 8.47 с (1Н, Н6). Спектр ЯМР 13С DEPTQ,
(C2), 155.8 (C3а), 161.1 (С7). Найдено, %: C 44.70;
δC, м. д.: 17.2 (CH2), 82.2 (С3), 108.0* к (С6Н, 3JCF
H 3.04; N 52.17. C8H6N8. Вычислено, %: C 44.86; H
3.9 Гц), 111.9 (С≡N), 116.0 (C≡N), 119.1 к (CF3, 1JCF
2.82; N 52.32. M 214.19.
275.0 Гц), 128.3* (2С, Ph), 129.4* (2С, Ph), 132.7*
(C4, Ph), 134.3 к (C7, 2JCF 38.0 Гц), 134.5 (C1, Ph),
ИНФОРМАЦИЯ ОБ АВТОРАХ
151.6 (C2 или С3а), 151.8 (C3а или С2), 160.0 (С5).
Семенова Аминат Манафовна, ORCID: https://
Найдено, %: C 58.65; H 2.56; N 20.34. C16H8F3N5.
orcid.org/0000-0003-0639-9916
Вычислено, %: C 58.72; H 2.46; N 21.40. M 327.26.
Гаджиахмедова Ядигар Рамидиновна, ORCID:
7-(E)-Стирил-5-фенил-2-(цианометил)-
4,5-дигидропиразоло[1,5-a]пиримидин-3-карбо-
нитрил (12). Смесь 0.5 г (3.4 ммоль) пиразола 1
Беспалов Александр Валерьевич, ORCID:
и 0.80 г (3.4 ммоль) дибензальацетона в 5 мл ледя-
ной АсОН кипятили 15 ч. При этом наблюдалось
Доценко Виктор Викторович, ORCID: http://
образование осадка из кипящего раствора. Реакци-
orcid.org/0000-0001-7163-0497
онную массу охлаждали и выдерживали 48 ч при
Аксенов Николай Александрович, ORCID:
20°С. Осадок отфильтровывали, промывали EtOH,
петролейным эфиром и сушили при 60°С. Выход
Аксенова Инна Валерьевна, ORCID: http://
39%, темно-оранжевый порошок. ИК спектр, ν,
orcid.org/0000-0002-8083-1407
см-1: 3230 ср (N-H), 2260 сл, 2208 с (C≡N), 1650
сл, 1603 с, 1591 с (C=С, С=N). Спектр ЯМР 1Н, δ,
БЛАГОДАРНОСТЬ
м. д.: 4.16 с (2Н, CH2CN), 6.92-6.93 м (1Н, Н5), 7.22
д (1Н, Ph-CH=CH, 3J 16.1 Гц), 7.33-7.37 м (1Н, H4,
Аналитические исследования выполнены с
Ph), 7.40-7.43 м (2Н, H3, Н5, Ph), 7.50-7.58 м (7Н,
использованием научного оборудования Центра
H6, NH, H-Ph), 7.63 д (2Н, H2, Н6, Ph, 3J 7.3 Гц),
коллективного пользования Национального иссле-
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
429
довательского центра «Курчатовский институт» -
12.
Elkholy A., Al-Qalaf F., Elnagdi M.H. // Arkivoc. 2008.
ИРЕА при финансовой поддержке Министерства
Vol. xiv. P. 124. doi 10.3998/ark.5550190.0009.e14
13.
Kankanala J., Marchand C., Abdelmalak M., Aihara H.,
образования и науки России (соглашение № 075-
Pommier Y., Wang Z. // J. Med. Chem. 2016. Vol. 59.
11-2021-070 от 19.08.2021).
N 6. P. 2734. doi 10.1021/acs.jmedchem.5b01973
ФОНДОВАЯ ПОДДЕРЖКА
14.
Metwally N.H., Deeb E.A. // Synth. Commun. 2018. Vol. 48.
N 13. P. 1614. doi 10.1080/00397911.2018.1457162
Работа выполнена при финансовой поддержке
15.
Metwally N.H., Abdallah M.A., Almabrook S.A. // J.
Министерства образования и науки Российской
Heterocycl. Chem. 2017. Vol. 54. N 1. P. 347. doi
Федерации (тема 0795-2020-0031).
10.1002/jhet.2590
16.
Hassan M.I., Hassane A.M.A. // Egypt. J. Chem.
КОНФЛИКТ ИНТЕРЕСОВ
2019. Vol. 62, Part 1. P. 103. doi 10.21608/
EJCHEM.2019.14725.1907
Авторы заявляют об отсутствии конфликта
17.
Golubev P., Karpova E.A., Pankova A.S., Sorokina M.,
интересов.
Kuznetsov M.A. // J. Org. Chem. 2016. Vol. 81. N 22.
P. 11268. doi 10.1021/acs.joc.6b02217
СПИСОК ЛИТЕРАТУРЫ
18.
Ragab E.A., Metwally N.H., Mohamed M.S. //
1.
Elnagdi M.H., Abdel-Galil F.M., Riad B.Y., Elge-
Synth. Commun. 2017. Vol. 47. N 2. P. 148. doi
meie G.E.H. // Heterocycles. 1983. Vol. 20. N 12.
10.1080/00397911.2016.1257722
19.
Elghandour A.H.H., Elmoghayar M.R.H.,
P. 2437. doi 10.3987/R-1983-12-2437
2.
Abu Elmaati T.M., El-Taweel F.M. // J. Heterocycl.
Ramiz M.M.M. // J. Prakt. Chem. 1988. Vol. 330. N 4.
Chem. 2004. Vol. 41. N 2. P. 109. doi 10.1002/
P. 657. doi 10.1002/prac.19883300424
jhet.5570410201.
20.
Naik N.S., Shastri L.A., Shastri S.L., Chougala B.M.,
3.
Fichez J., Busca P., Prestat G. In: Targets in
Shaikh F., Madar J.M., Kulkarni R.C., Dodamani S.,
Heterocyclic Systems / Eds O.A. Attanasi, P. Merino,
Jalalpure S., Joshi S.D., Sunagar V. // ChemistrySelect.
D. Spinelli. Roma: Italian Society of Chemistry, 2017.
2019. Vol. 4. N 1. P. 285. doi 10.1002/slct.201802927
Vol. 21. P. 322. doi 10.17374/targets.2018.21.322
21.
Abdelmoniem A.M., Ghozlan S.A., Abdelmoniem D.M.,
4.
Anwar H.F., Elnagdi M.H. // Arkivoc. 2009. Vol. i.
Elwahy A.H., Abdelhamid I.A. // J. Heterocycl. Chem.
P. 198. doi 10.3998/ark.5550190.0010.107
2018. Vol. 55. N 12. P. 2792. doi 10.1002/jhet.3346
5.
Aggarwal R., Kumar V., Kumar R., Singh S.P. //
22.
Elnagdi M.H., El-Moghayar M.R., Fleita D.H.,
Beilstein J. Org. Chem. 2011. Vol. 7. N 1. P. 179. doi
Hafez E.A., Fahmy S.M. // J. Org. Chem. 1976. Vol. 41.
10.3762/bjoc.7.25
N 24. P. 3781. doi 10.1021/jo00886a002
6.
Aggarwal R., Kumar S. // Beilstein J. Org. Chem. 2018.
23.
Bulychev Y.N., Korbukh I.A., Preobrazhenskaya M.N. //
Vol. 14. N 1. P. 203. doi 10.3762/bjoc.14.15
Chem. Heterocycl. Compd. 1981. Vol. 17. N 4. P. 392.
7.
Shaabani A., Nazeri M.T., Afshari R. // Mol. Divers.
doi 10.1007/BF00503346
2019. Vol. 23. N 3. P. 751. doi 10.1007/s11030-018-
24.
Elnagdi M.H., Erian A.W. // Lieb. Ann. Chem. 1990.
9902-8
Vo l .
1990. N
12. P.
1215. doi
10.1002/
8.
Chebanov V.A., Gura K.A., Desenko S.M. In: Synthesis
jlac.1990199001219
of Heterocycles via Multicomponent Reactions I.
25.
Al-Azmi A. // Curr. Org. Chem. 2019. Vol. 23. N 6.
Topics in Heterocyclic Chemistry book series / Eds
P. 721. doi 10.2174/1385272823666190410145238
R.V.A. Orru, E. Ruijter . Heidelberg: Springer, 2010.
26.
Castillo J.C., Portilla J. In: Targets in Heterocyclic
Vol. 23. P. 41. doi 10.1007/7081_2009_21
Systems / Eds O.A. Attanasi, P. Merino, D. Spinelli.
9.
Marinozzi M., Marcelli G., Carotti A. // Mini Rev. Med.
Roma: Italian Society of Chemistry, 2018. Vol. 22.
Chem. 2015. Vol. 15. N 4. P. 272. doi 10.2174/1389557
P. 194. doi 10.17374/targets.2019.22.194
515666150312154536
27.
Salem M.A., Helal M.H., Gouda M.A., Abd El-
10.
Taylor E.C., Hartke K.S. // J. Am. Chem. Soc. 1959.
Gawad H.H., Shehab M.A.M., El-Khalafawy A. //
Vol. 81. N 10. P. 2452. doi 10.1021/ja01519a044
Synth. Commun. 2019. Vol. 49. N 14. P. 1750. doi
11.
Доценко В.В., Кривоколыско С.Г., Семенова А.М. //
10.1080/00397911.2019.1604967
ХГС. 2018. Т. 54. № 11. С. 989; Dotsenko V.V.,
28.
Cherukupalli S., Karpoormath R., Chandrasekaran B.,
Krivokolysko S.G., Semenova A.M. // Chem. Heterocycl.
Hampannavar G.A., Thapliyal N., Palakollu V.N. // Eur.
Compd. 2018. Vol. 54. N. 11. P. 989. doi 10.1007/
J. Med. Chem. 2017. Vol. 126. P. 298. doi 10.1016/j.
s10593-018-2383-y
ejmech.2016.11.019
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
430
СЕМЕНОВА и др.
29.
Arias-Gómez A., Godoy A., Portilla J. // Molecules.
Russ. Chem. Bull. Int. Ed. 2021. Vol. 70. N 3. P. 600.
2021. Vol. 26. N 9. P. 2708. doi 10.3390/
doi 10.1007/s11172-021-3131-5
molecules26092708
46.
Hauck S., Hiesinger K., Hosseini S.K., Achenbach J.,
30.
Metwally N.H., Mohamed M.S., Deeb, E.A. // Res.
Biondi R.M., Proschak E., Zörnig M., Odadzic D. //
Chem. Intermed. 2021. Vol. 47. P. 5027. doi 10.1007/
Bioorg. Med. Chem. 2016. Vol. 24. N 22. P. 5717. doi
s11164-021-04564-x
10.1016/j.bmc.2016.09.015
31.
Radini I.A.M., Ibrahim D.A., Khidre R.E. // Acta Pol.
47.
Yoshida M., Mori A., Inaba A., Oka M., Makino H.,
Pharm. Drug Res. 2019. Vol. 76. N 3. P. 453. doi
Yamaguchi M., Fujita H., Kawamoto T., Goto M.,
10.32383/appdr/102651
Kimura H., Baba A., Yasuma T. // Bioorg. Med.
32.
Bondock S., Khalifa W., Fadda A.A. // Synth.
Chem. 2010. Vol. 18. N 24. P. 8501. doi 10.1016/j.
Commun. 2006. Vol. 36. N 11. P. 1601. doi
bmc.2010.10.035
10.1080/00397910600591763
48.
Abe M., Seto M., Gogliotti R.G., Loch M.T., Bollinger K.A.,
33.
Danagulyan G.G., Mkrtchyan A.D., Panosyan G.A. //
Chang S., Engelberg E.M., Luscombe V.B., Harp J.M.,
Chem. Heterocycl. Compd. 2005. Vol. 41. N 4. P. 485.
Bubser M., Engers D.W., Jones C.K., Rodriguez A.L.,
doi 10.1007/s10593-005-0176-6
Blobaum A.L., Conn P.J., Niswender C.M., Linds-
34.
Metwally N.H., Mohamed M.S., Ragb E.A. // Bioorg.
ley C.W. // ACS Med. Chem. Lett. 2017. Vol. 8. N 10.
Chem. 2019. Vol. 88. Paper N 102929. doi 10.1016/j.
P. 1110. doi 10.1021/acsmedchemlett.7b00317
bioorg.2019.102929
49.
Емелина Е.Е., Петров А.А., Фирсов А.В. // ЖОрХ.
35.
Kolosov M.A., Beloborodov D.A., Orlov V.D., Dotsenko
2003. Т. 39. № 2. С. 299; Emelina E.E., Petrov A.A.,
V.V. // New J. Chem. 2016. Vol. 40. N 9. P. 7573. doi
Firsov A.V. // Russ. J. Org. Chem. 2003. Vol. 39. N 2.
10.1039/c6nj00336b
P. 277. doi 10.1023/A:1025512924673
36.
Семенова А.М., Оганесян Р.В., Доценко В.В., Чи-
50.
Liu J., Zhao J., Lu J.-F. // J. Chem. Soc. Pak. 2020.
горина Е.А., Аксенов Н.А., Аксенова И.В., Нетре-
Vol. 42. N 4. P. 591. doi 10.52568/000663/
ба Е.Е. // ЖОХ. 2019. Т. 89. № 1. С. 25; Semeno-
JCSP/42.04.2020
va A.M., Oganesyan R.V., Dotsenko V.V., Chigori-
51.
Ячевский Д.С., Чижов Д.Л., Чарушин В.Н. // ЖОрХ.
na E.A., Aksenov N.A., Aksenova I.V., Netreba E.E. //
2006. Т. 42. № 1. С. 148; Yachevskii D.S., Chizhov D.L.,
Russ. J. Gen. Chem. 2019. Vol. 89. N 1. P. 19. doi
Charushin, V.N. // Russ. J. Org. Chem. 2006. Vol. 42.
10.1134/S1070363219010043
N 1. P. 142. doi 10.1134/S1070428006010234
37.
Ledenyova I.V., Didenko V.V., Dotsenko V.V., Shikha-
52.
Balicki R. // Pol. J. Chem. 1981. Vol. 55. N. 10. P. 1995.
liev K.S. // Tetrahedron Lett. 2014. Vol. 55. N 6. P. 1239.
53.
Ho Y.-W., Yao C.-T. // J. Chin. Chem. Soc. 2003. Vol. 50.
doi 10.1016/j.tetlet.2014.01.010
N 2. P. 283. doi 10.1002/jccs.200300043
38.
Dolzhenko A.V., Dolzhenko A.V., Chu W.-K. //
54.
Ho Y.-W. // J. Chin. Chem. Soc. 1999. Vol. 46. N 6.
Heterocycles. 2008. Vol. 75. P. 1575. doi 10.3987/REV-
P. 955. doi 10.1002/jccs.199900131
08-629
55.
Емелина Е.Е., Петров А.А., Фирсов А.В. // ЖОрХ.
39.
Singh S., Mandal M.K., Masih A., Saha A., Ghosh S.K.,
2001. Т. 37. № 6. С. 899; Emelina E.E., Petrov A.A.,
Bhat H.R., Singh U.P. // Arch. Pharm. 2021. Vol. 354.
Firsov A.V. // Russ. J. Org. Chem. 2001. Vol. 37. N 6.
N 6. Paper N e2000363. doi 10.1002/ardp.202000363
P. 852. doi 10.1023/A:1012417816375
40.
Wichmann J., Woltering T.J. Pat. WO 2005040171
56.
Neese F. // WIREs Comput. Mol. Sci. 2012. Vol. 2.
(2005).
N 1. P. 73. doi 10.1002/wcms.81
41.
Cohen C. Pat. DE 2900288 (1979). Germany // С. А.
57.
Neese F. // WIREs Comput. Mol. Sci. 2018. Vol. 8. N 1.
1979. Vol. 91. 157770.
Paper N e1327. doi 10.1002/wcms.1327
42.
Liu J., Ge H.-G., Lu J.-F. // J. Chem. Res. 2015. Vol. 39.
58.
Becke A.D. // Phys. Rev. (A). 1988. Vol. 38. P. 3098. doi
N 1. P. 4. doi 10.3184/174751915X14206280982837
10.1103/PhysRevA.38.3098
43.
Aggarwal R., Masan E., Kaushik P., Kaushik D.,
59.
Lee C., Yang W., Parr R.G. // Phys. Rev. (B). 1988.
Sharma C., Aneja K.R. // J. Fluor. Chem. 2014. Vol. 168.
Vol. 37. P. 785. doi 10.1103/PhysRevB.37.785
P. 16. doi 10.1016/j.jfluchem.2014.08.017
60.
Grimme S., Ehrlich S., Goerigk L. // J. Comput. Chem.
44.
Kokorekin V.A., Neverov S.V., Kuzina V.N., Petro-
2011. Vol. 32. N 7. P. 1456. doi 10.1002/jcc.21759
syan V.A. // Molecules. 2020. Vol. 25. N 18. Paper
61.
Weigend F., Ahlrichs R. // Phys. Chem. Chem. Phys.
N 4169. doi 10.3390/molecules25184169
2005. Vol. 7. P. 3297. doi 10.1039/B508541A
45.
Кокорекин В.А., Ходонов В.М., Неверов С.В., Грам-
62.
Allouche A.-R. // J. Comput. Chem. 2011. Vol. 32. N 1.
матикова Н.Э., Петросян В.А. // Изв. АН. Сер. xим.
P. 174. doi 10.1002/jcc.21600
2021. № 3. С. 600; Kokorekin V.A., Khodonov V.M.,
63.
Tomasi J., Mennucci B., Cammi R. // Chem. Rev. 2005.
Neverov S.V., Grammatikova N.É., Petrosyan V.A. //
Vol. 105. N 8. P. 2999. doi 10.1021/cr9904009
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022
НОВЫЕ РЕАКЦИИ ГЕТЕРОЦИКЛИЗАЦИИ
431
64. Lipinski C.A., Lombardo F., Dominy B.W., Feeney P.J. //
Document on Acute oral Toxicity Testing. ENV/JM/
Adv. Drug. Delivery Rev. 1997. Vol. 23. N 1-3. P. 4. doi
10.1016/S0169-409X(96)00423-1
officialdocuments/publicdisplaydocumentpdf/?cote=e
65. Lipinski C.A. // Drug Discov. Today: Technologies. 2004.
nv/jm/mono(2001)4&doclanguage=en
Vol. 1. N 4. P. 337. doi 10.1016/j.ddtec.2004.11.007
73. Yang J., Kwon S., Bae S.H., Park K.M., Yoon C.,
66. Lipinski C.A., Lombardo F., Dominy B.W., Feeney P.J. //
Lee J.H., Seok C. // J. Chem. Inf. Model. 2020. Vol. 60.
Adv. Drug. Delivery Rev. 2012. Vol. 64. Suppl. P. 4. doi
N 6. P. 3246. doi 10.1021/acs.jcim.0c00104
10.1016/j.addr.2012.09.019
74. GalaxyWEB. A web server for protein structure
67. Sander T. OSIRIS Property Explorer. Idorsia
prediction, refinement, and related methods.
chemistry.org/prog/peo/
Computational Biology Lab, Department of Chemistry,
68. Daina A., Michielin O., Zoete V. // Sci. Rep. 2017.
Vol. 7. Article N 42717. doi 10.1038/srep42717
seoklab.org/index.html
69. Cheng F., Li W., Zhou Y., Shen J., Wu Z., Liu G.,
75. Ko J., Park H., Heo L., Seok C. // Nucleic Acids Res.
Lee P.W., Tang Y. // J. Chem. Inf. Model. 2012. Vol. 52.
2012. Vol. 40. Iss. W1. P. W294. doi 10.1093/nar/gks493
N 11. P. 3099. doi 10.1021/ci300367a
76. Pettersen E.F., Goddard T.D., Huang C.C., Couch G.S.,
70. Lagunin A., Zakharov A., Filimonov D., Poroikov V. //
Greenblatt D.M., Meng E.C., Ferrin T.E. // J. Сomput.
Mol. Informatics. 2011. Vol. 30. N 2-3. P. 241. doi
Chem. 2004. Vol. 25. N 13. P. 1605. doi 10.1002/
10.1002/minf.201000151
jcc.20084
71. GUSAR Online. Laboratory for Structure-Function
77. UCSF Chimera. Visualization system for exploratory
Based Drug Design, Institute of Biomedical Chemistry
research and analysis developed by the Resource for
gusar/references.html
Biocomputing, Visualization, and Informatics at the
72. OECD Environment, Health and Safety Publications
University of California, San Francisco, US. https://
Series on Testing and Assessment No 24. Guidance
New Heterocyclisation Reactions of 5-Amino-3-(cyanomethyl)-
1H-pyrazole-4-carbonitrile with Some 1,3-Dielectrophilic Agents
A. M. Semenovaa, Ya. R. Gadzhiakhmedovaa, A. V. Bespalova, V. V. Dotsenkoa,b,*,
N. A. Aksenovb, and I. V. Aksenovab
a Kuban State University, Krasnodar, 350040 Russia
b North Caucasus Federal University, Stavropol, 355009 Russia
*e-mail: victor_dotsenko_@mail.ru
Received December 7, 2021; revised January 27, 2022; accepted January 28, 2022
5-Amino-3-(cyanomethyl)-1H-pyrazole-4-carbonitrile enters into condensation reactions with β-diketones and
dibenzalacetone to form 2-(cyanomethyl)pyrazolo[1,5-a]pyrimidine-3-carbonitrile derivatives. The reaction
with cyanoguanidine in an acid medium leads to the formation of 2,4-diamino-7-(cyanomethyl)pyrazolo[1,5-a]-
[1,3,5]triazine-8-carbonitrile. Structure of the obtained compounds was confirmed by spectral data, as well
as the results of a quantum chemical study of possible reaction routes for the reaction of 5-amino-3-(cyano-
methyl)-1H-pyrazole-4-carbonitrile with benzoyltrifluoroacetone. Bioavailability parameters were predicted for
the obtained products in silico, and possible protein targets were predicted by protein-ligand docking.
Keywords: 3(5)-aminopyrazoles, dicyandiamide, pyrazolo[1,5-a]pyrimidines, calculated biological activity,
quantum chemical studies
ЖУРНАЛ ОБЩЕЙ ХИМИИ том 92 № 3 2022