Журнал аналитической химии, 2019, T. 74, № 4, стр. 243-253
Применение хроматографических и электрофоретических методов в метаболомных исследованиях
Л. А. Карцова 1, *, С. А. Соловьёва 1
1 Санкт-Петербургский государственный университет, Институт химии
198504 Санкт-Петербург, Старый Петергоф, Университетский просп., 26, Россия
* E-mail: kartsova@gmail.com
Поступила в редакцию 10.01.2018
После доработки 24.10.2018
Принята к публикации 24.10.2018
Аннотация
Живой организм представляет собой биохимически уникальную систему, его жизненные функции могут быть исследованы с помощью аналитических измерений. Характеристические хроматографические или электрофоретические профили биологически активных веществ уже более 15 лет являются объектами активных биомедицинских исследований в связи с возможностью экспрессного получения диагностически важной информации. В представленном обзоре рассматриваются целевые и нецелевые метаболомные исследования методами хроматографии и капиллярного электрофореза.
Исследования в области метаболомики направлены на выявление изменений эндогенных метаболитов посредством получения хроматографических или электрофоретических профилей биологически активных соединений с последующей хемометрической обработкой результатов [1–3]. Для мочи по сравнению с другими биологическими жидкостями характерны легкость пробоотбора, многообразие метаболитов и способность отражать дисбалансы биохимических путей в организме. Мочевая метаболомика стала важным инструментом идентификации неинвазивных биомаркеров, которые позволяют обнаруживать метаболические изменения в ответ на конкретное заболевание или терапевтическое вмешательство. Гипотеза о метаболическом профиле как индивидуальной характеристике нашла свое подтверждение в начале 1950-х гг. в работе [4], в которой методом бумажной хроматографии были получены различные метаболические профили образцов мочи здоровых доноров и больных шизофренией. В 1970-х гг. опубликованы работы по различию уровней экскреций сахаров [5] и органических кислот [6], характеристическим профилям образцов крови [7]. Позднее сообщалось [8, 9] о получении газохроматографических профилей летучих органических соединений (ЛОС) в равновесной паровой фазе образцов мочи и выдыхаемом воздухе. Характеристические профили биологических объектов получали также методами ЯМР [10] и капиллярного электрофореза (КЭ) [11].
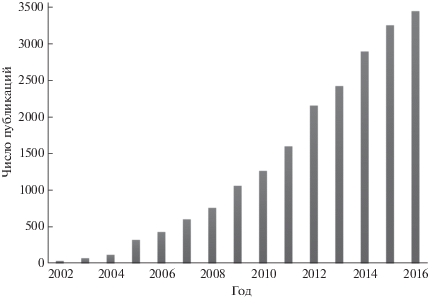
Метаболомика изучает профили низкомолекулярных метаболитов (М ≤ 1.5 кДа) в биологическом объекте, являющихся промежуточными или конечными продуктами обмена веществ [12–14]. Сам термин “метаболомика” введен в 1998 г. [15], и с каждым годом интерес к этой науке непрерывно растет (рис. 1).
Метаболомные исследования активно используют в биомедицине. Наряду с геномикой, протеомикой, липидомикой и др., метаболомика позволяет расширить возможности персонализированной медицины [16, 17], целью которой является прогнозирование индивидуальной восприимчивости к конкретному заболеванию, его протеканию и лечению. От особенностей организма зависит эффективность и безопасность терапевтического лечения, поэтому для подбора правильной дозировки лекарственных средств проводят мониторинг действующих веществ и их метаболитов в биологических объектах [18].
Одна из сфер приложения метаболомики – поиск специфических биомаркеров различных заболеваний. Наличие или отсутствие конкретных аналитов, изменение их соотношения позволяют выявить конкретную патологию на ранней стадии заболевания [19] или диагностировать степень его развития [20–22]. Опубликованы обзоры, посвященные поиску биомаркеров по характеристическим профилям биологически активных веществ в онкологии [23], гастроэнтерологии [24], нефрологии [25, 26], репродуктологии [27]; при нейродегенеративных [28], аллергических [29] и ревматических [30] заболеваниях и патологиях сердечно-сосудистой системы [31], диабете [32], туберкулезе [33], атеросклерозе [34] и др.
Не менее важным является применение метаболомики в нутрициологии (научная дисциплина по различным аспектам питания) с целью идентификации компонентов пищевых продуктов [35, 36] в биологических объектах, исследования их физиологической активности [37, 38] и путей метаболизма [39–41], оценки изменений в метаболоме [42–44] и рисков возникновения определенных заболеваний [45, 46] при употреблении конкретных пищевых продуктов. Метаболомика интенсивно применяется и в токсикологии при исследовании пагубного влияния тяжелых металлов [47], полициклических ароматических углеводородов [48], N-нитрозоаминов [49] и др. на живые организмы.
Различают целевые [14] и нецелевые [15] метаболомные исследования. В целевых исследованиях выявляют конкретные группы эндогенных метаболитов в различных образцах. Так, описано [50–56] различие в балансе стероидных гормонов (по сравнению с “нормой”) при синдроме Иценко−Кушинга, гиперплазии коры надпочечников, синдроме поликистозных яичников; незаменимых аминокислот – при колоректальном раке, раке легких и молочных желез и др. В нецелевой метаболомике (или метаболическом профилировании) получают характеристический профиль с максимально возможным количеством метаболитов и выделяют среди них те, которые доминируют в исследуемых физиологических процессах [57]. Например, установлены различия в уровнях глутатиона и мочевой кислоты при болезни Паркинсона [58]; нуклеозидов, карнитина, дипептидов при колоректальной опухоли и карциноме [59]; аминокислот, сахаров, спиртов при болезни Баттена [60] и др.
Эксперимент в метаболомике (рис. 2) включает отбор пробы биологического образца и его пробоподготовку, хроматографический и/или электрофоретический анализ образцов с получением характеристических профилей, хемометрическую обработку результатов с последующим выявлением потенциальных биомаркеров, идентификацию биомаркеров с помощью баз данных метаболома человека (Human Metabolome Database [61], METLIN Metabolomics Database [62] и др.) и анализ метаболического пути конкретного биологически активного соединения для уточнения физиологии заболевания.
Наиболее распространенными биологическими объектами в метаболомике являются плазма [54, 63–65] и сыворотка [41, 51, 53, 66–68] крови, моча [43, 69–71], что в существенной степени объясняется их доступностью и возможностью получения большого объема информации о состоянии организма. Со стремлением к неинвазивным методам диагностики вырос интерес к анализу и других объектов: выдыхаемому воздуху [72, 73], слюне [74, 75], поту [76], слезной жидкости [77, 78]. Опубликованы работы по метаболомному исследованию спинномозговой жидкости [79], экстрактов гомогенатов тканей [80, 81].
Стратегия пробоподготовки биологических объектов в целевом и нецелевом вариантах существенно различается. В случае целевого метаболического исследования, где известны физико-химические характеристики аналитов, процедура подготовки пробы к анализу должна быть селективной и воспроизводимой, а степень извлечения аналитов – максимальной. Так, в работе [69] предложен способ определения саркозина, аланина, лейцина и пролина – биомаркеров рака простаты в моче – методом дисперсионной жидкостно–жидкостной микроэкстракции с изобутилхлорформиатом в качестве дериватизирующего агента с последующим хроматографическим разделением образующихся производных. Напротив, в нецелевом варианте пробоподготовка должна быть неселективной и обеспечивать очистку пробы от мешающих компонентов с сохранением максимально возможного исходного состава метаболома, чтобы обнаружить диагностические маркеры [82]. Простым и экспрессным способом подготовки образцов крови и мочи к метаболическому профилированию является осаждение белков органическими растворителями [83–85] или ультрафильтрация [86]. В работе [87] отмечено, что фосфолипиды и другие эндогенные компоненты могут значительно влиять на разделение аналитов и подавлять ионизацию при масс-спектрометрическом детектировании, поэтому предложено после депротеинизации образца проводить твердофазную экстракцию, выделяя 3 фракции: фосфолипиды, липиды и полярные компоненты, в результате чего возможное число определяемых метаболитов увеличивается более чем в 2 раза.
На метаболический состав биологических объектов влияет процедура отбора и хранения. Например, метаболиты образцов мочи подвергаются бактериальному воздействию [88–90], причем наибольшее изменение происходит при комнатной температуре в течение 12 ч [89]. Если необходимо длительное хранение образцов, рекомендуется температура ниже –20°С [88]. Эффективно также добавление консервантов для предотвращения роста бактерий [89] и краткосрочное хранение образцов при +4°С [89, 90]. На метаболом плазмы крови влияет природа антикоагулянта [90]. В отличие от образцов мочи, хранение плазмы крови при комнатной температуре в течение 16 ч приводит к незначительным потерям в метаболическом профиле [91]. Как и для других биологических жидкостей, для длительного хранения образцов плазмы крови требуются температуры ниже –20°С [92, 93], при этом на метаболом влияет и количество циклов заморозки–разморозки [92].
Получение характеристических профилей биологических объектов, содержащих большое количество метаболитов с различными физико-химическими свойствами и широким диапазоном концентраций, представляет собой серьезную аналитическую задачу. Традиционными методами для метаболомных исследований является спектроскопия ЯМР [77, 94], масс-спектрометрия (МС) [95, 96]. В качестве гибридных методов анализа отдают предпочтение газовой (ГХ–МС) [97–100] и жидкостной (ВЭЖХ–МС) [51, 59, 64, 65, 82, 101] хромато-масс-спектрометрии, а также методу КЭ с МС-детектированием [66, 67, 75], что обусловлено возможностью идентификации доминирующих диагностических маркеров и количественного определения концентраций метаболитов в пробе.
ПОЛУЧЕНИЕ ХРОМАТОГРАФИЧЕСКИХ И ЭЛЕКТРОФОРЕТИЧЕСКИХ ПРОФИЛЕЙ БИОЛОГИЧЕСКИХ ОБРАЗЦОВ ДЛЯ ДИАГНОСТИКИ РАЗЛИЧНЫХ ЗАБОЛЕВАНИЙ
Обращенно-фазовую (ОФ) ВЭЖХ успешно применяют как в целевом, так и в нецелевом метаболомных исследованиях. Например, для диагностики шизофрении методом ОФ ВЭЖХ–МС получены характеристические профили 47 свободных жирных кислот в образцах сыворотки крови [68]. Известно, что оптические изомеры эндогенных метаболитов обладают различной биологической активностью и могут одновременно служить диагностическими маркерами ряда заболеваний [102]. В работе [103] для обнаружения методом ОФ ВЭЖХ–МС/МС энантиомеров карбоновых кислот в слюне разделяли их диастереомерные производные. Найдено, что концентрации D-молочной кислоты и соотношение D,L-стереоизомеров существенно выше в образцах больных сахарным диабетом. В работе [104] методом двумерной (2D) ВЭЖХ определяли в плазме крови D-энантиомеры аминокислот в виде флуоресцирующих производных. При этом аналиты сначала разделяли на обращенно-фазовом сорбенте, а затем на хиральной стационарной фазе. У пациентов с хронической болезнью почек обнаружено в плазме крови 16 D-аминокислот. Установлено, что концентрации D-серина, D-пролина и D-аспарагина связаны с функцией почек; D-аланина и D-пролина – с возрастом; D-аспарагиновой кислоты и D-пролина – с наличием сахарного диабета; D-серина и D-аспарагина – с прогрессированием хронической болезни почек.
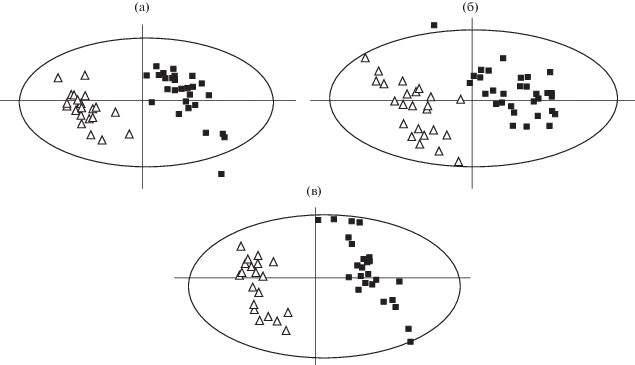
Альтернативой ОФ ВЭЖХ для разделения высокополярных и ионных метаболитов является гидрофильная хроматография (Hydrophilic Interaction Liquid Chromatography, HILIC). В работе [65] получены метаболические профили образцов плазмы крови и мочи методом HILIC–MC с целью поиска маркеров хронической болезни почек на разных стадиях. Метод HILIC используют для определения эндогенных метаболитов и в сочетании с ОФ ВЭЖХ. Так, в работе [101] методы ОФ ВЭЖХ–МС и HILIC–МС использовали для выявления биомаркеров рака молочной железы в слюне. Показано, что одновременная математическая обработка результатов двух методов позволяет достичь более четкого разделения кластеров норма/патология (рис. 3).
Рис. 3.
Графики счетов, полученные методом дискриминантного анализа с регрессией на латентные структуры, образцов слюны здоровых людей (△) и людей с патологией рака молочной желез (◼) по характеристическим профилям, полученным методами ОФ ВЭЖХ–МС (а), HILIC–МС (б) и при объединении результатов двух методов (в) [101].
Этот же подход реализован для метаболического профилирования водного (для полярных компонентов) и органического (для липидов) экстрактов гомогенатов образцов тканей почек с целью выявления карциномы почек [105]. В работе [64] для оптимизации условия получения более полного метаболического профиля образцов мочи и плазмы крови варьировали 5 типов стационарных фаз в условиях ОФ ВЭЖХ и HILIC, что существенно увеличило число обнаруженных метаболитов по сравнению с применением только ОФ ВЭЖХ.
Режим двумерной ВЭЖХ позволяет одновременно определять эндогенные метаболиты в широком диапазоне их полярностей. Так, методом 2D-ВЭЖХ–МС/МС с использованием двух различных ОФ сорбентов идентифицировали 28 модифицированных нуклеозидов – продуктов метаболизма РНК, являющихся диагностических маркерами рака молочной железы на ранней стадии [106]. Сочетание HILIC и ОФ ВЭЖХ обеспечивает более высокую ортогональность, благодаря чему в моче удалось одновременно определить более 570 полярных и 260 неполярных метаболитов, 11 из которых позволили четко различить образцы с раком легких и без данной патологии [107]. В [108] предложена схема комплексного подхода к анализу плазмы крови, включая определение метаболитов и липидов, путем сочетания ОФ ВЭЖХ и HILIC. В результате обнаружены аминокислоты, карнитины, желчные кислоты, свободные жирные кислоты, фосфолипиды, лизофосфолипиды и сфингомиелины. Общая продолжительность анализа составило 30 мин.
Газохроматографические метаболомные исследования нередко требуют предварительной дериватизации компонентов биологического образца с применением реакций ацилирования [97–99] или силилирования. Разработан [98] автоматизированный способ определения в моче потенциальных биомаркеров раковых опухолей (диаминовалериановой и γ-аминомасляной кислот, путресцина, кадаверина, спермина), основанный на in situ дериватизации этилхлорформиатом, выделении производных твердофазной микроэкстракцией (ТФМЭ) и определении методом ГХ–МС. Обнаружено значительное различие в содержаниях путресцина и диаминовалериановой кислоты в образцах мочи здоровых добровольцев и больных с меланомой, раком печени и легких. Предложен [99] способ определения стероидов и токоферолов в плазме крови и амниотической жидкости с помощью реакций трифторалкилхлорформиатов со вторичными ациклическими гидроксильными функциональными группами в их структурах. В работе [97] при исследовании образцов мочи получен профиль метаболитов с амино- и карбоксильными функциональными группами, в качестве реагента дериватизации использовали 1,1,1,2,2,3,3-гептафторбутилхлорформиат. Образующиеся слабополярные производные путем жидкостно–жидкостной микроэкстракции переводили в октановую фазу и определяли методом ГХ–МС. В нецелевой метаболомике чаще используют реакцию силилирования N,O-бис(триметилсилил)трифторацетамидом или его N-метилпроизводным [100, 109]. Отмечено [97], что данная реакция эффективнее для определения методом ГХ–МС О-силилированных производных, что позволяет получать метаболические профили биологических объектов практически без потерь компонентов. Например, данный подход применен [110] к ГХ–МС-профилированию сыворотки крови с целью выявления видов рака желудочно-кишечного тракта. Обнаружены такие доминирующие диагностические маркеры, как малоновая кислота и L-серин в случае рака пищевода, 3-гидроксиприпионовая и пировиноградная кислоты – рака желудка и L-аланин и L-глутамин – колоректального рака. Предложена [111] автоматизированная процедура силилирования метаболитов плазмы крови для метаболического профилирования методом ГХ–МС, что привело к значительному сокращению продолжительности анализа и лучшей воспроизводимости результатов.
Метод ГХ–МС востребован при определении ЛОС. Так, анализ выдыхаемого воздуха с предварительным концентрированием ЛОС сорбционными трубками [112, 113] или ТФМЭ [114] позволяет проводить неинвазивную диагностику различных патологий. По компонентам, обнаруженным в выдыхаемом воздухе, оказалось возможным отличить пациентов с раком легких от здоровых доноров и больных с другими патологиями легких, включая астму и хроническое обструктивное заболевание легких [114]. Обнаружены такие диагностические маркеры, как диметилсульфид, 1,4-пентадиен, этилацетат, метилциклопентан, 2-пропанол, изобутан и 2,4-диметилгептан, по которым можно выделить группы норма/патология. Получены характеристические профили альдегидов в выдыхаемом воздухе для диагностики рака легких [115]; чувствительность и специфичность этого метода сопоставимы с данными компьютерной томографии. В работе [116] в качестве биомаркеров рака легких предложены 23 ЛОС и разработана диагностическая модель, способная отличить больного или здорового человека с высокими показателями чувствительности (96%) и специфичности (97%).
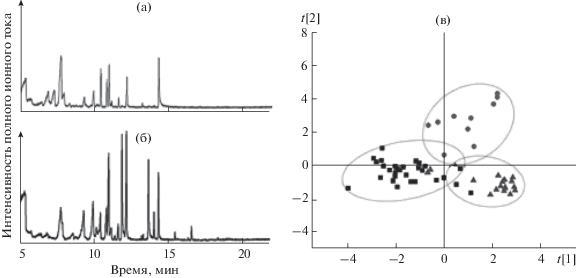
Летучие органические соединения над конденсированной фазой мочи также являются важнейшими специфическими биомаркерами. Для диагностики рака простаты ЛОС над образцами мочи извлекали ТФМЭ и определяли методом ГХ–МС [70]. Выявлены 4 биомаркера: 2,6-диметил-7-октен-2-ол, пентаналь, 3-октанон и 2-октанон; точность диагностического критерия составила 63–64%. При совместном учете указанных четырех биомаркеров и простат-специфического антигена точность повышена до 74%. Выявленные при парофазном анализе ЛОС мочи позволили идентифицировать образцы больных с карциномой почек [117]. Отмечено, что значение pH биологической жидкости сильно влияет на эффективность ТФМЭ, более представительные метаболические профили получены в кислой среде. В работе [118] диагностировали туберкулез легких по характеристическим газохроматографическим профилям ЛОС с предварительной ТФМЭ над образцами мочи (рис. 4). Авторы обнаружили значительное увеличение содержания о-ксилола, изопропилацетата и снижение уровней пентанола-3, диметилстирола и цимола для образцов мочи туберкулезных больных по сравнению со здоровыми донорами. Важно отметить, что по этим диагностическим маркерам возможно также отличить туберкулез легких от хронического обструктивного заболевания и рака легких.
Рис. 4.
Хроматографические профили летучих компонентов равновесной паровой фазы образцов мочи здоровых доноров (а) и больных туберкулезом (б) и график счетов (метод дискриминантного анализа с регрессией на латентные структуры) характеристических профилей образцов мочи здоровых людей с отрицательной (⚫) и положительной (▲) туберкулиновой пробой и больных туберкулезом (◼) (в) [117].
Электрофоретические профили биологически активных веществ широко используют в метаболомике, особенно для определения полярных метаболитов биологических объектов (сахаров, аминокислот, фосфорилированных метаболитов, нуклеотидов, нуклеозидов и др.). Сравнительный анализ показал [119], что метод КЭ–МС эффективно дополняет другие аналитические платформы при обнаружении полярных и ионогенных метаболитов, которые не могут быть детектированы с помощью методов ГХ–МС, ВЭЖХ–МС, ЯМР или прямой МС.
В большинстве публикаций, касающихся поиска диагностических маркеров различных патологий методом КЭ–МС, используют сочетания капиллярного зонного электрофореза при низком и высоком pH фонового электролита для исследования положительно и отрицательно заряженных метаболитов. Так, данный подход реализован при метаболическом профилировании сыворотки крови для выявления заболеваний печени [66]. Обнаружены три метаболита (триптофан, глутамин, 2-гидроксимасляная кислота), по которым можно отличить здорового от больного гепатоцеллюлярной карциномой или циррозом. Получены [81] электрофоретические профили аминокислот в образцах экстрактов гомогенатов тканей с колоректальной карциномой и аденомой. Установлено, что данные патологии можно различить по содержанию метионина, тирозина, валина и изолейцина. Проведено метаболомное исследование образцов сыворотки крови и слюны с целью поиска диагностических маркеров лобно-височной деменции, болезни Альцгеймера и деменции с тельцами Леви [67]. Уровни 6 метаболитов в сыворотке крови (β-аланин, креатинин, гидроксипролин, глутамин, изоцитрат и цитидин) и 2 в слюне (аргинин и тирозин) значительно различались у здоровых и больных данными нейродегенеративными заболеваниями. Методом многомерного анализа показано, что сыворотка крови является более представительным биологическим объектом, чем слюна для диагностики данных патологий. Осуществлен поиск специфичных биомаркеров сыворотки крови для отличия больных гриппом и больных энцефалопатией, провоцированной гриппом [120]. В течение острой фазы заболевания энцефалопатией уровни трех метаболитов (янтарная кислота, ундекановая кислота и кинуренин) оказались значительно выше, а двух других метаболитов (декановая кислота и цистин) – ниже по сравнению с группой больных гриппом. В работе [75] в электрофоретических профилях образцов слюны больных раком полости рта, груди и поджелудочной железы обнаружено 57 метаболитов, по которым можно надежно отличить каждое из этих заболеваний от образцов здоровых добровольцев.
* * *
Таким образом, несмотря на значительный прогресс, достигнутый в клинической диагностике, интенсивный поиск дополнительных биомаркеров различных заболеваний по-прежнему остается крайне актуальным. Одновременное определение сотен эндогенных метаболитов представляет собой серьезную проблему. Ценным инструментом для обнаружения биомаркеров как на ранних стадиях заболевания, так и на более поздних являются целевая и нецелевая метаболомика. Целевой анализ наиболее часто проводят методом ВЭЖХ–МС с использованием режима “мониторинга заданных реакций”. Однако применение этой стратегии ограничено относительно низким содержанием метаболитов. Разработаны новые подходы для расширения возможностей целевого исследования метаболитов на основе таких технологий, как мониторинг параллельных реакций, обеспечивающий высокую чувствительность и избирательность при определении десятков и сотен аналитов в сложных матрицах [121–123]. Метаболическое профилирование (нецелевая метаболомика) обеспечивает получение характеристических профилей с определением доминирующих в исследуемых физиологических процессах биомаркеров.
Список литературы
Sniehotta M., Schiffer E., Zurbig P., Novak J., Mischak H. CE – a multifunctional application for clinical diagnosis // Electrophoresis. 2007. V. 28. № 9. P. 1407.
Карцова Л.А., Объедкова Е.В. Хроматографические и электрофоретические профили биологически активных соединений для диагностики различных заболеваний // Журн. аналит. химии. 2013. Т. 68. № 4. С. 316. (Kartsova L.A., Obedkova E.V. Chromatographic and electrophoretic profiles of biologically active compounds for the diagnosis of various diseases // J. Analyt. Chem. 2013. V. 68. № 4. P. 291.)
Kartsova L.A., Bessonova E.A. Biomedical applications of capillary electrophoresis // Russ. Chem. Rev. 2015. V. 84. № 8. P. 860.
Williams R.J. Biochemical Institute Studies IV. Individual Metabolic Patterns and Human Disease: An Exploratory Study Utilizing Predominantly Paper Chromatographic Methods. Austin: The University of Texas Press, 1951. P. 7.
Young D.S. High pressure column chromatography of carbohydrates in the clinical laboratory // Am. J. Clin. Pathol. 1970. V. 53. P. 803.
Burtis C.A., Goldstein G., Scott C.D. Fractionation of human urine by gel chromatography // Clin. Chem. 1970. V. 16. № 3. P. 201.
Williams G.Z., Young D.S., Stein M.R., Cotlove E. Biological and analytic components of variation in long-term studies of serum constituents in normal subjects. I. Objectives, subject selection, laboratory procedures, and estimation of analytic deviation // Clin. Chem. 1970. V.16. № 12. P. 1016.
Pauling L., Robinson A.B., Teranishi R., Cary P. Quantitative analysis of urine vapor and breath by gas-liquid partition chromatography // Proc. Natl. Acad. Sci. U.S.A. 1971. V. 68. № 10. P. 2374.
Horning E.C., Horning M.G. Metabolic profiles: gas-phase methods for analysis of metabolites // Clin. Chem. 1971. V. 17. № 8. P. 802.
Hoult D.I., Busby S.J., Gadian D.G., Radda G.K., Richards R.E., Seeley P.J. Observation of tissue metabolites using 31P nuclear magnetic resonance // Nature. 1974. V. 252. № 5481. P. 285.
Dolnik V., Dolnikov J. Capillary zone electrophoresis of organic acids in serum of critically ill children // J. Chromatogr. A. 1995. V. 716. № 1–2. P. 269.
Johnson C.H., Ivanisevic J., Siuzdak G. Metabolomics: beyond biomarkers and towards mechanisms // Nat. Rev. Mol. Cell Biol. 2016. V. 17. № 7. P. 451.
Griffiths W.J., Koal T., Wang Y., Kohl M., Enot D.P., Deigner H.P. Targeted metabolomics for biomarker discovery // Angew. Chem. Int. Ed. Engl. 2010. V. 49. № 32. P. 5426.
Alonso A., Marsal S., Julià A. Analytical methods in untargeted metabolomics: State of the art in 2015 // Front. Bioeng. Biotechnol. 2015. V. 3. P. 23.
Oliver S.G., Winson M.K., Kell D.B., Baganz F. Systematic functional analysis of the yeast genome // Trends Biotechnol. 1998. V. 16. № 9. P. 373.
Wishart D.S. Emerging applications of metabolomics in drug discovery and precision medicine // Nat. Rev. Drug Discov. 2016. V. 15. № 7. P. 473.
Дедов И.И., Тюльпаков А.Н., Чехонин В.П., Баклаушев В.П., Арчаков А.И., Мошковский С.А. Персонализированная медицина: современное состояние и перспективы // Вестн. РАМН. 2012. Т. 67. № 12. С. 4.
Kim H.-J., Seo K.-A., Kim H.-M., Jeong E.-S., Ghim J.L., Lee S.H., Lee Y.M., Kim D.H., Shin J.-G. Simple and accurate quantitative analysis of 20 anti-tuberculosis drugs in human plasma using liquid chromatography–electrospray ionization–tandem mass spectrometry // J. Pharm. Biomed. Anal. 2015. V. 102. P. 9.
Gowda G.N., Zhang S., Gu H., Asiago V., Shanaiah N., Raftery D. Metabolomics-based methods for early disease diagnostics // Expert Rev. Mol. Diagn. 2008. V. 8. № 5. P. 617.
Kobayashi T., Yoshida T., Fujisawa T., Matsumura Y., Ozawa T., Yanai H., Iwasawa A., Kamachi T., Fujiwara K., Kohno M., Tanaka N. A Metabolomics-based approach for predicting stages of chronic kidney disease // Biochem. Biophys. Res. Commun. 2014. V. 445. № 2. P. 412.
Wang W., Feng B., Li X., Yin P., Gao P., Zhao X., Lu X., Zheng M., Xu G. Urinary metabolic profiling of colorectal carcinoma based on online affinity solid phase extraction-high performance liquid chromatography and ultra performance liquid chromatography-mass spectrometry // Mol. Biosyst. 2010. V. 6. № 10. P. 1947.
Shah V.O., Townsend R.R., Feldman H.I., Pappan K.L., Kensicki E., Vander Jagt D.L. Plasma metabolomic profiles in different stages of CKD // Clin. J. Am. Soc. Nephrol. 2013. V. 8. № 3. P. 363.
Armitage E.G., Barbas C. Metabolomics in cancer biomarker discovery: Current trends and future perspectives // J. Pharm. Biomed. Anal. 2014. V. 87. P. 1.
Preter V.D., Verbeke K. Metabolomics as a diagnostic tool in gastroenterology // World J. Gastrointest. Pharmacol. Ther. 2013. V. 4. № 4. P. 97.
Weiss R.H., Kim K. Metabolomics in the study of kidney diseases // Nat. Rev. Nephrol. 2011. V. 8. № 1. P. 22.
Rhee E.P. Metabolomics and renal disease // Curr. Opin. Nephrol. Hypertens. 2015. V. 24. № 4. P. 371.
Egea R.R., Puchalt N.G., Escriva M.M., Varghese A.C. OMICS: Current and future perspectives in reproductive medicine and technology // J. Hum. Reprod. Sci. 2014. V. 7. № 2. P. 73.
Botas A., Campbell H.M., Han X., Maletic-Savatic M. Metabolomics of neurodegenerative Diseases // Int. Rev. Neurobiol. 2015. V. 122. P. 53.
Scrivo R., Casadei L., Valerio M., Priori R., Valesini G., Manetti C. Metabolomics approach in allergic and rheumatic diseases // Curr. Allergy Asthma Rep. 2014. V. 14. № 6. P. 445.
Guma M., Tiziani S., Firestein G.S. Metabolomics in rheumatic diseases: Desperately seeking biomarkers // Nat. Rev. Rheumatol. 2016. V. 12. № 5. P. 269.
Kordalewska M., Markuszewski M.J. Metabolomics in cardiovascular diseases // J. Pharm. Biomed. Anal. 2015. V. 113. P. 121.
Sas K.M., Karnovsky A., Michailidis G., Pennathur S. Metabolomics and diabetes: Analytical and computational approaches diabetes // Diabetes. 2015. V. 64. № 3. P. 718.
Preez I.D., Luies L., Loots D.T. Metabolomics biomarkers for tuberculosis diagnostics: Current status and future objectives // Biomark. Med. 2017. V. 11. №. 2. P. 179.
Goonewardena S.N., Prevette L.E., Desai A.A. Metabolomics and atherosclerosis // Curr. Atheroscler. Rep. 2010. V. 12. № 4. P. 267.
Urpi-Sarda M., Boto-Ordóñez M., Queipo-Ortuño M.I., Tulipani S., Corella D., Estruch R., Tinahones F.J., Andres-Lacueva C. Phenolic and microbial-targeted metabolomics to discovering and evaluating wine intake biomarkers in human urine and plasma // Electrophoresis. 2015. V. 36. № 36. P. 2259.
Pujos-Guillot E., Hubert J., Martin J.F., Lyan B., Quintana M., Claude S., Chabanas B., Rothwell J.A., Bennetau-Pelissero C., Scalbert A., Comte B., Hercberg S., Morand C., Galan P., Manach C. Mass spectrometry-based metabolomics for the discovery of biomarkers of fruit and vegetable intake: citrus fruit as a case study // J. Proteome Res. 2013. V. 12. № 4. P. 1645.
Wang M., Chen L., Liu D., Chen H., Tang D., Zhao Y. Metabolomics highlights pharmacological bioactivity and biochemical mechanism of traditional Chinese medicine // Chem. Biol. Interact. 2017. № 273. V. 273. P. 133.
Paudel L., Wyzgoski F.J., Giusti M.M., Johnson J.L., Rinaldi P.L., Scheerens J.C., Chanon A.M., Bomser J.A., Miller A.R., Hardy J.K., Reese R.N. NMR-Based metabolomic investigation of bioactivity of chemical constituents in black raspberry (Rubus occidentalis L.) fruit extracts // J. Agric. Food Chem. 2014. V. 62. № 8. P. 1989.
Chen H.H., Tseng Y.J., Wang S.Y., Tsai Y.S., Chang C.S., Kuo T.C., Yao W.J., Shieh C.C., Wu C.H., Kuo P.H. The metabolome profiling and pathway analysis in metabolic healthy and abnormal obesity // Int. J. Obes. 2015. V. 39. № 8. P. 1241.
Chang A.Y., Lalia A.Z., Jenkins G.D., Dutta T., Carter R.E., Singh R.J., Nair K.S. Combining a nontargeted and targeted metabolomics approach to identify metabolic pathways significantly altered in polycystic ovary syndrome // Metabolism. 2017. V. 71. P. 52.
Chen T., He P., Tan Y., Xu D. Biomarker identification and pathway analysis of preeclampsia based on serum metabolomics // Biochem. Biophys. Res. Commun. 2017. V. 485. № 1. P. 119.
Medina S., Ferreres F., García-Viguera C., Horcajada M.N., Orduna J., Savirón M., Zurek G., Martínez-Sanz J.M., Gil J.I., Gil-Izquierdo A. Non-targeted metabolomic approach reveals urinary metabolites linked to steroid biosynthesis pathway after ingestion of citrus juice // Food Chem. 2013. V. 136. № 2. P. 938.
Rasmussen L.G., Winning H., Savorani F., Toft H., Larsen T.M., Dragsted L.O., Astrup A., Engelsen S.B. Assessment of the effect of high or low protein diet on the human urine metabolome as measured by NMR // Nutrients. 2012. V. 4. № 2. P. 112.
Jacobs D.M., Fuhrmann J.C., Dorsten F.A., Rein D., Peters S., Velzen E.J.J., Hollebrands B., Draijer R., Duynhoven J., Garczarek U. Impact of short-term intake of red wine and grape polyphenol extract on the human metabolome // J. Agric. Food Chem. 2012. V. 60. № 12. P. 3078.
Guertin K., Loftfield E., Boca S., Sampson J., Moore S., Xiao Q., Huang W., Xiong X., Freedman N., Cross A., Sinha R. Serum biomarkers of habitual coffee consumption may provide insight into the mechanism underlying the association between coffee consumption and colorectal cancer // Am. J. Clin. Nutr. 2015. V. 101. № 5. P. 1000.
Wittenbecher C., Muhlenbruch K., Kroger J., Jacobs S., Kuxhaus O., Floegel A., Fritsche A., Pischon T., Prehn C., Adamski J., Joost H., Boeing H., Schulze M.B. Amino acids, lipid metabolites, and ferritin as potential mediators linking red meat consumption to type 2 diabetes // Am. J. Clin. Nutr. 2015. V. 101. № 6. P.1241.
Gao Y., Lu Y., Huang S., Gao L., Liang X., Wu Y., Wang J., Huang Q., Tang L., Wang G., Yang F., Hu S., Chen Z., Wang P., Jiang Q., Huang R., Xu Y., Yang X., Ong C.N. Identifying early urinary metabolic changes with long-term environmental exposure to cadmium by mass spectrometry-based metabolomics // Environ. Sci. Technol. 2014. V. 48. № 11. P. 6409.
Wang Z., Zheng Y., Zhao B., Zhang Y., Liu Z., Xu J., Chen Y., Yang Z., Wang F., Wang H., He J., Zhang R., Abliz Z. Human metabolic responses to chronic environmental polycyclic aromatic hydrocarbon exposure by a metabolomic approach // J. Proteome Res. 2015. V. 14. № 6. P. 2583.
Hecht S.S., Stepanov I., Carmella S.G. Exposure and metabolic activation biomarkers of carcinogenic tobacco-specific nitrosamines // Acc. Chem. Res. 2016. V. 49. № 1. P. 106.
Объедкова Е.В., Карцова Л.А., Киранов Д.О., Великанова Л.И., Легин А.В. Получение характерных профилей стероидных гормонов методом обращенно-фазовой ВЭЖХ // Журн. аналит. химии. 2014. Т. 69. № 2. С. 214. (Obedkova E.V., Kartsova L.A., Kirsanov D.O., Velikanova L.I., and Legin A.V. Generation of characteristic profiles of steroid hormones by reversed-phase HPLC // J. Analyt. Chem. 2014. V. 69. № 2. С. 200.)
Rossi C., Calton L., Hammond G., Brown H.A., Wallace A.M., Sacchetta P., Morris M. Serum steroid profiling for congenital adrenal hyperplasia using liquid chromatography–tandem mass spectrometry // Clin. Chim. Acta. 2010. V. 411. № 3–4. P. 222.
Handelsman D.J., Teede H.J., Desai R., Norman R.J., Moran L.J. Performance of mass spectrometry steroid profiling for diagnosis of polycystic ovary syndrome // Hum. Reprod. 2017. V. 32. № 2. P. 418.
Leichtle A.B., Nuoffer J., Ceglarek U., Kase J., Conrad T., Witzigmann H., Thiery J., Fiedler G.M. Serum amino acid profiles and their alterations in colorectal cancer // Metabolomics. 2012. V. 8. № 4. P. 643.
Zhao Q., Cao Y., Wang Y., Hu C., Hu A., Ruan L., Bo Q., Liu Q., Chen W., Tao F., Ren M., Ge Y., Chen A., Li L. Plasma and tissue free amino acid profiles and their concentration correlation in patients with lung cancer // Asia Pac. J. Clin. Nutr. 2014. V. 23. № 3. P. 429.
Kim H.J., Jang S.H., Ryu J.S., Lee J.E., Kim Y.C., Lee M.K., Jang T.W., Lee S.Y., Nakamura H., Nishikata N., Mori M., Noguchi Y., Miyano H., Lee K.Y. The performance of a novel amino acid multivariate index for detecting lung cancer: a case control study in Korea // Lung Cancer. 2015. V. 90. № 3. P. 522.
Gu Y., Chen T., Fu S., Sun X., Wang L., Lu Y., Ding S., Ruan G., Teng L., Wang M. Perioperative dynamics and significance of amino acid profiles in patients with cancer // J. Transl. Med. 2015. V. 13. № 35. P. 1.
Трифонова O.П., Лохов П.Г., Арчаков А.И. Метаболомное профилирование крови // Биомед. химия. 2014. Т. 60. № 3. С. 281.
Bogdanov M., Matson W.R., Wang L., Matson T., Saunders-Pullman R., Bressman S.S., Flint Beal M. Metabolomic profiling to develop blood biomarkers for Parkinson’s disease // Brain. 2008. V. 131. № 2. P. 389.
Wang W., Feng B., Li X., Yin P., Gao P., Zhao X., Lu X., Zheng M., Xu G. Urinary metabolic profiling of colorectal carcinoma based on online affinity solid phase extraction-high performance liquid chromatography and ultra-performance liquid chromatography-mass spectrometry // Mol. Biosyst. 2010. V. 6. № 10. P. 1947.
Pears M.R., Cooper J.D., Mitchison H.M., Mortishire-Smith R.J., Pearce D.A., Griffin J.L. High resolution 1H NMR-based metabolomics indicates a neurotransmitter cycling deficit in cerebral tissue from a mouse model of Batten disease // J. Biol. Chem. 2005. V. 280. № 52. P. 42508.
Wishart D.S., Knox C., Guo A.C., Eisner R., Young N., Gautam B., Hau D.D., Psychogios N., Dong E., Bouatra S., Mandal R., Sinelnikov I., Xia J., Jia L., Cruz J.A., Lim E., Sobsey C.A., Shrivastava S., Huang P., Liu P., Fang L., Peng J., Fradette R., Cheng D., Tzur D., Clements M., Lewis A., De Souza A., Zuniga A., Dawe M., Xiong Y., Clive D., Greiner R., Nazyrova A., Shaykhutdinov R., Li L., Vogel H.J. Forsythe I. HMDB: a knowledgebase for the human metabolome // Nucleic Acids Res. 2009. V. 37.
Smith C.A., O’Maille G., Want E.J., Qin C., Trauger S.A., Brandon T.R., Custodio D.E., Abagyan R., Siuzdak G. METLIN: a metabolite mass spectral database // Ther. Drug Monit. 2005. V. 27. № 6. P. 747.
Lau S.K., Lee K.C., Curreem S.O., Chow W.N., To K.K., Hung I.F., Ho D.T., Sridhar S., Li I.W., Ding V.S., Koo E.W., Wong C.F., Tam S., Lam C.W., Yuen K.Y., Woo P.C. Metabolomic profiling of plasma from patients with tuberculosis by use of untargeted mass spectrometry reveals novel biomarkers for diagnosis // J. Clin. Microbiol. 2015. V. 53. № 12. P. 3750.
Contrepois K., Jiang L., Snyder M. Optimized analytical procedures for the untargeted metabolomic profiling of human urine and plasma by combining hydrophilic interaction and reverse-phase liquid chromatography – mass spectrometry // Mol. Cell Proteomics. 2015. V. 14. № 6. P. 1684.
Boelaert J., Lynen F., Glorieux G., Schepers E., Neirynck N., Vanholder R. Metabolic profiling of human plasma and urine in chronic kidney disease by hydrophilic interaction liquid chromatography coupled with time-of-flight mass spectrometry: a pilot study // Anal. Bioanal. Chem. 2017. V. 409. № 8. P. 2201.
Zeng J., Yin P., Tan Y., Dong L., Hu C., Huang Q., Lu X., Wang H., Xu G. Metabolomics study of hepatocellular carcinoma: discovery and validation of serum potential biomarkers by using capillary electrophoresis-mass spectrometry // J. Proteome Res. 2014. V. 13. № 7. P. 3420.
Tsuruoka M., Hara J., Hirayama A., Sugimoto M., Soga T., Shankle W.R., Tomita M. Capillary electrophoresis-mass spectrometry-based metabolome analysis of serum and saliva from neurodegenerative dementia patients // Electrophoresis. 2013. V. 34. № 19. P. 2865.
Yang X., Sun L., Zhao A., Hu X., Qing Y., Jiang J., Yang C., Xu T., Wang P., Liu J., Zhang J., He L., Jia W., Wan C. Serum fatty acid patterns in patients with schizophrenia: A targeted metabonomics study // Transl. Psychiatry. 2017. V. 7. № 7. P. 1176.
Shamsipur M., Naseri M.T., Babri M. Quantification of candidate prostate cancer metabolite biomarkers in urine using dispersive derivatization liquid–liquid microextraction followed by gas and liquid chromatography–mass spectrometry // J. Pharm. Biomed. Anal. 2013. V. 81–82. P. 65.
Khalid T., Aggio R., White P., Costello B.D.L., Persad R., Al-Kateb H., Jones P., Probert C.S., Ratcliffe N. Urinary volatile organic compounds for the detection of prostate cancer // PLoS One. 2015. V. 10. № 11.
Won A.J., Kim S., Kim Y.G., Kim K.B., Choi W.S., Kacew S., Kim K.S., Jung J.H., Lee B.M., Kim S., Kim H.S. Discovery of urinary metabolomic biomarkers for early detection of acute kidney injury // Mol. Biosyst. 2016. V. 12. № 1. P. 133.
Pouline M.P.O., Bruin S., Weda H., Knobel H.H., Schultz M.J., Bos L.D. Exhaled breath metabolomics for the diagnosis of pneumonia in intubated and mechanically-ventilated intensive care unit (ICU)-patients // Int. J. Mol. Sci. 2017. V. 18. № 2. P. 449.
Fu X., Li M., Knipp R.J., Nantz M.H., Bousamra M. Noninvasive detection of lung cancer using exhaled breath // Cancer Med. 2014. V. 3. № 1. P. 174.
Mikkonen J.J., Singh S.P., Herrala M., Lappalainen R., Myllymaa S., Kullaa A.M. Salivary metabolomics in the diagnosis of oral cancer and periodontal diseases // J. Periodontal. Res. 2016. V. 51. № 4. P. 431.
Sugimoto M., Wong D.T., Hirayama A., Soga T., Tomita M. Capillary electrophoresis mass spectrometry-based saliva metabolomics identified oral, breast and pancreatic cancer-specific profiles // Metabolomics. 2010. V. 6. № 1. P. 78.
Calderon-Santiago M., Priego-Capote F., Turck N., Robin X., Jurado-Gamez B., Sanchez J.C., Luque de Castro M.D. Human sweat metabolomics for lung cancer screening // Anal. Bioanal. Chem. 2015. V. 407. № 18. P. 5381.
Galbis-Estrada C., Martinez-Castillo S., Morales J.M., Vivar-Llopis B., Monleon D., Diaz-Llopis M., Pinazo-Duran M.D. Differential effects of dry eye disorders on metabolomic profile by 1H nuclear magnetic resonance spectroscopy // J. Biomed. Biotechnol. 2014. V. 2014. P. 1.
Chen L., Zhou L., Chan E.C.Y., Neo J., Beuerman R.W. Characterization of the human tear metabolome by LC-MS/MS // J. Proteome Res. 2011. V. 10. № 10. P. 4876.
Li Z., Du B., Li J., Zhang J., Zheng X., Jia H., Xing A., Sun Q., Liu F., Zhang Z. Cerebrospinal fluid metabolomic profiling in tuberculous and viral meningitis: Screening potential markers for differential diagnosis // Clin. Chim. Acta. 2017. V. 466. P. 38.
Huang Q., Tan Y., Yin P., Ye G., Gao P., Lu X., Wang H., Xu G. Metabolic characterization of hepatocellular carcinoma using non-targeted tissue metabolomics // Cancer Res. 2013. V. 73. № 16. P. 4992.
Gao P., Zhou C., Zhao L., Zhang G., Zhang Y. Tissue amino acid profile could be used to differentiate advanced adenoma from colorectal cancer // J. Pharm. Biomed. Anal. 2016. V. 118. P. 349.
Vuckovic D. Current trends and challenges in sample preparation for global metabolomics using liquid chromatography–mass spectrometry // Anal. Bioanal. Chem. 2012. V. 403. № 6. P. 1523.
Bruce S.J., Tavazzi I., Parisod V., Rezzi S., Kochhar S., Guy P.A. Investigation of human blood plasma sample preparation for performing metabolomics using ultrahigh performance liquid chromatography/mass spectrometry // Anal. Chem. 2009. V. 81. № 9. P. 3285.
Nagana Gowda G.A., Raftery D. Quantitating metabolites in protein precipitated serum using NMR spectroscopy // Anal. Chem. 2014. V. 86. № 11. P. 5433.
Qiu Y., Cai G., Su M., Chen T., Zheng X., Xu Y., Ni Y., Zhao A., Xu L.X., Cai S., Jia W. Serum metabolite profiling of human colorectal cancer using GC-TOFMS and UPLC-QTOFMS // J. Proteome Res. 2009. V. 8. № 10. P. 4844.
Tiziani S., Einwas A.H., Lodi A., Ludwig C., Bunce C.M., Viant M.R., Günther U.L. Optimized metabolite extraction from blood serum for H-1 nuclear magnetic resonance spectroscopy // Anal. Biochem. 2008. V. 377. № 1. P. 16.
Skov K., Hadrup N., Smedsgaard J., Frandsen H. LC–MS analysis of the plasma metabolome — a novel sample preparation strategy // J. Chrom. B. 2015. V. 978–979. P. 83.
Ferna’ndez-Peralbo M.A., Luque de Castro M.D. Preparation of urine samples prior to targeted or untargeted metabolomics mass-spectrometry analysis // Trends Anal. Chem. 2011. V. 41. P. 75.
Roux A., Thevenot E.A., Seguin F., Olivier M., Junot C. Impact of collection conditions on the metabolite content of human urine samples as analyzed by liquid chromatography coupled to mass spectrometry and nuclear magnetic resonance spectroscopy // Metabolomics. 2015. V. 11. № 5. P. 1095.
Bernini P., Bertini I., Luchinat C., Nincheri P., Staderini S., Turano P. Standard operating procedures for pre-analytical handling of blood and urine for metabolomic studies and biobanks // J. Biomol. NMR. 2011. V. 49. № 3–4. P. 231.
Yang W., Chen Y., Xi C., Zhang R., Song Y., Zhan Q., Bi X., Abliz Z. Liquid chromatography-tandem mass spectrometry-based plasma metabonomics delineate the effect of metabolites' stability on reliability of potential biomarkers // Anal. Chem. 2013. V. 85. № 5. P. 2606.
Fliniaux O., Gaillard G., Lion A., Cailleu D., Mesnard F., Betsou F. Influence of common preanalytical variations on the metabolic profile of serum samples in biobanks // J. Biomol. NMR. 2011. V. 51. № 4. P. 457.
Yin P., Lehmann R., Xu G. Effects of pre-analytical processes on blood samples used in metabolomics studies // Anal. Bioanal. Chem. 2015. V. 407. № 17. P. 4879.
Pérez-Rambla C., Puchades-Carrasco L., García-Flores M., Rubio-Briones J., López-Guerrero J.A., Pineda-Lucena A. Non-invasive urinary metabolomic profiling discriminates prostate cancer from benign prostatic hyperplasia // Metabolomics. 2017. V. 13. № 5. P. 52.
González-Domínguez R., García-Barrera T., Gómez-Ariza J.L. Using direct infusion mass spectrometry for serum metabolomics in Alzheimer’s disease // Anal. Bioanal. Chem. 2014. V. 406. № 28. P. 7137.
González-Domínguez R., García-Barrera T., Vitorica J., Gómez-Ariza J.L. Application of metabolomics based on direct mass spectrometry analysis for the elucidation of altered metabolic pathways in serum from the APP/PS1 transgenic model of Alzheimer’s disease // J. Pharm. Biomed. Anal. 2015. V. 107. P. 378.
Husek P., Svagera Z., Hanzlíkováa D., Rimnácová L., Zahradnícková H., Opekarová I., Simek P. Profiling of urinary amino-carboxylic metabolites by in situ heptafluorobutyl chloroformate mediated sample preparation and gas chromatography–mass spectrometry // J. Chromatogr. A. 2016. V. 1443. P. 211.
Ferreira A.M.C., Corderoa B.M., Pozas Á.P.C., Pavóna J.L.P. Use of microextraction by packed sorbents and gas chromatography-mass spectrometry for the determination of polyamines and related compounds in urine // J. Chromatogr. A. 2016. V. 1444. P. 32.
Rimnácová L., Husek P., Simek P. A new method for immediate derivatization of hydroxyl groups by fluoroalkyl chloroformates and its application for the determination of sterols and tocopherols in human serum and amniotic fluid by gas chromatography–mass spectrometry // J. Chromatogr. A. 2014. V. 1339. P. 154.
Savolainen O.I., Sandberg A., Ross A.B. A simultaneous metabolic profiling and quantitative multimetabolite metabolomic method for human plasma using gas chromatography tandem mass spectrometry // J. Proteome Res. 2016. V. 15. № 1. P. 259.
Zhong L., Cheng F., Lu X., Duan Y., Wang X. Untargeted saliva metabonomics study of breast cancer based on ultra-performance liquid chromatography coupled to mass spectrometry with HILIC and RPLC separations // Talanta. 2016. V. 158. P. 351.
Червяков А.В., Захарова М.Н., Пестов Н.Б. Роль D‑аминокислот в патогенезе нейродегенеративных заболеваний и при нормальном старении // Анналы клинической и экспериментальной неврологии. 2014. Т. 8. № 2. С. 51.
Takayama T., Kuwabara T., Maeda T., Noge I., Kitagawa Y., Inoue K., Todoroki K., Min J.Z., Toyooka T. Profiling of chiral and achiral carboxylic acid metabolomics: synthesis and evaluation of triazine-type chiral derivatization reagents for carboxylic acids by LC-ESI-MS/MS and the application to saliva of healthy volunteers and diabetic patients // Anal. Bioanal. Chem. 2015. V. 407. № 3. P. 1003.
Kimura T., Hamase K., Miyoshi Y., Yamamoto R., Yasuda K., Mita M., Rakugi H., Hayashi T., Isaka Y. Chiral amino acid metabolomics for novel biomarker screening in the prognosis of chronic kidney disease // Sci. Rep. 2016. V. 6.
Leuthold P., Schaeffeler E., Winter S., Büttner F., Hofmann U., Mürdter T.E., Rausch S., Sonntag D., Wahrheit J., Fend F., Hennenlotter J., Bedke J., Schwab M., Haag M. Comprehensive metabolomic and lipidomic profiling of human kidney tissue: a platform comparison // J. Proteome Res. 2017. V. 16. № 2. P. 933.
Willmann L., Erbes T., Krieger S., Trafkowski J., Rodamer M., Kammerer B. Metabolome analysis via comprehensive two-dimensional liquid chromatography: identification of modified nucleosides from RNA metabolism // Anal. Bioanal. Chem. 2015. V. 407. № 13. P. 3555.
Yang Q., Shi X., Wang Y., Wang W., He H., Lu X., Xu G. Urinary metabonomic study of lung cancer by a fully automatic hyphenated hydrophilic interaction/RPLC-MS system // J. Sep. Sci. 2010. V. 33. № 10. P. 1495.
Wang S., Zhou L., Wang Z., Shi X., Xu G. Simultaneous metabolomics and lipidomics analysis based on novel heart-cutting two-dimensional liquid chromatography-mass spectrometry // Anal. Chim. Acta. 2017. V. 966. P. 34.
Fiehn O., Garvey W.T., Newman J.W., Lok K.H., Hoppel C.L., Adams S.H. Plasma metabolomic rrofiles reflective of glucose homeostasis in non-diabetic and type 2 diabetic obese african-american women // PLoS One. 2010. V. 5. № 12.
Ikeda A., Nishiumi S., Shinohara M., Yoshie T., Hatano N., Okuno T., Bamba T., Fukusaki E., Takenawa T., Azuma T., Yoshida M. Serum metabolomics as a novel diagnostic approach for gastrointestinal cancer // Biomed. Chromatogr. 2012. V. 26. № 5. P. 548.
Zarate E., Boyle V., Rupprecht U., Green S., Villas-Boas S.G., Baker P., Pinu F.R. Fully automated trimethylsilyl (TMS) derivatisation protocol for metabolite profiling by GC-MS // Metabolites. 2017. V. 7. № 1. P. 1.
Basanta M., Jarvis R.M., Xu Y., Blackburn G., Tal-Singer R., Woodcock A., Singh D., Goodacre R., Thomas C.L., Fowler S.J. Non-invasive metabolomic analysis of breath using differential mobility spectrometry in patients with chronic obstructive pulmonary disease and healthy smokers // Analyst. 2010. V. 135. № 2. P. 315.
Basanta M., Ibrahim B., Dockry R., Douce D., Morris M., Singh D., Woodcock A., Fowler S.J. Exhaled volatile organic compounds for phenotyping chronic obstructive pulmonary disease: a cross-sectional study // Respir. Res. 2012. V. 13. № 1. P. 72.
Rudnicka J., Walczak M., Kowalkowski T., Jezierski T., Buszewski B. Determination of volatile organic compounds as potential markers of lung cancer by gas chromatography–mass spectrometry versus trained dogs // Sens. Actuators B. 2014. V. 202. P. 615.
Fuchs P., Loeseken C., Schubert J.K., Miekisch W. Breath gas aldehydes as biomarkers of lung cancer // Int. J. Cancer. 2010. V. 126. № 11. P. 2663.
Wang Y., Hu Y., Wang D., Yu K., Wang L., Zou Y., Zhao C., Zhang X., Wang P., Ying K. The analysis of volatile organic compounds biomarkers for lung cancer in exhaled breath, tissues and cell lines // Cancer Biomark. 2012. V. 11. № 4. P. 129.
Monteiro M., Carvalh M., Henrique R., Jeronimo C., Moreira N., Bastos M. L., Pinho P.G. Analysis of volatile human urinary metabolome by solid-phase microextraction in combination with gas chromatography–mass spectrometry for biomarker discovery: Application in a pilot study to discriminate patients with renal cell carcinoma // Eur. J. Cancer. 2014. V. 50. № 11. P. 1993.
Banday K.M., Pasikanti K.K., Chan E.C.Y., Singla R., Rao K.V.S., Chauhan V.S., Nanda R.K. Use of urine volatile organic compounds to discriminate tuberculosis patients from healthy subjects // Anal. Chem. 2011. V. 83. № 14. P. 5526.
Kok M.G.M., Somsen G.W., Jong G.J. The role of capillary electrophoresis in metabolic profiling studies employing multiple analytical techniques // Trends Anal. Chem. 2014. V. 61. P. 223.
Torii Y., Kawano Y., Sato H., Sasaki K., Fujimori T., Kawada J., Takikawa O., Lim C.K., Guillemin G.J., Ohashi Y., Ito Y. Quantitative metabolome profiling reveals the involvement of the kynurenine pathway in influenza-associated encephalopathy // Metabolomics. 2016. V. 12. № 5. P. 84.
Savolainen O.I., Sandberg A., Ross A.B. A simultaneous metabolic rrofiling and quantitative multimetabolite metabolomic method for human plasma using gas-chromatography tandem mass spectrometry // J. Proteome Res. 2016. V. 15. № 1. P. 259.
Zhou J., Yin Y. Strategies for large-scale targeted metabolomics quantification by liquid chromatography-mass spectrometry // Analyst. 2016. V. 141. № 23. P. 6362.
Novotny M.V., Soini H.A., Mechref Y. Biochemical individuality reflected in chromatographic, electrophoretic and mass-spectrometric profiles // J. Chroma-togr. B. Anal. Technol. Biomed. Life Sci. 2008. V. 866. № 1–2. P. 26.
Дополнительные материалы отсутствуют.
Инструменты
Журнал аналитической химии