Журнал аналитической химии, 2020, T. 75, № 12, стр. 1148-1152
Изучение влияния напряжения электрического поля на эффективность разделения одноцепочечных фрагментов ДНК при капиллярном гель-электрофорезе в полимерах на основе линейного поли-N,N,-диметилакриламида
Д. В. Бочарова a, *, Я. И. Алексеев a, В. Е. Курочкин a
a Институт аналитического приборостроения Российской академии наук
198095 Санкт-Петербург, ул. Ивана Черных, 31/33, Россия
* E-mail: souldarydary@gmail.com
Поступила в редакцию 21.11.2019
После доработки 13.03.2020
Принята к публикации 06.05.2020
Аннотация
Анализ фрагментов ДНК методом капиллярного гель-электрофореза в настоящее время широко используется в практике научно-исследовательских и испытательных лабораторий, прежде всего, для высокоточной и высокочувствительной идентификации организмов по следам их ДНК, выделенной из исследуемого биологического материала и размноженной с помощью полимеразной цепной реакции. Постоянно возрастающий поток экспертиз с использованием данного метода диктует необходимость оптимизации протоколов исследования с целью сокращения продолжительности анализа без потери эффективности разделения. Изучено влияние напряжения электрического поля на эффективность разделения одноцепочечных фрагментов ДНК в диапазоне длин от 60 до 1200 нуклеотидов в ряду полимеров на основе линейного поли-N,N-диметилакриламида. Экспериментально определены наилучшие значения напряжения электрического поля, которые следует рекомендовать для фрагментного анализа ДНК при работе на первом отечественном генетическом анализаторе Нанофор 05 с исследованными полимерами.
В настоящее время капиллярный гель-электрофорез с детектированием лазер-индуцируемой флуоресценции рутинно используют при анализе нуклеиновых кислот для решения двух основных задач – расшифровки первичной структуры ДНК по методу Сенгера (секвенирование ДНК) [1], а также для определения длин фрагментов ДНК в исследуемом образце по стандарту с известными длинами (фрагментный анализ ДНК или генетическое типирование ДНК) [2].
При секвенировании ДНК длины разделяемых фрагментов составляют от нескольких десятков до 1000 нуклеотидов и более [3]. При фрагментом анализе ДНК в зависимости от задачи исследования диапазон разделяемых фрагментов может варьировать в пределах от 20–25 нуклеотидов (длина праймеров, используемых для полимеразной цепной реакции) до 450–500 нуклеотидов (идентификация личности и популяционные исследования [4], идентификация сортов и гибридов растений [5]), либо в более широких пределах – до 1200–1500 нуклеотидов, например, при генетическом VNTR-типировании бактерий (типировании вариабельных тандемных повторов) [6].
В капиллярном гель-электрофоретическом анализе нуклеиновых кислот исторически использовали различные типы полимеров: полиэтиленоксид, поливинилпирролидон, полиэтиленгликоль, гидроксиэтилцеллюлоза, поли-N-акрилоламинопропанол [7]. Однако на практике, благодаря ряду существенных преимуществ, а именно максимальному снижению электроосмотического потока и высокой эффективности разделения фрагментов ДНК при сравнительно невысокой вязкости, наиболее широко в настоящее время используют полимерные гели на основе линейного поли-N,N-диметилакриламида (ПДМА) [8].
Известно, что важнейшей характеристикой, определяющей качество разделения фрагментов ДНК при капиллярном электрофорезе, является разрешающая способность полимерного геля [9]. На нее в различной степени влияют характеристики разделяющего фрагменты ДНК полимерного геля: прежде всего, содержание полимера в растворе [10], концентрация денатурирующего агента – мочевины [11], а также условия гель-электрофореза – напряжение электрического поля в капиллярах [8–10] и их температура [3, 8, 12, 13].
Цель данного исследования – изучение аналитических возможностей отечественного генетического анализатора Нанофор 05 [14] при анализе одноцепочечных ДНК в полимерах на основе линейного поли-N,N-диметилакриламида в широком диапазоне значений напряжения электрического поля и экспериментальное определение оптимальных параметров гель-электрофореза.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Реагенты. Использовали полимеры ПДМА-4 (ООО “СИНТОЛ”, Россия), POP-4 (ThermoFisher Scientific, США), NimaPOP-4 (Nimagen, Голландия), формамид Hi-Di (ThermoFisher Scientific, США), буфер для электрофореза ТАПС (ООО “СИНТОЛ”, Россия), спектральный калибратор СК–5 (ООО “СИНТОЛ”, Россия), стандарт длин фрагментов ДНК СД-1200 (ООО “СИНТОЛ”, Россия).
Приборы и материалы. Капиллярный гель-электрофорез с детектированием лазер-индуцируемой флуоресценции проводили на Генетическом анализаторе Нанофор 05 (Институт аналитического приборостроения РАН, Россия). Для исследования использовали капилляры внутренним диаметром 50 мкм, внешним диаметром 192 мкм, длиной 46 см (длина до окна детектора – 36 см) (Polymicro Technologies, США). Капилляры были собраны в линейки из 8 капилляров каждая (Институт аналитического приборостроения РАН, Россия).
Подготовка проб и анализ данных. 2 мкл раствора стандарта длин ДНК СД-1200 смешивали с 78 мкл деионизованного формамида и раскапывали по 10 мкл в стрип на 8 пробирок емк. 0.2 мл. Электрофорез проводили в тройном повторе по стандартному протоколу: температура капилляров – 60°С, напряжение префореза – 15 кВ, время префореза – 3 мин, напряжение ввода пробы – 3 кВ, время ввода пробы – 5 с, варьируя полимерный гель и значения напряжения электрического поля от 6 до 15 кВ с шагом в 1 кВ. Стандарт длин СД-1200 состоит из следующих 69 одноцепочечных фрагментов ДНК: 60, 70, 80, 90, 100, 114, 120, 140, 160, 180, 200, 214, 220, 240, 250, 260, 280, 300, 314, 320, 340, 360, 380, 400, 414, 420, 440, 460, 480, 500, 514, 520, 540, 560, 580, 600, 614, 620, 640, 660, 680, 700, 714, 720, 740, 760, 780, 800, 820, 840, 850, 860, 880, 900, 920, 940, 960, 980, 1000, 1020, 1040, 1060, 1080, 1100, 1120, 1140, 1160, 1180, 1200 нуклеотидов. Каждый фрагмент содержит на 5'-конце флуоресцентный краситель Sy-660 (λмакс.эмиссии = = 660 нм), позволяющий детектировать проходящие через окно детектора разделенные фрагменты ДНК в виде пиков на соответствующем данному красителю спектральном канале генетического анализатора Нанофор 05. Электрофореграмма разделения стандарта длин фрагментов ДНК СД-1200 в 8 капиллярах линейки приведена на рис. 1.
Рис. 1.
Результат электрофоретического разделения стандарта длин ДНК СД-1200 в полимере ПДМА-4 при напряженности электрического поля 6 кВ. А–Н – капилляры (снизу вверх).

Экспериментальные данные анализировали с помощью программы “TstSequenator” (Институт аналитического приборостроения РАН, Россия). Данная программа автоматически находит пики, соответствующие фрагментам стандарта длин СД-1200 и рассчитывает времена выхода пиков и значения их ширины на полувысоте.
Рассчитанные значения времен выхода и ширин пиков на полувысоте использовали для расчета разрешающей способности [15–17] полимерного геля при заданном значении напряжения электрического поля с помощью программы Excel по формуле:
где ∆T – расстояние между максимумами двух соседних пиков; W1 и W2 – ширины соседних пиков на полувысоте; ΔМ – разница в количестве нуклеотидов между фрагментами ДНК, соответствующими данным пикам.Найденные значения R для каждых двух соседних пиков в каждом капилляре использовали для нахождения среднего значения разрешающей способности Rср по всем 8 капиллярам линейки по формуле (2) с отбрасыванием наилучшего и наихудшего значений:
(2)
${{R}_{{{\text{ср}}}}} = \frac{{\sum\limits_8 {\left( {\begin{array}{*{20}{c}} {{{R}_{{\text{A}}}}} \\ {{{R}_{{\text{H}}}}} \end{array}} \right)} --{\text{min}}R--{\text{max}}R}}{6},$В качестве границы, соответствующей разрешению в один нуклеотид, использовали значение Rср = 0.5, экспериментально установленное для разных полимеров и разных условий электрофореза.
РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
Из рис. 2 видно, что для полимера ПДМА-4 длины фрагментов ДНК, для которых Rср = 0.5, находятся в диапазоне от 560 (при напряжении электрофореза 15 кВ) до 660 нуклеотидов (при напряжении электрофореза 12 кВ).
Рис. 2.
Зависимость среднего разрешения Rср от длины фрагментов ДНК СД-1200 для значений напряжения (кВ): 6 (1), 7 (2), 8 (3), 9 (4), 10 (5), 11 (6), 12 (7), 13 (8), 14 (9), 15 (10). Полимер ПДМА-4.
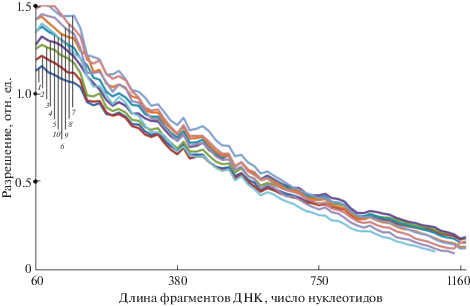
Рассчитанные значения длин фрагментов ДНК, соответствующих границе разрешения в один нуклеотид (Rср = 0.5) для полимеров ПДМА-4, POP-4 и NimaPOP-4 приведены в табл. 1. Как видно, наилучшим значением напряжения электрического поля для полимера ПДМА-4 является 12 кВ, для полимера POP-4 – 13 кВ, для полимера NimaPOP-4 – 8 кВ. При этих значениях достигается граница разрешения в один нуклеотид для фрагментов ДНК с максимальной длиной, которая составляет для полимера ПДМА-4 650 нуклеотидов, для полимера POP-4 698 нуклеотидов, а для полимера NimaPOP 4766 нуклеотидов
Таблица 1.
Длины фрагментов ДНК с Rср = 0.5 (число нуклеотидов) для исследованных полимеров (жирным шрифтом выделены максимальные длины ДНК)
| Напряжение, кВ | ПДМА-4 | POР-4 | NimaPOP-4 |
|---|---|---|---|
| 6 | 568.90 ± 0.09 | 654,52 ± 0.04 | 698.69 ± 0.07 |
| 7 | 558.9 ± 0.2 | 651.48 ± 0.06 | 684.86 ± 0.07 |
| 8 | 565.1 ± 0.2 | 676.6 ± 0.4 | 765.5 ± 0.3 |
| 9 | 631.4 ± 0.1 | 650.07 ± 0.07 | 762.8 ± 0.1 |
| 10 | 622.2 ± 0.2 | 655.3 ± 0.1 | 713.19 ± 0.08 |
| 11 | 619.73 ± 0.05 | 644.4 ± 0.1 | 693.0 ±0.1 |
| 12 | 649.5 ± 0.3 | 682.3 ± 0.4 | 678.7 ± 0.1 |
| 13 | 617.34 ± 0.08 | 697.9 ± 0.2 | 665.4 ± 0.2 |
| 14 | 584.6 ±0.2 | 658.3 ± 0.2 | 641.0 ± 0.2 |
| 15 | 551.9 ± 0.8 | 646.3 ± 0.2 | 622.25 ± 0.06 |
Важной характеристикой для пользователей, особенно для лабораторий, проводящих массовые анализы, является время, затрачиваемое на анализ образцов. В табл. 2 приведены значения времен выхода пиков фрагментов ДНК с длиной, соответствующей пороговому значению средней разрешающей способности Rср = 0.5.
Таблица 2.
Времена выхода фрагментов ДНК с Rср = 0.5 при наилучших значениях напряжений электрофореза для разных полимеров
| Характеристика | Значение | ||
|---|---|---|---|
| тип геля | ПДМА-4 | POP-4 | NimaPOP-4 |
| Длина фрагмента с Rср = 0.5, число нуклеотидов | 649.5 ± 0.3 | 697.9 ± 0.2 | 765.5 ± 0.3 |
| Напряжение электрофореза, кВ | 12 | 13 | 8 |
| Время выхода фрагмента с Rср = 0.5, с | 725 ± 2 | 1115 ± 4 | 2157 ± 7 |
Ниже приведены значения времени выхода самого высокомолекулярного фрагмента ДНК стандарта длиной 1200 нуклеотидов для трех полимеров при одинаковом напряжении электрического поля 12 кВ – лучшем значении для полимера отечественного производителя:
| Тип геля | ПДМА-4 | POP-4 | NimaPOP-4 |
|---|---|---|---|
| Время выхода фрагмента длиной 1200 нуклеотидов, с | 976 ± 3 | 1646 ± 9 | 1703 ± 10 |
Таким образом, из полученных экспериментальных данных видно, что изученные полимеры обеспечивают высокую эффективность разделения фрагментов ДНК в диапазоне длин от 60 до 1200 нуклеотидов. При этом полимеры POP-4 и NimaPOP-4 обладают несколько лучшими характеристиками разрешения, нежели ПДМА-4, но при этом время выхода фрагментов ДНК, соответствующих среднему разрешению Rср = 0.5 для них существенно выше. Чем длиннее анализируемые фрагменты ДНК, тем больше различие во времени их выхода, а следовательно, и во времени анализа для самого быстро делящего полимера ПДМА-4 и для медленно делящих полимеров POP-4, NimaPOP-4. Это различие достигает при напряжении электрофореза 12 кВ для фрагмента ДНК длиной 1200 нуклеотидов величины 670 с (при сравнении полимеров ПДМА-4 и POP-4) и 730 с (при сравнении полимеров ПДМА-4 и Ni maPOP-4).
* * *
Изучено влияние напряжения электрического поля на эффективность разделения одноцепочечных фрагментов ДНК в диапазоне длин от 60 до 1200 нуклеотидов методом капиллярного гель-электрофореза в полимерах на основе линейного поли-N,N-диметилакриламида: ПДМА-4, POP-4 и NimaPOP-4 на генетическом анализаторе Нанофор 05. Определены наилучшие значения напряжения электрического поля, которые следует рекомендовать для фрагментного анализа ДНК в изученных полимерах.
Работа выполнена по Государственному заданию Министерства науки и высшего образования Российской Федерации № 075-00780-19-00 по теме № 0074-2019-0017.
Список литературы
Slatko B.E., Kieleczawa J., Ju J., Gardner A.F., Hendrickson C.L., Ausubel F.M. “First Generation” automated DNA sequencing technology // Curr. Protocols Mol. Biol. 2011. V. 96. № 1. P. 7.2.1. https://doi.org/10.1002/0471142727.mb0702s96
Selva L., del Amo E., Brotons P., Muñoz-Almagro C. Rapid and easy identification of capsular serotypes of Streptococcus pneumoniae by use of fragment analysis by automated fluorescence-based capillary electrophoresis // J. Clin. Microbiol. 2012. V. 50. № 11. P. 3451. https://doi.org/10.1128/JCM.01368-12
Carrilho E., Ruiz-Martinez M.C., Berka J., Smirnov I., Goetzinger W., Miller A.W., Brady D., Karger B.L. Rapid DNA sequencing of more than 1000 basesper run by capillary electrophoresis using replaceable linear polyacrylamide solutions // Anal. Chem. 1996. V. 68. P. 3305. https://doi.org/10.1021/ac960411r
Al-Snan N.R., Messaoudi S., R. Babu S., Bakhiet M. Population genetic data of the 21 autosomal STRs included in Global Filer kit of a population sample from the Kingdom of Bahrain // PLoS ONE. 2019.V. 14. № 8. P. 1. DOI e0220620
Анискина Ю.В., Малиновская Е.В., Шалаева Т.В., Мицурова В.С., Родионова Д.А., Харченко П.Н., Шилов И.А. Технология генетической идентификации сортов и гибридов сорго на основе мультилокусного микросателлитного анализа // Биотехнология. 2018. Т. 37. № 2. С. 54. https://doi.org/10.21519/0234-2758-2018-34-2-54-69
Селянинов Ю.О., Егорова И.Ю., Алексеев Я.И., Казанцев А.В., Монахова Ю.А., Колбасов Д.В. Фенотипические, биохимические и молекулярно-генетические свойства штаммов Bacillusanthracis, выделенных во время вспышек сибирской язвы на территории Российской Федерации (2014–2016 годы) // Сельскохозяйственная биология. 2018. Т. 53. № 2. С. 404. https://doi.org/10.15389/agrobiology.2018.2.404rus
Albarghouthi M.N., Barron A.E. Polymeric matrices for DNA sequencing by capillary electrophoresis // Electrophoresis. 2000. V. 21. P. 4096. https://doi.org/10.1002/1522-2683(200012)21:18<4096:: AIDELPS4096>3.0.CO;2-W
Geiger M., Hogerton A.L., Bowser M.T. Capillary Electrophoresis // Anal. Chem. 2012. V. 84. № 2. P. 577. https://doi.org/10.1021/ac203205a
Quesada M. Replaceable polymers in DNA sequencing by capillary electrophoresis // Curr. Opinion Biotechnol. 1997. V. 8. № 1. P. 82. https://doi.org/10.1016/s0958-1669(97)80162-0
Manabe T., Chen N., Terabe S., Yohda M., Endo I. Effects of linear polyacrylamide concentrations and applied voltages on the separation of oligonucleotides and DNA sequencing fragments by capillary electrophoresis // Anal. Chem. 1994. V. 66. № 23. P. 4243. https://doi.org/10.1021/ac00095a020
Rosenblum B.B., Oaks F., Menchen S., Johnson B. Improved single-strand DNA sizing accuracy in capillary electrophoresis // Nucleic Acids Research. 1997. V. 25. № 19. P. 3925. https://doi.org/10.1093/nar/25.19.3925
Zhang J., Fang Y., HouJ Y., Ren H. J.,Jiang R., Roos P., Dovichi N.J. Use of non-cross-linked polyacrylamide for four-color DNA sequencing by capillary electrophoresis separation of fragments up to 640 bases in length in two hours // Anal. Chem. 1995. V. 67. № 24. P. 4589. https://doi.org/10.1021/ac00120a026
Klepárnik K., Foret F., Berka J., Goetzinger W., Miller A.W., Karger B.L. The use of elevated column temperature to extend DNA sequencing read lengths in capillary electrophoresis with replaceable polymer matrices // Elecatrophoresis. 1996. V. 17. № 12. P. 1860. https://doi.org/10.1002/elps.1150171210
Алексеев Я.И., Белов Ю.В., Малюченко О.П., Монахова Ю.А., Натыров А.Н., Орехов В.А., Коновалов С.В., Курочкин В.Е., Петров А.И. Генетический анализатор для фрагментного анализа ДНК // Научное приборостроение. 2012. Т. 22. № 4. С. 86.
Luckey J.A., Norris T.B., Smith L.M. Analysis of resolution in DNA sequencing by capillary gel electrophoresis // J. Phys. Chem. 1993. V. 97. № 12. P. 3067. https://doi.org/10.1021/j100114a038
Heller C. Influence of electric field strength and capillary dimensions on the separation of DNA // Electrophoresis. 2000. V. 21. № 3. P. 593. https://doi.org/10.1002/(sici)1522-2683(20000201)21:3<593::aid-elps593>3.0.co;2-u
Беленький Б.Г. Высокоэффективный капиллярный электрофорез. Санкт-Петербург: Наука, 2009. С. 314.
Дополнительные материалы отсутствуют.
Инструменты
Журнал аналитической химии


