Журнал аналитической химии, 2022, T. 77, № 5, стр. 393-405
Количественное определение мРНК Бета-лактамаз в РНК-транскриптах резистентных к антибиотикам бактерий с использованием колориметрических биочипов
М. Ю. Рубцова a, *, А. А. Филиппова a, Н. К. Фурсова b, В. Г. Григоренко a, Г. В. Преснова a, М. М. Уляшова a, А. М. Егоров a
a Московский государственный университет имени М.В. Ломоносова, химический факультет
119991 Москва, Ленинские горы, 1, стр. 3, Россия
b Государственный научный центр прикладной микробиологии и биотехнологии
Территория “Квартал А”
142279 Московская обл., Серпухов, 24, пос. Оболенск, г. о., Россия
* E-mail: mrubtsova@gmail.com
Поступила в редакцию 12.07.2021
После доработки 26.08.2021
Принята к публикации 28.08.2021
- EDN: KHVNRC
- DOI: 10.31857/S0044450222050140
Аннотация
Разработана методика количественного определения мРНК сериновых бета-лактамаз ТЕМ-типа у бактерий, устойчивых к бета-лактамным антибиотикам. Методика включает несколько стадий: выделение фракции общей РНК из бактериальной культуры, получение ДНК-мишени бета-лактамазы в последовательных реакциях обратной транскрипции и полимеразной цепной реакции с введением метки – биотина и гибридизацию ДНК-мишени на колориметрических биочипах. Оптимизированы условия пробоподготовки для увеличения выхода анализируемой ДНК-мишени. Градуировочная кривая для определения количества мРНК построена с использованием стандартного образца мРНК бета-лактамазы ТЕМ-1, полученного методом транскрипции in vitro. Преимуществом использования стандартного образца, соответствующего полноразмерному гену blaТЕМ-1, является прохождение им всех стадий анализа параллельно с исследуемыми образцами с одинаковой эффективностью. Предел обнаружения мРНК бета-лактамазы ТЕМ-1 составил 0.40 ± 0.05 амоль/мл, диапазон определяемых концентраций – от 1.0 амоль/мл до 2000 фмоль/мл, относительное стандартное отклонение не превышало 12%. Продолжительность анализа после получения бактериальной культуры составляет около 7 ч. Разработанная методика может быть использована для изучения условий экспрессии генов бета-лактамаз у резистентных к антимикробным препаратам бактерий.
Широкое распространение резистентных к антибиотикам бактерий – возбудителей инфекционных заболеваний – является одной из актуальных проблем современной биологии, медицины и фармакологии. Она обострилась в связи с появлением мульти- и пан-резистентных бактерий, одновременно устойчивых к нескольким классам, и даже практически ко всем антибиотикам [1–3]. Механизмы резистентности бактерий разнообразны и включают процессы c участием ферментов, модифицирующих бактериальные мишени антибиотиков, изменяющих структуру самих антибиотиков, нарушающих процессы их проникновения в бактериальную клетку и активный вывод из нее [4, 5]. Проблема устойчивости к антибиотикам является очень широкой и касается всех микроорганизмов, включая грамотрицательные и грамположительные бактерии. Сочетание нескольких механизмов устойчивости у патогенных бактерий существенно осложняет выбор антимикробных препаратов, особенно при лечении внутрибольничных инфекций, которые развиваются очень быстро у ослабленных пациентов.
Одним из наиболее распространенных видов резистентности у грамотрицательных бактерий является их устойчивость к бета-лактамным антибиотикам – классу антимикробных препаратов, наиболее часто используемых в здравоохранении. Так, на долю пенициллинов широкого спектра и цефалоспоринов приходится соответственно 39 и 20% от всех используемых в клинической практике антибиотиков [6, 7]. Основным механизмом резистентности является гидролиз антибиотиков бета-лактамазами (БЛ), образующими суперсемейство из более 2800 ферментов и различающимися по строению активного центра, субстратной специфичности и чувствительности к ингибиторам [8]. Мультирезистентные бактерии часто являются носителями генов нескольких типов БЛ.
Для исследования устойчивых к бета-лактамным антибиотикам бактерий, в том числе мультирезистентных, активно используются молекулярно-генетические методы, основанные на идентификации генов, кодирующих БЛ [9, 10]. В результате постоянно протекающих процессов эволюции резистентности в экосистемах, в том числе госпитальных, отмечается феномен развития гетерорезистентности, связанной с формированием смешанной популяции бактерий, резистентных и чувствительных к антимикробным препаратам [11, 12]. Для изучения механизмов формирования генетической и фенотипической гетерорезистентности популяций патогенов, влияния внешних факторов на подавление и активацию генов антибиотикорезистентности у бактерий необходимы методы количественного определения экспрессирующихся генов через выявление специфичных мРНК в РНК-транскриптах бактерий. Эти методы должны обладать высокими специфичностью и чувствительностью, а также широким динамическим диапазоном, позволяющим определять как низкие, так и высокие уровни экспрессии генов. В связи с разнообразием типов ферментов, обусловливающих антибиотикорезистентность, методы определения мРНК должны обладать высокой производительностью и возможностью одновременного определения большого набора разных мРНК.
В настоящее время для определения специфичных мРНК в РНК-транскриптах бактерий применяют полуколичественные методы с использованием внутренних контролей: изменение экспрессии изучаемого гена определяется по отношению к уровню экспрессии контрольного гена (генов). Для этих целей активно развиваются две технологии: полимеразная цепная реакция в режиме реального времени (ПЦР-РВ) [13–15] и использование биочипов высокой плотности [16]. Обе технологии включают пробоподготовку, состоящую из выделения общей РНК из бактериальной культуры и получения кДНК в реакции обратной транскрипции (ОТ). Количество одновременно определяемых генов в одной реакции методом ПЦР-РВ ограничено количеством флуорофоров, которые могут быть использованы в качестве метки [17]. Биочипы высокой плотности характеризуются наиболее высокой мультиплексностью – от десятков до сотен и более тысяч вариантов одновременно идентифицируемых генов, которая определяется количеством иммобилизованных на биочипе специфичных олигонуклеотидных зондов.
Технология ПЦР-РВ позволяет оценить количество мРНК или числа копий гена в одной бактериальной клетке, однако для этого необходимо знать эффективность стадий ОТ и ПЦР, что не всегда можно определить экспериментально [18]. Биочипы высокой плотности активно применяются для изучения механизмов резистентности бактерий, они показали свою эффективность для определения профилей изменения РНК-транскриптомов [19, 20]. Однако, поскольку результаты определения специфичных мРНК являются относительными, данная технология позволяет зарегистрировать только существенные изменения в экспрессии генов. Более тонкие эффекты, связанные с незначительными изменениями концентраций отдельных мРНК, трудно зафиксировать из-за сложностей многоступенчатой нормировки результатов анализа.
В последние годы для идентификации устойчивости к антимикробным препаратам у бактерий активно развивается технология колориметрических биочипов низкой плотности, включающих не более нескольких десятков специфичных олигонуклеотидных зондов [21, 22]. Колориметрические биочипы показали свою эффективность для идентификации генов различных БЛ, в том числе у клинических патогенных штаммов с множественной резистентностью к антибиотикам [23–25]. Данные биочипы представляются более перспективными для практического использования в клинической практике по сравнению с биочипами высокой плотности, поскольку не требуют дорогостоящего оборудования для проведения анализа и получения результатов. Подобные биочипы могут входить в состав автоматизированных устройств для “диагностики по месту лечения” (point of care), что существенно упрощает их применение в клинических лабораториях [26].
До настоящего времени биочипы низкой плотности применялись в основном для идентификации генов антибиотикорезистентности в геномах бактерий. Поскольку интенсивность окрашивания зон биочипа определяется с высокой точностью с использованием оптических сканеров высокого разрешения, данная технология может применяться и для количественного определения нуклеиновых кислот.
Цель данного исследования – разработка методики определения мРНК БЛ, основанной на комбинации реакций ОТ и ПЦР с гибридизацией полученной ДНК-мишени на колориметрических биочипах в лунках 96-луночных планшетов. Для определения исследуемой мРНК строили градуировочную зависимость с использованием набора стандартных образцов мРНК, полученной методом транскрипции in vitro. Задачи работы включали оптимизацию отдельных стадий методики, построение градуировочной зависимости с использованием стандартных образцов мРНК и применение разработанной методики для определения мРНК БЛ в контрольном и клинических образцах грамотрицательных бактерий.
В качестве объекта исследования выбрали сериновые БЛ класса А ТЕМ-типа. Данное семейство БЛ включает более 240 ферментов, характеризующихся высокой консервативностью последовательностей с единичными аминокислотными заменами [27]. БЛ ТЕМ-1 обеспечивает устойчивость бактерий к пенициллинам и цефалоспоринам I поколения. Часть мутантов этого фермента, имеющих единичные аминокислотные замены, относится к БЛ расширенного спектра (БЛРС), обусловливающих резистентность бактерий ко всем пенициллинам и цефалоспоринам. БЛ ТЕМ-типа достаточно широко распространены среди клинических штаммов бактерий, а также почвенных бактерий, они являются удобной моделью для изучения механизмов ферментативного гидролиза и резистентности бактерий к бета-лактамным антибиотикам [28]. Интерес к определению мРНК БЛ ТЕМ-типа обусловлен появлением данных об увеличении экспрессии ферментов этого типа у грамотрицательных бактерий при применении бета-лактамов в комбинации с бета-лактамными ингибиторами [29, 30].
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Бактериальные штаммы. В работе использовали лабораторные штаммы Escherihia сoli – продуценты рекомбинантных БЛ ТЕМ-1, ТЕМ-2, ТЕМ-12, ТЕМ-18, ТЕМ-129, ТЕМ-160, содержащие плазмиду pET 24d c клонированными генами соответствующих БЛ под контролем промотора бактериофага T7 со средним уровнем копийности по методике, описанной в работе [31], аббревиатуры ферментов соответствуют номерам мутантных форм БЛ ТЕМ-типа в международной базе данных NCBI Beta-Lactamase Data Resources (https://www.ncbi.nlm.nih.gov/pathogens/beta-lactamase-data-resources/); клинические штаммы грамотрицательных бактерий, выделенные из клинического материала (моча, эндотрахеальный аспират) (E. coli B-1350/18, E. coli B-2607/18, Klebsiella. pneumoniae B-1088/17). Клинические штаммы получены из Государственной коллекции патогенных микроорганизмов “ГКПМ-Оболенск”. Их видовую идентификацию проводили на приборах VITEK-2 Compact (bioMerieux, Франция) и MALDI Biotyper (Bruker, Германия). Интерпретацию результатов проводили согласно рекомендациям Европейского комитета по тестированию антимикробной чувствительности (The European Committee on Antimicrobial Susceptibility Testing), EUCAST v.10.0 [32].
Культивирование бактерий, выделение нуклеиновых кислот, выделение периплазматических фракций, определение в них ферментативной активности БЛ. Бактерии культивировали в трех повторах на жидкой питательной среде LB (Becton Dickinson, США) при 37°С в течение 18–24 ч, лабораторные штаммы – в присутствии 50 мг/л канамицина, клинические штаммы – в присутствии разных концентраций ампициллина (0, 8, 100, 250 мг/л).
Плазмидную ДНК выделяли из культуры бактериальных клеток по методике [33] с использованием набора Plasmid Midiprep 2.0 (Евроген, Россия). Фракцию общей РНК выделяли из бактериальных клеток набором РНК-экстран (Синтол, Россия) по протоколу компании-производителя. Качество полученной РНК контролировали методом электрофореза в 1%-ном агарозном геле по наличию двух полос 18S и 28S недеградированной рРНК. Примесь ДНК удаляли ДНКазой I (Thermo Scientific, Литва): инкубировали образец с ДНКазой (2 е. а./мкл) в течение 30 мин при 37°С, реакцию останавливали 4 мМ ЭДТА (конечная концентрация) и инкубировали 10 мин при 65°С. Полноту удаления ДНК контролировали методом ПЦР с использованием праймеров к фрагменту 16S бактериальной ДНК по отсутствию продукта амплификации [34]. Концентрацию нуклеиновых кислот определяли спектрофотометрически на спектрофотометре UV-1602 (Shimadzu, Япония).
Для получения периплазматических фракций использовали методику [35]: 10 мл бактериальной культуры осаждали центрифугированием при 3500 g в течение 10 мин на центрифуге (Eppendorf 5810/5810R, Германия) и замораживали осадок биомассы при –20°С. Клетки разрушали осмотическим шоком в растворе сахарозы (20%-ная сахароза, 10 мМ Трис-HCl, 1 мМ ЭДТА, pH 8.0), затем центрифугировали при 12000 g в течение 15 мин при температуре +4°С и отделяли супернатант. Ферментативную активность БЛ определяли по скорости гидролиза ампициллина: в кювете объемом 1 мл смешивали 50 мМ Na-фосфатного буферного раствора с рН 7.0 с 35 мкл периплазматической фракции и раствором ампициллина в конечной концентрации 75 мкМ, затем регистрировали кинетические кривые расхода ампициллина при длине волны 235 нм (∆ε235 = = 670 М–1 см–1). Ферментативную активность определяли как отношение изменения оптической плотности за единицу времени к молярному коэффициенту поглощения согласно формуле:
где $\Delta OD$ – изменение оптической плотности за промежуток времени t, $\Delta {{\varepsilon }}$ – молярный коэффициент поглощения.Получение мРНК БЛ ТЕМ-1 методом транскрипции in vitro. Амплифицировали фрагмент плазмиды, включающий полноразмерный ген БЛ ТЕМ-1 и участки промотора и терминатора Т7 с использованием прямого (5'-TCCGGATATAGTTCCTCCTTTCA-3') и обратного (5'-AGATCTCGATCCCGCGAA-3') праймеров, в качестве матрицы использовали фракцию общей ДНК, выделенной из клеток E. coli – продуцентов БЛ ТЕМ-1. Синтез мРНК проводили с использованием набора T7-Transcription (Биолабмикс, Россия) по методике [36] с увеличенными концентрациями фермента и dNTP: 1 мкл ПЦР-продукта смешивали в объеме 50 мкл с буфером для транскрипции, смесь dNTP в конечной концентрации 1 мМ и Т7 РНК-полимеразой (3 е. а./мкл) инкубировали в течение 2 ч при 37°С. Излишки ДНК удаляли с помощью ДНказы (2 е. а./мкл, 37°С, 30 мин). Концентрацию РНК определяли спектрофотометрически.
Получение первой цепи кДНК в реакции обратной транскрипции. Первую цепь кДНК получали с использованием реакции обратной транскрипции по методике [37], в которой меняли состав праймеров: 2 мкл образца общей РНК и праймеры в конечной концентрации 0.5 мкМ (набор праймеров случайного состава длиной 10 оснований (Random(dNTP)10), включающий олигонуклеотид для взаимодействия с поли-dАTP фрагментом на 3'-конце мРНК (Oligo(dTTP)15), или специфический обратный праймер для БЛ ТЕМ типа (5'-TTAATCAGTGAGGCACCTAT-3')) прогревали (70°С, 2 мин), затем охлаждали на льду и добавляли 5× буфер (250 мМ Трис-HCl, 250 мМ KCl, 20 мМ MgCl2, pH 8.3), смесь dNTP в конечной концентрации 1 мМ, 2 мМ дитиотриэтола и 1 мкл ревертазы MMLV вируса лейкемии летучих мышей (Евроген, Россия). Смесь инкубировали (42°С, 1 ч) и останавливали реакцию прогреванием (70°С, 10 мин).
Гибридизационный анализ биотинилированной ДНК-мишени на биочипах. 96-луночные планшеты (Greiner Bio-one Maxisorb, Германия) модифицировали поли-D-лизином (200 мкг/мл в фосфатном буферном растворе (ФБ), 50 мкл/лунку) в термомиксере Thermomixer Comfort (Eppendorf AG, Гамбург, Германия) (37°C, 1 ч), отмывали ФБ (2 раза по 10 мин). Затем в лунки добавляли глутаровый альдегид (1%-ный раствор в ФБ, 50 мкл/лунку), инкубировали (37°C, 1 ч), отмывали ФБ (2 раза по 10 мин). Олигонуклеотидные зонды, модифицированные на 5'-конце аминогруппой и линкером из 13 dTTP, растворяли в буферном растворе (160 мМ Na2SO4, 130 мМ Na2HPO4) до конечной концентрации 20 пмоль/мкл и наносили в виде матрицы (6 × 6) в лунки планшетов с использованием робота XactII™ (LabNextInc., США). Последовательности олигонуклеотидных зондов для идентификации БЛ разных типов приведены в табл. 1. Каждый специфичный зонд был нанесен в шести повторах, контрольные зонды – в трех повторах. Средний размер зоны биочипа с зондом составлял 300 мкм, расстояние между зонами составляло 500 мкм. После нанесения зондов планшет прогревали (60°C, 20 мин), отмывали ФБ (2 раза по 10 мин) и блокировали в растворе состава 1% бычьего сывороточного альбумина (БСА) и 1% казеина (37°C, 1 ч).
Таблица 1.
Последовательности олигонуклеотидных зондов для гибридизационного анализа генов бета-лактамаз на биочипах
| Тип бета-лактамазы (молекулярный класс) | Зонд (участок нуклеотидной последовательности гена, н.) | Олигонуклеотидная последовательность 5'–3' |
|---|---|---|
| TEM (класс A) | ID1-TEM (362–381) | CAGTGCTGCCATAACCATG |
| ID2-TEM (409–429) | CTGACAACGATCGGAGGACCG | |
| ID3-TEM (598–619) | GCTTCCCGGCAACAATTAATAG | |
| ID4-TEM (565–589) | СGCAAACTATTAACTGGCGAACTAC | |
| SHV (класс A) | ID-SHV | GTTGATCCGCTCCGTGCTG |
| CTX-M (класс A) | ID-CTX-M | TATATCGCGGTGATCTGGCC |
| OXA (класс D) | ID-OXA-48 | TGCTGTATCACAATAAGTTACACGTATC |
| NDM (класс B) | ID-NDM | TCCAAGTCGCTCGGCAATCTC |
| – | Положительный контроль гибридизации (ПКГ) | TGATTGGACGAGTCAGGAGC |
| – | Отрицательный контроль гибридизации (ОКГ) | TCTAGACAGCCACTCATA |
| – | Контрольный зонд для гибридизации (ПКГ-Бт*) | Биотин-GCTCCTGACTCGTCCAATC |
Образец первой цепи кДНК использовали в качестве матрицы в реакции ПЦР с введением биотина [38]. Размер амплифицируемых полноразмерных генов БЛ составлял 851 п. о. Затем полученные ампликоны фрагментировали ДНКазой I (0.1 е. а./30 нг ДНК) в объеме 50 мкл при комнатной температуре в течение 5 мин. Реакцию останавливали добавлением 3 мМ ЭДТА и инкубировали 10 мин при 65°С. Размер полученных фрагментов составлял от 50 до 150 п. н. Гибридизацию проводили в термомиксере: 50 мкл ДНК, меченной биотином, в буферном растворе SSPE (0.2 М NaH2PO4, 3.0 М NaCl, 20 мМ ЭДТА, 0.1% додецилсульфата натрия, рН 7.4) и 1 нМ биотинилированного контрольного олигонуклеотида инкубировали в лунках планшета (45°C, 2 ч). Затем планшет отмывали ФБ (45°C, 10 мин), инкубировали с конъюгатом стрептавидин-пероксидаза хрена (Имтек, Россия) в ФБ, содержащем 1% БСА (37°C, 30 мин), отмывали раствором ФБ (комнатная температура, три раза по 10 мин) и добавляли субстратный раствор (5 мМ 3,3',5,5' – тетраметилбензидина, 1 мМ H2O2, 0.5% декстрансульфата натрия в 0.1 М ацетатном буферном растворе, pH 5.5).
Количественная обработка результатов гибридизационного анализа. Поверхность биочипов в лунках планшета сканировали на оптическом сканере Perfection V750 Pro (Epson, Германия) с разрешением 4800 dpi, цветные изображения получали в формате TIFF файлов (16 bit). Далее цветные изображения переводили в монохромные черно-белые с использованием программного обеспечения Adobe Photoshop CS3 и определяли интенсивность окрашивания каждой зоны биочипа и локального фонового окрашивания вокруг нее с использованием ScanArray Express (версия 3.0).
Интенсивность окрашивания (I) определяли как среднее арифметическое значение интенсивностей окрашивания зоны за вычетом среднего локального фонового окрашивания по формуле:
где Ii – интенсивность окрашивания зоны биочипа, Iфi – локальное фоновое значение, n – число повторов одной зоны на биочипе.Относительную интенсивность окрашивания зоны рассчитывали как отношение среднего значения интенсивности окрашивания данной зоны к среднему значению окрашивания контрольной зоны по формуле (3):
где Iотн – среднее значение относительной интенсивности окрашивания зоны, I – среднее значение интенсивности окрашивания зоны со специфическим зондом для данного типа БЛ, Iконтр– среднее значение интенсивности окрашивания зоны положительного контроля гибридизации (ПКГ).Статистическую обработку данных проводили с использованием t-критерия Стьюдента (p < 0.05).
Построение градуировочной кривой для определения мРНК БЛ. Определение мРНК БЛ ТЕМ-1 в стандартных образцах проводили с использованием разработанной методики, включающей последовательные стадии ОТ, ПЦР и гибридизации на биочипе, как описано выше. Строили градуировочную зависимость относительной интенсивности окрашивания специфических зон биочипа от концентрации стандартных образцов мРНК.
Предел обнаружения мРНК БЛ рассчитывали по формуле:
где $I{\text{от}}{{{\text{н}}}_{0}}$ – значение относительной интенсивности окрашивания зоны биочипа со специфическим зондом при гибридизации образца, не содержащего мРНК БЛ ТЕМ-типа; σ – стандартное отклонение фонового сигнала.РЕЗУЛЬТАТЫ И ИХ ОБСУЖДЕНИЕ
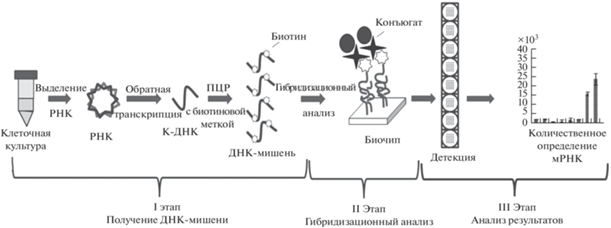
Определение специфичных мРНК БЛ включает несколько этапов: пробоподготовку меченной биотином ДНК-мишени, гибридизацию ее на биочипе с иммобилизованными специфическими олигонуклеотидными зондами и детекцию метки-биотина в дуплексах ДНК, образовавшихся в отдельных зонах биочипа (рис. 1). Пробоподготовка заключается в выделении фракции общей РНК из клеточной культуры, получении кДНК в реакции ОТ и амплификации анализируемого гена БЛ методом ПЦР с одновременным введением метки-биотина. Стадия детектирования включает выявление биотина конъюгатом стрептавидин-пероксидаза хрена и последующее колориметрическое определение пероксидазы. В результате ферментативной реакции образуется нерастворимый окрашенный продукт синего цвета, адсорбирующийся в соответствующей зоне биочипа. Изменение интенсивности окраски зон биочипа пропорционально изменению концентрации мРНК исследуемого гена в РНК-транскрипте.
Рис. 1.
Схема метода определения мРНК БЛ методом гибридизационного анализа на колориметрических биочипах низкой плотности.
Оптимизация условий определения мРНК включала оптимизацию определения ДНК БЛ на биочипах и оптимизацию методики пробоподготовки.
Оптимизация гибридизационного анализа на колориметрических биочипах для определения генов БЛ ТЕМ-типа. Для выявления генов БЛ ТЕМ-типа на биочипах протестировали четыре олигонуклеотидных зонда, последовательность которых соответствовала четырем консервативным участкам нуклеотидных последовательностей (362–381, 409–429, 565–589 и 598–619 п. н.) генов ферментов данного семейства. Биочипы включали специфичные зонды (ID1-TEM, ID2-TEM, ID3-TEM, ID4-TEM) и два контрольных зонда (положительный ПКГ и отрицательный ОКГ). Последовательности зондов приведены в табл. 1. В качестве контрольных зондов использовали последовательности олигонуклеотидов, которые не встречаются у бактерий.
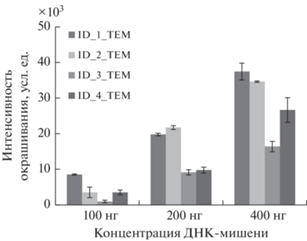
При гибридизации образца ДНК БЛ ТЕМ-1, меченной биотином, со специфичными олигонуклеотидными зондами интенсивность окрашивания зависела от типа зонда, поскольку последние имели разную структуру и длину и характеризовались соответственно разной стабильностью ДНК-дуплексов (рис. 2). Для всех зондов наблюдали прямо пропорциональную зависимость интенсивности окрашивания зон биочипа от количества ДНК-мишени. Для дальнейшей работы выбрали зонд ID1-TEM, соответствующий в нуклеотидной последовательности гена участку 362–381 п. н., обеспечивающий наиболее высокие значения интенсивностей окрашивания зон биочипа в диапазоне концентраций ДНК-мишени от 100 до 400 нг/биочип.
Рис. 2.
Интенсивность окрашивания зон биочипа с иммобилизованными олигонуклеотидными зондами при гибридизации меченной биотином ДНК БЛ ТЕМ-1.
Для определения ДНК БЛ изготовили биочипы, включающие шесть специфичных олигонуклеотидных зондов, соответствующих наиболее клинически значимым типам БЛ различных молекулярных классов (ТЕМ-, SHV-, CTX-M-типов (класс А), NDM типа (класс B), OXA-48 типа (класс D)). Структуру идентификационных зондов выбрали ранее по консервативным участкам нуклеотидных последовательностей генов соответствующих БЛ (табл. 1) [24]. Помимо специфичных зондов, каждый биочип включал два контрольных зонда. Схема расположения зондов на биочипе представлена на рис. 3.
Рис. 3.
Расположение специфичных и контрольных олигонуклеотидных зондов на биочипе в лунке 96-луночного планшета.

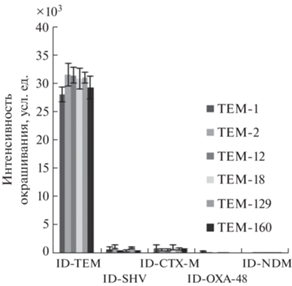
Биочипы применили для определения ДНК БЛ ТЕМ-типа с использованием образцов ДНК, выделенных из клеток E. coli – продуцентов разных вариантов БЛ (ТЕМ-2, -12, -18, -129, -160), различающихся единичными аминокислотными заменами. Ферменты ТЕМ-12, -18, -129 и -160 являются БЛРС и содержат замены ключевых аминокислот, приводящие к расширению спектра субстратной специфичности и устойчивости к ингибиторам БЛ бета-лактамной структуры. Результаты гибридизационного анализа (рис. 4) показали высокую специфичность определения БЛ данного типа: окрашивание зон биочипа со специфичным зондом ID1-TEM характеризовалось высокой интенсивностью, при этом интенсивность окрашивания зон с зондами другой специфичности, в том числе с зондами для наиболее близких структурно БЛ класса А (SHV-и CTX-M-типов), была низкой. Необходимо отметить хорошую сходимость значений интенсивности при гибридизации образцов ДНК, соответствующих разным вариантам БЛ ТЕМ-типа, разброс значений не превышал 11%, что позволяет использовать гибридизационный анализ на колориметрических биочипах для определения нуклеиновых кислот.
Рис. 4.
Интенсивность окрашивания зон биочипа с иммобилизованными зондами разной специфичности после гибридизации 400 нг ДНК-мишени, полученной из образцов ДНК разных БЛ ТЕМ-типа (ТЕМ-1, -2, -12, -18, -129, -160).
Получение кДНК и меченной биотином ДНК-мишени для гибридизационного анализа. Для оптимизации методики пробоподготовки ДНК-мишени изучали условия получения кДНК и амплификации генов БЛ методом ПЦР с одновременным введением метки-биотина. На первом этапе определили оптимальное количество циклов ПЦР, необходимое для получения достаточной интенсивности окрашивания специфичных зон биочипа. В качестве матрицы использовали образец ДНК, выделенный из клеток штамма E. coli – продуцента рекомбинантной БЛ ТЕМ-1. Проводили ПЦР в условиях, разработанных ранее для мультиплексной амплификации БЛ разных классов в одной реакции, в течение разного количества циклов реакции. Далее ампликоны гибридизовали на биочипе, определяли интенсивность окрашивания специфичных зон биочипа и нормировали ее на интенсивность окрашивания зоны положительного контроля. Анализ зависимости относительных интенсивностей окрашивания зон биочипа от числа циклов (рис. 5а) показал, что выход реакции начинает увеличиваться после 20 цикла и достигает максимальных значений после 30 цикла. В дальнейшей работе использовали 30 циклов для амплификации ДНК-мишеней.
Рис. 5.
(а): Относительная интенсивность окрашивания специфичных зон биочипа после гибридизации ПЦР-продуктов, полученных с использованием разного числа циклов амплификации. (б): Электрофореграмма продуктов последовательных реакций ОТ и ПЦР гена БЛ ТЕМ-1, в качестве матрицы использован образец общей РНК, выделенный из клеток E. coli-продуцентов (1 × 107 КОЕ/мл). М – смесь ДНК-маркеров известной длины. Реакцию ОТ проводили с использованием смеси праймеров случайного состава длиной 10 оснований и праймера из 15 dTTP (1) и специфического праймера для БЛ ТЕМ-типа (2).
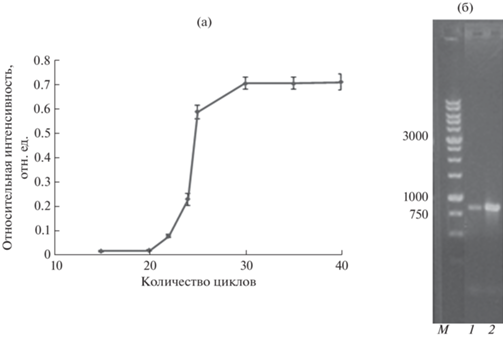
Получение кДНК проводили в реакции ОТ с использованием в качестве матрицы фракции общей РНК, выделенной из лабораторного штамма E. coli. Для увеличения эффективности данной стадии изучали состав праймеров, в качестве которых использовали набор праймеров случайного состава длиной 10 оснований и праймера, включающего последовательность из поли-dTTP для взаимодействия с поли-dATP фрагментом на 3'-конце мРНК, а также праймер, специфичный для БЛ ТЕМ-типа, выбранный по консервативному концевому участку нуклеотидной последовательности гена. Полученные кДНК использовали в качестве матрицы ПЦР, размер продуктов которой анализировали электрофоретически (рис. 5б). Сравнение эффективности использования в реакции ОТ набора случайных праймеров и специфичного праймера показывает преимущества последнего, что объясняется направленным синтезом кДНК нужной специфичности и отсутствием синтеза нецелевых продуктов. Таким образом, найдены условия последовательных стадий ОТ и ПЦР, в которых выход ДНК-мишени увеличен в несколько раз по сравнению со стандартными методиками с использованием наборов праймеров случайного состава.
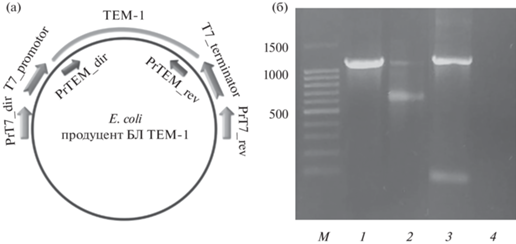
Определение мРНК БЛ ТЕМ-1 в лабораторном штамме E. coli. Для получения стандартного образца мРНК использовали плазмиду pET24 BL21 с клонированным геном blaТЕМ-1, выделенную из клеток штамма E. coli – продуцентов рекомбинантной БЛ ТЕМ-1. Методом ПЦР амплифицировали продукт размером 1140 п. н., включающий полноразмерный ген blaТЕМ-1, совместно с участками промотора и терминатора бактериофага Т7 (рис. 6а). Этот продукт ПЦР использовали в качестве матрицы для синтеза специфичной мРНК данной БЛ в реакции транскрипции in vitro. Размер и чистоту полученного образца мРНК оценивали методом электрофореза в агарозном геле (рис. 6б). Основным продуктом реакции являлась одноцепочечная мРНК БЛ ТЕМ-1 размером 1095 н. Образец содержал также примесь ДНК-матрицы размером 1140 п.н., которую удаляли обработкой ДНКазой, полноту удаления контролировали методом электрофореза. Очищенный от примесей ДНК образец использовали для приготовления стандартных образцов мРНК БЛ ТЕМ-1 с различными концентрациями (от 16 500 до 0.0016 фмоль/мл).
Рис. 6.
(а): Строение фрагмента плазмиды pET 24 BL21, использованного для получения мРНК БЛ ТЕМ-1. (б): Электрофореграммы продуктов отдельных стадий синтеза мРНК БЛ ТЕМ-1: М – смесь ДНК-маркеров известной длины; 1 – двухцепочечный ПЦР-продукт размером 1140 п. о. – фрагмент плазмиды pET 24 BL 21, включающий ген БЛ ТЕМ-1 c участками промотора и терминатора бактериофага Т7; 2 – одноцепочечная РНК БЛ ТЕМ-1 размером 1095 о., полученная в реакции транскрипции in vitro с примесью ДНК-матрицы размером 1140 п. о.; 3 – положительный контроль ПЦР – амплификация полноразмерного гена БЛ ТЕМ-1; 4 – отрицательный контроль ПЦР.
Градуировочная зависимость для определения мРНК БЛ ТЕМ-типа, построенная с использованием стандартных образцов, приведена на рис. 7а. Предел обнаружения мРНК БЛ ТЕМ-1 составил 0.40 ± ± 0.05 амоль/мл, диапазон определяемых концентраций – от 1.0 амоль/мл до 2000 фмоль/мл, относительное стандартное отклонение не превышало 12%. Таким образом, разработана методика определения специфичных мРНК БЛ, которая позволила количественно определять мРНК с высокими чувствительностью и точностью.
Рис.7.
(а): Градуировочная зависимость интенсивности окрашивания зон биочипа от концентрации мРНК БЛ ТЕМ-1; (б): определение концентрации мРНК ТЕМ-1 в образцах клеток лабораторного штамма E. coli; (в): определение ферментативной активности БЛ ТЕМ-1 в реакции гидролиза ампициллина в периплазматических фракциях клеток лабораторного штамма E. coli.
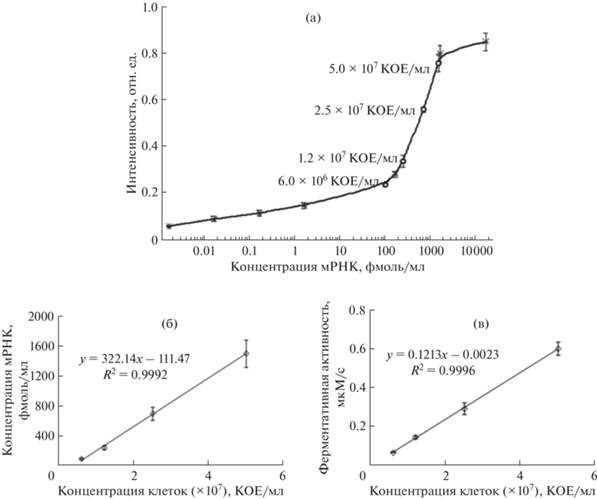
Разработанную методику применили для определения мРНК БЛ ТЕМ-1 в клетках лабораторного штамма E. сoli – продуцента данной БЛ. Из образцов бактериальных клеток с разной концентрацией (от 6 × 106 КОЕ/мл до 5 × 107 КОЕ/мл) выделили фракции общей РНК, в которых определили специфичную мРНК (рис. 7б). Для контроля экспрессии БЛ из образцов клеток E. coli с такой же концентрацией клеток были получены периплазматические фракции и в них определена ферментативная активность БЛ ТЕМ-1 в реакции гидролиза ампициллина (рис. 7в). Сравнение полученных результатов показало, что концентрация специфичной мРНК линейно зависит от концентрации клеток, что хорошо коррелирует с уровнем ферментативной активности в периплазматической фракции этих же клеток.
Определение мРНК БЛ ТЕМ-1 в клинических образцах грамотрицательных бактерий с разной чувствительностью к бета-лактамным антибиотикам. Разработанную методику применили для определения мРНК БЛ ТЕМ-1 в клинических штаммах E. coli и K. pneumoniae с разным уровнем чувствительности к бета-лактамным антибиотикам (табл. 2). Два штамма (E. coli B-1350/18 и K. pneumoniae B-1088/17) были устойчивыми к ампициллину и чувствительными к остальным бета-лактамам, штамм E. coli B-2607/18 был чувствительным ко всем бета-лактамным антибиотикам и использовался как контрольный. Наличие генов БЛ в препаратах плазмидной ДНК, выделенных из клеток клинических штаммов, определяли методом гибридизационного анализа на биочипах: у бактерий штаммов E. coli B-1350/18 и K. pneumoniae B-1088/17 обнаружили ген blaТЕМ-1, в то время как в контрольном штамме E. coli B-2607/18 гены БЛ отсутствовали (табл. 2).
Таблица 2.
Характеристика клинических штаммов грамотрицательных бактерий, использованных в работе
| Вид бактерий | Штамм | Источник выделения | МПК антибиотиков, мг/л | Плазмидно-локализован- ные гены БЛ | |||
|---|---|---|---|---|---|---|---|
| AMP | CAZ | MER | AZT | ||||
| E. coli | B-1350/18 | Моча | 256 | 0.5 | 0.25 | 0.5 | blaTEM-1 |
| K. pneumoniae | B-1088/17 | Аспират | ≥256 | 1 | 0.25 | 0.25 | blaTEM-1 |
| E. coli | B-2607/18 (контроль) | Аспират | 4 | 0.25 | 0.25 | 0.25 | – |
Выбранные клинические штаммы культивировали в присутствии разных концентраций ампициллина и в отсутствие антибиотика. Концентрации ампициллина выбрали согласно рекомендациям EUCAST: 8 мг/л – концентрация, соответствующая пороговой величине чувствительности к ампициллину у резистентных штаммов; 100 мг/л – стандартная концентрация, используемая при лечении ампициллином, 250 мг/л – повышенная концентрация антибиотика. Показано, что результаты культивирования бактерий зависят от типа штамма и концентрации антибиотика в среде культивирования (табл. 3). В отсутствие антибиотика бактерии всех исследованных штаммов выросли до одинаковой концентрации. При увеличении концентрации антибиотика клетки K. pneumoniae B-1088/17 росли только при низких концентрациях антибиотика (8 мг/л), при увеличении концентрации антибиотика от 100 мг/л клетки не росли, хотя по данным фенотипических тестов минимальная подавляющая концентрация (МПК) ампициллина для данного штамма превышала значение 256 мг/л. Клетки штамма E. coli B-1350/18 росли в присутствии всех использованных концентраций ампициллина, включая концентрацию, близкую к значению МПК.
Таблица 3.
Результаты определения концентрации клеток грамотрицательных бактерий, выращенных в присутствии различных концентраций ампициллина
| Вид бактерий | Штамм | Концентрация клеток, выращенных в среде без антибиотика, КОЕ/мл | Концентрация клеток, выращенных в присутствии ампициллина, КОЕ/мл | ||
|---|---|---|---|---|---|
| 8 мг/л | 100 мг/л | 250 мг/л | |||
| E. coli | B-1350/18 | 1 × 109 | 8 × 108 | 6 × 108 | 5 × 108 |
| K. pneumoniae | B-1088/17 | 1 × 109 | 1 × 109 | Нет роста | Нет роста |
| E. coli | B-2607/18 (контроль) | 1 × 109 | Нет роста | Нет роста | Нет роста |
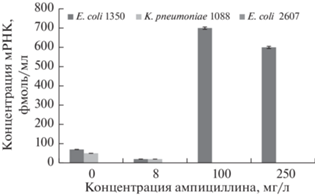
Из бактериальных культур, выросших в присутствии антибиотика, выделяли общую РНК и анализировали описанным выше методом гибридизационного анализа на биочипах. Интенсивность окрашивания специфичных зон биочипа нормировали на интенсивность окрашивания зоны положительного контроля. Результаты определения концентрации мРНК БЛ ТЕМ-1 в РНК-транскриптах клинических штаммов приведены на рис. 8. Культивирование штамма E. coli B-1350/18 в присутствии повышенных концентраций ампициллина (более 100 мг/л) индуцировало увеличение экспрессии гена blaТЕМ-1 примерно в 10 раз. Культивирование в присутствии пониженных концентраций ампициллина (8 мг/л) снижало экспрессию гена blaТЕМ-1 у обоих резистентных к ампициллину штаммов. Экспрессии гена blaТЕМ-1 у чувствительного к ампициллину штамма E. coli B-2607/18 не наблюдали.
* * *
Разработанная нами методика определения специфичных мРНК на колориметрических биочипах характеризуется высокой чувствительностью и существенно более высокой точностью по сравнению с методом ПЦР-РВ благодаря применению классического подхода биоаналитической химии – определения мРНК по градуировочной кривой, построенной с использованием стандартных образцов мРНК той же специфичности, в отличие от используемых ранее методов нормирования на результат определения мРНК референсного или контрольного гена [18, 39]. Стоит подчеркнуть, что выбор гена, экспрессия которого не зависит от условий эксперимента, представляет сложную задачу, использование нескольких контрольных генов существенно усложняет анализ и обработку результатов [40, 41].
К преимуществам использованного нами подхода, основанного на анализе стандартных образцов, относится размер молекул мРНК, соответствующий полноразмерному гену БЛ. Ранее показано, что использование в качестве стандартов ДНК конструкций, представляющих собой небольшой фрагмент определяемого гена, не позволяет правильно определять количество мРНК в транскриптомном анализе бактерий из-за различий в эффективности синтеза меченых мРНК разного размера [42]. Другим преимуществом использования стандартных образцов мРНК, проходящих все стадии анализа вместе с исследуемыми образцами, является отсутствие необходимости определения эффективности отдельных стадий пробоподготовки (ОТ и ПЦР), что требуется при количественном определении экспрессии генов методом ПЦР-РВ [18, 43]. Точное определение эффективности этих стадий часто невозможно, так как она зависит от концентрации мРНК, структуры праймеров и выбранных генов.
Мы показали применимость разработанного подхода для определения экспрессии генов БЛ в РНК-транскриптах клинических штаммов семейства Enterobacteriaceae, выращенных в присутствии различных концентраций ампициллина, которые соответствуют концентрациям, используемым в клинической практике. Следует отметить высокую точность результатов определения мРНК в широком диапазоне концентраций, достигнутую благодаря использованию стандартных образцов для построения градуировочной зависимости. Таким образом, использование новой методики позволяет перейти от полуколичественного анализа транскриптов генов, принятому в ПЦР-РВ, к количественному, что необходимо при изучении молекулярных механизмов индукции генов антибиотикорезистентности у бактерий с множественной устойчивостью к антибиотикам.
Размещение биочипов в лунках 96-луночного планшета позволяет существенно увеличить производительность методики. В данной работе определяли мРНК БЛ одного типа, однако используемый принцип мультианализа с размещением олигонуклеотидных зондов разной специфичности позволит определять на одном биочипе до десяти типов транскриптов генов. В настоящее время продолжительность анализа составляет около 7 ч, в дальнейшем она может быть сокращена при совмещении стадий ОТ и ПЦР и автоматизации процессов отмывки.
Таким образом, разработанная методика определения специфичных мРНК БЛ в РНК-транскриптах бактериальных клеток может быть использована для контроля экспрессируемых БЛ мультирезистентными к антибиотикам штаммами в клинических лабораториях, а также для изучения механизмов формирования устойчивости бактерий к антибиотикам. Дальнейшая оптимизация методики будет состоять в объединении стадий ОТ и ПЦР и увеличении количества определяемых специфичных мРНК БЛ.
Работа выполнена при финансовой поддержке Госзадания МГУ им. М.В. Ломоносова по теме АААА-А21-121011290089-4 и РФФИ (Грант 19-34-50071).
Список литературы
Eichenberger E.M., Thaden J.T. Epidemiology and mechanisms of resistance of extensively drug resistant gram-negative bacteria // Antibiotics (Basel). 2019. V. 8. № 2. P. 37.
World Health Organization. Antibiotic resistance. https://www.who.int/news-room/fact-sheets/detail/antibiotic-resistance (01.07.2021).
Mulani M.S., Kamble E.E., Kumkar S.N., Tawre M.S., Pardesi K.R. Emerging strategies to combat ESKAPE pathogens in the era of antimicrobial resistance: A review // Front. Microbiol. 2019. V. 10. P. 539.
Wright G.D. Bacterial resistance to antibiotics: Enzymatic degradation and modification // Adv. Drug Deliv. Rev. 2005. V. 57. № 10. P. 1451.
Егоров А.М., Уляшова М.М., Рубцова М.Ю. Бактериальные ферменты и резистентность к антибиотикам // Acta naturae. 2018. Т. 10. № 4. С. 33. (Egorov A.M., Ulyashova M.M., Rubtsova M.Y. Bacterial enzymes and antibiotic resistance // Acta naturae. 2018. V. 10. № 4. P. 33.)
Klein E.Y., Van Boeckel T.P., Martinez E.M., Pant S., Gandra S., Levin S.A., Goossens H., Laxminarayan R. Global increase and geographic convergence in antibiotic consumption between 2000 and 2015 // Proc. Nat. Acad. Sci. USA. 2018. V. 115. № 15. P. 3463.
King D.T., Sobhanifar S., Strynadka N.C.J. The Mechanisms of resistance to β-lactam antibiotics / Handbook of Antimicrobial Resistance. 2017. P. 177.
Bush K. Past and present perspectives on β-lactamases // Antimicrob. Agents Chemother. 2018. V. 62. № 10. Article e01076-18.
Edwards T., Williams C., Teethaisong Y., Sealey J., Sasaki S., Hobbs G., Cuevas L.E., Evans K., Adams E.R. A highly multiplexed melt-curve assay for detecting the most prevalent carbapenemase, ESBL, and AmpC genes // Diagn. Microbiol. Infect. Dis. 2020. V. 97. № 4. Article 115076.
Dallenne C., Da Costa A., Decré D., Favier C., Arlet G. Development of a set of multiplex PCR assays for the detection of genes encoding important beta-lactamases in Enterobacteriaceae // J. Antimicrob. Chemother. 2010. V. 65. № 3. P. 490.
Band V.I., Weiss D.S. Heteroresistance: A cause of unexplained antibiotic treatment failure? // PLoS Pathog. 2019. V. 15. № 6. Article e1007726.
Andersson D.I., Nicoloff H., Hjort K. Mechanisms and clinical relevance of bacterial heteroresistance // Nat. Rev. Microbiol. 2019. V. 17. № 8. P. 479.
Kjeldsen T.S.B., Overgaard M., Nielsen S.S., Bortolaia V., Jelsbak L., Sommer M., Guardabassi L., Olsen J.E. CTX-M-1 β-lactamase expression in Escherichia coli is dependent on cefotaxime concentration, growth phase and gene location // J. Antimicrob. Chemother. 2015. V. 70. № 1. P. 62.
Maurya A.P., Chanda D.D., Bora D., Talukdar A.D., Chakravarty A., Bhattacharjee A. Transcriptional response of multiple ESBL gene within Escherichia coli under oxymino-cephalosporin stress // Microb. Drug Resist. 2017. V. 23. № 2. P. 133.
Balabanian G., Rose M., Manning N., Landman D., Quale J. Effect of porins and blaKPC expression on activity of imipenem with relebactam in Klebsiella pneumoniae: Can antibiotic combinations overcome resistance? // Microb. Drug Resist. 2018. V. 24. № 7. P. 877.
Lietard J., Ameur D., Damha M.J., Somoza M.M. High-density RNA microarrays synthesized in situ by photolithography // Angew Chem. Int. Ed. Engl. 2018. V. 57. № 46. P. 15257.
Wang Y., Quc J., Baa Q., Donga J., Zhang L., Zhanga H., Wue A., Wangf D., Xia Z., Peng D., Shuf Y., Caoc B., Jiang T. Detection and typing of human-infecting influenza viruses in China by using a multiplex DNA biochip assay // J. Virol. Methods. 2016. V. 234. P. 178.
Bustin S., Nolan T. Talking the talk, but not walking the walk: RT-qPCR as a paradigm for the lack of reproducibility in molecular research // Eur. J. Clin. Invest. 2017. V. 47. № 10. P. 756.
Hughes T.R., Marton M.J., Jones A.R., Roberts C.J., Stoughton R., Armour C.D., Friend S.H. Functional discovery via a compendium of expression profiles // Cell. 2000. V. 102. № 1. P. 109.
Feng J., Billal D.S., Lupien A., Racine G., Winstall E., Légaré D., Ouellette M. Proteomic and transcriptomic analysis of linezolid resistance in streptococcus pneumoniae // J. Proteome Res. 2011. V. 10. № 10. P. 4439.
Al-Rubaye D.S., Henihan G., Al-Abasly A.K.A., Seagar A.L., Al-Attraqchi A.A.F., Schulze H., Hashim D.S., Kamil J.K., Laurenson I.F., Bachmann T.T. Genotypic assessment of drug-resistant tuberculosis in Baghdad and other Iraqi provinces using low-cost and low-density DNA microarrays // J. Med. Microbiol. 2016. V. 65. № 2. P. 114.
Moure R., Tudó G., Medina R., Vicente E., Caldito J.M., Codina M.G., Coll P., Español M., Gonzalez-Martin J., Rey-Jurado E., Salvadó M., Tórtola M.T., Alcaide F. Detection of streptomycin and quinolone resistance in mycobacterium tuberculosis by a low-density DNA array // Tuberculosis (Edinb). 2013. V. 93. № 5. P. 508.
Naas T., Cuzon G., Truong H., Bernabeu S., Nordmann P. Evaluation of a DNA microarray, the check-points ESBL/KPC array, for rapid detection of TEM, SHV, and CTX-M extended-spectrum beta-lactamases and KPC carbapenemases // Antimicrob. Agents Chemother. 2010. V. 54. № 8. P. 3086.
Rubtsova M.Yu., Ulyashova M.M., Edelstein M.V., Egorov A.M. Oligonucleotide microarrays with horseradish peroxidase-based detection for the identification of extended-spectrum β-lactamases // Biosens. Bioelectron. 2010. V. 26. № 4. P. 1252.
Рубцова М.Ю., Уляшова М.М., Поболелова Ю.И., Преснова Г.В., Егоров А.М. Биочип для одновременной идентификации генов бета-лактамаз и карбапенемаз, обуславливающих устойчивость бактерий к бета-лактамным антибиотикам // Прикладная биохимия и микробиология. 2020. Т. 56. № 2. С. 1. (Rubtsova M.Yu., Ulyashova M.M., Pobolelova Yu.I., Presnova G.V., Egorov A.M. Biochip for simultaneous identification of beta-lactamase and carbapenemase genes conferring the bacterial resistance to beta-lactam antibiotics // Appl. Biochem. Microbiol. 2020. V. 56. № 2. P. 130.)
Díaz-González M., Pablo Salvador J., Bonilla D., Pilar Marco M., Fernández-Sánchez C., Baldi A. A microfluidic device for the automated electrical readout of low-density glass-slide microarrays // Biosens. Bioelectron. 2015. V. 74. P. 698.
Palzkill T. Structural and mechanistic basis for extended-spectrum drug-resistance mutations in altering the specificity of TEM, CTX-M, and KPC β-lactamases // Front. Mol. Biosci. 2018. V. 5. P. 1.
Pimenta A.C., Fernandes R., Moreira I.S. Evolution of drug resistance: insight on TEM β-lactamases structure and activity and β-lactam antibiotics // Mini Rev. Med. Chem. 2014. V. 14. № 2. P. 111.
Singh N.S., Singhal N., Kumar M., Virdi J.S. Exploring the genetic mechanisms underlying amoxicillin-clavulanate resistance in waterborne Escherichia coli // Infect. Genet. Evol. 2021. V. 90. Article 104767.
Hubbard A.T.M., Mason J., Roberts P., Parry C.M., Corless C., van Aartsen J., Howard A., Bulgasim I., Fraser A.J., Adams E.R., Roberts A.P., Edwards T. Piperacillin/tazobactam resistance in a clinical isolate of Escherichia coli due to IS26-mediated amplification of blaTEM-1B // Nat. Commun. 2020. V. 11. № 1. P. 4915.
Grigorenko V.G., Andreeva I.P., Rubtsova M.Yu., Deygen I.M., Antipin R.L., Majouga A.G., Egorov A.M., Beshnova D.A., Kallio J., Hackenberg C., Lamzin V.S. Novel non-β-lactam inhibitor of β-lactamase TEM-171 based on acylated phenoxyaniline // Biochimie. 2017. V. 132. P. 45.
https://www.eucast.org/fileadmin/src/media/PDFs/ EUCAST_files/Breakpoint_tables/v_10.0_Breakpoint_Tables.pdf (01.07.2021).
Birnboim H.C., Doly J. A rapid alkaline lysis procedure for screening recombinant plasmid DNA // Nucleic Acids Res. 1979. V. 7. P. 1513.
Greisen K., Loeffelholz M., Purohit A., Leong D. PCR primers and probes for the 16S rRNA gene of most species of pathogenic bacteria, including bacteria found in cerebrospinal fluid // J. Clin. Microbiol. 1994. V. 32. № 2. P. 335.
Nossal N.G., Heppel L.A. The release of enzymes by osmotic shock from Escherichia coli in exponential phase // J. Biol. Chem. 1966. V. 241. № 13. P. 3055.
Schenborn E.T., Mierendorf R.C. Jr. A novel transcription property of SP6 and T7 RNA polymerases: Dependence on template structure // Nucleic Acids Res. 1985. V. 13. № 17. P. 6223.
Verma I.M. Reverse transcriptase / The Enzymes / Ed. Boyer P.D. N.Y.: Academic Press Inc., 1981. V. 14. P. 87.
Поболелова Ю.И., Уляшова М.М., Рубцова М.Ю., Егоров А.М. Мультиплексная ПЦР для совместной амплификации генов бактериальных ферментов карбапенемаз молекулярных классов А, В и D // Биохимия. 2014. Т. 79. № 6. С. 718. (Pobolelova Yu.I., Ulyashova M.M., Rubtsova M.Yu., Egorov A.M. Multiplex PCR for joint amplification of carbapenemase genes of molecular classes A, B, and D // Biochemistry (Moscow). 2014. V. 79. № 6. P. 566.)
Livak K.J., Schmittgen T.D. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-delta delta C(T)) method // Methods. 2001. V. 25. № 4. P. 402.
Bustin S.A. The reproducibility of biomedical research: Sleepers awake // Biomol. Detect. Quantif. 2014. V. 2. P. 35.
Bustin S.A., Benes V., Garson J., Hellemans J., Huggett J., Kubista M., Mueller R., Nolan T., Pfaffl M.W., Shipley G. et al. The need for transparency and good practices in the qPCR literature // Nat. Methods. 2013. V. 10. P. 1063.
Курбатов Л.К., Згода В.Г. Использование ДНК-матрицы в качестве контроля при проведении транскриптомного анализа на стандартных микрочипах Agilent Technologies // Биомедицинская химия. 2016. Т. 62. № 6. С. 715. (Kurbatov L.K., Zgoda V.G. A possibility to use the DNA-based probes as internal standards for Agilent Technologies microarray transcriptomic analysis // Biomed. Khim. 2016. V. 62. № 6. P. 715.)
Bustin S.A. Quantification of mRNA using real-time reverse transcription PCR (RT-PCR): trends and problems // J. Mol. Endocrinol. 2002. V. 29. P. 23.
Дополнительные материалы отсутствуют.
Инструменты
Журнал аналитической химии