Биоорганическая химия, 2021, T. 47, № 3, стр. 327-340
Матричная сборка днк-наноструктур из разветвленных олигонуклеотидов
А. А. Фокина 1, 2, Ю. Е. Полетаева 3, Е. А. Буракова 1, 2, А. Ю. Бакулина 1, 4, Т. С. Зацепин 5, 6, Е. И. Рябчикова 1, 3, Д. А. Стеценко 1, 2, *
1 ФГАОУВО “Новосибирский государственный университет”
630090 Новосибирск, ул. Пирогова, 2, Россия
2 ФГБУН Федеральный исследовательский центр “Институт цитологии и генетики” СО РАН
630090 Новосибирск, просп. акад. Лаврентьева, 10, Россия
3 ФГБУН “Институт химической биологии и фундаментальной медицины” СО РАН
630090 Новосибирск, просп. акад. Лаврентьева, 8, Россия
4 ФБУН ГНЦ ВБ “Вектор” Роспотребнадзора
630559 р.п. Кольцово, Новосибирская обл., Россия
5 Сколковский институт науки и технологий
121205 Московская обл., Инновационный центр “Сколково”, Большой бульвар, 30, стр. 1, Россия
6 Московский государственный университет им. М.В. Ломоносова
119991 Москва, Ленинские горы, 1, стр. 3, Россия
* E-mail: d.stetsenko@nsu.ru
Поступила в редакцию 17.10.2020
После доработки 26.11.2020
Принята к публикации 20.12.2020
Аннотация
Ранее нами был предложен способ построения трехмерных наноконструкций на основе нуклеиновых кислот, основанный на матричной сборке с использованием в качестве матрицы и строительных блоков разветвленных олигонуклеотидов, в состав которых включены линкеры ненуклеотидной природы, в частности точки разветвления, соединяющие две или три олигонуклеотидные цепи. В данной работе мы осуществили синтез разветвленных олигонуклеотидных матриц (для сборки ДНК-тетраэдра и ДНК-куба) и разветвленного олигонуклеотидного строительного блока, а также предприняли попытку матричной сборки ДНК-тетраэдра.
ВВЕДЕНИЕ
Одним из наиболее динамично развивающихся направлений химии нуклеиновых кислот выступает ДНК-нанотехнология [1, 2], которая рассматривает молекулу ДНК как уникальный материал для конструирования разнообразных наноархитектур [3], что открывает возможность создания на основе ДНК динамических наноустройств [4], наномашин [5] и нанороботов [6, 7]. В основе данного применения лежит присущая полинуклеотидам способность к комплементарному уотсон-криковскому связыванию, а также к образованию структур более высокого порядка [3]. Предложенный в 2006 г. метод ДНК-оригами [8], состоящий в “укладке” (folding) протяженной одноцепочечной молекулы ДНК, например, кольцевого генома фага М13, с помощью сотен олигонуклеотидов-“скрепок”, комплементарных уникальным участкам ДНК, за прошедшие полтора десятилетия развился в практическую технологию формирования двумерных и трехмерных ДНК-наноструктур с потенциалом применения в биомедицине для биоанализа [9], создания биосенсоров [10], биоимиджинга [11], доставки лекарств [12, 13] и иммунотерапии [14]. В основе большинства современных вариантов ДНК-оригами лежит сочетание компьютерных алгоритмов программируемой самосборки молекул ДНК [15, 16] с методами манипуляции протяженными ДНК из арсенала молекулярной биологии [17]. Однако использованию методов химической модификации участков нуклеиновых кислот при рациональном конструировании наноархитектур, на наш взгляд, все еще уделяется недостаточно внимания [18]. Это приводит к усложнению и увеличению размеров и массы ДНК-модулей для сборки наноархитектур, что может вызывать дополнительные проблемы при проникновении в клетку [19]. Представляется вероятным, что объемные ДНК-модули могут быть заменены относительно простыми синтетическими модифицированными олигонуклеотидами, в частности разветвленными олигонуклеотидами. Это позволило бы расширить интервал размеров получаемых ДНК-наноструктур в направлении нижней границы диапазона (~10 нм). Таким образом, дополнение метода ДНК-оригами приемами из арсенала твердофазного олигонуклеотидного синтеза и химической модификации нуклеиновых кислот с целью создания новых технологий конструирования трехмерных нанообъектов и функциональных наноустройств представляется актуальной задачей.
В ранее опубликованной работе мы предложили способ конструирования дискретных многогранников на основе нуклеиновых кислот (“энкаэдров”) по принципу матричной сборки из разветвленных олигонуклеотидных блоков, формирующих вершины и ребра энкаэдра, с использованием в качестве матрицы (каркаса) разветвленного олигонуклеотида (РО), структура которого определяется формой целевого многогранника, например, тетраэдра, куба, октаэдра и т.п. [20]. Наиболее подходящим способом получения соответствующих РО представляется комбинация автоматизированного твердофазного синтеза (с использованием ненуклеозидных фосфитамидных реагентов различной функциональности) с постсинтетической конъюгацией в растворе, например, при помощи методов клик-химии [21].
Ранее нами был обоснован расчетами выбор последовательностей олигонуклеотидов для конструирования разветвленных олигонуклеотидных матриц (РОМ) для сборки ДНК-тетраэдра, ДНК-куба и ДНК-аналога фуллерена С24, а также разветвленного олигонуклеотидного строительного блока (РОСТ), общего для всех этих топологий [20]. Был определен набор необходимых ненуклеозидных разветвляющих и модифицирующих реагентов для получения матриц и разветвленного строительного блока.
Настоящая работа была сосредоточена на решении двух основных задач: 1) получение разветвленных олигонуклеотидов (РО), служащих для построения олигонуклеотидных матриц для сборки ДНК-тетраэдра и ДНК-куба, и разветвленного олигонуклеотидного строительного блока (РОСТ), общего для обоих многогранников; 2) практическая отработка подходов к конструированию конкретных энкаэдров (на примере ДНК-тетраэдра) на основе направленной самосборки разветвленных олигонуклеотидных блоков на каркасе разветвленной олигонуклеотидной матрицы (РОМ).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Простейшей моделью для отработки условий матричной сборки энкаэдров служит ДНК-тетраэдр. Предложенная последовательность операций матричной сборки ДНК-тетраэдра изложена в работе Bakulina et al. [20]. В качестве “четырехвалентной” (т.е. формирующей четыре вершины будущего тетраэдра) разветвленной олигонуклеотидной матрицы (РОМ-1) для сборки ДНК-тетраэдра выступает димер разветвленного олигонуклеотида Y (“вилки”), формирующийся за счет самокомплементарного олиго-2'-O-метилрибонуклеотида ОН1 и содержащий четыре одноцепочечных олигодезоксирибонуклеотида ОН2 (рис. 1а). Первый шаг сборки – гибридизация матрицы РОМ-1 с разветвленным олигонуклеотидным строительным блоком – “звездочкой” РОСТ-1 (рис. 1е), формирующая четыре вершины тетраэдра, с образованием дуплексов ОН2:ОН3.
Рис. 1.
Структуры и последовательности разветвленных олигонуклеотидов (РО), полученных на первом этапе работы: (а) – “вилка” Y – мономер для сборки “четырехвалентной” разветвленной олигонуклеотидной матрицы (РОМ-1); (б) – 5-азидогексаноильный и (в) – 1-гексинильный разветвленные олигонуклеотиды (РО-азид и РО-гекс соответственно); (г) – побочный продукт, образующийся в результате неполного протекания клик-реакции циклоприсоединения CuAAC лишь по одной из двух 1-гексинильных групп РО-гекс; (д) – мономер для сборки “восьмивалентной” разветвленной олигонуклеотидной матрицы РОМ-2; (е) – “звездочка” – разветвленный олигонуклеотидный строительный блок (РОСТ-1). Обозначения: Ŷ – остаток симметричного удвоителя (symmetric doubler); Ψ – остаток симметричного утроителя (trebler); р – фосфодиэфирная группа –P(=O)(–O–)–; ОН – олигонуклеотид; РЭ – размыкаемый элемент, вертикальной стрелкой указана расщепляемая дисульфидная связь; Гекс – остаток 1-гексина; Азид – 6-азидогексаноильная группа; Tri – остаток 1,2,3-триазола с заместителями в положениях 1 и 5 (слева направо); горизонтальной стрелкой указано направление последовательности “обратного” олигодезоксирибонуклеотида ОН3 – от 5'- к 3'‑концу. Дезоксирибонуклеотиды обозначены прописными буквами, 2'-O-метилрибонуклеотиды – курсивом, последовательность ОН3, собранная из “обратных” 5'-фосфитамидов в направлении 5'–3', подчеркнута; опущены префиксы d и r для обозначения олигодезоксирибонуклеотидов и олиго-2'-О-метилрибонуклеотидов соответственно.

В результате гибридизации самокомплементарных олигодезоксирибонуклеотидов ОН4 из состава “звездочки” РОСТ-1 (рис. 1е) образуются ребра тетраэдра, при этом формируется пирамидальная супрамолекулярная двуцепочечная ДНК-конструкция в виде ДНК-тетраэдра, вершины которого соединены внутренним каркасом из матрицы РОМ-1. В качестве побочных продуктов возможно получение изомерных конструкций, имеющих отличную от тетраэдра топологию.
Для конструирования матрицы РОМ-1 был получен разветвленный олигонуклеотид Y (рис. 1а). Как следует из анализа электрофореграмм денатурирующего и нативного гель-электрофореза, матрица РОМ-1 образуется путем димеризации “вилки” Y за счет аутогибридизации самокомплементарного олиго-2'-O-метилрибонуклеотида ОН1 (рис. 2).
Рис. 2.
(а) – Электрофоретический анализ олигонуклеотидов в 20%-ном ПААГ, содержащем 7 М мочевину, с окрашиванием красителем Stains-All. Дорожки: 1 – “вилка” Y после 2 мин прогрева при 95°С и быстрого нанесения на гель под напряжением; 2 – маркер длины D157 (одноцепочечный олигодезоксирибонуклеотид длиной 34 нт); 3 – “вилка” Y (0.1 о.е.); 4 – “вилка” Y (0.05 о.е.). ВР – краситель бромфеноловый синий; (б) – нативный электрофорез в 15%-ном ПААГ, окрашивание красителем Stains-All. Дорожки: 1 – D157 после 2 мин прогрева при 95°С и быстрого нанесения на гель под напряжением; 2 – “вилка” Y в 1 M NaCl; 3 – “вилка” Y без 1 M NaCl; 4 – “вилка” Y без 1 M NaCl после 2 мин прогрева при 95°С с последующей инкубацией 10 мин при 0°С.

Для матричной сборки ДНК-куба требуется “восьмивалентная” разветвленная матрица РОМ-2, формирующая восемь вершин куба (например, рис. 1д). Попытка прямого твердофазного синтеза олигонуклеотида, соответствующего данной топологии матрицы, не увенчалась успехом из-за низкого выхода полноразмерного продукта после проведения двух конденсаций симметричного удвоителя (Ŷ) подряд. Поэтому было принято решение конструировать матрицу РОМ-2 с помощью постсинтетической конъюгации в растворе с использованием одного из наиболее популярных подходов клик-химии [21], а именно реакции катализируемого Сu(I) [2+3] циклоприсоединения азидов и алкинов (CuAAC) [22, 23].
Для получения мономера матрицы РОМ-2 (рис. 1д) были синтезированы два разветвленных олигонуклеотида: РО-азид, содержащий одну 6‑азидогексаноильную группу, введенную по 3'-концу ОН2 через аминолинкер С7 (рис. 1б), и РО-гекс, содержащий два остатка 1-гексина, введенные посредством конденсации коммерческого 1-гексинильного фосфитамида вслед за симметричным удвоителем на 5'-конце ОН1 (рис. 1в).
Конъюгация РО-азид и РО-гекс (4 : 1) по реакции CuAAC была осуществлена по методике, разработанной ранее [24]. Однако по данным гель-электрофореза (данные не приведены) основным продуктом реакции выступал монозамещенный конъюгат РО-гекс с одной молекулой РО-азид (рис. 1г) наряду с множественными полосами, вероятно, соответствующими деградации олигонуклеотидов при воздействии солей меди(I) и (II) даже в присутствии лиганда THPTA (трис-гидроксипропилтриазолилметиламин).
Чтобы избежать деградации олигонуклеотида в присутствии солей меди, было решено использовать для синтеза нового варианта матрицы РОМ-2′ реакцию “безмедного клика”, а именно некаталитическую реакцию алкин-азидного циклоприсоединения, промотируемую напряжением в производном циклооктина (SPAAC) [25–28]. Для решения этой задачи был синтезирован новый набор разветвленных олигонуклеотидов (рис. 3). Получение бициклононинового (BCN) производного (рис. 3г) протекало с высоким выходом из соответствующего 3'-аминогексильного предшественника и коммерческого активированного карбоната бицикло[6.1.0]нонина-4. Однако с очисткой диазидного компонента от примеси соответствующего монопроизводного возникли трудности. Полученные реакционные смеси не удавалось эффективно разделить ни с помощью офВЭЖХ, ни с помощью электрофореза в ПААГ. После перебора нескольких вариантов синтеза наилучший результат показало бис-азидоацетильное производное РО-азид' (рис. 3б), полученное с помощью коммерческого аминолинкера с последующей обработкой N-гидроксисукцинимидным эфиром азидоуксусной кислоты, приготовленным in situ.
Рис. 3.
Структуры и последовательности разветвленных олигонуклеотидов (РО), полученных на втором этапе работы: (а) – “вилки” Y – мономера для сборки “четырехвалентной” разветвленной олигонуклеотидной матрицы РОМ-1' для ДНК-тетраэдра; (б) – PO-азид' – азидного фрагмента “восьмивалентной” матрицы (РОМ-2′) для сборки ДНК-куба; (в) – РО-С6-амино – исходного материала для синтеза бициклоциклононинового (BCN) фрагмента матрицы РОМ-2′ (РО-BCN) (г); (д) – “звездочки” – разветвленного олигонуклеотидного строительного блока РОСТ-2′. Обозначения: Ŷ – остаток симметричного удвоителя (symmetric doubler); Ψ – остаток симметричного утроителя (trebler); р – фосфодиэфирная группа –P(=O)(–O–)–; ОН – олигонуклеотид; РЭ – размыкаемый элемент, вертикальной стрелкой указана расщепляемая дисульфидная связь; С6 Aмино – 6-аминогексильная группа; Aмино – остаток аминолинкера; BCN – остаток бицикло[6.1.0]нонина-4; Aзид' – азидоацетильная группа; Т* – остаток тимидина с фотолабильной защитной группой (NPOM). Дезоксирибонуклеотиды обозначены прописными буквами, 2'-O-метилрибонуклеотиды – курсивом, последовательность ОН3, собранная из “обратных” 5'-фосфитамидов в направлении 5'–3', подчеркнута, направление последовательности указано горизонтальной стрелкой; опущены префиксы d и r для обозначения олигодезоксирибонуклеотидов и олиго-2'-О-метилрибонуклеотидов соответственно.

Предпринятый синтез мономера матрицы РОМ-2' из РО-азид' и PO-BCN в соотношении 1 : 4 по реакции “безмедного клика” (SPAAC) (рис. 4) протекал значительно медленнее, чем каталитическая версия (CuAAC). В результате после 72 ч при 40°С, что значительно жестче условий, принятых в работе Jawalekar et al. [28] (комнатная температура, 12 ч), на электрофореграмме можно было зарегистрировать в основном образование монозамещенного продукта и лишь следовых количеств целевого дизамещенного олигонуклеотида (рис. 5). Таким образом, и этот путь конструирования “восьмивалентной матрицы” РОМ-2′ не привел к успеху, и отработка условий для матричной сборки ДНК-куба требует дальнейших исследований.
Рис. 4.
Схема синтеза “восьмивалентной” матрицы РОМ-2' для ДНК-куба с помощью реакции “безмедного клика” (SPAAC) [14] между разветвленными олигонуклеотидами, содержащими азидоацетильные группы (Aзид′) и остатки бицикло[6.1.0]нонина-4 (BCN), между которыми происходит реакция [2+3]циклоприсоединения с образованием 1,4,5-тризамещенного 1,2,3-триазола. Обозначения у фигурных скобок слева соответствуют рис. 5.
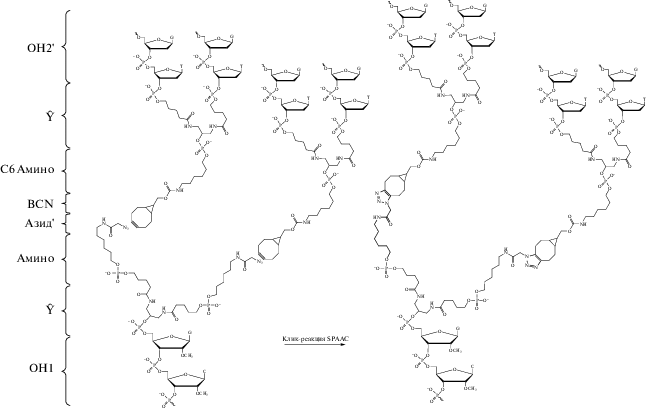
Рис. 5.
Электрофореграмма реакции “безмедного” клика (SPAAC). Обозначения полос: 1 – Азид'; 2 – BCN; 3, 4 – продукты циклоприсоединения моно- (основной) и бис- (следы) соответственно.

Далее был осуществлен синтез разветвленных олигонуклеотидных строительных блоков – “звездочек” РОСТ-1 и РОСТ-2 (рис. 1е и рис. 3д соответственно), которые, по нашим расчетам, могут использоваться для сборки как ДНК-тетраэдра, так и ДНК-куба [20]. Наибольшие проблемы вызвало введение размыкаемого элемента, а именно дисульфидной связи, с помощью коммерческого дисульфидного фосфитамида. Выход олигонуклеотида после 6 мин конденсации свежеприготовленного 0.15 М раствора дисульфидного фосфитамида в абсолютном ацетонитриле не превышал 40% по офВЭЖХ. Увеличение времени конденсации до 15 мин не привело к повышению выхода целевого компонента. При синтезе “звездочек” неудовлетворительная конденсация дисульфидного фосфитамида вызывала наибольшие потери продукта.
С использованием матрицы РОМ-1 и разветвленного олигонуклеотидного блока РОСТ-1 в соотношении 1 : 4 была предпринята матричная сборка ДНК-тетраэдра. Анализ реакционной смеси с помощью нативного гель-электрофореза в агарозе показал наличие продукта, соответствующего по молекулярной массе расчетной массе ДНК-тетраэдра (рис. 6). В то же время в смеси присутствовало значительное количество полимерных продуктов большой молекулярной массы, не обладающих подвижностью в геле. Образование высокомолекулярных продуктов можно объяснить неконтролируемой полимеризацией “звездочки” РОСТ-1 за счет гибридизации самокомплементарных олигонуклеотидов ОН4 (рис. 1е).
Рис. 6.
Анализ продуктов матричной сборки ДНК-тетраэдра с помощью электрофореза в 1%-ном агарозном геле. Дорожки: 1 – ДНК-маркеры 100–1000 нт; 2 – реакционная смесь после сборки ДНК-тетраэдра из РОМ-1 и РОСТ-1 (4 экв.); а – интенсивная полоса с размером между 200 и 400 нт; б – высокомолекулярные продукты, не вошедшие в гель (предположительно агрегаты конкатемерной природы).

С помощью просвечивающей электронной микроскопии было показано присутствие наряду с полимерными продуктами дискретных продуктов с линейными размерами <50 нм, которые могут соответствовать агрегатам “звездочки” РОСТ-1 (рис. 7).
Рис. 7.
Микрофотографии образцов олигонуклеотидов, полученные методом просвечивающей электронной микроскопии: (а) – РОМ-1; (б) – РОСТ; (в) и (г) – продукты матричной сборки ДНК-тетраэдра из РОМ-1 и РОСТ-1 в соотношении 1 : 4; [РОСТ-1] = 0.3 мкМ. Негативное контрастирование ацетатом уранила.

Полученные результаты убедительно продемонстрировали необходимость использования защитной группы, которая препятствовала бы преждевременной аутогибридизации “звездочки”. В качестве защитной группы нами была использована фотолабильная 1-(2-нитропиперонил)-этоксиметильная группа (NPOM) в положении 3 остатка тимидина, предложенная ранее для сходных целей Deuters et al. [29–31]. Исследование скорости удаления NPOM-группы с остатка тимидина в составе модельного олигонуклеотида D574 при облучении УФ-светом (λ = 365 нм) показало (табл. 1), что для >90%-ного отщепления в оптимизированных условиях требуется не менее 4 ч (рис. 8, дорожки 4 и 5). Длина волны была подобрана с целью минимизации возможного образования фотодимера тимидина и других нежелательных фотопродуктов. Как видно из электрофореграммы на рис. 8 (дорожки 1–3), облучение “звездочки” РОСТ-2 (олигонуклеотид D570), содержащей по три остатка NPOM-T(Т*) на ветвь (рис. 3д), в Трис-ацетатном буфере (ТАЕ, рН 7.6) УФ-светом (λ = 365 нм) в течение 5 ч и последующая обработка 100 мМ дитиотреитом (DTT) в течение 1 ч для разрыва дисульфидной связи, как и ожидалось, приводили к снижению электрофоретической подвижности олигонуклеотида.
Таблица 1.
Удаление фотолабильной 1-(2-нитропиперонил)-этоксиметильной группы NPOM с модельного олигонуклеотида 5'-TGTTT*GGCGC-3' (D574) при УФ-облучении (λ = 365 нм)
| Время облучения, ч | Степень удаления,%** |
|---|---|
| 1 | 38 |
| 3 | 76 |
| 4 | 88 |
| 4*** | 91 |
Рис. 8.
Электрофореграмма олигонуклеотидов: D570 (“звездочка” РОСТ-2, рис. 5д) до (дорожка 1) и после (дорожка 2) облучения УФ-светом (λ = 365 нм) и после обработки DTT (дорожка 3); D574 до (дорожка 4) и после (дорожка 5) облучения УФ-светом (λ = 365 нм). Условия: 15%-ный ПААГ, 8 М мочевина, 1× ТВЕ, pH 8.3, окрашено красителем Stains-All. ВР – краситель бромфеноловый синий.

Сборку ДНК-тетраэдра на основе матрицы РОМ-1′ и “звездочки” РОСТ-2 (рис. 3а и 3д соответственно) проводили поэтапно в следующей последовательности: отжиг – инкубация – УФ-облучение (λ = 365 нм) – восстановление DTT. Наличие продукта проверяли при помощи электрофореза в нативных условиях: в 8%-ном ПААГ или 0.75%-ном агарозном геле (условия – см. “Эксперим. часть”) (рис. 9). В указанных условиях не удалось достоверно зарегистрировать образование целевой наноструктуры. Однако, как видно из рис. 9а, матрица РОМ-1' присутствует в виде димера, что согласуется с концепцией сборки.
Рис. 9.
Электрофореграммы процесса матричной сборки ДНК-тетраэдра: (а) – 8%-ный нативный ПААГ, 1× ТАЕ, pH 7.6, 10 мM MgCl2, окрашено Stains-All; дорожки: 1 – D544 (0.05 o.e.); 2 – D570 (0.05 o.e.); 3 – D570 (800 нМ); 4–6 – D544 : D570 1 : 2, 1 : 4 и 1 : 8 соответственно; [D544] = 100 нM; (б) – 8%-ный нативный ПААГ, 1× ТАЕ, pH 7.6, окрашено Stains-All; дорожки: 1 – D544 (0.05 o.e.); 2, 3 – D544 : D570 1 : 8, 12.5 или 20 мМ MgCl2 соответственно; [D544] = 1 мкM; (в) – 0.75%-ный агарозный гель, 1× ТАЕ, pH 7.6, 10 мM MgCl2, окрашено EtBr, D544 : D570 1 : 8, [D544] = 1 мкМ, 12.5 мM MgCl2 и 20 мM MgCl2, 1 – без облучения при 365 нм, 2 – 5.5 ч облучения при 365 нм, 3 – 5.5 ч облучения и обработка 100 мM DTT в течение 1 ч; (г) – 0.75%-ный агарозный гель, 1× ТАЕ, pH 7.6, окрашено EtBr; дорожки: 1 – ДНК-маркеры 100–1000 нт; 2 – D570; 3 – D544 : D570 1 : 8, 12.5 мМ MgCl2; 4 – D544 : D570 1 : 8, 20 мМ MgCl2; [D544] = 1 мкM. ВР – краситель бромфеноловый синий.

На заключительном этапе работы было решено провести сборку комплекса при соотношении РОМ-1' и POCT-2 (олигонуклеотиды D544 и D570 соответственно) 1 : 8 и повышенной до 1 мкМ концентрации матрицы. В данных условиях нам удалось зафиксировать образование комплекса матрицы и “звездочки” до облучения (рис. 10б и 10г). Однако после облучения УФ-светом нам не удалось достоверно зарегистрировать образование наноструктур в нативном 8%-ном ПААГ. При попытке зафиксировать образование наноструктур в 0.75%-ном агарозном геле с целью увеличения стабильности комплексов в буфер для электрофореза добавляли MgCl2 до концентрации 10 мМ. Как видно из рис. 9в, это привело к ухудшению разрешения полос ДНК в геле. Тем не менее можно отметить, что интенсивность полос при окрашивании бромистым этидием (EtBr), который преимущественно связывается с двуцепочечной ДНК, в случае облученных комплексов D544–D570 значительно выше по сравнению с полосами необлученных образцов при одинаковом количестве нанесенных образцов (рис. 9в). Это свидетельствует в пользу образования ДНК-наноструктур, содержащих большое число двуцепочечных участков, что соответствует концепции сборки.
Рис. 10.
Фотографии просвечивающей электронной микроскопии дискретных нанообъектов, полученных в процессе сборки ДНК-тетраэдра (обведены кружками). Для отдельных нанообъектов (указаны стрелками) указан размер. На врезке – увеличенное изображение нанообъекта. Негативное контрастирование ацетатом уранила.

Препараты, полученные на финальной стадии сборки ДНК-тетраэдра и очищенные в агарозном геле, содержали дискретные нанообъекты размерами 10–12 нм, что хорошо согласуется с результатами компьютерного моделирования, опубликованными нами ранее [20] (рис. 10). Эти нанообъекты могут представлять собой искомые ДНК-тетраэдры. Визуализацию олигонуклеотидных структур в просвечивающем электронном микроскопе осуществляли с использованием иона уранила ${\text{UO}}_{2}^{{2 + }}$, который специфически связывается с фосфатными группами ДНК в соотношении 1 : 2 [32, 33]. В качестве контрастирующего реагента для визуализации ДНК-наноcтруктур предпочтительнее использовать формиат уранила UO2(HCOO)2, дающий более тонкое “зерно” [34–36]. В данной работе для выявления ДНК-наноструктур мы использовали ацетат уранила UO2(CH3COO)2 [37], поскольку формиат уранила производится только в США, и его экспорт запрещен. В результате оказалось невозможным однозначно подтвердить с помощью просвечивающей электронной микроскопии, что топология полученных нанообъектов соответствует ДНК-тетраэдру. Для этого требуются дополнительные исследования.
ЭКСПЕРИМЕНТАЛЬНАЯ ЧАСТЬ
Для офВЭЖХ использовали ацетонитрил UHPLC grade Supergradient (Panreac, Испания). Раствор ацетата триэтиламмония (ТЕАА, 2 М, рН 7.0) был приготовлен из триэтиламина ACS grade (Panreac, Испания) и ледяной уксусной кислоты (о.с.ч., Реахим, Россия). Дихлоруксусная кислота, иод, 0.25 М раствор 4,5-дицианимидазола (DCI) в безводном ацетонитриле, красители Stains-All, ксиленцианол FF и бромфеноловый синий (BP) были приобретены у компании Sigma-Aldrich (США), перхлорат натрия – у фирмы Acros Organics (США), дихлорметан, тетрагидрофуран, пиридин и триэтиламин – у фирмы Panreac (Испания). Формамид, акриламид, N,N'-метилен-бис-акриламид, мочевина, трис(гидроксиметил)-аминометан (Трис), борная кислота, динатриевая соль этилендиаминтетрауксусной кислоты (Na2ЭДТА) были приобретены у компании Диаэм (Россия). Концентрированный водный раствор аммиака (о.с.ч.), уксусная кислота (о.с.ч.) и ацетон (о.с.ч.) были производства Реахим (Россия). Все реактивы были наивысшей степени чистоты, доступной у соответствующих коммерческих поставщиков. Ацетонитрил (UHPLC grade Supergradient, Panreac, Испания) для синтеза олигонуклеотидов кипятили 6 ч над CaH2 в атмосфере аргона, затем перегоняли в атмосфере аргона и хранили под аргоном над молекулярными ситами 3 Å. Бидистиллированную воду подготавливали непосредственно в лаборатории.
Для центрифугирования небольших объемов растворов использовали микроцентрифугу MiniSpin Рlus (Eppendorf, ФРГ). Химические реакции проводили с использованием термошейкера Thermomixer Compact (Eppendorf, ФРГ). Растворы встряхивали с помощью вортекса BioVortexV1 (Biosan, Эстония). Гель-электрофорез проводили с использованием установки для электрофореза компании Bio-Rad (США). Небольшие объемы растворов олигонуклеотидов (до 1.5 мл) концентрировали с помощью вакуумного концентратора Savant SpeedVac DNA120ОР (Thermo Fisher Scientific, США).
Олигонуклеотиды были получены с помощью автоматического ДНК/РНК-синтезатора ASM-800 (Биоссет, Новосибирск, Россия) согласно модифицированным протоколам фосфитамидного синтеза в стандартных реакторах объемом 25–50 мкл в масштабе 0.2–0.4 мкМ из соответствующих 5'‑DMTr-3'-β-цианэтил-N,N-диизопропилфосфитамидов 2'-дезокси- и 2'-O-метилрибонуклеозидов (Sigma-Aldrich, США), а также 3'-DMTr-5'-β-цианэтил-N,N-диизопропилфосфитамидов 2'-дезоксирибонуклеозидов (“обратных” фосфитамидов, ChemGenes, США) и соответствующих полимерных носителей на основе пористого стекла с размером пор 1000 Å с привитыми 2'-дезокси- (Sigma-Aldrich, США) или 2'-O-метилрибонуклеозидами (Link Technologies, Великобритания). Для введения узлов разветвления были использованы коммерчески доступные ненуклеозидные разветвляющие β-цианэтил-N,N-диизопропилфосфитамиды: симметричный удвоитель (symmetric doubler) (Glen Research, США, кат. № 10-1920) и симметричный утроитель (trebler) (GlenResearch, США, кат. № 10-1922). Для введения размыкаемого элемента (РЭ) в РОСТ использовали дисульфидный фосфитамид С6 (C6 S-S modifier) (GlenResearch, США, кат. № 10-1936). Для введения алкинильной группы по 5'-концам разветвленного олигонуклеотида (“вилки”) использовали β‑цианэтил-N,N-диизопропиламидофосфит 5‑гексин-1-ола (GlenResearch, США, кат. № 10‑1908). Для постсинтетического введения 6-азидогексаноильной группы по 3'-концевой аминогруппе разветвленного олигонуклеотида использовали полимерный носитель 3'-Amino-Modifier C7 CPG 1000 (Link Technologies, Великобритания). Для введения 6-аминогексильной группы по 5'-концам разветвленного олигонуклеотида применяли β-цианэтил-N,N-диизопропиламидофосфит 6-N-монометокситритил-(MMTr)-аминогексанола (Sigma-Аldrich, США). Все фосфитамиды растворяли в сухом ацетонитриле до концентрации 0.1 или 0.15 М (для doubler, trebler и C6 S-S modifier). Время конденсации варьировало от 0.5 мин для 3'-фосфитамидов дезоксирибонуклеозидов, 6 мин для 5'-фосфитамидов дезоксирибонуклеозидов (“обратных”), фосфитамидов 2'-O-метилрибонуклеозидов, аминолинкерного и дисульфидного фосфитамидов и фосфитамида 5-гексин-1-ола, и до 30 мин в случае удвоителя и утроителя. При введении обоих типов разветвителей время конденсации увеличивали до 30 мин, а масштаб синтеза поднимали с 200 до 400 нM.
Для аналитической ВЭЖХ использовали хроматограф Agilent 1220 (Agilent Technologies, США) c УФ-детекцией при длине волны 260 нм, колонкой ZORBAX Eclipse XDB-C18 5 мкм 4.6 × 150 мм (Agilent Technologies, США). Элюцию осуществляли в градиенте ацетонитрила 0–60% в 20 мМ ТЕАА, рН 7.0, за 30 мин и скорости потока 1 мл/мин. Выделение олигонуклеотидов проводили с помощью хроматографа Waters 600E (Waters Corp., США) c УФ-детекцией при длинах волн 190, 260 и 280 нм и колонкой ZORBAX Eclipse Prep HT XDB-C18 7 мкм 21.2 × 150 мм (Agilent Technologies, США) в градиенте ацетонитрила 0–60% в 20 мМ ТЕАА, рН 7.0, за 30 мин при скорости потока 21 мл/мин.
Олигонуклеотиды были синтезированы в режиме без сохранения 5'-DMTr-группы (“DMTr OFF”) с выделением с помощью препаративного гель-электрофореза в 20%-ном ПААГ толщиной 2–3 мм в денатурирующих условиях и обессоливанием на колонке NAP-25 с сорбентом Sephadex G-25 (GE Healthcare, Великобритания) в виде натриевой соли. Для контроля качества олигонуклеотидов проводили аналитический электрофорез в 20%-ном ПААГ толщиной 0.4 мм в аналогичных условиях: акриламид – N,N′-метилен-бис-акриламид (30 : 1), 8 М мочевина, 90 мМ Трис-борат, pH 8.3, 2 мМ Na2ЭДTA при напряжении 50 В/см. Олигонуклеотиды наносили в растворе, содержащем 8 М мочевину, 0.05% ксиленцианола FF и 0.05% бромфенолового синего. Визуализацию полос проводили окрашиванием геля раствором красителя Stains-All (500 мг/л) в формамиде с последующей отмывкой дистиллированной водой.
Концентрацию олигонуклеотидов определяли по оптической плотности раствора с помощью УФ-спектрофотометра NanoDrop 2000с (Thermo Fisher Scientific, США).
Молекулярные массы олигонуклеотидов определяли с помощью масс-спектрометрии MALDI-TOF на приборе Ultraflex III TOF/TOF (Bruker Daltonics, Германия). Масс-спектры олигонуклеотидов получали в линейном режиме отрицательных ионов прибора и в диапазоне m/z от 500 Да до 16 кДа. Параметры измерения включали также частоту импульсов 25 Гц, ускоряющее напряжение 25.0 кВ и время задержки экстракции ионов 120 нс. Образцы олигонуклеотидов растворяли в водном буфере до концентрации 0.1 мM, содержащем 20 мM ТЕАА и 60% ацетонитрила. Объем анализируемого образца составлял 10 мкл. Смешивали 0.7 мкл образца на подложке (Ground Steel или Anchor Chip) с 0.7 мкл раствора матрицы, приготовленного согласно базовому протоколу производителя (Bruker Daltonics, Германия). Матрицей для ионизации олигонуклеотидов в отрицательном режиме служил 2,6-дигидроксиацетофенон (2,6-DHAP) с добавлением дигидроцитрата аммония. Итоговый масс-спектр получали суммированием 8–20 одиночных спектров по 100 импульсов лазера на точку. Калибровку прибора проводили с использованием калибровочных стандартов фирмы Bruker Daltonics (Германия), а также набора олигодезоксирибонуклеотидов с известными массами. Молекулярные массы олигонуклеотидов рассчитывали, используя наборы экспериментальных значений m/z, определенные для каждого анализируемого образца.
Сборку ДНК-тетраэдра регистрировали при помощи электрофореза в нативных условиях (8%-ный ПААГ, акриламид : метилен-бис-акриламид 30 : 1, или 0.75%-ный агарозный гель) (рис. 10). Сборку осуществляли поэтапно следующим образом. Проводили отжиг олигонуклеотидов D544 (матрица РОМ-1') и D570 (“звездочка” РОСТ-2) в 1× ТАЕ, 10 мМ MgCl2 при 95°С в течение 8 мин с последующим охлаждением до комнатной температуры и дальнейшей инкубацией в течение суток. Полученный комплекс облучали УФ-светом с длиной волны 365 нм в течение 5 ч и оставляли на сутки при комнатной температуре. На последнем этапе обрабатывали комплекс 100 мМ DTT в течение 1 ч. Концентрация матрицы D544 составляла 100 нМ, концентрация олигонуклеотида D570 – 200, 400 или 800 нМ (рис. 9а).
Для электронно-микроскопического исследования образцы сорбировали на медные сетки, покрытые формваровой пленкой, в течение 2 мин. После отбора излишков жидкости фильтровальной бумагой образцы контрастировали 0.5%-ным водным раствором уранилацетата (EMS, США) в течение 10–15 с. Образцы изучали в просвечивающем электронном микроскопе JEM-1400 (JEOL, Япония), цифровые изображения получали с помощью камеры бокового ввода Veleta (EMSIS, Германия).
ЗАКЛЮЧЕНИЕ
Осуществлен синтез серии разветвленных олигонуклеотидов для использования в качестве матриц и строительных блоков при матричной сборке ДНК-тетраэдра и ДНК-куба. Был проведен процесс матричной сборки ДНК-тетраэдра, в результате которой были получены дискретные нанообъекты, соответствующие по линейным размерам предполагаемому ДНК-тетраэдру. Размер полученных нанообъектов хорошо согласуется с предсказанным ранее с помощью метода компьютерного моделирования [20].
Список литературы
Seeman N., Sleiman H. // Nat. Rev. Mater. 2018. V. 3. P. 17068. https://doi.org/10.1038/natrevmats.2017.68
Seeman N.C. // Nano Lett. 2020. V. 20. P. 1477–1478. https://doi.org/10.1021/acs.nanolett.0c00325
Templated DNA Nanotechnology: Functional DNA Nanoarchitectonics // Ed. Govindaraju T. Pan Stanford Publishing Pte. Ltd., 2019. ISBN: 978-0-429-42866-1 (eBook).
Rajwar A., Kharbanda S., Chandrasekaran A.R., Gupta S., Bhatia D. // ACS Appl. Bio Mater. 2020. V. 3. P. 7265–7277. https://doi.org/10.1021/acsabm.0c00916
Angell C., Kai M., Xie S., Dong X., Chen Y. // Advanced Healthcare Materials. 2018. V. 7. P. 1701189. https://doi.org/10.1002/adhm.201701189
Li S., Jiang Q., Liu S., Zhang Y., Tian Y., Song C., Wang J., Zou Y., Anderson G.J., Han J.-Y., Chang Y., Liu Y., Zhang C., Chen L., Zhou G., Nie G., Yan H., Ding B., Zhao Y. // Nat. Biotechnol. 2018. V. 36. P. 258–264. https://doi.org/10.1038/nbt.4071
Ma W., Zhan Y., Zhang Y., Shao X., Xie X., Mao C., Cui W., Li Q., Shi J., Li J., Fan C., Lin Y. // Nano Lett. 2019. V. 19. P. 4505–4517. https://doi.org/10.1021/acs.nanolett.9b01320
Rothemund P.W.K. // Nature. 2006. V. 440. P. 297–302. https://doi.org/10.1038/nature04586
DNA Nanotechnology for Bioanalysis: from Hybrid DNA Nanostructures to Functional Devices // Eds. Arrabito G., Wang L. World Scientific Publishing Europe Ltd., 2018. ISBN: 9781786343796
Yu S., Chen T., Zhang Q., Zhoua M., Zhu X. // Analyst. 2020. V. 145. P. 3481–3489. https://doi.org/10.1039/D0AN00159G
Muhammad M., Baig F.A., Lai W.-F., Akhtar M.F., Saleem A., Ahmed S.A., Xia X.-H. // Nano-Structures & Nano-Objects. 2020. V. 23. P. 100523. https://doi.org/10.1016/j.nanoso.2020.100523
Hu Q., Li H., Wang L., Gu H., Fan C. // Chem. Rev. 2019. V. 119. P. 6459–6506. https://doi.org/10.1021/acs.chemrev.7b00663
Xu F., Xia Q., Wang P. // Front. Chem. 2020. V. 8. P. 751. https://doi.org/10.3389/fchem.2020.00751
Chi Q., Yang Z., Xu K., Wang C., Liang H. // Front Pharmacol. 2019. V. 10. P. 1585. https://doi.org/10.3389/fphar.2019.01585
Tapio K., Bald I. // Multifunct. Mater. 2020. V. 3. P. 032001. https://doi.org/10.1088/2399-7532/ab80d5
Loretan M., Domljanovic I., Lakatos M., Rüegg C., Acuna G.P. // Materials. 2020. V. 13. P. 2185. https://doi.org/10.3390/ma13092185
Bush J., Singh S., Vargas M., Oktay E., Hu C.-H., Veneziano R. // Molecules. 2020. V. 25. P. 3386. https://doi.org/10.3390/molecules25153386
Madsen M., Gothelf K.V. // Chem. Rev. 2019. V. 119. P. 6384–6458. https://doi.org/10.1021/acs.chemrev.8b00570
Balakrishnan D., Wilkens G.D., Heddle J.D. // Nanomedicine (Lond.). 2019. V. 14. P. 911–925. https://doi.org/10.2217/nnm-2018-0440
Bakulina A.Yu., Rad’kova Z.V., Burakova E.A., Benassi E., Zatsepin T.S., Fokina A.A., Stetsenko D.A. // Russ. J. Bioorg. Chem. 2019. V. 45. P. 608–618. https://doi.org/10.1134/S1068162019060062
Kolb H.C., Finn M.G., Sharpless K.B. // Angew. Chem. Int. Ed. 2001. V. 40. P. 2004–2021. https://doi.org/10.1002/1521-3773(20010601)40:11<2004: :AID-ANIE2004>3.0.CO;2-5
Tornøe C.W., Christensen C., Meldal M. // J. Org. Chem. 2002. V. 67. P. 3057–3064. https://doi.org/10.1021/jo011148j
Rostovtsev V.V., Green L.G., Fokin V.V., Sharpless K.B. // Angew. Chem. Int. Ed. 2002. V. 41. P. 2596–2599. https://doi.org/10.1002/1521-3773(20020715)41:14<2596: :AID-ANIE2596>3.0.CO;2-4
Васильева С.В., Буракова Е.А., Жданова Л.Г., Анисименко М.С., Стеценко Д.А. // Биоорг. химия. 2017. Т. 43. С. 51–58. [Vasilyeva S.V., Burakova E.A., Zhdanova L.G., Anisimenko M.S., Stetsenko D.A. // Russ. J. Bioorg. Chem. 2017. V. 43. P. 43–49.] https://doi.org/10.1134/S1068162017010113
Jayaprakash K.N., Geng Peng C., Butler D., Varghese J.P., Maier M.A., Rajeev K.G., Manoharan M. // Org. Lett. 2010. V. 12. P. 5410–5413. https://doi.org/10.1021/ol102205j
Van Delft P., Meeuwenoord N.J., Hoogendoorn S., Dinkelaar J., Overkleeft H.S., van der Marel G.A., Filippov D.V. // Org. Lett. 2010. V. 12. P. 5486–5489. https://doi.org/10.1021/ol102357u
Marks I.S., Sung Kang J., Jones B.T., Landmark K.J., Cleland A.J., Taton T.A. // Bioconjug. Chem. 2011. V. 22. P. 1259–1263. https://doi.org/10.1021/bc1003668
Jawalekar A.M., Malik S., Verkade J.M.M., Gibson B., Barta N.S., Hodges J.C., Rowan A., van Delft F.L. // Molecules. 2013. V. 18. P. 7346–7363. https://doi.org/10.3390/molecules18077346
Young D.D., Deiters A. // Org. Biomol. Chem. 2007. V. 5. P. 999–1005. https://doi.org/10.1039/B616410M
Lusic H., Young D.D., Lively M.O., Deiters A. // Org. Lett. 2007. V. 9. P. 1903–1906. https://doi.org/10.1021/ol070455u
Deiters A. // Curr. Opin. Chem. Biol. 2009. V. 13. P. 678–686. https://doi.org/10.1016/j.cbpa.2009.09.026
Zobel R.C., Beer M. // J. Biophys. Biochem. Cytol. 1961. V. 10. P. 335–346. https://doi.org/10.1083/jcb.10.3.335
Beer M., Zobel C.R. // J. Mol. Biol. 1961. V. 3. P. 717–726. https://doi.org/10.1016/S0022-2836(61)80076-4
Ketterer Ph., Willner E.M., Dietz H. // Sci. Adv. 2016. V. 2. e1501209. https://doi.org/10.1126/sciadv.1501209
Nickels Ph.C., Hoiberg H.C., Simmel S.S., Holzmeister Ph., Tinnefeld Ph., Liedl T. // ChemBioChem. 2016. V. 17. P. 1093–1096. https://doi.org/10.1002/cbic.201600059
Wang W., Chen S., An B., Huang K., Bai T., Xu M., Bellot G., Ke Y., Xiang Y., Wei B. // Nat. Commun. 2019. V. 10. P. 1067. https://doi.org/10.1038/s41467-019-08647-7.10:1067
Smith D.M., Schüller V., Forthmann C., Schreiber R., Tinnefeld Ph., Liedl T. // J. Nucleic Acids. 2011. Article ID 360954. https://doi.org/10.4061/2011/360954
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


