Биоорганическая химия, 2022, T. 48, № 3, стр. 255-265
Carbohydrate Structure Database и другие углеводные базы данных как важнейший элемент гликоинформатики
Ф. В. Тоукач 1, *, А. И. Ширковская 1
1 Институт органической химии им. Н.Д. Зелинского РАН
119334 Москва, Ленинский просп., 47, Россия
* E-mail: netbox@toukach.ru
Поступила в редакцию 08.11.2021
После доработки 22.11.2021
Принята к публикации 12.12.2021
- EDN: WQDOLB
- DOI: 10.31857/S0132342322030198
Аннотация
Углеводы – один из наиболее химически разнообразных классов биомакромолекул. Объем накопленной информации о них многократно превышает уровень, позволяющий ориентироваться в этом океане данных без специальных средств – баз данных (БД) гликомики и прогностических сервисов, использующих данные из этих баз. Существующие БД не полностью совместимы друг с другом как по покрытию, так и по форматам данных и возможностям, предоставляемым пользователям, и направлены на решение отдельных задач. Главные проблемы нынешних БД – наличие ошибок, пробелы в полноте покрытия и отсутствие общепризнанного углеводного языка. Наиболее востребованы углеводные БД с широким покрытием для обеспечения единого информационного пространства данных по структуре, свойствам и функциям углеводов, связанных с таксономией и свойствами их природных источников. В рамках проекта Carbohydrate Structure Database (CSDB) была создана архитектура БД, направленная на создание расширяемого проекта гликоинформатики с непрерывной поддержкой и регулярным обновлением данных. Она была реализована в программном продукте, лишенном основных недостатков других БД гликомики. За 15 лет своего существования CSDB стала основным источником данных по углеводам микроорганизмов и платформой для множества сервисов углеводной тематики. Проект нацелен на создание современной и всеобъемлющей базы природных углеводов со свободным доступом, ежегодным обновлением и дополнением содержимого, поиском и устранением ошибок (в том числе в публикациях), появлением новых сервисов.
СОДЕРЖАНИЕ
ВВЕДЕНИЕ ………………...............................255
СУЩЕСТВУЮЩИЕ БАЗЫ ДАННЫХ ГЛИКОМИКИ…..................................................256
КРИТЕРИИ ОЦЕНКИ УГЛЕВОДНЫХ БАЗ ДАННЫХ...........................................................256
УГЛЕВОДНЫЕ НОТАЦИИ………...............258
ИНТЕРФЕЙС И ИНТЕГРАЦИЯ БАЗ ДАННЫХ……….....................................................259
CARBOHYDRATE STRUCTURE DATABASE………….................................................259
ТЕХНИЧЕСКАЯ РЕАЛИЗАЦИЯ CSDB.......262
ЗАКЛЮЧЕНИЕ………………………................262
СПИСОК ЛИТЕРАТУРЫ………………. ........263
ВВЕДЕНИЕ
Углеводы – один из наиболее химически разнообразных классов биомакромолекул. С открытием гликозилирования белков и выяснением роли углеводных антигенов в межклеточных взаимодействиях интерес к ним непрерывно возрастает. К настоящему времени объем накопленной информации об углеводах многократно превысил уровень, позволяющий ориентироваться в этом океане данных без специальных средств. Поэтому прогресс гликобиологии во многом зависит от наличия единого информационного пространства данных по структуре, свойствам и функциям углеводов, связанных с таксономией и свойствами их природных источников. Основное средство создания такого пространства – базы данных (БД) гликомики и прогностические сервисы, использующие данные из этих баз. В отличие от геномики и протеомики, стандарты идентификации структур и протоколы обмена информацией в гликомике были унифицированы лишь в последние годы; этот процесс еще полностью не завершен. Появившиеся проекты новой области биоинформатики – гликоинформатики – не полностью совместимы друг с другом как по покрытию, так и по форматам данных и возможностям, предоставляемым химикам, биологам, генетикам, фармацевтам. Каждый из таких проектов направлен на решение своего класса задач, тем не менее прослеживается явная тенденция к взаимной интеграции. Более детальный разбор проблем гликоинформатики и их возможных решений опубликован в виде мини-обзора [1].
СУЩЕСТВУЮЩИЕ БАЗЫ ДАННЫХ ГЛИКОМИКИ
Базы данных гликомики можно разделить на структурные, протеомные, базы функций и вспомогательные. На рис. 1 представлены наиболее значимые базы данных, ориентированные на углеводные структуры.
Рис. 1.
Обзор существующих структурно-центрических углеводных баз данных. Пиктограммы отражают основные типы накопленных данных в соответствии с обозначениями в нижней части рисунка. Стрелки показывают направления заимствования данных. Приблизительное покрытие по структурам/публикациям (в тысячах записей) указано черными числами. Основные преимущества и недостатки приведены синим шрифтом. Проекты с платным доступом обозначены зеленым символом $. Закончившиеся проекты, тем не менее оказавшие влияние на существующие, обозначены красным восьмиугольником с белой чертой. Рисунок заимствован с адаптированием из работы Egorova, Toukach (2018) [1].

Наиболее востребованы углеводные БД с широким покрытием: GLYCOSCIENCES [2–4] (импорт CCSD/ CarbBank [5, 6] + углеводы млекопитающих + данные ЯМР), UniCarbKB [7, 8] (O- и N-гликаны млекопитающих, включая данные GlycoSuite [9, 10] и Glycobase/GlycoStore (NIBRT) [11, 12], KEGG Glycan [13] (в основном импорт CCSD/Carbbank), Carbohydrate Structure Database [14–17] (CSDB; углеводы прокариот, растений и грибов + данные ЯМР). Также следует отметить специализированные базы: портал GlyGen [18, 19] (структурно-протеомная база гликанов, унаследовавшая, как и UniCarbKB, данные GlycoSuite и архитектуру дизайн-проекта EuroCarbDB [7, 20]), ECODAB [21] (О-антигены Escherichia сoli), Monosaccharide DB [22, 23] (моносахариды), мета-репозиторий структур GlyTouCan [24, 25], созданный как источник универсальных идентификаторов углеводов, GlycoBase-Lille (углеводы амфибий и других животных + данные ЯМР). Исторически первой универсальной углеводной БД была CCSD/CarbBank [6], претендовавшая на полноту покрытия по всем структурам, опубликованным до 1996 г., в котором прекратилась ее поддержка. Поскольку сбор и оцифровка первичных данных из публикаций – наиболее трудоемкая часть работы по созданию БД, почти все современные проекты в том или ином виде используют данные CarbBank, а также элементы идеологии этой базы.
КРИТЕРИИ ОЦЕНКИ УГЛЕВОДНЫХ БАЗ ДАННЫХ
Отличительные особенности, как и критерии оценки углеводных баз данных: представленные типы информации, полнота покрытия, качество данных, функциональность (а также стабильность и производительность), интерфейс пользователя, возможность интеграции с другими проектами и, косвенно, внутренняя архитектура БД.
Типы информации, хранение и обработка которых необходимы для углеводной базы, – это как минимум первичная структура молекул, их таксономические и библиографические аннотации. Часто БД также включают экспериментальные данные, например, ЯМР- или масс-спектры. Возможность записи биохимической, генетической, медицинской и другой информации, как правило, присутствует, но покрытие по этим полям оставляет желать лучшего. Таксономические и библиографические аннотации также есть не во всех базах или не для всех записей. В тех базах, где есть спектры ЯМР, ЯМР-покрытие составляет 5–35% структур. На рис. 1 основные типы представленной информации обозначены иконками. Для первичных баз, получающих данные с помощью собственных усилий по аннотированию, иконки снабжены меткой ORIG; вторичные базы импортируют структуры из других проектов, надстраивая их производными данными.
Полнота покрытия существенно увеличивает полезность БД, т.к. в таком случае даже отрицательный ответ на поисковый запрос представляет собой значимую научную информацию. Полнота покрытия лимитируется невозможностью автоматизации процесса поиска статей с первичными данными. В настоящее время на полное (>80%) покрытие в рамках выбранного класса соединений претендуют только бактериальная и грибная части CSDB. Покрытие остается актуальным при своевременном обновлении базы; приемлемым можно считать период между публикацией и попаданием в базу ~1–2 года. Универсальное решение для повышения актуальности данных – это требование редакций журналов обязательно размещать описываемые структуры в базах данных перед публикацией, с предоставлением ID записи. Такой подход давно реализован в геномике, но отсутствует в гликомике из-за недостаточной стандартизации языков описания структур, корни которой лежат в высокой химической вариативности углеводов.
Процесс заполнения баз данными не поддается полной автоматизации не только на уровне отбора источников данных, но и на уровне интерпретации текстов публикаций. Как следствие, все химические и биологические БД содержат ошибки (перечислены в порядке распространения): привнесенные операторами, перекочевавшие из других БД, присутствующие в публикациях изначально, возникшие из-за несовершенства архитектуры БД и программных ошибок в импортерах и автоаннотаторах. По результатам нашего направленного исследования, большинство записей в CarbBank содержит ошибки [26], причем более трети записей – две и более ошибок, наиболее частая из которых – неверная таксономическая привязка структуры. Также обнаружены значительные пробелы в полноте покрытия. Поскольку большинство современных проектов использует данные CarbBank, эти ошибки проявляются и в них. Некоторые типы ошибок можно выявить (и иногда – исправить) автоматически, и такой контроль ведется в нескольких проектах, однако для достижения действительно высокого качества данных необходим ретроспективный экспертный анализ публикаций и курирование экспертами-гликобиологами.
Функциональность БД – это ее способность обрабатывать поисковые запросы разных типов, комбинировать их в разных логических сочетаниях, уточнять их с использованием данных других типов. Например, “найти все опубликованные за период 2001–2005 гг. структуры, содержащие такой-то фрагмент, а также связанный с моносахаридом лизин либо аланин, кроме синтетических и тех, которые найдены в гамма-протеобактериях, после чего вывести их ЯМР-спектры”. Также к функциональности относятся сопутствующие сервисы углеводной тематики (генерирование конформационных карт, предсказание спектров, поиск структурных закономерностей, статистическая обработка и кластеризация данных из базы и т.д.).
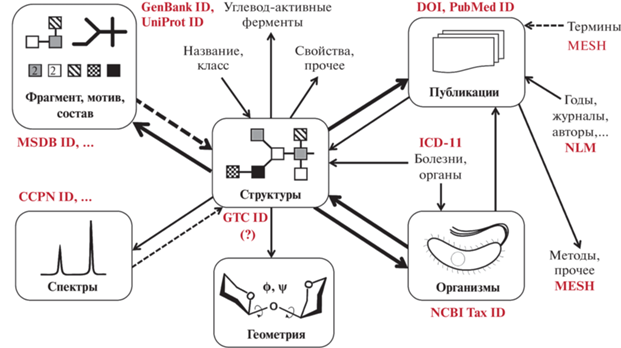
На рис. 2 представлены основные пользовательские запросы, позволяющие переходить от одних типов данных об углеводах к другим. Для многих данных существуют общепризнанные стандартные индексы. Для первичной структуры биогликанов роль такого индекса может выполнять GlyTouCan ID. В отличие от простых в реализации схем поиска по библиографии, ключевым словам, фрагментам текстов, таксономии и проч., поиск структур, содержащих указанный фрагмент, а также поиск структур или спектров, “похожих” на указанные, – задача, требующая предварительных исследований, изощренного программирования и значительных вычислительных ресурсов. В этой связи становится значимой внутренняя архитектура БД, правильность проектирования которой критична для достижения разумной скорости обработки структурных запросов. На этапе становления гликоинформатики в 2010-х гг. исследовательский коллектив GLYCOSCIENCES сформулировал “Десять заповедей построения углеводной базы данных”, объединивших опыт немецкой и российской групп и реализовавшихся в углеводной нотации GlycoCT [27] и дизайн-проекте GlycomeDB [28] (предшественник GlyTouCan). Ключевые положения этого документа включают использование таблицы связности для внутреннего представления структур, максимально возможную индексацию, минимальное количество свободнотекстовых данных (которыми, к сожалению, “грешат” почти все проекты) и однозначный контролируемый словарь для множества типов данных, в первую очередь – для названий остатков. Попытка вывести словарь мономеров из зоны ответственности конкретных проектов была сделана в рамках базы MonosaccharideDB [22]. Дальнейшее совершенствование этих правил Консорциумом по гликоинформатике (https://glic.glycoinfo.org) и консультативной группой по гликоинформатике при NCBI (https://www.ncbi.nlm.nih.gov/glycans/glyag.html) включало стандартизацию представления углеводов в статьях и компьютерных ресурсах (SNFG [29, 30]) и курс на использование семантической паутины (semantic web, модель Resource Description Framework [31]) для получения неявно заданных знаний, не зависящих от конкретных баз [32–34]. Адаптация этой модели к химии и биологии углеводов выразилась в появлении онтологий GlycoRDF (общая) [35] и GlycoCoO (гликоконъюгаты) [36].
Рис. 2.
Взаимосвязь между типами информации об углеводах, представленными в Carbohydrate Structure Database (CSDB). Однозначные переходы, применяемые при обработке запросов, показаны сплошными стрелками; переходы, в которых возможно применение нечеткой логики (выявление паттернов по аналогии или частичному соответствию и т.д.) – пунктирными стрелками. Толстые стрелки соответствуют наиболее распространенным запросам пользователей. Любые запросы могут комбинироваться с помощью булевских операций “И”, “ИЛИ”, “НЕ”.
УГЛЕВОДНЫЕ НОТАЦИИ
Возможность правильной обработки структурной информации напрямую связана со способом записи углеводных структур [37]. Несовершенство и несовместимость форматов этих записей долгое время были камнем преткновения для развития гликоинформатики. Языки записи структур, используемые для внутреннего представления данных и/или для пользовательского интерфейса, оцениваются по следующим критериям:
1) однозначность (строгие правила для записи каждой химически различной структуры единственным образом);
2) способность обрабатывать максимально возможное число реально существующих структур (полимерные, олигомерные и комбинированные углеводы, гликолипиды, гликопротеины), в том числе с неуглеводными компонентами и с поддержкой всевозможных “особых случаев” (нестандартные остатки, связи через фосфор и серу, циклические эфиры, амидные и сложноэфирные связи и т.д.);
3) способность работать с неполными (частично определенными) структурами как на уровне остатков, их конфигураций и позиций замещения, так и на уровне топологий и стехиометрии боковых цепей;
4) “машиночитаемость” (без необходимости сложного синтаксического анализа (parsing), как, например, в случае с языком Extended IUPAC) и “человекочитаемость” (необходима для контроля ошибок, неизбежно возникающих при “человеческой” работе с данными), включая понятные гликобиологам названия остатков;
5) совместимость с существующими форматами и атомарными моделями (наличие конвертеров, облегчающих освоение языка и переход между БД);
6) независимость от ресурсов, курируемых вручную, таких как словари мономеров, лигандов и т.д.
В настоящее время указанными характеристиками в наибольшей степени обладают языки CSDB Linear [37], GlycoCT [27] и WURCS [38, 39]. Однако первый не поддерживает некоторые топологии, а два других “нечеловекочитаемы”. В отличие от ситуации в геномике и протеомике, общепризнанного углеводного языка до сих пор не существует, кроме несовершенного для данного применения IUPAC. Один из упомянутых языков может стать таким стандартом в будущем.
ИНТЕРФЕЙС И ИНТЕГРАЦИЯ БАЗ ДАННЫХ
Сложившиеся представления о качественном продукте гликоинформатики подразумевают, что интерфейс пользователя (как и интерфейс администраторов) должен быть интуитивно понятным, хорошо документированным и находиться в бесплатном доступе для научной общественности через интернет. Понятность касается в том числе форматов ввода и вывода структур, которым пользователю не придется специально учиться. В этом аспекте чрезвычайно плодотворна реализация ввода фрагментов структур с помощью самостоятельных сервисов, в том числе специальных редакторов, имеющих программный интерфейс, позволяющий любой БД пользоваться интерфейсом других БД. Краткий обзор современных углеводных редакторов опубликован в статье, посвященной редактору CSDB/SNFGeditor [40].
Интеграция между проектами гликоинформатики подразумевает не только общий интерфейс поисковых запросов, но и возможность автоматического обмена данными. Это касается и взаимодействия с неуглеводными базами данных: библиографическими (например, NCBI PubMed) [41], таксономическими (NCBI Taxonomy) [42], генетическими (NCBI GenBank) [43], протеомными (UniProt) [44] и др. Первыми проектами, разработавшими протоколы автоматического обмена данными об углеводах [45], были GLYCOSCIENCES и Bacterial CSDB, после чего стандартизация форматов и разработка программных сервисов гликомики значительно ускорились.
Особняком стоит EuroCarbDB [20], которая была профинансирована как БД, полностью лишенная недостатков и обеспечивающая широчайшую функциональность при разумной цене обслуживания, но на деле ограничилась разработкой подходов (без их реального воплощения, которое выступает “узким местом” БД из-за человеческого фактора) и импортом CCSD/CarbBank. На противоположном конце идеологической иерархии находится мета-репозиторий GlyTouCan, который заведомо не предоставляет собственных данных, но интегрируется со множеством других проектов, импортируя их данные и выступая по сути “базой баз”, обеспечивающей межпроектную работу в едином интерфейсе.
Более глубокий анализ состояния гликоинформатики и современных инициатив в ней, углеводных баз и сопутствующих проектов можно найти в обзорах и сборниках [46–51].
CARBOHYDRATE STRUCTURE DATABASE
В рамках проекта Carbohydrate Structure Database (CSDB) мы поставили цель спроектировать архитектуру БД и реализовать ее в программном продукте, который был бы лишен основных недостатков других БД гликомики, а также обеспечить постоянную поддержку, не требующую серьезных финансовых вложений, и регулярное обновление данных. Ключевые особенности CSDB – полнота покрытия и полностью верифицируемое содержимое. За 15 лет своего существования CSDB стала основным источником данных по углеводам микроорганизмов и платформой для множества сервисов углеводной тематики. Проект нацелен на создание современной и всеобъемлющей базы природных углеводов, которая идеологически заменит собой Carbbank.
Коллектив CSDB проводит систематическую работу по информатизации гликомики [1, 52]. Во взаимодействии с мировым сообществом гликоинформатиков сформированы критерии качества программ и сервисов в этой области, созданы стандарты и онтологии компьютерного представления и визуализации углеводных данных, разработана платформа CSDB, включающая тематические базы данных и расчетные модули, позволяющие делать выводы на основании обработки всех данных, в том числе проводить скрининг и получать статистические данные. На рис. 3 представлены основные возможности и скриншоты отдельных инструментов базы. Все возможности проекта бесплатно доступны гликохимикам и гликобиологам через интернет (http://csdb.glycoscience.ru). Основные вехи его развития с разбивкой по годам приведены на сайте проекта в разделе “История изменений” (http://csdb.glycoscience.ru/help/about.html#changelog). В частности, нововведения 2020– 2021 гг. – интеграция с Международным классификатором болезней (ICD-11) и базой KEGG, модуль 3D-моделирования, SNFG-совместимый графический редактор структур, модуль работы с конформациями дисахаридов, сервисная база данных по агликонам и гликоэпитопам.
Рис. 3.
Основные возможности и скриншоты отдельных инструментов Carbohydrate Structure Database (CSDB). Посередине сверху – главная страница веб-портала CSDB со ссылками на основные инструменты: верхний ряд – поиск по фрагменту структуры, составу, организму, публикации, спектрам ЯМР; нижний ряд – сервис предсказания спектров ЯМР, предсказание структуры по экспериментальным данным, анализ распределения структурных фрагментов, кластеризация таксонов по их гликомам, активность гликозилтрансфераз, примеры использования. В нижнем ряду слева направо скриншоты: редактор структур CSDB/SNFG editor, кластеризация таксонов, таблица с конформационными данными дисахаридов, предсказанный двумерный спектр ЯМР HSQC, интерактивная конформационная карта.

Ниже перечислены важнейшие компоненты CSDB.
1) База данных природных углеводов бактерий, архей, грибов, растений и простейших [15, 16, 53]. По прокариотам и грибам база обеспечивает покрытие, близкое к полному (т.е. включает ~90% всех опубликованных данных, что делает даже отрицательный ответ на поисковый запрос значимой научной информацией). База содержит данные о первичной структуре гликанов и гликоконъюгатов, их таксономическую привязку вплоть до штаммов и серогрупп, подробные библиографические аннотации, спектры ЯМР и отчасти – биохимические, генетические, медицинские и другие аннотации, а также ссылки на другие базы (NCBI PubMed [41], NCBI Taxonomy [42], ICD-11 [54], MeSH [55], PubChem [56], GlyTouCan [24, 25], Thomson Reuters DCI [57]). Данные попадают в базу на основании отбора в библиографических базах и последующего аннотирования статей. Временной промежуток между публикацией и размещением в базе составляет ~1 год.
2) База данных конформаций гликозидных мостиков в олигосахаридах и родственных структурных фрагментах, заполненная данными низкотемпературной молекулярной динамики с явным учетом растворителя [58], и данными, импортированными из базы GlycoMapsDB [59]. Интерфейс базы позволяет изучать, визуализировать и экспортировать карты энергий и заселенностей с размерностью до 4.
3) База данных гликозилтрансфераз с экспериментально подтвержденной активностью и с привязкой к полным структурам и штаммам. Этот модуль предоставляет близкое к полному покрытие до 2020 г. по трем наиболее изученным представителям каждого царства: бактерии E. coli, дрожжам Saccharomyces сerevisiae и растению Arabidopsis thaliana [60–62].
4) База данных структурных компонентов природных углеводов (моносахаридов, полиолов, аминокислот, жирных кислот, распространенных агликонов и других молекулярных строительных блоков) с подробными структурно-химическими аннотациями вплоть до 3D-моделей.
5) Многочисленные инструменты поиска, фильтрации [63, 64], сопоставления, статистической обработки [65] и кластеризации структурных, таксономических, биосинтетических, конформационных, библиографических, ЯМР-спектроскопических и прочих данных по нескольким десяткам критериев.
6) Визуальный онлайн-редактор углеводных структур [40], позволяющий собирать сложные биомолекулы с помощью интуитивных операций в браузере. Редактор способен экспортировать результат во все современные форматы гликоинформатики, в атомарные форматы (MOL, PDB), структурные формулы, трехмерные модели, имеет собственные генератор возможных изомеров для структур с неопределенностями, генератор и визуализатор геометрии [66], оптимизированные для молекулярной механики сложных углеводов и опирающиеся на предварительно заполненную сервисную базу конформаций мономерных остатков.
7) Модуль предсказания одно- и двумерных спектров ЯМР произвольных углеводов [67], предсказания структуры по спектрам [68] и данным других экспериментов. Предсказание спектров базируется на собственной теории [69] и использовании базы данных, обеспечивает среднюю точность для структурных компонентов биогликанов на уровне 0.07 м.д. (1H) и 0.6 м.д. (13С). Для сложных углеводов и гликоконъюгатов достигнутая точность существенно превосходит показатели аналогов, в том числе нейросетевых и квантовомеханических расчетов на высоких уровнях теории [70], при этом на 3–4 порядка превосходя их по скорости. Такая производительность позволяет использовать разработанный инструмент для потоковой генерации спектров и их автоматического сравнения с экспериментальными спектрами.
8) Интеграция с другими проектами [34] глико-, хемо- и биоинформатики на уровне программного интерфейса, кросс-ссылок, универсальных идентификаторов, RDF-онтологии [35], договоренностей о стандартах и форматах данных. Используемая собственная углеводная нотация (язык CSDB Linear [37]) поддерживает подавляющее большинство структурных особенностей биогликанов, в том числе однозначно описывает их неуглеводные компоненты. Она имеет трансляторы на другие углеводные языки (GlycoCT, WURCS, Sweet-DB, SNFG) и из них (GlycoCT).
9) Инструменты автоматизации аннотирования оригинальных публикаций, выявления ошибок в публикациях и в базах данных, программы для повышения эффективности и снижения стоимости ретроспективного анализа публикаций коллективом CSDB.
10) Подробная справочная система для пользователей, администраторов и аннотаторов, включающая руководство пользователя (раздел “Help” в главном меню сайта проекта), учебник по решению наиболее распространенных задач [64] (http://csdb.glycoscience.ru/help/examples.html), справочные данные для разработчиков (http://csdb.glycoscience.ru/help/dbdocs.html), быструю контекстную справку по элементам интерфейса.
Каждый год в CSDB обновляются и дополняются данные, проводятся поиск и устранение ошибок (в том числе в публикациях других авторов), появляются новые сервисы. Планируемое дальнейшее развитие CSDB включает расширение покрытия гликанами животных, достижение полного покрытия по простейшим, формализацию поиска гликоэпитопов, совершенствование конформационного модуля и дальнейшее заполнение его сервисной базы данными о разветвленных трисахаридах, расширение покрытия базы гликозилтрансфераз ферментами патогенов группы ESKAPE и другие задачи.
ТЕХНИЧЕСКАЯ РЕАЛИЗАЦИЯ CSDB
Технически CSDB представляет собой платформу для компьютерных сервисов с оригинальным стандартизированным API. Данные, импортированные из других баз (CarbBank, GlycomeDB, GlycoMapsDB, GlycoEpitopeDB) и полученные из литературы, после всесторонней проверки и валидирования попадают в “человекочитаемые” текстовые файлы (дампы), одновременно выступающие резервными копиями. Все редактирование данных в базе происходит на уровне этих файлов. Ежегодно дампы импортируются в реляционную базу данных на СУБД MySQL 5, где получают дополнительные представления, ускоряющие многокритериальный поиск: таблицу связности на уровне мономерных остатков, сервисные базы, денормализацию. Скрипты импорта, обработки, поиска данных и обслуживания базы написаны на языке PHP 5.6; работа с атомарными химическими моделями – на Python 3.5 + RDKIT + openBabel; веб-интерфейс пользователя – на Javascript + DHTML. Для молекулярно-динамических расчетов используется Tinker 8.3. Проект развернут на выделенном аппаратном веб-сервере (64 ядра, 96 Gb RAM) под управлением Windows Server 2016, он же используется как вычислительный сервер для обработки ресурсоемких задач пользователей и фонового обсчета конформаций. Длительные задачи (например, выявление ошибок в дампах, подготовленных аннотаторами, или поиск структурных гипотез, наиболее подходящих экспериментальным данным) выполняются в отложенном формате с уведомлением пользователя по мере готовности результатов.
ЗАКЛЮЧЕНИЕ
Для эффективной работы с накопленным в настоящее время объемом информации об углеводах необходимо единое информационное пространство данных по структуре, свойствам и функциям углеводов, связанных с таксономией и свойствами их природных источников. Основное средство создания такого пространства – базы данных (БД) гликомики и прогностические сервисы, использующие данные из этих баз. За последние 10 лет информатизированность гликохимии и гликобиологии существенно возросла, появились общепризнанные стандарты и сервисы, увеличилась их совместимость друг с другом, и началось налаживание взаимодействия с компьютерными проектами смежных областей. Это постепенно приближает гликомику к уровню информационной обеспеченности, сравнимому с существующим в геномике и протеомике, несмотря на большую вариативность и гетерогенность объектов исследования.
Коллектив Carbohydrate Structure Database (CSDB) провел обширное исследование существующих инициатив гликоинформатики для выявления их преимуществ и недостатков. Полученная информация легла в основу создания архитектуры БД и ее реализации в программном продукте CSDB, ключевые особенности которого – полнота покрытия и полностью верифицируемое содержимое. CSDB содержит данные о первичной структуре гликанов и гликоконъюгатов, их таксономическую привязку вплоть до штаммов, подробные библиографические аннотации, спектры ЯМР и отчасти – биохимические, генетические, медицинские и другие аннотации. Встроенные надстройки позволяют проводить анализ путей биосинтеза посредством базы гликозилтрансфераз, получать конформационные карты олигосахаридов, предсказывать и относить спектры ЯМР 13С, 1Н, 2D, предсказывать структуру по спектрам и другим данным, проводить кластеризацию таксонов на основании их гликомов, распределять фрагменты по таксонам и положению в структурах, а также классифицировать мономеры. Данные в базе ежегодно обновляются и дополняются, проводятся поиск и устранение ошибок, появляются новые сервисы.
Список литературы
Egorova K.S., Toukach P.V. // Angew. Chem. Int. Ed. Engl. 2018. V. 57. P. 14986–14990. https://doi.org/10.1002/anie.201803576
Lütteke T. // In: A Practical Guide to Using Glycomics Databases / Ed. Aoki-Kinoshita K.F. Tokyo: Springer, 2017. P. 335–350. https://doi.org/10.1007/978-4-431-56454-6_16
Bohm M., Bohne-Lang A., Frank M., Loss A., Rojas-Macias M.A., Lutteke T. // Nucl. Acids Res. 2019. V. 47. P. D1195–D1201. https://doi.org/10.1093/nar/gky994
Lütteke T., Bohne-Lang A., Loss A., Goetz T., Frank M., von der Lieth C.W. // Glycobiology. 2006. V. 16. P. 71R–81R. https://doi.org/10.1093/glycob/cwj049
Doubet S., Bock K., Smith D., Darvill A., Albersheim P. // Trends in Biochemical Sciences. 1989. V. 14. P. 475–477. https://doi.org/10.1016/0968-0004(89)90175-8
Doubet S., Albersheim P. // Glycobiology. 1992. V. 2. P. 505–507. https://doi.org/10.1093/glycob/2.6.505
Campbell M.P., Peterson R., Mariethoz J., Gasteiger E., Akune Y., Aoki-Kinoshita K.F., Lisacek F., Packer N.H. // Nucleic Acids Res. 2014. V. 42. P. D215–D221. https://doi.org/10.1093/nar/gkt1128
Campbell M.P., Packer N.H. // Biochim. Biophys. Acta. 2016. V. 1860. P. 1669–1675. https://doi.org/10.1016/j.bbagen.2016.02.016
Cooper C.A., Joshi H.J., Harrison M.J., Wilkins M.R., Packer N.H. // Nucleic Acids Res. 2003. V. 31. P. 511–513. https://doi.org/10.1093/nar/gkg099
Cooper C.A., Harrison M.J., Wilkins M.R., Packer N.H. // Nucleic Acids Res. 2001. V. 29. P. 332–335. https://doi.org/10.1093/Nar/29.1.332
Zhao S., Walsh I., Abrahams J.L., Royle L., Nguyen-Khuong T., Spencer D., Fernandes D.L., Packer N.H., Rudd P.M., Campbell M.P. // Bioinformatics. 2018. V. 34. P. 3231–3232. https://doi.org/10.1093/bioinformatics/bty319
Campbell M.P., Royle L., Radcliffe C.M., Dwek R.A., Rudd P.M. // Bioinformatics. 2008. V. 24. P. 1214–1216. https://doi.org/10.1093/bioinformatics/btn090
Aoki-Kinoshita K.F., Kanehisa M. // In: Glycoinformatics / Eds. Lütteke T., Frank M. New York: Humana Press, 2015. P. 97–107. https://doi.org/10.1007/978-1-4939-2343-4_7
Toukach P.V., EgorovaK.S. // In: Glycoscience: Biology and Medicine / Eds.Taniguchi N., Endo T., Hart G., Seeberger P., Wong C.H. Tokyo: Springer, 2015. P. 241–250. https://doi.org/10.1007/978-4-431-54841-6_24
Toukach P.V., Egorova K.S. // Nucleic Acids Res. 2016. V. 44. P. D1229–D1236. https://doi.org/10.1093/nar/gkv840
Toukach P.V., Knirel Y.A. // Glycoconjugate J. 2005. V. 2. P. 216–217.
Toukach P.V. // J. Chem. Inf. Model. 2011. V. 51. P. 159–170. https://doi.org/10.1021/ci100150d
York W.S., Mazumder R., Ranzinger R., Edwards N., Kahsay R., Aoki-Kinoshita K.F., Campbell M.P., Cummings R.D., Feizi T., Martin M., Natale D.A., Packer N.H., Woods R.J., Agarwal G., Arpinar S., Bhat S., Blake J., Castro L.J.G., Fochtman B., Gildersleeve J., Goldman R., Holmes X., Jain V., Kulkarni S., Mahadik R., Mehta A., Mousavi R., Nakarakommula S., Navelkar R., Pattabiraman N., Pierce M.J., Ross K., Vasudev P., Vora J., Williamson T., Zhang W. // Glycobiology. 2020. V. 30. P. 72–73. https://doi.org/10.1093/glycob/cwz080
Kahsay R., Vora J., Navelkar R., Mousavi R., Fochtman B.C., Holmes X., Pattabiraman N., Ranzinger R., Mahadik R., Williamson T., Kulkarni S., Agarwal G., Martin M., Vasudev P., Garcia L., Edwards N., Zhang W., Natale D.A., Ross K., Aoki-Kinoshita K.F., Campbell M.P., York W.S., Mazumder R. // Bioinformatics. 2020. V. 36. P. 3941–3943. https://doi.org/10.1093/bioinformatics/btaa238
von der Lieth C.W., Freire A.A., Blank D., Campbell M.P., Ceroni A., Damerell D.R., Dell A., Dwek R.A., Ernst B., Fogh R., Frank M., Geyer H., Geyer R., Harrison M.J., Henrick K., Herget S., Hull W.E., Ionides J., Joshi H.J., Kamerling J.P., Leeflang B.R., Lutteke T., Lundborg M., Maass K., Merry A., Ranzinger R., Rosen J., Royle L., Rudd P.M., Schloissnig S., Stenutz R., Vranken W.F., Widmalm G., Haslam S.M. // Glycobiology. 2011. V. 21. P. 493–502. https://doi.org/10.1093/glycob/cwq188
Rojas-Macias M.A., Ståhle J., Lütteke T., Widmalm G. // Glycobiology. 2015. V. 25. P. 341–347. https://doi.org/10.1093/glycob/cwu116
Lutteke T., von der Lieth C.W. // Glycobiology. 2005. V. 15. P. 1209–1210. https://doi.org/10.1093/glycob/cwj039
Lütteke T. // In: A Practical Guide to Using Glycomics Databases / Ed. Aoki-Kinoshita K.F. Tokyo: Springer, 2017. P. 29–40. https://doi.org/10.1007/978-4-431-56454-6_3
Fujita A., Aoki N.P., Shinmachi D., Matsubara M., Tsuchiya S., Shiota M., Ono T., Yamada I., Aoki-Kinoshita K.F. // Nucleic Acids Res. 2021. V. 49. P. D1529–D1533. https://doi.org/10.1093/nar/gkaa947
Tiemeyer M., Aoki K., Paulson J., Cummings R.D., York W.S., Karlsson N.G., Lisacek F., Packer N.H., Campbell M.P., Aoki N.P., Fujita A., Matsubara M., Shinmachi D., Tsuchiya S., Yamada I., Pierce M., Ranzinger R., Narimatsu H., Aoki-Kinoshita K.F. // Glycobiology. 2017. V. 27. P. 915–919. https://doi.org/10.1093/glycob/cwx066
Egorova K.S., Toukach P.V. // J. Chem. Inf. Model. 2012. V. 52. P. 2812–2814. https://doi.org/10.1021/ci3002815
Herget S., Ranzinger R., Maass K., Lieth C.W. // Carbohydr. Res. 2008. V. 343. P. 2162–2171. https://doi.org/10.1016/j.carres.2008.03.011
Ranzinger R., Herget S., von der Lieth C.W., Frank M. // Nucleic Acids Res. 2011. V. 39. P. D373–D376. https://doi.org/10.1093/nar/gkq1014
Varki A., Cummings R.D., Aebi M., Packer N.H., Seeberger P.H., Esko J.D., Stanley P., Hart G., Darvill A., Kinoshita T., Prestegard J.J., Schnaar R.L., Freeze H.H., Marth J.D., Bertozzi C.R., Etzler M.E., Frank M., Vliegenthart J.F., Lutteke T., Perez S., Bolton E., Rudd P., Paulson J., Kanehisa M., Toukach P., Aoki-Kinoshita K.F., Dell A., Narimatsu H., York W., Taniguchi N., Kornfeld S. // Glycobiology. 2015. V. 25. P. 1323–1324. https://doi.org/10.1093/glycob/cwv091
Neelamegham S., Aoki-Kinoshita K., Bolton E., Frank M., Lisacek F., Lutteke T., O’Boyle N., Packer N.H., Stanley P., Toukach P., Varki A., Woods R.J., Group S.D. // Glycobiology. 2019. V. 29. P. 620–624. https://doi.org/10.1093/glycob/cwz045
Willighagen E.L., Brandle M.P. // J. Cheminform. 2011. V. 3. P. 15. https://doi.org/10.1186/1758-2946-3-15
Aoki-Kinoshita K.F., Aoki N.P., Fujita A., Fujita N., Kawasaki T., Matsubara M., Okuda S., Shikanai T., Shinmachi D., Solovieva E., Suzuki Y., Tsuchiya S., Yamada I., Narimatsu H. // Perspectives in Science. 2017. V. 11. P. 18–23. https://doi.org/10.1016/j.pisc.2016.05.012
Katayama T., Wilkinson M.D., Aoki-Kinoshita K.F., Kawashima S., Yamamoto Y., Yamaguchi A., Okamoto S., Kawano S., Kim J.D., Wang Y., Wu H., Kano Y., Ono H., Bono H., Kocbek S., Aerts J., Akune Y., Antezana E., Arakawa K., Aranda B., Baran J., Bolleman J., Bonnal R.J., Buttigieg P.L., Campbell M.P., Chen Y.A., Chiba H., Cock P.J., Cohen K.B., Constantin A., Duck G., Dumontier M., Fujisawa T., Fujiwara T., Goto N., Hoehndorf R., Igarashi Y., Itaya H., Ito M., Iwasaki W., Kalas M., Katoda T., Kim T., Kokubu A., Komiyama Y., Kotera M., Laibe C., Lapp H., Lutteke T., Marshall M.S., Mori T., Mori H., Morita M., Murakami K., Nakao M., Narimatsu H., Nishide H., Nishimura Y., Nystrom-Persson J., Ogishima S., Okamura Y., Okuda S., Oshita K., Packer N.H., Prins P., Ranzinger R., Rocca-Serra P., Sansone S., Sawaki H., Shin S.H., Splendiani A., Strozzi F., Tadaka S., Toukach P., Uchiyama I., Umezaki M., Vos R., Whetzel P.L., Yamada I., Yamasaki C., Yamashita R., York W.S., Zmasek C.M., Kawamoto S., Takagi T. // J. Biomed. Semantics. 2014. V. 5. P. 5. https://doi.org/10.1186/2041-1480-5-5
Aoki-Kinoshita K.F., Bolleman J., Campbell M.P., Kawano S., Kim J.D., Lutteke T., Matsubara M., Okuda S., Ranzinger R., Sawaki H., Shikanai T., Shinmachi D., Suzuki Y., Toukach P., Yamada I., Packer N.H., Narimatsu H. // J. Biomed. Semantics. 2013. V. 4. P. 39. https://doi.org/10.1186/2041-1480-4-39
Ranzinger R., Aoki-Kinoshita K.F., Campbell M.P., Kawano S., Lutteke T., Okuda S., Shinmachi D., Shikanai T., Sawaki H., Toukach P., Matsubara M., Yamada I., Narimatsu H. // Bioinformatics. 2015. V. 31. P. 919–925. https://doi.org/10.1093/bioinformatics/btu732
Yamada I., Campbell M.P., Edwards N., Castro L.J., Lisacek F., Mariethoz J., Ono T., Ranzinger R., Shinmachi D., Aoki-Kinoshita K.F. // Glycobiology. 2021. V. 31. P. 741–750. https://doi.org/10.1093/glycob/cwab013
Toukach P.V., Egorova K.S. // J. Chem. Inf. Model. 2020. V. 60. P. 1276–1289. https://doi.org/10.1021/acs.jcim.9b00744
Tanaka K., Aoki-Kinoshita K.F., Kotera M., Sawaki H., Tsuchiya S., Fujita N., Shikanai T., Kato M., Kawano S., Yamada I., Narimatsu H. // J. Chem. Inf. Model. 2014. V. 54. P. 1558–1566. https://doi.org/10.1021/ci400571e
Matsubara M., Aoki-Kinoshita K.F., Aoki N.P., Yamada I., Narimatsu H. // J. Chem. Inf. Model. 2017. V. 57. P. 632–637. https://doi.org/10.1021/acs.jcim.6b00650
Bochkov A.Y., Toukach P.V. // J. Chem. Inf. Model. 2021. V. 61. P. 4940–4948. https://doi.org/10.1021/acs.jcim.1c00917
Lu Z. // Database. 2011. V. 2011. P. baq036. https://doi.org/10.1093/database/baq036
Federhen S. // Nucleic Acids Res. 2012. V. 40. P. D136–D143. https://doi.org/10.1093/nar/gkr1178
Benson D.A., Cavanaugh M., Clark K., Karsch-Mizrachi I., Lipman D.J., Ostell J., Sayers E.W. // Nucleic Acids Res. 2013. V. 41. P. D36–D42. https://doi.org/10.1093/nar/gks1195
The Uniprot Consortium // Nucleic Acids Res. 2017. V. 45. P. D158–D169. https://doi.org/10.1093/nar/gkw1099
Toukach P., Joshi H.J., Ranzinger R., Knirel Y., von der Lieth C.W. // Nucleic Acids Res. 2007. V. 35. P. D280–D286. https://doi.org/10.1093/nar/gkl883
Li X., Xu Z., Hong X., Zhang Y., Zou X. // Int. J. Mol. Sci. 2020. V. 21. P. 6727. https://doi.org/10.3390/ijms21186727
Abrahams J.L., Taherzadeh G., Jarvas G., Guttman A., Zhou Y., Campbell M.P. // Curr. Opin. Struct. Biol. 2020. V. 62. P. 56–69. https://doi.org/10.1016/j.sbi.2019.11.009
Scherbinina S.I., Toukach P.V. // Int. J. Mol. Sci. 2020. V. 21. P. 7702. https://doi.org/10.3390/ijms21207702
Copoiu L., Malhotra S. // Curr. Opin. Struct. Biol. 2020. V. 62. P. 132–139. https://doi.org/10.1016/j.sbi.2019.12.020
A Practical Guide to Using Glycomics Databases / Ed. Aoki-Kinoshita K.F. Tokyo: Springer, 2017. 370 p. https://doi.org/10.1007/978-4-431-56454-6
Aoki-Kinoshita K.F. // Molecular & Cellular Proteomics. 2013. V. 12. P. 1036–1045. https://doi.org/10.1074/mcp.R112.026252
Тоукач Ф.В. // Информационные технологии в структурной гликохимии и гликобиологии. Дисс. докт. хим. наук. Москва: ФГБУН Институт органической химии им. Н.Д. Зелинского РАН, 2019.
Egorova K.S., Toukach P.V. // Carbohydr. Res. 2014. V. 389. P. 112–114. https://doi.org/10.1016/j.carres.2013.10.009
ICD-11: in Praise of Good Data// The Lancet Infectious Diseases. 2018. V. 18. P. 813. https://doi.org/10.1016/s1473-3099(18)30436-5
Baumann N. // Int. J. Clin. Pract. 2016. V. 70. P. 171–174. https://doi.org/10.1111/ijcp.12767
Kim S., Thiessen P.A., Bolton E.E., Chen J., Fu G., Gindulyte A., Han L., He J., He S., Shoemaker B.A., Wang J., Yu B., Zhang J., Bryant S.H. // Nucleic Acids Res. 2016. V. 44. P. D1202–D1213. https://doi.org/10.1093/nar/gkv951
Pavlech L.L. // J. Med. Library Association. 2016. V. 104. P. 88–90. https://doi.org/10.3163/1536-5050.104.1.020
Stroylov V., Panova M., Toukach P. // Int. J. Mol. Sci. 2020. V. 21. P. 7626. https://doi.org/10.3390/ijms21207626
Frank M., Lutteke T., von der Lieth C.W. // Nucleic Acids Res. 2007. V. 35. P. 287–290. https://doi.org/10.1093/nar/gkl907
Egorova K.S., Toukach P.V. // Glycobiology. 2017. V. 27. P. 285–290. https://doi.org/10.1093/glycob/cww137
Egorova K.S., Knirel Y.A., Toukach P.V. // Glycobiology. 2019. V. 29. P. 285–287. https://doi.org/10.1093/glycob/cwz006
Egorova K.S., Smirnova N.S., Toukach P.V. // Glycobiology. 2021. V. 31. P. 524–529. https://doi.org/10.1093/glycob/cwaa107
Egorova K.S., Toukach P.V. // In: A Practical Guide to Using Glycomics Databases / Ed. Aoki-Kinoshita K.F. Tokyo: Springer, 2017. P. 75–113. https://doi.org/10.1007/978-4-431-56454-6_5
Toukach P.V., Egorova K.S. // In: Glycoinformatics / Eds. Lütteke T., Frank M. New York: Humana Press, 2015. P. 55–85. https://doi.org/10.1007/978-1-4939-2343-4_5
Egorova K.S., Kondakova A.N., Toukach P.V. // Database. 2015. V. 2015. P. bav073. https://doi.org/10.1093/database/bav073
Chernyshov I.Y., Toukach P.V. // Bioinformatics. 2018. V. 34. P. 2679–2681. https://doi.org/10.1093/bioinformatics/bty168
Kapaev R.R., Toukach P.V. // J. Chem. Inf. Model. 2016. V. 56. P. 1100–1104. https://doi.org/10.1021/acs.jcim.6b00083
Kapaev R.R., Toukach P.V. // Bioinformatics. 2018. V. 34. P. 957–963. https://doi.org/10.1093/bioinformatics/btx696
Kapaev R.R., Egorova K.S., Toukach P.V. // J. Chem. Inf. Model. 2014. V. 54. P. 2594–2611. https://doi.org/10.1021/ci500267u
Kapaev R.R., Toukach P.V. // Anal. Chem. 2015. V. 87. P. 7006–7010. https://doi.org/10.1021/acs.analchem.5b01413
Дополнительные материалы отсутствуют.
Инструменты
Биоорганическая химия


