Биология моря, 2020, T. 46, № 5, стр. 329-337
Разнообразие и антимикробная активность бактерий, выделенных из донных осадков Чукотского моря
Л. А. Романенко 1, *, В. В. Куриленко 1, Н. Ю. Чернышева 1, К. В. Гузев 1, В. В. Михайлов 1, 2
1 Тихоокеанский институт биоорганической химии (ТИБОХ) им. Г.Б. Елякова ДВО РАН
690022 Владивосток, Россия
2 Дальневосточный федеральный университет
690950 Владивосток, Россия
* E-mail: lro@piboc.dvo.ru
Поступила в редакцию 06.06.2019
После доработки 02.10.2019
Принята к публикации 03.10.2019
Аннотация
Из образцов донных осадков Чукотского моря выделено и исследовано 248 штаммов гетеротрофных бактерий. На основании филогенетического анализа гена 16S рРНК были идентифицированы представители 33 родов, принадлежащие типам Proteobacteria, Bacteroidetes, Firmicutes и Actinobacteria. Восемь групп бактерий показали уровень сходства гена 16S рРНК не более 97–98% и могут представлять новые таксоны. Скрининг антимикробной активности выявил 40 штаммов, способных ингибировать рост двух и более индикаторных микроорганизмов. Активные штаммы представлены бактериями родов Bacillus, Paenibacillus, Terribacillus, Virgibacillus (тип Firmicutes), Streptomyces, Pseudonocardia, Nocardiopsis (Actinobacteria) и в меньшей степени грамотрицательными бактериями родов Shewanella, Psychrobacter (класс Gammaproteobacteria), Massilia (класс Betaproteobacteria), Arenibacter (тип Bacteroidetes). Обнаружение фрагментов генов поликетидсинтаз (PKS-1) и нерибосомных пептидсинтетаз (NRPS) методом ПЦР с использованием вырожденных праймеров в 23 активных штаммах свидетельствует о том, что исследуемые бактерии могут служить источниками антимикробных метаболитов поликетидной природы и/или нерибосомных пептидов.
Морские донные осадки являются природным источником биологического разнообразия, местом обитания и инкубатором для многих представителей морской биоты. Микроорганизмы донных осадков – это важный компонент морских экосистем, представляющий особый интерес для научных исследований (Lauro, Bartlett, 2008). Показано, что бактерии морской среды, в частности донных осадков, способны синтезировать разнообразные биологически активные вещества (БАВ) с антимикробными, противоопухолевыми и другими свойствами (Debbab et al., 2010; Penesyan et al., 2010; Pettit, 2011; Bhatnagar, Kim, 2012). Изучение микроорганизмов, изолированных из не исследованных ранее и малоизученных источников различных биотопов морской среды, является актуальным. Микроорганизмы донных осадков шельфа Чукотского моря исследованы фрагментарно (Yuan et al., 2014). Цель настоящей работы – изучение филогенетического разнообразия и антимикробной активности гетеротрофных бактерий, изолированных из образцов донных осадков Чукотского моря.
МАТЕРИАЛ И МЕТОДИКА
Пробы донных осадков отбирали в экспедиционном рейсе НИС “Академик Опарин” в сентябре 2016 г. в российском секторе Чукотского моря на 11 станциях в координатах: № 1 (71°21.44′ N, 175° 36.70′ W; глубина 36 м), № 2 (70°59.60′ N, 177°35.8′ W; 29 м), № 2/1 (71°01.18′ N, 177°42.18′ W; 23 м), № 5 (70°50.0′ N, 179°42.0′ W; 18 м), № 6 (70°31.01′ N, 177°25.92′ W; 50 м), № 11 (71°20.0′ N, 173°04.90′ W; 50 м), № 14 (69°54.02′ N, 171°00.46′ W; 44 м), № 16 (68°11.02′ N, 169°10.83′ W; 58 м), № 17 (67°25.98′ N, 169°37.30′ W; 50 м), № 18 (67°30.99′ N, 171°21.80′ W; 48 м), № 20 (67°32.44′ N, 173°36.72′ W; 42 м).
Штаммы бактерий выделяли методом прямого посева суспензии образцов грунта в разведениях на среды Морской агар 2216 (Marine Agar 2216), R2А (R2А Agar), Триптиказо-соевый агар (Tryptic soya agar, все – BD Difco) и др., как описано ранее (Romanenko et al., 2013a). Выделенные бактерии помещены на хранение в Коллекцию морских микроорганизмов (КММ) ТИБОХ ДВО РАН.
Исследование антимикробной активности выделенных штаммов в отношении тестовых микроорганизмов Escherichia coli K-12 CL588, Enterococcus faecium CIP 104105, Staphylococcus aureus CIP 65.8T, Staphylococcus epidermidis CIP 81.55T, Bacillus subtilis CIP 52.65T, Xanthomonas sp. pv. badrii LMG 546 и Candida albicans KMM 455 проводили, как описано ранее (Romanenko et al., 2008).
Нативную ДНК выделяли при помощи Genomic DNA Purification Kit (Fermentas). Фрагмент гена 16S рРНК амплифицировали с универсальными праймерами 8F (5'-AGAGTTTGATCCTGGCTCAG-3') и 1492R (5'-ACGGCTACCTTGTTACGACTT-3'), как описано в предыдущей работе (Romanenko et al., 2019). Амплификацию фрагментов генов PKS-1 и NRPS проводили с использованием олигонуклеотидных праймеров MDPQQRF/HGTGTR и A3F/A7R соответственно, как описано ранее (Santos et al., 2015). Ожидаемые длины фрагментов генов для PKS-1 и NRPS составляли соответственно 700–800 и 600–700 п.н.
Очищенные ПЦР фрагменты гена 16S рРНК секвенировали с использованием ABI PRISM 3130xl Genetic Analyzer (Applied Biosystems, США) и Big Dye v.3.1 sequencing kit (Applied Biosystems, США). Процент сходства генов 16S рРНК с гомологичными генами типовых штаммов рассчитывали с помощью Eztaxon service (Kim et al., 2012). Филогенетический анализ проводили с использованием MEGA 6 (Tamura et al., 2013). Полученные нуклеотидные последовательности генов 16S рРНК были депонированы в базе данных DDBJ/EMBL/GenBank под номерами доступа LC379505-LC379542.
РЕЗУЛЬТАТЫ
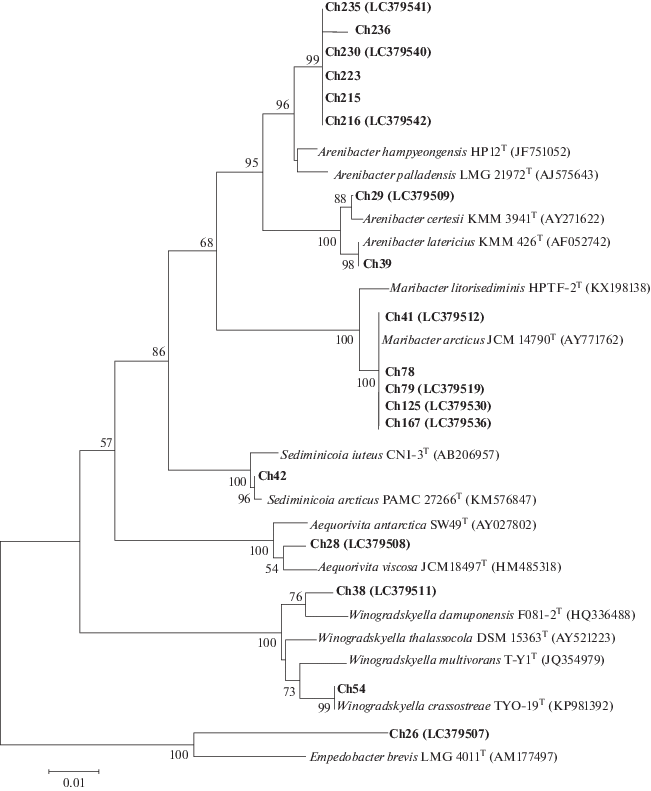
Из образцов донных осадков, отобранных в Чукотском море на 11 станциях на глубине от 18 до 58 м, были выделены 248 штаммов бактерий. Бактериальные изоляты сначала группировали по морфологическим свойствам колоний, КОН-тесту, данным микроскопии и физиологическим тестам; из них более 70 штаммов были отобраны для секвенирования гена 16S рРНК. Филогенетические позиции изученных штаммов представлены в дендрограммах (рис. 1–3). На основании анализа гена 16S рРНК исследованные штаммы были отнесены к четырем типам: Proteobacteria (34%), Bacteroidetes (26%), Firmicutes (21%) и Actinobacteria (19%), среди которых протеобактерии были доминантной группой. Класс Gammaproteobacteria был представлен бактериями родов Shewanella, Psychrobacter, Pseudoalteromonas и Halomonas. Среди представителей Alphaproteobacteria были идентифицированы штаммы трех родов – Loktanella, Sphingorhabdus и Ahrensia. Класс Betaproteobacteria был представлен родами Hydrogenophaga и Massilia (рис. 1). Вторыми по численности были штаммы филума Bacteroidetes семейства Flavobacteriaceae, включая Winogradskyella, Arenibacter и Maribacter как преобладающие группы, а также Sediminicola и Aequorivita как минорные группы (рис. 2). Таксономический состав выделенных бактерий, отнесенных к типу Firmicutes, характеризовался большим разнообразием и включал представителей девяти родов: Bacillus, Sporosarcina, Paenisporosarcina, Domibacillus, Fictibacillus, Paenibacillus, Terribacillus, Oceanobacillus и Virgibacillus. Многочисленными и разнообразными по родовому составу были также штаммы Actinobacteria, представленные бактериями 10 родов: Streptomyces, Rhodococcus, Aeromicrobium, Pseudonocardia, Amycolatopsis, Salinibacterium, Agrococcus, Arthrobacter, Cellulosimicrobium и Nocardiopsis (рис. 3). Филогенетический анализ показал, что большинство исследованных штаммов имели высокий уровень сходства (99–100%) в нуклеотидных последовательностях гена 16S рРНК с ранее известными видами бактерий, выделенными из морских и/или полярных источников. В то же время необходимо отметить, что штаммы родов Massilia (класс Betaproteobacteria), Fictibacillus и Terribacillus (Firmicutes) являются наземными микроорганизмами и редко выделяются из морских источников. По меньшей мере 8 групп, представленные штаммами Ch16, Ch26, Ch30, Ch38, Ch 100, Ch158/Ch160, Ch28 и Ch215/Ch235, показали уровень сходства гена 16S рРНК не более 97–98% и могут представлять новые таксоны. Так, штамм Ch26 имел только 92% сходства гена 16S рРНК с типовым штаммом Empedobacter brevis; штамм Ch100 показал 90% сходства с Pseudonocardia ammonioxydans, образуя удаленную монофилетическую ветвь среди актинобактерий. Филогенетически близкий к Winogradskyella damuponensis (97.2% сходства гена 16S рРНК) штамм Ch38 был предложен нами как новый вид Winogradskyella profunda (см.: Romanenko et al., 2019).
Рис. 1.
Филогенетическое дерево бактерий, изолированных из донных осадков Чукотского моря, отнесенных к типу Proteobacteria. Здесь и на рис. 2 и 3: филогенетическое дерево построено на основе сравнительного анализа нуклеотидных последовательностей гена 16S рРНК с использованием алгоритма neighbor-joining; статистическую достоверность ветвления оценивали с помощью bootstrap-анализа 1000 альтернативных деревьев. Масштаб соответствует двум нуклеотидным заменам на 100 нуклеотидов.
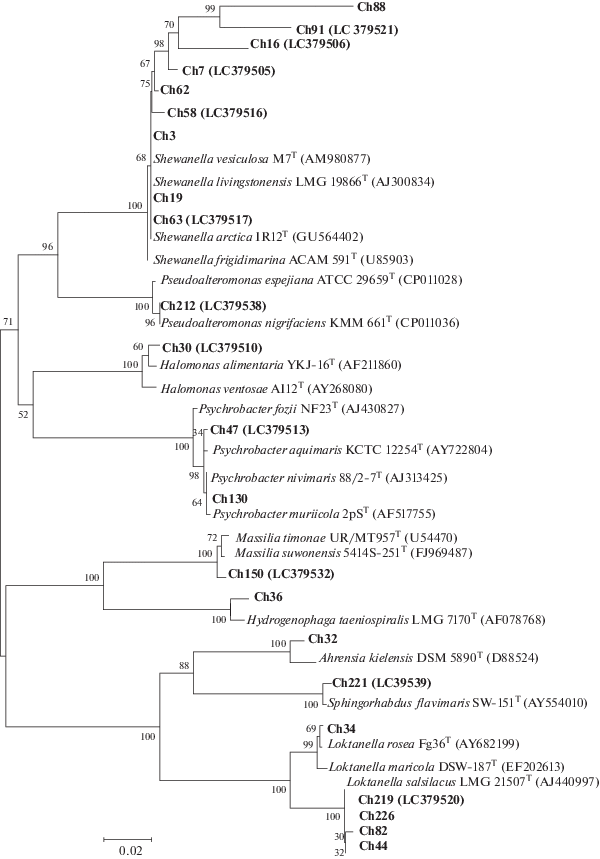
Рис. 2.
Филогенетическое дерево представителей Actinobacteria и Firmicutes, выделенных из донных осадков Чукотского моря. Масштаб соответствует двум нуклеотидным заменам на 100 нуклеотидов.
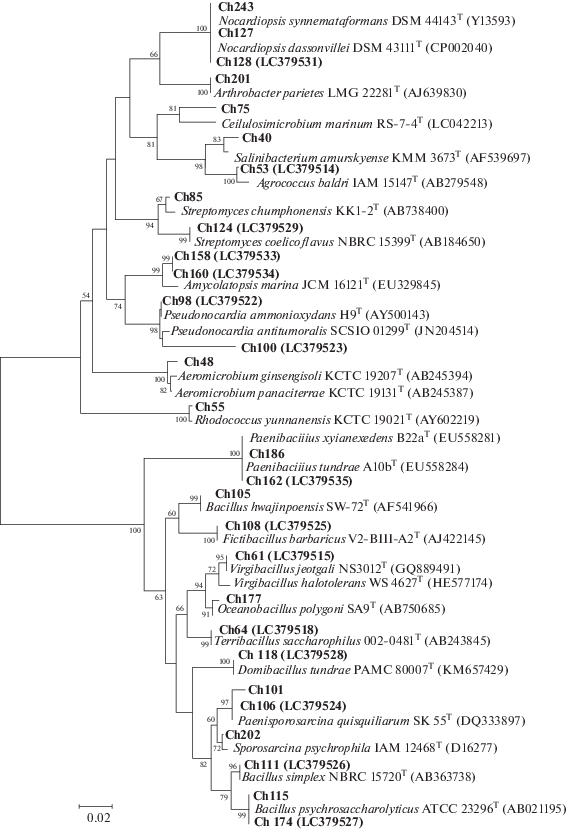
Рис. 3.
Филогенетическое дерево штаммов Bacteroidetes, изолированных из донных осадков Чукотского моря. Масштаб соответствует одной нуклеотидной замене на 100 нуклеотидов.
Скрининг антимикробной активности выявил 40 штаммов, которые были активны в отношении двух и более тест-культур. Большинство выделенных штаммов ингибировали рост B. subtilis, S. aureus, E. faecium, Xanthomonas sp. pv. badrii и C. albicans. Активность в отношении S. epidermidis и E. coli была незначительной. Активные штаммы были представлены грамположительными бактериями родов Bacillus, Paenibacillus, Terribacillus, Virgibacillus (тип Firmicutes), Streptomyces, Pseudonocardia, Cellulosimicrobium, Nocardiopsis (Actinobacteria) и в меньшей степени грамотрицательными бактериями Shewanella, Psychrobacter, Pseudoalteromonas (класс Gammaproteobacteria), Massilia (класс Betaproteobacteria) и Arenibacter (тип Bacteroidetes). Наиболее активные штаммы (табл. 1) были исследованы на наличие генов синтеза вторичных метаболитов: поликетидсинтаз (PKS-1) и нерибосомных пептидсинтетаз (NRPS) методом ПЦР с использованием вырожденных праймеров. Фрагменты гена NRPS были обнаружены в 10 штаммах следующих родов: Shewanella (Ch7, Ch18, Ch58), Loktanella (Ch44), Aeromicrobium (Ch48), Virgibacillus (Ch61), Cellulosimicrobium (Ch75), Streptomyces (Ch85) и Paenibacillus (Ch162, Ch186) (рис. 4). Фрагменты гена PKS-1 присутствовали в 16 штаммах родов Shewanella (Ch3), Arenibacter (Ch39, Ch216, Ch223, Ch235), Loktanella (Ch44), Massilia (Ch150), Virgibacillus (Ch61), Terribacillus (Ch64), Bacillus (Ch111), Streptomyces (Ch124), Oceanobacillus (Ch177), Amycolatopsis (Ch158, Ch160), Paenibacillus (Ch162) и Nocardiopsis (Ch243) (рис. 5).
Таблица 1.
Антимикробная активность бактерий, выделенных из донных осадков Чукотского моря
| Штамм | Филогенетически близкий вид | Сходство, % |
Зона ингибирования роста, мм | Фрагменты гена | |||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| Bacillus subtilis | Staphylococcus aureus | Staphylococcus epidermidis | Enterococ-cus faecium | Escherichia coli | Xanthomonas sp. pv. badrii | Candida albicans | NRPS | PKS-I | |||
| Ch3 | Shewanella livingstonensis | 98 | 12 | 14 | 14 | W* | 12 | – | – | – | + |
| Ch7 | Shewanella vesiculosa | 98 | 12 | 15 | 15 | 15 | 12 | – | – | + | – |
| Ch18 | Shewanella vesiculosa | 98 | 20 | 15 | – | W | – | 15 | 18 | + | – |
| Ch39 | Arenibacter latericius | 98 | – | – | – | 12 | – | – | 14 | – | + |
| Ch44 | Loktanella salsilacus | 98 | W | 12 | 12 | – | – | – | – | + | + |
| Ch48 | Aeromicrobium ginsengisoli | 98 | W | 12 | – | W | – | – | – | + | – |
| Ch58 | Shewanella arctica | 99 | 15 | 15 | – | 12 | – | W | 20 | + | – |
| Ch61 | Virgibacillus jeotgali | 97 | 12 | 12 | – | 12 | – | – | 12 | + | + |
| Ch64 | Terribacillus goriensis | 96 | W | W | – | 20 | – | – | 12 | – | + |
| Ch75 | Cellulosimicrobium marinum | 97 | 15 | 30 | – | 20 | – | – | 12 | + | – |
| Ch85 | Streptomyces chumphonensis | 98 | 28 | 32 | W | 35 | – | 20 | 30 | + | – |
| Ch111 | Bacillus simplex | 99 | – | W | – | – | – | W | 15 | – | + |
| Ch124 | Streptomyces coelicoflavus | 99 | 20 | 32 | 15 | 22 | – | 13 | 16 | – | + |
| Ch150 | Massilia timonae | 99 | 15 | 28 | – | W | – | – | W | – | + |
| Ch158 | Amycolatopsis marina | 97 | 22 | – | – | 14 | – | – | 12 | – | + |
| Ch160 | Amycolatopsis marina | 97 | 14 | 12 | – | – | – | 12 | 12 | – | + |
| Ch162 | Paenibacillus tundrae | 99 | W | – | – | 20 | – | 15 | 12 | + | + |
| Ch177 | Oceanobacillus polygoni | 99 | – | W | – | 14 | – | 15 | – | – | + |
| Ch186 | Paenibacillus tundrae | 99 | W | – | – | W | – | – | 12 | + | + |
| Ch216 | Arenibacter hampyeongensis | 98 | W | 15 | W | 12 | – | – | 20 | – | + |
| Ch223 | Arenibacter hampyeongensis | 98 | – | W | – | 15 | – | – | 12 | – | + |
| Ch235 | Arenibacter hampyeongensis | 98 | – | – | – | W | W | 12 | 15 | – | + |
| Ch243 | Nocardiopsis dassonvillei | 99 | 14 | 32 | – | 15 | – | 12 | 15 | – | + |
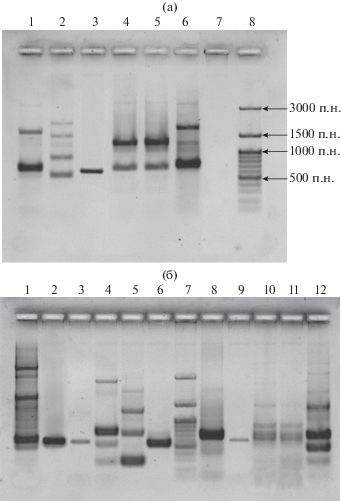
Рис. 4.
Обнаружение фрагментов генов NRPS в ДНК активных штаммов методом ПЦР. Штаммы бактерий: 2 – Ch7; 3 – Ch18; 4 – Ch44; 5 – Ch48; 6 – Ch58; 7 – Ch61; 8 – Ch75; 9 – Ch85; 10 – Ch162; 11 – Ch186; 12 – Sphingomonas molluscorum KMM 3882T (положительный контроль); 13 – отрицательный контроль; 1, 14 – ДНК маркер (от 100 до 3000 п.н.). Стрелками указаны размеры фрагментов маркерной ДНК.
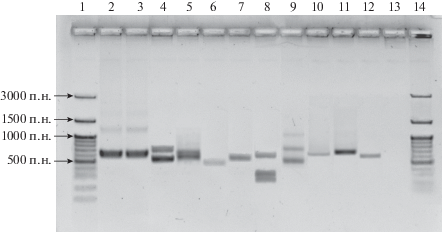
Рис. 5.
Обнаружение фрагментов генов PKS-1 в ДНК активных штаммов методом ПЦР. Штаммы бактерий: а) 1 – Ch243; 2 – Ch177; 3 – Ch216; 4 – Ch223; 5 – Ch235; 6 – Paenibacillus profundus KMM 9420T (положительный контроль); 7 – отрицательный контроль; 8 – ДНК маркер (от 100 до 3000 п.н.). Стрелками указаны размеры фрагментов маркерной ДНК. б) 1 – Сh3; 2 – Ch39; 3 – Ch44; 4 – Ch61; 5 – Ch64; 6 – Ch111; 7 – ДНК маркер (от 100 до 3000 п.н.); 8 – Ch124; 9 – Ch150; 10 – Ch158; 11 – Ch160; 12 – Ch162.
ОБСУЖДЕНИЕ
Полученные результаты по таксономическому составу бактерий донных осадков Чукотского моря согласуются с результатами исследований микроорганизмов донных осадков, полученных при использовании молекулярных методов на основе ПЦР-амплификации и создании библиотек клонов генов 16S рРНК и/или культуральных методов анализа (Li et al., 2009; Kouridaki et al., 2010). Показано, что Gammaproteobacteria и грамположительные бактерии с высоким содержанием Г + Ц были доминантными группами в микробных сообществах донных осадков двух заливов Японского моря (Urakawa et al., 1999). В исследовании Сюй с соавторами (Xu et al., 2008) Gammaproteobacteria (22.8%) и Alphaproteobacteria (16.5%) преобладали среди бактерий донных осадков северо-восточной части Тихого океана. При изучении бактерий донных осадков Японского моря были обнаружены Proteobacteria (38%), Firmicutes (27%), Actinobacteria (21%) и Bacteroidetes (14%) (Romanenko et al., 2013a). Следует отметить, что бактерии семейства Flavobacteriaceae, представители которого широко распространены в морской среде, часто выделяются из образцов донных осадков (Alonso et al., 2007).
В процессе скрининга было выявлено 40 штаммов с антимикробной активностью, большинство из них относятся к типам Firmicutes (Bacillus, Paenibacillus, Terribacillus, Virgibacillus) и Actinobacteria (Streptomyces, Pseudonocardia, Cellulosimicrobium, Nocardiopsis). Штаммы Proteobacteria и Bacteroidetes были менее активны. Анализ литературных данных и собственные исследования показывают, что многие представители родов Bacillus, Paenibacillus, Streptomyces и Nocardiopsis являются штаммами-антагонистами и служат продуцентами БАВ с антимикробными свойствами (Bull, Stach, 2007; Romanenko et al., 2013б). Юань с соавторами (Yuan et al., 2014) изучали родовой состав и активность штаммов Actinobacteria, изолированных из образцов донных осадков Чукотского моря (Канадский бассейн) в летний период 2010 г. Среди выделенных актинобактерий были идентифицированы штаммы 14 родов, в том числе Arsenicicoccus, Citricoccus, Kocuria и Saccharopolyspora, а также редко обнаруживаемый в морской среде род Microlunatus; антимикробную активность показали штаммы Streptomyces, Nocardiopsis и Microlunatus (Yuan et al., 2014). В нашем исследовании мы не обнаружили штаммов из пяти указанных выше родов актинобактерий, но штаммы Streptomyces и Nocardiopsis были также наиболее активными и ингибировали рост наибольшего числа индикаторных микроорганизмов. Активными были штаммы Amycolatopsis и Cellulosimicrobium, но показали ограниченный спектр антимикробной активности. Сведения об антимикробной активности бактерий родов Shewanella и Psychrobacter очень ограничены и отсутствуют для штаммов Arenibacter (тип Bacteroidetes). Мы не обнаружили данных по антимикробной активности бактерий Shewanella, выделенных из морских донных осадков, а также об активности шеванелл, входящих в кластер S. vesiculosa/S. frigidimarina. Показано, что штаммы Shewanella с антимикробной активностью присутствуют в микробных сообществах, ассоциированных с морскими беспозвоночными, где представлены видами S. baltica и S. putrefaciens (см.: Heindl et al., 2010; Pham et al., 2016).
Многие биологически активные метаболиты морских бактерий относятся к двум разнообразным классам соединений: поликетиды (ПК) и нерибосомные пептиды (НРП), которые конструируются с помощью мультимодулярных ферментов поликетидсинтаз (PKS) и нерибосомных пептидсинтетаз (NRPS) (см.: Williams, 2013). Показано, что бактерии из морских источников синтезируют около 70% НРП, которые обладают антимикробным, противовирусным, цитостатическим, противомалярийным или противопаразитарным действием, а также используются как стимуляторы роста животных и природные инсектициды (Agrawal et al., 2017). Анализ литературных данных свидетельствует о том, что метод ПЦР-скрининга генов PKS и NRPS используется для исследования потенциальной способности микроорганизмов различных таксономических групп синтезировать ПК и НРП. ПЦР-скрининг показал наличие генов PKS-1 и/или NRPS в 23 активных штаммах, что подтверждает возможность исследуемых штаммов синтезировать вещества поликетидной природы и нерибосомные пептиды. Для дальнейшего исследования планируется выделение метаболитов, продуцируемых активными штаммами, установление их химической структуры и изучение биологической активности.
Настоящее исследование вносит вклад в изучение биоразнообразия и антимикробной активности культивируемых микроорганизмов донных осадков Чукотского моря. Проведенное исследование показывает, что донные осадки Чукотского моря могут служить источником выделения новых таксонов морских бактерий и штаммов-продуцентов антимикробных веществ.
Список литературы
Agrawal S., Acharya D., Adholeya A. et al. Nonribosomal Peptides from Marine Microbes and Their Antimicrobial and Anticancer Potential // Front. Pharmacol. 2017. V. 8. P. 828. https://doi.org/10.3389/fphar.2017.00828
Alonso C., Warnecke F., Amann R., Pernthaler J. High local and global diversity of flavobacteria in marine plankton // Environ. Microbiol. 2007. V. 9. № 5. P. 1253–1266.
Bhatnagar I., Kim S.K. Pharmacologically prospective antibiotic agents and their sources: A marine microbial perspective // Environ. Toxicol. Pharmacol. 2012. V. 34. № 3. P. 631–643.
Bull A.T., Stach J.E.M. Marine actinobacteria: new opportunities for natural product search and discovery // Trends in Microbiology. 2007. V. 15. № 11. P. 491–499.
Debbab A., Aly A.H., Lin W.H., Proksch P. Bioactive Compounds from Marine Bacteria and Fungi // Microb. Biotech. 2010. V. 3. № 5. P. 544–563.
Heindl H., Wiese J., Thiel V., Imhoff J.F. Phylogenetic diversity and antimicrobial activities of bryozoan-associated bacteria isolated from Mediterranean and Baltic Sea habitats // Syst. Apll. Microbiol. 2010. V. 33. № 2. P. 94–104.
Kim O.S., Cho Y.J., Lee K. et al. Introducing EzTaxon-e: a prokaryotic 16S rRNA Gene sequence database with phylotypes that represent uncultured species // Int. J. Syst. Evol. Microbiol. 2012. V. 62. P. 716–721.
Kouridaki I., Polymenakou P.N., Tselepides A. et al. Phylogenetic diversity of sediment bacteria from the deep Northeastern Pacific Ocean: a comparison with the deep Eastern Mediterranean Sea // Int. Microbiol. 2010. V. 13. № 3. P. 143–150.
Lauro F.M., Bartlett D.H. Prokaryotic lifestyles in deep sea habitats // Extremophiles. 2008. V. 12. P. 15–25.
Li H., Yu Y., Luo W. et al. Bacterial diversity in surface sediments from the Pacific Arctic Ocean // Extremophiles. 2009. V. 13. № 2. P. 233–246.
Penesyan A., Kjelleberg S., Egan S. Development of novel drugs from marine surface associated microorganisms // Mar. Drugs. 2010. V. 8. P. № 3. 438–459.
Pettit R.K. Culturability and secondary metabolite diversity of extreme microbes: expanding contribution of deep sea and deep-sea vent microbes to natural product discovery // Mar. Biotechnol. 2011. V. 13. P. 1–11.
Pham T.M., Wiese J., Wenzel-Storjohann A., Imhoff J.F. Diversity and antimicrobial potential of bacterial isolates associated with the soft coral Alcyonium digitatum from the Baltic Sea // ResearchGate, in Antonie van Leeuwenhoek. 2016. V. 109. № 1. P. 105–119.
Romanenko L.A., Uchino M., Kalinovskaya N.I., Mikhailov V.V. Isolation, phylogenetic analysis and screening of marine mollusc-associated bacteria for antimicrobial, hemolytic and surface activities // Microbiol. Res. 2008. V. 163. № 6. P. 633–644.
Romanenko L.A., Tanaka N., Kalinovskaya N.I., Mikhailov V.V. Antimicrobial potential of deep surface sediment associated bacteria from the Sea of Japan // WJMB. 2013a. V. 29. P. 1169–1177.
Romanenko L.A., Tanaka N., Svetashev V.I., Kalinovskaya N.I. Paenibacillus profundus sp. nov., a deep sediment bacterium that produces isocumarin and peptide antibiotics // Arch. Microbiol. 2013b. V. 195. P. 247–254.
Romanenko L.A., Kurilenko V.V., Guzev K.V. et al. Winogradskyella profunda sp. nov. isolated from the Chukchi Sea bottom sediments // Arch. Microbiol. 2019. V. 201. № 1. P. 45–50.
Santos O.C.S., Soares A.R., Machado F.L.S. et al. Investigation of biotechnological potential of sponge-associated bacteria collected in Brazilian coast // Lett. Appl. Microbiol. 2015. V. 60. № 2. P. 140–147.
Tamura K., Stecher G., Peterson D. et al. MEGA6: Molecular Evolutionary Genetics Analysis Version 6.0 // Mol. Biol. Evol. 2013. V. 30. № 12. P. 2725–2729.
Urakawa H., Kita-Tsukamoto K., Ohwada K. Microbial diversity in marine sediments from Sagami Bay and Tokyo Bay, Japan, as determined by 16S rRNA gene analysis // Microbiology. 1999. V. 145. P. 3305–3315.
Williams G. Engineering polyketide synthases and nonribosomal peptide synthetases // Curr. Opin. Struct. Biol. 2013. V. 23. № 4. P. 603–612.
Xu H., Wu M., Wang X. et al. Bacterial diversity in deep-sea sediment from northeastern Pacific Ocean // Acta Ecologica Sinica. 2008. V. 28. № 2. P. 479–485.
Yuan M., Yu Y., Li H.R. et al. Phylogenetic diversity and biological activity of Actinobacteria isolated from the Chukchi shelf marine sediments in the Arctic Ocean // Mar. Drugs. 2014. V. 12. № 3. P. 1281–1297.
Дополнительные материалы отсутствуют.


