Биология моря, 2023, T. 49, № 2, стр. 135-142
Сравнительный филогеографический анализ изменчивости митохондриальной ДНК четырех видов рыб (Salmonidae и Cyprinidae) в Северо-Западной Пацифике
Вл. А. Брыков 1, *, В. В. Маляр 1
1 Национальный научный центр морской биологии им. А.В. Жирмунского (ННЦМБ) ДВО РАН
690041 Владивосток, Россия
* E-mail: vlbrykov@mail.ru
Поступила в редакцию 31.05.2022
После доработки 12.11.2022
Принята к публикации 24.11.2022
- EDN: DVBEAX
- DOI: 10.31857/S0134347523020031
Аннотация
Исследована генетическая изменчивость митохондриальных генов COI и cytb в выборках крупночешуйной красноперки Tribolodon hakonensis (Günther, 1877) и мелкочешуйной красноперки Tribolodon brandtii (Dybowski, 1872) у побережья о-ва Сахалин и материкового побережья Японского моря. Выявленные закономерности сопоставлены с данными, полученными на сходном ареале для сахалинского тайменя Parahuсho perryi (Brevoort, 1856) и кеты Oncorhynchus keta (Walbaum, 1792). Сравнительный анализ показал, что формирование внутривидовых линий мтДНК у четырех видов рыб связано с палеоклиматическими изменениями в регионе северо-западной Пацифики в период плейстоцена и в значительной степени синхронизировано по времени. Во многих случаях рассчитанное время дивергентной эволюции генеалогических линий мтДНК совпадает, что говорит о влиянии сходных факторов эволюции на исследованные виды.
Сравнительный анализ генетической изменчивости видов в географических регионах позволяет выяснить наличие внутривидовой структуры, определить время дивергенции основных генеалогических линий и выявить наиболее существенные факторы, которые влияли в прошлом или влияют в настоящее время на популяционно-генетическую структуру видов (Mayr, 1963; Avise, 2000).
Благодаря материнскому наследованию, быстрой эволюции и, как правило, отсутствию рекомбинаций, митохондриальная ДНК (мтДНК) представляет собой наиболее удобную молекулу для эволюционного и популяционного анализа (Brown et al., 1979). Филогеографический подход, основанный на анализе изменчивости мтДНК, оказался универсальным для понимания путей и механизмов формирования таксонов и отдельных внутривидовых единиц (Templeton et al., 1995; Avise, 2000). Ограничение генетического обмена между популяциями приводит к появлению различий в мтДНК особей в разных популяциях в результате накопления мутаций и стохастических процессов, а продолжительное независимое существование популяций во времени – к возникновению отличающихся филогрупп мтДНК. В дальнейшем при формировании видов эти различия увеличиваются (Avise et al, 1998). Филогеографический подход на основе анализа изменчивости мтДНК позволяет реконструировать события дивергенции внутри видов и групп видов и отнести их к тому или иному историческому периоду, используя молекулярные часы (Avise, 2000; Crête-Lafrenière et al., 2012).
Цель настоящей работы – анализ генетической изменчивости мтДНК у двух эндемичных дальневосточных полупроходных видов рыб, красноперок Tribolodon hakonensis (Günther, 1877) и Tribolodon brandtii (Dybowski, 1872) (семейство Cyprinidae) и сравнение полученных данных с ранее опубликованными результатами по проходным видам: кете Oncorhynchus keta (Walbaum, 1792) и сахалинскому тайменю Parahuсho perryi (Brevoort, 1856).
МАТЕРИАЛ И МЕТОДИКА
Рыб отлавливали ставной сетью или на удочку. Анализ включал особей мелкочешуйной красноперки Tribolodon brandtii из выборок материкового побережья Японского моря (зал. Восток) и о-ва Сахалин (соединенное протокой с морем оз. Тунайча, побережье Охотского моря), а также выборки крупночешуйной красноперки Tribolodon hakonensis с материкового побережья Японского моря (зал. Восток) и о-ва Сахалин (устье р. Ильинка).
Кусочки тканей плавников фиксировали в 96%-ном этаноле. Выделение ДНК, условия амплификации и определение нуклеотидных последовательностей фрагментов генов COI и cytb мтДНК описаны ранее (Маляр, Брыков, 2016).
Множественное выравнивание и анализ нуклеотидных последовательностей исследуемых фрагментов генов осуществляли с использованием программного обеспечения MEGA 5.05 (Tamura et al., 2011) по алгоритму CLUSTAL W (Thompson et al., 1994). Выравненные последовательности генов для каждой особи исследуемых видов объединяли в комбинированные последовательности. Подбор оптимальной модели нуклеотидного замещения для каждого раздела данных осуществляли в программе jModelTest 2.1.7 (Darriba et al., 2012), используя байесовский информационный критерий (BIC).
Для визуализации разных филогрупп мтДНК построены медианные сети гаплотипов в программе SplitsTree4 (Huson, Bryant, 2006) по алгоритму MJ (Median-Joining) (Bandelt et al., 1999). Значения гаплотипического и нуклеотидного разнообразия для каждой популяции, а также распределение различий между гаплотипами (mismatch distribution) рассчитывали, используя программу DnaSP v5.10 (Librado, Rozas, 2009).
Внутривидовые генеалогии и датировки дивергенции устанавливали с помощью байесовского подхода в пакете программ BEAST 1.10.4 (Drummond et al., 2012) на базе высокопроизводительного кластера CIPRES (Miller et al., 2010) при следующих условиях: диапазон значений скоростей эволюции с минимальной границей 0.76% за 1 млн лет (Zardoya, Doadrio, 1999) и максимальной границей на уровне 1.7% за 1 млн лет (Stepien et al., 2000); значения параметра ucld.mean с нормальным распределением; модель нуклеотидного замещения для последовательностей COI крупночешуйной и мелкочешуйной красноперки – HKY, cytb крупночешуйной красноперки – TrN, мелкочешуйной красноперки – TrN+I; модель молекулярных часов – свободные часы с некоррелированным логарифмически нормальным распределением; длина цепи Маркова равна 30 млн поколений с отбором каждого тысячного состояния и значением burn-in 10%, суммирование деревьев по алгоритму maximum clade credibility.
Дополнительные филогенетические реконструкции комбинированных последовательностей кеты проводили по методу ближайшего связывания (NJ) и максимального правдоподобия (ML) в программе MEGA 5.05.
Последовательности фрагментов генов COI и cytb крупночешуйной и мелкочешуйной красноперок депонированы в GenBank NCBI под номерами KY615261–KY615279, KY615280–KY615298; KY615299–KY615318 и KY615319–KY615338.
РЕЗУЛЬТАТЫ
Крупночешуйная красноперка Tribolodon hakonensis
Длина полученных нуклеотидных последовательностей составила 1623 нуклеотидные пары. Анализ последовательностей у исследованных особей выявил наличие 12 гаплотипов мтДНК (табл. 1). Выборка крупночешуйной красноперки из р. Ильинка характеризуется наибольшим количеством гаплотипов (9). В материковой выборке насчитывается всего 3 гаплотипа.
Таблица 1.
Встречаемость гаплотипов мтДНК в исследуемых популяциях крупночешуйной красноперки Tribolodon hakonensis
| Гаплотип | Популяции | Число особей | |
|---|---|---|---|
| зал. Восток | р. Ильинка | ||
| TH1 | 0 | 2 (100) | 2 |
| TH2 | 0 | 1 (100) | 1 |
| TH3 | 0 | 1 (100) | 1 |
| TH4 | 0 | 1 (100) | 1 |
| TH5 | 0 | 1 (100) | 1 |
| TH6 | 7 (100) | 0 | 7 |
| TH7 | 0 | 1 (100) | 1 |
| TH8 | 0 | 1 (100) | 1 |
| TH9 | 0 | 1 (100) | 1 |
| TH10 | 0 | 1 (100) | 1 |
| TH11 | 1 (100) | 0 | 1 |
| TH12 | 1 (100) | 0 | 1 |
Гаплотипы, обнаруженные в выборках р. Ильинка и зал. Восток, не перекрываются и встречаются только в одной из выборок. В материковой популяции выделяется наиболее частый гаплотип TH6, объединяющий 7 особей выборки. Гаплотипы сахалинской популяции из р. Ильинка (TH1–TH5, TH7–TH10) практически все уникальны.
Распределение гаплотипов подтверждается значениями гаплотипической и нуклеотидной изменчивости. Наиболее высокие значения гаплотипической (0.978 ± 0.054) и нуклеотидной (0.00704) изменчивости выявлены в выборке из р. Ильинка о-ва Сахалин, в материковой выборке из зал. Восток эти значения меньше практически в два раза, 0.417 ± 0.191 и 0.00027 соответственно.
В результате использования различных алгоритмов филогенетического анализа получены деревья с общей топологией (рис. 1б). Анализ показал наличие трех выраженных кластеров особей Tribolodon hakonensis с достоверными значениями бутстреп-поддержки и апостериорных вероятностей. В кластер А вошли все исследованные гаплотипы особей с о-ва Сахалин (р. Ильинка), в то время как кластер B объединил гаплотипы особей материкового побережья (зал. Восток). Клада А включает субклады А1 и А2 с высокими показателями достоверности по всем использованным алгоритмам.
Рис. 1.
Генеалогическая сеть гаплотипов, полученная на основании данных анализа нуклеотидных последовательностей генов COI и cytb мтДНК крупночешуйной красноперки Tribolodon hakonensis (а), и байесовское консенсусное филогенетическое дерево, отражающее генетическую изменчивость популяций и время дивергенции основных генеалогических линий Tribolodon hakonensis (б). Размеры окружностей пропорциональны числу особей с указанным гаплотипом. Доля особей кеты с материкового побережья Японского моря отмечена серым цветом. На ветвях сети указано количество нуклеотидных замен между группами гаплотипов. Обозначения гаплотипов как в табл. 1. Достоверность ветвления указана в виде величин апостериорных вероятностей (BI) и бутстреп-индексов (% от 1000 реплик) объединения соседей (NJ) и максимального правдоподобия (ML) (PP/BP/BP).
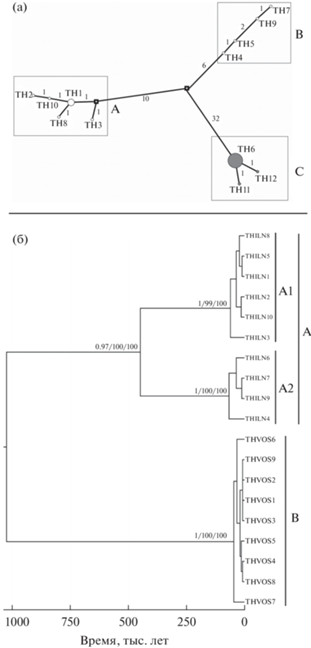
При реконструкции сети гаплотипов выделены три группы особей T. hakonensis (А, B и С) (рис. 1а). Филогруппы А и С содержат по одному центральному гаплотипу (TH1 и TH6). Остальные гаплотипы отличаются от них 1–2 нуклеотидными заменами. Филогруппа B объединяет гаплотипы TH4, TH5, TH9 и TH7, которые различаются 1–2 нуклеотидными заменами. Все гаплотипы из филогруппы C характерны для особей крупночешуйной красноперки из зал. Восток (материковое побережье), в то время как филогруппы A и B встречаются у особей в популяции из р. Ильинка (о-в Сахалин). Гаплотипы группы A отделяются 16 нуклеотидными заменами от ближайшего гаплотипа из филогруппы B. Материковая филогруппа C в значительной степени удалена от групп A и B на 42 и 38 нуклеотидных замен соответственно.
Мелкочешуйная красноперка Tribolodon brandtii
Длина полученных нуклеотидных последовательностей составила 1564 нуклеотидные пары. Анализ нуклеотидных последовательностей у исследованных особей мелкочешуйной красноперки выявил 6 гаплотипов (табл. 2). Популяция мелкочешуйной красноперки материкового побережья из зал. Восток включает наибольшее количество гаплотипов (5), характерных только для данной популяции. При этом чаще всего встречается гаплотип TB1, тогда как гаплотипы TB2, TB3 TB4 и TB6 уникальны. Особи из оз. Тунайча характеризуются одним гаплотипом TB5. Общих гаплотипов среди исследованных особей из выборок материкового побережья и о-ва Сахалин не обнаружено.
Таблица 2.
Встречаемость гаплотипов мтДНК в исследуемых популяциях мелкочешуйной красноперки Tribolodon brandtii
| Гаплотип | Популяции | Число особей | |
|---|---|---|---|
| зал. Восток | оз. Тунайча | ||
| TB1 | 8 (100) | 0 | 8 |
| TB2 | 1 (100) | 0 | 1 |
| TB3 | 1 (100) | 0 | 1 |
| TB4 | 1 (100) | 0 | 1 |
| TB5 | 0 | 8 (100) | 8 |
| TB6 | 1(100) | 0 | 1 |
Высокие значения гаплотипической (0.576 ± 0.163) и нуклеотидной (0.00227) изменчивости характерны для популяции из зал. Восток. В сахалинской выборке из оз. Тунайча генетической изменчивости не выявлено.
Топология филогенетических деревьев, реконструированных по объединенным последовательностям мтДНК мелкочешуйной красноперки, была идентичной при использовании разных алгоритмов построения (рис. 2б). Анализ выявляет наличие трех обособленных кластеров особей T. brandtii. Клада B включает особей красноперок из оз. Тунайча (о-в Сахалин). В кладе А выборки из материковой популяции обнаруживаются субклады А1 и A2, формирование которых поддержано относительно слабо. Только в случае байесовской филограммы значение апостериорных вероятностей оказалось близким к достоверным (0.94).
Рис. 2.
Генеалогическая сеть гаплотипов, полученная на основании данных анализа нуклеотидных последовательностей генов COI и cytb мтДНК мелкочешуйной красноперки Tribolodon brandtii (а), и байесовское консенсусное филогенетическое дерево, отражающее генетическую изменчивость популяций и время дивергенции основных генеалогических линий Tribolodon brandtii (б). Размеры окружностей пропорциональны числу особей с указанным гаплотипом. Доля особей кеты с материкового побережья Японского моря отмечена серым цветом. На ветвях сети указано количество нуклеотидных замен между группами гаплотипов. Обозначения гаплотипов как в табл. 1. Достоверность ветвления указана в виде величин апостериорных вероятностей (BI) и бутстреп-индексов (% от 1000 реплик) объединения соседей (NJ) и максимального правдоподобия (ML) (PP/BP/BP).
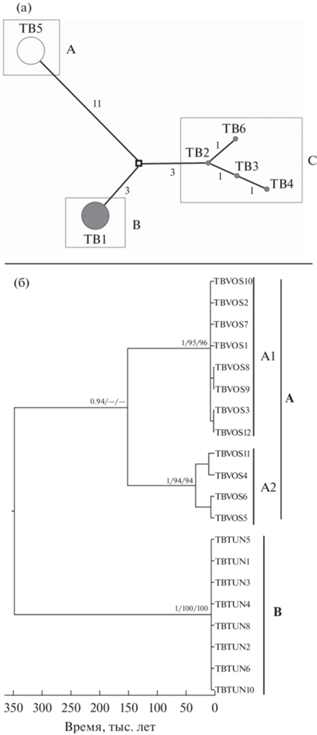
Полученные сети гаплотипов свидетельствуют о присутствии среди особей мелкочешуйной красноперки трех филогрупп (A, B и C) (рис. 2а). Особи материкового побережья из зал. Восток формируют группы B и C с центральными гаплотипами TB1 и TB2 соответственно. Данные группы отличаются в среднем шестью нуклеотидными заменами. Гаплотипы в филогруппе C отличаются 1–2 нуклеотидными заменами. Группа A включает всех представленных особей мелкочешуйной красноперки с о-ва Сахалин и отделяется от материковых групп 14 нуклеотидными заменами.
ОБСУЖДЕНИЕ
Закономерности пространственного распределения генетической изменчивости
Характеристика и сравнение распределения генетической изменчивости у двух видов красноперок рода Tribolodon (семейство Cyprinidae) и двух видов лососевых рыб (семейство Salmonidae) (Маляр, Брыков, 2016), имеющих разные истории происхождения и в значительной степени отличающихся биологическими характеристиками, ареалами распространения и численностью, позволили выявить как особенности, так и сходство их формирования и распространения на исследованном ареале.
Известно, что общий уровень генетической изменчивости определяется несколькими параметрами: временем существования вида, ареалом, численностью и историческими изменениями численности (Martin, Palumbi, 1993; Avise, 2000). Несмотря на то, что исследованные нами виды отличаются по этим параметрам, общие значения гаплотипической изменчивости внутри видов высоки. Для крупночешуйной красноперки характерно более длительное существование как вида среди других таксонов в роде Tribolodon (приблизительно 9 млн лет, при этом среднее значение времени дивергенции внутри рода составляет около 4–6 млн лет) (Sasaki et al., 2007; Watanabe et al., 2018). Как следствие, у данного вида более высокие оценки гаплотипической и нуклеотидной изменчивости (0.871 ± 0.00525 и 0.01528 соответственно), чем у мелкочешуйной красноперки (0.705 ± 0.00488 и 0.00542). Исследование биологии этих рыб на юге Приморского края и результаты мечения позволяют предположить, что дальневосточным красноперкам свойственны определенные места нагула, т.е. они, вероятно, обладают способностью различать водоемы и возвращаться после нагула в одни и те же реки для нереста и зимовки (Большаков, 2013).
При сравнении генетической изменчивости у четырех видов рыб из разных географических локальностей обнаруживаются определенные закономерности. Так, например, уровень нуклеотидного разнообразия в сахалинских популяциях тайменя Parahuсho perryi на порядок превышает нуклеотидное разнообразие популяций материкового побережья. Одна из внутривидовых линий мтДНК локализована исключительно в популяции зал. Набильский, а популяция из оз. Айнское характеризуется наивысшим гаплотипическим разнообразием на о-ве Сахалин (Маляр, Брыков, 2016).
Сходная закономерность обнаруживается у крупночешуйной красноперки, уровень гаплотипической изменчивости сахалинской выборки которой в два раза превышает значения для популяции материкового побережья (табл. 1). ПДРФ-анализ множества выборок крупночешуйной красноперки Tribolodon hakonensis при сравнении географически удаленных популяций также показал значительные различия, связанные с изоляцией расстоянием и, следовательно, с ограничением потока генов. По результатам AMOVA теста 98% изменчивости приходилось на внутрипопуляционную компоненту (Брыков и др., 2013).
Значительная удаленность материковой филогруппы C в сети гаплотипов обусловлена присутствием “южной формы”, выделяемой некоторыми авторами в отдельный близкий вид Tribolodon sp. (Гудков и др., 2010; Рязанова, Полякова, 2012; Маляр, 2017) (рис. 1а). Относительная молодость вида выражается низким уровнем гетерогенности мтДНК (зал. Восток, табл. 1), что также соответствует данным по расширенным выборкам в предыдущих работах (Семина и др., 2006; Брыков и др., 2013). У первоописанного вида T. hakonensis (“северная форма”) в популяциях о-ва Сахалин выявляются две сильнодивергировавшие эволюционные линии (филогруппы A и B, рис. 1а).
Иной характер распределения показателей генетического разнообразия обнаружен в популяциях кеты и мелкочешуйной красноперки, у которых высокий уровень гаплотипического и нуклеотидного разнообразия зафиксирован в материковых популяциях Приморья и существенно более низкий − в сахалинских (Маляр, Брыков, 2016).
Распределение генетического разнообразия в выборках мелкочешуйной красноперки совпадает с полученными ранее данными ПДРФ-анализа мтДНК. По данным Брыкова с соавторами (2013), значительная часть изменчивости приходилась на различия между сахалинской выборкой (зал. Анива) и материковыми популяциями.
Высокая изменчивость особей материковой выборки и топология филогенетических реконструкций мелкочешуйной красноперки подтверждают существование предкового рефугиума на территории материкового побережья Японского моря (рис. 2б). Наличие материковых филогрупп гаплотипов B и C (рис. 2а) указывает на присутствие как минимум двух предковых линий мелкочешуйной красноперки. Ранее наличие сходной популяционно-генетической структуры вида было установлено с помощью методов ПДРФ-анализа мтДНК и изменчивости аллозимов (Брыков и др., 2013; Sakai et al., 2002).
Как следует из полученных ранее данных, у кеты филогруппы в значительной степени разделены в пространстве. Показано, что все выявленные гаплотипы из одной филогруппы обнаруживаются в материковых популяциях Японского моря, а большая часть гаплотипов другой филогруппы – в выборке Охотского моря (Маляр, Брыков, 2016). Более полное исследование изменчивости мтДНК кеты, включающее бόльшую часть ареала, выявило четыре статистически значимо выраженные группы популяций: побережье Японского моря, побережье Охотского моря и западная зона Берингова моря, северо-запад Аляски и зал. Аляска (Yoon et al., 2008).
Так как наибольшее генетическое разнообразие у видов встречается в регионах, расположенных в местах возникновения видов или в предполагаемых предковых рефугиумах, то центром происхождения (или рефугиумом в период оледенений) для сахалинского тайменя и крупночешуйной красноперки, вероятно, был о-в Сахалин (Охотское море), а для кеты и мелкочешуйной красноперки – материковое побережье Японского моря.
Таким образом, сравнительный анализ гаплотипических сетей у четырех видов показал наличие, по крайней мере, двух больших филогрупп гаплотипов. Образование филогрупп определялось изоляцией популяций в прошлой истории вида, а генетический обмен между ними отсутствовал или был незначителен на протяжении многих поколений. Такой сценарий формирования двух и более филогрупп подтверждается в случае кеты и мелкочешуйной красноперки, где выявленные филогруппы до сих пор в значительной мере разобщены в пространстве. Иная картина наблюдается в случае крупночешуйной красноперки и тайменя, где отличающиеся филогруппы встречаются в одних и тех же выборках. Для такого распределения мы предполагаем тот же аллопатрический сценарий формирования филогрупп, в ходе которого накопление мутаций происходило в генетически независимых популяциях. Однако впоследствии, в силу палеогеологических событий в регионе и биологических особенностей видов (например, бóльшая подвижность или меньший уровень “хоминга”), произошло объединение особей с отличающимися филогруппами мтДНК в одну панмиксную популяцию. Таким образом, события географического разделения в прошлом отразились в митохондриальном геноме видов.
Датировка этапов дивергенции филогрупп мтДНК гаплотипов
Байесовские хронограммы исследованных видов рода Tribolodon позволяют выделить несколько этапов дивергенции (табл. 3). Начальный этап формирования эволюционных линий крупночешуйной красноперки связан с дивергенцией “северной” (о-в Сахалин) и “южной” (материковое побережье, Приморье) форм около 1 млн лет назад, что соответствует видовому уровню. Второй этап датирован 450 тыс. лет назад, третий – около 70–10 тыс. лет. У мелкочешуйной красноперки первый этап соответствует 350 тыс. лет назад, второй – 150 тыс. лет и третий – около 35–10 тыс. лет.
Полученные оценки времени дивергенции филогенетических линий мтДНК у дальневосточных красноперок соответствуют плейстоценовой эпохе. Как отмечалось в предыдущих наших работах, в периоды глубоких климатических изменений плейстоцена в регионе северо-западной Пацифики заметно менялись основные характеристики Охотского и Японского морей. Снижение уровня океана в результате осцилляций климата и частичная или полная изоляция от океана с опреснением внутренних морей могли быть основными факторами, определяющими дивергенцию пресноводных и морских рыб. В результате подобных преобразований менялись такие значимые факторы как температура и соленость (Korotky et al., 1997; Каплин, Селиванов, 1999).
При сравнении полученных датировок образования генеалогических линий красноперок с данными по другим видам выявляются общие временные этапы (табл. 3). Так, у сахалинского тайменя и крупночешуйной красноперки близкие значения периодов дивергенции, которые датируются 450–430 тыс. лет назад и приходятся на миндельский (окский) ледниковый период (478–424 тыс. лет назад). У сахалинского тайменя и кеты – практически идентичные значения в 220 тыс. лет, датируемые рисским (днепровским) ледниковым периодом (347–130 тыс. лет назад) (Маляр, Брыков, 2016). Близок к этим оценкам период дивергенции сахалинских и материковых популяций у мелкочешуйной красноперки – 350 тыс. лет, приуроченный к окончанию межледниковой эпохи на границе миндель-рисс. Предпоследний период дивергенции и формирования внутривидовых линий мтДНК у трех из четырех обсуждаемых видов приходится на период 150−70 тыс. лет назад и совпадает с вюрмским (валдайским) оледенением (110–12 тыс. лет назад). Последний этап “взрывного” образования гаплотипов приходится на период 70−10 тыс. лет назад и, очевидно, связан с отступлением ледников и расширением ареалов у исследуемых видов в голоцене (Маляр, 2017).
Таким образом, полученные нами данные дают основание считать, что палеоклиматические и, возможно, палеогеологические процессы в прошлой истории региона оказывали значительное влияние на дивергентную эволюцию ихтиофауны. В ряде случаев обнаруживаются близкие временные значения периодов внутривидовой дивергенции и сходные закономерности распределения генетической изменчивости у разных видов. Это может свидетельствовать, что в эти периоды факторы дивергенции влияли на разные виды сходным образом.
Список литературы
Большаков С.Г. Некоторые особенности биологии, рост и возраст мелкочешуйной Tribolodon brandtii и крупночешуйной T. hakonensis дальневосточных красноперок на юге Приморья // Изв. ТИНРО. 2013. С. 127–144.
Брыков Вл.А., Полякова Н.Е., Семина А.В. Сравнительный анализ изменчивости митохондриальной ДНК у четырех видов дальневосточных красноперок рода Tribolodon (Pisces, Cyprinidae) // Генетика. 2013. Т. 49. № 3. С. 355–365.
Гудков П.К., Полякова Н.Е., Семина А.В., Назаркин М.В. Сравнительный морфологический анализ крупночешуйной красноперки Tribolodon hakonensis Günther, 1877 (Cyprinidae, Cypriniformes) // Вопр. ихтиологии. 2010. Т. 50. № 6. С. 772–776.
Каплин П.А., Селиванов А.О. Изменения уровня морей России и развитие берегов: прошлое, настоящее, будущее. М.: ГЕОС. 1999. 299 с.
Маляр В.В., Брыков В.А. Генетическая изменчивость у анадромных рыб кеты Oncorhynchus keta (Walbaum, 1792) и сахалинского тайменя Parahuсho perryi (Brevoort, 1856) из северо-западной Пацифики как отражение осцилляций палеоклимата // Биол. моря. 2016. Т. 42. № 4. С. 289–299.
Маляр В.В. Сравнительная филогеография четырех видов рыб семейств Salmonidae и Cyprinidae в Японском и Охотском морях: Автореф. дис. … канд. биол. наук. Владивосток. 2017. 26 с.
Рязанова И.Н., Полякова Н.Е. Дифференциация крупночешуйной красноперки Tribolodon hakonensis (Pisces: Cyprinidae) на российской части ареала по данным кариологического анализа и ПЦР-ПДРФ-анализа митохондриальной ДНК // Генетика. 2012. Т. 48. № 2. С. 225–234.
Семина А.В., Полякова Н.Е., Брыков Вл.А. Генетический анализ выявляет криптический вид у дальневосточных красноперок рода Tribolodon // Докл. Академии наук. 2006. Т. 407. № 4. С. 571–573.
Avise J.C. Phylogeography. The History and Formation of Species. Cambridge: Harvard University Press. 2000. 447 p.
Avise J.C., Walker D.E., Johns G.C. Speciation durations and Pleistocene effects on vertebrate phylogeography // Proc. R. Soc. Lond., Ser. B: Biol. Sci. 1998. V. 265. № 1407. P. 1707–1712.
Bandelt H.-J., Forster P., Röhl A. Median-joining networks for inferring intraspecific phylogenies // Mol. Biol. Evol. 1999. V. 16. № 1. P. 37–48.
Brown W.M., George M., Wilson A.C. Rapid evolution of animal mitochondrial DNA // Proc. Natl. Acad. Sci. 1979. V. 76. № 4. P. 1967–1971.
Crête-Lafrenière A., Weir L.K., Bernatchez L. Framing the Salmonidae family phylogenetic portrait: a more complete picture from increased taxon sampling // PLoS One. 2012. V. 7. № 10. P. e46662.
Darriba D., Taboada G.L., Doallo R., Posada D. jModelTest 2: more models, new heuristics and parallel computing // Nat. Methods. 2012. V. 9. № 8. P. e772.
Drummond A.J., Suchard M.A., Xie D., Rambaut A. Baye-sian phylogenetics with BEAUti and the BEAST 1.7 // Mol. Biol. Evol. 2012. V. 29. № 8. P. 1969–1973.
Huson D.H., Bryant D. Application of phylogenetic networks in evolutionary studies // Mol. Biol. Evol. 2006. V. 23. № 2. P. 254–267.
Korotky A., Grebennikova T., Razjigaeva N. et al. Marine terraces of western Sakhalin Island // Catena. 1997. V. 30. № 1. P. 61–81.
Librado P., Rozas J. DnaSP v5: A software for comprehensive analysis of DNA polymorphism data // Bioinformatics. 2009. V. 25. № 11. P. 1451–1452.
Martin A.P., Palumbi S.R. Body size, metabolic rate, gene-ration time, and the molecular clock // Proc. Natl. Acad. Sci. U.S.A. 1993. V. 90. № 9. P. 4087–4091.
Mayr E. Animal Species and Evolution. Cambridge: Harvard University Press. 1963. 797 p.
Miller M.A., Pfeiffer W., Schwartz T. Creating the CIPRES Science Gateway for inference of large phylogenetic trees // Proc. Gateway Computing Environments Workshop (GCE). New Orleans. 2010. P. 1–8.
Sakai H., Goto A., Jeon S.R. Speciation and dispersal of Tribolodon species (Pisces, Cyprinidae) around the Sea of Japan // Zool. Sci. 2002. V. 19. № 11. P. 1291–1303.
Sasaki T., Kartavtsev Y.P., Chiba S.N. et al. Genetic divergence and phylogenetic independence of Far Eastern species in subfamily Leuciscinae (Pisces: Cyprinidae) inferred from mitochondrial DNA analyses // Genes and Genetic Systems. 2007. V. 82. № 4. P. 329–340.
Stepien C.A., Dillon A.K., Patterson A.K. Population gene-tics, phylogeography, and systematics of the thornyhead rockfish (Sebastolobus) along the deep continental slopes of the North Pacific Ocean // Canadian J. Fish. Aqua. Sci. 2000. V. 57. № 8. P. 1701–1717.
Tamura K., Peterson D., Peterson N. et al. MEGA5: Mole-cular Evolutionary Genetics Analysis using Maximum Likelihood, Evolutionary Distance, and Maximum Parsimony Methods // Mol. Biol. Evol. 2011. V. 28. № 10. P. 2731–2739.
Templeton A.R., Routman E., Phillips C.A. Separating population structure from population history: a cladistic analysis of the geographic distribution of mitochondrial DNA haplotypes in the Tiger Salamander, Ambystoma tigrinum // Genetics. 1995. V. 140. № 2. P. 767–782.
Thompson J.D., Higgins D.G., Gibson T.J. CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, positions specific gap penalties and weight matrix choice // Nucleic Acids Res. 1994. V. 22. № 22. P. 4673–4680.
Watanabe K., Sakai H., Sanada T., Nishida M. Comparative phylogeography of diadromous and freshwater daces of the genus Tribolodon (Cyprinidae) // Ichthyol. Res. 2018. V. 65. P. 383–397.
Yoon M., Sato S., Seeb J.E. et al. Mitochondrial DNA variation and genetic population structure of chum salmon Oncorhynchus keta around the Pacific Rim // J. Fish Biol. 2008. V. 73. № 5. P. 1256–1266.
Zardoya R., Doadrio I. Molecular evidence on the evolutionary and biogeographical patterns of European cyprinids // J. Mol. Evol. 1999. V. 49. № 2. P. 227–237.
Дополнительные материалы отсутствуют.



