Биотехнология, 2023, T. 39, № 5, стр. 61-69
Геномная характеристика бактерий Bacillus subtilis PY79 и NCIB 3610 как потенциальных продуцентов сурфактина
В. С. Трефилов 1, *, В. А. Лабанов 2, М. Г. Хренова 1, Т. В. Панова 1, В. А. Родин 1, В. Ю. Савицкая 1, Е. А. Кубарева 3, М. Э. Зверева 1
1 Химический факультет, Московский государственный университет имени М.В. Ломоносова
119991 Москва, Россия
2 Факультет биоинженерии и биоинформатики, Московский государственный университет
имени М.В. Ломоносова
119991 Москва, Россия
3 НИИ Физико-химической биологии имени А.Н. Белозерского, Московский
государственный университет имени М.В. Ломоносова
119992 Москва, Россия
* E-mail: trefilov.vadik@gmail.com
Поступила в редакцию 05.10.2023
После доработки 30.10.2023
Принята к публикации 02.11.2023
Аннотация
Сурфактин является одним из мощных поверхностно-активных веществ биологического происхождения и обладает широким спектром потенциальных сфер применения в промышленности и медицине. Главным фактором, ограничивающим его активное использование, является высокая стоимость производства. Из-за сложности структуры химический синтез сурфактина нерентабелен, поэтому основным подходом к его получению является микробиологический синтез. В данной работе способность синтезировать сурфактин продемонстрирована для штамма Bacillus subtilis NCIB 3610, обладающего свойствами, потерянными большинством модельных лабораторных штаммов B. subtilis. Методом нанопорового секвенирования получены последовательности геномов B. subtilis NCIB 3610 и B. subtilis PY79 и проведен их сравнительный анализ с геномом штамма B. subtilis 168, генно-модифицированные производные которого являются запатентованными продуцентами сурфактина. Показано отсутствие в штамме NCIB 3610 каких-либо мутаций, способных помешать биосинтезу сурфактина. В штамме PY79 выявлены мутации в генах, участвующих в клеточных процессах, конкурирующих с биосинтезом сурфактина. Таким образом, полученные в данной работе результаты позволяют рассматривать B. subtilis NCIB 3610 и PY79 как альтернативные штаммы-основы для генно-инженерного конструирования потенциального продуцента сурфактина.
Поверхностно-активные вещества (ПАВ) представляют собой органические амфифильные соединения, содержащие как гидрофильные, так и гидрофобные части (“голова” и “хвост” соответственно), что позволяет использовать их в качестве чистящих, диспергирующих, эмульгирующих и пенообразующих средств и обеспечивает широкое применение в повседневной жизни и промышленности [1]. К сожалению, все широко используемые на сегодняшний день ПАВ являются синтетическими соединениями, медленно разлагающимися в природе. Вследствие этого их масштабное распространение ведет к увеличению риска серьезных экологических проблем. Разработка путей снижения уровня загрязнения окружающей среды этими соединениями является актуальной задачей.
Перспективным подходом к уменьшению уровня биосферного загрязнения является замена синтетических ПАВ на ПАВ биологического происхождения, или биоПАВ. Последние имеют целый ряд преимуществ, таких как биоразлагаемость, низкая токсичность, биодоступность и щадящее действие на кожу [1, 2]. Кроме того, большинство биоПАВ обладают антимикробной активностью, что позволяет рассматривать их как основу для разработки различных противомикробных препаратов [3].
Одним из наиболее известных биоПАВ является сурфактин [4], состоящий из остатка β-гидроксижирной кислоты (С12-С18) и гептапептидного кольцевого фрагмента следующего строения: LGlu1-LLeu2-DLeu3-LVal4-LAsp5-DLeu6-LLeu7 [5]. При этом гидрофобную часть молекулы составляют углеводородная цепь β-гидроксижирной кислоты и остатки гидрофобных аминокислот (Val, Leu), а гидрофильную – отрицательно заряженные остатки Asp и Glu [4]. Хотя Аримой и др. [6] сурфактин впервые был выделен в 1968 г., он по-прежнему является предметом изучения многих научных групп вследствие широкого спектра возможного применения в различных областях, включая пищевую, косметическую, нефтяную, сельскохозяйственную промышленности, а также благодаря его потенциальной противоопухолевой, противомикробной, противовирусной и тромболитической активности [7–10]. Однако повсеместное использование этого биоПАВ ограничено из-за его высокой стоимости. Сурфактин имеет слишком сложную структуру для химического синтеза, поэтому его производство основано на микробиологическом подходе, однако количество сурфактина, секретируемого запатентованными на сегодняшний день штаммами-продуцентами, недостаточно велико [11]. Таким образом, разработка штаммов с высоким уровнем продукции сурфактина является актуальной задачей современной биотехнологии.
Сурфактин синтезируется несколькими штаммами бактерий рода Bacillus, однако впервые он был обнаружен в культуральной жидкости Bacillus subtilis IAM 1213 [6].В. subtilis – палочковидная почвенная бактерия. Она повсеместно распространена в природе, и часто используется в качестве модельного организма при изучении грамположительных бактерий, а также для биотехнологического производства различных ферментов и рекомбинантных белков, таких как амилаза, липаза и щелочная полигалактуронатлиаза (PGL). На сегодняшний день запатентовано несколько генно-модифицированных штаммов-продуцентов сурфактина, штаммы-основы которых относятся к виду B. subtilis, например, B. subtilis THY-7, SD901 и 168 [12]. Интересно, что последний, являясь одним из наиболее изученных штаммов вида B. subtilis, из-за мутации в гене sfp, кодирующем 4′-фосфопантетеинилтрансферазу, необходимую для активации нерибосомальной пептидной синтетазы сурфактина, не способен производить сурфактин (фенотип sfp–). Замена в геноме мутантной формы гена sfp на интактную приводит к появлению сурфактин-продуцирующей активности (sfp+) [13]. Внесение в B. subtilis 168 sfp+ дополнительных генных модификаций, таких как нокаут генов, участвующих в конкурирующих с биосинтезом сурфактина процессах, или замена промоторов на более сильные (например, P43), активирующая транскрипцию генов, положительно влияющих на эффективность биосинтеза сурфактина, позволили получить и запатентовать новый штамм – B. subtilis 168, BAsfp Δeps ΔsipW Δdhb Δpks Δpps ΔcodY, Pp43-swrC, pta:: Pp43-acrB, Pp43-liaIHGFSR, Pp43-fabHB, Pp43-fabD-accDABC, Pp43-fabF-fabG-fabZ-fabI, fadB::Pp43-tesA, amyE::Pp43-BKD, ldh::Pp43-lipALM, Pp43-alsS ilvD ilvC leuABCD, Pp43-pfkA, Pp43-gapA, Pp43-pgk, Pp43-pdhABCD, BAcomQXPA, BAPsrfA-srfAA. На сегодняшний день он обладает одним из самых высоких уровней продукции сурфактина среди известных штаммов-продуцентов (титр вещества был увеличен с 0.4 до 12.8 г/л) [14]. Однако такое количество не способно обеспечить существенное снижение конечной стоимости данного соединения, что ограничивает его использование и обуславливает необходимость проведения дальнейшего поиска штаммов B. subtilis, обладающих бóльшим потенциалом к секреции сурфактина, чем имеющиеся продуценты.
Объектом данного исследования являлись два штамма B. subtilis – NCIB 3610 и PY79. NCIB 3610 – природный, “дикий” штамм, родственный штамму 168, однако, в отличие от него, демонстрирующий множество фенотипов, потерянных лабораторными штаммами при их продолжительном культивировании, таких как роящаяся подвижность (“swarming motility”), образование слизистого слоя (“slime layer”), наличие экзополисахаридной капсулы, продукция антимикробных соединений и др. [15]. Благодаря этому штамм B. subtilis NCIB 3610 более защищен от воздействия бактериофагов и неблагоприятных условий среды, а также может быть использован как штамм-продуцент более широкого круга соединений. Штамм PY79 является одним из наиболее часто используемых в лабораторной практике штаммов B. subtilis, происходящим от B. subtilis 168 [16]. Показано, однако, что в геноме штамма PY79, по сравнению с геномом штамма 168, присутствует несколько областей больших делеций, точное влияние которых на метаболизм клетки остается неясным [17]. Имеющиеся геномные различия между данными штаммами могли бы способствовать более эффективному биосинтезу сурфактина штаммом PY79 по сравнению со штаммом 168, что позволило бы сконструировать генно-модифицированный штамм-продуцент на его основе с бóльшим, чем у штамма на основе B. subtilis 168, выходом сурфактина.
Целью данной работы было: 1) определение способности бактерий B. subtilis NCIB 3610 и PY79 продуцировать сурфактин, 2) проведение полногеномного секвенирования ДНК этих штаммов методом длинных прочтений, позволяющего учесть возможные геномные делеции и перестройки и 3) сравнение методами биоинформатического анализа полученных геномов с геномом штамма B. subtilis 168, не способного производить сурфактин, но послужившего основой для создания запатентованного генно-инженерного штамма-продуцента сурфактина.
УСЛОВИЯ ЭКСПЕРИМЕНТА
Культивирование штаммов в жидких средах
Используемые штаммы B. subtilis были любезно предоставлены научной группой проф. Роланда К. Хартманна (Philipps University of Marburg, Марбург, Германия). Их культивировали в среде LB (5 г/л NaCl, 10 г/л пептона, 5 г/л дрожжевого экстракта, рН 7.0) при 37°C с непрерывным перемешиванием при 220 об./мин. Все питательные среды были приготовлены с использованием дистиллированной воды и стерилизовались автоклавированием при 121°С и 1 атм. Твердая агаризованная среда LB дополнительно содержала 1.5% (v/v) агара. Клеточные суспензии были получены инокулированием 3 мл питательной среды LB единичными колониями, выросшими на чашках Петри с твердой питательной средой (14 ч, 37°С), и последующей инкубацией в клеточном шейкере-инкубаторе (14 ч, 37°С, 220 об./мин, конечная оптическая плотность суспензии OD600 = 7.0 – 7.3).
Гемолитический тест на твердой питательной среде
Для определения способности штаммов В. subtilis PY79 и NCIB 3610 синтезировать сурфактин был использован гемолитический тест на твердой питательной среде, основанный на способности этого соединения разрушать эритроциты крови [18, 19]. В чашках Петри на агаризованную среду LB, содержащую 5% (v/v) стерильной дефибринированной овечьей крови (“ЭКОлаб”, Россия), наносили по 10 мкл клеточной суспензии изучаемых штаммов, высушивали на воздухе (10–15 мин), затем инкубировали при 37°С в течение 48 ч. В ходе эксперимента вокруг колоний сурфактин-продуцирующих бактерий в результате гемолиза образуются обесцвеченные зоны, по диаметру которых оценивали его интенсивность. Для каждой клеточной линии эксперимент проводили в трех биологических репликах на отдельных чашках Петри.
Выделение геномной ДНК, нанопоровое секвенирование и сборка геномов бактерий B. subtilis PY79 и NCIB 3610
Выделение геномной ДНК
Геномную ДНК из бактерий B. subtilis PY79 и NCIB 3610 для полногеномного секвенирования выделяли с помощью набора Monarch Genomic DNA Purification Kit T3010 (New England Biolabs, Великобритания) в полном соответствии с протоколом производителя для грам-положительных бактерий. Оценку количества и контроль качества полученной гДНК проводили с помощью анализа длин фрагментов методом электрофореза в 1%-ном агарозном геле (“ДНК-технология”, Россия), спектрофотометра для микрообъемов NanoDrop (Thermo Fisher Scientific, США) и флуориметра Qubit 4.0 (Thermo Fisher Scientific).
Нанопоровое секвенирование и сборка геномов
Нанопоровое секвенирование образцов проводили по технологии секвенирования геномной ДНК Oxford Nanopore Technologies (ONT, Великобритания) в полном соответствии с протоколом Native barcoding genomic DNA (SQK-LSK109) на приборе MinION (ONT) с использованием ячейки R9.4.1 (ONT). На первом этапе проводили репарацию ДНК и подготовку концов к лигированию баркодов и адаптеров с помощью набора NEBNext Companion Module for Oxford Nanopore Technologies Ligation Sequencing E7180S (New England Biolabs, США) с последующей очисткой на магнитных частицах Agencourt AMPure XP beads (Beckman Coulter, США). Далее к образцам ДНК путем лигирования добавляли уникальные баркоды с использованием набора Native barcoding Expansion 1–12 (EXP-NBD104, ONT) и лигазы Blunt/TA Ligase Master Mix (New England Biolabs), а затем проводили очистку на магнитных частицах Agencourt AMPure XP beads (Beckman Coulter). Полученные образцы с присоединенными баркодами объединяли в эквимолярных количествах в библиотеку и проводили лигирование адаптеров с использованием лигазы Quick T4 DNA Ligase (New England Biolabs) и реагентов из набора SQK-LSK 109 (ONT) в соответствии с протоколом. Образец очищали на магнитных частицах Agencourt AMPure XP beads (Beckman Coulter), используя буфер для удерживания длинных фрагментов ДНК. Далее ячейку секвенатора готовили к загрузке образца с использованием реактивов из набора SQK-LSK 109 (ONT), как описано в протоколе, и затем наносили на ячейку подготовленную библиотеку. Ячейку помещали в прибор и начинали процесс секвенирования. По результатам запуска для образца PY79 получено 195 Мб из 38 425 “ридов” и для образца NCIB 3610 получено 634.8 Мб из 86 328 “ридов”.
Первичными данными нанопорового секвенирования являются сигналы тока, которые связаны с размером гетероциклического основания конкретного нуклеотида, проходящего через нанопору. Сигналы, записанные в формате FAST5, на первом этапе переводятся в формат FASTQ, несущий явную информацию о конкретном нуклеотиде в каждом положении прочитанных нуклеотидных звеньев и качестве его определения. Для преобразования первичных данных использовали программу Guppy basecaller (ONT). Дебаркодирование образцов проводили с помощью этой же программы в ходе первичной обработки данных. Все данные с качеством Q < 7.5, полученном из оценки программой Guppy basecaller, не участвовали в дальнейшем анализе. Для сборки генома без референса использовали программу Flye [20] с последующей корректировкой ошибок программой Medaka [21].
Биоинформатический анализ геномных последовательностей и идентификация мутаций, потенциально имеющих влияние на процесс биосинтеза сурфактина
Собранные геномы B. subtilis штаммов NCIB 3610 и PY79 сравнивали вместе с депонированным в базе данных NCBI (https://www.ncbi.nlm.nih.gov/) референсным геномом B. subtilis 168 (NC_000964.3) с помощью алгоритма MAFFT [22] в программе Jalview (https://www.jalview.org/). Этот алгоритм обладает достаточной скоростью множественного “выравнивания” полных геномов и одновременно достаточной точностью для “выравнивания” геномов близкородственных бактерий. Далее на языке программирования C++ в интегрированной среде разработки (IDE) Visual Studio 2022 нами была написана программа, с помощью которой искали различия между “выравненными” геномами. Из конечного набора обнаруженных различий были исключены делеции и вставки одного нуклеотида, если они находились внутри или на концах тандемных повторов того же нуклеотида, делеция или вставка которого была обнаружена. Данное решение обуславливается особенностью нанопорового секвенирования, имеющего большую вероятность ошибки при прочтении подобных тандемных повторов [23].
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Анализ способности штаммов B. subtilis PY79 и NCIB 3610 продуцировать сурфактин
Чтобы выяснить, способны ли штаммы B. subtilis NCIB 3610 и PY79 синтезировать сурфактин, мы использовали гемолитический тест на твердых питательных средах. Согласно полученным результатам, В. subtilis NCIB 3610 такой способностью обладает. Наблюдаемые зоны гемолиза являются четкими и ярко выраженными, что свидетельствует об относительно высокой эффективности процесса биосинтеза сурфактина в данном штамме (рис. 1а). В. subtilis PY79 является производным штамма 168, демонстрирующим фенотип sfp–. Исходя из этого, мы ожидали отсутствие у него гемолитической активности. Выполненные эксперименты подтвердили неспособность данного штамма продуцировать сурфактин (рис. 1b). Отметим, что первые признаки гемолиза для клеток штамма NCIB 3610 появились после 24 ч их выдерживания при +37°С, в то время как для штамма PY79 зоны гемолиза не проявились даже через 4 дня инкубации.
Рис. 1.
Результаты гемолитического теста по определению способности клеток продуцировать сурфактин. а – B. subtilis NCIB 3610, b – B. subtilis PY79. Темные пятна – результат разрастания клеточного пятна в течение 4 дней культивации. Четкие, хорошо оформленные участки гемолиза свидетельствует о достаточно высокой эффективности биосинтеза сурфактина. Fig. 1. The results of a hemolytic test for estimation the cell ability to produce surfactin. a – B. subtilis NCIB 3610, b – B. subtilis PY79. Dark spots are the result of the cell colonies growth for 4 days of incubation. Presence of clear, well-formed hemolysis areas indicates a quite high efficiency of surfactin biosynthesis.

Сравнительный биоинформатический анализ геномов B. subtilis NCIB 3610 и PY79
Для оценки потенциала использования бактерий B. subtilis NCIB 3610 и PY79 в микробиологическом синтезе сурфактина был проведен биоинформатический анализ. Геномы штаммов сравнивали с аннотированным в базе данных NCBI геномом B. subtilis 168 для определения различий в последовательностях генов, каким-либо образом связанных с биосинтезом этого соединения в клетках B. subtilis. Согласно данным литературы, выделяют три клеточные системы, функционирование которых негативно влияет на эффективность продукции сурфактина за счет конкуренции этих процессов за ресурсы клетки, аминокислоты и жирные кислоты:
• образование биопленок,
• синтез поликетидов,
• синтез соединений, осуществляемый нерибосомальными пептидными синтетазами.
Было показано, что нокаут генов, участвующих в данных процессах, приводит к повышению уровня продукции сурфактина [14]. Напротив, стимуляция систем биосинтеза аминокислот и жирных кислот, а также увеличение уровня транскрипции генов, отвечающих за производство и секрецию сурфактина, положительно влияет на клеточный уровень его продукции [13]. Исходя из этого, был составлен перечень известных на данный момент генов, влияющих на биосинтез сурфактина в клетках B. subtilis (Таблица S1 , дополнительный материал), согласно которому мы проводили сравнительный анализ штаммов PY79 и NCIB 3610 со штаммом 168.
Мы обнаружили, что B. subtilis NCIB 3610 содержит интактную последовательность гена sfp. Мутацией, приводящей к потери белком Sfp ферментативной активности, является вставка Т в позиции 467, характерная для B. subtilis 168 и приводящая к сдвигу открытой рамки считывания (ОРС) [24]. В отличие от B. subtilis 168, в штамме NCIB 3610 указанный нуклеотид отсутствует (табл. 1). Это соответствует нашим экспериментальным данным и обнаруженной сурфактин-синтезирующей активности NCIB 3610 (рис. 1а).
Таблица 1.
Различия в генах, ассоциированных с синтезом сурфактина, штамма B. subtilis NCIB 3610 относительно референсного генома B. subtilis 168 и их последствия Table 1. Differences in gene sequences, associated with the synthesis of surfactin for B. subtilis NCIB 3610 strain relative to the genome of B. subtilis 168 used as reference and their consequences
| Ген | Мутация | Последствия |
|---|---|---|
| codY | 1690644 (A → G) | Аминокислотная замена 176I → 176V |
| perR | 944916 (– → TG) 944917 (A → T) 944918 (T → C) 944919 (C → G) 944921 (T → A) 944922 (T → A) 944923 (A → T) |
Сдвиг ОРС изменение С-концевой последовательности белка 144NH145 → 144MSK146 |
| sfp | 407668 (T → –) | Генотип sfp+ |
Следующим геном, в котором обнаружены мутации, был ген perR, кодирующий независимый активатор транскрипции оперона srfA (субъединицы сурфактин-синтетазы) [25]. В частности, относительно штамма 168 в B. subtilis NCIB 3610 в гене perR были обнаружены вставка из 2 нуклеотидов и 6 однонуклеотидных замен, приводящие, однако, к замене только двух С-концевых аминокислотных остатков. Согласно данным базы PDB (https://www.rcsb.org/, PDB ID 2FE3), концевые аминокислотные остатки (а.о.) находятся в области сильной разупорядоченности пространственной структуры белка и не участвуют в связывании ни ДНК, ни ионов металлов, необходимых для проявления белком ДНК-связывающей активности. Поэтому мы полагаем, что данные мутации не оказывают влияния на активность белка PerR. Еще одна мутация была обнаружена в гене codY, который кодирует глобальный ГТФ-зависимый ингибитор транскрипции генов (в том числе и srfA), необходимый клетке в стационарной фазе роста [26]. Однако в данном случае происходит замена изолейцина на валин, что ввиду большой схожести этих аминокислот, скорее всего не приведет к серьезным изменениям активности данного белка [27]. Таким образом, по сравнению с B. subtilis 168 геном штамма NCIB 3610 не только кодирует интактный ген sfp, но и не содержит никаких серьезных нарушений с точки зрения биосинтеза сурфактина, что, вместе с бóльшей устойчивостью к неблагоприятным условиям и патогенам, характеризует штамм B. subtilis NCIB 3610 как потенциально полезный для конструирования на его основе штамма-продуцента сурфактина.
В случае штамма PY79, различий в гене sfp между ним и штаммом 168 обнаружено не было, что коррелирует с неспособностью штамма PY79 синтезировать сурфактин. Однако между геномами данных бактерий были обнаружены другие различия, также позволяющие рассматривать штамм PY79 как подходящий на роль штамма-основы генно-модифицированного суперпродуцента сурфактина (табл. 2).
Таблица 2.
Различия в генах, ассоциированных с синтезом сурфактина, штамма B. subtilis PY79 относительно референсного генома B. subtilis 168 и их последствия Table 2. Differences in gene sequences, associated with the synthesis of surfactin for B. subtilis PY79 strain relative to the genome of B. subtilis 168 used as reference and their consequences
| Ген | Мутация | Последствия |
|---|---|---|
| comQ | 3256130 (C → –) | сдвиг ОРС изменение С-концевой последовательности белка 260EIYKQKCISAIEQLNLDKEK KELIKECLLSYTKGDTRCKT299 → 260KYINRNVFLL269 |
| dhbF | 3284686 (G → –) | сдвиг ОРС уменьшение длины белка 2378 а.о. → 1000 а.о. |
| epsC | 3527377 (C → T) | аминокислотная замена 276A → 276V |
| epsH | 3521611 (G → –) | сдвиг ОРС уменьшение длины белка 344 а.о. → 202 а.о. |
| epsJ | Делеция (478944-480864) |
нокаут, генотип epsJ– |
| epsK | нокаут, генотип epsK– | |
| pksJ | 1806775 (– → A) | сдвиг ОРС уменьшение длины белка 5043 а.о. → 4681 а.о. |
| pksN | 1835095 (C → –) | нокаут, генотип pksN– |
| ppsC | Делеция (1969507-1978426) |
нокаут, генотип ppsC– |
| ppsD | нокаут, генотип ppsD– |
В частности, в штамме PY79 присутствуют мутации, способные заметно нарушить функционирование упоминавшихся выше систем биосинтеза:
• различных поликетидов,
•пептидов фенгицина и бациллибактина (синтезируются нерибосомальным способом),
• а также экзополимерных соединений, необходимых для формирования биопленки.
За эти процессы в клетках B. subtilis отвечают опероны pks, pps, dhb и eps соответственно [14, 24, 28, 29]. Все они негативно влияют на процесс биосинтеза сурфактина за счет конкуренции с ним за аминокислоты, жирные кислоты и другие клеточные ресурсы. Таким образом, можно предположить, что в штамме B. subtilis PY79 (при восстановлении в нем фенотипа sfp+) за счет меньшей конкуренции биосинтез сурфактина будет протекать эффективнее, чем в штамме 168, что положительно отразится на количестве конечного продукта. Единственным различием, способным негативно повлиять на синтез сурфактина в штамме PY79 по сравнению с 168, является делеция одного нуклеотида в гене comQ. Данный ген кодирует белок ComQ, необходимый для производства сигнального гексапептида ComX, стимулирующего положительную регуляцию транскрипции оперона srfA белком ComA [30]. Обнаруженная мутация приводит к изменению С-концевой последовательности белка, а именно, замене последних 9 а.о. (с 260 по 269) и укорочению белка на 30 а.о. (с 270 по 299). Однако, судя по данным литературы, перечисленные аминокислотные остатки не входят в каталитический центр ComQ, поэтому данная мутация вряд ли приведет к значительной потере активности фермента [31].
Таким образом, нами проведена геномная характеристика двух штаммов B. subtilis – PY79 и NCIB 3610 – как альтернативных базовых штаммов для конструирования промышленного суперпродуцента сурфактина. На рис. 2 схематично представлены функции генов, в которых были обнаружены отличия по сравнению со штаммом 168.
Рис. 2.
Роль генов, в последовательностях которых обнаружены мутации в бактериях B. subtilis PY79 и NCIB 3610, в процессе биосинтеза сурфактина. Гены/опероны, в которых были найдены мутации при сравнении геномов B. subtilis PY79/NCIB 3610 с референсным геномом B. subtilis 168, выделены красной рамкой. Процессы образования биопленки, синтеза фенгицина, бациллибактина и поликетидов (опероны eps, pps, dhb и pks соответственно) конкурируют с сурфактин-синтетазой, кодируемой srfA опероном, за аминокислоты и жирные кислоты в бактериальной клетке. Гены perR и codY кодируют активатор и ингибитор транскрипции srfA, белки Sfp и ComQ участвуют в активации сурфактин-синтетазы и биосинтезе сигнального пептида ComX, необходимого для ComA-зависимой регуляции srfA, соответственно. Fig. 2. The role of genes with mutations which have been detected in B. subtilis PY79 and NCIB 3610 compared with B. subtilis 168 in surfactin biosynthesis process. The genes/operons in which mutations have been found are pointed out by red frames. The processes of biofilm formation, fengicine, bacillibactine and polyketide synthesis (operons eps, pps, dhb and pks respectively) compete with the srfA-encoded surfactin synthase for amino acids and fatty acids in the bacterial cells. The perR and codY genes encode the srfA transcription activator and inhibitor, the Sfp and ComQ proteins are involved in the activation of the surfactin synthase and biosynthesis of the ComX signaling peptide required for ComA-dependent srfA regulation respectively.
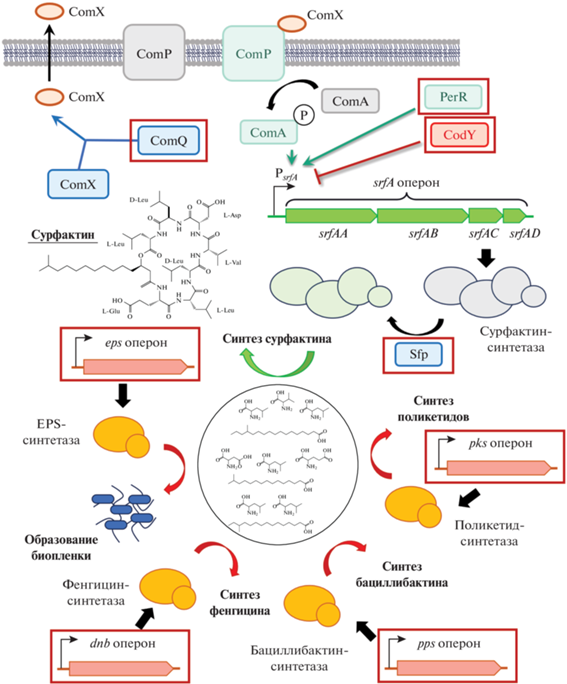
Согласно полученным результатам, оба штамма, PY79 и NCIB 3610, обладают высоким потенциалом биотехнологического применения и могут служить основой для получения эффективного продуцента сурфактина. Для этого необходимо дальнейшее детальное изучение процесса биосинтеза этого соединения и его секреции в данных штаммах.
Список литературы
Singh A., Van Hamme J.D., Ward O.P. Surfactants in microbiology and biotechnology: Part 2. Application aspects. Biotechnology Advances, 2007, 25, 99–121.https://doi.org/10.1016/j.biotechadv.2006.10.004
Pardhi D.S., Panchal R.R., Raval V.H., Joshi R.G., Poczai P., Almalki W.H., Rajput K.N. Microbial surfactants: A journey from fundamentals to recent advances. Front. Microbiol., 2022, 13, 982603. https://doi.org/10.3389/fmicb.2022.982603
Stein T. Bacillus subtilis antibiotics: structures, syntheses, and specific functions. Mol. Microbiol., 2005, 56, 845–857. https://doi.org/10.1111/j.1365-2958.2005.04587.x
Кисиль О.В., Трефилов В.С., Садыкова В.С., Зверева М.Э., Кубарева Е.А. Сурфактин: биологическая активность и возможность применения в сельском хозяйстве. Прикладная биохимия и микробиология, 2023, 59, 1, 3–16.https://doi.org/10.31857/S0555109923010026
Théatre A., Cano-Prieto C., Bartolini M., Laurin Y., Deleu M., Niehren J., Jacques P. The surfactin-like lipopeptides from Bacillus spp.: Natural biodiversity and synthetic biology for a broader application range. Front. Bioeng. Biotechnol., 2021, 9, 623701. https://doi.org/10.3389/fbioe.2021.623701
Arima K., Kakinuma A., Tamura G. Surfactin, a crystalline peptide lipid surfactant produced by Bacillus subtilis: Isolation, characterization and its inhibition of fibrin clot formation. Biochem. Biophys. Res. Commun., 1968, 31, 488‒494. https://doi.org/10.1016/0006-291X(68)90503-2
Seydlová G., Svobodová J. Review of surfactin chemical properties and the potential biomedical applications. Cent. Eur. J. Med., 2008, 3, 123‒133.https://doi.org/10.2478/s11536-008-0002-5
Bais H.P., Fall R., Vivanco J.M. Biocontrol of Bacillus subtilis against infection of Arabidopsis roots by Pseudomonas syringae is facilitated by biofilm formation and surfactin production. Plant Physiol., 2004, 134, 307‒319. https://doi.org/10.1104/pp.103.028712
Yang Z., Zu Y., Zhu J., Jin M., Cui T., Long X. Application of biosurfactant surfactin as a pH-switchable biodemulsifier for efficient oil recovery from waste crude oil. Chemosphere, 2020, 240, 124946.https://doi.org/10.1016/j.chemosphere.2019.124946
Liu J.F., Mbadinga S.M., Yang S.Z., Gu J.D., Mu B.Z. Chemical structure, property and potential applications of biosurfactants produced by Bacillus subtilis in petroleum recovery and spill mitigation. Int. J. Mol. Sci., 2015, 16, 4814‒4837. https://doi.org/10.3390/ijms16034814
Yeh M.S., Wei Y.H., Chang J.S. Bioreactor design for enhanced carrier-assisted surfactin production with Bacillus subtilis. Process Biochem., 2006, 41, 1799‒1805. https://doi.org/10.1016/j.procbio.2006.03.027
Qun Wu, Yan Xu, Method for Promoting Bacillus subtilis to synthesize surfactin based on Multi-gene Synergy, US20200131229A1, USA, 2020 (пaтeнт).
Xia L., Wen J. Available strategies for improving the biosynthesis of surfactin: a review. Crit. Rev. Biotechnol., 2022, 1‒18. https://doi.org/10.1080/07388551.2022.2095252
Wu Q., Zhi Y., Xu Y. Systematically engineering the biosynthesis of a green biosurfactant surfactin by Bacillus subtilis 168. Metab. Eng., 2019, 52, 87‒97. https://doi.org/10.1016/j.ymben.2018.11.004
Nye T.M., Schroeder J.W., Kearns D.B., Simmons L.A. Complete genome sequence of undomesticated Bacillus subtilis strain NCIB 3610. Genome Announce, 2017, 5, 10‒1128. https://doi.org/10.1128/genomeA.00364-17
Yazgan A., Özcengiz G., Marahiel M.A. Tn10 insertional mutations of Bacillus subtilis that block the biosynthesis of bacilysin. Biochim. Biophys. Acta, Gene Struct. Expr., 2001, 1518, 87‒94. https://doi.org/10.1016/s0167-4781(01)00182-8
Zeigler D.R., Prágai Z., Rodriguez S., Chevreux B., Muffler A., Albert T., Perkins J.B. The origins of 168, W23, and other Bacillus subtilis legacy strains. J. Bacteriol., 2008, 190, 6983‒6995. https://doi.org/10.1128/JB.00722-08
Youssef N.H., Duncan K.E., Nagle D.P., Savage K.N., Knapp R.M., McInerney M.J. Comparison of methods to detect biosurfactant production by diverse microorganisms. J. Microbiol. Methods, 2004, 56, 339‒347. https://doi.org/10.1016/j.mimet.2003.11.001
Dufour S., Deleu M., Nott K., Wathelet B., Thonart P., Paquot M. Hemolytic activity of new linear surfactin analogs in relation to their physico-chemical properties. Biochim. Biophys. Acta, Gen. Subj., 2005, 1726, 87‒95. https://doi.org/10.1016/j.bbagen.2005.06.015
Kolmogorov M., Yuan J., Lin Y., Pevzner P.A. Assembly of long, error-prone reads using repeat graphs. Nat. Biotechnol., 2019, 37, 540‒546.https://doi.org/10.1038/s41587-019-0072-8
Huang Y.T., Liu P.Y., Shih P.W. Homopolish: a method for the removal of systematic errors in nanopore sequencing by homologous polishing. Genome Biol., 2021, 22, 1‒17. https://doi.org/10.1186/s13059-021-02282-6
Katoh K., Rozewicki J., Yamada K.D. MAFFT online service: multiple sequence alignment, interactive sequence choice and visualization. Briefings in Bioinf., 2019, 20, 1160‒1166. https://doi.org/10.1093/bib/bbx108
Lu H., Giordano F., Ning Z. Oxford Nanopore MinION sequencing and genome assembly. Genomics, Proteomics Bioinf., 2016, 14, 5, 265‒279. https://doi.org/10.1016/j.gpb.2016.05.004
Nakano M.M., Corbell N., Besson J., Zuber P. Isolation and characterization of sfp: a gene that functions in the production of the lipopeptide biosurfactant, surfactin, in Bacillus subtilis, Mol. Gen. Genet., 1992, 232, 313‒321. https://doi.org/10.1007/BF00280011
Hayashi K., Ohsawa T., Kobayashi K., Ogasawara N., Ogura M. The H2O2 stress-responsive regulator PerR positively regulates srfA expression in Bacillus subtilis. J. Bacteriol., 2005, 187, 19, 6659‒6667. https://doi.org/10.1128/JB.187.19.6659-6667.2005
Molle V., Nakaura Y., Shivers R.P., Yamaguchi H., Losick R., Fujita Y., Sonenshein A.L. Additional targets of the Bacillus subtilis global regulator CodY identified by chromatin immunoprecipitation and genome-wide transcript analysis. J. Bacteriol., 2003, 185, 6, 1911‒1922. https://doi.org/10.1128/JB.185.6.1911-1922.2003
Takano K., Ogasahara K., Kaneda H., Yamagata Y., Fujii S., Kanaya E., Yutani K. Contribution of hydrophobic residues to the stability of human lysozyme: calorimetric studies and X-ray structural analysis of the five isoleucine to valine mutants. J. Mol. Biol., 1995, 254, 1, 62‒76. https://doi.org/10.1006/jmbi.1995.0599
Tsuge K., Ano T., Hirai M., Nakamura Y., Shoda M. The genes degQ, pps, and lpa-8 (sfp) are responsible for conversion of Bacillus subtilis 168 to plipastatin production. Antimicrob. Agents Chemother., 1999. 43, 9, 2183‒2192. https://doi.org/10.1128/AAC.43.9.2183
Kearns D.B., Chu F., Branda S.S., Kolter R., Losick R. A master regulator for biofilm formation by Bacillus subtilis. Mol. Microbiol., 2005, 55, 3, 739‒749. https://doi.org/10.1111/j.1365-2958.2004.04440.x
Schneider K.B., Palmer T.M., Grossman A.D. Characterization of comQ and comX, two genes required for production of ComX pheromone in Bacillus subtilis. J. Bacteriol., 2002, 184, 2, doi: 410‒419. https://doi.org/10.1128/JB.184.2.410-419.2002
Hirooka K., Shioda S., Okada M. Identification of critical residues for the catalytic activity of ComQ, a Bacillus prenylation enzyme for quorum sensing, by using a simple bioassay system. Biosci. Biotechnol. Biochem., 2020, 84, 2. 347‒357. https://doi.org/10.1080/09168451.2019.1685371
Дополнительные материалы отсутствуют.


