Биология внутренних вод, 2020, № 3, стр. 244-252
Углеводородокисляющие бактерии из городского озера: идентификация и филогенетический анализ
Т. Р. Кравцова a, *, В. В. Ильинский a, И. В. Лазебная b, О. Е. Лазебный c, А. Ю. Акулова a, И. В. Мошарова a, d, О. А. Кокшарова e
a Московский государственный университет им. М.В. Ломоносова
Москва, Россия
b Институт общей генетики им. Н.И. Вавилова Российской академии наук
Москва, Россия
c Институт биологии развития им. Н.К. Кольцова Российской академии наук
Москва, Россия
d Институт океанологии им. П.П. Ширшова Российской академии наук
Москва, Россия
e Институт физико-химической биологии им. А.Н. Белозерского, МГУ
Москва, Россия
* E-mail: skypoy-92@mail.ru
Поступила в редакцию 27.05.2019
После доработки 30.08.2019
Принята к публикации 23.09.2019
Аннотация
Впервые из бактериопланктона оз. Белое (г. Москва) выделены культивируемые углеводородокисляющие бактерии – штаммы 2012B и 2012С. Жирно-кислотный профиль штаммов 2012В и 2012C представлен в диапазоне C14:0–C19:0. Спектр наиболее представленных жирных кислот штамма 2012В включает в себя C15:0 (54%), C16:0 (17%), C17:0 (10%) и 10-метил С18:0 (3.5%), липидный спектр штамма 2012C представлен жирными кислотами C15:0 (45%), C16:0 (32%) и C17:0 (9%). По биохимическим и морфологическим характеристикам штамм 2012B наиболее сходен с представителями рода Rhodococcus spp. (Actinobacteria, Nocardiaceae). Проведенный филогенетический анализ с использованием нуклеотидных последовательностей генов 16S рРНК (KP779 654.1) и alkВ (KR422 620.1) показал, что штамм 2012B принадлежит к виду Rhodococcus qingshengii. Нуклеотидная последовательность гена 16S рРНК штамма 2012С (MG966 152) имеет наибольшее сходство (99.57%) с последовательностями Pseudomonas psychrotolerans и P. oryzihabitans.
ВВЕДЕНИЕ
Городские озера – “экологические барометры здоровья города” (Ravikumar et al., 2013), а бактериопланктонные сообщества – их неотъемлемые компоненты. Загрязняющие вещества, среди которых полициклические ароматические углеводороды, поступающие из выхлопных газов автомобилей, гидрофобные хлорорганические соединения, как хлордан, дильдрин, полихлорированные бифенилы (Long et al., 2003), легко попадают в городские озера по водным стокам и накапливаются в виде донных отложений.
Наиболее дешевый способ биоремедиации – использование УОБ, активно участвующих в естественной биодеградации нефтяных и других углеводородных загрязнений (Brooijmans et al., 2009). В водной среде УОБ утилизируют 0.003–100% углеводородов (Das, Chandran, 2011), что приводит к ускорению очистки водных систем и позволяет обеспечить сбалансированность биотопа. Показано, что в лабораторных условиях микробному сообществу, состоящему из двух изолятов Pseudomonas aeruginosa (Schroeter, 1872; Migula, 1900) и Rodococcus sp. (EU259 892), удалось разложить до 90% углеводородов за 6 нед. в жидкой культуре (Cameotra, Singh, 2008).
Таксономический состав УОБ, обитающих в городских озерах России, в литературе фактически не описан, а видовой таксономический состав культивируемых УОБ, выделенных из московских озер, ранее не исследовали. Более полувека назад сообщалось лишь о том, что в водах оз. Белое обнаружены бактерии, способные окислять метан, гексан и нафталин (Кузнецов, 1952).
Известно, что на долю культивируемых УОБ приходится <1% общего числа бактерий в естественном бактериоценозе. В связи с этим идентификация новых культивируемых природных штаммов УОБ, безусловно, влияющих на состояние всей биоты городского оз. Белое, представляет особый интерес.
Цель работы – охарактеризовать выделенные впервые из городского оз. Белое культивируемые штаммы УОБ, провести филогенетический анализ и определить их таксономическое положение.
МАТЕРИАЛ И МЕТОДЫ ИССЛЕДОВАНИЯ
Характеристика вод оз. Белое. Озеро природного происхождения – наиболее глубокое из трех озер природно-исторического парка. Его глубина 4.2–13.5 м, площадь 0.2 км2 (Россолимо, 1925). В 1930-х гг. озеро уже имело статус эвтрофного, а в его донных осадках отмечены процессы анаэробной деструкции органического вещества (Кузнецов, 1970). В 1986 г. оз. Белое вошло в черту г. Москвы.
За период наблюдений температура поверхностного слоя воды в озере варьировала от 0.1–0.5 до 27–30°С. Значения рН колебались от нейтральных 7.22 до слабощелочных 7.99, пики подщелачивания воды ‒ от 9.11 до 8.62. Концентрация ионов аммония изменялась в пределах 0.05–1.73 мг/л, нитратов – 0.22–4.73 мг/л, концентрация нитритов – 0.003–0.12 мг/л, фосфатов – 0.02–0.33 мг/л. Содержание легкоразлагаемого органического вещества, оцененное по параметрам величин перманганатной окисляемости, достигало 4.41–7.13 мг О2/л.
Таким образом, по содержанию биогенных элементов степень сапробности оз. Белое в разные сезоны года варьировала от ксеносапробной до полисапробной. Постоянное присутствие в воде биогенных элементов выводит их из числа факторов, лимитирующих развитие гетеротрофных бактерий (Акулова и др., 2014).
Отбор проб. Пробы воды из оз. Белое для микробиологического анализа отбирали с глубины ~0.5 м в 5 м от берега, где глубина была ≥1.5 м.
Выделение чистых культур УОБ. Чистые культуры выращивали на 2%-ной агаризованной синтетической среде ММС (Mills et al., 1978) с добавлением летнего дизельного топлива, в состав которого входят 20–30% нафтеновых, 20–30% ароматических и до ≤~40% парафиновых углеводородов. Культуры бактерий хранили на среде ММС в холодильнике при температуре 4–6°С.
Морфологические признаки. Морфологию клеток бактериальных штаммов изучали с помощью светового микроскопа Nikon Eclipse E 200F 200 и сканирующего электронного микроскопа Camscan S-2 Cambridge (Великобритания). Образцы для микроскопии отбирали через 1, 3, 7, 16 и 30 сут роста культур на минеральной среде ММС и среде Мюллера (Hansen, Møller, 1975).
Ферментативную активность определяли по методике Сэмбрука (Sambrook et al., 1989). Биодеградацию аминокислот и сахаров оценивали по росту штаммов на среде М9 с углеводородами (Sambrook et al., 1989). Жирные кислоты анализировали по методике MIDI (Microbial Identification System) (Sasser, 2001).
Штаммы УОБ идентифицировали по определителю Берги (Bergey et al., 1989), а также с применением молекулярных методов.
Экстракция геномной ДНК. Выделение геномной ДНК из бактерий проводили по методике Кокшаровой и др. (Koksharova et al., 1998), aмплификацию нуклеотидной последовательности гена 16S рРНК – с помощью ПЦР. Для штамма 2012B использовали праймеры RodF (5′-CAGCAGCTCAACTGCTGGCT-3′) и RodR (5′-CATGCTCCGCCGCTTGTG-3′) (Fredriksson et al., 2013), для штамма 2012C – праймеры Ps-F (5′-GGTCTGAGAGGATGATCAGT-3′) и Ps-R (5′-TTAGCTCCACCTCGCGGC-3′) (Widmer et al., 1998). Температурно-временной режим для ПЦР: 94°C – 10 мин; 25 циклов (94°C – 45 c, 54°C – 45 c, 68°C – 2 мин); хранение при 4 °C. Продукты ПЦР визуализировали в 1.5%-ном агарозном геле. Амплификацию последовательности гена alkВ проводили с использованием праймеров alk-FI (5′-CATAATAAAGGGCATCACCG-3′) и alk-RI (5′-GATTTCATTCT-CGAAACTCC-3′) (Kohno et al., 2002).
Клонирование и секвенирование ПЦР продуктов. Фрагменты ДНК клонировали с помощью набора CloneJet PCRтм Cloning Kit # K1231 (Fermentas, EC). Секвенирование полученных продуктов ПЦР выполняли на автоматическом секвенаторе Applied Biosystems 3730 DNA Analyzer с использованием реагентов ABI PRISM ® BigDye™ Terminator v. 3.1 в Центре коллективного пользования “Геном” (Институт молекулярной биологии им. В.А. Энгельгардта РАН).
Анализ нуклеотидных последовательностей. Филогенетическую реконструкцию последовательностей проводили методом ближайшего соседа (Neighbor-Joining) (Saitou, Nei, 1987) с помощью программы Blast Tree View (Fast minimum evolution ‒ tree method). Для первичного сравнительного анализа полученных последовательностей использовали GenBank (NCBI) и BLAST (www.blast.ncbi.nlm.nih.gov) (Altschul et al., 1997). Последовательности проверяли на наличие химер с помощью сервиса Bellerophon (Huber et al., 2004). Индексы бутстрепа (1000 итераций) показаны рядом с ветвями (Felsenstein, 1985). Длины ветвей эквивалентны эволюционным расстояниям, используемым для построения филогенетического дерева. Для построения филогенетических деревьев применяли программу MEGA X (Kumar et al., 2018).
РЕЗУЛЬТАТЫ ИССЛЕДОВАНИЯ И ИХ ОБСУЖДЕНИЕ
Из выделенных культивируемых штаммов для определения таксономического положения выбрано два штамма УОБ, способных к использованию жидких углеводородов в качестве единственного источника углерода и энергии.
Характеристика штамма 2012В. Колонии клеток розового цвета, выпуклые, матовые, с ровным краем, сухие. Клетки грамположительные, аэробные, неподвижные, неспорообразующие (рис. 1).
На среде ММС с дизельным топливом размер клетки 1–2 мкм, на среде Мюллера – 3–5 мкм. Клетки каталазоположительны и кислотоустойчивы. Жирно-кислотный профиль штамма представлен жирными кислотами с длиной углеродной цепи C14:0–C19:0. Спектр наиболее представленных жирных кислот включал в себя C15:0 (54%), C16:0 (17%), C17:0 (10%) и 10-метил С18:0 (3.5%).
Штамм 2012В способен утилизировать сахара и ряд аминокислот: L-инозин, L-арабинозу, L-аспарагин, D-фруктозу, мальтозу, D-маннитол и D-сорбитол. Он не утилизирует лактозу, D-ксилозу, а из фунгицидов – беномил и карбендазим (табл. 1).
Таблица 1.
Сравнительная фенотипическая характеристика штамма Rhodococcus sp. 2012B с референтными штаммами рода Rhodococcus
| Показатели | R. sp. 2012B |
R. qingshengii sp. djl-6T (Xu et al., 2007) |
R. jialingiae sp. djl-6-2T (Wang et al., 2010) |
R. erythropolis sp. DSM 43 066T (Xu et al., 2007) |
|---|---|---|---|---|
| Источник изоляции образца | Оз. Белое (г. Москвa) |
Почва овощного поля, загрязненного карбендазимом (провинция Цзянсу, Китай) | Осадки сточных вод очистительных сооружений, производcтва карбендазима (Цзянсу, Китай) | нд |
| Цвет колонии | Розовый | Оранжевый | Бледно-розовый | Бледно-розовый |
| Max NaCl, % | 5 | 5 | 7 | 5 |
| pH | 6–9.2 | 5–8 | 4–9 | 5–8 |
| Углерод и азот как единственный источник питания | ||||
| L-аспарагин | + | – | – | + |
| Беномил | – | + | + | – |
| Карбендозим | – | + | + | – |
| L-инозин | + | нд | нд | нд |
| L-арабиноза | + | нд | нд | нд |
| D-фруктоза | + | – | + | + |
| L-молочная кислота | – | – | – | + |
| Мальтоза | + | – | – | + |
| D-маннитол | + | – | + | + |
| D-сорбитол | + | – | – | + |
| Сахароза | + | + | – | + |
| D-ксилоза | – | – | – | + |
Недавно продемонстрировано уникальное свойство штамма 2012В, способного к биодеструкции наноалмазов (Сафронова, Кокшарова, 2018). Попадание наноматериалов в почву и воду приводит к негативным последствиям. В этой связи данный штамм может рассматриваться как потенциальный деструктор и этих углеродсодержащих материалов.
ПЦР и филогенетический анализ штамма 2012В. В результате ПЦР получен ДНК фрагмент, содержащий нуклеотидную последовательность (1479 пн) гена 16S рРНК малой субъединицы бактериальной рибосомы.
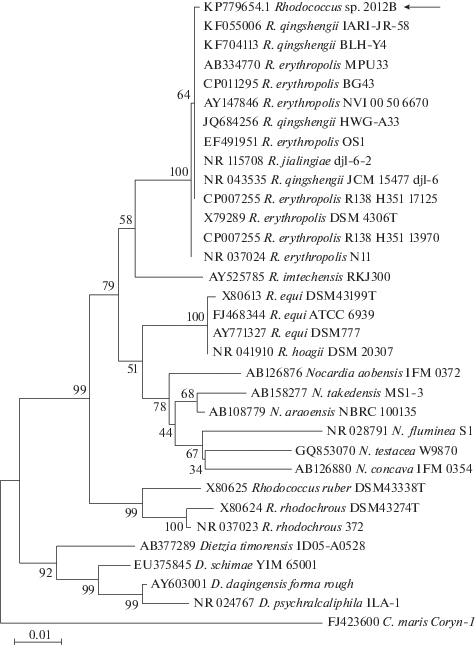
Филогенетический анализ позволил установить, что последовательность гена 16S рРНК попадает в отдельный кластер, сформированный бактериями Rhodococcus spр. и поддерживается тремя референтными штаммами – R. qingshengii sp. dj1-6T (NR_043535.1), R. jialingiae sp. dj1-6-2T (NR_115 708.1) и R. erythropolis sp. DSM 43 066T (X79 289) с гомологией >99.9% и со штаммом R. qingshengii BLH-Y4 (KF704113) со степенью сходства (99.93%) (рис. 2). Все последовательности гена 16S рРНК, входящие в кластер с последовательностью штамма Rhodococcus sp. 2012B, принадлежат к представителям одной монофилетической группы.
Рис. 2.
Эволюционные отношения штамма Rhodococcus sp. 2012B (KP779 654.1) c представителями рода Rhodococcus по фрагменту гена 16S рРНК (1286 пн). Эволюционные расстояния рассчитаны методом, описанным в работе Wang et al. (2010), и выражены в единицах количества замен оснований на сайт. Изменение скорости замены оснований в исследованной последовательности моделировали с помощью гамма-распределения (параметр формы = 0.44). В качестве аутгруппы использовали вид Corynebacterium maris.
Также видовую идентификацию проводили с помощью сравнения морфологических и физиолого-биохимических признаков с референтными штаммами – R. qingshengii dj1-6T, R. jialingiae dj1-6-2T и R. erythropolis DSM 43066T (Wang et al., 2010; Xu et al., 2007) (табл. 1).
Для уточнения результатов филогенетического анализа, проведенного с использованием рибосомального гена, в качестве дополнительного гена-маркера использовали белок-кодирующую последовательность alkB гена. Продукт этого гена – алканмонооксигеназа, обеспечивающая окисление n-алканов с длиной углеводородной цепи C6–C17 и катализирующая реакцию деградации углеводородов (Чернявская и др., 2012; van Beilen, Funhoff, 2007).
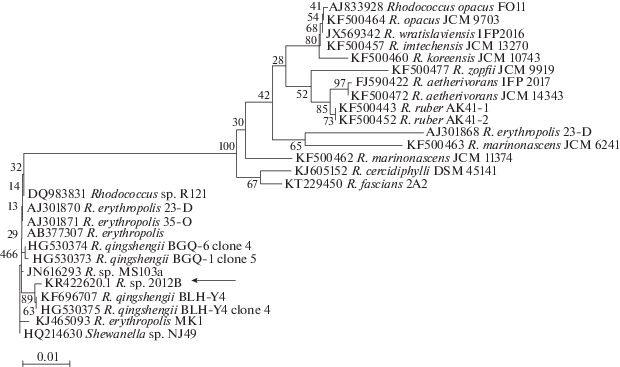
С помощью ПЦР для штамма 2012В получена нуклеотидная последовательность длиной 333 пн, соответствующая фрагменту гена alkB, которая позволила построить филогенетическое дерево (рис. 3). Видно, что ближайший родственник штамма Rhodococcus sp. 2012В – штамм R. qingshengii BLH-Y4 (KF696707) (идентичность 97.3%), выделенный из почвы Цинхай-Тибетского плато (Хu et al., 2007). Таким образом, уровень сходства последовательностей гена 16SрРНК и alkB показал, что по молекулярным таксономическим критериям (Stackebrandt, 2011) штамм 2012В можно отнести к виду Rhodococcus qingshengii sp. nov. (Xu et al., 2007).
Рис. 3.
Эволюционные отношения штамма Rhodococcus sp. 2012B (KR422 620.1) с представителями рода Rhodococcus по фрагменту гена аlkB (272 пн). Эволюционные расстояния рассчитаны с использованием трехпараметрического метода (van Beilen, Funhoff, 2007) и выражены в единицах количества замен оснований на сайт. Изменение скорости замены оснований в исследованной последовательности моделировали с помощью гамма-распределения (параметр формы = = 0.41). В качестве аутгруппы использовали штамм NJ49 рода Shewanella.
Характеристика штамма 2012С. Клетки грамотрицательные, аэробные, одиночные палочки (рис. 4). На среде ММС с дизельным топливом образуют выпуклые блестящие колонии бежевого цвета с ровным краем, размер клеток 2–3 мкм. На среде Мюллера клетки крупнее (3–5 мкм). Клетки флюоресцируют в проходящем свете. Бактерии давали положительный ответ на каталазу и отрицательный – на кислотоустойчивость.
Состав жирных кислот характеризуется преобладанием длинноцепочечных насыщенных и полиненасыщенных жирных кислот с длиной углеродной цепи C14:0–C19:0, где наиболее представлено жирным кислотами C15:0 (45%), C16:0 (32%) и C17:0 (9%).
Штамм 2012С способен утилизировать сахара и аминокислоты (L-аспарагин, L-инозин, D-мальтоза, D-сорбитол, L-арабиноза и D-арабитол); не утилизирует лактозу и фунгициды беномил и карбендозим.
ПЦР и филогенетический анализ штамма 2012С. В результате ПЦР получен фрагмент ДНК, содержащий нуклеотидную последовательность (936 пн) гена малой субъединицы бактериальной рибосомы 16S рРНК.
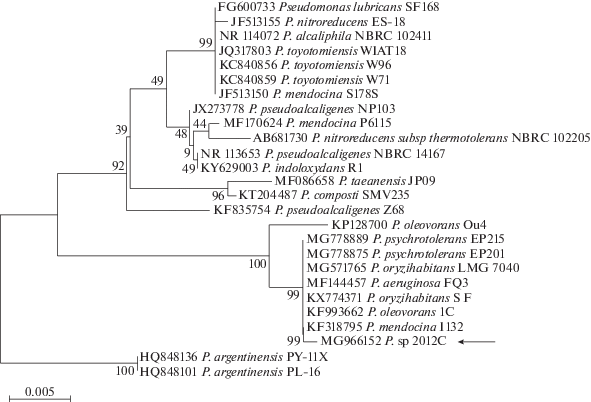
Установлено, что нуклеотидная последовательность гена 16SрРНК наиболее сходна с таковыми различных представителей сем. Pseudomonadaceae и позволяет отнести штамм 2012С к роду Pseudomonas (Proteobacteria, Pseudomonadaceae) (рис. 5). Филогенетический анализ показал, что штамм 2012С сформировал таксономически однородный кластер, в котором представлены штаммы рода Pseudomonas с гомологией 99.57%, при этом в кластере объединились бактерии из разных экосистем и разных географических зон. Так, P. mendocina (KF318 795) выделен из ризосферной почвы в Индии, P. psychrotolerans (MG778 875) – из бразильского цветка пассифлоры. P. oryzihabitans (MG571 765.1) из загрязненной почвы в Саудовской Аравии обладает способностью деградировать углеводороды.
Рис. 5.
Эволюционные отношения штамма Pseudomonas sp. 2012C (MG966 152) с представителями рода Pseudomonas по фрагменту гена 16S рРНК (917 пн). Эволюционные расстояния рассчитаны с использованием двух параметрического метода Кимуры (Long et al., 2003) и выражены в единицах количества замен оснований на сайт. Изменение скорости замены оснований в исследованной последовательности моделировали с помощью гамма-распределения (параметр формы = 0.44).
Культивирование штамма 2012С проводили на твердой синтетической среде ММС, содержащей в качестве единственного источника углерода летнее дизельное топливо, что свидетельствует о способности штамма 2012С деградировать алифатические углеводороды. С помощью ПЦР при использовании специфических праймеров (Kohno et al., 2002) получена нуклеотидная последовательность длиной ~220 пн, соответствующая фрагменту гена alkB. Полученные результаты дают возможность предположить, что в геноме Pseudomonas sp. 2012С присутствуют гены, кодирующие ферменты, отвечающие за окисление алканов с “короткой” углеродной цепью. Таким образом, филогенетический анализ позволил определить штамм Pseudomonas sp. 2012С как наиболее близкий к видам P. psychrotolerans и P. oryzihabitans.
Выводы. Из пресного водоема, находящегося в черте г. Москвы, выделены и охарактеризованы культивируемые углеводородокисляющие штаммы. Видовое таксономическое положение штамма Rhodococcus 2012В определено как Rh. qingshengii, штамм и Pseudomonas sp. 2012С наиболее сходны с видами P. psychrotolerans и P. oryzihabitans. Новые штаммы могут стать основой для создания биопрепаратов, стимулирующих процессы естественного очищения среды от нефтяных углеводородов, в том числе, от наиболее устойчивых полициклических ароматических соединений.
Список литературы
Акулова А.Ю., Ильинский В.В., Мошарова И.В. и др. 2014. Состояние гетеротрофного бактериопланктона прибрежья озер Святое и Белое природно-исторического парка “Косинский” (город Москва) в 2011 году // Известия Самарского научного центра РАН. Т. 16. № 1. С. 1185.
Кузнецов С.И. 1952. Роль микроорганизмов в круговороте веществ в озерах. Москва: Наука.
Кузнецов С.И. 1970. Микрофлора озер и ее геохимическая деятельность. Ленинград: Наука.
Россолимо Л.Л. 1925. Морфометрия Косинских озер // Труды лимнологической станции в Косине. № 2. С. 3.
Сафронова Н.А., Кокшарова О.А. 2018. Бактерия Rhodococcus sp. – потенциальный деструктор детонационных наноалмазов // Российские нанотехнологии. № 7–8. С. 88.
Чернявская М.И., Эльгаммуди А.А., Титок М.А. 2012. Первичная характеристика бактерий – деструкторов нефти // Вестник Белорусского государственного университета. Т. 2. № 3. С. 44.
Altschul S.F., Madden T.L., Schäffer A.A. et al. 1997. Gapped BLAST and PSI-BLAST: a new generation of protein database search programs // Nucleic Acids Res. V. 25. № 17. P. 3389.
Bergey D.H., Krieg N.R. Holt J.G. 1989. Bergey’s manual of systematic bacteriology. Baltimore, MD: Williams & Wilkins. V. 4. P. 2648.
Brooijmans R.J.W., Pastink M.I., Siezen R.J. 2009. Hydrocarbon-degrading bacteria: the oil-spill clean-up crew // Microb. Biotechnol. V. 2. № 6. P. 587.
Cameotra S.S., Singh P. 2008. Bioremediation of oil sludge using crude biosurfactants // Int. Biodeterior. Biodegrad. V. 62. P. 274.
Das N., Chandran P. 2011. Microbial degradation of petroleum hydrocarbon contaminants: an overview // Biotechnol. Res. Internat. V. 2011. P. 1. 13.
Felsenstein J. 1985. Confidence limits on phylogenies: An approach using the bootstrap // Evolution. V. 39. P. 783.
Fredriksson N.J., Hermansson M., Wilén Britt-Marie. 2013. The choice of PCR primers has great impact on assessments of bacterial community diversity and dynamics in a wastewater treatment plant // PLoS One. V. 8. № 10.
Hansen J., Møller I. 1975. Percolation of starch and soluble carbohydrates from plant tissue for quantitative determination with anthrone // Anal. Biochem. V. 68. P. 87.
Huber T., Faulkner G., Hugenholtz P. 2004. Bellerophon: a program to detect chimeric sequences in multiple sequence alignments // Bioinformatics. V. 20. P. 2317.
Kimura M. 1980. A simple method for estimating evolutionary rate of base substitutions through comparative studies of nucleotide sequences // J. Mol. Evol. V. 16. P. 111.
Kohno T., Sugimoto Y., Sei K., Mori K. 2002. Design of PCR primers and gene probes for general detection alkane-degrading bacteria // Microbiol. Environ. V. 17. № 3. P. 114.
Koksharova O., Shubert M., Shestakov S., Cerff R. 1998. Genetic and biochemical evidence for distinct key functions of two highly divergent gapdh genes in catabolic and anabolic carbon flow of the cyanobacterium Synechocystis sp. // Plant Mol. Biol. V. 36. P. 183.
Kumar S., Stecher G., Li M. et al. 2018. MEGA X: Molecular Evolutionary Genetics Analysis across computing platforms // Mol. Biol. Evol. V. 35. P. 1547.
Long G.R., Ayers M.A., Callender E., Van Metre P.C. 2003. Trends in chemical concentration in sediment cores from three lakes in New Jersey and one lake on Long Island, New York. U.S. // Geological Survey. Water-Res. Inv. Rep. V. 02-4272. P. 32.
Mills A.L., Breule C., Colwell R.R. 1978. Enumeration of petroleum – degrading marine and estuarine microorganisms by the most probable number method // Can. J. Microbiol. V. 24. P. 552.
Ravikumar P., Mehmood M.A., Somashekar R.K. 2013. Water quality index to determine the surface water quality of Sankey tank and Mallathahalli lake, Bangalore urban district, Karnataka, India // Appl. Water Sci. V. 3. № 1. P. 247.
Saitou N., Nei M. 1987. The neighbor-joining method: A new method for reconstructing phylogenetic trees // Mol. Biol. Evol. V. 4. P. 406.
Sambrook J., Fritsch E.F., Maniatis T. 1989. Molecular cloning: a Laboratory manual. New York: Cold Spring Harbor Laboratory Press.
Sasser M. 2001. Identification of bacteria by gas chromatography of cellular fatty acids // MIDI Technical Note 101.
Stackebrandt E. 2011. Molecular taxonomic parameters // Microbiol. V. 32. P. 59.
van Beilen J.B., Funhoff E.G. 2007. Alkane hydroxylases involved in microbial alkane degradation // Appl. Microbiol. Biotechnol. V. 74. P. 13.
Wang Z., Xu J., Li Y. et al. 2010. Rhodococcus jialingiae sp. nov., anactinobacterium isolated from sludge of a carbendazim wastewater treatment facility // Int. J. Syst. Evol. Microbiol. V. 60. P. 378.
Widmer F., Seidler R.J., Gillevet P.M. et al. 1998. A Highly Selective PCR Protocol for Detecting 16S rRNA Genes of the Genus Pseudomonas (Sensu stricto) in Environmental Samples // Appl. Env. Microbiol. V. 64. № 7. P. 2545.
Xu J.L., He J., Wang Z.C. et al. 2007. Rhodococcus qingshengii sp. nov., a carbendazim-degrading bacterium // Int. J. Syst. Evol. Microbiol. V. 57. P. 2754.
Дополнительные материалы отсутствуют.
Инструменты
Биология внутренних вод




