Биология внутренних вод, 2022, № 5, стр. 567-585
Эволюционные преобразования альбумина на примере модельных видов бесчелюстных рыбообразных и костных челюстноротых рыб (обзор)
А. М. Андреева *
Институт биологии внутренних вод им И.Д. Папанина Российской академии наук
пос. Борок, Некоузский р-н, Ярославская обл, Россия
* E-mail: aam@ibiw.ru
Поступила в редакцию 19.03.2022
После доработки 05.05.2022
Принята к публикации 17.05.2022
- EDN: UNKDMP
- DOI: 10.31857/S0320965222050023
Аннотация
На примере модельных представителей низших водных позвоночных: бесчелюстных рыбообразных (Agnatha, Cyclostomata) и челюстноротых костных лопастеперых (Sarcopterygii) и лучеперых (Actinopterygii: Chondrostei, Holostei, Teleostei) рыб рассмотрены вопросы организации альбумина – белка, принадлежащего суперсемейству альбуминоидов; проведен сравнительный анализ полной длины и длины кодирующей и некодирующей последовательностей гена альбумина, количества экзонов, длины интронов и наличия в них повторяющихся элементов ДНК; наличия/отсутствия генов-паралогов, их хромосомной принадлежности и состава синтеничных групп; длины аминокислотной последовательности, количества доменов и S‒S-связей в структуре альбумина. Обсуждены вопросы появления предкового гена альбуминоидов, “расширения” и “сужения” репертуара альбуминоидов в разных группах позвоночных, появления кластера генов-паралогов в составе одной хромосомы в ветви Sarcopterygii и генов-паралогов на разных хромосомах в ветви Actinopterygii у представителей хрящевых ганоидов и костистых рыб; удлинение гена и белковой цепи альбумина в линии миног и потери гена альбумина в наиболее массовых группах костистых рыб – костнопузырных Ostariophysi и колючеперых Acanthopterygii. Перечисленные сценарии были рассмотрены в свете их согласованности с событиями полногеномных, локальных и сегментных дупликаций. На примере группы костистых рыб обсуждалась проблема компенсации функций “утерянного” гена альбумина за счет множественных осмотически активных белков плазмы.
ВВЕДЕНИЕ
Альбумин принадлежит суперсемейству альбуминоидов, в состав которого также входят витамин D связывающий белок, альфа- фетопротеин и афамин. Альбуминоиды локализованы преимущественно в жидкостях организма и участвуют в транспорте ряда важных лигандов. Интерес к альбумину обусловлен его ключевым вкладом в гомеостаз физиологических процессов у высших позвоночных, который реализуется в виде транспортной и осмотической функций. По сравнению с другими альбуминоидами, альбумин транспортирует максимально широкий спектр лигандов – неорганические катионы, жирные кислоты, билирубин, витамины, гормоны, пептиды, белки и другие соединения, обеспечивая пластический и энергетический обмен позвоночных (Curry et al., 1998; Otterbein et al., 2002; Jerkovic et al., 2005; Ascenzi et al., 2013; Malik et al., 2013). Имея небольшие размеры глобулы и высокий титр в плазме крови (~60% общего белка), альбумин создает ее коллоидно-осмотическое давление, поддерживая волемию сосудов и осмотический гомеостаз внутренней среды организма (Dziegielewska et al., 1980; Byrnes, Gannon, 1990; Gray, Doolittle, 1992; Metcalf et al., 1998a, 1998b; Xu, Ding, 2005; Majorek et al., 2012; Anguizola et al., 2013).
Тестирование 183 видов амниот, амфибий и рыб на наличие альбумина выявило 100%-ное присутствие этого белка в первых двух группах организмов, в то время как среди рыб его имела лишь половина отобранных видов (Li et al., 2017). Например, среди костистых рыб (Teleostei) альбумины обнаружены у лососеобразных (Salmoniformes) (Nynca et al., 2017), а в семействе карповых рыб они не найдены (Noel et al., 2010). Подобные факты потери генов связывают с событиями полногеномных дупликаций (Noel et al., 2010; Braasch et al., 2014, 2016). В ходе эволюции позвоночные пережили несколько масштабных и менее крупных дупликационных событий, затронувших целые геномы или их ограниченные участки (Ohno, 1970). Все мультигенные семейства, включая альбуминоиды, формировались под влиянием этих процессов (Hoegg et al., 2004; Noel et al., 2010; Озернюк, Мюге, 2013; Soshnikova et al., 2013; Braasch et al., 2014, 2016). Параллельно с удвоением генов и геномов, в некоторых мультигенных семействах происходило “сужение” репертуара в виде потери отдельных генов (Braasch et al., 2016; Pasquier et al., 2016). Потеря гена альбумина в семействе карповых рыб – один из примеров последствий геномных перестроек. Однако, до сих пор нет ясности в вопросе масштабов этой потери у Teleostei, – ограничивается ли она только карповыми рыбами или охватывает более широкий спектр отрядов?
В обзоре рассмотрены пути эволюционных преобразований альбумина в составе суперсемейства альбуминоидов, на примере модельных представителей низших водных позвоночных – бесчелюстных рыбообразных (Agnatha) и костных челюстноротых рыб (Gnatostomata: Osteichthyes). Обсуждается связь этих сценариев с событиями полногеномных, локальных и сегментных дупликаций, а также проблема потери гена альбумина и компенсации этой потери в наиболее массовых группах костистых рыб – костнопузырных Ostariophysi и колючеперых Acanthopterygii.
Материал и методы исследования
Для поиска альбуминов были отобраны 26 видов, принадлежащих 20 отрядам, относящимся к бесчелюстным рыбообразным (Agnatha: Cyclostomata, 1 отряд) и костным челюстноротым рыбам (19 отрядов) – лопастеперым и лучеперым (Gnatostomata: Sarcopterygii, Actinopterygii) (табл. 1). При отборе объектов использовали сведения о наличии геномов в DB Genomes NCBI (Genome) и полногеномном секвенировании (BioProject), а также перечень модельных объектов из обзорной работы (Ravi, Venkatesh, 2018) с идентификаторами BioProject accession для 60 видов лучеперых рыб.
Таблица 1.
Список идентификаторов генов и белков у тестируемых объектов
| Таксон, группа | Вид | Наименование и идентификатор | Хромосома | |
|---|---|---|---|---|
| ген | белок | |||
| Cyclostomata | Petromyzon marinus (морская минога) | SDS-1 103091717 |
Serum albumin SDS-1-like XP_032800944 |
– |
| ALB или AS 103091689 |
Аlbumin (фрагменты) XP_032800109 |
– | ||
| Lethenteron camtschaticum (японская минога) | – | Plasma albumin BAF47283 |
– | |
| Сoelacanths | Latimeria chalumnae (латимерия) | Serum albumin-like 102347143 |
Serum albumin-like XP_006011457 |
– |
| Chondrostei | Acipenser ruthenus (стерлядь) | Serum albumin 2-like 117420476 |
Serum albumin 2-like XP_034777466.1 |
chr1 |
| Serum albumin 2-like 117408315 |
Serum albumin 2-like XP_033869101.2 |
chr2 | ||
| Holostei | Lepisosteus oculatus (панцирная щука) | Serum albumin 2-like 102693995 | Serum albumin 2-like XP_015195668 | LG2 |
| Teleostei Salmoniformes |
Salmo salar (лосось атлантический) | alb1 100136575 |
Serum albumin 1 | – |
| alb2 100136922 |
Serum albumin 2 | ssa18 | ||
| 106570911 | Albumin 2-like | ssa15 | ||
| Salmo trutta (кумжа) | Serum albumin 2 115193491 |
Serum albumin 2 | chr5 | |
| Serum albumin 2 115200616 |
Serum albumin 2 XP_029619662.1 |
chr9 | ||
| Serum albumin 2 115193491 |
Serum albumin 2 XP_029553400.1 |
chr18 | ||
| Oncorhynchus mykiss (Parasalmo mykiss) (радужная форель) |
Albumin 1 100499622 |
Albumin 1 XP_036844031.1 |
chr1 | |
| – | Albumin 2 isoform X1 XP_036792380.1 |
chr11 | ||
| Albumin 1 110531464 |
Albumin 1 XP_036816797.1 |
chr23 | ||
| Salvelinus namaycush (озерный голец) | Albumin 2-like 120046859 |
Albumin 2-like XP_038848354.12 |
chr4 | |
| Albumin 2-like 120063464 |
Albumin 2-like XP_038869773.1 |
chr18 | ||
| Albumin 2 120039988 |
albumin 2-like XP_038841236.1 |
– | ||
| Esox lucius (щука обыкновенная) | Serum albumin 2 ID:105008514 |
Serum albumin 2 XP_034148111 |
chr5 | |
| Serum albumin 2 ID:105014523 |
Serum albumin 2 XP_012992300 |
chr13 | ||
| Teleostei Ostariophysi |
Danio rerio Electrophorus electricus, Chanos chanos Pygocentrus nattereri, Ictalurus punctatus |
– | – | – |
| Teleostei Acanthopterygii |
Periophthalmodon schlosseri, Mugil cephalus, Neolamprologus bifasciatus, Odontesthes bonariensis, Poeciliopsis retropinna, Seriola lalandi, Cynoglossus semilaevis, Thunnus orientalis, Tautogolabrus adspersus, Sebastes nebulosus, Takifugu flavidus |
– | – | – |
| Mammalia* | Homo sapiens | ID:213 | NP_000468.1 | chr4 |
Систематика и латинские видовые названия даны согласно “Аннотированному каталогу круглоротых и рыб…” (Решетников, 1998). Для радужной форели использовали видовое название Parasalmo mykiss (Решетников, 1998) и Oncorhynchus mykiss. Последний вариант (O. mykiss) используется в базах данных Proteins, Genes (NCBI) и KEGG.
По костистым рыбам сформирован расширенный список модельных объектов, в который входят: (1) низшие Teleostei из двух семейств (Salmonidae, Esocidae) отряда Salmoniformes; (2) костнопузырные (Ostariophysi) из пяти отрядов – Cypriniformes, Gymnotiformes, Gonorynchiformes, Characiformes, Siluriformes; и (3) высшие костистые колючеперые (Acanthopterygii) из десяти отрядов – Gobiiformes, Mugiliformes, Cichliformes, Atheriniformes, Cyprinodontiformes, Carangiformes, Pleuronectiformes, Labriformes, Perciformes и Tetraodontiformes. В качестве референсной группы использовали млекопитающих (Homo sapiens) (табл. 1).
Сведения об аминокислотных последовательностях, доменной организации и S-S-связях альбумина и других альбуминоидов тестируемых видов получены из DB Proteins NCBI. Для сравнения использовали полные последовательности (исключение – фрагменты альбумина озерного гольца и миноги). Сведения об организации генов альбумина – экзон-интронной структуре; полной длине; длине кодирующей и некодирующей последовательностей; генах- паралогах, их хромосомной принадлежности и синтеничных группах – получены из DB Genes и Genome Data Viewer (GDV) NCBI. Для поиска близкородственных совпадений последовательностей в виде пяти лучших совпадений и расширенного списка гомологов, множественного выравнивания и количественной оценки сходства последовательностей использовали инструмент SmartBLAST и/или веб-ресурс KEGG. Выравнивание и сравнение сходных по положению в структуре генов альбумина последовательностей интронов, на наличие в них коротких повторяющихся последовательностей ДНК у тестируемых видов, проводили с помощью программы MEGA 6 (Tamura et al., 2013).
Наименования белков/генов альбуминоидов, обозначения параметров их организации даны в соответствии с DB Genes и Proteins NCBI: для альбумина и витамин D-связывающего белка низших позвоночных – ALB/alb и DBP/dbp соответственно; для альбумина, альфа- фетопротеина, афамина и витамин D-связывающего белка человека – ALB/ALB, AFP/AFP, AFM/AFM и DBP/DBP соответственно; СDS (coding sequence) – кодирующая последовательность ДНК; SINEs (short interspersed nuclear element) – короткие диспергированные повторы; LINEs (long interspersed nuclear element) – длинные диспергированные повторы; D – домен; L, а.о. – длина белковой цепи в аминокислотных остатках; L, нт – длина последовательности ДНК в нуклеотидах.
Наименования событий полногеномных, таксонспецифичных и других дупликаций даны в соответствии с принятыми в литературе обозначениями: WGD (whole genome duplication) – полногеномная дупликация; AsGD (acipenseric-specific WGD) – полногеномная дупликация у осетровых рыб; TGD (teleost WGD) – полногеномная дупликация у костистых рыб; CyGD (cyprinidae WGD) – полногеномная дупликация у карповых рыб; SaGD (salmonidae WGD) – полногеномная дупликация у лососевых рыб; SD – сегментная дупликация; LD – локальная дупликация; 1R, 2R, 3R – раунды WGD (Ohno, 1970; Freeling, Thomas, 2006; Braash et al., 2016).
Полногеномные, локальные и сегментные дупликации у позвоночных
Большинство биологов считают дупликацию генов и геномов с последующей дивергенцией источником эволюционного процесса, стимулировавшим появление и радиацию крупных таксонов животных и растений, например позвоночных и покрытосеменных (Freeling, Thomas, 2006). Дупликации генов и геномов достигаются с помощью дупликаций разного масштаба – WGD, SD и LD. При WGD амплифицируются протяженные участки ДНК (онологи). При SD под влиянием хромосомных перестроек амплифицируются менее протяженные геномные сегменты; при межхромосомных перестройках они могут оказаться на разных хромосомах. LD обычно происходят по типу тандемных повторов, в результате которых на хромосоме появляется кластер расположенных друг за другом родственных генов. Во всех перечисленных случаях происходит дупликация генома, группы генов или отдельных генов, в результате чего из исходных (предковых) генов получаются гомологичные им копии. Гомологичные гены в составе одного генома, образовавшиеся путем дупликации предкового гена, являются паралогами; гомологичные гены в составе геномов разных организмов, появившиеся в результате эволюции одного и того же предкового гена, который был в геноме общего предка этих организмов, являются ортологами (Ravi, Venkatesh, 2018).
В 1970 г. Сусуму Оно (Ohno, 1970) вынес на обсуждение гипотезу 2R, согласно которой анцестральный геном позвоночных до их диверсификации прошел через два раунда полногеномных дупликаций – 1R (WGD1) и 2R (WGD2). Позднее челюстноротые рыбы прошли через третий раунд полногеномной дупликации (3R, WGD3), давший начало таксону костистых рыб (Teleostei) и потому обозначенный как TGD (Ohno, 1970; Noеl et al., 2010; Braasch et al., 2016; Pasquier et al., 2016). Среди тетрапод дупликации геномов произошли у амфибий и рептилий (Sauria); у птиц и млекопитающих устойчивые полиплоидные формы не обнаружены (Васильев, 1985; Uno et al., 2013; Evans et al., 2017; Ravi, Venkatesh, 2018) (рис. 1).
Рис. 1.
Схема эволюции позвоночных с отметкой основных раундов полногеномных дупликаций (WGD1, WGD2, WGD3) относительно этапов дивергенции Vertebrata на Agnatha и Gnatostomata, и расхождения последних на Sarcopterygii и Actinopterygii. Esoc – Esocidae; Salm – Salmonidae. Звездочками отмечены дупликации в отдельных отрядах в составе таксонов. Пояснения в тексте.

Большинство авторов считают, что первые два раунда дупликаций имели место до разделения бесчелюстных и челюстноротых ~500–550 млн лет назад (Holland et al., 1994; Kuraku et al., 2009a, 2009b; Van de Peer et al., 2017). Другая точка зрения поддерживает сценарий, в котором первый раунд отнесен к периоду до расхождения бесчелюстных и челюстноротых, а второй – к периоду после их расхождения (Nakatani et al., 2021). Третий раунд дупликаций произошел, предположительно, ~230–400 (Venkatachalam et al., 2017) или ~300–450 млн лет назад (Taylor et al., 2001).
Анализ геномов у круглоротых указывает на высокую вероятность независимых от челюстноротых событий полиплоидизации (гексаплоидизации) в линии миног (Smith, Keinath, 2015; Nakatani et al., 2021). Независимые события полиплоидизации произошли у осетрообразных в семействах Acipenseridae и Polyodontidae (Crow et al., 2012; Du et al., 2020; Cheng et al., 2021). В ветви лучеперых рыб после события TGD и появления таксона Teleostei, дупликации геномов произошли у карповых (СyGD) (Ma et al., 2014) и лососевых рыб (SaGD) (Alexandrou et al., 2013; Berthelot et al., 2014; Животовский, 2015). Полиплоиды также обнаружены в отдельных отрядах колючеперых, например, у губанообразных Labriformes, лабиринтообразных Anabantiformes и некоторых других (Васильев, 1985; Felip et al., 2009; Cioffi et al., 2015) (рис. 1).
Помимо гипотезы 2R, предложены модели, рассматривающие вклад в эволюционный процесс как полногеномных, так и локальных и сегментных дупликаций (Freeling, Thomas, 2006). Например, в модели “1R + SD” учитывается вклад множественных сегментных дупликаций в геномные перестройки миног до события 1R и после него (Asrar et al., 2013; Smith, Keinath, 2015; Nakatani et al., 2021). Предполагается, что в возникновение Salmonidae внесли вклад как полногеномные дупликации TGD и SaGD, так и SD (Васильев, 1977; Животовский, 2015).
Организация генов альбуминоидов на хромосомах
Среди позвоночных только амниоты имеют полный набор из четырех генов альбуминоидов. У млекопитающих все четыре гена – DBP, ALB, AFP и AFM – находятся на одной хромосоме (рис. 2). У челюстноротых рыб обнаружены гены двух альбуминоидов – alb и dbp. Бесчелюстные имеют только один альбуминоид – альбумин; ген dbp у них отсутствует (Fanali et al., 2012). Подобно млекопитающим, у панцирной щуки и стерляди гены alb и dbp расположены на одной хромосоме в противоположной транскрипционной ориентации (рис. 2б). У низших костистых рыб обнаружено 2–3 гена-паралога альбумина: у щуки обыкновенной – 2 гена, у лососевых – 3. Причем, гены alb и dbp могут находиться как на одной, так и на разных хромосомах. У щуки гены-паралоги находятся на двух разных хромосомах. У микижи 3 гена-паралога альбумина находятся на хромосомах 1, 11 и 23; на хромосоме 11 расположены гены и альбумина и dbp; на хромосомах 1 и 23 – только гены альбумина.
Рис. 2.
Положение на хромосоме генов альбуминоидов в составе синтеничных групп у человека (а), стерляди (б), лосося атлантического (в) и щуки обыкновенной (г). Стрелки показывают направление транскрипции. un – ген не определен. Наименования генов и пояснения в тексте.

У млекопитающих и рыб синтеничные группы генов, включающие альбумин, различны. Не совпадают они у панцирной щуки, стерляди и лососеобразных. В то же время в отряде лососеобразных обнаружены две устойчивые синтеничные группы, включающие гены-паралоги альбумина (рис. 2в, 2г). У щуки обыкновенной, которая рассматривается как наиболее близкий к общим предкам лососеобразных вид (Rondeau et al., 2014), синтеничная группа на хромосоме 13 включает, с учетом альбумина, пять генов: E3 ubiquitin protein lipase K CMF1 like – solute carrier family 20 member – albumin – methylenetetrahydrofolate dehydrogenase (NADP+ dependent) 2 like – epithelial mitogen (epigen). Другая синтеничная группа (также из пяти генов) на хромосоме 5 включает другие гены: poly(A) polymerase gamma – retinal homeobox protein Rx1-like – albumin – v-rel avian reticuloendotheliosis vital oncogene homolog – peroxisomal biogenesis factor 13 (рис. 2в).
У лосося атлантического синтеничная группа на хромосоме ssa15 совпадает (кроме одного гена) с последовательностью генов щуки на хромосоме 13, но только в обратном порядке: epigen – methylenetetrahydrofolate dehydrogenase (NADP + dependent) 2 like – albumin – sodium dependeter phosphate transporter 2 – Un – E3 ubiquitin protein lipase K CMF1 like (рис. 2г). Другая синтеничная группа на неустановленной хромосоме лосося (Un) полностью совпадает с порядком генов у щуки на хромосоме 5 (рис. 2г).
Обе синтеничные группы встречаются на разных хромосомах у кумжи Salmo trutta озерного гольца Salvelinus namaycush радужной форели Oncorhynchus mykiss в виде полных или укороченных вариантов. При этом, в каждом случае отмечаются примеры преобразования синтеничных групп в результате инверсий единичных генов, нескольких генов или полных групп сцепления.
Экзон-интронная организация генов альбумина
Длина кодирующей последовательности СDS гена альбумина у челюстноротых относительно постоянна (табл. 2). У всех протестированных видов она организована в виде 14–16 экзонов; минимальное число экзонов – 13 – имеет ген альбумина озерного гольца Salvelinus namaycush на хромосоме 4. За исключением двух крайних экзонов, остальные кодируют полипептидную цепь из трех доменов. Длины полных последовательностей гена варьируют существенно за счет разной длины интронов – некодирующих участков ДНК (табл. 2, 3, рис. 3). У миноги все перечисленные показатели превосходят таковые у челюстноротых рыб.
Таблица 2.
Параметры организации гена и полипептидной цепи альбумина у модельных видов бесчелюстных рыбообразных и челюстноротых рыб (по DB Genes и Proteins NCBI от 14.02.2022 г.)
| Вид | ID гена, хромосома | L1, нт | СDS, нт | Экзоны | L2, а.о. | D | S‒S |
|---|---|---|---|---|---|---|---|
| Petromyzon marinus | 103091717, – | 110 143 | 3495 | 24 | 1423 | 7 | 41 |
| Latimeria chalumnae | 102347143, – | 58 773 | 1842 | 15 | 613 | 3 | – |
| Acipenser ruthenus | 117420476, chr1 | 42 995 | 2337 | 14 | 778 | 3 | – |
| 117408315, chr2 | 13 084 | 1839 | 15 | 612 | 3 | – | |
| Lepisosteus oculatus | 102693995, LG2 | 14 504 | 1842 | 15 | 613 | 3 | – |
| Salmo salar | 106570911, ssa15 | 20 122 | 1827 | 15 | 608 | 3 | 18 |
| 100136575, – | 22 205 | 1827 | 14 | 608 | 3 | 18 | |
| – , ssa18 | – | – | – | – | – | – | |
| Esox lucius | 105008514, chr5 | 12 201 | 1827 | 16 | 608 | 3 | – |
| 105014523, chr13 | 10 641 | 1833 | 14 | 610 | 3 | – | |
| Homo sapiens | 213, chr4 | 17 196 | 1830 | 15 | 609 | 3 | 17 |
Примечание. L1, L2 – длина полной нуклеотидной последовательности гена в нуклеотидах (нт) и аминокислотной последовательности в аминокислотных остатках (а.о.) соответственно; CDS – длина кодирующей последовательности в нуклеотидах (нт); D – число альбуминовых доменов, S‒S – число дисульфидных связей. “–” – данные отсутствуют.
Таблица 3.
Положение и длина интронов в генах альбумина Gnatostomata (по DB Genes NCBI от 01.04.2022 г.)
| Положение интрона между экзонами | Длина интрона, нт | |||||||
|---|---|---|---|---|---|---|---|---|
| L.c. (Un) |
A.r. (chr 2) |
L.o. (LG2) |
S.s. (ssa15) |
S.s. (Un) |
E.l. (chr 5) |
E.l. (chr 13) |
H.s. (chr 4) |
|
| 1ex и 2ex | 7796 | 469 | 724 | 467 | 6894 | 1165 | 377 | 709 |
| 2ex и 3ex | 1058 | 1829 | 701 | 7411 | 3019 | 817 | 1500 | 1455 |
| 3ex и 4ex | 2439 | 279 | 720 | 3139 | 183 | 2665 | 1001 | 1832 |
| 4ex и 5ex | 1415 | 2083 | 1152 | 136 | 4919 | 705 | 84 | 549 |
| 5ex и 6ex | 5470 | 699 | 108 | 327 | 189 | 107 | 327 | 824 |
| 6ex и 2ex | 3422 | 510 | 552 | 111 | 109 | 133 | 417 | 1586 |
| 7ex и 2ex | 7030 | 1827 | 905 | 284 | 605 | 95 | 602 | 1294 |
| 8ex и 2ex | 1013 | 765 | 4144 | 491 | 201 | 314 | 1917 | 1400 |
| 9ex и 2ex | 6868 | 457 | 303 | 4010 | 1418 | 502 | 503 | 1088 |
| 10ex и 11ex | 2570 | 946 | 247 | 778 | 303 | 352 | 258 | 1177 |
| 11ex и 12ex | 6536 | 332 | 760 | 158 | 1017 | 215 | 193 | 418 |
| 12ex и 13ex | 4996 | 131 | 1706 | 163 | 195 | 1468 | 277 | 1195 |
| 13ex и 14ex | 3908 | 759 | 169 | 240 | 930 | 109 | 283 | 614 |
| 14ex и 15ex | 206 | 81 | 333 | 770 | ||||
| 15ex и 16ex | 1214 | |||||||
Примечание. L.c. – латимерия, A.r. – стерлядь, L.o.– панцирная щука, S.s. – лосось атлантический, E.l. – щука обывновенная, H.s.– человек. В скобках ‒ обозначение хромосомы, Un –хромосома не установлена. Заливкой выделены интроны, по которым проводили поиск и сравнение повторяющихся последовательностей.
Рис. 3.
Графическое представление экзон-интронной организации генов альбумина у морской миноги (Petromyzon marinus), латимерии (Latimeria chalumnae), стерляди (Acipenser ruthenus), панцирной щуки (Lepisosteus oculatus), атлантического лосося (Salmo salar), щуки обыкновенной (Esox luceus) и человека (Homo sapiens). Экзоны представлены в виде темных вертикальных боксов разной толщины в зависимости от их длины, интроны – в виде горизонтальных линий между ними. D1–D3 – домен. Использованы графические данные DB Genes NCBI.
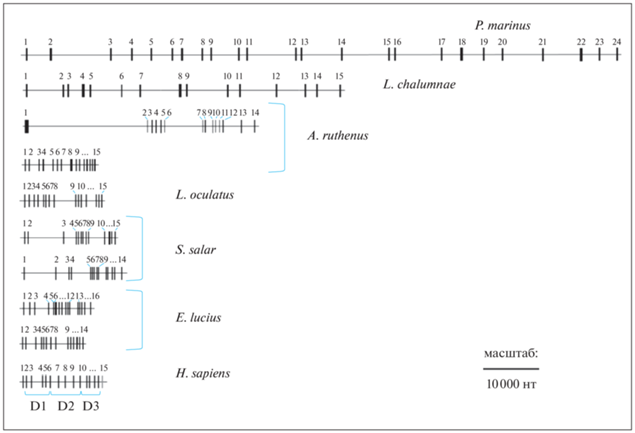
Сравнение длин соответствующих интронов в генах альбумина челюстноротых рыб и человека не выявило ни одного совпадения (табл. 3). Большинство представленных в DB NCBI последовательностей интронов начинаются с динуклеотида GT и заканчиваются AG (сайты сплайсинга); заметим, что в ряде представленных последовательностей (интроны между вторым и третьим экзонами гена альбумина щуки обыкновенной Esox Lucius на хромосоме 5 и гена альбумина панцирной щуки Lepisosteus oculatus эти сайты отсутствуют.
Сравнение повторяющихся элементов в структуре интронов генов альбумина рыб и человека, расположенных между экзонами 2 и 3 и выделенных в табл. 3 розовым цветом, не выявило совпадений. В интронах гена альбумина человека имеются 11 SINEs и 6 LINEs – коротких и длинных диспергированных повторов ДНК. Они не следуют в линейном порядке друг за другом, а разбросаны по разным участкам последовательности (Nishio, Dugaiczyk, 1996; Nishio et al., 1996). Cреди SINEs наиболее известны специфичные для приматов Alu – повторы, названные по наименованию рестриктазы Alu, обработка которой ДНК и привела к их обнаружению: aattgcttgtgtttctt, gatgtatg, gaatactcattcat и другие. В гене альбумина человека выявлено 4 Alu-последовательности.
У рыб в составе интронов гена альбумина обнаружены другие короткие повторы. Так, интрон, расположенный между экзонами 1 и 2, содержит tg-повторы разной длины – от 40 до 124 п.н. у лососевых и более короткие – у латимерии. Самый длинный второй интрон (7411 п.н.) в гене альбумина лосося на хромосоме ssa15 содержит 13 копий коротких повторов agagtatggc, 10 копий ccttggtaat и 11 копий aaccaggtcc. Перечисленные последовательности отсутствуют у гена-паралога альбумина лосося, а также в генах альбумина радужной форели, латимерии и человека.
Область промотора гена альбумина у тестируемых видов представлена в DB NCBI только последовательностью у лосося атлантического. Она состоит из 410 п.н. (Salmo salar albumin promoter region: GenBank X79487.1). У человека область промотора детально исследована (Kajiyama et al., 2006). Она состоит из 406 п.н. (albumin (ALB) 5' regulatory region; RefSeq NC_000004.12). И у лосося, и у человека промоторы генов альбумина содержат ТАТА-мотивы. Заметим, что у многих позвоночных промоторы ряда генов могут содержать другие инициирующие элементы (Carninci et al., 2006). Промоторный мотив ТАТА-бокс гена альбумина лосося состоит из шести нуклеотидов (ТАТААА), у человека – из семи (ТАТАААА). Регуляторные области генов альбумина лосося и человека гомологичны, что подтверждается специфическим связыванием фактора транскрипции HNF1 лосося промоторными участками генов альбумина рыб и млекопитающих (Deryckere et al., 1995).
Организация полипептидной цепи альбумина
Полипептидная цепь альбумина и других альбуминоидов состоит из трех альбуминовых доменов, являющихся структурными и функциональными единицами (Li et al., 2017). Домены содержат сайты связывания разнообразных эндогенных и экзогенных лигандов, включая семь сайтов связывания жирных кислот (Ghuman et al., 2005). Каждый домен содержит ~190 аминокислотных остатков (а.о.). У млекопитающих каждый домен стабилизирован 5–6 S‒S- связями; полная полипептидная цепь имеет 17 S‒S-связей (Saber et al., 1977). Спирализованные области полипептидной цепи придают ей конформационную гибкость, а S‒S-связи поддерживают необходимую жесткость пространственной структуры. Подобно млекопитающим, рыбы сохраняют консервативную трехдоменную организацию альбуминов, а также консервативное количество аминокислотных остатков и S-S-связей. У миног все показатели пропорционально возрастают (табл. 2, рис. 4).
Рис. 4.
Графическое изображение альбуминовых доменов (D) полипептидной цепи альбумина человека (HSA) (RefSeq NP_000468.1) (а) и японской миноги Lеthenteron camtschaticum (GenBank BAF47283.1) (б); положения цистеинов (С77‒С193) и S‒S-связей в домене (D1) HSA (RefSeq NP_000468.1) (в). Пояснения в тексте.

Консервативность перечисленных параметров поддерживается при относительно высоких показателях сходства аминокислотных последовательностей на внутриродовом и внутриотрядном уровнях. В то же время, сходство последовательностей у представителей разных отрядов оказывается минимальным. Так, сходство альбумина SDS-1 морской миноги с альбуминами того же вида и японской миноги достигает ~77–99%, а с альбуминами млекопитающих лишь ~22–24%. Сходство альбумина стерляди (612 а.о.) с альбуминами осетрообразных – ~81–99%; с альбуминами панцирникообразных, многоперообразных и лососевых ~58, ~49–51 и ~43% соответственно; с AFP и AFM амниот ~28–35% и с DBP позвоночных <28%. Такая же закономерность прослеживается для альбумина лосося атлантического: высокое сходство последовательностей на внутриродовом (~78–99%) и внутриотрядном (~64–93%) уровнях, более низкое – на межотрядном уровне (~41–49% с альбуминами панцирной щуки и осетрообразных) и минимальное – с альбуминоидами амниот (~23–27%) и DBP (~20–22%).
Для панцирной щуки максимальное сходство (93%) также отмечено на внутриотрядном уровне с альбумином миссисипского панцирника Atractosteus spatula; более низкие показатели – с альбуминами осетрообразных (~58–60%) и лососеобразных (~43–49%) и минимальное сходство – с альбуминами млекопитающих (~31–33%), AFP и AFM амниот (~26–32%) и DBP (~21–22%).
Альбумины морской миноги имеют особый статус, обусловленный большой длиной полипептидной цепи (1423 а.о.), наличием семи альбуминовых доменов и пропорционального размеру белка числа S‒S-связей (41), в целом, сохраняя сходство с альбуминоидами челюстноротых. Высокий уровень сходства последовательностей альбуминов миног на внутриотрядном уровне по сравнению с альбуминами других позвоночных, вероятно, объясняется их независимым происхождением от одно- или двухдоменного предшественника, в отличие от альбуминов других позвоночных, ведущих начало от трехдоменного предшественника (Gray, Doolittle, 1992). Гомология аминокислотных последовательностей альбуминов миног и альбуминоидов челюстноротых сохраняется на всей протяженности миножьей аминокислотной цепи (рис. 5).
Рис. 5.
Множественное выравнивание в SmartBLAST последовательностей альбуминов японской речной миноги Lethenteron camtschaticum (а) и морской миноги Petromyzon marinus (б) с пятью ближайшими гомологами альбуминоидами ALB, VDB, AFP и AFM человека Homo sapiens, мыши Mus musculus и рыбки данио Danio rerio. Вверху – шкала длины аминокислотных последовательностей, справа – их длина; рядом с наименованием белка и вида указан идентификатор ID.

Эволюционные преобразования альбумина и их связь с дупликацией геномов/генов
Трехдоменные альбуминоиды произошли из однодоменного предшественника. Первоначально этот вывод сделан на основе анализа аминокислотной последовательности бычьего сывороточного альбумина и, прежде всего, по расположению S‒S-мостиков, характеризующихся 3-кратным повторением в аминокислотной последовательности (Brown, 1976). Позднее выяснилось, что последовательности альбуминоидов других видов млекопитающих имели сходную архитектуру S‒S-связей и трехдоменную структуру (Gorin et al., 1981; Jagodzinski et al., 1981; Morinaga et al., 1983 и др.; Minghetti et al., 1985).
Начало альбуминоидам дал древний ген, кодирующий “полудоменный” белок (½D) (Gibbs, Dugaiczyk, 1987). Он имел в своей структуре пять экзонов. Из него произошел ген с семью экзонами, кодирующий однодоменный (1D) белок. На его основе появился ген, кодирующий двухдоменный белок (2D), и содержащий 11 экзонов, и далее – анцестральный ген из 15 экзонов, кодирующий трехдоменный белок – предшественник альбуминоидов (3D). Удлинение древнего гена произошло в результате двух событий негомологичной рекомбинации (неравный кроссинговер) и одного события гомологичной рекомбинации. Предполагается, что анцестральный ген был дуплицирован, дав начало двум протогенам-паралогам – прото-ALB и прото-DBP. Из прото-ALB появились гены ALB, которые дали начало генам AFP и AFM, а из прото-DBP появились гены витамин D-связывающего белка позвоночных (Eiferman et al., 1981; Ohno, 1981; Sargent et al., 1981; Gibbs, Dugaiczyk, 1987; Gray, Doolittle, 1992; Noel et al., 2010). Ген DBP на начальных этапах эволюции потерял два экзона (12-й и 13-й) (Nishio, Dugaiczyk, 1996; Malik et al., 2013). DBP не обнаружен у миног (Gray, Doolittle, 1992), однако, представлен почти во всех других группах позвоночных и за ~500 млн лет их эволюции хорошо сохранил свою структуру (Bouillon et al., 2020). Ген альбумина имеют все позвоночные, кроме некоторых костистых рыб; AFP и AFM имеют только амниоты (Noel et al., 2010).
События дупликации генов, которые привели к общей для альбуминоидов трехдоменной структуре, предположительно, происходили между ответвлением миног от ствола позвоночных (~450 млн лет назад) и появлением DBP (Gray, Doolittle, 1992). Альбумин миног произошел, скорее всего, в результате дупликации гена, кодирующего одно- или двудоменный белок, независимо от событий, ведущих к трехдоменному альбумину, и до появления гена DBP (Gray, Doolittle, 1992). В линии миног произошло удлинение полипептидной цепи альбумина до семи доменов. По аналогии с моделью Гиббса и Дужека (Gibbs, Dugaiczyk, 1987), можно предположить, что это удлинение могло быть результатом негомологичных рекомбинаций в виде неравного кроссинговера. Литературные данные указывают на наличие у миног двух альбуминов – AS и SDS-1, предположительно, кодируемых генами-паралогами (Filosa et al., 1998). Недавние исследования указывают на высокую вероятность независимых от челюстноротых событий полиплоидизации в ветви миног в виде гексаплоидизации и сегментарных дупликаций (Smith, Keinath, 2015; Nakatani et al., 2021), что косвенно поддерживает точку зрения о возможности наличия у миног генов-паралогов альбумина.
После события 2R эволюция альбумина на основе трехдоменного предшественника в разных ветвях челюстноротых происходила по разным сценариям. Латимерия (Sarcopterygii) и панцирная щука (Holostei) имели типичный для позвоночных диплоидный геном, сходный с геномом человека (Ravi, Venkatesh, 2018), их альбумины детерминированы одним геном. У Tetrapoda (Sarcopterygii) произошли дупликационные события, затронувшие альбуминоиды. Так, у амфибий ~40 млн лет назад произошла полногеномная дупликация, в результате чего лягушки имеют гены альбумина на гомеологичных хромосомах. У амниот полиплоиды появились в группе Sauria (ящерицы); у птиц и Mammalia стабильные полиплоиды не обнаружены (Васильев, 1985). Между тем у всех амниот есть гены AFP и AFM, отсутствующие у других Vertebrata. Они появились, предположительно, вследствие локальной – тандемной дупликации анцестрального гена альбуминоидов и появления предкового гена AFP/AFM (Noel et al., 2010). Другая точка зрения рассматривает AFM как промежуточное звено между ALB и AFP (Fasano et al., 2007). Таким образом, у тетрапод эволюция альбумина шла в двух направлениях: появление множественных копий гена альбумина в составе гомеологичных хромосом у аллотетраплоидов (амфибии) и тандемная дупликацая гена альбумина с появлением генов-паралогов AFP и AFM, в результате чего возник кластер ALB/AFP/AFM, от которого на отдалении 1.5 млн п.н. расположен ген DBP (амниоты). Появление AFP/AFM произошло, вероятно, после 2R и расхождения амфибий и рептилий ~320 млн лет назад (Noel et al., 2010).
В другой ветви челюстноротых – Actinopterygii – в отряде осетрообразных у стерляди выявлено две копии гена альбумина, появившиеся, вероятно, вследствие специфичной для Acipenseriformes дупликации генома. На отрезке времени ~230–450 млн лет назад, параллельно со временем расхождения амфибий и рептилий, в ветви Actinopterygii произошло событие TGD, давшее начало таксону Teleostei и стимулировавшее диверсификацию этой группы. В результате реорганизации геномов наиболее массовые группы костистых – колючеперые и костнопузырные – утратили ген альбумина, а низшие костистые – лососевые и щуковые – получили дополнительные копии этого гена.
На основе представленных данных предлагается схема эволюции альбумина в группах Agnatha и Gnatostomata (рис. 6), дополненная событиями полногеномных и сегментарных дупликаций, рекомбинаций и дупликаций гена альбумина, в том числе у тетрапод в группе Amniota.
Рис. 6.
Схема эволюционных преобразований предшественника альбуминоидов и альбумина под влиянием WGD: у бесчелюстных (а) и челюстноротых до события TGD (б) и после него (в). Схема дополнена событиями сегментарных и локальных (тандемных) дупликаций, а также событием потери генов альбумина. Пояснения в тексте.

Эволюционная судьба генов-паралогов альбумина после дупликационных событий
Сусумо Оно (Ohno, 1970) предположил, что наиболее часто после дупликации один из двух генов-паралогов теряет функциональность. Одной из причин потери может быть накопление вредных мутаций, которые приводят к вырождению, псевдогенизации, нефункциональному состоянию гена. Псевдогены характеризуются определенной степенью гомологии с функциональными генами и изменениями организации, препятствующими их нормальной транскрипции и трансляции. Бóльшая часть копий теряет функциональность в процессе ре-диплоидизации, следующей за дупликационным событием. Анализ последствий TGD на примере мультигенных семейств Teleostei указывает на высокий процент утраты их генов после дупликации геномов (Brunet et al., 2006; Braash et al., 2016). Так, у Danio rerio сохраняется ~20% всех дуплицированных генов, остальные теряют функциональность (Postlethwait et al., 2000), а в целом, по группе костистых, этот показатель достигает ~17% (Braasch, Postlethwait, 2012). Примером потери гена как результата TGD, вероятно, является и отсутствие гена альбумина у колючеперых и костнопузырных рыб. Это событие не имеет ничего общего с отсутствием DBP у миног. В первом случае речь идет о возможной утрате генов альбумина, так как до события TGD альбумины были во всех группах костных челюстноротых. Во втором случае отсутствие DBP объясняется тем, что ответвление бесчелюстных произошло до появления трехдоменного предшественника всех альбуминоидов, а значит, и до появления прото-DBP.
В то же время, некоторые группы рыб, например Salmonidae, демонстрируют включение почти половины всех дуплицированных генов в процессы суб- и неофункционализации (Berthelot et al., 2014). Эти два сценария реализуются в эволюции генов-паралогов, которые сохранились после дупликационных событий. При субфункционализации происходит разделение функций исходного гена между паралогами. Модель “дупликации–вырождения–дополнения” или DDC (Duplication-Degeneration-Complementation (Force et al., 1999) предлагает возможный сценарий распределения функций предкового гена между двумя паралогами. Согласно ей, накопление мутаций происходит в обеих копиях. Это ведет к снижению их функциональности, в результате чего обе копии в сумме воспроизводят эффект одного (исходного) гена. Если мутации касаются регуляторных областей, то это будет отражаться на паттернах экспрессии паралогов. Если мутации касаются белок-кодирующих областей, то они могут привести к распределению функций исходного гена между его дочерними копиями. Примеры этих сценариев описаны в ряде экспериментальных работ и обзоров (Озернюк, Мюге, 2013; Каменская, Брыков, 2020; Байрамов и др., 2021; Gu, Xia, 2019 и др.).
Примеряя перечисленные сценарии к альбуминоидам, отметим, что первоначально кодируемый предковым геном трехдоменный анцестральный белок, вероятно, выполнял функцию транспорта. Его дупликация привела к появлению двух паралогов – прото-ALB и прото-DBP – давших начало генам альбумина и витамин D-связывающего белка. Последующая их субфункционализация могла проходить в виде специализации каждого из них на транспорте определенного круга лигандов: широкого – в случае альбумина и узкого – в случае DBP. Возможно, что сужение функциональности DBP произошло вследствие потери им двух экзонов на ранних этапах эволюции. Примером неофункционализации может служить приобретение одним из двух паралогов, а именно альбумином, новой (осмотической) функции. Это могло произойти из-за повышения уровня экспрессии кодирующего его гена, возможно, в результате мутаций в его регуляторной области. Соответствующий уровень экспрессии обеспечил высокий титр альбумина в плазме, оказавшийся достаточным для создания им коллоидно-осмотического давления плазмы. Поскольку среди всех альбуминоидов только альбумин имеет максимально высокий титр в плазме позвоночных, именно он и был закреплен отбором в качестве идеального белкового фактора, контролирующего гомеостаз внеклеточной жидкости в организме позвоночных.
Другой пример субфункционализации касается альбумина у миног и тетрапод. Паралоги гена альбумина SDS-1 и AS разделили функцию транспорта лигандов между разными стадиями онтогенеза (Filosa et al., 1998), подобно генам-паралогам ALB и AFP у млекопитающих, разделившим функцию транспорта между эмбриональным и постнатальным этапами развития.
Еще один пример суб- и неофункционализации демонстрируют лососевые, чьи геномы прошли через полногеномные и сегментные дупликации и испытали не менее шести событий множественного слияния хромосом (Makhrov, 2017). Почти половина предковых генов (48%) сохранилась у лососевых в виде копий, значительная часть которых имеет дивергентные профили (уровни) экспрессии (Berthelot et al., 2014). Данное обстоятельство указывает на высокую вероятность мутаций в регуляторных областях паралогов и на то, что у лососевых почти половина генов-паралогов вовлечена в процессы суб- и неофункционализации. Возможно, что в эти процессы вовлечены и гены-паралоги альбумина.
После дупликационных событий геномы проходят через период ре-диплоидизации или возврата к диплоидному состоянию (Ravi, Venkatesh, 2018). Этим, вероятно, и объясняется причина несовпадения в количествах реальных и ожидаемых генов-паралогов у полиплоидов. Установлено (Ramberg et al., 2021), что у Salmo salar среднее число паралогов для ряда генов, кодирующих ферментные белки, – три. Такое же число паралогов установлено и для альбумина у S. salar. По-видимому, это число больше двух из-за того, что лососевые пережили (помимо 1R и 2R) две полногеномные дупликации – TGD и SaGD. В то же время оно меньше ожидаемой величины “четыре”, возможно, вследствие процесса ре-диплоидизации полиплоидного генома, который лососевые пережили в позднем меловом периоде и эоцене (Gundappa et al., 2021). Между тем, у щуки обыкновенной обнаруживается только два, а не три гена-паралога альбумина, что может объясняется только одним (помимо 1R и 2R) дупликационным событием в виде TGD в эволюции Esocidae.
После приведенных примеров той или иной стратегии в эволюции генов-паралогов возникает закономерный вопрос – какие факторы определяют ее выбор?
Факторы, влияющие на эволюционную судьбу генов-паралогов
Эволюционная стратегия генов-паралогов зависит от способа дупликации (WGD или SD) и типа амплифицируемых фрагментов ДНК (Freeling, Thomas, 2006). При полногеномных дупликациях ДНК копируется в виде длинных копий – онологов, объединяющих множественные группы генов. В таком виде сохраняются гены, вовлеченные в процессы регуляции онтогенеза, оогенеза, клеточного цикла, сигнальных каскадов и другие. В составе онологов гены часто объединены в так называемые “функциональные модули”. Кодируемые генами таких модулей белки, как правило, организованы в надмолекулярные функциональные комплексы. Считается, что дупликация таких модулей приводит к скачку приспособленности организмов. В то же время, при сегментных дупликациях отдельные гены, не объединенные в функциональные модули, сохраняются в виде коротких копий ДНК. Такой способ амплификации не приводит к скачку приспособленности, но вносит определенный вклад в модуляцию биологических процессов. В виде коротких копий сохраняются гены, отвечающие за консервативные функции, например за метаболизм ДНК, активность нуклеаз и др. (Bowers et al., 2003; Papp et al., 2003; Yang et al., 2003; Maere et al., 2005; Freeling, Thomas, 2006). Представления о роли баланса генов в составе длинных и коротких копий ДНК основаны на том, что баланс соблюдается в онологах, в то время как локальные дупликации, например тандемные, из-за риска несбалансированности предпочтительно охватывают гены, кодирующие мономерные белки или гены, слабо связанные на хромосоме (Freeling, Thomas, 2006).
Возвращаясь к альбуминам лососеобразных (щуковых и лососевых рыб), отметим, что информация о наличии их в составе функциональных модулей отсутствует. Однако, оказалось, что они стабильно присутствуют в составе двух синтеничных групп из пятии-шести генов. Одна такая “щучья” группа генов на chr 5 полностью воспроизводится на одной из хромосом лосося атлантического, кумжи и микижи. Другая “щучья” группа генов на chr 13 полностью воспроизводится на chr 18 озерного гольца, и в виде инвертированной последовательности генов – на хромосоме ssa15 лосося; при этом, у лосося один из пяти генов щуки заменяется на другой – “теряется”. Инверсии отдельных или нескольких генов из этих двух синтеничных групп щуки, замены одних генов на другие – эти события имеют место и у других лососеобразных. Ни полиплоидизация, ни характерные для лососеобразных интенсивные хромосомные перестройки, которые обнаружены и у предков костистых (Braash et al., 2016), не “разорвали” эти группы, а только перестроили их.
Сравнительный анализ геномов костистых рыб, панцирной щуки, слоновой акулы Callorhinchus milii и других челюстноротых показал, что для костистых характерны короткие консервативные синтеничные группы или блоки (Ravi, Venkatesh, 2018). Для одиночных генов, кодирующих мономерные белки, не организованных в кластеры в виде тандемных повторов и не входящих в состав функциональных модулей, установлена еще одна закономерность – такие гены теряются чаще, чем связанные гены в составе функциональных модулей онологов (Freeling, Thomas, 2006). Не исключено, что подобный сценарий потери гена альбумина мог реализоваться в группах Ostariophysi и Acanthopterygii.
Благодаря WGD, костистые рыбы совершили глобальный скачок в эволюционном развитии, в то время как в процессе сегментных дупликаций и дупликаций отдельных генов может достигаться более “тонкая” подгонка механизмов, направленных на поддержание сбалансированной работы удвоенного генома и, в конечном счете, на гомеостаз физиологических функций.
Проблема компенсации функции альбумина в группах Acanthopterygii и Ostariophysi
Учитывая роль сывороточного альбумина как одного из ключевых факторов физиологического гомеостаза у высших позвоночных, его потеря у представителей низших позвоночных могла бы привести к неблагоприятным последствиям. Однако, Acanthopterygii и Ostariophysi относятся к наиболее массовым и эволюционно продвинутым группам позвоночных, а это, в свою очередь, позволяет предположить, что осмотическую и транспортную функции альбумина у них взяли на себя какие-то другие белки плазмы.
Анализ белкового состава плазмы крови костистых рыб, включая Acanthopterygii и Ostariophysi, выявил широкий спектр белков с высоким электроотрицательным потенциалом, способных связывать неорганические катионы и диполи воды и проявляющих таким образом осмотическую активность. Оказалось, что в плазме крови альбумин-содержащих и “безальбуминовых” костистых рыб присутствует не один, а несколько белков в составе осмотически активной белковой фракции: 1) альбумин; 2) белок тепловой акклимации (warm temperature acclimated 65 kDa protein), проявляющий свойства гемопексина; 3) ингибиторы сериновых и цистеиновых протеиназ; 4) аполипопротеины АроА-I и Аро-14 в составе липопротеинов высокой плотности; и 5) множественная дополнительная анодная фракция из низкомолекулярных белков с высокоотрицательным поверхностным потенциалом (Andreeva, 2019, 2020, 2021). В составе последней имеются не только “истинные” белки плазмы – внеклетоточные белки, выполняющие в системе кровообращения специфические функции, но и так называемые “транзитные” белки, чей пул постоянно пополняют внутриклеточные белки, попадающие в кровь вследствие разрушения клеток (Andreeva, 2021).
Доля осмотически активных белков плазмы костистых рыб из вышеперечисленных групп достигает ~50–60% тотального белка, что сопоставимо с содержанием альбумина в плазме млекопитающих. Это предполагает сопоставимый с Mammalia вклад этих белков в коллоидно-осмотическое давление плазмы у “безальбуминовых” костистых рыб. Не пострадала у Acanthopterygii и Ostariophysi и транспортная функция плазмы крови. Так, сравнение функций транспорта жирных кислот между альбуминами и липопротеинами высокой плотности позволяет предположить их более эффективное воплощение с участием липопротеиновых частиц (Andreeva, 2019).
Причины эволюционного успеха полиплоидов на примере Teleostei или почему у костистых рыб потеря альбумина прошла “незамеченной”
Главные изменения, возникшие у Teleostei по сравнению с более древними лучеперыми рыбами, и, по-видимому определившие их эволюционный успех, затронули механизмы локомоции, питания и плодовитости (Romer, Parsons, 1986). Teleostei отличаются от других позвоночных высочайшим разнообразием формы тела, образа жизни, типа питания и высокой плодовитостью. Поскольку геномы представителей костистых рыб прошли через дополнительный раунд WGD и таксонспецифичные события SaGD, CyGD и др., прогрессивные черты этой группы в значительной степени связывают с событиями дупликации геномов. Среди дуплицированных генов особое место занимают HOX-гены. После отделения Acipenseriformes и Semionotiformes, у предков костистых произошла дупликация четырех кластеров НОХ-генов. Полагают, что именно это событие отразилось на формообразовательных процессах, морфологии организма и послужило причиной радиации предковой группы, давшей начало новому таксону (Озернюк, Мюге, 2013; Soshnikova et al., 2013).
Другая группа генов, чьи дупликации имели важные последствия для эволюции Teleostei, – гены, кодирующие факторы транскрипции. Они представлены разными мультигенными семействами – Sox, Bmp, Gtf3a, Hif, HNF1 и др., вовлеченными в регуляцию экспрессии сотен других генов, сигнальные каскады, контроль процессов эмбриогенеза, онтогенеза, оогенеза, клеточного цикла и др. (Озернюк, Мюге, 2013; Pelster, Egg, 2018; Rojo-Bartolome et al., 2020). Эволюционному успеху Teleostei, несомненно, способствовали и гены, кодирующие белки промежуточного обмена. Прежде всего, это белки, участвующие в липидном обмене, составляющем основу энергетики рыб. Особая роль отводится транспортным белкам, отвечающим за обеспечение тканевых обменных процессов всеми требуемыми лигандами, включая альбуминоиды (Sharma et al., 2006).
Для выживания полиплоидных организмов важно состояние кластеров генов рРНК, поскольку экспрессия дуплицированных генов лимитируется белок-синтезирующей способностью рибосом, зависящей от эффективности пополнения пулов различных фракций рРНК. При сравнении экспрессии кластеров этих генов у аллотетраплоидного (4n = 200) и диплоидного (2n = 100) серебряного карася Carassius auratus четкой корреляции не выявлено, хотя замечены различия в экспрессии 5S и 45S рРНК (Zhao et al., 2021). На примере Danio rerio и полиплоидных окунеобразных показано влияние дупликационных событий и хромосомных перестроек на перераспределение, накопление и гомеостаз рРНК, обеспечивающих необходимый уровень экспрессии востребованных дуплицированных генов (Cioffi et al., 2015; Rojo-Bartolome et al., 2020). В отличие от костистых рыб и других эукариот, в семействе Acipenseridae на примере девяти видов североамериканских осетровых показана множественность аллелей гена 18S рРНК, связанная, предположительно, с полиплоидизацией этой группы (Krieger et al., 2000, 2006; Krieger, Fuerst, 2002). Существенная индивидуальная вариация гена 18S рРНК отсутствует у большинства других видов, для которых установлены его последовательности. Существует предположение, что высокий полиморфизм этих последовательностей у осетровых может быть связан с низкой скоростью согласованной эволюции в этой группе, замедлившей потерю любой возникшей полиморфной вариации. Данное обстоятельство позволяет объяснить отсутствие подобной вариабельности у костистых рыб быстрыми темпами эволюционных преобразований в этой группе (Ravi, Venkatesh, 2018).
ЗАКЛЮЧЕНИЕ
Проведенный поиск предполагает различия в сценариях эволюции генов альбумина у бесчелюстных и челюстноротых позвоночных. В линии миног эволюция альбумина проходила под влиянием одного или двух раундов WGD и независимых событий гексаплоидизации и сегментных дупликаций. Взяв начало не от трехдоменного предшественника, а от более коротких одно- или двухдоменных белков, альбумины миног достигли двукратного превосходства по длине аминокислотной последовательности над альбуминами челюстноротых. Два гена-паралога, разделившие между собой функцию транспорта лигандов у молоди и половозрелых особей миног, очень напоминают паралогов – альбумин и альфа-фетопротеин – у амниот, разделивших транспортную функцию исходного гена между эмбриональной и постнатальной стадиями. Не затронутые третьей полногеномной дупликаций челюстноротые рыбы (латимерия и панцирная пятнистая щука) имеют, как и млекопитающие, по одному гену альбумина. В то же время у стерляди, в эволюции которой, помимо первых раундов WGD, присутствовала специфичная для Aciprnseriformes полногеномная дупликация, обнаружено два гена-паралога альбумина. В группе костистых рыб, третий раунд WGD и таксонспецифичные полногеномные дупликации привели, с одной стороны, к появлению генов-паралогов альбумина у низших Teleostei, с другой стороны, к потере альбумина в наиболее массовых и эволюционно продвинутых группах Acanthopterygii и Ostariophysi, в результате у них произошло “сужение” репертуара альбуминоидов до одного белка – DBP.
Наличие секвенированных геномов при отсутствии аннотаций или слабой поддержке в виде аннотирования последовательностей некоторых анализируемых видов не позволяют считать результаты поиска окончательными. Однако, обширные литературные данные по идентификации белков плазмы и ее тканевых фильтратов у модельных видов Acanthopterygii и Ostariophysi убеждают в отсутствии альбуминов в составе протеомов плазмы (Dietrich et al., 2014, 2021; Vilchez et al., 2016; Banerjee et al., 2017; Schrama et al., 2017 и др.). Не обнаружены альбумины и у ряда немодельных объектов, демонстрирующих высокий уровень идентичности с белками модельных Acanthopterygii и Ostariophysi (Andreeva et al., 2015, 2017, 2019). Эти факты позволяет считать утрату гена альбумина событием, выходящим за рамки одного семейства карповых рыб.
Консервативность организации полипептидных цепей альбумина во всех группах челюстноротых – сохранение их длины, числа доменов и S‒S-связей, и пропорциональное сохранение этих особенностей при увеличении длины альбумина у бесчелюстных миног, – вероятно, объясняется тем, что организованный таким образом белок успешно выполняет возложенные на него функции транспорта и осмотической активности. Все эти консервативные черты поддерживаются отбором при относительно низком сходстве аминокислотных последовательностей на межотрядном уровне, редко превышающем 40%. При этом поддержка консервативной организации полипептидной цепи достигается высоким оборотом интронов. Нами не отмечено ни одного примера совпадения длин соответствующих интронов в генах-паралогах и ортологах альбумина. Динамичный оборот интронов демонстрируют паралоги альбумина у стерляди и у низших костистых рыб – у них не выявлено совпадений длин соответствующих интронов и повторов ДНК. Между тем, для генов млекопитающих с консервативными белок-кодирующими областями отмечен дефицит приобретения и оборота интронов (Roy et al., 2003). Учитывая, что интроны включают в себя и регуляторные элементы, их высокий оборот в генах альбумина костистых рыб, вероятно, способствовал процессам их суб- и неофункционализации.
Другой эволюционный сценарий преобразований альбумина у костистых рыб – в виде его утраты вследствие WGD – не помешал таксону Teleostei в освоении и завоевании новых экологических ниш. На фоне динамичных преобразований геномов, высокого уровня внутри- и межхромосомных перестроек, повышенной скорости эволюции белок-кодирующих последовательностей, а также более высокой по сравнению с другими позвоночными скорости оборота интронов и регуляторных элементов (Ravi, Venkatesh, 2018) – утрата альбумина у Teleostei прошла “незамеченной”, а функцию альбумина взяли на себя другие осмотически активные белки.
Список литературы
Байрамов А.В., Ермакова Г.В., Кучерявый А.В., Зарайский А.Г. 2021. Геномные дупликации как основа эволюционного успеха позвоночных // Онтогенез. Т. 52(3). С. 170.https://doi.org/10.31857/S0475145020030052
Васильев В.П. 1977. О полиплоидии у рыб и некоторые вопросы эволюции кариотипов лососевых (Salmonidae) // Журн. общ. биологии. Т. 38. С. 380.
Васильев В.П. 1985. Эволюционная кариология рыб. Москва: Наука.
Животовский Л.А. 2015. Эволюционная история тихоокеанских лососей и форелей // Тр. ВНИРО. Т. 157. С. 4.
Каменская Д.Н., Брыков В.А. 2020. Гены гормона роста у рыб: структура и дивергенция // Биология моря. Т. 46(4). С. 219. https://doi.org/10.31857/S0134347520040038
Озернюк Н.Д., Мюге Н.С. 2013. Крупномасштабные дупликации генов и дивергенция паралогичных генов на примере рыб // Генетика. Т. 49(1). С. 73.https://doi.org/10.7868/S0016675813010086
Решетников Ю.С. 1998. Аннотированный каталог круглоротых и рыб континентальных вод России. Москва: Наука.
Alexandrou M., Swartz B., Matzke N., Oakley T. 2013. Genome duplication and multiple evolutionary origins of complex migratory behavior in Salmonidae // Mol. Phylogenet. Evol. V. 69(3). P. 514.https://doi.org/10.1016/j.ympev.2013.07.026
Andreeva A.M., Lamash N.E., Serebryakova M.V. et al. 2015. Reorganization of low-molecular-weight fraction of plasma proteins in the annual cycle of Cyprinidae // Biochemistry (Moscow). V. 80(2). P. 208. https://doi.org/10.1134/S0006297915020078
Andreeva A.M., Serebryakova M.V., Lamash N.E. 2017. Oligomeric protein complexes of apolipoproteins stabilize the internal fluid environment of organism in redfins of the Tribolodon genus [Pisces; Cypriniformes, Cyprinidae] // Comp. Biochem. Physiol, Part D: Genomics Proteomics. V. 22. P. 90. https://doi.org/10.1016/j.cbd.2017.02.007
Andreeva A.M. 2019. The Strategies of Organization of the Fish Plasma Proteome: with and without Albumin // Russian Journal of Marine Biology. V. 45(4). P. 263.https://doi.org/10.1134/S1063074019040023
Andreeva A.M., Vasiliev A.S., Toropygin I.Y. et al. 2019. Involvement of apolipoprotein A in maintaining tissue fluid balance in goldfish Carassius auratus // Fish Physiol. Biochem. V. 45(5). P. 1717. https://doi.org/10.1007/s10695-019-00662-1
Andreeva A.M. 2020. Structural Organization of Plasma Proteins as a Factor of Capillary Filtration in Pisces // Inland Water Biology. V. 13. № 4. P. 664. https://doi.org/10.1134/S1995082920060036
Andreeva A.M. 2021. Organization and Function of Osmotically Active Fraction of Fish (Pisces) Plasma Proteome // Inland Water Biology. V. 14. № 4. P. 449. https://doi.org/10.1134/S1995082921040039
Anguizola J., Matsuda R., Barnaby O.S. et al. 2013. Review: Glycation of human serum albumin // Clin Chim Acta. V. 425. P. 64. https://doi.org/10.1016/j.cca.2013.07.013
Ascenzi P., di Masi A., Leboffe L. et al. 2013. Molecular phylogenetic analyses of albuminoids reveal the molecular evolution of allosteric properties // IUBMB Life. V. 65(6). P. 544. https://doi.org/10.1002/iub.1164
Asrar Z., Haq F., Abbasi A.A. 2013. Fourfold paralogy regions on human HOX-bearing chromosomes: role of ancient segmental duplications in the evolution of vertebrate genome // Mol. Phylogenet Evol. V. 66(3). P. 737. https://doi.org/10.1016/j.ympev.2012.10.024
Banerjee S., Mahanty A., Mohanty S. et al. 2017. Identification of potential biomarkers of hepatotoxicity by plasma proteome analysis of arsenic-exposed carp Labeo rohita // J. Hazard Mater. V. 336. P. 71. https://doi.org/10.1016/j.jhazmat.2017.04.054
Berthelot C., Brunet F., Chalopin D. et al. 2014. The rainbow trout genome provides novel insights into evolution after whole-genome duplication in vertebrates // Nat. Commun. V. 5. P. 3657. https://doi.org/10.1038/ncomms4657
Bouillon R., Schuit F., Antonio L. Rastinejad F. 2020. Vitamin D Binding Protein: A Historic Overview // Front Endocrinol (Lausanne). V. 10. P. 910. https://doi.org/10.3389/fendo.2019.00910
Bowers J.E., Chapman B.A., Ron J., Paterson A.H. 2003. Unravelling angiosperm genome evolution by phylogenetic analysis of chromosomal duplication events // Nature. V. 422. P. 433. https://doi.org/10.1038/nature01521
Braasch I., Postlethwait J.H. 2012. Polyploidy in Fish and the Teleost Genome Duplication // Polyploidy and Genome Evolution. P. 341. Berlin: Springer. https://doi.org/10.1007/978-3-642-31442-1_17
Braasch I., Gehrke A.R., Smith, J.J. et al. 2016. The spotted gar genome illuminates vertebrate evolution and facilitates human-teleost comparisons // Nat. Genet. V. 48(4). P. 427. https://doi.org/10.1038/ng.3526
Braasch I., Guiguen Y., Loker R. et al. 2014. Connectivity of vertebrate genomes: Paired-related homeobox (Prrx) genes in spotted gar, basal teleosts, and tetrapods // Comp. Biochem. Physiol. C Toxicol. Pharmacol. V. 163. P. 24. https://doi.org/10.1016/j.cbpc.2014.01.005
Brown J.D. 1976. Structural origins of mammalian albumin // Fed. Proc. Gene. V. 20. P. 415.
Brunet F.G., Crollius H., Paris M. et al. 2006. Gene loss and evolutionary rates following whole-genome duplication in teleost fishes // Mol. Biol. Evol. V. 23(9). P. 1808. https://doi.org/10.1093/molbev/msl049
Byrnes L., Gannon F. 1990. Atlantic salmon (Salmo salar) serum albumin: cDNA sequence, evolution, and tissue expression // DNA Cell Biol. V. 9(9). P. 647. https://doi.org/10.1089/dna.1990.9.647
Carninci P., Sandelin A., Lenhard B. et al. 2006. Genome-wide analysis of mammalian promoter architecture and evolution // Nat Genet. V. 38(6). P. 626. https://doi.org/10.1038/ng1789
Cheng P., Huang Y., Lv Y. et al. 2021. The American Paddlefish Genome Provides Novel Insights into Chromosomal Evolution and Bone Mineralization in Early Vertebrates // Mol. Biol. Evol. V. 38(4). P. 1595. https://doi.org/10.1093/molbev/msaa326
Cioffi M., Bertollo L.A., Villa M.A. et al. 2015. Genomic Organization of Repetitive DNA Elements and Its Implications for the Chromosomal Evolution of Channid Fishes (Actinopterygii, Perciformes) // PLoS One. V. 10(6). e0130199. https://doi.org/10.1371/journal.pone.0130199
Crow K.D., Smith C.D., Cheng J.F. et al. 2012. An independent genome duplication inferred from HOX paralogs in the American paddlefish a representative basal ray-finned fish and important comparative reference // Genome Biol Evol. V. 4(9). P. 937. https://doi.org/10.1093/gbe/evs067
Curry S., Mandelkow H., Brick P., Franks N. 1998. Crystal structure of human serum albumin complexed with fatty acid reveals an asymmetric distribution of binding sites // Nat. Struct. Biol. V. 5(9). P. 827. https://doi.org/10.1038/1869
Deryckere F., Byrnes L., Wagner A. et al. 1995. Salmon HNF1: cDNA sequence, evolution, tissue specificity and binding to the salmon serum albumin promoter // J. Mol. Biol. V. 247(1). P. 1. https://doi.org/10.1006/jmbi.1994.0115
Dietrich M.A., Arnold G.J., Nynca J. et al. 2014. Characterization of carp seminal plasma proteome in relation to blood plasma // J. Proteomics. V. 98. P. 218. https://doi.org/10.1016/j.jprot.2014.01.005
Dietrich M.A., Irnazarow I., Adamek M. et al. 2021. 2D-DIGE proteomic analysis of blood plasma reveals changes in immune- and stress-associated proteins following hormonal stimulation of carp males // Fish Shellfish Immunol. V. 118. P. 354. https://doi.org/10.1016/j.fsi.2021.09.018
Du K., Stöck M., Kneitz S. et al. 2020. The sterlet sturgeon genome sequence and the mechanisms of segmental rediploidization // Nat. Ecol. Evol. V. 4(6). P. 841–852. https://doi.org/10.1038/s41559-020-1166-x
Dziegielewska K.M., Evans C.A., Fossan G. et al. 1980. Proteins in cerebrospinal fluid and plasma of fetal sheep during development // J Physiol. V. 300. P. 441. https://doi.org/10.1113/jphysiol.1980.sp013171
Eiferman F.A., Young P.R., Scott R.W., Tilghman S.M. 1981. Intragenic amplification and divergence in the mouse alpha-fetoprotein gene // Nature. V. 294(5843). P. 713. https://doi.org/10.1038/294713a0
Evans B., Upham N., Golding G. et al. 2017. Evolution of the Largest Mammalian Genome // Genome Biol. Evol. V. 9(6). P. 1711. https://doi.org/10.1093/gbe/evx113
Fanali G., Ascenzi P., Bernardi G., Fasano M.J. 2012. Sequence analysis of serum albumins reveals the molecular evolution of ligand recognition properties // Biomol. Struct. Dyn. V. 29(6). P. 691. https://doi.org/10.1080/07391102.2011.672632
Fasano M., Fanali G., Leboffe L., Ascenzi P. 2007. Heme binding to albuminoid proteins is the result of recent evolution // IUBMB Life. V. 59(7). P. 436. https://doi.org/10.1080/15216540701474523
Felip A., Carrillo M., Zanuy S. 2009. Older triploid fish retain impaired reproductive endocrinology in the European sea bass Dicentrarchus labrax // J. Fish Biol. V. 75(10). P. 2657. https://doi.org/10.1111/j.1095-8649.2009.02458.x
Filosa M.F., Adam I., Robson P. et al. 1998. Partial clone of the gene for AS protein of the lamprey Petromyzon marinus, a member of the albumin supergene family whose expression is restricted to the larval and metamorphic phases of the life cycle // J. Exp. Zool. V. 282(3). P. 301.
https://doi.org/10.1002/(sici)1097-010x(19981015)282:3<301::aid-jez3>3.0.co;2-q
Force A., Lynch M., Pickett F.B. et al. 1999. Preservation of duplicate genes by complementary, degenerative mutations // Genetics. V. 151. P. 1531. https://doi.org/10.1093/genetics/151.4.1531
Freeling M., Thomas B.C. 2006. Gene-balanced duplications, like tetraploidy, provide predictable drive to increase morphological complexity // Genome Res. V. 16(7). P. 805. https://doi.org/10.1101/gr.3681406
Ghuman J., Zunszain P.A., Petitpas I. et al. 2005. Structural basis of the drug-binding specificity of human serum albumin // J. Mol. Biol. V. 353(1). P. 38. https://doi.org/10.1016/j.jmb.2005.07.075
Gibbs P.E., Dugaiczyk A. 1987. Origin of structural domains of the serum-albumin gene family and a predicted structure of the gene for vitamin D-binding protein // Mol. Biol. Evol. V. 4(4). P. 364. https://doi.org/10.1093/oxfordjournals.molbev.a040447
Gorin M.B., Cooper D.L., Eiferman F. et al. 1981. The evolution of alpha-fetoprotein and albumin, I. A comparison of the primary amino acid sequences of mammalian alpha-fetoprotein and albumin // J. Biol. Chem. V. 256(4). P. 1954.
Gray J.E., Doolittle R.F. 1992. Characterization, primary structure, and evolution of lamprey plasma albumin // Protein Science. V. 1(2). P. 289. https://doi.org/10.1002/pro.5560010211
Gu L., Xia C. 2019. Cluster expansion of apolipoprotein D (ApoD) genes in teleost fishes // BMC Evolutionary Biology. V. 19(9). P. 2.
Gundappa M.K., To T.H., Grønvold L. et al. 2021. Genome-Wide Reconstruction of Rediploidization Following Autopolyploidization across One Hundred Million Years of Salmonid Evolution // Mol. Biol. Evol. V. 39(1). https://doi.org/10.1093/molbev/msab310
Hoegg S., Brinkmann H., Naylor J.S. et al. 2004. Phylogenetic timing of the fish specific duplication correlates with the diversification of teleost fish // J. Mol. Evol. V. 59. P. 190.
Holland P.W., Garcia-Fernàndez J., Williams N.A., Sidow A. 1994. Gene duplications and the origins of vertebrate development // Dev Suppl. P. 125.
Jagodzinski L.L., Sargent T.D., Yang M. et al. 1981. Sequence homology between RNAs encoding rat alpha-fetoprotein and rat serum albumin // Proc. Natl. Acad. Sci. USA. V.78(6). P. 3521. https://doi.org/10.1073/pnas.78.6.3521
Jerkovic L., Voegele A.F., Chwatal S. et al. 2005. Afamin is a novel human vitamin E-binding glycoprotein characterization and in vitro expression // J. Proteome Res. V. 4(3). P. 889. https://doi.org/10.1021/pr0500105
Kajiyama Y., Tian J., Locker J. 2006. Characterization of distant enhancers and promoters in the albumin-alpha-fetoprotein locus during active and silenced expression // J. Biol. Chem. V. 281(40). P. 30 122. https://doi.org/10.1074/jbc.M603491200
Krieger J., Fuerst P.A. 2002. Evidence of multiple alleles of the nuclear 18S ribosomal RNA gene in sturgeon (Family: Acipenseridae) // J. Appl. Ichthyol. V. 18. P. 290. https://doi.org/10.1046/J,1439-0426.2002.00377.X
Krieger J., Fuerst P.A., Cavender T.M. 2000. Phylogenetic relationships of the North American sturgeons (order Acipenseriformes) based on mitochondrial DNA sequences // Mol. Phylogenet. Evol. V. 16(1). P. 64. https://doi.org/10.1006/mpev.1999.0743
Krieger J., Hett A.K., Fuerst P.A. et al. 2006. Unusual intraindividual variation of the nuclear 18S rRNA gene is widespread within the Acipenseridae // J. Hered. V. 97(3). P. 218. https://doi.org/10.1093/jhered/esj035
Kuraku S., Meyer A., Kuratani S. 2009a. Timing of genome duplications relative to the origin of the vertebrates: did cyclostomes diverge before or after? // Mol. Biol. Evol. V. 26(1). P. 47. https://doi.org/10.1093/molbev/msn222
Kuraku S., Meyer A. 2009b. The evolution and maintenance of HOX gene clusters in vertebrates and the teleost-specific genome duplication // Int. J. Dev. Biol. V. 53(5–6). P. 765. https://doi.org/10.1387/ijdb.072533km
Li S., Cao Y., Geng F. 2017. Genome-wide identification and comparative analysis of albumin family in vertebrates // Evol. Bioinf. Online. V. 13. P. 1. https://doi.org/10.1177/1176934317716089
Ma W., Zhu Z., Bi X. et al. 2014. Allopolyploidization is not so simple: evidence from the origin of the tribe Cyprinini (Teleostei: Cypriniformes) // Curr. Mol. Med. V. 14(10). P. 1331. https://doi.org/10.2174/1566524014666141203101543
Makhrov A.A. 2017. A narrowing of the phenotypic diversity range after large rearrangements of the karyotype in Salmonidae: The relationship between saltational genome rearrangements and gradual adaptive evolution // Genes. V. 8. P. 297. https://doi.org/10.3390/genes8110297
Maere S., De Bodt S., Raes J. et al. 2005. Modeling gene and genome duplications in eukaryotes // Proc Natl Acad Sci. V. 102. P. 5454. https://doi.org/10.1073/pnas.0501102102
Majorek K.A., Porebski P.J., Dayal A., et al. 2012. Structural and immunologic characterization of bovine, horse, and rabbit serum albumins // Mol. Immunol. V. 52(3–4). P. 174. https://doi.org/. Epub 2012 Jun 6https://doi.org/10.1016/j.molimm.2012.05.011
Malik S., Fu L., Juras D.J. et al. 2013. Common variants of the vitamin D binding protein gene and adverse health outcomes // Crit. Rev. Clin. Lab. Sci. V. 50(1). P. 1. https://doi.org/10.3109/10408363.2012.750262
Metcalf V., Brennan S., Chambers G., George P. 1998a. The albumins of Chinook salmon (Oncorhynchus tshawytscha) and brown trout (Salmo trutta) appear to lack a propeptide // Arch Biochem Biophys. V. 350(2). P. 239. https://doi.org/10.1006/abbi.1997.0509
Metcalf V.J., Brennan S.O., Chambers G.K., George P.M. 1998b. The albumin of the brown trout (Salmo trutta) is a glycoprotein // Biochim. Biophys. Acta. V. 1386(1). P. 90.
Minghetti P.P., Law S.W., Dugaiczyk A. 1985. The rate of molecular evolution of alpha-fetoprotein approaches that of pseudogenes // Mol. Biol. Evol. V. 2(4). P. 347. https://doi.org/10.1093/oxfordjournals.molbev.a040350
Morinaga T., Sakai M., Wegmann T.G., Tamaoki T. 1983. Primary structures of human alpha-fetoprotein and its mRNA // Proc. Natl. Acad. Sci USA. V. 80(15). P. 4604. https://doi.org/10.1073/pnas.80.15.4604
Nakatani Y., Shingate P., Ravi V. et al. 2021. Publisher Correction: Reconstruction of proto-vertebrate, protocyclostome and proto-gnathostome genomes provides new insights into early vertebrate evolution // Nat. Commun. V. 12(1). P. 4704. https://doi.org/10.1038/s41467-021-25110-8
Nishio H., Dugaiczyk A. 1996. Complete structure of the human alpha-albumin gene, a new member of the serum albumin multigene family // Proc. Natl. Acad. Sci. USA. V. 93(15). P. 7557. https://doi.org/10.1073/pnas.93.15.7557
Nishio H., Heiskanen M., Palotie A. et al. 1996. Tandem arrangement of the human serum albumin multigene family in the sub-centromeric region of 4q: Evolution and chromosomal direction of transcription // J. Mol. Biol. V. 259. P. 113. https://doi.org/10.1006/jmbi.1996.0306
Noel E.S., Reis M., Arai Z., Ober E.A. 2010. Analysis of the Albumin/α-Fetoprotein/Afamin/Group specific component gene family in the context of zebrafish liver differentiation // Gene Expression Patterns. V. 10(6). P. 237. https://doi.org/10.1016/j.gep.2010.05.002
Nynca J., Arnold G., Fröhlich T., Ciereszko A. 2017. Proteomic identification of rainbow trout blood plasma proteins and their relationship to seminal plasma proteins // Proteomics. V. 17(11). P. 1. https://doi.org/10.1002/pmic.201600460
Ohno S. 1970. Evolution by Gene Duplication. Berlin; Heidelberg; New York: Springer-Verlag.
Ohno S. 1981. Original domain for the serum albumin family arose from repeated sequences // Proc. Natl. Acad. Sci. USA. V. 78(12). P. 7657. https://doi.org/10.1073/pnas.78.12.7657
Otterbein L.R., Cosio C., Graceffa P., Dominguez R. 2002. Crystal structures of the vitamin D-binding protein and its complex with actin: structural basis of the actin-scavenger system // Proc. Natl. Acad. Sci. USA. V. 99(12). P. 8003. https://doi.org/10.1073/pnas.122126299
Pelster B., Egg M. 2018. Hypoxia-inducible transcription factors in fish: expression, function and interconnection with the circadian clock // J. Exp. Biol. V. 221(13). jeb163709. https://doi.org/10.1242/jeb.163709
Papp B., Pal C., Hurst L.D. 2003. Dosage sensitivity and the evolution of gene families in yeast // Nature. V. 424. P. 194. https://doi.org/10.1038/nature01771
Pasquier J., Cabau C., Nguyen T. et al. 2016. Gene evolution and gene expression after whole genome duplication in fish: the PhyloFish database // BMC Genomics. V. 17(368). P. 1. https://doi.org/10.1186/s12864-016-2709-z
Postlethwait J.H., Woods I.G., Ngo-Hazelet P. et al. 2000. Zebrafish comparative genomics and the origins of vertebrate chromosomes // Genome Res. V. 10(12). P. 1890. https://doi.org/10.1101/gr.164800
Ramberg S., Hoyheim B., Ostbye T., Andreassen R. 2021. A de novo full- length mRNA transcriptome generated from hybrid- corrected PacBio long- reads improves the transcript annotation and identifies thousands of novel splice variants in Atlantic Salmon // Frontiers in Genetics. V. 12. P. 1‒22. https://doi.org/10.3389/fgene.2021.656334
Ravi V., Venkatesh B. 2018. The Divergent Genomes of Teleosts // Annu Rev. Anim. Biosci. V. 6. P. 47. https://doi.org/10.1146/annurev-animal-030117-014821
Rojo-Bartolomé I., de Souza J., de Cerio O., Cancio I. 2020. Duplication and subfunctionalisation of the general transcription factor IIIA (gtf3a) gene in teleost genomes, with ovarian specific transcription of gtf3ab // PLoS One. V. 15(1). e0227690. https://doi.org/10.1371/journal.pone.0227690
Romer A.S., Parsons T.S. 1986. The Vertebrate Body. Philadelphia: Saunders College Publishing.
Rondeau E.B., Minkley D.R., Leong J.S. et al. 2014. The genome and linkage map of the northern pike (Esox lucius): conserved synteny revealed between the salmonid sister group and the Neoteleostei // PLoS One. V. 9(7). https://doi.org/10.1371/journal.pone.0102089
Roy S.W., Fedorov A., Gilbert W. 2003. Large- scale comparison of intron positions in mammalian genes shows intron loss but no gain // PNAS USA. V. 100. P. 7158. https://doi.org/10.1073/pnas.1232297100
Saber M.A., Stockbauer P., Moravek L., Meloun B. 1977. Disulfide bonds in human serum albumin // Collect Czech Chem Commun. V. 42. P. 564. https://doi.org/10.1135/cccc19770564
Sargent T.D., Jagodzinski L.L., Yang M., Bonner J. 1981. Fine structure and evolution of the rat serum albumin gene // Mol. Cell. Biol. V. 1(10). P. 871. https://doi.org/10.1128/mcb.1.10.871-883
Schrama D., Richard N., Silva T.S. et al. 2017. Enhanced dietary formulation to mitigate winter thermal stress in gilthead sea bream (Sparus aurata): a 2D-DIGE plasma proteome study // Fish Physiol. Biochem. V. 43(2). P. 603. https://doi.org/10.1007/s10695-016-0315-2
Sharma M.K., Liu R.Z., Thisse C. et al. 2006. Hierarchical subfunctionalization of fabp1a, fabp1b and fabp10 tissue-specific expression may account for retention of these duplicated genes in the zebrafish (Danio rerio) genome // FEBS J. V. 273(14). P. 3216. https://doi.org/10.1111/j.1742-4658.2006.05330.x
Smith J.J., Keinath M.C. 2015. The sea lamprey meiotic map improves resolution of ancient vertebrate genome duplications // Genome Res. V. 25(8). P. 1081. https://doi.org/10.1101/gr.184135.114
Soshnikova N., Dewaele R., Janvier P. et al. 2013. Duplications of HOX gene clusters and the emergence of vertebrates // Dev. Biol. V. 378(2). P. 194. https://doi.org/10.1016/j.ydbio.2013.03.004
Tamura K., Stecher G., Peterson D., Filipski A., Kumar S. 2013. MEGA6: Molecular Evolutionary Genetics Analysis version 6.0 // Mol. Biol. Evol. V. 30. P. 2725. https://doi.org/10.1093/molbev/mst197
Taylor J.S., Van de Peer Y., Braasch I., Meyer A. 2001. Comparative genomics provides evidence for an ancient genome duplication event in fish // Philos. Trans. R. Soc. Lond. B. Biol. Sci. .V. 356(1414). P. 1661. https://doi.org/10.1098/rstb.2001.0975
Uno Y., Nishida C., Takaqi C. et al. 2013. Homoeologous chromosomes of Xenopus laevis are highly conserved after whole-genome duplication // Heredity (Edinb). V. 111(5). P. 430. https://doi.org/10.1038/hdy.2013.65
Van de Peer Y., Mizrachi E., Marchal K. 2017. The evolutionary significance of polyploidy // Nat. Rev. Genet. V. 18(7). P. 411. https://doi.org/10.1038/nrg.2017.26
Venkatachalam A.B., Parmar M.B., Wright J.M. 2017. Evolution of the duplicated intracellular lipid-binding protein genes of teleost fishes // Mol. Genet. Genomics. V. 292(4). P. 699. https://doi.org/10.1007/s00438-017-1313-5
Vílchez M.C., Pla D., Gallego V. et al. 2016. Identification of the major proteins present in the seminal plasma of European eel, and how hormonal treatment affects their evolution. Correlation with sperm quality // Comp. Biochem. Physiol. A Mol. Integr. Physiol. V. 201. P. 37. https://doi.org/10.1016/j.cbpa.2016.06.025
Xu Y., Ding Z. 2005. N-terminal sequence and main characteristics of Atlantic salmon (Salmo salar) albumin // Prep. Biochem. Biotechnol. V. 35(4). P. 283. https://doi.org/10.1080/10826060500218081
Yang J., Lusk R., Li W.-H. 2003. Organismal complexity, protein complexity, and gene duplicability // Proc. Natl. Acad. Sci. USA. V. 100. P. 15661. https://doi.org/10.1073/pnas.2536672100
Zhao C., Zhang Y., Qin H. et al. 2021. Organization and expression analysis of 5S and 45S ribosomal DNA clusters in autotetraploid Carassius auratus // BMC Ecol. Evol. V. 21(201). P. 2. https://doi.org/10.1186/s12862-021-01918-2
Дополнительные материалы отсутствуют.
Инструменты
Биология внутренних вод


