Ботанический журнал, 2022, T. 107, № 2, стр. 132-148
МОРФОЛОГИЧЕСКИЙ И МОЛЕКУЛЯРНО-ГЕНЕТИЧЕСКИЙ АНАЛИЗ РОДА VISCHERIA (EUSTIGMATACEAE, OCHROPHYTA) В АЛЬГОЛОГИЧЕСКОЙ КОЛЛЕКЦИИ ACSSI
А. Д. Темралеева 1, *, Е. А. Портная 1
1 Институт физико-химических и биологических проблем почвоведения Российской академии наук,
Федеральный исследовательский центр “Пущинский научный центр биологических исследований
Российской академии наук”
142290 Пущино, ул. Институтская, 2, Россия
* E-mail: temraleeva.anna@gmail.com
Поступила в редакцию 23.04.2021
После доработки 24.10.2021
Принята к публикации 16.11.2021
- EDN: SNCJGR
- DOI: 10.31857/S0006813622020119
Аннотация
Выполнен морфологический и молекулярно-генетический анализ четырех штаммов почвенных эустигматофитовых водорослей, выделенных из серых лесных почв Московской и Тульской областей России. По данным 18S рРНК и ITS2 филогении, изученные штаммы Альгологической коллекции ACSSI (Algal Collection of Soil Science Institute) являются представителями рода Vischeria. Морфологически они близки к V. magna, однако только один штамм ACSSI 026 с высокой статистической поддержкой кластеризовался с аутентичным штаммом SAG 2554. Остальные штаммы образовали отдельную независимую группу.
Систематика рода представляется проблемной из-за неразрешенности филогенетического дерева 18S рРНК и ITS2, неинформативности вариабельных регионов V4–V5 и V8–V9 гена 18S рРНК и безуспешности CBC-подхода, основанного на разделении близкородственных видов при наличии хотя бы одной CBC в консервативных регионах вторичной структуры ITS2. Предполагается, что увеличение количества штаммов эустигматофитовых водорослей, выделенных из различных биотопов, использование пластидных генов или глубокого секвенирования всего пластидного генома, изучение ультраструктурных, физиологических, биохимических характеристик позволит разработать концепцию вида у эустигматофитовых водорослей в целом и рода Vischeria в частности.
Пятьдесят лет назад Hibberd и Leedale (1971), исследовав 12 родов коккоидных желто-зеленых водорослей, обнаружили у них уникальные ультраструктурные и биохимические особенности, позволившие выделить их в новый класс водорослей – Eustigmatophyceae. В настоящее время эустигматофитовые водоросли представляют собой самостоятельную линию отдела Ochrophyta, состоящую из 102 таксономически признанных видов, принадлежащих 18 родам, восемь из которых являются монотипными (Guiry, Guiry, 2021). Это свидетельствует о недостаточной изученности таксономического разнообразия группы, большинство новых членов которой только предстоит описать. Полагают, что в настоящее время обнаружено только от 0.2 до 2% видов эустигматофитовых водорослей (Norton et al., 1996). Группа представлена, как правило, мелкими неподвижными одноклеточными, реже колониальными коккоидными водорослями различной формы (шаровидной, эллипсоидной, цилиндрической, яйцевидной, полиэдрической, звездчатой, веретеновидной или дисковидной), некоторые из них имеют ножку или разветвленные выступы. В клетке имеется обычно один желто-зеленый хлоропласт с одним или несколькими пиреноидами (реже без пиреноида). Типичными характеристиками является отсутствие хлорофилла с и присутствие виолаксантина. Воспроизведение происходит путем образования автоспор и зооспор с 1–2 передними различными по строению жгутиками (иногда присутствует задний голый жгутик). Прямых наблюдений полового процесса не описано, однако данные полногеномного секвенирования предполагают его присутствие у некоторых видов (Elias et al., 2017). Большинство эустигматофитовых живут в пресной воде, два рода морские (Nannochloropsis Hibberd, Microchloropsis Fawley, Jameson et Fawley), а некоторые представители встречаются в наземных экосистемах, в которых редко доминируют. Часть таксонов предпочитают обитать в условиях среды с pH менее 7. Большой интерес к эустигматофитовым водорослям со стороны биотехнологии обусловлен их способностью к синтезу липидов, в том числе полиненасыщенных жирных кислот (Pal et al., 2013; Gao et al., 2016, 2018; Wang et al., 2018), а также пигментов, стеролов, витаминов (Patterson et al., 1994; Durmaz, 2007; Li et al., 2012; Stoykova et al., 2019; Stoyneva-Gärtner et al., 2019; Martins et al., 2021). Оптимизируя лабораторные условия их культивирования, можно многократно увеличить продукцию нужных метаболитов (Cepák et al., 2014; Remias et al., 2020). Кроме того, описано эффективное применение штаммов эустигматофитовых водорослей для биоремедиации загрязненных сред (Fukuda et al., 2014; Upadhyay et al., 2016). Подобная востребованность группы говорит о необходимости поиска и открытия новых таксонов, инвентаризации и ревизии коллекционных штаммов, что требует их правильной идентификации.
Традиционно роды Eustigmatos Hibberd и Vische- ria Pascher различались наличием выростов на клеточной оболочке у последнего (Hibberd, 1981; Ettl, Gärtner, 1995). Хотя было отмечено, что старые культуры (свыше 6 месяцев культивирования) E. magnus (Petersen) Hibberd характеризовались присутствием клеток с выростами (Safiullina et al., 2014). Внутри рода Eustigmatos было описано четыре вида, различающиеся размерами вегетативных клеток и зооспор. Для E. vischeri Hibberd диаметр клеток составлял 7–9 мкм (максимально до 20–30 мкм), для E. polyphem (Pitschmann) Hibberd 14–25 мкм в диаметре и зооспоры до 24 мкм длиной, у E. magnus клетки 14–34 мкм в диаметре и зооспоры 12 мкм длиной, у E. calaminaris Trzcinska et Pawlik-Skowronska клетки 7–12 мкм в диаметре (редко до 18–30 мкм) и зооспоры 7–12 мкм длиной. Последняя ревизия на основе данных морфологии, 18S рРНК и ITS2-филогении объединила роды Eustigmatos и Vischeria в один (Krivenda et al., 2018). В настоящее время род Vischeria насчитывает 16 таксономически принятых видов, при этом для девяти из них (V. aculeata Pascher, V. aster Pascher, V. gemma Pascher, V. gibbosa Pascher, V. regularis Pascher, V. rimosa Pascher, V. tetraedroides Pascher, V. torta Pascher, V. undulata Pascher) отсутствуют депонированные нуклеотидные последовательности в GenBank, а длина последовательности 18S рРНК аутентичного штамма V. magna (Petersen) Kryvenda, Rybalka, Wolf et Friedl SAG 2554 не превышает 300 п.н. Таким образом, филогения данного рода остается все еще слабо изученной.
Целью настоящей работы является морфологический и молекулярно-генетический анализ четырех штаммов эустигматофитовых водорослей из Альгологической коллекции ACSSI.
МАТЕРИАЛЫ И МЕТОДЫ
Изоляция штаммов и условия культивирования. Штаммы ACSSI 012 и 081 были выделены из верхнего гумусового горизонта серой лесной почвы Московской области (54°50'03"N 37°34'24"E), штаммы ACSSI 013 и 026 – из второго гумусового горизонта в виде кротовин, серой лесной почвы Тульской области (54°23'30.1"′N 38°10'53.5"E). Из образцов стерильно отобранных почв готовили водно-почвенную суспензию и наносили ее на поверхность твердой питательной среды BG11 (1%-ный агар, pH = 7.0) на чашке Петри, одновременно образцы почвы добавляли к жидкой среде BG11 в 96-луночном культуральном планшете. Далее культуры были доведены до альгологически чистых с помощью многократного пересева. Культивирование штаммов проводили в климатостате KC-200, (Смоленское СКТБ СПУ, Россия) при стандартных условиях (температура +23–25°С, свет 60–75 μмоль фотонов м–2 с–1, фотопериод 12 ч.).
Морфологический анализ. Морфологию и жизненные циклы штаммов эустигматофитовых изучали методами световой микроскопии (светлое поле и интерференционный контраст) с помощью микроскопов Leica DM750 и Carl Zeiss Axio Scope A1 (Германия). Результаты наблюдений документированы рабочими рисунками и фотографиями, снятыми с помощью цветной цифровой камеры Carl Zeiss MRc 5 (Германия), морфологические характеристики приведены в табл. 1. Сроки наблюдения составляли от 2 недель до 6 месяцев. При морфологической идентификации штаммов учитывали тип организации таллома; форму и размеры клеток; наличие и толщину слизистых оболочек; количество и тип хлоропластов; наличие пиреноида и структуру крахмальной обкладки; способы размножения, присутствие гигантских клеток и шиповидных выростов. Для оценки размеров вегетативных клеток проводили 100 измерений для каждого штамма, для автоспор – 20. Расчеты минимального, максимального, среднего значения и стандартного отклонения выполняли в ПО Видеозавр (Россия). Каждый штамм был предварительно идентифицирован на основе данных морфологии.
Таблица 1.
Морфологическая характеристика штаммов ACSSI Table 1. Morphological features of ACSSI strains
| Штамм Strain |
Диаметр вегетатив-
ных клеток, мкм Diameter of vegeta- tive cells, µm |
Наличие гиган-тских клеток Presence of giant cells |
Наличие шипо-видных выростов Presence of cell wall projections |
Размеры автоспор, мкм Autospore size, µm |
Количество автоспор Number of autospores |
|---|---|---|---|---|---|
| V. magna ACSSI 012 | 8.4–11.9 (10.0 ± 0.87) | До 32 мкм Up to 32 µm |
Не наблюдали Not observed |
8.1–11.6 × 5.6–8.2 (9.8 ± 0.87 × 6.9 ± 0.75) |
2–4 |
| V. magna ACSSI 013 | 7.1–13.7 (10.1 ± 1.42) | Не наблюдали Not observed |
5.7–11.1 × 3.9–8.2 (9.1 ± 1.23 × 6.4 ± 1.08) |
||
| V. magna ACSSI 026 | 7.9–15.1 (10.7 ± 1.51) | 6.0–11.5 × 5.1–10.1 (9.0 ± 1.60 × 7.3 ± 1.40) |
2 | ||
| V. magna ACSSI 081 | 6.9–13.4 (9.6 ± 1.21) | До 24 мкм Up to 24 µm |
4.9–8.1 × 3.8–5.8 (6.4 ± 0.68 × 5.0 ± 0.54) |
2–6 |
Примечание. Для зрелых клеток указаны минимальные и максимальные значения диаметра, в скобках – среднее значение ± стандартное отклонение, для автоспор указаны минимальные и максимальные значения длины и ширины, в скобках – среднее значение ± стандартное отклонение.
Note. For mature cells, the minimum and maximum diameters are specified, with the mean ± standard deviation in parentheses. For autospores, the minimum and maximum values of length and width are specified, with the mean ± standard deviation in parentheses.
Экстракция ДНК, амплификация, секвенирование. Далее идентификация морфовидов подтверждалась с помощью молекулярно-генетических методов. Суммарную ДНК выделяли с помощью набора DNeasy Plant Mini Kit (Qiagen, США), следуя протоколу производителя. Для амплификации использовали готовую смесь для ПЦР Screen Mix-HS (Евроген, Россия). Праймеры и условия для амплификации выбранных маркеров указаны в табл. 2. Детекцию целевых ПЦР-продуктов проводили электрофоретически в 1%-ном агарозном геле. Для дальнейшей очистки ампликонов из геля применяли набор Cleanup Mini (Евроген, Россия). Секвенирование нуклеотидных последовательностей осуществляли на базе ЗАО “Синтол” (Россия).
Таблица 2.
Праймеры и условия амплификации эустигматофитовых водорослей Table 2. Primers and amplification conditions for eustigmatophyte algae
| Локус Locus |
Праймер Primer |
Последовательность (5'–3') Sequence |
Условия амплификации Amplification conditions |
Ссылка Reference |
|---|---|---|---|---|
| 18S рРНК 18S rRNA | 18S F | AACCTGGTTGATCCTGCCAGT | 95°C – 5 min; 95°C – 1 min, 55°C – 1 min, 72°C – 2 min, 25 cycles; 72°C – 5 min | Katana et al., 2001 Fawley et al., 2014 |
| Eustig R1 | GTTATAAACTCGTTGAACGCA | |||
| ITS2 | ITS-F-Visch | GCGCCGTTGGCTTCTAGCC | 94°C – 3 min; 94°C – 1 min, 53°C – 1 min, 72°C – 2 min, 35 cycles; 72°C – 10 min | Procházková, 2012 |
| ITS4-Eustig | TCCTCCGCTTAGTTATATGC |
Молекулярно-генетический анализ. Для филогенетического анализа штаммов был выполнен поиск гомологии нуклеотидных последовательностей по алгоритму BLAST в GenBank, составлены наборы из собственных и депонированных в GenBank последовательностей (табл. 3). Названия штаммов, нуклеотидные последовательности которых были использованы в этом исследовании, приведены в соответствии с их названиями в генетической базе данных. Всего в анализ вошло 86 последовательностей гена 18S рРНК длиной 1781 п.н. и 18 последовательностей спейсера ITS2 длиной 397 п.н. В программе BioEdit по алгоритму ClustalW было выполнено множественное выравнивание. Для выбора модели нуклеотидных замен использовали программу jModelTest. Реконструкцию филогенетических взаимосвязей осуществляли методом максимального правдоподобия (ML) в программе PhyML. Статистическая поддержка топологии дерева была оценена с помощью бутстрэп-анализа (1000 повторностей) и указана в узлах ветвей в виде процентов (рис. 2). Генетические различия между нуклеотидными последовательностями гомологичных генов охарактеризовали с помощью генетических дистанций. Мерой генетических различий являлся процент несовпадений нуклеотидов при попарном сравнении выровненных последовательностей, вычисление которого проводили в программе MEGA 5.0. Филогенетические деревья визуализировали с помощью программы FigTree v1.3.1. Границы вариабельных участков V4-V5 и V8-V9 гена 18S рРНК были обнаружены автоматическим поиском при выравнивании последовательностей и праймеров v4_Reuk454FWDI (CCAGCASCYGCGGTAATTCC) и v4-v5_1132r (CCGTCAATTHCTTYAART), V8_V8f (ATAACAGGTCTGTGATGCCCT) и V9_1510r (CCTTCYGCAGGTTCACCTAC), соответственно (Bradley et al., 2016).
Рис. 1.
Строение клеток штаммов Vischeria magna.
а – вегетативные клетки ACSSI 012, b – гигантские зрелые клетки ACSSI 012, с – вегетативные клетки ACSSI 013, d – вегетативные клетки ACSSI 026, e – вегетативные клетки ACSSI 081, f – гигантские зрелые клетки ACSSI 081. Шкала: 10 мкм.
Fig. 1. Cell structure of Vischeria magna strains.
a – vegetative cells ACSSI 012, b – giant mature cells ACSSI 012, c – vegetative cells ACSSI 013, d – vegetative cells ACSSI 026, e – vegetative cells ACSSI 081, f – giant mature cells ACSSI 081. Scale: 10 μm.
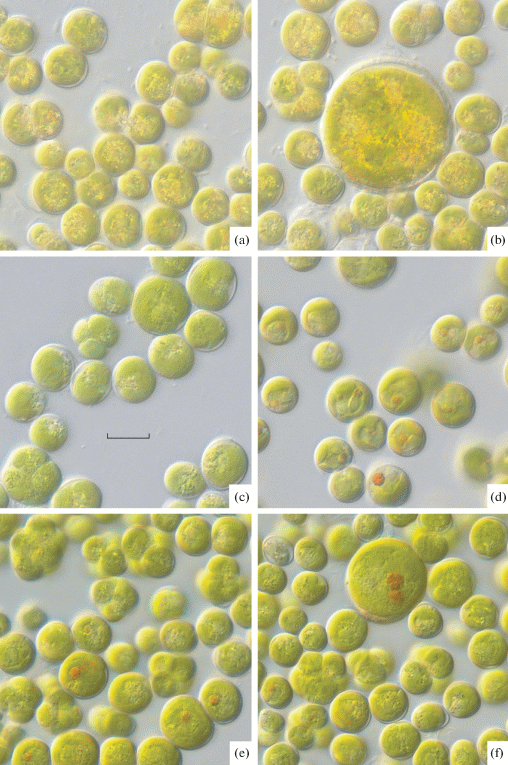
Таблица 3.
Список штаммов, использованных в филогенетическом анализе эустигматофитовых водорослей Table 3. List of strains used in the phylogenetic analysis of the eustigmatophyte algae
| N | Виды Species |
Номер штамма
при изоляции Strain number at isolation |
Коллекционный штамм Collection strain |
18S рРНК 18S rRNA |
ITS2 | Местообитание, страна Habitat, country |
|---|---|---|---|---|---|---|
| 1 | Eustigmatos vischeri | T8 | CCAP 860/7 | KJ713283 | KJ713283 | Почва, Австрия Soil, Austria |
| 2 | Eustigmatos magnus | – | SAG 2554* (= CCAP 860/4, UTEX 2351, CCMP 387) | – | MG596348 | Почва, Новая Зеландия Soil, New Zealand |
| 3 | Eustigmatos cf. polyphem | – | CAUP H4302 | JX865375 | – | Чехия Czech Republic |
| 4 | Vischeria punctata | 266 | SAG 887–1* | KY271664 | KY271673 | Почва, Швейцария Soil, Switzerland |
| 5 | Vischeria punctata | 316 | UTEX 86 | FJ858971 | – | Почва Soil |
| 6 | Eustigmatos magnus | – | SAG 36.89 | KY271662 | JX202557 | Почва, Непал Soil, Nepal |
| 7 | Uncultured eukaryote clone WS072.033 | – | – | KP404737 | – | Южно-Китайское море South China Sea |
| 8 | Uncultured eukaryote clone WS071.070 | – | – | KP404701 | – | Южно-Китайское море South China Sea |
| 9 | Eustigmatos calaminaris | E120 | CCALA 1014* | JX188078 | JX202554 | Почва, Польша Soil, Poland |
| 10 | Eustigmatos magnus | – | SAG 2506 | KY271665 | KY271678 | – |
| 11 | Eustigmatos vischeri | 241 | SAG 860–1* (= CCAP 860/1A; UTEX 310) | JX274590 | JX202556 | Почва, Швейцария Soil, Switzerland |
| 12 | Eustigmatos polyphem | ASIB 207 | SAG 38.84* (CCAP 860/8 ) | JX188077 | JX202558 | Почва, Кения Soil, Kenya |
| 13 | Vischeria stellata (T) | 185 | SAG 887–2* (CCAP 887/2B; UTEX 312) | KY271666 | KY271675 | Почва, Швейцария Soil, Switzerland |
| 14 | Vischeria helvetica | 255 | UTEX 49* (SAG 876–1; ATCC 30425; CCAP 861/1) | AF045051 | JX202559 | Почва Soil |
| 15 | Eustigmatos magnus | – | SAG 2370 | KY271663 | KY271677 | – |
| 16 | Chloridella neglecta | – | SAG 48.84 | KF848924 | KY271697 | Почва, Германия Soil, Germany |
| 17 | Vischeria helvetica | – | CCALA 514 | KF848920 | – | – |
| 18 | Eustigmatos magnus | – | SAG 2266 | KY271669 | KY271676 | – |
| 19 | Vischeria stellata | – | SAG 33.83 | KF848919 | KY271674 | Почва, бывшая Югославия Soil, former Yugoslavia |
| 20 | Vischeria helvetica | KGU-Y001 | – | AB731568 | – | Япония Japan |
| 21 | Eustigmatos vischeri | JNU4 | KT191017 | – | – | |
| 22 | Characiopsis saccata | – | SAG 15.97 (= ACOI 481) | KF848925 | – | Рисовое поле, Португалия Rice field, Portugal |
| 23 | Characiopsis acuta | – | ACOI 456 (SAG 14.97) | KY271644 | – | Пресные воды, Португалия Freshwater, Portugal |
| 24 | Eustigmatophyceae sp. | – | SAG 2220 | KY271668 | – | – |
| 25 | Characiopsis acuta | – | ACOI 1837 | KY271646 | – | Планктон, Португалия Plankton, Portugal |
| 26 | Dioxys sp. | – | ACOI 2029 | KY271650 | – | Португалия Portugal |
| 27 | Chlorobotrys gloeothece | – | ACOI 1114 | KY271649 | – | Пруд, Португалия Pond, Portugal |
| 28 | Uncultured stramenopile | clone OL10 | – | KX465211 | – | Озеро, Кения Lake, Kenya |
| 29 | Chlorobotrys regularis | – | ACOI 307 | KY271643 | – | Стоячая вода, Португалия Stagnant water, Portugal |
| 30 | Chlorobotrys regularis | – | ACOI 1089 | KY271648 | – | Пруд, Португалия Pond, Portugal |
| 31 | Vischeria punctata | – | IPPAS H–242 | MH979476 | – | – |
| 32 | Uncultured eukaryote clone WS072.034 | – | – | KP404738 | – | Южно-Китайское море South China Sea |
| 33 | Uncultured eukaryote clone WS071.072 | – | – | KP404703 | – | Южно-Китайское море South China Sea |
| 34 | Nannochloropsis sp. | JL2/4-1 | – | DQ977727 | – | Озеро, США Lake, USA |
| 35 | Characiopsis longipes | – | ACOI 1838 | KY271647 | – | Бассейн, Португалия Reservoir, Portugal |
| 36 | Nannochloropsis limnetica | AS3-9 | – | DQ977726 | – | Озеро, США Lake, USA |
| 37 | Nannochloropsis granulata | CCMP529 | – | U41092 | – | – |
| 38 | Nannochloropsis limnetica | CCMP505 | – | U41050 | – | – |
| 39 | Nannochloropsis oculata | CCMP225 | – | KU900229 | – | – |
| 40 | Nannochloropsis sp. | Tow 2/24 P-1w | – | DQ977728 | – | Пруд, США Pond, USA |
| 41 | Nannochloropsis sp. | NANNO-IOLR | – | AB025533 | – | – |
| 42 | Nannochloropsis oculata | CCMP525 | – | AF045044 | – | – |
| 43 | Nannochloropsis sp. | CCMP531 | – | U41094 | – | – |
| 44 | Nannochloropsis limnetica | – | SAG 18.99* | AF251496 | EU165325 | Пруд, Германия Pond, Germany |
| 45 | Uncultured eukaryote clone WS074.005 | – | – | KP404875 | – | Южно-Китайское море South China Sea |
| 46 | Nannochloropsis sp. | IOLR | – | AF067956 | – | – |
| 47 | Monodopsis unipapilla | – | SAG 8.83* | AM490827 | KY271699 | Почва, Австрия Soil, Austria |
| 48 | Monodopsis subterranea | – | SAG 848–1 (= CCAP 848/1; UTEX 151; ATCC 30593 ) | U41054 | KY271698 | Камни в реке, США Wet rock in a river, USA |
| 49 | Uncultured eukaryote clone WS071.073 | – | – | KP404704 | – | Южно-Китайское море South China Sea |
| 50 | Pseudotetraedriella kamillae | – | SAG 2056* | EF044311 | KY271700 | Озеро, Германия Lake, Germany |
| 51 | Microchloropsis salina | – | CCMP369 | U41093 | – | – |
| 52 | Monodopsis cf. guttula | – | CCALA 825 | KF848929 | – | – |
| 53 | Monodopsis sp. | 1 ME-2013 | CAUP 901 | KF848926 | – | Камни, Чехия Rocks, Czech Republic |
| 54 | Monodopsis guttula | CCALA 826 | KF848927 | – | – | |
| 55 | Microchloropsis gaditana | – | CCAP 849/5* (= CCMP1775) | AF045036 | Море, Испания Sea, Spain |
|
| 56 | Microchloropsis gaditana | – | CCMP526 | KF040086 | – | – |
| 57 | Microchloropsis gaditana | – | CCMP527 | M87328 | – | США USA |
| 58 | Uncultured phytoplankton clone Q3–25 | – | – | JQ420104 | – | Фитопланктон, Китай Phytoplankton, China |
| 59 | Nannochloropsis oceanica | – | CCALA 978 | KF010154 | – | Солоноватые воды, Аргентина Brackish water, Argentina |
| 60 | Pseudellipsoidion edaphicum | – | CAUP Q 401* | KF848933 | – | Торфяно-болотная почва, Чехия Peat-bog soil, Czech Republic |
| 61 | Nannochloropsis oceanica | MBIC10090 | – | AB052273 | DQ069777 | – |
| 62 | Nannochloropsis granulata | MBIC10054 | – | AB052272 | EU165324 | – |
| 63 | Nannochloropsis sp. | KMMCC EUS-16 | – | GQ122351 | – | Южная Корея South Korea |
| 64 | Eustigmatophyceae sp. | Tow 9/21 P–2w | – | KF757253 | – | Пруд, США Pond, USA |
| 65 | Eustigmatophyceae sp. | Mary 6/3 T–1w | – | KF757240 | – | Озеро, США Lake, USA |
| 66 | Eustigmatophyceae sp. | Mary 8/18 T–4d | – | KF757239 | – | Озеро, США Lake, USA |
| 67 | Nannochloropsis oceanica | – | CCAP 849/10 | KJ756836 | KJ713291 | – |
| 68 | Nannochloropsis gaditana | IVP | – | EF473733 | – | Италия Italy |
| 69 | Nannochloropsis gaditana | – | – | AF133819 | – | Лагуна, Италия Lagoon, Italy |
| 70 | Nannochloropsis salina | MBIC10063 | – | AB052278 | EU165326 | – |
| 71 | Nannochloropsis oceanica | – | CCAP 849/9 | KJ756835 | – | Море, Япония Sea, Japan |
| 72 | Nannochloropsis oceanica | – | CCAP 849/8 | KJ756834 | KJ713290 | Море, Китай Sea, China |
| 73 | Nannochloropsis gaditana | MBIC10118 | – | AB052269 | – | – |
| 74 | Nannochloropsis oculata | – | CCAP 849/1* (= SAG 38.85, UTEX 2164) | KJ756827 | KY271701 | Скалистая зона заплеска, Шотландия Supralitoral rock pool, Scotland |
| 75 | Nannochloropsis oculata | – | CCAP 849/7 | KJ756833 | KJ713289 | Озеро, Тунис Lake, Tunis |
| 76 | Pseudocharaciopsis ovalis | – | CAUP Q 302 | KF848932 | – | Почва, Чехия Soil, Czech Republic |
| 77 | Nannochloropsis salina | – | CCAP 849/2 | KJ756828 | KJ713285 | Море, Шотландия Sea, Scotland |
| 78 | Microchloropsis gaditana | – | CCAP 849/6 | KJ756832 | KJ713288 | Море, США Sea, USA |
| 79 | Microchloropsis salina | – | CCAP 849/4 | KJ756830 | KJ713287 | Море, Шотландия Sea, Scotland |
| 80 | Pseudocharaciopsis ovalis | – | CAUP Q 301 | KF848931 | – | Торфяно-болотная почва, Чехия Peat-bog soil, Czech Republic |
| 81 | Eustigmatophyceae sp. | Mary 8/18 T-3d | – | KF757238 | – | Озеро, США Lake, USA |
| 82 | Nannochloropsis australis | CS-416 | – | KT031997 | – | Море, Австралия Sea, Australia |
| 83 | Nannochloropsis australis | CS-759* | – | KT031998 | – | Море, Австралия Sea, Australia |
| 84 | Vischeria magna | – | ACSSI 012 | MK228871 | MK228867 | Серая лесная почва, Россия Grey forest soil, Russia |
| 85 | Vischeria magna | – | ACSSI 013 | MK228872 | MK228868 | Кротовина, серая лесная почва, Россия Mole drain, grey forest soil, Russia |
| 86 | Vischeria magna | – | ACSSI 026 | – | MK228869 | Кротовина, серая лесная почва, Россия Mole drain, grey forest soil, Russia |
| 87 | Vischeria magna | – | ACSSI 081 | MK228873 | MK228870 | Серая лесная почва, Россия Grey forest soil, Russia |
| Mediophyceae, Bacillariophyta (внешняя группа) Mediophyceae, Bacillariophyta (outgroup) |
||||||
| 88 | Thalassiosira pseudonana | – | CCMP 1007 | HF565127 | HF565127 | Море, США Sea, USA |
Примечание. Прочерк означает отсутствие данных, * – аутентичный штамм, (T) – типовой вид.
Note. Dash – data missing, * – authentic strain, (T) – type species.
Аббревиатуры коллекций культур / Abbreviations of culture collections: ACOI – Coimbra Collection of Algae, Португалия / Portugal; ACSSI – Algal Collection of Soil Science Institute, Россия / Russia; ATCC – American Type Culture Collection, США / USA; CAUP – the Culture Collection of Algae of Charles University, Чехия / Czech Republic; CCALA – the Culture Collection of Autotrophic Organisms, Чехия / Czech Republic; CCAP – the Culture Centre Algae and Protozoa, Великобритания / United Kingdom; CCMP – National Center for Culture of Marine Phytoplankton, США / USA; IPPAS – Culture Collection of Microalgae IPPAS, Россия / Russia; SAG – the Culture Collection of Algae at the University of Göttingen, Германия / Germany; UTEX – the Culture Collection of Algae at The University of Texas at Austin, США / USA.
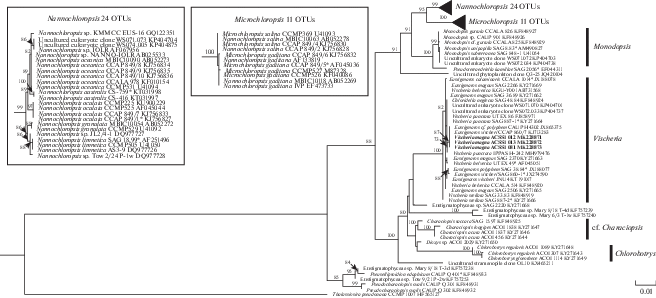
Рис. 2.
Укорененное филогенетическое дерево эустигматофитовых водорослей, построенное методом максимального правдоподобия (ML), на основе последовательностей гена 18S рРНК.
В качестве статистической поддержки узлов дерева указаны бутстреп-значения ML; значения <70% не показаны. Модель нуклеотидных замен: GTR + I + G. Жирным шрифтом выделены штаммы ACSSI, * отмечены аутентичные штаммы.
Fig. 2. Rooted phylogenetic tree of eustigmatophyte algae constructed by the maximum likelihood (ML) method, based on the 18S rRNA gene (1781 bp).
ML bootstrap values are specified as statistical support for tree nodes; values < 70% are not shown. Nucleotide Substitution Model: GTR + I + G. ACSSI strains are highlighted in bold, authentic strains are marked with asterisk (*).
В качестве инструмента разделения видов был использован подход, предложенный A. Coleman (2000, 2009), которая показала, что наличие хотя бы одной компенсаторной замены (compensatory base change, CBC) в консервативных регионах ITS2 (10 пар нуклеотидов для II шпильки и 18 пар – для III шпильки) у двух водорослей коррелирует с их полной половой несовместимостью. Напротив, замены в менее консервативных регионах (I и IV шпильки), а также полукомпенсаторные замены (hCBC) в консервативных регионах не были связаны со способностью скрещиваться. На основе мета-анализа большого числа данных Müller с соавт. (2007) установили, что наличие даже одной CBC в 93% исследованных случаев указывает на принадлежность организмов к разным видам. Для анализа вторичной структуры ITS2 была выполнена аннотация спейсера в ITS2-DataBase (http://its2.bioapps.biozentrum.uni-wuerzburg.de), его фолдинг с помощью RNAfold web server (http://rna.tbi.univie.ac.at/cgi-bin/RNAWebSuite/RNAfold.cgi), визуализация посредством программы PseudoViewer3 (Byun, Han, 2009), выравнивание ITS2 с учетом вторичной структуры и поиск CBC осуществляли в программе 4SALE.
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Морфологический анализ. Наблюдения за штаммами ACSSI 012, 013, 026 и 081 показали их морфологическое сходство с V. magna (≡ E. magnus): шаровидные одиночные клетки в среднем 9.6–10.7 мкм в диаметре, для штаммов ACSSI 012 и 081 отмечено присутствие “гигантских” клеток с максимальным диаметром – 32 и 24 мкм, соответственно. Клеточная оболочка гладкая, нескульптурированная, шиповидных выростов не наблюдали. Хлоропласт пристенный, лопастной с 1 полиэдрическим пиреноидом и большой красной глобулой (рис. 1). Размножение преимущественно 2–4 (ACSSI 012, ACSSI 013), 2 (ACSSI 013) или 2–6 (ACSSI 081) автоспорами. Автоспоры эллипсоидной и широкоовальной формы, самые крупные – у штаммов ACSSI 012 и 026, самые мелкие – у штамма ACSSI 081 (табл. 1). Половой процесс и зооспоры не наблюдали.
Молекулярно-генетический анализ. Проведенный 18S рРНК-анализ подтвердил принадлежность штаммов ACSSI к роду Vischeria со 100%-ной статистической поддержкой (рис. 2). Сестринскими являются род Chlorobotrys и Characiopsis-подобный род, представители которого принадлежат эустигматофитовым, а не желто-зеленым водорослям. На филогенетическом дереве некультивируемые клоны эустигматофитовых водорослей образуют несколько независимых филогенетических линий, которые, возможно, в будущем будут описаны как новые самостоятельные роды. Основным местообитанием проанализированных штаммов являются пресноводные пруды и озера. Изоляты двух родов (Nannochloropsis и Microchloropsis) были выделены в основном из лагун и открытого моря. Все штаммы рода Vischeria были изолированы из почв, исключение составляют некультивируемые клоны WS072.033 и WS071.070 из Южно-Китайского моря (табл. 3).
Кластеризация внутри рода Vischeria характеризовалась низкими бутстрэп-значениями, что свидетельствует о недостаточном филогенетическом сигнале гена, кодирующего 18S рРНК, на видовом уровне. Анализ вариабельных регионов V4-V5 и V8-V9, которые широко используются в качестве ДНК-баркодов при идентификации сообществ эукариотических организмов, показал наличие только двух замен у V. stellata (Chodat) Pascher SAG 887-2 и штаммов ACSSI, соответственно. Таким образом, данная работа подтвердила и дополнила вывод о том, что для эустигматофитовых водорослей ген 18S рРНК можно использовать для выделения высоких таксономических рангов (Fawley et al., 2014), например, порядков. Однако, ни целый ген, ни его вариабельные области V4-V5 и V8-V9 не подходят для разграничения видов, в том числе в метагеномных исследованиях. Поэтому для уточнения филогенетического статуса штаммов ACSSI был проанализирован более изменчивый молекулярно-генетический маркер – ITS2 (рис. 3). По данным ITS2-филогении все четыре изученных штамма ACSSI вошли в одну группу с аутентичным штаммом Eustigmatos magna SAG 2554 со статистической поддержкой 64%. Внутри данной группы штаммы ACSSI 012, 013 и 081 образовали первую подгруппу, штамм ACSSI 026 и SAG 2554 – вторую (рис. 3). Высокими поддержками характеризовались клада “V. punctata + V. helvetica” и “V. stellata” – 93 и 100% соответственно. Следует отметить, что часть штаммов, по морфологии идентифицированные как V. magna (≡ E. magnus), на дереве ITS2 образовывали самостоятельные филогенетические линии, не группируясь с аутентичным штаммом SAG 2554, и, следовательно, могут быть отдельными криптическими таксонами. Аналогично штамм CCAP 860/7б, идентифицированный как V. vischeri (≡ E. vischeri), не объединился с аутентичным штаммом SAG 860-1. Применение более вариабельного маркера ITS2 привело к снижению доли консервативных участков с 78% до 35% по сравнению с маркером 18S рРНК.
Рис. 3.
Укорененное филогенетическое дерево эустигматофитовых водорослей рода Vischeria, построенное методом максимального правдоподобия (ML), на основе анализа первичной структуры ITS2.
В качестве статистической поддержки узлов дерева указаны бутстреп-значения ML; значения <70% не показаны. Модель нуклеотидных замен: SYM. Жирным шрифтом выделены штаммы ACSSI, * отмечены аутентичные штаммы.
Fig. 3. Rooted phylogenetic tree of the genus Vischeria constructed by the maximum likelihood (ML) method, based on the analysis of the primary structure of ITS2 (397 bp).
ML bootstrap values are specified as statistical support for tree nodes; values <70% are not shown. Nucleotide Substitution Model: SYM. ACSSI strains are highlighted in bold, authentic strains are marked with asterisk (*).

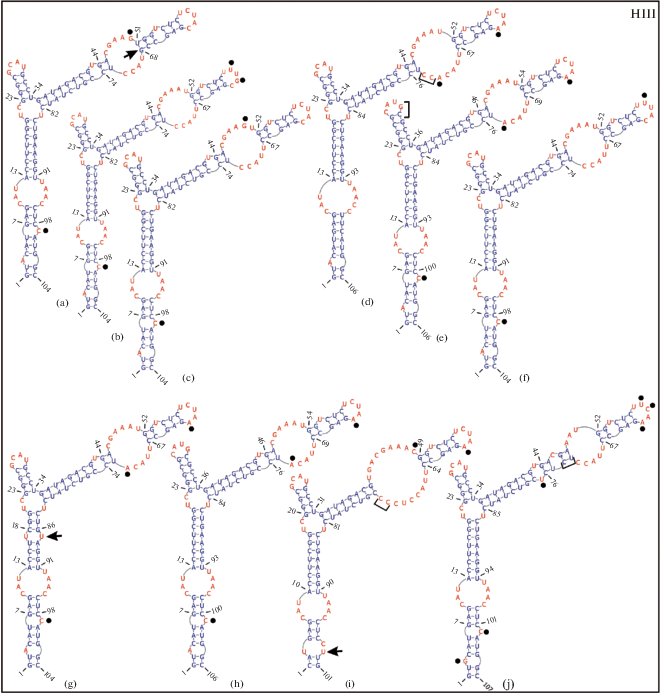
Тем не менее использование первичной структуры ITS2 не позволило разрешить филогению рода Vischeria. Поэтому далее мы изучили вторичную структуру ITS2 штаммов ACSSI и сравнили ее со всеми доступными аутентичными штаммами видов рода Vischeria. Длина спейсера составила 255 п.н., третья шпилька была самая длинная и разветвленная (рис. 5), что было показано для всех штаммов рода Vischeria. Во второй шпильке присутствовал U-U мисматч, на верхушке третьей шпильки – консервативный мотив GGUCGG (рис. 4, 5). При сравнении вторичных структур ITS2 четырех штаммов ACSSI с аутентичными штаммами видов V. calaminaris (Trzcinska et Pawlik-Skowronska) Kryvenda, Rybalka, Wolf et Friedl CCALA 1014, V. helvetica (Vischer et Pascher) Hibberd UTEX 49, V. magna SAG 2554, V. polyphem (Pitschmann) Kryvenda, Rybalka, Wolf et Friedl SAG 38.84, V. punctata Vischer SAG 887-1, V. stellata SAG 887-2, V. vischeri (Hibberd) Kryvenda, Rybalka, Wolf et Friedl SAG 860-1 не было обнаружено ни одной CBC ни между исследуемыми штаммами ACSSI, ни между штаммами из других коллекций. Тем не менее была найдена одна CBC во второй шпильке ITS2 при сравнении штаммов ACSSI с неаутентичным штаммом E. magnus SAG 2506 и одна CBC в четвертой шпильке ITS2 при сравнении с E. magnus SAG 36.89. Данные замены не учитываются при разделении видов. Таким образом, CBC-подход не позволяет разделить описанные виды рода Vischeria: V. magna, V. calaminaris, V. stellata, V. helvetica, V. punctata, V. vischeri и V. polyphem. Несмотря на отсутствие CBC, необходимо отметить другие различия, которые были обнаружены во всех четырех шпильках ITS2 (рис. 4, 5). Штамм ACSSI 026 и аутентичный штамм V. magna SAG 2554 отличались от других штаммов одной заменой С → U на вершине I шпильки ITS2 (рис. 4b, f). Штамм V. stellata SAG 887-2 отличался от остальных видов рода одной заменой С → U на неспаренной вершине I шпильки ITS2 (рис. 4i). Во II шпильке ITS2 штаммов V. punctata SAG 887-1 и V. stellata SAG 887-2 были обнаружены по одной hCBC (рис. 4h, i), а для штамма V. vischeri SAG 860-1 был характерен распаренный участок UUGCA, а не CUGCA как у остальных штаммов (рис. 4j). Кроме трех hCBC и мутаций неспаренных участков основания и верхушки III шпильки, были обнаружены вставки в коротком стебле разветвленной III шпильки GC у V. helvetica UTEX 49 (рис. 5e) и V. punctata SAG 887-1 (рис. 5 h), а также в длинном стебле разветвленной III шпильки СC у V. calaminaris CCALA 1014 (рис. 5 d) и V. stellata (рис. 5i), а также UC у V. vischeri SAG 860-1 (рис. 5j). Шпилька IV была наиболее вариабельной и имела одинаковую hCBC у штаммов ACSSI и V. magna SAG 2554 A-U (рис. 4a, b, c, f), U-U мисматч у V. helvetica UTEX 49 (рис. 4e) и V. punctata SAG 887-1 (рис. 4h), а также мутации на верхушке шпильки. Однако наличие нескольких паралогичных последовательностей ITS2 в пределах одного штамма (Kryvenda et al., 2018) затрудняет использование этого маркера для надежного разграничения видов рода Vischeria.
Рис. 4.
Сравнение вторичной структуры шпилек I, II, IV ITS2 штаммов ACSSI и аутентичных штаммов рода Vischeria.
Fig. 4. Comparison of the ITS2 secondary structure of helices I, II, IV for ACSSI strains and authentic strains of the genus Vischeria.
a – ACSSI 013, b – ACSSI 026, c – ACSSI 081, d – Vischeria calaminaris, e – V. helvetica, f – V. magna, g – V. polyphem, h – V. punctata, i – V. stellata, j – V. vischeri.
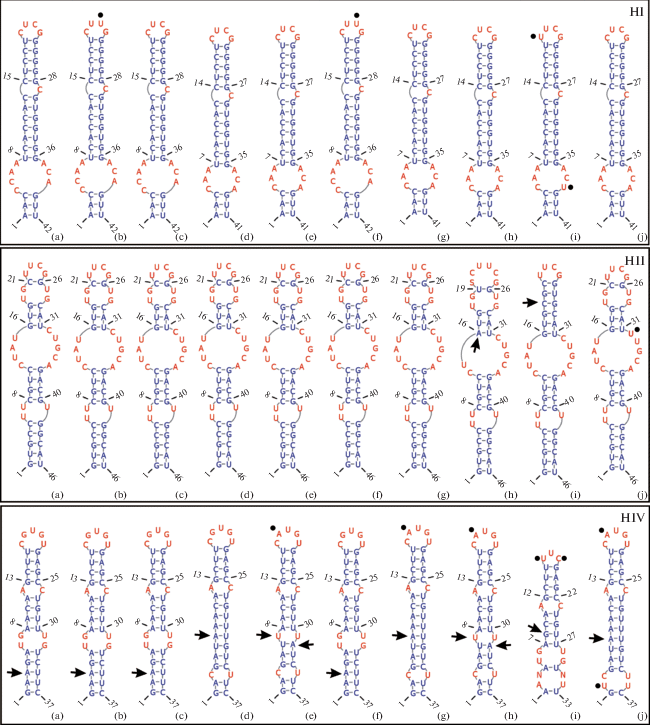
Рис. 5.
Сравнение вторичной структуры шпилек III ITS2 штаммов ACSSI и аутентичных штаммов рода Vischeria.
Fig. 5. Comparison of the ITS2 secondary structure of helix III for ACSSI strains and authentic strains of the genus Vischeria.
a – ACSSI 013, b – ACSSI 026, c – ACSSI 081, d – Vischeria calaminaris, e – V. helvetica, f – V. magna, g – V. polyphem, h – V. punctata, i – V. stellata, j – V. vischeri.
ЗАКЛЮЧЕНИЕ
Проведенный молекулярно-генетический анализ подтвердил, что все четыре изученных штамма ACSSI являются представителями рода Vischeria. Несмотря на то, что по морфотипу они были близки к V. magna, только ACSSI 026 с высокой статистической поддержкой кластеризовался с аутентичным штаммом SAG 2554. Систематика рода представляется проблемной из-за низкого разрешения ветвей на филогенетическом дереве 18S рРНК и ITS2. Используемые для других групп водорослей вариабельные регионы V4-V5 и V8-V9 гена 18S рРНК оказались неинформативны для видовой идентификации внутри рода. CBC-подход, основанный на разделении близкородственных видов при наличии хотя бы одной CBC в консервативных регионах ITS2, также не имел успеха. Другие обнаруженные различия во вторичной структуре ITS2 могут быть непоказательны вследствие внутригеномной вариации последовательностей ITS2 внутри одного штамма. Вероятно, увеличение объема выборки эустигматофитовых водорослей, использование пластидных генов, в частности rbcL, или глубокого секвенирования всего пластидного генома (Amaral et al., 2020), изучение ультраструктурных, физиологических, биохимических характеристик позволит надежно разделить виды рода Vischeria.
Список литературы
Amaral R., Fawley K.P., Nemcová Y., Sevcíková T., Lukesová A., Fawley M.W., Santos L.M.A., Eliás M. 2020. Toward modern classification of eumastigophytes, including the description of Neomonodaceae fam. nov. and three new genera. – J. Phycol. 56 (3): 630–648. https://doi.org/10.1111/jpy.12980
Byun Y., Han K. 2009. PseudoViewer3: generating planar drawings of large-scale RNA structures with pseudoknots. – Bioinformatics. 25 (11): 1435–1437. https://doi.org/10.1093/bioinformatics/btp252
Bradley I.M., Pinto A.J., Guest J.S. 2016. Design and evaluation of Illumina MiSeq-compatible, 18S rRNA gene-specific primers for improved characterization of mixed phototrophic communities. – Appl. Environ. Microbiol. 82 (19): 5878–5891. https://doi.org/10.1128/AEM.01630-16
Cepák V., Přibyl P., Kohoutková J., Kaštánek P. 2014. Optimization of cultivation conditions for fatty acid composition and EPA production in the eustigmatophycean microalga Trachydiscus minutus. – J. Appl. Phycol. 26 (1): 181–190. https://doi.org/10.1007/s10811-013-0119-z
Coleman A.W. 2009. Is there a molecular key to the level of “biological species” in eukaryotes? A DNA guide. – Mol. Phylogenet. Evol. 50: 197–203. https://doi.org/10.1016/j.ympev.2008.10.008
Coleman A.W. 2000. The significance of a coincidence between evolutionary landmarks found in mating affinity and a DNA sequence. – Protist. 151 (1): 1–9. https://doi.org/10.1078/1434-4610-00002
Durmaz Y. 2007. Vitamin E (α-tocopherol) production by the marine microalgae Nannochloropsis oculata (Eustigmatophyceae) in nitrogen limitation. – Aquaculture. 272: 717–722. https://doi.org/10.1016/j.aquaculture.2007.07.213
Elias M., Amaral R., Fawley K.P., Fawley M.W., Nemcova Y., Neustupa J., Pribyl P., Santos L.M.A., Sevcıkova T. 2017. Eustigmatophyceae. – In: Handbook of the Protists. Archibald J.M., Simpson A.G.B., Slamovits C.H. [Eds.] Springer International Publishing. Switzerland. P. 367–406.
Ettl H., Gärtner G. 1995. Syllabus der Boden-, Luft-, und Flechtenalgen. Stuttgart, Jena, New York, Gustav Fischer. 710 p.
Fawley K.P., Eliáš M., Fawley M.W. 2014. The diversity and phylogeny of the commercially important algal class Eustigmatophyceae, including the new clade Goniochloridales. – J. Appl. Phycol. 26: 1773–1782. https://doi.org/10.1007/s10811-013-0216-z
Fukuda S.Y., Iwamoto K., Atsumi M., Yokoyama A., Nakayama T., Ishida K., Inouye I., Shiraiwa Y. 2014. Global searches for microalgae and aquatic plants that can eliminate radioactive cesium, iodine and strontium from the radio-polluted aquatic environment: A bioremediation strategy. – J. Plant Res. 127 (1): 79–89. https://doi.org/10.1007/s10265-013-0596-9
Gao B., Xia S., Lei X., Zhang Z. 2018. Combined effects of different nitrogen sources and levels and light intensities on growth and fatty acid and lipid production of oleaginous eustigmatophycean microalga Eustigmatos cf. polyphem. – J. Appl. Phycol. 30: 215–229. https://doi.org/10.1007/s10811-017-1180-9
Gao B., Yang J., Lei X., Xia S., Li A., Zhang C. 2016. Characterization of cell structural change, growth, lipid accumulation, and pigment profile of a novel oleaginous microalga, Vischeria stellata (Eustigmatophyceae), cultured with different initial nitrate supplies. – J. Appl. Phycol. 28: 821–830. https://doi.org/10.1007/s10811-015-0626-1
Guiry M.D., Guiry G.M. 2021. AlgaeBase. World-wide electronic publication. National University of Ireland. Galway. http://www.algaebase.org
Hibberd D.J. 1981. Notes on the taxonomy and nomenclature of the algal classes Eustigmatophyceae and Tribophyceae (synonym Xanthophyceae). – Bot. J. Linn. Soc. 82 (2): 93–119. https://doi.org/10.1111/j.1095-8339.1981.tb00954.x
Hibberd D.J., Leedale G.F. 1971. A new algal class – The Eustigmatophyceae. – Taxon. 20 (4): 523–525. https://doi.org/10.1038/225758b0
Katana A., Kwiatowski J., Spalik K., Zakryś B., Szalacha E., Szymańska H. 2001. Phylogenetic position of Koliella (Chlorophyta) as inferred from nuclear and chloroplast small subunit rDNA. – J. Phycol. 37 (3): 443–451. https://doi.org/10.1046/j.1529-8817.2001.037003443.x
Kryvenda A., Rybalka N., Wolf M., Friedl T. 2018. Species distinctions among closely related strains of Eustigmatophyceae (Stramenopiles) emphasizing ITS2 sequence-structure data: Eustigmatos and Vischeria. – Eur. J. Phycol. 53 (4): 471–491. https://doi.org/10.1080/09670262.2018.1475015
Li Z., Sun M., Li Q., Li A., Zhang C. 2012. Profiling of carotenoids in six microalgae (Eustigmatophyceae) and assessment of their ß-carotene productions in bubble column photobioreactor. – Biotechnol. Lett. 34 (11): 2049–2053. https://doi.org/10.1007/s10529-012-0996-2
Martins C.B., Ferreira O., Rosado T. et al. 2021. Eustigmatophyte strains with potential interest in cancer prevention and treatment: partial chemical characterization and evaluation of cytotoxic and antioxidant activity. – Biotechnol Lett. https://doi.org/10.1007/s10529-021-03122-0
Müller T., Philippi N., Dandekar T., Schultz J., Wolf M. 2007. Distinguishing species. – RNA. 3 (9): 1469–1472. https://doi.org/10.1261/rna.617107
Norton T.A., Melkonian M., Andersen R.A. 1996. Algal biodiversity. – Phycologia. 35: 308–326. https://doi.org/10.2216/i0031-8884-35-4-308.1
Pal D., Khozin-Goldberg I., Didi-Cohen S., Solovchenko A., Batushansky A., Kaye Y., Sikron N., Samani T., Fait A., Boussiba S. 2013. Growth, lipid production and metabolic adjustments in the euryhaline eustigmatophyte Nannochloropsis oceanica CCALA 804 in response to osmotic downshift. – Appl. Microbiol. Biotechnol. 97 (18): 8291–8306. https://doi.org/10.1007/s00253-013-5092-6
Patterson G.W., Tsitsa-Tzardis E., Wikfors G.H., Smith B.C., Gladu, P. K. 1994. Sterols of Eustigmatophytes. – Lipids. 29 (9): 661–664. https://doi.org/10.1007/BF02536102
Procházková K. 2012. Diverzita a druhový koncept u komplexu Vischeria/Eustigmatos (Eustigmatophyceae): Diplomova prace. Praha. Karlova univerzita. 79 p.
Remias D., Nicoletti C., Krennhuber K. et al. 2020. Growth, fatty, and amino acid profiles of the soil alga Vischeria sp. E71.10 (Eustigmatophyceae) under different cultivation conditions. – Folia Microbiol. (Praha). 65 (6): 1017–1023. https://doi.org/10.1007/s12223-020-00810-8
[Safiullina et al.] Сафиуллина Л.М., Муратова К.Р., Закирова М.Б. 2014. Сравнительный анализ микроскопических почвенных водорослей Eustigmatos magnus и Vischeria helvetica (Eustigmatophyta). – Альгология. 24 (3): 270–273.
Stoykova P., Stoyneva-Gärtner M., Uzunov B., Gärtner G., Atanassov I., Draganova P., Borisova C. 2019. Morphological characterization and phylogenetic analysis of aeroterrestrial Vischeria/Eustigmatos strains with industrial potential. – Biotechnol. Biotechnol. Equip. 33 (1): 231–242. https://doi.org/10.1080/13102818.2018.1561212
Stoyneva-Gärtner M., Uzunov B., Gärtner G., Borisova C., Draganova P., Radkova M., Stoykova P., Atanassov I. 2019. Current bioeconomical interest in stramenopilic Eustigmatophyceae: a review. – Biotechnol. Biotechnol. Equip. 33 (1): 302–314. https://doi.org/10.1080/13102818.2019.1573154
Upadhyay A.K., Singh N.K., Singh R., Rai U.N. 2016. Amelioration of arsenic toxicity in rice: Comparative effect of inoculation of Chlorella vulgaris and Nannochloropsis sp. on growth, biochemical changes and arsenic uptake. – Ecotoxicol. Environ. Saf. 124: 68–73. https://doi.org/10.1016/j.ecoenv.2015.10.002
Wang F., Gao B., Huang L., Su M., Dai C., Zhang C. 2018. Evaluation of oleaginous eustigmatophycean microalgae as potential biorefinery feedstock for the production of palmitoleic acid and biodiesel. – Bioresour. Technol. 270: 30–37. https://doi.org/10.1016/j.biortech.2018.09.016
Дополнительные материалы отсутствуют.
Инструменты
Ботанический журнал


