Цитология, 2023, T. 65, № 1, стр. 102-110
Двойное ковалентное связывание биливердина в ближне-инфракрасных флуоресцентных белках предотвращает их протеолитическую деградацию
Ольга В. Степаненко 1, *, Олеся В. Степаненко 1
1 Институт цитологии РАН
194064 Санкт-Петербург, Россия
* E-mail: sov@incras.ru
Поступила в редакцию 24.08.2022
После доработки 22.09.2022
Принята к публикации 23.09.2022
- EDN: GLEWOB
- DOI: 10.31857/S0041377123010108
Аннотация
В настоящей работе мы проанализировали как двойное ковалентное связывание лиганда биливердина (BV) в ближне-инфракрасном флуоресцентном белке iRFP670, содержащем два ключевых остатка цистеина, влияет на устойчивость биомаркера к протеолитической деградации. Ранее выявлено, что ковалентное присоединение BV одновременно к двум ключевых остаткам цистеина является причиной наибольшего квантового выхода флуоресценции BV-содержащих ближне-инфракрасных флуоресцентных белков (NIR-FP) с двумя ключевыми остатками цистеина по сравнению с остальными BV-содержащими NIR-FP. Наши данные указывают на то, что ковалентное связывание BV в NIR-FP с двумя ключевыми остатками цистеина одновременно с двумя участками полипептидной цепи, которая, помимо этого, формирует узел типа восьмерки, приводит к экранированию в них многих сайтов расщепления протеолитическими ферментами трипсином и химотрипсином. Как следствие, при ковалентном связывании BV в NIR-FP одновременно с двумя ключевыми остатками цистеина не только стабилизируется их структура, но и может возрастать их устойчивость к протеолитической деградации, что определяет клеточную стабильность биомаркеров и важно для их использования в качестве флуоресцентных меток в клетке.
Генетически кодируемые белковые флуоресцентные маркеры успешно используются для изучения биологических процессов посредством селективного мечения ими целевых клеточных белков (Chernov et al., 2017; Rodriguez et al., 2017). Среди них ближне-инфракрасные (NIR) биомаркеры могут прижизненно использоваться в экспериментах на модельных животных, поскольку спектры поглощения и флуоресценции этих белков попадают в “окно прозрачности” биологических тканей, характеризующееся минимальным поглощением света компонентами клеток и водой (Weissleder, 2001). Mечение отдельных клеток с использованием NIR-биомаркеров позволило изучать биологические процессы в модельных животных, такие как метастазирование и канцерогенез (Yu et al., 2014), процессы регенерации, развитие паразитарных и вирусных инфекций, процессы, связанные с нарушением липидного метаболизма. Совместное использование спектрально различимых мономерных вариантов NIR-биомаркеров (Matlashov et al., 2020) и NIR-биосенсоров ключевых клеточных процессов на их основе (Komatsu et al., 2011; Shcherbakova et al., 2018a; Qian et al., 2019; Subach et al., 2019) с биомаркерами и биосенсорами видимого диапазона на основе GFP-подобных белков и с оптогенетическими инструментами на основе родопсинов и криптохромов, активность которых регулируется светом в сине-зеленой области спектра, значительно расширяет возможности изучения биологических процессов внутри клетки (Shcherbakova et al., 2018a). Использование NIR-биомаркеров с передовыми методами получения изображений (фотоакустической томографией, двух-фотонной микроскопией, регистрацией кривых затухания флуоресценции) позволяет достичь субмиллиметрового разрешения в глубоких тканях модельных животных (Li et al., 2018).
Большинство доступных в настоящее время ближне-инфракрасных (NIR) биомаркеров разработаны на основе ближне-инфракрасных флуоресцентных белков (NIR-FP), состоящих из двух доменов – PAS (Per-ARNT-Sim) и GAF (cGMP PDE/AC/FhlA), которые вместе образуют так называемый хромофор-связывающий домен полноразмерных бактериальных фитохромов (Rodriguez et al., 2017; Shcherbakova et al., 2018b). Природным лигандом бактериальных фитохромов и большинства NIR-биомаркеров на их основе является биливердин IXα (BV). Данный тетрапиррол синтезируется в клетках млекопитающих в достаточном количестве в результате деградации гема (Kapitulnik, Maines, 2012). Важной характеристикой NIR-биомаркеров при их использовании является яркость в клетке, которая определяется молекулярной яркостью белка и его стабильностью в клетке. Среди BV-содержащих NIR-FP наибольшим квантовым выходом флуоресценции и устойчивостью к химическим денатурантам обладают те из них, в которых присутствуют одновременно два ключевых остатка цистеина: консервативный для бактериальных фитохромов Cys15 в N-концевом участке PAS-домена (CysPAS) и Cys256, введенный в GAF-домен в положение, эквивалентное сайту связывания хромофора фоторецепторов растений и цианобактерий (CysGAF) (Stepanenko et al., 2022). Как было показано, CysGAF, наряду с CysPAS может выступать сайтом ковалентного связывания BV в NIR-FP (Shcherbakova et al., 2015; Stepanenko et al., 2016).
При кристаллографическом исследовании структуры мономерного белка miRFP670, который содержит два ключевых остатка цистеина, было выявлено ковалентное присоединение BV одновременно к этим двум остаткам (Baloban et al., 2017). Предполагают, что именно это и является причиной наибольшего квантового выхода флуоресценции и увеличения стабильности структуры (Stepanenko et al., 2017a) BV-содержащих NIR-FP с двумя ключевыми остатками цистеина по сравнению с остальными BV-содержащими NIR-FP. Эти предположения нашли подтверждение при анализе спектральных свойств биомаркера iRFP682 (Buhrke et al., 2019) и в наших недавних исследованиях спектральных свойств целого ряда NIR-FP, в которых BV связан одновременно с CysPAS и CysGAF (CysGAF-BV-CysPAS) (Stepanenko et al., 2022). Использование специальной методики для обогащения препаратов NIR-FP молекулами белка, содержащими хромофор CysGAF-BV-CysPAS, позволило нам надежно определить спектральные характеристики этого вида молекул NIR-FP. Тем не менее, связывание BV одновременно с двумя цистеиновыми остатками NI FP до сих пор не было подтверждено наиболее прямым методом масс-спектроскопии.
Мы предположили, что ковалентное присоединение BV одновременно к двум ключевым остаткам цистеина в NIR-FP может делать их малочувствительными к действию протеолитических ферментов. На примере iRFP670 мы провели анализ устойчивости NIR-FP с двумя ключевыми остатками цистеина к действию протеолитических ферментов. Эти исследования позволили нам установить причины неэффективности метода масс-спектроскопии в отношении NIR-FP, в которых BV одновременно присоединен ковалентно к двум остаткам цистеина. Нами выявлены структурные элементы, определяющие высокую устойчивость этих NIR-FP к протеолитическому расщеплению.
МАТЕРИАЛ И МЕТОДИКА
Материалы. Гидрохлорид гуанидина (GdnHCl), компоненты буферных растворов и трипсин из поджелудочной железы быка производства Sigma (США) использованы без дополнительной очистки. Химотрипсин и сурфактант ProteaseMAX™ (усилитель трипсина) были приобретены у Promega (США).
Выделение и очистка белков. Ген целевого белка iRFP670 с полигистидиновой меткой на N-конце клонирован в вектор pBAD/His-B (Invitrogen, США) по сайтам BglII и EcoRI, которым трансформировали штамм бактерий Escherichia сoli LMG 194. Белок iRFP670 получен и очищен, как описано ранее (Stepanenko et al., 2016, 2019). Белок iRFP670 в апо- (т.е. в отсутствие хромофора) и холоформе (т.е. в комплексе с BV) получен при экпрессии только гена целевого белка и совместно с геном гемоксигеназы (НО) с вектора pWA23h-HO соответственно. Фермент HO обеспечивает синтез BV. Для экспрессии биомаркеров в апоформе использовали среду RM (состав (мМ): 48 Na2HPO4, 22 KH2PO4, 19 NH4Cl, 8.5 NaCl, 2% казаминовых кислот, 1 MgCl2 и 1 тиамина), содержащую ампицилин. В состав культуральной среды для экспрессии целевого белка в холоформе входили ампицилин и канамицин. Для индукции экспрессии генов, кодирующих iRFP670 и НО, использовали арабинозу и рамнозу соответственно. Клеточный лизат очищали с помощью афинной хроматографии на колонках His-GraviTrap (GE Healthcare, Швеция) и ион-обменной хроматографии на колонке MonoQ (GE HealthCare, Швеция). Чистоту полученных препаратов белка контролировали электрофоретически (Laemmli, 1970).
Биохимические измерения. Ковалентное связывание BV с iRFP670 анализировали по цинк-индуцированной флуоресценции BV и путем окрашивания кумасси синим образцов белка, разделенных с помощью SDS-PAGE-электрофореза (Berkelman, Lagarias, 1986). Содержание NIR-FP с ковалентно присоединенным BV оценивали с помощью программы для обработки изображений ImageJ по общей интенсивности полос, визуализируемых при окрашивании кумасси синим.
Образцы, обработанные протеазой, анализировали с помощью электрофореза в полиакриламидном геле Tricine-SDS (16/6%), используемого для разделения пептидов в широком диапазоне молекулярных масс (Schagger, 2006). В качестве маркеров использовали пептиды с известной молекулярной массой до 40 кДа. Образцы белка инкубировали с протеазой в соотношении 20 : 1 в течение ночи при 37°С для трипсина и 50°С для химотрипсина (оптимальная температура для активности фермента). Некоторые образцы предварительно инкубировали с мочевиной в концентрации 5 М при 50°С в течение 30 мин перед добавлением протеазы. В качестве альтернативы образцы предварительно инкубировали с 0.2, 0.1 и 0.025% препарата ProteaseMAX™ при 37°C в течение 7 ч без мочевины или с добавлением 2 M мочевины. Подготовленные таким образом образцы смешивали с протеазами в соотношении 20 : 1 и инкубировали в течение ночи при 4, 20, 37 или 50°С.
Анализ пространственной структуры белка. Для анализа расположения сайтов протеолитического расщепления в iRFP670 использовали рентгеноструктурные данные PAS-GAF-доменов бактериального фитохрома RpBphP2 (файл 4E04.ent (Bellini, Papiz, 2012)), депонированные в базе данных PDB (Dutta et al., 2008).
РЕЗУЛЬТАТЫ И ОБСУЖДЕНИЕ
Мономерные (Baloban et al., 2017) и димерные (Stepanenko et al., 2022) NIR-FP с двумя ключевыми цистеиновыми остатками в доменах PAS и GAF содержат в своем составе две формы хромофора примерно в эквимолярном соотношении: BV, ковалентно связанный через атом С32 с CysGAF и BV, одновременно связанный ковалентно через атом С31 с CysGAF и через атом С32 с CysPAS (CysGAF-BV-CysPAS). Показанная нами ранее возможность получения NIR-FP с увеличенным содержанием хромофора CysGAF-BV-CysPAS с использованием метода селективной денатурации мономеров с CysGAF-BV в составе исходных NIR-FP указывает на то, что скрепление доменов PAS и GAF через одновременное связывание цистеиновых остатков в них с хромофором стабилизирует структуру NI-FP. На стабильность биомаркера в клетке помимо стабильности его структуры будет влиять его устойчивость к действию протеолитических ферментов. Поэтому мы проанализировали влияние протеаз, трипсина и химотрипсина, на структуру NIR-FP, содержащих оба ключевых цистеиновых остатка в доменах PAS и GAF, на примере белка iRFP670.
Устойчивость iRFP670 в холоформе к протеолизу. Белок iRFP670 имеет 33 и 22 сайта расщепления трипсином и химотрипсином соответственно (рис. 1). Белок после воздействия протеаз анализировали с использованием метода Tricine-SDS-электрофореза в полиакриламидном геле, позволяющего разделить пептиды в широком диапазоне молекулярных масс (Schagger, 2006). При расчете размеров пептидов, полученных при расщеплении iRFP670 протеазами, мы учитывали присутствие двух видов хромофоров в мономерах этого белка – CysGAF-BV и CysGAF-BV-CysPAS (табл. 1). Следует отметить, что пригодные для анализа методом масс-спектрометрии пептиды, учитывая ограничения метода (мелкие пептиды лучше выявляются), можно ожидать при полном расщеплении iRFP670 трипсином (пептиды 2.79, 1.88 и 0.91 кДа, содержащие ключевые цистеиновые остатки; см. табл. 1). Однако анализ образцов iRFP670, обработанных как трипсином, так и химотрипсином, не позволил выявить методом масс-спектрометрии искомые пептиды (данные не представлены).
Рис. 1.
Выравнивание аминокислотной последовательности iRFP670 относительно последовательности RpBphP2-PAS-GAF дикого типа. Пурпурным цветом выделены консервативные остатки Cys (С) в домене PAS (Cys15) и уникальный остаток Cys в домене GAF белка iRFP670 (Cys256). Аминокислоты Phe, Tyr и Trp, предшествующие расщепляемой химотрипсином амидной связи, и Lys и Arg, предшествующие расщепляемой трипсином амидной связи, выделены красным и зеленым соответственно.

Таблица 1.
Молекулярная масса пептидов, полученных при полном расщеплении iRFP670 в холоформе и апоформе химотрипсином (расщепление по остаткам F, Y и W) и трипсином (расщепление по остаткам K и R)
| Протеаза | Мол. масса пептида, кДа |
|---|---|
| iRFP670 в холоформе | |
| Химотрипсин | 7.13а, * (A-BV-Б); 5.04б (A); 4.34; 3.48; 2.69; 2.26; 2.09в, * (Б-BV); 2.08; 1.80; 1.74; 1.58; 1.46; 1.12; 1.02; 0.96; 0.82; 0.79; 0.62; 0.6; 0.35; 0.32; 0.26; 0.17 |
| Трипсин | 3.31; 2.79 а, * (A-BV-Б); 2.49; 2.40; 2.38; 2.33; 2.12; 1.99; 1.88 в, * (Б-BV); 1.40; 1.25; 1.18; 1.03; 0.99; 0.96; 0.91б (A); 0.91; 0.77; 0.68; 0.66; 0.65; 0.62; 0.60; 0.52; 0.50; 0.47; 0.39; 0.38; 0.37; 0.25; 0.17; 0.15 |
| iRFP670 в апоформе | |
| Химотрипсин, полное расщепление | 5.04б (A); 4.34; 3.48; 2.69; 2.26; 2.08; 1.80; 1.74; 1.58; 1.51г (Б); 1.46; 1.12; 1.02; 0.96; 0.82; 0.79; 0.62; 0.6; 0.35; 0.32; 0.26; 0.17 |
| Химотрипсин, частичное расщепление | 6.41 или 4.97д |
(а) – BV, ковалентно связанный с двумя цистеинами – CysPAS в пептиде A и CysGAF в пептиде Б (A-BV-Б); (б) – пептид А содержит CysPAS (A); (в) пептид Б содержит CysGAF, к которому ковалентно пришит BV в белке в холоформе (Б-BV); (г) – пептид Б содержит CysGAF (Б); (д) – пептид Б, образующийся, если полипептидная цепь не расщепляется по сайтам Y213, F247. Нумерация соответствует выравниванию последовательностей iRFP670 относительно RpBphP2 (4E04 file в базе данных PDB (Bellini, Papiz, 2012)); (*) – соответствующие пептиды содержат ковалентно присоединенный BV и могут быть идентифицированы по Zn-индуцированной флуоресценции.
Мы обнаружили, что электрофореграммы iRFP670 в холоформе после обработки протеазами содержали только полосы, соответствующие целевому нерасщепленному белку (рис. 2). Предварительная инкубация биомаркера в денатурирующих условиях (5 М мочевины) для дестабилизации его структуры (Proctor, Cunningham, 1988; Mine et al., 2004) не способствовала расщеплению белка протеазами (рис. 2). Это свидетельствует о высокой устойчивости iRFP670 в холоформе к действию протеаз.
Рис. 2.
Расщепление iRFP670 в холоформе в разных условиях. Tricine-SDS-электрофорез образцов белка с последующим окрашиванием кумасси синим. Проверены следующие условия: гель 1 (дорожки 1 и 2) – расщепление белка химотрипсином при 50°C без и с предварительной инкубацией образцов в присутствии 5 М мочевины; дорожка 3 – расщепление белка трипсином при 37°C. Гель 2 (дорожки 1, 2 и 3 – расщепление белка химотрипсином при 50°С с предварительной инкубацией образцов в присутствии 2 М мочевины и сурфактанта ProteaseMAX™ в концентрациях 0.2, 0.1, и 0.025%; дорожки 4, 5 и 6 – расщепление белка химотрипсином при 50°С с предварительной инкубацией образцов в присутствии сурфактанта ProteaseMAX™ в концентрациях 0.2, 0.1, и 0.025%. Гель 3 (дорожки 1, 2 и 3) – расщепление белка трипсином при 37°С с предварительной инкубацией образцов в присутствии 2 М мочевины и сурфактанта ProteaseMAX™ в концентрациях 0.2, 0.1 и 0.025%; дорожки 4, 5, и 6 – расщепление белка трипсином в 37°С с предварительной инкубацией образцов в присутствии сурфактанта ProteaseMAX™ в концентрациях 0.2, 0.1 и 0.025%. Гель 4 (дорожки 1, 2 и 3) – расщепление белка химотрипсином при 4, 37 и 50°C с предварительной инкубацией образцов в присутствии 0.2% сурфактанта. M – маркерные пептиды, К – контрольные образцы исходного белка. Активность протеаз (Eакт) подтверждается их саморасщеплением при оптимальной температуре. Растворы протеаз, активность которых блокировали (добавлением ингибитора PMSF в концентрации 1 мМ при 4°C) содержат только пептиды с мол. массой около 14 и 10 кДа для химотрипсина и 23.3 кДа для трипсина, что соответствует интактным субъединицам фермента (Eинт).
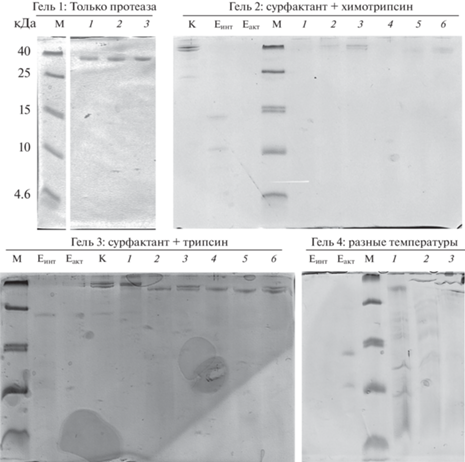
Условия для расщепления iRFP670. Нам удалось подобрать условия, при которых наблюдается лишь частичный гидролиз iRFP670. Мы обнаружили, что частичное расщепление iRFP670 происходит при его обработке химотрипсином, но не трипсином, в присутствии агента ProteaseMAX™ Surfactant (Trypsin Enhancer) (Shin et al., 2009; Diaz-Gonzalez et al., 2015; Krey et al., 2015) (рис. 2). Считается, что этот препарат увеличивает эффективность ряда протеолитических ферментов за счет дестабилизации структуры анализируемых белков и увеличения их растворимости (Diaz-Gonzalez et al., 2015). В растворе iRFP670, расщепленного химотрипсином, присутствуют в основном пептиды с молекулярной массой около 14–15 кДа и выше (рис. 3). Эти пептиды содержат в своем составе ковалентно присоединенный хромофор, о чем свидетельствует Zn-индуцированная флуоресценция соответствующих им электрофоретических полос (рис. 3). Также можно предполагать, что в анализируемом растворе присутствуют следовые количества пептидов с молекулярной массой около 12 и 7–10 кДа с ковалентно присоединенным хромофором в их составе (рис. 3).
Рис. 3.
Расщепление белка iRFP670 в холоформе (две панели слева) и апоформе (правая панель) химотрипсином. Tricine-SDS-электрофорез образцов белка с последующим окрашиванием кумасси синим и регистрацией цинк-индуцированной флуоресценции (Zn). В лунки загружали следующие образцы: М – маркерные пептиды, К – контрольные образцы исходного белка, дорожки 1 и 2 – образцы iRFP670, расщепленные при 4 и 20°С. Между панелями указаны ожидаемые размеры обнаруженных пептидов и характер связывания с ними биливердина (BV) (см. также табл. 2).

Основываясь на кристаллографических данных о структуре фитохромов, мы провели анализ расположения сайтов расщепления химотрипсином в iRFP670, чтобы понять причины его частичного протеолиза (табл. 2, рис. 4). Мы обнаружили, что часть потенциальных сайтов расщепления химотрипсином в последовательности белка может быть стерически недоступна для действия протеазы. Так, недоступность сайта расщепления Y213 может быть связана с его расположением вблизи от одного из пересечений полипептидной цепи белка, формирующей узел (рис. 4).
Таблица 2.
Расчетный размер BV-содержащих пептидов, полученных при частичном и полном расщеплении iRFP670 в холоформе химотрипсином (расщепление по остаткам F, Y и W)
| Мол. масса петида с BV, кДа | Последовательность пептида Б* при полном расщеплении, или в случае, когда некоторые сайты расщепления пропускаются протеазой | |
|---|---|---|
| А-BV-Ба | BV-Б | |
| Частичное расщеплениеб | ||
| 12.04 | 6.99 | PASLVPQQARLLY213LKNAIRVVSDSRGISSRIVPEHDASGAALDLSF247AHLRSISPCysGAFHLEF260в |
| 14.28 | 9.24 | PASLVPQQARLLY213LKNAIRVVSDSRGISSRIVPEHDASGAALDLSF247AHLRSISPCysGAFHLEF260LRNMGVSASMSLSIIIDGTLW281 |
| 15.22 | 10.18 | PASLVPQQARLLY213LKNAIRVVSDSRGISSRIVPEHDASGAALDLSF247AHLRSISPCysGAFHLEF260LRNMGVSASMSLSIIIDGTLW281GLIICHHY289 |
| Полное расщепление | ||
| 7.13 | 2.09 | AHLRSISPCysGAFHLEF260 |
(а) – пептид A содержит CysPAS (размер 5.04 кДа, последовательность: MARKVDLTSCysPASDREPIHIPGSIQPCGCLLACDAQAVRITRITENAGAF52). Пептид Б содержит CysGAF; (б) – сайты расщепления, пропущенные протеазой, выделены жирным шрифтом; (в) – нумерация соответствует выравниванию последовательности iRFP670 относительно RpBphP2 (файл 4E04 в базе данных PDB (Bellini, Papiz, 2012)); (*) – часть потенциальных сайтов расщепления химотрипсином в последовательности белка может быть стерически недоступна для действия протеазы, что приводит к формированию пептида Б разной молекулярной массы, в то время как молекулярная масса пептида А не изменяется.
Рис. 4.
Расположение сайтов протеолитического расщепления в структуре iRFP670, потенциально недоступных для химотрипсина. а – Пространственная структура белка. б – Схематическое изображение структуры белка показывает топологию узла. Рисунок создан на основе рентгеноструктурных данных доменов PAS и GAF белка RpBphP2 из Rhodopseudomonas palustris (PDB: файл 4e04 (Bellini, Papiz, 2012)) с помощью графических программных пакетов VMD (Visual Molecular Dynamics) (Hsin et al., 2008), Raster3D (Merritt, Bacon, 1997) и ICM Pro (Abagyan et al., 1994). Показаны сайты расщепления химотрипсином Y213, F247, F260, и W281 (в виде Ван-дер-Ваальсовых сфер показаны атомы СА соответствующих остатков). Изображен связанный биливердин (BV).

Сайт расщепления F247 находится вблизи другого пересечения полипептидной цепи в петле, через которую протягивается N-концевой сегмент белка (рис. 4). Следует заметить, что эти два сайта расщепления находятся с N-конца от ключевого остатка CysGAF. Расчет показывает, что если гидролиз по сайтам Y213 и F247 не происходит, то в анализируемом растворе iRFP670 после его обработки химотрипсином должны присутствовать пептиды с ковалентно присоединенным хромофором с молекулярной массой 6.99 (пептид Б с ковалентно присоединенным хромофором: Б-BV) и 12.04 кДа (пептид А и пептид Б, связанные через ковалентно присоединенный хромофор: Б-BV-А) (табл. 2). Доступ протеазы к сайту расщепления F260, расположенному с C-конца от CysGAF, может быть ограничен из-за ковалентного связывания BV в биомаркере (рис. 4), что приведет к появлению в анализируемом растворе пептидов с ковалентно присоединенным хромофором размером 9.24 (пептид Б-BV) и 14.28 кДа (пептид Б-BV-А) (табл. 2).
Еще один сайт расщепления W281 с C-конца от CysGAF находится глубоко внутри глобулы белка в домене GAF (рис. 4). В случае отсутствия гидролиза полипептидной цепи iRFP670 по всем перечисленным сайтам, включая Y213, F247, F260 и W281, размер ожидаемых фрагментов белка составит 10.18 кДа (пептид Б-BV) и 15.22 кДа (пептид Б-BV-А) (табл. 2). Следует заметить, что при расщеплении мономеров iRFP670, содержащих хромофор CysGAF-BV, должен образовываться пептид с ключевым остатком CysPAS который не несет ковалентно присоединенного хромофора, его молекулярная масса составляет порядка 5.04 кДа (табл. 1).
Основываясь на проведенном структурном анализе, присутствие на электрофореграмме iRFP670 после его обработки химотрипсином интенсивных Zn-позитивных полос, соответствующих пептидам с размером 14–15 кДа, и едва выраженных Zn-позитивных полос, соответствующих пептидам с размером 12 и 7–10 кДа, а также Zn-негативной полосы, соответствующей пептиду размером 5 кДа, можно интерпретировать в пользу практически полного ингибирования расщепления мономерного белка с хромофором CysGAF-BV-CysPAS, но не мономера с CysGAF-BV, расщепление которого, по всей видимости, идет до конца (рис. 3). Полученные нами данные хорошо согласуются с данными из литературы о наличии в димерных и мономерных NIR-FP с двумя ключевыми остатками цистеина двух видов хромофора (Baloban et al., 2017; Stepanenko et al., 2017b). Большой размер хромофор-содержащих пептидов, полученных при расщеплении iRFP670 химотрипсином, также хорошо укладывается в предположение о возможности одновременного ковалентного присоединения BV к остаткам цистеина в обоих доменах в NIR-FP.
Анализ влияния химотрипсина на структуру апобелка iRFP670 (не связанного с хромофором) в присутствии ProteaseMAX™ Surfactant показал, что в растворе белка после такого воздействия присутствуют короткие пептиды, размером между 4.6 и 10 кДа (рис. 3). Согласно анализу аминокислотной последовательности iRFP670 в этот диапазон попадает только пептид А с молекулярной массой 5.04 кДа (табл. 1). При полном гидролизе протеазой всей остальной полипептидной цепи белка образуются пептиды молекулярной массы не более 4.34 кДа (табл. 1). В случае неполного гидролиза апобелка iRFP670 из-за недоступности сайтов расщепления Y213 и F247, или только F247, находящихся у пересекающихся участков полипептидной цепи в составе узла, могут формироваться пептиды молекулярной массы 6.41 и 4.97 кДа (варианты пептида Б) соответственно (табл. 1). Такая ситуация, вероятно, и реализуется в нашем случае (рис. 3).
Подводя итоги, заметим, что частичный гидролиз iRFP670 может быть достигнут только с помощью химотрипсина, действие которого усиливается поверхностно-активным веществом ProteaseMAX™ (Trypsin Enhancer). Следует отметить, что и в этом случае эффективность расщепления белка низка, так что концентрация и размер образующихся пептидов не позволяют обнаружить их масс-спектрометрическим методом. Мы считаем это важным результатом, показывающим, что NIR-FP, содержащие хромофор в форме CysGAF-BV-CysPAS, слабо чувствительны к исследуемым протеазам.
ЗАКЛЮЧЕНИЕ
Полученные данные указывают на возможную функцию узла в составе полипептидной цепи хромофор-связывающего домена фитохромов, заключающуюся в повышении устойчивости этих белков против протеолитической деградации. В совокупности со стабилизирующим действием узла, одновременное ковалентное связывание BV с обоими доменами NIR-биомаркеров с двумя ключевыми остатками цистеина может вносить вклад в устойчивость этих белков к действию различных, в том числе клеточных, протеаз.
Список литературы
Abagyan R., Totrov M., Kuznetsov D. 1994. ICM-A New Method for protein modeling and design: applications to docking and structure prediction from the distorted native conformation. J. Comput. Chem. V. 15. P. 488.
Baloban M., Shcherbakova D.M., Pletnev S., Pletnev V.Z., Lagarias J.C., Verkhusha V.V. 2017. Designing brighter near-infrared fluorescent proteins: insights from structural and biochemical studies. Chem. Sci. V. 8. P. 4546. https://doi.org/10.1039/c7sc00855d
Bellini D., Papiz M.Z. 2012. Dimerization properties of the RpBphP2 chromophore-binding domain crystallized by homologue-directed mutagenesis. Acta Crystallogr. D Biol. Crystallogr. V. 68. P. 1058. https://doi.org/10.1107/S0907444912020537
Berkelman T.R., Lagarias J.C. 1986. Visualization of bilin-linked peptides and proteins in polyacrylamide gels. Anal. Biochem. V. 156. P. 194. https://doi.org/10.1016/0003-2697(86)90173-9
Buhrke D., Tavraz N.N., Shcherbakova D.M., Sauthof L., Moldenhauer M., Velazquez Escobar F., Verkhusha V.V., Hildebrandt P., Friedrich T. 2019. Chromophore binding to two cysteines increases quantum yield of near-infrared fluorescent proteins. Sci. Rep. V. 9. P. 1866. https://doi.org/10.1038/s41598-018-38433-2
Chernov K.G., Redchuk T.A., Omelina E.S., Verkhusha V.V. 2017. Near-Infrared fluorescent proteins, biosensors, and optogenetic tools engineered from phytochromes. Chem. Rev. V. 117. P. 6423. https://doi.org/10.1021/acs.chemrev.6b00700
Diaz-Gonzalez F., Milano M., Olguin-Araneda V., Pizarro-Cerda J., Castro-Cordova P., Tzeng S.C., Maier C.S., Sarker M.R., Paredes-Sabja D. 2015. Protein composition of the outermost exosporium-like layer of Clostridium difficile 630 spores. J. Proteomics. V. 123. P. 1. https://doi.org/10.1016/j.jprot.2015.03.035
Dutta S., Burkhardt K., Swaminathan G.J., Kosada T., Henrick K., Nakamura H., Berman H.M. 2008. Data deposition and annotation at the worldwide protein data bank. Methods Mol. Biol. V. 426. P. 81. https://doi.org/10.1007/978-1-60327-058-8_5
Hsin J., Arkhipov A., Yin Y., Stone J.E., Schulten K. 2008. Using VMD: an introductory tutorial. Curr. Protoc. Bioinformatics. Chapter 5: Unit 5.7.
Kapitulnik J., Maines M.D. 2012. The role of bile pigments in health and disease: effects on cell signaling, cytotoxicity, and cytoprotection. Front. Pharmacol. V. 3. P. 136. https://doi.org/10.3389/fphar.2012.00136
Komatsu N., Aoki K., Yamada M., Yukinaga H., Fujita Y., Kamioka Y., Matsuda M. 2011. Development of an optimized backbone of FRET biosensors for kinases and GTPases. Mol. Biol. Cell. V. 22(23). P. 4647. https://doi.org/10.1091/mbc.E11-01-0072
Krey J.F., Sherman N.E., Jeffery E.D., Choi D., Barr-Gillespie P.G. 2015. The proteome of mouse vestibular hair bundles over development. Sci. Data. V. 2. P. 150047. https://doi.org/10.1038/sdata.2015.47
Laemmli U.K. 1970. Cleavage of structural proteins during the assembly of the head of bacteriophage T4. Nature. V. 227. P. 680. https://doi.org/10.1038/227680a0
Li L., Shemetov A.A., Baloban M., Hu P., Zhu L., Shcherbakova D.M., Zhang R., Shi J., Yao J., Wang L.V., Verkhusha V.V. 2018. Small near-infrared photochromic protein for photoacoustic multi-contrast imaging and detection of protein interactions in vivo. Nat. Commun. V. 9. P. 2734. https://doi.org/10.1038/s41467-018-05231-3
Matlashov M.E., Shcherbakova D.M., Alvelid J., Baloban M., Pennacchietti F., Shemetov A.A., Testa I., Verkhusha V.V. 2020. A set of monomeric near-infrared fluorescent proteins for multicolor imaging across scales. Nat. Commun. V. 11. P. 239. https://doi.org/10.1038/s41467-019-13897-6
Merritt E.A., Bacon D.J. 1997. Raster3D: photorealistic molecular graphics. Methods Enzymol. V. 277. P. 505. https://doi.org/10.1016/S0076-6879(97)77028-9
Mine Y., Ma F., Lauriau S. 2004. Antimicrobial peptides released by enzymatic hydrolysis of hen egg white lysozyme. J. Agric. Food Chem. V. 52(5). P. 1088. https://doi.org/10.1021/jf0345752
Proctor V.A., Cunningham F.E. 1988. The chemistry of lysozyme and its use as a food preservative and a pharmaceutical. Crit. Rev. Food Sci. Nutr. V. 26. P. 359. https://doi.org/10.1080/10408398809527473
Qian Y., Piatkevich K.D., Mc Larney B., Abdelfattah A.S., Mehta S., Murdock M.H., Gottschalk S., Molina R.S., Zhang W., Chen Y., Wu J., Drobizhev M., Hughes T.E., Zhang J., Schreiter E. et al. 2019. A genetically encoded near-infrared fluorescent calcium ion indicator. Nat. Methods. V. 16. P. 171. https://doi.org/10.1038/s41592-018-0294-6
Rodriguez E.A., Campbell R.E., Lin J.Y., Lin M.Z., Miyawaki A., Palmer A.E., Shu X., Zhang J., Tsien R.Y. 2017. The growing and glowing toolbox of fluorescent and photoactive proteins. Trends Biochem. Sci. V. 42. P. 111. https://doi.org/10.1016/j.tibs.2016.09.010
Schagger H. 2006. Tricine-SDS-PAGE. Nat. Protoc. V. 1. P. 16. https://doi.org/10.1038/nprot.2006.4
Shcherbakova D.M., Baloban M., Pletnev S., Malashkevich V.N., Xiao H., Dauter Z., Verkhusha V.V. 2015. Molecular basis of spectral diversity in near-infrared phytochrome-based fluorescent proteins. Chem. Biol. V. 22. P. 1540. https://doi.org/10.1016/j.chembiol.2015.10.007
Shcherbakova D.M., Cox Cammer N., Huisman T.M., Verkhusha V.V., Hodgson L. 2018a. Direct multiplex imaging and optogenetics of Rho GTPases enabled by near-infrared FRET. Nat. Chem. Biol. V. 14. P. 591. https://doi.org/10.1038/s41589-018-0044-1
Shcherbakova D.M., Stepanenko O.V., Turoverov K.K., Verkhusha V.V. 2018b. Near-infrared fluorescent proteins: multiplexing and optogenetics across scales. Trends Biotechnol. V. 36. P. 1230. https://doi.org/10.1016/j.tibtech.2018.06.011
Shin J.B., Pagana J., Gillespie P.G. 2009. Twist-off purification of hair bundles. Methods Mol. Biol. V. 493. P. 241. https://doi.org/10.1007/978-1-59745-523-7_14
Stepanenko O.V., Baloban M., Bublikov G.S., Shcherbakova D.M., Stepanenko O.V., Turoverov K.K., Kuznetsova I.M., Verkhusha V.V. 2016. Allosteric effects of chromophore interaction with dimeric near-infrared fluorescent proteins engineered from bacterial phytochromes. Sci. Rep. V. 6. P. 18750. https://doi.org/10.1038/srep18750
Stepanenko O.V., Kuznetsova I.M., Turoverov K.K., Stepanenko O.V. 2022. Impact of double covalent binding of BV in NIR FPs on their spectral and physicochemical properties. Int. J. Mol. Sci. V. 23. P. 7347. https://doi.org/10.3390/ijms23137347
Stepanenko O.V., Stepanenko O.V., Bublikov G.S., Kuznetsova I.M., Verkhusha V.V., Turoverov K.K. 2017a. Stabilization of structure in near-infrared fluorescent proteins by binding of biliverdin chromophore. J. Mol. Str. V. 1140. P. 22. https://doi.org/10.1016/j.molstruc.2016.10.095
Stepanenko O.V., Stepanenko O.V., Kuznetsova I.M., Shcherbakova D.M., Verkhusha V.V., Turoverov K.K. 2017b. Interaction of biliverdin chromophore with near-infrared fluorescent protein BphP1-FP engineered from bacterial phytochrome. Int. J. Mol. Sci. V. 18. P. 1009. https://doi.org/10.3390/ijms18051009
Stepanenko O.V., Stepanenko O.V., Shpironok O.G., Fonin A.V., Kuznetsova I.M., Turoverov K.K. 2019. Near-Infrared markers based on bacterial phytochromes with phycocyanobilin as a chromophore. Int. J. Mol. Sci. V. 20. P. 6067. https://doi.org/10.3390/ijms20236067
Subach O.M., Barykina N.V., Anokhin K.V., Piatkevich K.D., Subach F.V. 2019. Near-infrared genetically encoded positive calcium indicator based on GAF-FP bacterial phytochrome. Int. J. Mol. Sci. V. 20. P. 3488. https://doi.org/10.3390/ijms20143488
Weissleder R. 2001. A clearer vision for in vivo imaging. Nat. Biotechnol. V. 19. P. 316. https://doi.org/10.1038/86684
Yu D., Gustafson W.C., Han C., Lafaye C., Noirclerc-Savoye M., Ge W.P., Thayer D.A., Huang H., Kornberg T.B., Royant A., Jan L.Y., Jan Y.N., Weiss W.A., Shu X. 2014. An improved monomeric infrared fluorescent protein for neuronal and tumour brain imaging. Nat. Commun. V. 5. P. 3626. https://doi.org/10.1038/ncomms4626
Дополнительные материалы отсутствуют.


